ABSTRACT
Polymorphism Identification of ILSTS028, ILSTS052 and ILSTS056 Microsatellite DNA Loci on Katingan Cattle in Central Kalimantan
Erwinsyah, Jakaria and C. Sumantri
This study aimed to determine the genetic diversity of Katingan cattle population in Central Kalimantan by DNA microsatellite markers ILSTS028, ILSTS052 and ILSTS056 loci. The 70 blood samples used in this study originated from three subpopulation of Pendahara sub populations (26 samples), Buntut Bali (13 samples) and Tumbang Lahang (31 samples), and other samples were Bali cattle (11 samples), Madura cattle (1 sample), Limousin cattle (3 samples) and PO cattle (6 samples). The amplification of microsatellite DNA marker was done by PCR (Polymerase Chain Reaction). The PCR product was then electrophoresed using 6% polyacrilamide gel followed by silver staining. The data was analyzed to get allele frequency, genotype frequency and heterozygosity value. Microsatellite DNA of loci showed high polymorphisms. ILSTS028 locus had 20 alleles in Katingan cattle with the highest allele frequency was allele H (0.1691) in Tumbang Lahang population and the lowest allele C, D, E and K (0.0167) in Tumbang Lahang population. The highest allele frequency of Bali, PO, Limousin and Madura cattle was alleles H and R in Madura cattle (0.5000). The lowest allele frequency was alleles I, K and L (0.0455) in Bali cattle. ILSTS052 locus had 13 alleles in Katingan cattle with the highest allele frequency was allele C (0.5000) in Buntut Bali populations and lowest allele G, H, J, M and N (0.0208) in Pendahara population. Allele B had the highest frequency in Limousin cattle (1.0000) and alleles E and I the lowest frequency in Bali cattle (0.0455). ILSTS056 locus had 19 alleles in Katingan cattle with the highest allele frequency was allele M (0.2692) in Buntut Bali population and the lowest is allele B, O, Q and U (0.0167) in Tumbang Lahang population. Alleles D and I in Limuosin cattle and alleles A and D in Madura cattle had the highest frequency (0.5000) and alleles D, L and M the lowest frequency in Bali cattle (0.0455). The heterozygosity value from ILSTS028, ILSTS052 and ILSTS056 locus of Katingan cattle with the others cattle were 0.989, 0.629 and 1.000, respectively.
3 PENDAHULUAN
Latar Belakang
Ternak lokal merupakan kebanggaan bagi Indonesia, karena ternak lokal merupakan sumber daya genetik ternak bagi bangsa. Ternak lokal memiliki beberapa keunggulan yaitu daya adaptasi tinggi walaupun kondisi pakan berkualitas rendah, lingkungan marjinal, pemeliharaan secara ekstensif tradisional dan memiliki keunggulan tahan terhadap beberapa penyakit dan parasit. Ternak lokal juga memiliki beberapa kekurangan yaitu produktivitas rendah dibandingkan ternak impor. Peningkatan dan perbaikan mutu genetik ternak lokal agar dapat dimanfaatkan dengan optimal.
Pelestarian terhadap sumber daya genetik ternak asli atau ternak lokal sangat penting karena merupakan bagian dari komponen keanekaragaman hayati untuk memenuhi kebutuhan pangan, pertanian, dan perkembangan sosial masyarakat di masa yang akan datang. Beberapa alasan pelestarian sumber daya genetik ternak Indonesia penting dilakukan yaitu lebih dari 60% bangsa ternak di dunia berada di negara berkembang, konservasi ternak asli atau ternak lokal tidak menarik bagi petani, secara umum tidak ada program pemantauan yang sistematis dan tidak tersedia informasi deskriptif dasar sebagian sumber daya genetik ternak, dan sedikit sekali bangsa-bangsa ternak asli maupun ternak lokal yang telah digunakan dan dikembangkan secara aktif (FAO, 2001). Populasi sapi dari tahun 2005 sampai 2010 dilaporkan oleh Mayulu et al. (2010) selalu meningkat tiap tahunnya dari 11.045.900 ekor pada tahun 2005 menjadi 14.763.000 ekor pada tahun 2010.
Sapi Katingan adalah salah satu jenis sapi yang terdapat di Kalimantan Tengah sejak puluhan tahun dan sudah beradaptasi dengan lingkungan setempat. Sapi Katingan dipelihara oleh suku Dayak dan hidup disepanjang daerah aliran sungai Katingan. Sapi Katingan dipelihara secara ekstensif tradisional oleh masyarakat suku Dayak. Menurut Utomo et al. (2010), sapi tersebut dinamakan sapi Katingan dikarenakan lokasi pemeliharaan yang menyebar di sepanjang daerah aliran sungai Katingan. Masyarakat Dayak sendiri menyebutnya sapi lokal atau kadang-kadang sapi ”Helu” (sapi jaman dulu), tidak pernah mereka menamakannya sapi Katingan. Istilah sapi Katingan diberikan untuk membedakan dengan sapi lokal lain (Utomo, 2011). Penamaan sapi diberikan sesuai lokasi habitatnya sebagaimana pada
4 sapi-sapi lokal lain (Abdullah, 2008; Sarbaini, 2004; Sun et al., 2008). Penelitian ini dilakukan karena keterbatasan informasi mengenai data-data yang berhubungan dengan aspek fenotipik dan genetik pada sapi Katingan.
Tujuan
Tujuan penelitian ini adalah untuk mengidentifikasi keragaman sapi Katingan di Kalimantan Tengah dengan menggunakan penanda molekuler DNA mikrosatelit lokus ILSTS028, ILSTS052 dan ILSTS056.
5 TINJAUAN PUSTAKA
Sumber Daya Genetik Ternak Lokal
Keanekaragaman ternak sapi di Indonesia terbentuk dari sumber daya genetik ternak asli dan impor. Impor ternak sapi Ongole (Bos indicus) atau Zebu yang dimulai pada awal abad ke-20 memegang peranan penting dalam pengembangan peternakan di Indonesia. Ongole murni pertama kali dibawa ke Pulau Sumbawa yang kemudian disebut sebagai Sumba Ongole (SO) dan selanjutnya dibawa ke tempat-tempat lain di Indonesia untuk disilangkan dengan sapi asli Jawa dan membentuk Peranakan Ongole (PO) dan sapi Madura (Utoyo, 2002). Proses perkembangan sapi di Indonesia telah menghasilkan sumber daya genetik ternak yang lebih beragam, yaitu mulai dari sapi asli seperti sapi Bali, juga sapi hasil silangan yang telah menjadi sapi lokal seperti sapi Pesisir, sapi Aceh, sapi Madura, sapi Sumba Ongole (SO) dan sapi Peranakan Ongole (PO) (Utoyo, 2002; Martojo, 2003).
Sumber daya genetik ternak merupakan kerangka dasar acuan bagi pertanian dan pengembangan varietas dan bangsa ternak untuk masa yang akan datang. Keanekaragaman bangsa ternak asli maupun yang beradaptasi secara lokal berlimpah dan dapat menyelamatkan petani dalam menghadapi iklim yang sulit dan wilayah yang marjinal. Sumber daya genetik ternak lokal dapat dimanfaatkan dengan biaya (input) minimum dan memegang peranan penting dalam budaya masyarakat pedesaan (FAO, 2001).
Keanekaragaman genetik ternak lokal memiliki beberapa manfaat, yaitu (1) untuk keberlanjutan dan peningkatan produksi pangan, (2) untuk memaksimalkan produktivitas lahan dan sumber daya pertanian, (3) untuk pencapaian pertanian berkelanjutan dan (4) untuk pemenuhan keanekaragaman baik yang telah maupun yang akan diketahui manfaatnya bagi kehidupan sosial masyarakat. Ketersediaan keanekaragaman genetik ternak khususnya ternak sapi akan memberikan keberhasilan dalam strategi pemuliaan untuk masa yang akan datang (FAO-AAAS, 1994).
6 tidak akan dapat diganti meskipun dengan kemajuan bioteknologi hingga sampai saat ini, sehingga pelestarian sumber daya genetik ternak perlu dilakukan.
Pelestarian sumber daya genetik ternak asli atau ternak lokal sangat penting karena merupakan bagian dari komponen keanekaragaman hayati untuk memenuhi kebutuhan pangan, pertanian, dan perkembangan sosial masyarakat di masa yang akan datang. Beberapa alasan pelestarian sumber daya genetik ternak Indonesia penting dilakukan, yaitu (1) lebih dari 60% bangsa ternak di dunia terdapat di negara berkembang, (2) konservasi ternak asli atau ternak lokal tidak menarik bagi petani, (3) secara umum tidak terdapat program pemantauan yang sistematis dan tidak tersedia informasi deskriptif dasar sebagian sumber daya genetik ternak yang ada dan (4) sedikit sekali bangsa-bangsa ternak asli maupun ternak lokal yang telah digunakan dan dikembangkan secara aktif (FAO, 2001).
Sapi Lokal Kalimantan Tengah
Kalimantan Tengah harus mendatangkan ternak sapi potong dari luar provinsi sekitar 3.000 ekor setiap tahun sebab produksi lokal hanya mampu memenuhi sekitar 45%-50% dari total kebutuhan. Pemerintah menargetkan pencapaian swasembada daging sapi secara nasional pada tahun 2014 sehingga untuk Provinsi Kalimantan Tengah sapi potong ditargetkan mencapai 27.000 ekor pada tahun 2014. Saat ini prediksi populasi sapi potong di Kalimantan Tengah pada tahun 2010 baru mencapai 14.000 ekor. Kondisi ini membuat pesimis berbagai pihak sehingga perlu berbagai upaya dan kerja keras semua pihak untuk menggunakan potensi sumberdaya lokal yang ada di daerah dimanfaatkan secara optimal, baik sumberdaya genetik maupun sumberdaya lahan dan pakan lokal. Kalimantan Tengah memiliki sapi lokal yang oleh masyarakat setempat (suku Dayak) dinamakan juga sapi lokal, berbeda dengan sapi lokal lainnya disebut sesuai dengan nama asal dari sapi tersebut, misalnya sapi Bali. Sapi lokal Kalimantan Tengah belum memiliki nama, namun beberapa orang menyebut sesuai dengan nama daerah aliran sungai tempat sapi tersebut hidup. Sapi-sapi tersebut hanya dipelihara oleh masyarakat setempat (suku Dayak). Sapi-Sapi-sapi lokal lainnya seperti sapi Bali (dominan), sapi Madura dan sapi PO kebanyakan dipelihara oleh masyarakat pendatang (transmigrasi) (Adrial, 2010).
Asal-usul sapi lokal Kalimantan Tengah sampai saat ini masih belum diketahui secara pasti. Sapi-sapi tersebut dipelihara secara ekstensif di padang
7 gembalaan yang relatif luas dalam bentuk ranch-ranch. Keberadaan sapi sudah puluhan tahun dan sudah beradaptasi dengan lingkungan sekitarnya dengan lahan tergolong asam dan miskin mineral. Populasi sapi lokal di Kabupaten Katingan dilaporkan sekitar 1.500 ekor. Pengembangan sapi lokal mengalami beberapa hambatan, salah satunya adalah masih sedikitnya informasi terutama data dasar tentang sistem produksi dan reproduksi, keadaan lingkungan, daya tampung lahan dan keterampilan petani yang mengelola. Informasi-informasi tersebut sangat penting karena berkaitan dengan keberhasilan pelestarian dan pengembangannya di masa yang akan datang (Adrial, 2010).
Sapi lokal Kalimantan Tengah memiliki potensi besar sebagai ternak potong, karena sapi ini mampu beradaptasi dengan lingkungan Kalimantan Tengah yang asam dan miskin mineral, mempunyai produktivitas yang cukup baik pada kondisi pemeliharaan ekstensif tradisional, relatif tahan terhadap berbagai macam penyakit dan parasit serta mempunyai kemampuan reproduksi yang tinggi. Potensi ini belum dimanfaatkan secara optimal, bahkan banyak orang Kalimantan Tengah yang tidak mengetahui bahwa Kalimantan Tengah memiliki sapi lokal yang potensial untuk dikembangkan (Adrial, 2010).
Ciri Spesifik Sapi Katingan
Karakteristik yang menonjol pada sapi Katingan yang hidup di sepanjang daerah aliran sungai Katingan, Kalimantan Tengah yang membedakan dengan sapi lokal lainnya di Indonesia adalah bentuk tanduk yang sebagian besar melengkung ke depan pada sapi betina dewasa. Bentuk tanduk pada sapi Katingan jantan tumbuh normal seperti umumnya sapi lokal yang lain, yaitu ke samping atas serta tonjolan pada kepala bagian atas diantara dua tanduk. Tonjolan hanya ditemukan pada sapi betina (Utomo et al., 2010).
Warna bulu mata bervariasi seperti berwarna hitam, coklat kemerahan, coklat keputihan, bahkan ditemukan warna putih. Teracak pada sapi Katingan ditemukan dua warna, yaitu warna hitam (dominan) dan warna coklat kemerahan (Utomo et al., 2010).
.
8 Bentuk Tubuh
Ukuran tubuh dewasa sapi Katingan jantan tidak selalu lebih besar dibandingkan dengan sapi betina. Sapi jantan maupun sapi betina mempunyai gumba yang cukup jelas terlihat. Karakteristik morfologik ini sama seperti pada sapi PO dan Madura (Utomo et al., 2010).
Ukuran gumba pada sapi jantan jauh lebih tinggi ketika tumbuh dewasa. Gelambir ditemukan baik pada sapi jantan maupun sapi betina. Tampilan gelambir pada sapi jantan lebih tebal dan lebih berat dibandingkan dengan gelambir pada sapi betina. Gelambir dijumpai mulai dari bawah kerongkongan sampai bawah dada di antara dua kaki depan (Utomo et al., 2010).
Karakteristik Kualitatif
Warna bulu sapi hanya ditentukan berdasarkan warna utama atau warna dasar untuk memudahkan dalam pengelompokan warna, karena dari warna dasar tersebut ditemukan warna lain di bagian-bagian tertentu tubuh sapi (Utomo et al., 2010). Keragaman warna bulu sapi Katingan dapat dilihat pada Gambar 1.
Sumber : Utomo et al. (2010)
Gambar 1. Keragaman Warna Bulu Sapi Katingan Betina (a, b, c) dan Jantan (d, e, f)
Variasi warna bulu sapi Katingan betina di daerah aliran sungai Katingan didominasi oleh warna coklat kemerahan, yaitu 27%, diikuti berturut-turut cokelat keputihan (14,1%), cokelat warna sapi Bali (13,8%), hitam (12,5%), cokelat keruh atau kusam (9,6%), cokelat merah bata (9,3%), kehitaman (7,1%), putih kecokelatan
a
d
e
f
c
b
e
9 (5,5%) dan putih keabuan (4,5%). Variasi warna bulu sapi Katingan jantan didominasi warna hitam sebesar 27%, diikuti berturut-turut cokelat keputihan (14,8%), cokelat keputihan dan kemerahan (14,8%), cokelat kemerahan (13,1%), kehitaman (12,3%), cokelat keputihan punuk hitam (10,7%), cokelat merah bata (9,8%), dan cokelat merah bata punuk hitam (7,8%). Menurut Fries dan Ruvinsky (1999), warna yang lebih gelap pada leher dan kepala pada sapi jantan merupakan warna tipe sapi liar (Utomo et al., 2010).
Variasi bentuk tanduk sapi Katingan betina adalah bentuk tanduk melengkung ke depan, pendek dan kecil yang tidak melekat pada tulang kepala sehingga kalau dipegang akan goyang, melengkung menyamping ke depan, melengkung ke bawah, menyamping horizontal, tidak bertanduk (Utomo et al., 2010). Variasi pertumbuhan (bentuk) tanduk pada sapi jantan dewasa hanya dua, yaitu bentuk tanduk ke arah samping atas dan melengkung ke atas (Gambar 2).
Sumber : Utomo et al. (2010)
Gambar 2. Keragaman Bentuk Tanduk Sapi Katingan Jantan
Tonjolan pada kepala sebagian besar ditemukan pada sapi betina (82,93%-97,56%), sedangkan pada jantan tidak ditemukan. Tinggi rendah tonjolan bervariasi, dari tinggi hingga rendah (Gambar 3). Tonjolan di kepala tidak ditemukan pada sapi
4 5 6
10 PO, Bali dan Madura, sehingga tonjolan ini dapat dijadikan sebagai penciri pada sapi Katingan (Utomo et al., 2010).
Sumber : Utomo et al. (2010)
Gambar 3. Variasi Tonjolan (Tinggi Rendah) pada kepala Sapi Katingan Betina Karakteristik Kuantitatif
Rata-rata bobot badan sapi betina di daerah aliran sungai Katingan asal Buntut Bali 201,8 kg, asal Pendahara 208,9 kg dan asal Tumbang Lahang 217,1 kg. Rata-rata bobot badan sapi jantan dari Buntut Bali sebesar 299,9 kg, Pendahara 250,5 kg dan dari Tumbang Lahang 261,1 kg (Utomo et al., 2010).
DNA Mikrosatelit
Mikrosatelit adalah rangkaian pola nukleotida antara dua sampai enam pasang basa yang berulang secara berurutan. Mikrosatelit biasa digunakan sebagai penanda genetik untuk menguji kemurnian galur, studi filogenik, lokus pengendali sifat kuantitatif dan forensik. Mikrosatelit diamplifikasi menggunakan teknik Polymerase Chain Reaction (PCR) dengan beberapa pasang mikrosatelit. Hasil PCR dideteksi menggunakan teknik elektroforesis gel poliakrilamid (PAGE) yang dilanjutkan dengan pewarnaan perak (silver staining).
Mikrosatelit atau sekuen berulang sederhana (Short Tandem Repeats / STRs) adalah sekuen DNA genom dengan unit ulangan 1-6 pb (pasang basa) dengan panjang ulangan 10-100 pb, namun ada yang ditemukan lebih panjang lagi (Bennet, 2000; Goldenstein et al., 1995) atau sekuen yang berulang secara berurutan yang disusun berbagai kombinasi dari empat basa DNA yaitu adenin (A), cytosine (C), guanin (G), dan thymin (T) (Tautz, 1993). Pola ulangan DNA mikrosatelit dapat
berupa mono-, di-, tri- maupun tetra-nukleotida berulang (Tautz, 1993). Ulangan yang paling banyak adalah ulangan CA (atau GT pada untai lain) yang diduga ada
11 sekitar 35.000. Sekuen tersebut terdapat pada genom haploid manusia. Sekuen ini ditemukan di setiap 100 pb (Valdes et al., 1993). Ulangan nukleotida yang paling sering ditemukan pada mamalia adalah GT/AC (Hoelzel, 1998).
Mikrosatelit merupakan indikator pusat gen ideal (Valdes et al., 1993). Jumlahnya berlimpah, bersifat kodominan, memiliki polimorfik tinggi dan tersebar hampir di seluruh genom serta mudah ditemukan (Lehmann et al., 1996). Karakteristik tersebut menjadikan mikrosatelit sebagai penanda yang ideal untuk mengukur tingkat keragaman populasi.
Mikrosatelit terdapat melimpah dalam genom dan mudah ditemukan, sehingga sering digunakan dalam pemetaan genom (Weber, 1990), analisis keterpautan pada semua spesies serta dapat digunakan dalam rekonstruksi filogenik manusia (Goldenstein et al., 1995). Mikrosatelit juga digunakan sebagai penciri genetik (Lehmann et al., 1996) atau dapat digunakan sebagai penanda (marker) yang ideal untuk mengukur tingkat keragaman populasi (Valdes et al., 1993) karena memiliki jumlah alel yang tinggi serta ekspresi pola pitanya kodominan sehingga dengan mudah dapat membedakan individu homozigot dengan individu heterozigot. Marker ini juga bermanfaat dalam pemuliaan ternak seperti dalam mengidentifikasi ternak, asal usul, penentuan garis keturunan dan mengevaluasi sumberdaya genetik (Ciampolini et al.,1995).
Keragaman DNA Mikrosatelit
Keragaman mikrosatelit ditunjukan oleh variasi dalam jumlah pengulangan sekuen inti. Tingkat keragaman mikrosatelit secara positif berhubungan dengan panjang dari sekuen berulang (Weber, 1990). Perbedaan alel yang dihasilkan disebabkan perbedaan jumlah pengulangan basa nitrogen (Bennet, 2000).
Mikrosatelit dengan panjang pengulangan kurang dari 20 pb, berpeluang kecil polimorfik. Keragaman mikrosatelit ini berkaitan dengan ketidakstabilan lokus. Keragaman yang tinggi dari lokus mikrosatelit dihasilkan dari kecepatan mutasi yang tinggi yaitu diperkirakan pada kisaran 10-3 – 10-5 lokus/gamet/generasi (Lehmann et al., 1996). Rekombinasi yang tidak seimbang dan DNA polimerase slippage diduga menjadi penyebab ketidakstabilan dan keragaman dari mikrosatelit (Maskur, 2001).
Tipe dan kemurnian pengulangan merupakan bentuk dari keragaman mikrosatelit. Kategori mikrosatelit berdasarkan kemurnian pengulangan yaitu (1)
12 mikrosatelit berulang sederhana (perfect repeats) yang terdiri dari sekuen tanpa disisipi oleh penyela sepanjang unit berulangnya, (2) mikrosatelit berulang komplek (imperfect repeats) terdiri atas sekuen dengan satu atau lebih penyela dalam unit berulangnya dan (3) mikrosatelit berulang campuran (compound repeats) terdiri atas rangkaian perfect atau imperfect repeats berdampingan dengan sebuah rangkaian sekuen simple repeats yang lain (Weber, 1990).
Keragaman dalam mikrosatelit dapat dideteksi dengan menggunakan teknologi PCR. Sekuen pengapit yang khas disebut sebagai primer digunakan untuk mengamplifikasi daerah mikrosatelit. Primer pada mikrosatelit bersifat khas sehingga primer tersebut hanya mengamplifikasi dan dapat divisualisasikan menggunakan pewarnaan perak yang lebih sensitif dibandingkan pewarnaan dengan Ethidium Bromida (Tegelstrom, 1992). Banyak mikrosatelit yang ditemukan bersifat polimorf karena terpaut dengan daerah penyandi gen yang memiliki runutan cenderung lestari (conserved) sehingga sangat ideal untuk analisis keterpautan (Muladno, 2000).
Penanda Genetik
Penanda genetik adalah sebuah sifat genetik yang mudah diamati dan mempunyai pola penurunan sederhana (Kerje, 2003). Penanda genetik dapat diidentifikasi dengan berbagai teknik RFLP (Restriction Fragment Length Polymorphisms), RAPD (Random Amplyfied Polymorphism DNA), DNA minisatelit dan DNA mikrosatelit. Penanda genetik digunakan untuk mengukur respon genetik terhadap seleksi alam dan seleksi buatan (Gomez-Raya et al., 2002) dan untuk mengetahui keragaman genetik (Nei dan Kumar, 2000).
Analisis sekuens DNA yang memilki akurasi tinggi dimulai pada tahun 1977 melalui metode Sanger atau metode Maxam dan Gilbert. Metode tersebut berkembang menjadi suatu metode dengan dua prosedur yang berbeda (Brown, 1999), yaitu metode Sanger (Chain Termination Method) dan metode Maxam-Gilbert (Chemical Degradation Method) sehingga analisis genom menjadi lebih berkembang. Metode tersebut memiliki beda yaitu perkembangan. Metode Sanger lebih berkembang dibandingkan dengan metode Maxam-Gilbert karena lebih mudah, praktis dan efisien dilakukan (Muladno, 2002). Bersamaan dengan perkembangan teknik PCR dan teknik pendukung lain, maka proses perunutan atau sekuensing DNA secara keseluruhan dapat lebih cepat.
13 MATERI DAN METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Genetika dan Molekuler Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini dilaksanakan selama tujuh bulan, mulai Oktober 2010 sampai Mei 2011.
Materi Sampel Ternak
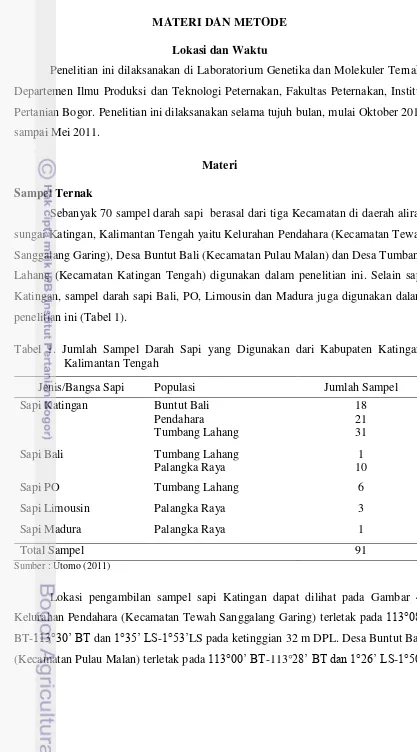
Sebanyak 70 sampel darah sapi berasal dari tiga Kecamatan di daerah aliran sungai Katingan, Kalimantan Tengah yaitu Kelurahan Pendahara (Kecamatan Tewah Sanggalang Garing), Desa Buntut Bali (Kecamatan Pulau Malan) dan Desa Tumbang Lahang (Kecamatan Katingan Tengah) digunakan dalam penelitian ini. Selain sapi Katingan, sampel darah sapi Bali, PO, Limousin dan Madura juga digunakan dalam penelitian ini (Tabel 1).
Tabel 1. Jumlah Sampel Darah Sapi yang Digunakan dari Kabupaten Katingan, Kalimantan Tengah
Jenis/Bangsa Sapi Populasi Jumlah Sampel
Sapi Katingan Buntut Bali 18
Pendahara 21
Tumbang Lahang 31
Sapi Bali Tumbang Lahang 1
Palangka Raya 10
Sapi PO Tumbang Lahang 6
Sapi Limousin Palangka Raya 3
Sapi Madura Palangka Raya 1
Total Sampel 91
Sumber : Utomo(2011)
14 LS pada ketinggian 27 m DPL. Desa Tumbang Lahang (Kecamatan Katingan Tengah) terletak pada 112°44’ BT-113°19’ BT dan 1°07’ LS-1°43’ LS pada ketinggian 30 m DPL. Sungai Katingan memiliki panjang 650 km yang melalui ketiga lokasi penelitian.
Sumber : Bhermana (2010)
Gambar 4. Peta Lokasi Pengambilan Sampel Sapi Katingan Bahan-bahan
Bahan-bahan yang digunakan dalam penelitian ini, adalah :
1) Bahan-bahan untuk pengambilan sampel, yaitu sampel darah, kapas, dan alkohol 96%.
2) Bahan-bahan untuk isolasi dan pemurnian DNA, yaitu SDS (sodium dosesil sulfat), proteinase-K, STE (Sodium Tris-EDTA), CIAA (Chloroform Iso Amil
15 Alkohol), larutan phenol, ethanol absolut, NaCl, TE (Tris EDTA) dan DW (destilated water).
3) Bahan-bahan untuk amplifikasi DNA, yaitu campuran destilated water (DW), primer, dNTP, MgCl2, buffer dan taq DNA polimerase.
4) Bahan-bahan untuk elektroforesis, yaitu larutan akrilamid, TEMED (N,N,N’,N’ -tetramethylethylenediamine), DW (destilated water), APS (ammonium peroxodisulfat) 10%, marker 20 bp dan larutan 5x TBE (tris boric acid-EDTA). 5) Bahan-bahan untuk silver staining (pewarnaan perak), yaitu DW, AgNO3,
NaOH, formaldehid, asam asetat dan amonia.
6) Primer yang digunakan, yaitu ILSTS028, ILSTS052 dan ILSTS056. Alat-alat
Alat-alat yang digunakan dalam penelitian ini dilihat dari cara penggunaannya, yaitu :
1) Alat-alat untuk pengambilan sampel seperti tabung venoject, spoit, rak tabung; alat yang umum digunakan seperti timbangan analitik, gelas ukur, gelas piala, freezer, pengaduk kaca, pipet ukur, autoclave, pisau atau silet dan sarung tangan.
2) Alat untuk ekstraksi dan pemurnian DNA seperti tabung Eppendorf, rak tabung Eppendorf, pipet mikro, tip, vortex, centrifuge, inkubator dan sarung tangan. 3) Alat-alat untuk PCR seperti tabung Eppendorf 0,2 µl, rak tabung, vortex,
centrifuge, mesin PCR dan sarung tangan.
4) Alat-alat elektroforesis seperti mesin elektroforesis, spiser, gelas ukur, pipet ukur, bulp, gelas Erlenmeyer, waterbath dan sarung tangan.
Prosedur Penelitian
Pengambilan Sampel
Pengambilan sampel darah sapi Katingan menggunakan disposible syringe 10 ml dari masing-masing sebanyak lima ml dari vena jugularis. Darah dimasukkan ke dalam tube test 12 ml yang telah diisi alkohol 96% sebanyak lima ml dan diberi label. Ketika darah ditambahkan alkohol, tabung segera dikocok dengan kuat agar darah benar-benar bercampur dengan alkohol dan tidak menggumpal, kemudian disimpan di dalam box plastik sampai darah akan diisolasi.
16 Ekstraksi dan Purifikasi DNA Genom
Ekstraksi dan purifikasi DNA dilakukan dengan metode Sambrook et al. (1989), yaitu : sebanyak 200 µl sampel darah dalam EtOH dipindahkan ke tabung 1,5 µl kemudian ditambahkan 1.000 µl DW/TE. Larutan dikocok kuat atau vortex dan didiamkan sekitar lima menit lalu disentrifugasi pada kecepatan 8.000 rpm selama sekitar lima menit. Bagian supernatan dibuang, kemudian diulangi lagi tahapan tersebut sampai bagian supernatan yang kedua dibuang. Setelah itu sebanyak 40 µl SDS 10%, 10 µl proteinase-K 5 mg/ml, dan 1 x STE sampai 400 µl ditambahkan setelahnya. Larutan dikocok dalam inkubator pada suhu 55 ºC selama dua jam, kemudian tambahkan 400 µl larutan phenol, 400 µl kloroform iso amil alkohol (CIAA), dan DNA diendapkan dengan 40 µl 5 m NaCl. Larutan dikocok pelan pada suhu ruang selama satu jam, kemudian disentrifugasi pada kecepatan 12.000 rpm selama lima menit. Bagian DNA (bening) dipindahkan dengan menggunakan pipet ke tabung 1,5 µl baru sebanyak 400 µl, kemudian ditambahkan 800 µl EtOH absolute dan 40 µl 5 M NaCl. Larutan dibekukan selama semalam. Endapan yang dihasilkan setelah proses pembekuan dilanjutkan dengan menambahkan 400 μl 70 % etanol dan disentrifugasi pada kecepatan 12.000 rpm selama lima menit. Etanol dibuang dan diuapkan dengan menggunakan pompa vakum, selanjutnya DNA dilarutkan dengan 80 μl 80% buffer TE.
Amplifikasi DNA Mikrosatelit
Primer yang digunakan untuk menganalisis keragaman DNA mikrosatelit sapi Katingan adalah ILSTS028, ILSTS052 dan ILSTS056 (Tabel 2).
Tabel 2. Informasi tentang Tiga Pasang Primer Pengapit DNA Mikrosatelit
Lokus Sekuen Primer Suhu
Annealing
Motif Ulangan
Lokasi dikromosom ILSTS028 F-5′ tccagattttgtaccagacc 3′
R-5′ gtcatgtcatacctttgagc 3′ 55 (CA)7 11
ILSTS052 F-5′ ctgtcctttaagaacaaacc 3′
R-5′ tgcaacttaggctattgacg 3′ 55 (CA)12 21
ILSTS056 F-5′ gctactgagtgatggtaggg 3′
R-5′ aatatagccctggaggatgg 3′ 55 (CA)11 12
Keterangan : F=Forward primer; R=Reverse
Sumber : Kathiravan et al. (2009)
17 Campuran PCR terdiri atas sampel darah 1 µl dengan tiga primer yaitu ILSTS028, ILSTS052 dan ILSTS056 sebanyak 0,05 µl; dNTP sebanyak 0,1 µl; MgCl2 sebanyak 0,25 µl; 10 kali buffer sebanyak 1,25 µl; DW sebanyak 9,3 µl dan taq DNA sebanyak 0,05 µl. Tabung Eppendorf yang berisi campuran sampel dan bahan PCR tersebut kemudian diputar dengan mesin vortex, lalu dimasukkan ke dalam mesin PCR. Mesin PCR kemudian dioperasikan dengan program perubahan suhu sebagai berikut : siklus pertama adalah denaturasi awal pada 94 ºC selama lima menit, diikuti dengan 35 siklus yang masing-masing terdiri atas denaturasi (94 ºC selama 20 detik), penempelan primer (55-60 ºC selama 30 detik), pemanjangan (72 ºC selama 45 detik), dan diakhiri dengan satu siklus berikutnya yaitu pemanjangan pada 72 ºC selama lima menit. Setelah siklus terakhir selesai, mesin PCR dimatikan dan tabung Eppendorf diambil untuk disimpan pada suhu ruang atau pada suhu 4 ºC atau dapat dianalisis lebih lanjut.
Elektroforesis Gel Poliakrilamida Vertikal (Vertical PAGE)
Alat dan bahan dalam pembuatan gel dipersiapkan. Elektroforesis gel akrilamid dikerjakan dengan menggunakan vertical electrophoresis apparatus. Poliakrilamid gel elektroforesis (PAGE) yang digunakan 6%. Komponen-komponen berikut dicampurkan dalam satu gelas ukur,: 30% Akrilamida sebanyak 5 ml, 5 kali TBE sebanyak 2,5 ml, DW 17,3 ml, Temed sebanyak 15 µl dan 10% APS sebanyak 150 µl. Semua larutan tersebut dicampur dengan menggoyang-goyangkan hingga homogen (menggunakan sarung tangan). Larutan tersebut segera dituangkan ke dalam cetakan gel kemudian ditutupi larutan gel dengan tutup cetakan yang telah disediakan. Sisir dipasang untuk membuat lubang tempat sampel (sumur) dan dibiarkan pada suhu ruangan sehingga gel membeku.
Elektroforesis akrilamid dijalankan pada tegangan 100 volt selama lebih kurang dua jam. Setiap sumur pada gel diisi dengan produk PCR sebanyak dua µl yang dicampur dengan 0,25 µl larutan pemberat (loading dye). Satu sumur gel terakhir diisi dengan 1 µl DNA marker 20 bp sebagai ukuran standar pita-pita DNA hasil amplifikasi, setelah elektroforesis selesai dilakukan pewarnaan perak (silver staining). Pewarnaan perak menggunakan empat larutan yaitu larutan A (DW 200 ml; AgNO3 0,2 g; 10 N NaOH 80 µl dan ammonia 800 µl), DW sebanyak 200 ml, larutan B (DW 200 ml, NaOH 6 g, formaldehid 200 µl) dan larutan C (DW 100 µl,
18 100 µl asetat). Gel kemudian dimasukkan ke dalam larutan A dan didiamkan selama lebih kurang delapan menit, kemudian gel dicuci dengan DW selama dua menit. Larutan B dipanaskan terlebih dahulu di dalam waterbath pada suhu 60-65 ºC sampai siap digunakan. Selanjutnya gel direndam dan digoyang-goyangkan perlahan dalam larutan B sampai muncul pita. Setelah pita muncul, gel dicuci dengan larutan C. Selesai mencuci dengan larutan C kemudian gel dibungkus dengan plastik mika dan diberi nomor pada setiap sampel.
Penentuan Posisi Pita DNA
Penentuan genotipe dilakukan dengan cara memperhatikan dan menghitung jumlah pita pada gel elektroforesis. Heterozigot ditunjukkan dengan dua pita, sedangkan homozigot satu pita. Penentuan pita yang paling bawah diberi sandi A, B, C dan seterusnya sampai pita paling atas. Pita yang memiliki laju sama merupakan alel yang homolog (Sumantri et al. 2008).
Analisis Data
Analisis data dilakukan dengan mempelajari pola pita (alel) yang terdapat pada sapi Katingan, sapi Bali, PO, Madura dan Limousin. Analisis data DNA mikrosatelit menggunakan software POPGENE Versi 32 antara lain adalah frekuensi alel, frekuensi genotipe dan derajat heterozigositas.
Frekuensi Alel
Frekuensi alel lokus ILSTS028, ILSTS052 dan ILSTS056 dihitung dengan menggunakan rumus Nei dan Kumar (2000):
Keterangan : j ≠ 1
xi = frekuensi alel ke-i
nij = jumlah individu untuk genotip ij nii = jumlah individu untuk genotip ii N = jumlah sampel
19 Frekuensi Genotipe
Frekuensi genotipe ditentukan dengan cara membagi jumlah sampel sapi Katingan yang memiliki genotipe tipe tertentu dengan seluruh jumlah sapi yang diamati dengan menggunakan rumus Nei dan Kumar (2000):
Keterangan :
xi = frekuensi genotipe ke-i ni = jumlah individu bergenotipe i N = jumlah individu sampel Derajat Heterozigositas
Derajat heterozigositas ditentukan dengan menggunakan rumus Nei dan Kumar (2000):
Keterangan :
= nilai heterozigositas xi = frekuensi alel ke-i n = jumlah individu
Rataan heterozigositas pada setiap lokus dihitung dengan menggunakan rumus Nei dan Kumar (2000):
Keterangan :
= rataan heterozigositas semua lokus hj = heterozigositas lokus ke-j
r = jumlah lokus
20 HASIL DAN PEMBAHASAN
Amplifikasi DNA Mikrosatelit
Amplifikasi DNA dilakukan dengan tiga macam primer yaitu ILSTS028, ILSTS052 dan ILSTS056 serta masing-masing lokus menganalisis 70 sampel DNA. Hasil amplifikasi menunjukkan lokus ILSTS028 dapat mengamplifikasi 68 sampel darah sapi Katingan yaitu 31 sampel dari Tumbang Lahang, 24 sampel dari Pendahara dan 13 sampel dari Buntut Bali. Lokus ILSTS052 dapat mengamplifikasi 68 sampel darah sapi katingan yaitu 31 sampel dari Tumbang Lahang, 24 sampel dari Pendahara dan 13 sampel dari Buntut Bali. Lokus ILSTS056 dapat mengamplifikasi 69 sampel darah sapi katingan yaitu 30 sampel dari Tumbang Lahang, 26 sampel dari Pendahara dan 13 sampel dari Buntut Bali . Sampel darah sapi Bali, Madura, PO dan Limousin yang berasal dari Kalimantan Tengah masing-masing sebanyak 11, 1, 6 dan 3 sampel dari lokus ILSTS028, ILSTS052 dan ILSTS056 juga berhasil diamplifikasi.
Suhu annealing setiap lokus berbeda-beda berdasarkan beberapa kali optimasi. Suhu annealing lokus ILSTS028 dan ILSTS052 adalah 55 0C dan berbeda dengan suhu annealing lokus ILSTS056 sebesar 60 0C. Perbedaan suhu annealing yang digunakan oleh Kathiravan et al. (2009) pada penelitiannya disebabkan ternak yang digunakan adalah kerbau Marathwada, sedangkan pada penelitian ini adalah sapi Katingan (sapi lokal Kalimantan Tengah). Suhu annealing menentukan ketebalan pita DNA yang diperoleh dari elektroforesis. Kisaran temperatur penempelan yang digunakan antara 36-72 0C, namun suhu yang biasa digunakan antara 50-60 0C (Muladno, 2002).
Amplifikasi DNA mikrosatelit pada setiap populasi yang menggunakan lokus ILSTS028, ILSTS052 dan ILSTS056 menghasilkan sifat polimorfik yang tinggi. Hal ini menunjukkan bahwa jumlah mikrosatelit berlimpah, bersifat kodominan, memiliki polimorfik tinggi dan tersebar hampir di seluruh genom serta mudah ditemukan (Lehmann et al.,1996). Karakteristik tersebut menjadikan mikrosatelit sebagai penanda yang ideal untuk mengukur tingkat keragaman populasi.
21 perak, tetapi sering kali ditemukan pita-pita tambahan yang bukan termasuk pita target. Pita-pita tambahan tersebut dihasilkan dari beberapa proses, seperti penyelipan selama amplifikasi PCR. Kemunculan pita-pita tersebut juga mencerminkan mikroheterogenitas dalam panjangan ulangan dinukleotida secara in-vitro (Litt dan Luty, 1989). Dua molekul DNA untai ganda hasil amplifikasi pada siklus pertama menjadi DNA target dan dilipatgandakan menjadi empat molekul DNA dan selanjutnya empat molekul baru ini dilipatgandakan jumlahnya menjadi delapan dan seterusnya (Muladno, 2002).
Keragaman DNA Mikrosatelit
Hasil penelitian menunjukkan bahwa lokus ILSTS052 dan ILSTS056 bersifat polimorfisme. Hal ini didasarkan pada jumlah unit ulangan yang lebih dari 10 ulangan tetapi berbeda dengan lokus ILSTS028 yang hanya memiliki jumlah unit ulangan kurang dari 10 ulangan. Menurut Winaya (2000), polimorfisme akan semakin tinggi apabila unit ulangan tergandakan lebih dari 10 kali lipat. Hasil pengukuran lokus dari ketiga sub populasi sapi Katingan menghasilkan jumlah alel yang beragam. Lokus ILSTS028 menghasilkan 20 alel, lokus ILSTS052 menghasilkan 13 alel dan lokus ILSTS056 menghasilkan 19 alel. Hal ini menunjukkan bahwa keragaman genetik dapat dilihat dari jumlah alel di setiap lokus dan heterozigositasnya (Sun et al., 2008).
Lokus ILSTS028
Lokus ILSTS028 memiliki 20 macam alel dari ketiga populasi sapi katingan yaitu alel A, B, C, D, E, F, G, H, I, J, K, L, M, N, O, P, Q, R, S dan T. Pemberian simbol alel dengan menggunakan urutan abjad sesuai dengan ukuran alel. Lokus ILSTS028 memiliki jumlah alel terbanyak dibandingkan jumlah alel lokus-lokus lainnya. Jumlah alel yang dihasilkan menunjukkan bahwa populasi sapi Katingan memiliki tingkat keragaman genetik yang relatif tinggi. Menurut Karthickeyan et al. (2009), meningkatnya jumlah alel pada lokus yang berbeda akan meningkatkan rata-rata keragaman genetik dalam populasi. Hasil amplifikasi PCR sapi Katingan terhadap lokus ILSTS028 menghasilkan alel yang sebagian ditampilkan pada Gambar 5.
22 Keterangan : M = Marker
a = Sapi Bali b = Sapi PO
Gambar 5. Penentuan Genotipe Lokus ILSTS028 pada Sapi Katingan dan Sapi Lokal Lainnya
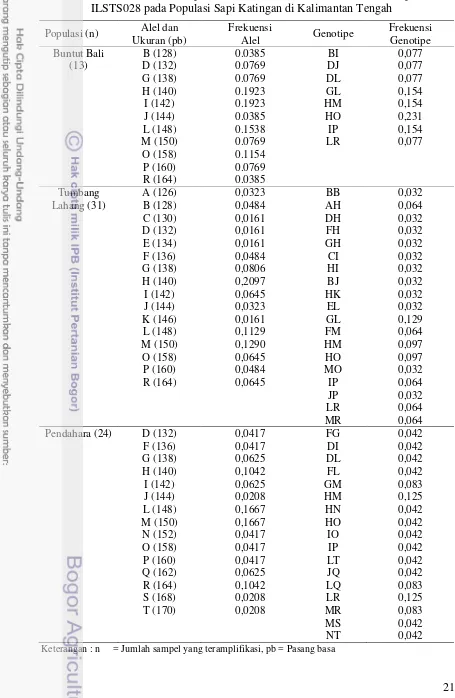
Data mengenai jumlah alel dan frekuensi alel untuk masing-masing populasi dapat dilihat pada Tabel 3. Jumlah alel lokus ILSTS028 pada populasi Buntut Bali, Tumbang Lahang dan Pendahara berturut-turut menghasilkan 11 alel, 16 alel dan 15 alel. Jumlah alel lebih banyak ditemukan pada populasi Tumbang Lahang karena sampel darah lebih banyak diambil pada populasi Tumbang Lahang dibandingkan dengan populasi Buntut Bali dan Pendahara, tetapi frekuensi alel tertinggi dan terendah lokus ILSTS028 ditemukan pada populasi Tumbang Lahang. Frekuensi alel tertinggi adalah alel H sebesar 0,2097 dan frekuensi alel terendah adalah alel C, D, E dan K sebesar 0,0167. Frekuensi genotipe tertinggi pada ketiga populasi Buntut Bali, Tumbang Lahang dan Pendahara berturut-turut adalah genotipe HO sebesar 0,231, LG sebesar 0,129 serta MH dan RL sebesar 0,125. Frekuensi genotipe terendah pada populasi Buntut Bali adalah genotipe BI, DJ, dan DL sebesar 0,077; dan pada populasi Tumbang Lahang adalah genotipe BB, DH, FH, GH, CI, HI, BJ, HK, EL, MO dan JP sebesar 0,032; serta pada populasi Pendahara adalah genotipe FG, DI, DL, FL, HN, HO, IO, IP, LT, JQ, MS dan NT sebesar 0,042.
M 49 50 51 52 53 54 55 56 57 58 59 60
MR FNb GL FL LQ DL FM DLa DLa DL FH LR (+) (-)
100 bp 300 bp
200 bp
150 bp
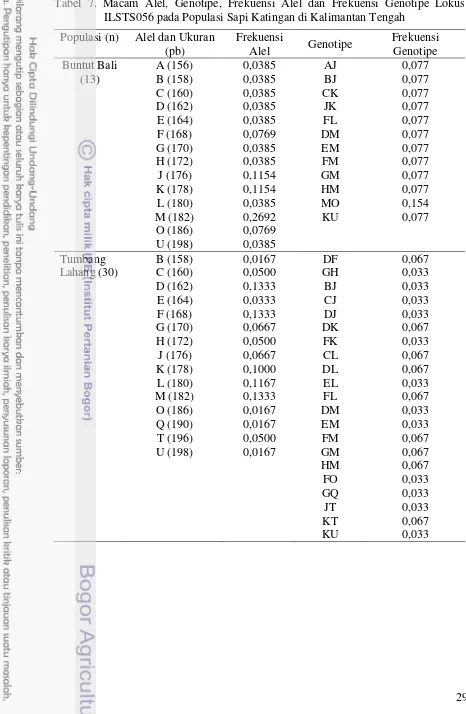
23 Tabel 3. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe Lokus
ILSTS028 pada Populasi Sapi Katingan di Kalimantan Tengah
Populasi (n) Alel dan
Keterangan : n = Jumlah sampel yang teramplifikasi, pb = Pasang basa
24 Frekuensi alel dan jenis alel pada lokus ILSTS028 yang sangat bervariasi dan beragam dapat dilihat pada Gambar 6. Lokus ILSTS028 menghasilkan beberapa alel yang hanya ditemukan pada populasi tertentu yang disebut alel spesifik.
Gambar 6. Frekuensi dan Macam Alel pada Lokus ILSTS028
Alel spesifik yang ditemukan pada populasi Tumbang Lahang yaitu alel A, C, E dan K. Alel spesifik yang ditemukan pada populasi Pendahara yaitu alel N, Q, S dan T, alel yang spesifik tidak ditemukan pada populasi Buntut Bali. Hal ini menyebabkan tidak terdapat alel khusus di dalam populasi Buntut Bali sebagai penciri dari sapi Katingan dari Buntut Bali. Alel spesifik dapat digunakan sebagai pembeda antara ketiga populasi (Sarbaini, 2004).
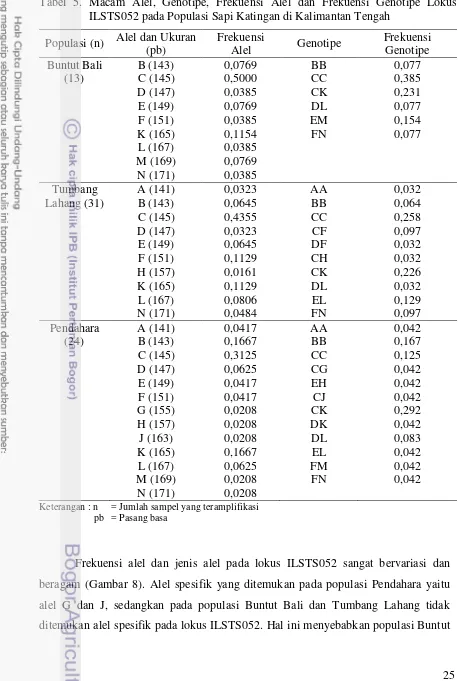
Informasi mengenai jumlah, macam dan frekuensi alel sapi Katingan dan sapi lokal di Kabupaten Katingan pada lokus ILSTS028 dapat dilihat pada Tabel 4. Frekuensi alel tertinggi yang ditemukan pada lokus ILSTS028 dari ketiga populasi adalah alel H sebesar 0,1691. Frekuensi alel tertinggi terdapat pada sapi Bali, PO dan Limousin berturut-turut yaitu 0,2727 (alel L), 0,1667 (alel I, J, M dan P) dan 0,3333 (alel L dan R). Sapi Madura hanya memiliki dua jenis alel dan memiliki nilai frekuensi alel yang sama besar. Frekuensi alel tertinggi pada masing-masing populasi dapat mengindikasikan bahwa alel-alel mendominasi alel lainnya di dalam populasi tersebut. Frekuensi genotipe tertinggi terdapat pada sapi Katingan, Bali dan Limousin berturut-turut yaitu HM (0,1176), DL (0,3636) dan LR (0,6667). Populasi sapi PO tidak memiliki nilai frekuensi genotipe tertinggi dikarenakan nilai frekuensi genotipe yang sama pada semua macam genotipe.
Macam Alel
25
Macam Alel Frekuensi
26 Lokus ILSTS052
Lokus ILSTS052 dari ketiga populasi sapi Katingan menghasilkan 13 macam alel yaitu alel A, B, C, D, E, F, G, H, J, K, L, M dan N. Hasil amplifikasi PCR sapi Katingan terhadap lokus ILSTS052 disajikan pada Gambar 7.
Keterangan : M = Marker a = Sapi Bali b = Sapi PO
Gambar 7. Penentuan Genotipe Lokus ILSTS052 pada Sapi Katingan dan Sapi Lokal Lainnya
Data mengenai jumlah alel dan frekuensi alel untuk masing-masing populasi dapat dilihat pada Tabel 5. Jumlah alel lokus ILSTS052 pada populasi Buntut Bali, Tumbang Lahang dan Pendahara berturut-turut menghasilkan 9, 10 dan 13 alel. Jumlah alel lebih banyak ditemukan pada populasi Pendahara. Frekuensi alel tertinggi yang ditemukan pada populasi Buntut Bali adalah alel C sebesar 0,5000 dan frekuensi alel terendah yang ditemukan pada populasi Pendahara adalah alel G, H, J, M dan N sebesar 0,0208. Frekuensi genotipe tertinggi pada ketiga populasi Buntut Bali, Tumbang Lahang dan Pendahara berturut-turut adalah genotipe CC sebesar 0,385, CC sebesar 0,258 dan CK sebesar 0,292. Frekuensi genotipe terendah pada populasi Buntut Bali adalah genotipe BB, DL dan FN sebesar 0,077 dan pada populasi Tumbang Lahang adalah genotipe AA, DF, CH dan DL sebesar 0,032 sedangkan pada populasi Pendahara adalah genotipe AA, CG, EH, CJ, DK, EL, FM dan FN sebesar 0,042.
M 61 62 63 64 65 66 67 68 69 70 71 72
CJb CJ ELa EL FMa EL EL EL EL FM ELb FMa
150bp 200bp
100bp 300bp
(+) (-)
27 Tabel 5. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe Lokus
ILSTS052 pada Populasi Sapi Katingan di Kalimantan Tengah Populasi (n) Alel dan Ukuran
(pb)
Keterangan : n = Jumlah sampel yang teramplifikasi
pb = Pasang basa
Frekuensi alel dan jenis alel pada lokus ILSTS052 sangat bervariasi dan beragam (Gambar 8). Alel spesifik yang ditemukan pada populasi Pendahara yaitu alel G dan J, sedangkan pada populasi Buntut Bali dan Tumbang Lahang tidak ditemukan alel spesifik pada lokus ILSTS052. Hal ini menyebabkan populasi Buntut
28 Bali dan Tumbang Lahang tidak memiliki alel spesifik sebagai penciri dari sapi Katingan pada populasi tersebut.
Gambar 8. Frekuensi dan Macam Alel Lokus ILSTS052
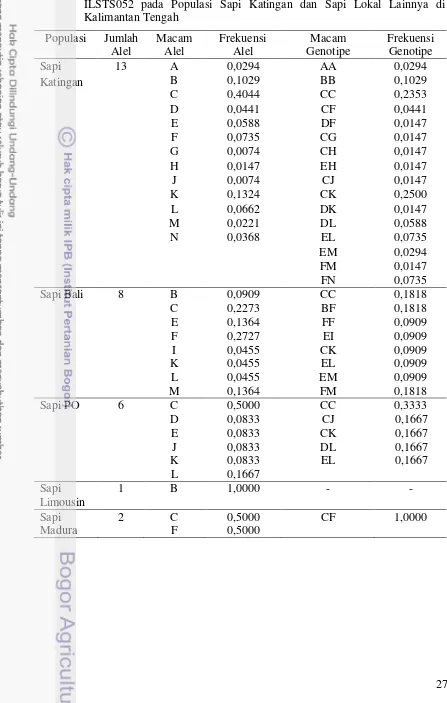
Berdasarkan Tabel 6, frekuensi alel C tertinggi pada lokus ILSTS052 dari ketiga populasi yaitu sebesar 0,4044. Frekuensi alel tertinggi pada sapi Bali dan PO berturut-turut yaitu 0,2727 (alel F) dan 0,5000 (alel C). Sapi Limousin hanya memiliki satu macam alel saja, sedangkan sapi Madura hanya terdapat dua macam alel dan memiliki nilai frekuensi alel yang sama besar. Tinggi rendah frekuensi alel sapi Limousin dan sapi Madura tidak dapat dibandingkan. Hal ini dikarenakan jumlah sampel yang digunakan terbatas. Frekuensi alel tertinggi pada masing-masing populasi dapat menyatakan bahwa alel-alel mendominasi alel lainnya di dalam populasi tersebut. Frekuensi genotipe tertinggi pada sapi Katingan, Bali dan PO berturut-turut adalah CK sebesar 0,2500, serta CF, BF dan FM sebesar 0,1818 dan CC sebesar 0,3333. Sapi Limousin memiliki satu macam alel (alel B) dan sapi Madura memiliki satu macam genotipe (CF), sehingga kedua populasi sapi tersebut tidak memiliki nilai frekuensi genotipe tertinggi pada lokus ILSTS052. Informasi mengenai jumlah, macam alel, genotipe, frekuensi alel dan frekuensi genotipe pada sapi Katingan dan sapi lokal lainnya lokus ILSTS052 dapat dilihat pada Tabel 6.
Macam Alel
30 Lokus ILSTS056
Lokus ILSTS056 dari ketiga populasi sapi Katingan menghasilkan 19 macam alel yaitu alel A, B, C, D, E, F, G, H, J, K, L, M, N, O, P, Q, R, T dan U. Hasil amplifikasi PCR sapi Katingan terhadap lokus ILSTS052 disajikan pada Gambar 9.
Keterangan : M = Marker a = Sapi Bali
Gambar 9. Penentuan Genotipe Lokus ILSTS056 pada Sapi Katingan dan Sapi Lokal Lainnya
Jumlah alel dan frekuensi alel pada sapi Katingan pada setiap populasi dapat dilihat pada Tabel 7. Jumlah alel lokus ILSTS056 pada populasi Buntut Bali, Tumbang Lahang dan Pendahara berturut-turut menghasilkan 14, 15 dan 17 alel. Frekuensi alel tertinggi ditemukan pada populasi Buntut Bali yaitu alel M sebesar 0,2692 dan frekuensi alel terendah ditemukan pada populasi Tumbang Lahang yaitu alel B, O, Q dan U masing-masing sebesar 0,0167. Frekuensi genotipe tertinggi pada ketiga populasi Buntut Bali, Tumbang Lahang dan Pendahara berturut-turut adalah genotipe MO sebesar 0,154, serta DF, DK, CL, DL, FL, FM, GM, HM dan KT sebesar 0,067 dan FL sebesar 0,115.
200 bp
160 bp
100 bp 300 bp
M 1 2 3 4 5 6 7 8 9 10 11 12
EKa CK CK CL CG KR EK KR GM FN GM GM (-)
(+)
31 Tabel 7. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe Lokus
ILSTS056 pada Populasi Sapi Katingan di Kalimantan Tengah Populasi (n) Alel dan Ukuran
32 Populasi (n) Alel dan Ukuran
(pb)
Keterangan : n = Jumlah sampel yang teramplifikasi
pb = Pasang basa
Alel spesifik yang ditemukan pada populasi Pendahara yaitu alel N, P dan R. Alel spesifik yang ditemukan pada populasi Tumbang Lahang yaitu alel Q. Alel spesifik pada lokus ILSTS056 tidak ditemukan pada populasi Buntut Bali, sehingga di dalam populasi Buntut Bali tidak terdapat alel spesifik sebagai penciri dari sapi Katingan. Semua macam alel ditemukan pada populasi Pendahara (kecuali alel N dan Q). Hal ini mengindikasikan populasi Pendahara memiliki keragaman yang tinggi dibandingkan dengan populasi Tumbang Lahang dan Buntut Bali (Gambar 10). Frekuensi alel dan jenis alel pada lokus ILSTS056 sangat bervariasi dan beragam (Gambar 10).
33 Gambar 10. Frekuensi dan Macam Alel Lokus ILSTS056
Informasi mengenai jumlah, macam dan frekuensi alel pada sapi Katingan dan sapi lokal lainnya lokus ILSTS056 dapat dilihat secara lengkap pada Tabel 8. Frekuensi alel tertinggi lokus ILSTS056 pada sapi Katingan yaitu alel K sebesar 0,1377. Keterbatasan sampel darah yang dianalisis menyebabkan Sapi Limosin dan sapi Madura hanya menunjukkan dua jenis alel dan memiliki nilai frekuensi alel yang sama besar, sehingga pada sapi Limousin dan sapi Madura tidak dapat dibandingkan tinggi rendahnya frekuensi alel. Frekuensi alel tertinggi pada masing-masing populasi mengindikasikan bahwa alel-alel mendominasi alel lainnya di dalam populasi tersebut. Frekuensi genotipe pada sapi Katingan adalah FL sebesar 0,0869, sedangkan pada sapi Bali dan PO memiliki nilai frekuensi yang sama. Sapi Limousin dan Madura pada lokus ILSTS056 hanya memiliki satu macam genotipe karena sampel yang digunakan terbatas.
Macam Alel
35
Nilai Heterozigositas bervariasi antara 0,00 hingga 1,00. Nilai heterozigositas dari ketiga lokus yang digunakan dalam penelitian ini disajikan pada Tabel 9.
36 Nilai heterozigositas lokus ILSTS028 pada populasi Buntut Bali dan Pendahara bernilai ( ) = 1,00; sedangkan nilai heterozigositas pada populasi Tumbang Lahang bernilai ( ) = 0,97. Hal ini membuktikan bahwa lokus ILSTS028 pada populasi Buntut Bali dan Pendahara memiliki keragaman yang sangat tinggi dibandingkan dengan populasi Tumbang Lahang. Heterozigositas lokus ILSTS052 pada populasi Buntut Bali bernilai ( ) = 0,54; bernilai ( ) = 0,67 pada populasi Pendahara dan pada populasi Tumbang Lahang bernilai ( ) = 0,64. Heterozigositas lokus ILSTS056 pada ketiga populasi bernilai ( ) = 1,00. Hal ini menunjukkan bahwa pada ketiga populasi tersebut memiliki keragaman genetik sangat tinggi. Heterozigositas pada sapi lokal lain pada umumnya bernilai ( ) = 1,00. Hal ini menunjukkan bahwa sapi-sapi lokal yang berada di Kalimantan Tengah memiliki tingkat keragaman yang tinggi. Nilai heterozigositas pada sapi Limousin lokus ILSTS052 sebesar ( ) = 0. Hal ini disebabkan oleh hanya ditemukan satu macam alel (alel B) pada sapi Limousin lokus ILSTS052. Prahasta (2001) menyatakan bahwa semakin banyak sampel yang digunakan pada suatu lokus maka makin besar nilai heterozigositas. Hal tersebut berbeda dengan hasil penelitian ini bahwa hanya lokus tertentu yang dapat menentukan tinggi rendahnya suatu nilai heterozigositas. Rataan heterozigositas pada masing-masing populasi yaitu sebesar 0,8462 (Buntut Bali), sebesar 0,8889 (Pendahara) dan sebesar 0,8710 (Tumbang Lahang). Takezaki dan Nei (1996) menekankan untuk mengukur suatu keragaman genetik dapat dilihat dari rataan heterozigositas pada lokus-lokus mikrosatelit yaitu antara 0,3 dan 0,8. Peneletian ini telah sesuai dengan kriteria tersebut. Rataan Heterozigositas ( ) dari masing-masing lokus dapat dilihat pada Tabel 10.
Tabel 10. Rataan Heterozigositas ( ) dari Populasi Sapi Katingan dan Sapi Lokal Lainnya
No. Lokus Jumlah Sampel (n) Heterozigositas ( )
1 ILSTS028 89 0,989
2 ILSTS052 89 0,629
3 ILSTS056 90 1,000
Rataan Heterozigositas ( ) 0,873
Nilai heterozigositas pada lokus ILSTS028 sebesar ( ) = 0,989, lokus ILSTS052 sebesar ( ) = 0,629 dan lokus ILSTS056 sebesar ( ) = 1,000. Tabel 10 menunjukkan bahwa lokus ILSTS056 memiliki nilai heterozigositas ( ) tertinggi
37 dibandingkan dengan kedua lokus lainnya. Rataan Heterozigositas ( ) dari semua lokus memiliki nilai yang cukup tinggi yaitu 0,873. Rataan heterozigositas ( ) yang tinggi pada populasi menunjukkan bahwa sapi-sapi tersebut mengandung alel-alel sapi lain (Abdullah, 2008). Informasi mengenai Rataan Heterozigositas ( ) pada beberapa bangsa sapi di Indonesia dapat dilihat pada Tabel 11.
Tabel 11. Informasi Mengenai Rataan Heterozigositas ( ) pada Beberapa Bangsa Sapi di Indonesia
Bangsa Ternak Lokus ( ) Referensi
Sapi Pesisir 6 lokus 0,86 Harmayanti (2004) Sapi Bali 16 lokus 0,33 Winaya et al. (2007) Sapi Madura 16 lokus 0,31 Winaya et al. (2007)
Sapi PO 6 lokus 0,73 Abdullah (2008)
Sapi Aceh 16 lokus 0,62 Abdullah (2008)
Sapi Katingan 15 lokus 0,56 Utomo (2011) Sapi Katingan* 3 lokus 0,87 Hasil Penelitian Keterangan: (*) = Terdiri atas Sapi Katingan, Bali, PO, Madura dan Limousin
38 KESIMPULAN DAN SARAN
Kesimpulan
Sapi Katingan memiliki lokus ILSTS028, ILSTS052 dan ILSTS056 yang bersifat polimorfik dengan jumlah alel masing-masing sebanyak 20, 13 dan 19 alel. Alel spesifik ditemukan di setiap populasi sapi Katingan yang dapat digunakan sebagai pembeda genetik antar populasi. Rataan heterozigositas tertinggi terdapat pada lokus ILSTS056 sebesar ( ) = 1,000 dan terendah terdapat pada lokus ILSTS052 sebesar ( ) = 0,629.
Saran
IDENTIFIKASI KERAGAMAN DNA MIKROSATELIT LOKUS
ILSTS028, ILSTS052 DAN ILSTS056 PADA SAPI KATINGAN
DI KALIMANTAN TENGAH
SKRIPSI ERWINSYAH
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
IDENTIFIKASI KERAGAMAN DNA MIKROSATELIT LOKUS
ILSTS028, ILSTS052 DAN ILSTS056 PADA SAPI KATINGAN
DI KALIMANTAN TENGAH
SKRIPSI ERWINSYAH
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
ERWINSYAH. D14070201. 2011. Identifikasi Keragaman DNA Mikrosatelit Lokus ILSTS028, ILSTS052 dan ILSTS056 pada Sapi Katingan di Kalimantan Tengah. Skripsi. Mayor Ilmu Produksi dan Teknologi Peternakan. Institut Pertanian Bogor.
Pembimbing Utama : Dr. Jakaria, S.Pt., M.Si.
Pembimbing Anggota : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.
Sapi Katingan adalah salah satu jenis sapi lokal yang terdapat di Kalimantan Tengah dan sudah beradaptasi dengan lingkungan sekitarnya. Sapi Katingan umumnya dipelihara oleh suku Dayak dan hidup disepanjang daerah aliran sungai Katingan. Informasi sapi Katingan saat ini sangat terbatas, sehinga dilakukan strategi pelestarian dan pengembangan sapi Katingan di Kalimantan Tengah. Sapi Katingan bukan hanya mempunyai nilai kultural yang tinggi, selain itu juga memiliki peranan terhadap pendapatan keluarga yaitu sebesar 18-28%, terbesar kedua setelah komoditas karet. Tujuan penelitian ini yaitu untuk mengidentifikasi keragaman genetik sapi lokal di Kalimantan Tengah dengan menggunakan penanda molekuler DNA mikrosatelit yang menggunakan lokus ILSTS028, ILSTS052 dan ILSTS056.
Penelitian dilaksanakan di Laboratorium Genetika dan Molekuler Ternak selama tujuh bulan, mulai Oktober 2010 sampai Mei 2011. Sampel darah sapi Katingan yang digunakan sebanyak 70 unit yang berasal dari tiga Kecamatan di daerah aliran sungai Katingan, Kalimantan Tengah yaitu Kelurahan Pendahara (Kecamatan Tewah Sanggalang Garing) sebanyak 21 ekor, Desa Buntut Bali (Kecamatan Pulau Malan) sebanyak 18 ekor, dan Desa Tumbang Lahang (Kecamatan Katingan Tengah) sebanyak 31 ekor serta sampel darah sapi Bali, Madura, PO dan Limousin masing-masing sebanyak 11, 1, 6 dan 3 sampel. Amplifikasi DNA dilakukan dengan tiga macam primer yaitu ILSTS028, ILSTS052 dan ILSTS056 dengan masing-masing suhu annealing ILSTS028 dan ILSTS052 adalah 55 0C, dan suhu annealing ILSTS056 adalah 60 0C.
Data dianalisis berdasarkan jumlah alel, frekuensi alel dan heterozigositas. Hasil analisis menunjukkan bahwa lokus ILSTS028 menghasilkan 20 alel (A, B, C, D, E, F, G, H, I, J, K, L, M, N, O, P, Q, R, S dan T), lokus ILSTS052 menghasilkan 13 alel (A, B, C, D, E, F, G, H, J, K, L, M dan N), dan lokus ILSTS056 menghasilkan 19 alel (A, B, C, D, E, F, G, H, J, K, L, M, N, O, P, Q, R, T dan U). Nilai heterozigositas lokus ILSTS028 pada populasi Buntut Bali dan Pendahara bernilai ( )=1,00 sedangkan nilai heterozigositas pada populasi Tumbang Lahang bernilai ( )=0,96. Nilai heterozigositas lokus ILSTS052 pada populasi Buntut Bali bernilai ( )=0,54, pada populasi Pendahara bernilai ( )=0,67 dan pada populasi Tumbang Lahang bernilai ( )=0,64. Nilai Heterozigositas lokus ILSTS056 pada ketiga populasi bernilai sama yaitu ( )=1,000; hal ini menunjukkan bahwa keragaman genetik pada ketiga populasi tersebut sangat tinggi. Nilai rataan heterozigositas pada ketiga lokus sebesar ( )= 0,873.
ABSTRACT
Polymorphism Identification of ILSTS028, ILSTS052 and ILSTS056 Microsatellite DNA Loci on Katingan Cattle in Central Kalimantan
Erwinsyah, Jakaria and C. Sumantri
This study aimed to determine the genetic diversity of Katingan cattle population in Central Kalimantan by DNA microsatellite markers ILSTS028, ILSTS052 and ILSTS056 loci. The 70 blood samples used in this study originated from three subpopulation of Pendahara sub populations (26 samples), Buntut Bali (13 samples) and Tumbang Lahang (31 samples), and other samples were Bali cattle (11 samples), Madura cattle (1 sample), Limousin cattle (3 samples) and PO cattle (6 samples). The amplification of microsatellite DNA marker was done by PCR (Polymerase Chain Reaction). The PCR product was then electrophoresed using 6% polyacrilamide gel followed by silver staining. The data was analyzed to get allele frequency, genotype frequency and heterozygosity value. Microsatellite DNA of loci showed high polymorphisms. ILSTS028 locus had 20 alleles in Katingan cattle with the highest allele frequency was allele H (0.1691) in Tumbang Lahang population and the lowest allele C, D, E and K (0.0167) in Tumbang Lahang population. The highest allele frequency of Bali, PO, Limousin and Madura cattle was alleles H and R in Madura cattle (0.5000). The lowest allele frequency was alleles I, K and L (0.0455) in Bali cattle. ILSTS052 locus had 13 alleles in Katingan cattle with the highest allele frequency was allele C (0.5000) in Buntut Bali populations and lowest allele G, H, J, M and N (0.0208) in Pendahara population. Allele B had the highest frequency in Limousin cattle (1.0000) and alleles E and I the lowest frequency in Bali cattle (0.0455). ILSTS056 locus had 19 alleles in Katingan cattle with the highest allele frequency was allele M (0.2692) in Buntut Bali population and the lowest is allele B, O, Q and U (0.0167) in Tumbang Lahang population. Alleles D and I in Limuosin cattle and alleles A and D in Madura cattle had the highest frequency (0.5000) and alleles D, L and M the lowest frequency in Bali cattle (0.0455). The heterozygosity value from ILSTS028, ILSTS052 and ILSTS056 locus of Katingan cattle with the others cattle were 0.989, 0.629 and 1.000, respectively.
IDENTIFIKASI KERAGAMAN DNA MIKROSATELIT LOKUS
ILSTS028, ILSTS052, DAN ILSTS056 PADA SAPI KATINGAN
DI KALIMANTAN TENGAH
ERWINSYAH D14070201
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman DNA Mikrosatelit Lokus ILSTS028, ILSTS052 dan ILSTS056 pada Sapi Katingan di Kalimantan Tengah
Nama : Erwinsyah
NIM : D14070201
Menyetujui,
Tanggal Ujian: 1 Nopember 2011 Tanggal Lulus: Pembimbing Utama,
(Dr. Jakaria, S.Pt., M.Si.) NIP: 19660105 199303 1 001
Pembimbing Anggota,
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) NIP: 1951212 198603 1 004
Mengetahui: Ketua Departemen,
Ilmu Produksi dan Teknologi Peternakan
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 18 Maret 1989 di Jakarta. Penulis adalah anak pertama dari tiga bersaudara dari pasangan Bapak Sa’al dan Ibu Eni Wahyuni.
Penulis mengawali pendidikan dasar pada tahun 1995 di SD Negeri Pisangan 1 Ciputat, Tangerang. Pendidikan lanjutan tingkat pertama di SLTP N 1 Ciputat, Tangerang dimulai pada tahun 2001 dan diselesaikan pada tahun 2004. Penulis melanjutkan di SMA Keluarga Widuri, Lebak Bulus, Jakarta Selatan pada tahun 2004 dan diselesaikan pada tahun 2007.
KATA PENGANTAR
Segala puji serta syukur kehadirat Allah SWT yang telah menciptakan seluruh alam beserta isinya. Yang Maha Pengasih lagi Maha Penyayang kepada seluruh umat. Shalawat serta salam selalu tercurah kepada pembawa pelita saat dunia masih gelap dan penerang hati serta jalan manusia, kekasih Allah, Rasulullah SAW. Alhamdulilah penelitian dan penulisan skripsi ini dapat diselesaikan. Skripsi yang berjudul “Identifikasi Keragaman DNA Mikrosatelit Lokus ILSTS028, ILSTS052 dan ILSTS056 pada Sapi Katingan di Kalimantan Tengah” ini merupakan salah satu syarat untuk memperoleh gelar sarjana pada Fakultas Peternakan, Institut Pertanian Bogor.
Penelitian ini dilaksanakan selama tujuh bulan, yaitu dari bulan Oktober 2010 sampai dengan Mei 2011. Penelitian ini mengenalkan Sapi Katingan sebagai salah satu jenis sapi yang hidup di Kalimantan Tengah yang keberadaannya sudah puluhan tahun dan sudah beradaptasi dengan lingkungan sekitarnya. Sapi ini hanya dipelihara oleh suku Dayak saja dan hidup di sepanjang daerah aliran sungai (DAS) Katingan. Penelitian ini bertujuan untuk mengetahui keragaman genetik di dalam populasi ternak sapi lokal di daerah Kalimantan Tengah dengan menggunakan penanda molekuler DNA mikrosatelit pada lokus ILSTS028, ILSTS052 dan ILSTS056.
Bogor, Nopember 2011
DAFTAR ISI
Halaman RINGKASAN ... i
Frekuensi Genotipe ... 17 Derajat Heterozigositas ... 17 HASIL DAN PEMBAHASAN ... 18 Amplifikasi DNA Mikrosatelit ... 18 Keragaman DNA Mikrosatelit ... 19 Lokus ILSTS028 ... 19 Lokus ILSTS052 ... 24 Lokus ILSTS056 ... 28 Nilai Heterozigositas ... 33 KESIMPULAN DAN SARAN ... 36 Kesimpulan ... 36 Saran ... 36 UCAPAN TERIMAKASIH ... 37 DAFTAR PUSTAKA ... 38 LAMPIRAN ... 41
DAFTAR TABEL
Nomor Halaman 1. Jumlah Sampel Darah Sapi Katingan yang Digunakan dari
Kabupaten Katingan, Kalimantan Tengah ... 11 2. Informasi tentang Tiga Pasang Primer Pengapit DNA Mikrosatelit 14 3. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe
Lokus ILSTS028 pada Populasi Sapi Katingan di Kalimantan Tengah 21 4. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe
Lokus ILST028 pada Ketiga Populasi Sapi Katingan dan Sapi Lokal Lainnya di Kalimantan Tengah ... 23 5. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe
Lokus ILSTS052 pada Populasi Sapi Katingan di Kalimantan Tengah 25 6. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe
Lokus ILSTS052 pada Populasi Sapi Katingan dan Sapi Lokal Lainnya di Kalimantan Tengah ... 27 7. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe
Lokus ILSTS056 pada Populasi Sapi Katingan di Kalimantan Tengah 29 8. Macam Alel, Genotipe, Frekuensi Alel dan Frekuensi Genotipe
Lokus ILSTS056 pada Sapi Katingan dan Sapi Lokal lainnya di
Kalimantan Tengah ... 32 9. Nilai Heterozigositas pada Populasi Sapi Katingan dan Sapi Lokal
Lainnya dari Masing-masing Lokus ... 33 10.Rataan Heterozigositas ( ) dari Populasi Sapi Katingan dan Sapi
Lokal Lainnya ... 34 11.Informasi Mengenai Rataan Heterozigositas ( ) pada Beberapa Bangsa
1 DAFTAR GAMBAR
Nomor Halaman
1. Keragaman Warna Bulu Sapi Katingan Betina (a, b, c) dan Jantan
(d, e, f) ... 6 2. Keragaman Bentuk Tanduk Sapi Katingan Jantan ... 7 3. Variasi Tonjolan (Tinggi Rendah) pada Sapi Katingan Betina ... 8 4. Peta Lokasi Pengambilan Sampel Sapi Katingan ... 12 5. Penentuan Genotipe Lokus ILSTS028 pada Sapi Katingan dan Sapi
Lokal Lainnya ... 20 6. Frekuensi dan Macam Alel pada lokus ILSTS028 ... 22 7. Penentuan Genotipe Lokus ILSTS052 pada Sapi Katingan dan Sapi
Lokal Lainnya ... 24 8. Frekuensi dan Macam Alel Lokus ILSTS052 ... 26 9. Penentuan Genotipe Lokus ILSTS056 pada Sapi Katingan dan Sapi
2 DAFTAR LAMPIRAN
Nomor Halaman
1. Motif Ulangan Nukleotida Lokus ILSTS028, ILSTS052 dan
ILSTS056 ... 42 2. Macam, Ukuran Alel dan Genotipe Lokus ILSTS028 pada Sapi
Katingan, Kalimantan Tengah ... 43 3. Macam, Ukuran Alel dan Genotipe Lokus ILSTS052 pada Sapi
Katingan, Kalimantan Tengah ... 44 4. Macam, Ukuran Alel dan Genotipe Lokus ILSTS056 pada Sapi
3 PENDAHULUAN
Latar Belakang
Ternak lokal merupakan kebanggaan bagi Indonesia, karena ternak lokal merupakan sumber daya genetik ternak bagi bangsa. Ternak lokal memiliki beberapa keunggulan yaitu daya adaptasi tinggi walaupun kondisi pakan berkualitas rendah, lingkungan marjinal, pemeliharaan secara ekstensif tradisional dan memiliki keunggulan tahan terhadap beberapa penyakit dan parasit. Ternak lokal juga memiliki beberapa kekurangan yaitu produktivitas rendah dibandingkan ternak impor. Peningkatan dan perbaikan mutu genetik ternak lokal agar dapat dimanfaatkan dengan optimal.
Pelestarian terhadap sumber daya genetik ternak asli atau ternak lokal sangat penting karena merupakan bagian dari komponen keanekaragaman hayati untuk memenuhi kebutuhan pangan, pertanian, dan perkembangan sosial masyarakat di masa yang akan datang. Beberapa alasan pelestarian sumber daya genetik ternak Indonesia penting dilakukan yaitu lebih dari 60% bangsa ternak di dunia berada di negara berkembang, konservasi ternak asli atau ternak lokal tidak menarik bagi petani, secara umum tidak ada program pemantauan yang sistematis dan tidak tersedia informasi deskriptif dasar sebagian sumber daya genetik ternak, dan sedikit sekali bangsa-bangsa ternak asli maupun ternak lokal yang telah digunakan dan dikembangkan secara aktif (FAO, 2001). Populasi sapi dari tahun 2005 sampai 2010 dilaporkan oleh Mayulu et al. (2010) selalu meningkat tiap tahunnya dari 11.045.900 ekor pada tahun 2005 menjadi 14.763.000 ekor pada tahun 2010.
Sapi Katingan adalah salah satu jenis sapi yang terdapat di Kalimantan Tengah sejak puluhan tahun dan sudah beradaptasi dengan lingkungan setempat. Sapi Katingan dipelihara oleh suku Dayak dan hidup disepanjang daerah aliran sungai Katingan. Sapi Katingan dipelihara secara ekstensif tradisional oleh masyarakat suku Dayak. Menurut Utomo et al. (2010), sapi tersebut dinamakan sapi Katingan dikarenakan lokasi pemeliharaan yang menyebar di sepanjang daerah aliran sungai Katingan. Masyarakat Dayak sendiri menyebutnya sapi lokal atau kadang-kadang sapi ”Helu” (sapi jaman dulu), tidak pernah mereka menamakannya sapi Katingan. Istilah sapi Katingan diberikan untuk membedakan dengan sapi lokal lain (Utomo, 2011). Penamaan sapi diberikan sesuai lokasi habitatnya sebagaimana pada
4 sapi-sapi lokal lain (Abdullah, 2008; Sarbaini, 2004; Sun et al., 2008). Penelitian ini dilakukan karena keterbatasan informasi mengenai data-data yang berhubungan dengan aspek fenotipik dan genetik pada sapi Katingan.
Tujuan
Tujuan penelitian ini adalah untuk mengidentifikasi keragaman sapi Katingan di Kalimantan Tengah dengan menggunakan penanda molekuler DNA mikrosatelit lokus ILSTS028, ILSTS052 dan ILSTS056.
5 TINJAUAN PUSTAKA
Sumber Daya Genetik Ternak Lokal
Keanekaragaman ternak sapi di Indonesia terbentuk dari sumber daya genetik ternak asli dan impor. Impor ternak sapi Ongole (Bos indicus) atau Zebu yang dimulai pada awal abad ke-20 memegang peranan penting dalam pengembangan peternakan di Indonesia. Ongole murni pertama kali dibawa ke Pulau Sumbawa yang kemudian disebut sebagai Sumba Ongole (SO) dan selanjutnya dibawa ke tempat-tempat lain di Indonesia untuk disilangkan dengan sapi asli Jawa dan membentuk Peranakan Ongole (PO) dan sapi Madura (Utoyo, 2002). Proses perkembangan sapi di Indonesia telah menghasilkan sumber daya genetik ternak yang lebih beragam, yaitu mulai dari sapi asli seperti sapi Bali, juga sapi hasil silangan yang telah menjadi sapi lokal seperti sapi Pesisir, sapi Aceh, sapi Madura, sapi Sumba Ongole (SO) dan sapi Peranakan Ongole (PO) (Utoyo, 2002; Martojo, 2003).
Sumber daya genetik ternak merupakan kerangka dasar acuan bagi pertanian dan pengembangan varietas dan bangsa ternak untuk masa yang akan datang. Keanekaragaman bangsa ternak asli maupun yang beradaptasi secara lokal berlimpah dan dapat menyelamatkan petani dalam menghadapi iklim yang sulit dan wilayah yang marjinal. Sumber daya genetik ternak lokal dapat dimanfaatkan dengan biaya (input) minimum dan memegang peranan penting dalam budaya masyarakat pedesaan (FAO, 2001).
Keanekaragaman genetik ternak lokal memiliki beberapa manfaat, yaitu (1) untuk keberlanjutan dan peningkatan produksi pangan, (2) untuk memaksimalkan produktivitas lahan dan sumber daya pertanian, (3) untuk pencapaian pertanian berkelanjutan dan (4) untuk pemenuhan keanekaragaman baik yang telah maupun yang akan diketahui manfaatnya bagi kehidupan sosial masyarakat. Ketersediaan keanekaragaman genetik ternak khususnya ternak sapi akan memberikan keberhasilan dalam strategi pemuliaan untuk masa yang akan datang (FAO-AAAS, 1994).