IDENTIFIKASI KERAGAMAN GEN
CALPASTATIN
(CAST|
Alu
I) PADA BANGSA SAPI INDONESIA
DENGAN METODE PCR-RFLP
SKRIPSI AHMAD FURQON
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
Ahmad Furqon. D14080130. 2012. Identifikasi Keragaman Gen Calpastatin (CAST|AluI) pada bangsa sapi Indonesia dengan metode PCR RFLP . Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Muladno, M.SA. Pembimbing Anggota : Dr. Jakaria, S.Pt., M.Si.
Gen calpastatin merupakan salah satu gen yang berhubungan dengan keempukan daging yang berfungsi sebagai penghambat (inhibitor) enzim calpain. Identifikasi keragaman gen calapastatin (CAST|AluI) pada sapi lokal dengan menggunakan metode PCR RLFP masih terbatas. Tujuan penelitian ini adalah untuk mengetahui keragaman gen CAST|AluI pada sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh dan sapi Katingan dengan metode Polymerase Chain Reaction - Restriction Fragment Length Polymorphism (PCR-RFLP).
Sampel darah sapi yang digunakan sebanyak 282 sampel yang terdiri atas sapi Bali (102 sampel), sapi Madura (68 sampel), sapi Pesisir (49 sampel), sapi Katingan (50 sampel) dan sapi Aceh (14 sampel). Identifikasi keragaman gen CAST|AluI menggunakan pendekatan Polymerase Chain Reaction - Restriction Fragment Length Polymorphism (PCR-RFLP) dengan menggunakan enzim restriksi
AluI. Data genotipe gen CAST|AluI dianalisis dengan pendekatan frekuensi genotipe dan alel serta pendugaan nilai heterozigositas.
Hasil amplifikasi gen CAST|AluI pada sapi lokal Indonesia menghasilkan fragmen sepanjang 624 pb dan tiga genotipe yang berbeda yaitu GG (474 pb), GC (474 pb dan 324 pb) dan CC (324 pb). Frekuensi genotipe GG tertinggi ditemukan pada semua bangsa sapi Indonesia, tetapi genotipe CC tidak ditemukan pada sapi Aceh. Frekuensi alel G tertinggi ditemukan pada populasi sapi Bali (85,8%), sapi Madura (80,1%), sapi Pesisir (78,6%), sapi Aceh (80,8%), dan sapi Katingan (85,0%). Frekuensi alel C sapi Bali, Madura, Pesisir, Aceh dan Katingan berturut-turut adalah 14,2%, 19,9%, 21,4%, 19,2%, dan 15,0%. Pendugaan nilai heterozigositas tertinggi terdapat pada sapi Pesisir dan sapi Aceh, sedangkan nilai heterozigositas terendah terdapat pada sapi Bali, sapi Madura dan sapi Katingan. Identifikasi keragaman gen CAST|AluI menunjukkan bahwa bangsa sapi lokal Indonesia bersifat polimorfik dengan ditemukannya 3 genotipe (GG, GC, CC) dan 2 alel (G dan C). Adanya keragaman gen CAST|AluI dapat dijadikan sumber informasi dasar dalam penyeleksian ternak unggul yang berhubungan dengan sifat keempukan daging pada sapi pedaging.
ABSTRACT
Identification of Calpastatin Gene (CAST|AluI) Polymorphisms in Indonesian Cattle Breed Using PCR-RFLP Technique
Furqon, A., Muladno, and Jakaria
Calpastatin (CAST) is dependent proteases and a specific inhibitor of the μ-calpain and m-calpain function, were found in mammalian tissues. Calpastatin (CAST) gene is a gene that have functions in inhibiting muscle protein degradation and responsible for muscle hypertrophy. Thepurpose of this study is to identify polymorphism of the Calpastatin (CAST) gene in Indonesian local cattles (Bali, Madura, Pesisir, Aceh and Katingan). DNA polymorphisms within the calpastatin gene may lead to the phenotypic differences of cattle growth traits. A 624 bp of Indonesian local cattles calpastatin gene was successfully amplified using Polymerase Chain Reaction (PCR) technique. PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Length Polymorphism) method will be used to identify polymorphism of calpastatin genes in local cattles. An AluI restriction enzyme will cut the PCR product into two different length fragments that are 474 bp and 324 bp and revealed two alleles system, G and C and three genotypes GG, GC and CC. All of Indonesian local cattles population are polymorphic in calpastatin gene. The highest GG genotype frequency was found in all Indonesian local cattles, but the CC genotype was not found in Aceh Cattle. The G allele frequency was found in Bali Cattle population (85,8%), Madura Cattle population (80,1%), Pesisir Cattle population (78,6%), Aceh Cattle population (80,8%), and Katingan Cattle population (85,0%). The allele C frequencies of Bali, Madura, Pesisir, Aceh and Katingan Cattle population are 14,2%, 19,9%, 21,4%, 19,2%, and 15,0%, respectively. The higest heterozygosity value was found in Pesisir cattle and Aceh cattle, while the lower heterozygosity value was found in Bali, Madura, and Katingan cattles population. Based on the analysis, Indonesian cattle breed (bali cattle, madura cattle, pesisir cattle aceh cattle, and katingan cattle) are polymorphic in calpastatin gene with three genotipes (GG, GC, CC) and two alelles (G and C).
IDENTIFIKASI KERAGAMAN GEN
CALPASTATIN
(CAST|
Alu
I) PADA BANGSA SAPI INDONESIA
DENGAN METODE PCR-RFLP
AHMAD FURQON D14080130
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman Gen Calpastatin (CAST|AluI) pada Bangsa Sapi Indonesia dengan Metode PCR-RFLP
Nama : Ahmad Furqon NIM : D14080130
Menyetujui,
Mengetahui, Ketua Departemen
Ilmu Produksi dan Teknologi Peternakan
Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. NIP. 19591212 198603 1 004
Tanggal Ujian: 8 Agustus 2012 Tanggal Lulus: Pembimbing Utama
Prof. Dr. Ir. Muladno, MSA. NIP. 19610824 198603 1 001
Pembimbing Anggota
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 18 Mei 1991 di Jakarta. Penulis adalah anak pertama dari tiga bersaudara dari pasangan Bapak Luthfi dan Ibu Sumiati.
Penulis mengawali pendidikan dasar pada tahun 1996 di Madrasah Ibtidaiyyah Al Ittihadiyyah sampai kelas 3 kemudian pindah ke SDN Sukatani 7 pada tahun 1998 dan diselesaikan pada tahun 2002. Pendidikan menengah tingkat pertama dimulai pada tahun 2002 dan diselesaikan pada tahun 2005 di SMPN 11 Depok. Penulis melanjutkan pendidikan menengah tingkat atas di SMAN 4 Depok pada tahun 2005 dan diselesaikan pada tahun 2008.
Penulis diterima di Institut Pertanian Bogor pada tahun 2008 melalui jalur Undangan Seleksi Masuk IPB (USMI) dan diterima di Departemen Ilmu Produksi dan Teknologi Peternakan. Penulis pernah menjadi anggota dalam organisasi jurnalistik kampus Majalah Emulsi pada tahun 2009-2011. Selain itu, penulis juga pernah menjadi anggota Animal Breeding and Genetic Student Community
KATA PENGANTAR
Puji dan syukur senantiasa penulis panjatkan kepada Allah SWT atas nikmat, rahmat, dan karunia yang telah diberikan sehingga penelitian dan skripsi ini dapat terselesaikan. Shalawat dan salam semoga senantiasa terlimpah kepada Rasulullah Muhammad SAW, keluarga, para sahabat dan pengikutnya. Skripsi yang berjudul
Identifikasi Keragaman Gen Calpastatin (CAST|AluI) pada bangsa sapi Indonesia dengan metode PCR RFLP merupakan salah satu syarat untuk memperoleh gelar sarjana pada Fakultas Peternakan, Institut Pertanian Bogor.
Ternak sapi pedaging merupakan salah satu komoditas yang memiliki berbagai manfaat. Namun, perkembangan populasi dan produktivitas sapi pedaging di Indonesia belum optimal sehingga berpotensi untuk ditingkatkan. Beberapa cara yang dapat dilakukan untuk meningkatkan produktivitas ternak antara lain dengan perbaikan pakan, manajemen pemeliharaan, seleksi, dan persilangan. Seleksi pada ternak dapat dilakukan pada level DNA, salah satunya dengan teknik PCR-RFLP yang digunakan untuk mendeteksi keragaman gen yang berhubungan dengan sifat ekonomis seperti sifat keempukan daging.
Penulis menyadari bahwa dalam penulisan ini masih banyak kekurangan. Kritik dan saran yang membangun sangat diharapkan demi kesempurnaan tulisan ini. Semoga tulisan ini dapat bermanfaat bagi pembaca dan menjadi pedoman dasar untuk penelitian serupa pada masa yang akan datang. Amin.
Bogor, September 2012
DAFTAR ISI
KESIMPULAN DAN SARAN ……….. 22
Kesimpulan ………..
Saran ………...
22 22
UCAPAN TERIMA KASIH ………... 23
DAFTAR PUSTAKA ………... 24
DAFTAR TABEL
Nomor Halaman
1. Karakteristik Fenotipik Bangsa Sapi Indonesia ………... 4 2. Jumlah Sampel Ternak Sapi Indonesia...………….... 8 3. Hasil Identifikasi Genotipe Gen Calpastatin pada Bangsa Sapi
Indonesia……… 16
4. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen CAST|AluI
pada Bangsa Sapi Indonesia... 17 5. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen CAST|AluI
pada Bos taurus... 18 6. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas
Harapan (He) gen CAST|AluI pada Bangsa Sapi Indonesia... 19
DAFTAR GAMBAR
Nomor Halaman
1. Karakteristik Fenotipik Bangsa Sapi Indonesia. (a) Sapi Bali Betina (b) Sapi Bali Jantan (c) Sapi Madura (d) Sapi Aceh (e)
Sapi Pesisir (f) Sapi Katingan... 5
2. Struktur Gen Calpastatin Sapi ………... 7
3. Posisi Penempelan Primer pada Sekuen Gen Calpastatin CAST|AluI…………... 10
4. Penentuan Genotipe Gen Calpastatin…………... 12
5. Hasil Amplifikasi Gen Calpastatin pada Gel Agarose 1,5%... 14
DAFTAR LAMPIRAN
Nomor Halaman
1. Sekuen gen Calpastatin Bos taurus dengan Nomor Akses
GenBank AF117813... 29 2. Sekuen gen Calpastatin Bos taurus dengan Nomor Akses
GenBank AY834765... 31 3.
4.
Sekuen gen Calpastatin Bos taurus dengan Nomor Akses GenBank AY834775... Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen Gen CAST pada Bangsa Sapi Indonesia...
35
41
PENDAHULUAN Latar Belakang
Sapi potong merupakan hewan ternak yang dipelihara untuk diambil daging sebagai hasil utamanya. Banyak bangsa sapi potong yang terdapat di dunia, beberapa diantaranya terdapat di Indonesia. Beberapa bangsa sapi potong yang terdapat di Indonesia adalah sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh dan sapi Katingan. Kelima sapi tersebut memiliki sifat dan karakteristik yang berbeda. Bangsa sapi tersebut sangat potensial untuk dikembangkan sebagai sumberdaya genetik ternak lokal Indonesia. Namun, keberadaan bangsa sapi Indonesia masih kurang mendapat perhatian serius dari pemerintah dan masyarakat sehingga pengembangan dan pemanfaatan bangsa sapi Indonesia belum optimal.
Keempukan daging (meat tenderness) merupakan faktor yang penting di dalam penentuan kualitas daging. Beberapa faktor diidentifikasi berhubungan dengan proses pengempukan daging setelah pemotongan seperti lama rigormortis, pH, dan enzim keempukan daging. Perubahan fisiologi struktur otot setelah pemotongan merupakan proses fisiologis yang kompleks. Dua enzim yang berperan terhadap keempukan daging adalah enzim calpastatin yang dihasilkan oleh gen CAST dan
calpain yang dihasilkan oleh gen calsium-activative neural protease (CAPN 1) (Koohmarie, 1966). Calpastatin merupakan enzim protease utama dan bersifat inhibitor spesifik terhadap fungsi µ-calpain dan m-calpain. Aktivitas calpastatin
meningkat ketika aktivitas degradasi protein pada jaringan otot hewan hidup menurun. Calpastatin berfungsi untuk menghambat proses degradasi protein sel otot. Selain itu, diduga kuat terkait dengan sifat pertumbuhan otot dan keempukan daging pada mamalia (Morgan et al., 1993).
2
Tujuan
TINJAUAN PUSTAKA Sapi Lokal Indonesia
Indonesia merupakan salah satu negara yang memiliki sumberdaya genetik ternak. Sapi merupakan salah satu jenis ternak di Indonesia yang memiliki potensi besar terutama sebagai ternak penghasil daging. Direktorat Jendral Peternakan (2008) melaporkan bahwa keberadaan sapi lokal Indonesia pada beberapa tahun terakhir ini masih kurang mendapat perhatian serius baik dari pemerintah maupun masyarakat. Dilihat dari potensi yang dimiliki, sapi lokal Indonesia mampu secara sosial dan budaya berinteraksi dengan masyarakat sejak lama.
Sumberdaya genetik ternak adalah semua yang termasuk dalam spesies, bangsa, dan strain (galur) ternak yang secara ekonomi, ilmiah, dan budaya penting bagi umat manusia baik dalam bentuk makanan maupun produksi (Food Agriculture Organization, 1999). Departemen Pertanian (2006) menyatakan bahwa sumberdaya genetik ternak adalah substansi yang terdapat dalam bentuk individu suatu populasi rumpun ternak secara genetik unik, terbentuk dalam proses domestikasi dari masing-masing spesies yang memiliki potensial serta dapat dimanfaatkan dan dikembangkan baik untuk menciptakan rumpun atau galur unggul.
Sumberdaya ternak sapi Indonesia saat ini terdiri dari tiga kelompok yaitu ternak asli, ternak impor dan ternak yang telah beradaptasi. Beberapa bangsa sapi yang menjadi sumberdaya adalah sapi Aceh, sapi Madura, sapi Pesisir, dan sapi Peranakan Ongole. Keragaman sapi di Indonesia terbentuk dari sumberdaya genetik asli dan impor. Sehubungan dengan pentingnya nilai konservasi pada kelompok ternak, maka beberapa bangsa sapi menjadi target konservasi sekaligus pemanfaatannya (Utoyo, 2002). Salah satu jenis sapi lokal Indonesia adalah sapi Bali. Sapi Bali dipercaya sebagai salah satu sapi lokal Indonesia yang diperoleh dari hasil domestikasi Banteng (Namikawa, 1980). Martojo (2003) menyatakan bahwa hasil domestikasi spesies Bos (Bibos) banteng adalah sapi Bali (Bos sundaicus) atau (Bos javanicus) yang sekarang telah menjadi bangsa ternak asli Indonesia.
4 Tabel 1. Karakteristik Fenotipik Bangsa Sapi Indonesia
Ternak Karakteristik Literatur
Sapi Madura Berukuran sedang, pertulangan bagus, berotot bagi sapi jantan, tanduk kecil ke atas atau ke samping, kaki dan teracak kuat, gumba berkembang baik pada jantan, terdapat lingkaran putih di sekitar moncong. Bobot badan 248 kg putih), temperamen jinak, berpunuk kecil sampai sedang, bertanduk pendek mengarah ke luar, bobot badan lebih berukuran sedang (jantan), memiliki warna cokelat yang lebih gelap pada bagian depan dibandingkan bagian belakang tubuhnya. Bobot badan 302 kg
Namikawa. et al.
(1982), Otsuka et al.
(1980), Adrial (2010)
Sapi Katingan Ukuran tubuh jantan tidak terlalu besar dibandingkan betina, memilki gumba yang cukup jelas, tonjolan pada kepala bagian atas pada betina, tanduk melengkung ke depan
Utomo et al. (2010)
5
(a) (b)
(c) (d)
(e) (f)
Gambar 1. Karakteristik Fenotipik Bangsa Sapi Indonesia. (a) Sapi Bali Betina (b) Sapi Bali Jantan (c) Sapi Madura (d) Sapi Pesisir (e) Sapi Aceh dan (f) Sapi Katingan.
Sumber : Republika, 2011
Penanda Molekuler
Penanda molekuler merupakan pemanfaatan dari keragaman pada tingkat
6 terjadinya proses seleksi (Vignal et al., 2002). Tipe dasar perubahan dalam DNA meliputi substitusi, delesi, inersi, dan inversi (Nei dan Kumar, 2000).
Single nucleotide polymorphims (SNP) merupakan penanda yang memiliki perbedaan satu nukleotida dalam sekuen DNA, dan diperkirakan bahwa satu SNP terjadi setiap 250-1000 bp. Perbedaan tersebut disebabkan oleh terjadinya proses substitusi, sehingga biasanya memiliki dua kemungkinan pada posisi yang sama dalam sekuen DNA (Vignal et al., 2002). Frekuensi mutasi dan stabilitas yang tinggi menyebabkan SNP sering digunakan sebagai penanda molekuler dalam penelitian tentang genetika populasi dan pemetaan gen untuk penyakit kompleks (Ye et al., 2001). Metode yang digunakan dalam menganalisis adanya SNP antara lain PCR-RFLP dan sequencing.
Polymerase Chain Reaction (PCR) merupakan suatu teknik untuk menggandakan jumlah molekul DNA scara in vitro. Proses ini berjalan dengan bantuan enzim polymerase dan primer. Primer merupakan oligonukleotida spesifik pada DNA template. Enzym polymerase merupakan enzim yang dapat mencetak urutan DNA baru. Hasil PCR dapat langsung divisualisasikan dengan elektroforesis atau dapat digunakan untuk analisis lebih lanjut (Williams, 2005).
Restriction Fragment Lenght Polymorphism (RFLP) merupakan salah satu teknik penciri genetik (genetic marker) yang dikembangkan oleh Botstein et al. (1980) yang digunakan untuk mengetahui adanya keragaman sekuens DNA. Mullis
et al. (1986) menyatakan bahwa penggunaan teknik RFLP menjadi lebih intensif setelah teknik RFLP dikombinasikan dengan teknologi PCR yang digunakan hingga saat ini.
PCR-RFLP merupakan metode analisis lanjutan dari produk PCR. Metode PCR-RFLP memanfaatkan perbedaan pola pemotongan enzim pemotong yang berbeda pada tiap-tiap mikroorganisme. Analisis RFLP sering digunakan untuk mendeteksi lokasi genetik dalam kromosom (Orita et al., 1989), ataupun untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
Gen Calpastatin
7 dengan proses pengempukan daging setelah pemotongan seperti lama rigormortis, pH,dan sistem enzim keempukan daging. Perubahan fisiologi struktur otot setelah pemotongan merupakan proses fisiologis yang kompleks. Dua enzim yang berperan terhadap keempukan daging adalah enzim calpastatin yang dihasilkan oleh gen CAST dan calpain yang dihasilkan oleh gen calsium-activative neural protease
(CAPN 1) (Koohmarie, 1966). Calpastatin merupakan enzim protease utama dan bersifat inhibitor spesifik terhadap fungsi µ-calpain dan m-calpain. Aktivitas
calpastatin meningkat ketika aktivitas degradasi protein pada jaringan otot hewan hidup menurun. Calpastatin berfungsi untuk menghambat proses degradasi protein sel otot. Selain itu, diduga kuat terkait dengan sifat pertumbuhan otot dan keempukan daging pada mamalia (Morgan et al., 1993).
Gen calpastatin pada ternak sapi (Bos taurus) terletak pada kromosom nomor 7 (Bishop et al., 1993) sedangkan pada domba nomor 5 (Hediger et al., 1991). Gen
calpastatin dengan simbol CAST terletak diantara dua penciri apit mikrosatelit MCM527 dan BMS1247 pada posisi lokus 5q15 – q21 antara 96,057-96,136 Mb (Gambar 2).
Gambar 2. Daerah Target Amplifikasi Gen Calpastatin
Sumber : Palmer et al., 2000
Kubiak et al. (2004) melaporkan bahwa terdapat keragaman gen Calpastatin
MATERI DAN METODE Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Pemuliaan dan Genetik Molekuler, Bagian Pemuliaan dan Genetik Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini dilaksanakan selama lima bulan yaitu mulai September 2011 sampai dengan Februari 2012.
Materi Sampel
Sampel darah sapi lokal Indonesia yang digunakan sebanyak 282 sampel terdiri atas sampel sapi Bali, sapi Madura, sapi Aceh, sapi Pesisir, dan sapi Katingan. Jumlah sampel dari masing-masing sapi lokal Indonesia yang digunakan dalam penelitian ini disajikan pada Tabel 2.
Tabel 2. Jumlah Sampel Ternak Sapi Indonesia
Ternak Sapi N Tahun Koleksi Asal
Sapi Pesisir 49 2006 Kab. Pesisir Selatan
Sapi Aceh 13 2010 Kab. Aceh Besar
Sapi Bali 102
2010 Balai Pembibitan
Ternak Unggul Sapi Bali
Sapi Katingan 50 2010 Daerah Aliran Sungai
Katingan
Sapi Madura 68 2011 Pulau Kangean
Total 282
Keterangan: N = jumlah individu
Bahan dan Alat
9 steril, sampel DNA, buffer, MgCl2, pasangan primer, enzim taq dan dNTP, enzim retriksi AluI dan buffer AluI. Bahan yang digunakan dalam elektroforesis adalah air destilasi, agarose, 0,5x TBE, EtBr, loading dye (0,01% Xylene cyanol, 0,01% Bromtimolblue, 50% gliserol), dan marker 100 bp. Bahan yang digunakan dalam
genotyping yaitu loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%) dan untuk membuat 1 lembar gel agarose 1% adalah sebagai berikut: agarose 0,3 g, 0,5 TBE 30 ml, dan 2,5 μl EtBr.
Alat yang digunakan antara lain autoclave, satu set pipet mikro dan tipnya,
vortexmixer, alat sentrifugasi, refrigerator, dan freezer, tabung PCR, mesin PCR, satu set alat pencetak gel, power supply 100 volt, microwave, stearer, magnet stearer, gelas ukur, dan tabung erlenmeyer.
Prosedur Pengambilan Sampel
Pengambilan sampel dilakukan pada lima bangsa sapi berbeda, yaitu sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh dan sapi Katingan. Sampel darah diambil melalui vena jugularis menggunakan jarum vacutainer tabung vacum yang ditambahakan alkohol 70%. Sampel disimpan dalam termos es atau lemari es sampai akan digunakan lebih lanjut.
Ekstraksi DNA
Ektraksi DNA yang dilakukan merupakan modifikasi dari metode Sambrook
et al. (1989). Sampel darah yang disimpan dalam alkohol 70% diambil sebanyak 200 µl, kemudian ditambahkan 1000 μl DW. Sampel divortex lalu didiamkan selama 5 menit. Setelah itu disentrifuse pada kecepatan 8000 rpm selama 5 menit, lalu supernatan dibuang, dan diulangi seperti proses sebelumnya, kemudian ditambahkan 10 µl proteinase-K yang berfungsi untuk menghancurkan protein, 350 µl 1xSTE (sodium tris-EDTA) dan 40 µl 10% SDS (sodium dodesil sulfat) yang berfungsi untuk melisiskan membran sel. Campuran tersebut kemudian diinkubasi pada suhu 550C selama 2 jam sambil dikocok pelan menggunakan alat pemutar (tilting).
10 DNA yang larut dalam fase air dipisahkan dari fase fenol dengan alat sentrifugasi pada kecepatan 12.000 rpm selama 5 menit. Setelah terbentuk fase DNA, diambil sebanyak 40 µl pada fase DNA untuk dipindahkan ke tabung baru 1,5 ml. Kemudian ditambahkan 5M NaCl sebanyak 40 µl dan etanol absolut sebanyak 800 µl. Molekul DNA kemudian dimalamkan (over night) pada suhu -200C.
Molekul DNA kemudian dipisahkan dari etanol absolut dengan cara sentrifugasi pada kecepatan 12.000 rpm selama 5 menit, kemudian supernatan yang diperoleh dibuang. Endapan yang terbentuk kemudian dicuci dengan menambahkan 70% etanol sebanyak 800 µl dan disentrifugasi kecepatan 12.000 rpm selama 5 menit. Supernatan yang diperoleh kemudian dibuang sehingga didapatkan endapan molekul DNA. Endapan tersebut didiamkan sampai kering. Lalu endapan DNA disuspensikan dalam 100 µl 80% buffer TE (tris EDTA).
Amplifikasi DNA
Sekuen primer yang digunakan dalam penelitian ini berdasarkan Palmer et al. (1998), yaitu primer forward(AF146) 5’ TGGGGCCCAATGACGCCATCGATG 3’
yang terletak di ekson 1C dan primer reverse (AF147) 5’ GGTGGAGCAGCAC TTCTGATCACC 3’ yang terletak di ekson 1D (Gambar 3), dengan panjang produk PCR 624 pb.
Forward
1 tggggcccaa tgatgccatc gatgccttgt catccgactt cacctgcagt tcccctacag
61 ctgatgcaaa gaaaactgag aaagaggtat ggtttttaat gcccttaggg aagcttgtta 121 gaaactacct cccactttaa gacaacaact tttttttaaa cttcattttt cacttcactg 181 cgtcttcatt gctgtgttcg ggctttctct agttggggca agcgaggcct gttctctatt 241 tgcaattttt aggcttctgc agggggctcc tcttgttgct gggccggggc tctaggtgca 301 caggcttcat ttgttgtggc tcgagggctc taaaccacag gctcattggt cttggcgcac 361 gggcatggtt accccaatgc atttgggatc tcccctggcc agggagcaaa cctgtttccc 421 ctgcattgca aggcggcctc ttaaccgctg gccaccaggg aagccccaaa atgccaaggc 481 tttttacttc tggttcttac cgtttggttc atatttttcc ttcatctgcc agtcaaacct 541 tcttctgtat tttattttcc agaaatctac agaagaggct ttaaaagctc agtcagctgg 601 ggtgatcaga agtgctgctc cacc
Reverse
Keterangan :
: primer forward
: primer reverse
agct : situs pemotongan enzim AluI
11 Amplifikasi DNA yang dilakukan merupakan modifikasi dari metode Sambrook et al. (1989). Pereaksi untuk amplifikasi gen DNA secara umum dilakukan menggunakan campuran yang terdiri dari 1 µl sampel DNA yang sudah diekstraksi, 10,85 µl DW, 0,3 µl primer, 0,05 µl taq polymerase, 1,5µl buffer, 0,3 µl dNTP dan 1,00 µl MgCl2. Campuran tersebut kemudian dimasukkan ke dalam mesin PCR dengan kondisi suhu denaturasi awal 950C selama 5 menit, 35 siklus (denaturasi 950C selama 30 detik, annealing 600C selama 45 detik dan elongasi 720C selama 1 menit) dan elongasi akhir 720 C selama 5 menit.
Elektroforesis
Elektroforesis yang dilakukan merupakan modifikasi dari metode Sambrook
et al. (1989). Elektroforesis produk PCR dilakukan menggunakan 5 μl produk PCR pada gel agarose 1% dengan tegangan 100 volt selama 30 menit. Gel dibuat dengan cara memanaskan agarose 0.3 g yang dilarutkan dalam larutan 0,5xTBE 30 ml selama 5 menit dalam microwave. Kemudian ditambahkan 2,5 μl EtBr pada saat distearer sampai didapatkan larutan jernih. Larutan yang masih cair dituangkan ke dalam pencetak gel serta menempatkan sisir di dekat tepian gel dan gel dibiarkan mengeras. Apabila gel sudah mengeras, sisir dicabut sehingga akan terbentuk sumur-sumur yang digunakan untuk menempatkan sebanyak 5μl produk PCR dicampur dengan loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%). Gel ditempatkan ke dalam gel tray elektroforesis yang sudah terisi larutan buffer dan dialiri listrik, molekul DNA yang bermuatan negatif pada pH netral akan bergerak (migrasi) ke arah positif (anode). Setelah elektroforesis selesai gel agarose diambil untuk dilihat panjang pita DNA dengan menggunakan sinar Ultraviolet yaitu dengan menarik garis lurus antara posisi pita dari masing-masing sampel DNA yang ingin diukur dengan posisi pita DNA marker, kita dapat mengestimasi ukuran sampel DNA karena ukuran DNA pengukur telah diketahui.
Genotyping
12 2% dengan tegangan 100 volt selama 30 menit. Selanjutnya dilakukan visualisasi di bawah mesin UV-Transilluminator. Pita DNA yang muncul pada tahap ini dibandingkan dengan marker untuk mengetahui panjang pita tersebut. Genotipe gen
calpastatin ditentukan berdasarkan panjang pita DNA yang muncul (Gambar 4). Penentuan genotipe (genotyping) mengacu pada penelitian Kubiak et al. (2004) yang menghasilkan tiga genotipe yaitu GG, GC, dan CC. Genotipe GG ditunjukkan dengan satu fragmen (pita) DNA dengan panjang 324 pb. Genotipe GC ditunjukkan dengan dua fragmen DNA yaitu 324 dan 474 pb. Genotipe CC ditunjukkan dengan satu fragmen DNA sepanjang 474 pb.
M 1 2 3
Genotipe GG GC CC
Gambar 4. Penentuan Genotipe Gen Calpastatin. M = marker; 1-13 = Pemotongan dengan enzim AluI.
Rancangan dan Analisis Data
Keragaman genotipe pada masing-masing sampel dapat dilihat dari pita-pita yang ditemukan. Masing-masing sampel dianalisis menggunakan pendekatan nilai frekuensi alel, frekuensi genotipe, dan nilai heterozigositas.
Frekuensi genotipe dan alel gen calpastatin dihitung berdasarkan rumus (Nei & Kumar, 2000) :
Xi =
Xii =
Keterangan :
xii = frekuensi genotipe ke-ii
xi = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii
13 N = jumlah individu sampel
Pendugaan nilai heterozigositas pengamatan (Ho) dan heterozigositas harapan (He) dihitung menggunakan rumus (Weir, 1996) :
∑
∑
Keterangan :
Ho = heterozigositas pengamatan (populasi)
nij = jumlah individu heterozigot
N = jumlah individu yang diamati He = nilai heterozigositas harapan
P1i = frekuensi alel ke-I pada lokus I
n = jumlah alel pada lokus ke-I
Pengujian nilai genotipe antara hasil pengamatan dan nilai harapan dapat diukur dengan menggunakan uji Chi-Kuadrat (Nei dan Kumar, 2000):
∑
Keterangan:
X2 = Chi-Kuadrat O = nilai pengamatan E = nilai harapan
Σ = sigma (jumlah dari nilai-nilai)
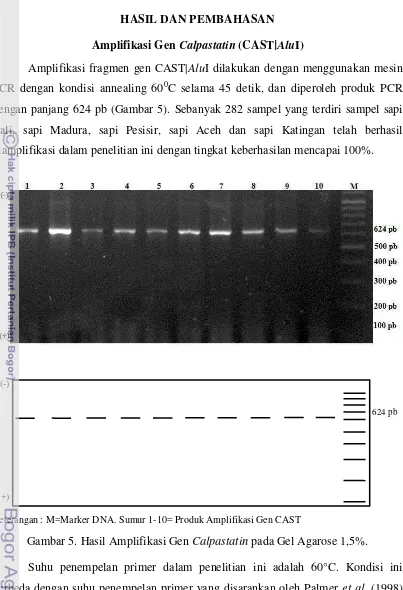
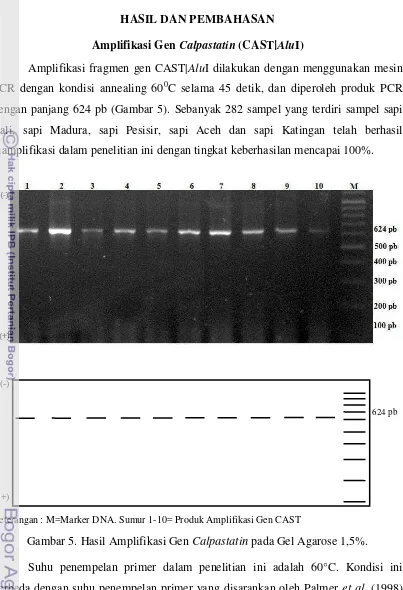
HASIL DAN PEMBAHASAN Amplifikasi Gen Calpastatin (CAST|AluI)
Amplifikasi fragmen gen CAST|AluI dilakukan dengan menggunakan mesin PCR dengan kondisi annealing 600C selama 45 detik, dan diperoleh produk PCR dengan panjang 624 pb (Gambar 5). Sebanyak 282 sampel yang terdiri sampel sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh dan sapi Katingan telah berhasil diamplifikasi dalam penelitian ini dengan tingkat keberhasilan mencapai 100%.
(-)
624 pb
(+)
Keterangan : M=Marker DNA. Sumur 1-10= Produk Amplifikasi Gen CAST
Gambar 5. Hasil Amplifikasi Gen Calpastatin pada Gel Agarose 1,5%.
Suhu penempelan primer dalam penelitian ini adalah 60°C. Kondisi ini berbeda dengan suhu penempelan primer yang disarankan oleh Palmer et al. (1998) yaitu 62°C. Menurut Muladno (2002), suhu penempelan primer (annealing) berkisar antara 360C sampai dengan 720C, namun suhu yang biasa digunakan 50-600C. Suhu
15 Keberhasilan amplifikasi gen sangat ditentukan oleh kondisi penempelan primer DNA genom (gen target) dan kondisi mesin thermocycler. Selain itu, keberhasilan dalam mengamplifikasi DNA tergantung pada interaksi komponen campuran PCR (Palumbi, 1986). Menurut Al Soud dan Radstrom (2001) keberhasilan amplifikasi dipengaruhi oleh adanya hemoglobin yang dapat menghambat kerja enzim taq polymerase.
Penentuan Genotipe Gen Calpastatin
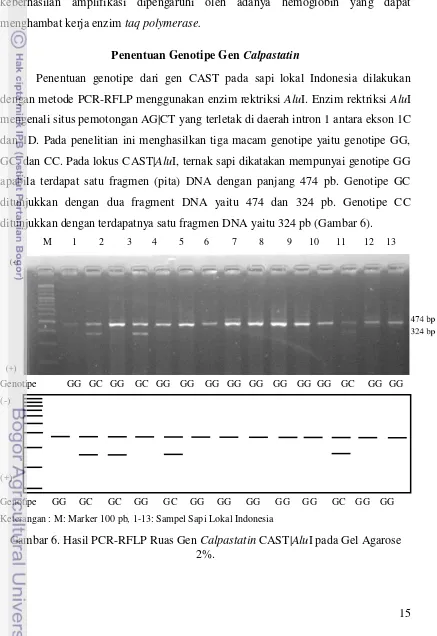
Penentuan genotipe dari gen CAST pada sapi lokal Indonesia dilakukan dengan metode PCR-RFLP menggunakan enzim rektriksi AluI. Enzim rektriksi AluI mengenali situs pemotongan AG|CT yang terletak di daerah intron 1 antara ekson 1C dan 1D. Pada penelitian ini menghasilkan tiga macam genotipe yaitu genotipe GG, GC, dan CC. Pada lokus CAST|AluI, ternak sapi dikatakan mempunyai genotipe GG apabila terdapat satu fragmen (pita) DNA dengan panjang 474 pb. Genotipe GC ditunjukkan dengan dua fragment DNA yaitu 474 dan 324 pb. Genotipe CC ditunjukkan dengan terdapatnya satu fragmen DNA yaitu 324 pb (Gambar 6).
M 1 2 3 4 5 6 7 8 9 10 11 12 13
Genotipe GG GC GG GC GG GG GG GG GG GG GG GG GC GG GG
(-)
(+)
Genotipe GG GC GC GG GC GG GG GG GG GG GC GG GG
Keterangan : M: Marker 100 pb, 1-13: Sampel Sapi Lokal Indonesia
16 Berdasarkan sekuen DNA ruas gen CAST yang diamplifikasi terdapat empat titik pemotongan AluI yang menghasilkan fragmen dengan panjang 61, 52, 474, 10, dan 27 pb yang dikenal dengan alel G, sedangkan alel C memiliki panjang fragmen 324 pb. Menurut Kubiak et al. (2004), keragaman gen calpastatin sapi disebabkan oleh adanya mutasi titik yang terjadi pada posisi antara basa ke-61 (SNP|AluI) ( nomor akses GenBank AF117813). Subtitusi G-C pada titik mutasi mengubah asam amino Serine menjadi asam amino Threonine (Ser-Thr). Kode AGC untuk serine (Ser) dan kode ACC threonine (Thr). Terjadinya subtitusi basa nitrogen (transversi) G – C menyebabkan munculnya situs pemotongan yang baru untuk enzim restriksi
AluI.
Mutasi yang terjadi pada fragmen gen CAST|AluI adalah mutasi subtitusi tipe transversi yaitu terjadi perubahan basa purin (guanin) menjadi basa pirimidin (sitosin). Mutasi transversi terjadi karena adanya subtitusi antara satu basa purin (adenin atau guanin) menjadi satu basa pirimidin (timin atau sitosin) ataupun sebaliknya. Menurut Palmer et al. (1998) terjadinya mutasi pada ruas gen CAST menyebabkan keragaman gen calpastatin yang mempengaruhi kualitas karkas domba, sedangkan pada sapi Angus jantan dapat mempengaruhi sifat pertumbuhannya (Chung et al., 1999).
Ternak sapi dengan genotipe homozigot (GG atau CC) berarti bahwa kedua tetua masing-masing menyumbangkan gen (alel) yang sama. Sapi dengan genotipe heterozigot (GC) menunjukkan bahwa ternak tersebut memiliki kombinasi gen yang berbeda dari kedua tetuanya. Keragaman gen CAST|AluI pada sapi lokal Indonesia ditunjukkan dengan jumlah genotipe yang muncul dari masing-masing bangsa (Tabel 3).
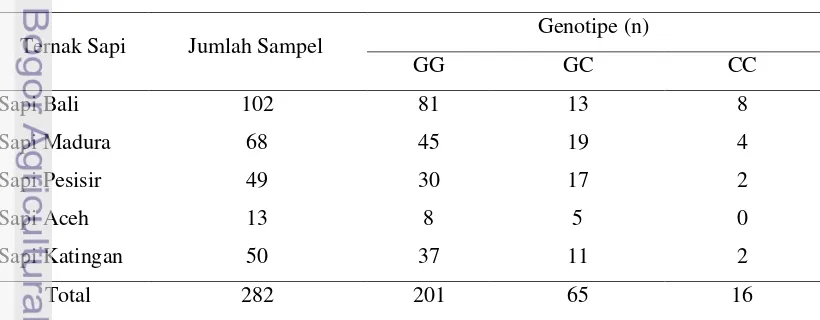
Tabel 3. Hasil Identifikasi Genotipe Gen Calpastatin pada Bangsa Sapi Indonesia
Ternak Sapi Jumlah Sampel Genotipe (n)
GG GC CC
Sapi Bali 102 81 13 8
Sapi Madura 68 45 19 4
Sapi Pesisir 49 30 17 2
Sapi Aceh 13 8 5 0
Sapi Katingan 50 37 11 2
Total 282 201 65 16
17 Berdasarkan hasil identifikasi genotipe gen calpastatin pada bangsa sapi lokal Indonesia, ditemukan tiga macam genotipe yaitu GG, GC, dan CC pada sapi Bali, sapi Madura, sapi Pesisir dan sapi Katingan, sedangkan pada sapi Aceh hanya ditemukan dua macam genotipe yaitu GG dan GC. Hasil penelitian Kubiak et al.
(2004) menunjukkan bahwa amplifikasi PCR-RFLP gen CAST|AluI pada sapi Limousin, sapi Simmental, sapi Polish Friesian dan sapi Polish Red menghasilkan tiga genotipe yaitu GG, GC, dan CC, sedangkan pada sapi Red Angus, sapi Charolaise dan sapi Hereford menghasilkan dua genotipe yaitu GC dan CC.
Keragaman Gen Calpastatin
Hasil analisis frekuensi genotipe (GG, GC, dan CC) dan frekuensi ale (G, C) fragmen gen CAST|AluI pada bangsa sapi Indonesia disajikan pada Tabel 4.
Tabel 4. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen CAST|AluI pada Bangsa Sapi Indonesia
Ternak Sapi N Frekuensi Genotipe Frekuensi Alel GG GC CC G C
Sapi Bali 102 0,794 0,127 0,078 0,858 0,142
Sapi Madura 68 0,662 0,279 0,059 0,801 0,199
Sapi Pesisir 49 0,612 0,347 0,041 0,786 0,214
Sapi Aceh 13 0,615 0,385 0,000 0,808 0,192
Sapi Katingan 50 0,740 0,220 0,040 0,850 0,150
Total 282 0,713 0,230 0,057 0,885 0,115
Keterangan: N = jumlah individu
18 Limousin, Simmental, dan Polish Friesian. Pada populasi Hereford, frekuensi genotipe CC dan GC seimbang (Tabel 5).
Frekuensi alel adalah frekuensi relatif dari suatu alel dalam populasi atau jumlah suatu alel terhadap jumlah total alel yang terdapat dalam suatu populasi (Nei dan Kumar, 2000). Frekuensi alel G tinggi ditemukan pada sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh, dan sapi Katingan, sebaliknya frekuensi alel C rendah pada kelima populasi sapi tersebut sehingga cenderung tidak terdapat perbedaan terhadap frekuensi alel. Hal ini berbeda dengan hasil penelitian Kubiak et al. (2004) menunjukkan bahwa frekuensi alel Bos taurus tertinggi yaitu alel C dan terendah alel G (Tabel 5).
Tabel 5. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen CAST|AluI pada Bos taurus
Ternak Sapi N
Frekuensi Genotipe Frekuensi Alel
GG GC CC G C
Red Angus 9 - 0,111 0,889 0,07 0,93
Charolaise 12 - 0,167 0,833 0,42 0,58
Limousin 10 0,100 0,300 0,600 0,25 0,75
Simmental 9 0,111 0,556 0,333 0,39 0,61
Hereford 8 - 0,500 0,500 0,25 0,75 Polish Friesian 84 0,214 0,417 0,369 0,42 0,58
Polish Red 7 0,286 0,143 0,571 0,36 0,64
Total 138 0,159 0,362 0,479 0,31 0,69
Keterangan: N = jumlah individu
Sumber : Kubiak et al. (2004)
19 Keragaman gen CAST|AluI pada kelima sapi Indonesia bersifat polimorfik dengan ditemukannya frekuensi alel kurang dari 0,99. Menurut Nei (1987), suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama dengan atau kurang dari 0,99 atau lebih dari 1% (Nei dan Kumar, 2000). Tingginya frekuensi alel G pada semua populasi sapi lokal Indonesia diduga akibat seleksi dan manajemen perkawinan yang dilakukan oleh peternak. Seleksi merupakan suatu proses yang melibatkan kekuatan-kekuatan untuk menentukan ternak mana yang boleh berkembang biak pada generasi selanjutnya (Noor, 2010). Seleksi yang dilakukan peternak yaitu mempertahankan individu-individu yang memilki alel G dibandingkan dengan individu-individu yang memiliki alel C. Menurut Noor (2010), faktor-faktor yang mempengaruhi frekuensi gen adalah seleksi, mutasi, pencampuran populasi, silang dalam, silang luar dan
genetic drift.
Keseimbangan Gen dalam Populasi
Hasil uji chi-square (X2), terhadap genotipe fragmen gen CAST|AluI (Lampiran 4) menunjukkan bahwa frekuensi gen dalam keadaan seimbang pada populasi sapi Madura, sapi Pesisir, sapi Aceh, dan sapi Katingan. Ketidakseimbangan genotipe hanya terjadi pada populasi sapi Bali yang terdapat di Balai Pembibitan Ternak Unggul Sapi Bali. Keseimbangan frekuensi genotipe pada sapi Madura, sapi Pesisir, sapi Aceh, dan sapi Katingan menunjukkan bahwa pada populasi tersebut tidak terjadi seleksi terutama seleksi yang dilakukan terhadap gen CAST, sebaliknya ketidakseimbangan frekuensi genotipe sapi Bali di Balai Pembibitan Ternak Unggul disebabkan oleh seleksi yang secara tidak langsung berhubungan dengan gen CAST.
20
Pendugaan Nilai Heterozigositas
Pendugaan nilai heterozigositas pengamatan (Ho) dan nilai heterozigositas harapan (He) gen CAST|AluI pada bangsa sapi Indonesia tersaji pada Tabel 6.
Tabel 6. Nilai Heterozigositas Pengamatan (Ho) dan Heterozigositas Harapan (He) gen CAST|AluI pada Bangsa Sapi Indonesia
Ternak Sapi N Heterozigositas
Ho He
Bali 102 0,127 0,244
Madura 68 0,279 0,318
Pesisir 49 0,347 0,337
Aceh 13 0,385 0,311
Katingan 50 0,220 0,255
Keterangan: N = jumlah individu
Pada Tabel 5 menunjukkan bahwa nilai heterozigositas yang paling tinggi ditemukan pada sapi Aceh (0,385), sebaliknya nilai heterozigositas terendah ditemukan pada sapi Bali (0,127). Meskipun demikian, hasil yang diperoleh masih memiliki keterbatasan ukuran jumlah sampel yang digunakan terutama pada populasi sapi Pesisir, sapi Madura, sapi Aceh, dan sapi Katingan yang memiliki jumlah sampel relatif sedikit.
Menurut Diyono (2009), nilai heterosigositas dipengaruhi oleh jumlah sampel, jumlah alel dan frekuensi alel. Heterozigositas menggambarkan adanya variasi genetik pada suatu populasi. Semakin tinggi nilai heterozigositas pada suatu populasi maka tinggi pula variasi genetik pada populasi tersebut (Ferguson, 1980). Menurut Marson et al. (2005) pendugaan nilai heterozigositas dapat digunakan untuk mendapatkan gambaran variabilitas genetik pada suatu populasi. Keragaman genetik dapat diukur secara akurat dengan nilai heterosigositas (ĥ) (Nei, 1987).
KESIMPULAN DAN SARAN Kesimpulan
Keragaman gen calpastatin (CAST|AluI) pada sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh, dan sapi Katingan bersifat polimorfik. Identifikasi genotipe gen
calpastatin pada bangsa sapi lokal Indonesia, ditemukan tiga macam genotipe yaitu GG, GC, dan CC pada sapi Bali, sapi Madura, sapi Pesisir dan sapi Katingan, sedangkan pada sapi Aceh hanya ditemukan dua macam genotipe yaitu GG dan GC. Proporsi genotipe GG lebih tinggi pada semua populasi sapi Indonesia, sehingga alel G dapat dijadikan sebagai salah satu alel (penciri) spesifik pada semua bangsa sapi Indonesia.
Saran
UCAPAN TERIMA KASIH
Puji dan syukur senantiasa penulis panjatkan kepada Allah SWT atas nikmat, rahmat, dan karunia yang telah diberikan sehingga penelitian dan skripsi ini dapat terselesaikan. Shalawat dan salam semoga senantiasa terlimpah kepada Rasulullah Muhammad SAW, keluarga, para sahabat dan pengikutnya. Terima kasih sebesar-besarnya penulis ucapkan kepada Prof. Dr. Ir. Muladno, MSA. selaku pembimbing utama dan Dr. Jakaria, S.Pt., M.Si. selaku pembimbing anggota atas segala perhatian, bimbingan, arahan, dan motivasi yang telah diberikan.
Terima kasih penulis ucapkan kepada Ayah dan Ibu tercinta, Bapak Ahmad Lutfi dan Ibu Sumiati yang senantiasa mencurahkan kasih sayang, dukungan moral dan material, serta doa-doa yang selalu dipanjatkan untuk kesuksesan penulis. Kepada adik-adik tersayang, Ahmad Reza Khalafi dan Muhammad Hasby Ash Shidiq yang telah memberikan senyuman, motivasi, dan doanya. Terima kasih atas semuanya, atas keceriaan dan kebersamaannya. Terima kasih kepada Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. yang senantiasa memberikan motivasi dan masukan selama penulis berada di Laboratorium Genetika Molekuler Ternak Fapet IPB. Terima kasih kepada Dr. Ir. Henny Nuraini, M.Si. selaku pembimbing akademik yang selalu memberikan nasihat, semangat dan motivasi kepada penulis. Terima kasih penulis ucapkan kepada Dr. Ir. Sri Darwati, M.Si., Dr. Ir. Ahmad Darobin Lubis, M.Sc., dan Ir. Lucia Cyrilla, M.Si. atas masukan terhadap skripsi penulis.
Terima kasih penulis ucapkan kepada kak Eryk, Pak Andi, Irine, Ima, Ferdy, dan David yang telah banyak membantu penulis selama penelitian. Kepada Cicha, Gya, Febi, Eka, Isyana, Tessa, Rey, Sri, Adil, Menix sebagai teman-teman seperjuangan terbaik selama penulis melakukan penelitian di Laboratorium LGMT, Yogi, Dina, Yudi, Huda, Catur, Ade, Aldi, Angga, Sugma, Riza, Artadi, Luthfi, Gigih, Putri, Silvi, Ninta, Munthe, dan Dewi, serta teman-teman IPTP 45, penulis mengucapkan banyak terima kasih untuk bantuan, kebersamaan, dan kekeluargaan yang telah terbina.
DAFTAR PUSTAKA
Adrial. 2010. Sapi Lokal Kalimantan Tengah Sumberdaya Lokal Potensi yang Belum Termanfaatkan Secara Optimal. Balai Pengkajian Teknologi Pertanian Kalimantan Tengah.
Al-Soud, W. A. & P. Radstrom. 2001. Purification and characterization of PCR inhibitory components in bloods cells. J. Clin. Microbiol. 39: 485-493.
Barendse, W. 2002. Assessing Lipid Metabolisme International Patent Application PCT/AU98/00882, International Patent Publication WO 99/23428.
Bishop, M. D., M. Koohmaraie, J. Killefer & S. Kappes. 1993. Rapid communication: restriction lenght fragment polymorphisms in the bovine calpastatin gene. J.Anim.Sci. 71: 2277.
Bourdon, R. M. 2000. Understanding Animal Breeding. 2nd Edition. Prentice Hall Inc. Upper Saddle River, New Jersey. USA.
Chung, H. Y., M. E. Davis, H. C. Hines & D. M. Wulf. 1999. Effect of the calpain proteolysis and calpain genotype on meat tenderness of angus bulls. J. Anim. Sci. 77: 31-38.
Departemen Pertanian. 2006. Peraraturan Menteri Pertanian Nomor 35/Permentan/ OT.140/8/2006. Pedoman Pelestarian dan Pemanfaatan Sumberdaya Genetik Ternak. Deptan, Jakarta.
Direktorat Jendral Peternakan. 2008. Buku Statistik Peternakan 2008. Jakarta: Ditjenak, Departemen Pertanian Republik Indonesia.
Diyono, R. 2009. Karakteristik ukuran tubuh dan polimorfisme gen GH, GHRH, Pit-1 pada populasi kerbau di Banten. Tesis. Bogor. Program Pascasarjana, Institut Pertanian Bogor.
Ferguson, A. 1980. Biochemical Systematics and Evolution Lecturer in Zoology. The Queens University of Belfast. London.
Food Agriculture Organization. 1999. The Global Strategy for The Management of Farm Animal Genetic Resources. Rome.
Gillespie, J. H. 1998. Population Genetics, A Concies Guide. The Johns Hopkins University Press. London.
Gunawan. 1998. Upaya peningkatan mutu genetik sapi aceh. Pidato Pengukuran dalam Jabatan Guru Besar Tetap Fakultas Pertanian Universitas Syiah Kuala. Disampaikan pada Rapat Senat Terbuka Universitas Syiah Kuala, Sabtu 28 Maret 1998, Banda Aceh.
25 Hartl, D. L. & A. G. Clark. 1997. Principle of Population Genetic. Sinauer
Associates, Sunderland, M. A.
Haryana, I. G. N. R. 1989. Beberapa aspek biologi reproduksi sapi bali jantan muda. Disertasi. Program Pasca Sarjana. Institut Pertanian Bogor.
Hediger, R., H. A. Ansari & G. F. Stranzinger. 1991. Chromosome banding and gene localizations support extensive conservation of chromosome structure between cattle and sheep. Cytogenet. Cell Genet. 57: 127-134.
Huitema, H. 1986. Peternakan di Daerah Tropis Arti Ekonomi dan Kemampuannya. Penelitian di Beberapa Daerah di Indonesia. Terjemahan. Peni Hardjosworo. Yayasan Obor Indonesia. PT. Gramedia. Jakarta.
Kirby, G. W. M. 1979. Bali Cattle in Australia World Animal Review. FAO. 31:24-29.
Koohmaraie, M., S. D. Shackelford, T. L. Wheeler, S. M. Lonergan & M. E. Doumit. 1995. A muscle hyperthrophy condition in lamb (callipyge): characterization of effects on muscle growth and meat quality traits. J. Anim. Sci. 73: 3596-3607.
Kubiak, E. J., J. R. Stanislaw, W. Krystyna, S. Tomasz & S. Tomasz. 2004. A nofel RFLP|Alu1 polymorphism of the bovine calpastatin (CAST) gene and its association with selected trait of beef. Animal Science Papers and Reports. 22 (2): 195-204.
Li, W. H. & D. Graur. 1991. Fundamental of Moleculer Evolution. Sinauer Associates Inc. Publisher. Sunderlad, Massachusetts.
Ma’sum, K. 1992. Hasil penelitian sapi madura di sub Balai Penelitian Ternak Grati. Pertemuan Ilmiah Hasil Penelitian dan Pengembangan Sapi Madura. Sub Balai Penelitian Ternak Grati. Pasuruan; 45-54.
Machado, M. A., I. Schuster, M. L. Martinez & A. L. Campos. 2003. Genetic diversity of four breed using microsatellite markers. Rev. Bras. De Zool. 32:93-98.
Marson, E. P., B. S. Jose, V. M. Flavio, C. Julio, P. E. Joanir, G. Luis & B. M. Gerson. 2005. Genetic characterization of European Zebu composite bovine using RFLP markers. Genet. Mol. Res. 4 : 496-505.
Martojo, H. 2003. Indigenous Bali cattle: The best suited cattle breed for sustainable small farm in Indonesia. The Chinese Society of Animal Science. 112 Farm Road, Hsinhua, Tainan, Taiwan.
Moioli, B., F. Napolitano & G. Catillo. 2004. Genetic diversity between Piedmontese, Maremmana and Podolica cattle breeds. J. Hered. 95: 250-256.
26 Muladno. 2002. Seputar Teknologi Rekayasa Genetik. Pustaka Wira Usaha Muda,
Bogor
Namikawa, T., Y. Matsuda, K. Kondo, B. Pangestu & H. Martojo. 1980. Blood group and blood polymorphism of dufferent types of the cattle in Indonesia. The Origin and Phylogeny of Indonesia Native Livestock. Bogor. The Research Group of Overseas Scientific Survey. 35-45.
Namikawa T., J. Amano & H. Martojo. 1982. Coat color variations of Indonesia cattle. The Origin and Phylogeny of Indonesia Native Livestock (Part III). The Research Group of Overseas Scientific Survey. Tokyo, Japan. 35-42.
Nei, M. 1987. Molecular Evolutionary Genetics. Columbia University Press, New York.
Nei & Kumar. 2000. Moleculear Evolution and Phylogenetics. New York: Oxford University Press.
Noor, R. R. 2010. Genetika Ternak. Penebar Swadaya., Jakarta.
Orita, M., H. Iwahana, H. Kanazawa, K. Hayashi & T. Sekiya. 1989. Detection of polymorphism of human DNA by gel electrophoresis as single-strand conformation polymorphism. Proc Natl Acad Sci 86:2766-2770.
Page, B. T., E. Casas, R. L. Quaas, R. M. Thallman, T. L. Wheeler, M. Koohmaraie, S. D. Schackelford, S. N. White, J. W. Keele & T. P. L. Smith. 2004. Association of markers in the bovine CAPN1 with meat tenderness in large corssbred poulations that sample influential industry sires.J.Anim. Sci. 80 :3474 – 3481.
Page, B. T., E. Casas, M. P. Heaton, N. G. Cullen, D. L. Hyndman, C. A. Morris, A. M. Crawford, T. L. Weeler, M. Koohmaraie, J. W. Keele & T. P. L. Simth. 2002. Evaluation of singlenucleotide polymorphisms in CAPN1 for association with meat tenderness in cattle. J. Anim. Sci.80 : 3077-3085.
Palmer, B. R., N. Roberts, J. G. H. Hickford & R. Bickerstaffe. 1998. Rapid communication : PCR-RFLP for Msp1 and NcoI in the ovine calpastatin gene. American Society. Animal. Sci. 76 : 1499-1500.
Palumbi, S. R. 1986. Nucleic acid II: polymerase chain reaction. In: D.M. Hillis, C. Moritz dan B.K. Mable (Editor). Molecular Systematics. 2nd Edition. Sinauer Associates. Inc., Massachusetts USA.
Payne, W. J. & J. Hodges. 1997. Tropical Cattle : Origin, Breeds and Breeding Policies. Blackwell Science.
27 Sambrook J., E. F. Fritsch & T. Maniatis. 1989. Moleculer Cloning: a Laboratory
Manual. CSH Laboratory Press. USA.
Sarbaini. 2004. Kajian keragaman karakteristik eksternal dan DNA mikrosatelit sapi pesisir Sumatera Barat. Disertasi. Sekolah Pasca Sarjana, Institut Pertanian Bogor, Bogor.
Schenkel, F.S., S.P. Miller, X. Ye, S.S. Moore & J.D. Nkrumah. 2006. Association of single nucleotide polymorphism in the leptin gene with carcass and meat quality traits of beef cattle. J Anim Sci 83:2009-2020.
Soehadji. 1992. Kebijaksanaan pengembangan ternak potong di Indonesia tinjauan khusus sapi madura. Proceedings. Pertemuan Ilmiah Hasil Penelitian dan Pengembangan Sapi Madura. Sub Balai Penelitian Ternak Grati. Pasuruan; 1-12.
Sumantri, C., A. Anggraeni., A. Farajallah., & D. Perwitasari. 2007. Keragaman mikrosatelit DNA sapi perah FH di balai pembibitan ternak unggul Baturraden. Jurnal Ilmu Ternak dan Veteriner 12 (2): 124-133.
Tambasco, D. D., Paz, M. M. Alencar, Freitas, L. L. Coutinho, Packer & Regitano. 2003. Candidate genes for growth traits in beef cattle crosses Bos taurus x Bos indicus. J. Anim. Breed. Genet. 120: 51-60.
Utomo, B.N., R.R. Noor, C. Sumantri, I. Supriatna & E.D. Gunardi. 2010. Keragaman morfometrik dan fenotipik sapi Katingan di Kalimantan Tengah. JITV. 15 (in Press).
Utoyo, D.P. 2002. Management of Farm Domestic Animal Genetic Resource in Indonesia. Di dalam Animal Genetic Resource. Directorate Generale of Livestock Services. Ministry of Agriculture Indonesia. Jakarta.
Vignal, A., D. Milan, M. Sancristobal & A. Eggen. 2002. A review on SNP and other types of molecular markers and their use in animal genetics. Genet Sel Evol 34:275-305.
Weir, B. S. 1996. Genetic Data Analysis : Method for Discrete Population Genetic Data. Second ed. Sinauer Associates. Sunderland, MA USA.
Williams, J. L. 2005. The use of marker-assisted selection in animal breeding and biotechnology. Rev Sci Technol Int Epiz 24:379-391.
29 Lampiran 1. Sekuen gen Calpastatin Bos taurus dengan Nomor Akses
GenBank AF117813
LOCUS AF117813 624 bp DNA linear MAM 21-MAR-1999 DEFINITION Bos taurus calpastatin (CAST) gene, partial cds. ACCESSION AF117813
VERSION AF117813.1 GI:4455058 KEYWORDS .
SOURCE Bos taurus (cattle) ORGANISM Bos taurus
30
ORIGIN
1 tggggcccaa tgatgccatc gatgccttgt catccgactt cacctgcagt tcccctacag
61 ctgatgcaaa gaaaactgag aaagaggtat ggtttttaat gcccttaggg aagcttgtta
121 gaaactacct cccactttaa gacaacaact tttttttaaa cttcattttt cacttcactg
181 cgtcttcatt gctgtgttcg ggctttctct agttggggca agcgaggcct gttctctatt
241 tgcaattttt aggcttctgc agggggctcc tcttgttgct gggccggggc tctaggtgca
301 caggcttcat ttgttgtggc tcgagggctc taaaccacag gctcattggt cttggcgcac
361 gggcatggtt accccaatgc atttgggatc tcccctggcc agggagcaaa cctgtttccc
421 ctgcattgca aggcggcctc ttaaccgctg gccaccaggg aagccccaaa atgccaaggc
481 tttttacttc tggttcttac cgtttggttc atatttttcc ttcatctgcc agtcaaacct
31
Lampiran 2. Sekuen gen Calpastatin Bos taurus dengan Nomor Akses GenBank AY834765
LOCUS AH014526S01 2418 bp DNA linear MAM 19-APR-2005 DEFINITION Bos taurus calpastatin (CAST) gene, exons 1xa and 1xb. ACCESSION AY834765
Mammalia; Eutheria; Laurasiatheria; Cetartiodactyla; Ruminantia; Pecora; Bovidae; Bovinae; Bos.
REFERENCE 1 (bases 1 to 2418)
AUTHORS Raynaud,P., Jayat-Vignoles,C., Laforet,M.P., Leveziel,H. and Amarger,V.
TITLE Four promoters direct expression of the calpastatin gene JOURNAL Arch. Biochem. Biophys. 437 (1), 69-77 (2005)
PUBMED 15820218
REFERENCE 2 (bases 1 to 2418)
AUTHORS Raynaud,P., Jayat,C., Laforet,M.-P., Leveziel,H. and Amarger,V. TITLE Direct Submission
33
34
61 catgaaacat cattgcactt tcaaaacaag taaagccgca caaaacacac ccaggcccgt 121 gtgtgtacac acacacacac acacacacac gcacacacac acacaatcac aacacacgga 181 gtcacacaca gaccaatttg tttctgcaat ccgcttcctc atccagaagg tccaggcccc 241 gggccatctg agttggtaaa ttctcctcct agttaactca gagcagattg cagaaatgct 301 gtctaattct tgagattctt agggagtggg gcttgagatg tagacggggc cacgccccgg 361 tcccgctctc cttgcacaac tgcgagccaa gtctaggcaa gtcgggggga aaaccccgcc 421 atctccagcc cctcctctct gcgacccact ggggcaccaa ggcgacctcg ggtggggtgg 481 ggtgtccctg ggagaagata cgggacccag ggtgtgagtt gcaaacaggc agccccgggc 541 tgctgccgcc cgggcgctgc caactgcagg caggaagggg agggccctgc ccggcgaagg 601 ggagctctcg cgggtcgggg ctgggtcgga aaagctgcct cacaggcgcg cccgccagcc 661 cctccgcgcc ctcgctccct cccagcgctc cccggctcca gcctcccttc caggctccgc 721 cgcgcccgcc cggaggcagc gctcgcaccg gcctcgccat gtcccagccg ggcccgaagc 781 ccgccgcctc cccgcggccc cggcgcgctg cccgccacac ccaggaggtg agtggcgctc 841 ctaccgccgg ggttgagcgc ggggaggatc tcggggcccc ccgaagcctc gggtccacct 901 tcagaggagg gttagccttc ctggccttct gtccctgggc gccacccctc ccaggcccgg 961 agaaacatct ggagagagac gggtgtggac tcaggacctg cctctgggca ggtgaccgga 1021 ggggcccgcg gatggccctg ggtcccggcc aagttcactg ggcgcggcgc aagccacatt 1081 ggagtgggct tccctggcgg ctcagctggt gaagaatccg cctacaatgc gggagacctg 1141 ggctcgttgg gttgggaaga tcccctggag aagggaactg gctcccagta tcctggcctg 1201 gagaattccc tgcactggat agtccatggg tggcggagtc aggacacgac tgagcgactt 1261 ttgctcactt gtcactcatg cgttgggaaa agttggtgga tgagacaggg gtgggtgaga 1321 agcggataag ggtaaaagga gaatgaaatt catttgggac cccccgggat tccggagtct 1381 gtaagatcag cagaaaagtc tggtcttccg cccagtcagg gctgcagggc gtggctgcct 1441 ggagcaataa cacttcttcc ctaagcagct ttgagccaaa ccggcagggc ggggcgggga 1501 ggggcggggc aggggcgggg aggggcgggg agagggcggg gtgagccgga ggggcggggt 1561 ctgggtaggc cccgcccagc tgtcttgccc accccctgac agcctaggtg cttacagagt 1621 tagtcccagt caggtctgcg gcaggtggag tgcgaacccg tggccctttg ctgcgctgca 1681 cccgtgtcct cgccgggtcc ctcgggtctc tcgctgcgtc tctcggaaca catccatcgt 1741 cgccatggca tttgcaagct ggtggtacaa gacggtaaat aggagtgatc gtccctgggc 1801 aggactggga agggaatgtg ctctgtcttc ctgggctgta ggcgaggtca ctgttcagat 1861 tttcgggcga gggttgggga gcaggttgtg acctcctctc agaccttcag gggaggctcc 1921 gggagaggct gagacccacc ctgctgtgga atgtgggagc cagctcggac gtacacgtgc 1981 tagtcggcgt gagttcaggc tcacaagttg aatggcatag ggattgttgc tggacgaatt 2041 ctgccctcgg acgcggattc ctggatgaac gcggactcag aaagcgctca gctctagagt 2101 ttgtttttaa ctcataagta aagcacaaaa ctttcagagg cgtgttgttg ggcgtgtatt 2161 ttccagcgcc aagtagacag gtaactaaga tagtggggtg gggcaaggaa aaaaatccca 2221 aaactccaag gaacataaac aactgagatc aagacccttg ttgagaatga gagagagaga 2281 gggagactct atactttatt gcttggatcg ggggcaaaga tgttccattt atttaatgca 2341 ctgaattcct ctccgaatat gtaagtgtga tatccagtca agctggtact tgtaatgttt 2401 tgaaccagga agcatgct
35
Lampiran 3. Sekuen gen Calpastatin Bos taurus dengan Nomor Akses GenBank AY834775
LOCUS AH014526S11 3241 bp DNA linear MAM 19-APR-2005 DEFINITION Bos taurus calpastatin (CAST) gene, exons 29, 30 and complete cds,
Mammalia; Eutheria; Laurasiatheria; Cetartiodactyla; Ruminantia; Pecora; Bovidae; Bovinae; Bos.
REFERENCE 1 (bases 1 to 3241)
AUTHORS Raynaud,P., Jayat-Vignoles,C., Laforet,M.P., Leveziel,H. and Amarger,V.
TITLE Four promoters direct expression of the calpastatin gene JOURNAL Arch. Biochem. Biophys. 437 (1), 69-77 (2005)
PUBMED 15820218
REFERENCE 2 (bases 1 to 3241)
AUTHORS Raynaud,P., Jayat,C., Laforet,M.-P., Leveziel,H. and Amarger,V. TITLE Direct Submission
39
40
41
Lampiran 4. Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen Gen CAST pada Bangsa Sapi Indonesia
Lampiran 4a. Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen Gen CAST Sapi Bali
Pengamatan n
Genotipe Frekuensi Alel
Uji X2
GG GC CC G C
Jumlah diamati
(O) 102 81 13 8 0,858 0,142 23,251
Jumlah
diharapkan (E) 75,06 24,88 2,06 (n)
Keterangan : X2 tabel, db (n-1), α 5% = 5,99; n: nyata; tn: tidak nyata
Lampiran 4b. Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen Gen CAST Sapi Madura
Pengamatan n Genotipe Frekuensi Alel Uji X2
GG GC CC G C
Jumlah diamati
(O) 68 45 19 4 0,801 0,199 1,012
Jumlah
diharapkan (E) 43,68 21,64 2,68 (tn)
Keterangan : X2 tabel, db (n-1), α 5% = 5,99; n: nyata; tn: tidak nyata
Lampiran 4c. Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen Gen CAST Sapi Pesisir
Pengamatan n
Genotipe Frekuensi Alel
Uji X2
GG GC CC G C
Jumlah diamati
(O) 49 30 17 2 0,786 0,214 0,045
Jumlah
diharapkan (E) 30,25 16,50 2,25 (tn)
42 Lampiran 4d. Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen
Gen CAST Sapi Aceh
Pengamatan n
Genotipe Frekuensi Alel
Uji X2
GG GC CC G C
Jumlah diamati
(O) 13 8 5 0 0,808 0,192 0,737
Jumlah
diharapkan (E) 8,481 4,038 0,481 (tn)
Keterangan : X2 tabel, db (n-1), α 5% = 5,99; n: nyata; tn: tidak nyata
Lampiran 4e. Hasil Uji X2 Keseimbangan Hardy-Weinberg (p2+2pq+q2) Fragmen Gen CAST Sapi Katingan
Pengamatan n
Genotipe Frekuensi Alel
Uji X2
GG GC CC G C
Jumlah diamati
(O) 50 37 11 2 0,850 0,150 0,942
Jumlah
diharapkan (E) 36,13 12,75 1,125 (tn)
PENDAHULUAN Latar Belakang
Sapi potong merupakan hewan ternak yang dipelihara untuk diambil daging sebagai hasil utamanya. Banyak bangsa sapi potong yang terdapat di dunia, beberapa diantaranya terdapat di Indonesia. Beberapa bangsa sapi potong yang terdapat di Indonesia adalah sapi Bali, sapi Madura, sapi Pesisir, sapi Aceh dan sapi Katingan. Kelima sapi tersebut memiliki sifat dan karakteristik yang berbeda. Bangsa sapi tersebut sangat potensial untuk dikembangkan sebagai sumberdaya genetik ternak lokal Indonesia. Namun, keberadaan bangsa sapi Indonesia masih kurang mendapat perhatian serius dari pemerintah dan masyarakat sehingga pengembangan dan pemanfaatan bangsa sapi Indonesia belum optimal.
Keempukan daging (meat tenderness) merupakan faktor yang penting di dalam penentuan kualitas daging. Beberapa faktor diidentifikasi berhubungan dengan proses pengempukan daging setelah pemotongan seperti lama rigormortis, pH, dan enzim keempukan daging. Perubahan fisiologi struktur otot setelah pemotongan merupakan proses fisiologis yang kompleks. Dua enzim yang berperan terhadap keempukan daging adalah enzim calpastatin yang dihasilkan oleh gen CAST dan
calpain yang dihasilkan oleh gen calsium-activative neural protease (CAPN 1) (Koohmarie, 1966). Calpastatin merupakan enzim protease utama dan bersifat inhibitor spesifik terhadap fungsi µ-calpain dan m-calpain. Aktivitas calpastatin
meningkat ketika aktivitas degradasi protein pada jaringan otot hewan hidup menurun. Calpastatin berfungsi untuk menghambat proses degradasi protein sel otot. Selain itu, diduga kuat terkait dengan sifat pertumbuhan otot dan keempukan daging pada mamalia (Morgan et al., 1993).
2
Tujuan
TINJAUAN PUSTAKA Sapi Lokal Indonesia
Indonesia merupakan salah satu negara yang memiliki sumberdaya genetik ternak. Sapi merupakan salah satu jenis ternak di Indonesia yang memiliki potensi besar terutama sebagai ternak penghasil daging. Direktorat Jendral Peternakan (2008) melaporkan bahwa keberadaan sapi lokal Indonesia pada beberapa tahun terakhir ini masih kurang mendapat perhatian serius baik dari pemerintah maupun masyarakat. Dilihat dari potensi yang dimiliki, sapi lokal Indonesia mampu secara sosial dan budaya berinteraksi dengan masyarakat sejak lama.
Sumberdaya genetik ternak adalah semua yang termasuk dalam spesies, bangsa, dan strain (galur) ternak yang secara ekonomi, ilmiah, dan budaya penting bagi umat manusia baik dalam bentuk makanan maupun produksi (Food Agriculture Organization, 1999). Departemen Pertanian (2006) menyatakan bahwa sumberdaya genetik ternak adalah substansi yang terdapat dalam bentuk individu suatu populasi rumpun ternak secara genetik unik, terbentuk dalam proses domestikasi dari masing-masing spesies yang memiliki potensial serta dapat dimanfaatkan dan dikembangkan baik untuk menciptakan rumpun atau galur unggul.
Sumberdaya ternak sapi Indonesia saat ini terdiri dari tiga kelompok yaitu ternak asli, ternak impor dan ternak yang telah beradaptasi. Beberapa bangsa sapi yang menjadi sumberdaya adalah sapi Aceh, sapi Madura, sapi Pesisir, dan sapi Peranakan Ongole. Keragaman sapi di Indonesia terbentuk dari sumberdaya genetik asli dan impor. Sehubungan dengan pentingnya nilai konservasi pada kelompok ternak, maka beberapa bangsa sapi menjadi target konservasi sekaligus pemanfaatannya (Utoyo, 2002). Salah satu jenis sapi lokal Indonesia adalah sapi Bali. Sapi Bali dipercaya sebagai salah satu sapi lokal Indonesia yang diperoleh dari hasil domestikasi Banteng (Namikawa, 1980). Martojo (2003) menyatakan bahwa hasil domestikasi spesies Bos (Bibos) banteng adalah sapi Bali (Bos sundaicus) atau (Bos javanicus) yang sekarang telah menjadi bangsa ternak asli Indonesia.
4 Tabel 1. Karakteristik Fenotipik Bangsa Sapi Indonesia
Ternak Karakteristik Literatur
Sapi Madura Berukuran sedang, pertulangan bagus, berotot bagi sapi jantan, tanduk kecil ke atas atau ke samping, kaki dan teracak kuat, gumba berkembang baik pada jantan, terdapat lingkaran putih di sekitar moncong. Bobot badan 248 kg putih), temperamen jinak, berpunuk kecil sampai sedang, bertanduk pendek mengarah ke luar, bobot badan lebih berukuran sedang (jantan), memiliki warna cokelat yang lebih gelap pada bagian depan dibandingkan bagian belakang tubuhnya. Bobot badan 302 kg
Namikawa. et al.
(1982), Otsuka et al.
(1980), Adrial (2010)
Sapi Katingan Ukuran tubuh jantan tidak terlalu besar dibandingkan betina, memilki gumba yang cukup jelas, tonjolan pada kepala bagian atas pada betina, tanduk melengkung ke depan
Utomo et al. (2010)
5
(a) (b)
(c) (d)
(e) (f)
Gambar 1. Karakteristik Fenotipik Bangsa Sapi Indonesia. (a) Sapi Bali Betina (b) Sapi Bali Jantan (c) Sapi Madura (d) Sapi Pesisir (e) Sapi Aceh dan (f) Sapi Katingan.
Sumber : Republika, 2011
Penanda Molekuler
Penanda molekuler merupakan pemanfaatan dari keragaman pada tingkat
6 terjadinya proses seleksi (Vignal et al., 2002). Tipe dasar perubahan dalam DNA meliputi substitusi, delesi, inersi, dan inversi (Nei dan Kumar, 2000).
Single nucleotide polymorphims (SNP) merupakan penanda yang memiliki perbedaan satu nukleotida dalam sekuen DNA, dan diperkirakan bahwa satu SNP terjadi setiap 250-1000 bp. Perbedaan tersebut disebabkan oleh terjadinya proses substitusi, sehingga biasanya memiliki dua kemungkinan pada posisi yang sama dalam sekuen DNA (Vignal et al., 2002). Frekuensi mutasi dan stabilitas yang tinggi menyebabkan SNP sering digunakan sebagai penanda molekuler dalam penelitian tentang genetika populasi dan pemetaan gen untuk penyakit kompleks (Ye et al., 2001). Metode yang digunakan dalam menganalisis adanya SNP antara lain PCR-RFLP dan sequencing.
Polymerase Chain Reaction (PCR) merupakan suatu teknik untuk menggandakan jumlah molekul DNA scara in vitro. Proses ini berjalan dengan bantuan enzim polymerase dan primer. Primer merupakan oligonukleotida spesifik pada DNA template. Enzym polymerase merupakan enzim yang dapat mencetak urutan DNA baru. Hasil PCR dapat langsung divisualisasikan dengan elektroforesis atau dapat digunakan untuk analisis lebih lanjut (Williams, 2005).
Restriction Fragment Lenght Polymorphism (RFLP) merupakan salah satu teknik penciri genetik (genetic marker) yang dikembangkan oleh Botstein et al. (1980) yang digunakan untuk mengetahui adanya keragaman sekuens DNA. Mullis
et al. (1986) menyatakan bahwa penggunaan teknik RFLP menjadi lebih intensif setelah teknik RFLP dikombinasikan dengan teknologi PCR yang digunakan hingga saat ini.
PCR-RFLP merupakan metode analisis lanjutan dari produk PCR. Metode PCR-RFLP memanfaatkan perbedaan pola pemotongan enzim pemotong yang berbeda pada tiap-tiap mikroorganisme. Analisis RFLP sering digunakan untuk mendeteksi lokasi genetik dalam kromosom (Orita et al., 1989), ataupun untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
Gen Calpastatin
7 dengan proses pengempukan daging setelah pemotongan seperti lama rigormortis, pH,dan sistem enzim keempukan daging. Perubahan fisiologi struktur otot setelah pemotongan merupakan proses fisiologis yang kompleks. Dua enzim yang berperan terhadap keempukan daging adalah enzim calpastatin yang dihasilkan oleh gen CAST dan calpain yang dihasilkan oleh gen calsium-activative neural protease
(CAPN 1) (Koohmarie, 1966). Calpastatin merupakan enzim protease utama dan bersifat inhibitor spesifik terhadap fungsi µ-calpain dan m-calpain. Aktivitas
calpastatin meningkat ketika aktivitas degradasi protein pada jaringan otot hewan hidup menurun. Calpastatin berfungsi untuk menghambat proses degradasi protein sel otot. Selain itu, diduga kuat terkait dengan sifat pertumbuhan otot dan keempukan daging pada mamalia (Morgan et al., 1993).
Gen calpastatin pada ternak sapi (Bos taurus) terletak pada kromosom nomor 7 (Bishop et al., 1993) sedangkan pada domba nomor 5 (Hediger et al., 1991). Gen
calpastatin dengan simbol CAST terletak diantara dua penciri apit mikrosatelit MCM527 dan BMS1247 pada posisi lokus 5q15 – q21 antara 96,057-96,136 Mb (Gambar 2).
Gambar 2. Daerah Target Amplifikasi Gen Calpastatin
Sumber : Palmer et al., 2000
Kubiak et al. (2004) melaporkan bahwa terdapat keragaman gen Calpastatin