TIRAM MUTIARA (
Pinctada margaritifera
)
BERDASARKAN ANALISIS DNA MITOKONDRIA
RINI SUSILOWATI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Bogor, Agustus 2008
RINI SUSILOWATI C 151 060 151
Pearl Oyster (Pinctada margaritifera) Assesed by Mitochondrial DNA Analysis. under direction of Komar Sumantadinata, Dinar Tri Soelistyowati, and Achmad Sudradjat
The black-lipped pearl oyster (Pinctada margaritifera) is one of important species in pearl industry. The objective of this study was to determine of genetic diversity by mtDNA RFLP (Restriction Fragment Length Polymorphism) method from five populations. A total of 50 individual P. margaritifera were analyzed for genetic variation within a 750-base pair region of the mitochondrial DNA COI gene using RFLP analysis. The percentages of polymorphic loci and the genetic diversity were comparable among populations. Five populations of P. margaritifera formed three groups subpopulation with the values of Nei’s genetic distance were small among the Sumbawa and North Bali populations (0.017) and high between these and the South Sulawesi populations (0.142). The South Sulawesi populations possess uniqueness based on distribution haplotipe of BBCAA (60%) with the values of genetic diversity were lower (0.105) compared others populations (0.177-0.328). These case related to geographical condition, where characteristic of bay were semi-closed with high salinity.
margaritifera) Berdasarkan Analisis DNA Mitokondria. Dibimbing oleh Komar Sumantadinata, Dinar Tri Soelistyowati, dan Achmad Sudradjat
Tiram mutiara Pinctada margaritifera atau the black-lipped merupakan salah satu spesies penting yang digunakan dalam industri mutiara. Meskipun penggunaan stok benih hatchery meningkat, namun banyak perusahaan yang masih bergantung pada stok benih alam karena lebih efektif. Ketersediaan benih tiram mutiara alam terbatas, sementara eksploitasi semakin meningkat, hal ini akan mengakibatkan kelestarian populasi terancam, sehingga menuntut diperlukannya manajemen yang baik untuk tercapainya kelestarian populasi.
Salah satu manajemen untuk kelestarian populasi tiram mutiara ini adalah dengan budidaya. Kegiatan budidaya sebagai salah satu manajemen pelestarian populasi tidak terlepas dengan tersedianya stok induk unggul dan mampu menurunkan keturunan yang unggul pula. Salah satu upaya menghasilkan stok induk unggul yaitu dengan seleksi dan hibridisasi, yang dilakukan atas dasar informasi keragaman genetik.
Salah satu metode pengukuran keragaman genetik adalah analisis DNA mitokondria (mtDNA) dengan teknik PCR-RFLP. Penelitian ini bertujuan untuk memetakan keragaman genetik intrapopulasi dan perbedaan genetik interpopulasi dengan teknik mtDNA PCR–RFLP tiram mutiara Indonesia yang berasal dari Sumbawa, Bali Utara, Selat Sunda, Belitung, dan Sulawesi Selatan serta memetakan hubungan filogenetik dari lima populasi tiram mutiara tersebut.
Penelitian dilakukan dengan metode survei (pengambilan contoh) dan pengamatan di laboratorium. Pengambilan contoh dilakukan dari bulan Mei– Desember 2007. Lima puluh tiram mutiara dikoleksi dari lima lokasi yang mewakili sebaran tiram mutiara di Indonesia, yaitu Sumbawa, Bali Utara, Selat Sunda, Belitung, dan Sulawesi Selatan. Analisis laboratorium dilaksanakan dari bulan Februari–Juni 2008 di Laboratorium Rekayasa Genetik Loka Pemuliaan dan Teknologi Budidaya Air Tawar, Sukamandi. Analisis laboratorim meliputi ekstraksi DNA, amplifikasi mtDNA dengan menggunakan primer universal COI
forward: 5’-ATA ATG ATA GGA GGR TTT GG-3’ reverse: 5’-GCT CGT GTR CTA CRT CCA T-3’, dan digesti dengan enzim restriksi (FokI, HaeIII, NlaIV, DpnII, Eco0190I).
Hasil penelitian menunjukkan amplifikasi daerah COI mtDNA menghasilkan fragmen DNA berukuran sekitar 750 pb dan ditemukan pada semua populasi contoh tiram mutiara. Keragaman jumlah situs dan ukuran fragmen restriksi (RFLP) yang diperoleh dari hasil digesti mtDNA dengan lima enzim adalah 13 tipe restriksi (genotipe) yaitu FokI dengan empat tipe restriksi (A, B, C, D), HaeIII dengan tiga tipe restriksi (A, B, C), NlaIV dengan empat tipe restriksi (A, B, C, D), sedangkan DpnII dan Eco0190I menghasilkan masing-masing satu tipe restriksi (A) yang monomorfik, serta 34 fragmen restriksi. Analisis composite
berkisar antara 0,000−0,0016. Nilai tersebut lebih kecil dibandingkan dengan nilai
© Hak Cipta milik IPB, tahun 2008 Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau karya tulisan ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian,
penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB. Dilarang mengumumkan atau memperbanyak sebagian atau seluruh karya tulis dalam
BERDASARKAN ANALISIS DNA MITOKONDRIA
RINI SUSILOWATI
TESIS
sebagai salah satu syarat untuk memperoleh
gelar Magister Sains pada Program Studi Ilmu Perairan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
Halaman Pengesahan
Judul : KERAGAMAN GENETIK POPULASI TIRAM MUTIARA (Pinctada margaritifera) BERDASARKAN ANALISIS
DNA MITOKONDRIA
Nama : RINI SUSILOWATI
NRP : C151060151
Program Studi : ILMU PERAIRAN
Disetujui,
Komisi Pembimbing
Prof. Dr. Ir. Komar Sumantadinata, MSc Ketua
Dr. Ir. Dinar Tri Soelistyowati, DEA Prof. Riset. Dr. Achmad Sudradjat
Anggota Anggota
Diketahui,
Ketua Program Studi Ilmu Perairan Dekan Sekolah Pascasarjana IPB
Prof. Dr. Ir. Enang Harris, MS Prof. Dr. Ir. Khairil A. Notodiputro, MS
Bogor, Agustus 2008
RINI SUSILOWATI
PRAKATA
Puji syukur penulis panjatkan kehadirat Allah SWT atas kehendak-Nya
sehingga tesis dengan judul “Keragaman Genetik Populasi Tiram Mutiara
(Pinctada margaritifera) Berdasarkan Analisis DNA Mitokondria” ini dapat
penulis selesaikan.
Pada kesempatan ini, penulis ingin menyampaikan penghargaan dan ucapan
terima kasih yang sebesar-besarnya kepada Bapak Prof. Dr. Ir. Komar
Sumantadinata M.Sc., selaku ketua komisi pembimbing, Ibu Dr. Ir. Dinar Tri
Soelistyowati, DEA selaku anggota komisi pembimbing dan Prof. Riset. Dr.
Achmad Sudradjat selaku anggota komisi pembimbing yang telah memberikan
kesempatan untuk ikut dalam kegiatan penelitian, atas keikhlasan dan kesabaran
memberikan bimbingan, nasehat, arahan, dan dorongan semangat dalam
menyelesaikan tesis ini; Prof. Dr. Ir. Enang Harris, MS selaku Ketua Program
Studi Ilmu Perairan atas bimbingan selama penulis menjadi mahasiswa Program
Studi Ilmu Perairan, SPs IPB; Ibu Ir. Retna Utami, M.Sc dan Dr. Ir. Imron, M.Si
atas kesempatan yang diberikan kepada penulis untuk menggunakan fasilitas di
Laboratorium Rekayasa Genetik, Loka Riset Pemuliaan dan Teknologi Budidaya
Air Tawar, Sukamandi; Asosiasi Budidaya Mutiara Indonesia (ASBUMI) yang
telah membantu dalam penyediaan data untuk penelitian ini; Ayahanda Surono,
Ibunda Sutarti, Dek Hendry Tiyas TDR atas dukungan, do’a, dan nasehat selama
ini; ‘sam’ Ahmad Zahid dan rekan mahasiswa AIR ’06 atas dukungan, kritik,
saran dan kebersamaan selama ini; dan seluruh pihak yang telah membantu
penulis dalam menyelesaikan tesis ini.
Akhir kata, semoga tesis ini dapat bermanfaat dalam rangka pengembangan
RIWAYAT HIDUP
Penulis dengan nama lengkap Rini Susilowati dilahirkan di Madiun pada
tanggal 6 Mei 1981 dari pasangan Bapak Surono dan Ibu Sutarti. Pendidikan
formal ditempuh dari SD, SMP, dan SMU sejak tahun 1987–1999 di Ngawi.
Pendidikan Program Diploma (D3) diselesaikan tahun 2002 di Universitas
Airlangga pada Program Studi Budidaya Perikanan, Program Sarjana (S1)
diselesaikan pada tahun 2006 di Universitas Diponegoro pada Program Studi
Manajemen Sumberdaya Perikanan, dan pada tahun 2006 melanjutkan studi
Program Magister Sains (S2) pada Program Studi Ilmu Perairan Sekolah
Pascasarjana, Institut Pertanian Bogor.
Dalam rangka penyelesaian studi di Sekolah Pascasarjana IPB, penulis
melakukan penelitian yang berjudul “Keragaman Genetik Populasi Tiram
Mutiara (Pinctada margaritifera) Berdasarkan Analisis DNA Mitokondria”. Sebagian hasil penelitian ini telah disampaikan pada Simposium Nasional
Bioteknologi Akuakultur II pada tanggal 14 Agustus 2008 dengan judul
Karakteristik Genetika Tiram Mutiara (Pinctada margaritifera) Terkait dengan
DAFTAR ISI
2.2 Polimorfisme Genetik Tiram Mutiara... 7
2.2.1 Keragaman Genetik mtDNA... 8
2.2.2 Metode Analisis PCR – RFLP ... 11
2.3 Parameter Kualitas Air yang Berhubungan dengan Kelangsungan HidupTiram Mutiara ... 12
2.4 Perairan Pulau Panjang, Selat Alas ... 14
2.5 Teluk Pegametan, Bali ... 15
2.6 Perairan Pulau Handeuleum, Selat Sunda... 15
2.7 Perairan Selat Ru, Belitung ... 16
2.8 Perairan Teluk Awerange, Sulawesi Selatan ... 17
BAHAN DAN METODE PENELITAN... 19
3.1 Waktu dan Lokasi Pengambilan Contoh... 19
3.2 Metode ... 19
3.2.1 Metode Pengambilan Contoh... 19
3.2.2 Analisis DNA Mitokondria ... 19
3.3 Analisis Keragaman Genetik mtDNA... 21
HASIL DAN PEMBAHASAN... 23
4.1 Hasil ... 23
4.1.1 Amplifikasi Daerah Cytochrome Oxidase Subunit I (COI )... 23
4.1.2 Pola Restriksi Fragmen mtDNA Teramplifikasi... 23
4.1.3 Identifikasi Fragmen Restriksi (RFLP)... 26
4.1.4 Keragaman Genetik Intrapopulasi... 27
4.1.5 Perbedaan Genetik Interpopulasi ... 29
4.2 Pembahasan... 30
4.2.1 Keragaman Haplotipe ... 30
4.2.2 Filogenetis ... 33
4.2.3 Apikasi pada Budidaya Tiram Mutiara ... 36
KESIMPULAN DAN SARAN ... 37
DAFTAR TABEL
Tabel
1. Tipe ukuran fragmen digesti mtDNA daerah COI dengan
lima enzim restriksi (FokI, HaeIII, NlaIV, DpnII dan Eco0190I) ... 24
2. Distribusi genotipe (tipe restriksi) pada lima populasi tiram mutiara... 27
3. Distribusi frekuensi dan komposisi haplotipe pada lima populasi
DAFTAR GAMBAR
Gambar
1. Tiram mutiara (Pinctada margaritifera) ... 4
2. Siklus hidup tiram mutiara ... 6
3. Peta distribusi penyebaran tiram mutiara ... 7
4. Struktur DNA mitokondria ... 10
5. Fragmen mtDNA teramplifikasi dengan primer universal COI ... 23
6. Digesti mtDNA dengan FokI (4 tipe restriksi: A, B, C, D) ... 24
7. Digesti mtDNA dengan HaeIII(4 tipe restriksi: A, B, C) ... 25
8. Digesti mtDNA dengan NlaIV (4 tipe restriksi: A, B, C, D) ... 25
9. Digesti mtDNA dengan DpnII (1 tipe restriksi: A)... 26
10. Digesti mtDNA dengan Eco0190I(1 tipe restriksi: A)... 26
11. Dendrogram hubungan kekerabatan (filogeni) lima populasi tiram mutiara (Pinctada margaritifera)... 29
DAFTAR LAMPIRAN
Lampiran
1. Nama-nama Perusahaan Mutiara Indonesia ... 44
2. Jenis-jenis Tiram Mutiara ... 45
3. Lokasi Pengambilan Contoh Tiram Mutiara (Pinctada margaritifera) . 46
4. Nama enzim restriksi dan situs digestinya ... 47
5. Analisis deskriptif pada lima populasi tiram mutiara
(Pinctada margaritifera)... 48
6. Distribusi haplotipe pada lima populasi tiram mutiara (Pinctada
margaritifera)... 53 7. Analisis keseimbangan Hardy-Weinberg ... 54
8. Analisis jarak genetik (genetic distance) pada lima populasi tiram
mutiara (Pinctada margaritifera)... 56 9. Parameter kualitas air di lima lokasi pengambilan sampel dan kondisi perairan untuk kelangsungan hidup tiram mutiara
I. PENDAHULUAN
1.1Latar Belakang
Tiram mutiara Pinctada margaritifera atau the black-lipped merupakan
salah satu spesies penting yang digunakan dalam industri mutiara (Smith et al.
2003). Budidaya tiram mutiara di Indonesia pertama kali dilakukan pada tahun
1928 di Pulau Buton, Sulawesi Tenggara dan menghasilkan mutiara south sea
pearl pertama di dunia. Sampai tahun 2002 perusahaan mutiara yang terdaftar
sebagai anggota ASBUMI (Asosiasi Budidaya Mutiara Indonesia) sekitar 37
perusahaan yang tersebar di seluruh Indonesia (Poernomo 2002). Nama-nama dan
alamat perusahaan disajikan pada Lampiran 1.
Meskipun penggunaan stok benih hatchery meningkat, namun banyak
perusahaan yang masih bergantung pada stok benih alam karena lebih efektif
(Benzie et al. 2003). Ketersediaan benih tiram mutiara alam terbatas, sementara
eksploitasi semakin meningkat, hal ini akan mengakibatkan kelestarian populasi
terancam, sehingga menuntut diperlukannya manajemen yang baik untuk
tercapainya kelestarian populasi.
Salah satu manajemen untuk kelestarian populasi tiram mutiara ini adalah
dengan budidaya (Haig dan Nordstrom 1991). Kegiatan budidaya sebagai salah
satu manajemen pelestarian populasi tidak terlepas dengan tersedianya stok induk
yang unggul dan mampu menurunkan keturunan yang unggul pula (Alfsen 1987).
Salah satu upaya menghasilkan stok induk unggul yaitu dengan seleksi dan
hibridisasi yang dilakukan atas dasar informasi keragaman genetik.
Menurut Masyud (1992), kemampuan organisme beradaptasi terhadap
perubahan lingkungan alam ditentukan oleh keragaman genetiknya, keragaman
genetik dipandang sebagai bahan dasar bagi evolusi yang menyebabkan populasi
dapat beradaptasi terhadap perubahan-perubahan lingkungan. Sumantadinata
(1982) menyatakan bahwa evaluasi keragaman genetik penting dilakukan sebagai
dasar pengetahuan untuk pengembangan kegiatan budidaya dan manajemen
sumberdaya perikanan.
Beberapa penelitian terkait dengan genetik tiram mutiara populasi Indonesia
populasi Sumbawa dan Lombok (Widowati et al. 2007). Penelitian keragaman
genetik juga telah banyak dilakukan pada beberapa populasi dari genus Pinctada
diantaranya yaitu populasi P. albina, P. maculata, P. radiata (Haond et al. 2002),
P. martensii (Hosoi et al. 2004), P. imbricata (Yu et al. 2006).
Keragaman genetik berdasarkan distribusi geografis beberapa spesies tiram
mutiara populasi alam telah banyak dilaporkan di beberapa negara, misalnya
populasi alam P. margaritifera yang terpisah ratusan kilometer di Polynesia
Perancis menunjukkan perbedaan pada keragaman alelnya (Blanc dan Durand
1989). Hasil yang sama juga dilaporkan pada spesies P. margaritifera populasi
Jepang (Durand et al. 1993) dan Australia (Benzie dan Ballment 1994). Penelitian
yang dilakukan Evans et al. (2006) dan Smith et al. (2003) yang menggunakan
penanda microsatellite untuk P. maxima populasi Australia dan Indonesia, serta
penelitian genetik P. imbricata oleh Masaoka dan Kobayashi (2005).
Beberapa metode dapat diterapkan untuk memetakan struktur genetik, antara
lain metode meristik dan morfometrik, analisis polimorfisme protein dan
polimorfisme DNA. Secara umum metode polimorfisme DNA dianggap memiliki
tingkat akurasi yang tinggi dibandingkan dengan metode lain karena tidak
terpengaruh oleh perubahan lingkungan dan strukturnya dibedakan oleh urutan
basa yang khas dalam DNA. Analisis polimorfisme DNA dapat dilakukan dengan
berbagai macam teknik yaitu DNA mitokondria RFLP, DNA RAPD, DNA
microsatellite dan lain-lain.
1.2 Perumusan dan Pendekatan Masalah
Tiram mutiara (P. margaritifera) bersifat sedenter, namun populasinya
memiliki distribusi yang luas dengan kelimpahan yang tidak merata, diperkirakan
terdapat keragaman genetik populasi pada tiap unit populasinya. Oleh karena itu,
pengetahuan tentang keragaman genetik tiram mutiara ini perlu dipahami untuk
membantu dalam penentuan manajemen konservasi dan budidaya populasi ini.
Peranan keragaman genetik dalam budidaya merupakan salah satu indikator
dalam penentuan potensi genetik untuk seleksi dan hibridisasi dalam menentukan
populasi stok unggul. Populasi stok unggul ini diperlukan dalam manajemen stok
induk yang diharapkan dapat menghasilkan benih unggul untuk kelangsungan
informasi mengenai potensi genetik tiram mutiara lokal yang ada. Informasi
keragaman genetik dapat diperoleh melalui pemetaan DNA dan menganalisis
keragaman genetiknya.
Salah satu metode pengukuran keragaman genetik adalah analisis
mitokondria DNA (mtDNA) dengan teknik PCR-RFLP. Keunggulan metode ini,
antara lain pewarisan mtDNA berlangsung dari induk betina (maternal
inheritance) kepada keturunannya melalui gamet betina, dengan demikian pada
setiap generasi yang berasal dari induk betina yang sama akan memiliki pola
genetik yang sama, mtDNA mempunyai laju polimorfisme yang tinggi dengan
laju evolusinya sekitar 5−10 kali lipat dari DNA inti, bersifat klonal yaitu tidak
mengalami segregasi dan rekombinasi, mempunyai copy number yang tinggi
(Anderson et al. 1981).
Penelitian tentang polimorfisme mtDNA tiram mutiara menjadi tahap awal
dalam program pemuliaan yang sasarannya adalah untuk menentukan potensi
genetik tiram mutiara yang unggul terkait dengan pola adaptasi atau distribusi
penyebarannya. Informasi tersebut selanjutnya dapat digunakan dalam
pengelolaan sumber genetik tiram yang berkelanjutan, baik untuk pengembangan
produksi maupun konservasi.
1.3Tujuan dan Manfaat Penelitian
Penelitian ini bertujuan untuk memetakan keragaman genetik intrapopulasi
dan perbedaan genetik interpopulasi dengan teknik mtDNA PCR–RFLP tiram
mutiara Indonesia yang berasal dari Sumbawa, Bali Utara, Selat Sunda, Belitung,
dan Sulawesi Selatan serta memetakan hubungan filogenetik dari lima populasi
tiram mutiara tersebut.
Informasi mengenai keragaman genetik intrapopulasi dan perbedaan genetik
interpopulasi serta jarak genetik (genetic distance) tiram mutiara tersebut
diharapkan dapat dijadikan acuan dalam program pemuliaan yang sasarannya
adalah menghasilkan penyediaan induk tiram mutiara unggul yang
II. TINJAUAN PUSTAKA
2.1 Tiram Mutiara (Pinctada margaritifera)
2.1.1 Aspek Biologi Reproduksi
Tiram mutiara merupakan hewan bertubuh lunak atau moluska yang hidup
di laut, tubuhnya dilindungi oleh sepasang cangkang yang tipis dan keras,
termasuk dalam kelas Bivalva dan famili Pteriidae. Menurut Temkin (2006), nama
filum Moluska berasal dari bahasa latin molluscus yang berarti lunak merupakan
ilustrasi sinonim dari mollusc menjadi mollusca.
Pinctada margaritifera disebut juga “Black-lipped Pearl Oyster,”
merupakan salah satu spesies dari genus Pinctada yang dikenal dapat
menghasilkan mutiara berukuran besar, warnanya indah dan berkualitas tinggi.
Tiram mutiara (P. margaritifera) terlihat pada Gambar 1 dan beberapa jenis tiram
mutiara lainnya disajikan pada Lampiran 2. Klasifikasi tiram mutiara menurut
Temkin (2006) adalah sebagai berikut:
Filum : Mollusca
Kelas : Pelecypoda
Sub Kelas : Lamellibranchiata
Ordo : Anycomyaria
Famili : Pteriidae
Genus : Pinctada
Spesies : Pinctada margaritifera
Moluska diketahui menunjukkan variasi dalam kebiasaan siklus
reproduksinya, hal ini tergantung adanya sinyal dari lingkungannya.
Gametogenesis yang singkat dimulai sesudah pertumbuhan dan pematangan
gonad. Pematangan gamet dipengaruhi beberapa faktor eksogen seperti suhu,
periode bulan, kedalaman, faktor mekanis, kelimpahan dan ketersediaan pakan,
serta intensitas cahaya dan adanya faktor endogen yaitu genetik dan hormon
(Mackie 1984).
Menurut Dharmaraj et al. (1987) proses reproduksi diawali dengan fertilisasi
eksternal yang terjadi dalam air, selanjutnya telur dibuahi oleh sperma. Selama
proses pemijahan biasanya induk jantan memijah terlebih dahulu, kemudian
sekitar 45 menit kemudian diikuti induk betina mengeluarkan sel-sel telur. Hasil
pengamatan Saoruddin (2004) yang dilakukan di laboratorium menunjukkan
bahwa induk betina akan mengeluarkan telurnya 30–35 menit kemudian setelah
induk jantan memijah. Proses pembuahan segera terjadi setelah induk melakukan
pemijahan.
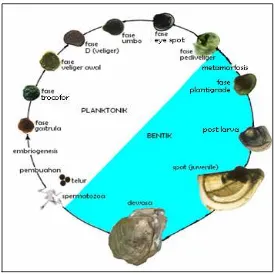
Menurut Haws dan Ellis (2000) tahapan perubahan siklus hidup pada
Gambar 2 dapat dijelaskan sebagai berikut: pada awal perubahan terjadi
penonjolan polar, lalu membentuk polar lube II yang merupakan awal proses
pembelahan sel. Proses pembelahan sel terjadi 40 menit setelah pembuahan. Lima
menit kemudian sel mulai membelah menjadi dua sel, 13 menit berikutnya sel
membelah menjadi empat sel. Pembelahan berikutnya menjadi 8, 16, 32 sel, dan
selanjutnya menjadi multisel atau fase morula setelah 2,5 jam. Fase blastula
dicapai setelah larva berumur 3,5 jam dengan gerakannya aktif berputar-putar.
Pada fase gastrula yang berumur tujuh jam bersifat fotonegatif dan bergerak
dengan silia.
Tahap berikutnya adalah fase trocofor yang dicapai beberapa menit setelah
silia menghilang. Terdapat flagela tunggal pada bagian anterior yang berfungsi
sebagai alat gerak. Beberapa jam kemudian trocofor berkembang menjadi veliger
(larva bentuk D). Fase ini dicapai setelah larva berumur 18–20 jam dan berukuran
70x80 µm, tumbuh organ mulut dan pencernaan, bersifat fotopositif sehingga
Gambar 2. Siklus hidup tiram mutiara (Haws dan Ellis 2000)
Fase umbo dicapai setelah larva berumur 12–14 hari dengan ukuran 130x
135µm yang ditandai dengan adanya tonjolan (umbo) pada bagian dorsal. Fase
bintik mata (eye spot) terjadi pada hari ke-16–17 dengan ukuran 200x190 µm.
Posisi eye spot berada di sebelah bawah primordia kaki. Fase pediveliger (umbo
akhir) yang berumur 18–20 hari dengan ukuran 210x200 µm. Larva ini mulai
mencari tempat untuk menempel atau menetap. Fase plantigrade merupakan fase
transisi atau fase akhir kehidupan planktonis larva, terjadi pada hari ke 20–22.
Ukuran larva plantigrade sekitar 130x210 µm yang ditandai dengan tumbuhnya
cangkang baru di sepanjang periphery dan memproduksi benang-benang bisus
untuk menempelkan diri pada substrat, selanjutnya akan berubah menjadi bentuk
spat atau bentuk tiram mutiara dewasa, hanya garis-garis pertumbuhannya masih
terlihat jelas atau transparan.
2.1.2 Distribusi Geografis
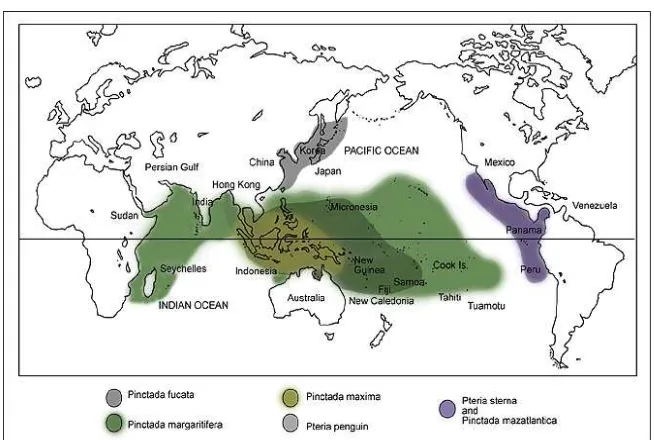
Menurut Martin (2004), penyebaran tiram mutiara terbatas pada ekosistem
terumbu karang. Hampir seluruh perairan laut di Indo-Pasifik bahkan hingga ke
mutiara. Populasi P. margaritifera di Indonesia banyak tersebar di wilayah
Indonesia Barat dan Tengah, seperti Laut Jawa, Sumbawa dan Kalimantan dan di
wilayah Indonesia Timur seperti Sulawesi, gugusan Laut Arafura dan Irian Jaya
(Gambar 3).
Gambar 3. Peta distribusi penyebaran tiram mutiara (Martin 2004)
2.2 Polimorfisme Genetik Tiram Mutiara
Polimorfisme tiram mutiara telah banyak dipelajari dan diteliti untuk
kepentingan konservasi dan pemuliabiakannya. Beberapa penelitian yang terkait
antara lain Haond et al. (2002) menganalisis heterosigositas P. margaritifera
populasi Polynesia Perancis dengan metode Direct Amplification of Length
Polymorphism (DALP) dan Exon Primed Intron Crossing (EPIC). Hasil analisis
menunjukkan nilai heterosigositas berkisar 0,44–0,69 (metode DALP) dan 0,29–
0,31 (metode EPIC). Metode DALP dan EPIC juga digunakan Haond et al. (2003)
untuk mengevaluasi keragaman genetik pada spat tiram mutiara P. margaritifera
cumingi pada populasi alam dan budidaya di Polynesia Perancis. Hasilnya
menunjukkan bahwa heterosigositas populasi berkisar antara 0,275–0,366 pada
sampel budidaya dan 0,294–0,366 pada sampel alam.
Karakteristik loci P. maxima dari Australia bagian utara menggunakan
microsatellite menunjukkan delapan loci polimorfik, jumlah alel per lokus 14–68
(2006) menganalisis karakteristik loci P. maxima dari Australia dan Indonesia
menggunakan microsatellite menunjukkan 6–17 alel dengan nilai
heterosigositasnya 0,172–0,813.
Benzie et al. (2003) menganalisis keragaman haplotipe P. maxima dari
Australia dan Indonesia yaitu populasi Madura dan Sumbawa menggunakan
metode mtDNA menghasilkan delapan haplotipe (62% dari seluruh haplotipe)
ditemukan di Australia dan tiga haplotipe (38%) ditemukan di Indonesia.
Pengamatan keragaman genetik pada P. maxima di kawasan Indonesia-Australia
Archipelago (IAA) dengan menggunakan metode microsatellite menghasilkan
nilai Fst sebesar 0,027 yang mengindikasikan bahwa sejarah biogeografi atau
faktor oseanografi terkait pada pembentukan struktur populasi genetik di kawasan
tersebut (Lind et al. 2007).
2.2.1 Keragaman Genetik mtDNA
Mitokondria adalah organel sel eukariot yang berfungsi sebagai organ
respirasi pembangkit energi dengan menghasilkan adenosin triphosphat (ATP).
Mitokondria ditemukan dalam jumlah banyak pada sel yang aktivitas
metabolismenya tinggi yaitu sel-sel kontraktil seperti sperma pada bagian
ekornya, sel otot jantung, dan sel yang aktif membelah seperti epitelium, akar
rambut, dan epidermis kulit. Mitokondria memiliki perangkat genetik sendiri yang
disebut DNA mitokondria (mtDNA), terletak pada matriks semi cair di bagian
paling dalam mitokondria. Satu mitokondria dapat mengandung puluhan mtDNA
(Toha 2001).
mtDNA yang berukuran relatif kecil dan terdapat dalam jumlah berlimpah
serta bersifat klonal dan maternal inheretance memungkinkan analisis mtDNA ini
potensial untuk mengetahui hubungan maternal antar individu, mempelajari
antropologi, serta biologi evolusi berbagai makhluk hidup. mtDNA sangat
potensial digunakan sebagai sistem untuk pengamatan hubungan genetik antar
spesies maupun di dalam spesies. Peranan mtDNA dalam studi keragaman genetik
dan biologi populasi pada hewan cukup besar, karena mtDNA memiliki derajat
polimorfisme yang tinggi (Toha 2001).
Teknik-teknik eksplorasi dalam biologi molekuler dapat dimanfaatkan untuk
yaitu dengan menggunakan teknik RFLP (Restriction Fragment Length
Polymorphism) dengan melihat variasi jumlah dan ukuran fragmen DNA yang
dihasilkan dari hasil digesti enzim restriksi pada genom mitokondria suatu
organisme. Selanjutnya tiap fragmen restriksi dapat ditelaah secara lebih
terperinci dengan mengetahui urutan basa-basa nukleotida penyusunnya dengan
melalui teknik sekuensing.
Umumnya genom mitokondria berbentuk sirkular dan berutas ganda yang
terdiri dari utas berat (heavy strand) dan utas ringan (light strand) (Gambar 4).
Berdasarkan jenis gennya, genom mitokondria ini dapat dibagi menjadi dua
bagian yaitu daerah penyandi (coding region) dan daerah bukan penyandi
(noncoding region). Daerah penyandi terdiri dari 37 gen, yaitu 13 gen penyandi
protein, dua gen penyandi rRNA dan 22 gen penyandi tRNA. Gen-gen tersebut
tersebar pada kedua utas mtDNA. Utas ringan dari mtDNA hewan umumnya
terdiri dari satu gen penyandi protein yaitu NADH Dehydrogenase 6 (ND 6) dan
delapan gen penyandi tRNA yaitu Glutamid acid (tRNA Glu), Proline (tRNA Pro),
Serine (tRNA Ser), Tyrosine (tRNA Tyr), Cysteine (tRNA Cys), Asparagine (tRNA Asn
), Alanine (tRNA Ala) dan Glutamine (tRNA Gln), sedangkan sisanya terdapat
pada utas berat, yaitu dua gen penyandi Dehydrogenase (ND): ND1, ND2, ND3,
ND4, ND5, ND4L; Cytochrome c Oxidase (CO): COI, COII, COIII; Cytochrome
b, ATPase 6 dan Leucine (tRNA Leu), Isoleucine (tRNA Ile), Methionine (tRNA Met
), Tryptophen (tRNA Trp), Aspartic acid (tRNA Asp), Lysine (tRNA Ser), Threonine
(tRNA Thr). Daerah bukan penyandi hanya terdiri dari daerah kontrol (control
region) yang memegang peranan penting dalam proses transkripsi dan replikasi
genom mitokondria (Anderson et al. 1981).
Berdasarkan hasil penelitian, diketahui bahwa bagian-bagian dari genom
mitokondria memiliki laju evolusi yang berbeda-beda. Beberapa bagian
mengalami perubahan yang sangat lambat (conserved), sedangkan bagian yang
lainnya mengalami perubahan yang sangat cepat, sehingga menjadi bagian yang
paling bervariasi (hypervariable) (Roderick 1996). Dikatakan pula bahwa
umumnya gen-gen yang terkonservasi dengan baik (berevolusi lambat) dapat
dijadikan dasar penelusuran asal muasal atau filogeni, sedangkan gen-gen yang
identifikasi galur-galur baru. Oleh sebab itu, maka analisis keragaman genetik
dapat dilakukan pada sebagian gen dari genom mitokondria hewan.
Gambar 4. Struktur DNA mitokondria (Lemire 2005) (ket: : penyandi protein, : penyandi rRNA, : penyandi tRNA)
Analisis keragaman genetik dengan mtDNA pada bivalva telah banyak
dilakukan diantaranya penelitian yang dilakukan oleh Smith et al. (2004) yang
menggunakan mtDNA dalam analisis heterosigositas pada Mytilidae di Selandia
Baru. Milbury et al. (2004) juga menggunakan metode ini untuk mengamati
perbaikan genetik Crassostrea virginica. Menurut Pie et al. (2006) menyatakan
metode ini juga efektif dalam membedakan spesies bivalva (Crassostrea
brasiliana, C. rhizophorae, dan C. gigas) yang dibudidayakan.
Beberapa penelitian pada genus Pinctada yang menggunakan metode
mtDNA telah banyak dilaporkan diantaranya yaitu Haond et al. (2003)
menggunakan dua gen mitokondria yaitu COI dan 12S rRNA dalam penelitiannya
yang mengamati tiram mutiara P. margaritifera populasi Pasifik Tengah.
Masaoka dan Kobayashi (2005) menggunakan mitokondria daerah 16S rRNA
pada spesies P. imbricata, P. martensii dan P. fucata populasi Jepang, Cina dan
COI untuk mengamati keragaman genetik populasi P. maxima Indonesia dan
Australia.
2.2.2 Metode Analisis PCR – RFLP
Prosedur awal dalam analisis mtDNA adalah memecah sel untuk
mengesktrak mtDNA. Tahap berikutnya adalah amplifikasi yaitu proses
perbanyakan (sintesis) sekuen mtDNA. Amplifikasi dapat dilakukan secara in
vitro dengan menggunakan teknik PCR (Polymerase Chain Reaction) atau in vivo
melalui teknik kloning pada sel hidup. Jaringan contoh yang digunakan dapat
berupa hati, otot, darah, sel kultur atau jaringan lain, baik dalam kondisi segar,
telah difiksasi ataupun beku (Erlich 1989).
Pada prinsipnya teknik PCR adalah proses enzimatis untuk memperbanyak
DNA dengan memanfaatkan sifat replikasi DNA dan perubahan fisik DNA
terhadap suhu. Proses ini dibantu dengan enzim Taq-DNA polymerase pada tahap
ekstensi polinukleotida primer (Zyskind dan Bernstein 1993). Replikasi terjadi
jika terdapat untai tunggal DNA yang bertindak sebagai cetakan (template) dan
basa nukleotida (dNTP). Enzim DNA polimerase membantu dalam pembentukan
DNA untai lainnya. Primer adalah potongan pendek DNA terdiri dari 20–30
nukleotida yang melakukan hibridisasi pita secara berpasangan dengan sekuen
tertentu yang mengapit (flanking) daerah DNA target amplifikasi pada tiap pita
DNA (Erlich 1989).
Pada kondisi normal, DNA berada dalam keadaan heliks ganda. Heliks ini
terdiri atas dua utas tunggal DNA yang saling berpasangan dan terikat
non-kovalen oleh ikatan hidrogen. Pada awal proses PCR, utas ganda ini didenaturasi
pada suhu tinggi hingga menjadi utas tunggal. Selanjutnya, suhu diturunkan untuk
memungkinkan primer utas tunggal menempel pada daerah target. DNA
polimerase kemudian digunakan untuk pemanjangan sekuen dengan bantuan
suplai empat basa nukleotida (dNTP; Adenin, Guanin, Timin, dan Sitosin) dan
buffer. Dengan cara ini dapat menghasilkan duplikat daerah target dan siklus ini
biasanya berlangsung 20–40 kali (Erlich 1989).
Kelebihan teknik PCR adalah proses isolasi cepat, jumlah sekuen DNA yang
dihasilkan dapat mencapai 300.000 kopi, sangat sensitif dalam mendeteksi sekuen
itu, suhu tinggi dalam sintesa DNA (75 °C) dapat meningkatkan ketelitian
sehingga meminimumkan ekstensi primer yang tidak sesuai dengan template
(Zyskind dan Bernstein 1993). Dalam prosedur PCR ini, struktur sekunder dari
template DNA yang dapat menghalangi aktivitas enzim polimerase juga akan
direduksi melalui denaturasi sekuen pada suhu tinggi, namun demikian beberapa
faktor harus diperhatikan supaya pita-pita yang dihasilkan baik dan utuh, antara
lain konsentrasi DNA, ukuran dan komposisi basa primer dan suhu hibridisasi
(Erlich 1989).
Metode RFLP merupakan suatu cara yang digunakan untuk mengetahui
perbedaan profil ukuran fragmen DNA dari individu yang berbeda, dengan
menggunakan enzim restriksi untuk memotong sekuen mtDNA teramplifikasi.
Hansen et al. (1997) menyatakan bahwa teknik RFLP menggunakan suatu enzim
restriksi endonuklease yang dapat memotong fragmen besar DNA pada situs
restriksi tertentu menjadi fragmen yang lebih kecil. Visualisasi hasil pemotongan
dapat menunjukkan pola fragmen yang khas tergantung jenis DNA dan enzim
yang digunakan. Organisme yang berbeda memiliki perbedaan urutan DNA,
sehingga teknik RFLP dapat membedakan jenis organisme pada tingkat spesies
atau strain dan telah banyak digunakan diantaranya pada moluska. Kombinasi
antara teknik PCR dan penggunaan enzim retriksi atau teknik sekuensing
nukleotida dapat menggambarkan karakter atau profil DNA individual yang
berguna untuk menjelaskan hubungan filogenetik populasional dalam takson.
2.3 Parameter Kualitas Air yang Berhubungan dengan Kelangsungan Hidup Tiram Mutiara
Kegiatan manajemen broodstock membutuhkan populasi calon induk yang
memiliki (viabilitas dan fertilitas) dan kebugaran potensial (potentialfitness) yang
bagus. Fitness terkait dengan peluang kelangsungan hidup (survival rate) atau
fekunditas dan kebugaran potensial (potential fitness) terkait dengan tingkat
polimorfisme (keragaman genetik) dan pola adaptasi melalui seleksi alam
terhadap perubahan lingkungan. Pola adaptasi terkait dengan kemampuan individu
menyediakan ragam genetik dalam mekanisme evolusi sehingga mampu bertahan
hidup (survive) dan terlibat dalam PRE (Populasi Reproduktif Efektif) serta
Faktor yang mempengaruhi fitness dalam populasiantara lain: pola breeding
yang dipengaruhi oleh Ne (populasi efektif), predasi atau eksploitasi terkait
dengan manajemen sistem budidaya dan lingkungan (geografi), seperti barier
geografi dan kondisi perairan.
Suhu adalah salah satu faktor yang sangat penting bagi kehidupan organisme
laut, karena mempengaruhi aktivitas metabolisme maupun perkembangbiakan
organisme tersebut. Suhu rata-rata organisme tiram mutiara dapat hidup adalah
25–28 °C (Tarwiyah, 2000). Pada suhu antara 26–29°C, tiram mutiara (P.
margaritifera) sangat aktif melakukan kegiatan metabolisme dan mampu tumbuh
dengan baik (Doroudi et al. 1999).
Salinitas air laut mempengaruhi penyebaran hewan bentos seperti bivalva,
karena organisme laut hanya dapat bertoleransi terhadap perubahan salinitas yang
kecil dan perlahan (Meade 1989). Menurut penelitian Doudori et al. (1999),
salinitas yang baik untuk pertumbuhan optimal tiram mutiara P. margaritifera
berkisar antara 27–32‰.
Oksigen terlarut umumnya menjadi faktor pembatas bagi kelangsungan
hidup organisme akuatik (Meade 1989). Hasil penelitian Dharmaraj et al. (1987)
tentang kebutuhan oksigen terlarut tiram mutiara P. fucata, menunjukkan bahwa
tiram berukuran 40–50 mm mengkonsumsi oksigen 1,339 µl/jam, ukuran 50–60
mm mengkonsumsi 1,650 µl/jam dan ukuran 60–70 mm mengkonsumsi 1,810
µl/jam.
Menurut Nayar dan Mahadevan (1987), pada prinsipnya habitat tiram
mutiara berada pada perairan dengan pH lebih tinggi dari 6,75; namun tiram
mutiara tidak bereproduksi bila pH lebih tinggi dari 9, pH air yang layak untuk
kehidupan tiram mutiara P. maxima berkisar antara 7,8–8,6; sedangkan pada pH
7,9–8,2 tiram mutiara dapat berkembang biak dan tumbuh dengan baik.
Faktor kecerahan suatu perairan berpengaruh terhadap kehidupan organisme
di dalamnya. Tinggi rendahnya tingkat kecerahan sangat dipengaruhi intensitas
cahaya matahari yang dapat menembus kedalaman lapisan perairan. Tiram
mutiara hidup pada dasar perairan, sehingga kecerahan perairan yang dibutuhkan
tiram mutiara pada umumnya mencapai dasar perairan. Kedalaman perairan untuk
2.4 Pulau Panjang, Selat Alas
Perairan Pulau Panjang yang termasuk dalam perairan Selat Alas terletak di
antara pulau Lombok dan pulau Sumbawa Propinsi Nusa Tenggara Barat (NTB),
dengan luas perairan sekitar 20541.695 km2; lebar mulut selat bagian utara sekitar
20.372 km, sedangkan di bagian selatan sekitar 35.188 km (Wasilun dan Amin
1989). Kedalaman perairan Selat Alas berkisar antara 16–268 m (Anonim 1990)
dan rata-rata kedalaman < 100 m (Suharsono et al. 1995).
Suhu permukaan laut Selat Alas pada musim tenggara relatif lebih rendah
dari pada musim barat laut dan kedua musim peralihan di antaranya. Nilai
rata-rata suhu pada musim tenggara (Juni–Agustus) sekitar 26 °C, sedangkan pada
musim barat laut (Desember–Februari) dan musim peralihan dua (September–
November) nilai rata-rata suhu sekitar 28 °C dan pada musim peralihan satu
(Maret–Mei) nilai rata-rata suhunya sekitar 29 °C (Hartati et al. 2001).
Konsentrasi oksigen terlarut di perairan Selat Alas baik pada musim barat
laut, tenggara, dan kedua musim peralihan diantaranya cukup mendukung untuk
kehidupan biota perairan, yaitu berkisar antara 3,7–4,4 mg/L, sedangkan salinitas
permukaan laut Selat Alas berkisar antara 33–34‰ (Hartati et al. 2001).
Hasil pengamatan Tasywiruddin (1999), pengukuran karakteristik perairan
Selat Alas menunjukkan kelimpahan plankton relatif sama baik di perairan Selat
Alas bagian dalam maupun Selat Alas bagian luar. Kisaran rata-rata kelimpahan
plankton di perairan Selat Alas bagian dalam adalah 8.115–143.888 individu/m3,
sedangkan perairan Selat Alas bagian luar adalah 84.000–14.263 individu/m3.
Secara oseanografis kondisi perairan Selat Alas dipengaruhi oleh
perubahan-perubahan yang terjadi di perairan Laut Flores dan Samudera Hindia. Pada musim
barat (Desember–Februari), massa air perairan Selat Alas berasal dari Laut Jawa,
sedangkan pada musim timur (Mei/Juni–September) terjadi perubahan arah arus
dari utara ke selatan Laut Flores (Hartati et al. 2001).
Pada musim timur (Mei–September) terjadi proses air naik (up welling) di
sepanjang pantai selatan Jawa–Sumbawa (Wyrtki 1961). Di sekitar perairan
tersebut, suhu air permukaan biasanya turun sampai 25 °C, karena massa air yang
dingin dan kaya akan nutrien dari lapisan bawah terangkat ke atas, sehingga
menyatakan bahwa nilai kisaran rata-rata kekeruhan di perairan Selat Alas yaitu
8,18–11,73 NTU. Hasil pengamatan Mahrus (1996) pH pada perairan Selat Alas
berkisar antara 6,5–7,4.
2.5 Teluk Pegametan, Bali Utara
Teluk Pegametan merupakan bagian perairan Selat Bali, sehingga
menyebabkan Teluk Pegametan lebih cenderung dipengaruhi oleh massa air dari
Selat Bali yaitu dari Samudera Hindia. Perubahan yang dialami Teluk Pegametan
akan sama dengan perubahan yang dialami oleh Selat Bali, pada saat musim angin
timur terjadi proses umbalan air atau naiknya zat hara yang kaya akan nutrien
akibat desakan Arus Pantai Jawa (APJ) dan Arus Khatulistiwa Selatan (AKS).
Menurut penelitian Hanafi et al. (2004) menyatakan pada Teluk Pegamtan
suhu permukaan laut berkisar 25,1–28 °C yang didominasi oleh suhu 26,1–28 °C.
Dikatakan pula bahwa suhu perairan Teluk Pegametan terukur dari 0–100 m di
bawah permukaan air laut, nilai berkisar antara 20–27,5 °C.
Berdasarkan pengamatan Hanafi et al. (2004), sebaran fitoplankton mulai
terlihat pada bulan Mei dibagian barat Teluk Pegametan, kemudian mulai
meningkat baik kualitas maupun kuantitasnya berkisar antara 0,7–3 mg/m3 begitu
pula pada bulan-bulan selanjutnya. Dikatakan pula puncak performansi kuantitas
dan kualitas sebaran fitoplankton terjadi pada bulan Agustus dan September dan
pada bulan November dan Desember sudah tidak terlihat lagi, selanjutnya
dikatakan pula bahwa nilai salinitas di perairan Teluk Pegametan berkisar antara
31,5–33‰, nilai salinitas rendah terkonsentrasi pada kedalaman 10 m, salinitas
kurang dari 30‰ terdapat pada kedalaman 2–3 m dan pada kedalaman 70 m
salinitas di atas 30,5‰.
2.6 Pulau Handeuleum, Selat Sunda
Selat Sunda merupakan selat penghubung antara Laut Jawa dan Samudera
Hindia yang memiliki keunikan sifat oseanografis tersendiri karena ditempati oleh
dua massa air yaitu dari Laut jawa dan Samudera Hindia. Bagian utara selat lebih
sempit dan lebih dangkal dibandingkan bagian selatan, pada bagian tersempit
hanya sekitar 24 km lebarnya dengan kedalaman kurang dari 80 m. melalui
Samudera Hindia lemah. Karakteristik oseanografi selat ini di bawah pengaruh
Laut Jawa dan Samudera Hindia (Hendiarty 2003).
Pengamatan dengan sensor SeaWiFS (Sea-viewing Wide Field-of-view
Sensor) oleh Hendiarty (2003), memperlihatkan bahwa perairan Selat Sunda pada
musim angin timur (Juni–September) terjadi fenomena pergerakan massa air Laut
Jawa menuju Samudera Hindia melalui Selat Sunda yang dicirikan dengan
kandungan klorofil-a perairan sebesar 0,8 mg/m3, sementara pada musim angin
barat (November–Maret) perairan tersebut pada musim barat umumnya kurang
subur yang dicirikan dengan kandungan klorofil-a yang lebih rendah.
Suhu rata-rata bulanan permukaan laut Selat Sunda relatif stabil sepanjang
tahun berkisar antara 28–29 oC, dengan suhu maksimum ditemui pada bulan Mei
dan suhu minimum pada bulan Oktober (Wyrtki 1961). Kondisi temperatur
permukaan pada kedalaman satu meter berkisar antara 28,32–29,68 oC (Paryono
2004).
Salinitas permukaan di perairan Selat Sunda berkisar antara 32,5–33,6‰,
salinitas minimum ditemui pada bulan Januari dan salinitas maksimum pada bulan
Agustus (Wyrtki 1961). Menurut Paryono (2004) salinitas Selat Sunda pada
kedalaman 1 m, berkisar antara 33,28–33,49‰, sedangkan menurut
Romimohtarto (1983), salinitas permukaan di selat bagian utara biasanya lebih
rendah daripada di bagian selatan, salinitas Selat Sunda bervariasi dari 31,5–
33,5‰. Rendahnya salinitas permukaan di selat bagian utara disebabkan oleh
masuknya massa air dari laut Jawa ke Selat Sunda hampir sepanjang tahun.
Salinitas dekat dasar di selat bagian selatan juga lebih tinggi daripada di bagian
utara. Kecerahan 80 cm sementara pH terukur antara 6,5–8,5 (Anonim 2007).
2.7 Selat Ru, Belitung
Selat Ru merupakan salah satu selat yang berada di antara Pulau Ru dan
Pulau Nado, di bagian barat Pulau Belitung. Secara keseluruhan Selat Ru
termasuk dalam Selat Gaspar yang berada di antara Pulau Bangka dan Belitung.
Perairan di Selat Ru dipengaruhi oleh kondisi perairan di Laut Cina Selatan dan
Selat Gaspar.
Hasil pengamatan Wati (2004) menyatakan pada musim barat konsentrasi
mulai dari Riau (Pulau Singkep) sampai ke selatan Sumatera Pulau Bangka dan
Belitung. Dikatakan pula pada musim timur arus dari Laut Jawa memberikan
sumbangan terhadap kesuburan fitoplankton di Selat Karimata. Hasil penelitian
Sofian (2004) menunjukkan variasi suhu pada Selat Ru 26,3–28 °C, salinitas 30–
32,6; pH 7,6–8; Kecerahan pada kedalaman 5–10 m adalah 40–100%.
2.8 Teluk Awerange, Sulawesi Selatan
Teluk Awerange berada di wilayah Kecamatan Soppeng Riaja Kabupaten
Barru. Sebelah utara teluk berbatasan dengan Desa Batu Pute, terdapat pelabuhan
rakyat Awerange, pemukiman nelayan dan pantai pasir putih, sebelah timur
berbatasan dengan Desa Siddo, sebelah timur pesisir Teluk Awerange terdapat
pemukiman nelayan, tambak tradisional dan semi intensif serta hutan bakau,
sebelah selatan berbatasan dengan Desa Lawallu, sebelah barat teluk berbatasan
dengan Selat Makassar yang merupakan sumber pemasukan air laut (Rusman
2003).
Menurut Rusman (3003), secara umum perairan di Teluk Awerange pada
musim kemarau dan musim hujan tidak mengalami perbedaan yaitu berkisar
antara 25–26 °C. Pada perairan yang dangkal tidak terdapat lapisan termoklin
yang dapat menyebabkan perbedaan suhu yang ekstrim di setiap kedalaman
perairan. Selain itu Teluk Awerange merupakan perairan yang semi tertutup
sehingga suhu perairan teluk hampir merata. Kedalaman Teluk Awerange berkisar
antara 1–40 m, perbedaan ini disebabkan karena kontur dasar perairan yang tidak
merata. Kedalaman terbesar berada di sekitar mulut dan bagian tengah perairan
teluk.
Hasil penelitian Rusman (2003) pada perairan Teluk Awerange
menunjukkan rata-rata kecerahan perairan adalah 60–100%. Pada musim hujan
penurunan kecerahan sebesar 20% dari kecerahan perairan musim kemarau. Arus
permukaan yang terjadi di perairan Teluk Awerange umumnya disebabkan oleh
pasang surut dan angin yang bertiup di permukaan. Kondisi geografis Teluk
Awerange dengan mulut teluk yang sempit dan terumbu karang merupakan
penghalang arus yang berasal dari perairan Selat Makassar. Rusman (2003)
melaporkan bahwa sepanjang tahun pergerakan massa air di Selat Makassar
Maret massa air Laut Jawa bergerak ke arah timur sedangkan pada bulan Mei–
September massa air bergerak ke arah barat.
Penelitian Rusman (2003) menunjukkan beberapa parameter kimia di
perairan Teluk Awerange yaitu pH berkisar antara 7,5–8,4; oksigen terlarut
berkisar antara 5–8,7 mg/l, salinitas 34–35‰. Hasil pengamatannya, bentos di
perairan Teluk Awerange pada 20 titik pengambilan contoh diperoleh sebanyak
105 spesies, dengan kisaran kepadatan antara 33–497667 ind/m3. Hal ini
memberikan gambaran bahwa perairan Teluk Awerange sangat kaya dengan
III. BAHAN DAN METODE PENELITIAN
3.1 Waktu dan Lokasi Pengambilan Contoh
Penelitian dilakukan dengan metode survei (pengambilan contoh) dan
pengamatan di laboratorium. Pengambilan contoh dilakukan dari bulan Mei–
Desember 2007 dan analisis laboratorium dilaksanakan dari bulan Februari–Juni
2008. Tiram mutiara contoh dikoleksi dari lima lokasi yang mewakili sebaran
tiram mutiara P. margaritifera di Indonesia, yaitu Sumbawa, Bali Utara, Selat
Sunda, Belitung, dan Sulawesi Selatan. Peta pengambilan contoh dapat dilihat
pada Lampiran 3.
3.2 Metode
3.2.1 Metode Pengambilan Contoh
Pengambilan contoh tiram mutiara dilakukan dengan penyelaman ke dasar
perairan atau dari tempat budidaya tiram setempat. Contoh tiram dari Sumbawa
berasal dari perairan Pulau Panjang, Selat Alas, tiram mutiara Bali Utara berasal
dari Teluk Pegametan, Bali, tiram mutiara Selat Sunda berasal dari perairan Pulau
Handeuleum, Selat Sunda, tiram mutiara Belitung berasal dari perairan Selat Ru,
sedangkan tiram mutiara Sulawesi Selatan berasal dari Teluk Awerange, Barru.
Jumlah contoh tiram mutiara adalah 10 ekor untuk masing-masing lokasi dengan
ukuran sekitar 10–30 cm. Contoh tiram mutiara yang dikoleksi disimpan dalam
larutan alkohol 70−100% untuk keperluan analisis DNA di laboratorium. Analisis
DNA contoh tiram mutiara dilakukan di Laboratorium Rekayasa Genetik Loka
Pemuliaan dan Teknologi Budidaya Air Tawar, Sukamandi.
Data sekunder mengenai sifat fisik dan kimia perairan lokasi sampling yang
mempengaruhi kelangsungan hidup dan distribusi tiram mutiara, meliputi
kedalaman, suhu, salinitas, oksigen terlarut, dan produksi primer, dikumpulkan
dari berbagai literatur.
3.2.2 Analisis DNA mitokondria
Ekstraksi DNA
Ekstrak DNA yaitu melakukan tahapan penghancuran sel, eliminasi protein
menghaluskan potongan daging sebanyak 20 mg/contoh dengan scapel kemudian
dimasukkan ke dalam tabung 1,5 ml yang telah diisi 250 µl larutan sel lisis yang
terdiri 10 µl Tris pH 8; 100 mM EDTA pH 8; 2% SDS, lalu ditambahkan 5 µl
protein kinase dan inkubasi pada suhu 55 oC selama 4–24 jam. Tahap berikutnya
adalah tahap eliminasi protein dan RNA yaitu dengan menambahkan 250 µl
amonium asetat ke dalam larutan kemudian dihomogenkan (aduk kencang) selama
lima menit hingga tercampur rata. Diaduk perlahan selama 15 menit pada suhu
ruang, selanjutnya dimasukkan dalam suhu 4 oC selama 10 menit. Tahap
selanjutnya adalah tahap pengendapan protein dengan sentrifugasi pada kecepatan
13.000 rpm selama 10 menit. Larutan supernatant (lapisan cair) dipindahkan ke
dalam tabung baru dan ditambahkan etanol 100% sebanyak dua kali volume
supernatant, lalu disentrifugasi kembali pada kecepatan 13.000 rpm selama 5
menit kemudian etanol dibuang, lalu ditambahkan alkohol 70% sebanyak 600 µl
dan disentrifugasi pada kecepatan 13.000 selama 3 menit. Lapisan etanol dibuang
dan dikeringanginkan selama 30 menit, ditambahkan 50 µl buffer T10E0.1 dan
inkubasi pada suhu 65 oC selama 1 jam (Nguyen et al. 2006).
Amplifikasi mtDNA dengan PCR
mtDNA diamplifikasikan menggunakan primer universal Cytochrome
Oxidase I (COI) forward: 5’-ATA ATG ATA GGA GGR TTT GG-3’ dan
reverse: 5’-GCT CGT GTR CTA CRT CCA T-3’ (Williams dan Benzie 1997).
Amplifikasi dilakukan menggunakan metode polymerase Chain Reaction (PCR)
dengan komposisi premiks yaitu dH2O 13,6 µl, 10 x Buffer 5 µl; 25 mM MgCl2
1,5 µl; 2,5 mM dNTP 0,5 µl; 10 pM primer masing-masing 1,6 µl; 5 unit/µl
Taq-DNA polymerase 0,2 µl; genom DNA sebanyak 1 µl, sehingga volume akhir tiap
mikrotube adalah 25 µl. Tube yang berisi contoh dimasukkan ke dalam mesin
PCR yang telah diprogram menurut Benzie et al. (2003) yaitu proses denaturasi
pada suhu 94 °C selama 1 menit, annealing pada suhu 45 °C selama 1 menit, dan
extension pada suhu 72 °C selama 1 menit, sebanyak 30 siklus. Hasil PCR dapat
langsung dianalisis dengan elektroforesis atau disimpan di dalam refrigerator.
Bahan-bahan yang digunakan dalam proses elektroforesis dengan agarose
hasil PCR, gel loading buffer dan penanda DNA.
Gel agarose dibuat dengan cara memanaskannya di atas hotplate dan diaduk
rata. Setelah gel larut dan berwarna bening, larutan tersebut dibiarkan sampai
hangat, kemudian teteskan etidium bromida sebanyak 0,1 ml; kemudian
dituangkan ke dalam cetakan yang telah terpasang sisir pembuat lubang dengan
ketebalan 3–5 mm, selanjutnya gel dibiarkan sampai membeku dan dimasukkan
ke dalam bak elektroforesis yang berisi buffer elektroforesis.
Contoh dan marker DNA masing-masing dicampur dengan gel loading
buffer lalu dimasukkan dalam lubang-lubang yang terdapat pada gel dengan
menggunakan mikropipet. Setelah running selama 5 jam atau migrasi sepanjang ¾
bagian dari panjang gel, kemudian keberadaan DNA dilihat dengan ultraviolet
illuminator.
Digesti mtDNA dengan enzim retriksi
Digesti mtDNA dilakukan menggunakan enzim restriksi. Tahapannya adalah
mencampur 15,5 µl dH2O; 2,0 µl DNA hasil amplifikasi PCR, buffer enzim 2,0 µl, enzim restriksi 0,5 µl sehingga total larutan 20 µl dalam tabung Eppendorf
steril dan dilakukan on ice karena enzim sangat sensitif terhadap suhu dan mudah
rusak kemudian campuran dihomogenkan dan diinkubasi pada suhu 37 oC selama
1–4 jam, selanjutnya elektroforesis untuk mengetahui hasil digesti enzim dan
untuk pengamatan jumlah dan ukuran fragmen restriksi (bend) menggunakan
ultraviolet illuminator
3.3Analisis Keragaman Genetik mtDNA
Fragmen hasil restriksi mtDNA teramplifikasi dapat ditera berdasarkan
marker. Setiap pola fragmen terdiri dari satu atau lebih fragmen DNA hasil digesti
sebagai genotipe individu dari setiap enzim restriksi. Genotipe individual dari
suatu enzim dicirikan dengan kode huruf (letter code), dan kombinasi genotipe
dari enzim restriksi tersebut merupakan tipe haplotipe dari setiap individu tiram.
Keragaman genetik intrapopulasi diukur berdasarkan parameter ‘diversitas
haplotipe’ (h). Diversitas haplotipe (h) dihitung menurut persamaan Nei (1987):
Keterangan: n: ∑ contoh, Xi: frekuensi haplotipe sampai ke-i dari suatu populasi
Perbedaan genetik interpopulasi diukur berdasarkan parameter jarak genetik
(genetic distance) (Nei 1987) yang dianalisis dengan menggunakan program
TFPGA dan hubungan filogenetik interpopulasi digambarkan dalam bentuk
dendrogram berdasarkan analisis kluster terhadap nilai jarak genetik menurut
IV. HASIL DAN PEMBAHASAN
4.1Hasil
Lima puluh contoh tiram mutiara P. margaritifera yang dianalisis berasal
dari lima lokasi yaitu di Sumbawa (SB), Bali Utara (BA), Selat Sunda (SS),
Belitung (BL), dan Sulawesi Selatan (SL).
4.1.1 Amplifikasi mtDNA pada Daerah Cytochrome Oxidase I (COI )
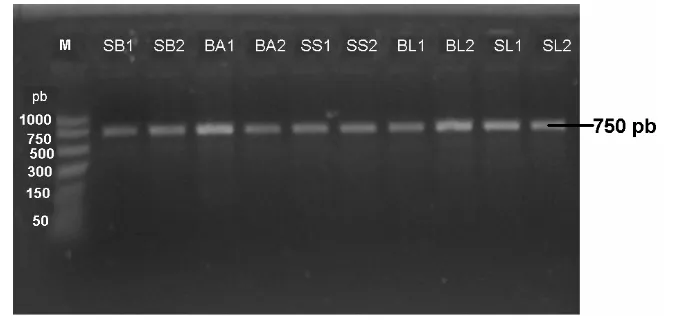
Amplifikasi daerah COI mtDNA pada tiram mutiara P. margaritifera
menggunakan primer universal COI menghasilkan fragmen DNA berukuran
sekitar 750 pb pada semua populasi contoh tiram mutiara yaitu populasi
Sumbawa, Bali Utara, Selat Sunda, Belitung, dan Sulawesi Selatan. Fragmen yang
teramplifikasi didigesti dengan enzim restriksi. Fragmen mtDNA teramplifikasi
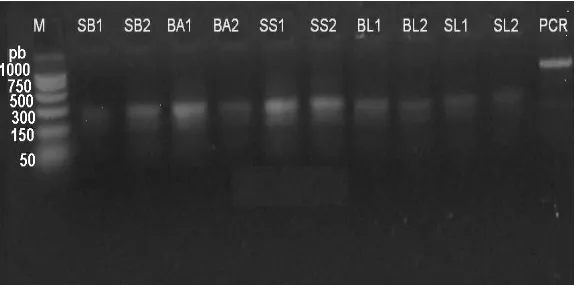
dengan primer universal COI disajikan pada Gambar 5.
Gambar 5. Fragmen mtDNA teramplifikasi dengan primer universal COI
Ket: M: marker, pb: pasangan basa, SB: Sumbawa, BA: Bali Utara, SS: Selat Sunda, BL: Belitung, SL: Sulawesi Selatan
4.1.2 Pola Restriksi Fragmen mtDNA Teramplifikasi
Pada penelitian ini telah dicoba 16 enzim restriksi (Lampiran 4), namun
hanya tiga enzim yang memberikan hasil digesti polimorfik yaitu FokI, HaeIII,
NlaIV, dua enzim menghasilkan digesti yang monomorfik yaitu DpnII dan
Eco0190I, dan 11 enzim lainnya tidak terjadi digesti. Keragaman jumlah situs dan
ukuran fragmen restriksi (RFLP) yang diperoleh dari hasil digesti mtDNA dengan
lima enzim adalah 13 tipe restriksi (genotipe) yaitu FokI dengan empat tipe
restriksi (A, B, C, D), HaeIII dengan tiga tipe restriksi (A, B, C), NlaIV dengan
masing-masing satu tipe restriksi (A) yang monomorfik, serta 34 fragmen restriksi
disajikan pada Tabel 1.
Tabel 1. Tipe fragmen restriksi mtDNA daerah COI dengan lima enzim restriksi (FokI, HaeIII, NlaIV, DpnII dan Eco0190I)
Enzim
Panjang
fragmen FokI HaeIII NlaIV DpnII Eco0190I
(pb) A B C D A B C A B C D A A
600 ▬▬ ▬▬ ▬▬
500 ▬▬ ▬▬ ▬▬
450 ▬▬
400 ▬▬ ▬▬
300 ▄▄▄ ▬▬ ▄▄▄ ▬▬ ▄▄▄ ▄▄▄
250 ▬▬ ▬▬ ▬▬
200 ▬▬
150 ▬▬ ▬▬ ▬▬ ▬▬ ▬▬ ▬▬
100 ▬▬ ▬▬
50 ▬▬ ▬▬ ▬▬
Jumlah 2 3 2 2 4 2 2 3 3 2 3 3 3
Keterangan: ▄▄▄ : pita ganda ▬▬ : pita tunggal
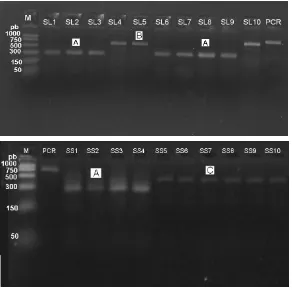
a. Digesti dengan enzim restriksi FokI
Digesti fragmen mtDNA teramplifikasi dengan enzim restriksi FokI
menghasilkan empat tipe yaitu A (600, 150 pb), B (300, 300, 150 pb), C (500, 250
pb), dan D (450, 300 pb). Digesti mtDNA dengan enzim FokI disajikan pada
Gambar 6.
Gambar 6. Digesti mtDNA dengan FokI(empat tipe restriksi: A, B, C, D)
Ket: M: marker, PCR: DNA amplifikasi, pb: pasangan basa, SS: Selat Sunda
b. Digesti dengan enzim restriksi HaeIII
Digesti fragmen DNA teramplifikasi dengan enzim restriksi HaeIII
B (600, 150 pb), C (500, 250 pb). Digesti mtDNA dengan enzim HaeIII dapat
dilihat pada Gambar 7.
Gambar 7. Digesti mtDNA dengan HaeIII (tiga tipe restriksi A, B, C)
Ket: M: marker, PCR: DNA amplifikasi, pb: pasangan basa, SL: Sulawesi Selatan, SS: Selat Sunda
c. Digesti dengan enzim restriksi NlaIV
Digesti mtDNA teramplifikasi dengan enzim restriksi NlaIV memberikan
empat tipe restriksi yaitu A (400, 250, 100 pb), B (500, 200, 50 pb), C (600, 150
pb), dan D (400, 300, 50 pb) (Gambar 8).
Gambar 8. Digesti mtDNA dengan NlaIV (empat tipe restriksi: A, B, C, D)
d. Digesti dengan enzim restriksi DpnII
Digesti DNA teramplifikasi dengan enzim restriksi DpnII menghasilkan tipe
restriksi yang monomorfik yaitu tipe A (300, 300, 150 pb) (Gambar 9).
Gambar 9. Digesti dengan DpnII (satu tipe restriksi: A)
Ket: M: marker, PCR: DNA amplifikasi, pb: pasangan basa, SB: Sumbawa, BA: Bali Utara, SS: Selat Sunda, BL: Belitung, SL: Sulawesi Selatan
e. Digesti dengan enzim restriksi Eco0190I
Digesti dengan enzim restriksi Eco0190I menghasilkan tipe restriksi
monomorfik yaitu tipe A (300, 300, 150 pb) (Gambar 10).
4.1.3 Identifikasi Fragmen Restriksi (RFLP)
Rekapitulasi identifikasi genotipe (tipe restriksi) mtDNA teramplifikasi hasil
digesti dengan lima enzim (FokI, HaeIII, NlaIV, DpnII, Eco0190I) disajikan pada
Tabel 2.
Gambar 10. Digesti dengan Eco0190I (satu tipe restriksi: A)
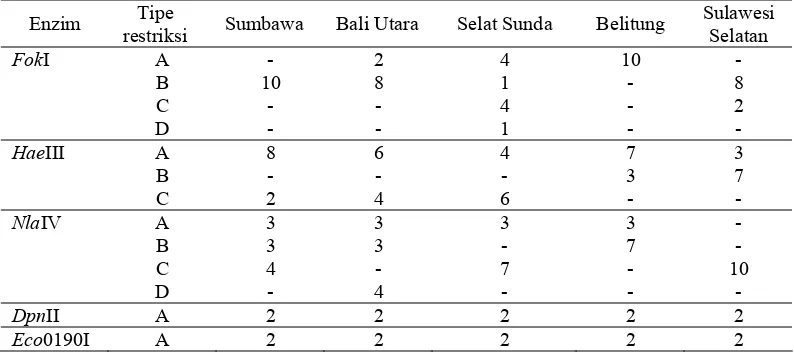
Tabel 2. Distribusi genotipe (tipe restriksi) pada lima populasi tiram mutiara
Enzim Tipe
restriksi Sumbawa Bali Utara Selat Sunda Belitung
Sulawesi
Enzim FokI menghasilkan tipe restriksi A monomorfik pada populasi
Belitung dan tipe B monomorfik pada populasi Sumbawa, sedangkan pada
populasi Bali Utara dan Sulawesi Selatan tipe B lebih dominan dan empat tipe
restriksi (A, B, C, D) pada populasi Selat Sunda.
Enzim HaeIII menghasilkan dua tipe restriksi pada semua populasi, namun
berbeda situs restriksi. Tipe A dominan ditemukan pada semua populasi, kecuali
pada populasi Selat Sunda dan Sulawesi Selatan, berturut-turut tipe C dan B lebih
dominan dari pada tipe A. Selain di Sulawesi Selatan tipe B ditemukan pula di
Belitung, sedangkan tipe C ditemukan juga pada populasi Sumbawa dan Bali
Utara.
Enzim NlaIV menghasilkan tipe restriksi C monomorfik pada populasi
Sulawesi Selatan dan dominan pada populasi Sumbawa dan Selat Sunda, dua tipe
restriksi ditemukan pada Selat Sunda dan Belitung dan tiga tipe restriksi pada
populasi Sumbawa dan Bali Utara. Tipe A terdapat pada seluruh populasi kecuali
Sulawesi Selatan, tipe B dominan pada populasi Belitung, dan tipe D hanya pada
populasi Bali Utara.
Enzim DpnII dan Eco0190I menghasilkan tipe restriksi A monomorfik pada
seluruh populasi.
4.1.4 Keragaman Genetik Intrapopulasi
Analisis composite haplotipe menghasilkan 24 composite haplotipe pada
seluruh populasi, tiap populasi terdiri dari tiga sampai delapan haplotipe yang
Lampiran 5) atau rata-rata 0,225±0,093. Distribusi composite haplotipe pada
kelima populasi tiram mutiara disajikan pada Tabel 3 dan Lampiran 6.
Tabel 3. Distribusi frekuensi dan composite haplotipe pada lima populasi tiram mutiara (Pinctada margaritifera)
No. Composite
Keragaman haplotipe 0,206 0,307 0,328 0,177 0,105
Composite haplotipe BBCAA terdapat dalam frekuensi tertinggi pada
populasi Sulawesi Selatan, dan tidak ditemukan pada populasi lainnya, demikian
pula AABAA pada Belitung dan CCCAA pada Selat Sunda, sedangkan BACAA
pada populasi Sumbawa dan Sulawesi Selatan. Composite haplotipe AAAAA
terdistribusi pada tiga populasi dari lima populasi yang dianalisis yaitu Bali Utara,
Selat Sunda, dan Belitung.
Berdasarkan analisis keseimbangan distribusi genotipik populasi menurut
Hardy-Weinberg menggunakan metode tests for Hardy-Weinberg equilibrium
dalam program TFPGA menghasilkan nilai p-value berkisar antara 0,000−0,0016
(Lampiran 7). Nilai tersebut lebih kecil dibandingkan dengan nilai p<0,01
sehingga dapat dikatakan frekuensi haplotipe intrapopulasi terdistribusi sesuai
4.1.5 Perbedaan Genetik Interpopulasi
Jarak genetik antar populasi (D) dan dendrogram hubungan kekerabatan
antar populasi (filogeni) pada lima populasi tiram mutiara menurut metode
UPGMA menggunakan program TFPGA disajikan pada Tabel 4, Gambar 11, dan
Lampiran 8.
Tabel 4. Jarak genetik interpopulasi tiram mutiara (Pinctada margaritifera)
Populasi Sumbawa Bali Utara Selat Sunda Belitung Sulawesi Selatan
Hasil perhitungan jarak genetik menunjukkan jarak terkecil (terdekat) antara
populasi Sumbawa dan Bali Utara yaitu sebesar 0,017; sedangkan jarak genetik
terbesar (terjauh) adalah antara populasi Belitung dan Sulawesi Selatan yaitu
sebesar 0,142.
Berdasarkan dendrogram kekerabatan lima populasi tiram mutiara (Gambar
11) menunjukkan pemisahan antara dua kelompok populasi yang terdiri dari
populasi Sumbawa dan Bali Utara yaitu sebesar (D=0,017) di satu pihak dengan
populasi Selat Sunda dan Belitung (D=0,069) di pihak lain terhadap populasi
Sulawesi Selatan yang memiliki tingkat kedekatan genetik paling jauh (D=0,142).
4.2 Pembahasan
4.2.1 Keragaman Haplotipe
Distribusi composite haplotipe BBCAA tertinggi ditemukan pada populasi
Sulawesi Selatan (60%), namun tidak ditemukan pada populasi lainnya, demikian
pula CCCAA (40%) pada populasi Selat Sunda dan AABAA (40%) pada populasi
Belitung. Populasi Bali Utara, Selat Sunda, dan Belitung dicirikan dengan
distribusi composite haplotipe AAAAA, dan populasi Sumbawa dan Sulawesi
Selatan dicirikan oleh distribusi composite haplotipe BACAA. Terkait dengan
kondisi lingkungan perairan yang berbeda pada lokasi pengambilan sampel
(Lampiran 9), diduga memberi pengaruh terhadap munculnya keragaman genetik
populasi tiram mutiara, seperti pada populasi Selat Sunda dan Bali Utara yang
memiliki ragam haplotipe yang tertinggi (0,328 dan 0,307). Hal ini terkait dengan
kemampuan individu menyediakan ragam genetik dalam mekanisme evolusi
sehingga bertahan hidup dan bereproduksi. Koehn et al. (1976) dan Koehn et al.
(1984) menyatakan bahwa perbedaan genetik dapat terjadi karena letak geografis,
perbedaan salinitas dan suhu. Perubahan keragaman alel yang mempengaruhi
keragaman genotipe juga disebabkan oleh migrasi, seleksi dan genetic drift
(Frankham et al. 2002).
Berdasarkan beberapa tipe haplotipe populasi Sumbawa (BAAAA, BCBAA,
BABAA) yang juga ditemukan pada populasi Bali Utara, menunjukkan kedekatan
secara geografis dan pertukaran populasi tiram mutiara terkait dengan budidaya
yang dilakukan oleh pembudidaya. Petani Sumbawa dan Bali Utara memperoleh
stok induk berasal dari wilayah timur, seperti Maluku dan Papua, sehingga
kemungkinan telah terjadi interbreeding antara populasi dari stok yang berbeda
dan introduksi keragaman jenis haplotipe baru. Menurut Hamzah et al. (2003),
induk tiram mutiara untuk kawasan tengah Indonesia tidak selamanya matang
gonad sepanjang tahun. Kawasan tengah Indonesia termasuk Sumbawa dan Bali
Utara, proses pematangan gonadnya mulai berlangsung dari awal bulan
September hingga Maret dan pada bulan April–Agustus telur kosong, sehingga
para pembudidaya mengalami kesulitan mencari induk lokal yang matang gonad
dan mereka umumnya mendatangkan dari daerah lain termasuk dari Maluku dan
Pada populasi Sumbawa dan Sulawesi Selatan yang secara geografis sangat
jauh, namun terdapat haplotipe yang sama BACAA pada populasi Sumbawa
(40%) yang juga ditemukan pada populasi Sulawesi Selatan (20%) diduga secara
genetik kedua populasi tersebut memiliki asal stok yang sama, terkait dengan
distribusinya yang secara geografis letak Selat Alas menghubungkan Laut Flores
di sebelah utara dan Samudera Hindia di sebelah Selatan, sehingga kontribusi
massa air selat tersebut dipengaruhi oleh Laut Flores dan Samudera Hindia.
Menurut Rusman (2003) menyatakan bahwa sepanjang tahun pergerakan massa
air di Selat Makassar mengarah ke selatan menuju Samudera Hindia melalui Selat
Lombok dan Laut Flores serta massa air oseanik masuk secara tetap ke Laut Jawa
melalui Selat Makassar yang berasal dari Samudera Pasifik dan Laut Flores yang
berasal dari Samudera Hindia. Adanya massa air oseanik dari Selat Makassar ini
kemungkinan arusnya membawa telur tiram dan larva fase planktonik. Menurut
Stenzel (1971) bahwa pada kehidupan organisme tiram yang hidup di perairan
laut, ketika pada masa planktonik dapat secara mudah terdispersi oleh arus,
misalnya pada larva genus Crassostrea di daerah Gulf-Stream dapat terdispersi
sampai 1000–1300 km pada kecepatan arus 2 m/detik.
Sebaran composite haplotipe AAAAA yang merupakan haplotipe umum
(common haplotipe) pada populasi Bali Utara, Selat Sunda, dan Belitung diduga
dipengaruhi oleh faktor oseanografi, geografis dan hubungannya dengan perairan
di sekitarnya (Laut Flores, Selat Makassar dan Laut Cina Selatan), aliran arus
pada perairan Selat Bali, Selat Sunda, dan Belitung masuk dalam perairan Laut
Jawa. Rusman (2003) menyatakan bahwa di perairan Laut Jawa terdapat dua
massa air yaitu massa air tercampur dan massa air oseanik. Massa air tercampur
berfluktuasi secara musiman dan dikendalikan oleh massa air dari Laut Cina
Selatan (oseanik), curah hujan dan run off dari daratan Kalimantan, sedangkan
massa air oseanik masuk secara tetap ke Laut Jawa melalui Selat Makassar yang
berasal dari Samudera Pasifik dan Laut Flores yang berasal dari Samudera Hindia.
Mengingat bahwa populasi tiram mutiara dalam siklus hidupnya mengalami stadia
planktonis selama 20–22 hari (Haws dan Ellis 2000), sehingga diduga telah terjadi
aliran gen pada ketiga populasi tersebut.