PREDIKSI PEPTIDA ANTIMIKROBA DARI HISTON H2A
DAN ANALISIS FILOGENETIK GEN COI DARI KODOK
BUDUK
Duttaphrynus melanostictus
DAN
Phyrinoidis asper
MUHAMMAD DAILAMI
G851130021
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis yang berjudul Prediksi Peptida Antimikroba dari Histon H2A dan Analisis Filogenetik Gen COI dari Kodok Buduk
Duttaphrynus melanostictus dan Phyrinoidis asper adalah benar karya saya dengan
arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2015
Muhammad Dailami
RINGKASAN
MUHAMMAD DAILAMI. Prediksi Peptida Antimikroba dari Histon H2A dan Analisis Filogenetik Gen COI dari Kodok Buduk Duttaphrynus melanostictus dan Phyrinoidis asper. Dibimbing oleh I MADE ARTIKA dan MIRZA DIKARI
KUSIRINI.
Peptida antimikroba merupakan kelompok senyawa yang dapat digunakan sebagai alternatif antibiotik konvensional untuk membasmi berbagai mikroba patogen. Kelompok hewan yang banyak menghasilkan peptida antimikroba adalah kelompok Anura, yaitu kodok dan katak. Dengan habitatnya yang ekstrim, anura
banyak terpapar dengan berbagai macam mikroba patogen dan sebagai pertahanannya anura menghasilkan berbagai macam peptida antimikroba yang bermanfaat bagi kesehatan manusia. Indonesia memiliki keanekaragaman amfibi yang tinggi, sehingga sangat berpotensi untuk menemukan peptida antimikroba baru dari Anura asal Indonesia, termasuk familyBufonidae yang banyak tersebar di
Indonesia. Duttaphrynus melanostictus dan Phyrinoidis asper adalah dua spesies
anggota family Bufonidae yang banyak ditemukan di Indonesia.
Tujuan dari penelitian ini yaitu untuk mengidentifikasi peptida antimikroba baru dengan menggunakan metode in silico berdasarkan urutan asam amino gen
penyandi histon H2A dari D. melanostictus dan P. asper. Selanjutnya identifikasi
spesies secara molekuler dan analisis pohon filogenetik dilakukan dengan menggunakan sekuens DNA fragmen gen sitokrom oksidase I.
Uji pendahuluan aktivitas antibakteri dari lendir kodok D. melanostictus dan P. asper menunjukkan hasil yang positif terhadap bakteri Echerichia coli dengan
menggunakan metode resazurin microtiter assay. Total 393 pasang basa nukleotida
yang menyandi 131 asam amino dari histon H2A berhasil diidentifikasi dan digunakan dalam prediksi peptida antimikroba. Prediksi peptida antimikroba dilakukan dengan menggunakan beberapa webserver dan didasarkan pada karakter
kimia fisika yang berupa berat molekul, muatan positif, titik isoelektrik, agregasi pada larutan dan dinding sel bakteri, serta stabilitas dari degradasi enzim peptidase. Sebanyak delapan fragmen peptida (20 asam amino) dari histon H2A D. melanostictus dan P. asper diidentifikasi memiliki potensi sebagai peptida
antimikroba. Hasil pemeringkatan menunjukkan bahwa peptida 4, 3 dan 6 memiliki potensi yang paling besar diantara kedelapan peptida tersebut. Struktur sekunder dari ketiga peptida tersebut berupa coil dan turn.
Total 668 pb nukleotida fragmen gen COI dari D. melanostictus dan P. asper
berhasil ditentukan urutan nukleotidanya. Identifikasi molekuler dengan menggunakan teknik homologi pada database NCBI dan Barcode of Life Database
menunjukkan bahwa kedua kelompok sampel yang digunakan adalah benar dari spesies D. melanostictus (100% identity) dan P. asper (99% identity). Hasil ini juga
didukung oleh pohon filogenetik yang dibuat dengan metode neighbor joining dan maximum likelihood. Pohon filogenetik menunjukkan bahwa kelompok sampel D. melanostictus berada dalam satu clade dengan sekuens D. melanostictus dari
GenBank. Untuk kelompok sampel P. asper berada dalam clade tersendiri dan tidak
ada sekuens dari P. asper yang telah dilaporkan pada GenBank sehingga tidak ada
Analisis genetika populasi dari D. melanostictus yang dibandingkan dengan
sekuens D. melanostictus dari beberapa Cina dan India pada data GenBank,
menunjukkan bahwa terdapat pemisahan clade antara D. melanostictus dari India
dan Cina. Sementara itu, sampel D. melanostictus dari Bogor berada dalam satu clade yang sama dengan sekuens dari India. Hal ini berarti bahwa sampel D. melanostictus dari Bogor memiliki hubungan kekerabatan yang lebih dekat pada D. melanostictus dari India dibandingkan dari Cina.
SUMMARY
MUHAMMAD DAILAMI. Antimicrobial Peptides Prediction from Histon H2A and Phylogenetic Analysis of COI Gene from Buduk Toads Duttaphrynus melanostictus and Phyrinoidis asper. Supervised by I MADE ARTIKA and
MIRZA DIKARI KUSRINI.
Antimicrobial peptides are group of compounds that can be used as an alternative of conventional antibiotic to combat many resistance microbial pathogen. Group of animal that produce many kinds of antimicrobial peptides is anura, which are frogs and toads. Life in extreme habitats make this animal potentially exposed to various microbial pathogens. As a defensive method, anura produce many kinds of antimicrobial peptides that beneficial for human health. Indonesia with high diversity of amphibi, potentially to discover new antimicrobial peptides from Indonesian anura, including from family Bufonidae that wide
distribute in Indonesia. Duttaphrynus melanostictus and Phyrinoidis asper are
species of Bufonidae that can be found in Indonesia.
The aim of this study is to identify new antimicrobial peptides by using in silico methods based on amino acid sequence that encodes histone H2A from D. melanostictus and P. asper. Furthermore we identify the toads species based on
molecular genetic and analyze the phylogenetic based on the sequences of cytochrome oxidase I gene.
Preliminary assay of antimicrobial activties of toad skin secretion shows a positive result by using resazurin microtiter assay (REMA) methods. A total 393 base pairs nukleotides encodes 131 amino acids from histone H2A successfully identified and were used for antimicrobial peptides prediction. In this study, antimicrobial peptides prediction was done by using several webserver and also selection based on their pshicochemical properties that includes molecular weight, positif charges, isoelectric point, aggregation in solution and membrane cell, also stability from peptidase degradation. A total eight peptide fragments (20 amino acids long) from histone H2A of D. melanostictus and P. asper were identified have
a potency to be an antimicrobial peptides. Scoring with the web server resulted on peptides 4, 3 and 6 to be the most potential as antimicrobial peptides compare to others fragments. Secondary structure of the three potent peptides are coil and turn.
A total 668 base pairs of Cytochrome oxidase I genes sequences from H2A
D. melanostictus and P. asper were identified. Species identification by molecular
genetic approach based on homology sequence with National Center for Biotechnology Information and Barcode of Life Database shows that two groups
of sampel are D. melanostictus (100% identity) and P. asper (99% identity). The
result was supported by phylogenetic tree that is constructed with neighbor joining and maximum likelihood. The tree shows that D. melanostictus sample is one clade
with the sequens of D. melanostictus from GenBank. For the P. asper sample, they
are sparated into one different clade from others species. There is no sequence cythochrome oxidase gen of P. asper already reported in GenBank, therefore no
comparative sequences can be used in phylogenetic reconstruction.
Population genetic analysis from D. melanostictus from Bogor compared to
melanostictus from Bogor, are one clades with sequence from India. This result
indicated that D. melanostictus from Bogor have closer relationship to sequence
from India than that from China.
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
PREDIKSI PEPTIDA ANTIMIKROBA DARI HISTON H2A
DAN ANALISIS FILOGENETIK GEN COI DARI KODOK
BUDUK
Duttaphrynus melanostictus
DAN
Phyrinoidis asper
MUHAMMAD DAILAMI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Biokimia
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis :.Prediksi Peptida Antimikroba dari Histon H2A dan Analisis Filogenetik Gen COI dari Kodok Buduk Duttaphrynus melanostictus dan Phyrinoidis asper
Nama : Muhammad Dailami NIM : G851130021
Disetujui oleh
Komisi Pembimbing
Dr Ir I Made Artika, M.App.Sc
Ketua Dr Mirza D. Kusrini, M.Si Anggota
Diketahui oleh
Ketua Program Studi Departemen Biokimia
Prof Dr drh Maria Bintang, MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, M.Sc.Agr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah Subhanahu Wa Ta’ala atas segala karunia-Nya sehingga karya ilmiah yang berjudul Prediksi Peptida Antimikroba dari Histon H2A dan Analisis Filogenetik Gen COI Kodok Buduk
Duttaphrynus melanostictus dan Phyrinoidis asper ini dapat diselesaikan tepat
waktu.
Terima kasih penulis ucapkan kepada Bapak Dr Ir I Made Artika, M.App,Sc dan Ibu Dr Mirza Dikari Kusrini, M.Si selaku pembimbing, serta Bapak Dodi Safari, Ph.D yang telah memfasilitasi peralatan laboratorium selama penelitian ini berlangsung. Di samping itu, penghargaan penulis sampaikan kepada rekan-rekan mahasiswa Himakova, Departemen Konservasi Sumberdaya Hutan dan Ekowisata, Fakultas Kehutanan IPB Darmaga yang telah membantu dalam proses penangkapan kodok. Terimakasih kepada seluruh teman-teman biokimia yang tidak dapat disebutkan satu persatu. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2015
DAFTAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xii
1 PENDAHULUAN
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 ANALISIS FILOGENETIK DAN PREDIKSI IN SILICO PEPTIDA
ANTIMIKROBA DARI HISTON H2A Duttaphrynus melanostictus DAN Phyrinoidis asper
Abstract 3
Pendahuluan 3
Metode Penelitian 4
Hasil dan Pembahasan 7
Simpulan 12
3 ANALISIS FILOGENETIK GEN SITOKROM OKSIDASE I DARI KODOK BUDUK Duttaphrynus melanostictus dan Phrynoidis asper
Abstract 13
Pendahuluan 13
Metode Penelitian 15
Hasil dan Pembahasan 16
Simpulan 22
4 PEMBAHASAN UMUM 24
5 SIMPULAN 27
DAFTAR PUSTAKA 28
LAMPIRAN 31
DAFTAR TABEL
2.1 Aktivitas antibakteri terhadap lendir kodok terhadap bakteri E. coli 8
2.2 Pengelompokan haplotype dan posisi titik polimorfisme 9
2.3 Peptida berpotensi sebagai AMPs beserta molecular properties dan
skor AMPs-nya berdasarkan 4 algoritma pada server CAMP R2 11
3.1 Posisi polimorfisme dari sampel P. asper 18
3.2 Daftar haplotype dari sampel D. melanostictus dan P. asper 18
3.3 Hasil BLAST pada database NCBI dan BOLD 18
3.4 Jarak genetik D. melanostictus dan P. asper dengan data GenBank 23
3.5 Jarak genetik antara kelompok D. melanostictus Bogor dengan data
GenBank 23
4.1 Keragaman haplotypeD. melanostictus dan P. asper berdasarkan
marka gen H2A dan COI 25
DAFTAR GAMBAR
2.1 Hasil Pengujian aktivitas ekstrak lendir kodok terhadap bakteri E.
coli dalam microplate 96 well. PA (P. asper), DM (D. melanostictus) 7
2.2 Hasil elektroforesis amplikon gen penyandi histon H2A dari D.
melanostictus dan P. asper 8
2.3 Hasil penerjemahan asam amino 9
2.4 Pohon Filogenetik dari sekuens histon H2A D. melanostictus dan P.
asper dengan beberapa sekuens pembanding 10
2.5 Gambar struktur 3D dari peptida berpotensi sebagai peptida
antimikroba 12
3.1 Hasil elektroforesis amplikon gen COI dari D. melanostictus (kode
DM) dan P. aser (kode PA), dengan marker (kode M) 16
3.2 Pohon Filogenetik dengan metode Neighbour joining dan Maximum
likelihood 20
3.3 Filogenetik kelompok D. melanostictus asal Bogor dengan sekuens
GenBank dengan asal lokais yang berbeda-beda 21
DAFTAR LAMPIRAN
1 Foto Sampel D. melanostictus dan P. asper 31
2 Urutan Nukleotida Fragmen Gen COI dari D. melanostictus dan P.
asper 32
1
1 PENDAHULUAN
Latar Belakang
Peptida antimikroba (Antimicrobial peptides, AMPs) merupakan salah satu
kelompok senyawa yang sangat berpotensi untuk dikembangkan dalam mengatasi masalah resistensi bakteri terhadap antibiotik konvensional (Singh dan Rai 2012). AMPs digunakan oleh berbagai organisme baik organisme tingkat rendah seperti bakteri maupun organisme tingkat tinggi seperti hewan dan tumbuhan (Bulet et al.
2004) sebagai pertahanan alami dari serangan mikroba patogen. Identifikasi peptida antimikroba baru terus dilakukan dari beragam sumber daya alam yang ada, baik dari lautan maupun dari hutan. Selain itu, penelitian mengenai peptida antimikroba juga dilakukan dengan menggunakan berbagai pendekatan seperti secara kimiawi, genetik dan bioteknologi maupun secara in silico atau bioinformatik.
Anura adalah kelompok hewan amfibi yang hidup pada lingkungan ekstrim
dan sangat rentan akan serangan berbagai mikroba patogen. Hal ini yang menyebabkan kelompok Anura harus mampu bertahan dan melindungi dirinya
dengan berbagai sistem pertahanan tubuh, yang salah satunya dengan mensekresikan lendir yang banyak mengandung AMPs. Terdapat 1900 AMPs yang telah dilaporkan dari kelompok Anura (Xu dan Lai 2015) dengan spektrum aktivitas
yang sangat luas, baik sebagai antibakteri, antifungi, antikanker dan juga antivirus. Indonesia yang memiliki keanekargaman Amfibi tinggi mencapai 16% dari total spesies dunia, tentunya sangat berpotensi untuk menemukan AMPs baru dari Anura
Indonesia (BAPPENAS 1993).
D. melanostictus dan P. asper merupakan spesies kodok yang termasuk dalam family Bufonidae yang banyak tersebar di seluruh Indonesia. Kedua spesies ini
memiliki bentuk morfologi yang mirip, dengan kulit berbintil kasar. Perbedaan mendasar terdapat pada garis parental hitam di bagian kepala. Selain itu, habitat P. asper yaitu di area hutan primer maupun sekunder yang dekat dengan aliran sungai
(Inger et al. 1974), sementara D. melanostictus lebih menyukai area sekitar
permukiman manusia. D. melanostictus sangat mudah beradaptasi dengan
lingkungan baru, sehingga jumlahnya relatif melimpah di sekitar pemukiman manusia. Beberapa peneliti juga telah melaporkan adanya aktivitas antibakteri dan antifungi dari senyawa aktif yang diisolasi dari D. melanostictus.
Rangkaian penelitian ini dibuat untuk mengidentifikasi senyawa peptida antimikroba dengan metode in silico dari sekuens gen histon H2A D. melanostictus
dan P. asper. Untuk mencapai tujuan tersebut, perlu dilakukan pengujian aktivitas
antibakteri dari ekstrak lendir D. melanostictus dan P. asper untuk memastikan
bahwa sampel yang digunakan memang benar memiliki aktivitas antibakteri. Selanjutnya dilakukan isolasi DNA, amplifikasi dan penentuan urutan nukleotida gen penyandi histon H2A yang nantinya akan diterjemahkan menjadi urutan asam amino yang menjadi dasar dalam identifikasi AMPs secara in silico. Setelah
2
Tujuan
Secara garis besar, tujuan dari penelitian ini adalah:
1. Mengidentifikasi AMPs baru yang berpotensi untuk dikembangkan dari sekuens histon H2A dengan menggunakan metode in silico.
2. Mengidentifikasi secara molekuler dan menganalisis hubungan kekerabatan
D. melanostictus dan P. asper berdasarkan marka gen COI.
Manfaat
Hasil penelitian ini diharapkan dapat memberikan informasi ilmiah berupa peptida antimikroba baru yang berpotensi untuk dikembangkan dari histon H2A D. melanostictus dan P. asper. Selain itu, memberikan informasi tentang sekuens gen
penyandi histon H2A dan COI dari kedua spesies tersebut dan hubungan filogenetik dari kedua spesies tersebut dengan spesies lain.
Ruang Lingkup Penelitian
Ruang lingkup penelitian ini adalah mendapatkan AMPs baru berdasarkan sekuens histon H2A dari D. melanostictus dan P. asper dan data sekuens gen
penyandi histon H2A dan gen COI beserta hubungan kekerabatannya berupa pohon filogenetik. Hasil penelitian ini disajikan dalam dua makalah, yaitu:
Makalah 1: Analisis Filogenetik dan Prediksi In silico Peptida Antimikroba dari
Histon H2A Duttaphrynus melanostictus dan Phyrinoidis asper.
Makalah 2: Analisis Filogenetik Gen Sitokrom Oksidase I dari Kodok Buduk
3
2 ANALISIS FILOGENETIK DAN PREDIKSI
IN SILICO
PEPTIDA ANTIMIKROBA DARI HISTON H2A
Duttaphrynus
melanostictus
DAN
Phyrinoidis asper
Abstract
Antimicrobial peptides of skin secretion of toads are promising compounds to combat wide spectrum of bacteria. Histone H2A is a type of DNA-binding protein that play as precursor of several antimicrobial peptides. In toads, family Bufonidae, buforin I and buforin II are examples of antimicrobial peptides derived from histone H2A. This study investigated the genetic diversity and phylogenetic analysis and in silico prediction of AMPs derived from histone H2A of Duttaphrynus melanostictus and Phyrinoidis asper, which were collected from
Bogor Agricultural University’s campus area. Skin secretion of D. melanostictus
and P. asper have antimicrobial activities against Escherichia coli. New set of
primers (Buf_fwd and Buf_rev) were designed by using primer BLAST, to amplify 393 nucleotide of Histone H2A gene that code 131 amino acid. Haplotype diversity of both species was very low. Phylogenetic analysis showed sample D. melanostictus and P. asper are separated to each other in two different clades.
Several short predicted peptides from histone H2A showed potency as an antimicrobial peptides based on in silico prediction. Psychochemical properties and
the 3D structure of potent antimicrobial peptides were described.
Keywords: Antimicrobial peptides, Histone H2A, In sillico prediction, Phylogenetic.
Pendahuluan
Resistensi bakteri terhadap antibiotik merupakan permasalahan global yang sedang dihadapi dunia saat ini. Song (2015) melaporkan adanya peningkatan laju resistensi berbagai patogen bakteri terhadap antibiotik konvensional di kawasan Asia. Hal ini disebabkan oleh penggunaan antibiotik yang tidak tepat baik pada sektor klinik maupun pada sektor peternakan. Oleh karenanya diperlukan alternatif antibiotik baru yang mampu membunuh bakteri yang telah resisten terhadap antibiotik konvensional sekaligus tidak menimbulkan resistensi selanjutnya.
Antimicrobial Peptides (AMPs) merupakan peptida pendek yang dapat digunakan
sebagai alternatif antibiotik konvensional, karena AMPs dapat membunuh bakteri yang telah resisten terhadap antibakteri konvensional (Singh dan Rai 2012) dan resistensi alami bakteri terhadap AMPs sangat jarang ditemukan (Sang dan Blecha 2008).
AMPs merupakan salah satu senyawa alami yang digunakan sebagai pertahanan suatu organisme dari serangan patogen. AMPs ini banyak ditemukan pada berbagai kelompok organisme mulai dari mikroba, baik bakteri maupun yeast
hingga organisme tingkat tinggi seperti hewan dan tumbuhan (Bulet et al. 2004).
4
bagian kulitnya yang mengandung AMPs untuk melindungi tubuh dari serangan mikroba patogen. Xu dan Lai (2015) merangkum seluruh peptida antimikroba dari amfibi (1900 AMPs) yang telah diteliti hingga tahun 2013 dan mengklasifikasikannya berdasarkan strukturnya menjadi 100 family peptida. Untuk
kelompok anura dari family Bufonidae, termasuk D. melanostictus dan P. asper,
masih sedikit peneliti yang melaporkan AMPs dari kedua spesies ini. Garg et al.
(2007) melaporkan adanya potensi aktivitas antimikroba dari lendir kodok D. melanostictus asal India dan aktivitas antifungi dari D. melanostictus juga telah
dilaporkan oleh Utami et al. (2010).
Buforin I dan II merupakan salah satu contoh AMPs yang diisolasi dari lambung bufo gargarizans. Buforin ini memiliki sekuens peptida yang identik
dengan histon H2A dan merupakan hasil pemotongan dari histon H2A oleh enzim endopeptidase. Kawasaki dan Iwamuro (2008) telah merangkum berbagai AMPs yang dihasilkan dari berbagai histon dari berbagai hewan. Hal ini menunjukkan bahwa histon merupakan salah satu sumber AMPs yang perlu dikaji lebih lanjut. Penapisan dan identifikasi AMPs secara konvensional merupakan penelitian yang panjang dan membutuhkan biaya mahal. Sebagai alternatif, screening dan
identifikasi AMPs dengan metode in silico dapat memberikan banyak keuntungan
baik dari segi biaya, waktu, efisiensi dan juga hasil yang diperoleh sangat menjanjikan. Beberapa penelitian eksplorasi AMPs secara in silico dari histon yang
telah dilakukan yaitu dari histon mikroba (Almaali 2014), dan dari histon Ikan (Chaitanya et al. 2014).
Oleh karena itu, penelitian ini bertujuan menguji aktivitas antibakteri dari lendir kodok buduk asal Bogor, merancang primer yang dapat mengamplifikasi gen penyandi histon H2A, kemudian menentukan urutan nukleotidanya serta melakukan analisis filogenetik gen penyandi histon H2A dari D. melanostictus dan P. asper asal Bogor, Indonesia. Selain itu, dalam penelitian ini juga dilakukan
prediksi in silico dari asam amino penyusun histon H2A untuk memperoleh
kandidat AMPs yang baru dari D. melanostictus dan P. asper.
Metode Penelitian
Waktu dan Tempat Penelitian
Penelitian ini dilakukan pada bulan Februari hingga Juni 2015 yang bertempat di Laboratorium Biokimia IPB dan Laboratorium Bakteriologi Molekuler, Lembaga Biologi Molekuler Eijkman, Jakarta.
Bahan dan Alat
Alat yang digunakan dalam penelitian ini terdiri dari: gelas ukur, labu erlenmeyer, neraca analitik, pipet mikro, tabung PCR, tabung eppendorf, rak tabung
PCR, mesin PCR (thermo cycler tipe 1148 ), heat block, microsentrifuge eppendorf,
pinset, gelas piala, lampu bunsen, magnetic stirrer, vortex, sentrifuge, seperangkat
alat elektroforesis (cetakan gel, lampu UV-Transluminator, kamera digital, tangki
dan power suplay elektroforesis), freeze dryer, cawan petri, inkubator, pH meter,
5
Bahan yang digunakan dalam penelitian ini yaitu: NaCl, NaCOOH, etanol 96%, aquades, DNAeasy blood and tissue kit (Qiagen), Molekuler Grade Water
(ddH2O), go taq green PCR-mix, oligonukleotida primer, agarosa, loading dye, gel red, Allumunium foil dan tisu, bakteri uji (E. coli), media Muller Hilton, media
dextrose agar, kloramfenikol, garam resazurin.
Pengambilan Sampel
Pengambilan sampel D. melanostictus dilakukan pada malam hari dengan
menangkap langsung di habitatnya yaitu di sekitar gedung kampus IPB Dramaga, Bogor dan untuk sampel P. asper ditangkap dari kebun percobaan Cikabayan, IPB,
Dramaga, Bogor. Identifikasi morfologi, dilakukan oleh ahli herpetofauna (Dr Ir Mirza D. Kusrini), Departemen Konservasi Sumberdaya Hutan dan Ekowisata.
Sekresi Lendir Kodok
Proses sekresi lendir kodok dilakukan sesuai dengan metode yang digunakan oleh Artika et al. (2015) dengan sedikit modifikasi. Stimulasi kimiawi dilakukan
dengan menginjeksi ephineprine-base (Kimia Farma Corp) pada bagian punggung
dengan menggunakan syringe 1 mL. Jumlah ephinephrine yang digunakan
divariasikan dari 0.01 mL/gram body weight (gbw), 0.015 mL/gbw dan 0,02
mL/gbw dari larutan stok dengan konsentrasi 1 mg/mL. Kemudian kodok direndam dalam buffer koleksi (50 mM NaCl, 25 mM NaCOOH, pH = 7) selama 15 menit
dengan volum bervariasi antara 75-200 mL. Hasil sekresi ditambahkan HCl 1% sebanyak 1 mL untuk setiap 9 mL buffer. Hasil sekresi dengan dosis yang sama
disatukan dan dikering bekukan dengan mengguanakan freeze dryer. Berat kering
ditimbang kemudian dilarutkan dalam akuades steril (1 mL) dan disterilkan menggunakan syringe 0.2 µm (Hyundai) dan digunakan dalam uji antibakteri.
Uji Aktivitas Antibakteri dengan Resazurin Microtiter Assay (REMA)
Pengujian aktivitas antibakteri dilakukan dengan menggunakan metode
resazurin microtiter assay yang dimodifikasi dari Sarker et al. (2007). Metode ini
didasarkan pada reduksi garam resazurin yang berwarna biru tua oleh bakteri yang menghasilkan produk berupa garam resofurin yang berwarna merah muda. Bakteri
Escherichia coli ditumbuhkan pada media Muller Hillton Agar selama 24 jam,
kemudian koloni tunggal disub-kultur dengan media yang sama. Bakteri dipanen dan dimasukkan dalam larutan NaCl 9% hingga mencapai 0.5 Macfarland (McF) atau setara dengan 3x108 sel. Pengujian dilakukan dengan menggunakan microplate
96 well. Setiap sumur berisi 30 µL media Muller Hillton Broth (MHB) konsentrasi
3.3x, 10 µL suspensi bakteri (0.5 McF) dan 50 µL sampel uji. Sebagai kontrol negatif digunakan media tanpa bakteri, H2O dan sebagai kontrol positif digunakan
kloramfenikol. Kemudian plate diinkubasi selama 18-20 jam, pada suhu 37oC.
6
Isolasi DNA dan Amplifikasi
DNA genom diisolasi dari sampel jaringan yang berasal dari ujung jari kaki dengan menggunakan DNAeasy Blood and Tissue Kit Qiagen. Fragmen gen yang
menyandi asam amino dari histon H2A sebagai precursor AMP diamplifikasi
dengan menggunakan primer Buf_fwd (5’-AAGAGAACGATGTCTGGACG-3’) dan Buf_rev2 (5’-TTAGAAGAGCCTTTGGTTCGGG-3’), yang didesain berdasarkan sekuens dari histon H2A dari Bufo gargarizans U70133.1 (Kim et al.
1996) dengan menggunakan primer3 pada Primer Blast NCBI (Ye et al. 2012).
Profil suhu yang digunakan dalam PCR adalah 95oC selama 5 menit, denaturasi
94oC selama 1 menit, annealing 50oC selama 1 menit, extention 72oC selama 1
menit, dan extention akhir 72oC selama 10 menit, jumlah siklus yang digunakan
yaitu 35 siklus. Hasil PCR divisualisasikan dengan elektroforesis gel agarose 1,5%, buffer TBE 1x, pewarna gel red dan marker 100 bp DNA Ladder (Biolabs Inc.). Sekuensing DNA dilakukan oleh PT. Genetika Science, Jakarta.
Analisis Filogenetik
Sekuens forward dan reverse dipastikan kebenarannya dengan mencocokkan
urutan nukleotida dengan elektroferogramnya menggunakan program MEGA 5 (Tamura et al. 2011) kemudian disejajarkan dengan menggunakan ClustalW.
Identifikasi sekuens nukleotida dilakukan dengan Basic Local Search Alignment Tools (BLAST) pada server NCBI (http://www.ncbi.nlm.nih.gov/blast). Deduksi
asam amino gen penyandi histon H2A dilakukan dengan menggunakan kode genetik standar yang tersedia pada software MEGA 5. Pohon filogenetik dibuat
dengan menggunakan metode Maximum likelihood menggunakan MEGA 5. Model test digunakan untuk mengetahui model subtitusi yang paling sesuai dengan dataset
yang digunakan. Metode bootstrap dengan 1000 replikasi digunakan untuk
membantu prediksi setiap percabangan yang terbentuk.
Prediksi Peptida Antimikroba
Prediksi secara in silico untuk memperoleh AMPs yang potensial dilakukan
dengan mengacu pada metode yang digunakan oleh Yoo et al (2015) dengan sedikit
modifikasi. Sekuens histon H2A diunggah ke server CAMP dan dipotong per 20 asam amino. Setiap fragmen peptida diprediksikan potensinya sebagai AMPs dengan menggunakan 4 algoritma yang tersedia pada server CAMP. Fragmen yang
berpotensi menjadi AMPs selanjutnya dihitung berat molekulnya dengan menggunakan EMBOSS PEPSTATS (Rice et al. 2000), titik isoelektrik (8≤PI≤12)
dan muatan positif juga dihitung serta dijadikan sebagai dasar seleksi frgamen peptida. Prediksi kemungkinan agregasi pada taraf in vitro maupun in vivo
dilakukan dengan menggunakan TANGO (AGG ≤ 500, 0 ≤ HELIX ≤ 25, 25 ≤ BETA ≤ 100) (Fernandez-Escamila et al. 2004) dan AGGRESCAN (-40 ≤ Na4vSS
≤ 60) (Conchillo-Sole et al. 2007). Kemungkinan didegradasinya kandidat AMPs
dalam sistem in vivo diprediksikan dengan menentukan adanya titik pemotongan
7
dengan server PEPstr (http://www.imtech.res.in/raghava /pepstr/) kemudian di visualisasikan dengan VMD 1.9.1 (Humphey et al. 1996).
Hasil dan Pembahasan
Aktivitas Antibakteri Lendir Kodok
Sebanyak 9 sampel D. melanostictus dan 9 sampel P. asper dikoleksi
lendirnya dengan stimulasi kimia menggunakan 3 variasi dosis (0.01 mg/gbw, 0.0015 mg/gbw dan 0.02 mg/gbw) dengan 3 individu untuk setiap dosis. Hasil pengujian aktivitas antibakteri disajikan pada Gambar 2.1 dan Tabel 2.1. Hasil pengujian ekstrak lendir kodok, menunjukkan bahwa lima ekstrak lendir kodok (PA1, PA4, DM1, DM4, DM7) memiliki aktivitas penghambatan pertumbuhan bakteri E. coli yang ditunjukkan dengan warna biru pada sumur uji. Warna biru ini
menunjukkan bahwa garam resazurin tidak mengalami reduksi menjadi resofurin (berwarna merah muda)(Sarker et al. 2007), karena tidak adanya bakteri yang
hidup. Sementara pada sampel PA7, terlihat adanya perubahan menjadi warna merah muda yang menandakan adanya pertumbuhan bakteri yang dapat mereduksi resazurin menjadi resofurin. Pada ekstrak lendir dari D. melanostictus, pemberian
variasi dosis tidak menyebabkan adanya perubahan aktivitas antibakteri. Sementara pada ekstrak lendir dari P. asper, ekstrak yang dihasilkan dengan pemberian epinephrine dengan dosis 0.02 mg/gbw ternyata tidak menunjukkan aktivitas
antibakteri.
Gambar 2.1 Hasil pengujian aktivitas ekstrak lendir kodok terhadap bakteri E. coli dalam microplate 96 well. PA (P. asper), DM (D.
melanostictus), Kl (kloramfenikol)
Desain Primer dan Amplikon yang Dihasilkan
Sepasang primer Buf_Fwd (5’-AAGAGAACGATGTCTGGACG-3’) dan Buf_rev2 (5’-TTAGAAGAGCCTTTGGTTCGGG-3’) dipilih dari beberapa alternatif primer yang diperoleh dari PrimerBlast NCBI. Primer yang didesain ini berhasil mengamplifikasi gen yang menyandi histon H2A dari D. melanostictus dan P. asper dengan produk amplikon mencapai 400-500 pb. Hasil elektroforesis dari
8
Tabel 2.1 Aktivitas antibakteri ekstrak lendir terhadap bakteri E. coli
No Sampel
diperoleh berkisar antara 400-500 pasang basa.
Urutan Nukleotida dan Deduksi Asam Amino Histon H2A
Sepanjang 393 pasang basa DNA yang menyandi 131 asam amino histon H2A yang dimulai dari kodon start (ATG) hingga kodon stop (TGA) dari kedua
spesies dipilih untuk digunakan dalam pembuatan pohon filogenetik dan deduksi asam amino. Semua sekuens histon H2A dari penelitian ini telah disubmit ke GenBank dengan kode akses KT264163 sampai KT264180 (18 sekuens). Hasil analisis BLAST pada database NCBI menunjukkan bahwa sekuens dari kedua
spesies adalah benar sekuens dari gen yang menyandi histon H2A, memiliki kemiripan 97% dengan data sekuens histon H2A dari B. gargarizans dengan kode
akses AF255739.1 dan 96% dengan dua sekuens lainnya dengan kode akses U70133.1 dan AF255740.1. Pada database GenBank, tidak ditemukan sekuens
histon H2A dari D. melanostictus dan P. asper, sehingga sekuens histon H2A dari
penelitian ini merupakan sekuens pertama yang dilaporkan dari D. melanostictus
dan P. asper asal Indonesia.
Sekuens D. melanostictus dan P. asper memiliki keragaman genetik gen
penyandi histon H2A yang sangat rendah. Hanya ditemukan dua haplotype dari
sembilan sampel D. melanostictus dan satu haplotype dari P. asper.
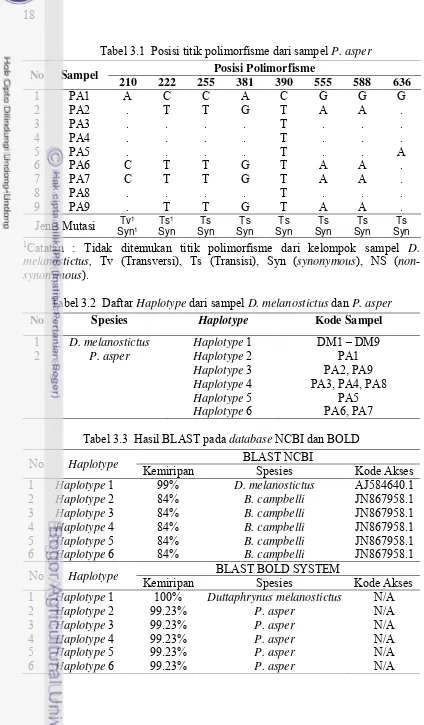
Pengelompokkan haplotype ini berdasarkan adanya 5 titik polimorfisme yang
9
purin ke pirimidin atau sebaliknya dan hanya satu subtitusi transisi (posisi 379) yaitu terjadi perubahan dari nukleotida G (purin) menjadi A (purin).
Tabel 2.2 Pengelompokkan haplotype dan posisi titik polimorfisme
Haplotype dan Sampel Posisi Nukleotida
57 124 168 376 379 Haplotype 1: D. melanostictus 1,2,3,4,6,7,9 C G C A G
Haplotype 2: D. melanostictus 5,8 C C C A G
Haplotype 3: P. asper 1 sampai 9 A G G C A
Tipe Subtitusi Tv1 Tv Tv Tv Ts Syn Non Syn Non Non
1 Tv (Transversi), Ts (Transisi), Syn (Synonymous), Non (Non Synonymous).
Deduksi asam amino dari sekuens gen penyandi histon H2A dilakukan berdasarkan tabel kode genetik standar. Hasil penerjemahan asam amino ini disajikan pada Gambar 2.3, dimana baris pertama merupakan urutan nukleotida, sedangkan baris kedua merupakan urutan asam amino. Perbedaan nukleotida dari ketiga haplotype ditandai dengan warna kuning dan merah jambu, sementara
perbedaan asam amino ditandai dengan warna merah pada baris asam amino. Berdasarkan hasil penerjemahan asam amino ini menunjukkan adanya 3 mutasi yang menyebabkan perubahan asam amino (Non synonymous), yaitu pada posisi
asam amino ke 42, 126 dan 127. Semua mutasi asam amino yang ditemukan terjadi akibat adanya mutasi nukleotida pada posisi kodon kedua dan pertama (Gambar 2.3).
10
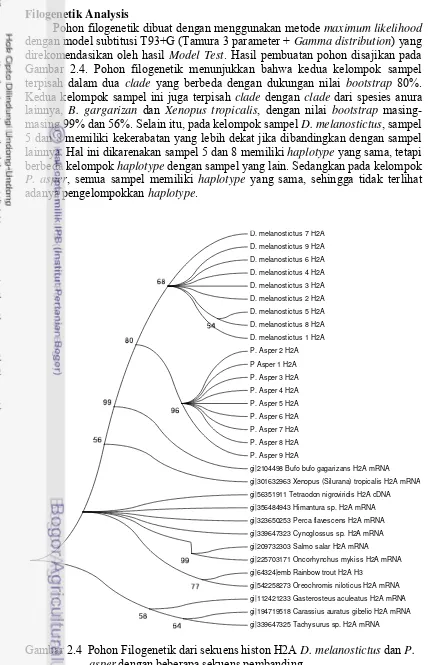
Filogenetik Analysis
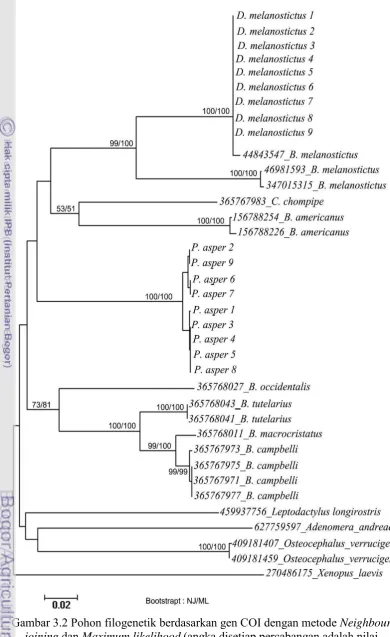
Pohon filogenetik dibuat dengan menggunakan metode maximum likelihood
dengan model subtitusi T93+G (Tamura 3 parameter + Gamma distribution) yang
direkomendasikan oleh hasil Model Test. Hasil pembuatan pohon disajikan pada
Gambar 2.4. Pohon filogenetik menunjukkan bahwa kedua kelompok sampel terpisah dalam dua clade yang berbeda dengan dukungan nilai bootstrap 80%.
Kedua kelompok sampel ini juga terpisah clade dengan clade dari spesies anura
lainnya, B. gargarizan dan Xenopus tropicalis, dengan nilai bootstrap
masing-masing 99% dan 56%. Selain itu, pada kelompok sampel D. melanostictus, sampel
5 dan 8 memiliki kekerabatan yang lebih dekat jika dibandingkan dengan sampel lainnya. Hal ini dikarenakan sampel 5 dan 8 memiliki haplotype yang sama, tetapi
berbeda kelompok haplotype dengan sampel yang lain. Sedangkan pada kelompok P. asper, semua sampel memiliki haplotype yang sama, sehingga tidak terlihat
adanya pengelompokkan haplotype.
Gambar 2.4 Pohon Filogenetik dari sekuens histon H2A D. melanostictus dan P. asper dengan beberapa sekuens pembanding.
D. melanostictus 7 H2A
gi|2104498 Bufo bufo gagarizans H2A mRNA
gi|301632963 Xenopus (Silurana) tropicalis H2A mRNA
gi|56351911 Tetraodon nigroviridis H2A cDNA
gi|356484943 Himantura sp. H2A mRNA
gi|323650253 Perca flavescens H2A mRNA
gi|339647323 Cynoglossus sp. H2A mRNA
gi|194719518 Carassius auratus gibelio H2A mRNA
11
Prediksi Peptida yang Potensial sebagai AMPs dari Sekuens Histon H2A Prediksi peptida yang berpotensi menjadi AMPs dilakukan dengan menggunakan dataset server collection of antimicrobial peptides release 2 (CAMP
R2)(Waghu et al. 2014). Total 130 asam amino digunakan sebagai template dan
diperoleh 111 fragmen sekuens peptida. Prediksi kandidat peptida yang berpotensi sebagai AMPs dilakukan dengan menggunakan 4 algoritma, yaitu search vector machine (SVM), random forest (RF), Artificial Neural Network (ANN) dan Discriminant analysis (DA). Total diperoleh 42 peptida yang berpotensi menjadi
AMP berdasarkan keempat algoritma tersebut. Peptida yang dinyatakan tidak berpotensi dari salah satu algoritma akan dihilangkan. Selanjutnya diperoleh 13 peptida yang memiliki muatan positif (Shai 2002) dan nilai titik isoelektriknya (PI) berkisar antara 8-12 (Yoo et al. 2015).
Agregasi peptida pada larutan dan pada dinding luar membran sel bakteri dilakukan dengan prediksi menggunakan software Tango (pada larutan) dan
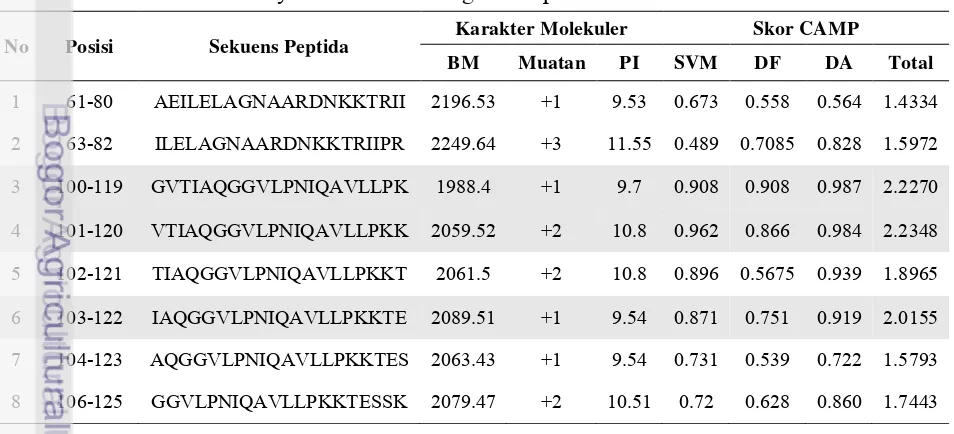
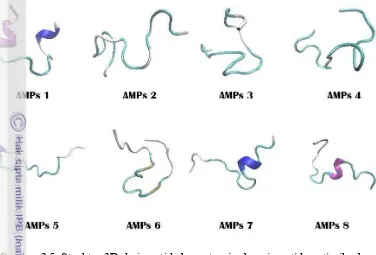
menggunakan Aggrescan (membran luar bakteri). Hasil analisis ini diperoleh 8 peptida yang memiliki potensi sebagai antimicrobial peptide (Tabel 2.3). Prediksi
struktur 3D disajikan pada Gambar 2.5. Selain itu, prediksi kemungkinan titik pemotongan enzim chymotrypsin dilakukan dengan software peptide cutter,
menunjukkan kedelapan sampel tidak memiliki potensi untuk dipotong oleh enzim tersebut. Pencarian motif PEST dari setiap peptida menggunakan EMBOSS EPESTFIND menunjukkan kedelapan sekuens tidak memiliki motif PEST. Hal ini berarti bahwa kestabilan peptida dalam sistem in vivo sangat baik, karena tidak
memiliki peluang untuk didegradasi oleh enzim penting dalam sistem in vivo.
Berdasarkan skor dari 4 algoritma yang diperoleh dari server CAMP pada tahap awal, diperoleh 3 peptida yang paling berpotensi untuk dikembangkan menjadi AMPs yaitu peptida 4,3 dan 6. Ketiga peptida tersebut perlu dikaji lebih lanjut dengan mensintesis dan menguji aktivitas antibakterinya secara in vitro. Modifikasi
struktur dari ketiga peptida tersebut juga perlu dilakukan, untuk melihat fungsi dari setiap asam amino yang ada.
Tabel 2.3 Peptida berpotensi sebagai AMPs beserta molecular properties dan
skor AMPs-nya berdasarkan 4 algoritma pada server CAMP R2
No Posisi Sekuens Peptida
Karakter Molekuler Skor CAMP
12
Gambar 2.5 Struktur 3D dari peptida berpotensi sebagai peptida antimikroba
Secara umum, struktur sekunder dari kedelapan AMPs didominasi oleh struktur coil dan turn. Beberapa AMPs memiliki sedikit dtruktur helix seperti pada
AMPs 8, 7, 5 dan 1. Tidak ditemukan adanya peptida yang memiliki struktur beta sheet. Peptida dengan nilai potensi yang tinggi yaitu AMPs 4, 3 dan 6, kesemuanya
tersusun atas struktur sekunde yang berupa coil dan turn.
Simpulan
Ekstrak lendir kodok D. melanostictus dan P. asper memiliki aktvitas
antibakteri terhadap E. coli. Sepasang primer Buf_Fwd dan Buf_Rev dapat
digunakan untuk mengamplifikasi gen penyandi histon H2A dari kedua spesies dengan baik. Pohon filogenetik menunjukkan kedua spesies terpisah dalam dua
clade yang berbeda. Keanekaragaman haplotype dari D. melanostictus lebih tinggi
dibandingkan P. asper. Sebanyak 8 fragmen peptida berpotensi sebagai peptida
antimikroba berdasarkan prediksi in silico. Berdasarkan skor dari CAMP diperoleh
13
3 ANALISIS FILOGENETIK GEN SITOKROM OKSIDASE I
DARI KODOK BUDUK
Duttaphrynus melanostictus
DAN
Phrynoidis asper
Abstract
Indonesia has high diversity of Amphibians. Amphibies have an important role in ecosystem and produce many bioactive peptides. However, the genetic information of amphibies from Indonesia is very limited, especially Duttaphrynus melanostictus and Phrynoidis asper. The aims of this study are to determine the
nucleotide sequence of cytochrome oxidase I (COI) from D. melanostictus and P. asper, to analyse their genetik diversity and their phylogenetic relationship. A total
668 base pairs of COI gene fragment was successfully amplified and their nucleotide sequence determined. P. asper (5 haplotypes) samples group have high
haplotype diversity compared to D. melanostictus (1 haplotype). The result of Basic
Local Alignment Search Tools (BLAST) to the NCBI and BOLD database, showed 99%-100% identity to sequence of D. melanostictus. The sequence of P. asper
showed 99.23% identity to sekuens P. asper in BOLD database. There was no
sequence of COI gene of P. asper in NCBI database. Genetic relationship among
species in family Bufonidae, indicated that D. melanostictus has closer relation to P. asper than to other species, inspite of their pharapyletic characteristic. For interen
species relationship of D. melanostictus, the data explains that D. melanostictus
from Bogor have closer relationship to sekuens D. melanostictus from India than D. melanostictus from China.
Keywords: COI gene, D. melanostictus, genetic diversity, phylogenetic relationship, Phyrinoidis asper
Pendahuluan
Indonesia merupakan negara yang memiliki keanekaragaman amfibi yang tinggi, sekitar 16% spesies amfibi (dari 1100 spesies diseluruh dunia) dapat ditemukan di Indonesia (BAPPENAS 1993). Secara global, jumlah populasi amfibi di dunia semakin menurun. Beberapa faktor yang diduga menjadi penyebab menurunnya jumlah amfibi di Indonesia yaitu penangkapan berlebih (untuk dikonsumsi dan diperdagangkan), hilangnya habitat, pencemaran, penyakit, spesies introduksi dan juga kecacatan (Kusrini 2007). Penurunan jumlah amfibi ini akan menyebabkan hilangnya banyak informasi ilmiah yang belum dikaji terkait amfibi asal Indonesia. Salah satu informasi penting yang masih jarang ditemukan adalah informasi genetik dari amfibi tersebut. Jika ditelusuri pada database NCBI, jumlah
sekuens atau informasi genetik dari kodok dan katak asal Indonesia masih sangat jarang.
14
invertebrata (kontrol keseimbangan ekosistem) serta menjadi salah satu indikator kualitas lingkungan (García-Muñoz et al. 2010). Selain itu, sekresi kulit anura
mengandung peptida bioaktif yang bermanfaat sebagai antimikroba, antikanker dan lainnya yang bermanfaat bagi kesehatan manusia. Beberapa senyawa peptida dari lendir anura yang memiliki aktifitas antimikroba yaitu senyawa peptida dari kelompok aurin, caerin, citropin, dahlein, maculatin, signiferin dan uperin (Pukala
et al. 2006). Xu dan Lai (2015) merangkum seluruh peptida antimikroba dari
ampfibi (1900 AMPs) yang telah diteliti hingga tahun 2013 dan mengklasifikasikannya berdasarkan strukturnya menjadi 100 family peptida.
Penelitian mengenai senyawa aktif dari lendir anura telah banyak dilakukan dan banyak dijadikan dasar dalam pengembangan obat berbasis antimicrobial peptides (AMPs). Akan tetapi, pengkajian mengenai sisi genetik dan biologi
molekuler dari kodok dan katak asal Indonesia masih sangat jarang, termasuk
family Bufonidae yang banyak tersebar di Indonesia. D. melanostictus dan P. asper
merupakan anggota family Bufonidae yang menarik untuk dikaji dari sisi
genetikanya. Kedua spesies ini memiliki bentuk morfologi yang mirip, dengan kulit berbintil kasar. Perbedaan mendasar terdapat pada garis parental hitam dibagian kepala. Selain itu, habitat P. asper yaitu di area hutan primer maupun sekunder yang
dekat dengan aliran sungai (Inger et al. 1974), sementara D. melanostictus lebih
menyukai area sekitar permukiman manusia. D. melanostictus sangat mudah
beradaptasi dengan lingkungan baru, sehingga jumlahnya relatif melimpah di sekitar pemukiman manusia.
Dalam studi genetika, gen cytochrome oxidase sub unit I (COI) merupakan
gen penting yang banyak dipelajari. Gen COI ini menyandi protein penting Sitokrom C Oksidase I yang berperan dalam proses transfer elektron pada saat sintesis Adenosine Triphosphate (ATP) dalam mitokondria. Gen COI dilaporkan
memiliki potensi laju mutasi yang rendah dibandingkan gen sitokrom b (Da Fonseska et al. 2008). Gen Sitokrom Oksidase Sub Unit I (COI) merupakan DNA barcode (Hebert et al. 2003) yang biasa digunakan sebagai acuan dalam identifikasi
genetik. Gen COI juga banyak digunakan dalam analisis pohon filogenetik, keragaman genetik, sejarah evolusi, maupun genetika populasi. Gen ini berada dalam genom mitokondria (DNA mitokondria). Beberapa keistimewaan DNA mitokondria yaitu diturunkan berdasarkan garis keturunan tetua betina, memiliki laju mutasi yang relatif lebih tinggi dibanding dengan DNA inti (Brown et al. 1979),
memiliki genom yang relatif pendek sehingga mudah untuk dipelajari (Solihin 1994).
Penelitian ini bertujuan untuk mengkaji sisi genetik dari D. melanostictus dan P. asper yang berupa urutan nukleotida gen COI, keanekaragaman genetik,
15
Metode Penelitian
Waktu dan Tempat Penelitian
Penelitian ini dilakukan pada bulan Februari hingga Juni 2015 yang bertempat di Laboratorium Biokimia IPB dan Laboratorium Bakteriologi Molekuler, Lembaga Biologi Molekuler Eijkman, Jakarta.
Bahan dan Alat
Alat yang digunakan dalam penelitian ini terdiri dari: pipet mikro, tabung PCR, tabung eppendorf, rak tabung PCR, mesin PCR (thermo cycler), heat block, microsentrifuge eppendorf, pinset, gelas piala, lampu bunsen, magnetic stirrer,
vortex, sentrifuge, seperangkat alat elektroforesis (cetakan gel, lampu
UV-Transluminator, kamera digital, tangki dan power suplay elektroforesis), penangas
air, autoklaf.
Bahan yang digunakan dalam penelitian ini yaitu: etanol 96%, aquades, DNAeasy blood and tissue kit (Qiagen), Molekuler Grade Water (ddH2O), go taq green PCR-mix, oligonukleotida primer, agarosa, loading dye, gel red, Allumunium
foil dan tisu.
Pengambilan Sampel
Pengambilan sampel D. melanostictus dilakukan pada malam hari dengan
menangkap langsung di habitatnya yaitu di sekitar gedung kampus IPB Dramaga, Bogor dan untuk sampel P. asper ditangkap dari kebun percobaan Cikabayan
kampus IPB, Dramaga, Bogor. Identifikasi morfologi, dilakukan oleh ahli herpetofauna (Dr Mirza D. Kusrini), Departemen Konservasi Sumberdaya Hutan dan Ekowisata.
Isolasi DNA dan Amplifikasi
DNA genom diisolasi dari sampel jaringan yang berasal dari ujung jari kaki dengan menggunakan DNAeasy Blood and Tissue Kit Qiagen. Fragmen gen COI
diamplifikasi menggunakan mesin PCR (Applied Biosystem) dengan primer Chmf4
(5’-TYTCWACWAAYCAYAAAGAYATCGG-3’) dan Chmr4 (5’-ACYTCRG GRTGRCCRAARAATCA-3’) (Chee et al. 2011), kode IUPAC untuk nukleotida
yang digunakan pada oligonukleotida primer terdapat pada Lampiran 3. Profil suhu yang digunakan dalam PCR adalah 95oC selama 5 menit, denaturasi 94oC selama 1
menit, annealing 50oC selama 1 menit, extention 72oC selama 1 menit, dan extention akhir 72oC selama 10 menit, jumlah siklus yang digunakan yaitu 35
siklus. Hasil PCR divisualisasikan dengan elektroforesis gel agarose 1,5%, buffer
16
Analisis Data
Sekuens forward dan reverse dipastikan kebenarannya dengan
mencocokkan urutan nukleotida dengan elektroferogramnya menggunakan program MEGA 5 kemudian disejajarkan dengan menggunakan ClustalW. Identifikasi spesies secara genetik dilakukan dengan Basic Local Search Alignment Tools (BLAST) pada server NCBI (http://www.ncbi.nlm.nih.gov/blast) dan BOLD
(barcode of life database). Untuk memperoleh sekuens dari taxa yang memiliki
kekerabatan terdekat dengan sekuens sampel dilakukan MOLE-BLAST. Deduksi asam amino gen COI dilakukan dengan menggunakan kode genetik mitokondria vertebrata yang tersedia pada software MEGA 5. Pohon filogenetik dibuat dengan
menggunakan dua metode: Neighbor joining (NJ) dan Maximum likelihood (ML)
menggunakan MEGA 5 (Tamura et al. 2011). Model test digunakan untuk
mengetahui model subtitusi yang paling sesuai dengan dataset yang digunakan (Nei dan Kumar et al. 2000). Metode bootstrap dengan 1000 replikasi digunakan untuk
mendukung setiap percabangan yang terbentuk pada pohon filogenetik (Nei dan Kumar et al. 2000). Jarak genetik dikalkulasikan dengan menggunakan metode pairwise distance (p-distance).
Hasil dan Pembahasan
Amplikon Gen COI
Hasil amplifikasi gen COI dari 18 sampel D. melanostictus dan P. asper
menggunakan primer universal COI untuk amfibi (ChmF4 dan ChmR4, Che et al.
2011) menunjukkan pita DNA pada gel agarose dengan panjang yang relatif
bervariasi antara 700-900 pasang basa (Gambar 3.1). Hal ini menunjukkan kurang spesifiknya daerah penempelan primer pada DNA template dari setiap individu,
sehingga produk amplikon yang diperoleh memiliki panjang pasang basa yang berbeda-beda. Akan tetapi, hasil sekuensing menunjukkan bahwa daerah target amplifikasi dari setiap individu adalah fragmen gen yang sama yaitu fragmen gen COI. Selain itu, spesifitas dari primer yang digunakan sangat berkaitan erat dengan sifat universal dari primer ini yang ditujukan untuk dapat mengamplifikasi fragmen gen COI dari berbagai spesies amfibi (Che et al. 2011).
Gambar 3.1 Hasil elektroforesis amplikon gen COI dari D. melanostictus (kode
17
Konsentrasi DNA hasil amplifikasi sangat tinggi, yang terlihat dari kecerahan pita DNA yang dibandingkan dengan kecerahan pita DNA marker. Konsentrasi amplikon diperkirakan lebih dari 100 ng/µL. Konsentrasi DNA amplikon akan sangat berpengaruh pada hasil sekuensing. Konsentrasi DNA yang disyaratkan dari setiap penyedia jasa sekuensing sangat bervariasi, namun umumnya meminta konsentrasi sekitar 100 ng. Berdasarkan kecerahan pita DNA sampel yang dibandingkan dengan kecerahan pita DNA marker (sesuai protokol produsen), dapat diprediksikan konsentrasi DNA amplikon yang diperoleh adalah lebih dari 97 ng (produk information). Hal ini didasarkan pada kecerahan pita ke 5 dan 10 dari DNA marker yang menunjukkan konsentrasi 97 ng (Sambrock et al.
1989), sementara pita DNA sampel, jauh lebih cerah jika dibandingkan DNA marker. Konsentrasi amplikon ini lebih dari cukup untuk dapat digunakan pada proses sekuensing.
Urutan Nukleotida Fragmen Gen COI
Sepanjang 668 pasang basa DNA fragmen gen COI dari D. melanostictus
dan P. asper berhasil ditentukan urutan nukleotidanya. Hasil pensejajaran sekuens
dari sampel D. melanostictus tidak ditemukan adanya titik polimorfisme. Untuk
sampel P. asper ditemukan adanya delapan titik polimorfisme (Tabel 3.1).
Kesemua titik polimorfisme tersebut berupa mutasi titik yang bersifat synonymous,
yaitu mutasi yang tidak menyebabkan terjadinya perubahan asam amino yang disandinya. Hal ini dikarenakan, satu jenis asam amino memiliki beberapa jenis kodon, sehingga terjadinya mutasi pada posisi kodon ke tiga memiliki peluang yang sangat kecil untuk dapat merubah asam amino yang disandinya. Jika ditinjau dari jenis basa purin dan pirimidinnya, dari delapan mutasi titik tersebut, tujuh diantaranya berupa mutasi transisi dan hanya satu yang berupa mutasi transversi. Mutasi transisi ini terlihat pada posisi nukleotida nomor 222, 255, 381, 390, 555, 588, 636 yang kesemuanya terjadi mutasi dari nukleotida yang memiliki basa purin menjadi nukleotida yang memiliki basa purin atau dari pirimidin ke pirimidin. Sedangkan untuk mutasi pada posisi 210, terjadi mutasi dari basa A (purin) menjadi C (pirimidin) sehingga mutasi ini disebut sebagai mutasi transversi.
Keanekaragaman haplotype dari P. asper lebih tinggi dari pada B. melaonstictus. Sembilan sampel D. melanostictus dapat dikelompokkan menjadi
satu haplotype dan untuk sampel P. asper terbagi menjadi 5 haplotype (Tabel 3.2).
Tingkat keragaman haplotype yang rendah ini menunjukkan bahwa gen COI dari D. melanostictus sangat conserve, yakni relatif stabil dari satu generasi ke generasi
berikutnya. Gen COI merupakan gen yang memiliki laju mutasi sangat rendah (Da Fonseska et al. 2008), sehingga variasi genetik dari gen ini sangat sedikit. Selain
itu, juga dimungkinkan sembilan sampel yang digunakan dalam penelitian ini masih berada dalam satu jalur keturunan betina. Sifat dari gen COI yang merupakan salah satu gen dari genom mitokondria adalah diturunkan hanya dari garis keturunan betina tanpa adanya hibridisasi dari genom mitokondria pejantan (Hutchison et al.
1974). Populasi P. asper yang di koleksi dari kampus IPB, memiliki keragaman haplotype yang tinggi, dimana ditemukan 5 haplotype dari sembilan sampel yang
dikoleksi. Hal ini berbeda dengan keragaman haplotype dari gen penyandi histon
H2A, dari sampel yang sama, D. melanostictus memiliki keragaman haplotype yang
18
Tabel 3.1 Posisi titik polimorfisme dari sampel P. asper
No Sampel Posisi Polimorfisme
210 222 255 381 390 555 588 636
Tabel 3.2 Daftar Haplotype dari sampel D. melanostictus dan P. asper
No Spesies Haplotype Kode Sampel
1 D. melanostictus Haplotype 1 DM1 – DM9
Tabel 3.3 Hasil BLAST pada database NCBI dan BOLD
No Haplotype BLAST NCBI
Kemiripan Spesies Kode Akses
1 Haplotype 1 99% D. melanostictus AJ584640.1
No Haplotype BLAST BOLD SYSTEM
Kemiripan Spesies Kode Akses
19
Hasil BLAST pada database GenBank (NCBI) menunjukkan sekuens dari
sampel D. melanostictus 1 sampai 9 identik dengan sekuens Bufo melanostictus
(kode akses AJ584640.1) dengan tingkat kemiripan mencapai 99%. Untuk sampel
P. asper, menunjukkan kemiripan tertinggi (84%) dengan sekuens B. campbelli
(kode akses JN867958.1). Identifikasi dengan menggunakan database BOLD
System (Barcoding of Life Database, http://www.boldsystems.org/) menunjukkan
bahwa sampel D. melanostictus identik dengan sekuens Duttaphrynus melanostictus dengan kemiripan 100%, dan untuk sampel P. asper identik dengan
sekuens P. asper dengan kemiripan 99.23%. Akan tetapi kedua sekuens yang mirip
tersebut, tidak bisa diakses maupun di download datanya, sehingga tidak bisa digunakan sebagai pembanding dalam pembuatan filogenetik.
Filogenetik analysis
MOLE-BLAST dari NCBI digunakan untuk melakukan multiple sekuens
BLAST dengan tujuan untuk mencari sekuens yang berkerabat dekat dengan D. melanostictus dan P. asper. Diperoleh sebanyak 112 sekuens dengan perbedaan
maksimal perbedaan 0.75 yang selanjutnya di dipilih 39 sekuens dari Bufonidae
(termasuk 18 sekuens sampel penelitian ini) dan juga diambil 4 sekuens dari family
lain yang masih memiliki kemiripan sangat tinggi (hasil MOLE-BLAST) dan satu sekuens dari family lain yang berkerabat jauh. Sekuens dari family lain digunakan
sebagai out group.
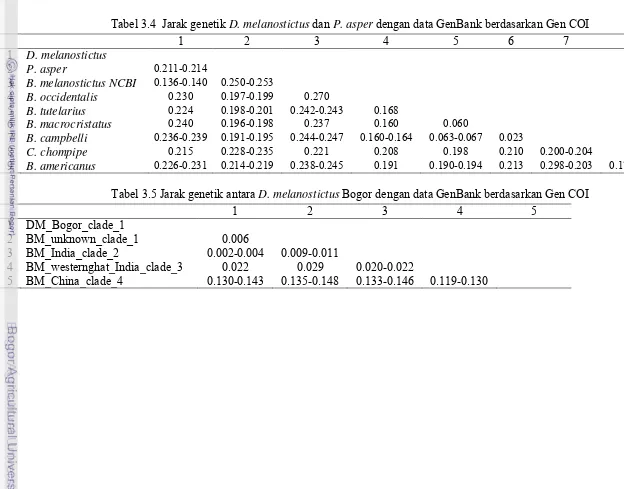
Pohon filogenetik dibuat dengan metode Neighbor Joining (NJ) dengan
model subtitusi standar yaitu Kimura 2 Parameter. Metode bootstrapt dengan 1000
replikasi digunakan sebagai parameter untuk menguji pohon filogenetik yang dihasilkan dengan metode NJ dan maximum likelihood (ML). Hasil pohon
filogenetik dengan nilai bootstrap dari metode NJ dan ML disajikan pada Gambar
3.2 dan jarak genetiknya (Genetic Distance) disajikan pada Tabel 3.4. Selain itu,
analisis pohon filogenetik dari kelompok D. melanostictus dengan perbandingan
data GenBank yang berasal dari beberapa negara yang berbeda disajikan pada Gambar 3.3 dan jarak genetiknya pada Tabel 3.5.
Pohon filogenetik menunjukkan bahwa semua spesies dari familyBufonidae
yang digunakan dalam pohon ini, berada dalam satu clade besar atau bersifat monophyletic. Semua sampel D. melanostictus berada dalam satu clade terpisah
dari spesies lain dari family Bufonidae, demikian juga dengan P. asper. Pada clade D. melanostictus, terbagi menjadi dua clade kecil yang teripsah dengan sangat jelas
dengan support bootstrapt 99% dan 100% untuk NJ dan ML. Pemisahan ini terjadi dikarenakan adanya perbedaan geografi dari sampel yang digunakan dengan sekuens dari GenBank. Sampel D. melanostictus asal kampus IPB (Indonesia)
memiliki kekerabatan yang lebih dekat dengan data GenBank dengan kode 44843547 dibandingkan dengan sekuens dengan kode 46981593 dan 347015315. Oleh karenanya, perlu dilakukan analisis hubungan filogenetik dari sampel D. melanostictus dari kampus IPB dengan sekuens dari lokasi lain yang ada di
GenBank.
Berdasarkan jarak genetiknya, D. melanostictus dari Bogor memiliki jarak
genetik terhadap C. chompipe, B. americanus dan P. asper masing-masing 0. 215,
0.226-0.231 dan 0.211-0.214. Hal ini berarti bahwa D. melanostictus memiliki
20
dan B. americanus. Frost et al. (2006) memisahkan ketiga spesies (D. melanostictus, P. asper dan B. americanus) menjadi tiga genus yang berbeda, yaitu
genus Duttaphrynus, Phyrinoidis, dan Anaxyrus. Sedangkan untuk Crepidophyrene chompipe, baru dideskripsikan pada tahun 2007, sehingga tidak termasuk dalam
pengelompokkan yang dilakukan oleh Frost et al. (2006). Meskipun D. melanostictus dan P. asper memiliki kekerabatan yang lebih dekat, namun kedua
spesies ini bersifat paraphyletic. Hal ini sesuai dengan pendapat Graybeal (1997)
yang menyatakan bahwa kedua spesies tersebut adalah paraphyletic berdasarkan
gen 12S, 16S, Cytochrome b dan c-mos (n-DNA).
Pada clade selanjutnya, terlihat bahwa B. occidentalis, B. tutelaris, B. macrocristatus, dan B. campbelly berada dalam satu clade yang sama atau bersifat monophyletic. Jarak genetik dari B. occidentalis terhadap B. tutelaris, B. macrocristatus, dan B. campbelly masing-masing yaitu 0.168, 0.160 dan
0.160-0.164. Pada penamaan baru, keempat spesies tersebut dikelompokkan dalam satu genus Cranopsis (Frost et al. 2006). Jarak genetik dari empat spesies ini yang masih
berada dalam satu genus, jauh lebih kecil jika dibandingkan 4 spesies (D. melanostictus, C. chompipe, B. americanus dan P. asper) yang berasal dari empat
genus yang berbeda. Dari beberapa informasi tersebut, terlihat bahwa, pohon filogenetik dari beberapa spesies dalam family Bufonidae yang dibangun dengan
menggunakan sekuens gen COI, memiliki hubungan genetik yang sesuai dengan klasifikasi yang dilakukan berdasarkan data morfologi dan kombinasi antara gen
16S, 12S dan Cytochrome b (Frost et al. 2006). Oleh karenanya, dapat dikatakan
bahwa, gen COI dari family Bufonidae, dapat mewakili ketiga gen diatas untuk
menjelaskan sejarah evolusi atau hubungan kekerabatan dari anggota Bufonidae sampai taraf spesies. Akan tetapi, sedikitnya sekuens COI yang tersedia pada data GenBank, menyebabkan analisis filogenetik dari seluruh spesies anggota family Bufonidae, belum dapat dilakukan. Sehingga perlu dilakukan sekuensing dari gen
COI dari spesies lain dalam family Bufonidae dan dibandingkan dengan data gen
lain yang ada di GenBank.
Pohon Filogenetik dari kelompok D. melanostictus asal kampus IPB dengan
sekuens dari lokasi lain, menunjukkan adanya 4 clade yang berbeda. Clade pertama
merupakan kelompok D. melanostictus dari Bogor dengan satu sekuens dari
genebank, namun sekuens tersebut tidak diketahui asal lokasinya. Berdasarkan pohon filogenetik tersebut, terlihat bahwa, sekuens D. melanostictus dari Bogor
memiliki hubungan kekerabatan yang lebih dekat dengan sekuens yang berasal dari India, dibandingkan dengan sekuens yang berasal dari Cina. Jarak genetik antara sampel dari Bogor dengan sekuens dari India (clade 2) yaitu antara 0.002-0.004.
Nilai ini sangat kecil jika dibandingkan dengan jarak antara sampel Bogor dengan sekuens Cina yang mencapai 0.130-0.143. Hal ini menunjukkan adanya aliran genetik (genetic drift) antara D. melanostictus asal Bogor dan India. Ada
kemungkinan bahwa D. melanostictus Bogor merupakan hasil introduksi dari India.
Hal ini didasarkan pada hipotesis Zhang et al. (2005) yang menyatakan bahwa, asal
21
Gambar 3.2 Pohon filogenetik berdasarkan gen COI dengan metode Neighbour joining dan Maximum likelihood (angka disetiap percabangan adalah nilai
22
Gambar 3.3 Filogenetik gen COI kelompok D. melanostictus asal Bogor dengan
sekuens GenBank dengan asal lokasi yang berbeda-beda.
Simpulan
Berukuran 668 pb, fragmen gen COI dari D. melanostictus dan P. asper
berhasil diamplifikasi dan diidentifikasi urutan nukleotidanya. Kelompok sampel
P. asper memiliki keanekaragaman haplotype yang lebih tinggi dibandingkan
dengan kelompok D. melanostictus. Hasil identifikasi molekuler berdasarkan gen
COI menunjukan sekuens D. melnostictus 100% identik dengan sekuens D. melanostictus dan sekuens P. asper 99% genetik dengan sekuens P. asper. Dalam
23
Tabel 3.4 Jarak genetik D. melanostictus dan P. asper dengan data GenBank berdasarkan Gen COI
1 2 3 4 5 6 7 8 9
1 D. melanostictus
2 P. asper 0.211-0.214
3 B. melanostictus NCBI 0.136-0.140 0.250-0.253
4 B. occidentalis 0.230 0.197-0.199 0.270
5 B. tutelarius 0.224 0.198-0.201 0.242-0.243 0.168
6 B. macrocristatus 0.240 0.196-0.198 0.237 0.160 0.060
7 B. campbelli 0.236-0.239 0.191-0.195 0.244-0.247 0.160-0.164 0.063-0.067 0.023
8 C. chompipe 0.215 0.228-0.235 0.221 0.208 0.198 0.210 0.200-0.204
9 B. americanus 0.226-0.231 0.214-0.219 0.238-0.245 0.191 0.190-0.194 0.213 0.298-0.203 0.179-0.183
Tabel 3.5 Jarak genetik antara D. melanostictus Bogor dengan data GenBank berdasarkan Gen COI
1 2 3 4 5
1 DM_Bogor_clade_1
2 BM_unknown_clade_1 0.006
3 BM_India_clade_2 0.002-0.004 0.009-0.011
4 BM_westernghat_India_clade_3 0.022 0.029 0.020-0.022
24
4 PEMBAHASAN UMUM
Ekstrak lendir kodok buduk D. melanostictus dan P. asper memiliki
kemapuan untuk membunuh bakteri E. coli yang dibuktikan dengan pengujian
aktivitas antibakteri menggunakan metode resazurin microtiter assay (REMA).
Dengan mengetahui adanya potensi ditemukannya senyawa aktif anti bakteri dari kedua spesies tersebut, menunjukkan bahwa kedua spesies ini sangat menjanjikan untuk dikaji potensi senyawa AMPs dari histon H2A-nya. Meskipun peneliti sebelumnya (Garg et.al 2007; Utami et al. 2010) telah melaporkan aktivitas
antibakteri dan antifungi dari D. melanostictus, namun potensi dari P. asper sendiri
belum diketahui. Selain itu, kemampuan kodok dalam menghasilkan senyawa antibakteri sangat dipengaruhi oleh faktor lingkungan dan makananya. Oleh karenanya, pengujian awal terkait aktivitas antibakteri dari kedua spesies ini sangat penting untuk dijadikan acuan dalam identifikasi senyawa AMPs dengan metode in silico.
Berdasarkan urutan nukleotida gen penyandi histon H2A (393 nukleotida, 131 asam amino), hanya ditemukan 5 titik polimorfisme yang disebabkan oleh subtitusi yang bersifat synonymous (3 titik polimorfisme) dan nonsynonymous (2
titik polimorfisme). Berdasarkan adanya polimorfisme tersebut, 18 sekuens (dari 2 spesies) dapat dikelompokkan menjadi 3 haplotipe. Karena sedikitnya mutasi yang ditemukan ini, sehingga dalam identifikasi peptida antimikroba, digunakan satu sekuens asam amino dari haplotype 1 untuk proses prediksi AMPs secara in silico.
Berdasarkan analisis tersebut, diperoleh delapan fragmen peptida dari histon H2A yang berpotensi dikembangkan menjadi AMPs. Tiga kandidat AMPs dengan nilai pemeringkatan paling tinggi menurut database CAMP adalah AMPs 4, 3 dan 6.
Ketiga AMPs tersebut memiliki struktur 3D yang berupa coil dan turn, serta tidak ditemukannya adanya struktur alpha maupun beta.
Identifikasi secara molekuler dengan menggunakan marka gen COI yang merupakan DNA barcoding, yaitu fragmen gen yang digunakan sebagai penciri
spesies, menunjukkan bahwa sekuens dari sampel D. melanostictus 100% identik
dengan sekuens D. melanostictus pada database BOLD. Sementara itu, sekuens P. asper 99% identik dengan sekuens P. asper. Untuk identifikasi dengan
menggunakan database NCBI, ditemukan sekuens sampel D. melanostictus 99%
identik dengan sekuens D. melanostictus dengan kode akses AJ584640.1.
Sementara untuk sekuens P. asper, belum adanya sekuens gen COI dari P. asper
yang telah di submit ke NCBI, sehingga hasil identifikasi menunjukkan hasil 84% identik dengan sekuens B. campbelli.
Analisis filogenetik dilakukan dengan menggunakan dua marka molekuler yaitu gen penyandi histon H2A (393 pb) dan juga gen COI (668 pb). Keragaman
haplotype dari kedua gen ini dirangkum dalam Tabel 4.1. Untuk gen penyandi
histon H2A, keragaman haplotype dari D. melanostictus (2 haplotype) lebih tinggi
dibandingkan pada P. asper (1 haplotype). Namun hal ini berbanding terbalik
dengan gen COI, dimana keragaman haplotypeP. asper (5 haplotype) lebih tinggi
dibandingkan dengan D. melanostictus (1 haplotype). Hal ini menunjukkan bahwa,