i
ANALISIS SEKUEN GEN SITOKROM OKSIDASE I DNA
MITOKONDRIA LALAT BUAH
Bactrocera sp
skripsi
sebagai salah satu syarat
untuk memperoleh gelar Sarjana Sains Biologi
Oleh Siti Nur Jannah
4411410039
JURUSAN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
UNIVERSITAS NEGERI SEMARANG
iv
Jannah S N. 2014. Analisis Sekuen Gen Sitokrom Oksidase I DNA Mitokondria Lalat buah Bactrocera sp. Skripsi, Jurusan Biologi FMIPA Universitas Negeri Semarang. Pembimbing Dr. Ir. Dyah Rini Indriyanti M.P.
Pada penelitian ini ditemukan spesies Bactrocera sp yang menyerang buah liar yang masih diragukan identitas spesiesnya. Bactrocera sp memiliki sebagian besar karakter morfologi sama dengan Bactrocera calumniata dan memiliki karakter sayap sama dengan Bactrocera cucurbitae. Bactrocera calumniata dengan Bactrocera cucurbitae masih terletak dalam subgenus yang sama Zeugodacus.
Penelitian ini bertujuan untuk mengetahui sekuen gen sitokrom oksidase I DNA mitokondria lalat buah Bactrocera sp dan untuk mengetahui hubungan antara karakter morfologi dengan gen sitokrom oksidase I.
Sampel berupa Bactrocera sp yang menyerang buah liar dan sebagai pembanding digunakan B. cucurbitae dan B. papayae. Sampel Bactrocera sp diambil dari hutan pegunungan Muria kudus. Sampel direaring sampai imago dan diidentifikasi morfologinya. Sampel B. papayae dan B. cucurbitae diambil dari biakan massal di Laboratorium Entomologi UGM yang telah teridentifikasi secara morfologi. Ketiga sampel diisolasi genom menggunakan Genomic DNA mini kit dan diamplifikasi PCR menggunakan primer mtD7 & mtD9. Produk amplifikasi PCR disekuensing dengan bantuan jasa lembaga Genetica Science di Singapura.
Tiga sampel hasil amplifikasi berhasil disekunsing dan menghasilkan panjang fragmen Cox I 427 dan 430 bp dan menunjukkan sedikit variasi
(conserve). Berdasarkan hasil BLAST menunjukan bahwa Bactrocera sp
mempunyai homologi 100 % dengan B. cucurbitae GenBank Acc Number DQ006875.1, sedangkan hasil BLAST dengan B.calumniata GenBank Acc Number GQ154088.1 dengan homologi 96%. Hal tersebut tidak sesuai dengan identifikasi morfologi yang menyebutkan bahwa sebagian besar karakter morfologi Bactrocera sp
sama dengan B.calumniata. Berdasarkan kontruksi filogeni pada Bactrocera sp
menunjukan bahwa Bactrocera sp mempunyai common ancestor yang sama dengan
B. cucurbitae yang berasal dari Swiss Eropa.
Identifikasi molekuler Bactrocera sp menggunakan gen sitokrom oksidase I DNA Mitokondria tidak sesuai dengan klasifikasi Bactrocera sp berdasarkan karakter morfologi. Gen mtCOI merupakan gen untuk mengatur fungsi respirasi sel dan bukan merupakan gen untuk mengatur karakter morfologi. Karakter morfologi yang berbeda pada Bactrocera sp dapat disebabkan oleh perkawinan silang antara B.calumniata
dengan B.cucurbitae dan proses adatasi pada kondisi lingkungan yang berbeda. Kata Kunci : Bactrocera sp, Sitokrom oksidase I, DNA mitokondria.
v
Puji syukur kehadirat Allah SWT yang telah melimpahkan rahmat dan hidayah-NYA sehingga penulis dapat menyelesaikan penyusunan skripsi yang
berjudul “Analisis sekuen gen sitokrom oksidase I DNA mitokondria lalat buah
Bactrocera sp”. Penelitian ini merupakan bagian dari penelitian payung Dr. Ir. Dyah Rini Indriyanti, M.P.
Penulisan skripsi ini tidak terlepas dari kesulitan dan hambatan, namun berkat bimbingan dan motivasi dari berbagai pihak skripsi ini dapat diselesaikan dengan baik. Atas selesainya penyusunan skripsi ini penulis mengucapkan terimakasih yang sebesar-besarnya kepada :
1. Rektor Universitas Negeri Semarang yang telah memberikan kesempatan pada penulis untuk dapat menimba ilmu di Universitas ini.
2. Dekan FMIPA Universitas Negeri Semarang yang telah memberi izin penulis sehingga dapat menyelesaikan penulisan skripsi ini.
3. Ketua Jurusan Biologi yang memudahkan jalan penulis dalam menyusun skripsi. 4. Ibu Dr. Ir. Dyah Rini Indriyanti, M.P. sebagai dosen pembimbing yang telah
meluangkan waktu guna memberikan bimbingan dan motivasi sehingga penulis dapat meyelesaikan skripsi ini.
5. Ibu Dr.drh. R. Susanti, M.P selaku dosen penguji utama yang telah banyak memberikan gagasan menarik dan ilmu pengetahuan yang belum pernah penulis pelajari.
6. Ibu Ir.Tuti Widianti, M.Biomed selaku dosen penguji ke dua yang memberikan banyak motivasi, bimbingan serta banyak ilmu dasar molekuler sehingga penulis dapat menyempurnakan skripsi ini yang tentunya banyak kekurangan.
7. Ibu Ari Yuniastuti SPt, M.kes yang memberikan banyak pengarahan dan ilmu sehingga penulis dapat menyelesaikan skripsi ini.
vi
10.Keluarga baru saya, Vivi, Sista, April dan teman-teman laboratorium virologi UGM yang telah membantu dalam analisis moluker.
11.Ayahanda Nur Salim serta Ibunda Siti dan Adikku Miftahul Anam Arois dan Nur Maulana Aeli Yudinkan sebagai penuntun dan penyemangat dalam kehidupanku, memberikan semangat dan dukungan dalam melanjutkan kehidupan dengan penuh tanggung jawab.
12.Mas Muhammad Kuriyanto yang selalu menemani mengambil sampel dengan mendaki gunung Muria Kudus sehingga penulis dapat menyelesaikan penelitian ini.
13.Ibu Indah dan keluarga selaku orang tua wali saya di Semarang yang selalu memberikan bantuan dan mempermudah dalam perjalanan kehidupan mahasiswa saya.
14.Sahabatku Annisa, Muna, Fahmi, Mei, Durroh, dan teman-teman biomi 2010 yang tidak dapat saya sebutkan satu persatu yang selalu memberi semangat, nasehat, kegalauan, dan canda tawa kepadaku.
Skripsi ini masih banyak kekurangannya, oleh sebab itu kritik dan saran sangat diharapkan. Atas segala bimbingan dan bantuan dari semua pihak, penulis berdoa semoga mendapat pahala dari Allah SWT.
Semarang, 27 Agustus 2014
vii
Halaman
HALAMAN JUDUL ... i
PERNYATAAN KEASLIAN SKRIPSI ... ii
ABSTRAK ... iii
PENGESAHAN ... iv
KATA PENGANTAR ... v
DAFTAR ISI ... vii
DAFTAR TABEL ... ix
DAFTAR GAMBAR ... x
DAFTAR LAMPIRAN ... xi
BAB I. PENDAHULUAN A.Latar Belakang ... 1
B. Rumusan Masalah ... 3
C.Penegasan Istilah ... 3
D.Tujuan Penelitian ... 3
E. Manfaat Penelitian ... 4
BAB II. TINJAUAN PUSTAKA A.Lalat buah (Bactrocera sp) ... 5
B.Identifikasi spesies secara molekuler ... 7
C.Filogenetika ... 15
BAB III. METODE PENELITIAN A.Lokasi dan Waktu Penelitian ... 20
B. Subjek penelitian ... 20
C.Rancangan Penelitian ... 21
D.Alat dan Bahan Penelitian ... 21
E. Langkah Kerja ... 22
viii
B. Pembahasan ... 47
BAB V. Simpulan dan Saran A.Simpulan ... 56
B. Saran ... 56
DAFTAR PUSTAKA ... 57
ix
Halaman
1. Beberapa spesies lalat buah yang diteliti oleh Zhang et al. (2010) ... 18
2. Alat dan bahan yang digunakan dalam penelitian... 21
3. Primer Oligonukleotida yang digunakan untuk amplifikasi PCR ... 26
4. Cocktail yang digunakan untuk amplifikasi PCR. ... 27
5. Rangkaian amplifikasi daerah sitokrom oksidase I DNA mitokondria ... 27
6. Daftar spesies yang dianalisis ... 29
7. Kandungan basa nukleotida Bactrocera sp dari progam Mega software 6.0. ... 32
8. Hasil analisis BLAST sekuen DNA gen Cox I. ... 33
x
Halaman
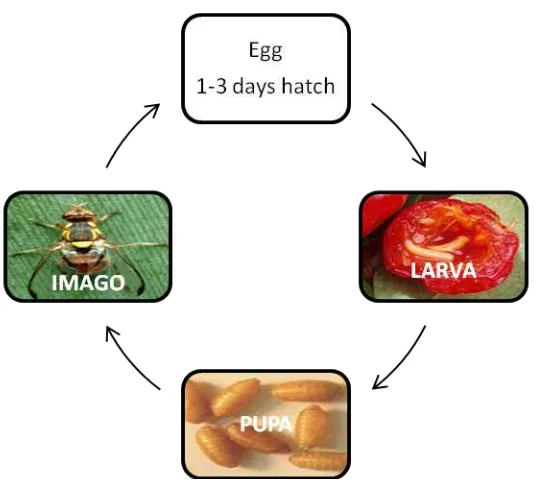
1. Siklus Hidup Bactrocera sp ... 6
2. Daerah gen mitokondria ... 10
3. Kladogram yang menggambarkan hubungan kekerabatan dari 27 spesies Bactrocera ... 17
4. Buah liar yang diserang Bactrocera sp ... 22
5. Rearing Bactrocera sp... 23
6. Hasil elektroforesis genom Bactrocera sp ... 30
7. Hasil amplifikasi PCR Bactrocera sp ... 31
8. Hasil analisis BLAST gen Cox I Bactrocera sp dengan GenBank B.calumniata Acc Number GQ154088.1 ... 34
9. Hasil analisis BLAST gen Cox IBactrocera sp dengan GenBank B. cucurbitae Acc Number DQ006875.1 ... 35
10.Hasil analisis BLAST gen Cox IB. cucurbitae dengan GenBank B. cucurbitae Acc Number DQ116244.1 ... 36
11.Hasil analisis BLAST gen Cox IBactrocera papaya dengan GenBank B. papayae Acc Number FJ903487.1 ... 37
12.Lokasi penempelan primer ... 37
13.Hasil aligment nukleotida dengan sampel koleksi GenBank ... 44
14.Hasil aligment asam amino dengan sampel koleksi genBank ... 46
xi
1. Hasil identifikasi morfologi Bactrocera sp yang menyerang buah liar,
Bactrocera cucurbitae dari buah pare, Bactrocera papayae dari buah
salak dan sebagai pembanding Bactrocera calumniata ... 63 2. Alat-Bahan yang digunakan dalam penelitian ... 64 3. Hasil kromatogram sekuen gen Cox I Bactrocera papayae yang
menyerang buah salak pada sekuen forward.. ... 65 4. Hasil kromatogram sekuen gen Cox I Bactrocera papayae yang
menyerang buah salak pada sekuen Reverse ... 66 5. Hasil kromatogram sekuen gen Cox I Bactrocera cucurbitae yang
menyerang buah pare pada sekuen forward.. ... 67 6. Hasil kromatogram sekuen gen Cox I Bactrocera cucurbitae yang
menyerang buah salak pada sekuen Reverse.. ... 68 7. Hasil kromatogram sekuen gen Cox I Bactrocera cucurbitae yang
menyerang buah liar pada sekuen forward.. ... 69 8. Hasil kromatogram sekuen gen Cox I Bactrocera cucurbitae yang
menyerang buah liar pada sekuen Reverse..... 70 9. Hasil consensus sekuen gen sitokrom oksidase I Bactrocera
cucurbitae yang menyerang buah liar (CL), Bactrocera cucurbitae
yang menyerang buah pare (CU) dan Bactrocera papayae yang
1 BAB I PENDAHULUAN
A. Latar Belakang
Indonesia merupakan salah satu negara yang disebut mega biodiversity
flora fauna setelah Brazil dan Madagaskar. Aneka flora dan fauna yang ada di dunia 25 % berada di Indonesia, dan dari setiap spesies tersebut terdiri dari ribuan plasma nutfah. Total keanekaragaman hayati di Indonesia adalah sebesar 325.350 jenis flora dan fauna (Ariyanto 2010).
Serangga sebagai salah satu komponen keanekaragaman hayati memiliki peranan penting dalam jaring makanan yaitu sebagai herbivor, karnivor, dan detrivor. Serangga herbivora merupakan faktor penyebab utama dalam kerugian hasil panen, baik secara langsung memakan jaringan tanaman atau sebagai vektor dari patogen tanaman (Ariyanto 2010).
Lalat buah (Bactrocera sp) merupakan salah satu jenis serangga yang melimpah spesiesnya (Zhang et al. 2010). Ada sekitar 4000 spesies lalat buah di dunia dan 35% di antaranya menyerang buah-buahan yang berkulit lunak dan tipis, termasuk di dalamnya buah-buahan komersial yang mempunyai nilai ekonomi tinggi. Muryati (2007) melaporkan bahwa selain menyerang buah-buahan yang lunak, sekitar 40% lalat buah juga hidup dan berkembang pada bunga tanaman famili Asteraceae (Compositae). Selebihnya hidup pada tanaman famili lainnya, misalnya Cucurbitaeceae, Solanaceae, atau menjadi pengorok pada daun, batang, dan jaringan akar.
Bactrocera sp banyak menyerang buah yang dikonsumsi, seperti apel, jambu air, papaya, mangga, belimbing, sirsak, alpukat dan cimpedak (CABI 2007). Hal ini terkait dengan kebutuhan nutrisi untuk berkembangnya telur, warna buah yang menarik dan gas metal-euginol yang dikeluarkan buah menjadi daya tarik tersendiri bagi masing-masing spesies Bactrocera sp. Hal yang menarik adalah Bactrocera sp juga menyerang buah yang tidak dikonsumsi manusia (buah liar). Buah liar tidak mempunyai organoleptik yang bagus sehingga manusia tidak mengkonsumsinya.
Lalat buah yang sering menjadi perhatian khusus bagi manusia adalah spesies yang menyerang tanaman holtikultura. Penelitian untuk mengidentifikasi
Bactrocera sp yang menyerang buah liar menggunakan analisis molekuler belum
banyak dilakukan.
Analisis molekuler pada Bactrocera sp berdasarkan kandungan DNA di dalamnya. Bila diperhatikan, tidak ada satu individu yang penampilannya sama dengan individu yang lain. Perbedaan ini disebabkan oleh perbedaan DNA. Karakter morfologi banyak disebabkan oleh interaksi genetik dan kondisi lingkungan, sehingga penanda morfolgi kurang akurat untuk mengidentifikasi organisme.
Pada penelitian ini ditemukan spesies Bactrocera sp yang menyerang buah liar yang masih diragukan identitas spesiesnya. Bactrocera sp memiliki sebagian besar karakter morfologi sama dengan Bactrocera calumniata dan memiliki karakter sayap sama dengan Bactrocera cucurbitae. Bactrocera calumniata
dengan Bactrocera cucurbitae yang masih terletak dalam subgenus yang sama
Zeugodacus. Oleh karena itu diperlukan analisis molekuler menggunakan gen sitokrom oksidase I DNA mitokondria untuk mengidentifikasi spesies
Bactrocera sp.
Alasan pemilihan gen sitokrom oxidase I adalah fragmen Cox I sering digunakan sebagai DNA barkoding untuk membedakan antara spesies. Fragmen
menyebabkan fragmen Cox I berguna untuk merekronstruksi keragaman filogenetik pada cabang evolusi di bawah tingkat spesies. Mitokondria mengalami evolusi cepat, tetapi ada bagiannya yaitu fragmen Cox I yang mengalami evolusi rendah, sehingga dipilih sebagai karakter genetika. Alasan lain dipilih sitokrom oksidase I adalah sedikit perbedaan basa nukleotida yang dijumpai diharapkan mampu mengidentifikasi spesies secara akuarat (Zein & Prawiradilaga 2013).
B. Rumusan Masalah
1. Bagaimana sekuen gen sitokrom oksidase I DNA mitokondria pada lalat buah
Bactrocera sp ?
2. Bagaimana hubungan karakter morfologi Bactrocera sp yang menyerang buah liar dengan gen sitokrom oksidase I?
C. Penegasan Istilah
Analisis sekuen gen yang dilihat adalah gen sitokrom oksidase I DNA mitokondria pada spesies Bactrocerasp.
Pada penelitian ini digunakan sampel Bactrocera sp. Bactrocera sp
menyerang buah liar dari Famili Cucurbitaeceae diperoleh dari pegunungan Muria Kudus. Sampel pembanding digunakan B. cucurbitae dan B. papayae
menyerang tanaman holtikultura diperoleh dari biakan massal di Laboratorium Entomologi Dasar di Fakultas Pertanian Universitas Gadjah Madja.
D.Tujuan Penelitian
1. Mengetahui sekuen gen sitokrom oksidase I DNA mitokondria pada lalat buah
Bactrocera sp.
E.Manfaat Penelitian
5 BAB II
TINJAUAN PUSTAKA
A.Lalat buah (Bactrocera sp)
Lalat buah (Bactrocera) dapat diklasifikasikan sebagai berikut (Siwi et al. 2006): Kingdom :Animalia
Phylum :Arthropoda Kelas : Hexapoda Ordo : Diptera Famili : Tephritidae Genus : Bactrocera
Spesies : Bactrocera sp
Lalat buah mengalami perubahan bentuk tubuh atau metamorfosis sempurna (holometabola) yaitu telur, larva, pupa, dan lalat dewasa dalam satu siklus kehidupannya. Telur lalat buah umumya berbentuk bulat panjang dengan warna putih atau putih kekuningan. Panjang telur antara 0,3 mm-0,8 mm dan lebar 0,2 mm. Telur tersebut akan menetas menjadi larva kira-kira 2 hari setelah diletakkan oleh induknya (Siwi et al. 2006). Larva umumnya berbentuk bulat panjang, berwarna putih keruh kekuningan, dan salah satu ujungnya runcing, mempunyai alat pengait dan bintik yang jelas. Larva lalat buah melewati tiga instar dalam waktu 7-10 hari hingga membentuk pupa. Lalat buah dewasa keluar dari permukaan tanah, mereka mengeraskan sayapnya terlebih dahulu sebelum terbang (Hou et al. 2006).
fungsi dan peranan betina dalam menghasilkan keturunan. Ukuran tubuh yang lebih besar memungkinkan betina untuk menyimpan telur dalam tubuhnya sebelum telur diletakan pada tubuh inangnya (Pujiastuti 2009).
Gambar 1. Siklus hidup Bactrocera sp (Pena et al. 2002) Menurut Drew (1989) ada beberapa spesies Bactrocera sp yang menyerang tanaman holtikultura. Diantaranya sub genus Zeugodacus dan Bactrocera.
Berikut contoh beberapa spesies Bactrocera sp. 1. Bactrocera (Zeugodacus) cucurbitae.
B. cucurbitae merupakan anggota genus Bactrocera dan subgenera
Zeugodacus. Menurut Dhillon et al. 2005 & Pinero et al. 2006 B. cucurbitae
2. Bactrocera (Zeugodacus) calumniata.
B. calumniata merupakan anggota genus Bactrocera dan subgenera
Zeugodacus. B. calumniata juga menyerang tanaman Cucurbitaeceae. B.
calumniata tertarik pada atraktan Cue lure. B. calumniata merupakan spesies yang kecil jumlahnya (Drew 1994).
3. Bactrocera (Bactrocera) papayae
B. papayae merupakan anggota sub genera Bactrocera yang paling melimpah jumlahnya. B. papayae bersifat polifag dan tertarik pada atraktan Metil eugenol. Menurut Muryati et al. (2007) B. papayae adalah salah satu lalat buah yang paling banyak merugikan karena banyak menyerang tanaman holtikultura.
B.Identifikasi Spesies Secara Molekuler
Identifikasi Bactrocera sp secara molekuler diperlukan beberapa komponen penting yang terkait prosedur yang digunakan, diantaranya adalah gen target dari sampel yang akan diamplifikasi. Metode yang digunakan untuk mengamplifikasi gen target menggunakan metode PCR dan metode sekuensing yang digunakan untuk melihat sekuen nukleotida dari gen targetsampel.
mutasi sinonim atau mutasi bisu yaitu mutasi pada kodon tanpa mengubah struktur protein yang diekspresinya. Berikut ini informasi mengenai DNA mitokondria dan gen sitokrom oksidase I.
1. DNA Mitokondria untuk Identifikasi Spesies
Mitokondria berbentuk sterik atau memanjang, strukturnya seperti batang dikelilingi oleh membran dalam dan membran luar. Membran luar mitokondria halus, sedangkan membran dalam melekuk menjadi lembaran atau tubuli yang disebut dengan cristae yang memanjang menuju ruangan dalam (matrix). Mitokondria ditemukan di seluruh sitoplasma dalam jumlah besar sekitar 1000 buah disetiap sel. Mitokondria banyak ditemukan pada sel-sel yang menggunakan sejumlah besar energi, sedangkan sel-sel yang kurang aktif mengandung lebih sedikit mitokondria (Wandia 2001).
DNA mitokondria tidak dapat mengalami rekombinasi sehingga terjadinya diversifikasi genetika hanya melalui mutasi. Hal tersebut menunjukkan bahwa perubahan gen yang terjadi di mitokondria terjadi pada waktu-waktu tertentu sehingga dapat digunakan untuk studi sejarah kehidupan organisme di masa lalu (Sabeti 2005).
DNA mitokondria terdiri dari 15.000–17.000 pasang basa. Setiap genom DNA mitokondria terdiri atas daerah coding dan noncoding. Daerah coding
mengambil proporsi 90% dari total genom sedangkan sisanya merupakan daerah
noncoding. Daerah coding mengandung 37 gen penyandi yang terdiri atas 22 gen penyandi transfer RNA (tRNA), dua gen penyandi ribosomal RNA (rRNA), dan 13 gen penyandi protein. Protein terdiri atas tiga subunit sitrokrom oksidase (CO I-III), tujuh subunit NADH-dehidrogenase, dua subunit ATPase dan sitokrom-b (cyt-b) yang nantinya akan mengkode pembentukan protein, terutama protein yang terlibat dalam transport elektron dan reaksi fosforilasi oksidatif dari mitokondria (Wibowo 2009).
satu diantaranya banyak mengandung basa guanin disebut rantai berat (heavy, H) dan rantai lainya mengandung basa guanin lebih sedikit sehingga disebut rantai ringan (Light, L). Hou et al. (2006) menambahkan bahwa penamaan tersebut berdasarkan pada perbedaan densitas tiap rantai, dimana rantai H memiliki berat molekul lebih besar dari pada rantai L karena rantai H memiliki banyak basa purin yang memiliki dua buah cincin pada strukturnya.
Fungsi utama dari mitokondria adalah penghasil energi melalui proses fosforilasi oksidatif. Pada proses fosforilasi oksidatif menghasilkan produk samping radikal oksigen yaitu Reactive oxygen spesies (ROS) (Wandia 2001). Hal ini dikarenakan mtDNA yang tidak memiliki system reparasi yang efisien, tidak memiliki protein histon dan terletak berdekatan dengan membran dalam mitokondria tempat berlangsungnya reaksi fosforilasi oksidatif. ROS merupakan agen oksidasi yang sangat tidak stabil sehingga dapat dengan mudah bereaksi
dengan DNA. Selain itu, DNA polymerase γ yang dimiliki mitokondria tidak
memiliki suatu proses perbaikan dan pengakuratan dalam replikasi DNA. Replikasi DNA yang tidak akurat menyebabkan mutasi mudah terjadi (Han et al. 2006).
2. Gen Sitokrom oksidase subunit I
Sitokrom oksidase I enzim yang larut dalam air yang berperan sebagai pembawa elektron dalam reaksi fosforilasi oksidatif pada mitokondria. Enzim ini memiliki struktur kompleks yang mengandung 13 subunit, antara lain lima fosfatidil etanolamina, tiga fosfatidil gliserol, dua asam kolat, dua gugus heme A, dan beberapa kofaktor ion logam, meliputi tiga atom tembaga, satu atom magnesium, dan satu atom seng. Enzim ini juga berperan pada reaksi terakhir pada rantai transpor elektron dan mentransfer elektron ke oksigen, ketika memompa proton melewati membran (Richter & Ludwig 2003).
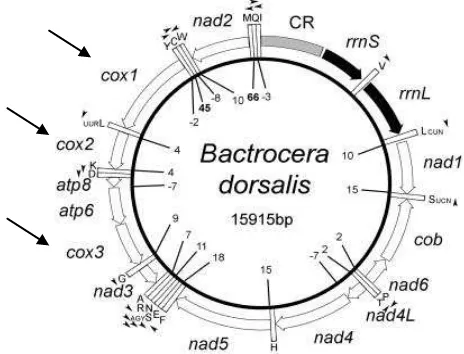
evolusi rendah (Zein & Prawiradilaga 2013). Daerah gen sitokrom oksidase ditunjukan pada Gambar 2.
Gambar 2. Daerah gen mitokondria. Gen penyandi sitokrom oksidase memiliki tiga subunit yaitu Cox I, Cox II, Cox III yang ditunjukan oleh anak panah.
Menurut Zhang et al. (2010) gen sitokrom oksidase subunit I pada
Bactrocera sp memiliki panjang nukleotida 689 bp. Gen sitokrom oksidase I pada Bactrocera sp memiliki kandungan basa (A+T) lebih banyak dibandingkan Thepritidae lainnya. Menurut penelitian yang dilakukan oleh Jamnongluk et al. (2003) kandungan basa (A+T) pada beberapa spesies Bactrocera sp sekitar 63 – 68 % dari 639 bp gen Cox I. Pada sekuen gen penyandi sitokrom oksidase subunit I Bactrocera sp terdapat 18 tempat variasi dari 213 asam amino.
3. Polymerase Chain Reaction (PCR)
primer forward dan yang berada setelah daerah target disebut primer reverse. Untuk dapat mencetak rangkaian tersebut dalam teknik PCR diperlukan juga dNTPs yang mencakup dATP, dCTP, dGTP dan dTTP. Amplifikasi sekuen DNA target dapat memperoleh 106-109 kali jumlah DNA target awal. Amplifikasi ini dapat menghasilkan lebih dari satu juta kali DNA asli (Muladno 2010 & Sudjadi 2008).
Konsep teknologi PCR mensyaratkan bagian tertentu sekuen DNA yang akan dilipatgandakan harus diketahui terlebih dahulu sebelum proses pelipatgandaan tersebut dilakukan. Reaksi pelipat gandaan suatu fragmen DNA dimulai dengan melakukan denaturasi DNA template (cetakan) sehingga rantai DNA yang berantai ganda (double stranded) akan terpisah menjadi rantai tunggal (single stranded). Denaturasi DNA dilakukan dilakukan dengan menggunakan suhu (95°C) selama1–2 menit, kemudian suhu diturunkan menjadi 55°C sehingga primer akan menempel (annealing) pada cetakan yang telah terpisah menjadi rantai tunggal. Primer akan membentuk jembatan hidrogen dengan cetakan pada daerah sekuen yang komplementer dengan sekuen primer. Suhu optimal untuk penempelan primer ± 55 °C. Amplifikasi akan lebih efisien jika dilakukan pada suhu yang lebih rendah 37°C, tetapi biasanya akan terjadi
mispriming yaitu penempelan primer pada tempat yang salah. Pada suhu yang lebih tinggi (55°C), spesifitas reaksi amplifikasi akan meningkat, tetapi secara keseluruhan efisiensinya akan menurun (Yuwono 2006).
Menurut Handoyo & Rudiretna (2001) komponen- komponen yang diperlukan pada proses PCR secara umum adalah DNA template, sepasang primer, dNTPs (deoxynucleotide triphosphates), buffer PCR, magnesium klorida (MgCl) dan enzim polimerase DNA.
a. Template DNA
dengan melakukan isolasi DNA kromosom atau DNA plasmid. (Handoyo & Rudiretna 2001). Konsentrasi template DNA harus dioptimasi. Jika konsentrasinya terlalu rendah, maka primer tidak dapat menemukan target. Sebaliknya bila konsentrasi template DNA terlalu tinggi akan meningkatkan kemungkinan salah target (mispriming). Selain itu kemurnian template DNA juga penting, karena dapat mempengaruhi hasil reaksi (Zein & Prawiradilaga 2013).
b. Primer (Oligonukleotida)
Primer yang digunakan dalam PCR ada dua yaitu oligonukleotida yang mempunyai sekuen yang identik dengan salah satu rantai DNA cetakan pada
ujung 5’-fosfat, dan oligonukleotida yang kedua identik dengan sekuen pada
ujung 3’-OH rantai DNA cetakan yang lain (Yuwono 2006).
Menurut Suryanto (2003), primer biasanya terdiri dari 10-20 nukleotida. Semakin panjang primer, maka harus spesifik daerah yang diamplifikasi. Jika suatu kelompok organisme memang berkerabat dekat, maka primer dapat digunakan untuk mengamplifikasi daerah tertentu yang sama dalam genom kelompok tersebut.
c. dNTPs (deoxynucleotide triphosphates)
dNTPs merupakan suatu campuran yang terdiri atas dATP (deoksiadenosin trifosfat), dTTP (deoksitimidin trifosfat), dCTP (deoksisitidin trifosfat) dan dGTP
d. Buffer PCR dan MgCl
Reaksi PCR hanya akan berlangsung pada kondisi pH tertentu. Oleh karena itu untuk melakukan proses PCR diperlukan buffer PCR. Fungsi buffer di sini adalah untuk menjaga pH medium. Selain buffer PCR diperlukan juga adanya ion Mg 2+ yang berasal dari MgCl2. MgCl2 bertindak sebagai kofaktor yang berfungsi menstimulasi aktivitas DNA polimerase. MgCl2 berperan meningkatkan interaksi primer dengan template DNA yang membentuk komplek terlarut dengan dNTP untuk membuat substrat yang akan dikenali oleh enzim taq DNA Polymerase. Konsentrasi Ion Mg 2+ yang terlalu tinggi akan menyebabkan denaturasi rantai DNA menjadi sulit. Konsentrasi di atas 10 mM akan mengakibatkan sifat enzimatik Taq DNA menjadi tidak efektif (Zein & Prawiradilaga 2013).
e. DNA Polymerase (Taq Polymerase)
Enzim Taq DNA polymerase adalah suatu enzim yang bersifat thermostabil yang diisolasi dari Thermus aquaticus. DNA polymerase adalah enzim yang mengkatalisis polimerasi DNA. Jumlah polimerase DNA yang digunakan tergantung pada panjang fragmen DNA yang akan diamplifikasi. Untuk panjang fragmen DNA kurang dari dua kilobasa diperlukan 1,25 – 2 unit per 50 uL campuran reaksi, sedangkan untuk panjang fragmen DNA lebih besar dari dua kilobasa diperlukan 3 – unit per 50 uL campuran reaksi (Zein & Prawiradilaga 2013).
4. Metode Sekuensing
Pada tahun 1977 metode sekuensing telah berkembang di Amerika yang dipelopori oleh Maxam and Gilbert dan pada tahu 1974 di Inggris oleh Sanger. Ada dua metode Sekuensing yaitu metode Maxam and Gilbert dan Sanger (Lilian
et al. 2002).
a. Metode Maxam and Gilbert
Metode ini didasarkan pada degradasi basa secara kimiawi. Pada metode ini DNA yang akan disekuensing ditandai dengan zat radioaktif. Fragment DNA yang sudah dilabeli merupakan subjek untuk pemecahan secara acak pada posisi basa adenine, sitosin, guanine dan timin menggunakan agen kimia spesifik. Degradasi senyawa kimia ini didasarkan pada tiga tahap: Perubahan basa nukleotida, penggantian dari basa yang telah mengalami perubahan pada molekul gulanya dan rantai DNA yang dipecah pada molekul gulanya. Hal ini akan menghasilkan sekumpulan fragmen bertanda radioaktif yang panjangnya tergantung pada jarak antara letak basa yang dihilangkan dengan ujung molekul bertanda radioaktif. Sekuens DNA dapat dibaca dari hasil pemisahan fragmen – fragmen yang terbentuk pada gel poliakrilamida (Lilian et al. 2002).
b. Metode Sanger
Pada metode Sanger, ekstensi rantai DNA dimulai pada situs spesifik pada DNA cetakan dengan menggunakan oligonukleotida pendek yang disebut
raadioaktif. Fragmen-fragmen DNA tersebut lalu dipisahkan menurut ukurannya dengan elektroforesis gel poliakrilamida (Lilian et al. 2002).
c. Filogenetika
Filogenetika menggambarkan klasifikasi secara taksonomi dari suatu organisme berdasarkan pada sejarah evolusi. Proses evolusi melibatkan proses rekombinan gen dan mutasi genetik pada spesies yang membentuk spesies yang baru. Sejarah evolusi suatu organisme dapat dilihat berdasarkan perubahan karakter organisme. Karakter merupakan dasar yang digunakan untuk menganalisis hubungan antara spesies (Schmidt 2003).
Tujuan dari penyusunan filogenetika salah satunya adalah untuk merekontruksi dengan tepat hubungan antara organisme dan menganalisis perbedaan yang terjadi dari satu nenek moyang kepada keturunannya. Filogenetika dapat menganalisis perubahan yang terjadi dalam evolusi organisme yang berbeda. Pohon filogenetik adalah pendekatan logis untuk menunjukkan hubungan evolusi organisme satu dengan yang lainya dapat digambarkan melalui sebuah poon filogenetik (Schmidt 2003). Sebelum menganalisis filogentika suatu organisme, terlebih dahulu mengetahui sejarah filogeni suatu organisme.
Bactrocera sp merupakan genus terbanyak dari jenis lalat buah (Diptera: Tephritidae) yang ditemukan di Benua Asia dan Australia dan merupakan salah satu generasi terbesar yaitu 500 spesies (Drew 1989; Drew & Hancock 2000). Sebelumnya Bactrocera sp merupakan anggota dari genus Dacus, hal itu disebabkan terjadi kekeliruan identifikasi. Genus Dacus merupakan spesies lalat buah asli dari Afrika, dan biasanya berasosiasi dengan bunga dari tanaman Cucurbitaecea dan kulit buah tanaman kacang-kacangan (Muryati 2007).
betina maupun jantan dilengkapi dengan glandular ceromae. Analisis sekuen DNA mitokondria kedua genus tersebut berkerabat dekat.
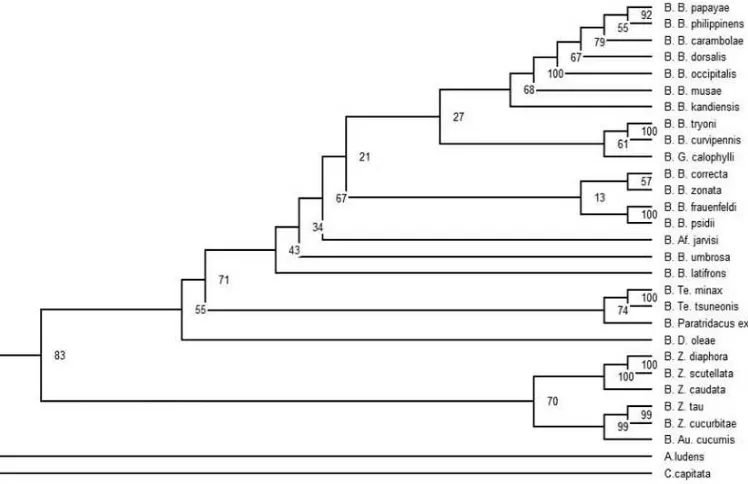
Menurut Zhang et al. (2010), yang membedakan 27 spesies (Tabel 1) dari delapan subgenera lalat buah berdasarkan gen Cox I dan 16S rRNA yaitu
Afrodacus, Austrodacus, Bactrocera, Daculus, Gymnodacus, Paratridacus,
Tetradacus, dan Zeugodacus. Bactrocera dan Zeugodacus merupakan parafiletik, sedangkan Austrodacus dan Zeugodacus yang menyerang tanaman Cucurbitaeceae tidak berkerabat dengan Afrodacus, Bactrocera, dan
Gymnodacus yang menyerang tanaman sefamili. Paratridacus yang merupakan saudara dari Tetradacus dan tujuh spesies dari Bactrocera termasuk clade
Gambar 3. Kladogram yang menggambarkan hubungan kekerabatan dari 27 spesies Bactrocera sp (Zhang et al. 2010).
Zhang et al. (2010) menambahkan bahwa Bactrocera dorsalis complex yang menduduki subgenus Bactrocera merupakan monofiletik. Bactrocera
dorsalis complex mempunyai bentuk dan sifat yang berbeda akan tetapi mereka
Tabel 1. Beberapa spesies lalat buah yang diteliti oleh Zhang et al.( 2010)
NO Spesies Sub genera
1 Bactrocera carambolae Bactrocera
2 Bactrocera correcta Bactrocera
3 Bactrocera dorsalis Bactrocera
4 Bactrocera latifrons, Bactrocera
5 Bactrocera musae, Bactrocera
6 Bactrocera papayae, Bactrocera
7 Bactrocera philippinensis Bactrocera
8 Bactrocera zonata , Bactrocera
9 Bactrocera jarvisi Afrodacus
10 Bactrocera cucumis Austrodacus
11 Bactrocera curvipennis Bactrocera
12 Bactrocera frauenfeldi Bactrocera
13 Bactrocera kandiensis, Bactrocera
14 Bactrocera occipitalis Bactrocera
15 Bactrocera psidii Bactrocera
16 Bactrocera tryoni Bactrocera
17 Bactrocera umbrosa Bactrocera
18 Bactrocera oleae Daculus
19 Bactrocera calophylli Gymnodacus
20 Bactrocera expandens Paratridacus
21 Bactrocera minax Tetradacus
22 Bactrocera tsuneonis Tetradacus
23 Bactrocera caudate Zeugodacus
24 Bactrocera diaphora , Zeugodacus
25 Bactrocera scutellata Zeugodacus
26 Bactrocera tau Zeugodacus
27 Bactrocera cucurbitae Zeugodacus
28 Anastrepha ludens Out group
20 BAB III
METODE PENELITIAN
A.Lokasi dan Waktu Penelitian
Penelitian ini dilakukan di lima lokasi yang berbeda.
1. Pengambilan buah liar dari Famili Cucurbitaceae yang terserang Bactrocera sp
dilakukan di pegunungan Muria Kabupaten Kudus. Pengambilan buah liar dilakukan pada bulan April 2013.
2. Rearing Bactrocera sp dari buah liar dan identifikasi morfologi imago
Bactrocerasp dilakukan di Laboratorium Biologi Universitas Negeri Semarang dan diperkuat di Laboratorium Entomologi UGM pada bulan Mei 2013.
3. Pengambilan sampel B. cucurbitae dan B. papayae dari biakan massal Laboratorium Entomologi Dasar Fakultas Pertanian Universitas Gadjah Mada dilakukan pada bulan April 2014.
4. Analisis molekuler sampel di Laboratorium Virology Fakultas Pertanian Universitas Gajah Mada pada April-Mei 2014.
5. Sekuensing nukleotida ketiga sampel dengan bantuan jasa Lembaga Genetica science dilakukan di Singapura.
B. Subjek Penelitian
Sampel dalam penelitian ini terdiri dari tiga jenis Bactrocera sp. Pertama spesimen Bactrocerasp yang terdiri dari imago dan larva yang berasal dari buah liar dari Famili Cucurbitaeceae. Sampel pembanding adalah Bactrocera papayae
C.Rancangan penelitian
Penelitian ini merupakan penelitian deskriptif observasional. Sampel yang digunakan dalam penelitian ini adalah Bactrocerasp yang menyerang buah liar, B. cucurbitae yang menyerang buah pare dan B. papayae yang menyerang buah salak. Ketiga sampel diidentifikasi morfologi dan dianalisis molekulernya.
D.Alat dan Bahan penelitian
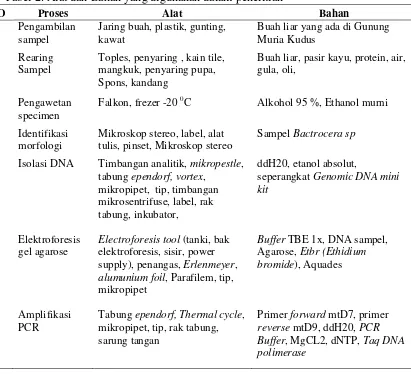
Alat dan bahan yang digunakan dalam penelitian ini disajikan dalam Tabel 2. Tabel 2. Alat dan Bahan yang digunakan dalam penelitian
NO Proses Alat Bahan
1 Pengambilan
sampel
Jaring buah, plastik, gunting, kawat
Buah liar yang ada di Gunung Muria Kudus
Buah liar, pasir kayu, protein, air, gula, oli,
3 Pengawetan
specimen
Falkon, frezer -20 0C Alkohol 95 %, Ethanol murni
4 Identifikasi
morfologi
Mikroskop stereo, label, alat tulis, pinset, Mikroskop stereo
Sampel Bactrocera sp
5 Isolasi DNA Timbangan analitik, mikropestle,
tabung ependorf, vortex,
Tabung ependorf, Thermal cycle,
mikropipet, tip, rak tabung, sarung tangan
Primer forward mtD7, primer
E. Langkah Kerja 1.Pengambilan Sampel
Sampel yang digunakan dalam penelitian ini terdiri dari tiga spesies yaitu
Bactrocera sp, B. cucurbitae dan B. papayae. B. cucurbitae dan B. papayae
diperoleh dari pembiakan massal Bactrocera sp di Laboratorium Entomologi Dasar Unniversitas Gadjah Mada, sedangkan Bactrocera diperoleh dari pengambilan buah liar (inang Bactrocerasp) famili Cucurbitaeceae.
Pengambilan Bactrocera sp yang menyerang buah liar dari hutan pegunungan Muria Kudus. Inang Bactrocera sp adalah buah liar yang tidak dikonsumsi manusia. Buah ini termasuk famili Cucurbitaeceae yang belum diketahui nama spesiesnya. Rasa buahnya sedikit pahit ketika muda dan sedikit hambar ketika sudah matang. Tanaman buah merambat dipohon yang tinggi untuk memperoleh banyak sinar matahari. Ukuran buah berdiameter 15 cm X 15 cm. Buah yang diambil sudah dalam keadaan busuk dan secara makro terlihat ada larva di dalam buah. Berikut adalah foto buah liar yang diserang Bactrocera sp yang diambil dari pegunungan Muria Kudus yang diperjelas pada Gambar 4. Larva yang ada dalam buah dibiakan (Rearing) di Laboratorium untuk diidentifikasi imagonya.
2. Rearing Bactrocera sp.
Buah yang terinfeksi larva lalat buah diambil dan direaring sampai larva menjadi imago Bactrocera sp (Gambar 5a). Buah yang mengandung lalat buah dapat diketahui dari bekas tusukan ovopositor lalat buah betina. Buah semakin lama semakin membusuk, kemudian diletakan dalam saringan kecil yang dibawahnya ada wadah penampung tetesan air yang berasal dari buah (Gambar 5b). Seperangkat wadah buah tersebut dimasukkan dalam wadah yang berisi limbah kayu dan kemudian ditutup dengan kain tile. Setelah larva berkembang menjadi pupa, kemudian disaring (5c) dan dipindahkan ke sangkar sampai menjadi imago yang digunakan untuk identifikasi selanjutnya (Gambar 5d).
Gambar 5. a) Buah yang sudah busuk karena diserang Bactrocerasp, b) Pembiakan larva, c) Pupa , d) Pembentukan imago
(Dokumen pribadi) .
3. Identifikasi spesies Bactrocera secara morfologi
Imago yang keluar dari pupa, kemudian diidentifikasi secara morfologi dengan menggunakan mikroskop stereo di Laboratrium Biologi Unniversitas Negeri Semarang dengan bantuan buku pedoman identifikasi lalat buah (Suputa
et al. 2006). Hasil identifikasi menunujukan bahwa Bactrocera sp termasuk
Bactrocera sp Subgenus Zeugodacus. Langkah selanjutnya untuk keperluan A
B c
analisis molekuler dilakukan pengawetan specimen dengan menyimpan imago
Bactrocerasp dalam etanol murni. Larva instar tiga disimpan dalam alcohol 95 %. Masing-masing awetan disimpan dalam frezer -20 0 C sampai spesimen dibutuhkan untuk isolasi.
4. Identifikasi Bactrocera sp secara molekuler
Identifikasi molekuler ketiga spesies Bactrocera dilakukan di Laboratorium Virologi Unniversitas Gadjah Mada. Analisis ini dilakukan melalui beberapa langkah yaitu Isolasi DNA, elektoforesis DNA, reaksi berantai PCR, kemudian dielektoforesis kembali, dan yang terakhir metode sekuensing. Berikut ini dijelaskan langkah-langkah analisis molekuler Bactrocera sp, B. cucurbitae dan B. papayae sebagai berikut.
a. Isolasi DNA
Isolasi DNA lalat buah menggunakan sampel yang berupa imago dan larva. Masing-masing jaringan sampel dilakukan isolasi DNA genom Bactrocera sp
berdasarkan protokol genomic DNA mini kit (tissue) yang tetera pada langkah sebagai berikut:
1) Sampel imago Bactrocera sp diambil jaringan toraknya menggunakan pinset. Jika menggunakan sampel larva, maka diambil larvanya.
2) Sampel ditimbang 30 mg dan dimasukan dalam tube 3) Sampel dihaluskan menggunakan micropestle
4) Pada proses penghalusan 200 µl GT Buffer ditambahkan pada sampel kemudian dihaluskan kembali.
5) Sampel yang telah diahaluskan ditambahkan 20 µl Proteinase K dan dihomogenkan menggunakan vortex.
6) Sampel diinkubasikan pada suhu 60°C selama 30 menit, selama inkubasi tube di bolak-balik setiap 5 menit
8) Sampel diinkubasikan kembali pada suhu 60°C selama 20 menit, selama diinkubasi tube di bolak-balik setiap 5 menit, pada proses ini Elution Buffer dipanaskan (100 µl per sampel) pada 60°C.
9) Sampel disentrifugasi pada 14.000-16.000 selama 2 menit
10) Sampel yang telah disentrifugasi diambil supernatannya sebanyak 1,5 ml dan dipindahkan pada tube baru.
11) Supernatan ditambahkan ethanol absolute sebanyak 200 µl dan di shaker selama 10 detik
12) GD kolom ditempatkan pada tube 2 ml
13) Cairan yang ada pada tube 1,5 ml dipindakan ke GD kolom, setelah itu disentrifugasi pada 14.000-16.000 selama 2 menit
14) GD kolom yang telah disentrifugasi dipindahakn pada tube 2 ml yang baru 15) W1 Buffer sebanyak 400 ul ditambahkan pada GD kolom
16) GD kolom disentrifugasi 14.000-16.000 selama 30 detik, kemudian dipindahkan tube 2 ml yang baru
17) 600 µl Wash Buffer ditambahkan pada GD kolom dan disentrifuse kembali pada 14.000-16.000 selama 30 detik
18) GD kolom dipindahkan pada tube 2 ml yang baru dan disentrifuse 14.000-16.000 selama 3 menit
19) GD kolom dipindahkan pada tube 1,5 ml
20) 100 µl Elution Buffer ditambahkan pada bagian tengah tube
21) GD kolom diinkubasi pada suhu kamar selama 5 menit dan disentrifugasi 14.000-16.000 selama 30 detik
22) GD kolom yang telah disentrifugasi dibuang, sedangkan tube pada suhu -20°C/-40°C.
Keberhasilan isolasi DNA ketiga sampel Bactrocera.sp, B. cucurbitae dan
b. Elektroforesis Gel agarose
Visualisasi DNA dilakukan dengan elektroforesis pada bak elektroforesis horisontal dengan menggunakan 1,5% gel agarosa. Gel agarosa dibuat dengan melarutkan agarosa dalam bufer 1X TBE dan dipanaskan sampai tercampur homogen dan larutan menjadi jernih. Larutan agarosa kemudian dituang ke dalam bak elektroforesis yang sebelumnya telah dipasang sisir cetakan dan ditunggu sampai menjadi keras (15-20 menit). Elektroforesis dilakukan selama 45 menit pada tegangan 50 volt (lama waktu running tergantung pada konsentrasi gel dan voltase). Setelah elektroforesis, gel agarosa direndam dalam larutan Etbr selama 5 detik kemudian direndam dalam aquades selama 15 menit untuk mencuci atau meminimalkan kontaminan Etbr. Setelah itu DNA divisualisasi menggunakan UV transiluminator. Hasil visualisasi DNA genom dilihat berdasarkan keberadaan pita DNA genom sampel. Hasil isolasi yang baik dapat dilanjutkan pada metode selanjutnya yaitu amplifikasi PCR.
c. Amplifikasi PCR
Fragmen Cox I yang panjangnya 500 bp diamplifikasi dengan primer
forward mtD7 dan primer reverse mtD9. Sekuen primer oligonukleotida dapat dilihat pada Tabel 3.
Tabel 3. Primer Oligonukleotida yang digunakan untuk amplifikasi PCR Nama Primer Sekuen
Amplifikasi PCR menggunakan master mix kappa dengan total cocktail 12,5 ul dalam setengah kali reaksi yang dijelaskan pada Tabel 4.
Tabel 4. Cocktail yang digunakan untuk amplifikasi PCR
No Bahan vol x reaksi
1 5x Kappa ekstrak buffer 2,5 ul
2 Mgcl2 0,875 ul
3 ddH20 6,375 ul
4 dNtp 0,375 ul
5 DNA Polimerase 0,125 ul 6 Primer Forward 0,625 ul 7 Primer Revers 0,625 ul
Total 12,5 ul
Tabung PCR yang sudah berisi larutan tersebut dimasukkan ke dalam mesin Termal cycle yang sudah diprogram untuk 35 siklus pada kondisi yang disajikan pada Tabel 5.
Tabel 5. Rangkaian amplifikasi daerah sitokrom oksidase I DNA mitokondria
Tahapan Proses Suhu Waktu
I Denaturasi 94 3 menit
Denaturasi 94 15 detik
II Annealing 53 15 detik
III Extension 70 1 menit
Extention 72 1 menit
Tiga mikroliter sampel dari produk PCR dan dua mikroliter loading dye
d. Elektroforesis Gel Agarose
Metode elektroforesis gel agarose digunakan untuk melihat keberhasilan amplifikasi PCR gen sitokrom oksidase I menggunakan primer forward dan
reverse berdasarkan laju migrasi fragmen DNA dalam gel agarose. DNA divisualisasi menggunakan elektroforesis horisontal dengan menggunakan 1,5% gel agarosa. Setelah elektroforesis, DNA divisualisasi di dalam UV transiluminator. Hasil yang baik pada amplifikasi PCR dapat dilanjutkan pada langkah selanjutnya yaitu sekuensing untuk melihat runutan nukleotida gen sitokrom oksidase I pada Bactrocerasp, B. cucurbitae dan B. papayae.
e. Sekuensing
Produk PCR yang baik dilanjutkan dengan tahap sekuensing. Sekuensing DNA dilakukan untuk mengetahui urutan nukleotida pada daerah cox1. Sekuensing dilakukan pada Lembaga Genetica science diSingapura.
f. Analisis Data
Data hasil sekuensing berupa ABI file dari Bactrocera sp, Bactrocera papayae dan Bactrocera cucurbitae dilakukan pengeditan secara manual menggunakan program BioEdit Versi 7.0.9. Hasil pengeditan sekuen dimasukkan dalam BLAST (Basic Local Alignment Search Tool) NCBI untuk melihat homologi dengan spesies terdekat. Pohon filogeni diperoleh dengan menggunakan metode neighbor-joining, dimana kalkulasi matrik jarak genetik dengan model Kimura-2 parameter yang diimplementasikan pada pairwise distance calculation dengan Bootstrap 1000 ulangan dalam program MEGA (Molecular Evolutionary Genetics Analysis) software Versi 6.0 (Tamura et al.
2013). Sekuen sampel yang dibandingkan dengan beberapa koleksi GenBank
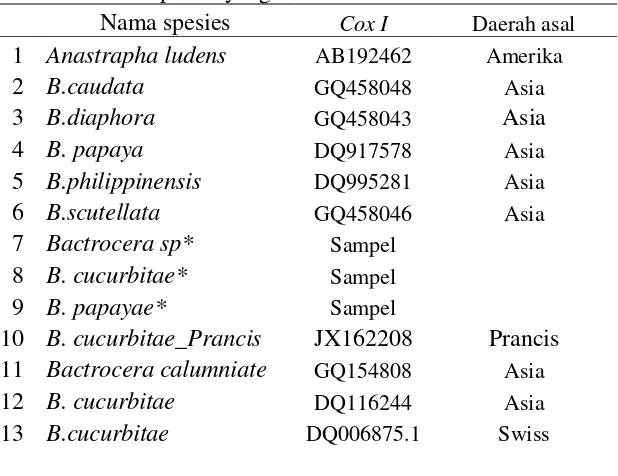
Tabel 6. Daftar spesies yang dianalisis.
Nama spesies Cox I Daerah asal
1 Anastrapha ludens AB192462 Amerika
2 B.caudata GQ458048 Asia
3 B.diaphora GQ458043 Asia
4 B. papaya DQ917578 Asia
5 B.philippinensis DQ995281 Asia
6 B.scutellata GQ458046 Asia
7 Bactrocerasp* Sampel
8 B. cucurbitae* Sampel
9 B. papayae* Sampel
10 B. cucurbitae_Prancis JX162208 Prancis
11 Bactrocera calumniate GQ154808 Asia
12 B. cucurbitae DQ116244 Asia
13 B.cucurbitae DQ006875.1 Swiss
Ket:* merupakan sampel dalam penelitian ini.
30 BAB IV
HASIL DAN PEMBAHASAN
A.Hasil Penelitian
1. Identifikasi Morfolgi pada Bactrocera yang menyerang buah liar.
Hasil identifikasi morfologi Bactrocera sp yang menyerang buah liar Famili (Cucurbitaeceae). Bactrocera sp yang ditemukan ini memiliki karakter morfologi hampir sama dengan B. calumniata tetapi mempunyai pola sayap sama dengan B. cucurbitae. Karakter morfologi tersebut berdasarkan kunci identifikasi mengacu pada pedoman Suputa et al. (2006). Hasil identifikasi Bactrocera sp
disajikan pada Lampiran 1.
2. Identifikasi Molekuler Bactrocera sp
Ekstraksi DNA sampel menggunakan metode Genomic DNA mini kit. Keberhasilannya dilihat menggunakan elektroforesis gel agarose.
a. Elektroforesis Gel Agarosa
Elektroforesis DNA hasil ekstraksi disajikan pada Gambar 6.
Gambar 6. Hasil elektroforesis genom Bactrocera. M=Marker, B. papayae
(No 1), B. cucurbitae (No 2), Larva Bactrocera sp (No 3), Imago Bactrocera sp (No 4).
Ekstraksi dengan Genomic DNA mini kit berhasil mengisolasi genom
Bactrocera sp, Bactrocera cucurbitae dan Bactrocera papayae. Hal tersebut dapat dilihat pada pita genom yang jelas, meskipun masih terdapat smear. Pada isolasi DNA genom Bactrocerasp digunakan larva Bactrocerasp karena sampel awetan imago Bactrocera sp terbatas jumlahnya dan untuk meminimalkan kekurangan sampel imago Bactrocerasp. Hasil ekstraksi ini menunjukan bahwa isolasi DNA genom berhasil dengan baik sehingga dapat digunakan untuk analisis selanjutnya yaitu Amplifikasi PCR.
b. Amplifikasi DNA Gen Sitokrom oxidase I mtDNA.
DNA sampel diamplifikasi menggunakan mesin PCR TaKaRa Thermal Cycler. Amplifikasi DNA sampel menggunakan primer forward mtD7 dan revers
mtD9 pada daerah gen cox1 Genom mitokondria Bactrocera sp, Bactrocera cucurbitae dan Bactrocera papayae menghasilkan segmen berukuran ± 500 bp (Gambar 8).
Hasil Visualisasi fragmen Cox I memperlihatkan fragmen Cox I dapat teramplifikasi menggunakan primer forward mtD7 dan primer reverse mtD9. Pita yang jelas dan tebal menunjukkan kondisi PCR yang optimal tercapai sehingga proses PCR dapat berlangsung dengan baik sehingga dapat diproses pada tahap selanjutnya yaitu Sekuensing. Pada tahap sekuensing dibutuhkan volume cocktail minimal 30 ul.
c. Hasil sekuen DNA
Hasil Sekuen gen sitokrom oksidase I Bactrocera sp diperoleh panjang ukuran gen 403 bp, B. cucurbitae 427 bp dan Bactrocera papayae 427 bp yang disajikan pada Lampiran 6. Sedangkan hasil analisis sekuen menggunakan progam Mega software 6.0 menunjukan persentase kandungan basa pada daerah
cox1Bactrocera sp, yang tercantum pada Tabel 7.
Tabel 7. Kandungan basa nukleotida Bactrocera sp dari progam Mega software 6.0.
No Nama spesies T A G C G+C A+T
1 B. cucurbitaee_Swiss* 36,6 28,8 16,0 18,6 34,6 65,4
2 Bactrocerasp** 37,2 25,7 16,6 20,5 37,0 63,0
3 B. cucurbitaee** 37,9 26,9 15,2 19,9 35,1 64,9
4 B. papayae** 35,8 29,0 15,7 19,4 35,1 64,9
5 B. caudate* 31,0 35,5 19,7 13,9 33,6 66,4
6 B. diaphora* 29,8 35,1 20,5 14,6 35,1 64,9
7 B. papaya* 34,3 39,2 10,2 16,2 26,5 73,5
8 B.philippinensis* 34.4 39,2 10,2 16,1 26,4 73,6
9 B.scutellata* 29,7 34,7 20,7 14,9 35,6 64,4
10 B.calmuniata* 36,3 28,6 16,9 18,2 35,1 64,9
11 B. cucurbitae_Ind* 36,7 27,8 16,6 18,9 35,5 64,5
12 B.cucurbitaee_Perancis* 34,4 28,3 17,9 19,4 37,3 62,7
13 Anastrepha ludens* 36,3 34,2 12,9 16,6 29,5 70,5
d. BLAST
Penentuan spesies dilakukan dengan analisis BLAST, yaitu membandingkan sekuen Bactrocera sp, B. cucurbitae dan B. papayae dengan database yang ada pada GeneBank. Hasil analisis BLAST dapat dilihat pada Tabel 8.
Tabel 8. Hasil analisis BLAST sekuen DNA gen sitokrom oksidase I Gen sitokrom oksidase I
Bactrocerasp Bactrocerasp B. cucurbitae B. papayae
Karakter CL CL CU LB Homologi 100 96% 100 100 Gaps 0% 0% 0% 0%
Acc.Number DQ006875.1 GQ154088.1 DQ116244.1 FJ903487.1 Homologi dari
nukleotida ke 284-718 bp 240-658 bp 189-613 bp 260-686 bp E-value 0.0 0.0 0.0 0.0 Ket: CL : Bactrocera sp yang menyerang buah liar.
CU : B. cucurbitae yang menyerang buah salak. LB : B. papayae yang menyerang buah pare.
Berdasarkan hasil analisis BLAST ditemukan banyak variasi nukleotida pada gen cox1 Bactrocera sp yang diteliti dengan GenBank Acc Number GQ154088.1 (Bactrocera calumniata) dengan tingkat kesamaan (homologi) yaitu 96% (Gambar 8). Bactrocera sp yang menyerang buah liar dilakukan BLAST ulang gen Cox I dengan GenBank Acc Number DQ006875.1 (B. cucurbitae) dengan tingkat kesamaan (homologi) yang tinggi yaitu 100% (Gambar 9).
Query 1 GAATGAATAATATAAGATTTTGATTATTACCTCCCTCTCTTACATTACTTTTAGTGAGCA 60 |||| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 240 GAATAAATAATATAAGATTTTGATTATTACCTCCCTCTCTTACATTACTTTTAGTGAGCA 299
Query 61 GTATAGTAGAAAACGGAGCTGGTACAGGTTGAACTGTTTATCCTCCCCTTTCATCAATTA 120 |||||||||||||||||||||||||||||||||||||||| |||||||||||||||||||
Sbjct 300 GTATAGTAGAAAACGGAGCTGGTACAGGTTGAACTGTTTACCCTCCCCTTTCATCAATTA 359
Query 121 TCGCTCATGGTGGAGCCTCAGTTGATTTAGCTATTTTTTCTCTACATTTAGCTGGTATTT 180 ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 360 TCGCTCATGGTGGAGCCTCAGTTGATTTAGCTATTTTTTCTCTACATTTAGCTGGTATTT 419
Query 181 CATCAATTTTAGGGGCCGTAAATTTCATTACTACAGTAATTAATATGCGATCAACAGGAA 240 |||||||||||||||| ||||||||||||||||||||||||||||| ||||||||||||
Sbjct 420 CATCAATTTTAGGGGCTGTAAATTTCATTACTACAGTAATTAATATACGATCAACAGGGA 479
Query 241 TCACATTTGACCGGATACCTTTATTCGTTTGAGCTGTAGTATTGACAGCTCTTCTTTTAC 300 | ||||||||||| ||||||||||||||||||||||||||||| ||||||||||||||||
Sbjct 480 TTACATTTGACCGAATACCTTTATTCGTTTGAGCTGTAGTATTAACAGCTCTTCTTTTAC 539
Query 301 TTCTATCTCTACCTGTGTTAGCCGGAGCTATTACTATACTTTTAACAGACCGAAATTTAA 360 |||||||||| || || ||||| |||||||||||||||||||||||||||||||| ||||
Sbjct 540 TTCTATCTCTCCCAGTATTAGCTGGAGCTATTACTATACTTTTAACAGACCGAAACTTAA 599
Query 361 ACACCTCTTTCTTCGACCCGGCTGGTGGTGGAGACCCTATTTTATACCAACATTTATTT 419 | ||| |||||||||||||| |||||||||||||| ||||||||||||||||| |||||
Sbjct 600 ATACATCTTTCTTCGACCCAGCTGGTGGTGGAGATCCTATTTTATACCAACACTTATTT 658
Gambar 8. Hasil analisis BLAST gen cox1Bactrocera sp dengan GenBank
Bactrocera calumniata Acc Number GQ154088.1
Keterangan : Angka sebelah kanan gambar menunjukkan urutan nukleotida, tanda : homologi (kesamaan),
Query 1 GAATGAATAATATAAGATTTTGATTATTACCTCCCTCTCTTACATTACTTTTAGTGAGCA 60 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 284 GAATGAATAATATAAGATTTTGATTATTACCTCCCTCTCTTACATTACTTTTAGTGAGCA 343
Query 61 GTATAGTAGAAAACGGAGCTGGTACAGGTTGAACTGTTTATCCTCCCCTTTCATCAATTA 120 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 344 GTATAGTAGAAAACGGAGCTGGTACAGGTTGAACTGTTTATCCTCCCCTTTCATCAATTA 403
Query 121 TCGCTCATGGTGGAGCCTCAGTTGATTTAGCTATTTTTTCTCTACATTTAGCTGGTATTT 180 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 404 TCGCTCATGGTGGAGCCTCAGTTGATTTAGCTATTTTTTCTCTACATTTAGCTGGTATTT 463
Query 181 CATCAATTTTAGGGGCCGTAAATTTCATTACTACAGTAATTAATATGCGATCAACAGGAA 240 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 464 CATCAATTTTAGGGGCCGTAAATTTCATTACTACAGTAATTAATATGCGATCAACAGGAA 523
Query 241 TCACATTTGACCGGATACCTTTATTCGTTTGAGCTGTAGTATTGACAGCTCTTCTTTTAC 300 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 524 TCACATTTGACCGGATACCTTTATTCGTTTGAGCTGTAGTATTGACAGCTCTTCTTTTAC 583
Query 301 TTCTATCTCTACCTGTGTTAGCCGGAGCTATTACTATACTTTTAACAGACCGAAATTTAA 360 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 584 TTCTATCTCTACCTGTGTTAGCCGGAGCTATTACTATACTTTTAACAGACCGAAATTTAA 643
Query 361 ACACCTCTTTCTTCGACCCGGCTGGTGGTGGAGACCCTATTTTATACCAACATTTATTTT 420 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 644 ACACCTCTTTCTTCGACCCGGCTGGTGGTGGAGACCCTATTTTATACCAACATTTATTTT 703
Query 421 GATTCTTTGGACACC 435 ||||||||||||||| Sbjct 704 GATTCTTTGGACACC 718
Gambar 9. Hasil analisis BLAST gen cox1 Bactrocera sp dengan GenBank
Bactrocera cucurbitae Acc Number DQ006875.1
Keterangan : Angka sebelah kanan gambar menunjukkan urutan nukleotida, tanda : homologi (kesamaan),
: merupakan variasi nukleotida.
Query 1 AATAATATAAGATTTTGATTATTACCTCCCTCTCTTACATTACTTTTAGTGAGCAGTATA 60 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 189 AATAATATAAGATTTTGATTATTACCTCCCTCTCTTACATTACTTTTAGTGAGCAGTATA 248
Query 61 GTAGAAAACGGAGCTGGTACAGGTTGAACTGTTTACCCTCCCCTTTCATCAATTATCGCT 120 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 249 GTAGAAAACGGAGCTGGTACAGGTTGAACTGTTTACCCTCCCCTTTCATCAATTATCGCT 308
Query 121 CATGGTGGAGCCTCAGTTGATTTAGCTATTTTTTCTCTACATTTAGCTGGTATTTCATCA 180 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 309 CATGGTGGAGCCTCAGTTGATTTAGCTATTTTTTCTCTACATTTAGCTGGTATTTCATCA 368
Query 181 ATTTTAGGGGCTGTAAATTTCATTACTACAGTAATTAATATACGATCAACAGGAATTACA 240 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 369 ATTTTAGGGGCTGTAAATTTCATTACTACAGTAATTAATATACGATCAACAGGAATTACA 428
Query 241 TTTGACCGAATACCTTTATTCGTTTGAGCTGTAGTATTAACAGCTCTTCTTTTACTTCTA 300 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 429 TTTGACCGAATACCTTTATTCGTTTGAGCTGTAGTATTAACAGCTCTTCTTTTACTTCTA 488
Query 301 TCTCTCCCAGTATTAGCTGGAGCTATTACTATACTTTTAACAGACCGAAACTTAAATACA 360 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 489 TCTCTCCCAGTATTAGCTGGAGCTATTACTATACTTTTAACAGACCGAAACTTAAATACA 548
Query 361 TCTTTCTTCGACCCAGCTGGTGGTGGAGATCCTATTTTATACCAACACTTATTTTGATTC 420 |||||||||||||||||||||||||| |||||||||||||||||||||||||||||||| Sbjct 549 TCTTTCTTCGACCCAGCTGGTGGTGGGGATCCTATTTTATACCAACACTTATTTTGATTT 608
Query 421 TTTGG 425 ||||| Sbjct 609 TTTGG 613
Gambar 10. Hasil analisis BLAST gen cox1 Bactrocera cucurbitae yang menyerang buah pare dengan Bactrocera cucurbitae GenBank Acc Number DQ116244.1
Keterangan : Angka sebelah kanan gambar menunjukkan urutan nukleotida, tanda : homologi (kesamaan),
Query 1 AATAATATAAGATTTTGATTATTACCTCCTTCCCTTACATTACTATTAGTAAGAAGTATA 60 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 260 AATAATATAAGATTTTGATTATTACCTCCTTCCCTTACATTACTATTAGTAAGAAGTATA 319
Query 61 GTAGAAAACGGAGCTGGTACAGGTTGAACAGTTTACCCACCCCTATCATCTGTTATTGCA 120 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 320 GTAGAAAACGGAGCTGGTACAGGTTGAACAGTTTACCCACCCCTATCATCTGTTATTGCA 379
Query 121 CACGGAGGAGCTTCAGTTGACCTAGCTATTTTTTCACTTCACTTAGCGGGTATTTCCTCA 180 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 380 CACGGAGGAGCTTCAGTTGACCTAGCTATTTTTTCACTTCACTTAGCGGGTATTTCCTCA 439
Query 181 ATTTTAGGAGCAGTAAATTTCATTACAACAGTAATTAATATACGATCGACAGGAATCACC 240 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 440 ATTTTAGGAGCAGTAAATTTCATTACAACAGTAATTAATATACGATCGACAGGAATCACC 499
Query 241 TTTGATCGAATACCTTTATTCGTTTGAGCAGTTGTATTAACAGCTTTATTACTTTTATTA 300 ||||||||||||||| |||||||||||||||||||||||||||||||||||||||||||| Sbjct 500 TTTGATCGAATACCTCTATTCGTTTGAGCAGTTGTATTAACAGCTTTATTACTTTTATTA 559
Query 301 TCATTACCAGTTTTAGCAGGGGCTATTACTATATTACTAACAGACCGAAACTTAAATACT 360 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 560 TCATTACCAGTTTTAGCAGGGGCTATTACTATATTACTAACAGACCGAAACTTAAATACT 619
Query 361 TCCTTTTTTGACCCTGCCGGAGGAGGAGATCCTATTCTTTACCAACATTTATTTTGATTC 420 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 620 TCCTTTTTTGACCCTGCCGGAGGAGGAGATCCTATTCTTTACCAACATTTATTTTGATTC 679
Query 421 TTTGGAC 427 ||||||| Sbjct 680 TTTGGAC 686
Gambar 11. Hasil analisis BLAST gen cox1 Bactrocera papayae yang menyerang buah salak dengan Bactrocera papayae GenBank Acc Number FJ903487.1
angka sebelah kanan gambar menunjukkan urutan nukleotida, tanda : homologi (kesamaan), : merupakan variasi nukleotida.
e. Lokasi penempelan primer
Lokasi penempelan primer MtD7 dan MtD9 pada gen sitokrom oksidase I menghasilkan ukuran fragmen ± 430 bp yang disajikan pada Gambar 12.
Around 250 bp Around 820 bp
± 1500 bp
f. Analisis Asam Amino
Alignment terhadap susunan asam amino sangat penting dilakukan mengingat adanya mutasi dari nukleotida pada daerah coding sequence, dikhawatirkan akan berpengaruh terhadap fungsi dari protein yang dibentuknya (Dale dan Park 2004). Hasil alignment susunan nukleotida terhadap sampel lainnya di GenBank dengan program Mega Software 6.0 disajikan pada Gambar 13 dan Hasil aligment asam amino pada Gambar 14.
#B.cucurbitae_JN635562 TCG CGA CAA TGG CTA TTT TCA ACG AAC CAT AAA GAT ATC
#B.cucurbitae_JN635562 GGA ACA TTA TAT TTT ATT TTC GGA GCT TGA GCA GGT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 ATA GTG GGA ACA TCT CTT AGA ATC TTA ATC CGG GCA GAA CTG #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 GGT CAC CCA GGA GCT TTA ATC GGA GAT GAT CAA ATC #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 TTC ATA GTG ATA CCT ATT ATA ATT GGA GGA TTT GGA
#B.cucurbitae_JN635562 AAT TGA CTA GTA CCC CTA ATA CTA GGA GCG CCA GAT ATA GCA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 TTC CCT CGA ATG AAT AAT ATA AGA TTT TGA TTA TTA #B.cucurbitae* --- --- --- --- ... ... ... ... ... ... ... ...
#B.cucurbitae_JN635562 CCC CCC TCT CTT ACA TTA CTT TTA GTG AGC AGT ATA GTA GAA #B.cucurbitae* ..T ... ... ... ... ... ... ... ... ... ... ... ... ...
#B.cucurbitae_JN635562 AAC GGA GCT GGT ACA GGT TGA ACT GTT TAT CCT CCC #B.cucurbitae* ... ... ... ... ... ... ... ... ... ..C ... ...
#B.cucurbitae_JN635562 CTT TCA TCA ATT ATC GCT CAT GGT GGA GCC TCA GTT GAT TTA #B.cucurbitae* ... ... ... ... ... ... ... ... ... ... ... ... ... ...
#B.cucurbitae_JN635562 ATT TTA GGG GCC GTA AAT TTC ATT ACT ACA GTA ATT AAT ATG
#B.cucurbitae_JN635562 CGA TCA ACA GGA ATC ACA TTT GAC CGG ATA CCT TTA #B.cucurbitae* ... ... ... ... ..T ... ... ... ..A ... ... ...
#B.cucurbitae_JN635562 TTC GTT TGA GCT GTA GTA TTG ACA GCT CTT CTT TTA CTT CTA #B.cucurbitae* ... ... ... ... ... ... ..A ... ... ... ... ... ... ...
#B.cucurbitae_JN635562 TCT CTA CCT GTA TTA GCC GGA GCT ATT ACT ATA CTT #B.cucurbitae* ... ..C ..A ... ... ..T ... ... ... ... ... ...
#B.cucurbitae_JN635562 TTA ACA GAC CGA AAT TTA AAC ACC TCT TTC TTC GAC CCG GCT #B.cucurbitae* ... ... ... ... ..C ... ..T ..A ... ... ... ... ..A ...
#B.cucurbitae_JN635562 GGT GGT GGA GAC CCT ATT TTA TAC CAA CAT TTA TTT #B.cucurbitae* ... ... ... ..T ... ... ... ... ... ..C ... ...
#B.cucurbitae_JN635562 GGA TTC GGT ATA ATT TCT CAT ATT ATT AGA CAA GAA
#B.cucurbitae_JN635562 TCG GGT AAA AAG GAA ACA TTT GGT TCC TTA GGT ATA ATT TAC #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 GCT ATA ATA GCA ATT GGA TTA CTT GGA TTT ATT GTA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 TGA GCC CAC CAT ATA TTT ACA GTA GGT ATA GAT GTT GAT ACT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 CGA GCT TAC TTC ACT TCA GCT ACA ATA ATT ATT GCT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 GTT CCT ACT GGA ATT AAA ATT TTC AGA TGA CTA GCC ACT CTT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 CCT ACT GGA ATT AAA ATT TTC AGA TGA CTA GCC ACT CTT CAT
#B.cucurbitae_JN635562 GGA ACA CAA TTA AAT TAT TCC CCA GCT ATG CTA TGA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 GCT TTA GGT TTT GTA TTT TTA TTC ACA GTT GGG GGA CTA ACT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 GGA GTA GTA TTA GCC AAC TCT TCT GTT GAT ATT ATT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 CTT CAT GAC ACT TAC TAC GTA GTA GCT CAT TTC CAT TAT GTT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 TTA TCA ATA GGA GCA GTA TTT GCT ATT ATA GCT GGA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 CCT AAG TGA TTA AAA AGT CAA TTT ATT ATT ATA TTT
#B.cucurbitae_JN635562 ATT GGT GTA AAC TTA ACT TTC TTC CCT CAA CAC TTC TTA GGA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 TTA GCA GGA ATA CCT CGA CGT TAC TCC GAC TAC CCA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 GAT GCT TAC ACA ACG TGA AAC GTA GTT TCT ACA ATT GGT TCA #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 TCA ATT TCT TTA CTA GGA ATT CTT TTC TTT TTA TTT #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbitae_JN635562 ATT ATT TGA GAA AGA TTA GTT ACT CAA CGT CAA GTA ATT TAC #B.cucurbitae* --- --- --- --- --- --- --- --- --- --- --- --- --- ---
#B.cucurbita_JN635562 CCA CCT GCT GAA CAT AGT TAT TCA GAA TTA CCT CTT TTA ACT
Gambar 13. Hasil aligment nukleotida dengan koleksi sampel GenBank. Ket : * merupakan sampel dalam penelitian ini.
#B.cucurbitae_JN635562 SRQWLFSTNH KDIGTLYFIF GAWAGMVGTS LSILIRAELG HPGALIGDDQ #B.cucurbitae* --- --- --- --- ---
#B.cucurbitae_JN635562 IYNVIVTAHA FVMIFFMVMP IMIGGFGNWL #B.cucurbitae* --- --- ---
#B.cucurbitae_JN635562 VPLMLGAPDM AFPRMNNMSF WLLPPSLTLL LVSSMVENGA GTGWTVYPPL #B.cucurbitae* --- ---... ... ... ...
#B.cucurbitae_JN635562 SSIIAHGGAS VDLAIFSLHL AGISSILGAV #B.cucurbitae* ... ... ...
#B.cucurbitae_JN635562 RNLNTSFFDP AGGGDPILYQ HLFWFFGHPE
#B.cucurbitae_JN635562 VYILILPGFG MISHIISQES GKKETFGSLG MIYAMMAIGL LGFIVWAHHM #B.cucurbitae* --- --- --- --- ---
#B.cucurbitae_JN635562 FTVGMDVDTR AYFTSATMII AVPTGIKIFS #B.cucurbitae* --- --- ---
#B.cucurbitae_JN635562 WLATLHGTQL NYSPAMLWAL GFVFLFTVGG LTGVVLANSS VDIILHDTYY #B.cucurbitae* --- --- --- --- ---
#B.cucurbitae_JN635562 VVAHFHYVLS MGAVFAIMAG FVHWYPLFTG #B.cucurbitae* --- --- ---
#B.cucurbitae_JN635562 LVLNPKWLKS QFIIMFIGVN LTFFPQHFLG LAGMPRRYSD YPDAYTTWNV #B.cucurbitae* --- --- --- --- ---
#B.cucurbitae_JN635562 IYPMQLSSSI EWLQNTPPAE HSYSELPLLT N--
Gambar 14. Hasil aligment asam amino dengan koleksi sampel GenBank. Ket : * merupakan sampel dalam penelitian ini.
g. Aligmant DNA
Berdasarkan neighbor - joining (NJ) (Saitou & Nei 1987 ) dengan
Bootstrap 1000x ulangan (Felsenstein 1985) diperoleh kontruksi filogeni (Nei & Kumar 2000) dari sampel Bactrocera sp yang menyerang buah liar, Bactrocera cucurbitae dan Bactrocera papayae dengan koleksi GenBank (Zhang et al. 2010) yang dimodifikasi pada Tabel 6.
B.cucurbitae_Swiss
Gambar 15. Kladogram fragmen Cox IBactrocera sp
Keterangan : * merupakan sampel dalam penelitian.
Bactrocerasp* merupakan sampel yang menyerang buah liar.
Angka pada kladogram menunjukkan nilai Bootstrap.