ANALISIS KERAGAMAN GENETIK PLASMA NUTFAH
KELAPA SAWIT (
Elaeis guineensis
Jacq.) ASAL ANGOLA
MENGGUNAKAN MARKA MIKROSATELIT
URIP SAYEKTI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Dengan ini saya menyatakan bahwa tesis yang berjudul Analisis Keragaman Genetik Plasma Nutfah Kelapa Sawit (Elaeis guineensis Jacq.) asal Angola Menggunakan Marka Mikrosatelit adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2014
Urip Sayekti
(Elaeis guineensis Jacq.) Asal Angola menggunakan Marka Mikrosatelit. Dibimbing oleh UTUT WIDYASTUTI dan NURITA TORUAN-MATHIUS.
Upaya peningkatan produktivitas dan karakter unggul lainnya dalam program pemuliaan kelapa sawit Indonesia mengalami kendala disebabkan oleh keragaman genetik yang rendah. Untuk meningkatkan keragaman genetik telah dilakukan eksplorasi plasma nutfah di Angola, Afrika Tengah. Tujuan penelitian ini adalah untuk menetapkan keragaman genetik tanaman kelapa sawit berdasarkan marka mikrosatelit pada populasi antar daerah dan antar aksesi dari kelima daerah distribusi spasial kelapa sawit asal Angola dan dibandingkan dengan keragaman populasi komersial Tenera (D x P), Dura (D) dan Pisifera (P).
Bahan tanam aksesi asal Angola yang digunakan sebanyak 27 aksesi atau 136 individu palma yang ditanam di kebun Palapa, Riau. Sementara bahan tanam populasi komersial digunakan sebanyak 34 tanaman (25 progeni) yang terdiri dari 20 tanaman (14 progeni) kelapa sawit tipe D, 1 progeni (4 tanaman) kelapa sawit tipe P dan 10 tanaman (10 progeni) kelapa sawit D x P yang berasal dari kebun Kandista dan Damimas, Riau, Sumatera. DNA diisolasi menggunakan protokol isolasi DNA kemudian diamplifikasi dengan PCR menggunakan 20 marka mikrosatelit. Analisis data dilakukan menggunakan perangkat lunak GenAlEx 6.5 untuk menganalisis keragaman alelik, Analisis Variasi Molekuler (AMOVA), analisis koefisien keragaman genetik (Gst) dan analisis persebaran individu dalam populasi menggunakan Principal Coordinate Analysis (PCoA). Perangkat lunak
Power Marker V 3.15 digunakan untuk menganalisis tingkat polimorfisme lokus
yang digunakan (PIC); dan perangkat lunak NTSYS V 2.1 digunakan untuk menganalisis dendogram kekerabatan yang dikonstruksi berdasarkan Unweighted
Pair Group Method with Arithmetic Mean (UPGMA). Seluruh analisis dilakukan
baik pada individu dalam seluruh populasi, antar populasi maupun antar subpopulasi berdasarkan daerah distribusi spasial di Angola.
Hasil penelitian menunjukkan nilai PIC 0.55 dengan 107 total alel terdeteksi. Koefisien keragaman genetik antar daerah distribusi spasial rendah antara 0.01 sampai 0.13. AMOVA menunjukkan bahwa sumber keragaman genetik terbesar disumbangkan oleh keragaman genetik antar individu baik di dalam aksesi maupun di dalam subpopulasi berdasarkan daerah distribusi spasial. Analisis kelompok UPGMA membagi seluruh individu menjadi enam kelompok pada koefisien kesamaan genetik 0.42 sampai 0.96 dimana pengelompokan tidak berdasarkan aksesi dan daerah distribusi spasial. Analisis sebaran individu dalam populasi menunjukkan bahwa sebagian individu dalam subpopulasi cenderung menyebar pada empat kuadran dan sebagian mengelompok pada dua kuadran. Individu D x P, D dan P membentuk kelompok yang terpisah dengan populasi asal Angola.
Populasi hasil eksplorasi asal Angola memiliki nilai parameter-parameter keragaman genetik yang lebih tinggi. Populasi tersebut memiliki potensi untuk diseleksi sebagai tetua pada skema program pemuliaan kelapa sawit saat ini.
Oil Palm (Elaeis guineensis Jacq.) Using Microsatellite Marker. Supervised by UTUT WIDYASTUTI and NURITA TORUAN-MATHIUS.
The effort to increase productivity and other elite characters of Indonesia oil palm breeding program is facing a problem because of the narrow of genetic diversity. To widen the genetic diversity, germplasm exploration has been done in Angola, Central Africa. The objectives of this research is to assess the genetic diversity of oil palm Angola-originated between accessions and spatial distribution regions compared to the commercial population of Tenera (D x P), Dura (D) and Pisifera (P) based on microsatellite markers. The Angola-originated plant materials used were 27 accessions consist of 136 palm planted in Palapa Estate Riau, Sumatera. And commercial population plant materials used were 34 palms (25 progenies) consist of 20 palms (14 progenies of D palm), 4 palms (1 progeny of P palm and 10 palms (10 progenies) of D x P palms planted in Kandista and Damimas Estate, Riau, Sumatera. DNA has been isolated and amplified using PCR based on 20 SSR markers. Data analysis has been done using GenAlEx 6.5 software to analyze allelic diversities, Analysis of Molecular Variance (AMOVA), Genetic diversity coefficient (Gst), and the individuals spread in the populations using Principal Coordinates Analysis (PCoA). Power Marker V 3.15 software has been used to analyze the level of loci polymorphism (PIC); and NTSYS ver. 2.1 has been used to analyze the genetic relationship based on Unweighted Pair Group Method with Arithmetic Mean (UPGMA). All the analysis has been used either on individual in all populations, between populations or between subpopulations based on spatial distribution region in Angola.
The result shows that the PIC value is 0.55 with 107 total numbers of alleles. Genetic diversity coefficient between spatial distribution regions is range from 0.01 to 0.13. AMOVA shows that the sources of highest diversity is given by diversities between individuals either in the subpopulations or based on spatial distribution regions. UPGMA grouping analysis has dividing all individuals into six groups on 0.42 to 0.96 similarity coefficient. The grouping of individuals is not based on the accessions and spatial distribution regions. The individuals spread analysis shows that a part of individuals tend to spread on four quadrants and part of them tend to agglomerate only on two quadrants. The D x P, D and P individuals tend to separate from the Angola-originated individuals.
The Angola-originated population has all the genetic diversities parameter values compared to the commercial population D x P, D and P. The Angola-originated population has the potencies to be selected as a parent material on the current oil palm breeding program scheme.
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB.
MENGGUNAKAN MARKA MIKROSATELIT
URIP SAYEKTI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
NRP : P051110151
Disetujui oleh,
Komisi Pembimbing
Dr Ir Utut Widyastuti Suharsono, MSi Dr Nurita Toruan-Mathius, MS
Ketua Anggota
Diketahui oleh
Ketua Program Studi Bioteknologi Dekan Sekolah Pascasarjana IPB
Prof Dr Suharsono, DEA Dr Ir Dahrul Syah, MscAgr
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Mei 2013 sampai Februari 2014 ini adalah keragaman genetik kelapa sawit dengan judul Analisis Keragaman Genetik Kelapa sawit asal Angola Menggunakan Marka Mikrosatelit. Sumber dana penelitian ini berasal dari PT SMART Tbk, Divisi Plant Production and
Biotechnology. Sebagian hasil penelitian ini sedang dalam proses publikasi di
Jurnal Agronomi Indonesia (JAI).
Terima kasih penulis ucapkan kepada Ibu Dr Ir Utut Widyastuti, MSi dan Ibu Dr Nurita Toruan-Mathius, MS selaku pembimbing yang telah banyak memberi arahan dan masukan mulai dari awal sampai akhir. Penghargaan dan terima kasih juga penulis sampaikan kepada Bapak Dr Ir Miftahudin, MSi atas kesediaannya menjadi penguji luar komisi, terima kasih kepada Bapak Prof Suharsono, DEA selaku Ketua Program Studi PS Bioteknologi.
Selain itu, penulis juga menyampaikan terima kasih kepada Bapak Jo Daud Dharsono selaku Head of Upstream PT SMART Tbk yang telah memberi izin dan kesempatan penulis untuk melanjutkan studi, Bapak Dr Tony Liwang selaku Division Head dan Ibu Lisa Muliani yang telah memberikan motivasi dan dukungan selama studi. Terima kasih juga penulis sampaikan kepada Bapak Yong Yit Yuan dan timnya Bapak Arnolly S. Ardi dan Bapak Yopy Dedywiryanto selaku Plant Breeder SMARTRI yang telah membantu dan mengarahkan pada proses pengambilan sampel daun. Terima kasih kepada Bapak Hairinsyah, MSi dan Bapak Hotdi atas bantuan dan izin dalam pemilihan dan pengambilan sampel tanaman komersial. Disamping itu penulis juga menyampaikan penghargaan kepada rekan staf khususnya Andree Sunanjaya Kusnandar, Hadi Septian Guna Putra dan Yogo Adhi Nugroho, seluruh staf Tissue culture dan Bioteknologi, karyawan Bioteknologi, Plant Production and Biotechnology Division PT SMART Tbk yang telah membantu dalam proses penelitian, pengumpulan dan analisis data. Terima kasih kepada seluruh sahabat dan rekan-rekan khususnya Bioteknologi 2011 atas kebersamaan selama ini.
Ungkapan terima kasih yang tak berhingga juga disampaikan kepada suami tercinta Amir Hamzah, STP, ayahanda Ngamin (Alm), ibunda Sugiyanti, kakak Slamet Widodo serta seluruh keluarga, atas doa, kesabaran, motivasi, dukungan dan kasih sayang yang tak terbalaskan; semoga selalu dalam rahmat dan lindungan Allah SWT. Amin.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2014
DAFTAR TABEL xiii
DAFTAR GAMBAR xiv
DAFTAR LAMPIRAN xv
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Hipotesis 2
2 TINJAUAN PUSTAKA 3
Asal dan Sejarah Kelapa Sawit 3
Geografi Zona 3
Budidaya Kelapa Sawit di Angola 4
Botani Kelapa Sawit 5
Marka Molekuler pada Kelapa Sawit 7
Analisis Keragaman Genetik Kelapa Sawit menggunakan Marka SSR
8
3 BAHAN DAN METODE 9
Tempat dan Waktu Penelitian 9
Bahan Tanaman 9
Pengambilan Sampel 10
Analisis Molekuler 11
Isolasi DNA 11
Penentuan Kualitas dan Kuantitas DNA 12
Amplifikasi DNA dengan PCR 12
Seleksi Primer Mikrosatelit 12
Optimasi Suhu Annealing 13
Elektroforesis DNA Hasil Amplifikasi 13
Analisis Data Molekuler 14
Skoring Data 14
Profil Hasil Karakterisasi Marka SSR 16 Analisis Tingkat Heterozigositas dan Analisis Polimorfisme 16 Analisis Variasi Molekuler (Analysis of Molecular Variance –
AMOVA)
17
Analisis Keragaman dan Kemiripan Genetik Individu 17 Analisis Koordinat Utama (Principal Coordinate Analysis –
PCoA)
17
4 HASIL DAN PEMBAHASAN 18
Isolasi DNA 18
Suhu Annealing optimum 18
(AMOVA)
Analisis Kelompok berdasarkan UPGMA populasi plasma nutfah asal Angola dan populasi D x P, D dan P
30
Analisis Kelompok berdasarkan UPGMA pada populasi asal Angola 31 Analisis Kelompok berdasarkan UPGMA pada populasi D x P, D dan P
32
Analisis Koordinat Utama/AKOORDA atau Principal Coordinate
Analysis (PCoA)
2 Daftar primer yang digunakan dalam percobaan 15 3 Suhu annealing optimum dan rata-rata panjang alel pada 20 lokus
mikrosatelit
20
4 Rata-rata jumlah alel, klasifikasi, tingkat heterozigositas dan polimorfisme 20 lokus mikrosatelit
21
5 Beberapa nilai peubah keragaman pada 20 lokus mikrosatelit 22 6 Klasifikasi alel berdasarkan frekuensi munculnya alel di dalam lokus
pada populasi plasma nutfah asal Angola dan populasi D x P, D dan P
24
7 Beberapa parameter keragaman genetik berdasarkan populasi asal Angola dan D x P, D dan P
24
8 Klasifikasi alel berdasarkan pada frekuensi alel 26 9 Beberapa parameter keragaman berdasarkan lima daerah distribusi
spasial dibandingkan dengan populasi D x P, D dan P
26
10 Matriks koefisien keragaman genetik (Gst) (Nei 1972) antar lima daerah distribusi spasial dan satu populasi D x P, D dan P
27
11 Pembagian total nilai keragaman genetik berdasarkan marka mikrosatelit pada aksesi-aksesi tipe liar asal Angola dan kelompok tipe D x P, D dan P berdasarkan AMOVA
29
12 Pengelompokan 170 individu berdasarkan daerah distribusi spasial dan tipe D, P dan D x P
31
1 Tipe Vegetasi di Angola 5 2 Peta Angola yang menunjukkan daerah eksplorasi kelapa sawit 10 3 Proses pemisahan DNA sampel pada elektroforesis menggunakan
sistem QIAxcel
16
4 Elektropherogram QIAxcel (atas) pita-pita SSR pada primer spesifik (atas) lokus mEgCIR3519 dan primer tidak spesifik (bawah) lokus mEgCIR0905 a dan b pada beberapa individu plasma nutfah kelapa sawit asal Angola
19
5 Dendogram UPGMA berdasarkan koefisien DICE untuk 170 individu dari populasi plasma nutfah asal Angola dan populasi D x P, D dan P berdasarkan pada jarak genetik Nei (1972)
33
6 Dendogram UPGMA berdasarkan koefisien DICE untuk 136 individu dari populasi plasma nutfah asal Angola berdasarkan pada jarak genetik Nei (1972)
34
7 Dendogram UPGMA berdasarkan koefisien DICE untuk 34 individu dari populasi D x P, D dan P berdasarkan pada jarak
genetik Nei (1972)
35
8 Posisi relatif 136 individu kelapa sawit tipe liar asal Angola, 20 individu tipe D, 10 individu tipe D x P dan 4 individu tipe P yang dipetakan pada keempat sumbu koordinat berdasarkan koordinat 1 dan 2
36
9 Posisi relatif individu-individu kelapa sawit berdasarkan daerah distribusi spasial dan tipe D x P, D, P yang dipetakan pada keempat sumbu koordinat
36
DAFTAR LAMPIRAN
1 Daftar aksesi kelapa sawit asal Angola 43
2 Daftar sampel tanaman D, P, dan D x P 44
3 Visualisasi hasil isolasi DNA daun muda kelapa sawit 45 4 Gambar hasil elektroforesis 20 primer mikrosatelit 50 5 Daftar kepemilikan alel masing-masing daerah distribusi spasial
setiap lokus
50
6 Nilai akar ciri dan vektor ciri pada tiga koordinat utama 170 individu kelapa sawit asal Angola dan D x P, D dan P
51
7 Reagen DNA genomik dari tanaman Kit NucleoSpin Plant IITM
Miniprep untuk 50 preps
52
8 Reagen Kimia untuk PCR Polymerase Chain Reaction yang digunakan untuk Master Mix DNA Genomik Kelapa Sawit untuk 1 x reaksi (15 µL) PCR
1 PENDAHULUAN
Latar Belakang
Program pemuliaan tanaman kelapa sawit mengakibatkan keragaman genetik menjadi lebih sempit. Untuk program seleksi yang efektif diperlukan sumber keragaman genetik yang luas. Kelapa sawit memiliki sifat reproduktif menyerbuk silang yang berimplikasi pada terdapatnya keragaman genetik yang cukup besar dalam populasinya. Ketersediaan sumber genetik baru dapat mendorong program pemuliaan kelapa sawit menjadi lebih efektif (Hardon et al. 1985).
Salah satu cara untuk memperluas keragaman genetik tanaman kelapa sawit adalah mencari sumber plasma nutfah baru melalui program eksplorasi. Beberapa karakter penting yang perlu diintroduksikan ke dalam program pemuliaan tanaman kelapa sawit saat ini diantaranya adalah kemampuan adaptasi pada lingkungan khusus seperti ketahanan terhadap kekeringan dan ketahanan terhadap penyakit diantaranya busuk pangkal batang yang disebabkan oleh cendawan Ganoderma boninense, pertumbuhan vegetatif yang lambat, tangkai buah yang panjang serta kandungan beta-karoten dan iodine mesokarp yang tinggi (Hartley 1988). Eksplorasi sumber gen baru pada tanaman kelapa sawit tipe liar asal Afrika sebagai sumber plasma nutfah telah dilakukan oleh beberapa peneliti dari Malaysia (Rajanaidu 1994ab), dan di Indonesia dilakukan oleh Indonesia
Palm Oil Board (IPOB).
Daerah Guinea sampai Angola diketahui sebagai daerah asal kelapa sawit, sehingga Angola menjadi salah satu negara tujuan eksplorasi yang berpotensi menghasilkan sumber gen baru. Walaupun di negara Angola itu sendiri tidak menjadi produsen utama kelapa sawit di dunia karena kelapa sawit tidak dibudidayakan secara intensif yang disebabkan oleh kondisi politik negara yang mengalami perang saudara dari tahun 1975 sampai 2002 dan pembangunan baru dimulai tahun 2006. Eksplorasi IPOB di Angola memperoleh 127 aksesi terpilih berdasarkan kriteria ketinggian tempat, tipe iklim, suhu dan curah hujan (Tirtawinata et al. 2010). Selama proses penyiapan bahan tanam di sebuah perusahaan perkebunan swasta nasional, tersisa hanya 48 aksesi yang telah ditanam di kebun plasma nutfah dan sedang dalam proses karakterisasi fenotipik.
Karakter fenotipik merupakan manifestasi dari faktor genetik, faktor lingkungan serta interaksi antara faktor genetik dan lingkungan. Oleh karena itu perlu dilakukan karakterisasi secara genetik untuk melengkapi karakterisasi fenotipik. Analisis genetik dalam upaya karakterisasi genetik dilakukan dengan menggunakan marka molekuler (DNA) sehingga analisis menjadi lebih mudah (Chakravarthi dan Naravaneni 2006). Marka molekuler merupakan alat yang sangat baik bagi pemulia dan ahli genetik untuk menganalisis genom tanaman berkaitan dengan keragaman genetik, klasifikasi plasma nutfah, dan sebagai alat bantu seleksi dalam program pemuliaan melalui penandaan gen. Kegiatan seleksi menjadi lebih efektif dan efisien karena seleksi hanya didasarkan pada sifat genetik tanaman, tidak dipengaruhi oleh faktor lingkungan (Azrai 2006).
basa nukleotida yang tersebar di dalam genom eukariot (Delseny et al. 1983). SSR mempunyai polimorfisme yang tinggi, sedikit variasi di sejumlah pengulangan
(repetability) (Amos et al. 1996). SSR menunjukkan pewarisan kodominan, yaitu
dapat membedakan homozigot dan heterozigot, serta bersifat spesifik (satu lokus setiap pasangan primer). SSR memiliki tingkat polimorfisme lebih tinggi dibandingkan dengan marka RFLP, RAPD dan AFLP (Powell et al. 1996).
Penelitian keragaman genetik kelapa sawit menggunakan marka mikrosatelit pertama kali dilakukan oleh Billotte et al. (2001). Selanjutnya marka tersebut dikembangkan secara luas untuk studi keragaman genetik plasma nutfah baru hasil introduksi dari Afrika, diantaranya dari Nigeria (Zulhermana et al.
2010), dan Kamerun (Ajambang et al. 2012). Salah satu perusahaan perkebunan swasta nasional juga telah menggunakan marka SSR terseleksi hasil penelitian Billotte et al. (2005) untuk membuat profil sidik jari DNA dari ortet kelapa sawit Tenera yang berasal dari origin Deli-AVROS.
Pada penelitian ini digunakan 20 primer mikrosatelit yang tersebar pada 16 kromosom yang berasal dari penelitian Billotte et al. (2005). Primer-primer ini telah dioptimasi oleh Hatorangan et al. (2010) dan Kusnandar et al. (2010) menghasilkan pita polimorfik pada kelapa sawit Tenera dan Kamerun yang berasal dari kebun percobaan yang berlokasi di Riau.
Tujuan penelitian
Tujuan penelitian ini adalah menetapkan keragaman genetik tanaman kelapa sawit berdasarkan marka mikrosatelit untuk:
(i) populasi antar daerah dan antar aksesi dari kelima daerah distribusi spasial hasil eksplorasi kelapa sawit di Angola dan dibandingkan dengan keragaman populasi komersial
(ii) individu di dalam populasi dari masing-masing daerah berdasarkan distribusi spasial di Angola
(iii) antar individu dari 170 tanaman kelapa sawit populasi asal Angola dan populasi D x P, D dan P.
Hipotesis
Terdapat keragaman genetik:
(i) populasi antar daerah distribusi spasial dan terdapat keragaman genetik antar aksesi dari kelima daerah distribusi spasial hasil eksplorasi kelapa sawit asal Angola serta keragaman genetik antara populasi kelapa sawit asal Angola dengan populasi D x P, D dan P
(ii) individu di dalam populasi dari masing-masing daerah distribusi spasial di Angola
2 TINJAUAN PUSTAKA
Asal dan Sejarah Kelapa SawitKelapa sawit saat ini dibudidayakan atau tumbuh liar di daerah ekuator dan tropis Afrika, Asia Tenggara dan Amerika Tengah. Kelapa sawit berasal dari daerah yang terletak antara Guinea dan Angola di Afrika Tengah. Terdapat tiga hal yang memperkuat pendapat tersebut yang ditinjau dari peristiwa sejarah, bahasa dan geologi (Mansjoer 1980). Elaeis berasal dari bahasa Yunani elaion yang berarti minyak dan guineensis yang berarti Guinea yang menunjukkan asal kelapa sawit dari Guinea (Pamin 1998).
Kelapa sawit tipe liar dan semi liar ditemukan tumbuh di sepanjang pantai dari daerah belahan Utara Senegal melalui Sierra Leone, Liberia, Pantai Gading, Ghana, Togo, Benin, Nigeria, Kamerun, Republik Rakyat Kongo, Angola sampai di belahan Selatan Republik Demokratik Kongo. Jajaran ini sedikit berada di Afrika Barat tetapi menjadi menyebar di Afrika Tengah dimana pusat asal dan persebaran kelapa sawit terkonsentrasi di hutan tropis Nigeria, Kamerun, Kongo dan Angola (Corley dan Tinker 2003).
Pedagang Eropa pada mulanya mengumpulkan biji kelapa sawit dari Afrika dan menanamnya di Amsterdam Botanic Garden, Belanda dan kemudian memindahkannya ke Kebun Raya Bogor Indonesia pada masa penjajahan Belanda pada tahun 1848. Empat palma ditanam di Bogor kemudian menjadi stok bibit untuk perkembangan industri kelapa sawit dunia. Keempat palma tersebut sama dan semuanya adalah Dura dan telah digunakan sebagai tanaman hias sampai saat ini.
Populasi tipe liar kelapa sawit dari Angola diyakini mengandung beberapa sifat atau karakter yang menarik yang disebabkan oleh persebaran wilayah dan kondisi lingkungan fisik. Dengan kondisi ini dimungkinkan akan dihasilkan plasma nutfah yang bermanfaat bagi program pemuliaan kelapa sawit Indonesia.
Geografi zona
Gambaran paling baru dari penyebaran kelapa sawit dipaparkan oleh Schad (1914) Dalam Corley dan Tinker (2003), diikuti oleh Zeven (1967). Dimulai dari sebagian besar daerah Utara di sepanjang pantai Barat Afrika, konsentrasi pertama kelapa sawit adalah terdapat pada dataran tinggi dari distrik Fouta Djallon dari Guinea, pada 10-11° lintang Utara. Daerah kelapa sawit Afrika kemudian berjalan melalui Sierra Leone, Liberia, Pantai Gading, Ghana, Togoland, Benin, Nigeria, Kamerun, Republik Rakyat Kongo dan Republik Demokratik Kongo (Zaire). Di Afrika Barat daerah kelapa sawit sedikit, karena penurunan yang drastis curah hujan di bagian Utara, tetapi di ekuatorial Afrika lebih banyak tersebar. Hal ini, di Nigeria tidak terdapat kelapa sawit semi liar pada 7° lintang Utara kecuali pada bagian yang mendukung area dimana terdapat daerah aliran air yang dangkal seperti dekat dataran tinggi Jos (Corley dan Tinker 2003).
ditemukan di perbatasan Kongo-Uganda, antara Danau Kivu dan Danau Tanganyika pada belakang pantai Timur dan tersebar sepanjang pantai (Corley dan Tinker 2003).
Republik Angola terletak di Pantai Barat Afrika dan Selatan ekuator. Angola merupakan negara terbesar di Afrika bagian Selatan dengan luas wilayah 1,246,700 km. Angola terbentang kira-kira 1,320 km dari Utara sampai Selatan antara 5°52' lintang Utara dan 18°00' lintang Selatan, dan 1,365 km dari Timur ke Barat E-W antara 11°41' bujur Barat dan 24°05' bujur Timur. Di sebelah Utara, Angola berbatasan dengan Republik Demokrasi Kongo (dulu Zaire), di bagian Timur berbatasan dengan Zambia, di sebelah Selatan dengan Namibia dan di sebelah Barat adalah garis pantai Atlantik sepanjang 1,650 km (The Government of
Angola 2001).
Secara umum Angola dapat dibagi menjadi tiga wilayah besar dimulai dari Barat: (1) daerah pantai yang relatif sempit terbentang mulai dari pantai sampai perbukitan dengan ketinggian 400 m dpl yang bervariasi lebarnya mulai dari 10 m hingga ratusan km, (2) sub dataran dengan berbagai macam lansekapdan (3) di bagian Timur merupakan dataran pedalaman yang dikelilingi oleh dataran tinggi yang dapat diklasifikasikan sebagai pegunungan. Dengan bentangan lokasi Angola di 5 ºC sampai 18ºC dan dataran 1,500 m dpl atau lebih tinggi (The Government of
Angola 2001).
Angola mempunyai lansekap hutan khatulistiwa yang padat, lahan tertutup semak belukar dan padang rumput, sejauh hamparan kosong Namibe dimana formasi tanaman tumbuh dengan kuat dan spesies lain yang tumbuh dari kelembaban kabut pesisir pantai. Iklim di Angola, yaitu tropis di belahan Utara dan subtropik di bagian Selatan, serta temperate di daerah hamparan kosong dataran tinggi. Terdapat dua perbedaan iklim panas dan lembab di daerah dengan curah hujan lebih tinggi dan lainnya lebih dingin dan lebih kering disebut sebagai
“cacimbo” atau iklim kering.
Vegetasi di Angola meliputi dataran pantai yang kering antara Sumbe dan sungai Cunene, membentang dari bawah sampai dataran tinggi di atas pada ketinggian 200 m sampai 500 m yang berupa padang rumput stepa didominasi oleh
Aristida papposa, Schmidtia kalahariensis dan Setaria sp tetapi sebagian besar
Acacia heteracantha, A. quintanilhae, Colophospermum mopane dan tanaman
berduri Terminalia prunioides dengan Crotalaria sp., Indigofera daleoides dan
Cassia italica daerah yang lebih rendah. Sabana dengan berbagai macam tingkat
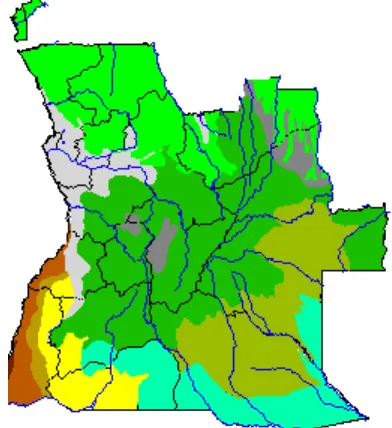
kerapatan didominasi oleh Brachystegia dan Julbernardia spp menutupi sebagian besar daerah dataran di pedalaman di wilayah Selatan dan Timur. Sementara hutan tertutup terletak di lembah sungai. Akan tetapi di sisi lembah yang lebih tinggi hutan ini umumnya didukung oleh sabana. Vegetasi pesisir Cabinda terutama sabana berumput dengan hutan lebat di perbukitan rendah pedalaman (Gambar 1).
Budidaya Kelapa Sawit di Angola
menggunakan ekstrak minyak kelapa sawit untuk konsumsi sendiri yaitu untuk memasak atau dijual di pasar tradisional.
Gambar 1 Tipe Vegetasi di Angola (Sumber: FAO 2014)
Beberapa daerah di Angola pernah memiliki perkebunan kelapa sawit tetapi telah ditinggalkan selama perang sipil yang berakhir tahun 2002. (Tirtawinata et al. 2010). Total area kelapa sawit yang dipanen di Angola hanya sekitar 0.058 juta hektar atau 58,000 hektar pada tahun 2010-2012 (USDA 2013).
Botani Kelapa Sawit
Dalam sistem kekerabatan, kelapa sawit diklasifikasikan sebagai berikut: Divisi : Spermatophyta
Kelas : Monocotyledonae
Ordo : Arecales
Famili : Arecaceae
Genus : Elaeis
Spesies : Elaeis guineensis Jacq.
Tanaman kelapa sawit merupakan tanaman palma monoesius yang termasuk dalam subfamili Cocoidae bersama dengan tanaman kelapa. Tanaman ini berasal dari Afrika dan Amerika. Genus Elaeis setidaknya memiliki empat spesies yang telah dideskripsikan, yaitu E. guineensis, E. oleifera, E. odora, dan E.
madagascariensis. E. oleifera dan E. odora dapat dijumpai di Amerika Selatan
sebagai daerah asal dari kedua spesies tersebut, sedangkan E. guineensis dan E.
madagascariensis berasal dari Afrika (Latiff 2000; Corley dan Tinker 2003).
Hanya dua spesies dari genus Elaeis yang dibudidayakan sebagai tanaman penghasil minyak, yaitu E. guineensis dan E. oleifera. Spesies yang umum dibudidayakan di Asia Tenggara adalah E. guineensis sedangkan E. oleifera
dibudidayakan di Amerika latin. E guineensis dikenal sebagai kelapa sawit
Mosaic of dry forest and grassland Wetter Zambezian woodland
Lowland rainforest and grassland
Bushland and thicket mosaics
Scrub woodland
North Zambezian woodland
Grassland on Kalahari sand
Woodland and shrubland Semi desert shrubland
komersial yang memiliki keunggulan utama pada kandungan minyak mentah
(Crude Palm Oil, CPO) yang tinggi namun kandungan asam lemak tak jenuh
sangat rendah antara 40% sampai 60%. Sedangkan E. oleifera memiliki kandungan CPO sangat rendah, tetapi memiliki persentase asam lemak tak jenuh sangat tinggi antara 70% sampai 83% dan pertumbuhan batang yang lambat (Irwansyah 2004).
Kelapa sawit tumbuh baik pada iklim tropis dengan rataan suhu minimum antara 20 oC sampai 23 oC dan suhu maksimum antara 28 oC sampai 32 oC, curah hujan sebanyak 2,000 mm/tahun, serta panjang penyinaran minimum 5 jam per hari. Kelembaban atmosfir dan konsentrasi CO2 yang tinggi juga diduga menjadi
faktor sangat penting untuk pertumbuhan kelapa sawit (Latiff 2000; Henson dan Chang 2000). Kelapa sawit dibudidayakan pada lahan bertopografi datar pada ketinggian lahan antara 0 sampai 600 mdpl. Kelapa sawit dapat tumbuh pada rentang pH tanah antara 4.0 sampai 6.5 dengan pH optimum pada kurang lebih 5.0 sampai 5.5.
Oleh karena sifat reproduktifnya yang menyerbuk silang dengan bantuan serangga atau angin, terdapat keragaman genetik yang cukup besar dalam populasi tanaman kelapa sawit. Beberapa fenotipe berkaitan dengan hasil panen yang diwariskan secara monogenik antara lain karakter warna eksokarp dan ketebalan cangkang biji. Kedua karakter ini merupakan karakter utama penciri kelompok genotipe berkaitan dengan daya hasil. Karakter warna berhubungan dengan kualitas panen, yaitu kandungan karotenoid. Sedangkan karakter ketebalan biji berhubungan dengan proporsi kandungan minyak mentah atau CPO (Latiff 2000). Kelapa sawit komersial dikenal dalam tiga kelompok, yaitu Dura, Pisifera, dan Tenera. Pengelompokan ini didasarkan pada ketebalan cangkang biji yang merupakan pewarisan sifat monogenik. Kelompok Dura (DD) memiliki cangkang tebal, dengan ketebalan antara 2 sampai 8 mm, kandungan mesokarp rendah sampai menengah dari kisaran 35 hingga 55%, di bagian luar tidak terdapat lingkaran sabut.
Tenera (Dd) memiliki ketebalan cangkang antara 0.5 sampai 4 mm dengan kandungan mesokarp menengah sampai tinggi antara 60% sampai 96%, terdapat lingkaran sabut pada bagian luarnya. Sedangkan Pisifera (dd) memiliki ketebalan cangkang sangat tipis atau bahkan tidak ada, bunga betina steril, buah gugur prematur, serta memiliki seks rasio lebih tinggi dibandingkan dengan Dura dan Tenera (Latiff 2000; Corley dan Tinker 2003).
Seperti halnya pada tanaman palma yang lain, pertumbuhan awal kelapa sawit setelah tahap bibit melibatkan formasi batang basal yang besar tanpa pemanjangan internodus. Terdapat sangat sedikit pertumbuhan ke atas pada tiga tahun pertama. Pada mulanya internodus mulai memanjang dan terbentuk batang
columnar. Walaupun masing-masing ruas batang mungkin dideskripsikan sebagai
internodus dan daun, node hanya mengindikasikan eksternal pada palma yang tua
dengan bekas luka daun; secara internal tidak terdapat batas antara internodus
yang berdekatan (Corley dan Tinker 2003).
berlanjut dari tunas daun atau primordial terpisah secara lateral dari meristem apikal. Perkembangan dari daun dimulai sangat lambat. Terdapat 40 sampai 60 daun dengan tunas apikal masing-masing tetap bersisa kira-kira dua tahun kemudian secara cepat berkembang ke dalam „spear‟ tengah dan akhirnya
membuka (Corley dan Tinker 2003).
Akar kelapa sawit dapat mencapai jarak horizontal 16 m dan jarak vertikal 8 m pada tanah dengan drainase yang baik. Akar sekunder berkembang dari akar primer dan akar tersier berkembang pada akar sekunder membentuk jaringan sistem perakaran. Absorpsi maksimum akar lebih dari 0.5 m di atas permukaan tanah dan pada radius 3.5 sampai 4.5 m di sekitar pohon dengan tanpa jaringan lignin pada ujung akar primer, sekunder dan tersier (Corley dan Tinker 2003).
Bunga kelapa sawit berumah satu dimana bunga jantan dan betina terpisah pada tanaman yang sama. Akan tetapi investigasi yang rinci pada bunga telah menunjukkan bahwa masing-masing primordial bunga termasuk organ jantan dan betina mampu menghasilkan bunga hermaprodit. Calon bunga mulai berinisiasi di dalam axil pada setiap daun tetapi beberapa infloresen mengalami aborsi sebelumnya. Sebuah infloresen biasanya memiliki kira-kira 250 spikelet dan masing-masing spikelet dapat memiliki antara 12 sampai 30 bunga untuk bunga betina. Bunga betina siap dibuahi pada kira-kira 36 sampai 48 jam. Bunga jantan terdiri dari 100,000 bunga masing-masing. Spikelet bunga jantan mulai membuka antara 2 hingga 4 hari dan bunga jantan viabel setelah enam hari (Corley dan Tinker 2003).
Agen penyerbuk utama pada kelapa sawit adalah serangga dari subfamili
Derelominae, yaitu spesies Elaeidobius kamerunicus yang ditemukan di Kamerun
dan sedikit di Malaysia. Selain itu juga terdapat agen-agen penyerbuk yang lain, yaitu angin, hujan dan serangga (Thrips hawaiiensis dan Mystrops costaricensis). Satu perkembangan yang mengejutkan adalah introduksi E. kamerunicus sebagai agen penyerbuk utama di Malaysia, Indonesia, Papua New Guinea dan Colombia. Keberadaan E. kamerunicus mampu meningkatkan hasil hingga 35% (Corley dan Tinker 2003).
Marka Molekuler pada Kelapa Sawit
Marka molekuler mempunyai akurasi yang tinggi dibandingkan dengan marka morfologi karena tidak terpengaruh dengan lingkungan. Selain itu marka ini dapat digunakan pada semua stadia pertumbuhan tanaman dan sangat membantu dalam program pemuliaan karena dapat mereduksi waktu. Marka molekuler telah banyak digunakan pada kelapa sawit. RAPD telah digunakan sebagai alat untuk memverifikasi hibrida kelapa sawit dari kultur embrio zigotik setengah dewasa (Thawaro dan Te-chato 2008). Syukri (2012) melakukan studi keragaman genetik menggunakan RAPD pada kelapa sawit asal Jawa Barat yang diduga mempunyai ketahanan terhadap Ganoderma.
Barcelos et al. (2002) meneliti hubungan dan keragaman genetik antara kelapa sawit asal Amerika dan Afrika dengan RFLP dan AFLP. Singh et al. (2008) juga menggunakan marka Restriction Fragment Length Polimorphism (RFLP) yang berasal dari klon-klon cDNA yang dibuat dari analisis genom kelapa sawit untuk menguji 321 kelapa sawit Tenera hasil selfing. Peta Quantitative Trait Loci
mesocarp dapat dibuat dari marka ini (Oil to Wet Mesocarp atau O/WM). Penelitian lain dilakukan oleh Maizura et al. (2006) menggunakan marka RFLP untuk menguji keragaman genetik kelapa sawit sebanyak 359 aksesi asal 11 negara-negara di Afrika, yaitu Nigeria, Kamerun, Kongo, Tanzania, Angola, Senegal, Sierra Leone, Guinea, Ghana, Madagaskar dan Gambia dengan Dura Deli sebagai varietas standar.
AFLP juga telah banyak digunakan pada kelapa sawit. AFLP digunakan untuk berbagai tujuan diantaranya untuk menganalisis genotipe normal dan abnormal klon-klon kelapa sawit (Toruan-Mathius et al. 2005), marka genetik yang berhubungan dengan warna buah viresen (Ying et al. 2007), bahkan bersama-sama dengan penanda RFLP digunakan sebagai peta keterpautan genetik pada Tenera hasil persilangan antara Dura Deli dengan Tenera Yangambi.
AFLP untuk studi karakterisasi keragaman genetik telah dilakukan pada Dura dibandingkan dengan Tenera dan E. oleifera oleh Galeano (2005). Penelitian ini dilakukan pada kelapa sawit asal Amerika dan Afrika. Purba et al. (2000) melakukan studi keragaman genetik kelapa sawit Indonesia menggunakan isoenzim dan AFLP serta konsekuensinya untuk program pemuliaan.
Analisis Keragaman Genetik Kelapa Sawit menggunakan Marka SSR
Mikrosatelit atau SSR adalah sekuen berulang yang terdiri dari dua (di), tiga (tri), empat (tetra) atau lima (penta) nukleotida. Lokus SSR sangat bervariasi di dalam ukurannya sehingga dapat digunakan untuk identifikasi genetik.
Marka mikrosatelit telah banyak digunakan pada kelapa sawit selain untuk eksplorasi genotipe-genotipe baru yang unggul juga banyak digunakan untuk memverifikasi kualitas di dalam manajemen produksi secara klonal. Untuk tujuan tersebut, Artutiningsih (2012) telah melakukan analisis kestabilan genetik ortet kelapa sawit Tenera dan klon-klon turunannya pada 90 tanaman yang berasal dari 10 genotipe. Inpuay et al. (2012) menggunakan marka mikrosatelit untuk menguji ketidakstabilan genetik plantlet dari kultur jangka panjang kelapa sawit melalui embriogenesis somatik sekunder. Demikian juga yang dilakukan oleh Thawaro dan Te-chato (2010) dan Singh et al. (2007).
Penelitian tentang keragaman genetik kelapa sawit menggunakan marka molekuler mikrosatelit pertama kali dilakukan oleh Billotte et al. (2001) pada genus Elaeis. Marka ini telah digunakan untuk studi keragaman genetik plasma-plasma nutfah baru hasil introduksi dari Afrika seperti Nigeria dan Kamerun. Zulhermana et al. (2010) yang menguji keragaman genetik ramet hasil kultur jaringan kelapa sawit klon Pisifera asal Nigeria intra dan antar populasi yang berasal dari 6 ortet terpilih. Hasil yang dicapai dapat menunjukkan adanya variasi somaklonal dengan perubahan susunan SSR dari ortet ke ramet. Hasil ini dapat digunakan untuk mengevaluasi hubungan genetik antara ramet dengan ortet asal, keseragaman ramet dalam satu ortet dan mendeteksi variasi somaklonal.
tandan dan berat tandan. Hatorangan et al. (2010) membuat profil sidik jari DNA ortet kelapa sawit sebagai sumber eksplan perbanyakan klonal menggunakan marka mikrosatelit.
3 BAHAN DAN METODE
Tempat dan Waktu PenelitianPenelitian ini dilaksanakan di Laboratorium Genomic and Transcriptomic,
Plant Production and Biotechnology Division, PT SMART Tbk, Bogor pada bulan
Mei 2013 sampai Februari 2014. Sampel daun diambil dari kebun percobaan kelapa sawit di Siak dan Damimas, Riau, Sumatera.
Kegiatan penelitian mencakup (i) penetapan bahan tanam dan pengambilan sampel, (ii) isolasi DNA, (iii) penghitungan kualitas dan kuantitas DNA, (iv) amplifikasi DNA dengan PCR yang didahului dengan optimasi suhu annealing, (v) elektroforesis DNA hasil amplifikasi, (vi) analisis keragaman genetik yang meliputi skoring dan analisis data molekuler.
Analisis data molekuler meliputi (a) skoring data, (b) profil hasil karakterisasi marka SSR, (c) analisis tingkat heterozigositas dan analisis polimorfisme, (d) analisis keragaman genetik dan struktur populasi yang meliputi perbandingan analisis keragaman genetik populasi plasma nutfah asal Angola dengan populasi D x P, D dan P; serta perbandingan analisis keragaman genetik berdasarkan daerah distribusi spasial dibandingkan dengan populasi D x P, D dan P, (e) analisis variasi molekuler/Analysis of Molecular Variance (AMOVA), (f) analisis keragaman dan kemiripan genetik individu, (g) analisis koordinat utama/Principal Coordinates Analysis (PCoA).
Bahan Tanaman
Bahan tanaman yang digunakan adalah daun muda tanaman kelapa sawit Dura dan Tenera aksesi hasil eksplorasi dari Angola serta Dura Deli (D) dan Tenera (D x P) tanaman komersial. Bahan tanaman yang berasal dari Angola terdiri dari 136 tanaman yang dikelompokkan menjadi 27 aksesi (masing-masing aksesi kurang lebih 5 tanaman) dari total 48 aksesi yang dieksplorasi pada tanggal 03 sampai 31 Juli 2010 oleh kerjasama Tim Indonesia Palm Oil Board (IPOB),
Malaysia Palm Oil Board (MPOB) dan Instituto Nacional do Cafe (INCA). Biji
hasil eksplorasi ini telah dikecambahkan dan ditanam pada 2011 di main nursery
kebun percobaan di Riau.
Penanaman di main nursery dilakukan sebanyak 7 batch dari bulan April 2011 sampai Juli 2011. Pemindahan dari main nursery ke kebun percobaan dilakukan pada bulan Oktober 2012 sehingga tanaman berumur kira-kira 8 bulan. Sedangkan tanaman kelapa sawit komersial jenis Tenera sebagian berumur 20 tahun dan sebagian diantaranya berumur 12 tahun (ditanam tahun 2001) serta tanaman induk Dura dan non induk dan Pisifera berumur antara 14-15 tahun (ditanam antara tahun 1998 hingga 1999).
terdiri dari 20 kelapa sawit tipe D (14 progeni), empat kelapa sawit tipe P (1 progeni) dan 10 kelapa sawit tipe D x P (10 progeni) (Lampiran 2).
Berdasarkan daerah distribusi spasial, pengambilan sampel kelapa sawit asal Angola dibagi menjadi lima daerah, yaitu Angola bagian Barat Laut, Barat, Utara, Tengah dan Selatan. Lima daerah distribusi spasial mencakup enam provinsi di Angola yang dieksplorasi pada kondisi geografis, iklim, medan dan tanah yang berbeda (Gambar 2 dan Tabel 1). Ketinggian tempat dari lima daerah berkisar pada hampir mendekati permukaan laut sampai 1,000 m dpl. Provinsi ini dipilih sebagai daerah yang paling potensial untuk keragaman kelapa sawit. Koleksi kelapa sawit mewakili sampel dari habitat aslinya termasuk hutan, area berbukit, pantai dan padang rumput yang luas/savanna (Tirtawinata et al. 2010).
Gambar 2 Peta Angola yang menunjukkan daerah eksplorasi kelapa sawit.
Warna merah: (1) Cabinda (2) Bengo (3) Uige (4) Kwanza Norte. Warna biru: (1) Cabinda (2) Kwanza Norte (3) Kwanza Sul (4) Benguela. (Sumber: Tirtawinata et al. 2010).
Tabel 1 Daftar informasi tanaman yang digunakan pada masing-masing populasi asal Angola
Asal Provinsi Angola bagian Jumlah aksesi Jumlah tanaman
Cabinda Barat Laut 2 10
Bengo Barat 3 15
Uige Utara 6 30
Kwanza Norte dan Kwanza Sul Tengah 12 61
Benguela Selatan 4 20
Daftar sampel tanaman yang lengkap disajikan di Tabel Lampiran 1
Pengambilan sampel
kemudian keduanya disatukan, ujung daun dipotong kurang lebih 5 cm. Sampel dibungkus dalam plastik diberi label dan ditempatkan di dalam cool box dan selanjutnya dibawa ke laboratorium untuk dilakukan analisis molekuler.
Analisis Molekuler
Isolasi DNA
Isolasi DNA dilakukan menggunakan metode Nucleospin Plant IITM
(Macherey-Nagel 2010). Sebanyak kurang lebih 100 mg (bobot basah) atau setara dengan 20 mg bobot kering daun kelapa sawit muda digerus di dalam mortar
porselein dengan ditambahkan Nitrogen cair. Daun muda digerus sampai halus. Ekstraksi DNA dari dalam sel dilakukan menggunakan bufer PL1 (SDS). Serbuk
sampel dimasukkan ke dalam tabung. Campuran yang berisi 400 µ Lbufer PL1 di
dalam Eppendorf divorteks secara menyeluruh. Selanjutnya ditambahkan 10 µL larutan RNAse A, divorteks, disuspensi kemudian diinkubasi selama 10 menit pada suhu 65 °C. Sampel yang sudah diberi RNAse kemudian disentrifus dengan kecepatan 11,000 x g selama 5 menit.
Filter NucleoSpinTM (cincin ungu) ditempatkan di dalam tabung baru (2 mL) dan lysate dimasukkan ke dalam kolom. Tabung kemudian disentrifugasi selama 2 menit dengan kecepatan 11,000 x g. Cairan bening atau supernatan dikumpulkan dan Filter NucleoSpinTM dibuang. Apabila tidak semua cairan melewati filter, tahap sentrifugasi dapat diulangi sekali lagi. Apabila pelet tidak nampak, supernatan bening dipindahkan ke dalam 1.5 mL tabung mikrosentrifugasi baru.
Sebanyak 450 µL bufer PC ditambahkan dan dicampur secara menyeluruh dengan dipipet ke atas dan ke bawah sebanyak 5 kali atau dengan divorteks. Setelah itu, pengikatan DNA dilakukan dengan menempatkan NucleoSpin Plant
IITM Column (cincin hijau) ke dalam tabung baru (2 mL) yang mampu memuat
maksimum 700 µL sampel. Sentrifugasi dilakukan selama 1 menit pada kecepatan 11,000 x g selanjutnya cairan yang tertampung di bawah dibuang. Maksimum kapasitas tabung NucleoSpin Plant IITM Column memuat 700 µL. Untuk sampel yang lebih banyak, tahap pemuatan dapat diulang.
Pencucian membran silika (kolom bercincin hijau) dilakukan dengan menambahkan 400 µL bufer PW1 untuk NucleoSpin Plant II Column. Kemudian
kolom disentrifugasi selama 1 menit pada 11,000 x g dan cairan yang tertampung dibuang. Pencucian kedua dilakukan dengan menambahkan 700 µL bufer PW2 ke
dalam NucleoSpin Plant II Column. Kolom disentrifugasi kembali selama 1 menit pada kecepatan 11,000 x g dan larutan yang tertampung dibuang kembali. Pencucian ketiga dilakukan dengan menambahkan bufer PW2 sebanyak 200 µL ke
dalam NucleoSpin Plant IITM Column. Kemudian kolom disentrifugasi selama 2 menit pada 11,000 x g untuk membuang bufer pencuci dan mengeringkan membran silika secara menyeluruh.
Untuk mengelusi DNA, NucleoSpin Plant IITM Column ditempatkan di dalam tabung mikrosentrifugasi baru volume 1.5 mL. Kemudian sebanyak 50 µL bufer PE dipipet ke dalam membran. Selanjutnya NucleoSpin Plant II Column
pada 11,000 x g untuk mengelusi DNA. Tahap ini diulangi kembali dengan menggunakan 50 µL bufer PE (65 °C) dan elusi dilakukan pada tabung yang sama.
Penentuan Kualitas dan Kuantitas DNA
Elektroforesis horizontal pada gel agarosa digunakan untuk mengkonfirmasi kemurnian DNA (Sambrook et al. 1989). Sebanyak 1% gel agarosa disiapkan (0.8 g di dalam 80 mL TAE) di dalam 1 x bufer TAE. Dengan menggunakan sisir, sumur berukuran kecil dibuat di dalam gel untuk memungkinkan dimasukkannya 3 µL DNA genom yang telah dicampur dengan 1
µL loading dye (bromofenol biru (Sigma B8026). Tangki dibungkus untuk
memungkinkan proses elektroforesis, kemudian elektroforesis dijalankan selama 30 menit pada voltage 100V. Kemudian gel dibuang dan direndam di dalam larutan ethidium bromide selama 10 menit untuk proses staining. Gel selanjutnya dibilas menggunakan akuades selama 10 menit sebelum dilihat di dalam
transluminator UV Gel DocTM Universal Hood 76S/09155..
Kuantifikasi DNA dilakukan dengan menggunakan Nanodrop 2000c
(Thermo Scientific) spektrofotometer. Larutan yang digunakan sebagai blanko
dalam mengukur konsentrasi DNA hasil isolasi adalah larutan yang sama yang digunakan sebagai pelarut DNA, yaitu elute solution (TE) dari kit NucleoSpin
Plant IITM. Sebanyak 1 µL TE diteteskan pada bagian pedestal (bagian alat untuk
meletakkan sampel). Konsentrasi DNA muncul pada layar komputer yang terhubung dalam satuan ng µL-1.
Konsentrasi DNA hasil isolasi biasanya masih tinggi. Oleh karena itu masih perlu diencerkan sebagai larutan kerja sebesar 20 ng menggunakan bufer TE. DNA stok disimpan pada freezer untuk penyimpanan jangka panjang.
Amplifikasi DNA dengan PCR
Kegiatan amplifikasi DNA mencakup seleksi primer mikrosatelit dan optimasi suhu annealing primer mikrosatelit. Selanjutnya suhu annealing optimum dari primer uji digunakan dalam reaksi amplifikasi dengan PCR pada bahan tanam yang diuji.
Seleksi primer mikrosatelit
Sebanyak 23 pasang primer spesifik mikrosatelit yang digunakan pada penelitian ini adalah primer yang telah dipublikasi (Billotte et al. 2005). Hatorangan et al. (2010) dan Kusnandar et al. (2010) masing-masing melakukan optimasi primer yang sudah dipublikasi tersebut dan melaporkan kondisi optimum untuk 16 primer serta 7 primer lainnya dan menghasilkan pita polimorfik pada kelapa sawit Tenera yang berasal dari kebun kelapa sawit di Riau. Primer dengan tingkat polimorfis yang tinggi dapat digunakan sebagai marka atau penanda.
Selain tingkat polimorfisme, alasan lain pemilihan 23 primer tersebut diantaranya (i) kandungan basa GC antara 20% sampai 60%, dan (ii) posisi pada
Optimasi suhu annealing
Kegiatan ini bertujuan untuk memperoleh suhu annealing yang optimum pada masing-masing primer mikrosatelit yang digunakan. Suhu annealing untuk berbagai macam primer ditetapkan menggunakan rumus berikut: Tm = 4 (G + C) + 2 (A + T) dan suhu annealing = Tm -4. Hanya primer yang mampu menghasilkan pita spesifik pada suhu annealing optimum yang akan digunakan untuk mengamplifikasi DNA uji pada percobaan selanjutnya.
Amplifikasi SSR dilakukan di dalam 100 sumur Veriti Thermal cycler
(Applied Biosystem) dengan 15 µL volume reaksi. PCR mix terdiri dari 30 µg DNA genom, 1.5 pmol primer forward, 1.5 pmol primer reverse, 0.225 X 10-3 µM MgCl2, 1.5 µL Dream Tag buffer (10 x), 1.5 µL dNTPs, 0.6 unit Taq DNA
polymerase dan 6.48 µL ddH2O. PCR dijalankan dalam tiga tahap, yang terdiri
dari tahap pertama adalah tahap denaturasi pada suhu 94 °C selama 5 menit. Tahap kedua, yaitu denaturasi selama 94 °C selama 5 detik, tahap annealing tergantung dari Tm primer selama 30 detik dan tahap ekstensi pada 72 °C selama 50 detik. Proses dijalankan selama 35 siklus sebelum suhu direduksi menjadi 4 °C.
Untuk memastikan keberhasilan amplifikasi tanpa mengetahui hasil ukuran pita SSR, dilakukan pengecekan produk PCR pada gel agarosa. Setiap produk PCR diloading ke dalam sumur yang telah disiapkan. Gel agarosa di running di dalam tangki elektroforesis selama 30 menit 100 V. Setelah selesai proses running, gel berisi amplifikasi pita SSR divisualisasi ke dalam Gel Doc. Pita-pita SSR masing-masing individu akan terlihat berpendar pada ukuran antara 100 sampai 300 pasang basa. Individu yang tidak menghasilkan amplifikasi pita SSR di PCR ulang hingga seluruh individu menghasilkan pita SSR pada semua lokus.
Elektroforesis DNA Hasil Amplifikasi
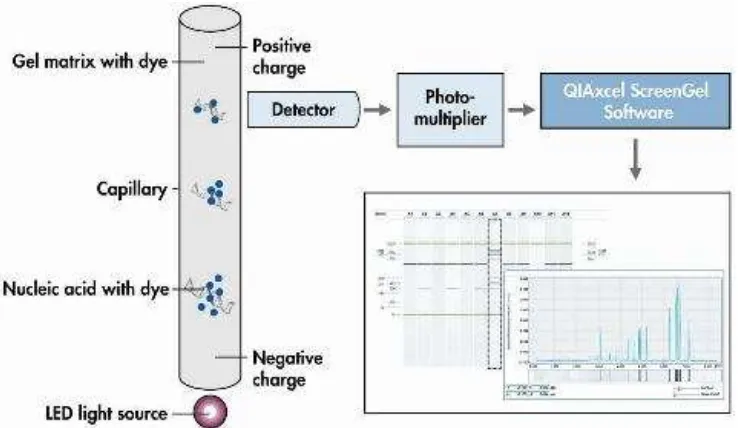
Elektroforesis dilakukan menggunakan sistem QIAxcel. Prinsip pemisahan pada QIAxcel dilakukan dalam kapiler cartridge gel dengan sampel secara otomatis masuk ke dalam kapiler. DNA yang bermuatan negatif bermigrasi melalui kapiler ke ujung bermuatan positif melalui detektor yang dapat mendeteksi dan mengukur signal dan kemudian mengubah signal emisi menjadi data elektronik yang selanjutnya akan terlihat sebagai gambar gel (Gambar 3).
Alat QIAxcel dan komputer yang sudah terhubung dihidupkan. QX
alignment marker dimasukkan ke dalam tray alat. Sampel hasil amplifikasi dalam
tabung mikro PCR 0.2 mL atau plate 96 well diletakkan dalam sampel tray alat. Tampilan “Instrument Control” memerlukan beberapa informasi yang perlu diisi.
Analisis Data Molekuler
Skoring Data
Skoring data dilakukan berdasarkan pada ukuran pita-pita DNA dan ada tidaknya pita DNA hasil amplifikasi dari individu-individu uji. Fragmen-fragmen DNA yang dihasilkan pada gel elektroforesis akan diskor secara manual sebagai data kodominan, data biner dan data genotipik berdasarkan pada kebutuhan data input masing-masing perangkat lunak yang digunakan.
Untuk data kodominan, setiap alel yang muncul diberi skor 1, 2, 3 dan seterusnya berdasarkan urutan alel sesuai ukuran berat molekul atau panjang pasang basa (bp) pada masing-masing lokus. Sebuah matriks excel akan dibuat dengan marka-marka yang diskor yang disusun sebagai baris dan sampel disusun sebagai kolom. Data kodominan akan digunakan sebagai data input pada analisis keragaman alelik, pengelompokan menggunakan metode Principal Coordinate
Analysis (PCoA) dan Analisis Variasi Molekuler/Analysis of Molecular Variance
(AMOVA) pada perangkat lunak GenAlEx versi 6.5.
Untuk data biner, setiap alel diberi nilai berdasarkan ada tidaknya suatu alel. Nilai 1 diberikan apabila ada alel dan nilai 0 apabila tidak ada alel. Data yang meragukan ada dan tidaknya dianggap sebagai data hilang dan diberi kode 999 pada input data. Selanjutnya seperti halnya dengan skoring data kodominan, data biner disusun pada matriks excel dengan marka sebagai baris dan sampel sebagai kolom. Data biner selanjutnya digunakan sebagai data input pada analisis kekerabatan dan kesamaan genetik pada perangkat lunak NTSYS versi 2.0.
Data genotipik digunakan sebagai data input pada perangkat lunak Power Marker versi 3.15 (Liu dan Muse 2005). Perangkat lunak ini mampu menganalisis nilai polimorfisme yang terkandung pada lokus-lokus (Polymorphic Information
Content/PIC). Data genotipik dibuat menggunakan huruf A, B, C, D dan
seterusnya berdasarkan urutan alel yang dihasilkan pada masing-masing lokus. Misalnya alel dengan ukuran paling besar diskor sebagai alel A, alel yang lebih kecil ukurannya dan berada pada urutan dibawah alel A disebut sebagai alel B dan seterusnya. Alel-alel hasil skoring dimasukkan ke dalam matriks excel untuk dianalisis.
Data kodominan digunakan sebagai data input pada analisis menggunakan perangkat lunak GenAlEx 6.5. Perangkat lunak ini digunakan untuk menganalisis frekuensi alel berupa AMOVA, koefisien perbedaan genetik (Gst), dan pengelompokan berdasarkan Analisis Koordinat Utama/AKOORDA atau
Principal Coordinate Analysis (PCoA). AMOVA merepresentasikan nilai
keragaman genetik berdasarkan korelasi frekuensi alel antar individu di dalam subpopulasi (Fis), nilai keragaman genetik berdasarkan korelasi frekuensi alel antar subpopulasi (Fst), dan nilai keragaman genetik berdasarkan frekuensi alel dalam populasi yang disebabkan oleh kedua faktor tersebut.
ke dalam matriks excel dengan baris sebagai individu dan kolom sebagai alel setiap lokus yang dianalisis.
Tabel 2 Daftar primer SSR yang digunakan dalam percobaan
Nama Lokus 5‟-3‟ Primer Linked Group mEgCIR0905 a & b F-CACCACATGAAGCAAGCAGT
Gambar 3 Proses pemisahan DNA sampel pada elektroforesis menggunakan sistem QIAxcel (Qiagen 2011)
Analisis data hanya dilakukan pada lokus-lokus yang menghasilkan amplifikasi spesifik dan tidak meragukan dalam skoring. Lokus dieliminasi dari analisis jika tidak menghasilkan amplifikasi yang konsisten atau meragukan di dalam interpretasi.
Profil hasil karakterisasi marka SSR
Keragaman alelik merupakan keragaman yang diukur dari keragaman alel-alelnya. Salah satu keragaman alelik adalah jumlah alel per lokus dan jumlah lokus polimorfik (Boer 2007). Menurut Marshall dan Brown (1975) keragaman alel pada lokus di dalam suatu populasi dapat dilihat dari klasifikasi alel diantaranya jumlah alel umum yaitu alel dengan frekuensi ≥5% dan jumlah alel khusus yaitu alel dengan frekuensi <5%. Sedangkan menurut Buchert et al. (1997) jumlah alel umum adalah alel dengan frekuensi ≥0.75, jumlah alel sedang adalah alel dengan frekuensi 0.75<P≤0.25, jumlah alel jarang adalah alel dengan frekuensi 0.25<P≤0.01 dan jumlah alel spesifik adalah alel dengan frekuensi <0.01.
A = Jumlah total semua alel pada semua lokus Jumlah lokus yang digunakan
Analisis Tingkat Heterozigositas dan Analisis Polimorfisme
nilai PIC menggunakan perangkat lunak Power Marker v 3.15 (Liu dan Muse 2005).
Salah satu kelebihan marka mikrosatelit sebagaimana marka kodominan lainnya, adalah dapat mendeteksi heterozigositas. Lokus yang heterozigot akan menghasilkan pita/alel lebih dari satu setiap primer dimana batasan jumlah alel yang dihasilkan bergantung dari jumlah ploidi individu yang dianalisis. Sedangkan lokus yang homozigot hanya akan menghasilkan satu pita/alel setiap primer. Tingkat heterozigot dihitung dengan rumus (Hidayatun 2005):
Heterozigositas (%) = Jumlah lokus heterozigot x 100% Total lokus yang digunakan
Analisis Variasi Molekuler (Analysis of Molecular Variance– AMOVA)
Analysis of Molecular Variance (AMOVA) mampu merepresentasikan
perbandingan antara keragaman antar individu di dalam aksesi, antar aksesi, di dalam daerah distribusi spasial maupun antar daerah distribusi. Selain itu AMOVA juga dapat digunakan untuk menganalisis keragaman individu di dalam populasi dan antar populasi baik populasi antar daerah dibandingkan dengan populasi D x P, D dan P maupun populasi antara plasma nutfah asal Angola dengan populasi D x P, D dan P. Analisis ini dilakukan menggunakan perangkat lunak GenAlEx v. 6.5 (Peakall dan Smouse 2006).
Analisis Keragaman dan Kemiripan Genetik Individu
Hasil skoring pita-pita yang muncul dan tidak muncul data biner pada matriks excel digunakan sebagai data input pada analisis kemiripan genetik. Estimasi kesamaan genetik dibuat dalam bentuk matriks dengan similarity for
qualitative data (SIMQUAL) yang merupakan sub program dari similarity and
dissimilarity. Pengelompokan dilakukan dengan sub program SAHN Clustering
dengan metode Unweighted Pair Group Method with Arithmetic Mean (UPGMA) pada bootsrap 1,000 menggunakan perangkat lunak Numerical Taxonomy and
Multivariate System (NTSYS)-pc v. 2.0 (Rohlf 2000).
Analisis Koordinat Utama (Principal Coordinate Analysis– PCoA)
Analisis Koordinat Utama/Principal Coordinate Analysis (PCoA) menggambarkan posisi relatif masing-masing individu yang dilakukan menggunakan perangkat lunak GenAlEx versi 6.5 (Peakall dan Smouse 2006). Data kodominan dihitung untuk mendapatkan nilai jarak genetik antar individu. Jarak genetik disajikan dalam tri matrix distance (Orloci 1978). Kemudian jarak pada matriks dihitung untuk mendapatkan eigen value/nilai akar ciri untuk seluruh koordinat yang mempengaruhi. Dua nilai akar ciri tertinggi pada koordinat 1 dan 2 digunakan untuk merepresentasikan kelompok individu-individu di dalam populasi.
Menurut Chae dan Warde (1995), ketika terdapat data yang hilang PCoA mampu memberikan hasil yang lebih baik dibandingkan dengan Principal
dibandingkan dengan PCA untuk mengidentifikasi objek-objek pada sebuah kelompok pada kondisi tertentu. Kemampuan pengelompokan/pembentukan klaster atau kemampuan algoritma pengelompokan aglomeratif meningkat pada penggunaan PCoA dan lebih rendah pada PCA. PCoA menggunakan data input berupa jarak dalam bentuk koefisien korelasi sedangkan PCA menggunakan data input berupa jarak Euclidean dimana keduanya berbeda.
Untuk pembentukan kelompok, penggunaan koefisien korelasi lebih sensitif terhadap perubahan/noise. PCoA menggambarkan pengelompokan berdasarkan tingkat perbedaan genetik (jarak genetik) sementara PCA menggambarkan pengelompokan berdasarkan tingkat kesamaan genetik. PCoA dapat digunakan untuk mengidentifikasi apabila terdapat individu yang sangat berbeda secara genetik/pencilan.
4 HASIL DAN PEMBAHASAN
Isolasi DNA
Konsentrasi DNA hasil isolasi yang diperoleh berkisar antara 14 sampai 308 ng µL-1. Konsentrasi DNA yang digunakan pada proses PCR sebanyak 20 ng µL-1. Isolasi DNA menggunakan metode SDS kit Nucleospin Plant II Macherey
Nagel menghasilkan pita yang tegas secara kualitatif pada genom akan tetapi
masih terdapat smear pada hasil elektroforesis horizontal gel agarosa.
Smear ini dapat berasal dari kontaminan komponen dan protein. Smear
menunjukkan kurang murninya kualitas DNA yang dihasilkan (Lampiran 3). Walaupun kualitas DNA yang dihasilkan kurang murni, amplifikasi pita-pita SSR masih dapat dilakukan dengan baik.
Suhu annealing optimum
Suhu annealing untuk primer yang diidentifikasi berkisar antara 50 °C sampai 55 °C. Sebanyak 11 primer mampu menghasilkan pita-pita yang jelas pada suhu annealing 51 °C. Hal ini menunjukkan bahwa kesebelas primer tersebut adalah primer yang spesifik. Primer-primer mikrosatelit yang spesifik mampu mengamplifikasi lokus tertentu yang spesifik pada kromosom.
Lokus merupakan lokasi suatu gen di dalam kromosom, sedangkan alel merupakan bentuk alternatif dari suatu gen. Alel pada SSR merupakan pita DNA yang muncul pada suatu lokus tertentu. Dalam hal ini, lokus pada SSR merupakan hasil amplifikasi primer. Lokus yang dihasilkan pada produk SSR adalah spesifik
(single locus) tetapi multi alelik/banyak alel.
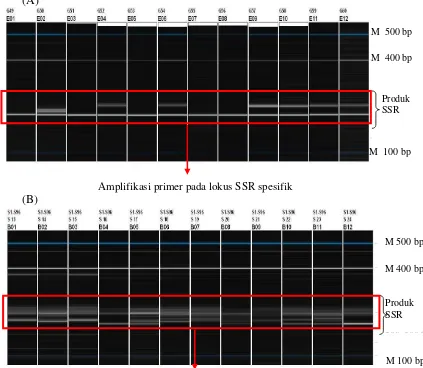
Alel terpanjang berukuran antara 240 sampai 311 pasang basa (bp) diperoleh dari lokus mEgCIR2518 dan paling pendek berukuran antara 115 sampai 169 bp berasal dari amplifikasi lokus mEgCIR0521 (Tabel 3). Tiga dari 23 primer yang digunakan, yaitu primer pada lokus mEgCIR3847, mEgCIR0905 dan mEgCIR3543 tidak menghasilkan pita spesifik, akan tetapi menghasilkan banyak pita pada produk PCR. Hal ini disebabkan ketiga primer tersebut tidak spesifik untuk amplifikasi. Oleh sebab itu ketiga lokus tersebut tidak digunakan untuk analisis tahap selanjutnya.
(A)
(B)
Gambar 4 Elektropherogram QIAxcel (atas) pita-pita SSR pada primer spesifik (atas) lokus mEgCIR3519 dan primer tidak spesifik (bawah) lokus mEgCIR0905 a dan b pada beberapa individu plasma nutfah kelapa sawit asal Angola. M 100 bp: marker ukuran 100 bp, M 400 bp: marker ukuran 400 bp, M 500: marker ukuran 500 bp.
M 500 bp
M 400 bp
M 100 bp Produk SSR
200-300 bp
M 500 bp
M 400 bp
M 100 bp Produk SSR
200-300 bp
Profil Pita Mikrosatelit, Tingkat Heterozigositas dan Polimorfisme
Dua puluh lokus mikrosatelit yang dianalisis menghasilkan 107 alel pada seluruh individu yang diuji dengan rata-rata jumlah alel sebanyak 5.35 alel per lokus (Tabel 4). Zulhermana et al. (2010) memperoleh jumlah alel sebanyak 3.67 pada keragaman genetik populasi klon kelapa sawit Pisifera asal Nigeria, Augustina et al. (2010) melaporkan jumlah alel sebanyak 3.85 pada keragaman genetik 22 aksesi Pisifera dari berbagai daerah asaldan Maizura et al. (2006) menghasilkan jumlah alel sebanyak 1.9 pada keragaman genetik aksesi kelapa sawit asal 11 negara-negara di Afrika.
Tabel 3 Suhu annealing optimum dan panjang alel pada 20 lokus mikrosatelit
Arias et al. (2012) melaporkan bahwa aksesi asal Kamerun mempunyai rata-rata jumlah alel 7.5 dan Wening et al. (2012) menunjukkan aksesi asal Ghana mempunyai rata-rata jumlah alel sebanyak 9.9 alel. Billotte et al. (2001) menghasilkan rata-rata jumlah alel sebanyak 5.3 pada karakterisasi keragaman genetik antar taxa beberapa genus Elaeis dari berbagai daerah asal. Penelitian Ajambang et al. (2012) menunjukkan bahwa aksesi plasma nutfah asal Kamerun mempunyai jumlah alel sebanyak 4.71. Jumlah alel hasil penelitian Billotte et al. (2001) dan Ajambang et al. (2012) sama dengan hasil penelitian ini.
Jumlah alel pada populasi kelapa sawit asal Angola lebih rendah dibandingkan dengan jumlah alel pada beberapa negara pusat penyebaran lainnya seperti Kamerun dan Ghana. Hal ini mungkin disebabkan oleh lokasi Angola yang berada di Selatan daerah pusat penyebaran kelapa sawit (dari 16º lintang utara di Senegal sampai 15º lintang Selatan di Angola (Henson 2012)) atau di sepanjang laut Atlantik Selatan (Corley dan Tinker 2003) sehingga pertukaran alel lebih sedikit terjadi di negara ini. Pola penyebaran alel pada negara-negara asal kelapa sawit berdasarkan penelitian Zulkifli et al. (2012) dan Maizura et al. (2006) menunjukkan bahwa kemungkinan pusat penyebaran kelapa sawit terletak di daerah sekitar Nigeria. Di negara tersebut jumlah alel yang dimiliki paling banyak dibandingkan dengan jumlah alel asal negara-negara lainnya.
Tabel 4 Rata-rata jumlah alel, klasifikasi, tingkat heterozigositas dan polimorfisme 20 lokus mikrosatelit
Parameter Keragaman Total Rata-rata
Jumlah alel (Na) 107 5.35
Jumlah dan frekuensi alel ≥ 5% (alel umum) 74 (69%) 3.70 Jumlah alel khusus <5% (alel khusus) 33 (31%) 1.65
Jumlah alel efektif (Ne) 2.85
Heterozigositasharapan (He) 0.60
Polymorphic Information Content (PIC) 0.55
Menurut Marshall dan Brown (1975) klasifikasi alel berdasarkan frekuensinya dibagi menjadi dua, yaitu alel umum dan alel khusus. Alel dikategorikan sebagai alel umum apabila mempunyai frekuensi ≥5% dan alel khusus apabila mempunyai frekuensi <5%. Dari 107 alel pada populasi yang diuji, 74 alel (69%) merupakan alel umum dan 33 alel (31%) merupakan alel khusus (Tabel 4).
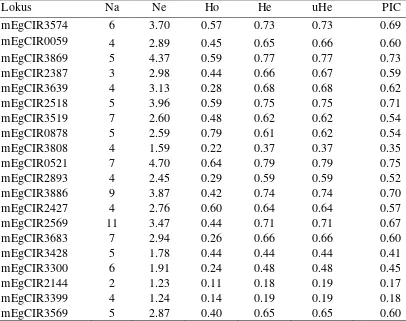
Rata-rata heterozigositas harapan (He) sebesar 0.60 dari maksimum nilai 1.0. Nilai ini menunjukkan bahwa populasi yang dianalisis memiliki keragaman genetik yang tinggi berdasarkan primer-primer mikrosatelit yang diuji (Tabel 4). Nilai heterozigositas harapan (He) masing-masing lokus berkisar antara 0.17 sampai 0.89. Sebanyak 5 lokus memiliki nilai He <0.5 dengan alel-alel yang dihasilkan memiliki tingkat heterozigositas yang rendah (Tabel 5).
Nilai heterozigositas menunjukkan jumlah penyebaran gen atau alel pada suatu populasi. Nilai heterozigositas pengamatan (Ho) biasanya lebih rendah dibandingkan dengan heterozigositas harapan (He) dengan asumsi bahwa populasi masih alami, menyerbuk bebas dan ukuran populasi cukup besar. Kondisi lainnya adalah populasi tidak mengalami seleksi, tidak terjadi mutasi yang menyebabkan hilangnya alel-alel tertentu di dalam populasi (genetic drift). Adanya pengurangan ukuran populasi (bottleneck effect) juga mempengaruhi penurunan nilai heterozigositas pengamatan (Ho) (Nei 1987).
Tabel 5 Beberapa nilai peubah keragaman pada 20 lokus mikrosatelit
Lokus Na Ne Ho He uHe PIC
mEgCIR3574 6 3.70 0.57 0.73 0.73 0.69
mEgCIR0059 4 2.89 0.45 0.65 0.66 0.60
mEgCIR3869 5 4.37 0.59 0.77 0.77 0.73
mEgCIR2387 3 2.98 0.44 0.66 0.67 0.59
mEgCIR3639 4 3.13 0.28 0.68 0.68 0.62
mEgCIR2518 5 3.96 0.59 0.75 0.75 0.71
mEgCIR3519 7 2.60 0.48 0.62 0.62 0.54
mEgCIR0878 5 2.59 0.79 0.61 0.62 0.54
mEgCIR3808 4 1.59 0.22 0.37 0.37 0.35
mEgCIR0521 7 4.70 0.64 0.79 0.79 0.75
mEgCIR2893 4 2.45 0.29 0.59 0.59 0.52
mEgCIR3886 9 3.87 0.42 0.74 0.74 0.70
mEgCIR2427 4 2.76 0.60 0.64 0.64 0.57
mEgCIR2569 11 3.47 0.44 0.71 0.71 0.67
mEgCIR3683 7 2.94 0.26 0.66 0.66 0.60
mEgCIR3428 5 1.78 0.44 0.44 0.44 0.41
mEgCIR3300 6 1.91 0.24 0.48 0.48 0.45
mEgCIR2144 2 1.23 0.11 0.18 0.19 0.17
mEgCIR3399 4 1.24 0.14 0.19 0.19 0.18
mEgCIR3569 5 2.87 0.40 0.65 0.65 0.60
Na: jumlah alel, Ne: jumlah alel harapan, Ho: heterozigositas pengamatan, He: heterozigositas harapan, uHe: heterozigositas tidak bias, PIC: Polimorphic Information Content.
Nilai PIC menggambarkan tingkat informatif lokus-lokus yang digunakan untuk menganalisis tingkat keragaman alel-alelnya. Nilai PIC diklasifikasikan menjadi tiga kelas, yaitu PIC>0.5 dikategorikan sebagai sangat informatif,
0.25≥PIC≥0.5 dikategorikan sebagai moderat informatif dan PIC<0.25