BIOTYPING ISOLAT LOKAL
Enterobacter sakazakii
(
Cronobacter
spp.) DAN KERAGAMAN GENETIK BEBERAPA
ISOLAT BERDASARKAN GEN
infB
IZA AYU SAUFANI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Biotyping Isolat Lokal Enterobacter sakazakii (Cronobacter spp.) dan Keragaman Genetik Beberapa Isolat Berdasarkan Gen infB adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Mei 2013 Iza Ayu Saufani NIM F251100081
RINGKASAN
IZA AYU SAUFANI. Biotyping Isolat Lokal Enterobacter sakazakii (Cronobacter spp.) dan Keragaman Genetik Beberapa Isolat Berdasarkan Gen infB. Dibimbing oleh RATIH DEWANTI-HARIYADI dan SRI HENDRASTUTI HIDAYAT.
E. sakazakii pada awalnya didefinisikan sebagai bakteri E. cloacae yang memiliki pigmen kuning, yang kemudian dapat diklasifikasikan dengan menggunakan reaksi biokimia, ketahanan terhadap antibiotik dan hibridisasi DNA. Pada tahun 2007, E. sakazakii digolongkan ke dalam Cronobacter spp. yang terdiri dari C. sakazakii, C. malonaticus, C. muytjensiii, C. dublinensis dan C. turicensis berdasarkan f-AFLP serta 4 reaksi biokimia Iversen (produksi asam dari dulsitol, produksi asam dari methyl-α-D-glucoside, produksi indol dan pemanfaatan malonat).
Isolat lokal yang diperoleh dari susu formula bubuk, makanan bayi, pati, tepung-tepungan dan rempah-rempah di Indonesia teridentifikasi sebagai Cronobacter spp. dan tidak memiliki keragaman genetik yang tinggi berdasarkan gen parsial 16S rRNA (Gitapratiwi et al 2012). Meskipun demikian, beberapa isolat menunjukkan ketahanan panas yang lebih tinggi dibandingkan lainnya (Seftiono 2012).
Tujuan penelitian ini adalah melakukan identifikasi dan klasifikasi berdasarkan sifat biokimia (biotyping) dengan menggunakan perangkat cepat RapID onE dan 4 uji biokimia Iversen (Iversen et al. 2007b), serta untuk mengetahui keragaman isolat-isolat lokal yang telah diketahui kinetika termalnya berdasarkan gen infB.
Dengan menggunakan RapID onE diketahui bahwa dari 19 isolat lokal yang dipelajari, 9 teridentifikasi sebagai E. sakazakii, 9 lainnya sebagai E. cloacae dan 1 teridentifikasi sebagai E. cancerogenus. Jika hasil identifikasi menggunakan perangkat cepat RapID onE dibandingkan dengan hasil berdasarkan perangkat cepat API 20E (Gitapratiwi 2011; Hamdani 2012), 8 dari 19 isolat uji sama-sama teridentifikasi sebagai E. sakazakii. Berdasarkan 4 reaksi biokimia Iversen diketahui bahwa 15 dari 19 isolat adalah Cronobacter spp. sementara 4 lainnya tidak bisa diklasifikasikan ke dalam Cronobacter spp. Dengan mencermati hasil reaksi pada RapID onE dan memilih reaksi yang memberikan hasil berbeda, maka dapat disimpulkan bahwa uji pyrohydonyl-β-naphthylamide dapat digunakan untuk menggolongkan keempat isolat tersebut ke dalam C. sakazakii.
Sepuluh dari 19 reaksi uji yang terdapat pada RapID onE dapat digunakan untuk mengelompokkan isolat-isolat lokal menjadi 16 biotipe. Jika hasil biotyping dengan perangkat RapID onE dibandingkan dengan 4 uji biokimiawi Iversen, maka biotipe I-VIII adalah spesies C. sakazakii; biotipe IX, X, XV dan XVI adalah spesies C. malonaticus; biotype XIII dan XIV adalah spesies C. muytjensii; serta biotipe XI, XII dan XVI adalah spesies C. turicensis.
inisiasi translasi IF2. Berdasarkan gen infB, isolat lokal Cronobacter spp. yang diuji dapat dibagi menjadi 2 kelompok yaitu isolat DES b7a, DES b10, DES c13, YR t2a dan E6 memiliki kerabatan yang dekat dengan isolat C. sakazakii sementara isolat YR c3a memiliki hubungan kekerabatan yang dekat dengan isolat C. malonaticus dan C. dublinensis.
SUMMARY
IZA AYU SAUFANI. Biotyping of Local Isolates Entrobacter sakazakii (Cronobacter spp.) and Genetic Variability among Several Isolates based on infB Gene. Supervised by RATIH DEWANTI-HARIYADI and SRI HENDRASTUTI HIDAYAT.
E. sakazakii was initially described as E. cloacae with yellow pigment, and later were classified based on their biochemical reactions, antibiotics susceprability and DNA-DNA hybridization. In 2007, E. sakazakii was reclassified into Cronobacter spp. consisting of C. sakazakii, C. malonaticus, C. muytjensii, C. dublinensis and C. turicensis based on f-AFLP and production of acid from dulcitol, production of indole, malonate utilization and production of acid from methyl-α-D-glucoside.
All local isolates obtained from powder infant formula, weaning foods, starch, flours and spices in Indonesia have been identified as Cronobacter spp. and were reported to have close genetic relatedness based on its partial gene sequence encoding for 16S rRNA. However, they have been reported to have different thermal resistance (Seftiono 2012).
This study aims to identify and classify local isolates of C. sakazakii based on their biochemical properties and genetic relatedness based on infB genes which known thermal kinetics.
Identification of the 19 local isolates using RapID onE suggested that 9 isolates were identified as E. sakazakii, 9 others were E. cloacae and 1 was E. cancerogenus. When the results were compared to the identification using API 20E (Gitapratiwi 2011; Hamdani 2012), 8 of the 19 isolates were identified as E. sakazakii by both kits. Based on classification using 4 biochemical reaction, 15 isolates can be identified as Cronobacter spp. whereas 4 isolates can not be identified. By evaluating biochemical reaction in the RapID onE test kit, it can be conclude that reaction of pyrohydonyl-β-naphthylamide can be used as the additional testing to classify the previously unclassified local isolates into C. sakazakii.
Genotypic characterization of infB gene among local isolates showed that all tested isolates where closely related. Based on the BLAST program, it was revealed that the contig sequence of the infB genes was as a translation initiation factor IF2. Based on the infB gene the local isolates can be classified into 2 groups. Isolat DES b7a, DES b10, DES c13, YR t3a and E6 belong to C. sakazakii, while isolate YR c3a is C. malonaticus and C. dublinensis.
© Hak Cipta Milik IPB, Tahun 2013
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Pangan
BIOTYPING ISOLAT LOKAL
Enterobacter sakazakii
(
Cronobacter
spp.) DAN KERAGAMAN GENETIK BEBERAPA
ISOLAT BERDASARKAN GEN
infB
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2013
Judul Tesis : Biotyping Isolat Lokal Enterobacter sakazakii (Cronobacter spp.) dan Keragamana Genetik Beberapa Isolat Berdasarkan Gen infB Nama : Iza Ayu Saufani
NIM : F251100081
Disetujui oleh Komisi Pembimbing
Dr. Ir. Ratih Dewanti-Hariyadi, MSc Ketua
Dr. Ir. Sri Hendrastuti Hidayat, MSc Anggota
Diketahui oleh
Ketua Program Studi Ilmu Pangan
Dr. Ir. Ratih Dewanti-Hariyadi, MSc
Dekan Sekolah Pascasarjana
Dr. Ir. Dahrul Syah, MSc. Agr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini dapat diselesaikan sebagai salah satu syarat memperoleh gelar Magister Sains. Penelitian yang dilaksanakan sejak bulan Juli 2012 selama 6 bulan di Laboratorium SEAFAST Center dapat penulis selesaikan dengan baik berkat bantuan dana penelitian dari SEAFAST Center IPB dan Direktorat Jenderal Perguruan Tinggi melalui hibah kompetensi 2011.
Penulis mengucapkan terima kasih dan penghargaan setinggi-tingginya kepada Ibu Dr. Ir. Ratih Dewanti-Hariyadi, MSc selaku ketua komisi pembimbing dan ibu Dr. Ir. Sri Hendrastuti Hidayat, MSc selaku anggota komisi pembimbing , serta ibu Dr. Ir. Harsi Dewantarikusumaningrum selaku penguji luar komisi yang telah memberikan bimbingan, kritik, saran dan motivasi yang membangun selama penelitian dan penulisan karya ilmiah ini. Selain itu penulis sampaikan terima kasih kepada seluruh staf dan laboran laboratorium SEAFAST Center IPB.
Ucapan terima kasih penulis sampaikan kepada papa, mama, kakak dan adik atas doa, kasih sayang, motivasi dan semangat yang konstruktif selama ini. Penulis juga mengucapkan terima kasih kepada keluarga besar IPN 2010, rekan-rekan sepenelitian dan teman-teman serta semua pihak yang tidak dapat penulis sebutkan satu persatu atas kerjasama selama penulis melaksanakan perkuliahan maupun penelitian.
Semoga karya ilmiah ini bermanfaat.
Bogor, Mei 2013
DAFTAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xii
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
2 TINJAUAN PUSTAKA 3
Enterobacter sakazakii (Cronobacter spp.) 3 Klasifikasi Enterobacter sakazakii (Cronobacter spp.) 4
Peran Gen infB dan Ketahanan Panas 7
3 METODE PENELITIAN 10
Lokasi Penelitian dan Waktu Penelitian 10 Bahan Penelitian dan Peralatan Penelitian 10
Prosedur Penelitian 11
4 HASIL DAN PEMBAHASAN 19
Identifikasi Biokimia Isolat Lokal Cronobacter spp. 19 Keragaman Genotip Gen infB Cronobacter spp. 27
5 SIMPULAN DAN SARAN 33
Simpulan 33
Saran 34
DAFTAR PUSTAKA 34
LAMPIRAN 39
DAFTAR TABEL
1 Peosedur isolasi Cronobacter spp. dari susu formula 4
2 Kriteria untuk klasifikasi bakteri 5
3 Uji biokimia untuk membedakan spesies Cronobacter 6 4 Daftar isolat Cronobacter spp. yang digunakan 10 5 Hasil identifikasi takson dan tingkat kemiripan isolat lokal Cronobacter
spp. dengan program ERIC RapID onE 19
6 Perbandingan identifikasi isolat lokal Cronobacter spp. menggunakan
RapID onE dengan API 20E 23
7 Klasifikasi isolat lokal Cronobacter spp. berdasarkan 4 reaksi biokimia
Iversen 24 8 Uji biokimia untuk membedakan spesies lokal Cronobacter spp. 25
9 Biotyping isolat lokal Cronobacter spp. berdasarkan 10 reaksi biokimia 26
10 Karakteristik fenotip Cronobacter spp. 27
11 Jumlah nukleotida hasil sekuensing gen infB menggunakan primer
infBf/infBr 30
12 Hasil BLAST gen infB dengan database 30
13 Persen homologi antar isolat lokal Cronobcater spp. yang diuji
berdasarkan gen infB 31
14 Persen homologi isolat lokal Cronobacter spp. dengan isolat luar 32
DAFTAR GAMBAR
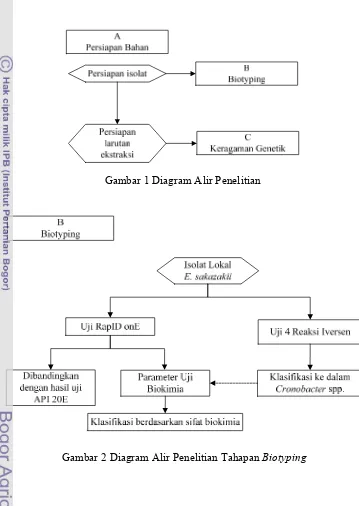
1 Diagram Alir Penelitian 12
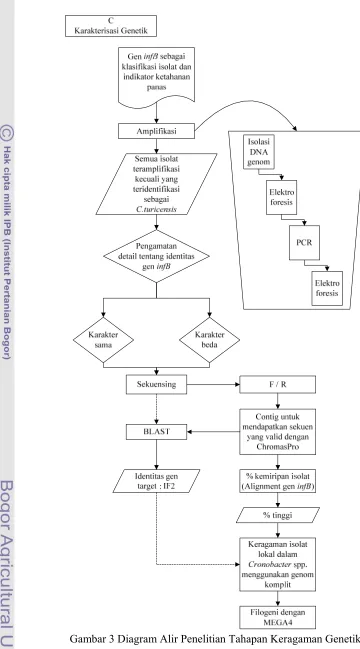
2 Diagram Alir Penelitian Tahapan Biotyping 12 3 Diagram Alir Penelitian Tahapan Keragaman Genetik 13 4 Penampilan Kerja Perangkat Lunak Eric (Remel) 15
5 Penanda DNA ladder 1kb 18
6 Visualisai DNA hasil amplifikasi gen infB menggunakan primer
infB-f/infB-r 29
7 Dendogram isolat lokal Cronobacter spp. dan isolat pembanding berdasarkan gen faktor translasi inisition 33
DAFTAR LAMPIRAN
1 Identifikasi Bakteri Berdasarkan Bergey’s Manual 39
2 Form Hasil Kerja RapID onE 42
3 Identifikasi Isolat Lokal Cronobacter spp. Berdasarkan RapID onE 43 4 Peta Sekuen Contig Isolat Cronobacter spp. 46 5 Sekuen Contig Isolat Lokal Cronobacter spp. 47
1
PENDAHULUAN
Latar Belakang
Enterobacter sakazakii (Cronobacter spp.) mula-mula digolongkan ke dalam genus Enterobacter dan dapat dibedakan dari E. cloacae karena memiliki pigmen kuning. Bakteri ini merupakan mikroorganisme berbahaya yang dapat menyebab beberapa kasus infeksi meningitis, septicemia dan necrotizing enterocolitis (NEC) pada bayi kelompok tertentu melalui konsumsi susu formula bubuk. FDA (2002a) melaporkan terjadinya kasus kematian bayi prematur akibat meningitis di Indiana US, sehingga dilakukan penarikan susu formula bubuk yang terkontaminasi oleh Cronobacter spp. dari pasaran.
Bakteri patogen ini bisa diisolasi dari bahan pangan seperti sayuran, buah-buahan, minuman tradisional, biji-bijian, bahan rempah dan susu formula serta lingkungan disekitar bahan pangan. (Jaradat et al. 2009). Di Indonesia Cronobacter spp. diisolasi dari susu formula bubuk, makanan bayi (Dewanti-Hariyadi et al. 2010; Estuningsih et al. 2006) dan produk pangan kering lainnya (Dewanti-Hariyadi et al. 2010; Hamdani 2012). Isolat-isolat lokal tersebut tidak memiliki keragaman genetik yang tinggi dan teridentifikasi sebagai Cronobacter spp. dengan polymerase chain reaction (PCR) berdasarkan gen parsial 16S rRNA (Gitapratiwi et al. 2012). Isolat-isolat lokal ini belum diketahui klasifikasinya berdasarkan sifat biokimiawi.
Berdasarkan beberapa literatur, klasifikasi Cronobacter spp. ada beberapa
jenis. Farmer et al. (1980) mengelompokkan Cronobacter spp. menjadi 15 biogrup berdasarkan produksi indol, motilitas pada suhu 36˚C, produksi asam
inositol, dulsitol, α-methyl-D-glukosidase, pemanfaatan malonat, ornitin, methyl red dan Vogas-Prokauer (VP). Kemudian Iversen et al. (2006) menambahkan biogrup 16 dari pengelompokkan yang dilakukan oleh Farmer et al. (1980) dengan profil reaksi positif terhadap inositol dan dulsitol serta reaksi negatif untuk indol. Nazarowec-White dan Farber (1999) mengelompokkan Cronobacter spp. menjadi 3 biogrup berdasarkan profil biokimia menggunakan kit API 20E. Selanjutnya, Iversen et al. (2007a) mengusulkan klasifikasi ulang bakteri patogen ini ke dalam genus Cronobacter berdasarkan pengujian biokimia dengan API 20E, ID32E, α-glukosidase dan produksi pigmen kuning serta berdasarkan gen dnaG dan gluA. Iversen et al. (2007b) menggunakan 4 rekasi biokimia (produksi asam dari dulsitol, produksi asam dari methyl-α-D-glucoside, produksi indol dan pemanfaatan malonat) untuk mengelompokkan Cronobacter spp. menjadi C. sakazakii, C. malonaticus, C. muytjensii, C. dublinensis dan C. turicensis.
2
berdasarkan metode Amplified Fragment Length Polymorphism dengan fluorescent (f-AFLP). Keragaman genetik isolat-isolat lokal Cronobacter spp. terlihat dari analisa filogeni oleh Gitapratiwi et al. (2012) melaporkan bahwa 10 isolat lokal yang diuji memiliki hubungan kekerabatan dengan C. sakazakii ATCC 29544. Begitu juga dengan Hamdani (2012) menyatakan bahwa 7 dari 12 isolat lokal Cronobacter spp. memiliki hubungan kekerabatan yang dekat dengan C. sakazakii, 4 isolat mirip dengan C. malonaticus dan 1 isolat lokal mirip dengan C. muytjensii. Hubungan kekerabatan isolat lokal tersebut dilakukan berdasarkan sekuen gen parsial 16S rRNA.
Pada penelitian Hedegaard et al. (1999) dinyatakan bahwa gen 16S rRNA tergolong multigenik rRNA (ditemukan beberapa kopi dalam satu genomnya). Oleh karena itu untuk memperoleh hasil analisa metode genetik yang lebih terpercaya, Hedegaard et al. (1999) melakukan uji terhadap gen lain yaitu mengidentifikasi Enterobacteriaceae berdasarkan sekuen parsial gen infB. Penelitian menggunakan gen infB tersebut dapat memisahkan spesies yang sama ke dalam beberapa klaster serta telah mampu mengkonfirmasi hasil klasifikasi hingga pada tingkat spesies. Selanjutnya Asakura et al. (2007) memanfaatkan keberadaan gen infB sebagai pembeda isolat E. sakazakii yang tahan panas dengan yang rentan terhadap panas.
Penelitian ini juga bertujuan untuk melakukan identifikasi dan klasifikasi bakteri Cronobacter spp. baik biotyping dengan menggunakan 4 uji biokimia Iversen dan perangkat cepat RapID onE. Disamping itu enam isolat lokal yang telah diketahui kinetika thermalnya, diklasifikasikan berdasarkan genetik menggunakan gen infB.
Tujuan Penelitian
Penelitian bertujuan untuk :
1. Melakukan identifikasi isolat-isolat lokal Cronobacter spp. dengan perangkat cepat RapID onE dan membandingkan dengan perangkat cepat API 20E. 2. Mengklasifikasikan isolat-isolat lokal Cronobacter spp. berdasarkan profil
biokimia melalui uji RapID onE.
3. Melakukan klasifikasi isolat-isolat lokal Cronobacter spp. berdasarkan produksi asam dari dulsitol, produksi asam dari methyl-α-D-glukosidase, produksi indol dan pemanfaatan malonat.
4. Mengetahui keragaman enam isolat Cronobacter spp. berdasarkan gen infB.
Manfaat Penelitian
2
TINJAUAN PUSTAKA
Enterobacter sakazakii (Cronobacter spp.)
Penamaan untuk E. sakazakii diberikan untuk menghormati ahli bakteriologi Jepang yang bernama Riichi Sakazaki. Penemuan E. sakazakii sebagai E. cloacae yang berpigmen kuning berawal dari laporan infeksi yang mengakibatkan sepsis dan meningitis di Inggris yang dilaporkan oleh Urmenyi dan Franklin pada tahun 1961 dan kasus meningitis yang terjadi di Denmark tahun 1965. Dalam kasus-kasus ini, bakteri E. cloacae berhasil diisolasi dari jaringan otak, sumsum tulang belakang dan spesimen lainnya. Pada tahun 1972 untuk pertama kalinya keluar bukti-bukti bahwa bakteri-bakteri yang digolongkan kedalam spesies E. cloacae ini masih beragam, ada yang menghasilkan pigmen kuning dan ada pula yang tidak menghasilkan pigmen kuning dan hubungan kekerabatannya hanya 50%. Nama E. sakazakii pertama kali digunakan pada pertemuan tahunan American Society for Microbiology pada tahun 1977, dan dideskripsikan E. sakazakii sebagai spesies baru oleh Farmer et al. (1980).
E. sakazakii mula-mula digolongkan sebagai anggota dari famili Enterobacteriaceae, genus Enterobacter (Nazarowec-White dan Farber 1997a). Bakteri ini merupakan bakteri gram negatif, tidak membentuk spora, berbentuk batang, bersifat anaerob fakultatif, sel motil dengan flagela peritrichous, serta memiliki ukuran dengan panjang 3 μm dan lebar 1 μm (Shaker et al. 2007; Adams dan Moss 2000; Kandhai 2010).
Sumber kontaminasi bakteri ini adalah dari klinis, lingkungan dan bahan pangan. E. sakazakii diisolasi dari cairan otak, darah, air ludah, kerongkongan, hidung, peralatan klinis, usus, kulit, luka, sumsum tulang belakang, mata dan telinga (Farmer et al. 1980). Keberadaan E. sakazakii pada lingkungan seperti tikus, serangga, air, tanah dan sampah (Iversen dan Forsythe 2003). Bakteri ini ditemukan juga pada produk-produk olahan pangan seperti susu UHT, keju, daging, sayuran, biji-bijian ataupun produk fermentasi (Gassem 1999, 2002; Iversen dan Forsythe 2003, 2004; Leclercq et al. 2002; Muytjens et al., 1988; Skaldal et al. 1993 dalam Shaker et al. 2007).
Bakteri ini dapat menginfeksi semua umur terutama bayi yang terlahir prematur kurang dari 36 minggu, berat lahir rendah, sistem kekebalan tubuh rendah dan bayi yang terlahir dari ibu yang terinfeksi HIV (Fiore et al. 2008). Kontamiasi E. sakazakii dapat terjadi secara intrinsik pada susu formula maupun secara ekstrinsik (Van Acker et al. 2001; Muytjens et al. 1983). Sifat invasif oleh E. sakazakii dapat mengakibatkan penyakit berbahaya seperti septis, meningitis dan necrotizing enterocolitis. Angka kematian untuk necrotizing enterocolitis berkisar antara 10-55% dan meningitis pada rentang antara 40-80% (Fiore et al. 2008).
4
Cronobacter spp. merupakan nama lain (sinonim) dari E. sakazakii. Bakteri ini digolongkan ke dalam genus baru Cronobacter, yang terdiri atas
spesies-spesies C. sakazakii, C. malonaticus, C. turicensis, C. muytjensii dan C. dublinensis (Iversen et al. 2007b). Metoda deteksi dan isolasi E. sakazakii
dapat juga digunakan untuk bakteri Cronobacter. Metoda isolasi Cronobacter spp. dari susu formula seperti pada Tabel 1.
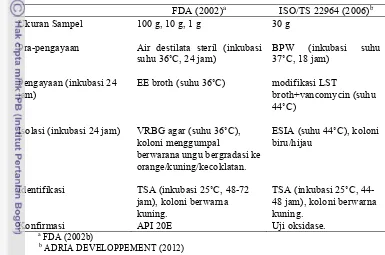
Tabel 1 Prosedur isolasi Cronobacter spp. dari susu formula
FDA (2002)a ISO/TS 22964 (2006)b
Ukuran Sampel
Pra-pengayaan
Pengayaan (inkubasi 24 jam)
Isolasi (inkubasi 24 jam)
Identifikasi
Konfirmasi
100 g, 10 g, 1 g
Air destilata steril (inkubasi suhu 36˚C, 24 jam)
EE broth (suhu 36˚C)
VRBG agar (suhu 36˚C), koloni menggumpal
berwarana ungu bergradasi ke orange/kuning/kecoklatan.
TSA (inkubasi 25˚C, 48-72 jam), koloni berwarna kuning.
API 20E
30 g
BPW (inkubasi suhu 37˚C, 18 jam) 48 jam), koloni berwarna kuning.
Uji oksidase.
a
FDA (2002b)
b
ADRIA DEVELOPPEMENT (2012)
Klasifikasi Enterobacter sakazakii (Cronobacter spp.)
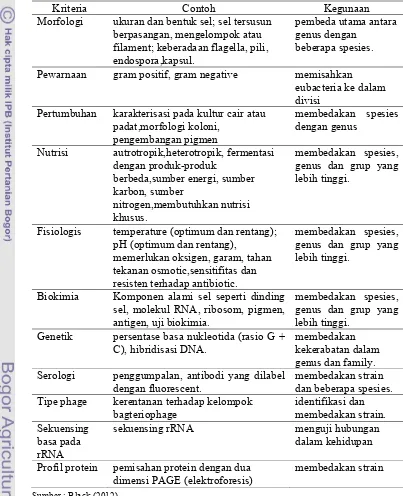
analisis sekuensing 16s rDNA) (Black 2012). Secara umum kriteria klasifikasi bakteri seperti terlihat pada Tabel 2.
Tabel 2 Kriteria untuk klasifikasi bakteri
Kriteria Contoh Kegunaan
Morfologi ukuran dan bentuk sel; sel tersusun berpasangan, mengelompok atau filament; keberadaan flagella, pili, endospora,kapsul.
pembeda utama antara genus dengan
beberapa spesies. Pewarnaan gram positif, gram negative memisahkan
eubacteria ke dalam divisi
Pertumbuhan karakterisasi pada kultur cair atau padat,morfologi koloni,
pengembangan pigmen
membedakan spesies dengan genus
Nutrisi autrotropik,heterotropik, fermentasi dengan produk-produk
berbeda,sumber energi, sumber karbon, sumber
nitrogen,membutuhkan nutrisi khusus.
membedakan spesies, genus dan grup yang lebih tinggi.
Fisiologis temperature (optimum dan rentang); pH (optimum dan rentang),
memerlukan oksigen, garam, tahan tekanan osmotic,sensitifitas dan resisten terhadap antibiotic.
membedakan spesies, genus dan grup yang lebih tinggi.
Biokimia Komponen alami sel seperti dinding sel, molekul RNA, ribosom, pigmen, antigen, uji biokimia.
membedakan spesies, genus dan grup yang lebih tinggi.
Genetik persentase basa nukleotida (rasio G + C), hibridisasi DNA.
membedakan kekerabatan dalam genus dan family. Serologi penggumpalan, antibodi yang dilabel
dengan fluorescent.
membedakan strain dan beberapa spesies. Tipe phage kerentanan terhadap kelompok
bagteriophage
sekuensing rRNA menguji hubungan dalam kehidupan Profil protein pemisahan protein dengan dua
dimensi PAGE (elektroforesis)
6
Klasifikasi Biokimia Enterobacter sakazakii (Cronobacter spp.)
Klasifikasi bakteri oleh Bergey’s manual dibedakan berdasarkan pewarnaan dan morfologi sel. Enterobacteriaceae merupakan kelompok bakteri gram negatif dan berbentuk batang. Famili Enterobacteriaceae dapat dibedakan dari Aeromonas, Pseudomonas dan Vibrio melalui pengujian oksidase. Bakteri yang tergolong ke dalam famili Enterobacteriaceae memiliki karakteristik negatif terhadap uji oksidase. Spesies-epesies di dalam famili Enterobacteriaceae dibedakan dengan pengujian fermentasi laktosa, produksi indol, uji MR-VP dan lisin decarboxylase seperti yang tertera pada Lampiran 1 (Holt 2000).
Farmer et al. (1980) mengelompokkan Cronobacter spp. menjadi 15 biogrup berdasarkan produksi indol, motilitas pada suhu 36 ˚C, produksi asam
inositol, dulsitol, α-methil-D-glukosidase, pemanfaatan malonat, ornitin, methyl red dan Vogas-Prokauer (VP). Nazarowec-White dan Farber (1999) mengelompokkan Cronobacter spp. menjadi 3 biotipe berdasarkan profil biokimia menggunakan perangkat cepat API 20E. Biotipe 1 memiliki karakteristik yang sama dengan E. sakazakii ATCC 29544. Biotipe 2 memiliki perbedaan pada uji inositol negatif dibandingkan dengan biotipe 1. Sedangkan untuk biotipe 3 memiliki karakteristik reaksi positif untuk uji VP. Iversen et al. (2006) menambahkan biogrup 16 dari pengelompokan yang dilakukan Farmer et al. (1980) dengan profil positif untuk reaksi inositol dan dulsitol positif tetapi menunjukkan reaksi negatif untuk indol. Iversen et al. (2007b) menggunakan 4 reaksi biokimia (dulsitol, produksi asam dari methyl-α-D-glucoside, produksi indol dan pemanfaatan malonat) untuk mengelompokkan Cronobacter spp. menjadi C. sakazakii, C. malonaticus, C. muytjensii, C. dublinensii, C. turicensis (Tabel 3).
Klasifikasi Genetik Enterobacter sakazakii (Cronobacter spp.)
Klasifikasi Cronobacter spp. secara genetik oleh Nazarowec-White dan Farber (1999) dilakukan berdasarkan ribotyping, pulsed-field gel electrophoresis (PFGE) dan randomly amplified polymorphic DNA (RAPD). Klasifikasi berdasarkan ribotyping tersebut, dapat mengelompokan E. sakazakii menjadi 10 ribotipe dengan menggunakan enzim restriksi endonuklease EcoR1.
Isolat yang tidak bisa dibedakan menggunakan metoda ribotyping dapat dibedakan berdasarkan RAPD. Cronobacter spp. terbagi atas 17 kelompok berdasarkan RAPD menggunakan primer UBC 245 (5’-CGC GTG CCA G-3’) dan 18 kelompok menggunakan primer UBC 282. Produk yang dihasilkan dari
Tabel 3 Uji biokimia untuk membedakan spesies Cronobacter
Dulsitol Indol Malonat AMGa
Cronobacter sakazakii - - - +
Cronobacter malonaticus - - + +
Cronobacter muytjensii + + + -
Cronobacter dublinensii - + v +
Cronobacter turicensis + - + +
Cronobacter genomospesies 1 + - + +
a
metil-α-D-glukosida
analisis dengan RAPD yakni berukuran 0.3-2.5 kb (Nazarowec-White dan Farber 1999). Analisa RAPD oleh Kim et al. (2008) menyatakan bahwa Cronobacter spp. menghasilkan 5 grup berdasarkan primer UBC 245 dan 6 grup berdasarkan primer UBC 282. Produk DNA yang dihasilkan oleh kedua primer tersebut berukuran 0.2-2.0 kb.
Klasifikasi berdasarkan PFGE dilakukan menggunakan enzim endonuklease Xba1 (5’-TCT AGA-3’) dan Spe1 (5’-ACT AGT-3’). Cronobacter spp. dapat dikelompokkan menjadi 18 kelompok menggunakan enzim endonuklease Xba1 dan 17 kelompok menggunakan enzim endonuklease Spe1 (Nazarowec-White dan Farber 1999). Klasifikasi oleh Kim et al. (2008) membagi Cronobacter spp. menjadi 8 grup berdasarkan PFGE. Keragaman genetik Cronobacter spp. menggunakan RAPD dan PFGE lebih diskriminatif dibandingkan menggunakan metoda ribotyping.
Iversen et al. (2004b) mengelompokkan Cronobacter spp. menjadi klaster 1, 2, 3 dan 4 berdasarkan gen parsial 16S rDNA dan hsp60. Sekuensing gen hsp60 menggunakan pasangan primer GGT AGA AGA AGG CGT GGT TGC-3’ dan 5’-ATG CAT TCG GTG GTG ATC ATC AG-3’. Berdasarkan gen 16S rDNA isolat Cronobacter spp. memiliki hubungan kekerabatan yang dekat dengan Citrobacter koseri (97.8%) dibandingkan dengan spesies Enterobacter lainnya. Pengelompokan isolat-isolat Cronobacter spp. berdasarkan gen 16S rDNA, disimpulkan bahwa klaster 1 dan 2 dapat dibedakan sebesar 1.6-1.9%. Klaster 2 memiliki hubungan yang lebih dekat dengan E. sakazakii. Klaster 3 memiliki hubungan yang dekat dengan E. pyrinus, E. hormaechei dan Citrobacter koseri. Klaster 4 memiliki hubungan sebesar 96.5% dengan E. sakazakii. Berdasarkan gen hsp60 klaster 1 memiliki hubungan sebesar 97.6-99.4% dengan E. sakazakii, klaster 2 sebesar 89.2-90.4%, klaster 3 sebesar 95.2% dan klaster 4 sebesar 88.8%.
Iversen et al. (2006) membandingkan sifat genotip dengan karakteristik biokimia sehingga menyatakan bahwa klaster 1 terdiri atas biogrup 1-5, 7-9,11,13 dan 14. Klaster 3 dengan biogrup 15 dan klaster 4 dengan biogrup 6, 10 dan 12, sedangkan klaster 2 terdiri atas bigrup baru 16. Iversen et al. (2007b) mengelompokkan Cronobacter spp. menjadi C. sakazakii, C. malonaticus, C. muytjensii, C. dublinensis, C. turicensis dan Cronobacter genomospesies 1.
Golongan C. sakazakii, C. malonaticus merupakan strain pada grup 1. C. muytjensii merupakan strain pada grup 3. C. dublinensis merupakan strain pada
grup 4. C. turicensis merupakan strain pada grup 2. Cronobacter genomospesies 1 yaitu strain NCTC 9529 dan E680 yang tergolong kedalam grup 2, namun memiliki perbedaan dengan C. turicensis berdasarkan Amplified Fragment Length Polymorphism (AFLP). AFLP merupakan teknik molekuler yang digunakan sebagai penanda genetik terhadap hasil amplifikasi DNA dari fragmen-fragmen DNA yang terbentuk akibat aktivitas enzim restriksi tertentu.
Peran Gen infB dan Ketahanan Panas
8
Proses translasi tersebut dimulai dengan pemisahan ribosom sub unit besar (50S) dan sub unit kecil (30S). IF3 akan berikatan dengan sub unit 30S yang mendukung pemisahan kedua sub unit tersebut. IF1 akan menstimulasi kerja IF3. Setelah kedua sub unit ribosom terpisah, asam amino metionin akan berikatan dengan inisiator tRNAfMet, sehingga fMet-tRNA dan mRNA akan saling berikatan dan masuk ke ribosom sub unit 30S. Inisiator tRNA tersebut berikatan langsung dengan kodon awal (sisi-P). Ikatan ini dipengaruhi juga oleh kerja IF1, yakni dengan memblok sisi-A. Tahapan translasi inisiasi ini, akan membuat interaksi antara kodon-antikodon sehingga terjadi perubahan konformasi sub unit 30S. IF1 dan IF3 bersifat relatif tidak stabil terhadap perubahan komformasi tersebut, sehingga IF1 dan IF3 terlepas dari 30S, selanjutnya IF2 membawa triplet mRNA, sub unit 30S dan tRNA-met secara bersamaan kemudian bergabung dengan sub unit 50S hingga terbentuk ribosom komplek 70S (Laursen et al. 2005; Madison et al. 2012).
Shazand et al. (1990) menyatakan gen infB pada bakteri Bacillus subtilis mengandung 2 148 pb (716 asam amino) dengan ATG sebagai kodon awal dan TGA sebagai kodon akhir. Pada Escherichia coli, dimana gen infBnya terletak pada operon yang sama dengan inisiator tRNA (metY), terminasi transkripsi (nusA) dan tiga protein open reading frame (ORF) yang fungsinya tidak diketahui. Pada Bacillus subtilis, gen infB tidak diikuti oleh nusA tetapi diapit oleh dua ORF (polC dan pscB). Gen infB terletak pada 145˚ kromosom Bacillus subtilis. Gen tersebut mengkode dua protein, IF2α dan IF2β yang homolog dengan IF2 pada Escherichia coli.
Hedegaard et al. (1999) menggunakan gen infB untuk mengkonfirmasi hasil klasifikasi dan untuk melihat keragaman pada level spesies di dalam genus Enterobacteriaceae. Isolat E. aerogenes diklasifikasikan sebagai isolat E. cloacae (EclAU9604) berdasarkan gen infBnya. Isolat Hafnia alvei (HalAU9601) juga diklasifikasikan ke dalam grup Escherichia coli berdasarkan analisis sekuen infB. Berdasarkan pohon filogeni, gen infB dapat memisahkan Escherichia coli menjadi dua klaster. Gen ini juga dapat memperlihatkan keragaman spesies dengan memisahkan Citrobacter diversus dan Citrobacter freundii, Klebsiella pneumonia dan Klebsiella oxytoca, E. cloacae dan E. aerogenes serta Erwinia herbicola dan Morganella morganii.
Baldwin et al. (2009) menyatakan bahwa lokus gen infB beserta 6 lokus gen lainnya (atpD, fusA, glnS, gltB, gyrB dan pps) digunakan untuk membedakan spesies C. sakazakii dan C. malonaticus menggunakan metoda multilocus sequence typing (MLST). C. sakazakii terdiri dari 12 kelompok sedangkan C. malonaticus terdiri atas 3 kelompok berdasarkan profil filogeni ketujuh lokus gen tersebut. Isolat yang tergolong C. sakazakii merupakan isolat-isolat pada biotipe 1, 2, 2a, 3, 4, 4a, 5, 7, 13 dan 13a, adapun C. malonaticus merupakan isolat-isolat di dalam biotipe 2a, 4a, 5, 5a, 8c, 9, 13a, 13b dan 14b. Hasil analisis MLST menunjukkan gen infB memiliki ukuran fragmen 441 bp.
panasnya, Cronobacter spp. terdiri dari 3 kelompok yaitu heat-resisten (kelas R), heat-sensitive (kelas S) dan intermediate (kelas M). Pengelompokkan ini dilakukan berdasarkan konsentrasi sel yang bertahan hidup setelah mendapatkan perlakuan pemanasan. Sel yang bertahan hidup lebih dari 104 sel/ml digolongkan ke dalam kelas R, kurang dari 102 sel/ml dikelompokkan ke dalam kelas S dan sel yang bertahan antara 102-104 sel/ml merupakan kelas M (Asakura et al. 2007).
Analisis terhadap Cronobacter spp. baik digunakan pada saat bakteri ini berada di fase stasioner, karena alasan kompetitif antara bakteri. Fase stasioner Cronobacter spp. berada pada suhu media 58˚C. Nilai D untuk Cronobacter spp. yang diperoleh pada suhu ini berada pada rentang 0.39-0.6 menit (Breewer et al. 2003). Menurut Edelson-Mammel dan Buchanan (2004) menyatakan bahwa nilai D untuk Cronobacter spp. bervariasi yaitu antara 0.51 hingga 10.4 menit pada suhu 58˚C. Ghassem et al. (2011) menyatakan bahwa rentang nilai D untuk isolat C. muytjensii strain ATCC 51329 dan isolat-isolat C. sakazakii dari bahan pangan berkisar antara 42.92 menit pada suhu 52˚C hingga 1.86 menit pada suhu 60˚C. Nazarowec-White dan Farber (1997b) menyatakan nilai D untuk Cronobacter spp. yaitu 4.2 menit pada suhu 58˚C. Nilai ini menindikasikan bahwa Cronobacter spp. merupakan bakteri yang tahan panas dibandingkan Enterobactericeae lainnya pada produk susu. Iversen et al. (2004a) menyatakan bahwa nilai D untuk Cronobacter spp. pada suhu 54˚C yaitu 10.2-16.4 menit. Al-Holy et al. (2009) menyatakan bahwa nilai D pada suhu 55˚C memiliki rentang yang jauh yakni 14.8 menit untuk isolat E. sakazakii ATCC 29004 hingga 1.5 menit untuk isolat E. sakazakii 55. Nilai D suhu 60˚C pada susu bubuk dilaporkan oleh Iversen et al. (2004a) sebesar 1.1 menit, dan nilai D pada suhu 62˚C yaitu antara 0.2-0.4 menit. Al-Holy et al. (2009) melaporkan bahwa nilai D pada suhu 63˚C yakni 0.88 menit.
Berdasarkan analisis nilai-nilai D tersebut, Iversen et al. (2004a) memperkirakan nilai D suhu 71.2˚C yakni 0.7 detik. Dengan demikian, Cronobacter spp. tidak mampu bertahan pada suhu pasteurisasi, karena suhu pasteurisasi yaitu 71.7 ˚C selama 15 detik. Pasteurisasi dengan metoda HTST (High Temperature Short Time) dapat mereduksi 6 log Cronobacter spp., sedangkan pemanasan selama 50 detik pada suhu 86.5˚C mampu mereduksi seluruh Cronobacter spp. (Al-Holy et al. 2009).
10
3
METODE PENELITIAN
Lokasi Penelitian dan Waktu Penelitian
Penelitian ini dilakukan di Laboratorium SEAFAST (South East Asia Food and Agricultural Science and Technology) Center, kampus IPB Darmaga, Bogor, Institut Pertanian Bogor pada bulan Juli sampai November 2012.
Bahan Penelitian dan Peralatan Penelitian
Bahan Penelitian
Isolat yang digunakan dalam penelitian ini adalah 19 isolat lokal Cronobacter spp. koleksi SEAFAST Center IPB (Tabel 4) yang telah
terkonfirmasi berdasarkan keberadaan sekuen parsial gen penyandi 16S rRNA dengan PCR. Untuk melihat keragaman genetik berdasarkan gen infBnya dipilih 6 isolat lokal (isolat DES b7a, DES b10, DES c13, YR t2a, YR c3a dan 6a) yang telah dilaporkan kinetika termalnya oleh Seftiono (2012).
Tabel 4 Daftar isolat Cronobacter spp. yang digunakan Nama Isolat Asal Isolat No. Aksesi
DES b7a Makanan bayib
Maizenab Maizenab Susu formulac Susu formulac Formula lanjutan bayid
Tepung berase Tepung terigue Tepung beras ketane
Gula haluse Pati singkonge
Cabai bubuke Cabai bubuke Cabai bubuke
Jintane Ketumbar bubuke
Pala bubuke Merica bubuke
-
Belum terdapat pada GeneBank
b
Bahan-bahan utama yang digunakan antara lain BHI broth (Oxoid Ltd., UK), TSA (Oxoid Ltd., UK), akuabides, phenol red broth, dulsitol dan perangkat cepat Rapid One untuk konfirmasi biokimia (Remel,USA). Bahan yang digunakan untuk isolasi DNA antara lain media pertumbuhan LB Broth (Miller Caisson Lab., USA), bahan-bahan ekstraksi antara lain Tris (Amersham Bioscience, Swedan), EDTA disodium salt (Amersham Bioscience, Swedan), 10% (w/v) Sodium dodecyl sulphate (SDS) (Promega Corporation, USA), 10 mg/ml proteinase K (Fermentas, US), Cethyiltrimethyl ammonium bromide (CTAB) (Merck, Darmstadt, Germany), NaCl, Phenol, Chloroform, Isoamyl alcohol (Applichem), sodium asetat (Merck, Darmstadt, Germany), isopropanol (Merck, Darmstadt, Germany), etanol 70%, dan HCl untuk pengaturan pH bufer. Bahan untuk campuran PCR yaitu akuabides steril, primer forward dan reverse (primer infB-f 5’-GCG TAA TAA ACT GTA GCA GGA A-3’ dan primer infB-r 5’-CGT TCT CTT CAG CCA TAC GAC-3’), DNA templet dari hasil ekstraksi DNA genom serta DreamTaq DNA polymerase (2× DreamTaq Green buffer, 0.4 mM dNTP, 4 mM MgCl2). Bahan yang digunakan untuk elektroforesis adalah medium agarosa (Sigma-Aldrich Chemie, Steinheim, Germany), bufer Tris-asetat-EDTA (TAE bufer), 0.5 µg/ml etidium bromida (Amersham Biosciences, Sweden), 6× loading dye dan penanda 1 kb DNA ladder (0.5 µg/µl #SM0311).
Peralatan Penelitian
Tabung reaksi bertutup beserta dudukannya, jarum ose, inkubator, pipet mikro beserta tip, vortex, eppendorf beserta kedudukannya, sentrifugasi dengan kekuatan 18 000 rpm, water bath shaker, freezer, laminar air flow (ESCO®), perangkat PCR Applied Biosystem 2720 Thermal Cycler (Foster City, California), timbangan analitik, erlenmeyer, gelas ukur, pipet volumetrik, labu takar, botol Schott Duran, pengaduk magnet, hotplate stirrer, perangkat elektroforesis (Bio-Rad), pH meter, plastik steril, bunsen, kertas parafilm, Geldoc XR (Bio-(Bio-Rad), autoklaf, termometer, perangkat dokumentasi, dan Spektrofotometer UV-Vis (Shimadzu).
Perangkat Lunak (Software)
Beberapa software yang digunakan dalam penelitian ini yaitu Eric (Remel) untuk konfirmasi hasil uji biokimia RapID onE, program ChromasPro untuk proses trimming dan contig sekuen DNA, program BLAST (Basic Local Alignment Search Tool) dari situs NCBI (www.ncbi.nih.nlm.gov) untuk menganalisis hasil perunutan, serta program MEGA4 (www.megasoftware.net) untuk membentuk pohon filogeni.
Prosedur Penelitian
12
tersebut. Untuk melihat hubungan kekerabatan isolat lokal maka dilakukan dengan analisa pohon filogeni. Tahapan amplifikasi gen diawali dengan tahapan isolasi DNA genom. Pada gambar 2 disajikan skema penelitian tahap biotyping, adapun tahap keragaman genetik dapat dilihat pada Gambar 3.
Gambar 1 Diagram Alir Penelitian
14
Klasifikasi Isolat Lokal dengan Biotyping
Persiapan Isolat Bakteri
Bakteri uji yang digunakan pada penelitian ini yaitu sebanyak 19 isolat lokal Cronobacter spp. koleksi SEAFAST Center. Semua isolat berasal dari susu formula bayi, makanan bayi, tepung-tepungan dan tanaman rempah. Pembagian isolat yang digunakan dapat terlihat pada Tabel 4. Isolat-isolat tersebut telah dikonfirmasi sebagai Cronobacter spp. menggunakan PCR terhadap gen parsial 16S rRNA (Gitapratiwi et al. 2012; Hamdani 2012). Selanjutnya isolat lokal pada media DFI agar dan CES agar diambil 1 atau 2 loop kemudian disegarkan pada media BHI broth, diinkubasi pada suhu 37˚C selama 24 jam. Satu ose isolat dari BHI broth dikultur pada media agar miring yang berisi TSA. Setelah diinkubasi pada suhu 37˚C selama 24 jam, kultur dapat digunakan sebagai kultur kerja.
Identifikasi dan Klasifikasi Isolat Lokal Cronobacter spp. dengan RapID onE
Biotyping menggunakan RapID onE merupakan perangkat cepat yang berguna untuk mengidentifikasi sifat biokimia bakteri Enterobacteriaceae. Perangkat ini berupa strip yang terdiri atas 18 sumur berisi kompartemen uji yang telah dikeringkan. Satu sumur pada RapID onE mewakili satu pengamatan uji biokimia. Pada prinsipnya, perangkat cepat RapID onE bekerja berdasarkan perubahan warna pada masing-masing sumur yang mengindikasikan reaksi positif atau negatif suatu sampel. Indikator warna untuk 19 uji biokimia dapat dilihat pada form laporan kerja RapID onE (Lampiran 2).
Masing-masing isolat dari agar miring diambil 1 hingga 2 loop untuk disuspensikan dengan larutan Remel. Kemudian larutan dituang ke dalam ujung strip dan diratakan. Setelah diinkubasi pada suhu 37˚C selama 4 jam maka dapat diamati reaksi-reaksi warna pada masing-masing sumur. Untuk mengamati reaksi indol, maka ditetesi larutan indol pada sumur yang bertuliskan ADON. Pembacaan reaksi indol setelah 2 menit ditetesi larutan indol.
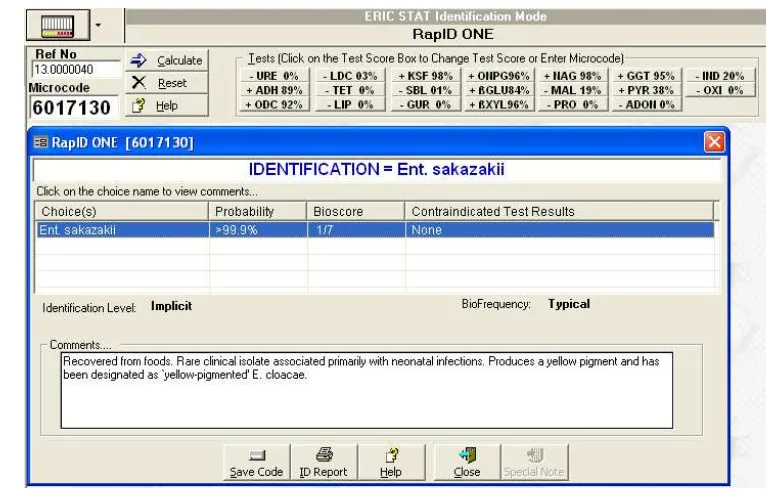
Hasil pembacaan RapID onE kemudian diinterpretasikan dengan menggunakan perangkat lunak Eric (Remel). Perangkat lunak akan bekerja dengan menghasilkan kombinasi angka, sehingga diperoleh hasil identifikasi spesies berdasarkan database perangkat tersebut serta persentase kemiripannya. Gambar 4 disajikan penampilan analisa salah satu isolat lokal Cronobacter spp. dengan perangkat lunak Eric (Remel). Hasil identifikasi RapID onE dibandingkan dengan hasil API 20E yang telah diperoleh pada penelitian Gitapratiwi (2011) dan Hamdani (2012).
memiliki karakteristik biokimia yang sama akan diklasifikasikan ke dalam satu biotipe.
Gambar 4 Penampilan Kerja Perangkat Lunak Eric (Remel)
Klasifikasi Isolat Lokal Cronobacter spp. dengan 4 Reaksi Biokimia Iversen
Dalam melakukan klasifikasi menggunakan 4 reaksi biokimia Iversen, dilakukan pengujian terhadap produksi asam dari dulsitol, methyl-α-D-glukosidase, produksi indol dan pemanfaatan malonat. Modifikasi metoda Iversen et al. (2007b) pada penelitian ini yakni tiga reaksi diantaranya diperoleh dari hasil uji pada RapID onE yaitu malonat, indol dan p-nitrophienyl-β-D-glukoside. Pengujian terhadap produksi asam dari dulsitol dilakukan berdasarkan metoda Iversen et al. (2007b) yaitu phenol red broth dilarutkan dengan larutan steril dulsitol. Konsentrasi dulsitol yang digunakan pada medium adalah 0.5% (m/v). Kemudian media diinokulasi pada tabung reaksi dan diamati perubahan warna dari merah menjadi kuning untuk hasil positif setelah diinkubasi 24 jam pada suhu 37 °C.
Selanjutnya isolat-isolat lokal akan dikelompokkan ke dalam C. sakazakii, C. malonaticus, C. muytjensii, C. dublinensis dan C. turicensis. Klasifikasi ini dilakukan berdasarkan pengamatan reaksi positif atau negatif dari keempat uji biokimia Iversen yang disesuaikan dengan pola Iversen et al. (2007b) seperti pada Tabel 3.
Hasil pengamatan sifat biokimia isolat Kombinasi angka
16
Keragaman Genetik
Persiapan Bahan Pereaksi Ekstraksi DNA
Pembuatan larutan untuk isolasi DNA dan bufer yang dibutuhkan yaitu 0.5 M EDTA pH 8 (garam disodium EDTA.2H2O dilarutkan di dalam akuabides dan ditepatkan hingga pH 8 dengan NaOH. Kemudian larutan ditambahkan akuabides hingga tepat 50 ml dan larutan disterilisasi dengan autoclave). Larutan 1 M Tris dibuat dengan cara melarutkan garam tris dengan akuabides dan diatur pH 8 dengan HCl. Selanjutnya ditambahkan akuabides hingga 50 ml dan disterilisasi dengan akuabides. Untuk membuat larutan TE pH 8, maka 1 ml EDTA 0.5 M ditambahkan dengan 5 ml Tris 1 M lalu ditambahkan akuabides hingga 500 ml. larutan disterilisasi dengan autoclave.
Bufer CTAB yang digunakan yaitu 10% CTAB dalam 0.7 M NaCl. Sebanyak 4.1 gram garam NaCl ditimbang dan dilarutkan dalam 80 ml akuabides. Kemudian ditambahkan secara perlahan 10 gram CTAB. Larutan dipanaskan sambil distirrer. Pemanasan dilakukan suhu 65˚C. Selanjutnya ditepatkan hingga 100 ml.
Larutan PCI dibuat dengan perbandingan phenol : chloroform : isoamyl alcohol = 25 : 24 : 1, sedangkan CIA dengan perbandingan chloroform : isoamyl alcohol = 24 : 1. Untuk larutan 3 M sodium asetat dibuat dengan melarutkan 408.1 gram sodium asetat 3H2O dalam 800 ml akuabides. Pengaturan pH disesuaikan menjadi 5.2 dengan menambahkan asetat glasial. Selanjutnya ditambahkan akuabides hingga volume 1 liter dan disterilisasi dengan autoclave.
Larutan-larutan lain yang diperlukan yaitu SDS 10% (w/v), proteinase K 10 mg/ml, isopropanol dan etanol 70%. Bufer 50× TAE dan EtBr diperlukan untuk visualisasi DNA pada gel agarosa. Bufer TAE dibuat dengan 242 gram tris dilarutkan ke dalam 700 ml akuabides, kemudian ditambahkan 57.1 ml asam asetat dan 100 ml EDTA 0.5 M pH 8. Larutan tersebut ditepatkan hingga 1 liter dan disterilisasi dengan autoclave.
Ekstraksi DNA
DNA ditambah dengan 500 μl etanol 70% dan disentrifugasi pada 13 500 rpm selama 10 menit. Pelet DNA dikeringkan dan diresuspensi dalam 100μl TE.
Amplifikasi DNA
Amplifikasi DNA hasil ekstraksi dilakukan secara in vitro dengan teknik PCR menggunakan mesin thermocycler (Foster City, California). Segmen DNA yang diamplifikasi adalah gen infB E. sakazakii. DNA target diamplifikasi dengan menggunakan 2 pasang primer spesifik infB-f 5’-GCG TAA TAA ACT GTA GCA GGA A-3’ dan primer infB-r 5’-CGT TCT CTT CAG CCA TAC GAC-3’. Primer dipilih berdasarkan Asakura et al. (2007) yang mengamplifikasi gen infB pada isolat E. sakazakii.
Total pereaksi yang digunakan adalah sebanyak 25 µl, terdiri atas 9 µl air bebas ion, 9 µl 2× DreamTaq Green DNA polymerase (0.4 mM dNTP, 4 mM MgCl2), 0.8 µl primer forward 10 µM, 0.8 µl primer reverse 10 µM dan 5.4 µl DNA hasil ekstraksi.
Proses amplifikasi dilakukan pada kondisi suhu pra-denaturasi 94˚C selama 4 menit, dilanjutkan 30 siklus yang terdiri atas tahap denaturasi DNA 94˚C selama 50 detik, penempelan primer (annealing) pada suhu 61˚C selama 1 menit dan sintesis DNA target (elongasi) pada suhu 72˚C selama 50 detik. Proses diakhiri dengan sintesis DNA akhir (post-PCR) pada suhu 72˚C selama 4 menit.
Elektroforesis dan Pewarnaan DNA
Elektroforesis dilakukan dengan menggunakan gel agarosa 1%. Agarosa bubuk LE-500 ditimbang dengan timbangan analitik sebanyak 0.4 g. Dilarutkan dengan menambahkan larutan bufer TAE 1× sebanyak 40 ml. Larutan tersebut dipanaskan dengan hotplate stirrer hingga jernih (kira-kira 10 menit). Ditunggu agak dingin dan larutan siap dituang ke gel tray dan dipasang sisir untuk membuat sumur-sumur pada agar dan ditunggu hingga beku.
Gel agarosa yang telah beku dipindahkan ke dalam electrophoresis chamber dengan sebelumnya melepaskan sisir pembuat sumur. Sebanyak 9 µl DNA dicampur dengan 1 µl loading dye dengan cara meneteskan pada kertas parafin. Campuran DNA dan loading dye diambil dan dimasukkan ke dalam sumur-sumur gel agarosa dengan perlahan. Diikuti dengan penenda 1 kb pada masing-masing sumur berbeda. Kemudian alat elektroforesis dijalankan dengan tegangan 110 volt selama 30 menit. DNA merupakan molekul bermuatan negatif sehingga bila diletakkan di medan listrik maka DNA akan bermigrasi dari kutub negatif ke kutub positif. Kecepatan pergerakan DNA tergantung pada ukuran molekul DNA, kerapatan gel, dan arus listrik yang diberikan untuk memigrasikan molekul DNA. Pewarna (loading dye) ditambahkan untuk memudahkan peletakan sampel DNA ke dalam sumur. Loading dye juga berfungsi agar DNA dapat bergerak ke arah anoda dengan laju yang diperkirakan sehingga dapat digunakan sebagai tanda migrasi DNA.
18
Hasil Elektroforesis
Hasil elektroforesis bersifat kualitatif dalam bentuk pita-pita pada permukaan agarosa. Ukuran produk DNA diperoleh dengan membandingkan fragmen DNA masing-masing sampel dengan penenda DNA ladder (Gambar 5).
Perunutan (Sekuensing) Fragmen Gen infB
Produk hasil PCR dimurnikan sebelum proses sekuensing. Pemurnian produk PCR perlu dilakukan untuk mengurangi pengotor-pengotor berupa Mg2+, DNA template dan dNTP yang berlebih yang dapat mengganggu proses perunutan basa nukleotida sehingga dapat menimbulkan kesalahan dalam pembacaan hasil sekuensing. Proses pemurnian dan sekuensing fragmen Gen infB dilakukan menggunakan jasa sekuensing di 1st Base Pte Ltd, Malaysia.
Alignment Gen infB Isolat Lokal Cronobacter spp.
Produk hasil sekuensing dilanjutkan pada tahapan trimming dan contig menggunakan program ChromasPro. Proses trimming dilakukan untuk membetulkan hasil sekuen yang kurang tepat dan menghilangkan yang tidak dibutuhkan. Basa yang tidak sesuai dengan kromatogramnya akan ditandai dengan blok hitam secara otomatis. Proses contig dilakukan untuk menggabungkan dua sekuen hasil forward dan reverse. Hasil sekuen contig yang diperoleh akan dilinkkan ke program BLAST dari NCBI (the National Center of Biotechnology Information) pada situs http://www.ncbi.nlm.nih.gov. Hasil BLAST merupakan identitas dari gen yang diamplifikasi berdasarkan tingkat kemiripannya dengan database yang tersedia.
Program BLAST nukleotida juga digunakan untuk melihat tingkat kemiripan antar isolat lokal Cronobacter spp. ataupun tingkat kemiripan dengan isolat lain dalam Cronobacter. Sekuen contig gen infB sampel disepadankan dengan beberapa genom komplit Cronobacter spp. yang ada di data Gen Bank.
Selanjutnya program MEGA4 (Molecular Evolutionary Genetics Analysis) untuk membentuk pohon filogeni.
4
HASIL DAN PEMBAHASAN
Identifikasi Biokimia Isolat Lokal Cronobacter spp.
Identifikasi berdasarkan RapID onE
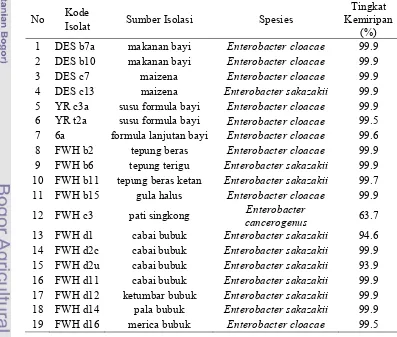
Dari hasil identifikasi menggunakan RapID onE dapat disimpulkan bahwa dari 19 isolat uji ada 9 isolat yang teridentifikasi sebagai E. sakazakii (FWH d2c, FWH d14, FWH d1, FWH b11, FWH d2u, FWH d11, FWH b6, FWH d12 dan DES c13) dengan tingkat kemiripan antara 93.9 hingga 99.9%. Sembilan dari 19 isolat lainnya (FWH b2, FWH d16, FWH b15, DES c7, 6a, YR c3a, YR t2a, DES b10 dan DES b7a) teridentifikasi sebagai E. cloacae dengan tingkat kemiripan 99.5-99.9%, sementara itu satu isolat (FWH c3) yang teridentifikasi sebagai E. cancerogenus (63.7%) (Tabel 5).
Tabel 5 Hasil identifikasi takson dan tingkat kemiripan isolat lokal Cronobacter spp. dengan program ERIC RapID onE
No Kode
Isolat Sumber Isolasi Spesies
Tingkat Kemiripan
(%) 1 DES b7a makanan bayi Enterobacter cloacae 99.9 2 DES b10 makanan bayi Enterobacter cloacae 99.9 3 DES c7 maizena Enterobacter cloacae 99.9 4 DES c13 maizena Enterobacter sakazakii 99.9 5 YR c3a susu formula bayi Enterobacter cloacae 99.9 6 YR t2a susu formula bayi Enterobacter cloacae 99.5 7 6a formula lanjutan bayi Enterobacter cloacae 99.6 8 FWH b2 tepung beras Enterobacter cloacae 99.9 9 FWH b6 tepung terigu Enterobacter sakazakii 99.9 10 FWH b11 tepung beras ketan Enterobacter sakazakii 99.7 11 FWH b15 gula halus Enterobacter cloacae 99.9 12 FWH c3 pati singkong Enterobacter
20
Kemampuan bakteri dalam memfermentasi karbohidrat tertentu, dapat digunakan untuk mengidentifikasi spesies berbeda. Fermentasi karbohidrat seperti adonitol maupun sorbitol pada medium pengujian dapat diamati melalui perubahan warna media. Hal ini dapat terjadi karena bakteri yang memfermentasi adonitol maupun sorbitol akan menghasilkan produk akhir berupa asam atau asam dan gas, yang mengakibatkan penurunan pH media, sehingga komponen indikator yang terdapat di dalam media akan menimbulkan perbedaan warna.
Stephan et al. (2008) menyatakan perbedaan profil biokimia isolat E. sakazakii dan E. cloacae yakni reaksi negatif dalam memfermentasi mucate
dan sorbitol untuk isolat E. sakazakii. Menurut Iversen et al. (2007b) bahwa E. sakazakii digolongkan ke dalam Cronobacter spp. karena memiliki karakteristik reaksi negatif terhadap sorbitol.
Sorbitol dan adonitol adalah parameter pembeda spesies E. sakazakii dengan E. cloacae berdasarkan analisis RapID onE. Reaksi negatif terhadap sorbitol merupakan karakteristik dari E. sakazakii, adapun E. cloacae memiliki karakteristik positif terhadap sorbitol. Hal ini terlihat pada isolat-isolat FWH d16, FWH b15, DES c7, 6a, YR c3a, YR t2a, DES b10 dan DES b7a memberikan reaksi biokimia positif terhadap produksi sorbitol, sedangkan isolat-isolat FWH d2c, FWH d14, FWH d1, FWH b11, FWH d2u, FWH d11, FWH b6, FWH d12 dan DES c13 negatif (Lampiran 3).
Salah satu manfaat sorbitol dilaporkan oleh De Barros dan Celligoi (2006) yakni sebagai respon untuk melindungi sel bakteri dari lingkungan berbahaya, seperti tekanan osmotik yang tinggi. Namun, Breeuwer et al. (2003) menyatakan bahwa penambahan sorbitol pada media tumbuh tidak mempengaruhi ketahanan hidup E. sakazakii akibat adanya tekanan osmotik. Laporan tersebut mendukung hasil penelitian ini bahwa E. sakazakii memiliki karakteristik tidak mampu memfermentasi sorbitol. Dengan demikian sorbitol tidak akan dimanfaatkan oleh E. sakazakii untuk bertahan hidup jika mendapat tekanan dari lingkungannya.
Perbedaan karakteristik E. sakazakii dengan E. cloacae berdasarkan RapID onE pada isolat FWH b2 adalah reaksi biokimia adonitolnya. Dimana isolat FWH b2 memiliki reaksi biokimia positif terhadap adonitol, sehingga teridentifikasi sebagai E. cloacae. Untuk teridentifikasi sebagai E. sakazakii, maka isolat ini harus menghasilkan reaksi adonitol yang negatif.
Isolat FWH c3 diindikasikan sebagai spesies E. cancerogenus (63.7%).
Faktor pembeda isolat ini dari spesies E. sakazakii yaitu keberadaan p-Nitrophenil-β-D-xyloside. Isolat FWH c3 menunjukkan reaksi negatif untuk
p-Nitrophenil-β-D-xyloside, sedangkan isolat lain yang terindikasi sebagai E. sakazakii memiliki reaksi biokimia positif terhadap p-Nitrophenil-β-D-xyloside.
pentosa fosfat atau memiliki gen penyandi xylose reductase dan xylitol dehydrogenase.
Dari pengujian biokimiawi pada penelitian ini diketahui bahwa E. sakazakii memiliki karakteristik umum reaksi positif terhadap arginin, ornitin, gula aldehid,
β-galaktosidase, p-nitrophienyl-β-D-xyloside, T-glutamyl-β-naphtylamide, pyrohidonyl-β-naphtylamide, penggunaan sitrat, produksi asetoin (uji Voges-Proskauer), fermentasi sakarosa, melibiosa, amigladin, arabinosa, ramnosa dan manitol. Sebaliknya E. sakazakii memberikan reaksi negatif terhadap produksi indol, produksi H2S, adonitol, prolin-β-naphthylamide, p-nitrophenyl-β -D-glucoramide, asam lemak ester, aliiphatic thiol, fermentasi sorbitol dan urea.
Jika isolat-isolat lokal yang teridentifikasi sebagai E. sakazakii dibandingkan dengan biogrup Farmer et al. (1980), maka isolat FWH d2c, FWH d2u dan FWH d12 adalah biogrup 1; isolat d14 dan FWH b6 adalah biogrup 2 karena memiliki persamaan karakteristik biokimia inositol negatif; FWH d1 merupakan biogrup 11 karena memiliki karakteristik biokimia inositol negatif dan dulsitol positif; dan isolat FWH d11 adalah biogrup 15 karena memiliki persamaan reaksi indol, malonat dan dulsitol positif.
Bakteri E. cloacae memiliki karakteristik memberikan reaksi positif terhadap arginin, ornitin, sorbitol, gula aldehid, β-galaktosidase, p-nitrophienyl-β -D-xyloside, T-glutamyl-β-naphtylamide, p-nitrophenyl-n-acetyl-β-D-gLucoamide, penggunaan sitrat, produksi asetoin (uji Voges-Proskauer), fermentasi inositol, sakarosa, melibiosa, amigladin, arabinosa, ramnosa dan manitol. Bakteri ini meberikan reaksi negatif terhadap oksidase, lisin, urea, produksi indol, asam lemak ester, aliphatic thiol, p-nitrophenyl-β-D-glucoramide, prolin-β -naphthylamide dan produksi H2S. Menurut Stephan et al. (2008) perbedaan karakteristik E. cloacae dan E. cancerogenus yaitu fermentasi melibiosa, sakarosa, sorbitol dan methyl-α-galaktopyranoside. Dibandingkan dengan isolat lokal pada penelitian ini, hanya sorbitol yang memberikan perbedaan karakteristik antara E. cloacae dan E. cancerogenus.
Karakteristik biokimia isolat E. cancerogenus yaitu memiliki reaksi positif terhadap arginin, ornitin, gula aldehid, p-nitrophenyl-n-acetyl-β-D-gLucoamide,
β-galaktosidase, fermentasi glukosa, T-glutamyl-β-naphtylamide, pyrohidonyl-β -naphtylamide, penggunaan sitrat, produksi aseton (uji Voges-Proskauer), fermentasi sakarosa, melibiosa, amigladin, arabinosa, ramnosa dan manitol. E. cancerogenus memberikan reaksi negatif terhadap lisin, urea, oksidase, produksi indol, aliphatic thiol, asam lemak ester, p-nitrophenyl-β-D-glucoramide, p-nitrophienyl-β-D-xyloside, malonat, prolin-β-naphthylamide, produksi H2S dan fermentasi inositol. Stephan et al. (2008) melaporkan bahwa E. cancerogenus memiliki karakteristik fenotip sebagai berikut reaksi positif terhadap uji Voges-Prokauer, ornitin, arginin, motilitas, hidolisis askulin, memfermentasi mucate dan fukosa serta reaksi-reaksi negatif terhadap fermentasi melibiosa, sakarosa, arabitol, sorbitol dan methyl-α-galaktopyranoside. Isolat yang sama-sama teridentifikasi sebagai E. cancerogenus berdasarkan Stephan et al. (2008) maupun berdasarkan penelitian ini memiliki perbedaan karakteristik biokimia dalam memfermentasi melibiosa dan sakarosa.
sama-22
sama berasal dari cabai bubuk dan teridentifikasi sebagai E. sakazakii, memiliki perbedaan profil biokimia dalam pembentukan urease. El-Sharoud et al. (2009) juga menemukan perbedaan profil biokimia antar isolat dalam satu spesies, dimana terdapat 3 dari 12 isolat C. sakazakii memiliki reaksi positif terhadap keberadaan malonat, dan 1 dari 12 isolat memiliki reaksi positif terhadap dulsitol dan indol. Oleh karena itu klasifikasi isolat lokal Cronobacter spp. terdiri atas beberapa biotipe berdasarkan sifat biokimiawinya.
Perbandingan Hasil Identifikasi Isolat Cronobacter spp. dengan RapID onE dan API 20E
Identifikasi dengan RapID onE memberikan hasil yang berbeda dengan hasil identifikasi dengan menggunakan API 20E, ternyata hanya 8 dari 19 isolat uji (FWH b6, FWH b11, FWH d1, FWH d2c, FWH d2u, FWH d11, FWH d12 dan FWH 14) sama-sama teridentifikasi sebagai E. sakazakii. Tujuh dari 19 isolat uji (DES b7a, DES b10, YR c3a, YR t2a, FWH b2, FWH b15 dan FWH d16) teridentifikasi sebagai E. cloacae dengan RapID onE tetapi teridentifikasi sebagai E. sakazakii dengan API 20E. Dua isolat uji lainnya (DES c7 dan DES c13) teridentifikasi sebagai bakteri non Entrobacter dengan API 20E tetapi berdasarkan RapID onE isolat DES c7 teridentifikasi sebagai E. cloacae dan isolat DES c13 teridentifikasi sebagai E. sakazakii (Tabel 6).
Perbedaan kombinasi parameter uji biokimia oleh kedua perangkat cepat ini menghasilkan identifikasi spesies bakteri yang berbeda pula. Dari 30 uji biokimia yang dijadikan sebagai parameter penentuan hasil identifikasi, hanya 8 uji (sebesar 27%) yang sama-sama digunakan oleh kedua perangkat cepat tersebut. Sebelas parameter uji biokimia yang digunakan pada RapID onE adalah aliphatic thiol, fatty acid ester,sugar aldehid, p-nitrophenyl-β-D-glucoramide, p-nitrophenyl-n-acetyl-β-D-gLucoamide, p-nitrophienyl-β-D-xyloside, malonat, prolin-β-naphthylamide, T-glutamyl-β-naphtylamide, pyrohidonyl-β-naphtylamide dan adonitol. Parameter uji biokimia yang terdapat pada API 20E adalah penggunaan sitrat, produksi H2S, gelatinase, uji VP, fermentasi inositol, sakrosa, melibiosa, amigladin, arabinosa, ramnosa dan manitol.
Isolat FWH c3 tergolong sebagai spesies E. cloacae berdasarkan API 20E, namun tergolong E. cancerogenus berdasarkan RapID onE. Hal ini disebabkan karena perbedaan interpretasi oleh kedua kit. Hasil dengan API 20E menunjukkan reaksi positif terhadap fermentasi glukosa, namun reaksi negatif terhadap p-Nitophenyl-β-D-glucoside menggunakan Rapid onE. Pengujian fermentasi glukosa maupun p-Nitophenyl-β-D-glucoside bertujuan untuk mengetahui aktivitas penggunaan glukosa pada isolat FWH c3 dalam proses pengidentifikasian bakteri. Aktivitas penggunaan glukosa dengan API 20E dilakukan secara kualitatif. Tube pada perangkat cepat API 20E terkandung senyawa glukosa, sehingga isolat yang mampu memfermentasi glukosa, akan memanfaatkan senyawa tersebut dan menghasilkan asam. Kondisi asam akan mengubah indikator fenil merah menjadi kuning. Dalam hal ini membuktikan isolat FWH c3 bersifat fermentatif terhadap glukosa. Akan tetapi pada perangkat cepat RapID onE menggunakan pendekatan kuantitatif yakni menggunakan
substrat p-Nitrophenyl-β-D-glucoside untuk memonitor aktivitas enzim
β-D-glukosa. Hasil uji negatif pada kit ini mengindikasikan bahwa isolat FWH c3 tidak memiliki aktivitas enzim β-glukosidase atau dimungkinkan juga aktivitasnya yang sangat rendah sehingga tidak terdeteksi oleh perangkat cepat tersebut.
Tabel 6 Perbandingan identifikasi isolat lokal Cronobacter spp. menggunakan RapID onE dengan API 20E
Kode Isolat
RapID onE API 20Ea
Spesies
DES b7a Enterobacter
cloacae 99.9
Enterobacter
sakazakii 99.9
DES b10 Enterobacter
cloacae 99.9
Enterobacter
sakazakii 99.9
DES c7 Enterobacter
cloacae 99.9 Pantoea spp. 3 44.4
DES c13 Enterobacter
sakazakii 99.9 Rahnella aquatilis 40.2
YR c3a Enterobacter
cloacae 99.9
Enterobacter
sakazakii 18.5
YR t2a Enterobacter
cloacae 99.5
FWH b2 Enterobacter
cloacae 99.9
Enterobacter
sakazakii 98.4
FWH b6 Enterobacter
sakazakii 99.9
Enterobacter
sakazakii 51.9
FWH b11 Enterobacter
sakazakii 99.7
Enterobacter
sakazakii 98.4
FWH b15 Enterobacter
cloacae 99.9
Enterobacter
sakazakii 98.4
FWH c3 Enterobacter
cancerogenus 63.7
Enterobacter
cloacae 69.1
FWH d1 Enterobacter
sakazakii 94.6
Enterobacter
sakazakii 99.1
FWH d2c Enterobacter
sakazakii 99.9
Enterobacter
sakazakii 99.9
FWH d2u Enterobacter
sakazakii 93.9
Enterobacter
sakazakii 99.9
FWH d11 Enterobacter
sakazakii 99.9
Enterobacter
sakazakii 99.9
FWH d12 Enterobacter
sakazakii 99.9
Enterobacter
sakazakii 99.9
FWH d14 Enterobacter
sakazakii 99.9
Enterobacter
sakazakii 51.9
FWH d16 Enterobacter
cloacae 99.5
Enterobacter
sakazakii 98.4
a
Meutia (2008); Gitapratiwi (2011); Hamdani (2012)
b
24
Kemungkinan lain diasumsikan bahwa isolat FWH c3 hanya mampu memfermentasi glukosa dalam bentuk siklik a trans (α-D-glukosa) dan diperoleh dari hasil hidrolisis enzim α-glukosidase. Hal ini lah yang mengakibatkan terjadinya perbedaan interpretasi oleh kedua kit (API 20E dan RapID onE) pada isolat FWH c3.
Klasifikasi Cronobacter spp.Berdasarkan 4 Reaksi Biokimia Iversen
Klasifikasi isolat-isolat lokal Cronobacter spp. dengan 4 reaksi biokimia Iversen dikelompokkan menjadi lima kelompok. Kelompok pertama isolat DES b7a, YR t2a, FWH d16, FWH d1, FWH d14 dan FWH d2c tergolong ke dalam spesies C. sakazakii. Kelompok kedua isolat DES b10, YR c3a, FWH b2, FWH b11 dan FWH b15 tergolong C. malonaticus. Kedua kelompok ini dibedakan terhadap reaksi malonat, dimana C. malonaticus memiliki reaksi positif terhadap
malonat. Isolat DES c13 dan FWH d11 digolongkan ke dalam kelompok C. muytjensii. Kelompok keempat isolat DES c7 dan FWH d2u sebagai C. turicensis. Isolat-isolat tersebut memiliki reaksi dulsitol positif. Sedangkan
kelompok kelima yakni isolat-isolat yang tidak teridentifikasi sebagai Cronobacter spp. berdasarkan 4 reaksi biokimia Iversen. Hal ini karena isolat-isolat 6a, FWH c3, FWH b6 dan FWH d12 menghasilkan reaksi negatif terhadap Tabel 7 Klasifikasi isolat lokal Cronobacter spp. berdasarkan 4 reaksi biokimia
Iversen
No Kode
Isolat
AMG Mal Ind Dul
Klasifikasi
(+/-) (+/-) (+/-) (+/-)
1 DES c13 - + + + Cronobacter muytjensii
2 DES b7a + - - - Cronobacter sakazakii subsp. sakazakii
3 DES b10 + + - - Cronobacter sakazakii subsp. malonaticus
4 YR t2a + - - - Cronobacter sakazakii subsp. sakazakii
5 YR c3a + + - - Cronobacter sakazakii subsp. malonaticus
6 6a - - - - td
7 DES c7 + + - + Cronobacter turicensis
8 FWH b15 + + - - Cronobacter sakazakii subsp. malonaticus
9 FWH d16 + - - - Cronobacter sakazakii subsp. sakazakii
10 FWH b2 + + - - Cronobacter sakazakii subsp. malonaticus
11 FWH c3 - - - - td
12 FWH b6 - - - - td
13 FWH d11 - + + + Cronobacter muytjensii
14 FWH d2u + + - + Cronobacter turicensis
15 FWH b11 + + - - Cronobacter sakazakii subsp. malonaticus
16 FWH d1 + + - + Cronobacter turicensis
17 FWH d14 + - - - Cronobacter sakazakii subsp. sakazakii
18 FWH d2c + - - - Cronobacter sakazakii subsp. sakazakii
19 FWH d12 - - - - td
produksi asam dari methyl-α-D-glukosidase, produksi indol dan pemanfaatan malonat (Tabel 7).
Berdasarkan analisis 19 uji biokimia pada RapID onE, maka diketahui bahwa reaksi pyrohydonyl-β-naphthylamide membentuk reaksi yang berbeda pada 4 spesies dan dapat digunakan sebagai uji tambahan untuk membedakan isolat lokal. Chagla et al. (1993) menyatakan bahwa L-pyrrolidonyl-β -naphthylamide merupakan substrat yang digunakan untuk mendeteksi aktivitas pyrrolidonyl peptidase yang dapat membedakan bakteri gram negatif dan gram positif. Dengan melakukan kombinasi dengan reaksi malonat, produksi asam dulsitol dan produksi indol serta pyrohydonyl-β-naphthylamide (Tabel 8), maka isolat-isolat lokal yang belum teridentifikasi ke dalam kelompok Cronobacter spp. (6a, FWH c3, FWH b6 dan FWH d12) dapat diklasifikasikan ke dalam kelompok C. sakazakii.
Tabel 8 Uji biokimia untuk membedakan spesies lokal Cronobacter spp.
Dulsitol Indol Malonat PYRa
Cronobacter sakazakii - - - +
Cronobacter malonaticus - - + -
Cronobacter muytjensii + + + +
Cronobacter dublinensis - + v v
Cronobacter turicensis + - + +
apyrohydonyl-β-naphthylamide
Biotyping Isolat Lokal Cronobacter spp. Berdasarkan RapID onE dan Perbandingannya Berdasarkan 4 Reaksi Biokimia
Dari 19 uji biokimia pada RapID onE, 9 diantaranya tidak digunakan sebagai parameter pembeda antar isolat lokal. Uji arginin, ornitin, sugar aldehid, 0-nitrophenyl-β-D-galactosidase dan T-glutamyl-β-naphtylamide menghasilkan reaksi positif untuk semua isolat lokal. Adapun pengujian untuk urease, aliphatic thiol, p-nitrophenyl-β-D-glucoramide dan prolin-β-naphthylamide memberikan reaksi negatif terhadap semua isolat lokal. Sepuluh dari 19 reaksi biokimia yang diujikan pada RapID onE memiliki pola spesifik dalam mengelompokkan isolat lokal Cronobacter spp. Reaksi-reaksi tersebut diantaranya pemanfaatan malonat, sorbitol, metabolisme substrat p-nitrophienyl-β-D-glukoside, pyrohidonyl-β -naphtylamide, p-nitrophenyl-n-acetyl-β-D-gLucoamide, produksi lisin, indol, fermentasi adonitol, produksi asam lemak ester dan memetabolisme substrat p-nitrophienyl-β-D-xyloside.
p-26
nitrophienyl-β-D-xyloside. Biotipe IV merupakan isolat dengan karakteristik rekasi negatif terhadap malonat, sorbitol dan positif terhadap p-nitrophienyl-β -D-glukoside dan lisin. Biotipe V merupakan isolat yang memiliki karakteristik reaksi
negatif terhadap malonat, sorbitol dan lisin serta reaksi positif terhadap p-nitrophienyl-β-D-glukoside. Biotipe VI merupakan isolat yang memiliki
karakteristik reaksi negatif terhadap malonat, p-nitrophienyl-β-D-glukoside dan positif terhadap sorbitol. Biotipe VII merupakan isolat yang memiliki karakteristik reaksi negatif terhadap malonat dan adonitol serta positif terhadap sorbitol. Biotipe VIII merupakan isolat yang memiliki karakteristik reaksi negatif terhadap malonat serta positif terhadap sorbitol dan adonitol. Biotipe IX merupakan isolat yang memiliki karakteristik reaksi negatif terhadap sorbitol, pyrohidonyl-β -naphtylamide serta positif terhadap malonat dan lisin. Biotipe X merupakan isolat yang memiliki reaksi positif terhadap malonat serta negatif terhadap sorbitol, pyrohidonyl-β-naphtylamide dan lisin. Biotipe XI merupakan isolat yang memiliki
reaksi negatif terhadap sorbitol dan p-nitrophenyl-n-acetyl-β-D-glucoamide serta positif terhadap malonat. Biotipe XII merupakan isolat yang memiliki karakteristik reaksi negatif terhadap sorbitol dan indol serta positif terhadap malonat dan p-nitrophenyl-n-acetyl-β-D-glucoamide. Biotipe XIII merupakan isolat yang memiliki karakteristik reaksi negatif terhadap sorbitol serta positif terhadap malonat dan indol. Biotipe XIV merupakan isolat yang memiliki karakteristik reaksi negatif terhadap sorbitol dan p-nitrophienyl-β-D-glukoside serta positif terhadap malonat. Biotipe XV merupakan isolat yang memiliki karakteristik reaksi negatif terhadap p-nitrophienyl-β-D-glukoside serta positif terhadap malonat dan sorbitol. Sedangkan biotipe XVI merupakan isolat yang memiliki karakteristik reaksi positif terhadap malonat, sorbitol dan
p-nitrophienyl-β-D-glukoside.
Jika hasil biotyping dengan perangkat RapID onE dibandingkan dengan 4 uji biokimiawi Iversen et al. (2007b), maka biotipe I-VIII adalah spesies C. sakazakii; biotipe IX, X, XV dan XVI adalah spesies C. malonaticus; biotipe XIII dan XIV adalah spesies C. muytjensii; serta biotipe XI, XII dan XVI adalah
spesies C. turicensis. Masing-masing kelompok tersebut (C. sakazakii, C. malonaticus, C. muytjensii dan C. turicensis) dapat dibedakan juga dengan
karakteristik fenotip seperti pada Tabel 10.
Keragaman Genotip Gen infBCronobacter spp.
Kekuatan dasar dari analisis mikroorganisme menggunakan metoda genetik karena kestabilan yang tinggi dari sekuen nukleotida pada DNA. Untuk melakukan isolasi DNA tersebut digunakan metoda ekstraksi phenol-chloroform.
Tabel 10 Karakteristik fenotip Cronobacter spp.
KARAKTER
C.sakazakii subsp. sakazakii
C.sakazakii subsp. malonaticus
C.muytjensii C.turicensis
MAL - + + +
SBL v v - (-)
βGLU v (+) v +
PYR + - + (+)
NAG (+) (+) + (+)
LDC (-) (-) - -
IND - - + -
ADON (-) (-) - -
LIP - - v -
βXYL (+) + + +