KOMPUTASI PARALEL
MULTIPLE SEQUENCE
ALIGNMENT
MENGGUNAKAN
MESSAGE PASSING
INTERFACE
RAMDAN SATRA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Komputasi Paralel Multiple Sequence Alignment menggunakan Message Passing Interface adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
RAMDAN SATRA. Komputasi Paralel Multiple Sequence Alignment menggunakan Message Passing Interface. Dibimbing oleh WISNU ANANTA KUSUMA dan HERU SUKOCO.
Multiple sequence alignment (MSA) adalah teknik untuk menemukan kesamaan dalam banyak sekuen. Teknik ini sangat penting untuk mendukung banyak tugas bioinformatika seperti mengidentifikasi single nucleotide polymorphism (SNP), membuat phylogenetic tree, dan metagenome fragments binning. Algoritma sederhana untuk MSA adalah Algoritma Star.
Algoritma ini terdiri atas tiga tahap, yaitu menjajarkan semua kemungkinan pasangan sekuen, menentukan sekuen star yang dipilih dari sekuen yang memiliki nilai penjajaran maksimum, dan menjajarkan semua sekuen terhadap sekuen star. Setiap pasangan sekuen dijajarkan dengan menggunakan teknik pemrograman dinamis. Kompleksitas penjajaran sekuen DNA menggunakan teknik pemrograman dinamis mengikuti fungsi eksponensial. Waktu komputasi meningkat secara eksponensial seiring dengan meningkatnya jumlah dan panjang sekuen DNA. Penelitian ini bertujuan untuk mempercepat perhitungan MSA menggunakan message passing interface (MPI).
Evaluasi kinerja dari metode yang diusulkan dilakukan dengan menghitung speedup. Percobaan dilakukan dengan menggunakan data Glycine max-kromosom-9-BBI dengan jumlah 64 sekuen yang memiliki panjang seragam, yaitu 800 base pair (bp). Sekuens ini dihasilkan dengan cara memotong secara acak sekuen referensi dari Glycine max-kromosom-9-BBI yang bersumber dari national center for biotechnology information (NCBI). Hasil penelitian menunjukkan bahwa teknik yang diusulkan mampu menghasilkan speedup sebesar tiga kali, dengan menggunakan lima komputer. Selain itu, dapat ditarik kesimpulan bahwa peningkatan jumlah komputer akan meningkatkan speedup. Kata kunci : multiple sequence alignment, message passing interfaces, komputasi
SUMMARY
RAMDAN SATRA. Multiple Sequence Alignment Parallel Computing using Message Passing Interface. Supervised by WISNU ANANTA KUSUMA and HERU SUKOCO.
Multiple sequence alignment (MSA) is a technique for finding similarity in many sequences. This technique is very important to support many Bioinformatics task such as identifying single nucleotide polymorphism (SNP), generating phylogenetic tree, and metagenome fragments binning. The simplest algorithm in MSA is star algorithm.
This algorithm consists of aligning all possible pairs of sequences, finding a sequence Star chosen from sequence that has maximum alignment score, and aligning all sequences referred to the sequence Star. Each of pairwise alignments is conducted using dynamic programming technique. The complexity of DNA multiple sequence alignment using dynamic programming technique is very high. The computation time is increased exponentially due to the increasing of the number and the length of DNA sequences. This research aims to accelerate computation of star multiple sequence alignment using message passing interfaces (MPI).
The performance of the proposed method was evaluated by calculating speedup. Experiment was conducted using 64 sequences of 800 base pair Glycine-max-chromosome-9-BBI fragments yielded by randomly cut from reference sequence of Glycine-max-chromosome-9-BBI taken from national center for biotechnology information (NCBI). The results showed that the proposed technique could obtain speed up three times using five computers when aligning 64 sequences of Glycine-max-chromosome-9-BBI fragments. Moreover, the increasing of the number of computers would significantly increase speed up of the proposed technique.
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Komputer
pada
Program Studi Ilmu Komputer
KOMPUTASI PARALEL
MULTIPLE SEQUENCE
ALIGNMENT
MENGGUNAKAN
MESSAGE PASSING
INTERFACE
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2014
Judul Tesis : Komputasi Paralel Multiple Sequence Alignment menggunakan Message Passing Interface
Nama : Ramdan Satra NIM : G651120591
Disetujui oleh Komisi Pembimbing
DrEng Wisnu Ananta Kusuma, ST MT Ketua
DrEng Heru Sukoco, SSi MT Anggota
Diketahui oleh
Ketua Program Studi Ilmu Komputer
DrEng Wisnu Ananta Kusuma, ST MT
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Desember 2013 ini ialah Pemrosesan Paralel Penjajaran Sekuen DNA, dengan judul Komputasi Paralel Multiple Sequence Alignment menggunakan Message Passing Interface.
Terima kasih penulis ucapkan kepada Bapak Dr Wisnu Ananta Kusuma dan Bapak Dr Heru Sukoco selaku pembimbing. Di samping itu, penghargaan penulis sampaikan kepada Bapak Muhammad Adi Puspo Sujiwo yang telah membantu dalam pembuatan Cluster Komputer dan kepada bapak Dr. Ir. Yandra Arkeman M.Eng yang membantu memfasilitasi beberapa unit Komputer. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 3
Sequence alignment 3
Dynamic Programming Algorithm 3
Needleman-Wunsch Algorithm 4
Multiple Sequence Alignment (MSA) 7
Star Alignment 7
Komputasi Paralel 9
Pemrograman Paralel 10
Message Passing Interface (MPI) 12
Penjadwalan Round Robin 12
3 METODE 13
Tahapan Penelitian 13
Bahan 15
Alat 15
4 HASIL DAN PEMBAHASAN 16
Persiapan Data 16
Desain Aplikasi Sekuensial 16
Desain Aplikasi Paralel 21
Analisis Hasil 22
a. Waktu eksekusi penjajaran sekuen 22
b. Analisa Speedup 23
c. Analisa Efisiensi 24
5 SIMPULAN DAN SARAN 25
DAFTAR PUSTAKA 26
DAFTAR GAMBAR
1 Ilustrasi penjajaran global dan lokal 3
2 Sequence alignment 3
3 Matriks Am,n 4
4 Pengisian cell matriks Am,n 5
5 Jalur global sequence alignment yang mungkin 5
6 Jalur global sequence alignment yang optimal 6
7 Hasil global sequence alignment 6
8 Matriks identitas pasangan sekuen 7
9 Skor kemiripan penjajaran sekuen 8
10 Pemilihan sekuen star 8
11 Realignment 9
12 Arsitektur flynn's taxonomy (Qiunn 2003) 10
13 Tahapan foster's methodology (Quinn 2003) 11
14 Ilustrasi distributed memory dengan MPI (Quinn 2003) 12 15 Ilustrasi algoritma penjadwalan round robin (RR) 12 16 Ilustrasi distribusi proses jika jumlah pasangan sekuen lebih banyak
dibanding jumlah komputer atau Kn > Pn 14
17 Ilustrasi pembagian data sekuen dengan MPI 14
18 Tahapan fungsi pembacaan data sekuen DNA 16
19 Format file .TXT 16
20 Tahapan fungsi inisialisasi matriks 17
21 Tahapan fungsi perhitungan skor kemiripan sekuen 18
22 Tahapan fungsi pemilihan sekuen star 19
23 Tahapan fungsi penjajaran sekuen star dengan sekuen lainnya 20 24 Pengembangan aplikasi sekuensial menjadi aplikasi paralel 22
25 Grafik waktu eksekusi penjajaran sekuen 23
26 Speedup komputasi paralel 23
27 Grafik nilai efisiensi penggunaan prosesor terhadap jumlah data
sekuen 24
DAFTAR LAMPIRAN
1 Hasil pengujian aplikasi MSA sekuensial 28
1
PENDAHULUAN
Latar Belakang
Pada bioinformatika, terdapat masalah yang sangat mendasar dan penting yaitu masalah penjajaran sekuen (sequence alignment). Penjajaran sekuen dapat didefinisikan sebagai teknik untuk menemukan bagian mana dari sekuen yang sama dan bagian mana yang berbeda (Junior 2003). Penjajaran sekuen dapat dilakukan terhadap dua sekuen (pairwise sequence) atau banyak sekuen (multiple sequence). Multiple sequence alignment (MSA) adalah teknik menjajarkan beberapa sekuen biologi (Liu et al. 2009). MSA digunakan dalam analisis filogenetik untuk menilai asal-usul spesies. MSA digunakan juga untuk pencarian single nucleotide polymorphism (SNP) untuk pemuliaan tanaman genetika berbasis molekuler (Sujiwo dan Kusuma 2013).
Kompleksitas komputasi penjajaran sekuen yaitu O(LN), dengan L merupakan panjang tiap sekuen dan N adalah jumlah sekuen (Lloyd 2010). Semakin panjang dan banyak data sekuen yang akan dijajarkan maka semakin lama waktu komputasinya. Waktu optimal penjajaran sekuen meningkat secara eksponensial seiring peningkatan jumlah dan panjang sekuen (Edgar dan Batzoglou 2006).
Penelitian untuk mengoptimalkan komputasi penjajaran sekuen telah banyak dilakukan, di antaranya menggunakan algoritme dynamic programming dengan kompleksitas O(mn) oleh Zhou dan Chen (2013). Selanjutnya penelitian menggunakan algoritme heuristic seperti FASTA dan FASTP oleh Pearson et al. (1988), BLAST oleh Altschul et al. (1997), Pertsemlidis et al. (2001), Rangwala et al. (2005), dan Kanchanamala et al. (2012). Myers et al. (1988) dan Sandes et al. (2011) menggunakan algoritme linear space untuk mengoptimalkan kompleksitas penjajaran sekuens menjadi O(n).
Selain itu penelitian dengan menggunakan komputasi parael untutk menyelesaikan permasalahan MSA juga telah dilakukan, antara lain menggunakan MPI (Sunarto et al. 2013) dan GPU-CUDA (Liu et al. 2009; Sujiwo dan Kusuma 2013; Ling et al. 2009; Khajeh-Saeed et al. 2010; Siriwardena et al. 2010; Gudyś et al. 2011; Li J et al. 2012; dan Korpar 2013).
2
Perumusan Masalah
Rumusan masalah penelitian ini adalah bagaimana mengoptimalkan proses komputasi multiple sequence alignment menggunakan komputasi paralel dengan message passing inteface.
Tujuan Penelitian
Tujuan penelitian ini adalah untuk mendapatkan waktu proses komputasi multiple sequence alignment yang optimal dengan menggunakan komputasi paralel.
Manfaat Penelitian
Manfaat penelitian ini adalah untuk mempercepat waktu komputasi penjajaran sekuen (sequence alignment) DNA, RNA atau protein percepatan proses penjajaran sekuen maka akan mempercepat dalam menemukan keanekaragaman sifat pada makhluk hidup dan mengetahui sifat-sifat unggul pada makhluk hidup.
Ruang Lingkup Penelitian
Ruang lingkup pada penelitian ini adalah sebagai berikut :
1. Penerapannya pada cluster tertutup dengan pengaturan IP address static khusus untuk jaringan cluster komputer lab. NCC.
2. Data sekuen maksimal panjang 850 base pair (bp) dan jumlah 64 sekuen dengan format FASTA didapatkan dari NCBI.
3
2
TINJAUAN PUSTAKA
Sequence alignment
Penjajaran sekuens (sequence alignment) adalah salah satu operasi dasar dalam biologi komputasi.. Salah satu algoritme yang efisien untuk penjajaran sekuens adalah dengan menggunakan algoritme dynamic programming (Charter et al. 2000). Ada dua metode yang digunakan untuk penjajaran dua sekuens dengan pendekatan dynamic programming yaitu penjajaran global yang menggunakan algoritme needleman-wunsch dan penjajaran lokal yang menggunakan algoritme smith waterman (Huang et al. 2002) (Junior 2003) (Charter et al. 2000) (Khajeh-Saeed et al. 2010) (Siriwardena et al. 2010). Ilustrasi penjajaran global dan lokal ditunjukan pada Gambar 1. Penjajaran sekuens digunakan untuk menemukan kesamaan biologis antara manusia, tikus, sapi dan lainnya. Penjajaran digunakan untuk melihat kesamaan elemen pada sekuens yang ditunjukan dengan garis vertikal antar nukleotida pada dua sekuens seperti yang ditunjukan Gambar 2.
Gambar 1 Ilustrasi penjajaran global dan lokal
Gambar 2 Sequence alignment
Dynamic Programming Algorithm
4
paling maksimal dan setelah semua matriks terisi dilakukan pelacakan kembali (trace back) yang dimulai dari skor tertinggi pada bagian ujung kanan bawah matriks.
Needleman-Wunsch Algorithm
Algoritma needleman-wunsch ini pertama kali diusulkan oleh Saul B. Needleman dan Christian D. Wunsch pada tahun 1970. Algoritma needleman-wunsch melakukan penjajaran sekuens global, yaitu melihat tingkat kemiripan yang paling tinggi dari penjajaran sekuen global yang ditunjukkan oleh nilai skor penjajarannya (Global … 2013)(Siriwardena et al. 2010).
Tahapan algoritma needleman-wunsch adalah sebagai berikut : 1. Inisialisasi matriks
Pada tahap ini pairwise sequence dibuat menjadi matriks M x N, di mana M adalah sekuens pertama dan N adalah sekuens kedua, sebagai contoh, ada sepasang sekuens, di mana ATGCGC adalah sequence 1 dengan panjang M dan AGACT adalah sequence 2 dengan panjang N, maka kedua sekuens
Gambar 3 Matriks Am,n Variabel yang digunakan :
m menjelaskan baris dan n menjelaskan kolom.
A nilai matriks untuk setiap cell dinyatakan sebagai (Am,n)
S adalah skor setiap cell dinyatakan sebagai (Sm,n)
W sebagai gap alignment
2. Pengisian nilai tiap cell matriks (matrix filling)
Pengisian cell matriks untuk matriks Am,n, didapatkan dengan menggunakan persamaan (1) di bawah ini :
(1)
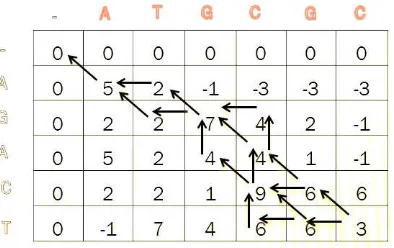
5 yang berbeda jauh antara nilai matching, mismatch dan gap. Tujuan pemberian nilai ini adalah untuk mendapatkan nilai cell yang jauh berbeda dengan cell lainnya. Perbedaan nilai pada setiap cell akan memudahkan dalam menentukan hasil penjajaran sekuen. Semakin besar perbedaan skor nilai matching dibanding mismatch maka akan semakin baik untuk mengetahui hasil penjajaran. Dalam contoh kasus ini, untuk elemen sekuen yang sesuai (matching) diberikan nilai +5 , tidak sesuai (mismatch) diberikan nilai -3 dan gap dikenakan penalti -4. Pengisian cell kolom 1 dan baris 1 matriks A dengan menggunakan persamaan (1) maka didapatkan hasil sebagai berikut:
A1,1 = Maksimum[A1-1,1-1+S1,1,A1,1+W,A1-1,1+W+0] = Maksimun[A0,0+S1,1,A1,1+W,A0,1+W+0] = Maksimum[0+(5),0+(-3),0+(-3)+0] = Maksimum[5,-3,-3,0]
A1,1 = 5
Nilai untuk semua cell matriks Am,n apabila semua telah dihitung dapat dilihat pada Gambar 4.
A T G C G C 0 0 0 0 0 0 0 A 0 5 2 -1 -3 -3 -3 G 0 2 2 7 4 2 -1 A 0 5 2 4 4 1 -1 C 0 2 2 1 9 6 6 T 0 -1 7 4 6 6 3 Gambar 4 Pengisian cell matriks Am,n 3. Pelacakan kembali (trace back)
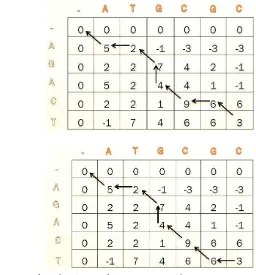
Pelacakan kembali (trace back) merupakan tahap terakhir dari algoritme Needleman-Wunsch, pada tahap ini mencari semua kemungkinan jalur dalam penjajaran sekuen global. Pelacakan jalur dimulai dari cell baris dan kolom terakhir, kemudian memilih cell yang tertinggi nilainya secara vertikal, horizontal atau diagonal. Pada contoh kasus ini jalur yang memungkinkan untuk hasil penjajaran dapat dilihat pada Gambar 5.
6
Setelah mendapatkan beberapa jalur global maka dipilih jalur global yang paling maksimal nilainya. Penentuan jalur yang memiliki nilai maksimal adalah dengan menjumlahkan setiap langkah berdasarkan skor matching, mismatch dan gap yang telah ditentukan sebelumnya. Pada tahap untuk kasus ini didapatkan dua jalur global yang memiliki nilai maksimal seperti ditunjukan pada Gambar 6.
Gambar 6 Jalur global sequence alignment yang optimal
Penentuan sekuen hasil penjajaran memiliki aturan penulisan sebagai berikut :
i. apabila arah jalurnya diagonal maka karakter sekuen 1 dan sekuen 2 tetap, ii. apabila arah jalurnya horizontal maka karakter sekuen 1 tetap dan sekuen 2
ditulis gap (-),
iii. apabila arah jalurnya vertical maka sekuen 1 ditulis gap (-) dan karakter sekuen 2 tetap.
Sesuai aturan di atas didapatkan hasil global sequence alignment pada kasus ini dapat dilihat pada Gambar 7.
7
Multiple Sequence Alignment (MSA)
MSA merupakan masalah yang sangat mendasar dalam biologi molekuler (Gupta et al. 1995). Metode penjajaran banyak sekuens (multiple sequence alignment) merupakan pengembangan dari penjajaran dua sekuens. Adapun pengertian penjajaran dua sekuen (pairwise sequence alignment) adalah pencarian kemiripan yang terbaik dalam penjajaran global dan lokal pada dua sekuens protein (amino acid) atau DNA (nucleicacid) (Oladele et al. 2009).
Star Alignment
Metode star alignment untuk penjajaran banyak sekuens (multiple sekuens alignment) dapat dilakukan dalam tiga tahap (Sujiwo dan Kusuma 2013).
1. Perhitungan pairwise similarity score untuk penjajaran sekuens menggunakan algoritme Needleman-Wunsch.
2. Pemilihan sekuens yang akan menjadi Star, dilanjutkan dengan memutakhirkan sekuens Star dengan menjajarkan ulang terhadap sekuen lainnya.
3. Realignment adalah penjajaran ulang untuk semua sekuens terhadap sekuen Star.
Ilustrasi penjajaran multi sekuen adalah sebagai berikut : Misalnya data sekuen DNA yang digunakan adalah :
Seq 1 = ATGG Seq 2 = ATGC
Seq 3 = ATCC Seq 4 = AGCG
Tahap pertama adalah dengan melakukan perhitungan nilai kemiripan semua pasangan sekuen DNA. Algoritme perhitungan ini memiliki kompleksitas O( ), di mana n adalah banyak sekuen. Pada tahap ini dimulai dengan menghitung banyak pasangan sekuen menggunakan persamaan (2) di bawah ini:
(2) Dengan n adalah banyak sekuen.
Hasil penentuan pasangan sekuen ditampilkan dalam matriks identitas, kemudian setiap pasangan sekuen disimbolkan dengan Kn. Menggunakan data di atas maka matriks identitasnya sebagai berikut (Gambar 8):
Seq 1 Seq 2 Seq 3 Seq 4
Seq 1 K1 K2 K3
Seq 2 K1 K4 K5
Seq 3 K2 K4 K6
Seq 4 K3 K5 K6
8
Hasil penentuan pasangan sekuen DNA adalah : K1 = Seq 1 (ATGG) dan Seq 2 (ATGC)
K2 = Seq 1 (ATGG) dan Seq 3 (ATCC) K3 = Seq 1 (ATGG) dan Seq 4 (AGCG)
K4 = Seq 2 (ATGC) dan Seq 3 (ATCC) K5 = Seq 2 (ATGC) dan Seq 4 (AGCG) K6 = Seq 3 (ATCC) dan Seq 4 (AGCG) Setiap pasangan sekuen di atas dijajarkan menggunakan algoritme Needleman Wunsch. Algoritme ini menggunakan pendekatan Dynamic Programing untuk mencari kemiripan global dari dua sekuen.
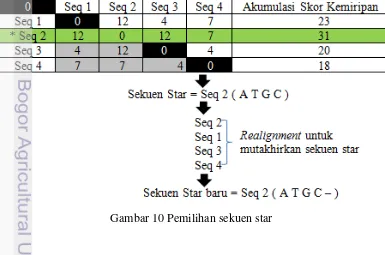
Hasil perhitungan skor kemiripan untuk semua pasangan sekuen diperlihatkan pada Gambar 9.
0 Seq 1 Seq 2 Seq 3 Seq 4
Seq 1 0 12 4 7
Seq 2 12 0 12 7
Seq 3 4 12 0 4
Seq 4 7 7 4 0
Gambar 9 Skor kemiripan penjajaran sekuen
Tahap kedua dari algoritme star adalah pemilihan sekuen star. Penentuan sekuen Star dengan memilih akumulasi skor kemiripan penjajaran sekuen yang tertinggi dibanding dengan sekuen lainnya. Sekuen star akan dimutakhirkan dengan menjajarkannya terhadap sekuen lainnya. Tujuan pemutakhiran sekuen Star adalah untuk mendapatkan hasil penjajaran yang optimal. Sekuen star akan dijajarkan dengan sekuen lainnya dan memungkinkan adanya perubahan karakter dengan adanya gap pada sekuen star. Pada contoh kasus ini sekuen 2 sebagai sekuen star akan dijajarkan dengan sekuen 1, kemudian sekuen Star baru dari hasil penjajaran sebelumnya akan dijajarkan dengan sekuen 3, proses ini dilakukan untuk semua sekuen. Kompleksitas tahap ini adalah O(n), dengan n adalah jumlah sekuen. Ilustrasi pemilihan sekuen star diperlihatkan pada Gambar 10.
9 Tahap ketiga dari algoritme star adalah realignment. Pada tahapan ini dilakukan penjajaran ulang semua sekuen terhadap sekuen star. Sekuen star yang akan dijajarkan merupakan sekuen star yang telah dimuktakhirkan. Pada contoh kasus ini adalah sekuen 2 sebagai sekuen Star yang telah dimuktakhirkan akan dijajarkan dengan sekuen 1, sekuen 3 dan sekuen 4. Kompleksitas pada tahapan ini adalah O(n), dengan n adalah jumlah sekuens. Ilustrasi realignment diperlihatkan pada Gambar 11.
Gambar 11 Realignment
Komputasi Paralel
Komputasi paralel adalah penggunaan beberapa komputer yang saling terhubung jaringan komputer untuk mengurangi waktu yang dibutuhkan dalam memecahkan masalah komputasi tunggal. Skema klasifikasi komputasi paralel yang paling terkenal adalah flynn's taxonomy lihat Gambar 12. Ada empat kategori untuk mengklasifikasikan komputer menurut Hence Flynn's (Quinn 2003), yaitu :
1. SISD (single instruction single data)
Komputer dengan arsitektur single instruction single data hanya memiliki satu intruksi untuk satu data. Sebuah CPU hanya menjalankan satu intruksi saja dan tidak dapat diterapkan untuk komputasi paralel.
2. SIMD (single instruction multiple data)
Single instruction multiple data memungkinkan komputer untuk mengolah banyak data dengan satu intruksi. Processor array dan processor pipeline vector merupakan kategori SIMD. Prosesor larik (processor array) memungkinkan satu prosesor memroses satu intruksi, bahkan beberapa prosesor mampu melakukan proses secara bersamaan dengan elemen data yang berbeda. Sebuah pipelined vector processor bergantung pada kecepatan clock untuk mengeksekusi operasi yang sama pada elemen data.
3. MISD (multiple instruction single data)
Multiple instruction single data memungkinkan komputer untuk mengeksekusi banyak intruksi untuk satu aliran data. Komputer ini belum pernah diciptakan hanya sekedar prototype.
4. MIMD (multiple instruction multiple data)
10
Gambar 12 Arsitektur flynn's taxonomy (Qiunn 2003)
Pemrograman Paralel
Pemrograman paralel adalah bahasa pemrograman yang membolehkan pembuat program untuk membagi komputasi dengan tugas berbeda yang dijalankan secara bersama-sama oleh prosesor yang berbeda. Peengembangan perangkat lunak untuk komputasi paralel ada 4 cara yaitu : (Quinn 2003).
1. Mengembangkan compiler yang sudah ada dengan mengubah program sekuensial menjadi program paralel.
2. Menambahkan fungsi baru pada bahasa pemrograman yang sudah ada yang memungkinkan pengguna untuk mengekspresikan paralelisme.
3. Menambahkan layer paralel di bahasa pemrograman sekuensial. 4. Membuat bahasa dan compiler paralel yang baru.
11
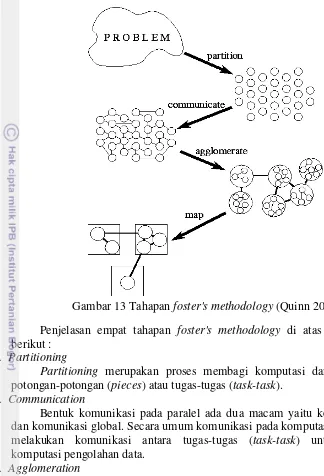
Gambar 13 Tahapan foster's methodology (Quinn 2003)
Penjelasan empat tahapan foster's methodology di atas adalah sebagai berikut :
1. Partitioning
Partitioning merupakan proses membagi komputasi dan data menjadi potongan-potongan (pieces) atau tugas-tugas (task-task).
2. Communication
Bentuk komunikasi pada paralel ada dua macam yaitu komunikasi lokal dan komunikasi global. Secara umum komunikasi pada komputasi paralel adalah melakukan komunikasi antara tugas-tugas (task-task) untuk melakukan komputasi pengolahan data.
3. Agglomeration
Agglomeration merupakan proses mengabungkan task-task. Penggabungan task dibutuhkan untuk mengatur pemberian beban kerja untuk prosesor. Ketika data yang diproses besar maka dibutuhkan pengelompokan task untuk mengolah data yang nantinya akan dibagi ke semua prosesor.
4. Mapping
12
Message Passing Interface (MPI)
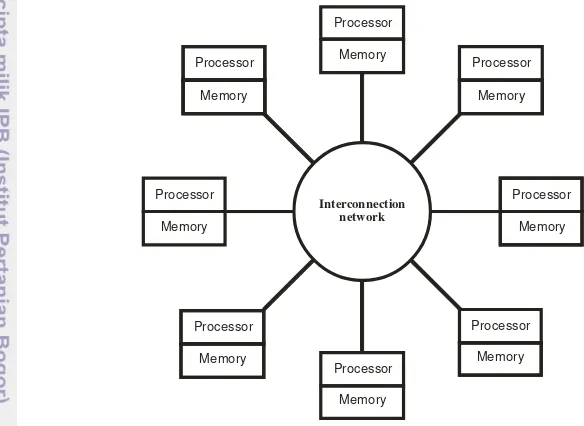
MPI adalah bahasa pemrograman paralel dan merupakan bahasa tingkat tinggi, yang berfungsi sebagai protokol komunikasi antara komputer pada program paralel (Kurniawan 2010). MPI merupakan spesifikasi standar untuk message-passing library (Quinn 2003). MPI dapat mempermudah dalam pengelolaan paralelisasi, dan MPI library tersedia gratis untuk berbagai bahasa pemrograman. MPI memungkinkan koordinasi beberapa proses pada lingkungan distributed memory (Pineda et al. 1998). Ilustrasi MPI yang digunakan untuk distributed memory dalam suatu jaringan komputer dapat dilihat pada Gambar 14.
Gambar 14 Ilustrasi distributed memory dengan MPI (Quinn 2003)
Penjadwalan Round Robin
Round robin (RR) merupakan algoritma penjadwalan yang sederhana dengan pembagian proses yang sama, dirancang khusus untuk pembagian waktu (time-sharing) (Behera et al. 2012). Setiap proses pada penjadwalan round robin diberikan porsi yang sama dan proses dikerjakan sesuai urutan membentuk lingkaran. Ilustrasi algoritma penjadwalan round robin (RR) dapat dilihat pada Gambar 15.
13
3
METODE
Tahapan Penelitian
Secara umum penelitian ini dimulai dengan mengumpulkan data penelitian, kemudian mendesain aplikasi sekuensial untuk multiple sequence alignment menggunakan algoritme star alignment. Kemudian penerapan komputasi paralel (cluster Komputer) dan analisa hasil komputasi pada cluster Komputer. Penjelasan setiap tahapan penelitian adalah sebagai berikut :
1. Persiapan data penelitian
Data penelitian diperoleh dari national center for biotechnology information (NCBI). NCBI merupakan tempat penyimpanan informasi genetic. Data genetik bisa didapatkan dengan mengunjungi situs NCBI dengan alamat url adalah sebagai berikut www.ncbi.nlm.nih.gov/genbank/. 2. Desain aplikasi sekuensial MSA dengan algoritma star
Pada tahap ini peneliti membuat aplikasi sekuensial MSA menggunakan bahasa pemograman C.
3. Desain aplikasi paralel pada cluster komputer dengan MPI
Pada tahapan ini dikembangkan aplikasi sekuensial menjadi aplikasi paralel. Paralisasi hanya dilakukan pada proses pairwise similarity score, karena tahap ini merupakan tahapan yang memiliki kompleksitas tertinggi dibandingkan tahapan lainnya dalam algoritma Star. Kompleksitas pairwise similarity score adalah O(n2), di mana n adalah jumlah sekuen. Pemrograman paralel didesain mengunkan foster's methodology.
14
Penggabungan Proses (T1) Penggabungan Proses (T2) (Tn)
Gambar 16 Ilustrasi distribusi proses jika jumlah pasangan sekuen lebih banyak dibanding jumlah komputer atau Kn > Pn
Paralelisasi MSA dilakukan dengan MPI menggunakan komunikasi point to point dengan operasi blocking send and receive. Skema paralelisasi untuk MSA adalah dengan menjadikan satu komputer sebagai pembagi data dan komputer lainnya yang akan memproses data. Komputer pembagi yang dinamakan rank 0 akan mengirimkan data pasangan sekuen menggunakan MPI_Send(). Komputer pemroses data yang disimbolkan dengan rank 1..n akan menerima data pasangan sekuen dengan perintah MPI_Recv(). Komputer pemroses data akan mengirimkan data hasil perhitungan skor kemiripan pasangan sekuen ke rank 0 menggunakan MPI_Send(). Komputer rank 0 akan menerima data hasil perhitungan skor kemiripan dengan perintah MPI_Recv(). Ilustrasi pembagian data sekuen DNA dengan MPI dapat dilihat pada Gambar 17.
Rank 0
MPI_Recv(Skor Kemiripan K1) / MPI_Send(Skor Kemiripan K1)
MPI_Recv(Skor Kemiripan K2) / MPI_Send(Skor Kemiripan K2)
MPI_Recv(Skor Kemiripan Kn) / MPI_Send(Skor Kemiripan Kn)
Gambar 17 Ilustrasi pembagian data sekuen dengan MPI 4. Analisa hasil.
Pada tahap ini membandingkan hasil komputasi multiple sequence alignment pada satu komputer dibandingkan dengan beberapa komputer dengan melihat waktu eksekusi, speedup dan efficiency.
Untuk mendapatkan speedup komputasi pada pemrograman paralel adalah dengan membandingakan waktu eksekusi secara sekuensial dengan waktu eksekusi paralel (Quinn 2003). Adapun efficiency komputasi pemrograman paralel didapatkan dengan membandingkan waktu eksekusi secara sekuensial dengan waktu eksekusi paralel dikalikan penggunaan prosesor (Quinn 2003). Untuk mempermudah memahami speedup dan efficiency dapat dilihat pada rumus (2) dan (3).
Pembagi data
15
Keterangan : S = Speedup E = Efficiency
Ts = Waktu eksekusi sekuensial Tp = Waktu eksekusi paralel P = Banyak Prosesor.
Bahan
Pada penelitian ini menggunakan data FASTA Glycine-max-chromosome-9-BBI (Kedelai). Sumber data adalah diperoleh dari NCBI (National Center for Biotechnology Information). Data dibagi menjadi 18 bagian berdasarkan dari sekuen referensi Glycine-max-chromosome-9-BBI,
Alat
16
4
HASIL DAN PEMBAHASAN
Persiapan Data
Data FASTA Glycine-max-chromosome-9-BBI dibagi menjadi 3, 4, 8, 12, 14, 16, 20, 24, 28, 32, 36, 40, 44, 48, 52, 56, 60 dan 64 berdasarkan sekuen referensi Glycine-max-chromosome-9-BBI.
DesainAplikasi Sekuensial
Desain aplikasi sekuensial untuk MSA dengan algoritma star menggunakan bahasa C yang terdiri dari beberapa tahapan yaitu :
a. Pembuatan fungsi pembacaan data sekuen b. Pembuatan fungsi inisialisasi matriks
c. Pembuatan fungsi perhitungan skor kemiripan sekuen d. Pembuatan fungsi pemilihan sekuen star
e. Pembuatan fungsi penjajaran sekuen star dengan sekuen lainnya Penjelasan tiap tahapan adalah sebagai berikut :
1. Pembuatan fungsi pembacaan data sekuen DNA dengan format pembacaan file .TXT yang berisikan karakter ATGC. Tahapan perancangan dapat dilihat pada Gambar 18.
Pembacaan data inputan
Tampilkan data dan siap untuk diproses Deklarasi variable untuk menampung inputan keyboard
Gambar 18 Tahapan fungsi pembacaan data sekuen DNA Adapun format file.TXT dapat dilihat pada Gambar 19.
Gambar 19 Format file .TXT Keterangan :
17 Pseudocode fungsi pembacaan data sekuen
{Deklarasi Variable} Jumlah_Sekuen
Panjang_Sekuen Temp_Data_Sekuen
{Pembacaan inputan Keyboard}
Perulangan Mulai dari = 0 Jumlah_Sekuen
Mulai
Temp_Data_Sekuen Alokasi Memory = (Panjang_Sekuen +
1)
Baca Inputan Keyboard Temp_Data_Sekuen
Berhenti
{Pencetakan data}
Perulangan Mulai dari = 0 Jumlah_Sekuen
Mulai
Cetak Temp_Data_Sekuen
Berhenti
2. Pembuatan fungsi inisialisasi matriks.
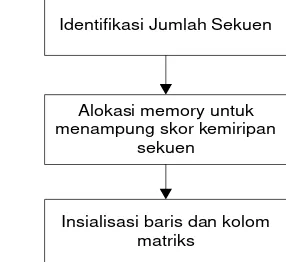
Fungsi inisialisasi matriks merupakan tahapan awal perhitungan skor kemiripan sekuen. Pada tahapan ini akan mengalokasikan memory sebanyak panjang sekuen. Adapun tahapan fungsi insialisasi matriks dapat dilihat pada Gambar 20.
Identifikasi Jumlah Sekuen
Alokasi memory untuk menampung skor kemiripan
sekuen
Insialisasi baris dan kolom matriks
Gambar 20 Tahapan fungsi inisialisasi matriks Pseudocode fungsi inisialisasi matriks
{Deklarasi Variabel} Matriks_Skor
{Pembuatan matriks untuk menampung skor}
Perulangan mulai dari = 0 Jumlah_Sekuen
Mulai
Matriks_Skor Alokasi Memory = (Jumlah_Sekuen + 1)
Berhenti
{Inisialisasi baris dan kolom matriks skor}
Perulangan mulai dari index = 0 Jumlah_Sekuen
Mulai
Matriks_Skor [0][index] = 0
18
3. Pembuatan fungsi perhitungan skor kemiripan sekuen.
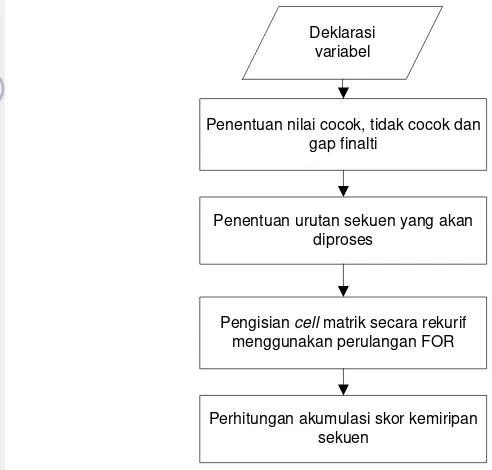
Fungsi perhitungan skor kemiripan 2 pasang sekuen akan melakukan perhitungan untuk tiap cell matriks. Setiap cell melakukan perhitungan nilai integer dengan alokasi memory untuk tiap cell adalah 2 byte. Adapun tahapan dari fungsi perhitungan skor kemiripan 2 pasang sekuen dapat dilihat pada Gambar 21.
Pengisian cell matrik secara rekurif
menggunakan perulangan FOR Deklarasi
variabel
Perhitungan akumulasi skor kemiripan sekuen
Penentuan nilai cocok, tidak cocok dan gap finalti
Penentuan urutan sekuen yang akan diproses
Gambar 21 Tahapan fungsi perhitungan skor kemiripan sekuen Pseudocode fungsi perhitungan skor kemiripan 2 pasangan sekuen {Deklarasi variabel}
Vektor_Akumulasi_Skor, Kolom, Baris Urutan_Sekuen1, Urutan_Sekuen2
{Penentuan nilai cocok, tidak cocok dan gap finalti} Match = 5
Mismatch = -3 Gap = -4
{Penentuan urutan sekuen yang akan diproses}
Perulangan mulai dari Kolom = 0 Jumlah_Sekuen
Mulai
Perulangan mulai dari Baris = Baris+1 Jumlah_Sekuen
Mulai
{Inisialisasi urutan pembacaan sekuen} Urutan_Sekuen1 = Kolom-1;
Urutan_Sekuen2 = Baris-1;
{Perhitungan skor kemiripan secara rekursif}
Perulangan mulai dari Baris = 1 Total_Baris
Mulai
Perulangan mulai dari Kolom =1 Total_Kolom
Jika (Urutan_Sekuen1[Baris-1] = Urutan_Sekuen2[Kolom-1] Maka
Skor_Diagonal = Matriks_Skor[Baris-1][Kolom-1] + Match Selainnya
Skor_Diagonal = Matriks_Skor[Baris-1][Kolom-1] +
19 Skor_Vertikal = Matriks_Skor[Baris-1][Kolom] + Gap Skor_Horizontal = Matriks_Skor[Baris][Kolom-1] + Gap Jika Skor_Vertikal <= Skor_Diagonal >= Skor_Horizontal Maka
Matriks_Skor = Skor_Diagonal
Selainnya Jika Skor_Vertikal >= Skor_Horizontal Maka
{Perhitungan akumulasi skor kemiripan sekuen}
Perulangan mulai dari Baris = 1 Jumlah_Sekuen
Mulai
Vektor_Akumulasi_Skor[Baris] = 0;
Perulangan mulai dari Kolom = 1 Jumlah_Sekuen
Vektor_Akumulasi_Skor[Baris] = Matriks_Skor [Baris][Kolom] +
Vektor_Akumulasi_Skor[Baris] Berhenti
4. Pembuatan fungsi pemilihan sekuen star.
Fungsi pemilihan sekuen star terdiri dari 2 tahapan utama yaitu penyeleksian sekuen star yang memiliki akumulasi skor kemiripan tertinggi terhadap sekuen lainnya dan pemutakhiran sekuen star. Adapun tahapan pemilihan sekuen star dapat dilihat pada Gambar 22.
Pencarian sekuen yang memiliki skor kemiripan tertinggi sebagai star
Pembuatan jalur untuk traceback
Penentuan jalur optimal dan penulisan sekuen star terbaru
Gambar 22 Tahapan fungsi pemilihan sekuen star Pseudocode fungsi pemilihan sekuen star
{Deklarasi Variabel} Skor_Maksimal
Urutan_Sekuen_Tertinggi Matriks_Traceback
{Insialisasi awal Skor Maksimal} Skor_Maksimal = 0
Perulangan mulai dari index = 0 Jumlah_Sekuen
Mulai
Jika Vektor_Akumulasi_Skor[index] = Skor_Maksimal Maka
20
Berhenti
{Pemutakhiran sekuen star}
Perulangan mulai dari index = 0 Jumlah_Sekuen
Mulai
Jika Ururan_Sekuen[index] != Urutan_Sekuen_Tertinggi Maka
Panggil (Fungsi perhitungan skor kemiripan pasangan sekuen)
{Pengisian jalur pada Matriks_Traceback}
Jika Skor_Vertikal <= Skor_Diagonal >= Skor_Horizontal Maka
Matriks_ Traceback = Diagonal ( \\ )
Selainnya Jika Skor_Vertikal >= Skor_Horizontal Maka
Matriks_ Traceback = Vertikal ( | ) Selainnya
Matriks_Skor = Horizontal ( - ) Berhenti
Berhenti
{Penentuan jalur optimal dan penulisan karakter sekuen star} Apabila Kondisi Matriks_Traceback != NULL
Maka
Jika Matriks_Traceback = Diagonal ( \\ ) Maka
Karakter_Sekuen_Star_Baru = Karakter_Sekuen_Star_Lama Jika Matriks_ Traceback = Vertikal ( | )
Maka
Karakter_Sekuen_Star_Baru = Karakter_Sekuen_Star_Lama Jika
Matriks_Skor = Horizontal ( - ) Maka
Karakter_Sekuen_Star_Baru = Gap ( _ ) Berhenti
5. Pembuatan fungsi penjajaran sekuen star dengan sekuen lainnya.
Fungsi penjajaran sekuen terhadap sekuen star merupkan tahapan teakhir dari desain aplikasi MSA sekuensial. Tahap ini memiliki langkah yang sama dengan proses pemutakhiran sekuen star dengan penambahan penulisan karakter sekuen hasil penjajaran. Adapun tahapan fungsi penjajaran sekuen star dengan sekuen lainnya dapat dilihat pada Gambar 23.
Penjajaran sekuen star dengan sekuen lainnya
Hasil penjajaran Pembacaan urutan sekuen star
Penentuan urutan sekuen yang akan dijajarkan dengan sekuen star
21 Pseudocode fungsi penjajaran sekuen star dengan sekuen lainnya
Perulangan mulai dari index = 0 Jumlh_Sekuen
Mulai
Panggil (Fungsi perhitungan skor kemiripan pasangan sekuen) Jika Matriks_Traceback = Diagonal ( \\ )
Maka
Karakter_Sekuen_Lainnya = Karakter_Sekuen[index] Jika Matriks_ Traceback = Vertikal ( | )
Maka
Karakter_Sekuen_Lainnya = Karakter_Sekuen[index] Jika
Matriks_Skor = Horizontal ( - ) Maka
Karakter_Sekuen_Lainnya = Gap ( _ ) Berhenti
{Cetak hasil penjajaran sekuen}
Perulanga mulai dari index = 0 Jumlah_Sekuen
Mulai
Jika Sekuen[index] = Sekuen_Star Maka
Cetak Sekuen_Star Cetak Sekuen [index] Berhenti
Desain Aplikasi Paralel
22
Penjajaran sekuen star terbaru dengan sekuen lainnya Kirim data pasangan sekuen dengan
perintah MPI_Send() Ya
Perhitungan skor kemiripan pasangan sekuen
Kirim data hasil perhitungan skor kemiripan dengan
perintah MPI_Send() Menerima hasil perhitungan skor
kemiripan sekun dengan perintah MPI_Recv()
MPI_Finalize MPI_Finalize
Aplikasi Paralel
Gambar 24 Pengembangan aplikasi sekuensial menjadi aplikasi paralel
Analisis Hasil
a. Waktu eksekusi penjajaran sekuen
23 sebagai pembagi data dan 3 komputer sebagai pemroses data. Skenario ke tiga adalah 1 komputer sebagai pembagi data dan 4 komputer sebagai pemroses data. Hasil pengujian waktu eksekusi dapat dilihat pada Gambar 25.
Gambar 25 Grafik waktu eksekusi penjajaran sekuen
Hasil eksekusi penjajaran sekuen di atas menunjukan bahwa waktu eksekusi paralel lebih baik dibandingkan waktu eksekusi sekuensial. Penambahan jumlah komputer akan meningkatkan kecepatan waktu eksekusi paralel. Gambar 25 memperlihatkan perbedaan waktu eksekusi perhitungan skor kemiripan sekuen Glycine-max-chromosome-9-BBI dengan panjang 811-850 bp. Jumlah sekuen 3, 4, 8 dan 12 waktu eksekusinya tidak berbeda jauh antara sekuensial dan paralel. Akan tetapi jumlah sekuen 16, 20, 24 sampai dengan 64 terlihat perbedaan waktu eksekusi yang sangat tinggi. Gambar 25 memperlihatkan waktu eksekusi sekuensial semakin lama seiring dengan peningkatan jumlah sekuen, dan waktu eksekusi paralel semakin cepat untuk jumlah sekuen yang banyak.
b. Analisa Speedup
Speedup merupakan percepatan komputasi secara paralel dibandingkan dengan komputasi secara sekuensial. Pada penelitian ini membandingkan komputasi penjajaran sekuen secara paralel menggunakan 2, 3 dan 4 PC. Hasil komputasi paralel dengan 2, 3 dan 4 PC (Personal Computer) membuktikan bahwa penambahan jumlah komputer (PC) mempengaruhi waktu eksekusi paralel. Speedup komputasi paralel meningkat seiring dengan penambahan jumlah komputer. Grafik speedup komputasi paralel untuk 2, 3 dan 4 PC diperlihatkan pada Gambar 26.
24
Gambar 26 memperlihatkan penigkatan speedup komputasi paralel pada jumlah data antara 16 sampai 64 sekuen.dengan panjang 811-850 bp. Perbandingan kecepatan komputasi sekuensial dibanding komputasi paralel dengan 3 PC mencapai rasio 1,89. Speedup meningkat mecapai rasio 2,84 dengan 4 PC dan terjadi peningkatkan speedup mencapai rasio 3,84 pada 5 PC. Hasil ini menunjukan bahwa penambahan jumlah komputer akan meningkatkan speedup komputasi paralel dibanding sekuensial.
c. Analisa Efisiensi
Analisa efisiensi adalah melihat efisiensi penambahan prosesor atau PC terhadap ukuran data sekuen yang digunakan. Pengukuran efisiensi bertujuan untuk mengetahui pengaruh penambahan prosesor dalam memproses data sekuen. Peningkatan jumlah prosesor akan menurunkan nilai efisiensi, dan sebaliknya peningkatan ukuran data akan meningkatkan nilai efisiensi (Maria 2008). Nilai efisiensi akan konstan apabila penambahan data sekuen seiring dengan penambahan jumlah prosesor. Nilai efisiensi konstan menunjukan bahwa kinerja sistem paralel scalable dengan ukuran data. Scalable adalah kemampuan sistem paralel yang dapat mempertahankan kinerja seiring dengan penambahan jumlah prosesor berdasarkan metrik tertentu (Maria 2008) dalam memproses data dengan ukuran tertentu. Hasil perhitungan nilai efisiensi dapat dilihat pada Gambar 27.
Gambar 27 Grafik nilai efisiensi penggunaan prosesor terhadap jumlah data sekuen
25
5
SIMPULAN DAN SARAN
26
DAFTAR PUSTAKA
Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Oxford University Press. 25(17).
Behera HS, Swain KB, Parida AK, Sahu G. 2012. A New Proposed Round Robin with Highest Response Ratio Next (RRHRRN) Scheduling Algorithm for Soft Real Time Systems. International Journal of Engineering and Advanced Technology (IJEAT). 1(3). 200-206.
Charter K, Schaeer J, Szafron D. 2000. Sequence Alignment using FastLSA. The Department of Computing Science at the University of Alberta Canada.
Edgar RC, Batzoglou S. 2006. Multiple sequence alignment. Current opinion in structural biology, 16(3):368-373. doi : 10.1016/j.sbi.2006.04.004
Global alignment of two sequences-Needleman-Wunsch Algorithm 2013, [internet]. [diunduh 2013 Oktober 12]. Tersedia pada
http://amrita.vlab.co.in/?sub=3&brch=274 &sim= 1431& cnt=1.
Gudyś A, Deorowicz S. 2011. A parallel GPU-designed algorithm for the constrained pairwise sequence alignment problem. Di dalam: Man-Machine Interactions 2, AISC 103. Berlin (DE): Springer. hlm 361-368.
Gupta SK, Kececioglu JD dan Schäffer AA. 1995. Improving the Practical Space and Time Efficiency of the Shortest-Paths Approach to Sum-of-Pairs Multiple Sequence Alignment. Journal Of Computational Biology. 2(3): hlm. 459. Huang KF, Yang CB, Tseng KT. 2002. An Efficient Algorithm For Multiple
Sequence Alignment. Proc. of the 19th Workshop on Combinatorial Mathematics and Computation Theory.
Junior SAC. 2003. Sequence Alignment Algorithms. Department of Computer Science School of Physical Sciences & Engineering Kingís College London. Kanchanamala P, Rao AA, Rao PS, Sridhar GR. 2012. Influence of Blast, Fasta
and Wu-Blast algorithms on sequence alignments and 3-D structure prediction of DPP-IV. Journal of Bioinformatics & Research. 1(1).
Khajeh-Saeed A, Poole S, Perot JB. 2010. Acceleration of the Smith–Waterman algorithm using single and multiple graphics processors. Journal of Computational Physics. 229 (2010) 4247–4258. doi:10.1016/j.jcp.2010.02.009.
Korpar, Matija, Šikić, Mile. 2013. SW# - GPU enabled exact alignments on genome scale. Bioinformatic Oxford Journal.
Kurniawan A. 2010. Pemograman Paralel dengan MPI dan C. Penerbit Andi Yogyakarta 2010. ISBN : 9789792917109.
Li J, Ranka S, Sahni S. 2012. Pairwise Sequence Alignment for Very Long Sequences on GPUs. Department of Computer and Information Science and Engineering University of Florida Gainesville.
27 Ling C, Benkrid K, Hamada T. 2009. A Parameterisable and Scalable
Smith-Waterman Algorithm Implementation on CUDA-compatible GPUs. IEEE 7th Symposium on Application Specific Processors (SASP).
Lloyd GS. 2010. Parallel Multiple Sequence Alignment: An Overview. Maria A. Kartawidjaja. 2008. Analisis Kinerja Perkalian Matriks Paralel
Menggunakan Metrik Isoefisiensi. TESLA.10(2). 51-52.
Myers EW, Miller W. 1988. Optimal alignments in linear space. Oxford Univ Pres.
Oladele TO, Bamigbola OM, Bewaji CO. 2009. On efficiency of sequence alignment algorithms. AfricanScientist. 9(1).
Quinn MJ. 2003. Parallel Programing in C with MPI and OpenMP. McGraw-Hill Companies, Inc. ISBN 007-282256-2.
Pearson WR, Lipman DJ. 1988. Improved tools for biological sequence comparison. Proceeding Of the Nasional Academy of Sciences of the United States of America. 85(8)
Pertsemlidis A, Fondon JW. 2001. Tutorial Having a BLAST with bioinformatics (and avoiding BLASTphemy). BioMed Central Ltd.
Pineda AC, Smith B. 1998. MPI Tutorial. The University of New Mexico.
Rangwala H, Lantz E, Musselman Roy, Pinnow K, Smith B, Wallenfelt B. 2005. Massively Parallel BLAST for the Blue Gene/L. Citeseer.
Siriwardena TRP, Ranasinghe DN. 2010. Global Sequence Alignment using CUDA compatible multi-core GPU. IEEE Journal. Page 201 – 206. ISBN 978-1-4244-8549-9. doi : 10.1109/ICIAFS.2010.5715660.
Steeman Q. 2012. The Vehicle Routing Problem With Drop Yards: A Dynamic Programming Approach. Thesis Industrial Engineering & Management University of Twente.
Sujiwo MAP dan Kusuma W. 2013. Multiple Sequence Alignment with Star Method in Graphical Processing Unit using CUDA. International Seminar on Sciences (ISS).
Sunarto AA, WA Kusuma, H Sukoco. Paralelisme Of Star Alignment. 2013. IEEE International Conference on Instrumentation, Comunications, Information Technologi, and Biomedical Engineering.
Sandes EFO, de Melo ACMA. 2011. Smith-Waterman Alignment of Huge Sequences with GPU in Linear Space. IEEE International Parallel & Distributed Processing Symposium.
28
Lampiran 1 Hasil pengujian aplikasi MSA sekuensial
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-3.fasta waktu proses pengisian cell matrik = 0.032075 dtk
waktu penentuan dan update sekuen star = 0.017447 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.017880 dtk waktu total = 0.067402 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-4.fasta waktu proses pengisian cell matrik = 0.055050 dtk
waktu penentuan dan update sekuen star = 0.027630 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.028758 dtk waktu total = 0.111438 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-8.fasta waktu proses pengisian cell matrik = 0.232598 dtk
waktu penentuan dan update sekuen star = 0.064453 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.068011 dtk waktu total = 0.365062 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-12.fasta
waktu proses pengisian cell matrik = 0.538907 dtk waktu penentuan dan update sekuen star = 0.102073 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.110916 dtk waktu total = 0.751896 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-16.fasta
waktu proses pengisian cell matrik = 0.977081 dtk waktu penentuan dan update sekuen star = 0.142992 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.158084 dtk waktu total = 1.278158 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-20.fasta
waktu proses pengisian cell matrik = 1.548911 dtk waktu penentuan dan update sekuen star = 0.186291 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.206911 dtk waktu total = 1.942113 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-24.fasta
waktu proses pengisian cell matrik = 2.251927 dtk waktu penentuan dan update sekuen star = 0.230258 dtk
29 $ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-28.fasta
waktu proses pengisian cell matrik = 3.076540 dtk waktu penentuan dan update sekuen star = 0.275387 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.312242 dtk waktu total = 3.664169 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-32.fasta
waktu proses pengisian cell matrik = 4.042671 dtk waktu penentuan dan update sekuen star = 0.322441 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.370586 dtk waktu total = 4.735698 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-36.fasta
waktu proses pengisian cell matrik = 5.135911 dtk waktu penentuan dan update sekuen star = 0.370936 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.428911 dtk waktu total = 5.935757 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-40.fasta
waktu proses pengisian cell matrik = 6.360388 dtk waktu penentuan dan update sekuen star = 0.420146 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.486188 dtk waktu total = 7.266721 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-44.fasta
waktu proses pengisian cell matrik = 7.701725 dtk waktu penentuan dan update sekuen star = 0.469428 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.542717 dtk waktu total = 8.713870 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-48.fasta
waktu proses pengisian cell matrik = 9.178471 dtk waktu penentuan dan update sekuen star = 0.519858 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.601717 dtk waktu total = 10.300046 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-52.fasta
waktu proses pengisian cell matrik = 10.385301 dtk waktu penentuan dan update sekuen star = 0.558599 dtk
30
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-56.fasta
waktu proses pengisian cell matrik = 12.539642 dtk waktu penentuan dan update sekuen star = 0.623290 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.726846 dtk waktu total = 13.889778 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-60.fasta
waktu proses pengisian cell matrik = 14.429251 dtk waktu penentuan dan update sekuen star = 0.676661 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.792865 dtk waktu total = 15.898778 dtk
$ ./msa-sekuensial < data/Glycine/Glycine-max-chromosome-9-BBI-test-64.fasta
waktu proses pengisian cell matrik = 16.443364 dtk waktu penentuan dan update sekuen star = 0.730532 dtk
31 Lampiran 2 Hasil pengujian aplikasi MSA paralel dengan 3 PC
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-3.fasta
Banyak kombinasi pasangan sekuen = 3 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 0.024446 dtk waktu penentuan dan update sekuen star = 0.017941 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.017738 dtk waktu total = 0.060125 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-4.fasta
Banyak kombinasi pasangan sekuen = 6 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 0.039041 dtk waktu penentuan dan update sekuen star = 0.028291 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.028545 dtk waktu total = 0.095877 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-8.fasta
Banyak kombinasi pasangan sekuen = 28 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 0.128907 dtk waktu penentuan dan update sekuen star = 0.066157 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.067431 dtk waktu total = 0.262495 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-12.fasta
Banyak kombinasi pasangan sekuen = 66 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 0.291107 dtk waktu penentuan dan update sekuen star = 0.104779 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.109975 dtk waktu total = 0.505861 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-16.fasta
Banyak kombinasi pasangan sekuen = 120 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 0.520108 dtk waktu penentuan dan update sekuen star = 0.146759 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.156822 dtk waktu total = 0.823689 dtk
data/Glycine/Glycine-max-32
chromosome-9-BBI-test-20.fasta
Banyak kombinasi pasangan sekuen = 190 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 0.818783 dtk waktu penentuan dan update sekuen star = 0.190978 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.205441 dtk waktu total = 1.215202 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-24.fasta
Banyak kombinasi pasangan sekuen = 276 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 1.185027 dtk waktu penentuan dan update sekuen star = 0.236163 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.257535 dtk waktu total = 1.678726 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-28.fasta
Banyak kombinasi pasangan sekuen = 378 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 1.622433 dtk waktu penentuan dan update sekuen star = 0.282483 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.309931 dtk waktu total = 2.214847 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-32.fasta
Banyak kombinasi pasangan sekuen = 496 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 2.129304 dtk waktu penentuan dan update sekuen star = 0.330488 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.367907 dtk waktu total = 2.827700 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-36.fasta
Banyak kombinasi pasangan sekuen = 630 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 2.704004 dtk waktu penentuan dan update sekuen star = 0.380034 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.425764 dtk waktu total = 3.509803 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-40.fasta
33 waktu proses pengisian cell matrik = 3.348212 dtk
waktu penentuan dan update sekuen star = 0.430262 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.482796 dtk waktu total = 4.261271 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-44.fasta
Banyak kombinasi pasangan sekuen = 946 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 4.055652 dtk waktu penentuan dan update sekuen star = 0.480970 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.538784 dtk waktu total = 5.075406 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-48.fasta
Banyak kombinasi pasangan sekuen = 1128 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 4.838349 dtk waktu penentuan dan update sekuen star = 0.532884 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.597514 dtk waktu total = 5.968748 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-52.fasta
Banyak kombinasi pasangan sekuen = 1378 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 5.484156 dtk waktu penentuan dan update sekuen star = 0.572249 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.643050 dtk waktu total = 6.699454 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-56.fasta
Banyak kombinasi pasangan sekuen = 1540 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 6.614786 dtk waktu penentuan dan update sekuen star = 0.638398 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.722411 dtk waktu total = 7.975595 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-60.fasta
Banyak kombinasi pasangan sekuen = 1770 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 7.624032 dtk waktu penentuan dan update sekuen star = 0.694821 dtk
34
waktu total = 9.109212 dtk
$ mpirun -np 3 -f hosts3 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-64.fasta
Banyak kombinasi pasangan sekuen = 2016 Banyak komputer = 3 - 1 = 2
waktu proses pengisian cell matrik = 8.699073 dtk waktu penentuan dan update sekuen star = 0.747635 dtk
35 Lampiran 3 Hasil pengujian aplikasi MSA paralel dengan 4 PC
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-3.fasta
Banyak kombinasi pasangan sekuen = 3 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.017253 dtk waktu penentuan dan update sekuen star = 0.017917 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.017730 dtk waktu total = 0.052900 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-4.fasta
Banyak kombinasi pasangan sekuen = 6 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.028503 dtk waktu penentuan dan update sekuen star = 0.028312 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.028533 dtk waktu total = 0.085348 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-8.fasta
Banyak kombinasi pasangan sekuen = 28 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.091121 dtk waktu penentuan dan update sekuen star = 0.066079 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.067434 dtk waktu total = 0.224635 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-12.fasta
Banyak kombinasi pasangan sekuen = 66 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.199108 dtk waktu penentuan dan update sekuen star = 0.104743 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.109981 dtk waktu total = 0.413832 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-16.fasta
Banyak kombinasi pasangan sekuen = 120 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.351066 dtk waktu penentuan dan update sekuen star = 0.146745 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.156813 dtk waktu total = 0.654624 dtk
data/Glycine/Glycine-max-36
chromosome-9-BBI-test-20.fasta
Banyak kombinasi pasangan sekuen = 190 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.548206 dtk waktu penentuan dan update sekuen star = 0.191026 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.205378 dtk waktu total = 0.944610 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-24.fasta
Banyak kombinasi pasangan sekuen = 276 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 0.796891 dtk waktu penentuan dan update sekuen star = 0.236006 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.257557 dtk waktu total = 1.290454 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-28.fasta
Banyak kombinasi pasangan sekuen = 378 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 1.085998 dtk waktu penentuan dan update sekuen star = 0.282403 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.309993 dtk waktu total = 1.678394 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-32.fasta
Banyak kombinasi pasangan sekuen = 496 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 1.422739 dtk waktu penentuan dan update sekuen star = 0.330478 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.367917 dtk waktu total = 2.121135 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-36.fasta
Banyak kombinasi pasangan sekuen = 630 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 1.808011 dtk waktu penentuan dan update sekuen star = 0.379983 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.425679 dtk waktu total = 2.613673 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-40.fasta
37 waktu proses pengisian cell matrik = 2.235190 dtk
waktu penentuan dan update sekuen star = 0.430151 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.482759 dtk waktu total = 3.148101 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-44.fasta
Banyak kombinasi pasangan sekuen = 946 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 2.703796 dtk waktu penentuan dan update sekuen star = 0.481674 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.539644 dtk waktu total = 3.725114 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-48.fasta
Banyak kombinasi pasangan sekuen = 1128 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 3.222652 dtk waktu penentuan dan update sekuen star = 0.532569 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.597609 dtk waktu total = 4.352830 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-52.fasta
Banyak kombinasi pasangan sekuen = 1378 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 3.793804 dtk waktu penentuan dan update sekuen star = 0.572327 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.643276 dtk waktu total = 5.009407 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-56.fasta
Banyak kombinasi pasangan sekuen = 1540 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 4.414007 dtk waktu penentuan dan update sekuen star = 0.638328 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.722466 dtk waktu total = 5.774801 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-60.fasta
Banyak kombinasi pasangan sekuen = 1770 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 5.071873 dtk waktu penentuan dan update sekuen star = 0.693032 dtk
38
waktu total = 6.553291 dtk
$ mpirun -np 4 -f hosts4 ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-64.fasta
Banyak kombinasi pasangan sekuen = 2016 Banyak komputer = 4 - 1 = 3
waktu proses pengisian cell matrik = 5.790389 dtk waktu penentuan dan update sekuen star = 0.748102 dtk
39 Lampiran 4 Hasil pengujian aplikasi MSA paralel dengan 5 PC
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-3.fasta
Banyak kombinasi pasangan sekuen = 3 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.019443 dtk waktu penentuan dan update sekuen star = 0.017960 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.017724 dtk waktu total = 0.055128 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-4.fasta
Banyak kombinasi pasangan sekuen = 6 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.026037 dtk waktu penentuan dan update sekuen star = 0.028335 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.028553 dtk waktu total = 0.082925 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-8.fasta
Banyak kombinasi pasangan sekuen = 28 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.070029 dtk waktu penentuan dan update sekuen star = 0.066134 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.067437 dtk waktu total = 0.203600 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-12.fasta
Banyak kombinasi pasangan sekuen = 66 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.151491 dtk waktu penentuan dan update sekuen star = 0.104755 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.109993 dtk waktu total = 0.366240 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-16.fasta
Banyak kombinasi pasangan sekuen = 120 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.268061 dtk waktu penentuan dan update sekuen star = 0.146787 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.156792 dtk waktu total = 0.571640 dtk
data/Glycine/Glycine-max-40
chromosome-9-BBI-test-20.fasta
Banyak kombinasi pasangan sekuen = 190 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.417370 dtk waktu penentuan dan update sekuen star = 0.190907 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.205422 dtk waktu total = 0.813699 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-24.fasta
Banyak kombinasi pasangan sekuen = 276 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.600906 dtk waktu penentuan dan update sekuen star = 0.236071 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.257678 dtk waktu total = 1.094655 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-28.fasta
Banyak kombinasi pasangan sekuen = 378 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 0.818885 dtk waktu penentuan dan update sekuen star = 0.282356 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.310078 dtk waktu total = 1.411319 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-32.fasta
Banyak kombinasi pasangan sekuen = 496 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 1.068426 dtk waktu penentuan dan update sekuen star = 0.330378 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.367864 dtk waktu total = 1.766667 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-36.fasta
Banyak kombinasi pasangan sekuen = 630 Banyak komputer = 5 - 1 = 4
waktu proses pengisian cell matrik = 1.359025 dtk waktu penentuan dan update sekuen star = 0.380009 dtk
waktu realigment sekuen star dengan sekuen lainnya = 0.425773 dtk waktu total = 2.164807 dtk
$ mpirun -np 5 -f hosts ./msa-paralel-mpi < data/Glycine/Glycine-max-chromosome-9-BBI-test-40.fasta