1
PENDAHULUAN
Kambing, Capra hircus, merupakan anggota kelas Mamalia, ordo Artiodaktila, sub ordo Ruminansia dan famili Bovidae (Devendra & McLeroy 1982). Kambing banyak digunakan sebagai hewan ternak penghasil daging, susu, dan serat (Ensminger 2002). Di Indonesia, memelihara ternak kambing masih tergolong sebagai usaha sampingan (small entrepreneurship) oleh petani penggarap lahan, yang biasanya memelihara kambing sebanyak 2-10 ekor (Devandra & Burns 1994).
Kambing sebagai hewan ternak pertama kali dimasukkan ke Jawa pada tahun 1700-an oleh Pemerintahan Hindia Belanda, yang kemudian menyebar ke berbagai wilayah di Indonesia. Di setiap wilayah, kemudian ternak kambing berkembang membentuk kambing-kambing lokal Indonesia yang dicirikan dengan karakter teradaptasi ke kondisi kelokalannya. Karakter lokal yang umum ditemukan di seluruh wilayah Indonesia adalah menurunnya laju pertumbuhan dan ukuran tubuh, bersifat prolifik (kemampuan beranak lebih dari satu ekor), dan lebih tahan terhadap parasit dan iklim tropis. Sebagai hewan ternak, salah satu campur tangan peternak yang menonjol adalah membantu proses perkawinan untuk memperoleh ternak-ternak yang lebih baik. Biasanya peternak-ternak menyukai ternak jantan unggul untuk dikawinkan dengan induk betina yang dipunyainya secara turun-menurun di suatu wilayah. Perkawinan berbantuan manusia tersebut akan meningkatkan keragaman ternak kambing, baik antar wilayah maupun di dalam suatu wilayah. Untuk mengungkapkan hal tersebut, dilakukan analisis keragaman ternak kambing yang ada di beberapa wilayah di Indonesia berdasarkan genom mitokondria.
Genom mitokondria (mtDNA) vertebrata merupakan molekul DNA yang berbentuk sirkular, dengan ukuran 15-20 ribu pasang basa. Genom mitokondria ini berisi 22 gen yang menyandikan tRNA, 2 gen menyandikan rRNA, 13 gen menyandikan protein, dan sebuah ruas yang disebut Daerah Pengendali (control region atau d-loop) sebagai tempat dimulainya replikasi dan transkripsi (Avise 1994). Variasi panjang mtDNA pada vertebrata lebih disebabkan oleh duplikasi gen daripada adanya tambahan gen. Genom mitokondria memperlihatkan pewarisan sifat genetik melalui garis maternal, artinya mtDNA hanya diwariskan dari induk betina. Selain itu, mtDNA tidak mengalami
rekombinasi dan banyak dilaporkan mempunyai laju mutasi yang cepat (MacHugh & Bradley 2001). Atas dasar ini, mtDNA menjadi populer sebagai penanda molekuler keragaman genetik pada berbagai hewan.
Keragaman genetik mtDNA dapat diketahui dengan cara mengamplifikasi mtDNA pada sekuen tertentu secara in vitro. Metode ini dikenal sebagai Polymerase Chain Reaction (PCR). Keragaman mtDNA dapat diketahui dengan memotong hasil amplifikasi PCR menggunakan enzim restriksi, yang dikenal dengan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Variasi panjang hasil pemotongan ini yang akan memperlihatkan keragaman mtDNA akibat perbedaan-perbedaan situs enzim restriksi yang digunakan. Selain itu, keragaman mtDNA dapat diketahui dengan metode sekuensing, suatu metode untuk mengetahui runutan nukleotida, yang dilanjutkan dengan analisis penyejajaran (alignment) antar sampel.
Penelitian ini bertujuan untuk mengetahui keragaman kambing lokal berdasarkan Daerah Pengendali mtDNA.
METODE
Bahan
Sampel darah kambing, berupa buffy coat, yang digunakan pada penelitian ini merupakan koleksi beku Dr. Bess Tiesnamurti, Puslitbangnak Bogor. Sampel tersebut berasal dari Sumatera Utara (22 sampel), Muara-kab. Tapanuli Utara (30), Samosir (31), Marica- Sulawesi Selatan (30), dan Gembrong-Bali (13).
Metode
Ekstraksi dan isolasi DNA. Ekstraksi DNA dilakukan menggunakan kit ekstraksi DNA (Real Genomics™ Genomic DNA Extraction Kit – Real Biotech Corporation), dengan prosedur sesuai dengan petunjuk produsen untuk buffy coat.
Amplifikasi Daerah Pengendali. Ruas Daerah Pengendali pada DNA mitokondria diamplifikasi menggunakan mesin Thermocycler (TaKaRa PCR Thermal Cycler MP4 – TaKaRa Biomedicals). Amplifikasi ruas ini menggunakan pasangan primer AF22
(5’-GCG TAC GCA ATC TTA CGA
1
PENDAHULUAN
Kambing, Capra hircus, merupakan anggota kelas Mamalia, ordo Artiodaktila, sub ordo Ruminansia dan famili Bovidae (Devendra & McLeroy 1982). Kambing banyak digunakan sebagai hewan ternak penghasil daging, susu, dan serat (Ensminger 2002). Di Indonesia, memelihara ternak kambing masih tergolong sebagai usaha sampingan (small entrepreneurship) oleh petani penggarap lahan, yang biasanya memelihara kambing sebanyak 2-10 ekor (Devandra & Burns 1994).
Kambing sebagai hewan ternak pertama kali dimasukkan ke Jawa pada tahun 1700-an oleh Pemerintahan Hindia Belanda, yang kemudian menyebar ke berbagai wilayah di Indonesia. Di setiap wilayah, kemudian ternak kambing berkembang membentuk kambing-kambing lokal Indonesia yang dicirikan dengan karakter teradaptasi ke kondisi kelokalannya. Karakter lokal yang umum ditemukan di seluruh wilayah Indonesia adalah menurunnya laju pertumbuhan dan ukuran tubuh, bersifat prolifik (kemampuan beranak lebih dari satu ekor), dan lebih tahan terhadap parasit dan iklim tropis. Sebagai hewan ternak, salah satu campur tangan peternak yang menonjol adalah membantu proses perkawinan untuk memperoleh ternak-ternak yang lebih baik. Biasanya peternak-ternak menyukai ternak jantan unggul untuk dikawinkan dengan induk betina yang dipunyainya secara turun-menurun di suatu wilayah. Perkawinan berbantuan manusia tersebut akan meningkatkan keragaman ternak kambing, baik antar wilayah maupun di dalam suatu wilayah. Untuk mengungkapkan hal tersebut, dilakukan analisis keragaman ternak kambing yang ada di beberapa wilayah di Indonesia berdasarkan genom mitokondria.
Genom mitokondria (mtDNA) vertebrata merupakan molekul DNA yang berbentuk sirkular, dengan ukuran 15-20 ribu pasang basa. Genom mitokondria ini berisi 22 gen yang menyandikan tRNA, 2 gen menyandikan rRNA, 13 gen menyandikan protein, dan sebuah ruas yang disebut Daerah Pengendali (control region atau d-loop) sebagai tempat dimulainya replikasi dan transkripsi (Avise 1994). Variasi panjang mtDNA pada vertebrata lebih disebabkan oleh duplikasi gen daripada adanya tambahan gen. Genom mitokondria memperlihatkan pewarisan sifat genetik melalui garis maternal, artinya mtDNA hanya diwariskan dari induk betina. Selain itu, mtDNA tidak mengalami
rekombinasi dan banyak dilaporkan mempunyai laju mutasi yang cepat (MacHugh & Bradley 2001). Atas dasar ini, mtDNA menjadi populer sebagai penanda molekuler keragaman genetik pada berbagai hewan.
Keragaman genetik mtDNA dapat diketahui dengan cara mengamplifikasi mtDNA pada sekuen tertentu secara in vitro. Metode ini dikenal sebagai Polymerase Chain Reaction (PCR). Keragaman mtDNA dapat diketahui dengan memotong hasil amplifikasi PCR menggunakan enzim restriksi, yang dikenal dengan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Variasi panjang hasil pemotongan ini yang akan memperlihatkan keragaman mtDNA akibat perbedaan-perbedaan situs enzim restriksi yang digunakan. Selain itu, keragaman mtDNA dapat diketahui dengan metode sekuensing, suatu metode untuk mengetahui runutan nukleotida, yang dilanjutkan dengan analisis penyejajaran (alignment) antar sampel.
Penelitian ini bertujuan untuk mengetahui keragaman kambing lokal berdasarkan Daerah Pengendali mtDNA.
METODE
Bahan
Sampel darah kambing, berupa buffy coat, yang digunakan pada penelitian ini merupakan koleksi beku Dr. Bess Tiesnamurti, Puslitbangnak Bogor. Sampel tersebut berasal dari Sumatera Utara (22 sampel), Muara-kab. Tapanuli Utara (30), Samosir (31), Marica- Sulawesi Selatan (30), dan Gembrong-Bali (13).
Metode
Ekstraksi dan isolasi DNA. Ekstraksi DNA dilakukan menggunakan kit ekstraksi DNA (Real Genomics™ Genomic DNA Extraction Kit – Real Biotech Corporation), dengan prosedur sesuai dengan petunjuk produsen untuk buffy coat.
Amplifikasi Daerah Pengendali. Ruas Daerah Pengendali pada DNA mitokondria diamplifikasi menggunakan mesin Thermocycler (TaKaRa PCR Thermal Cycler MP4 – TaKaRa Biomedicals). Amplifikasi ruas ini menggunakan pasangan primer AF22
(5’-GCG TAC GCA ATC TTA CGA
2
sampai ke bagian awal gen tRNA Phe. Komposisi reaksi amplifikasi bervolume 25 µl adalah sampel DNA sebagai cetakan 2 µl (10-100 ng), 2,5nM Primer 2 µl; Taq polimerase (New England Biolabs) 1 unit beserta bufernya, 2,5mM MgCl2 2 µl, 2mM dNTP 4 µl, dan volume diatur dengan air steril sampai 25 µl. Proses amplifikasi dilakukan pada kondisi suhu pra-denaturasi 94oC selama 5 menit, dilanjutkan 30 siklus dengan tahap denaturasi 94oC selama 1 menit, penempelan primer (annealing) pada suhu 55oC selama 1 menit, dan síntesis DNA ruas target pada suhu 72oC selama 2 menit. Proses diakhiri dengan síntesis DNA akhir pada suhu 72oC selama 10 menit dan penyimpanan pada suhu 10oC sampai mesin dimatikan. Kualitas hasil amplifikasi dipastikan dengan elektroforesis gel poliakrilamid 6 %, diikuti dengan pewarnaan perak (Farajallah et al. 1998).
Pemotongan dengan enzim restriksi. Metode RFLP (Restriction Fragment Length Polymorphism) digunakan untuk mengetahui keragaman ruas target berdasarkan polimorfisme situs restriksi. Enzim yang digunakan dalam penelitian ini adalah AluI (AG↓CT), DdeI (C↓TNAG), MboI (↓GATC), BamHI (G↓GATCC), dan HaeIII (GG↓CC). Kondisi reaksi pemotongan dari setiap enzim restriksi mengikuti petunjuk teknis produsen, yaitu menggunakan bufer yang menyertai setiap enzim restriksi dan menginkubasinya pada suhu 37oC selama semalam. Hasil pemotongan dipisah-pisahkan dengan gel poliakrilamid 6 % dalam bufer 1xTBE (Tris 0,5 M; Asam Borat 0,65 M; EDTA 0,02 M) pada tegangan 180mV selama 80 menit. Pola Hasil pemotongan oleh enzim restriksi kemudian divisualisasikan dengan pawarnaan perak. Pola pita hasil pemotongan tiap enzim restriksi digunakan untuk menentukan haplotipe mtDNA atau disebut sebagai genotiping. Genotiping dilakukan dengan cara menentukan ukuran panjang potongan DNA berdasarkan jarak migrasi pada gel poliakrilamid yang diacukan pada DNA ladder 100 base pair (Biorad).
Sekuensing Produk PCR ruas target. Sekuensing dilakukan terhadap dua sampel yang berasal dari Bali (N30) dan Sulawesi Selatan (N123). Sekuensing dengan metode big dye determinator dilakukan menggunakan ABI Prism 3700 (lembaga biologi molekuler PT. CHAROEN POKPHAND INDONESIA).
Analisis Nukleotida. Untuk mengetahui keragaman kambing, hasil sekuen sampel dialignment dengan beberapa sekuen Daerah Pengendali mtDNA kambing yang tersedia di
GenBank (http://ncbi.nlm.nih.gov) mengguna-kan program ClustalX 1.83. Penghitungan komposisi nukleotida, jarak genetik, dan konstruksi pohon filogeni dilakukan meng-gunakan program MEGA versi 3 (Kumar et al. 2004) berdasarkan data perbedaan-perbedaan nukleotida.
HASIL
Amplifikasi Ruas Target
Amplifikasi menggunakan pasangan primer AF22 dan AF24 memberikan hasil sebesar 1700 pasang basa (pb) (Gambar 1). Produk amplifikasi dengan menggunakan pasangan primer ini meliputi bagian akhir gen Cyt b sampai sebelum gen tRNA Phe. Ukuran DNA hasil amplikasi tersebut sesuai dengan perkiraan hasil amplifikasi dari desain primer. Selain itu, kontrol primer internal AF23 (3’ -GTA GCT GGA CTT AAC TGC AT-5’) yang didesain berada di bagian tengah ruas AF22 dan AF24 memberikan konfirmasi yang lebih pasti bahwa hasil amplifikasi yang dimaksud adalah ruas DNA target. Dari seluruh sampel (n=126), 102 sampel di antaranya menunjukkan hasil DNA yang teramplifikasi.
PCR-RFLP
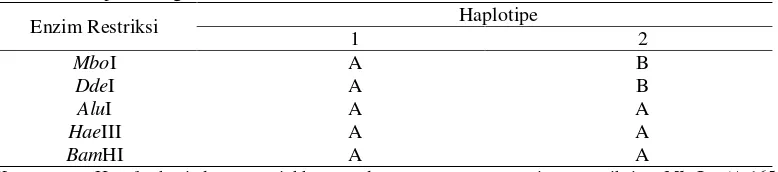
Hasil pemotongan menggunakan lima jenis enzim restriksi, yaitu DdeI (C↓TNAG), MboI (↓GATC), HaeIII (GG↓CC), AluI (AG↓CT), dan BamHI (G↓GATCC) terhadap DNA hasil amplifikasi disajikan dalam Tabel 1. Ada dua haplotipe kambing yang ditemukan berdasarkan lima enzim restriksi. Haplotipe pertama mewakili hampir semua sampel yang dianalisis, yaitu 98 ekor kambing yang menyebar di lima wilayah. Sedangkan haplotipe kedua hanya diwakili oleh empat ekor kambing yang mewakili Wilayah Marica Sulawesi Selatan. Dengan kata lain, sebagian besar (96,08 % dari sampel yang digunakan) ternak kambing di Indonesia bersifat monomorfik berdasarkan situs-situs pemotongan yang digunakan dalam penelitian ini.
2
sampai ke bagian awal gen tRNA Phe. Komposisi reaksi amplifikasi bervolume 25 µl adalah sampel DNA sebagai cetakan 2 µl (10-100 ng), 2,5nM Primer 2 µl; Taq polimerase (New England Biolabs) 1 unit beserta bufernya, 2,5mM MgCl2 2 µl, 2mM dNTP 4 µl, dan volume diatur dengan air steril sampai 25 µl. Proses amplifikasi dilakukan pada kondisi suhu pra-denaturasi 94oC selama 5 menit, dilanjutkan 30 siklus dengan tahap denaturasi 94oC selama 1 menit, penempelan primer (annealing) pada suhu 55oC selama 1 menit, dan síntesis DNA ruas target pada suhu 72oC selama 2 menit. Proses diakhiri dengan síntesis DNA akhir pada suhu 72oC selama 10 menit dan penyimpanan pada suhu 10oC sampai mesin dimatikan. Kualitas hasil amplifikasi dipastikan dengan elektroforesis gel poliakrilamid 6 %, diikuti dengan pewarnaan perak (Farajallah et al. 1998).
Pemotongan dengan enzim restriksi. Metode RFLP (Restriction Fragment Length Polymorphism) digunakan untuk mengetahui keragaman ruas target berdasarkan polimorfisme situs restriksi. Enzim yang digunakan dalam penelitian ini adalah AluI (AG↓CT), DdeI (C↓TNAG), MboI (↓GATC), BamHI (G↓GATCC), dan HaeIII (GG↓CC). Kondisi reaksi pemotongan dari setiap enzim restriksi mengikuti petunjuk teknis produsen, yaitu menggunakan bufer yang menyertai setiap enzim restriksi dan menginkubasinya pada suhu 37oC selama semalam. Hasil pemotongan dipisah-pisahkan dengan gel poliakrilamid 6 % dalam bufer 1xTBE (Tris 0,5 M; Asam Borat 0,65 M; EDTA 0,02 M) pada tegangan 180mV selama 80 menit. Pola Hasil pemotongan oleh enzim restriksi kemudian divisualisasikan dengan pawarnaan perak. Pola pita hasil pemotongan tiap enzim restriksi digunakan untuk menentukan haplotipe mtDNA atau disebut sebagai genotiping. Genotiping dilakukan dengan cara menentukan ukuran panjang potongan DNA berdasarkan jarak migrasi pada gel poliakrilamid yang diacukan pada DNA ladder 100 base pair (Biorad).
Sekuensing Produk PCR ruas target. Sekuensing dilakukan terhadap dua sampel yang berasal dari Bali (N30) dan Sulawesi Selatan (N123). Sekuensing dengan metode big dye determinator dilakukan menggunakan ABI Prism 3700 (lembaga biologi molekuler PT. CHAROEN POKPHAND INDONESIA).
Analisis Nukleotida. Untuk mengetahui keragaman kambing, hasil sekuen sampel dialignment dengan beberapa sekuen Daerah Pengendali mtDNA kambing yang tersedia di
GenBank (http://ncbi.nlm.nih.gov) mengguna-kan program ClustalX 1.83. Penghitungan komposisi nukleotida, jarak genetik, dan konstruksi pohon filogeni dilakukan meng-gunakan program MEGA versi 3 (Kumar et al. 2004) berdasarkan data perbedaan-perbedaan nukleotida.
HASIL
Amplifikasi Ruas Target
Amplifikasi menggunakan pasangan primer AF22 dan AF24 memberikan hasil sebesar 1700 pasang basa (pb) (Gambar 1). Produk amplifikasi dengan menggunakan pasangan primer ini meliputi bagian akhir gen Cyt b sampai sebelum gen tRNA Phe. Ukuran DNA hasil amplikasi tersebut sesuai dengan perkiraan hasil amplifikasi dari desain primer. Selain itu, kontrol primer internal AF23 (3’ -GTA GCT GGA CTT AAC TGC AT-5’) yang didesain berada di bagian tengah ruas AF22 dan AF24 memberikan konfirmasi yang lebih pasti bahwa hasil amplifikasi yang dimaksud adalah ruas DNA target. Dari seluruh sampel (n=126), 102 sampel di antaranya menunjukkan hasil DNA yang teramplifikasi.
PCR-RFLP
Hasil pemotongan menggunakan lima jenis enzim restriksi, yaitu DdeI (C↓TNAG), MboI (↓GATC), HaeIII (GG↓CC), AluI (AG↓CT), dan BamHI (G↓GATCC) terhadap DNA hasil amplifikasi disajikan dalam Tabel 1. Ada dua haplotipe kambing yang ditemukan berdasarkan lima enzim restriksi. Haplotipe pertama mewakili hampir semua sampel yang dianalisis, yaitu 98 ekor kambing yang menyebar di lima wilayah. Sedangkan haplotipe kedua hanya diwakili oleh empat ekor kambing yang mewakili Wilayah Marica Sulawesi Selatan. Dengan kata lain, sebagian besar (96,08 % dari sampel yang digunakan) ternak kambing di Indonesia bersifat monomorfik berdasarkan situs-situs pemotongan yang digunakan dalam penelitian ini.
3
Tabel 1 Pola pemotongan enzim restriksi
Enzim Restriksi Haplotipe
1 2
MboI A B
DdeI A B
AluI A A
HaeIII A A
BamHI A A
Keterangan: Huruf kapital menunjukkan pola pemotongan enzim restriksi. MboI (A:165+ 650+140+175+570,B:(800+300+340+250),DdeI(A:750+320+300+250+80,B:700+600+160+ 150),AluI (A:280+800+320+310), HaeIII (A: 710+500+60+150+350), BamHI (A:1100+600).
Sekuensing
Dua produk PCR disekuensing mengguna-kan primer AF22 untuk mengetahui urutan nukleotidanya, yaitu kambing dari Sulawesi Selatan (N123) dan dari Gembrong Bali (N30). Dua sampel ini mewakili untuk tiap haplotipe. N30 merupakan sampel yang masuk ke dalam haplotipe 1 dan N123 ter-masuk ke dalam haplotipe 2. Hasil sekuensing satu arah yang diperoleh adalah sepanjang 672 nuklotida (nt) untuk sampel N30 dan 647 nt untuk sampel N123. Alignment dua sampel menggunakan program ClustalX 1.83 dan telah diedit dengan program MEGA3 meng-hasilkan 647 nt (gambar 2).
Berdasarkan hasil alignment dengan beberapa individu di GenBank dapat diketahui bahwa hasil sekuensing kedua sampel tersebut meliputi sekuen dari gen Cyt b, tRNA Thr, tRNA Pro, dan Daerah Pengendali. Gen Cyt b berada pada lokasi basa ke 1 s.d. 251. Gen Cyt b diakhiri oleh stop codon AGA, seperti pada Babi Sus scrofa (Ursing & Arnason 1998) dan Domba Ovis aries (Hiendleder et al. 1998). Pada gen ini terdapat empat transisi basa, yaitu pada posisi 44, 111, 153 berupa transisi G-A dan pada posisi 189 berupa transisi C-T. Gen tRNA Thr dan Pro, penyandi tRNA Trionin dan Prolin, relatif lebih stabil. Gen tRNA Thr dan tRNA Pro ini terdapat pada posisi basa ke 252-389. Daerah Pengendali terdapat pada posisi basa ke 390-648. Pada Daerah Pengendali ini terdapat satu transisi G-A pada basa ke 629.
Analisis Daerah Pengendali mtDNA Proses alignment dilanjutkan dengan membandingkan sekuen Daerah Pengendali dua sampel (N30 & N123) dengan 62 individu kambing yang ada di GenBank. Hasil alignment menghasilkan sekuen sepanjang 259 nt. Dari 259 nt yang telah dialignment, jumlah nukleotida yang sama untuk semua sampel adalah 229 nt dan jumlah yang berbeda 30 nt. Tiga puluh nukleotida yang
4 Gambar 2 Hasil Alignment sampel dari Gembrong-Bali (N30) dan Marica-Sulawesi Selatan (N123).
#N30 GTGTCCTAAT CTTAGTACTT GTACCCTTCC TCCACACATC TAAGCAACGA AGCATAATAT TCCGCCCAAT CAGCCAATGC [ 80] #N123 ... ... ... ... ...A... ... ... ... [ 80]
#N30 ATATTCTGAA TCCTGGTAGC AGATCTATTA ACACTCACAT GAATTGGAGG ACAGCCAGTC GAACATCCCT ACGTTATTAT [160] #N123 ... ... ... G... ... ... ... ..A... [160]
#N30 TGGACAACTA GCATCTATCA TATATTTCCT CATCATTCTA GTAATAATAC CAGCAGCTAG CACCATTGAA AACAACCTTC [240] #N123 ... ... ...T. ... ... ... ... ... [240]
#N30 TAAAATGAAG ACAAGTCTTT GTAGTACAAT CAATACACTG GTCTTGTAAA CCAGAAAAGG AGAATAGCCA ATCTCCCTAA [320] #N123 ... ... ... ... ... ... ... ... [320]
#N30 GACTCAAGGA AGAAGCCATA GCCTCACTAT CAGCACCCAA AGCTGAAATT CTATTTAAAC TATTCCCTGA ACCACTATTA [400] #N123 ... ... ... ... ... ... ... ... [400]
#N30 ACCACATCTA TTAATATACC CCCAAAAATA TTAAGAGCCT CCCCAGTATT AAATTTACTA AAAATTTCAA ATATACAACA [480] #N123 ... ... ... ... ... ... ... ... [480]
#N30 CAAACTTCCC ACTCCACAAG CTTACAGACA TGCCAACAAC CCACACGTAT AAAAACATCC CAATCCTAAC CCAACTTAGA [560] #N123 ... ... ... ... ... ... ... ... [560]
#N30 TACCCACACA AACGCCAACA CCACACAATG TTACGCGTAT GCAAGTACAT TACACCGCTC GCCTACACGC AAATACATTT [640] #N123 ... ... ... ... ... ... ...A. ... [640]
#N30 ACTAACAT [648] #N123 ... [648]
DdeI
MboI
AluI
DdeI
AluI
DdeI
DdeI
5
Tabel 2 Rata-rata jarak genetik antar populasi kambing di berbagai negara Negara
Negara
Indonesia Laos Pakistan Cina Unknown* Jepang
Indonesia [0.004] [0.007] [0.006] [0.008] [0.007]
Laos 0.006 [0.007] [0.007] [0.008] [0.008]
Pakistan 0.017 0.020 [0.003] [0.004] [0.002]
Cina 0.018 0.020 0.012 [0.004] [0.002]
Unknown* 0.019 0.021 0.010 0.010 [0.003]
Jepang 0.015 0.017 0.006 0.007 0.004
Keterangan : Jarak genetik : di bawah diagonal, Standar deviasi : di atas diagonal *: sekuen Daerah Pengendali yang tersedia di GenBank tanpa keterangan asal sampel
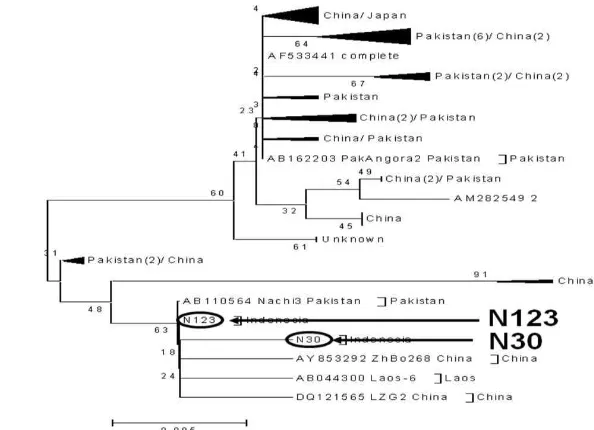
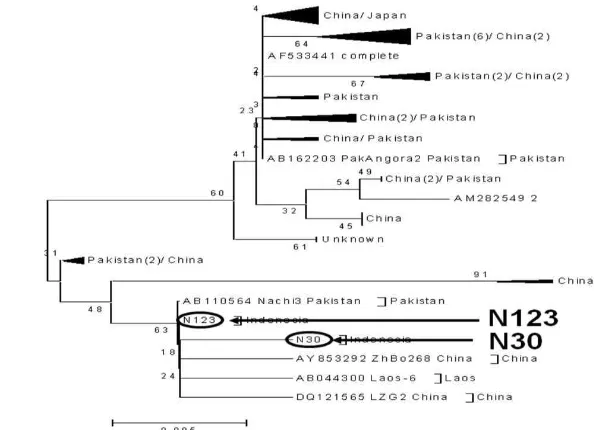
Gambar 3 Hasil rekonstruksi pohon filogeni dengan metode Neighbor Joining (bootstrap 250x). Kambing Gembrong dari Bali
mengelompok dengan kambing dari Laos dan Cina pada semua metode konstruksi pohon filogeni, sedangkan kambing dari Sulawesi Selatan berada pada posisi pangkal dari kelompok tersebut (Gambar 3). Pada posisi cabang yang lebih ke pangkal bisa ditemukan dua individu kambing dari Cina dengan laju mutasi yang sangat tinggi. Hal ini mengindikasikan kambing-kambing tersebut telah mengalami tekanan seleksi (oleh manusia) yang sangat besar.
PEMBAHASAN
Hasil PCR-RFLP yang sebagian besar monomorfik menunjukkan bahwa ternak kambing yang ada di Indonesia kemungkinan besar berasal dari induk betina yang sama. Yaitu, kambing awal yang masuk ke Indonesia sekitar tahun 1700-an. Selain itu, bisa disebutkan bahwa pola migrasi kambing di Indonesia berdasarkan kambing jantan.
Pada umumnya, untuk meningkatkan kualitas ternak (upgrading), para petani mendatangkan pejantan-pejantan unggul dari luar wilayah-nya. Dalam proses itu, induk-induk betina secara turun-temurun sejak awal masuknya tetap berada di wilayahnya. Hasil pemotongan enzim DdeI dan MboI menghasilkan pola polimorfik hanya pada empat sampel yang berasal dari Marica Sulawesi Selatan, hal ini kemungkinan disebabkan adanya percampur-an dengpercampur-an kambing Ettawah ypercampur-ang masuk ke Indonesia sekitar tahun 1900-an.
6
basa di antara kedua sampel. Sampel N123 terjadi perubahan basa G-A yang menyebabkan enzim restriksi DdeI tidak mengenali situs ini.
Daerah Pengendali digunakan sebagai dasar analisis keragaman pada penelitian ini karena banyak dilaporkan bahwa daerah ini dapat digunakan sebagai marker dalam analisis keragaman, seperti pada paus (Hoelzel 1991), rusa (Randi et al. 1998), domba (Wu et al. 2003), kerbau (Kierstein 2004), juga pada kambing (Luikart et al. 2001, Joshi et al. 2004, Meadows et al. 2005). Daerah Pengendali berukuran ±1000 pb sebagai tempat inisiasi replikasi dan transkripsi genom mitokondria (Avise 1994). Daerah ini merupakan bagian dari genom mitokondria yang tidak menyandikan protein (non-coding sequence), sehingga tingkat mutasi pada daerah ini relatif tinggi.
Hasil rekonstruksi pohon filogeni menunjukkan bangsa kambing ternak lokal di seluruh dunia berkerabat dekat, karena rata-rata jarak genetik atau tingkat kesamaan gen sebesar 98,8 % (Tabel 2). Hal ini berarti kambing-kambing ternak lokal yang ada di seluruh dunia berasal dari satu induk. Luikart et al. (2001) menulis bahwa kambing me-miliki struktur filogeografik yang lemah, keragaman mtDNA pada kambing hanya sekitar 10 % di beberapa benua di dunia lama, sedangkan sapi mencapai lebih dari 50 %. Hal ini memperlihatkan bahwa kambing dipindah-pindahkan secara ekstensif dan sebagai bukti bahwa kambing merupakan hewan yang penting dalam sejarah migrasi dan perdagang-an yperdagang-ang dilakukperdagang-an oleh mperdagang-anusia. Kambing termasuk salah satu hewan yang pertama kali diternakkan, yaitu sekitar 10000 – 6000 tahun sebelum masehi, banyak penulis menulis bahwa tempat awal domestikasi kambing di daerah pegunungan Asia Barat atau dikenal dengan Fertile Crescent (Devandra & McLeroy 1982; Devandra & Burns 1994; Zeder & Hesse 2000; Ensminger 2002). Daerah yang disebut sebagai Fertile Crescent merupakan pusat domestikasi sebagian besar hewan dan tanaman pertanian yang ada saat ini (Zedder & Hesse 2000). Para ilmuan percaya kambing Benzoar (Capra aegagrus) merupakan progenitor bagi kambing-kambing ternak lokal saat ini, dengan bukti banyak ditemukan fosil di situs-situs arkeologi di daerah Asia Barat (Pegler 1965; Devandra & McLeroy 1982; Devandra & Burns 1994; MacHugh & Bradley 2001; Ensminger 2002). Luikart et al. (2001), yang mempelajari kekerabatan kambing-kambing di dunia lama
berdasarkan keragaman Daerah Pengendali, menulis bahwa Capra hircus memiliki kekerabatan yang tinggi dengan Capra aegagrus (rata-rata 61,3 substitusi).
Kambing ternak lokal (Capra hircus) terdapat di seluruh dunia, khususnya di daerah tropis (Ensminger 2002), karena kambing ini menyukai daerah tropis yang kering (Devendra & McLeroy 1982). Kambing di Indonesia dikenal ada beberapa bangsa, antara lain kambing Kacang, Gembrong, Costa, dan Peranakan Ettawah. Sampel yang digunakan pada penelitian ini termasuk ke dalam bangsa kambing Gembrong, untuk sampel yang berasal dari Bali, dan bangsa kambing Kacang, seperti sampel yang berasal dari Sumatera Utara, Tapanuli, Samosir, dan Sulawesi Selatan. Walaupun tidak menutup kemungkinan adanya percampuran dengan kambing Ettawah yang berasal dari India.
Kambing ternak lokal di Indonesia, sebagian besar digunakan sebagai ternak penghasil daging atau susu. Kambing Kacang memiliki ciri rambut pendek yang berwarna putih, hitam, coklat, atau kombinasi ketiga-nya, hewan jantan dan betina memiliki tanduk, telinga pendek dan tegak, pada jantan selalu terdapat janggut, sedangkan betina jarang ditemukan adanya janggut. Hewan dewasa berukuran panjang 47-55 cm, tinggi pundak 55,3-55,7 cm, mempunyai berat 20 kg (betina) dan 25 kg (jantan). Kambing Gembrong terdapat di daerah kawasan timur Pulau Bali. Ciri khas kambing ini adalah memiliki rambut yang panjang, dengan panjang rambut berkisar 15-25 cm. Rambut panjang ini terdapat pada jantan, sedangkan betina hanya berkisar 2-3 cm. Rambut yang panjang ini biasa dipergunakan oleh nelayan setempat untuk membuat umpan (Devandra & Burns 1994). Kambing Gembrong secara umum memiliki ukuran tubuh yang lebih besar daripada kambing Kacang (http://peternakan.litbang.deptan.go.id).
i
KERAGAMAN GENETIK KAMBING LOKAL
BERDASARKAN GENOM MITOKONDRIA
NICO HIMAWAN
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
7
SIMPULAN
Kambing ternak lokal di beberapa daerah di Indonesia mempunyai situs restriksi dari 5 enzim yang seragam (monomorfik), kecuali empat individu kambing dari Marica. Hal ini dapat diduga bahwa kambing-kambing ternak lokal di Indonesia berasal dari induk yang sama, dengan melihat sifat dari mtDNA. Hasil alignment ternyata memperkuat hipotesis awal, bahwa sebagian basar kambing yang ada di dunia berasal dari induk yang sama.
SARAN
Berkaitan dengan seragamnya kambing-kambing ternak di dunia dalam hal mtDNA menyebabkan produk metabolisme seluler pada kambing-kambing ini juga sama, sehingga untuk meningkatkan kualitas ternak, peternak dari Indonesia dapat mendatangkan kambing ternak unggul dari luar negeri. Penelitian lanjutan dapat dilakukan dengan memperpanjang hasil sekuensing, sehingga seluruh sekuen dari Daerah Pengendali dapat terpetakan atau difokuskan pada keragaman dari garis paternal, yaitu dengan kromosom Y.
DAFTAR PUSTAKA
Avise JC.1994. Molecular Markers, Natural History, and Evolution. New York: Chapman & Hall.
Campbell NA, Reece JB, Mitchell LJ. 2002. Biologi. Lestari R et al., penerjemah; Amalia, Simarmata L, Hardani HW, editor. Jakarta: Erlangga. Terjemahan dari: Biology.
Devandra C, Burns. 1994. Produksi Kambing di daerah Tropis. Putra H, penerjemah. Bandung: Penerbit ITB. Terjemahan dari: Goat Production in the Tropics.
Devandra C, McLeroy GB. 1982. Goat and Sheep Production in the Tropics. Inggris: Longman Group,Ltd.
Ensminger ME. 2002. Sheep and Goat Science. Ed ke-6. Illinois: Interstate Publishers,Inc.
Hiendleder S, Lewalski H, Wassmuth R, Janke A. 1998. The complete mitochondrial DNA sequence of the domestic sheep (Ovis aries) and comparison with the other major ovine haplotype. J. Mol. Evol. 47:441-448. Hoelzel AR, Hancock JM, Dover GA. 1991.
Evolution of the cetacean mitochondrial
d-loop region. Mol. Biol. Evol. 8(3):475-493.
Joshi MB et al. 2004. Phylogeography and origin of indian domestic goat. Mol. Biol. Evol. 21(3):454-462.
Kierstein G et al. 2004. Analysis of mitochondrial d-loop region casts new light on domestic water buffalo (Bubalus bubalis) phylogeny. J. Mol. Phyl. Evol. 30:308-324.
Krettek A, Gullberg A, Arnason U. 1995. Sequence analysis of the complete mitochondrial DNA molecule of the hedgehog, Erinaeus eropaeus, and the phylogenetic of the lipotyphla. J. Mol. Evol 41:952-957.
Kumar S, Tamura K, Nei M. 2004. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Briefings in Bioinformatics 5:150-163.
Luikart G et al. 2001. Multiple meternal origins and weak phylogeographic structure in domestic goats. Proc. Nact. Acad. Sci 98(10):5927-5932.
MacHugh DE, Bradley DG. 2001. Livestock genetic origins: goat buck the trend. Proc. Nact. Acad. Sci 98(10):5382-5384.
Meadows JRS et al. 2005. Mitochondrial sequence reveals high levels of gene flow between breeds of domestic sheep from asia and Europe. J. Heredity: 96(5):494-501.
Pegler HSH. 1965. The Book of the Goat. Inggris: Billing and Sons,Ltd.
Randi E, Pierpaoli M, Danilkin A. 1998. Mitochondrial DNA polymorphism in population of siberian and european roe deer (Capreolus pygargus and C. capreolus). Heredity 80:429-437.
Ursing BM, Arnason U. 1998. The complete mitochondrial DNA sequence of the pig (Sus scrofa). J. Mol. Evol. 47:302-306. Valverde JR, Batuecas B, Maratilla C, Marco
R, Ganesse R. 1994. The complete mitochondrial DNA sequence of the crustacean Artemia franciscana. J. Mol. Evol 39:400-408.
Wu CH, Zhang YP, Bunch TD, Wang S, Wang W. 2003. Mitochondrial control region sequence variation within the argali wild sheep (Ovis ammon): evolution and conservation relevance. Mammalia 67:109-118.
i
KERAGAMAN GENETIK KAMBING LOKAL
BERDASARKAN GENOM MITOKONDRIA
NICO HIMAWAN
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ii
ABSTRAK
NICO HIMAWAN. Keragaman Genetik Kambing Lokal Berdasarkan Genom Mitokondria. Dibimbing oleh ACHMAD FARAJALLAH dan BESS TIESNAMURTI.
Kambing (Capra hircus) dimasukkan pertama kali ke Indonesia sebagai hewan ternak sekitar tahun 1700-an, yang kemudian berkembang manjadi ternak lokal. Peningkatan mutu ternak kambing sebagian besar dilakukan dengan cara mengawinkan ternak betina dengan jantan unggul yang didatangkan dari daerah lain. Penelitian ini dilakukan untuk mengetahui keragaman genetik kambing ternak lokal berdasarkan Daerah Pengendali DNA mitokondria. Sampel yang digunakan adalah kambing ternak lokal yang berasal dari Sumatera Utara, Muara-Tapanuli Utara, Samosir, Marica-Sulawesi Selatan, dan Gembrong-Bali. Amplifikasi DNA mitokondria secara in vitro dilakukan dengan metode Polymerase Chain Rection (PCR). Produk amplifikasi yang dihasilkan sebesar 1700 pasang basa. Produk amplifikasi tersebut dipotong dengan lima enzim restriksi, yaitu MboI, DdeI, AluI, HaeIII, dan BamHI. Dari 102 sampel yang dianalisis, ditemukan dua haplotipe. Haplotipe 1 terdapat pada seluruh wilayah, sedangkan haplotipe 2 hanya terdapat pada empat sampel yang berasal dari Marica Sulawesi Selatan. Hasil sekuensing yang telah dialignment dengan beberapa individu kambing yang terdapat pada GenBank, menunjukkan bahwa kambing-kambing ternak lokal di dunia memiliki kekerabatan yang dekat (98,8 %) dan berasal dari induk yang sama.
ABSTRACT
NICO HIMAWAN. Genetic Diversity of Native Goat Based on Mitochondrial Genome. Supervised by ACHMAD FARAJALLAH and BESS TIESNAMURTI.
iii
KERAGAMAN GENETIK KAMBING LOKAL
BERDASARKAN GENOM MITOKONDRIA
NICO HIMAWAN
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
iv
Judul skripsi : Keragaman Genetik Kambing Lokal Berdasarkan Genom Mitokondria Nama : Nico Himawan
NIM : G34103060
Disetujui
Diketahui
Dekan Fakultas Matematika dan Ilmu Pengetahuan Alam Institut Pertanian Bogor,
Prof. Dr. Ir. Yonny Koesmaryono, M.S. NIP. 131473999
Tanggal Lulus :
Pembimbing I,
Dr. Ir. Achmad Farajallah, M.Si. NIP. 131878947
Pembimbing II,
v
RIWAYAT HIDUP
Penulis dilahirkan di Semarang pada tanggal 17 September 1985 dari ayah Djaka Sarwana dan ibu Kurniati. Penulis merupakan putra pertama dari dua bersaudara.
Tahun 2003 penulis lulus dari SMA Negeri 1 Semarang dan pada tahun yang sama lulus seleksi masuk IPB melalui jalus Undangan Seleksi Masuk IPB. Penulis memilih Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam.
vi
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul yang dipilih dalam penelitian ini ialah Keragaman Genetik Kambing Lokal Berdasarkan Genom Mitokondria.
Terima kasih penulis ucapkan kepada Bapak Dr. Ir. Achmad Farajallah, M.Si dan Ibu Dr. Bess Tiesnamurti selaku pembimbing. Terima kasih penulis sampaikan kepada Bapak Dr. Ir. Aris Tjahjoleksono selaku penguji. Di samping itu, ucapan terima kasih juga penulis tujukan kepada Bapak Ketut, Ibu Rika Raffiudin, Bapak Adi serta semua dosen dan staf zoologi. Ungkapan terima kasih juga penulis sampaikan kepada Bapak, Ibu, Adik, dan yang terkasih Hilda ‘Neng’ Farida atas dukungan, doa, dan kasih sayang yang telah diberikan kepada penulis. Tak lupa kepada Lusi, Mbak Retno, Ogi, Mbak Zul, Wildan, Indra, Rut, Wafa, serta teman-teman zoologi, Biologi 40, teman-teman dari Fapet (Rohmat, Astri, Dani, Eryk, Dian), dan teman-teman dari UNIB (Sipri, Aan, Puji), terima kasih atas bantuan dan pertemanan yang telah terjalin selama ini.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2007
vii
DAFTAR ISI
Halaman
DAFTAR TABEL ...viii
DAFTAR GAMBAR ...viii
DAFTAR LAMPIRAN ...viii
PENDAHULUAN... 1
METODE ... 1
Bahan ... 1
Metode ... 1
Ekstraksi dan isolasi DNA ... 1
Amplifikasi Daerah Pengendali ... 1
Pemotongan dengan enzim restriksi ... 2
Sekuensing Produk PCR ruas target ... 2
Analisis Nukleotida ... 2
HASIL ... 2
Amplifikasi Ruas Target ... 2
PCR-RFLP ... 2
Sekuensing ... 3
Analisis Daerah Pengendali mtDNA ... 3
PEMBAHASAN ... 5
SIMPULAN ... 7
SARAN ... 7
DAFTAR PUSTAKA ... 7
viii
DAFTAR TABEL
Halaman 1 Pola pemotongan enzim restriksi ... 3 2 Rata-rata jarak genetik antar populasi kambing di berbagai negara ... 5
DAFTAR GAMBAR
Halaman 1 Hasil amplifikasi Daerah Pengendali mtDNA. ... 2 2 Hasil Alignment sampel dari Gembrong-Bali (N30) dan Marica-Sulawesi Selatan (N123). ... 4 3 Hasil rekonstruksi pohon filogeni dengan metode Neighbor Joining (bootstrap 250x). ... 5
DAFTAR LAMPIRAN
1
PENDAHULUAN
Kambing, Capra hircus, merupakan anggota kelas Mamalia, ordo Artiodaktila, sub ordo Ruminansia dan famili Bovidae (Devendra & McLeroy 1982). Kambing banyak digunakan sebagai hewan ternak penghasil daging, susu, dan serat (Ensminger 2002). Di Indonesia, memelihara ternak kambing masih tergolong sebagai usaha sampingan (small entrepreneurship) oleh petani penggarap lahan, yang biasanya memelihara kambing sebanyak 2-10 ekor (Devandra & Burns 1994).
Kambing sebagai hewan ternak pertama kali dimasukkan ke Jawa pada tahun 1700-an oleh Pemerintahan Hindia Belanda, yang kemudian menyebar ke berbagai wilayah di Indonesia. Di setiap wilayah, kemudian ternak kambing berkembang membentuk kambing-kambing lokal Indonesia yang dicirikan dengan karakter teradaptasi ke kondisi kelokalannya. Karakter lokal yang umum ditemukan di seluruh wilayah Indonesia adalah menurunnya laju pertumbuhan dan ukuran tubuh, bersifat prolifik (kemampuan beranak lebih dari satu ekor), dan lebih tahan terhadap parasit dan iklim tropis. Sebagai hewan ternak, salah satu campur tangan peternak yang menonjol adalah membantu proses perkawinan untuk memperoleh ternak-ternak yang lebih baik. Biasanya peternak-ternak menyukai ternak jantan unggul untuk dikawinkan dengan induk betina yang dipunyainya secara turun-menurun di suatu wilayah. Perkawinan berbantuan manusia tersebut akan meningkatkan keragaman ternak kambing, baik antar wilayah maupun di dalam suatu wilayah. Untuk mengungkapkan hal tersebut, dilakukan analisis keragaman ternak kambing yang ada di beberapa wilayah di Indonesia berdasarkan genom mitokondria.
Genom mitokondria (mtDNA) vertebrata merupakan molekul DNA yang berbentuk sirkular, dengan ukuran 15-20 ribu pasang basa. Genom mitokondria ini berisi 22 gen yang menyandikan tRNA, 2 gen menyandikan rRNA, 13 gen menyandikan protein, dan sebuah ruas yang disebut Daerah Pengendali (control region atau d-loop) sebagai tempat dimulainya replikasi dan transkripsi (Avise 1994). Variasi panjang mtDNA pada vertebrata lebih disebabkan oleh duplikasi gen daripada adanya tambahan gen. Genom mitokondria memperlihatkan pewarisan sifat genetik melalui garis maternal, artinya mtDNA hanya diwariskan dari induk betina. Selain itu, mtDNA tidak mengalami
rekombinasi dan banyak dilaporkan mempunyai laju mutasi yang cepat (MacHugh & Bradley 2001). Atas dasar ini, mtDNA menjadi populer sebagai penanda molekuler keragaman genetik pada berbagai hewan.
Keragaman genetik mtDNA dapat diketahui dengan cara mengamplifikasi mtDNA pada sekuen tertentu secara in vitro. Metode ini dikenal sebagai Polymerase Chain Reaction (PCR). Keragaman mtDNA dapat diketahui dengan memotong hasil amplifikasi PCR menggunakan enzim restriksi, yang dikenal dengan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Variasi panjang hasil pemotongan ini yang akan memperlihatkan keragaman mtDNA akibat perbedaan-perbedaan situs enzim restriksi yang digunakan. Selain itu, keragaman mtDNA dapat diketahui dengan metode sekuensing, suatu metode untuk mengetahui runutan nukleotida, yang dilanjutkan dengan analisis penyejajaran (alignment) antar sampel.
Penelitian ini bertujuan untuk mengetahui keragaman kambing lokal berdasarkan Daerah Pengendali mtDNA.
METODE
Bahan
Sampel darah kambing, berupa buffy coat, yang digunakan pada penelitian ini merupakan koleksi beku Dr. Bess Tiesnamurti, Puslitbangnak Bogor. Sampel tersebut berasal dari Sumatera Utara (22 sampel), Muara-kab. Tapanuli Utara (30), Samosir (31), Marica- Sulawesi Selatan (30), dan Gembrong-Bali (13).
Metode
Ekstraksi dan isolasi DNA. Ekstraksi DNA dilakukan menggunakan kit ekstraksi DNA (Real Genomics™ Genomic DNA Extraction Kit – Real Biotech Corporation), dengan prosedur sesuai dengan petunjuk produsen untuk buffy coat.
Amplifikasi Daerah Pengendali. Ruas Daerah Pengendali pada DNA mitokondria diamplifikasi menggunakan mesin Thermocycler (TaKaRa PCR Thermal Cycler MP4 – TaKaRa Biomedicals). Amplifikasi ruas ini menggunakan pasangan primer AF22
(5’-GCG TAC GCA ATC TTA CGA
2
sampai ke bagian awal gen tRNA Phe. Komposisi reaksi amplifikasi bervolume 25 µl adalah sampel DNA sebagai cetakan 2 µl (10-100 ng), 2,5nM Primer 2 µl; Taq polimerase (New England Biolabs) 1 unit beserta bufernya, 2,5mM MgCl2 2 µl, 2mM dNTP 4 µl, dan volume diatur dengan air steril sampai 25 µl. Proses amplifikasi dilakukan pada kondisi suhu pra-denaturasi 94oC selama 5 menit, dilanjutkan 30 siklus dengan tahap denaturasi 94oC selama 1 menit, penempelan primer (annealing) pada suhu 55oC selama 1 menit, dan síntesis DNA ruas target pada suhu 72oC selama 2 menit. Proses diakhiri dengan síntesis DNA akhir pada suhu 72oC selama 10 menit dan penyimpanan pada suhu 10oC sampai mesin dimatikan. Kualitas hasil amplifikasi dipastikan dengan elektroforesis gel poliakrilamid 6 %, diikuti dengan pewarnaan perak (Farajallah et al. 1998).
Pemotongan dengan enzim restriksi. Metode RFLP (Restriction Fragment Length Polymorphism) digunakan untuk mengetahui keragaman ruas target berdasarkan polimorfisme situs restriksi. Enzim yang digunakan dalam penelitian ini adalah AluI (AG↓CT), DdeI (C↓TNAG), MboI (↓GATC), BamHI (G↓GATCC), dan HaeIII (GG↓CC). Kondisi reaksi pemotongan dari setiap enzim restriksi mengikuti petunjuk teknis produsen, yaitu menggunakan bufer yang menyertai setiap enzim restriksi dan menginkubasinya pada suhu 37oC selama semalam. Hasil pemotongan dipisah-pisahkan dengan gel poliakrilamid 6 % dalam bufer 1xTBE (Tris 0,5 M; Asam Borat 0,65 M; EDTA 0,02 M) pada tegangan 180mV selama 80 menit. Pola Hasil pemotongan oleh enzim restriksi kemudian divisualisasikan dengan pawarnaan perak. Pola pita hasil pemotongan tiap enzim restriksi digunakan untuk menentukan haplotipe mtDNA atau disebut sebagai genotiping. Genotiping dilakukan dengan cara menentukan ukuran panjang potongan DNA berdasarkan jarak migrasi pada gel poliakrilamid yang diacukan pada DNA ladder 100 base pair (Biorad).
Sekuensing Produk PCR ruas target. Sekuensing dilakukan terhadap dua sampel yang berasal dari Bali (N30) dan Sulawesi Selatan (N123). Sekuensing dengan metode big dye determinator dilakukan menggunakan ABI Prism 3700 (lembaga biologi molekuler PT. CHAROEN POKPHAND INDONESIA).
Analisis Nukleotida. Untuk mengetahui keragaman kambing, hasil sekuen sampel dialignment dengan beberapa sekuen Daerah Pengendali mtDNA kambing yang tersedia di
GenBank (http://ncbi.nlm.nih.gov) mengguna-kan program ClustalX 1.83. Penghitungan komposisi nukleotida, jarak genetik, dan konstruksi pohon filogeni dilakukan meng-gunakan program MEGA versi 3 (Kumar et al. 2004) berdasarkan data perbedaan-perbedaan nukleotida.
HASIL
Amplifikasi Ruas Target
Amplifikasi menggunakan pasangan primer AF22 dan AF24 memberikan hasil sebesar 1700 pasang basa (pb) (Gambar 1). Produk amplifikasi dengan menggunakan pasangan primer ini meliputi bagian akhir gen Cyt b sampai sebelum gen tRNA Phe. Ukuran DNA hasil amplikasi tersebut sesuai dengan perkiraan hasil amplifikasi dari desain primer. Selain itu, kontrol primer internal AF23 (3’ -GTA GCT GGA CTT AAC TGC AT-5’) yang didesain berada di bagian tengah ruas AF22 dan AF24 memberikan konfirmasi yang lebih pasti bahwa hasil amplifikasi yang dimaksud adalah ruas DNA target. Dari seluruh sampel (n=126), 102 sampel di antaranya menunjukkan hasil DNA yang teramplifikasi.
PCR-RFLP
Hasil pemotongan menggunakan lima jenis enzim restriksi, yaitu DdeI (C↓TNAG), MboI (↓GATC), HaeIII (GG↓CC), AluI (AG↓CT), dan BamHI (G↓GATCC) terhadap DNA hasil amplifikasi disajikan dalam Tabel 1. Ada dua haplotipe kambing yang ditemukan berdasarkan lima enzim restriksi. Haplotipe pertama mewakili hampir semua sampel yang dianalisis, yaitu 98 ekor kambing yang menyebar di lima wilayah. Sedangkan haplotipe kedua hanya diwakili oleh empat ekor kambing yang mewakili Wilayah Marica Sulawesi Selatan. Dengan kata lain, sebagian besar (96,08 % dari sampel yang digunakan) ternak kambing di Indonesia bersifat monomorfik berdasarkan situs-situs pemotongan yang digunakan dalam penelitian ini.
3
Tabel 1 Pola pemotongan enzim restriksi
Enzim Restriksi Haplotipe
1 2
MboI A B
DdeI A B
AluI A A
HaeIII A A
BamHI A A
Keterangan: Huruf kapital menunjukkan pola pemotongan enzim restriksi. MboI (A:165+ 650+140+175+570,B:(800+300+340+250),DdeI(A:750+320+300+250+80,B:700+600+160+ 150),AluI (A:280+800+320+310), HaeIII (A: 710+500+60+150+350), BamHI (A:1100+600).
Sekuensing
Dua produk PCR disekuensing mengguna-kan primer AF22 untuk mengetahui urutan nukleotidanya, yaitu kambing dari Sulawesi Selatan (N123) dan dari Gembrong Bali (N30). Dua sampel ini mewakili untuk tiap haplotipe. N30 merupakan sampel yang masuk ke dalam haplotipe 1 dan N123 ter-masuk ke dalam haplotipe 2. Hasil sekuensing satu arah yang diperoleh adalah sepanjang 672 nuklotida (nt) untuk sampel N30 dan 647 nt untuk sampel N123. Alignment dua sampel menggunakan program ClustalX 1.83 dan telah diedit dengan program MEGA3 meng-hasilkan 647 nt (gambar 2).
Berdasarkan hasil alignment dengan beberapa individu di GenBank dapat diketahui bahwa hasil sekuensing kedua sampel tersebut meliputi sekuen dari gen Cyt b, tRNA Thr, tRNA Pro, dan Daerah Pengendali. Gen Cyt b berada pada lokasi basa ke 1 s.d. 251. Gen Cyt b diakhiri oleh stop codon AGA, seperti pada Babi Sus scrofa (Ursing & Arnason 1998) dan Domba Ovis aries (Hiendleder et al. 1998). Pada gen ini terdapat empat transisi basa, yaitu pada posisi 44, 111, 153 berupa transisi G-A dan pada posisi 189 berupa transisi C-T. Gen tRNA Thr dan Pro, penyandi tRNA Trionin dan Prolin, relatif lebih stabil. Gen tRNA Thr dan tRNA Pro ini terdapat pada posisi basa ke 252-389. Daerah Pengendali terdapat pada posisi basa ke 390-648. Pada Daerah Pengendali ini terdapat satu transisi G-A pada basa ke 629.
Analisis Daerah Pengendali mtDNA Proses alignment dilanjutkan dengan membandingkan sekuen Daerah Pengendali dua sampel (N30 & N123) dengan 62 individu kambing yang ada di GenBank. Hasil alignment menghasilkan sekuen sepanjang 259 nt. Dari 259 nt yang telah dialignment, jumlah nukleotida yang sama untuk semua sampel adalah 229 nt dan jumlah yang berbeda 30 nt. Tiga puluh nukleotida yang
4 Gambar 2 Hasil Alignment sampel dari Gembrong-Bali (N30) dan Marica-Sulawesi Selatan (N123).
#N30 GTGTCCTAAT CTTAGTACTT GTACCCTTCC TCCACACATC TAAGCAACGA AGCATAATAT TCCGCCCAAT CAGCCAATGC [ 80] #N123 ... ... ... ... ...A... ... ... ... [ 80]
#N30 ATATTCTGAA TCCTGGTAGC AGATCTATTA ACACTCACAT GAATTGGAGG ACAGCCAGTC GAACATCCCT ACGTTATTAT [160] #N123 ... ... ... G... ... ... ... ..A... [160]
#N30 TGGACAACTA GCATCTATCA TATATTTCCT CATCATTCTA GTAATAATAC CAGCAGCTAG CACCATTGAA AACAACCTTC [240] #N123 ... ... ...T. ... ... ... ... ... [240]
#N30 TAAAATGAAG ACAAGTCTTT GTAGTACAAT CAATACACTG GTCTTGTAAA CCAGAAAAGG AGAATAGCCA ATCTCCCTAA [320] #N123 ... ... ... ... ... ... ... ... [320]
#N30 GACTCAAGGA AGAAGCCATA GCCTCACTAT CAGCACCCAA AGCTGAAATT CTATTTAAAC TATTCCCTGA ACCACTATTA [400] #N123 ... ... ... ... ... ... ... ... [400]
#N30 ACCACATCTA TTAATATACC CCCAAAAATA TTAAGAGCCT CCCCAGTATT AAATTTACTA AAAATTTCAA ATATACAACA [480] #N123 ... ... ... ... ... ... ... ... [480]
#N30 CAAACTTCCC ACTCCACAAG CTTACAGACA TGCCAACAAC CCACACGTAT AAAAACATCC CAATCCTAAC CCAACTTAGA [560] #N123 ... ... ... ... ... ... ... ... [560]
#N30 TACCCACACA AACGCCAACA CCACACAATG TTACGCGTAT GCAAGTACAT TACACCGCTC GCCTACACGC AAATACATTT [640] #N123 ... ... ... ... ... ... ...A. ... [640]
#N30 ACTAACAT [648] #N123 ... [648]
DdeI
MboI
AluI
DdeI
AluI
DdeI
DdeI
5
Tabel 2 Rata-rata jarak genetik antar populasi kambing di berbagai negara Negara
Negara
Indonesia Laos Pakistan Cina Unknown* Jepang
Indonesia [0.004] [0.007] [0.006] [0.008] [0.007]
Laos 0.006 [0.007] [0.007] [0.008] [0.008]
Pakistan 0.017 0.020 [0.003] [0.004] [0.002]
Cina 0.018 0.020 0.012 [0.004] [0.002]
Unknown* 0.019 0.021 0.010 0.010 [0.003]
Jepang 0.015 0.017 0.006 0.007 0.004
Keterangan : Jarak genetik : di bawah diagonal, Standar deviasi : di atas diagonal *: sekuen Daerah Pengendali yang tersedia di GenBank tanpa keterangan asal sampel
Gambar 3 Hasil rekonstruksi pohon filogeni dengan metode Neighbor Joining (bootstrap 250x). Kambing Gembrong dari Bali
mengelompok dengan kambing dari Laos dan Cina pada semua metode konstruksi pohon filogeni, sedangkan kambing dari Sulawesi Selatan berada pada posisi pangkal dari kelompok tersebut (Gambar 3). Pada posisi cabang yang lebih ke pangkal bisa ditemukan dua individu kambing dari Cina dengan laju mutasi yang sangat tinggi. Hal ini mengindikasikan kambing-kambing tersebut telah mengalami tekanan seleksi (oleh manusia) yang sangat besar.
PEMBAHASAN
Hasil PCR-RFLP yang sebagian besar monomorfik menunjukkan bahwa ternak kambing yang ada di Indonesia kemungkinan besar berasal dari induk betina yang sama. Yaitu, kambing awal yang masuk ke Indonesia sekitar tahun 1700-an. Selain itu, bisa disebutkan bahwa pola migrasi kambing di Indonesia berdasarkan kambing jantan.
Pada umumnya, untuk meningkatkan kualitas ternak (upgrading), para petani mendatangkan pejantan-pejantan unggul dari luar wilayah-nya. Dalam proses itu, induk-induk betina secara turun-temurun sejak awal masuknya tetap berada di wilayahnya. Hasil pemotongan enzim DdeI dan MboI menghasilkan pola polimorfik hanya pada empat sampel yang berasal dari Marica Sulawesi Selatan, hal ini kemungkinan disebabkan adanya percampur-an dengpercampur-an kambing Ettawah ypercampur-ang masuk ke Indonesia sekitar tahun 1900-an.
6
basa di antara kedua sampel. Sampel N123 terjadi perubahan basa G-A yang menyebabkan enzim restriksi DdeI tidak mengenali situs ini.
Daerah Pengendali digunakan sebagai dasar analisis keragaman pada penelitian ini karena banyak dilaporkan bahwa daerah ini dapat digunakan sebagai marker dalam analisis keragaman, seperti pada paus (Hoelzel 1991), rusa (Randi et al. 1998), domba (Wu et al. 2003), kerbau (Kierstein 2004), juga pada kambing (Luikart et al. 2001, Joshi et al. 2004, Meadows et al. 2005). Daerah Pengendali berukuran ±1000 pb sebagai tempat inisiasi replikasi dan transkripsi genom mitokondria (Avise 1994). Daerah ini merupakan bagian dari genom mitokondria yang tidak menyandikan protein (non-coding sequence), sehingga tingkat mutasi pada daerah ini relatif tinggi.
Hasil rekonstruksi pohon filogeni menunjukkan bangsa kambing ternak lokal di seluruh dunia berkerabat dekat, karena rata-rata jarak genetik atau tingkat kesamaan gen sebesar 98,8 % (Tabel 2). Hal ini berarti kambing-kambing ternak lokal yang ada di seluruh dunia berasal dari satu induk. Luikart et al. (2001) menulis bahwa kambing me-miliki struktur filogeografik yang lemah, keragaman mtDNA pada kambing hanya sekitar 10 % di beberapa benua di dunia lama, sedangkan sapi mencapai lebih dari 50 %. Hal ini memperlihatkan bahwa kambing dipindah-pindahkan secara ekstensif dan sebagai bukti bahwa kambing merupakan hewan yang penting dalam sejarah migrasi dan perdagang-an yperdagang-ang dilakukperdagang-an oleh mperdagang-anusia. Kambing termasuk salah satu hewan yang pertama kali diternakkan, yaitu sekitar 10000 – 6000 tahun sebelum masehi, banyak penulis menulis bahwa tempat awal domestikasi kambing di daerah pegunungan Asia Barat atau dikenal dengan Fertile Crescent (Devandra & McLeroy 1982; Devandra & Burns 1994; Zeder & Hesse 2000; Ensminger 2002). Daerah yang disebut sebagai Fertile Crescent merupakan pusat domestikasi sebagian besar hewan dan tanaman pertanian yang ada saat ini (Zedder & Hesse 2000). Para ilmuan percaya kambing Benzoar (Capra aegagrus) merupakan progenitor bagi kambing-kambing ternak lokal saat ini, dengan bukti banyak ditemukan fosil di situs-situs arkeologi di daerah Asia Barat (Pegler 1965; Devandra & McLeroy 1982; Devandra & Burns 1994; MacHugh & Bradley 2001; Ensminger 2002). Luikart et al. (2001), yang mempelajari kekerabatan kambing-kambing di dunia lama
berdasarkan keragaman Daerah Pengendali, menulis bahwa Capra hircus memiliki kekerabatan yang tinggi dengan Capra aegagrus (rata-rata 61,3 substitusi).
Kambing ternak lokal (Capra hircus) terdapat di seluruh dunia, khususnya di daerah tropis (Ensminger 2002), karena kambing ini menyukai daerah tropis yang kering (Devendra & McLeroy 1982). Kambing di Indonesia dikenal ada beberapa bangsa, antara lain kambing Kacang, Gembrong, Costa, dan Peranakan Ettawah. Sampel yang digunakan pada penelitian ini termasuk ke dalam bangsa kambing Gembrong, untuk sampel yang berasal dari Bali, dan bangsa kambing Kacang, seperti sampel yang berasal dari Sumatera Utara, Tapanuli, Samosir, dan Sulawesi Selatan. Walaupun tidak menutup kemungkinan adanya percampuran dengan kambing Ettawah yang berasal dari India.
Kambing ternak lokal di Indonesia, sebagian besar digunakan sebagai ternak penghasil daging atau susu. Kambing Kacang memiliki ciri rambut pendek yang berwarna putih, hitam, coklat, atau kombinasi ketiga-nya, hewan jantan dan betina memiliki tanduk, telinga pendek dan tegak, pada jantan selalu terdapat janggut, sedangkan betina jarang ditemukan adanya janggut. Hewan dewasa berukuran panjang 47-55 cm, tinggi pundak 55,3-55,7 cm, mempunyai berat 20 kg (betina) dan 25 kg (jantan). Kambing Gembrong terdapat di daerah kawasan timur Pulau Bali. Ciri khas kambing ini adalah memiliki rambut yang panjang, dengan panjang rambut berkisar 15-25 cm. Rambut panjang ini terdapat pada jantan, sedangkan betina hanya berkisar 2-3 cm. Rambut yang panjang ini biasa dipergunakan oleh nelayan setempat untuk membuat umpan (Devandra & Burns 1994). Kambing Gembrong secara umum memiliki ukuran tubuh yang lebih besar daripada kambing Kacang (http://peternakan.litbang.deptan.go.id).
7
SIMPULAN
Kambing ternak lokal di beberapa daerah di Indonesia mempunyai situs restriksi dari 5 enzim yang seragam (monomorfik), kecuali empat individu kambing dari Marica. Hal ini dapat diduga bahwa kambing-kambing ternak lokal di Indonesia berasal dari induk yang sama, dengan melihat sifat dari mtDNA. Hasil alignment ternyata memperkuat hipotesis awal, bahwa sebagian basar kambing yang ada di dunia berasal dari induk yang sama.
SARAN
Berkaitan dengan seragamnya kambing-kambing ternak di dunia dalam hal mtDNA menyebabkan produk metabolisme seluler pada kambing-kambing ini juga sama, sehingga untuk meningkatkan kualitas ternak, peternak dari Indonesia dapat mendatangkan kambing ternak unggul dari luar negeri. Penelitian lanjutan dapat dilakukan dengan memperpanjang hasil sekuensing, sehingga seluruh sekuen dari Daerah Pengendali dapat terpetakan atau difokuskan pada keragaman dari garis paternal, yaitu dengan kromosom Y.
DAFTAR PUSTAKA
Avise JC.1994. Molecular Markers, Natural History, and Evolution. New York: Chapman & Hall.
Campbell NA, Reece JB, Mitchell LJ. 2002. Biologi. Lestari R et al., penerjemah; Amalia, Simarmata L, Hardani HW, editor. Jakarta: Erlangga. Terjemahan dari: Biology.
Devandra C, Burns. 1994. Produksi Kambing di daerah Tropis. Putra H, penerjemah. Bandung: Penerbit ITB. Terjemahan dari: Goat Production in the Tropics.
Devandra C, McLeroy GB. 1982. Goat and Sheep Production in the Tropics. Inggris: Longman Group,Ltd.
Ensminger ME. 2002. Sheep and Goat Science. Ed ke-6. Illinois: Interstate Publishers,Inc.
Hiendleder S, Lewalski H, Wassmuth R, Janke A. 1998. The complete mitochondrial DNA sequence of the domestic sheep (Ovis aries) and comparison with the other major ovine haplotype. J. Mol. Evol. 47:441-448. Hoelzel AR, Hancock JM, Dover GA. 1991.
Evolution of the cetacean mitochondrial
d-loop region. Mol. Biol. Evol. 8(3):475-493.
Joshi MB et al. 2004. Phylogeography and origin of indian domestic goat. Mol. Biol. Evol. 21(3):454-462.
Kierstein G et al. 2004. Analysis of mitochondrial d-loop region casts new light on domestic water buffalo (Bubalus bubalis) phylogeny. J. Mol. Phyl. Evol. 30:308-324.
Krettek A, Gullberg A, Arnason U. 1995. Sequence analysis of the complete mitochondrial DNA molecule of the hedgehog, Erinaeus eropaeus, and the phylogenetic of the lipotyphla. J. Mol. Evol 41:952-957.
Kumar S, Tamura K, Nei M. 2004. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Briefings in Bioinformatics 5:150-163.
Luikart G et al. 2001. Multiple meternal origins and weak phylogeographic structure in domestic goats. Proc. Nact. Acad. Sci 98(10):5927-5932.
MacHugh DE, Bradley DG. 2001. Livestock genetic origins: goat buck the trend. Proc. Nact. Acad. Sci 98(10):5382-5384.
Meadows JRS et al. 2005. Mitochondrial sequence reveals high levels of gene flow between breeds of domestic sheep from asia and Europe. J. Heredity: 96(5):494-501.
Pegler HSH. 1965. The Book of the Goat. Inggris: Billing and Sons,Ltd.
Randi E, Pierpaoli M, Danilkin A. 1998. Mitochondrial DNA polymorphism in population of siberian and european roe deer (Capreolus pygargus and C. capreolus). Heredity 80:429-437.
Ursing BM, Arnason U. 1998. The complete mitochondrial DNA sequence of the pig (Sus scrofa). J. Mol. Evol. 47:302-306. Valverde JR, Batuecas B, Maratilla C, Marco
R, Ganesse R. 1994. The complete mitochondrial DNA sequence of the crustacean Artemia franciscana. J. Mol. Evol 39:400-408.
Wu CH, Zhang YP, Bunch TD, Wang S, Wang W. 2003. Mitochondrial control region sequence variation within the argali wild sheep (Ovis ammon): evolution and conservation relevance. Mammalia 67:109-118.
8
9
Lampiran 1 Peta Situs Restriksi Daerah Pengendali mtDNA
MboI AluI HaeIII
DdeI
MboI MboI
DdeI AluI
BamHI
MboI HaeIII
HaeIII DdeI AluI
HaeIII
DdeI
1080 280
165
1370 1410
1440 1460
1620 1400
1260
1300 1130
1200 955
1070
1100
1090 700
750 815 710 800
MboI DdeI
MboI DdeI
MboI
DdeI
9
Haplotipe 1
10
Lampiran 2 Gambar pola restriksi pada gel poliakrilamid
AluI 280 800
320 310 M
M
M
750
300 250
80
700
600
160 320
150
DdeI
A B
HaeIII 710
500
150
60 350
BamHI 600 M
1100
M
250 340
300 800
570
175
140 650
165 B
A
ii
ABSTRAK
NICO HIMAWAN. Keragaman Genetik Kambing Lokal Berdasarkan Genom Mitokondria. Dibimbing oleh ACHMAD FARAJALLAH dan BESS TIESNAMURTI.
Kambing (Capra hircus) dimasukkan pertama kali ke Indonesia sebagai hewan ternak sekitar tahun 1700-an, yang kemudian berkembang manjadi ternak lokal. Peningkatan mutu ternak kambing sebagian besar dilakukan dengan cara mengawinkan ternak betina dengan jantan unggul yang didatangkan dari daerah lain. Penelitian ini dilakukan untuk mengetahui keragaman genetik kambing ternak lokal berdasarkan Daerah Pengendali DNA mitokondria. Sampel yang digunakan adalah kambing ternak lokal yang berasal dari Sumatera Utara, Muara-Tapanuli Utara, Samosir, Marica-Sulawesi Selatan, dan Gembrong-Bali. Amplifikasi DNA mitokondria secara in vitro dilakukan dengan metode Polymerase Chain Rection (PCR). Produk amplifikasi yang dihasilkan sebesar 1700 pasang basa. Produk amplifikasi tersebut dipotong dengan lima enzim restriksi, yaitu MboI, DdeI, AluI, HaeIII, dan BamHI. Dari 102 sampel yang dianalisis, ditemukan dua haplotipe. Haplotipe 1 terdapat pada seluruh wilayah, sedangkan haplotipe 2 hanya terdapat pada empat sampel yang berasal dari Marica Sulawesi Selatan. Hasil sekuensing yang telah dialignment dengan beberapa individu kambing yang terdapat pada GenBank, menunjukkan bahwa kambing-kambing ternak lokal di dunia memiliki kekerabatan yang dekat (98,8 %) dan berasal dari induk yang sama.
ABSTRACT
NICO HIMAWAN. Genetic Diversity of Native Goat Based on Mitochondrial Genome. Supervised by ACHMAD FARAJALLAH and BESS TIESNAMURTI.
7
SIMPULAN
Kambing ternak lokal di beberapa daerah di Indonesia mempunyai situs restriksi dari 5 enzim yang seragam (monomorfik), kecuali empat individu kambing dari Marica. Hal ini dapat diduga bahwa kambing-kambing ternak lokal di Indonesia berasal dari induk yang sama, dengan melihat sifat dari mtDNA. Hasil alignment ternyata memperkuat hipotesis awal, bahwa sebagian basar kambing yang ada di dunia berasal dari induk yang sama.
SARAN
Berkaitan dengan seragamnya kambing-kambing ternak di dunia dalam hal mtDNA menyebabkan produk metabolisme seluler pada kambing-kambing ini juga sama, sehingga untuk meningkatkan kualitas ternak, peternak dari Indonesia dapat mendatangkan kambing ternak unggul dari luar negeri. Penelitian lanjutan dapat dilakukan dengan memperpanjang hasil sekuensing, sehingga seluruh sekuen dari Daerah Pengendali dapat terpetakan atau difokuskan pada keragaman dari garis paternal, yaitu dengan kromosom Y.
DAFTAR PUSTAKA
Avise JC.1994. Molecular Markers, Natural History, and Evolution. New York: Chapman & Hall.
Campbell NA, Reece JB, Mitchell LJ. 2002. Biologi. Lestari R et al., penerjemah; Amalia, Simarmata L, Hardani HW, editor. Jakarta: Erlangga. Terjemahan dari: Biology.
Devandra C, Burns. 1994. Produksi Kambing di daerah Tropis. Putra H, penerjemah. Bandung: Penerbit ITB. Terjemahan dari: Goat Production in the Tropics.
Devandra C, McLeroy GB. 1982. Goat and Sheep Production in the Tropics. Inggris: Longman Group,Ltd.
Ensminger ME. 2002. Sheep and Goat Science. Ed ke-6. Illinois: Interstate Publishers,Inc.
Hiendleder S, Lewalski H, Wassmuth R, Janke A. 1998. The complete mitochondrial DNA sequence of the domestic sheep (Ovis aries) and comparison with the other major ovine haplotype. J. Mol. Evol. 47:441-448. Hoelzel AR, Hancock JM, Dover GA. 1991.
Evolution of the cetacean mitochondrial
d-loop region. Mol. Biol. Evol. 8(3):475-493.
Joshi MB et al. 2004. Phylogeography and origin of indian domestic goat. Mol. Biol. Evol. 21(3):454-462.
Kierstein G et al. 2004. Analysis of mitochondrial d-loop region casts new light on domestic water buffalo (Bubalus bubalis) phylogeny. J. Mol. Phyl. Evol. 30:308-324.
Krettek A, Gullberg A, Arnason U. 1995. Sequence analysis of the complete mitochondrial DNA molecule of the hedgehog, Erinaeus eropaeus, and the phylogenetic of the lipotyphla. J. Mol. Evol 41:952-957.
Kumar S, Tamura K, Nei M. 2004. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Briefings in Bioinformatics 5:150-163.
Luikart G et al. 2001. Multiple meternal origins and weak phylogeographic structure in domestic goats. Proc. Nact. Acad. Sci 98(10):5927-5932.
MacHugh DE, Bradley DG. 2001. Livestock genetic origins: goat buck the trend. Proc. Nact. Acad. Sci 98(10):5382-5384.
Meadows JRS et al. 2005. Mitochondrial sequence reveals high levels of gene flow between breeds of domestic sheep from asia and Europe. J. Heredity: 96(5):494-501.
Pegler HSH. 1965. The Book of the Goat. Inggris: Billing and Sons,Ltd.
Randi E, Pierpaoli M, Danilkin A. 1998. Mitochondrial DNA polymorphism in population of siberian and european roe deer (Capreolus pygargus and C. capreolus). Heredity 80:429-437.
Ursing BM, Arnason U. 1998. The complete mitochondrial DNA sequence of the pig (Sus scrofa). J. Mol. Evol. 47:302-306. Valverde JR, Batuecas B, Maratilla C, Marco
R, Ganesse R. 1994. The complete mitochondrial DNA sequence of the crustacean Artemia franciscana. J. Mol. Evol 39:400-408.
Wu CH, Zhang YP, Bunch TD, Wang S, Wang W. 2003. Mitochondrial control region sequence variation within the argali wild sheep (Ovis ammon): evolution and conservation relevance. Mammalia 67:109-118.
8
9
Lampiran 1 Peta Situs Restriksi Daerah Pengendali mtDNA
MboI AluI HaeIII
DdeI
MboI MboI
DdeI AluI
BamHI
MboI HaeIII
HaeIII DdeI AluI
HaeIII
DdeI
1080 280
165
1370 1410
1440 1460
1620 1400
1260
1300 1130
1200 955
1070
1100
1090 700
750 815 710 800
MboI DdeI
MboI DdeI
MboI
DdeI
9
Haplotipe 1
10
Lampiran 2 Gambar pola restriksi pada gel poliakrilamid
AluI 280 800
320 310 M
M
M
750
300 250
80
700
600
160 320
150
DdeI
A B
HaeIII 710
500
150
60 350
BamHI 600 M
1100
M
250 340
300 800
570
175
140 650
165 B
A