iv
ABSTRACT
RANNY DWITA OLIVIA. Genetic Diversity of Sengon Population (Paraserianthes falcataria (L.) Nielsen) in Community Forest in Java based on RAPD Marker. Under supervision of ULFAH JUNIARTI SIREGAR.
Paraserianthes falcataria (L.) Nielsen, with local name “sengon” has been planted widely as community forest in Java because of its easy and fast growing nature having multipurposes from its leaves, stems and roots. As sengon has been grown in monoculture, the plantation often has problems such as, susceptible to stem borer pest, Xystrocera festiva, damping off, and karat puru disease. Therefore, resistant sengon is required, which is obtained from breeding programs. A good breeding program requires high genetic diversity. The objective of this research is to study the genetic diversity of sengon populations in some community forests in Java.
Sengon leaf samples were taken from 9 populations in Java namely: from Cianjur, Sukabumi, Garut, Kuningan, Tasikmalaya, and Subang (West Java), Wonosobo (Central Java), Kediri and Lumajang (East Java). From each population 25 trees were randomly sampled. Sengon’s DNA was extracted using CTAB method and GenElute Plant Genomic DNA miniprep Kit from SIGMA. The DNA samples were amplified using 5 random primers, i.e. OPA-2, OPA-3, OPB-10, OPY-5 and OPU-5. Generated data was then analyzed using POPGENE, NTSYS and GenAlex program. Genetic diversity parameters measured consist of heterozygosity (He), percentage of polymorphic locus (PLP), the number of observed alleles (na), the number of effective alleles (ne), and genetic distance.
The value of expected heterozygosity (He) of whole population was 0.2349 indicated that sengon populations in Java have high genetic diversity. Most of the genetic variation (82%) was found within the population, while the differences among populations was only 18%. Kediri and Garut population had longest genetic distance (0.0857), and the closest genetic distance (0.0123) was found between Tasikmalaya and Lumajang population. Dendrogram based on the genetic distances showed that the distribution pattern of sengon in Java had occurred randomly, because some populations belonged to the same area (e.g. West Java) appeared to have close genetic relationship with population from other provinces.
BAB 1
PENDAHULUAN
1.1. Latar Belakang
Indonesia merupakan negara kepulauan yang memiliki sumber daya alam melimpah dan tanah yang subur sehingga cocok sebagai tempat tumbuh dari berbagai jenis tanaman. Keadaan ini, mengantarkan Indonesia sebagai negara yang memiliki tingkat biodiversitas yang tinggi. Indonesia berada di daerah yang beriklim tropis, yang memiliki hutan tropis ketiga terbesar di dunia. Dari hutan tersebut, kita dapat mengambil manfaatnya baik dari segi kayu maupun non kayu.
Paraserianthes falcataria (L) Nielsen yang dikenal dengan nama lokal sengon banyak ditanam pada hutan rakyat di Jawa, karena tergolong pohon yang cepat tumbuh (Santoso 1992), multiguna baik itu daun, batang, dan sistem perakaran. Akan tetapi sengon ditanam secara monokultur sehingga tanaman ini mempunyai masalah yaitu, mudah terserang hama dan penyakit seperti hama penggerek batang Xystrocera festiva, damping off, dan karat puru. Untuk itu diperlukan sengon unggul yang dihasilkan dari program pemuliaan. Untuk melaksanakan program pemuliaan, dibutuhkan keragaman genetik yang tinggi.
Keragaman genetik yang tinggi pada suatu populasi menunjukkan potensi populasi tersebut untuk beradaptasi pada berbagai kondisi lingkungan. Penanda genetik dari analisis DNA digunakan sebagai alat bantu dalam mempelajari keragaman genetik dan mengidentifikasi genotipe suatu sampel. Informasi yang dihasilkan berguna dalam penentuan hubungan kekerabatan dan filogenetik populasi/individu setelah terjadi evolusi karena pengaruh waktu dan tempat. Dalam penelitian ini digunakan penanda molekuler karena penanda ini bersifat stabil dan tidak terpengaruh lingkungan. Saat ini telah berkembang berbagai jenis penanda molekuler diantaranya adalah isoenzim, RFLP, SSR, AFLP, dan RAPD (Random Amplified Polymorphic DNA).
2 diantaranya dapat dengan cepat mendeteksi polimorfisme fragmen DNA, relatif mudah dilakukan dan hanya memerlukan sejumlah kecil DNA. Penggunaan penanda RAPD memungkinkan dapat mendeteksi polimorfisme fragmen DNA yang diseleksi dengan menggunakan satu primer bersifat acak.
RAPD lebih bersifat sederhana, hal ini dikarenakan teknik RAPD tidak memerlukan informasi awal mengenai urutan DNA genom organisme yang diuji maupun tidak memerlukan probe DNA yang spesifik (Williams et al. 1990). Akan tetapi walaupun RAPD relatif cepat, murah, dan mudah untuk dilaksanakan, konsistensi hasil PCR menjadi perhatian sejak dipublikasikannya teknik ini. Primer RAPD dapat tidak cocok secara sempurna pada urutan penempelan primer, akibatnya amplifikasi pada beberapa siklus mungkin tidak terjadi, sehingga band tetap samar atau bahkan amplifikasi tidak terjadi jika primer tidak berhasil menempel pada DNA cetakan (Lamboy 1994, diacu dalam Pharmawati 2009).
Menurut Padmalatha dan Prasad (2006) diacu dalam Pharmawati (2009), tidak menempelnya primer pada DNA secara sempurna, diakibatkan karena tidak tepatnya konsentrasi komponen─komponen PCR-RAPD. Disamping itu, kualitas DNA juga dapat mempengaruhi. Adanya kandungan polifenol dan metabolit sekunder lain seperti tannin, terpen dapat menurunkan kemurnian DNA dan menghambat penempelan primer.
1.2. Tujuan Penelitian
Tujuan dari penelitian ini adalah untuk mengetahui keragaman genetik di dalam dan antar populasi tanaman Sengon (P. falcataria) pada beberapa hutan rakyat di Jawa.
1.3. Manfaat Penelitian
BAB II
TINJAUAN PUSTAKA
2.1. Deskripsi Sengon (Paraserianthes falcataria (L.) Nielsen)
2.1.1. Taksonomi dan Tata Nama
Taksonomi dari sengon adalah sebagai berikut: Kingdom : Plantae
Divisi : Spermatophyta Sub divisi : Angiospermae Kelas : Dicotyledonae Bangsa : Fabales
Suku : Leguminoceae Sub famili : Mimosoidae Marga : Paraserianthes
Jenis : falcataria (L.) Nielsen
2.1.2. Ekologi dan Penyebaran Alami
Berdasarkan sejarah, sengon adalah jenis asli dari kepulauan sebelah Timur Indonesia yakni di sekitar Maluku dan Irian Jaya, merupakan jenis pionir, terutama terdapat di hutan hujan dataran rendah. Tahun 1870-an pohon ini menyebar keseluruh kawasan Asia Tenggara mulai dari Myanmar sampai Filipina. Habitat alami pohon sengon ditemukan di kepulauan Maluku, Papua Nugini, Kepulauan Solomon, dan Bismark. Tahun 1871 tanaman ini mulai dimasukkan ke Jawa tepatnya ke Kebun Raya Bogor (Atmosuseno 1998). Sejak itulah sengon mulai ditanam diberbagai tempat di Jawa sebagai tanaman pelindung perkebunan.
Sengon dapat tumbuh dengan kisaran suhu 20─33oC dalam pertumbuhannya. Namun demikian, suhu optimum yang diperlukan oleh sengon berkisar antara 22─29oC. Sengon tumbuh mulai dari pantai hingga ketinggian 1600 mdpl, dengan ketinggian optimum 0─800 mdpl. Sengon dapat beradaptasi dengan iklim monsoon dan lembab dengan curah hujan 200─2700 mm/tahun dengan bulan kering sampai 4 bulan (Atmosuseno 1998).
4 pengamatan di lapangan, tanaman sengon dapat tumbuh baik pada tanah regosol, aluvial, dan latosol. Tanah-tanah tersebut bertekstur lempung berpasir atau lempung berdebu dengan kemasaman tanah sekitar pH 6─7 (Santoso 1992). Sengon merupakan salah satu spesies yang paling cepat tumbuh di dunia, mampu tumbuh 8 m/tahun dalam tahun pertama penanaman.
2.1.3. Ciri Morfologi
Tinggi pohon sengon dapat mencapai 40 m dengan tinggi bebas cabang 20 m. Pohon sengon memiliki ciri-ciri, yaitu tidak berbanir, kulit licin, berwarna kelabu muda, bulat agak lurus. Diameter pohon dewasa bisa mencapai 100 cm atau lebih. Tajuk berbentuk perisai, jarang selalu hijau. Daun majemuk, panjang dapat mencapai 40 cm, terdiri dari 15─25 helai daun. Daunnya kecil-kecil dan mudah rontok. Bunga tersusun dalam bentuk malai. Bunganya kecil sekitar 0,5─1 cm, berwarna putih kekuning-kuningan dan sedikit berbulu. Setiap kuntum bunga yang mekar berisi bunga jantan dan betina. Adapun cara penyerbukannya dibantu dengan perantaraan angin dan serangga.
Buah sengon berbentuk polong, pipih, tipis, dan panjangnya sekitar 6─12 cm. Setiap polong buah berisi 15─30 biji. Biji tersebut biasanya terlepas dari polongnya yang terbuka bila terlalu masak. Bentuk biji dari sengon ini mirip perisai kecil, dan jika sudah tua biji tersebut berwarna cokelat kehitam-hitaman, agak keras dan berlilin (Santoso 1992).
Perakaran sengon relatif menguntungkan dibandingkan akar pohon lainnya. Akar tunggangnya cukup kuat menembus ke dalam tanah. Semakin besar pohonnya semakin dalam akar tunggangnya menembus ke dalam tanah. Sementara itu, akar rambut dari pohon sengon tidak terlalu besar, tidak rimbun, dan tidak menonjol ke permukaan tanah. akar rambut sengon justru dimanfaatkan oleh pohon induk untuk menyimpan nitrogen. Maka dari itu, tanah disekitar pohon sengon akan menjadi subur (Santoso 1992).
2.1.4. Kegunaan dan Manfaat
5 industri. Adapun pohon sengon memiliki karakteristik masa tebang pohon yang relatif cepat, teknik budi daya mudah diaplikasikan, dapat tumbuh di berbagai jenis tanah, kayu serba guna, dapat membantu menyuburkan tanah dan memperbaiki kualitas lahan (Siregar 2008).
Kayu sengon banyak diusahakan untuk berbagai keperluan dalam bentuk kayu olahan berupa papan-papan dengan ukuran tertentu sebagai bahan baku pembuat peti, papan penyekat, industri korek api, pensil, papan partikel, serta bahan baku industri pulp dan paper.
Daun sengon merupakan pakan ternak yang sangat baik dan mengandung protein tinggi. Selain sebagai pakan ternak, daun sengon yang berguguran akan menjadi pupuk hijau yang baik bagi tanah dan tanaman disekitarnya. Sementara itu, tajuk pohonnya yang rindang dapat dimanfaatkan sebagai pohon penaung di beberapa areal perkebunan (Siregar 2008). Kulit kayu sengon yang memiliki tannin dapat digunakan sebagai penyamak. Selain itu, sengon sebagai tanaman hutan juga memiliki jasa ekologis, diantaranya tegakan murninya yang dapat menahan erosi tanah dan air, serta berfungsi sebagai naungan pada penanaman campuran dengan teh, kopi dan cokelat (ICRAF 2006, diacu dalam Dwiyanti 2009).
2.2. Dasar─Dasar Genetika Molekuler
2.2.1. PCR (Polymerase Chain Reaction)
Kary B. Mullis mengembangkan Polymerase Chain Reaction (PCR) pada tahun 1983, untuk merevolusi metodologi dari biologi molekuler. PCR yaitu suatu proses yang didasarkan pada reaksi enzimatik in-vitro dalam amplifikasi DNA pada target tertentu dengan cara mensintesis molekul DNA baru yang berkomplemen dengan molekul DNA target dengan bantuan enzim dan oligonukleotida sebagai primer dalam suatu thermocycler.
6 Dalam uji PCR, terdapat tiga langkah temperatur terkendali yang dapat dilihat, dan siklus dari proses PCR ini dapat diulang berkisar antara 25─50 siklus. Menurut Bernard (1998) diacu dalam Siregar et al. (2008), PCR merupakan suatu teknik untuk memperbanyak potongan DNA spesifik. Ada empat komponen utama yang dibutuhkan untuk melakukan proses PCR yaitu: (i) DNA target, (ii) primer, (iii) DNA polymerase dan (iv) dNTP. Menurut Weising et al. (2005) ada lima komponen penting yang dibutuhkan untuk PCR yaitu: (i) Buffer, yang biasanya terdiri dari Tris-HCl, KCl dan MgCl2, (ii) DNA polymerase, (iii) dNTP,
(iv) primer dan (v) DNA target.
Selektivitas reaksi dalam proses PCR ini ditentukan oleh pemilihan primer. Primer yang terdiri dari potongan kecil DNA yang dihasilkan secara buatan dimana biasanya terdiri antara 10─25 nukleotida (oligonukleotida) melengkapi urutan template yang mengapit wilayah yang ditargetkan. Untuk memperhitungkan amplifikasi yang eksponensial, primer harus menempel pada arah yang berlawanan. Amplifikasi yang paling efisien untuk mengikat dua primer tidak lebih dari berkisar 4 kb. Namun, produk amplifikasi lebih dari 10 kb dapat diperoleh pada kondisi yang optimal (Weising et al. 2005).
7 2.2.2. Penanda Genetik
Menurut Siregar dan Kusmana (2002), analisis keragaman suatu tanaman dapat dilakukan dengan melakukan pengukuran terhadap performa fenotipe atau melalui penanda tertentu. Sifat fenotipe suatu tanaman dipengaruhi oleh dua faktor yaitu genotipe dan lingkungan. Genotipe adalah informasi genetik yang mengontrol fenotipe yang diamati. Fenotipe suatu tanaman akan berbeda dengan tanaman yang lain.
Penanda genetik merupakan alat terpenting untuk mempelajari sistem genetik pada banyak organisme. Penanda genetik banyak diterapkan pada program pemuliaan dan konservasi sumberdaya genetik hewan dan tumbuhan. Adapun kegunaan dari penanda genetik ini antara lain: identifikasi klon-klon, identifikasi hibrid, pengukuran keragaman genetik antar dan dalam populasi, pengamatan sistem reproduksi (meliputi sistem perkawinan dan aliran gen), bukti selektifitas (berkaitan dengan praktek pengelolaan hutan atau perubahan lingkungan) dan identifikasi lokus sifat kuantitatif atau Quantitative Train Loci (QTLs) (Finkeldey 2005).
Menurut Karsinah et al. (2002), penanda genetik merupakan teknik yang efektif dalam analisis genetik dan telah diaplikasikan secara luas dalam pemuliaan tanaman.
RAPD dihasilkan melalui proses amplifikasi DNA secara in-vitro dengan Polymerase Chain Reaction (PCR) yang dikembangkan oleh Williams et al. 1990. Menurut Bernard (1998) diacu dalam Siregar et al. (2008), PCR merupakan suatu teknik untuk memperbanyak potongan DNA spesifik. Adapun menurut Finkeldey (2005), PCR adalah suatu metode untuk menggandakan atau mengamplifikasi DNA yang diisolasi pada sebuah tabung reaksi kecil dengan melalui replikasi berulang. Empat komponen utama yang dibutuhkan untuk melakukan proses yaitu: DNA target, primer, DNA polymerase, dan dNTP (Bernard 1998 dalam Siregar et al. 2008). Titik awal dari reaksi (primer) adalah oligonukleotida, yakni potongan kecil DNA yang dihasilkan secara buatan (biasanya terdiri antara 10─25 nukleotida). Sekuensi basa dari primer dapat dipilih secara bebas.
8 rendah (0,5–50 ng), tidak membutuhkan radioisotip, dan tidak terlalu membutuhkan keahlian untuk pelaksanaannya.
Teknik RAPD akan mendeteksi polimorfisme DNA yang diakibatkan oleh tidak munculnya amplifikasi pada suatu lokus. Hal ini disebabkan oleh perbedaan urutan pada titik pertemuan primer. Ini mengakibatkan primer tidak dapat menempel pada bagian tersebut sehingga tidak terjadi amplifikasi. Oleh karena itu, hanya ada dua kemungkinan alel pada penanda RAPD, yaitu timbulnya pita pendek sebagai hasil amplifikasi atau tidak adanya pita karena tidak adanya amplifikasi. Penanda yang demikian disebut dominant marker. Pita yang berbeda ukurannya dari suatu primer RAPD diasumsikan berasal dari lokus yang berbeda. Metode RAPD ini mampu mendeteksi sekuen nukleotida dengan hanya menggunakan suatu primer atau nukleotida yang disusun secara acak. Dengan teknik ini keragaman genetik suatu populasi dapat dianalisis.
Secara teoritis jumlah fragmen yang diamplifikasi tergantung pada panjang primer dan ukuran genom target. Pada kebanyakan tanaman, primer dengan panjang antara 9─10 nukleotida dapat menghasilkan antara 2─10 produk amplifikasi.
2.3. Keragaman Genetik
Keragaman genetik adalah suatu besaran yang mengukur variasi fenotipe yang disebabkan oleh faktor-faktor genetik (Finkeldey 2005). Keragaman genetik juga merupakan perbedaan gen individu dalam suatu populasi dan berhubungan dengan kemampuan beradaptasinya suatu individu selama proses perkembangannya. Dalam suatu individu memiliki beberapa keragaman diantaranya keragaman provenan (keragaman geografis), keragaman antar tempat tumbuh (lokal), dan keragaman antar pohon. Variasi genetik terjadi karena adanya gen-gen yang bersegresi dan berinteraksi dengan gen-gen lain. Keragaman genetik dari suatu tanaman merupakan hasil dari perkembangbiakan secara seksual. Perkembangbiakan seksual terjadi karena adanya proses reduksi jumlah kromosom diploid (2n) dan sel tetua menjadi haploid (n) dalam gamet (Crowder 2006).
BAB III
BAHAN DAN METODE
3.1. Tempat dan Waktu
Tempat penelitian analisis DNA dilakukan di Common Laboratory SEAMEO BIOTROP dan laboratorium Silvikultur Fakultas Kehutanan Institut Pertanian Bogor. Penelitian ini dilaksanakan pada bukan Februari 2011 sampai dengan Januari 2012.
3.2. Bahan dan Alat Penelitian
3.2.1. Bahan Penelitian
Bahan tanaman yang digunakan berupa daun sengon yang berasal dari tegakan yang diambil secara acak di beberapa hutan rakyat di Jawa. Adapun daerah-daerah pengambilan sampel dapat dilihat pada Tabel 1.
Tabel 1 Lokasi tempat pengambilan sampel daun sengon pada hutan rakyat di Jawa
Provinsi Kota/ Kabupaten Jumlah Sampel
Jawa Barat
Cianjur 25
Sukabumi 25
Garut 25
Subang 25
Kuningan 25
Tasikmalaya 25
Jawa Tengah Wonosobo 25
Jawa Timur Kediri 25
Lumajang 25
Total 225
11 dalam visualisasi DNA yaitu, agarose, aqudest, buffer TAE, loading dye, DNA marker, Etbr atau SYBR safe gel stain DNA. Alat-alat yang digunakan dalam penelitian ini dapat dilihat secara lengkap pada Lampiran 2.
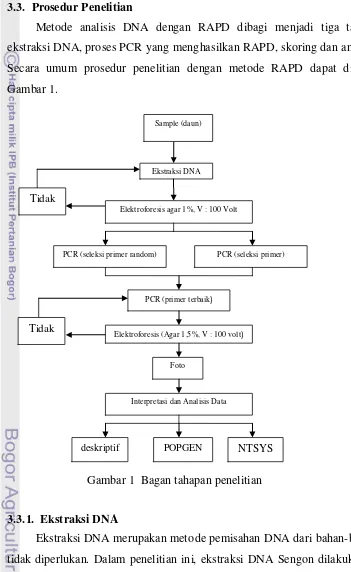
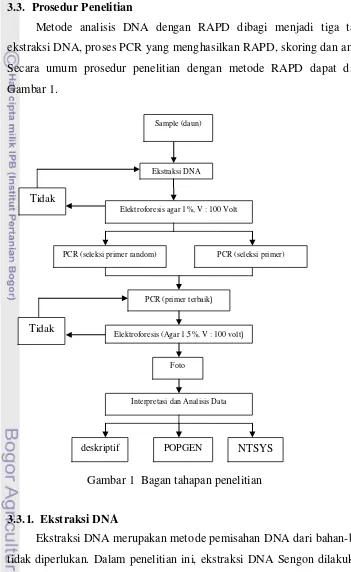
3.3. Prosedur Penelitian
Metode analisis DNA dengan RAPD dibagi menjadi tiga tahap yaitu ekstraksi DNA, proses PCR yang menghasilkan RAPD, skoring dan analisis data. Secara umum prosedur penelitian dengan metode RAPD dapat dilihat pada Gambar 1.
3.3.1. Ekstraksi DNA
Ekstraksi DNA merupakan metode pemisahan DNA dari bahan-bahan yang tidak diperlukan. Dalam penelitian ini, ekstraksi DNA Sengon dilakukan dengan dua metode, yaitu metode CTAB dan GenElute Plant Genomic DNA Miniprep Kit
Gambar 1 Bagan tahapan penelitian
Ekstraksi DNA
Interpretasi dan Analisis Data Foto
PCR (primer terbaik)
Elektroforesis (Agar 1,5%, V : 100 volt)
PCR (seleksi primer random)
Elektroforesis agar 1%, V : 100 Volt
PCR (seleksi primer) Sample (daun)
Tidak
Tidak
12 dari SIGMA. Metode CTAB menggunakan larutan buffer CTAB (Tris-HCL 1M, NaCl 5M, EDTA 0,5 M dan CTAB 10%), dipilih karena lebih murah dan mudah dilakukan (Rogers and Bendich 1994, diacu dalam Aritonang et al. 2007). Adapun langkah awal yang dilakukan dari 2 metode ini hampir sama yaitu, sampel daun Sengon 0,25─0,5 g digerus dengan bantuan nitrogen cair. Hasil gerusan dimasukkan ke dalam tube 2 ml yang telah diisi 1 ml buffer ekstrak CTAB dan 1% β-mercaptoethanol. Campuran divortex ± 2 menit, kemudian diinkubasi 650C selama 30 menit (setiap 10 menit campuran dihomogenkan dengan membolak-balikan tabung). Larutan ekstrak DNA dipurifikasi dengan menambahkan 750 µ l Chloroform, lalu campuran divortex dan disentrifugasi pada kecepatan 11.000 rpm selama 10 menit. Proses sentrifugasi dilakukan untuk memisahkan bahan-bahan kimia atau fase organik dari fase air berupa supernatan. Dari kedua fase tersebut yang digunakan untuk tahap selanjutnya adalah fase air yang berisi benang-benang nukleat. Supernatan dipipet kedalam tube 2 ml yang baru, proses purifikasi dilakukan sebanyak dua kali, hal ini bertujuan untuk memperoleh DNA yang memiliki tingkat kemurnian tinggi. DNA diendapkan dengan penambahan 1 ml isopropanol dingin. Kemudian dihomogenkan perlahan sampai terbentuk benang-benang putih. Campuran diinkubasi pada suhu -20oC selama minimal 30 menit, kemudian pelet di sentrifugasi pada kecepatan 11.000 rpm selama 10 menit. Setelah itu pelet DNA dilarutkan dengan buffer TE.
13 3.3.2. PCR (Polymerase Chain Reaction)
Proses amplifikasi DNA pada PCR membutuhkan bahan campuran yang terdiri dari buffer PCR, MgCl2, dNTP yang digunakan sebagai sumber nukleotida
pada proses PCR (gabungan dATP, dGTP, dCTP dan dTTP), Taq DNA polymerase, DNA hasil isolasi dan Nuclease Free Water (Tabel 2). Sebelum dilakukan proses amplifikasi PCR harus dilakukan pengenceran DNA tamplate dengan Nuclease Free Water. Hal ini tergantung dari tebal dan tipisnya DNA genomik hasil ekstraksi.
Mengacu pada Promega (2003), jumlah DNA yang diperlukan dalam proses PCR sangat sedikit yaitu sekitar 1 μl atau 10 ng/μl. Untuk mengetahui konsentrasi DNA hasil ekstraksi dapat ditetapkan dengan melakukan elektroforesis menggunakan gel agarose. Hasil amplifikasi DNA dengan PCR ditentukan oleh primer oligonukleotida yang digunakan.
Tabel 2 Komponen bahan untuk reaksi PCR.
No Nama Bahan 1 Sampel Reaksi
1 2 3 4 5 6 7 DNA target Primer PCR buffer MgCl2 dNTP
Taq DNA polymerase Nuclease free water
2 µl 1,5 µl 5 µl 2,5 µl 0,5 µl 0,2 µl 13,3 µl
Total Volume 25 µl
Proses PCR dilakukan dengan menggunakan primer hasil seleksi. Adapun tahapan PCR yang dilakukan dapat dilihat pada Tabel 3.
Tabel 3 Tahapan proses PCR
Tahapan Suhu Waktu Jumlah Siklus (oC) (menit)
14 3.3.3. Seleksi Primer
Primer adalah rantai pendek DNA yang dihasilkan secara buatan yang biasanya terdiri antara 10–25 nukleotida (Finkeldey 2005). Dalam teknik RAPD, umumnya primer yang digunakan berupa oligonukleotida yang memiliki panjang sebesar 10-mer yang dipilih secara acak dan minimum memiliki basa G dan C. Primer yang mempunyai panjang kurang dari 10-mer dapat digunakan tetapi akan menghasilkan produk amplifikasi yang lebih sedikit dan diperlukan metode pewarnaan yang lebih sensitif untuk mendeteksinya.
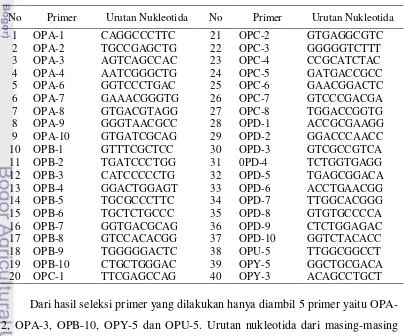
Seleksi primer dimaksudkan untuk mencari primer acak yang menghasilkan penanda polimorfik, karena tidak semua primer nukleotida dapat menghasilkan produk amplifikasi (primer positif) dan dari primer positif tidak semuanya menghasilkan fragmen DNA polimorfik. Pada kegiatan ini dilakukan seleksi terhadap primer, yaitu golongan OPO, OPY, OPA, OPB, OPC dan OPD yang diproduksi oleh Operon Technology. Urutan basa primer yang dicobakan dapat dilihat pada Tabel 4.
Tabel 4 Urutan basa nukleotida 41 primer (Operon Technology) yang dicobakan.
No Primer Urutan Nukleotida No Primer Urutan Nukleotida
1 OPA-1 CAGGCCCTTC 21 OPC-2 GTGAGGCGTC
2 OPA-2 TGCCGAGCTG 22 OPC-3 GGGGGTCTTT
3 OPA-3 AGTCAGCCAC 23 OPC-4 CCGCATCTAC
4 OPA-4 AATCGGGCTG 24 OPC-5 GATGACCGCC
5 OPA-6 GGTCCCTGAC 25 OPC-6 GAACGGACTC
6 OPA-7 GAAACGGGTG 26 OPC-7 GTCCCGACGA
7 OPA-8 GTGACGTAGG 27 OPC-8 TGGACCGGTG
8 OPA-9 GGGTAACGCC 28 OPD-1 ACCGCGAAGG
9 OPA-10 GTGATCGCAG 29 OPD-2 GGACCCAACC
10 OPB-1 GTTTCGCTCC 30 OPD-3 GTCGCCGTCA
11 OPB-2 TGATCCCTGG 31 0PD-4 TCTGGTGAGG
12 OPB-3 CATCCCCCTG 32 OPD-5 TGAGCGGACA
13 OPB-4 GGACTGGAGT 33 OPD-6 ACCTGAACGG
14 OPB-5 TGCGCCCTTC 34 OPD-7 TTGGCACGGG
15 OPB-6 TGCTCTGCCC 35 OPD-8 GTGTGCCCCA
16 OPB-7 GGTGACGCAG 36 OPD-9 CTCTGGAGAC
17 OPB-8 GTCCACACGG 37 OPD-10 GGTCTACACC
18 OPB-9 TGGGGGACTC 38 OPU-5 TTGGCGGCCT
19 OPB-10 CTGCTGGGAC 39 OPY-5 GGCTGCGACA
20 OPC-1 TTCGAGCCAG 40 OPY-3 ACAGCCTGCT
15 primer adalah OPA-2 (TGCCGAGCTG), OPA-3 (AGTCAGCCAC), OPB-10 (CTGCTGGGAC), OPY-5 (GGCTGCGACA) dan OPU-5 (TTGGCGGCCT). Hasil dari proses PCR tersebut dianalisis dengan melakukan proses elektroforesis menggunakan 1,5% gel agarose dalam larutan buffer TAE 1x dan distaining dengan menggunakan SYBR safe gel stain DNA.
3.4. Skoring Fragmen RAPD
Hasil PCR yang telah dielektroforesis dilakukan pengambilan foto dan dianalisis dengan melakukan skoring. Profil pita DNA hasil analisis RAPD diskoring dengan ada atau tidaknya hasil amplifikasi. Jika terdapat pita maka genotipe tersebut dinilai 1 dan jika tidak terdapat pita pada genotipe yang lain dinilai 0. Contoh proses skoring dapat dilihat pada Gambar 2.
Gambar 2 Cara analisis DNA dengan skoring
3.5. Data Analisis
Hasil skoring yang diperoleh bertujuan untuk memperoleh nilai untuk menghitung keragaman genetik seluruh populasi dan diferensiasi antar populasi dengan menggunakan program POPGEN 32. Pendugaan hubungan kekerabatan dilakukan berdasarkan jumlah pita polimorfik yang dimiliki bersama (Nei dan Lei 1979, diacu dalam Yunanto 2006). Analisis pengelompokan berdasarkan metode UPGMA (Unweighted Pair-Group with Aritmatic Average) dengan software NTSYS Versi 2.0. Analysis of Molecular Variance (AMOVA) berdasarkan Program GenAlEx.
Parameter variasi genetik yang diamati dalam penelitian ini adalah sebagai berikut (Finkeldey 2005):
1. Persentase Lokus Polimorfik (PLP) = x 100%
Keterangan :
16 2. Jumlah alel yang diamati (ne) =
3. Jumlah alel yang efektif (na) =
Keterangan : pi = frekuensi genetik tipe ke i 4. Heterozigositas harapan =
Keterangan : pi = frekuensi genetik tipe ke i
5. Diferensiasi genetik (Gst) (Nei 1973) =
Keterangan :
BAB IV
HASIL DAN PEMBAHASAN
4.1. Kualitas DNA Hasil Ekstraksi
Hasil dari uji kualitas DNA menentukan langkah selanjutnya pada tahapan PCR. Uji kualitas DNA ini juga dilakukan untuk menentukan perbandingan pengenceran DNA yang digunakan sebagai salah satu bahan pada proses PCR. Beberapa sampel yang diekstraksi dengan metode CTAB pada daun sengon yang dilakukan pada penelitian ini memperlihatkan pola pita yang tebal dan membutuhkan pengenceran (Gambar 3a) yang mengidentifikasikan masih terkontaminasi oleh bahan-bahan kimia sisa proses ekstraksi dan protein, polisakarida dan RNA (Qiagen 2001, diacu dalam Mulyadiana 2010) berbeda halnya dengan pita yang dihasilkan dengan mengunakan GenElute Plant Genomic DNA Miniprep Kit dari SIGMA (Gambar 3b).
Gambar 3 Contoh hasil ekstraksi DNA sengon: (a) DNA sengon yang diekstraksi dengan buffer CTAB dan dilakukan pengenceran, (b) DNA sengon yang diekstraksi dengan GenElute Plant Genomic DNA miniprep Kit dari SIGMA
Salah satu keuntungan analisis keragaman menggunakan teknik molekuler yang memanfaatkan teknologi PCR adalah kuantitas DNA yang dibutuhkan hanya sedikit. Selain itu, untuk RAPD tingkat kemurnian DNA yang dibutuhkan tidak perlu terlalu tinggi dengan kata lain teknik ini toleran terhadap tingkat kemurnian. Akan tetapi dibutuhkan senyawa-senyawa kontaminan yang dapat mengganggu reaksi PCR seperti polisakarida dan metabolit sekunder (Prana et al 2003).
Keberadaan polisakarida dan senyawa metabolit sekunder dalam sel tanaman sering menyulitkan dalam isolasi asam nukleat. Selain itu, metabolit sekunder dan polisakarida juga dapat menghambat kerja enzim.
18 Salah satu masalah yang dihadapi ketika mengekstraksi daun sengon adalah adanya kandungan polisakarida yang tinggi dalam tanaman ditandai dengan kekentalan pada hasil isolasi DNA. Oleh karena itu, untuk mengatasi hal ini maka perlu dilakukan pengenceran. Setiap sampel DNA mempunyai perlakuan pengenceran yang berbeda, tergantung dari kualitas DNA. Pengenceran ini dilakukan agar tahap PCR primer dapat menempel pada pita DNA dan dapat teramplifikasi. Akan tetapi, ekstraksi DNA dari jaringan tanaman dengan tingkat kemurnian yang tinggi seringkali sulit diperoleh.
4.1.1. PCR ( Polymerase Chain Reaction)
4.1.1.1. Seleksi Primer
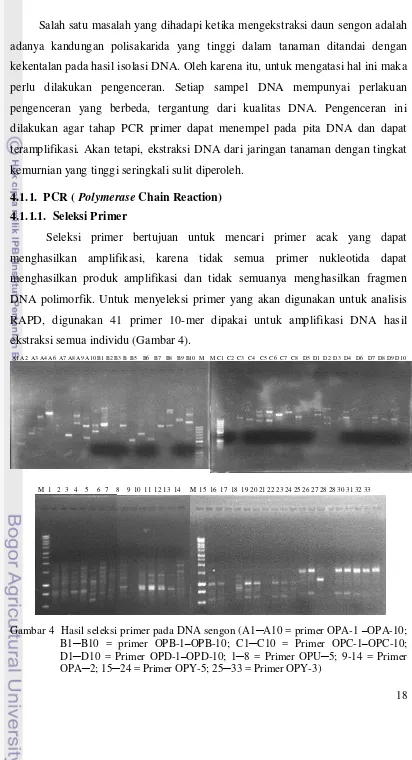
Seleksi primer bertujuan untuk mencari primer acak yang dapat menghasilkan amplifikasi, karena tidak semua primer nukleotida dapat menghasilkan produk amplifikasi dan tidak semuanya menghasilkan fragmen DNA polimorfik. Untuk menyeleksi primer yang akan digunakan untuk analisis RAPD, digunakan 41 primer 10-mer dipakai untuk amplifikasi DNA hasil ekstraksi semua individu (Gambar 4).
A1 A2 A3 A4 A6 A7 A8 A9 A10 B1 B2 B3 B B5 B6 B7 B8 B9 B10 M M C1 C2 C3 C4 C5 C6 C7 C8 D5 D1 D2 D3 D4 D6 D7 D8 D9 D10
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 M 15 16 17 18 19 20 21 22 23 24 25 26 27 28 28 30 31 32 33
19 Berdasarkan foto hasil seleksi pada Gambar 4, dapat dilihat kualitas pita pada DNA tanaman Sengon untuk masing-masing primer. Dari 41 primer yang diseleksi terdapat 20 primer yang menghasilkan pita polimorfik. Tidak semua pita yang dihasilkan menunjukkan kualitas yang bagus. Ada beberapa pita yang kurang jelas, sehingga hal ini mengakibatkan keraguan dalam menginterpretasikan serta menganalisis pita DNA Sengon. Hal ini bisa disebabkan karena kurang murninya DNA genom yang dihasilkan, proses pengenceran dan komposisi bahan-bahan yang kurang tepat.
DNA yang kurang murni dan terlalu kental dapat menghambat amplifikasi saat PCR karena primer tidak dapat menempel pada DNA target. Selain itu, banyaknya kontaminan yang berasal dari bahan kimia yang belum tercuci sempurna dapat menghambat primer untuk menempel pada DNA target pada tahap annealing.
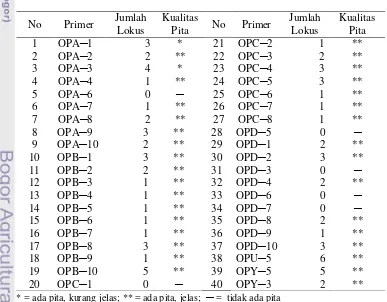
Dalam penelitian ini dipilih 5 primer terbaik untuk dilakukan interpretasi dan analisis pada penelitian ini yaitu 2, OPY-5, OPU-5, OPB-10 dan OPA-3. Kualitas pita tersebut disajikan pada Tabel 5.
Tabel 5 Kualitas pita pada DNA tanaman Sengon
No Primer Jumlah Lokus
Kualitas
Pita No Primer
Jumlah Lokus
Kualitas Pita
1 OPA─1 3 * 21 OPC─2 1 **
2 OPA─2 2 ** 22 OPC─3 2 **
3 OPA─3 4 * 23 OPC─4 3 **
4 OPA─4 1 ** 24 OPC─5 3 **
5 OPA─6 0 ─ 25 OPC─6 1 **
6 OPA─7 1 ** 26 OPC─7 1 **
7 OPA─8 2 ** 27 OPC─8 1 **
8 OPA─9 3 ** 28 OPD─5 0 ─
9 OPA─10 2 ** 29 OPD─1 2 **
10 OPB─1 3 ** 30 OPD─2 3 **
11 OPB─2 2 ** 31 OPD─3 0 ─
12 OPB─3 1 ** 32 OPD─4 2 **
13 OPB─4 1 ** 33 OPD─6 0 ─
14 OPB─5 1 ** 34 OPD─7 0 ─
15 OPB─6 1 ** 35 OPD─8 2 **
16 OPB─7 1 ** 36 OPD─9 1 **
17 OPB─8 3 ** 37 OPD─10 3 **
18 OPB─9 1 ** 38 OPU─5 6 **
19 OPB─10 5 ** 39 OPY─5 5 **
20 OPC─1 0 ─ 40 OPY─3 2 **
20 4.1.2.2. Analisis RAPD Populasi Sengon di Jawa
Amplifikasi PCR menggunakan 5 primer (OPA-2, OPB-10, OPU-5, OPA-3 dan OPY-5) berkisar antara 1─10 pita. Analisis DNA Sengon dengan penanda RAPD dilakukan melalui skoring dari semua individu yang berasal dari 9 populasi, dengan jumlah 225 individu.
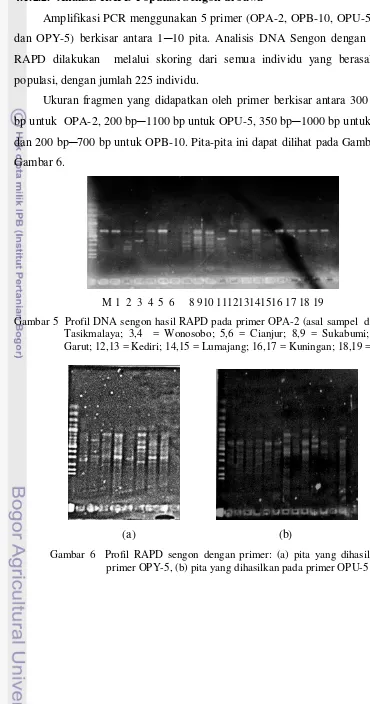
Ukuran fragmen yang didapatkan oleh primer berkisar antara 300 bp─900 bp untuk OPA-2, 200 bp─1100 bp untuk OPU-5, 350 bp─1000 bp untuk OPY-5, dan 200 bp─700 bp untuk OPB-10. Pita-pita ini dapat dilihat pada Gambar 5 dan Gambar 6.
M 1 2 3 4 5 6 8 910 111213141516 17 18 19
Gambar 5 Profil DNA sengon hasil RAPD pada primer OPA-2 (asal sampel dari: 1,2 = Tasikmalaya; 3,4 = Wonosobo; 5,6 = Cianjur; 8,9 = Sukabumi; 10,11 = Garut; 12,13 = Kediri; 14,15 = Lumajang; 16,17 = Kuningan; 18,19 = Subang)
(a) (b)
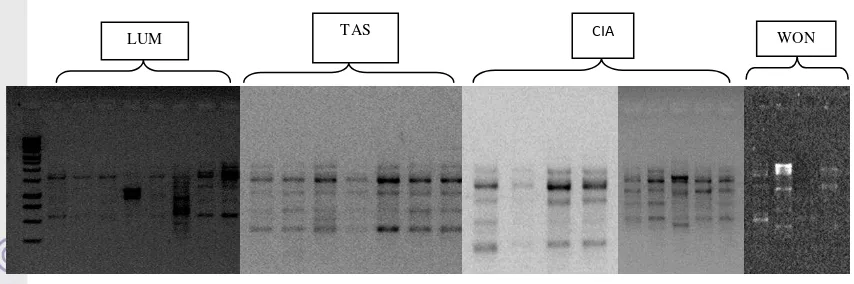
21 Gambar 7 Profil RAPD sengon dengan primer pita yang dihasilkan pada pita OPB-10 (asal sampel dari: LUM = Lumajang; TAS = Tasikmalaya; CIA = Cianjur; WON = Wonosobo)
4.2 Variasi Genetik
4.2.1 Variasi Genetik Dalam Populasi
Menurut Finkeldey (2005), Parameter yang digunakan untuk menandakan keragaman genetik dalam populasi, yaitu Persentase Lokus Polimorfik (PLP), jumlah alel yang diamati (na), jumlah alel efektif (ne) dan variasi genetik (He). Hasil analisis menunjukkan rata-rata jumlah alel yang diamati pada populasi Sengon di Pulau Jawa adalah 2,0000 dan rata-rata jumlah alel efektif adalah 1,3280. Adapun nilai rata-rata keragaman genetik (He) pada total populasi Sengon di Pulau Jawa sebesar 0,2349 dan Persen Lokus Polimorfis (PLP) pada populasi Sengon dalam penelitian mencapai 100%. Hasil analisis ini dapat dilihat pada Tabel 6.
Tabel 6 Hasil pengukuran keragaman genetik 9 populasi Sengon di Jawa.
No Populasi Jumlah
Sampel Na Ne He PLP
1 Cianjur 25 1,9815 1,4462 0,2757 98,15 %
2 Garut 25 2,0000 1,2555 0,1602 100,00 %
3 Sukabumi 25 2,0000 1,4693 0,2761 100,00 % 4 Tasikmalaya 25 2,0000 1,2675 0,1830 100,00 %
5 Subang 25 2,0000 1,2808 0,1941 100,00 %
6 Kuningan 25 2,0000 1,2128 0,1485 100,00 % 7 Wonosobo 25 2,0000 1,1675 0,1328 100,00 %
8 Kediri 25 2,0000 1,4892 0,2946 100,00 %
9 Lumajang 25 2,0000 1,2458 0,1785 100,00 % Rata─rata 2,0000 1,3280 0,2349 100,00 % Na = jumlah alel yang diamati; Ne = jumlah alel yang efektif; He = keragaman genetik; PLP = Presentasi Lokus Polimorfik
22 Dari Tabel 6 dapat dilihat nilai He dari masing-masing populasi sengon cukup beragam berkisar antara 0,1328─0,2946 dengan nilai rata-rata He populasi Sengon di Jawa sebesar 0,2349.
Menurut Weising et al. (2005), dominant marker seperti RAPD hanya dapat memproduksi dua alel pada masing-masing lokus. Maka dari itu nilai He maksimum adalah 0,5. Dari hasil analisis DNA dalam penelitian ini, dapat dikatakan bahwa keragaman genetik populasi sengon di Jawa tergolong tinggi. Hasil ini hampir mendekati dari hasil penelitian keragaman genetik Sengon yang dilakukan dengan penanda RAPD oleh Dwiyanti (2009) sebesar 0,2813 dan Widyastuti (2007) sebesar 0,1852 (dengan 6 populasi di Jawa, yaitu Cianjur, Kuningan, Tasikmalaya, Banjarnegara, Temanggung dan Lumajang). Hasil ini tidak berbeda jauh dengan penelitian keragaman genetik dengan penanda isoenzim oleh Gunawan (2005) sebesar 0,235 dan Wulan (2003) sebesar 0,172 (dengan 9 populasi di Jawa, yaitu Lumajang, Banjarnegara, Wonosobo, Garut, Tasikmalaya, Cianjur, Kuningan, Sukabumi dan Subang).
Populasi Kediri (Jawa Timur) memiliki nilai He tertinggi dari populasi lainnya di penelitian ini yaitu sebesar 0,2946, sedangkan He terendah terdapat pada populasi Wonosobo (Jawa Tengah) yaitu 0,1328.
Menurut Saidman et al. (1997) diacu dalam Widyastuti (2007), analisis keragaman genetik suatu populasi dengan penanda genetik yang berlainan sebenarnya tidak menghasilkan nilai keragaman yang berbeda. Perbedaan nilai keragaman yang diperoleh dari beberapa penelitian terhadap populasi Sengon lebih disebabkan karena adanya perbedaan sampel yang diambil.
4.2.2. Jarak Genetik Antar Populasi
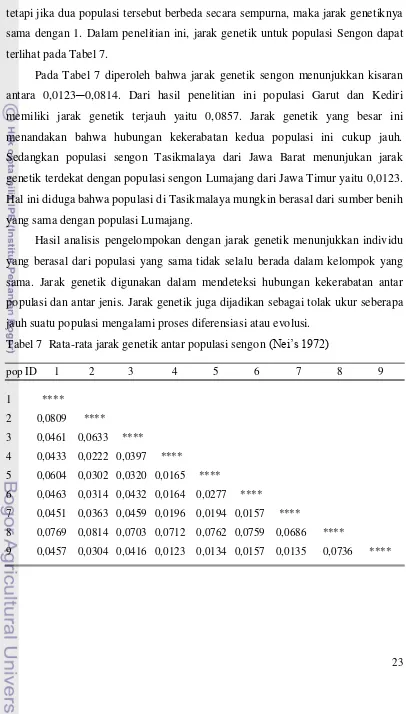
23 dengan 0 maka dua populasi tersebut tidak memiliki perbedaan (identik), akan tetapi jika dua populasi tersebut berbeda secara sempurna, maka jarak genetiknya sama dengan 1. Dalam penelitian ini, jarak genetik untuk populasi Sengon dapat terlihat pada Tabel 7.
Pada Tabel 7 diperoleh bahwa jarak genetik sengon menunjukkan kisaran antara 0,0123─0,0814. Dari hasil penelitian ini populasi Garut dan Kediri memiliki jarak genetik terjauh yaitu 0,0857. Jarak genetik yang besar ini menandakan bahwa hubungan kekerabatan kedua populasi ini cukup jauh. Sedangkan populasi sengon Tasikmalaya dari Jawa Barat menunjukan jarak genetik terdekat dengan populasi sengon Lumajang dari Jawa Timur yaitu 0,0123. Hal ini diduga bahwa populasi di Tasikmalaya mungkin berasal dari sumber benih yang sama dengan populasi Lumajang.
Hasil analisis pengelompokan dengan jarak genetik menunjukkan individu yang berasal dari populasi yang sama tidak selalu berada dalam kelompok yang sama. Jarak genetik digunakan dalam mendeteksi hubungan kekerabatan antar populasi dan antar jenis. Jarak genetik juga dijadikan sebagai tolak ukur seberapa jauh suatu populasi mengalami proses diferensiasi atau evolusi.
Tabel 7 Rata-rata jarak genetik antar populasi sengon (Nei’s 1972)
pop ID 1 2 3 4 5 6 7 8 9 1 ****
2 0,0809 ****
3 0,0461 0,0633 ****
4 0,0433 0,0222 0,0397 ****
5 0,0604 0,0302 0,0320 0,0165 ****
6 0,0463 0,0314 0,0432 0,0164 0,0277 ****
7 0,0451 0,0363 0,0459 0,0196 0,0194 0,0157 ****
8 0,0769 0,0814 0,0703 0,0712 0,0762 0,0759 0,0686 ****
24 Gambar 8 Dendrogram jarak genetik 9 populasi sengon di Pulau Jawa berdasarkan Nei’s
(1972)
Gambar 8 diatas juga menunjukkan Tasikmalaya dan Lumajang memiliki jarak genetik paling dekat. Dari dendrogram terdapat 2 kelompok besar dalam populasi tersebut. Kelompok pertama terdiri atas Tasikmalaya, Lumajang, Subang, Kuningan, Wonosobo, Garut, Sukabumi dan Cianjur, sedangkan kelompok kedua adalah Kediri. Kediri memiliki hubungan jarak genetik yang jauh dengan 8 populasi lainnya. Dendrogram diatas menunjukkan bahwa semua populasi mengelompok secara acak.
Jika 9 populasi tersebut dikelompokkan berdasarkan 3 wilayah bagian yaitu Jawa Barat, Jawa Tengah, dan Jawa Timur dapat terlihat pada dendrogram diatas bahwa pola penyebarannya tidak dipengaruhi oleh sebaran geografis. Hal ini terbaca dari Dendrogram bahwa Jawa Timur memiliki hubungan jarak genetik yang dekat dengan Jawa Barat begitu juga dengan Jawa Tengah yang memiliki jarak genetik yang dekat dengan Jawa Barat. Akan tetapi Jawa Timur khususnya daerah Kediri memiliki jarak genetik paling jauh.
25 populasi yang terdapat pada satu daerah yang sama (Jawa Barat) ternyata memiliki jarak yang dekat dengan provinsi lain.
Berdasarkan hasil Analysis of Molecular Variance (AMOVA) pada Tabel 8, keragaman genetik yang tersimpan didalam populasi adalah sebesar 82% sedangkan keragaman genetik antar populasi adalah 18%. Dari hasil yang didapat menunjukkan bahwa sebagian besar variasi genetik tersimpan di dalam populasi dibandingkan keragaman genetik antar populasi.
Tabel 8 Hasil Analysis of Molecular Variance (AMOVA)
Sumber Keragaman Derajat Bebas
Jumlah Kuadrat
Kuadrat
Tengah Est.Var. % Antar Populasi 8 410,276 51,284 1,737 18 Dalam Populasi 216 1695,120 7,848 7,848 82
Total 224 2105,396 9,585 100
Menurut Hamrick dan Godt (1996) diacu dalam Yunanto (2010), pada umumnya variasi genetik yang tersimpan didalam populasi lebih besar sedangkan perbedaan antara populasi hanya sedikit.
Menurut Namkoong et al. (1996) diacu dalam Finkeldey (2005) salah satu indikator genetik dalam pelaksanaan manajemen hutan lestari adalah besarnya keragaman genetik. Keragaman genetik merupakan modal dasar bagi suatu jenis tanaman untuk tumbuh, berkembang, dan bertahan hidup untuk generasi berikutnya. Adapun faktor-faktor yang mempengaruhi keragaman genetik antara lain adalah seleksi alam, pergeseran genetik (genetic drift), perpindahan gen (gen flow), dan mutasi.
Keragaman genetik tanaman bersumber dari dua hal yaitu proses meiosis dan mutasi. Meiosis adalah proses rekombinasi gen melalui segregasi acak. Meiosis hanya melibatkan keragaman genetik yang telah ada di dalam populasi atau jenis yang bersangkutan. Mutasi merupakan perubahan genetik yang terjadi dikarenakan penyimpangan yang terjadi pada proses pewarisan sifat dan merupakan sumber keragaman baru dalam populasi tanaman.
26 adaptabilitas (Finkeldey dan Hattemer 2007, diacu dalam Rimbawanto 2008). Variasi genetik yang tinggi akan menghasilkan sifat resisten atau tahan terhadap kondisi lingkungan, sehingga serangan hama dan penyakit dapat dihindari.
Jika dilihat dari hasil keragaman genetik masing-masing populasi dapat diketahui bahwa populasi Kediri memiliki nilai keragaman genetik yang paling tinggi. Hal ini berarti Kediri memiliki kemampuan adaptasi yang baik terhadap lingkungannya.
Adanya keragaman genetik yang tinggi pada populasi Sengon bertujuan dalam pelaksanaan program pemuliaan pohon untuk menghasilkan keturunan yang baik. Keragaman genetik merupakan kunci dalam program pemuliaan pohon. Hal ini dikarenakan adanya maksimalisasi perolehan genetik akan sifat tertentu. Program pemuliaan pohon berguna untuk memelihara dan meningkatkan variabilitas genetik di dalam suatu populasi. Studi keragaman genetik memiliki manfaat penting untuk pemuliaan pohon yaitu dalam membantu seleksi buatan, membantu menyiapkan uji provenans dan pengendali persilangan dan aktivitas silvikultur. Seleksi merupakan proses pemuliaan pohon dan dasar untuk perbaikan dalam mendapatkan kultivar unggul yang baru, keragaman genetik yang tinggi merupakan suatu syarat efektifnya program seleksi.
Dalam penelitian ini keragaman genetik populasi Sengon sebesar 0,2349, sehingga dari hasil yang didapat tersebut menunjukkan bahwa keragaman populasi Sengon yang cukup tinggi mendukung dalam kegiatan pemuliaan.
27 Selain itu, keragaman genetik yang tinggi juga menguntungkan dalam pelaksanaan konservasi tanaman hutan untuk pelestarian keragaman genetik. Adapun hal-hal yang melandasi pentingnya upaya konservasi yaitu, mempertahankan keberadaan individu pohon dan populasi, mempertahankan potensi evolusi dari jenis dan populasi tanaman, mempertahankan kemampuan suatu jenis atau populasi untuk merespon perubahan lingkungan yang tergantung pada keragaman genetik dan memberikan peluang pemanfaatan sumber genetik bagi berbagai tujuan lain.
Dari hasil penelitian ini, keragaman genetik yang tinggi yang diperlukan untuk tujuan konservasi dapat diwakili oleh populasi Kediri karena mempunyai keragaman genetik yang tinggi diantara 8 populasi lainnya. Maka dari itu, populasi Kediri harus dipertahankan agar keberlangsungan Sengon dimasa yang akan datang dapat dipertahankan.
BAB V
KESIMPULAN DAN SARAN
5.1. Kesimpulan
1. Keragaman genetik sengon di Jawa tergolong tinggi dengan nilai rata-rata He = 0,2349. Nilai He terendah terdapat pada populasi Wonosobo yaitu 0,1328 dan nilai He tertinggi terdapat pada populasi Kediri yaitu 0,2946. 2. Populasi sengon Tasikmalaya dan Lumajang memiliki hubungan
kekerabatan terdekat dengan jarak genetik sebesar 0,0123. Sedangkan kekerabatan terjauh terdapat antara populasi Garut dan Kediri dengan jarak genetik 0,0814.
3. Sebagian besar variasi genetik tersimpan di dalam populasi yaitu sebesar 82%, sedangkan perbedaan antar populasi hanya sebesar 18%.
5.2. Saran
Berdasarkan analisis data yang didapat dapat diberikan beberapa saran : 1. Untuk pelaksanaan kegiatan konservasi tanaman, dilihat dari keragaman
genetik yang tinggi, populasi Sengon yang berasal dari Sukabumi dan Kediri cukup mewakili. Sedangkan populasi yang memiliki keragaman yang rendah cukup diwakili oleh populasi Sengon Wonosobo.
KERAGAMAN GENETIK POPULASI SENGON (
Paraserianthes
falcataria
(L) NIELSEN) PADA HUTAN RAKYAT DI JAWA
BERDASARKAN PENANDA
RAPD
RANNY DWITA OLIVIA
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
DAFTAR PUSTAKA
Aritonang KV, Siregar IZ, Yunanto T. 2007. Manual Analisis Genetik Tanaman Hutan di Laboratorium Silvikultur Fakultas Kehutanan Institut Pertanian Bogor. Bogor: Laboratorium Silvikultur IPB.
Atmosuseno BS. 1998. Budidaya, Kegunaan, dan Prospek Sengon. Bogor: Penebar Swadaya..
Crowder LV. 1986. Genetika Tumbuhan. Yogyakarta: UGM─Press.
Dwiyanti FG. 2009. Keragaman sengon solomon (Paraserianthes falcataria (L) Nielsen) pada uji keturunan di hutan percobaan Cirangsad [skripsi]. Bogor. Departemen Silvikultur, Fakultas Kehutanan. Institut Pertanian Bogor.
Finkeldey R. 2005. Pengantar Genetika Hutan Tropis. Djamhuri E, Siregar IZ, Siregar UJ, Kertadikara AW, penerjemah. Bogor: Fakultas Kehutanan, Institut Pertanian Bogor. Terjemahan dari: An Indtroduction to Tropical Forest Genetics.
Gunawan R. 2005. Struktur dan variasi genetik bibit tanaman sengon, mangium, durian dan rambutan untuk GNRHL berdasarkan isoenzim [skripsi]. Bogor: Departemen Manajemen Hutan, Fakultas Kehutanan. Institut Pertanian Bogor.
Husnaeni A. 2008. Variasi genetik jati pada hutan tanaman di Jawa berdasarkan penanda RAPD (Random Ampliflied Polymorphic DNA) [skripsi]. Bogor: Departemen Silvikultur, Fakultas Kehutanan. Institut Pertanian Bogor.
Karsinah, Sudarsono, Setyobudi L, Aswidinnoor H. 2002. Keragaman genetik plasma nutfah Jeruk berdasarkan analisis penanda RAPD. Jurnal Bioteknologi Pertanian 7(1):8─16.
Mulyadiana A. 2010. Keragaman genetik Shorea laevis Ridl. di Kalimantan berdasarkan penanda mikrosatelit [skripsi]. Bogor: Departemen Silvikultur, Fakultas Kehutanan. Institut Pertanian Bogor.
Pharmawati M. 2009. Optimalisasi ekstraksi DNA dan PCR─RAPD pada Grevilla spp. (Proteaceaceae). Jurnal Biologi XIII (1):12─16.
Prana TK, Hartati NS. 2003. Identifikasi sidik jari DNA Talas (Colocasia esculente L. Schott) Indonesia dengan teknik RAPD (Random Amplified Polimorphic DNA): Skrining Primer dan Optimalisasi Kondisi PCR. Jurnal Natur Indonesia 5(2):107─112.
[Promega]. 2003. PCR Core System. USA: Promega Corporation.
30 Santoso HB. 1992. Budidaya Sengon. Yogyakarta: Kanisius.
Siregar IZ, Yunanto T, Ratnasari J. 2008. Prospek Bisnis, Budi Daya, Panen & Pascapanen Kayu Sengon. Jakarta: Penebar Swadaya.
Siregar IZ, Yunanto T, Pamoengkas P. 2008. Implikasi genetik metode pembiakan tanaman Shorea joharensis Foxw pada sistem silvikultur Tebang Pilih Tanam Jalur (TPTJ). Jurnal Biodiversitas 9(4):250─254.
Siregar UJ, Kusmana C. 2002. Studi keragaman Rhizophora mucronata Lamk. pada hutan alam di Sumatera Utara berdasarkan analisis isoenzim [laporan akhir penelitian]. Bogor: Fakultas Kehutanan, Institut Pertanian Bogor.
Weising K, Nybom H, Wolff K, Kahl G. 2005. DNA Fingerprinting in Plants Principles, Methods and Applications. Boca Raton: CRC Press.
Widyastuti DE. 2007. Keragaman genetik dengan penanda RAPD, fenotipa pertumbuhan dan pendugaan heritabilitas pada sengon (Paraserianthes falcataria (L.) Nielsen) [tesis]. Bogor: Program Pascasarjana, Institut pertanian Bogor.
William JGK, Kubelik AR, Livak KJ, Rafalski JA, Tingey SV. 1990. DNA polimorphisms amplified by arbitary primers are useful as genetic markers. Nucleic Acid Research 18:6531─6535.
Wulan R. 2003. Struktur dan Keragaman genetik populasi sengon (Paraserianthes falcataria (L) Nielsen) pada hutan rakyat di Jawa.[tesis]. Bogor: Program Pascasarjana, Institut Pertanian Bogor.
Yunanto T. 2006. Implifikasi genetik sistem silvikultur TPTJ pada jenis Shorea johorensis di HPH PT. Sari Bumi Kusuma berdasarkan Random Amplified Polymorphic DNA (RAPD) [skripsi]. Bogor: Departement Manajemen Hutan, Fakultas Kehutanan, Institut Pertanian Bogor.
KERAGAMAN GENETIK POPULASI SENGON (
Paraserianthes
falcataria
(L) NIELSEN) PADA HUTAN RAKYAT DI JAWA
BERDASARKAN PENANDA
RAPD
RANNY DWITA OLIVIA
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
ii
KERAGAMAN GENETIK POPULASI SENGON (
Paraserianthes
falcataria
(L) NIELSEN) PADA HUTAN RAKYAT DI JAWA
BERDASARKAN
PENANDA
RAPD
RANNY DWITA OLIVIA
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar
Sarjana Kehutanan pada
Departemen Silvikultur
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
iii
RINGKASAN
RANNY DWITA OLIVIA. Keragaman Genetik Populasi Sengon (Paraserianthes falcataria (L) NIELSEN) pada Hutan Rakyat di Jawa Berdasarkan Penanda RAPD. Dibimbing oleh ULFAH JUNIARTI SIREGAR.
Paraserianthes falcataria (L) Nielsen yang dikenal dengan nama lokal sengon banyak ditanam pada hutan rakyat di Jawa, karena tergolong pohon yang cepat tumbuh dan multiguna, baik dari daun, batang, dan sistem perakaran. Kebanyakan sengon ditanam secara monokultur sehingga pertanaman ini mempunyai masalah yaitu, mudah terserang hama dan penyakit seperti hama penggerek batang Xystrocera festiva, damping off, dan karat puru. Untuk itu diperlukan sengon unggul yang tahan hama dan penyakit, yang dihasilkan dari program pemuliaan. Untuk melaksanakan program pemuliaan, maka diperlukan keragaman genetik yang tinggi. Tujuan dari penelitian ini adalah untuk mengetahui keragaman genetik di antara populasi tanaman sengon pada beberapa hutan rakyat di Jawa.
Sampel tanaman sengon diambil dari 9 populasi yaitu, Cianjur, Sukabumi, Garut, Kuningan, Subang, Tasikmalaya dari Jawa Barat, Wonosobo dari Jawa Tengah, Kediri dan Lumajang dari Jawa Timur, dengan jumlah 25 sampel perpopulasi. DNA Sengon diekstraksi dengan menggunakan metode CTAB dan GenElute Plant Genomic DNA miniprep Kit dari SIGMA. Pada proses PCR, DNA tersebut diamplifikasi menggunakan primer OPA-2, OPA-3, OPB-10, OPY-5, dan OPU-5. Analisis data dilakukan dengan program POPGENE, NTSYS, dan GenAlex. Parameter yang diukur adalah heterozigositas harapan (He), Presentase Lokus Polimorfik (PLP), jumlah alel yang diamati (na), jumlah alel efektif (ne), dan jarak genetik.
Nilai dugaan heterozigositas harapan (He) seluruh populasi adalah 0,2349, menunjukkan bahwa populasi sengon di Jawa memiliki keragaman genetik yang tinggi. Sebagian besar variasi genetik tersimpan di dalam populasi yaitu sebesar 82%, sedangkan perbedaan antar populasi hanya sebesar 18%. Populasi Garut dan Kediri memiliki jarak genetik terjauh (0,0857) sementara jarak genetik terdekat (0,0123) terdapat pada populasi Tasikmalaya dan Lumajang. Dendrogram jarak genetik menunjukkan bahwa pola penyebaran sengon di Jawa terjadi secara acak, karena populasi yang terdapat pada satu daerah yang sama (Jawa Barat) ternyata memiliki jarak yang dekat dengan provinsi lain.
iv
ABSTRACT
RANNY DWITA OLIVIA. Genetic Diversity of Sengon Population (Paraserianthes falcataria (L.) Nielsen) in Community Forest in Java based on RAPD Marker. Under supervision of ULFAH JUNIARTI SIREGAR.
Paraserianthes falcataria (L.) Nielsen, with local name “sengon” has been planted widely as community forest in Java because of its easy and fast growing nature having multipurposes from its leaves, stems and roots. As sengon has been grown in monoculture, the plantation often has problems such as, susceptible to stem borer pest, Xystrocera festiva, damping off, and karat puru disease. Therefore, resistant sengon is required, which is obtained from breeding programs. A good breeding program requires high genetic diversity. The objective of this research is to study the genetic diversity of sengon populations in some community forests in Java.
Sengon leaf samples were taken from 9 populations in Java namely: from Cianjur, Sukabumi, Garut, Kuningan, Tasikmalaya, and Subang (West Java), Wonosobo (Central Java), Kediri and Lumajang (East Java). From each population 25 trees were randomly sampled. Sengon’s DNA was extracted using CTAB method and GenElute Plant Genomic DNA miniprep Kit from SIGMA. The DNA samples were amplified using 5 random primers, i.e. OPA-2, OPA-3, OPB-10, OPY-5 and OPU-5. Generated data was then analyzed using POPGENE, NTSYS and GenAlex program. Genetic diversity parameters measured consist of heterozygosity (He), percentage of polymorphic locus (PLP), the number of observed alleles (na), the number of effective alleles (ne), and genetic distance.
The value of expected heterozygosity (He) of whole population was 0.2349 indicated that sengon populations in Java have high genetic diversity. Most of the genetic variation (82%) was found within the population, while the differences among populations was only 18%. Kediri and Garut population had longest genetic distance (0.0857), and the closest genetic distance (0.0123) was found between Tasikmalaya and Lumajang population. Dendrogram based on the genetic distances showed that the distribution pattern of sengon in Java had occurred randomly, because some populations belonged to the same area (e.g. West Java) appeared to have close genetic relationship with population from other provinces.
v
PERNYATAAN
Dengan ini saya menyatakan bahwa skripsi berjudul Keragaman Genetik Populasi Sengon (Paraserianthes falcataria (L) NIELSEN) pada Hutan Rakyat di Jawa Berdasarkan Penanda RAPD adalah benar-benar hasil karya saya sendiri dengan bimbingan dosen dan belum pernah digunakan sebagai karya ilmiah pada perguruan tinggi atau lembaga manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Bogor, Maret 2012
vi
LEMBAR PENGESAHAN
Judul Skripsi : Keragaman Genetik Populasi Sengon (Paraserianthes falcataria (L) NIELSEN) pada Hutan Rakyat di Jawa Berdasarkan Penanda RAPD
Nama : Ranny Dwita Olivia NRP : E44070029
Menyetujui: Dosen Pembimbing,
Dr. Ir. Ulfah Juniarti Siregar, M.Agr NIP. 19580606 198303 2 001
Mengetahui:
Ketua Departemen Silvikultur Fakultas Kehutanan Institut Pertanian Bogor
Prof. Dr. Nurheni Wijayanto, MS NIP. 19601024 198403 1 009
vii
KATA PENGANTAR
Dengan mengucapkan Alhamdulillah, penulis mengucapkan puji dan syukur kehadirat Allah SWT yang telah memberikan rahmat dan karuniaNya sehingga karya ilmiah ini dapat diselesaikan dengan baik.
Karya ilmiah ini penulis persembahkan untuk kedua orang tua, kakak, dan adik penulis atas doa dan kasih sayang, serta menjadi sumber inspirasi dalam menyelesaikan karya ilmiah ini. Penyusunan karya ilmiah ini tentunya tidak lepas dari bantuan berbagai pihak. Oleh karena itu penulis menyampaikan penghargaan dan rasa terimakasih kepada:
1. Dr. Ir. Ulfah Juniarti Siregar, M.Agr selaku pembimbing skripsi yang telah memberikan arahan dan masukan dalam pelaksanaan dan penyusunan karya ilmiah ini.
2. Dr. Ir. Endes N. Dahlan, MS selaku dosen penguji dan Prof. Dr. Ir. Nurheni Wijayanto, MS selaku ketua sidang pada ujian komprehensif yang telah memberikan masukan yang begitu berarti kepada penulis. 3. Teknisi Common Laboratory, Seameo Biotrop (Mba Anidah) untuk
bimbingan dan arahan selama penelitian berlangsung.
4. Seluruh dosen dan staf Departemen Silvikultur untuk ilmu yang telah diberikan serta kerja sama baik selama pekuliahan maupun dalam penyusunan karya ilmiah ini.
5. Keluarga besar penulis Nenek, Om, tante, abang, serta adik-adikku atas doa dan kasih sayang, dukungan, dan menjadi inspirasi bagi penulis. 6. Sahabat terbaikku Ridha, Susan, Mas Hendro, Dyah Ayu, Anditya,
Gya, dan seluruh keluarga besar silvikultur 44 (khususnya Laswi, Aziz, Anin, Cyntia, Hafizah, Nifa, Eri, dan Eka), teman-teman kosan Dwi regina yang telah memberi semangat dan keceriaan serta tawa dalam setiap langkah penulis.
7. Mba Ana, Mayun, dan Juma atas dukungan dan kerjasamanya selama penelitian berlangsung.
Penulis menyadari bahwa masih banyak kekurangan dalam penulisan skripsi ini. Oleh karena itu penulis sangat mengharapkan saran dan kritik yang membangun. Semoga skripsi ini dapat digunakan sebagai referensi untuk kepentingan ilmu pengetahuan dan bermanfaat dalam pelaksanaan penelitian berikutnya.
Bogor, Maret 2012
viii
RIWAYAT HDUP
Penulis dilahirkan di Bukittinggi, Sumatera Barat pada tanggal 27 Januari 1989 sebagai anak kedua dari tiga bersaudara. Darah Minang yang kental mengalir dari kedua orangtua penulis, Bapak Muryadi (alm) dan Ibu Yenita K.
Penulis mulai menapaki jenjang pendidikan formal di TK Masyithah Bukittinggi pada tahun 1994, dilanjutkan pada tahun 1995 di SD Jam’iyyatul Hujjaj Bukittinggi, dan lulus pada tahun 2001. Kemudian pada tahun yang sama melanjutkan ke SLTPN 4 Bukittinggi dan lulus pada tahun 2004. Pada tahun 2004 penulis melanjutkan ke SMUN 1 Bukittinggi dan lulus pada tahun 2007. Pada tahun yag sama penulis diterima di Institut Pertanian Bogor melalui program Undangan Seleksi Masuk IPB (USMI) di Program Studi Silvikultur, Fakultas Kehutanan, Institut Pertanian Bogor.
Selama menuntut ilmu di Institut Pertanian Bogor, penulis aktif dalam berbagai kegiatan, di antaranya Badan Eksekutif Mahasiswa Keluarga Mahasiswa (BEM KM) Institut Pertanian Bogor (2008−2009), Tree Grower Community (TGC) (2008−2010), dan Bina Desa IPB (2008−2009).
Penulis juga mengikuti Praktek Pengenalan Ekosistem Hutan (PPEH) di Gunung Sawal dan Pangandaran pada tahun 2009, Praktek Pengelolaan Hutan (PPH) di Hutan Pendidikan Gunung Walat (HPGW) tahun 2010 serta Praktek Kerja Profesi Di PT. Newmont Nusa Tenggara tahun 2011.
DAFTAR ISI
x
xi
DAFTAR TABEL
Halaman 1 Lokasi tempat pengambilan sampel daun sengon pada hutan rakyat di
Jawa. ... 10
2 Komponen bahan untuk reaksi PCR. ... 13
3 Tahapan proses PCR ... 13 4 Urutan basa nukleotida 40 primer (Operon Technology) yang dicobakan. ... 14
5 Kualitas pita pada DNA tanaman sengon ... 19
xii
DAFTAR GAMBAR
Halaman 1 Bagan tahapan penelitian... 11
2 Cara analisis DNA dengan skoring ... 15
3 Contoh hasil ekstraksi DNA sengon: (a) DNA sengon yang diekstraksi dengan buffer CTAB dan dilakukan pengenceran, (b) DNA sengon yang diekstraksi dengan GenElute Plant Genomic DNA miniprep Kit dari SIGMA ... 17 4 Hasil seleksi primer pada DNA sengon (A1─A10 = primer OPA-1 –
OPA-10; B1─B10 = primer OPB-1–OPB-10; C1─C10 = Primer OPC-1– OPC-10; D1─D10 = Primer OPD-1–OPD-10; 1─8 = Primer OPU─5; 9-14 = Primer OPA─2; 15─24 = Primer OPY-5; 25─33 = Primer OPY-3) ... 18 5 Profil DNA sengon hasil RAPD pada primer OPA-2 (asal sampel dari: 1,2
= Tasikmalaya; 3,4 = Wonosobo; 5,6 = Cianjur; 8,9 = Sukabumi; 10,11 = Garut; 12,13 = Kediri; 14,15 = Lumajang; 16,17 = Kuningan; 18,19 = Subang) ... 20
6 Profil RAPD sengon dengan primer: (a) pita yang dihasilkan pada primer OPY-5, (b) pita yang dihasilkan pada primer OPU-5 ... 20
7 Profil RAPD sengon dengan primer pita yang dihasilkan pada pita OPB-10 (asal sampel dari: LUM = Lumajang; TAS = Tasikmalaya; CIA = Cianjur; WON = Wonosobo) ... 21
xiii
DAFTAR LAMPIRAN
Halaman
1 Bahan-bahan yang digunakan pada RAPD ... 32
2 Alat-alat yang digunakan dalam RAPD ... 33
3 Hasil analisis RAPD populasi sengon Cianjur ... 34
4 Hasil analisis RAPD populasi sengon Garut ... 35
5 Hasil analisis RAPD populasi sengon Sukabumi ... 36
6 Hasil analisis RAPD populasi sengon Tasikmalaya ... 37
7 Hasil analisis RAPD populasi sengon Subang ... 38
8 Hasil analisis RAPD populasi sengon Kuningan... 39
BAB 1
PENDAHULUAN
1.1. Latar Belakang
Indonesia merupakan negara kepulauan yang memiliki sumber daya alam melimpah dan tanah yang subur sehingga cocok sebagai tempat tumbuh dari berbagai jenis tanaman. Keadaan ini, mengantarkan Indonesia sebagai negara yang memiliki tingkat biodiversitas yang tinggi. Indonesia berada di daerah yang beriklim tropis, yang memiliki hutan tropis ketiga terbesar di dunia. Dari hutan tersebut, kita dapat mengambil manfaatnya baik dari segi kayu maupun non kayu.
Paraserianthes falcataria (L) Nielsen yang dikenal dengan nama lokal sengon banyak ditanam pada hutan rakyat di Jawa, karena tergolong pohon yang cepat tumbuh (Santoso 1992), multiguna baik itu daun, batang, dan sistem perakaran. Akan tetapi sengon ditanam secara monokultur sehingga tanaman ini mempunyai masalah yaitu, mudah terserang hama dan penyakit seperti hama penggerek batang Xystrocera festiva, damping off, dan karat puru. Untuk itu diperlukan sengon unggul yang dihasilkan dari program pemuliaan. Untuk melaksanakan program pemuliaan, dibutuhkan keragaman genetik yang tinggi.
Keragaman genetik yang tinggi pada suatu populasi menunjukkan potensi populasi tersebut untuk beradaptasi pada berbagai kondisi lingkungan. Penanda genetik dari analisis DNA digunakan sebagai alat bantu dalam mempelajari keragaman genetik dan mengidentifikasi genotipe suatu sampel. Informasi yang dihasilkan berguna dalam penentuan hubungan kekerabatan dan filogenetik populasi/individu setelah terjadi evolusi karena pengaruh waktu dan tempat. Dalam penelitian ini digunakan penanda molekuler karena penanda ini bersifat stabil dan tidak terpengaruh lingkungan. Saat ini telah berkembang berbagai jenis penanda molekuler diantaranya adalah isoenzim, RFLP, SSR, AFLP, dan RAPD (Random Amplified Polymorphic DNA).
2 diantaranya dapat dengan cepat mendeteksi polimorfisme fragmen DNA, relatif mudah dilakukan dan hanya memerlukan sejumlah kecil DNA. Penggunaan penanda RAPD memungkinkan dapat mendeteksi polimorfisme fragmen DNA yang diseleksi dengan menggunakan satu primer bersifat acak.
RAPD lebih bersifat sederhana, hal ini dikarenakan teknik RAPD tidak memerlukan informasi awal mengenai urutan DNA genom organisme yang diuji maupun tidak memerlukan probe DNA yang spesifik (Williams et al. 1990). Akan tetapi walaupun RAPD relatif cepat, murah, dan mudah untuk dilaksanakan, konsistensi hasil PCR menjadi perhatian sejak dipublikasikannya teknik ini. Primer RAPD dapat tidak cocok secara sempurna pada urutan penempelan primer, akibatnya amplifikasi pada beberapa siklus mungkin tidak terjadi, sehingga band tetap samar atau bahkan amplifikasi tidak terjadi jika primer tidak berhasil menempel pada DNA cetakan (Lamboy 1994, diacu dalam Pharmawati 2009).
Menurut Padmalatha dan Prasad (2006) diacu dalam Pharmawati (2009), tidak menempelnya primer pada DNA secara sempurna, diakibatkan karena tidak tepatnya konsentrasi komponen─komponen PCR-RAPD. Disamping itu, kualitas DNA juga dapat mempengaruhi. Adanya kandungan polifenol dan metabolit sekunder lain seperti tannin, terpen dapat menurunkan kemurnian DNA dan menghambat penempelan primer.
1.2. Tujuan Penelitian
Tujuan dari penelitian ini adalah untuk mengetahui keragaman genetik di dalam dan antar populasi tanaman Sengon (P. falcataria) pada beberapa hutan rakyat di Jawa.
1.3. Manfaat Penelitian
BAB II
TINJAUAN PUSTAKA
2.1. Deskripsi Sengon (Paraserianthes falcataria (L.) Nielsen)
2.1.1. Taksonomi dan Tata Nama
Taksonomi dari sengon adalah sebagai berikut: Kingdom : Plantae
Divisi : Spermatophyta Sub divisi : Angiospermae Kelas : Dicotyledonae Bangsa : Fabales
Suku : Leguminoceae Sub famili : Mimosoidae Marga : Paraserianthes
Jenis : falcataria (L.) Nielsen
2.1.2. Ekologi dan Penyebaran Alami
Berdasarkan sejarah, sengon adalah jenis asli dari kepulauan sebelah Timur Indonesia yakni di sekitar Maluku dan Irian Jaya, merupakan jenis pionir, terutama terdapat di hutan hujan dataran rendah. Tahun 1870-an pohon ini menyebar keseluruh kawasan Asia Tenggara mulai dari Myanmar sampai Filipina. Habitat alami pohon sengon ditemukan di kepulauan Maluku, Papua Nugini, Kepulauan Solomon, dan Bismark. Tahun 1871 tanaman ini mulai dimasukkan ke Jawa tepatnya ke Kebun Raya Bogor (Atmosuseno 1998). Sejak itulah sengon mulai ditanam diberbagai tempat di Jawa sebagai tanaman pelindung perkebunan.
Sengon dapat tumbuh dengan kisaran suhu 20─33oC dalam pertumbuhannya. Namun demikian, suhu optimum yang diperlukan oleh sengon berkisar antara 22─29oC. Sengon tumbuh mulai dari pantai hingga ketinggian 1600 mdpl, dengan ketinggian optimum 0─800 mdpl. Sengon dapat beradaptasi dengan iklim monsoon dan lembab dengan curah hujan 200─2700 mm/tahun dengan bulan kering sampai 4 bulan (Atmosuseno 1998).
4 pengamatan di lapangan, tanaman sengon dapat tumbuh baik pada tanah regosol, aluvial, dan latosol. Tanah-tanah tersebut bertekstur lempung berpasir atau lempung berdebu dengan kemasaman tanah sekitar pH 6─7 (Santoso 1992). Sengon merupakan salah satu spesies yang paling cepat tumbuh di dunia, mampu tumbuh 8 m/tahun dalam tahun pertama penanaman.
2.1.3. Ciri Morfologi
Tinggi pohon sengon dapat mencapai 40 m dengan tinggi bebas cabang 20 m. Pohon sengon memiliki ciri-ciri, yaitu tidak berbanir, kulit licin, berwarna kelabu muda, bulat agak lurus. Diameter pohon dewasa bisa mencapai 100 cm atau lebih. Tajuk berbentuk perisai, jarang selalu hijau. Daun majemuk, panjang dapat mencapai 40 cm, terdiri dari 15─25 helai daun. Daunnya kecil-kecil dan mudah rontok. Bunga tersusun dalam bentuk malai. Bunganya kecil sekitar 0,5─1 cm, berwarna putih kekuning-kuningan dan sedikit berbulu. Setiap kuntum bunga yang mekar berisi bunga jantan dan betina. Adapun cara penyerbukannya dibantu dengan perantaraan angin dan serangga.
Buah sengon berbentuk polong, pipih, tipis, dan panjangnya sekitar 6─12 cm. Setiap polong buah berisi 15─30 biji. Biji tersebut biasanya terlepas dari polongnya yang terbuka bila terlalu masak. Bentuk biji dari sengon ini mirip perisai kecil, dan jika sudah tua biji tersebut berwarna cokelat kehitam-hitaman, agak keras dan berlilin (Santoso 1992).
Perakaran sengon relatif menguntungkan dibandingkan akar pohon lainnya. Akar tunggangnya cukup kuat menembus ke dalam tanah. Semakin besar pohonnya semakin dalam akar tunggangnya menembus ke dalam tanah. Sementara itu, akar rambut dari pohon sengon tidak terlalu besar, tidak rimbun, dan tidak menonjol ke permukaan tanah. akar rambut sengon justru dimanfaatkan oleh pohon induk untuk menyimpan nitrogen. Maka dari itu, tanah disekitar pohon sengon akan menjadi subur (Santoso 1992).
2.1.4. Kegunaan dan Manfaat
5 industri. Adapun pohon sengon memiliki karakteristik masa tebang pohon yang relatif cepat, teknik budi daya mudah diaplikasikan, dapat tumbuh di berbagai jenis tanah, kayu serba guna, dapat membantu menyuburkan tanah dan memperbaiki kualitas lahan (Siregar 2008).
Kayu sengon banyak diusahakan untuk berbagai keperluan dalam bentuk kayu olahan berupa papan-papan dengan ukuran tertentu sebagai bahan baku pembuat peti, papan penyekat, industri korek api, pensil, papan partikel, serta bahan baku industri pulp dan paper.
Daun sengon merupakan pakan ternak yang sangat baik dan mengandung protein tinggi. Selain sebagai pakan ternak, daun sengon yang berguguran akan menjadi pupuk hijau yang baik bagi tanah dan tanaman disekitarnya. Sementara itu, tajuk pohonnya yang rindang dapat dimanfaatkan sebagai pohon penaung di beberapa areal perkebunan (Siregar 2008). Kulit kayu sengon yang memiliki tannin dapat digunakan sebagai penyamak. Selain itu, sengon sebagai tanaman hutan juga memiliki jasa ekologis, diantaranya tegakan murninya yang dapat menahan erosi tanah dan air, serta berfungsi sebagai naungan pada penanaman campuran dengan teh, kopi dan cokelat (ICRAF 2006, diacu dalam Dwiyanti 2009).
2.2. Dasar─Dasar Genetika Molekuler
2.2.1. PCR (Polymerase Chain Reaction)
Kary B. Mullis mengembangkan Polymerase Chain Reaction (PCR) pada tahun 1983, untuk merevolusi metodologi dari biologi molekuler. PCR yaitu suatu proses yang didasarkan pada reaksi enzimatik in-vitro dalam amplifikasi DNA pada target tertentu dengan cara mensintesis molekul DNA baru yang berkomplemen dengan molekul DNA target dengan bantuan enzim dan oligonukleotida sebagai primer dalam suatu thermocycler.
6 Dalam uji PCR, terdapat tiga langkah temperatur terkendali yang dapat dilihat, dan siklus dari proses PCR ini dapat diulang berkisar antara 25─50 siklus. Menurut Bernard (1998) diacu dalam Siregar et al. (2008), PCR merupakan suatu teknik untuk memperbanyak potongan DNA spesifik. Ada empat komponen utama yang dibutuhkan untuk melakukan proses PCR yaitu: (i) DNA target, (ii) primer, (iii) DNA polymerase d