PENGKLONAN DAN KARAKTERISASI GEN
GST12
DARI
KEDELAI KULTIVAR LUMUT DAN SLAMET
Oleh:
Syahnada Jaya Sy
G34102011
DEPARTMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
SYAHNADA JAYA SY. Pengklonan dan Karakterisasi gen
GST12 dari Kedelai Kultivar Lumut dan Slamet. Dibimbing oleh UTUT WIDYASTUTI SUHARSONO dan ARIS TJAHJOLEKSONO.GST12 diketahui sebagai salah satu gen yang berperan dalam respon ketahanan terhadap cekaman Alumunium (Al). cDNA LGST12 dan SGST12 telah berhasil diisolasi dari akar tanaman kedelai kultivar Lumut dan Slamet. Hasil penelitian sebelumnya diketahui bahwa pada kultur air pH rendah dan Al tinggi LGST12 tidak terinduksi sedangkan SGST12 terinduksi. Penelitian ini bertujuan untuk mengkarakterisasi kedua gen tersebut sebagai langkah awal eksplorasi dan isolasi gen. cDNA kedua kultivar telah dicek keberadaannya kemudian diklon ke dalam pGEM®-T easy. Plasmid rekombinan dalam Escherichia coli galur DH5 telah berhasil diperoleh. Pengurutan cDNA LGST12 dan SGST12 dengan DNA sekuenser ABI Prism Model 310 yang masing-masing menghasilkan 722 pb dan menyandikan 237 asam amino. Analisis kesejajaran lokal kedua cDNA terhadapbagian GST12 (AF243367)yang berada di GenBank menunjukan persentase kesamaan sebesar 85 % untuk LGST12 dan 92 % untuk SGST12 dengan Score Bits secara berurutan 908 dan 1146 dan E-value kedua cDNA tersebut 0 (nol). Hasil penelusuran daerah asam amino terkonservasi menunjukkan bahwa cDNA GST12 memiliki daerah domain asam amino terkonservasi GST tipe GST_N_Tau sedangkan cDNA SGST12 selain memiliki daerah domain asam amino terkonservasi GST_N_Tau juga memiliki daerah domain asam amino terkonservasi GST_C_Tau. Berdasarkan penjajajaran asam amino cDNA LGST12 dan SGST12 terhadap GST12
dari beberapa spesies menggunakan MAFFT v6. 500a diperoleh pohon filogenetik yang menunjukkan kedekatan kekerabatan kedua cDNA terhadap GST12 dari Glycine max (AF243367).
ABSTRACT
SYAHNADA JAYA SY. Cloning and Characterization of GST12 Gene from Soybean Lumut Cultivar and Slamet. Supervised by UTUT WIDYASTUTI SUHARSONO and ARIS TJAHJOLEKSONO.
GST12 is one of genes that has important role in Aluminum (Al) stress. cDNA LGST12 and
SGST12 are already isolated from soybean root of Lumut cultivar (cv.) and Slamet. The recent research reported that LGST12 was not induced and SGST12 was induced by low pH and Al stressed water culture system. The objection of this research is to characterize GST12 genes from Lumut cv. and Slamet soybean which have difference response against both of stress. Both of cDNA were re-confirmed by specific primers PCR. Thus, The PCR products are inserted into pGEM®-T easy and the plasmid was successfully introduced into E. coli DH5. Sequencing of
LGST12 and SGST12 by using ABI Prism 310 automated DNA sequencer showed that LGST12
and SGST12 have 722 base pairs encoding 237 amino acids. Basic local alignment analysis based on nucleotide with other GST12 from GenBank showed that LGST12 is 85 % identical and
SGST12 is 92% identical to part of GST12 from Glycine max (AF243367) with bit score 908 and 1146 respectively, and E-value is 0 (zero). LGST12 has big similarity with GST_N_Tau conserved domain type only, however cDNA SGST12 does not only has big similarity on GST_N_Tau, but it also has big similarity on GST_C_Tau conserved domain type. The amino acids alignment analysis is used for comparing the sequences with some species using the MAFFT
PENGKLONAN DAN KARAKTERISASI GEN
GST12
DARI
KEDELAI KULTIVAR LUMUT DAN SLAMET
Skripsi
Sebagai salah satu syarat memperoleh gelar Sarjana Sains
pada Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
Oleh :
Syahnada Jaya Sy
G34102011
DEPARTMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi
: Pengklonan
dan Karakterisasi Gen
GST12
dari Kedelai
Kultivar Lumut dan Slamet
Nama
: Syahnada Jaya Sy
NIM
:
G34102011
Menyetujui:
Mengetahui :
Tanggal Lulus:
Pembimbing I,
Dr. Ir. Utut Widyastuti Suharsono
NIP 131851279
Pembimbing II,
Dr. Aris Tjahjoleksono, DEA
NIP131664392
Dekan Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
PRAKATA
Alhamdulillah, puji dan syukur penulis sampaikan kepada Allah SWT karena hanya dengan rahmat, hidayah dan izin-Nya skripsi ini dapat diselesaikan. Penelitian dilakukan mulai bulan Februari 2006 sampai Mei 2007, di Laboratorium BIORIN (Biotechnology Research Indonesia-The Netherlands), Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB (PPSHB-IPB), Kampus IPB Darmaga, Bogor.
Ucapan terima kasih penulis sampaikan kepada Dr. Utut Widyastuti, MSi. dan Dr. Aris Tjahjoleksono, DEA. selaku pembimbing yang telah memberi arahan, bantuan dan waktu selama penelitian. Terima kasih juga penulis sampaikan kepada Dr. Aris Tri Wahyudi, MSi. atas masukan untuk perbaikan karya ilmiah ini dan Dr. Suharsono DEA, selaku kepala PPSHB-IPB yang telah memberikan kesempatan untuk melakukan penelitian di lab BIORIN serta kepada seluruh staf dan karyawan atas bantuan yang telah diberikan selama penulis melakukan penelitian. Terima kasih kepada pak Mulya dan mbak Pepi yang telah turut memberikan arahan dan bantuan selama di laboratorium.
Ungkapan terima kasih dan rasa hormat yang mendalam juga penulis sampaikan kepada keluarga besar Abu Senin dan Djamaluddin tercinta: Bapak Syaifullah AS, Ibu Adna Huda Djamal, Kak Syafrianto Sy dan keluarga, Ayuk Syafrida Sy, S E. Ak dan keluarga. dan Ayuk Syafrianti Sy, Spd I yang selalu memberikan doa, bantuan, perhatian dan kasih sayang. Juga kepada para sahabat yang selalu memberi inspirasi dan semangat: Rafli, Odan, Dwi, Kakak yang selalu mememberi inspirasi dan penyedia jurnal yang sedang berjuang di negeri orang (Bro Fadly (Korsel-SNU), Abang (Finlandia-Erasmus Mundus), Mas Aqil (Australia), Mas Jaya (Jerman-Erasmus Mundus), Mas Anuraga (Jerman-DAAD), Bro Marwan (Italia-Bologa), Uda Reflinur (Korsel-SNU) teman-teman SR dan BPA angkatan 2004-2005, teman-teman BEM TPB 2002, teman-teman Natrium, teman-teman PA 2004, teman-teman Biologi 39, teman-teman Al-izzah, Kodama-kun, Romone, Kenji-kun, Amirouche, Khalid, Haidir serta adik-adik asrama TPB angkatan 2004/05 dan 2005/06, juga ditujukan kepada teman-teman kelompok penelitian padi dan penyakit blast (Mbak Ela, Mbak Rina, Nindya, Dona, Zahro), Melastoma (Uzy, Mas Firda, Mas Yasier, Mbak Ulfa, Mbak Agustin, Mbak Zendi, Pak Hadi, Mbak Ratna), Kedelai (Mas Huda, Mbak Muti, Mbak Kiki, Mbah, Ibu Seh), keragaman Jarak dan Nanas (Ibu Panca dan Ibu Puji) dan Kultur jaringan Cabai, Kedelai dan Kelapa Sawit (Mbak Budi, Hakiim, Popi, Ammay, Sari) serta para kandidat Doktor (Pak Muzuni, Ibu Sri Lis, Mbak Hanum, Pak Ulung) yang telah meluangkan waktunya untuk saling menasehati, diskusi sehingga penelitian ini menyenangkan.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2007
Syahnada Jaya Sy
RIWAYAT HIDUP
Penulis dilahirkan di Kabupaten Rejang Lebong, Provinsi Bengkulu pada tanggal 16 Juli 1983 dari ayah Syaifullah AS dan ibu Adna Huda Djamal. Penulis merupakan putra keempat dari empat bersaudara.
Pada tahun 2002 penulis lulus dari SMU I Curup Bengkulu. Pada tahun yang sama penulis diterima di Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI).
Pada tahun ajaran 2002/2003 penulis aktif di kelembagaan Badan Eksekutif Mahasiswa Tingkat Persiapan Bersama (BEM-TPB) sebagai sekretaris umum dan kelembagaan Asrama TPB-IPB. Penulis dipercaya menjadi ketua penyambutan mahasiswa baru tingkat IPB pada tahun ajaran 2003/2004 serta ketua penyambutan mahasiswa baru tingkat Departemen Biologi pada tahun 2004/2005. Penulis mendapat beasiswa kerja Senior Residence asrama TPB-IPB selama dua periode pada tahun ajaran 2004/2005 dan 2005/2006. Penulis sekarang aktif di yayasn Bulan Sabit Merah Indonesia (BSMI) cabang Bogor. Penulis pernah menjadi finalis presentasi dan poster dalam ajang kompetisi Pekan Ilmiah Mahasiswa Nasional XIX (PIMNAS) Malang untuk kategori Program Kreatifitas Mahasiswa Teknologi (PKM-T). Selain itu penulis juga aktif mengikuti berbagai pelatihan ilmiah dan kepemimpinan. Pada bulan Juni-Agustus 2005 penulis melaksanakan praktik lapang dengan judul Pembenihan Ikan Bawal Air Tawar (Colossoma macropomum) Di PT Agung Fish Farm Kecamatan Cibungbulang, Kabupaten Bogor. Pada tahun 2004/2005 penulis aktif sebagai asiten Biologi Dasar untuk mahasiswa TPB dan Genetika Dasar pada semester ganjil tahun ajaran 2006/2007 untuk mahasiswa Biokimia serta matrikulasi mahasiswa pasca sarjana Beasiswa Utusan Daerah (BUD) Departemen Agama (DEPAG) pada tahun ajaran 2007/2008.
DAFTAR ISI
Halaman
DAFTAR GAMBAR ... vii
DAFTAR LAMPIRAN ... vii
PENDAHULUAN
Latar Belakang ... 1
Tujuan ... 1
BAHAN METODE Waktu dan tempat ... 2
Bahan ... 2
Metode ... 2
Penentuan Konsentrasi gen GST12 dari Stok Produk PCR ... 2
Pengklonan GST12 ... 2
Seleksi E. coli DH5α yang mengandung vektor rekombinan ... 3
Analisis gen sisipan ... 3
Pengurutan DNA dan analisis urutan DNA ... 3
HASIL DAN PEMBAHASAN
Hasil ... 4Konfirmasi GST12 dari Stok cDNA ... 4
Pengklonan GST12 ... 4
Pengurutan DNA dan analisis urutan DNA ... 6
Pembahasan ... 6
SIMPULAN ... 8
UCAPAN TERIMA KASIH ... 8
DAFTAR PUSTAKA ... 8
LAMPIRAN ...
10DAFTAR GAMBAR
Halaman
1 Peta fisik plasmid pGEM®-T Easy yang digunakan sebagai vektor pengklonan ... 2 2 cDNA LGST12 (a); cDNA SGST12 (b). 1= LGST12 berukuran sekitar 720 pb, 2= SGST
12 berukuran sekitar 720 pb. Elektroforesis pada 1.5 % (b/v) agarosa. M= 50 ng; M1= 100 ng; M2= marker 100 bp (RBC). Tanda panah: gen GST12 ... 4
3 Penampilan koloni E. coli DH5 yang diduga membawa plasmid rekombinan (koloni putih)
pada media seleksi (ampisilin, X-gal, dan IPTG) ... 4 4 PCR koloni putih E. coli yang membawa GST12. Elektroforesis pada 1.5 % (b/v) agarosa.
Kultivar Lumut: dari 5 koloni putih hanya dijumpai 1 koloni positif pembawa plasmid rekombinan (a); kultivar Slamet: dari 6 koloni putih dijumpai 5 koloni positif pembawa plasmid rekombinan (b). M= Marker 100 bp (RBC). Tanda panah: gen GST12 ... 4 5 Elektroforesis plasmid utuh rekombinan (a). Elektroforesis produk PCR GST12 dari plasmid
utuh rekombinan (b). 1= LGST12, 2= SGST12. Elektroforesis pada 1.5% (b/v) agarosa. M= Marker 50 ng; M1= Marker 100 bp (RBC). Tanda panah: gen GST12 ... 4 6 Penjajaran (alignment) asam amino LGST12 dan SGST12 terhadap GST12 Glycine max
(AF243367) ... 5 7 Pohon filogenetik berdasarkan urutan asam amino GST dari berbagai spesies ... 5 8 Perbandingan daerah domain asam amino terkonservasi antara LGST12danSGST12
terha-dapGST12 Glycine max (AF243367) dari GenBank ... 6
DAFTAR
LAMPIRAN
Halaman
1 Output sekuensing dengan menggunakan mesin sekuensing ABI PRISM 310 Genetic Analyzer ... 11 2 Hasil sekuensing dua arah menggunakan primer GST12F dan GST12R menghasilkan urutan nukleotida GST12 Glycine max (AF243367), cDNA LGST12 dan SGST12 leng-
PENDAHULUAN
Latar Belakang
Kedelai merupakan salah satu komoditi palawija yang digunakan secara luas di Indonesia sebagai sumber protein nabati. Produksi kedelai tahun 2006 (angka sementara) sekitar 749.04 ribu ton biji kering, mengalami penurunan sekitar 59.32 ribu ton (7.34 %) dibandingkan dengan produksi tahun 2005 (BPS 2006). Penurunan produksi kedelai tersebut menurut BPS (2006) disebabkan oleh adanya penyusutan luas panen dan penurunan produktivitas tanaman kedelai. Luas panen kedelai tahun 2006 menurun 39.93 ribu hektar (6.42 %) dibandingkan dengan tahun 2005. Sementara produktivitas tahun 2006 turun sebesar 0.13 kuintal/hektar (1.00 %) dibandingkan dengan tahun 2005. Bahkan pada tahun 2006 Indonesia mengimpor kedelai sebesar 1 370 000 ton (USDA 2007).
Usaha jangka panjang untuk mening-katkan produksi kedelai dapat dilakukan melalui ekstensifikasi dan perbaikan genetik (Suharsono 2002). Ekstensifikasi telah lama dilakukan di Indonesia, namun belum mem-berikan hasil yang memuaskan. Tanah asam menjadi salah satu faktor penghambat ekstensifikasi. Menurut Mulyani et al. (2003), Indonesia masih memiliki lahan potensial untuk perluasan areal tanam kedelai yang tersebar di Sumatera, Kalimantan, dan Papua. Lahan seluas 18.2 juta hektar sebagian besar bersifat asam dengan kandungan Alumunium (Al) tinggi sehingga masih belum dapat dimanfaatkan sebagai lahan produktif hingga diperolehnya kultivar tanaman yang dapat tumbuh dengan baik pada jenis tanah tersebut.
Pada tanah asam (pH < 5) ion Al3+ men-jadi faktor penghambat pertumbuhan akar tanaman kedelai sehingga mempengaruhi pertumbuhan kedelai secara keseluruhan (Kochian 1995, Matsumoto 2000, Srimulyani 2007). Sifat racun ion-ion Al tersebut juga dapat berupa penghambatan sintesis RNA (Matsumoto et al. 1997), peningkatan perok-sidasi fosfolipid dan protein pada membran (Cakmak & Horst 1991, Yamamoto et al. 2001). Ion-ion Al juga menyebabkan peng-hambatan influks ion Ca2+ sehingga meng-ganggu homeostatis Ca2+ pada apeks akar (Rengel 1992).
Pada Arabidopsis thaliana salah satu gen yang berhubungan dengan sistem pertahanan terhadap cekaman Al adalah gen GST yang menyandikan Glutathione S-Transferase (Richard et al. 1998). Glutathione S-Transferase (GST) merupakan kelompok enzim sitoplasmik. Setiap sub unit GST merupakan suatu dimerik dengan massa molekul sekitar 24-30 kD. Gen GST dapat dibagi menjadi tiga tipe berdasarkan kemiripan sekuennya yaitu GmGST (Glycine max Glutathione S-transferase) tipe I, GmGST tipe II dan GmGST tipe III (Droog et al. 1995; Board et al. 1997; McGonigle et al. 2000). GmGST tipe III atau kelas Tau (Hock & Elstner 2005) memiliki anggota paling besar dimana GmGST 12 termasuk dalam kelas ini. Berdasarkan hasil uji Expressed Sequence Taq (EST) diketahui bahwa GmGST tipe III diekspresikan paling melimpah di bagian akar dan daun kedelai dibandingkan dengan GmGST tipe I dan II (McGonigle et al. 2000).
Pada mamalia dan beberapa tanaman, gen GST ditemukan berperan dalam detoksifikasi metabolit yang disebabkan oleh cekaman oksidatif melalui peredaman molekul-molekul reaktif dengan menambah-kan GSH (Wilce et al. 1994; Sheehan et al. 2001). GST juga terlibat dalam menanggapi berbagai cekaman kimia dan berperan dalam sistem antioksidasi (Ulmasov et al.
1995). Selain fungsi endogenous tersebut, GST juga memiliki peran penting dalam proses detoksifikasi herbisida yang sebagian besar merupakan kelompok senyawa-senyawa xenobiotik (Dixon et al. 1998).
cDNA GST12 kedelai kultivar (var.) Lumut yang bersifat peka (Sunarto 1995) dan var. Slamet yang bersifat toleran terhadap cekaman Al (Jusuf et al. 1999) telah berhasil diisolasi dari bagian akar. Kedua var. tesebut memiliki pola ekpsresi GST12 yang berbeda selama diberi perlakuan cekaman Al dan pH rendah di dalam kultur air (Mashuda 2006; Sawitri 2007). LGST12
(gen GST12 yang diisolasi dari akar kedelai var. Lumut) diinduksi pada jam ke-8 hingga jam ke-72 dalam semua jenis cekaman Al (Mashuda 2006), sedangkan SGST12 (gen GST12 yang diisolasi dari akar kedelai var. Slamet) tidak terinduksi (Sawitri 2007). Oleh karena itu untuk melihat lebih jauh perbedaan yang ada pada gen GST12 dari kedua var. Lumut dan Slamet perlu dilakukan pengklonan dan karakterisasi.
Tujuan
BAHAN DAN METODE
Waktu dan Tempat
Penelitian dilaksanakan mulai Februari 2006 sampai dengan Mei 2007 di laborato-rium
Biotechnology Research Indonesia-The Netherlands (BIORIN) dan Genetika Molekular dan Selular Tanaman (GMST), Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB) IPB Darmaga.
BAHAN
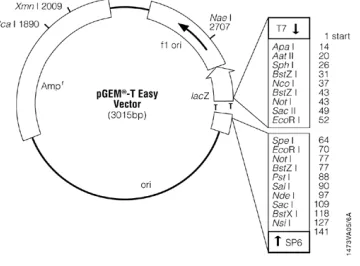
Produk PCR LGST12 (gen GST12 yang diisolasi dari akar kedelai var. Lumut) (Sawitri 2007) dan produk PCR serta cDNA SGST12 (gen GST12 yang diisolasi dari akar kedelai var. Slamet) (Mashuda 2006) yang keduanya diisolasi dari akar kedelai yang ditumbuhkan pada kultur air. Plasmid pGEM®-T Easy (Gambar 1) (Promega) digunakan sebagai vektor pengklonan dan E. coli galur DH5α sebagai inang vektor rekombinan.
Gambar 1 Peta fisik plasmid pGEM®-T Easy yang digunakan sebagai vektor pengklonan
Primer spesifik gen GST12 didesain berdasarkan GST12 (AF243367). Primer forward
(GST12F) dari gen GST12 terletak pada 8 nukleotida sebelum kodon awal
(5’CCATAGCAATGGCAGAGCAAG3’) dan primer reverse (GST12R) terletak pada 11 nukleotida sebelum kodon akhir sampai 6 nukleotida setelah kodon akhir (5’TAT-ATATCATTCTGTGGCAG3’) (Lampiran 2) digunakan sebagai alat verifikasi keberadaan gen
GST12 dari stok cDNA yang tersedia. Metode
Penelitian ini terdiri dari beberapa tahap yaitu penentuan kosentrasi gen GST12 dari stok cDNA. Pengklonan gen GST12 ke dalam plasmid pGEM®-T Easy, transformasi genetik E. coli DH5α dengan vektor rekombinan, analisis urutan nukleotida, dan deduksi asam amino.
Penentuan Konsentrasi gen GST12 dari Stok Produk PCR
cDNA LGST12 dan cDNA SGST12 digunakan sebagai cetakan PCR untuk konfirmasi keberadaan gen GST12. Komposisi PCR adalah 0.5 l cDNA LGST12 dan SGST12 (stok di lab BIORIN), 1 l 10x buffer Taq, 0.1 mM dNTP, 10 pmol primer GST12F, 10 pmol primer GST12R, 4% DMSO (Dimethyl Sulfoxide), 1 U enzim Taq DNA polymerase (Gen Scr Inc.) dan ddH2O hingga volume akhir mencapai 10 l. Kondisi PCR yang digunakan adalah 95 0C selama 5 menit untuk pra-PCR. Siklus PCR dilakukan sebanyak 37 kali dimulai dengan denaturasi pada suhu 94 0
C selama 30 detik, penempelan primer LGST12 55 0C selama 30 detik (52 0C untuk primer
SGST12), pemanjangan 72 0C selama 2 menit. Kemudian dilanjutkan dengan pasca-PCR pada suhu 72 0C selama 5 menit dan pendinginan dilakukan pada 15 0C selama 10 menit (Mashuda 2006; Sawitri 2007).
Pengklonan gen GST12 menggunakan prosedur promega (1996) yang dimodifikasi dengan cara mencampurkan 5 l 2x buffer rapid ligasi, 1 l vektor pGEM®-T Easy (10 ng), 1 l T4 DNA ligase (3 U/ l), dan 0.7 l (10 ng/ l) cDNA LGST12 (0.35 l (20ng/ l) cDNA SGST12). Kemudian diinkubasi pada suhu 4 0C. Untuk mendapatkan hasil pengklonan yang optimal jumlah insert (cDNA LGST12 atau SGST12) yang dibutuhkan dihitung dengan menggunakan rumus berikut:
Produk ligasi diintroduksikan ke dalam E. coli galur DH5α melalui transformasi dengan
prosedur Suharsono (2002). Transformasi diawali dengan pembuatan bakteri kompeten dengan membiakkan satu koloni E. coli DH5α dalam 2 ml LB (Lauria Broth) (1% (b/v) bakto tripton, 0.5% (b/v) bakto ekstrak khamir, 1% NaCl (b/v)) dan diinkubasi pada suhu 37 0C dan digoyang dengan kecepatan 250 rpm selama semalam di dalam inkubator bergoyang, ES-20 (Biosan). Kemudian sebanyak 100 l E. coli DH5α disubkulturkan dalam 30 ml LB dengan kondisi yang
sama hingga mencapai densitas bakteri 4-7 x 107 sel/ml (OD600 = 0.4-0.5). Sel bakteri dipanen dengan cara disentrifugasi pada kecepatan 3 000 rpm pada suhu 4 0C selama 10 menit dengan menggunakan tabung falkon 15 ml. Pelet yang diperoleh selanjutnya ditambahkan buffer transformasi TB (Transformation Buffer) (10 mM PIPES, 15 mM CaCl2.2H20, 250 mM KCl, 55 mM MnCl2.4H2O, pH 6.7) sebanyak 0.033 x volume larutan biakan. Pelet diresuspensi dengan menggunakan pipet dan diinkubasi dalam es selama 10 menit. Selanjutnya disentrifugasi dengan kecepatan 3 000 rpm pada suhu 4 0C selama 10 menit. Pelet yang diperoleh diresuspensikan kembali dengan penambahan buffer TB 41,25 l TB (0.5 x volume larutan biakan). Setelah benar-benar tersuspensikan kemudian ditambah 60 l DMSO dan diinkubasi di dalam es selama 10 menit sehingga diperoleh suspensi sel bakteri kompeten. Sebanyak 50 l suspensi sel kompeten ditambah dengan 10 l (50-100 ng) produk ligasi kemudian diinduksi dalam es selama 20-25 menit. Selanjutnya diberi perlakukaan kejutan panas 42 0C selama 45 detik dan segera dimasukan kembali ke dalam es selam 5 menit. Kemudian ditambah 100 l media YT (Yeast Tripton) (16 g/l bakto tripton, 10 g/l bakto ekstrak khamir, 5 g/l NaCl, pH 7.0). Setelah diinkubasi dalam inkubator bergoyang pada suhu 37 0C selama 20 menit, suspensi tersebut disebar secara merata pada media LB padat yang mengandung 100 mg/l ampisilin dan 10 l 100 mM IPTG (Isopropiltio-β-galaktosida) serta 50 l 2% (b/v) X-gal (5-bromo-4-choloro-3-indoly-β-D-galactoside), kemudian diinkubasi pada suhu 37 0C selama semalam.
Seleksi E. coli DH5α yang mengandung vektor rekombinan
E. coli DH5α yang mengandung vektor rekombinan diseleksi berdasarkan warna koloninya. Koloni putih yang tumbuh pada media yang mengandung ampisilin tersebut berpotensi sebagai koloni yang mengandung vektor rekombinan. Adanya sisipan gen GST12 di dalam plasmid rekombinan yang terdapat dalam koloni putih dideteksi secara cepat dengan melakukan PCR koloni. Pada PCR tersebut, sejumlah kecil koloni bakteri putih digunakan sebagai bagian dari campuran di dalam tabung PCR. Lisis sel bakteri untuk menghasilkan DNA cetakan terjadi di dalam tabung PCR. Untuk itu, koloni putih diambil dengan menggunakan tusuk gigi steril kemudian disuspensikan ke dalam tabung PCR yang telah berisi 6.5 l ddH2O. Kemudian tusuk gigi tersebut dioleskan pada media LA (Lauria agar) (1 g/l bakto tripton, 0.5 g/l bakto ekstrak khamir, 1 g/l NaCl, Agar 25g/l) + ampisilin (50 g/ml) sebagai replika koloni. Selanjutnya pada suspensi ditambah campuran PCR (komposisi dan kondisi PCR sama dengan tahap penentuan kosentrasi gen GST12 dari stok cDNA). Selanjutnya campuran dalam tabung PCR dipanaskan pada suhu 95 0C selama 10 menit dan diinkubasi pada suhu 15 0C selama 5 menit, kemudian diamplifikasi dengan mesin PCR.
Analisis gen sisipan
Plasmid pGEM®-T Easy rekombinan di dalam E. coli DH5α di isolasi dengan mengikuti prosedur Suharsono (2002). Satu koloni E. coli DH5α ditumbuhkan dalam 30 ml LB yang ditambah dengan 60 l ampisilin (50 g/ml) pada inkubator bergoyang (250 rpm) selama 16 jam pada suhu 37 0C. Kultur bakteri dimasukan ke dalam tabung falkon 15 ml, kemudian disentrifugasi (Jouan BR-4i) pada suhu 4 0C dengan kecepatan 4 000 rpm selama 10-15 menit. Endapan ditambah dengan 600 l larutan buffer suspensi sel (50 mM Tri-HCl pH 7.5 dan 10 mM EDTA). Suspensi sel ditambah dengan 400 l buffer lisis (0.2 M NaOH dan 1% SDS) dan dibolak-balik
vektor (ng) x ukuran insert (Kb)
Jumlah insert (ng)= 3/1
secara perlahan beberapa kali kemudian segera ditambah dengan 400 l larutan buffer netralisasi (1.32 M natrium asetat pH 4.8), divorteks dan disentrifugasi pada suhu 4 0C dengan kecepatan 10 000 rpm selama 10 menit. Supernatan diekstrak dengan penambahan PCI (fenol-kloroform-isoamilalkohol dengan perbandingan 25:24:1) sebanyak 1x volume supernatan. Kemudian divorteks dan disentrifugasi pada suhu 20 0C dengan kecepatan 10 000 rpm selama 10 menit. Fase jernih (bagian di atas PCI) diendapkan dengan penambahan natrium asetat 2 M pH 5.2 sebanyak 0.1x volume dan etanol absolut sebanyak 2x volume awal (fase jernih). Larutan diinkubasi pada suhu -20 0C selama 2 jam kemudian diendapkan dengan sentrifugasi 10 000 rpm pada suhu 4 0C selama 25 menit. Endapan (DNA plasmid) dibilas dengan menggunakan alkohol 70% (v/v) dengan bantuan sentrifugasi 10 000 rpm selama 10 menit pada suhu 4 0C. Endapan dikeringkan dan disuspensikan dengan ddH2O. Suspensi DNA palsmid ditambah RNAse (100 g/ml) sebanyak 0.1 x volume suspensi dan diinkubasi pada suhu 37 0C selama semalam. Kemudian ditambah dengan ddH2O hingga volume akhir menjadi 500 l. Plasmid hasil isolasi tersebut akan digunakan sebagai cetakan PCR untuk mendapatkan gen LGST12 dan SGST12 yang digunakan dalam analisis selanjutnya.
Pengurutan DNA dan analisis urutan DNA
Pengurutan DNA (DNA sequencing) dilakukan menggunakan DNA sequencer ABI PRISM 310 Genetic Analyzer Urutan nukleotida dari gen GST12 dianalisis kemiripan dan homologinya
dengan gen GST12 lainnya dengan menggunakan program
BLAST2(Basic_Local_Alignment_Search_Tools) (http://www.ebiac.uk/blast2). Analisis kesamaan dan filogenetik berdasarkan deduksi asam amino GST12 dari spesies lain dilakukan dengan menggunakan program MAFFT (Multiple Alignment Fast Fourier Transform) ver.5.8 (Katoh et al.
2005). Analisis daerah terkonservasi dan domain dilakukan dengan menggunakan program
conserved domain NCBI (www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb.cgi).
HASIL DAN PEMBAHASAN Hasil
Konfirmasi GST 12 dari Stok cDNA
Konfirmasi keberadaan GST12 dari stok cDNA dan produk PCR dari penelitian sebelumnya (Mashuda 2006; Sawitri 2007) dengan menggunakan primer spesifik GST12 (AF243367) menunjukan hasil positif, ukuran pita yang diperoleh sekitar 720 pb (Gambar 1).
M M1 M2 1 M M1 M2 1 2
(a) (b)
Gambar 2 cDNA LGST12 (a); cDNA SGST12 (b). 1= LGST12 berukuran sekitar 720 pb, 2=
SGST12 berukuran sekitar 720 pb. Elektroforesis pada 1.5% (b/v) agarosa. M= 50 ng; M1= 100 ng; M2= Marker 100 bp (RBC) Tanda panah: gen GST12
Pengklonan gen GST12
Setelah amplikon GST12 diligasikan dengan plasmid pGEM®-T Easy dan kemudian diintroduksikan ke dalam E. coli DH5α serta diinkubasi overnight, dijumpai dua jenis koloni yaitu koloni putih dan biru.
720pb
Koloni putih
Gambar 3 Penampilan koloni E. coli DH5α yang diduga membawa plasmid rekombinan (koloni
putih) pada media seleksi (ampisilin, X-gal, dan IPTG)
PCR koloni dilakukan untuk memastikan adanya sisipan amplikon di dalam beberapa koloni
E. coli putih yang tumbuh pada media seleksi. Koloni putih yang produk PCR-nya berupa amplikon yang berukuran sama dengan hasil konfirmasi keberadaan GST12 sebelum-nya yaitu sekitar 720 pb akan dipilih sebagai koloni yang akan digunakan pada proses analisi selanjutnya (Gambar 4).
M 1 2 3 4 5 M 1 2 3 4 5 6
(a) (b)
Gambar 4 PCR koloni putih E. coli yang membawa GST12. Elektroforesis pada 1.5% (b/v) agarosa. Kultivar Lumut: dari 5 koloni putih hanya dijumpai 1 koloni positif pembawa plasmid rekombinan (a); kultivar Slamet: dari 6 koloni putih dijumpai 5 koloni positif pembawa plasmid rekombinan (b). M= Marker 100 bp (RBC). Tanda panah: gen GST12
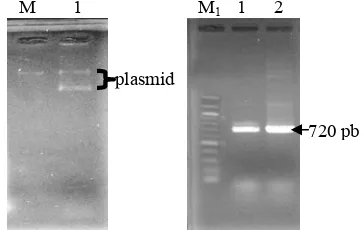
DNA plasmid rekombinan telah diisolasi dari koloni putih yang terpilih melalui PCR koloni. Keberadaan amplikon sisipan kembali dipastikan dengan PCR menggunakan plasmid hasil isolasi sebagai cetakan. Hasil elektroforesis menunjukkan hal yang sama dengan elektroforesis hasil PCR koloni (Gambar 4) yaitu menunjukkan amplikon yang berukuran sekitar 720 pb (Gambar 5). M 1 M1 1 2
720 pb 720 pb
Gambar 5 Elektroforesis plasmid utuh rekombinan (a). Elektroforesis produk PCR GST12 dari plasmid utuh rekombinan (b). 1= LGST12, 2= SGST12. Elektroforesis pada 1.5% (b/v) agarosa. M= Marker 50 ng; M1= Marker 100 bp (RBC). Tanda panah: gen
GST12
Pengurutan cDNA dan analisis urutan cDNA
Hasil sekuensing menunjukkan bahwa panjang cDNA dari kodon awal sampai kodon akhir kedua cDNA LGST12 dan SGST12 adalah 722 pb. Analisis cDNA dilakukan untuk melihat kesamaan dan kekerabatan LGST12 dan SGST12 terhadap gen GST12 koleksi GenBank. cDNA
LGST12 memiliki kesamaan sebesar 85 % dan SGST12 sebesar 92 % terhadap bagian GST12 dari
Glycine max (AF243367). Kedua sekuen gen tersebut memiliki nilai bit score (BS)di atas 50 bits
(908 dan 1146 bits) dan nilai expectation value (E-value) sebesar 0 (nol). Nilai BS dan nilai E-value merupakan nilai yang menunjukan signifikansi kemiripan sekuen (Claverie & Notredame 2003). Sekuen atau urutan nukleotida cDNA LGST12 dan SGST12 diterjemahkanmenjadi 237 asam amino. Urutan asam amino tersebut kemudian dianalisis kekerabatannya terhadap beberapa sekuen asam amino dari beberapa spesies yang tersedia di GenBank dengan menggunakan
(b) (a)
Gambar 6 Penjajaran (alignment) asam amino LGST12 dan SGST12 terhadap GST12 Glycine max (AF243367)
Keterangan: Size = 12 sequences × 76 sites; Method = NJ ÆConserved sites (76 aa) with un-rooted tree ; Model = JTT ; Alpha = ∞; Bootstrap re-sampling = 100 (50-1000)ÆThe number of sequences must be <1000 for Poisson model, or <100 for other models.
Keterangan: Size = 12 sequences × 76 sites; Method = NJ ÆConserved sites (76 aa) with un-rooted tree ; Model = JTT ; Alpha = ∞; Bootstrap re-sampling = 100 (50-1000)ÆThe number of sequences must be <1000 for Poisson model, or <100 for other models.
Gambar 7 Pohon filogenetik berdasarkan urutan asam amino GST dari berbagai spesies
MAFFT v6. 500a (Gambar 6). Hasil analisis ditampilkan dalam bentuk pohon filogenetik (Gambar 7).
Penelusuran daerah domain terkonservasi dilakukan dengan menggunakan program
conserved domain NCBI untuk daerah N-terminal dan C-terminal pada kelompok GST_Tau. Hasil penelusuran menunjukkan bahwa LGST12 hanya memiliki N terminal GST_Tau, sedangkan SGST12 selain memiliki GST_C_Tau juga memiliki GST_N_Tau (Gambar 8).
Pembahasan
Konfirmasi GST 12 dari Stok cDNA
Selain untuk memastikan keberadaan gen target yang akan diklon dan dianalisis, hasil
konfirmasi ini juga menunjukkan kosentrasi cDNA GST12 yang tersimpan dalam stok. Kosentrasi cDNA GST12 dari masing-masing stok didapatkan dengan cara membandingkan visualisasi kecerahan pita amplikon GST12 terhadap marker lamda ( ) pada gel elektroforesis. Hasil elektroforesis menunjukkan kosentrasi GST12 pada stok cDNA var. Lumut adalah 10 ng/ l sedangkan var. Slamet adalah 20 ng/ l.
Pengklonan gen GST12
Hasil seleksi koloni pada media seleksi yang mengandung antibiotik ampisilin, X-gal dan IPTG menunjukkan adanya koloni E. coli yang berwarna putih. Namun, tidak semua koloni putih membawa plasmid rekombinan (Gambar 4).
Plasmid pGEM®-T Easy selain sebagai vektor pembawa juga digunakan sebagai marker penyeleksi karena mengadung gen lacZ yang menyandikan β-galaktosidase (β-gal) yaitu suatu enzim yang dapat mendegradasi disakarida laktosa menjadi monosakarida glukosa dan galaktosa yang disertai dengan perubahan warna media. X-gal adalah senyawa khromogenik yang memiliki struktur mirip dengan laktosa, media akan menjadi biru ketika β-gal memotong struktur X-gal tersebut. Amplikon GST12 menyisip pada gen lacZ, menyebabkan gen lacZ tidak dapat diekspresikan sehingga β-gal tidak dihasilkan dan koloni E. coli berwarna putih (tidak menghasilkan perubahan warna menjadi biru). Hal sebaliknya terjadi jika amplikon tidak menyisip pada gen lacZ maka koloni E. coli akan menjadi biru karena gen LacZ diekspresi menjadi β-gal.
Pengurutan cDNA dan analisis urutan cDNA
Sekuensing dua arah yang menggunakan primer GST12F dan GST12R menghasilkan urutan nukleotida cDNA LGST12 dan SGST12 lengkap, dimulai dari kodon awal ATG yang merupakan bagian dari primer GST12F dan kodon akhir TGA yang merupakan bagian dari primer GST12R
(Lampiran 1 dan 2).
Setelah sekuen LGST12 dan SGST12 diterjemahkan menjadi asam amino (Lampir-an 3) dengan menggunakan program expasytranslate tool (http://www.expasy.ch/cgi-bin/
dna_aa). Dengan menggunakan program conserved domain NCBI (Marchel-Bauer et al. 2007; www.ncbi.nlm.nih.gov/Structure/cdd
/wrpsb.cgi), sekuen LGST12 dan SGST12 memiliki daerah domain terkonservasi yang berbeda. Program ini berkerja mirip dengan sistem kerja program BLAST, namun berbeda pada basis data yang digunakannya. Pada program ini basis data yang digunakan berupa koleksi daerah domain terkonservasi protein yang telah didaftarkan dalam database conserve domain NCBI.
Protein LGST12 hanya direkomendasikan untuk dimasukkan ke dalam GST tipe GST_N_Tau (cd03058) saja. Menurut Dixon et al. (2003), daerah domain terkonservasi GST tipe ini terdiri atas 3 feature spesifik yaitu GSH binding site (G-site), dimer interfa, dan C-terminal. Sedangkan
Gambar 8 Perbandingan daerah domain asam amino terkonservasi antara LGST12 dan SGST12 terhadapGST12 Glycine max (AF243367) dari GenBank
GST12 ( Glycine max) (EM_PL:AF243367 98 238 60 80 1 LGST12 10 80 SGST12
10 85 120 220
GST_N_Tau GST_C_Tau
GST12 ( Glycine max) (EM_PL:AF243367 98 238 60 80 1 LGST12 10 80 SGST12
10 85 120 220
GST_N_Tau GST_C_Tau
237
237
GST12 ( Glycine max) (EM_PL:AF243367 98 238 60 80 1 LGST12 10 80 SGST12
10 85 120 220
GST_N_Tau GST_C_Tau
GST12 ( Glycine max) (EM_PL:AF243367 98 238 60 80 1 LGST12 10 80 SGST12
10 85 120 220
GST_N_Tau GST_C_Tau
237
protein SGST12 selain direkomendasikan untuk dimasukan ke dalam GST tipe GST_N_Tau (cd03058) juga direkomedasikan untuk dimasukkan ke dalam GST_C_Tau (cd03185). GST_C_Tau juga terdiri dari 3 feature spesifik yaitu dimer interfa, substrat (H-site), dan N-terminal (Riechers et al. 1997).
GST_N_Tau pada protein LGST12 dijumpai pada posisi asam amino 10 sampai urutan ke-80. Sedangkan pada SGST12, GST_N_Tau dijumpai pada posisi yang hampir sama dengan LGST12 yaitu pada asam amino ke-10 sampai ke-85 (Gambar 8). Hal tersebut bersesuaian dengan laporan Mannervik dan Danielson (1998), yang menyatakan bahwa asam amino yang berada pada posisi 60-80 merupakan daerah yang sangat terkonservasi. Selain itu, daerah ini juga mengandung katalis tirosin, serin atau sistein yang akan berinteraksi dengan grup tiol GSH (Dirr et al. 1994). Menurut Dixon et al. (1998), daerah ini merupakan sisi pengikatan spesifik untuk GSH atau disebut G-site yang terbentuk dari kelompok residu asam amino yang konservatif di dalam domain ujung polipeptida. Dari penjelasan tersebut dapat disimpulkan bahwa kedua protein LGST12 dan protein SGST12 masing-masing memiliki G-site. Sedangkan GST_C_Tau yang hanya direkomendasikan untuk SGST12 dijumpai pada posisi asam amino ke-120 sampai 220 (Gambar 8). Daerah tersebut menurut Dixon et al. (1998) merupakan situs pengikatan substrat hidrofobik H-site yang strukturnya paling bervariasi dan terbentuk dari residu domain ujung karboksil; sehingga dapat disimpulkan bahwa protein LGST12 dan protein SGST12 berbeda dalam hal keberadaan GST_C_Tau-nya. Perbedaan tersebut kemungkinan akan menyebabkan adanya perbedaan kelengkapan feature untuk menjalankan fungsi GST12 secara utuh. Karena menurut Dixon et al.
(1998), hanya GST dari kelas yang sama saja yang akan membentuk dimer, dimana dimer tersebut terdiri dari sisi katalik yang tersusun dari dua komponen yaitu G-site dan H-site. Perbedaan ini diduga menjadi faktor penting penyebab perbedaan respon kedua var. kedelai terhadap cekaman Al dan pH rendah di lapangan.
Hasil analisis kesejajaran lokal dengan menggunakan BLAST2-NCBI menunjukkan bahwa cDNA LGST12 dan SGST12 memiliki nilai kemiripan yang tinggi terhadap bagian GST12 dari
Glycine max (AF243367). Tingkat keyakinan nilai kemiripan tersebut selain ditunjukkan oleh nilai persentase kemiripan yang tinggi juga harus disertai dengan nilai BS dan E-value yang dimiliki kedua sekuen. Menurut Claverie dan Notredame (2003) nilai BS suatu sekuen harus di atas 50 bits
agar dapat diyakini tingkat kemiripannya terhadap sekuen pembanding. Sedangkan E-value adalah nilai pengulangan pengurutan hingga diperoleh pengurutan terbaik. Jika suatu sekuen memiliki nilai E-value semakin mendekati nilai 0 (nol) maka akan semakin dapat disimpulkan kemiripannya terhadap pasangan pengurutannya. Sebaliknya, jika suatu sekuen memiliki nilai E-value diatas 0.001 maka pengurutan sekuen tersebut sulit untuk disimpulkan kemiripannya. Hasil serupa diperoleh dalam analisis kesamaan dengan menggunakan MAFFT berdasarkan nukleoti-da dan asam amino. cDNA LGST12 dan SGST12 memiliki kedekatan kekerabatan dengan gen GST12 dari kedelai (Glycine max) yang telah didaftarkan di dalam GenBank bahkan terhadap beberapa sekuen gen yang telah dipatenkan. Berdasarkan kedekatan kekerabatan dan kesamaan ini, dapat diduga bahwa cDNA LGST12 dan SGST12 memiliki peran yang sama dengan gen-gen GST12 yang telah dilaporkan sebelumnya yaitu menyan-dikan protein yang memiliki peranan dalam detoksifikasi herbisida, cekaman kimia (dalam hal ini berupa cekaman Al), dan dalam sistem antioksidasi.
SIMPULAN
cDNA LGST12 dan cDNA SGST12 memiliki kesejajaran lokal yang tinggi dengan bagian gen
GST12 dari Glycine max (AF243367). Analisis kesamaan asam amino menunjukkan bahwa protein LGST12 dan protein SGST12 memiliki kedekatan kekerabatan dengan protein GST12
Glycine max meskipun memiliki perbedaan dalam hal letak, jumlah dan komposisi asam amino untuk masing-masing domain. Protein LGST12 direkomendasikan untuk dimasukkan ke dalam daerah domain terkonservasi tipe GST_N_Tau saja sedangkan SGST12 selain direkomendasikan untuk dimasukkan ke dalam daerah domain terkonservasi tipe GST_N_Tau juga ke dalam daerah domain terkonservasi tipe GST_C_Tau.
SARAN
UCAPAN TERIMA KASIH
Terima kasih penulis ucapkan kepada Proyek Hibah Bersaing XII Dirjen Dikti atas nama Dr. Utut Widyastuti, MSi. yang telah membiayai penelitian ini serta Bogor International Club yang juga turut membantu.
DAFTAR PUSTAKA
Board PG, Baker RT, Chelvanayagam G, Jermiin LS. 1997. Zeta, a novel class of glutathione S-transferases in a range of species from plants to humans. Biochem J 328:929-935.
[BPS] Badan Pusat Statistik. 2006. Harvested area, yield rate and production of soybean by province, 2006. Jakarta: BPS.
Cakmak I, Horst WJ. 1991. Effect of aluminum on lipid peroxidation, superoxide dismutase, catalase and peroxidase activities in root tips of soybean (Glycine max). Physiol Plant
83:463-468.
Claverie JM, Notredame C. 2003. Bioinformatics For Dummies. Willey Publishing Inc. New York.
Dirr HW, Reinemer P, Huber R. 1994. X-ray cristal structure of cytosolic glutathione S-transferases implications for protein architecture, substrate recognition and catalytic function. Eur J Biochem 220:645-661.
Dixon DP, Cole DJ, Edwards R. 1998. Glutathion-mediated detoxification system in plant. Curr Opin Plant Biol 1:258-266.
Dixon DP, McEwen AG, Lapthorn AJ, Edwards R. 2003. Forced evolution of a herbicide detoxifying glutathione transferase. J Biol Chem 26:23930-23935.
Droog FNJ, Hooykes PJJ, van der Zaal BJ. 1995. 2,4-Dichlorophenoxyacetic acid and related chlorinated compounds inhibit two auxin-regulated type III tobacco glutathione S-transferases. Plant Mol Biol 42:79-92.
Hock B, Elstner EF editor. 2005. Plant Toxicology (fourth edition). Marcell Dekker. New York. Jusuf M, Suharsono, Sopandie D. 1999. Molecular biology of soybean tolerance to aluminum
stress. Report of Graduated Team Research Grant. Urge Project, Batch II. Jakarta: Directorate General of Higher Education.
Katoh K, Kuma K, Toh H, Muyata T. 2005. MAFFT version 5: improvement in accuracy of multiple sequence alignment. Nuc Ac Res 2:511-518.
Kochian LV. 1995. Cellular mechanism of aluminum toxicity and resistance in plants. Annu Rev Plan Physiol Plant Mol bio 46:237-260.
Ma JF. 2000. Role of organic acid in detoxcification of aluminum in higher plants. Plant Cell Physiol 41:383-390.
Mannervik B, Danielson UH. 1998. Glutathione transferase-structure and catalytic activity. CRC
Crit Rev BiochemMol Biol 23:283-337.
Marchler-Bauer A et al. 2007. CDD: a conserved domain database for interactive domain family analysis. Nucleic Acids Res 35:D237-40.
Mashuda. 2006. Ekspresi gen Gα pada kedelai Slamet yang mendapat cekaman alumunium [tesis]. Bogor: Program Pasca Sarjana, Institut Pertanian Bogor.
Matsumoto H. 2000. Cell biology of aluminum toxicity and tolerance in higher plants. Int Rev Cytol 200:1-46.
Matsumoto H, Morimura S, Takahashi E. 1997. Binding of aluminum to DNA in pea root nuclei.
Plant Cell Physiol 18:987-993.
McGonigle B, Keeler SJ, Lau S-MC, Koeppe MK, O’keefe DP. 2000. A genomic approach to the comprehensive analysis of the Glutathione S-transferases gene family in soybean and maize.
Plant Physiol 124:105-1120.
Mulyani A, Hikmatullah, Subagyo H. 2003. Karakteristik dan potensi tanah masam dan kering di Indonesia. Prosding Simposium Nasional Pemberdayaan Tanah Masam Buku I. Bandar Lampung 29-30 September 2003. Puslitbang Tanah dan Agroklimat Balitbang Deptan Bogor.
Promega. 1996. Protocol and application guide. Ed. Ke-3. USA: Promega Co.
Rengel Z. 1992. Disturbance of cell Ca2+ homeostasis as a primary trigger of Al toxicity syndrome.
Riechers DE, Irzyk GP, Jones SS, Fuerst EP. 1997. Partial characterization of glutathione S-transferase from wheat (Triticum spp.) and purification of a safener-induce glutathione S-transferase from Triticum tauschii. Plant Physiol 4:1461-1470.
Richards KD, Schott EJ, Sharma YK, Davis KR, Gardner RC. 1998. Aluminum induce oxidative stress genes in Arabidopsis thaliana. Plant Physiol 116:409-418.
Sawitri SM. 2007. Ekspresi gen Gα dan GST pada kedelai kultivar Lumut yang mendapatkan cekaman Aluminum. [makalah seminar]. Bogor: Program Pasca Sarjana, Institut Pertanian Bogor.
Sheehan D, Meade G, Foley VM, Dowd CA. 2001. Structure, function and evaluation of glutathione transferase: implication of non mammalian members of an ancient enzyme superfamily. Biochem J 360:1-16.
Srimulyani T. 2007. Keterlibatan protein heterotrimerik G α terhadap cekaman aluminium kedelai (Glycine max (L) Merryl) melalui studi histokimia [skripsi]. Bogor: Institut Pertanian Bogor. Suharsono. 2002. Konstruksi pustaka genom kedelai kultivar Slamet. Hayati 3:67-70.
Sunarto. 1995. Pemulian kedelai untuk toleransi terhadap tanah masam dan keracunan alumunium.
Jurnal Tanaman Industri dan Pangan. XII: 2.
Ulmasov T, Ohmiya A, Hagen G, Guifoyle T. 1995. The soybean GH2/4 gene that encode a glutathione S-transferase has a promoter that is activated by wide range of chemical agents.
Plan Physiol 108:919-927.
[USDA] United State Department of Agriculture. 2007. Oilseed: world markets and trade. Washington: USDA.
Yamamoto Y, Kobayashi Y, Matsumoto H. 2001. Lipid peroxidation is an early symptom triggered by aluminum, but not the primary of elongation inhibition in pea poots. Plant Physiol 125:199-20.
Wilce MCJ, Parker MW. 1994. Structure and function of glutathione S-transferase. Biochim
LAMPIRAN
Lampiran 1 Output sekuensing dengan menggunakan mesin sekuensing AB1 PRISM 310 Genetic Analyzzer
SP6_LGST12
Lanjutan Lampiran 1 T7_LGST12
Lanjutan Lampiran 1 T7_SGST12
Lampiran 2 Hasil sekuensing dua arah menggunakan primer GST12F
dan GST12R menghasilkan urutan nukleotida GST12 Glycine max
(AF243367), cDNA LGST12 dan
SGST12 lengkap mengandung
kodon ATG dan TGA
>GST12 (GST 12 mRNA Glycine max) (EM_PL:AF243367)
CCATAGCAAT GGCAGAGCAA
GACAAGGTGA TCCTACACGG
GATGTGGGCC AGCCCTTATG CCAAGAGGGT GGAATTGGCC CTTAATTTTA AGGGCATACC CTATGAGTAT GTTGAAGAAG ACTTGAGAAA TAAGAGTGAT TTGCTTCTAA AGTACAACCC TGTTCACAAG AAGGTTCCTG TACTTGTTCA TAATGGAAAG GCCATTGCTG AATCCATGGT GATCCTTGAG TATATTGATG AAACATGGAA AGATGGTCCT AAACTGCTTC CAAGTGATTC TTACAAACGA GCCCAAGCTC GATTCTGGTG TCATTTCATC CAGGATCAGT TAATGGAGAG CACTTTTCTA GTAGTCAAAA CTGATGGAGA AGCACAACAA AAGGCCATTG ACCACGTGTA TGAGAAACTG AAAGTGCTAG AAGATGGAAT GAAGACCTAT CTGGGAGAAG GCAATGCTAT TATCTCTGGT GTTGAAAACA ACTTTGGAAT CCTTGACATT GTGTTTTGTG CTTTATATGG TGCCTACAAG GCTCATGAAG AAGTTATTGG CCTCAAGTTC ATAGTGCCAG AAAAGTTTCC TGTGTTGTTT TCTTGGTTGA TGGCTATTGC TGAGGTTGAA GCTGTGAAAA TTGCAACTCC TCCACATGAA AAAACAGTGG GAATTCTTCA GTTGTTCAGG CTGTCTGCAC TGAAATCTTC TTCTGCCACA GAATGATATA TA
>LGST12
CCATAGCAAT GGCAGAGCAA
GACAAGGTGA TCCTACACGG
GATGTGGGCC AGCCCTTATG CCAAGAGGGT GGGATTGGCC CTTAATTTTA AGGGCATACC CTATGAGTAT GTTGAAGAAG ACTTGAGAAA TAAGAGTGAT TTGCTTCTAA AGTACAACCC TGTTCACAAG AAGGTTCCTG TACTATGTTC ATATATGGAA AAGGCCATTG CTGAATCCAT GGTGATCCTT GAGTATATAT TGAATGGAAA CCATGGAATT ATTTTAAAAA CGACCCCAAG TTTTGTTTTT TGGTTTTCAT TTTTCCCAGG ATCAGTTAAT TGGGAGAGCA CTTTTTTAGT AGTCAAAACT GATGGGAGAA GCCCCAACCA AAAGGCCCAT TGTCCACCTT GTATGAGAAA ACTGAAAGTG GCTAGAAGGA TGGGAAGGAA GGCCCTTTCT GGGAGAGGGC AATGCTATTA TCTCTGGGTG TTGAAAAACA ACTTAGGAAT CCTTGACGTG TATGTGCTTT TATATGGTGC CTACAAGGCT CATGACAGAT ATTGGCCTCA AGTTCATAGT GCCAGAAAAG TTTTCCCTGT GTTGTCTTCT TGGTTGATGG CTATTGCTGA GGTTGACAGC GTGTGCAAAT TGCAACTCCT CCACATGAAA AAACAGTGGG AACTCTTCAG TTGTTCACAG GCTGTCTGCA CTCTGAATCT CTCTGCCACA GAATGAAAAA TG
CCATAGCAAT GGCAGAGCAA
GACAAGGTGA TCCTACACGG
GACGTGGTCC AGCCCTTATG CCAAGAGGGT GGGATTGGCC CTTAATTTTA AGGGCATACC CTATGAGTAT GTTGAAGAAG ACTTGAGAAA TAAGAGTGAT TTGCTTCTAA AGTACAACCC TGTTCACAAG AAGGTTCCTG TACTTGTTCA TAATGGAAAG GCCATTGCTG AATCCCATGG TGATCCTTTG AGTTATATTG ATGAAGACAT GGAAAGATGG ATCCTAAACT GCTTCCAAGT GATTCTTAAC AAACGAGGGC CATCGATTTA TGGATGTTAT ATTCTATCCA TGGATCAACT AAGGGGACAA GCCTTTTTCT ATAATCCTGA TGGAGAAGCA CAACAAAAGG CCATTGACCA CGTGTATGAG AAACTGAAAG TGCTAGAAGA TGGAATGAAG ACCTATCTGG GAGAGGGCAA TGCGGTGTTG AAAACAACTT TGGAATCCTT GACATTGTGT TTTGTGCTTT ATATGGTGCC TACAAGGCTC ATGAAGAAGT TATTGGCCTT CAAGTTCATA GTGCCAGAAA AGTTTCCTGT GTTGTCTTTC TTGGTTGATG GCTATTGCAT GAGGTTTGAA GCTGTGAAAA TTGCAACTCC TCCACATGAA AAAACAGTGG GAATTCTTCA GTTGTTCAGG CTGTCTGCAC TGAAATCTTC TTCTGCCACA GAATGATATA TA
Lampiran 3 Asam amino hasil terjemahan sekuen GST12 Glycine max
(AF243367), LGST12 dan
SGST12 menggunakan program expasy translate tool
>aaGST12
MAEQDKVILH GMWASPYAKR
VELALNFKGI PYEYVEEDLR NKSDLLLKYN PVHKKVPVLV HNGKAIAESM VILEYIDETW KDGPKLLPSD SYKRAQARFW CHFIQDQLME STFLVVKTDG EAQQKAIDHV YEKLKVLEDG MKTYLGEGNA IISGVENNFG ILDIVFCALY GAYKAHEEVI GLKFIVPEKF PVLFSWLMAI AEVEAVKIAT PPHEKTVGIL QLFRLSALKS SSATE >aaLGST
MTMAEQDKVI LHGMWASPYA
KRVGLALNFK GIPYEYVEED LRNKSDLLLK YNPVHKKVPV LCSYMEKAIA ESMVILEYIL NGNHGIILKT TPSFVFWFSF FPGSVNWEST FLVVKTDGRS PNQKAHCPPC MRKLKVARRM GRKALSGRGQ CYYLWVLKNN LGILDVYVLL YGAYKAHDRY WPQVHSARKV FPVLSSWLMA IAEVDSVCKL QLLHMKKQWE LFSCSQAVCT LNLSATE >aaSGST12
MTMAEQDKVI LHGTWSSPYA
KRVGLALNFK GIPYEYVEED LRNKSDLLLK YNPVHKKVPV LVHNGKAIAE SHGDPLSYID EDMERWILNC FQVILNKRGP SIYGCYILSM DQLRGQAFFY NPDGEAQQKA IDHVYEKLKV LEDGMKTYLG EGNAVLKTTL ESLTLCFVLY MVPTRLMKKL LAFKFIVPEK FPVLSFLVDG YCMRFEAVKI ATPPHEKTVG ILQLFRLSAL KSSSATE
Keterangan: ATG: Penyandi kodon AWAL (Metionin); TGA: Penyandi kodon STOP; (Primer GST12F) (Primer GST12R)
PENGKLONAN DAN KARAKTERISASI GEN
GST12
DARI
KEDELAI KULTIVAR LUMUT DAN SLAMET
Oleh:
Syahnada Jaya Sy
G34102011
DEPARTMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
S
YAHNADA JAYA SY. Pengklonan dan Karakterisasi gen GST12 dari Kedelai Kultivar Lumut dan Slamet. Dibimbing oleh UTUT WIDYASTUTI SUHARSONO dan ARIS TJAHJOLEKSONO.GST12 diketahui sebagai salah satu gen yang berperan dalam respon ketahanan terhadap cekaman Alumunium (Al). cDNA LGST12 dan SGST12 telah berhasil diisolasi dari akar tanaman kedelai kultivar Lumut dan Slamet. Hasil penelitian sebelumnya diketahui bahwa pada kultur air pH rendah dan Al tinggi LGST12 tidak terinduksi sedangkan SGST12 terinduksi. Penelitian ini bertujuan untuk mengkarakterisasi kedua gen tersebut sebagai langkah awal eksplorasi dan isolasi gen. cDNA kedua kultivar telah dicek keberadaannya kemudian diklon ke dalam pGEM®-T easy. Plasmid rekombinan dalam Escherichia coli galur DH5 telah berhasil diperoleh. Pengurutan cDNALGST12 dan SGST12 dengan DNA sekuenser ABI Prism Model 310 yang masing-masing menghasilkan 722 pb dan menyandikan 237 asam amino. Analisis kesejajaran lokal kedua cDNA terhadap bagian GST12 (AF243367) yang berada di GenBank menunjukan persentase kesamaan sebesar 85 % untuk LGST12 dan 92 % untuk SGST12 dengan Score Bits secara berurutan 908 dan 1146 dan E-value kedua cDNA tersebut 0 (nol). Hasil penelusuran daerah asam amino terkonservasi menunjukkan bahwa cDNA GST12 memiliki daerah domain asam amino terkonservasi GST tipe GST_N_Tau sedangkan cDNA SGST12 selain memiliki daerah domain asam amino terkonservasi GST_N_Tau juga memiliki daerah domain asam amino terkonservasi GST_C_Tau. Berdasarkan penjajajaran asam amino cDNA LGST12 dan SGST12 terhadap GST12
dari beberapa spesies menggunakan MAFFT v6. 500a diperoleh pohon filogenetik yang menunjukkan kedekatan kekerabatan kedua cDNA terhadap GST12 dari Glycine max (AF243367).
ABSTRACT
SYAHNADA JAYA SY. Cloning and Characterization of GST12 Gene from Soybean Lumut Cultivar and Slamet. Supervised by UTUT WIDYASTUTI SUHARSONO and ARIS TJAHJOLEKSONO.
GST12 is one of genes that has important role in Aluminum (Al) stress. cDNA LGST12 and
SGST12 are already isolated from soybean root of Lumut cultivar (cv.) and Slamet. The recent research reported that LGST12 was not induced and SGST12 was induced by low pH and Al stressed water culture system. The objection of this research is to characterize GST12 genes from Lumut cv. and Slamet soybean which have difference response against both of stress. Both of cDNA were re-confirmed by specific primers PCR. Thus, The PCR products are inserted into pGEM®-T easy and the plasmid was successfully introduced into E. coli DH5. Sequencing of
LGST12 and SGST12 by using ABI Prism 310 automated DNA sequencer showed that LGST12
andSGST12 have 722 base pairs encoding 237 amino acids. Basic local alignment analysis based on nucleotide with other GST12 from GenBank showed that LGST12 is 85 % identical and
SGST12 is 92% identical to part of GST12 from Glycine max (AF243367) with bit score 908 and 1146 respectively, and E-value is 0 (zero). LGST12 has big similarity with GST_N_Tau conserved domain type only, however cDNA SGST12 does not only has big similarity on GST_N_Tau, but it also has big similarity on GST_C_Tau conserved domain type. The amino acids alignment analysis is used for comparing the sequences with some species using the MAFFT
PENDAHULUAN
Latar Belakang
Kedelai merupakan salah satu komoditi palawija yang digunakan secara luas di Indonesia sebagai sumber protein nabati. Produksi kedelai tahun 2006 (angka sementara) sekitar 749.04 ribu ton biji kering, mengalami penurunan sekitar 59.32 ribu ton (7.34 %) dibandingkan dengan produksi tahun 2005 (BPS 2006). Penurunan produksi kedelai tersebut menurut BPS (2006) disebabkan oleh adanya penyusutan luas panen dan penurunan produktivitas tanaman kedelai. Luas panen kedelai tahun 2006 menurun 39.93 ribu hektar (6.42 %) dibandingkan dengan tahun 2005. Sementara produktivitas tahun 2006 turun sebesar 0.13 kuintal/hektar (1.00 %) dibandingkan dengan tahun 2005. Bahkan pada tahun 2006 Indonesia mengimpor kedelai sebesar 1 370 000 ton (USDA 2007).
Usaha jangka panjang untuk mening-katkan produksi kedelai dapat dilakukan melalui ekstensifikasi dan perbaikan genetik (Suharsono 2002). Ekstensifikasi telah lama dilakukan di Indonesia, namun belum mem-berikan hasil yang memuaskan. Tanah asam menjadi salah satu faktor penghambat ekstensifikasi. Menurut Mulyani et al. (2003), Indonesia masih memiliki lahan potensial untuk perluasan areal tanam kedelai yang tersebar di Sumatera, Kalimantan, dan Papua. Lahan seluas 18.2 juta hektar sebagian besar bersifat asam dengan kandungan Alumunium (Al) tinggi sehingga masih belum dapat dimanfaatkan sebagai lahan produktif hingga diperolehnya kultivar tanaman yang dapat tumbuh dengan baik pada jenis tanah tersebut.
Pada tanah asam (pH < 5) ion Al3+ men-jadi faktor penghambat pertumbuhan akar tanaman kedelai sehingga mempengaruhi pertumbuhan kedelai secara keseluruhan (Kochian 1995, Matsumoto 2000, Srimulyani 2007). Sifat racun ion-ion Al tersebut juga dapat berupa penghambatan sintesis RNA (Matsumoto et al. 1997), peningkatan perok-sidasi fosfolipid dan protein pada membran (Cakmak & Horst 1991, Yamamoto et al. 2001). Ion-ion Al juga menyebabkan peng-hambatan influks ion Ca2+ sehingga meng-ganggu homeostatis Ca2+ pada apeks akar (Rengel 1992).
Pada Arabidopsis thaliana salah satu gen yang berhubungan dengan sistem pertahanan terhadap cekaman Al adalah gen GST yang menyandikan Glutathione S-Transferase (Richard et al. 1998). Glutathione S-Transferase (GST) merupakan kelompok enzim sitoplasmik. Setiap sub unit GST merupakan suatu dimerik dengan massa molekul sekitar 24-30 kD. Gen GST dapat dibagi menjadi tiga tipe berdasarkan kemiripan sekuennya yaitu GmGST (Glycine max Glutathione S-transferase) tipe I, GmGST tipe II dan GmGST tipe III (Droog et al. 1995; Board et al. 1997; McGonigle et al. 2000). GmGST tipe III atau kelas Tau (Hock & Elstner 2005) memiliki anggota paling besar dimana GmGST 12 termasuk dalam kelas ini. Berdasarkan hasil uji Expressed Sequence Taq (EST) diketahui bahwa GmGST tipe III diekspresikan paling melimpah di bagian akar dan daun kedelai dibandingkan dengan GmGST tipe I dan II (McGonigle et al. 2000).
Pada mamalia dan beberapa tanaman, gen GST ditemukan berperan dalam detoksifikasi metabolit yang disebabkan oleh cekaman oksidatif melalui peredaman molekul-molekul reaktif dengan menambah-kan GSH (Wilce et al. 1994; Sheehan et al. 2001). GST juga terlibat dalam menanggapi berbagai cekaman kimia dan berperan dalam sistem antioksidasi (Ulmasov et al.
1995). Selain fungsi endogenous tersebut, GST juga memiliki peran penting dalam proses detoksifikasi herbisida yang sebagian besar merupakan kelompok senyawa-senyawa xenobiotik (Dixon et al. 1998).
cDNA GST12 kedelai kultivar (var.) Lumut yang bersifat peka (Sunarto 1995) dan var. Slamet yang bersifat toleran terhadap cekaman Al (Jusuf et al. 1999) telah berhasil diisolasi dari bagian akar. Kedua var. tesebut memiliki pola ekpsresi GST12 yang berbeda selama diberi perlakuan cekaman Al dan pH rendah di dalam kultur air (Mashuda 2006; Sawitri 2007). LGST12
(gen GST12 yang diisolasi dari akar kedelai var. Lumut) diinduksi pada jam ke-8 hingga jam ke-72 dalam semua jenis cekaman Al (Mashuda 2006), sedangkan SGST12 (gen GST12 yang diisolasi dari akar kedelai var. Slamet) tidak terinduksi (Sawitri 2007). Oleh karena itu untuk melihat lebih jauh perbedaan yang ada pada gen GST12 dari kedua var. Lumut dan Slamet perlu dilakukan pengklonan dan karakterisasi.
Tujuan
BAHAN DAN METODE
Waktu dan Tempat
Penelitian dilaksanakan mulai Februari 2006 sampai dengan Mei 2007 di laborato-rium
Biotechnology Research Indonesia-The Netherlands (BIORIN) dan Genetika Molekular dan Selular Tanaman (GMST), Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB) IPB Darmaga.
BAHAN
Produk PCR LGST12 (gen GST12 yang diisolasi dari akar kedelai var. Lumut) (Sawitri 2007) dan produk PCR serta cDNA SGST12 (gen GST12 yang diisolasi dari akar kedelai var. Slamet) (Mashuda 2006) yang keduanya diisolasi dari akar kedelai yang ditumbuhkan pada kultur air. Plasmid pGEM®-T Easy (Gambar 1) (Promega) digunakan sebagai vektor pengklonan dan E. coli galur DH5α sebagai inang vektor rekombinan.
Gambar 1 Peta fisik plasmid pGEM®-T Easy yang digunakan sebagai vektor pengklonan
Primer spesifik gen GST12 didesain berdasarkan GST12 (AF243367). Primer forward
(GST12F) dari gen GST12 terletak pada 8 nukleotida sebelum kodon awal
(5’CCATAGCAATGGCAGAGCAAG3’) dan primer reverse (GST12R) terletak pada 11 nukleotida sebelum kodon akhir sampai 6 nukleotida setelah kodon akhir (5’TAT-ATATCATTCTGTGGCAG3’) (Lampiran 2) digunakan sebagai alat verifikasi keberadaan gen
GST12 dari stok cDNA yang tersedia. Metode
Penelitian ini terdiri dari beberapa tahap yaitu penentuan kosentrasi gen GST12 dari stok cDNA. Pengklonan gen GST12 ke dalam plasmid pGEM®-T Easy, transformasi genetik E. coli DH5α dengan vektor rekombinan, analisis urutan nukleotida, dan deduksi asam amino.
Penentuan Konsentrasi gen GST12 dari Stok Produk PCR
cDNA LGST12 dan cDNA SGST12 digunakan sebagai cetakan PCR untuk konfirmasi keberadaan gen GST12. Komposisi PCR adalah 0.5 l cDNA LGST12 dan SGST12 (stok di lab BIORIN), 1 l 10x buffer Taq, 0.1 mM dNTP, 10 pmol primer GST12F, 10 pmol primer GST12R, 4% DMSO (Dimethyl Sulfoxide), 1 U enzim Taq DNA polymerase (Gen Scr Inc.) dan ddH2O hingga volume akhir mencapai 10 l. Kondisi PCR yang digunakan adalah 95 0C selama 5 menit untuk pra-PCR. Siklus PCR dilakukan sebanyak 37 kali dimulai dengan denaturasi pada suhu 94 0
C selama 30 detik, penempelan primer LGST12 55 0C selama 30 detik (52 0C untuk primer
SGST12), pemanjangan 72 0C selama 2 menit. Kemudian dilanjutkan dengan pasca-PCR pada suhu 72 0C selama 5 menit dan pendinginan dilakukan pada 15 0C selama 10 menit (Mashuda 2006; Sawitri 2007).
Pengklonan gen GST12 menggunakan prosedur promega (1996) yang dimodifikasi dengan cara mencampurkan 5 l 2x buffer rapid ligasi, 1 l vektor pGEM®-T Easy (10 ng), 1 l T4 DNA ligase (3 U/ l), dan 0.7 l (10 ng/ l) cDNA LGST12 (0.35 l (20ng/ l) cDNA SGST12). Kemudian diinkubasi pada suhu 4 0C. Untuk mendapatkan hasil pengklonan yang optimal jumlah insert (cDNA LGST12 atau SGST12) yang dibutuhkan dihitung dengan menggunakan rumus berikut:
Produk ligasi diintroduksikan ke dalam E. coli galur DH5α melalui transformasi dengan
prosedur Suharsono (2002). Transformasi diawali dengan pembuatan bakteri kompeten dengan membiakkan satu koloni E. coli DH5α dalam 2 ml LB (Lauria Broth) (1% (b/v) bakto tripton, 0.5% (b/v) bakto ekstrak khamir, 1% NaCl (b/v)) dan diinkubasi pada suhu 37 0C dan digoyang dengan kecepatan 250 rpm selama semalam di dalam inkubator bergoyang, ES-20 (Biosan). Kemudian sebanyak 100 l E. coli DH5α disubkulturkan dalam 30 ml LB dengan kondisi yang
sama hingga mencapai densitas bakteri 4-7 x 107 sel/ml (OD600 = 0.4-0.5). Sel bakteri dipanen dengan cara disentrifugasi pada kecepatan 3 000 rpm pada suhu 4 0C selama 10 menit dengan menggunakan tabung falkon 15 ml. Pelet yang diperoleh selanjutnya ditambahkan buffer transformasi TB (Transformation Buffer) (10 mM PIPES, 15 mM CaCl2.2H20, 250 mM KCl, 55 mM MnCl2.4H2O, pH 6.7) sebanyak 0.033 x volume larutan biakan. Pelet diresuspensi dengan menggunakan pipet dan diinkubasi dalam es selama 10 menit. Selanjutnya disentrifugasi dengan kecepatan 3 000 rpm pada suhu 4 0C selama 10 menit. Pelet yang diperoleh diresuspensikan kembali dengan penambahan buffer TB 41,25 l TB (0.5 x volume larutan biakan). Setelah benar-benar tersuspensikan kemudian ditambah 60 l DMSO dan diinkubasi di dalam es selama 10 menit sehingga diperoleh suspensi sel bakteri kompeten. Sebanyak 50 l suspensi sel kompeten ditambah dengan 10 l (50-100 ng) produk ligasi kemudian diinduksi dalam es selama 20-25 menit. Selanjutnya diberi perlakukaan kejutan panas 42 0C selama 45 detik dan segera dimasukan kembali ke dalam es selam 5 menit. Kemudian ditambah 100 l media YT (Yeast Tripton) (16 g/l bakto tripton, 10 g/l bakto ekstrak khamir, 5 g/l NaCl, pH 7.0). Setelah diinkubasi dalam inkubator bergoyang pada suhu 37 0C selama 20 menit, suspensi tersebut disebar secara merata pada media LB padat yang mengandung 100 mg/l ampisilin dan 10 l 100 mM IPTG (Isopropiltio-β-galaktosida) serta 50 l 2% (b/v) X-gal (5-bromo-4-choloro-3-indoly-β-D-galactoside), kemudian diinkubasi pada suhu 37 0C selama semalam.
Seleksi E. coli DH5α yang mengandung vektor rekombinan
E. coli DH5α yang mengandung vektor rekombinan diseleksi berdasarkan warna koloninya. Koloni putih yang tumbuh pada media yang mengandung ampisilin tersebut berpotensi sebagai koloni yang mengandung vektor rekombinan. Adanya sisipan gen GST12 di dalam plasmid rekombinan yang terdapat dalam koloni putih dideteksi secara cepat dengan melakukan PCR koloni. Pada PCR tersebut, sejumlah kecil koloni bakteri putih digunakan sebagai bagian dari campuran di dalam tabung PCR. Lisis sel bakteri untuk menghasilkan DNA cetakan terjadi di dalam tabung PCR. Untuk itu, koloni putih diambil dengan menggunakan tusuk gigi steril kemudian disuspensikan ke dalam tabung PCR yang telah berisi 6.5 l ddH2O. Kemudian tusuk gigi tersebut dioleskan pada media LA (Lauria agar) (1 g/l bakto tripton, 0.5 g/l bakto ekstrak khamir, 1 g/l NaCl, Agar 25g/l) + ampisilin (50 g/ml) sebagai replika koloni. Selanjutnya pada suspensi ditambah campuran PCR (komposisi dan kondisi PCR sama dengan tahap penentuan kosentrasi gen GST12 dari stok cDNA). Selanjutnya campuran dalam tabung PCR dipanaskan pada suhu 95 0C selama 10 menit dan diinkubasi pada suhu 15 0C selama 5 menit, kemudian diamplifikasi dengan mesin PCR.
Analisis gen sisipan
Plasmid pGEM®-T Easy rekombinan di dalam E. coli DH5α di isolasi dengan mengikuti prosedur Suharsono (2002). Satu koloni E. coli DH5α ditumbuhkan dalam 30 ml LB yang ditambah dengan 60 l ampisilin (50 g/ml) pada inkubator bergoyang (250 rpm) selama 16 jam pada suhu 37 0C. Kultur bakteri dimasukan ke dalam tabung falkon 15 ml, kemudian disentrifugasi (Jouan BR-4i) pada suhu 4 0C dengan kecepatan 4 000 rpm selama 10-15 menit. Endapan ditambah dengan 600 l larutan buffer suspensi sel (50 mM Tri-HCl pH 7.5 dan 10 mM EDTA). Suspensi sel ditambah dengan 400 l buffer lisis (0.2 M NaOH dan 1% SDS) dan dibolak-balik
vektor (ng) x ukuran insert (Kb)
Jumlah insert (ng)= 3/1
secara perlahan beberapa kali kemudian segera ditambah dengan 400 l larutan buffer netralisasi (1.32 M natrium asetat pH 4.8), divorteks dan disentrifugasi pada suhu 4 0C dengan kecepatan 10 000 rpm selama 10 menit. Supernatan diekstrak dengan penambahan PCI (fenol-kloroform-isoamilalkohol dengan perbandingan 25:24:1) sebanyak 1x volume supernatan. Kemudian divorteks dan disentrifugasi pada suhu 20 0C dengan kecepatan 10 000 rpm selama 10 menit. Fase jernih (bagian di atas PCI) diendapkan dengan penambahan natrium asetat 2 M pH 5.2 sebanyak 0.1x volume dan etanol absolut sebanyak 2x volume awal (fase jernih). Larutan diinkubasi pada suhu -20 0C selama 2 jam kemudian diendapkan dengan sentrifugasi 10 000 rpm pada suhu 4 0C selama 25 menit. Endapan (DNA plasmid) dibilas dengan menggunakan alkohol 70% (v/v) dengan bantuan sentrifugasi 10 000 rpm selama 10 menit pada suhu 4 0C. Endapan dikeringkan dan disuspensikan dengan ddH2O. Suspensi DNA palsmid ditambah RNAse (100 g/ml) sebanyak 0.1 x volume suspensi dan diinkubasi pada suhu 37 0C selama semalam. Kemudian ditambah dengan ddH2O hingga volume akhir menjadi 500 l. Plasmid hasil isolasi tersebut akan digunakan sebagai cetakan PCR untuk mendapatkan gen LGST12 dan SGST12 yang digunakan dalam analisis selanjutnya.
Pengurutan DNA dan analisis urutan DNA
Pengurutan DNA (DNA sequencing) dilakukan menggunakan DNA sequencer ABI PRISM 310 Genetic Analyzer Urutan nukleotida dari gen GST12 dianalisis kemiripan dan homologinya
dengan gen GST12 lainnya dengan menggunakan program
BLAST2(Basic_Local_Alignment_Search_Tools) (http://www.ebiac.uk/blast2). Analisis kesamaan dan filogenetik berdasarkan deduksi asam amino GST12 dari spesies lain dilakukan dengan menggunakan program MAFFT (Multiple Alignment Fast Fourier Transform) ver.5.8 (Katoh et al.
2005). Analisis daerah terkonservasi dan domain dilakukan dengan menggunakan program
conserved domain NCBI (www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb.cgi).
HASIL DAN PEMBAHASAN Hasil
Konfirmasi GST 12 dari Stok cDNA
Konfirmasi keberadaan GST12 dari stok cDNA dan produk PCR dari penelitian sebelumnya (Mashuda 2006; Sawitri 2007) dengan menggunakan primer spesifik GST12 (AF243367) menunjukan hasil positif, ukuran pita yang diperoleh sekitar 720 pb (Gambar 1).
M M1 M2 1 M M1 M2 1 2
(a) (b)
Gambar 2 cDNA LGST12 (a); cDNA SGST12 (b). 1= LGST12 berukuran sekitar 720 pb, 2=
SGST12 berukuran sekitar 720 pb. Elektroforesis pada 1.5% (b/v) agarosa. M= 50 ng; M1= 100 ng; M2= Marker 100 bp (RBC) Tanda panah: gen GST12
Pengklonan gen GST12
Setelah amplikon GST12 diligasikan dengan plasmid pGEM®-T Easy dan kemudian diintroduksikan ke dalam E. coli DH5α serta diinkubasi overnight, dijumpai dua jenis koloni yaitu koloni putih dan biru.
720pb
Koloni putih
secara perlahan beberapa kali kemudian segera ditambah dengan 400 l larutan buffer netralisasi (1.32 M natrium asetat pH 4.8), divorteks dan disentrifugasi pada suhu 4 0C dengan kecepatan 10 000 rpm selama 10 menit. Supernatan diekstrak dengan penambahan PCI (fenol-kloroform-isoamilalkohol dengan perbandingan 25:24:1) sebanyak 1x volume supernatan. Kemudian divorteks dan disentrifugasi pada suhu 20 0C dengan kecepatan 10 000 rpm selama 10 menit. Fase jernih (bagian di atas PCI) diendapkan dengan penambahan natrium asetat 2 M pH 5.2 sebanyak 0.1x volume dan etanol absolut sebanyak 2x volume awal (fase jernih). Larutan diinkubasi pada suhu -20 0C selama 2 jam kemudian diendapkan dengan sentrifugasi 10 000 rpm pada suhu 4 0C selama 25 menit. Endapan (DNA plasmid) dibilas dengan menggunakan alkohol 70% (v/v) dengan bantuan sentrifugasi 10 000 rpm selama 10 menit pada suhu 4 0C. Endapan dikeringkan dan disuspensikan dengan ddH2O. Suspensi DNA palsmid ditambah RNAse (100 g/ml) sebanyak 0.1 x volume suspensi dan diinkubasi pada suhu 37 0C selama semalam. Kemudian ditambah dengan ddH2O hingga volume akhir menjadi 500 l. Plasmid hasil isolasi tersebut akan digunakan sebagai cetakan PCR untuk mendapatkan gen LGST12 dan SGST12 yang digunakan dalam analisis selanjutnya.
Pengurutan DNA dan analisis urutan DNA
Pengurutan DNA (DNA sequencing) dilakukan menggunakan DNA sequencer ABI PRISM 310 Genetic Analyzer Urutan nukleotida dari gen GST12 dianalisis kemiripan dan homologinya
dengan gen GST12 lainnya dengan menggunakan program
BLAST2(Basic_Local_Alignment_Search_Tools) (http://www.ebiac.uk/blast2). Analisis kesamaan dan filogenetik berdasarkan deduksi asam amino GST12 dari spesies lain dilakukan dengan menggunakan program MAFFT (Multiple Alignment Fast Fourier Transform) ver.5.8 (Katoh et al.
2005). Analisis daerah terkonservasi dan domain dilakukan dengan menggunakan program
conserved domain NCBI (www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb.cgi).
HASIL DAN PEMBAHASAN Hasil
Konfirmasi GST 12 dari Stok cDNA
Konfirmasi keberadaan GST12 dari stok cDNA dan produk PCR dari penelitian sebelumnya (Mashuda 2006; Sawitri 2007) dengan menggunakan primer spesifik GST12 (AF243367) menunjukan hasil positif, ukuran pita yang diperoleh sekitar 720 pb (Gambar 1).
M M1 M2 1 M M1 M2 1 2
(a) (b)
Gambar 2 cDNA LGST12 (a); cDNA SGST12 (b). 1= LGST12 berukuran sekitar 720 pb, 2=
SGST12 berukuran sekitar 720 pb. Elektroforesis pada 1.5% (b/v) agarosa. M= 50 ng; M1= 100 ng; M2= Marker 100 bp (RBC) Tanda panah: gen GST12
Pengklonan gen GST12
Setelah amplikon GST12 diligasikan dengan plasmid pGEM®-T Easy dan kemudian diintroduksikan ke dalam E. coli DH5α serta diinkubasi overnight, dijumpai dua jenis koloni yaitu koloni putih dan biru.
720pb
Koloni putih
Gambar 3 Penampilan koloni E. coli DH5α yang diduga membawa plasmid rekombinan (koloni
putih) pada media seleksi (ampisilin, X-gal, dan IPTG)
PCR koloni dilakukan untuk memastikan adanya sisipan amplikon di dalam beberapa koloni
E. coli putih yang tumbuh pada media seleksi. Koloni putih yang produk PCR-nya berupa amplikon yang berukuran sama dengan hasil konfirmasi keberadaan GST12 sebelum-nya yaitu sekitar 720 pb akan dipilih sebagai koloni yang akan digunakan pada proses analisi selanjutnya (Gambar 4).
M 1 2 3 4 5 M 1 2 3 4 5 6
(a) (b)
Gambar 4 PCR koloni putih E. coli yang membawa GST12. Elektroforesis pada 1.5% (b/v) agarosa. Kultivar Lumut: dari 5 koloni putih hanya dijumpai 1 koloni positif pembawa plasmid rekombinan (a); kultivar Slamet: dari 6 koloni putih dijumpai 5 koloni positif pembawa plasmid rekombinan (b). M= Marker 100 bp (RBC). Tanda panah: gen GST12
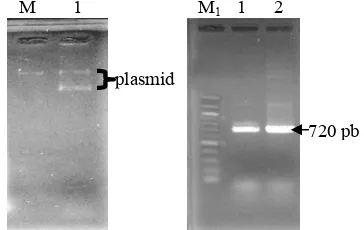
DNA plasmid rekombinan telah diisolasi dari koloni putih yang terpilih melalui PCR koloni. Keberadaan amplikon sisipan kembali dipastikan dengan PCR menggunakan plasmid hasil isolasi sebagai cetakan. Hasil elektroforesis menunjukkan hal yang sama dengan elektroforesis hasil PCR koloni (Gambar 4) yaitu menunjukkan amplikon yang berukuran sekitar 720 pb (Gambar 5). M 1 M1 1 2
720 pb 720 pb
[image:30.612.88.546.87.258.2]
Gambar 5 Elektroforesis plasmid utuh rekombinan (a). Elektroforesis produk PCR GST12 dari plasmid utuh rekombinan (b). 1= LGST12, 2= SGST12. Elektroforesis pada 1.5% (b/v) agarosa. M= Marker 50 ng; M1= Marker 100 bp (RBC). Tanda panah: gen
GST12
Pengurutan cDNA dan analisis urutan cDNA
Hasil sekuensing menunjukkan bahwa panjang cDNA dari kodon awal sampai kodon akhir kedua cDNA LGST12 dan SGST12 adalah 722 pb. Analisis cDNA dilakukan untuk melihat kesamaan dan kekerabatan LGST12 dan SGST12 terhadap gen GST12 koleksi GenBank. cDNA
LGST12 memiliki kesamaan sebesar 85 % dan SGST12 sebesar 92 % terhadap bagian GST12 dari
Glycine max (AF243367). Kedua sekuen gen tersebut memiliki nilai bit score (BS)di atas 50 bits
(908 dan 1146 bits) dan nilai expectation value (E-value) sebesar 0 (nol). Nilai BS dan nilai E-value merupakan nilai yang menunjukan signifikansi kemiripan sekuen (Claverie & Notredame 2003). Sekuen atau urutan nukleotida cDNA LGST