KARAKTERISTIK GEN CYTOCHROME OXIDASE SUBUNIT I (COI)
PADA ULAT SUTERA LIAR (Attacus atlas L.) ASAL JAWA BARAT
DEWI MURNI PUSPITO SARI
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKAN DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
DEWI MURNI PUSPITO SARI. Karakteristik Gen Cytochrome Oxidase Subunit I (COI) pada Ulat Sutera Liar (Attacus atlas L.) Asal Jawa Barat. Di bawah bimbingan DEDY DURYADI SOLIHIN dan SURIANA.
Attacus atlas merupakan ulat sutera liar Indonesia yang tersebar luas, mulai dari Nangroe Aceh Darusalam (NAD) hingga Papua. Berdasarkan basis data genetik di GenBank, belum ada data lengkap genom spesies tersebut, terutama asal Indonesia. Penelitian ini bertujuan mengetahui karakteristik gen COI A. atlas asal Provinsi Jawa Barat. DNA genomik diekstraksi dari otot larva A. atlas asal Bogor dan Purwakarta. DNA total hasil ekstraksi dilanjutkan pada proses amplifikasi dan sekuensing DNA pada gen COI. Gen COI A. atlas disejajarkan dengan gen COI spesies lain untuk membandingkan sekuen nukleotida gen COI. Data sekuen gen COI spesies lain diperoleh dari GenBank. Cricula trifenestrata, Antheraea pernyi, Bombyx mori, dan Bombyx mandarina merupakan spesies yang termasuk dalam pensejajaran. Enam sampel berhasil diamplifikasi dan menghasilkan produk amplifikasi sebesar 595 nt, yang kemudian dianalisis dengan software MEGA 4.0. Penelitian ini telah mampu mengkarakterisasi gen COI A. atlas dan produk sepanjang 508 nt. Runutan nukleotida gen COI pada spesies A. atlas bersifat monomorfik. Nukleotida tersebut bersifat lestari pada level spesies. Ada 31 situs nukleotida diagnostik yang merupakan penciri bagi spesies A. atlas. Pengelompokkan terpisah jelas antara sesama anggota famili Saturniidae (A. atlas, C. trifenestrata, dan A. pernyi) dengan anggota famili Bombycidae (B. mori dan B. mandarina). Analisis hubungan kekerabatan pada A. atlas menunjukkan bahwa A. atlas menempati nodus sendiri, sedangkan C. trifenestrata lebih dekat dengan A. pernyi.
Kata Kunci: Attacus atlas, COI, karakteristik
ABSTRACT
DEWI MURNI PUSPITO SARI. Characteristic of Cytochrome Oxidase Subunit I (COI) Gene in Wild Silkworm (Attacus atlas. L) from West Java Province. Supervised by DEDY DURYADI SOLIHIN and SURIANA.
Attacus atlas is a silkworm of Indonesia, distributing from Nangroe Aceh Darusalam (NAD) to West Papua. Based on genetic databases from GenBank, there is no genome complete data of this species, especially Indonesian origin. This study was aimed to characterize A. atlas COI gene originated from West Java Province. Genomic DNA was extracted from the tissue of A. atlas larvae from Bogor and Purwakarta. DNA extraction were then followed with DNA amplification and sequencing of COI gene. A. atlas COI gene were aligned along with other spesies COI gene to compare nucleotide sequences of COI gene. COI gene sequence data of the others were acquired from GenBank. Cricula trifenestrata, Antheraea pernyi, Bombyx mori, and Bombyx mandarina were among species included in the alignment. Six samples were amplified and the amplification product was 595 nt, which was then analyzed with MEGA 4.0 software. This study was able to characterize A. atlas COI gene and product was 508 nt. Nucleotide sequences of the gene was monomorfic and conserved in the species level. There are 31 diagnostic nucleotide sites characterizing A. atlas. Group separation was distinct between the member of Saturniidae family (A. atlas, C. trifenestrata, and A. pernyi) with the member of Bombycidae family (B. mori and B. mandarina). Phylogenetic analysis of A. atlas showed that A. atlas possesses separate nodes, while the position of C. trifenestrata closes to A. pernyi.
KARAKTERISTIK GEN CYTOCHROME OXIDASE SUBUNIT I (COI)
PADA ULAT SUTERA LIAR (Attacus atlas L.) ASAL JAWA BARAT
DEWI MURNI PUSPITO SARI
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKAN DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi : Karakteristik Gen
Cytochrome Oxidase
Subunit I (COI) pada
Ulat Sutera Liar (
Attacus atlas
L.) asal Jawa Barat
Nama
: Dewi Murni Puspito Sari
NIM
: G34070104
Disetujui,
Pembimbing I
Pembimbing II
Dr. Ir. Dedy Duryadi Solihin, DEA
Dr. Suriana, M.Si
NIP 195611021984031003
NIP 196805221992032001
Diketahui,
Ketua Departemen Biologi
Fakultas Matematika dan Ilmu Pengetahuan Alam
Dr. Ir. Ence Darmo Jaya Supena, M.Sc
NIP 196410021989031002
PRAKATA
Puji dan syukur penulis panjatkan kehadirat Allah SWT atas segala rahmat dan berkatNya sehingga penulis dapat menyelesaikan karya ilmiah yang berjudul Karakteristik Gen Cytochrome Oxidase Subunit 1 (COI) pada Ulat Sutera Liar (Attacus atlas L.) asal Jawa Barat. Penelitian dilaksanakan mulai bulan Februari 2011 hingga Mei 2012 di Laboratorium Biologi Molekuler, Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB) dan Laboratorium Terpadu Departemen Biologi, Institut Pertanian Bogor (IPB).
Penulis mengucapkan terima kasih kepada Bapak Dr. Ir. Dedy Duryadi Solihin, DEA dan Ibu Dr. Suriana, M.Si selaku pembimbing atas segala dukungan, saran dan bimbingan selama proses penelitian dan penyusunan skripsi. Terima kasih penulis sampaikan kepada Ibu N.A. Butet, Bapak Heri, dan Ibu Harini N, atas segala bantuan dan doa, terima kasih kepada seluruh staf Laboratorium Biologi Molekuler PPSHB IPB dan seluruh staf Departemen Biologi. Penulis haturkan terima kasih yang sebesar-besarnya kepada kedua orang tua, Bapak dan Ibu, serta adik Aprilia Sulistyowati atas segala doa, dukungan, dan kasih sayangnya selama ini. Salam hangat penuh cinta penulis sampaikan kepada teman-teman Biologi 44 (khususnya temen seperjuangan: Dini, Ratna, Lydia, Gita, Fery, Danis, Aryo), para sahabat (Anin, Tya, Tiara, Vero, Yosh, Icil, Ana, Anggi, Lia, Wendi, Uchi, Rani, Yusi, Fitri, Sita), M. Adrian Pratama, Aditya R. Syahreza, Keluarga Besar Saman Bungong Puteh, Rumah Muslimah Aisyah dan Pondok Cantik, atas segala kebersamaan dan kenangan indah yang takkan pernah terlupakan.
Penulis menyadari bahwa karya ilmiah ini masih jauh dari sempurna. Oleh karena itu, kritik dan saran yang membangun sangat diharapkan. Semoga karya ilmiah ini dapat bermanfaat bagi yang membutuhkan.
2
RIWAYAT HIDUP
Penulis dilahirkan di Wonogiri pada tanggal 13 Maret 1989 dari ayahanda Partino dan ibunda Suyati. Penulis merupakan anak pertama dari dua bersaudara.
Pada tahun 2007, penulis diterima sebagai mahasiswa Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam (FMIPA), Institut Pertanian Bogor (IPB) melalui jalur Seleksi Penerimaan Mahasiswa Baru (SPMB).
Selama menjadi mahasiswi, penulis aktif dalam kegiatan mahasiswa Saman Bungong Puteh sejak tahun 2008-2011. Selain itu, penulis juga sempat mengikuti kegiatan Studi Lapang di
Cangkuang, Sukabumi, Jawa Barat dengan judul laporan “Tinjauan Ekofisiologi dari Struktur Lanskap dan Vegetasi di Wana Wisata Cangkuang”. Penulis juga mengikuti kegiatan Praktek
Lapang di PT. Ultrajaya Milk Industry and Trading Company, Tbk. pada bulan Juli 2010 dengan
3
DAFTAR ISI
Halaman
DAFTAR GAMBAR ... vii
DAFTAR LAMPIRAN... vii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan... 2
Waktu dan Tempat ... 2
BAHAN DAN METODE ... 2
Bahan ... 2
Metode ... 2
Isolasi DNA ... 2
Amplifikasi dan Visualisasi Fragmen DNA ... 2
Perunutan Produk PCR (Sekuensing) DNA A. atlas Gen COI ... 3
Pensejajaran Runutan Nukleotida (Alignment) Gen COI A. atlas ... 3
Analisis Filogeni ... 3
HASIL DAN PEMBAHASAN ... 3
Hasil ... 3
Isolasi DNA Total ... 3
Amplifikasi Gen COI ... 3
Sekuens dan Alignment DNA A. atlas Gen COI ... 4
Analisis Filogeni A. atlas Gen COI ... 5
Pembahasan ... 6
Variabilitas gen COI... 6
SIMPULAN ... 7
SARAN ... 7
4
DAFTAR GAMBAR
Halaman
1 Hasil isolasi DNA total otot A. atlas pada gel agarosa 1.2%...4 2 Hasil amplifikasi daerah COI pada gel agarosa 1.5%...4 3 Elektroforesis DNA hasil pre-test produk PCR pada gel agarosa 1%...4
4 Konstruksi pohon filogeni berdasarkan runutan 508 nukleotida COI dengan metode Neighboor-Joining, bootstrapped 1000x, model p-distance……….5
DAFTAR LAMPIRAN
Halaman
1 Lokasi pengambilan sampel A. atlas Bogor dan Purwakarta……….10 2 Pensejajaran berganda nukleotida (508 nt) pada gen COI A. atlas dengan C. trifenestrata, B. mori, B. mandarina, dan A. pernyi (GenBank)………..11 3 Situs nukleotida diagnostik gen COI A. atlas...19 4 Perbandingan situs nukleotida diagnostik gen COI antara famili Saturniidae dengan
Bombycidae………20
1
PENDAHULUAN
Latar BelakangSebagai negara yang terkenal dengan kekayaan alam dan biodiversitasnya, Indonesia memiliki berbagai jenis hewan dan satwa yang sangat potensial untuk dikembangkan. Ulat sutera alam asli Indonesia merupakan salah satu contohnya. Empat jenis ulat sutera alam Indonesia (ordo Lepidoptera, famili Saturniidae), yakni Attacus atlas, Cricula trifenestrata, Antheraea pernyi, dan Philosamia ricini berpotensi untuk dikembangkan. Permintaan pasar terhadap benang sutera domestik yang berasal dari ulat sutera alam sampai saat ini sangat tinggi dan belum tercukupi. Masyarakat di daerah Yogyakarta, Gunung Kidul dan Bali telah memanfaatkan sutera alam dari jenis A. atlas dan C. trifenestrata (Solihin & Fuah 2010). A. atlas merupakan salah satu hewan Indonesia yang penyebarannya cukup luas (Solihin & Fuah 2010). Menurut Peigler (1989), peta penyebaran A. atlas tersebar di beberapa daerah di Indonesia, mulai dari Nangro Aceh Darusalam (NAD) hingga Papua.
Attacus atlas termasuk serangga holometabola yaitu serangga yang mengalami metamorfosis sempurna. Siklus hidupnya terbagi menjadi empat tahap yaitu telur, larva atau ulat, pupa atau kepompong dan imago atau ngengat dewasa (Gullan & Cranston 2000). Lamanya siklus hidup spesies tersebut dari telur hingga imago rata-rata 64-88 hari bagi A. atlas (Solihin & Fuah 2010). Keunggulan dari A. atlas ini antara lain memiliki sifat polifagus (makan dan hidup pada berbagai pohon inang) dan polivoltin (beberapa generasi dalam satu tahun) (Solihin & Fuah 2010).
Ulat sutera liar A. atlas (Lepidoptera: Saturniidae) adalah salah satu jenis serangga yang mempunyai nilai ekonomi tinggi karena warna benang sutera yang menarik yaitu coklat keemasan, lebih mengkilat dan harga jual kokon yang tinggi (Rianto 2009). Banyak komoditi olahan yang dapat diproduksi dari bahan ulat sutera liar ini, yaitu industri tekstil (industri tenun dan pakaian), makanan, obat-obatan, kosmetik, seni dan kerajinan tangan (Awan 2007).
Gen cytochrome oxidase subunit I (COI) merupakan salah satu gen yang terdapat dalam genom mitokondria. Dalam studi molekuler, gen COI digunakan sebagai penanda genetik untuk mempelajari karakteristik genetik antar spesies maupun
antar individu. COI juga dapat digunakan sebagai DNA barcoding karena sedikit sekali delesi dan insersi dalam sekuennya, serta variasi juga sedikit (Hebert et al. 2003), merekonstruksi filogenetik pada cabang evolusi tingkat spesies (Palumbi 1996) dan telah berhasil mengidentifikasi maupun membedakan spesies berbagai Invertebrata dan Vertebrata, dari Lepidoptera sampai burung dan dalam geografi yang berbeda (Hajibabaei et al. 2007). Ketepatan pemilihan gen sebagai penanda genetik berdasarkan beberapa kriteria, diantaranya gen ada di semua organisme, memiliki cara kerja yang sama, serta mampu membedakan dengan spesies lain. Karakteristik utama yang perlu diperhatikan sebagai penanda genetik adalah laju substitusi nukleotida ataupun asam amino pada daerah-daerah tertentu. Salah satu kelebihan gen COI sebagai penanda analisis filogeni adalah asam amino pada fragmen COI jarang mengalami substitusi. Namun, basa-basa pada triple kodonnya masih berubah dan bersifat silent (Lynch & Jarrell 1993).
Filogeni molekuler sudah banyak dikaji pada ulat sutera domestikasi (Bombyx mori) dengan penanda genetik cytochrome b (Li et al. 2005), 12S rRNA, 16S rRNA (Arunkumar et al. 2006) dan COI (Arunkumar et al. 2006; Li et al. 2009). Pada ulat sutera liar (Famili Saturniidae), khususnya pada beberapa spesies Antheraea juga sudah dilakukan pengkajian molekuler dengan penanda genetik 12S rRNA, 16S rRNA (Arunkumar et al. 2006), dan COI (Arunkumar et al. 2006; Mahendran et al. 2006). Selain itu, kajian molekuler juga telah dilakukan pada ulat sutera liar C. trifenestrata asal Indonesia dengan penanda genetik gen fibroin (Suriana 2011). Di Indonesia, kajian mengenai ulat sutera liar A. atlas masih terbatas, sehingga perlu dilakukan eksplorasi mengenai gen cytochrome oxidase subunit I (COI) pada spesies tersebut.
2
Tujuan
Penelitian bertujuan mengetahui karakteristik gen cytochrome oxidase subunit I (COI) pada ulat sutera liar Attacus atlas asal Jawa Barat.
Waktu dan Tempat
Penelitian ini dilaksanakan mulai bulan Februari 2011 hingga bulan Mei 2012 di laboratorium Biologi Molekuler, Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB) dan laboratorium terpadu Departemen Biologi.
BAHAN DAN METODE
BahanSampel yang digunakan adalah sampel otot A. atlas asal Bogor (AB) dan Purwakarta (AP). Lokasi pengambilan sampel disajikan pada Lampiran 1. Bahan ekstraksi dan purifikasi DNA yang digunakan yaitu larutan CTAB, proteinase K, larutan fenol, larutan CIAA, larutan NaCl, etanol absolut, etanol 70%, dan larutan TE-RNAse.
Metode Isolasi DNA
DNA genomik dari A. atlas masing-masing daerah yang berbeda individu (n= 3) diisolasi dari sampel otot melalui modifikasi metode Solihin (1997). Sampel otot yang
telah dihaluskan, ditambahkan 600 l larutan
CTAB, dimasukkan ke dalam ependorf dan
ditambahkan 10 l proteinase K 20 mg/ ml.
Sampel diinkubasi pada suhu 55o C selama semalam.
Suspensi yang telah diinkubasi pada suhu 55o C selama semalam ditambahkan 400 l
fenol, 400 l CIAA, dan 40 l NaCl.
Kemudian dikocok pelan pada suhu ruang selama 30 menit dan disentrifugasi pada kecepatan 13000 rpm selama 3 menit. Supernatan dipindahkan ke ependorf baru,
ditambahkan 400 l CIAA, dikocok pelan pada suhu ruang selama 20 menit, dan disentrifugasi pada kecepatan 13000 rpm selama 3 menit. Supernatan dipindahkan ke ependorf baru, ditambahkan 800 l etanol absolut, dan disimpan di freezer selama semalam. Sampel disentrifugasi pada kecepatan 12000 rpm selama 5 menit. Supernatan dibuang, ditambah dengan 800 µl etanol 70%, disentrifugasi pada kecepatan 12000 rpm selama 5 menit dan bagian supernatan dibuang. Sampel (endapan DNA) dikeringudarakan selama 15-30 menit. Endapan ditambahkan 35-50 µl larutan TE-RNAse 40 mg/ ml. Inkubasi pada suhu 37o C selama 15 menit. Sampel DNA disimpan
dalam freezer sampai akan digunakan pada tahap selanjutnya.
Kualitas DNA dilihat dengan dimigrasikan pada gel agarosa 1,2% menggunakan buffer 1xTAE (Tris-asetat 40 mM, EDTA 1 mM). Pewarnaan gel agarosa dengan ethidium bromide (0,8% g/ml) diamati dan difoto di bawah sinar ultra violet (200-400nm). Kuantitas DNA dapat diukur melalui uji spektrofotometri dengan rasio serapan pada panjang gelombang ( ) 260/280 lebih besar 1.8 dianggap murni (Sambrook et al. 1989).
Amplifikasi dan Visualisasi Fragmen DNA DNA total hasil purifikasi digunakan sebagai DNA cetakan untuk proses amplifikasi gen COI. Amplifikasi dilakukan melalui teknik Polymerase Chain Reaction (PCR) dengan menggunakan mesin thermocycler (ESCO). Fragmen DNA yang diamplifikasi yaitu gen COI pada A. atlas. Amplifikasi fragmen DNA daerah COI dilakukan dengan menggunakan pasangan primer yang didisain dari genom lengkap mitokondria Antheraea pernyi. Primer yang digunakan didesain khusus untuk gen COI famili Saturniidae berdasarkan Mahendran et al. (2006) dan Suriana (2011), yaitu primer forward dengan urutan nukleotida 5'-TGATCAAATTTATAATAC-3' dan primer reverse dengan urutan nukleotida 5'-GTAAAATTAAAATATAAAC-3'.
Komposisi perekasi PCR terdiri atas 2 µl sampel DNA, masing-masing 1 µl primer forward dan primer reverse dengan
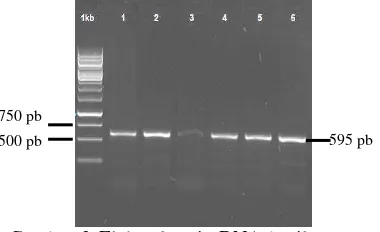
konsentrasi 20 ρmol, 1 µl MgCl2 50 mM, 5.5 µl ddH2O, 2 µl Enhancer, dan 12.5 µl Green Taq Polymerase Master Mix 2x dengan volume total reaksi adalah 25 µl. Kondisi reaksi PCR diawali dengan pre-denaturasi 94o C selama 5 menit. Siklus PCR dilakukan sebanyak 35 kali dengan kondisi denaturasi 94o C selama 45 detik, penempelan primer (annealing) 38o C selama 1 menit 30 detik, ekstensi atau elongasi 72o C selama 2 menit, dan diakhiri oleh post-elongasi 72o C selama 5 menit. Produk PCR yang nantinya dihasilkan sebesar 595 pasang basa (pb).
Kualitas produk PCR dilihat dengan dimigrasikan pada gel agarosa 1,5% menggunakan buffer 1xTAE (Tris-asetat 40 mM, EDTA 1 mM). Pewarnaan gel agarosa dengan ethidium bromide (0,8% g/ml) diamati dan difoto di bawah sinar ultra violet (300-400nm). Kuantitas DNA dapat diukur melalui uji spektrofotometri dengan rasio
3
lebih besar 1.8 dianggap murni (Sambrook et al. 1989).
Perunutan Produk PCR (Sekuensing) DNA A. atlas Gen COI
Pembacaan sekuen DNA sebagai produk PCR menjadi alat penting dan utama dalam biologi molekular karena dapat mengetahui komposisi nukleotida dan asam amino suatu gen, juga menganalisis kekerabatan dan jalur evolusinya (Albert et al. 1994). Prinsip analisa sekuensing DNA berbasis pada metode Sanger (1977) dan Maxam-Gilbert (Applied Biosystems 2012). DNA sequencing menggunakan metode PCR (Polymerase Chain Reaction) sebagai pijakannya. DNA yang akan ditentukan urutan basanya (A,C,G,T) dijadikan sebagai cetakan (template). Kemudian diamplifikasi menggunakan enzim dan bahan-bahan yang hampir sama dengan reaksi PCR, namun ada penambahan beberapa pereaksi tertentu. Proses ini dinamakan cycle sequencing. Saat proses ekstensi, enzim polimerase akan membuat rantai baru DNA salinan dari template dengan menambahkan dNTP-dNTP sesuai dengan urutan pada DNA cetakannya. Jika yang menempel adalah ddNTP, maka otomatis proses polimerisasi akan terhenti
karena ddNTP tidak memiliki gugus 3′-OH
yang seharusnya bereaksi dengan gugus 5′-P dNTP berikutnya membentuk ikatan fosfodiester. Pada akhir cycle sequencing, yang dihasilkan adalah fragmen-fragmen DNA dengan panjang bervariasi. Jika fragmen-fragmen tersebut dipisahkan dengan elektroforesis, maka akan terpisah-pisah dengan jarak antar fragmennya satu basa-satu basa. Urutan basa DNA dapat ditentukan dengan proses pemisahan fragmen pada gel elektroforesis dalam satu lajur, menggunakan label fluorescent dengan empat warna yang berbeda untuk setiap reaksi cycle sequencing (Applied Biosystems 2012).
Perbedaan cycle sequencing dengan PCR biasa diantaranya primer yang digunakan hanya satu untuk satu arah pembacaan, dan adanya penambahan ddNTPs (dideoxy-Nucleotide Triphosphate) sebagai modifikasi
dari dNTPs dengan menghilangkan gugus 3′ -OH pada ribosa (Applied Biosystems 2012).
Produk PCR gen COI penelitian ini berupa pita tunggal yang berukuran 595 pb. Produk PCR dilakukan sekuensing di PT Genetika Science secara lengkap, baik dengan primer forward maupun reverse, untuk memperoleh urutan DNA.
Pensejajaran Runutan Nukleotida
(Alignment) Gen COI A. atlas
Runutan nukleotida gen COI A. atlas dengan primer forward dan reverse diedit dan dianalisis untuk mendapatkan sekuens DNA dari gen COI. Pensejajaran runutan nukleotida dilakukan dengan menggunakan program Clustal W yang terdapat pada software MEGA 4.0 (Tamura et al. 2007). Runutan nukleotida gen COI A. atlas disejajarkan dengan ingroup (Famili Saturniidae) yaitu Cricula trifenestrata (Suriana 2011, GenBank: JF264728.1) dan Antheraea pernyi (GenBank: HQ264055.1), serta outgroup (Famili Bombycidae) yaitu Bombyx mori (GenBank: AF027953.1, AF167260.1, AB083339.1, AB649185.1), dan Bombyx mandarina (GenBank: NC_003395, AY301620.2). Pensejajaran berganda dilakukan hanya pada nukleotida, sehingga dapat diketahui apakah ada perbedaan nukleotida pada ulat sutera liar yang diuji.
Analisis Filogeni
Analisis filogeni A. atlas dikonstruksi antara gen COI hasil penelitian ini dengan basis data gen COI C. trifenestrata, A. pernyi, B. mori, dan B. mandarina. Analisis filogeni menggunakan program MEGA 4 (Tamura et al. 2007) berdasarkan metode substitusi nukleotida dengan jarak genetik p-distance dan konstruksi pohon filogeni berdasarkan bootstrapped Neighbour-Joinning (NJ) dengan 1000 kali pengulangan.
HASIL DAN PEMBAHASAN
HasilIsolasi DNA Total
Enam sampel DNA A. atlas yang digunakan dalam penelitian ini terdiri atas 3 sampel asal Bogor dan 3 sampel asal Purwakarta. Gambar 1 menunjukkan hasil isolasi DNA total setelah dimigrasikan pada gel agarose 1.2% dan dilihat dengan UV transilluminator.
DNA total tersebut selanjutnya digunakan sebagai cetakan DNA untuk mengamplifikasi gen COI dengan menggunakan teknik PCR.
Amplifikasi Gen COI
4
pada gel agarosa 1.5% dan dilihat dengan UV transilluminator (300 nm).
1 2 3 4 5 6
Gambar 1 Hasil isolasi DNA total otot A. atlas pada gel agarosa 1.2%. keterangan: dari kolom 1 sampai kolom 6= AP 4, AP 6, AP 23, AB 52, AB 53, AB 54
1 2 3 4 5 6 7
Gambar 2 Hasil amplifikasi daerah COI pada gel agarosa 1.5%. Keterangan: dari kolom 1 sampai kolom 7= marker 100 bp, AP 4, AP 6, AP 23, AB 52, AB 53, AB 54
Sekuens dan Alignment DNA A. atlas Gen
COI
PT Genetika Science melakukan pre-test semua sampel produk PCR untuk cek kualitas sampel sebelum dilakukan proses cycle sequencing (Gambar 3). Perunutan DNA (sekuensing) dilakukan dari dua arah, yaitu forward dan reverse. Berdasarkan hasil sekuensing yang diperoleh, semua sampel memiliki panjang nukleotida kurang dari 595
nt. Dua sampel yaitu AP 4 dan AP 6 menunjukkan hasil sekuensing yang lebih baik dari empat sampel lainnya, yaitu AP 23, AB 52, AB 53 dan AB 54. Sampel AP 23 menghasilkan pita tunggal yang lebih tipis (Gambar 3) sehingga hasil sekuensing yang diperoleh memiliki beberapa basa nukleotida yang tidak terdeteksi.
Panjang DNA hasil sekuensing setelah dilakukan pensejajaran berganda, diperoleh hasil sekuen sepanjang 508 nukleotida (nt) dari yang seharusnya sepanjang 595 nt (Lampiran 2). Hasil pensejajaran 508 nt gen COI antar populasi A. atlas menunjukkan bahwa gen tersebut sangat conserve pada level spesies (tidak ada satupun nukleotida yang berbeda). Sekuens intra spesies A. atlas pada gen COI menunjukkan komposisi nukleotida yang terdiri dari 37.6% basa timin (T), 28.3% basa adenin (A), 18.9% basa sitosin (C), dan 15.2% basa guanin (G). Basa yang mendominasi yaitu T dan A.
Keenam sekuens A. atlas disejajarkan dengan C. trifenestrata (Suriana 2011, GenBank: JF264728.1), A. pernyi (HQ264055.1), B. mori (GenBank: AF027953.1, AF167260.1, AB083339.1, AB649185.1), dan B. mandarina (NC_003395, AY301620.2), diperoleh nilai conserved dan variabel sebesar 74.2% (377/508) dan 25.8% (131/508). Berdasarkan pensejajaran inter spesies A. atlas dengan spesies lain, terdapat 31 situs nukleotida COI A. atlas yang berbeda dengan spesies lain dan bersifat diagnostik, sehingga dapat dipergunakan sebagai penciri bagi spesies tersebut (Lampiran 3). Ada 15 situs nukleotida COI diagnostik yang merupakan perbandingan antara famili Saturniidae dengan Bombycidae (Lampiran 4), dan 11 situs nukleotida COI diagnostik untuk B. mandarina Japanese (famili Bombycidae) (Lampiran 5).
[image:12.595.107.297.82.811.2] [image:12.595.322.512.592.708.2]5
kolom 7= marker 1 kb, AP 4, AP 6, AP 23, AB 52, AB 53, AB 54
Analisis Filogeni A. atlas Gen COI
Hubungan kekerabatan famili Saturniidae (A. atlas, C. trifenestrata, dan A. pernyi) maupun famili Bombycidae (B. mori, dan B. mandarina) dapat dibandingkan berdasarkan jarak genetik p-distance dari basa-basa nukleotidanya. Nilai jarak genetik A. atlas, C. trifenestrata, A. pernyi, B. mori, dan B. mandarina, berkisar antara 0-0.183 (Lampiran 6). Spesies A. atlas dengan C. trifenestrata dan A. pernyi, masing-masing, memiliki jarak genetik sebesar 0.154 (15.4%) dengan 78 nt berbeda dan 0.140 (14%) dengan 71 nt berbeda. Sedangkan spesies A. atlas dengan B. mori dan B. mandarina, masing-masing, memiliki jarak genetik sebesar 0.152-0.157 (15.2-15.7%) dengan 77-79 nt berbeda dan 0.154-0.156 (15.4-15.6%) dengan 78-79 nt berbeda. Persentase jarak genetik antara A. atlas dengan A. pernyi lebih kecil dibandingkan dengan persentase jarak genetik antara A. atlas dengan C. trifenestrata, B. mori, dan B. mandarina. Perbedaan susunan basa nukleotida hasil pensejajaran pada gen COI dapat dilihat pada Lampiran 2. Matriks perbedaan rata-rata p-distance pada spesies ulat sutera A. atlas, C.
trifenestrata, A. pernyi, B. mori, dan B. mandarina, dapat dilihat pada Lampiran 6.
Analisis filogeni menunjukkan kekerabatan yang dekat dan tidak ada perbedaan komposisi nukleotida antara data sampel A. atlas gen COI dari Bogor dan Purwakarta. Konstruksi pohon filogeni A. atlas, C. trifenestrata, A. pernyi, B. mori, dan B. mandarina berdasarkan jarak genetik p-distance dari basa-basa nukleotida COI dapat dilihat pada Gambar 4. Pada Gambar 4 tersebut menunjukkan bahwa enam sampel A. atlas berada pada nodus yang sama dengan nilai boostrap 100%.
Ulat sutera liar famili Saturniidae berbeda kelompok dengan ulat sutera domestik origin maupun hasil persilangannya (famili Bombycidae).
Sampel A. atlas (Bogor dan Purwakarta) dengan C. trifenestrata (Bogor, Purwakarta, dan Bantul) dan A. pernyi terletak pada nodus A. Sedangkan A. atlas dengan B. mori dan B. mandarina terletak pada nodus terpisah, yaitu nodus B. Hal ini menunjukkan bahwa A. atlas memiliki hubungan kekerabatan terdekat dengan C. trifenestrata dan A. pernyi karena berada dalam satu famili, yaitu Saturniidae.
Gambar 4 Konstruksi pohon filogeni berdasarkan runutan 508 nukleotida COI dengan metode Neighboor-Joining, bootstrapped 1000x, model p-distance (AA= Attacus atlas; CT= Cricula trifenestrata).
AB52 A. atlas
AB53 A. atlas
AP4 A. atlas
AP6 A. atlas
AP23 A. atlas
AB54 A. atlas
A. pernyi
CP1 C. trifenestrata
CBG1 C. trifenestrata
CBAJ1 C. trifenestrata
B. mandarina (Japanese)
B. mandarina (Chinese)
B. mori Back ok jam (Korea)
B. mori Daizo (Chinese)
B. mori Aojuk u (Japanese)
B. mori BmoHap2A (European)
6
Pada nodus B, B. mandarina (Japanese) berbeda cluster dengan B. mandarina (Chinese) dan B. mori lain.
Pembahasan
Semua sampel (enam sampel) menghasilkan pita tunggal yang cukup tebal dengan ukuran 595 pb. Pita DNA yang tebal mengindikasikan bahwa konsentrasi DNA tersebut tinggi. Namun dalam penelitian tidak dilakukan pengukuran kuantitas DNA melalui uji spektrofotometri. Keberhasilan amplifikasi gen COI dipengaruhi oleh beberapa faktor, yaitu kemurnian DNA hasil purifikasi (Sentausa 2003), komposisi bahan pereaksi, dan kondisi reaksi PCR yang tepat, terutama pada proses penempelan primer (annealing). Suhu optimum penempelan primer pada saat amplifikasi sampel yaitu 38o C. Penggunaan suhu penempelan primer tersebut penting dalam proses amplifikasi, karena pada tahap ini memungkinkan primer forward dan reverse akan menempel secara spesifik pada kedua ujung DNA cetakan. Menurut Newton dan Graham (1997), jika suhu penempelan primer terlalu tinggi dari suhu optimum, menyebabkan primer tidak menempel dengan DNA cetakan. Jika suhu penempelan primer terlalu rendah dari suhu penempelan optimum menyebabkan mispriming, yaitu penempelan primer pada tempat yang salah pada DNA cetakan sehingga dihasilkan produk non spesifik. Oleh karena itu, dilakukan optimasi terhadap suhu penempelan primer.
Hambatan yang sering terjadi dalam proses amplifikasi pada penelitian ini, yaitu pita yang dihasilkan multistrand atau banyak pita yang bukan target. Untuk mengatasi hal tersebut dilakukan beberapa modifikasi pada komposisi dan kondisi PCR. Modifikasi yang dilakukan pada komposisi antara lain dengan menurunkan volume MgCl2, menambah volume taq polymerase dan volume DNA cetakan (apabila pita yang dihasilkan tipis). Modifikasi pada kondisi PCR dilakukan dengan menaikkan suhu penempelan primer sehingga lebih spesifik (Febriana 2011).
Variabilitas gen COI
Pendekatan filogenetik dengan jarak genetik antar sekuens dapat digunakan untuk menunjukkan kedekatan kekerabatan. Persentase jarak genetik spesies A. atlas dengan ingroup (A. atlas, C. trifenestrata dan A. pernyi) sebesar 14%-15.4%. Sedangkan persentase jarak genetik A. atlas dengan outgroup (B. mori dan B. mandarina) sebesar 15.2%-15.7%. Hal ini menunjukkan bahwa
semakin besar nilai atau persentase jarak genetik maka semakin besar perbedaan jumlah basa nukleotidanya. Sehingga semakin jauh hubungan kekerabatannya. Sebaliknya, semakin kecil nilai atau persentase jarak genetiknya, maka semakin sedikit perbedaan jumlah basa nukleotidanya. Semakin kecil perbedaan susunan nukleotidanya, maka semakin dekat hubungan kekerabatannya (Febriana 2011). Meskipun nilai perbedaannya tampak tidak signifikan, namun tetap menunjukkan bahwa A. atlas, C. trifenestrata, dan A. pernyi berada dalam satu famili Saturniidae, sedangkan B. mori dan B. mandarina berada dalam satu famili Bombycidae.
Rendahnya keragaman sekuen nukleotida intra spesies berdasarkan COI juga ditemukan pada B. mori (Li et al. 2009), ordo Hymenoptera pada spesies Diadegma (Weagner et al. 2006), dan pada nyamuk (Cywinska 2006). Namun demikian, gen COI ternyata berhasil memisahkan spesies kumbang pada genus Ophraella dengan keragaman antar spesies sebesar 24% (Funk et al. 1995). Gen COI juga mampu memisahkan famili dengan jelas antara famili Saturniidae dengan famili Bombycidae (Mahendran et al. 2006).
Sampel AP 4, AP 6, AP 23, AB 52, AB 53, dan AB 54 terletak pada satu nodus dengan C. trifenestrata (CBAJ1, CBG1, dan CP1) dan A. pernyi. Filogeni berdasarkan runutan nukleotida menunjukkan bahwa A. atlas menempati nodus sendiri dan lebih dekat dengan C. trifenestrata dan A. pernyi dibandingkan dengan B. mori dan B. mandarina. Konstruksi filogenetik menggunakan C. trifenestrata dan A. pernyi sebagai kerabat pada level famili (Saturniidae) dan kerabat jauhnya yaitu B. mori dan B. mandarina (Bombycidae). DNA mitokondria gen COI pada B. mori dan B. mandarina digunakan sebagai outgroup pada analisis filogeni. Penggunaan outgroup berfungsi sebagai faktor koreksi dalam penentuan karakter diantara ingroup yang ada (Maddison 1984).
7
pada semua individu baik di dalam maupun antar populasi. Hal ini dapat dikatakan bahwa nukleotida tersebut bersifat conserve pada level spesies. Keberadaan 31 situs nukleotida diagnostik yang merupakan penciri bagi spesies A. atlas menunjukkan proses evolusi spesifik pada A. atlas. Substitusi nukleotida yang terjadi pada A. atlas memiliki pola sebagai berikut: basa nukleotida C akan digantikan dengan T atau A pada spesies lain; basa nukleotida G digantikan dengan A atau T; basa nukleotida A digantikan dengan T atau C; sedangkan basa nukleotida T digantikan dengan C atau A (Lampiran 3).
Berdasarkan konstruksi pohon filogeni, semua sampel A. atlas, C. trifenestrata dan B. mandarina (Japanese) memiliki nilai boostrap 100% yang menunjukkan konstruksi pohon tidak akan berubah. Antar individu di dalam spesies yang sama memiliki gen COI yang sama, karena tidak ada variasi genetik pada intra dan inter populasinya, yang berarti tidak adanya perubahan nukleotida pada gen tersebut, baik sesama individu maupun antar populasi.
SIMPULAN
Penelitian ini telah mampu mengkarakterisasi gen COI A. atlas sepanjang 508 nt yang dapat dijadikan barcoding untuk spesies tersebut. COI tidak mampu membedakan individu, baik pada populasi yang sama maupun berbeda asal usul. Filogeni menunjukkan bahwa famili Saturniidae bersifat monophyletic. Spesies C. trifenestrata dan A. pernyi menunjukkan kekerabatan dekat dengan A. atlas. Ada 31 situs nukleotida diagnostik untuk A. atlas sebagai penciri spesies.
SARAN
Perlu dilakukan penelitian lebih lanjut pada level molekuler mengenai kajian penggunaan gen cytochrome b sebagai penanda genetik dengan menggunakan sampel yang berasal dari lokasi lain di Indonesia. Selain itu perlu adanya usaha mengeksplorasi keragaman genetik menggunakan gen yang lain.
DAFTAR PUSTAKA
Albert J, Wahlberg J, Leitner T, Escamilla D, Uhlen M. 1994. Analysis of a rape case by direct sequencing of the human immunodeficiency virus type 1 pol and gag genes. J. Virol 68: 5018-24.
Applied Biosystems. 2012. DNA sequencing and fragment analysis by capillary electrophoresis. http://www.appliedbiosystems.com. [13 September 2012].
Arunkumar KP, Muralidhar M, Nagaraju J. 2006. Molecular phylogeny of silkmoths reveals the origin of domesticated silkmoth, Bombyx mori from Chinese Bombyx mandarina and paternal inheritance of Antheraea proylei mitochondrial DNA. Mol Phylogenet and Evol 40:419–427.
Awan A. 2007. Domestikasi ulat sutera liar Attacus atlas (Lepidoptera: Saturniidae) dalam usaha meningkatkan persuteraan nasional [disertasi]. Bogor: Program Pascasarjana, Institut Pertanian Bogor. Cywinska AC, Hunter FF, Hebert PDN.
2006. Identifying Canadian mosquito species through DNA barcodes. Med and Veter Ent 20: 413-424.
Febriana A. 2011. Filogeni Berdasarkan Sekuens DNA Mitokondria Gen Cytochrome Oxidase I (Gen COI) pada Beberapa Bangsa Sapi Lokal Indonesia [skripsi]. Bogor: Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Funk DJ, Futuyama DJ, Orti G, Meyer A. 1995. Mitochondrial DNA sequence and multiple data sets: A phylogenetic study of phyotophagus beetles (Chrysomelidae: Ophraella). Mol Biol Evol 12: 627-640.
Gullan PJ, Cranston PS. 2000. The Insects An Outline of Entomology. Second Edition. London: Blackwell Science Ltd. Hajibabaei M G, Singer AC, Clare EL and Hebert PDN. 2007. Design and Applicability of DNA Arrays and DNA Barcodes in Biodiversity Bionitoring. Bio Med Centre Biol 5:24-30.
Hebert PDN, Ratnasingham S, dan De Waard JR. 2003. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species. Proc R Soc 270: 96–99.
Li AL, Zhao Q, Hang Z. 2005. Molecular phylogeny of the domesticated silkworm, Bombyx mori, based on the sequences of mitochondrial cytochrome b genes. J Genet 84:137-142.
8
Lynch M, Jarrel PE. 1993. A method for calibrating molecular clocks and its application to animal mitochondrial DNA. Genetics 135: 1197-1208.
Maddison WP, Donoghue MJ, Maddison DR. 1984. Outgroup analysis and parsimony. Syst Zool 33: 83-103. Mahendran B, Ghosh SK, Kundu, SC. 2006.
Molecular phylogeny of silk-producing based on 16S ribosomal RNA and cytochrome oxidase subunit I genes. J Genet 85:31-38.
Newton CR, Graham A. 1997. PCR Introduction to Biotechnique. Second Edition. Oxford: Bios Scientific Publisher Ltd.
Palumbi SR. 1996. Nucleic acids II: the polymerase chain reaction. Dalam Molecular systematic. Ed ke-2. Edited by Hillis DM, Moritz C, Mable BK. Massachusetts: Sinauer.
Peigler RS. 1989. A revision of the Indo-Australian genus Attacus. California: The Lepidoptera Research Foundation, Inc.
Rianto F. 2009. Performa Reproduksi Imago Attacus atlas L. yang Berasal dari Perkebunan Teh Purwakarta [skripsi]. Bogor: Fakultas Peternakan, Institut Pertanian Bogor.
Sambrook J, Fristsch EF, Maniatis T. 1989. Molecular Cloning: A Laboratory Manual. New York: Cold Spring Harbour Laboratory Press.
Sanger F, Nicklen S, Coulson AR. 1977. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci USA 74: 5463-5467.
Sentausa E. 2003. Studi DNA Prasejarah dari Tulang Manusia Situs Arkeologi Tadulako, Sulawesi Tengah [skripsi]. Bogor: Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Solihin DD. 1997. Isolasi dan Purifikasi Mitochondrian (mtDNA). Laboratorium Biologi Molekuler Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB. Bogor.
Solihin DD, Fuah AM. 2010. Budidaya Ulat Sutera Alam Attacus atlas. Jakarta: Penebar Swadaya.
Suriana. 2011. Karakteristik Genetik Cricula trifenestrata dari Jawa Barat dan DIY [disertasi]. Bogor: Program Pascasarjana, Institut Pertanian Bogor. Tamura K, Dudley J, Nei M, Kumar S.
2007. MEGA 4: Molecular evolutionary
genetics analysis software version 4. J Mol Biol Evol 24: 1596-1599.
9
10
11
Lampiran 2 Pensejajaran berganda nukleotida (508 nt) pada gen COI A. atlas dengan C. trifenestrata, A. pernyi, B. mori, dan B. mandarina (GenBank)
#MEGA
!Title COI A. atlas vs C. trifenestrata vs A. pernyi vs B. mori vs B. mandarina; !Format
DataType=Nucleotide NSeqs=16 NSites=508
Identical=. Missing=? Indel=-;
!Domain=Data;
[ 1 1111111112 2222222223 3333333334 4444444445 5555555556 6666666667 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas TATAGTAATA CCCATTATAA TCGGAGGATT TGGAAATTGG TTAGTACCTT TAATATTAGG AGCACCTGAT #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata ... ..T... .T... ...T...A ...A.C..C. ...C.T.. ...T... #CP1_C._trifenestrata ... ..T... .T... ...T...A ...A.C..C. ...C.T.. ...T... #CBAJ1_C._trifenestrata ... ..T... .T... ...T...A ...A.C..C. ...C.T.. ...T...
#A._pernyi ...C... ..T... .T... ...A ...A.T..A. ... ...T...
#B._mori_Backokjam_(Korea) ...T... ..T... .T... ...A ...T...C .T...C.... ...A...
#B._mori_Aojuku_(Japanese) ...T... ..T... .T... ...A ...T...C .T...C.... ...A...
#B._mori_Daizo_(Chinese) ...T... ..T... .T... ...A ...T...C .T...C.... ...A...
#B._mori_BmoHap2A_(European) ...T... ..T... .T... ...A ...T...C .T...C.... ...A...
#B._mandarina_(Japanese) ...T... ..T... .T... ...A ...T..CC .T...C.... ...A...
12
Lampiran 2 Lanjutan
[ 1 1111111111 1111111111 1111111111 1111111111 ] [ 7777777778 8888888889 9999999990 0000000001 1111111112 2222222223 3333333334 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas ATAGCTTTCC CTCGAATAAA TAATATAAGT TTTTGACTTC TTCCCCCTTC CTTAACATTA TTAATCTCTA #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata ... ... ... ...AT .A... A...GT.C.T ...T..C. #CP1_C._trifenestrata ... ... ... ...AT .A... A...GT.C.T ...T..C. #CBAJ1_C._trifenestrata ... ... ... ...AT .A... A...GT.C.T ...T..C.
#A._pernyi ... .A... ... ...AT .A...C.. T...CC.. ...C.
#B._mori_Backokjam_(Korea) ...A.... .A... ...A ...C. .A...C.. .C.T.T.... ...T..A.
#B._mori_Aojuku_(Japanese) ...A.... .A... ...A ...C. .A...C.. .C.T.T.... ...T..A.
#B._mori_Daizo_(Chinese) ...A.... .A... ...A ...C. .A...C.. .C.T.T.... ...T..A.
#B._mori_BmoHap2A_(European) ...A.... .A... ...A ...C. .A...C.. .C.T.T.... ...T..A.
#B._mandarina_(Japanese) ...A.... .A... ...A ...C. .A...C.. .C.T.T.... ...T..A.
13
Lampiran 2 Lanjutan
[ 1111111111 1111111111 1111111111 1111111111 1111111111 1111111112 2222222222 ] [ 4444444445 5555555556 6666666667 7777777778 8888888889 9999999990 0000000001 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas GAAGAATTGT CGAAAATGGA GCAGGAACAG GATGAACAGT TTACCCCCCT CTCTCCTCTA ATATTGCCCA #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata ....T... T... ..T... .T...G.. ...C ..T..T..C. ...T.. #CP1_C._trifenestrata ....T... T... ..T... .T...G.. ...C ..T..T..C. ...T.. #CBAJ1_C._trifenestrata ....T... T... ..T... .T...G.. ...C ..T..T..C. ...T..
#A._pernyi ... A... ..T...T. ... ...C ...A. ...T..
#B._mori_Backokjam_(Korea) ... A...T ... ... ...A ..T..A.... ....C..A..
#B._mori_Aojuku_(Japanese) ... A...T ... ... ...A ..T..A.... ....C..A..
#B._mori_Daizo_(Chinese) ... A...T ... ... ...A ..T..A.... ....C..A..
#B._mori_BmoHap2A_(European) ... A...T ... ... ...A ..T..A.... ....C..A..
#B._mandarina_(Japanese) ... A...T ... ... ...A ..T..A.... ...A..
14
Lampiran 2 Lanjutan
[ 2222222222 2222222222 2222222222 2222222222 2222222222 2222222222 2222222222 ] [ 1111111112 2222222223 3333333334 4444444445 5555555556 6666666667 7777777778 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas CGGGGGAAGC TCAGTAGATT TAGCTATTTT TTCTCTTCAT TTAGCTGGAA TTTCTTCAAT TTTAGGGGCT #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata .A.T....CT ..T..T.... ... C..C... C.T..A.... ... .C....C..A #CP1_C._trifenestrata .A.T....CT ..T..T.... ... C..C... C.T..A.... ... .C....C..A #CBAJ1_C._trifenestrata .A.T....CT ..T..T.... ... C..C... C.T..A.... ... .C....C..A
#A._pernyi T..A...TCT ...C .T... ...C... C.T..A..T. ....A... ...A..A
#B._mori_Backokjam_(Korea) TA.A...A ..C...C .T... ...A..A... ...A..T. ....A... .A....A..A
#B._mori_Aojuku_(Japanese) TA.A...A ..C...C .T... ...A..A... ...A..T. ....A... .A....A..A
#B._mori_Daizo_(Chinese) TA.A...A ..C...C .T... ...A..A... ...A..T. ....A... .A....A..A
#B._mori_BmoHap2A_(European) TA.A...A ..C...C .T... ...A..A... ...A..T. ....A... .A....A..A
#B._mandarina_(Japanese) .A.A...A ..T...CC .T... ...AT.A... ...A..T. ....A... .A....A..A
15
Lampiran 2 Lanjutan
[ 2222222222 2222222223 3333333333 3333333333 3333333333 3333333333 3333333333 ] [ 8888888889 9999999990 0000000001 1111111112 2222222223 3333333334 4444444445 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas ATTAATTTTA TTACAACAAT TATTAATATA CGAATAAATA ATTTGTCATT TGATCAAATA CCCCTATTTG #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata ....T... ...T.. ...C... ... .CC.T..C.. ... ..A... #CP1_C._trifenestrata ....T... ...T.. ...C... ... .CC.T..C.. ... ..A... #CBAJ1_C._trifenestrata ....T... ...T.. ...C... ... .CC.T..C.. ... ..A...
#A._pernyi ... ....T... ... ... ....A... ... ..TT...
#B._mori_Backokjam_(Korea) ... ... A... ...T... ..A.A... ...T.. ...T...
#B._mori_Aojuku_(Japanese) ... ... A... ...T... ..A.A... ...T.. ...T...
#B._mori_Daizo_(Chinese) ... ... A... ...T... ..A.A... ...T.. ...T...
#B._mori_BmoHap2A_(European) ... ... A... ...T... ..A.A... ...T.. ...T...
#B._mandarina_(Japanese) ...C. ... A... ...T... .CA.A... ...T.. ..TT...
16
Lampiran 2 Lanjutan
[ 3333333333 3333333333 3333333333 3333333333 3333333334 4444444444 4444444444 ] [ 5555555556 6666666667 7777777778 8888888889 9999999990 0000000001 1111111112 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas TTTGAGCGGT AGGAATCACT GCTTTTCTTT ATTATTATCA TTACCAGTTT TAGCTGGAGC TATTACTATA #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata ...T.. ...T... ...C.... .C.T...T C.T... ... ... #CP1_C._trifenestrata ...T.. ...T... ...C.... .C.T...T C.T... ... ... #CBAJ1_C._trifenestrata ...T.. ...T... ...C.... .C.T...T C.T... ... ...
#A._pernyi ...T.. T...T..A ...CTAC. TC.T... ...T.... ... ...
#B._mori_Backokjam_(Korea) .A...T.. .T.G..T..A ..A...TA.. ... C....C.... ... ...A...
#B._mori_Aojuku_(Japanese) .A...T.. ...G..T..A ..A...TA.. ... C....T.... ... ...A...
#B._mori_Daizo_(Chinese) .A...T.. ...G..T..A ..A...TA.. ... C....T.... ... ...A...
#B._mori_BmoHap2A_(European) .A...T.. ...G..T..A ..A...TA.. ... C....T.... ... ...A...
#B._mandarina_(Japanese) .A...T.. ...T..A ..A...TA.. ... ...T.... ... ...A...
17
Lampiran 2 Lanjutan
[ 4444444444 4444444444 4444444444 4444444444 4444444444 4444444444 4444444444 ] [ 2222222223 3333333334 4444444445 5555555556 6666666667 7777777778 8888888889 ] [ 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 ] #AP4_A._atlas CTTTTAACTG ACCGTAACCT TAATACCTCC TTTTTTGACC CTGCAGGGGG AGGAGATCCA ATTCTTTACC #AP6_A._atlas ... ... ... ... ... ... ... #AP23_A._atlas ... ... ... ... ... ... ... #AB52_A._atlas ... ... ... ... ... ... ... #AB53_A._atlas ... ... ... ... ... ... ... #AB54_A._atlas ... ... ... ... ... ... ... #CBG1_C._trifenestrata ...C. ....A..TT. A...T..T ...T. .A..T..A.. ...T...T ..C..C..T. #CP1_C._trifenestrata ...C. ....A..TT. A...T..T ...T. .A..T..A.. ...T...T ..C..C..T. #CBAJ1_C._trifenestrata ...C. ....A..TT. A...T..T ...T. .A..T..A.. ...T...T ..C..C..T.
#A._pernyi ...A. .T..A..TT. A...T..T ...C..T. ....T..T.. ... ...T.A..T.
#B._mori_Backokjam_(Korea) T.A...A. .T..AG..T. A...A..A ...T. ....T..A.. ...C... ...T.A..T.
#B._mori_Aojuku_(Japanese) T.A...A. .T..A...T. A...A..A ...T. ....T..A.. ...C... ...T.A..T.
#B._mori_Daizo_(Chinese) T.A...A. .T..A...T. A...A..A ...T. ....T..A.. ...C... ...T.A..T.
#B._mori_BmoHap2A_(European) T.A...A. .T..A...T. A...A..A ...T. ....T..A.. ...C... ...T.A..T.
#B._mandarina_(Japanese) T.A...A. .T..A...T. A...A..A ... ....T..A.. G... ...T.A..T.
18
Lampiran 2 Lanjutan
[ 4444444445 55555555] [ 9999999990 00000000] [ 1234567890 12345678] #AP4_A._atlas AACATCTTTT TTGATTTT #AP6_A._atlas ... ... #AP23_A._atlas ... ... #AB52_A._atlas ... ... #AB53_A._atlas ... ... #AB54_A._atlas ... ... #CBG1_C._trifenestrata ...T.A.. ... #CP1_C._trifenestrata ...T.A.. ... #CBAJ1_C._trifenestrata ...T.A.. ...
#A._pernyi ...T.A.. ...
#B._mori_Backokjam_(Korea) ...T.A.. ....A...
#B._mori_Aojuku_(Japanese) ...T.A.. ...
#B._mori_Daizo_(Chinese) ...T.A.. ...
#B._mori_BmoHap2A_(European) ...T.A.. ...
#B._mandarina_(Japanese) ...T.A.. ...
#B._mandarina_(Chinese) ...T.A.. ....C...
Keterangan:
19
Lampiran 3 Situs nukleotida diagnostik gen COI mitokondria A. atlas berdasarkan runutan 508 nt yang dibandingkan
Spesies
Situs nukleotida ke-
1 1 1 1 1 2 2 2 2 2 2 2 3 3 3 4 4 4 4 4 4 4 4 4 4 4 4 1 2 4 4 0 1 3 5 9 0 1 2 4 5 7 8 2 5 6 2 3 3 4 4 5 6 6 8 8 9 9 3 2 0 6 9 2 9 1 0 8 4 0 4 6 7 0 5 8 7 9 5 9 1 7 0 5 8 6 9 6 8
Attacus atlas C C G A T T T C T C G C T T G T G G C T T C T C C A G T C C T
C. trifenestrata T T A C A A C T C T T T C A C A T T T C A T A T T T A C T T A
A. pernyi T T A T A A C A C T A T C A A A A T T A A T A T T T T A T T A
B. mori Backokjam (Korea) T T A T C A A A A A A A A A A A A T T A A T A A A T A A T T A
B. mori Aojuku (Japanese) T T A T C A A A A A A A A A A A A T T A A T A A A T A A T T A
B. mori Daizo (Chinese) T T A T C A A A A A A A A A A A A T T A A T A A A T A A T T A
B. mori BmoHap2A (European) T T A T C A A A A A A A A A A A A T T A A T A A A T A A T T A
B. mandarina (Japanese) T T A T C A A A A A A A A A A A A T T A A T A A A T A A T T A
20
Lampiran 4 Perbandingan situs nukleotida diagnostik gen COI antara famili Saturniidae dengan Bombycidae berdasarkan runutan 508 nt yang dibandingkan
Spesies
Situs nukleotida ke-
1 1 1 1 2 3 3 3 3 3 4 4
5 6 7 0 2 2 6 4 0 1 3 5 7 1 2
0 7 6 0 2 4 0 7 1 4 8 2 3 7 1
Famili Saturniidae
A. atlas T T T T T A A T T A A T T T C
C. trifenestrata T T T T T A A T T A A T T T C
A. pernyi T T T T T A A T T A A T T T C
Famili Bombycidae
B. mori Backokjam (Korea) C A A A C T T A A T T A A A T
B. mori Aojuku (Japanese) C A A A C T T A A T T A A A T
B. mori Daizo (Chinese) C A A A C T T A A T T A A A T
B. mori BmoHap2A (European) C A A A C T T A A T T A A A T
B. mandarina (Japanese) C A A A C T T A A T T A A A T
21
Lampiran 5 Situs nukleotida diagnostik gen COI B. mandarina (Japanese) dalam famili Bombycidae berdasarkan runutan 508 nt yang dibandingkan
Spesies
Situs nukleotida ke-
2
2
2
2
2
3
3
3
4
4
4
1
2
2
4
8
2
4
9
5
7
9
1
3
9
5
9
2
3
1
9
1
B. mori
Backokjam (Korea)
T
T
C
T
C
T
T
C
C
T
A
B. mori
Aojuku (Japanese)
T
T
C
T
C
T
T
C
C
T
A
B. mori
Daizo (Chinese)
T
T
C
T
C
T
T
C
C
T
A
B. mandarina
(Chinese)
T
T
C
T
C
T
T
C
C
T
A
B. mandarina
(Japanese)
C
C
T
C
T
C
C
T
T
C
G
22
Lampiran 6 Matriks perbedaan rata-rata nukleotida p-distance (% nukleotida yang berbeda) berdasarkan metode pairwise distance daerah COI pada ulat sutera A. atlas, C. trifenestrata, A. pernyi, B. mori, dan B. mandarina (508 nt)
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16
[1]
[2] 0.000
[3] 0.000 0.000
[4] 0.000 0.000 0.000
[5] 0.000 0.000 0.000 0.000
[6] 0.000 0.000 0.000 0.000 0.000
[7] 0.154 0.154 0.154 0.154 0.154 0.154
[8] 0.154 0.154 0.154 0.154 0.154 0.154 0.000 [9] 0.154 0.154 0.154 0.154 0.154 0.154 0.000 0.000
[10] 0.140 0.140 0.140 0.140 0.140 0.140 0.128 0.128 0.128
[11] 0.157 0.157 0.157 0.157 0.157 0.157 0.183 0.183 0.183 0.134
[12] 0.152 0.152 0.152 0.152 0.152 0.152 0.177 0.177 0.177 0.126 0.008
[13] 0.152 0.152 0.152 0.152 0.152 0.152 0.177 0.177 0.177 0.126 0.008 0.000
[14] 0.152 0.152 0.152 0.152 0.152 0.152 0.177 0.177 0.177 0.126 0.008 0.000 0.000
[15] 0.154 0.154 0.154 0.154 0.154 0.154 0.175 0.175 0.175 0.130 0.035 0.028 0.028 0.028
[16] 0.156 0.156 0.156 0.156 0.156 0.156 0.181 0.181 0.181 0.130 0.022 0.016 0.016 0.016 0.033
Keterangan: [ 1] AP4_AA [ 2] AP6_AA [ 3] AP23_AA [ 4] AB52_AA [ 5] AB53_AA [ 6] AB54_AA
[ 7] CBG1_CT [ 8] CP1_CT [ 9] CBAJ1_CT [10] A. pernyi
[11] B. mori_Backokjam_(Korea) [12] B. mori_Aojuku_(Japanese)