OPTIMASI PARAMETER PADA
SUPPORT VECTOR MACHINE
UNTUK KLASIFIKASI FRAGMEN METAGENOME
MENGGUNAKAN ALGORITME GENETIKA
INNA SABILY KARIMA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Optimasi Parameter pada Support Vector Machine untuk Klasifikasi Fragmen Metagenome Menggunakan Algoritme Genetika adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
INNA SABILY KARIMA. Optimasi Parameter pada Support Vector Machine Untuk Klasifikasi Fragmen Metagenome Menggunakan Algoritme Genetika. Dibimbing oleh WISNU ANANTA KUSUMA dan IRMAN HERMADI.
Klasifikasi fragmen metagenome merupakan salah satu contoh dari proses binning yang bertujuan untuk mengklasifikasikan fragmen-fragmen ke dalam beberapa tingkat taksonomi. Salah satu metode machine learning yang dapat digunakan adalah Support Vector Machine (SVM). Pada masalah yang bersifat non linear, diperlukan kernel untuk memetakan vektor ciri ke dalam ruang berdimensi tinggi, sehingga masalah yang non linear tersebut dapat dipecahkan secara linear. Kernel yang sering digunakan adalah Radial Basis Function (RBF). Tantangan yang dihadapi adalah bagaimana menemukan parameter yang optimal sehingga dihasilkan model klasifikasi yang akurat.
Tujuan penelitian ini adalah melakukan optimasi parameter pada SVM dalam klasifikasi fragmen metagenome menggunakan Algoritme Genetika. Parameter yang akan dicari nilai optimalnya adalah parameter C untuk jarak margin dan gamma () untuk percepatan fungsi pada kernel RBF untuk mendapatkan akurasi classifier model klasifikasi yang optimal. Evaluasi dilakukan untuk membandingkan akurasi model klasifikasi dengan parameter yang dioptimasi dengan Algoritme Genetika dan akurasi yang dihasilkan dengan parameter yang ditentukan dengan menggunakan grid search. Hasil evaluasi menunjukan bahwa akurasi yang dihasilkan dengan parameter yang dioptimasi dengan Algoritme Genetika adalah 67.3% untuk fragmen berukuran 400bp dan 98.6% untuk fragmen berukuran 10 Kbp. Akurasi ini lebih tinggi dibandingkan akurasi dari model klasifikasi dengan parameter yang ditentukan dengan menggunakan grid search, yaitu sebesar 65.3% untuk fragmen berukuran 400 bp dan 95.4 untuk fragmen berukuran 10 Kbp.
SUMMARY
INNA SABILY KARIMA. Parameter Optimization Support Vector Machine (SVM) for classification of metagenome fragment Using Genetic Algorithm. Supervised by WISNU ANANTA KUSUMA and IRMAN HERMADI.
Classification of metagenome fragment is an example of the binning process which aims to classify fragments into several taxonomic levels. One of the methods of machine learning that can be used is the Support Vector Machine (SVM). In the non linear problem, it is necessary to map the kernel feature vector into a high dimensional space, so that the non-linear problem can be solved linearly. One of the most popular kernel used in the classification problem is the Radial Basis Function (RBF). The challenge is how to find the optimal parameters to produce accurate classification models.
The purpose of this study is to optimize the parameters of the SVM in classifying metagenome fragment using Genetic Algorithms. Parameters to be searched is the optimal value of C representing margin and gamma (), a kernel parameter of the RBF kernel to obtain the optimal classification models. The evaluation is conducted to compare the accuracy of classification model that uses optimized parameters yielded by Genetic Algorithms and those of being determined using the grid search technique. The evaluation results show that the accuracies of the resulting parameters optimized by Genetic Algorithm are 67.3% for fragment size of 400 bp and 98.6% for the 10 kbp fragment size. These accuracies are higher than those of using the grid search technique which obtain 65.3% for fragment size of 400 bp and 95.4% for the 10 kbp fragment size.
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Komputer
pada
Program Studi Ilmu Komputer
OPTIMASI PARAMETER PADA
SUPPORT VECTOR MACHINE
UNTUK KLASIFIKASI FRAGMEN METAGENOME
MENGGUNAKAN ALGORITME GENETIKA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2014
Judul Tesis : Optimasi Parameter pada Support Vector Machine untuk Klasifikasi Fragmen Metagenome Menggunakan Algoritme Genetika
Nama : Inna Sabily Karima NIM : G651120351
Disetujui oleh Komisi Pembimbing
Dr Wisnu Ananta Kusuma, MT Ketua
Irman Hermadi, SKom, MS, PhD Anggota
Diketahui oleh
Ketua Program Studi Ilmu Komputer
Dr Wisnu Ananta Kusuma, MT
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan September 2013 dengan judul Optimasi Parameter pada Support Vector Machine untuk Klasifikasi Fragmen Metagenome Menggunakan Algoritme Genetika.
Terima kasih penulis ucapkan kepada Bapak Dr Wisnu Ananta Kusuma, MT dan Bapak Irman Hermadi, Ssi, MS, PhD selaku pembimbing. Selain itu, penulis menyampaikan terima kasih kepada semua dosen dan staf Departemen Ilmu Komputer IPB yang telah membantu selama proses penelitian. Ungkapan terima kasih juga disampaikan kepada Papah dan Mamah, suami tercinta Rahmat Oktavian, S. Kom, M. Kom serta kedua adik saya Ainun dan Sofia, atas doa, perhatian dan kasih sayangnya. Teman sepembimbingan (Lailan, Abrar, Nita, Ramdhan), teman-teman Dwi Regina (Kak Marlinda, Frinsa, Mentari, Thoyyibah, Astrid, Lian, Erlisa), dan teman-teman seperjuangan angkatan 14 (Dhieka, Yesi, Nia, Vira, Khusnul, Gita) Ilmu Komputer IPB yang selalu bersama penulis dua tahun ini, terima kasih atas dukungannya yang diberikan kepada penulis.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 3
Manfaat Penelitian 3
Ruang Lingkup Penelitian 3
2 TINJAUAN PUSTAKA 3
Metagenom 3
Support Vector Machine 3
Optimasi 6
Grid Search 7
K-fold cross-validation 7
Algoritme Genetika 8
3 METODE 10
Alur Metode Penelitian 11
Penyiapan data 11
Ekstrasi Fitur 12
Scaling 12
Optimasi parameter Kenel RBF dengan Algoritme Genetika 13
Desain Kromosom 13
Pembentukan Populasi Awal 15
Evaluasi Fitness 16
Kriteria Pemberhentian 16
Seleksi 16
Crossover 17
Mutasi 17
Elitisme 17
Optimasi parameter Kenel RBF dengan Grid Search 18
Pengujian SVM 19
Analisis 19
4 HASIL DAN PEMBAHASAN 20
Penyiapan Data 20
Ekstrasi Fitur 20
Proses Support Vector Machine 21
Sensitivity dan Specificity 23
5 SIMPULAN DAN SARAN 25
Simpulan 25
Saran 25
DAFTAR PUSTAKA 26
LAMPIRAN 28
DAFTAR TABEL
1. Nilai eksponen di format floating point 32-bit 15
2. Hasil Optimasi Parameter SVM dengan algoritme genetika 22
DAFTAR GAMBAR
1. Alternatif bidang pemisah (Osuna et al. 2007) 4
2. Bidang pemisah terbaik dengan margin (m) terbesar (Osuna et al.
2007) 4
3. Soft margin hyperplane (Osuna et al. 2007) 5
4. Transformasi dari vektor input ke feature space (Osuna et al. 2007) 6
5. K-fold cross-validation (Refaeilzadeh et al. 2009) 8
6. Pengkodean (encoding) (a) biner (b) permutasi (Jacob 2001) 9
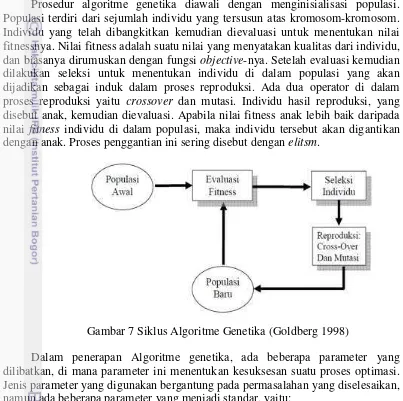
7. Siklus Algoritme Genetika (Goldberg 1998) 10
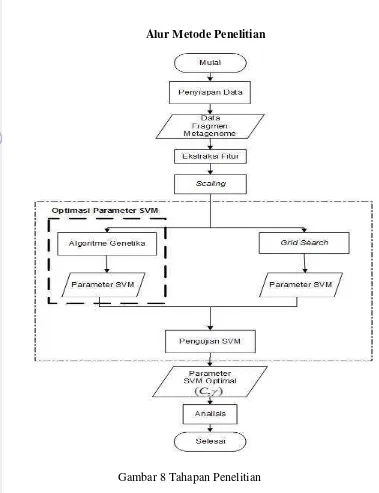
8. Tahapan Penelitian 11
9. Pola spaced k-mers dengan parameter w = 3 dan d = 0, 1, 2 (Kusuma
2012) 12
10. Optimasi parameter menggunakan algoritme Genetika 13
11. Desain kromosom insisalisasi parameter C dan 14
12. Format bilangan floating point 32-bit (Kahan 1997) 14
13. Estimasi Parameter Menggunakan Grid Search 18
14. Hasil akurasi berdasarkan panjang fragmen 22
15. Grafik perbandingan nilai akurasi klasifikasi Algoritme Genetika
dengan Grid Search 23
16. Sensitivity takson genus 24
17. Specificity takson genus 24
DAFTAR LAMPIRAN
1. Daftar nama organisme data latih 28
2. Daftar nama organisme data uji 37
3. Daftar tingkat taksonomi genus yang digunakan 42
4. Daftar hasil praproses data latih 43
1
PENDAHULUAN
Latar Belakang
Salah satu bidang kajian bioinformatika yang saat ini terus mengalami perkembangan adalah metagenomika (Chan et al. 2007; O’Malley 2012). Berbeda dengan studi yang mempelajari genom (genomika), pada metagenomika sekuens DNA dari komunitas mikrob tidak diperoleh dari pure clonal cultures dari individu tertentu melainkan diperoleh melalui proses sequencing secara langsung (McHardy et al. 2007). Metagenom yang berasal dari lingkungan mengandung berbagai organisme yang selanjutnya dilakukan proses assembly, yaitu proses perakitan penyesuaian dan penggabungan fragmen urutan DNA sequence menjadi urutan DNA yang sebenarnya. Karena mengandung fragmen-fragmen dari berbagai organisme, maka proses assembly menjadi lebih sulit. Kesalahan assembly akan dapat menyebabkan terbentuknya cymeric contigs, yaitu contigs yang dihasilkan dari fragmen-fragmen yang berasal dari organisme yang berbeda. Untuk memeperkecil kemungkinan munculnya cymeric contigs, maka proses binning diperlukan untuk mengelompokkan fragmen-fragmen tersebut sebelum melakukan proses assembly.
Ada dua pendekatan proses binning yaitu binning berdasarkan komposisi dan binning berdasarkan homologi (Wooley et al. 2010). Binning berdasarkan komposisi memiliki beberapa keunggulan dibandingkan pendekatan binning lainnya yang berdasarkan homologi. Binning berdasarkan komposisi merupakan jalan pintas (by pass) kebutuhan akan penjajaran sequences, vektor masukan yang dihasilkan dari ekstraksi ciri berupa pasangan basa (base pair) akan dihitung sebagai ciri komposisi, kemudian ciri tersebut akan digunakan sebagai masukan pada pembelajaran dengan contoh (supervised learning) atau pada pembelajaran secara observasi (unsupervised learning). Binning berdasarkan homologi merupakan proses pencarian penjajaran sekuens dengan membandingkan fragmen metagenom dengan basis data yang digunakan, yaitu National Centre for Biotechnology Information (NCBI) dan hasilnya akan disimpulkan pada tiap level taksonomi. Proses binning dalam persepktif bidang ilmu komputer dapat dilakukan dengan metode supervised atau unsupervised learning. Pada binning dengan metode supervised learning, fragmen-fragmen yang diklasifikasikan berdasarkan level taksonomi tertentu, misalnya yang paling rendah ialah level genus, masih sulit klasifikasi pada level species.
2
menggunakan 5-mers, yang berarti matriks fitur yang dihasilkan memiliki dimensi 45 = 1024. Proses ekstraksi fitur yang melibatkan dimensi yang besar ini memerlukan waktu komputasi yang tinggi. Kusuma dan Akiyama (2011) mengusulkan metode klasifikasi fragmen metagenom dengan menggunakan metode SVM. Characterization vector sebagai fiturnya yang digunakan dalam klasifikasi fragmen metagenom masih belum mempengaruhi hasil akurasi yang diperoleh cukup tinggi yaitu 78% untuk panjang fragmen 500 bp sampai dengan 87% untuk panjang fragmen 10 Kbp. Namun, ketika metode ini diterapkan pada dataset berukuran besar (374 organisme), akurasi yang diperoleh menurun secara signifikan, yaitu sebesar 30% untuk panjang fragmen 1 Kbp pada level genus. Ariny (2013) melakukan klasifikasi fragmen metagenom memperoleh hasil akurasi baik meskipun diterapkan pada fragmen dengan panjang yang kecil (400 bp), yaitu 82.1% pada level filum, 78.2% pada level kelas, 72% pada level order dan 65.3% untuk level genus. Berdasarkan penelitian Ariny (2013), metode klasifikasi fragmen metagenom dengan algoritme SVM memiliki akurasi lebih tinggi dibandingkan metode sebelumnya, namun waktu komputasinya lama pada saat training . Hal ini disebabakan penentuan nilai parameter C dan gamma () yang dilakukan dengan metode grid search. Metode grid search menguji semua kombinasi parameter C dan gamma (), kemudian memilih kombinasi yang memberikan hasil paling optimum dalam melakukan klasifikasi.
Support Vector Machine (SVM) mengklasifikasi data dengan class yang berbeda untuk menentukan sebuah hyperplane (Huang dan Wang 2006). Klasifikasi fragmen metagenom menggunakan metode SVM masih terdapat masalah yang diahadapi yaitu bagaimana cara mengatur parameter terbaik pada kernel. Pengaturan parameter yang tepat dapat meningkatkan akurasi klasifikasi SVM (Huang dan Wang 2006). Untuk itu parameter SVM perlu dioptimalkantermasuk paramater C dan parameter fungsi kernel termasuk gamma () untuk kernel Radial Basis Function (RBF). Algoritme grid merupakan alternatif mencari C yang terbaik dan gamma ketika menggunakan fungsi kernel RBF. Namun, metode ini memakan waktu yang lama dan tidak memiliki performa yang baik (Hsu dan Lin 2002). Algoritme genetika memiliki potensi untuk menghasilkan parameter SVM yang optimal pada saat yang sama. Algoritme genetika sangat baik untuk menyelesaikan permasalahan optimasi dan melakukan search point dengan mencari pola baru yang diharapkan memiliki nilai fitness yang lebih baik dari seluruh kromosom dan dapat meningkatkan kinerja pada classifier (Limai 2009). Penelitian ini bertujuan pada optimasi parameter C dan gamma () untuk classifer SVM. Untuk dataset yang digunakan data latih terdiri atas 381 organisme dan data uji terdiri dari 200 organisme. Fleksibilitas algoritme genetika untuk penyelesaian masalah optimasi yang tidak menuntut persyaratan yang ketat, seperti kekontinuan dan keterdifferensialan fungsi tujuan, memungkinkan metode ini dapat menyelesaikan masalah optimasi yang dilakukan pada penelitian ini.
Perumusan Masalah
3 semua kombinasi parameter C dan gamma ( ) terutama dengan ukuran data (bp) yang besar dapat mempengaruhi hasil akurasi klasifikasi.
Tujuan Penelitian
Tujuan penelitian ini adalah menggunakan algoritme genetika untuk optimasi parameter SVM dalam klasifikasi fragmen metagenom.
Manfaat Penelitian
Penentuan parameter SVM dengan algoritme genetika dapat meningkatkan hasil akurasi klasifikasi metagenom yang lebih baik dan mendapatkan parameter SVM yang optimal.
Ruang Lingkup Penelitian
Hal-hal yang membatasi penelitian adalah sebagai berikut :
1. Data mikrob diperoleh dari National Centre for Biotechnology Information (NCBI) yaitu 381 organisme.
2. Level takson yang digunakan yaitu genus.
3. Kernel yang digunakan pada SVM adalah kernel RBF.
4. Parameter SVM yang akan dioptimasi adalah C sebagai nilai cost dan sebagai perlambatan/percepatan fungsi kernel.
2
TINJAUAN PUSTAKA
Metagenom
Metagenom merupakan genom dari mikrob tanpa pengulturan mikrob. Istilah metagenom berasal dari konsep statistik meta-analisis (proses yang secara statistik mengombinasikan metode-metode analisis yang terpisah), serta genomik (analisis menyeluruh dari materi genetika suatu organisme). Metagenomics dikembangkan berdasarkan kemajuan terkini bidang biologi molukuler dan bioinformatika. Bioinformatika ini mempunyai peranan yang penting salah satunya yaitu untuk manajemen data biologi molekul, terutama sekuen DNA dan informasi genetika (Thontowi 2009).
SupportVector Machine
4
tetapi memiliki performansi yang lebih baik di berbagai bidang aplikasi seperti bioinformatika, pengenalan tulisan tangan, klasifikasi teks (Christianini 2001).
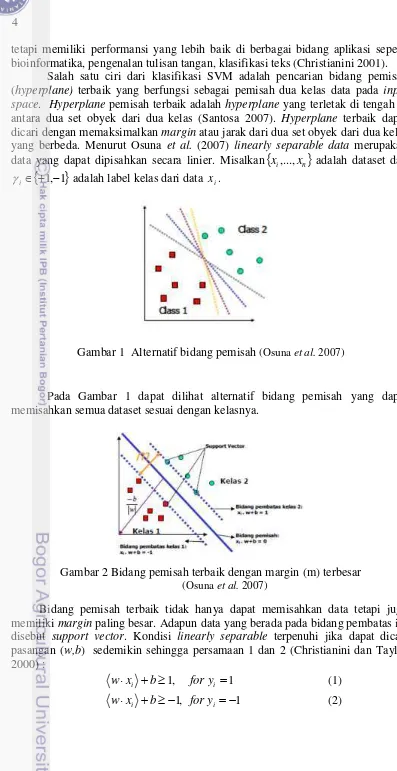
Salah satu ciri dari klasifikasi SVM adalah pencarian bidang pemisah (hyperplane) terbaik yang berfungsi sebagai pemisah dua kelas data pada input space. Hyperplane pemisah terbaik adalah hyperplane yang terletak di tengah di antara dua set obyek dari dua kelas (Santosa 2007). Hyperplane terbaik dapat dicari dengan memaksimalkan margin atau jarak dari dua set obyek dari dua kelas yang berbeda. Menurut Osuna et al. (2007) linearly separable data merupakan data yang dapat dipisahkan secara linier. Misalkan
xi,...,xn
adalah dataset dan
1,1
i
adalah label kelas dari data xi.
Pada Gambar 1 dapat dilihat alternatif bidang pemisah yang dapat memisahkan semua dataset sesuai dengan kelasnya.
Bidang pemisah terbaik tidak hanya dapat memisahkan data tetapi juga memiliki margin paling besar. Adapun data yang berada pada bidang pembatas ini disebut support vector. Kondisi linearly separable terpenuhi jika dapat dicari pasangan (w,b) sedemikin sehingga persamaan 1 dan 2 (Christianini dan Taylor 2000) :
1 ,
1
xi b foryi
w (1)
1 ,
1
xi b foryi
w (2)
Gambar 1 Alternatif bidang pemisah (Osuna et al. 2007)
5 dengan w adalah bidang normal dan b adalah posisi bidang relatif terhadap pusat koordinat. Kemudian, ruang hipotesis untuk data tersebut ialah set fungsi yang diberikan oleh Persamaan 3 (Christianini dan Taylor 2000) :
) (
, sign w x b
fwb (3)
Setelah dilakukan penyelesaian dengan formula Lagrangian menggunakan Lagrange multipier dan normalisasi parameter w, maka fungsi keputusan untuk menentukan kelas dari data uji x adalah pada Persamaan 4 (Christianini dan Taylor 2000) :
l i i ii x x b
y sign x f 1 ), ) , ( ( )
( (4)
dengan = koefisien Lagrange multipier.SVM pada Nonlinearly Separable Data untuk mengklasifikasikan data yang tidak dapat dipisahkan secara linier formula SVM harus dimodifikasi karena tidak akan ada solusi yang ditemukan. Pencarian bidang pemisah terbaik dengan dengan penambahan variabel i sering juga disebut soft margin hyperplane. Dengan demikian formula pencarian bidang pemisah terbaik berubah menjadi Persamaan 5 (Christianini dan Taylor 2000) :
0 1 ) . ( . . 2 1 min 1 2
i i i i n i i b x w y t s C w (5)C adalah parameter yang menentukan besar penalti akibat kesalahan dalam klasifikasi data dan nilainya ditentukan oleh pengguna.
Metode lain untuk mengklasifikasikan data yang tidak dapat dipisahkan secara linier adalah dengan mentransformasikan data ke dalam dimensi ruang fitur (feature space) diilustrasikan pada Gambar 4 sehingga dapat dipisahkan secara linier pada feature space.
6
Dengan metode ini, data dipetakan dengan menggunakan fungsi pemetaan (transformasi xk (xk) ke dalam feature space sehingga terdapat bidang pemisah yang dapat memisahkan data sesuai dengan kelasnya (Gambar 3). Feature space dalam praktiknya biasanya memiliki dimensi yang lebih tinggi dari vektor input (input space). Hal ini mengakibatkan komputasi pada feature space mungkin sangat besar, karena ada kemungkinan feature space dapat memiliki jumlah feature yang tidak terhingga. Selain itu, sulit mengetahui fungsi transformasi yang tepat. Untuk mengatasi masalah ini, pada SVM digunakan ”kernel trick”. Fungsi kernel yang umum digunakan adalah sebagai berikut (Osuna et al. 2007):
1. Kernel Linier
x x x x
K( i, ) Ti
2. Polynomial kernel
0 , ) (
) ,
(xi x xTi xr p K
3. Radial Basis Function (RBF)
0 ), | |
exp( ) ,
(x x x x 2
K i i
4. Sigmoid Kernel
) tanh(
) ,
(x x x x r
K i Ti
Optimasi
Optimasi merupakan suatu proses untuk mencari kondisi yang optimum, dalam arti paling menguntungkan (James dan Riggs 1988). Optimasi bisa berupa maksimasi atau minimasi. Jika berkaitan dengan masalah keuntungan, maka keadaan optimum adalah keadaan yang memberikan keuntungan maksimum (maksimasi). Jika berkaitan dengan masalah pengeluaran/pengorbanan, maka keadaan optimum adalah keadaan yang memberikan pengeluaran/pengorbanan minimum (minimasi). Hal-hal penting dalam studi optimasi meliputi:
1. Fungsi objektif dan decision variables 2. Kendala (constraints)
Secara umum, fungsi yang akan dimaksimumkan atau diminimumkan disebut fungsi objektif (objective function), sedangkan harga-harga yang berpengaruh dan bisa dipilih disebut variabel (perubah) atau decision variable.
7 Secara analitik, nilai maksimum atau minimum dari suatu persamaan: y = f (x) dapat diperoleh pada harga x yang memenuhi Persamaan 6 (Steven et al. 2003) :
0 )
( '
'
dx df dx dy x f
y (6)
Untuk fungsi yang sulit untuk diturunkan atau mempunyai turunan yang sulit dicari akarnya, proses optimasi dapat dilakukan secara numerik.
Grid Search
Algoritme grid search yaitu salah satu Algoritme umum yang sering digunakan untuk estimasi parameter, dengan prinsip kerjanya dengan menentukan beberapa nilai parameter pada rentang tertentu, kemudian memilih parameter pada nilai terbaik pada rentang tersebut dan melakukan pencarian berulang pada grid (rentang nilai) yang lebih kecil, dst. Fungsi grid search mengeluarkan nilai parameter terbaik yang dibutuhkan saat pembentukan model (tahap pelatihan) menggunakan kernel RBF dan polynomial. Parameter untuk kernel RBF adalah cost (c) dan gamma (γ), sedangkan untuk kernel polinomial adalah cost (c), gamma (γ), degree (d), dan koeff 0 (r). Akan tetapi, parameter r pada polinomial yang dipakai hanya nilai default-nya saja yaitu 0. Selain mengeluarkan nilai parameter terbaik, fungsi ini juga mengeluarkan akurasi 5-cross validation dari data latih.
Cross-validation merupakan metode statistika untuk mengevaluasi dan membandingkan algoritme pembelajaran dengan membagi data menjadi dua bagian. Satu bagian untuk melatih model dan bagian lainnya untuk memvalidasi model tersebut. Salah satu bentuk cross-validation adalah k-fold cross-validation. Kelemahan Algoritme grid search pada pencarian grid yang terlalu kecil dapat mengakibatkan overfitting ( Khotimah et al. 2010). Menurut Izenman (2008) overfitting adalah suatu kejadian di mana jumlah parameter yang masuk ke dalam model terlalu besar dibandingkan dengan ukuran data yang digunakan untuk membangun model (learning set). Model tersebut menghasilkan galat yang sangat kecil untuk data learning set, namun galat yang besar untuk data validasi.
K-fold cross-validation
8
Ilustrasi proses K-fold cross validation dapat dilihat pada Gambar 5. Metode ini melakukan perulangan sebanyak k kali untuk membagi sebuah himpunan contoh secara acak menjadi k-subset yang saling bebas. Setiap ulangan disisakan satu subset untuk pengujian, dan sisanya digunakan untuk pelatihan.
Algoritme Genetika
Algoritme genetika bisa dikatakan sebagai metode metaheuristik yang paling populer. Hal ini disebabkan karena algoritme genetika memiliki performa yang baik untuk berbagai macam jenis permasalahan optimisasi. Algoritme genetika diperkenalkan oleh Holland (1975) dalam bukunya “Adaptation in Natural and Artificial Systems”. Adaptasi menjadi prinsip yang penting di dalam algoritme genetika. Adaptasi adalah kemampuan untuk menyesuaikan diri dengan lingkungannya, dan di dalam algoritme genetika, adaptasi dinyatakan dengan proses memodifikasi struktur individu yang akan meningkatkan kinerja Algoritme genetika. Mekanisme kerja algoritme genetika mengikuti fenomena evolusi genetika yang terjadi dalam makhluk hidup. Ada 4 kondisi yang sangat mempengaruhi proses evolusi, yaitu:
1. Kemampuan organisme untuk melakukan reproduksi
2. Keberadaan populasi organisme yang bisa melakukan reproduksi 3. Keberagaman organisme dalam suatu populasi
4. Perbedaan kemampuan untuk survive
Secara umum struktur algoritme genetika sebagai berikut :
a. Populasi, istilah pada teknik pencarian yang dilakukan sekaligus atas sejumlah solusi yang mungkin
b. Kromosom, individu yang terdapat dalam satu populasi dan merupakan suatu solusi yang masih berbentuk simbol.
c. Generasi, populasi awal dibangun secara acak sedangkan populasi selanjutnya merupakan hasil evolusi kromosom-kromosom melalui iterasi. d. Fungsi Fitness, alat ukur yang digunakan untuk proses evaluasi kromosom.
Nilai fitness dari suatu kromosom akan menunjukkan kualitas kromosom dalam populasi tersebut.
9 e. Generasi berikutnya yang dikenal dengan anak (offspring) terbentuk dari gabungan 2 kromosom generasi sekarang yang bertindak sebagai induk (parent) dengan menggunakan operator penyilang (crossover).
f. Mutasi, operator untuk memodifikasi kromosom.
Menurut Goldberg (1989) algoritme genetika adalah suatu algoritme pencarian (searching) yang didasarkan pada mekanisme seleksi alam. Tujuannya untuk menentukan struktur-struktur yang disebut dengan individu berkualitas tinggi dalam suatu domain yang disebut populasi untuk mendapatkan solusi terbaik suatu persoalan. Golberg (1989) mengemukakan bahwa algoritme genetika mempunyai karakteristik-karakteristik yang perlu diketahui sehingga dapat dibedakan dari prosedur pencarian atau optimasi yang lain, yaitu:
a. Algoritme genetika dengan pengkodean dari himpunan solusi permasalahan berdasarkan parameter yang telah ditetapkan dan bukan parameter itu sendiri.
b. Algoritme genetika pencarian pada sebuah solusi dari sejumlah individu-individu yang merupakan solusi permasalahan bukan hanya dari sebuah individu.
c. Algoritme genetika informasi fungsi objektif (fitness), sebagai cara untuk mengevaluasi individu yang mempunyai solusi terbaik, bukan turunan dari suatu fungsi.
d. Algoritme genetika menggunakan aturan-aturan transisi peluang, bukan aturan-aturan deterministik.
Siklus dari algoritme genetika pertama kali dikenalakan oleh Goldberg, dimana gambaran siklus tersbut dapat dilihat pada Gambar 8. Menurut (Jacob 2001), ada empat prinsip dasar didalam algoritme genetika; (1) prinsip dualisme, (2) pengkodean diskrit (3) efek rekombinasi dan (4) building blok dasar. Di dalam system biologi, prinsip dualisme yang dimaksudkan adalah informasi genetik yang terdapat didalam DNA selain memiliki fungsi sebagai informasi yang dapat di replikasi, juga berfungsi sebagai suatu instruksi yang harus dieksekusi. Pada algoritme genetika, struktur genetik dari individu dimodifikasi berdasarkan operasi rekombinasi dan mutasi, sedangkan instruksi genetik dinyatakan secara terpisah melalui berbagai parameter di dalam algoritme genetika. Di dalam pengkodean biner, setiap string memiliki nilai 1 atau 0. Selain pengkodean biner, pengkodean permutasi merupakan jenis pengkodean yang cukup popular, tetapi hanya bisa diterapkan pada permasalahan pengurutan seperti travelling salesman problem. Di dalam pengkodean permutasi, angka di dalam string dinyatakan secara berurutan.
(a) Kromosom (b) Kromosom
10
Algoritme genetika meniru proses rekombinasi dari system biologi melalui dua operator utamanya, crossover dan mutasi. Crossover akan meneruskan sifat-sifat baik yang terdapat pada induk kepada anaknya sedangkan mutasi berfungsi untuk menjaga keberagaman dari populasi. Representasi penyelesaian di dalam algoritme genetika dinyatakan dengan kromosom. Satu kromosom biasanya menyatakan satu buah variabel penyelesaian dan setiap kromosom bisa terdiri dari beberapa gen.
Prosedur algoritme genetika diawali dengan menginisialisasi populasi. Populasi terdiri dari sejumlah individu yang tersusun atas kromosom-kromosom. Individu yang telah dibangkitkan kemudian dievaluasi untuk menentukan nilai fitnessnya. Nilai fitness adalah suatu nilai yang menyatakan kualitas dari individu, dan biasanya dirumuskan dengan fungsi objective-nya. Setelah evaluasi kemudian dilakukan seleksi untuk menentukan individu di dalam populasi yang akan dijadikan sebagai induk dalam proses reproduksi. Ada dua operator di dalam proses reproduksi yaitu crossover dan mutasi. Individu hasil reproduksi, yang disebut anak, kemudian dievaluasi. Apabila nilai fitness anak lebih baik daripada nilai fitness individu di dalam populasi, maka individu tersebut akan digantikan dengan anak. Proses penggantian ini sering disebut dengan elitsm.
Dalam penerapan Algoritme genetika, ada beberapa parameter yang dilibatkan, di mana parameter ini menentukan kesuksesan suatu proses optimasi. Jenis parameter yang digunakan bergantung pada permasalahan yang diselesaikan, namun ada beberapa parameter yang menjadi standar, yaitu:
a. Ukuran populasi (pop_size) b. Probabilitas crossover (pc) c. Probabilitas mutasi (pm)
3
METODE
Penelitian ini menggunakan metode algoritme genetika dalam optimasi parameter SVM. Algoritme genetika mempunyai potensi untuk membangkitkan parameter optimal SVM. Penelitian ini dilaksanakan dalam beberapa tahapan yang diilustrasikan pada Gambar 9.
11
Alur Metode Penelitian
Gambar 8 Tahapan Penelitian Penyiapan data
12
fragmen, sedangkan untuk data uji berjumlah 100 ribu fragmen. Panjang fragmen yang ditetapkan untuk setiap kali pengolahan yaitu 400 bp, 800 bp, 1 Kbp, 3 Kbp, 5 Kbp, dan 10 Kbp. Data latih dengan jumlah fragmen 9600 disiapkan sebagai data pendekatan pencarian parameter terbaik untuk kernel, sedangkan data latih dengan jumlah fragmen 320 ribu menjadi data masukan untuk pembuatan model. Penggunaan data latih kecil sebagai pendekatan pencarian paramater terbaik ini didasarkan pada percobaan yang dilakukan oleh McHardy (McHardy et al. 2007).
Ekstrasi Fitur
Tahapan selanjutnya adalah ekstrasi fitur. Pada tahapan ekstrasi fitur menggunakan data dari hasil praproses yang telah dilakukan oleh Ariny (2013) dengan menggunakan MetaSim. Metode ekstraksi fitur yang dilakukan dengan membaca frekuensi dari kombinasi nukleotida yang terbentuk ialah metode spaced k-mers. Terdapat 2 buah variabel yang berpengaruh pada metode ekstraksi fitur ini, yaitu w (weight of pattern) adalah banyaknya posisi yang cocok, dan d adalah jumlah posisi don’t care. Mengacu pada penelitian Kusuma (2012), pola terbaik spaced k-mers dengan nilai w = 3 dan d = 0, 1, 2. Ilustrasi perhitungan frekuensi pola kemunculan dapat dilihat pada Gambar 10.
3 2 , 1 , 0 w d
1111*111**11
AAA AAC .. GGG A*AA ... G*GG A*AA ... G **GG
64 Kombinasi 64 Kombinasi 64 Kombinasi
Template Fitur Fitur
192 Kombinasi
Gambar 9 Pola spaced k-mers dengan parameter w = 3 dan d = 0, 1, 2 (Kusuma 2012)
Metode ini memeriksa frekuensi nukleotida dari setiap fragmen DNA mulai dari AAA sampai GGG, A*AA sampai G*GG, dan A**AA sampai G**GG, sehingga didapat 192 dimensi fitur. Pada Gambar 10 pengertian dari simbol * (don’t care) pada fragmen DNA yang diperiksa adalah dapat berupa basa apapun, baik A, C, T, maupun G. Kemudian untuk simbol ** berarti diperbolehkan pasangan basa apapun mengisi 2 bit tersebut, sehingga kondisi ini dapat diisi oleh 24 pasang basa mulai dari AA, AC, AT, AG, dan seterusnya hingga GG.
Scaling
Prosedur tahapan algoritme genetika diawali dengan menginisialisasi populasi fragmen metagenom. Sebelum ke tahap selanjutnya yaitu evaluasi fitness, diperlukan analisis parameter yang berpengaruh terhadap classifier SVM yang disebut scaling. Tujuan scaling adalah untuk menghindari perkiraan angka yang besar yang mendominasi perkiraan angka lebih kecil. Propulasi terdiri dari beberapa individu yang tersusun atas kromosom-kromosom.
13 dibangkitkan kemudian dievaluasi pada tahap berikutnya untuk menentukan nilai fitness-nya. Data fragmen metagenom akan diubah bobotnya menjadi data yang memiliki rentang [0, 1] atau [-1,+1] menggunakan transformasi linear sederhana Persamaan 7 (Vesanto et al. 2000).
) min( ) max(
) min(
'
v v
v v
v
(7)
Dengan v adalah dataset, v'adalah nilai yang telah diskala, min (v) adalah nilai min dataset, max adalah nilai makasimum dataset.
Optimasi parameter Kenel RBF dengan Algoritme Genetika
Tahap berikutnya adalah optimasi parameter dengan Algoritme Genetika. Proses klasifikasi SVM dengan menggunakan kernel RBF membutuhkan parameter C dan (gamma) yang optimal agar hasil klasifikasi optimal. Kedua parameter ini dioptimasi dengan menggunakan algoritme genetika. Diagram alir optimasi parameter dengan menggunakan Algoritme Genetika ditunjukkan pada Gambar 11.
Gambar 10 Optimasi parameter menggunakan algoritme Genetika Desain Kromosom
14
dengan kromosom. Satu kromosom biasanya menyatakan satu buah variable penyelesaian dan setiap kromosom bisa terdiri dari beberapa gen. Kromosom mempunyai parameter berbeda sesuai fungsi kernel yang dipilih, pada penelitian ini fungsi kernel yang digunakan adalah kernel RBF. Parameter yang terdapat pada fungsi kernel RBF terdiri dari parameter C dan (gamma). Gambar 12 menunjukan desain kromosom gen yang dinyatakan dengan bit string. Untuk
C
n C C g
g1 ~ menyatakan nilai parameter C dan g1 ~gn menyatakan nilai parameter
(gamma).nCrepresentasi dari jumlah bit parameter C sedangkann representasi dari jumlah bit parameter .
1
C
g
ncg
1C
g
g
ni C
g
g
jGambar 11 Desain kromosom insisalisasi parameter C dan
Dalam penelitian ini nilai parameter C dan mempunyai rentang nilai yang mungkin terdapat bilangan yang sangat besar atau sangat kecil. Bilangan tersebut harus dapat direpresentasikan dengan tepat yaitu menggunakan floating-point. Bilangan floating-point direpresentasikan dengan mantissa yang berisi digit signifikan dan eksponen dari radix R. Format umum floating point pada persamaan (8).
Mantissa x Reksponen (8)
Represensasi bilangan floating point seringkali dinormalisasi terhadap radixnya, misalnya 1, 5 × 1044atau 1, 253 × 10− 36. Format bilangan floating-point biner telah distandarkan oleh IEEE 754-2008 (atau ISO/IEC/IEEE 60559:2011), yaitu meliputi format 16-bit (half), 32-bit (single-precision), 64-bit (double-precision), 80-bit (double-extended) dan 128-bit (quad-precision).
Bilangan floating-point 32-bit tersusun atas 1 bit Sign (S)
8 bit eksponen (E), dan 23 bit untuk mantissa (M)
15 Nilai ekstrem adalah untuk E=0 dan E=255
E=0 menyatakan bilangan NOL (jika M = 0) dan subnormal (jika M ≠ 0) E=255 menyatakan bilangan TAK TERHINGGA (jika M = 0) dan NAN/not-a-number (jika M ≠ 0);
Nilai normal adalah 1 ≤ E ≤ 254 yang menunjukkan nilai eksponen sebenarnya dari -126 sampai 127
Contoh: Emin(1) = − 126, E(50) = − 77 dan Emax(254) = 127;
Tabel 1 Nilai eksponen di format floating point 32-bit
Eksponen (E) Mantissa = 0 Mantissa 0 Persamaan
0 0, -0 126
2 .
0 ) 1
( s bitsignifikan
1-245 Nilai Ternormalisasi 127
2 .
0 ) 1
( s bitsignifikan
255 Bukan
bilangan (NAN = not-
a-number)
Saat nilai mantissa (M) dinormalisasi, most significant bit (MSB) selalu 1. Namun, bit MSB ini tidak perlu disertakan secara eksplisit di field mantisa (Tabel 1). Nilai mantissa yang sebenarnya adalah 1.M, sehingga nilai bilangan floating point-nya menjadi Persamaan 9 (Kahan 1997):
127 23 1 1 127 2 2 0 ) 1 ( 2 . 1 ) (
E i i S E m M B V (9)Di bilangan subnormal, nilai mantisa sebenarnya adalah 0.M, sehingga bilangan floating point-nya menjadi Persamaan 10 (Kahan 1997):
127 23 1 1 127 2 2 0 ) 1 ( 2 . 1 ) (
E i i S E m M B V (10)Dengan mantissa 23 bit ini ditambah 1 bit implisit, total presisi dari representasi floating point 32-bit ini adalah 24 bit atau sekitar 7 digit desimal (yaitu 24 × log10(2) = 7.225).
Pembentukan Populasi Awal
16
Evaluasi Fitness
Pada tahapan evaluasi fitness untuk setiap kromosom yang mewakili C dan gamma () yang dipilih, data training yang digunakan untuk melatih classifier SVM, sedangkan data testing digunakan untuk menghitung akurasi klasifikasi. Ketika klasifikasi akurasi diperoleh dari masing-masing kromosom akan dievalusai fungsi fitness dengan persamaan (11) (Huang dan Wang 2006).
accuracy
SVM
fitness
_
(11)Pada persamaan (10) menunjukan jumlah fitness yang dihasilkan sama dengan nilai akurasi pada klasifikasi dengan SVM pada persamaan (12) yang merupakan cara perhitungan akurasi untuk hasil klasifikasi dengan SVM.
% 100
x uji data
benar uji data Akurasi
(12)
Setelah dibangun model SVM dengan parameter C dan γ dari proses Algoritme Genetika maka model tersebut diuji dengan data uji yang sama untuk panjang fragmen yang berbeda. Hasil klasifikasi dibandingkan dengan kelas aktual sehinga didapatkan akurasi untuk model tersebut. Akurasi menjadi nilai fitness untuk menentukan baik tidaknya model SVM yang dihasilkan.
Kriteria Pemberhentian
Pada tahapan ini akan diperiksa kondisi populasi saat ini terhadap kriteria pemberhentian. Ketika kriteria sudah terpenuhi maka proses generate offspring dihentikan. Kriteria dikatakan sudah memenuhi apabila ( Hermadi et al.2014) :
1. Banyaknya generasi maksimum
2. Mencapai durasi maksimum (penenutuan durasi dilakukan sejak awal proses)
3. Nilai fitness terbaik sudah tidak mengalami peningkatan selama beberapa generasi terakhir (data fitness epsilon yang merupakan peningkatkan nilai fitness)
4. Sudah mencapai nilai fitness tertentu yang ditentukan sebelumnya. 5. Menggunakan kriteria berhenti dinamis setelah tingkat keyakinan
dari nilai fitness terbaik tercapai.
Kriteria di atas menandakan telah didapatkan parameter C & gamma () yang optimal. Apabila kriteria pemberhentian belum terpenuhi maka dilanjutkan proses generate offspring.
Seleksi
17 terpilih. Proses seleksi akan menghasilkan 25 kromosom yang berperan sebagai induk (Cox 2005).
Crossover
Proses crossover dilakukan dalam dua tahapan, yaitu memilih kromosom yang akan dikenai crossover dan melakukan crossover pada kromosom terpilih (Cox 2005). Pada tahap pertama, dibangkitkan suatu bilangan acak untuk setiap kromosom dalam populasi. Kemudian setiap bilangan tersebut dibandingkan dengan peluang crossover. Kromosom yang akan dipilih untuk dikenai crossover adalah kromosom yang memiliki bilangan acak lebih kecil dari nilai peluang crossover. Pada penelitian ini digunakan peluang crossover sebesar 60%. Selanjutnya, pada tahap kedua akan dibangkitkan sebuah variabel n yang berisi bilangan acak dari 1 sampai 10 untuk setiap pasang kromosom. Bilangan ini berguna untuk menentukan posisi gen untuk memulai proses crossover. Proses crossover dilakukan dengan menukar gen pertama sampai ke-n pada kromosom yang satu dengan gen ke-(n+1) sampai ke-10 pada kromosom yang lain.
Mutasi
Mutasi berfungsi untuk menjaga keberagaman dari populasi. Proses mutasi dilakukan pada offspring (kromosom anak) hasil dari proses crossover. Proses mutasi merupakan proses merubah nilai suatu gen dari suatu kromosom. Mutasi merupakan teknik ini bekerja pada satu kromosom berperan mengubah struktur kromosom.
Proses mutasi juga terdapat peluang mutasi yang merupakan kemungkinan sebuah kromosom akan terpilih untuk dilakukan mutasi. Persamaan 13 menunjukkan standar nilai peluang mutasi yang baik (Cox 2005).
) 1 , 01 , 0 (
N Max
Pm (13)
m
P = Peluang Mutasi ; N = Ukuran Populasi
Pada penelitian ini digunakan peluang mutasi sebesar 10% sehingga proses mutasi akan dilakukan pada 25 gen di setiap populasi. Proses pemilihan gen yang akan dimutasi dilakukan secara acak. Gen yang terpilih selanjutnya akan diisi dengan bilangan acak 0 atau 1.
Elitisme
18
Optimasi parameter Kenel RBF dengan Grid Search
Optimasi dengan grid search menggunakan data latih dengan jumlah fragmen 9600. Tahapan ini dilakukan dengan fungsi grid search yang sudah tersedia pada library SVM bernama LibSVM (Hsu et al. 2003) yang dapat diunduh pada alamat http://www.csie.ntu.edu.tw/~cjlin/libsvm. Fungsi grid search mengeluarkan nilai parameter terbaik yang dibutuhkan saat pembentukan model (tahap pelatihan) menggunakan kernel RBF. Parameter untuk kernel RBF ialah cost (c) dan gamma (γ). Algoritme Grid Search bertujuan sebagai algoritme pembanding dengan algoritme genetika. Diagram alir estimasi parameter dengan menggunakan algoritme grid search ditunjukan pada Gambar 15.Tahap klasifikasi SVM diawali dengan pelatihan SVM untuk data latih hasil ekstraksi fitur dengan jumlah fragmen 320 ribu. Dalam pelatihan ini, akan diterapkan pelatihan menggunakan kernel Gaussian radial basis function (RBF). Hasil dari pelatihan SVM ini ialah sebuah model yang dapat menguji data uji nantinya. Kemudian pengujian SVM akan memprediksi kelas untuk setiap fragmen data uji dan menghitung persentase banyaknya data uji yang telah diprediksi ke kelasnya dengan benar.
Mulai
Data Fragmen Metagenom
Scaling
Selesai Parameter
Optimal
) , (C
Menentukan nilai parameter pada rentang tertentu
Parameter ≤ 1
Training Klasifikasi SVM
Memilih Parameter Terbaik
Paramater 1 Optimal ?
Menentukan Nilai parameter pada rentang yang lebih kecil
Training Klasifikasi SVM
Paramater 2 Optimal ?
Kriteria memenuhi ? Tidak
Tidak
Ya Tidak
Ya ya
Tidak
Ya
[image:32.595.94.449.344.732.2]Optimasi SVM- Grid Search
19 Prinsip Grid Search yaitu memilih parameter terbaik dengan menentukan nilai parameter pada rentang tertentu untuk setiap parameter untuk menghitung performansi dengan k-fold cross validation, kemudian pilih nilai terbaik. Untuk rentang nilai parameter yang digunakan pada penelitian ini adalah menggunakan hasil penelitian Ariny (2013) yaitu dengan nilai sebagai berikut :
0.007813,0.031250,0.125000
10 , 8 , 2
C
(14)
Selanjutnya melakukan pencarian ulang pada grid (rentang) nilai yang lebih kecil. Kelemahan dari metode ini melakukan pencarian pada grid yang terlalu kecil yang mengakibatkan overfitting.
Pengujian SVM
Hasil dari pelatihan SVM sebelumnya ialah sebuah model yang akan diuji menggunakan hasil ekstraksi fitur dari data uji. Pengujian akan mengklasifikasikan data uji sebanyak 200 organisme ke dalam kelas genus. Semua organisme yang telah dikelaskan menghasilkan persentase hasil pengklasifikasiannya.
Analisis
Dari hasil pengoptimalan parameter SVM dengan algoritme genetika, parameter akan diujikan pada data training untuk setiap panjang fragmen sehingga dihasilkan model klasifikasi SVM. Kemudian akurasi untuk hasil klasifikasi dapat dihitung dengan Persamaan (15) :
% 100
uji data
benar uji data
akurasi (15)
Selain akurasi, juga dianalisis dari nilai sensitivity dan specificity yang dikelompokkan berdasarkan level genusnya. Persamaan untuk menghitung nilai sensitivity dan specificity, yaitu
en t t
∑true positi es ∑true positi es∑ false negati es
100%
(16)ec c t
∑true negati es ∑ true negati es∑ false positi es
100%
(17)20
4
HASIL DAN PEMBAHASAN
Penyiapan Data
Data yang digunakan pada penelitian ini adalah data metagenome yang diunduh dari situs National Centre for Biotechnology Information (NCBI). Data metagenome ini merupakan hasil sequences DNA mikroorganisme. Pada penelitian ini jumlah organisme yang digunakan terbatas pada 381 organisme untuk data latih dan 200 organisme untuk data uji. Daftar organisme untuk data latih dan data uji yang digunakan pada penelitian ini dapat dilihat pada Lampiran 1 dan Lampiran 2. Data yang telah diunduh dari situs NCBI akan diuraikan fragmennya meggunakan perangkat lunak MetaSim. MetaSim adalah perangkat lunak untuk mensimulasikan sequencer. Fail yang berisi sequences DNA mikroorganisme yang telah diunduh dari NCBI dimasukkan ke dalam perangkat lunak tersebut. Setelah memasukkan data dari NCBI ke dalam perangkat lunak MetaSim, proses selanjutnya adalah memilih beberapa sequences DNA mikroorganisme yang telah tersedia pada database sesuai dengan kebutuhan penelitian. Pada penelitian ini data yang disipakan utnuk data latih berjumlah 9600 dan 320 000 fragmen, sedangkan untuk data uji berjumlah 100 000 fragmen. Panjang fragmen yang ditentukan untuk setiap kali pengolahan yaitu 400 bp, 800 bp, 1 Kbp, 3 Kbp, 5 Kbp, dan 10 Kbp. Akan dilakukan 12 kali pengolahan untuk data latih dan 6 kali pengolahan untuk data uji, sehingga dihasilkan 18 fail FastA yang berisi fragmen sesuai dengan kebutuhan penelitian. Data latih dengan jumlah fragmen 9600 disiapkan sebagai data pendekatan pencarian parameter terbaik untuk kernel, sedangkan data latih dengan jumlah fragmen 320 ribu menjadi data masukan untuk pembuatan model. Penggunaan data latih kecil sebagai pendekatan pencarian paramater terbaik ini didasarkan pada percobaan yang dilakukan oleh McHardy et al. (2007).
Ekstrasi Fitur
21 Berikut contoh hasil ekstraksi fitur untuk data latih takson genus dengan jumlah fragmen 9600 dan panjang fragmen 400 bp :
Proses Support Vector Machine
Tahap selanjutnya setelah dilakukan ekstrasi fitur pada data fragmen metagenom adalah melakukan klasifikasi dengan SVM. Klasifikasi data terdiri dari 48 kelas. Pelatihan SVM menggunakan fungsi kernel Radial Basis Function. Kernel RBF memiliki parameter C dan yang harus dioptimalkan untuk menghasilkan model SVM yang baik. Model SVM yang baik dapat dilihat dari ketepatan antara kelas yang didapatkan dari model SVM dengan kelas yang sebenarnya. Pengoptimalan parameter kernel RBF pada penelitian ini menggunakan Algoritme Genetika. Parameter yang akan dihasilkan dari optimasi menggunakan algoritme Genetika akan menjadi masukan dalam pembentukan model SVM. Pelatihan dilakukan terhadap 6 panjang fragmen yang berbeda, yaitu panjang fragmen yaitu 400 bp, 800 bp, 1 Kbp, 3 Kbp, 5 Kbp, dan 10 Kbp. Model yang telah dihasilkan dari pelatihan sebelumnya digunakan untuk mengklasifikasikan data uji yang merepresentasikan fragmen metagenom dari organisme-organisme baru. Dari pengujian ini akan diperoleh akurasi dari setiap kelas yang ada pada level takson genus
Pengujian Hasil Optimasi dan Analisis Akurasi
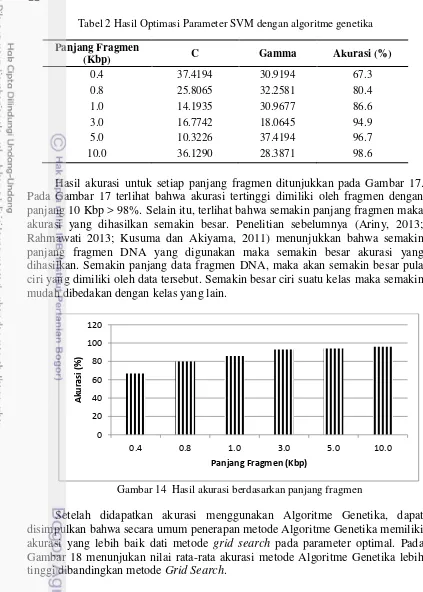
Analisis dilakukan dengan menguji data latih yang memiliki panjang fragmen berbeda. Data latih dari setiap panjang fragmen dijadikan model untuk membangun model klasifikasi dengan SVM. Parameter yang digunakan dalam membangun model ini diperoleh dari optimasi algoritme genetika. Data uji yang digunakan sama untuk setiap model klasifikasi. Confusion matrix merepresentasikan hasil klasifikasi untuk masing-masing kelas. Analisis dilakukan dengan akurasi (dalam hal ini sama dengan nilai fitness) terbaik yang dihasilkan dari optimasi dengan parameter SVM yaitu C dan gamma () dengan algoritme genetika. Hasil optimasi parameter SVM yang didapatkan pada setiap panjang fragmen terlihat pada Tabel 2.
22
Tabel 2 Hasil Optimasi Parameter SVM dengan algoritme genetika
Panjang Fragmen
(Kbp) C Gamma Akurasi (%)
0.4 37.4194 30.9194 67.3
0.8 25.8065 32.2581 80.4
1.0 14.1935 30.9677 86.6
3.0 16.7742 18.0645 94.9
5.0 10.3226 37.4194 96.7
10.0 36.1290 28.3871 98.6
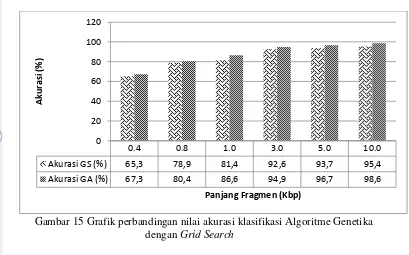
[image:36.595.116.466.110.232.2]Hasil akurasi untuk setiap panjang fragmen ditunjukkan pada Gambar 17. Pada Gambar 17 terlihat bahwa akurasi tertinggi dimiliki oleh fragmen dengan panjang 10 Kbp > 98%. Selain itu, terlihat bahwa semakin panjang fragmen maka akurasi yang dihasilkan semakin besar. Penelitian sebelumnya (Ariny, 2013; Rahmawati 2013; Kusuma dan Akiyama, 2011) menunjukkan bahwa semakin panjang fragmen DNA yang digunakan maka semakin besar akurasi yang dihasilkan. Semakin panjang data fragmen DNA, maka akan semakin besar pula ciri yang dimiliki oleh data tersebut. Semakin besar ciri suatu kelas maka semakin mudah dibedakan dengan kelas yang lain.
Gambar 14 Hasil akurasi berdasarkan panjang fragmen
Setelah didapatkan akurasi menggunakan Algoritme Genetika, dapat disimpulkan bahwa secara umum penerapan metode Algoritme Genetika memiliki akurasi yang lebih baik dati metode grid search pada parameter optimal. Pada Gambar 18 menunjukan nilai rata-rata akurasi metode Algoritme Genetika lebih tinggi dibandingkan metode Grid Search.
0 20 40 60 80 100 120
0.4 0.8 1.0 3.0 5.0 10.0
A
ku
rasi
(
%
)
[image:36.595.97.481.380.551.2]23
Gambar 15 Grafik perbandingan nilai akurasi klasifikasi Algoritme Genetika dengan Grid Search
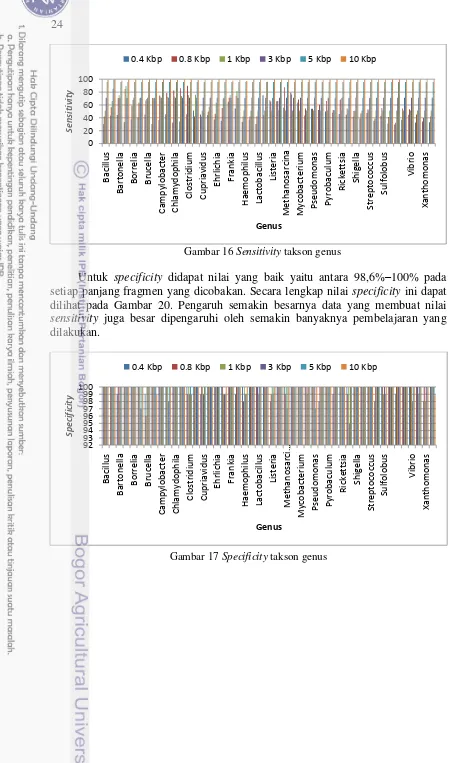
Sensitivity dan Specificity
Representasi sensitivity dan specificity terdapat pada Gambar 19, dan Gambar 20 Sensitivity merupakan proporsi dari hasil kelas positif yang benar dari seluruh data kelas positif (Manning et al. 2008). Dengan kata lain sensitivity dapat dikatakan sebagai kemampuan model dalam mengklasifikasikan data dalam suatu kelas secara benar diprediksi ke dalam kelasnya. Perhitungan sensitivity dan specificity pada penelitian ini dibatasi pada takson genus saja. Penelitian dengan ekstraksi fitur menggunakan spaced k-mers dan pengklasifikasian menggunakan metode SVM ini dapat menghasilkan sensitivity yang baik pada level takson genus, yang dapat dilihat pada Gambar 7. Nilai sensitivity yang didapat pada panjang fragmen 400 bp yaitu berada di antara 30.2%–79.3% dengan rata-rata sensitivity-nya 52,6%. Nilai sensitivity yang didapat pada panjang fragmen 10 Kbp yaitu berada di antara 87.1%–100% dengan rata-rata sensitivity-nya 95.3%. Nilai sensitivity ini menunjukkan bahwa setidaknya ada 73.2% data pada suatu kelas dapat diklasifikasikan ke kelas sebenarnya.
0.4 0.8 1.0 3.0 5.0 10.0
Akurasi GS (%) 65,3 78,9 81,4 92,6 93,7 95,4
Akurasi GA (%) 67,3 80,4 86,6 94,9 96,7 98,6
0 20 40 60 80 100 120
A
ku
rasi
(
%
)
24
Gambar 16 Sensitivity takson genus
Untuk specificity didapat nilai yang baik yaitu antara 98,6%–100% pada setiap panjang fragmen yang dicobakan. Secara lengkap nilai specificity ini dapat dilihat pada Gambar 20. Pengaruh semakin besarnya data yang membuat nilai sensitivity juga besar dipengaruhi oleh semakin banyaknya pembelajaran yang dilakukan.
Gambar 17 Specificity takson genus
0 20 40 60 80 100 Bac il lu s B arto n e ll a Borre li a Bru ce ll a Cam p y lo b act e r Chl a m y d o p h il a Cl o stri d iu m Cup ri av id u s E h rl ic h ia Fran ki a H aem o p h il u s La cto b aci ll u s Li steri a Me th an o sar ci na My cob act e ri u m Ps e u d o m o n as Py ro b a cu lu m Ri ckett si a Sh ige ll a Stre p to co ccus Su lf o lob u s Vi b ri o Xan th om on as S en si tivi ty Genus
0.4 Kbp 0.8 Kbp 1 Kbp 3 Kbp 5 Kbp 10 Kbp
92 93 94 95 96 97 98 99 100 Bac il lu s Ba rt o n el la Bo rr e li a Bru ce ll a Cam p y lo b act e r Chl a m y d o p h il a Cl o stri d iu m Cup ri av id u s E h rl ic h ia Fran ki a H aem o p h il u s La cto b aci ll u s Li steri a Me th an os arc i… My cob act e ri u m Ps e u d o m o n as Py ro b a cu lu m Ri ckett si a Sh ige ll a Stre p to co ccus Su lf o lob u s Vi b ri o Xan th o m o n as S pe ci fi ci ty Genus
25
5
SIMPULAN DAN SARAN
Simpulan
Simpulan dari hasil penelitian ini adalah :
1. Algoritme Genetika dapat digunakan untuk optimasi parameter RBF pada SVM.
2. Secara rata-rata nilai akurasi, sensitivity dan specificity yang dihasilkan algoritme genetika lebih besar dibandingkan algoritme Grid Search.
3. Hasil percobaan menunjukan nilai akurasi yang terbaik sebesar 98,6 % pada panjang fragmen 10 Kbp dengan parameter optimal C adalah 36.1290 dan adalah 28.3871.
Saran
26
DAFTAR PUSTAKA
Ariny. 2013. Klasifikasi Fragmen Metagenom Menggunakan Metode Support Vector Machine (SVM). [skripsi].Bogor(ID) : IPB.
Chan CK, Hsu AL, Tang SL, Halgamuge SK. 2007. Using Growing Self-Organizing Maps to Prove the Binning Process in Environmental Whole-Genome Shotgun Sequencing. Journal of Biomedicine and Biotechnology. 2008. doi:10.1155/2008/513701
Christianini, Nello. 2001. Support Vector and Kernel Machines. ICML tutorial. Cox E. 2005. Fuzzy Modeling and Genetic Algorithms for Data Mining and
Exploration. San Francisco (US): Morgan Kaufmann Publishers.
Cristianini N, and Taylor JS.2000. An Introduction to Support Vector Machines and other kernel-based learning methods;Cambridge University Press, UK. Fro¨hlich, H., & Chapelle, O. 2003. Feature selection for support vector machines
by means of genetic algorithms. Proceedings of the 15th IEEE international conference on tools with artificial intelligence. pp. 142–148. Sacramento, CA, USA
Goldberg, David E .1989. Genetic Algorithms in Search, Optimization and Machine Learning; Kluwer Academic Publishers;Boston, MA.
Harayama S, Kasai Y, Hara A. 2004. Microbial Communities in Oil-contaminated Seawater. Current Opinion in Biotechnology. 15:205-214
Hsu CW, Chang CC, Lin CJ. 2003. A practical guide to support vector classification [Internet]. [diunduh 2013 Sep 20]. Tersedia pada: http://www.csie.ntu.edu.tw/~cjlin.
Hsu, C. W., & Lin, C. J. 2002. A simple decomposition method for support vector machine. Machine Learning, 46(1–3), 219–314.
Huang CL, Wang CJ. 2006. A GA-based feature selection and parameters optimization for support vector machines.31(2006) 231-240; Taiwan.
Izenman AJ. 2008. Modern Multivariate Statistical Techniques (Regression, Classification, and Manifold Learning). USA: Springer.
James B, Riggs. 1988. An Introduction to Numerical Methods for Chemical Engineers.Texas: Texas Tech University Press, Chapter 6.
Joachims, T. (1998). Text categorization with support vector machines. In Proceedings of European conference on machine learning (ECML) (pp.137– 142). Chemintz, DE.
Kahan W. 1997. IEEE Standard for Floating-Point Arithmetic. Lecture Note from University of California.
Keerthi SS,Shevade SK, Bhattacharyya C and Murthy KRK.2001. Improvements to Platt’s SMO Algorithm for SVM Classifier Design, Neural Computation ;13; 637–649.
Khotimah BK, Arifin AZ dan Yuniarti Anny. 2010. Optimasi parameter pada klasifikasi Fuzzy Artmap berbobot berbasis algoritma genetika. Tesis. Fakultas Teknologi Informasi. ITS. Surabaya
Kusuma WA, Akiyama Y. 2011. Metagenome fragment binning based on characterization vector. Di dalam: International Conference on Bioinformatics and Biomedical Technology (ICBBT 2011); 2011 Mar 25–27; Sanya, China. Kusuma, WA. 2012. Combined approaches for improving the performance of de
27 fragments from next generation sequencer [disertasi]. Tokyo (JP): Tokyo Institute of Technology.
McHardy AC Rigoutsos I. 007. What’s in the mi : phy ogenetic c assification of metagenome sequence samples. Current Opinion in Microbiology. 10(5):499– 503. doi: 10.1016/j.mib.2007.08.004.
McHardy AC, Martín HG, Tsirigos A, Hugenholtz P, Rigoutsos I. 2007. Accurate phylogonetic classification of variabel-length DNA fragments. Nature Methods. 4(1):63–72. doi: 10.1038/nmeth976.
Meyerdierks A, Glockner FO. 2010. Metagenome Analysis. Advances in Marine Genomics. 1 : 33 – 71. doi : 10.1007/978-90-481-8639-6_2
O’Malley M. 2012. Metagenomics. Springer [Internet].[diunduh 2013 Sep 29]. Tersedia pada : http://www.maureenomalley.org/publications.html
Osuna EE, Freund R, Girosi F. 1997. Support vector machines: training and applications. AI Memo (1602).
Pontil, M., Verri, A. 1998. Support vector machines for 3D object recognition. IEEE Transactions on Pattern Analysis and Machine Intelligence, 20(6), 637– 646.
Rahmawati V. 2013. Perbandingan Ekstraksi Ciri K-Mers Dan Spaced K-Mers Pada Klasifikasi Fragmen Metagenome Dengan Naïve Bayes Classifier. [skripsi].Bogor(ID) : IPB.
Raymer, M. L., Punch, W. F., Goodman, E. D., Kuhn, L. A., Jain, A. K. 2000. Dimensionality reduction using genetic algorithms. IEEE Transactions on Evolutionary Computation, 4(2), 164–171.
Salcedo-Sanz, S., Prado-Cumplido, M., Pe´rez-Cruz, F., & Bouson˜o-Calzo´n, C.2002. Feature selection via genetic optimization Proceedings of the ICANN international conference on artificial neural networks, Madrid, Span pp. 547– 552. Salzberg, S. L. 1997. On comparing classifiers: Pitfalls to avoid and a recommended approach. Data Mining and Knowledge Discovery, 1, 317–327. Sastry K, Goldberg D, Kendall G. 2005. Genetic Algorithms. Search
Methodologies. 2005(1):97-125. doi: 10.1007/0-387-28356-0_4.
Scho˝lkopf, B., & Smola, A. J. 2000. Statistical learning and kernel methods.Cambridge, MA: MIT Press
Steven C, Chapra & Raymond P, Canale. 2003. Numerical Methods for Engineers: With Software and Programming Applications. 4th edition. New York: McGraw-Hill Company Inc, Part Four.
Tan PN, Steinbach M. dan Kumar V.2006. Introduction to Data Mining, Pearson Education, Inc., Boston.
Thontowi A. 2009. Pendekatan metagenomik dan bioinformatika untuk menganalisis komunitas mikroba laut Indonesia. SIGMA. 12(1):15-22.
Utami, N.D. 2008. Analisis Teknik Crossover Pada Penyelesaian Penjadwalan Praktikum Dengan Algoritma Genetika. Skripsi. Fakultas Matematika dan Ilmu Pengetahuan Alam. Universitas Brawijaya. Malang.
Vapnik, V. N. 1995. The nature of statistical learning theory. New York:Springer. Yu, G. X., Ostrouchov, G., Geist, A., & Samatova, N. F. (2003). An SVMbased
28
Lampiran 1 Daftar nama organisme data latih
No Nama Organisme No Nama Organisme
1 Bacillus amyloliquefaciens FZB42 23 Bartonella tribocorum CIP 105476
2 Bacillus anthracis str. 'Ames
Ancestor'
24 Bordetella avium 197N chromosome
3 Bacillus anthracis str. Ames
chromosome
25 Bordetella bronchiseptica RB50
4 Bacillus anthracis str. Sterne
chromosome
26 Bordetella parapertussis 12822
5 Bacillus cereus ATCC 10987
chromosome
27 Bordetella pertussis Tohama I
6 Bacillus cereus ATCC 14579 28 Bordetella petrii DSM 12804
7 Bacillus cereus E33L 29 Borrelia afzelii PKo
8 Bacillus cereus subsp. cytotoxis NVH 391-98
30 Borrelia duttonii Ly
9 Bacillus clausii KSM-K16 31 Borrelia garinii PBi chromosome
chromosome linear
10 Bacillus halodurans C-125
chromosome
32 Borrelia hermsii DAH chromosome
11 Bacillus licheniformis ATCC 14580 33 Borrelia recurrentis A1
12 Bacillus subtilis subsp. subtilis str. 168 chromosome
34 Borrelia turicatae 91E135
chromosome
13 Bacillus thuringiensis serovar
konkukian str. 97-27 chromosome
35 Bradyrhizobium japonicum USDA
110 chromosome
14 Bacillus thuringiensis str. Al Hakam chromosome
36 Bradyrhizobium sp. BTAi1
chromosome
15 Bacillus weihenstephanensis KBAB4 37 Bradyrhizobium sp. ORS278
chromosome
16 Bacteroides fragilis NCTC 9343
chromosome
38 Brucella abortus S19 chromosome 1
17 Bacteroides fragilis YCH46
chromosome
39 Brucella abortus bv. 1 str. 9-941 chromosome chromosome I
18 Bacteroides thetaiotaomicron VPI-5482 chromosome
40 Brucella canis ATCC 23365
chromosome I
19 Bacteroides vulgatus ATCC 8482
chromosome
41 Brucella melitensis biovar Abortus 2308 chromosome chromosome I
20 Bartonella bacilliformis KC583 42 Brucella melitensis bv. 1 str. 16M chromosome chromosome I
21 Bartonella henselae str. Houston-1 43 Brucella ovis ATCC 25840
chromosome chromosome I
22 Bartonella quintana str. Toulouse 44 Brucella suis 1330 chromosome
29 Lampiran 1 Lanjutan
No Nama Organisme No Nama Organisme
45 Brucella suis ATCC 23445 chromosome I
65 Burkholderia thailandensis E264 chromosome chromosome I 46 Burkholderia ambifaria AMMD
chromosome chromosome 1
66 Burkholderia vietnamiensis G4 chromosome chromosome 1 47 Burkholderia ambifaria MC40-6
chromosome chromosome 1
67 Burkholderia xenovorans LB400 chromosome 1
48 Burkholderia cenocepacia AU 1054 chromosome 3
68 Campylobacter concisus 13826 49 Burkholderia cenocepacia
HI2424 chromosome chromosome 1
69 Campylobacter curvus 525.92 chromosome
50 Burkholderia cenocepacia J2315 chromosome chromosome 1
70 Campylobacter fetus subsp. fetus 82-40
51 Burkholderia cenocepacia MC0-3 chromosome chromosome 1
71 Campylobacter hominis ATCC BAA-381
52 Burkholderia mallei ATCC 23344 chromosome chromosome 1
72 Campylobacter jejuni RM1221 53 Burkholderia mallei NCTC 10229
chromosome I
73 Campylobacter jejuni subsp. doylei 269.97
54 Burkholderia mallei NCTC 10247 chromosome I
74 Campylobacter jejuni subsp. jejuni NCTC 11168 chromosome 55 Burkholderia mallei SAVP1
chromosome I
75 Candidatus Phytoplasma australiense
56 Burkholderia multivorans ATCC 17616 chromosome chromosome 1
76 Candidatus Phytoplasma mali
57 Burkholderia phymatum STM815 chromosome chromosome 1
77 Chlamydophila abortus S26/3 58 Burkholderia phytofirmans PsJN
chromosome chromosome 1
78 Chlamydophila caviae GPIC 59 Burkholderia pseudomallei 1106a
chromosome I
79 Chlamydophila felis Fe/C-56 60 Burkholderia pseudomallei 1710b
chromosome chromosome I
80 Chlamydophila pneumoniae AR39
61 Burkholderia pseudomallei 668 chromosome I
81 Chlamydophila pneumoniae CWL029
62 Burkholderia pseudomallei
K96243 chromosome
chromosome 1
82 Chlamydophila pneumoniae J138
63 Burkholderia sp. 383 chromosome 1
83 Chlamydophila pneumoniae TW-183
64 Burkholderia sp. 383 chromosome chromosome 2
30
Lampiran 1 Lanjutan
No Nama Organisme No Nama Organisme
85 Chlorobium limicola DSM 245 chromosome
107 Clostridium tetani E88 chromosome
86 Chlorobium luteolum DSM 273 chromosome
108 Clostridium thermocellum ATCC 27405 chromosome
87 Chlorobium phaeobacteroides BS1 chromosome
109 Corynebacterium diphtheriae NCTC 13129 chromosome
88 Chlorobium phaeobacteroides DSM 266 chromosome
110 Corynebacterium efficiens YS-314
89 Chlorobium phaeovibrioides DSM 265 chromosome
111 Corynebacterium glutamicum ATCC 13032
90 Chlorobium tepidum TLS 112 Corynebacterium glutamicum R chromosome
91 Clostridium acetobutylicum ATCC 824
113 Corynebacterium jeikeium K411 92 Clostridium beijerinckii NCIMB
8052 chromosome
114 C