VARIASI DNA KLOROPLAS Shorea spp (Shorea acuminata
Dyer, Shorea leprosula Miq dan Shorea parvifolia Dyer)
BERDASARKAN PENANDA MIKROSATELIT
ZULFAHMI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
VARIASI DNA KLOROPLAS Shorea spp (Shorea acuminata
Dyer, Shorea leprosula Miq dan Shorea parvifolia Dyer)
BERDASARKAN PENANDA MIKROSATELIT
ZULFAHMI
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Ilmu Pengetahuan Kehutanan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
ABSTRACT
ZULFAHMI. Chloroplast DNA Variation in Shorea spp (Shorea acuminata Dyer, Shorea leprosula Miq and Shorea parvifolia Dyer) Based on Microsatellite Marker. Under supervision of ISKANDAR ZULKARNAEN SIREGAR and ULFAH JUNIARTI SIREGAR
Shorea acuminata, Shorea leprosula, and Shorea parvifolia are members of the Diptecarpaceae family. They are ecologically and commercially important in the Southeast Asia region. In this present study, we used chloroplast microsatellites (cpSSR) to study the variation within and among Shorea species populations and the distribution of chloroplast DNA haplotypes of Shorea species. Based on chloroplast microsatellite analysis, the following diagnostic haplotypes were detected; namely haplotype A, C, and D for S. leprosula, S. parvifolia, and S. acuminata, respectively. In general, haplotype variation of cpSSR was low in this studies. The highest within population variation of cpSSR was observed in S. acuminata. The highest genetic differentiation was detected in S. parvifolia (Gst = 0.58) followed by S. acuminata (Gst = 0.31) and S. leprosula (Gst = 0.14). The haplotypes distribute evenly among different populations and between islands, and do not correspond with geographical separation among populations and islands. Some haplotypes from one species were shared by other species, indicating the possibility of gene flow via interspecific hybridization, that occurred currently and/or in the past. Introgressive hybridization probably occurred among Shorea species due to co-existance in the forest stands and overlapping flowering times.
ABSTRAK
ZULFAHMI. Variasi DNA Kloroplas Shorea spp (Shorea acuminata Dyer, Shorea leprosula Miq dan Shorea parvifolia Dyer) Berdasarkan Penanda Mikrosatelit. Di bawah Bimbingan ISKANDAR ZULKARNAEN SIREGAR dan ULFAH JUNIARTI SIREGAR
Shorea acuminata, Shorea leprosula, dan Shorea parvifolia adalah anggota famili Dipterocarpaceae, yang secara ekologis dan ekonomis sangat penting di daerah Asia Tenggara. Dalam studi ini, digunakan kloroplas mikrosatelit (cpSSR) untuk mengetahui variasi genetika di dalam dan antar populasi jenis Shorea dan mengamati distribusi haplo tipe DNA kloroplas dari ketiga jenis tersebut. Hasil menunjukkan bahwa terdapat beberapa haplotipe diagnostik yang dideteksi, berturut-turut adalah haplotipe A, C, dan D untuk S. leprosula, S. parvifolia, dan
S. acuminata. Variasi cpSSR di dalam populasi tertinggi diamati pada S. acuminata. Secara umum, variasi haplotype cpSSR adalah rendah dalam studi ini. Diferensiasi genetik tertinggi dideteksi pada S. parvifolia (Gst = 0.58) diikuti oleh
S. acuminata (Gst = 0.31) dan S. leprosula (Gst = 0.14). Semua haplotipe menyebar merata pada populasi yang ada di setiap pulau, sehingga tidak ditemukan asosiasi antara distribusi haplotipe dengan pemisahan secara geografis. Beberapa haplotipe dari satu jenis ternyata juga ditemukan pada jenis lain, mengindikasikan kemungkinan adanya aliran gen melalui interspesifik hibridisasi yang terjadi sekarang dan/atau pada waktu lampau. Introgresif hibridisasi mungkin terjadi di antara jenis Shorea karena mereka tumbuh alami pada areal yang sama dalam hutan dan waktu pembungaan mereka yang tumpang tindih.
SURAT PERNYATAAN
Dengan ini saya menyatakan bahwa Tesis Variasi DNA Kloroplas Shorea
spp (Shorea acuminata, Dyer, Shorea leprosula, Miq dan Shorea parvifolia, Dyer) Berdasarkan Penanda Mikrosatelit adalah karya saya sendiri di bawah Bimbingan Dr. Ir. Iskandar Zulkarnaen Siregar, M.For.Sc dan Dr. Ir. Ulfah Juniarti Siregar, M.Agr belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan mau pun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka di bagian akhir Tesis ini.
Bogor, April 2006
Zulfahmi
Hak cipta milik Zulfahmi, tahun 2006 Hak cipta dilindungi
Judul Tesis : Variasi DNA Kloroplas Shorea spp (Shorea acuminata Dyer, Shorea leprosula Miq dan Shorea parvifolia Dyer) Berdasarkan Penanda Mikrosatelit.
Nama : Zulfahmi NIM : E051030211
Disetujui Komisi Pembimbing
Dr. Ir. Iskandar Z Siregar, M.For.Sc Dr. Ir. Ulfah J Siregar, M.Agr
Ketua Anggota
Diketahui
Ketua Program Studi Ilmu Dekan Sekolah Pascasarjana Pengetahuan Kehutanan
Dr. Ir. Dede Hermawan, M.Sc Prof. Dr. Ir. Syafrida Manuwoto, M.Sc.
PRAKATA
Puji dan syuk ur penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian adalah Variasi Genetik DNA Kloroplas dengan judul Tesis adalah Variasi DNA Kloroplas Shorea spp (Shorea acuminata, Dyer, Shorea leprosula, Miq dan Shorea parvifolia, Dyer) Berdasarkan Penanda Mikrosatelit.
Terima kasih penulis ucapkan kepada: Dr. Ir. Iskandar Zulkarnaen Siregar, M. For. Sc dan Dr. Ir. Ulfah Juniarti Siregar, M. Agr selaku dosen pembimbing, Prof. Dr. Reiner Finkeldey yang telah memberi kesempatan kepada penulis untuk melakukan penelitian di Institut Genetika Hutan dan Pemuliaan Pohon Hutan Universitas Goettingen Jerman, Dr. Oliver Gailing, Dr. Ludger Leinemann, Dr. Barbara Vornam, Prof. Dr. Martin Ziehe, Dr. Elizabeth Gillet yang telah membimbing penulis selama melakukan penelitian di Universitas Goettingen Jerman. Ucapan terima kasih juga penulis sampaikan kepada DFG (Deutsche Forschungsgemeinschaft) dan AUNP (Asean-EU University Network Programme) melalui proyek Genetic Variation of Shorea spp in Indonesian dan
Conservation and Sustainable Utilization of Plant Genetic Resources in SE-Asia
yang telah mendanai penelitian serta kepada Pemerintah Provinsi Riau atas Beasiswa yang diberikan penulis dala m menempuh pendidikan Magister di IPB-Bogor.
Ungkapan terima kasih juga disampaikan kepada Ayah, Ibu, Paman, abang, dan adik-adikku tercinta serta seluruh keluarga atas segala doa, kasih sayang dan pengorbanan serta istri tercinta (Rosmaina, S.P) atas semangat dan pengertian yang diberikan. Penulis juga mengucapkan terima kasih kepada Yanti Rachma yanti dan Ms. Olga Artes yang telah membantu pelaksanaan penelitian. Akhirnya penulis mengucapkan semoga karya ilmiah ini bermanfaat.
Bogor, April 2006
RIWAYAT HIDUP
i
DAFTAR ISI
Halaman
DAFTAR TABEL... ii
DAFTAR GAMBAR ... iii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan Penelitian... 3
Hipotesis ... 3
Manfaat Penelitian... 3
TINJAUAN PUSTAKA... 4
Shorea acuminata Dyer, S. leprosula Miq, dan S. parvifolia Dyer ... 4
Karakteristik DNA kloroplas dan Penggunaannya ... 6
Penanda Mikrosatelit ... 7
Penelitian Tentang Variasi Genetika Jenis Shorea spp ...11
BAHAN DAN METODE ...13
Tempat dan Waktu Penelitian...13
Alat dan Bahan Penelitian...13
Metode Penelitian...13
Koleksi Sampel ...13
Ekstraksi DNA dan PCR-cpSSR (Kloroplas Mikrosatelit)...15
Genescan dan Genotyping Hasil PCR...16
Analisis Data ...17
HASIL DAN PEMBAHASAN ...19
Haplotipe cpSSR...19
Variasi Haplotipe...21
Variasi Genetik di Dalam dan Antar Populasi ...23
Pembagian Haplotipe Antar Jenis ...29
Implikasi Terhadap Konservasi Genetika Shorea spp di Indonesia...33
SIMPULAN ...34
DAFTAR PUSTAKA ...35
Halaman 1 Nilai keragaman genetik beberapa jenis Shorea menggunakan penanda
isozim, RAPD, mikrosatelit dan AFLP ...12
2 Koleksi sampel berdasarkan populasi dan jumlah individu per populasi ...15
3 Sekuen DNA pasangan primer yang digunakan untuk amplifikasi cpSSR ...17
4 Definisi haplo tipe dan ukuran fragmen cpSSR...20
5 Perbedaan S. acuminata, S. leprosula dan S. parvifolia berdasarkan tipe mutasi ...21
6 Frekuensi haplotipe S. acuiminata, S. leprosula dan S. parvifolia pada setiap populasi ...22
7 Nilai keragaman genetika di dalam dan antar populasi pada S. acuminata, S. leprosula dan S. parvifolia...23
8 Analisis keragaman molekuler (AMOVA) ...24
iii
Halaman 1 Distribusi Dipterocarpaceae di Indonesia. Jumlah total jenis dalam setiap
pulau menurut Ashton (1982). ... 4
2 Diagram model slippage strand mispairing (SSM) pada mutasi mikrosatelit (Eisen 1999) ... 9
3 Bagan alur penelitian ...14
4 Lokasi pengambilan sampel di lapangan ...14
5 Lima primer yang berhasil diamplifikasi dengan PCR ...19
6 Hasil Genescan primer ccmp3 dan ccmp6 yang menunjukkan pola polimorfisme ...20
7 Distribusi geografis dan frekuensi haplotipe Shorea spp (S. acuminata, S. leprosula dan S. parvifolia) ...26
8 Dendogram UPGMA S. acuminata, S. leprosula dan S. parvifolia berdasarkan jarak genetik a Nei (1972)...28
9 Dendogram UPGMA S. acuminata, S. leprosula dan S. parvifolia berdasarkan jarak genetika Gregorious dan Roberds (1986) ...28
10 Waktu pembungaan pada beberapa jenis Shorea seksi Mutica famili Dipterocarpaceae di semenanjung Malaysia (Ashton 1988)...31
11 Mekanisme penangkapan kloroplas (chloroplast capture) pada Eucalyptus spp (Jackson et al. 1999) ...32
Halaman 1 Hasil GenescanS. acuminata, S. leprosula dan S. parvifolia menggunakan
PENDAHULUAN
Latar Belakang
Indonesia adalah salah satu negara dengan keanekaragaman jenis dipterokarp yang tinggi. Di sisi lain, laju deforestrasi hutan alam Indonesia adalah tinggi, diperkirakan mencapai 2.6 sampai 2.8 juta hektar per tahun (Kompas 2005), sehingga merupakan ancaman serius terhadap keberadaan jenis dipterokarp. Daerah-daerah di Sumatra dan Kalimantan yang di dominasi oleh jenis dipterokarp telah banyak mengalami kerusakan akibat penebangan illegal, kebakaran hutan, konversi lahan hutan menjadi lahan perkebunan, dan lain- lain sehingga perlu mendapat perhatian dan penanganan yang serius.
Penebangan hutan memberikan dampak negatif terhadap struktur genetik pohon hutan. Murawski et al. (1996) telah melaporkan adanya peningkatan derajat
selfing pada Shorea megistophylla setelah dilakukan penebangan pada hutan dipterokarp di Sri Lanka, sedangkan Obayashi et al. (2002) melaporkan bahwa terjadinya penurunan rata-rata outcrossing pada Shorea curtisii pada hutan bekas tebangan di Semananjung Malaysia.
Dalam studi ini, tiga jenis Shorea yaitu Shorea leprosula, Shorea parvifolia dan Shorea acuminata akan diteliti. Ketiga jenis Shorea tersebut dipilih karena beberapa alasan, pertama ketiga jenis tersebut termasuk ke dalam kelompok meranti merah yang bernilai ekonomis tinggi dan penting secara ekologis. Kedua, mereka menyebar luas di Indonesia, yaitu di Sumatra dan Kalimantan. Ketiga, S. leprosula dan S. parvifolia merupakan jenis dipterokarp yang cepat pertumbuhannya dibandingkan dengan jenis dipterokarp lainnya. Rata-rata riap diameter per tahun S. leprosula dan S. parvifolia masing- masing adalah 2.10 cm dan 1.87 cm (Suparna & Purnomo 2004) dan tinggi pohon S. leprosula
dan S. parvifolia pada umur dua tahun berturut-turut adalah 4.64 m dan 4.58 m (Soekotjo & Wardhana 2005), sehingga jenis ini dapat dikembangkan untuk pembangunan hutan tanaman meranti di Sumatra dan Kalimantan. Keempat
isoenzim, Rimbawanto & Isoda (2000) dengan penanda mikrosatelit, dan Indrioko (2005) dengan penanda PCR-RFLP dan mikrosatelit sehingga masih diperlukan lagi penelitian yang lebih banyak, untuk mendapatkan informasi keragaman genetika yang lebih detail.
Penggunaan metode penanda genetika dalam bidang kehutanan di Indonesia umumnya masih diarahkan untuk mendukung kegiatan konservasi sumber daya genetika dan pemuliaan pohon dari jenis-jenis unggulan, sedangkan untuk tujuan lain seperti melacak asal-usul kayu belum pernah dilakukan. Di era ekolabel, kayu yang berasal dari hutan yang dikelola secara lestari (sustainable forest management, SFM) merupakan produk yang sangat diinginkan oleh konsumen. Sertifikasi lacak balak (chain of custody) merupakan salah satu kegiatan utama sertifikasi ekolabel untuk memantau aliran kayu dari hutan ke tempat penimbunan sampai ke konsumen akhir. Sertifikasi lacak balak menuntut metode monitoring aliran kayu yang dapat diandalkan, sehingga kasus-kasus yang meragukan dapat dipecahkan. Penggunaan penanda molekuler seperti DNA merupakan salah satu metode yang dapat dikembangkan dan diterapkan di masa datang untuk sertifikasi lacak balak atau tujuan lain.
Salah satu tujuan konservasi sumber daya genetika adalah mencegah kepunahan suatu jenis. Untuk menyusun strategi konservasi sumber daya genetika diperlukan informasi tentang sejarah kehidupan (evolusi) tanaman dan pengetahuan tentang genetika populasi yang mengukur tingkat variabilitas genetika pada tingkat jenis dan populasi. Oleh karena itu, perlu diketahui variabilitas DNA kloroplas, mitokondria dan nuklear tanaman tersebut. Analisis polimorfisme genom yang diturunkan secara uniparental mungkin sangat berguna untuk mengumpulkan informasi tentang sejarah evolusi jenis dan populasi.
Dalam studi ini, DNA kloroplas dianalisis dengan menggunakan penanda mikrosatelit. DNA kloroplas dipilih sebagai penanda karena diturunkan secara
3
dibandingkan dengan penanda lainnya seperti isoenzim, RAPD dan RFLP (Powell
et al. 1995a; 1995b).
Tujuan Penelitian
Penelitian ini bertujuan untuk :
1. Mengetahui distribusi haplotipe DNA kloroplas S. acuminata, S. leprosula dan
S. parvifolia
2. Menduga keragaman genetika DNA kloroplas di dalam dan antar populasi pada S. acuminata, S. leprosula dan S. parvifolia.
Hipotesis
Hipotesis yang diuj i dalam penelitian ini adalah:
1. Masing- masing jenis Shorea memiliki haplotipe spesifik sebagai pembeda antar jenis.
2. Variasi DNA kloroplas pada S. acuminata, S. leprosula dan S. parvifolia
adalah rendah.
3. Adanya perbedaan DNA kloroplas yang spesifik antar populasi pada S. acuminata, S. leprosula dan S. parvifolia.
Manfaat Penelitian
Inventarisasi keragaman genetika pohon Shorea diperlukan sebagai informasi dasar untuk kegiatan konservasi sumber daya genetika, dan kegiatan perbaikan genetika pohon Shorea di masa datang. Di samping itu, data penelitian ini mungkin dapat digunakan sebagai penanda diagnostik DNA molekuler jenis
TINJAUAN PUSTAKA
Shorea acuminata Dyer, S. leprosula Miq, dan S. parvifolia Dyer
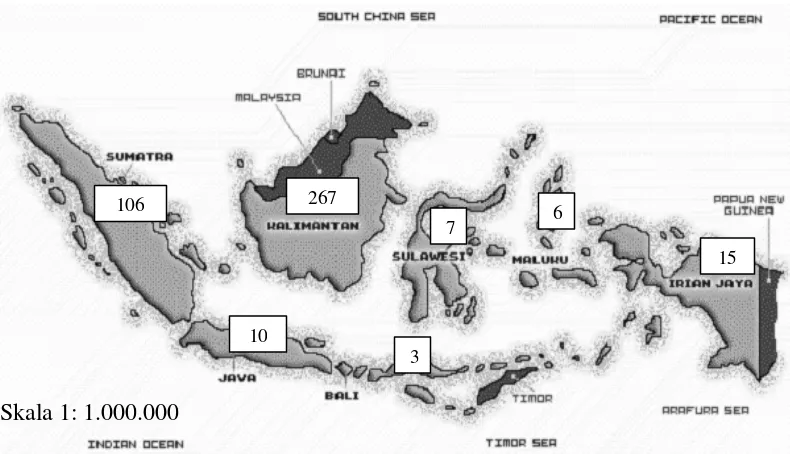
Famili Dipterocarpaceae merupakan salah satu famili yang bernilai ekonomis tinggi dan penting secara ekologis di Asia Tenggara. Distribusi alami dipterokarp di Indonesia me nurut Ashton (1982) seperti terlihat pada Gambar 1. Di Kalimantan (termasuk Malaysia dan Brunai Darussalam) ditemukan sembilan genus (Anisoptera, Cotylelobium, Dipterocarpus, Dryobalanops, Hopea,
Parashorea, Shorea, Vatica, dan Upuna) yang terdiri atas 267 jenis, Sumatra dengan delapan genus (Anisoptera, Cotylelobium, Dipterocarpus, Dryobalanops,
Hopea, Parashorea, Shorea, dan Vatica) terdiri atas 106 jenis, Jawa dan Nusa Tenggara dengan lima genus (Shorea, Hopea, Dipterocarpus, Anisoptera, dan
Vatica) terdiri atas 10 jenis, Sulawesi dengan empat genus (Shorea, Hopea,
Anisoptera, dan Vatica) terdiri atas 7 jenis, Maluku dengan empat genus (Shorea,
Hopea, Anisoptera, dan Vatica) terdiri atas 6 jenis dan Irian (termasuk Papua New Guinea) dengan tiga genus (Hopea, Anisoptera, and Vatica) terdiri atas 15 jenis.
Gambar 1 Distribusi Dipterocarpaceae di Indonesia. Jumlah total jenis dalam setiap pulau menurut Ashton (1982).
267 106
10
6 7
15
3
5
Berdasarkan sifat kayunya, Alrasjid et al. (1991) dan Newman et al. (1999) mengelompokkan meranti ke dalam empat kelompok, yaitu meranti merah, meranti putih, meranti kuning dan meranti balau. Kayu meranti termasuk ke dalam jenis kayu daun lebar yang ringan, tingkat keawetan yang rendah, dan sifat pengerjaannya relatif yang mudah. Kayu meranti banyak dipergunakan sebagai kayu pertukangan, kayu lapis, papan pertikel, peti pengepak, meubel, alat musik, bahan bangunan rumah dan perkapalan (Newman et al. 1999; Alrasjid et al. 1991)
Shorea adalah pohon dengan ketinggian mencapai 60 m dan diameter batang bisa mencapai 180 cm. Menurut Alrasjid et al. (1991), pohon ini umumnya menduduki lapisan tajuk teratas (stratum A) tetapi ada pula yang menduduki lapisan tajuk stratum B. Sebagian besar jenis Shorea tergolong jenis toleran dan semi toleran, sangat sedikit diantara mereka yang tergolong jenis intoleran. Pohon
Shorea tumbuh di tanah latosol, podsolik merah kuning dan podsolik kuning dengan tipe iklim A dan B, curah hujan diatas 2000 mm per tahun, basah dan kelembaban tinggi serta pada ketinggian mencapai 1750 m dari permukaan laut (Ashton 1982).
Alrasjid et al. (1991) menyatakan bahwa species Shorea memiliki malai bunga yang tumbuh pada ujung ranting atau ketiak daun, tiap daun memiliki dua helai daun penumpu, lima belas atau lebih benang sari dengan bakal buah bersel tiga masing- masing berisi dua bakal biji. Buah Shorea berbentuk bulat telur sampai hampir bulat, berukuran beberapa milimeter dengan panjang mencapai 6 cm dan berbiji satu. Shorea mulai berbunga pada umur enam tahun atau lebih, musim berbuah sekitar bulan Oktober sampai April tergantung pada keadaan cuaca yang memerlukan musim panas tertentu sehingga kebanyakan dari jenis ini tidak berbuah setiap tahun.
S. acuminata, S. leprosula dan S. parvifolia adalah jenis diploid (2n = 14) (Ashton 1982; Kajita et al. 1998), dan sistem reproduksi mereka adalah
outcrossing dengan polinator utama adalah thrips dan kumbang yang dapat bermigrasi pada jarak yang tidak terlalu jauh (Bawa 1998). Lee et al. (2000a) telah me laporkan bahwa rata-rata nilai outcrossing S. leprosula pada hutan alam adalah 0.84. Beberapa faktor yang mempengaruhi outcrossing adalah waktu pembungaan, kerapatan atau jumlah pohon yang berbunga serta jumlah dan jenis polinator (Lee et al. 2000a; Bawa 1998).
Ciri diagnostik secara morfologis untuk membedakan ketiga species tersebut (pada tanaman dewasa) adalah S. acuminata mempunyai daun dengan 7 sampai 10 pasang pertulangan sekunder (nerves), S. leprosula mempunyai 12 sampai 15 pasang pertulangan sekunder dan dibagian bawah daun ada domatia yang berwana krem, dan S. pavifolia mempunyai 9 sampai 13 pasang pertulangan sekunder, dan dibagian bawah daun ada petiole dan twigs yang berwarna coklat muda (Ashton 1982; Newman et al. 1999). Dalam studi filogenetika yang telah dilakukan oleh Indrioko (2005) menggunakan penanda PCR-RFLP berdasarkan cpDNA dengan 23 kombinasi primer dan enzim restriksi, ketiga jenis Shorea ini dapat dibedakan berdasarkan pola restriksi yang dihasilkan sebagai berikut:
rbcL/Alu I (1 1 0 0 1 0 0 1) untuk S. parvifolia dan trnLF/Hinf I untuk S. leprosula (4 0 1 1 0) dan S. acuminata (2 0 1 1 0).
Karakteristik DNA kloroplas dan penggunaannya.
Informasi genetika pada tanaman tinggi tersebar pada tiga genom yaitu nuklear, kloroplas dan mitokondria. Gen dalam genom kloroplas dan mitokondria diketahui sebagai gen sitoplasmik dan gen organel. Ketiga genom DNA pewarisannya berbeda, DNA nuklear selalu diturunkan secara biparental, DNA kloroplas dalam banyak Angiospermae diturunkan secara maternal (tidak selalu) dan secara paternal dalam Gymnospermae (Hamrick dan Nason 2000) tetapi ada bukti yang menyatakan bahwa DNA kloroplas diturunkan secara biparental dalam satu sampai tiga genus (Birky 1995) dan ada juga yang diturunkan secara paternal
7
secara maternal tetapi dalam beberapa kelompok Gymnospermae seperti (Araucariaceae, Cupreassaceae, Taxodiaceae) diturunkan secara paternal (Birky 1995), dan diturunkan secara biparental juga telah diamati pada jenis Pinaceae dan Taxaceae.
DNA kloroplas berbentuk sirkular, berukuran kecil sekitar 120 sampai 160 kilo pasang basa. Genom kloroplas dibagi ke dalam dua daerah, pertama daerah
large single copy (LSC), dan kedua adalah daerah small single copy (SSC) yang dipisahkan oleh inverted repeat (IR) (Grivet et al. 2001). Panjang daerah LSC diperkirakan sekitar 89200 bp pada Quercus robur dan 86686 bp dalam tembakau (Grivet et al. 2001). DNA kloroplas menga ndung rRNA, tRNA dan sekitar 50 sampai 100 gen yang mengkodekan enzim untuk fotosintesis.
Karakteristik DNA kloroplas adalah sebagai berikut: i) genom berukuran kecil dan secara struktur stabil, ii) Genom lebih konservatif dengan rata-rata subsitusi nukleotida yang rendah, iii) Genom tidak mengalami rekombinasi dan diturunkan secara uniparental (Clegg et al. 1994; Provan et al. 2001). DNA kloroplas telah digunakan untuk studi filogenetika pada beberapa tanaman, seperti studi filogenetika pada Dipterocarpaceae (Indrioko 2005; Kamiya et al. 2005; Dayandan et al. 1999; Kajita et al. 1999), studi ekologi dan sejarah evolusi tanaman (Heuertz et al. 2004; Vendramin et al. 1998) dan studi populasi genetika (Deguilloux et al. 2004a; Petit et al. 2002; Lian et al. 2003). Baru-baru ini DNA kloroplas telah digunakan sebagai alat untuk identifikasi asal geografis kayu, seperti yang telah dilakukan pada kayu Oak (Quercus patria (Matt.) Liebl) dan
Quercus robur (Deguilloux et al. 2002; 2003; 2004b) dan red ceder (Thuja plicata) (White et al. 2000).
Penanda mikrosatelit
sebagai simple sequence repeats (SSRs), short tandem repeat (STR), variable number tandem repeat (VNTR) dan simple sequence length polymorphism
(SSLP). Banyaknya istilah ini, cenderung membingungkan terutama ketika melakukan stud i literatur, tetapi istilah mikrosatelit sekarang telah menjadi umum untuk menggambarkan motif DNA pendek yang berulang (Hancock 1999).
Mikrosatelit mempunyai karakteristik sebagai berikut: tingkat polimorfisme yang tinggi, kodominan, dan diwariskan mengikuti hukum mendel (Powell et al. 1996; Hancock 1999). Bila satu primer yang spesifik telah didisain, lokus SSR dapat diamplifikasi dari sedikit sampel DNA dengan PCR (Ujino et al.
1998). Mikrosatelit telah diaplikasikan untuk: i) Identifikasi forensik (Balding 1999), bertujuan untuk mengkaitkan sampel darah, sperma, jaringan rambut atau daging dari kasus kriminal, ii) Diagnosis dan identifikasi penyakit, seperti deteksi kanker (Moxon et al. 1999), iii) Studi populasi genetika, untuk mengamati variasi dan membuat kesimpulan tentang struktur populasi, ha nyutan genetik (genetic drift), dan genetic bottlenecks, iv) Konservasi biologi, untuk mengamati perubahan dalam populasi, pengaruh fragmentasi dan interaksi populasi yang berbeda serta untuk identifikasi populasi yang baru terbentuk.
Rata-rata mutasi mikrosatelit lebih tinggi (10-6 sampai 10-2 kejadian per lokus per generasi) dibandingkan dengan rata-rata mutasi pada gen yang mengkodekan loci (Li et al. 2002). Mutasi menghasilkan perubahan dalam jumlah unit ulangan dan itu diamati sebagai variasi panjang mikrosatelit. Ada dua jenis mekanisme yang terlibat untuk menerangkan tingginya rata-rata mutasi mikrosatelit. Pertama, rekombinasi diantara kromosom DNA homolog melalui
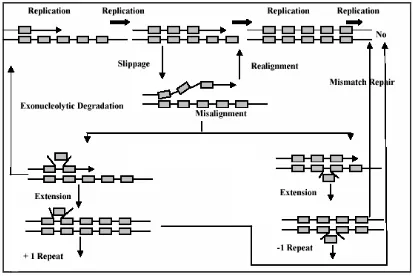
unequal crossing over (UCO) atau dengan konversi gen yang menghasilkan ketidaksempurnaan susunan dan menyebabkan adanya peningkatan ulangan dalam mikrosatelit. Kedua, slippage strand mispairing (SSM) yang terjadi selama replikasi DNA seperti yang ditunjukkan pada Gambar 2. Peristiwa ini dimulai dengan slipnya DNA polimerase selama replikasi yang menyebabkan template
9
dapat keluar dari loops DNA ganda. (Schloetterer dan Tautz, 1992; Schloetterer 1998; Eisen 1999).
Dari dua jenis mekanisme mutasi yang disebutkan diatas, banyak peneliti menyatakan bahwa SSM selama replikasi DNA adalah penyebab utama ketidakstabilan mikrosatelit (Eisen 1999). Rata-rata mutasi mikrosatelit dipengaruhi oleh sifat mikrosatelit, seperti: jumlah ulangan, motif ulangan sekuen, panjang unit ulangan, sekuen flanking, dan interuption dalam mikrosatelit, rata-rata transkripsi dan rata-rata-rata-rata rekombinasi (Schloetterer 2000), GC content, suhu, metilasi dan siklus sel (Eisen 1999), posisi kromosom, seks dan genotipe (Li et al.
2002).
Gambar 2 Diagram model slippage strand mispairing (SSM) pada mutasi mikrosatelit (Eisen 1999)
Slippage strand mispairing (SSM) selama replikasi DNA dapat dikoreksi oleh exonucleolytic proofreading dan mismatch repair. Exonucleolytic proofreading adalah proses pengujian untaian DNA yang salah, ya ng dibuat oleh DNA polimerase selama sintesis DNA. Jika kesalahan ditemukan, exonuclease
untaian DNA yang baru, dengan back-up DNA polimerase. Kesalahan yang dibuat oleh DNA polimerase tidak akan menjadi mutasi semuanya, sebab kesalahan itu akan diperbaiki (dihapus) oleh proofreading. Exonucleolytic proofreading mendeteksi kesalahan dengan memonitor DNA yang telah direplikasi, apakah membentuk struktur DNA double helik yang normal dengan untaian templatenya. Struktur DNA yang tidak normal akan merangsang (trigger) aktivitas exonuclease. Proofreading dipengaruhi oleh GC content dan sekuen DNA.
Mismatch repair berperan dalam mengenali dan memperbaiki kembali basa yang muncul karena salah dalam penggabungan. Mismatch repairs
memainkan peranan kunci dalam meregulasi kestabilan mikrosatelit, perbedaan dalam perbaikan loops oleh mismatch repairs menyebabkan banyaknya variasi mikrosatelit di dalam dan diantara species (Eisen 1999).
Ada beberapa permasalahan dalam menggunakan penanda mikrosatelit. Permasalahan ini dapat dikelompokkan ke dalam problem praktik dan problem data. Problem praktik meliputi: i) Pemilihan primer untuk mikrosatelit, banyak jenis primer yang telah didisain untuk analisis mikrosatelit pada tanaman. Primer-primer itu perlu diskrining dan dioptimasi sebelum diaplikasikan pada jenis tanaman tertentu tertentu, karena setiap tanaman mempunyai karakteristik spesifik yang berbeda satu sama lain. ii) Slippage selama proses amplifikasi, termopolimerase dapat slip sehingga menghasilkan produk yang berbeda dalam ukurannya. iii) Ukuran produk amplifikasi berbeda dari ukuran produk sebenarnya. Ketidakakuratan dalam identifikasi ale l mungkin juga disebabkan oleh Taq polimerase yang menambah nukleotida adenosin sampai ujung 3’ produk amplifikasi. Ginot et al. (1996) menyatakan untuk mengatasi permasalahan ini adalah dengan menambah polimerase pfu selama atau setelah proses PCR, atau dengan menggunakan polimerase DNA T4 setelah PCR.
11
2002). Homoplasi dalam mikrosatelit kloroplas dianggap sebagai sebuah pembatas utama, ketika digunakan sebagai penanda genetika (Provan et al. 2001). Para peneliti secara umum telah menganggap bahwa tingkat homoplasi cukup rendah pada mikrosatelit menggunakan DNA kloroplas (Cuenca et al. 2003).
Penelitian Tentang Variasi Genetika Jenis Shorea dan Manfaatnya
Keragaman genetika adalah perlengkapan yang penting bagi pohon hutan untuk dapat bertahandalam waktu lama. Tanpa keragaman genetika, pohon hutan sulit beradaptasi terhadap perubahan lingkungan sehingga mereka dapat punah, oleh karena itu keragaman ini sangat penting untuk dipelihara.
Finkeldey (1998) menyatakan bahwa evolusi adalah perubahan struktur genetika populasi, paling sedikit terjadi pada satu gen lokus. Proses evolusi akan mengakibatkan terjadinya keragaman genetika pada organisme tidak terkecuali pada pohon hutan. Proses evolusi tersebut meliputi: mutasi (mutation), migrasi (migration), hanyutan genetika (genetic drift), seleksi (selection) dan sistem perkawinan (mating system) (Finkeldey 1998; Ayala 1976).
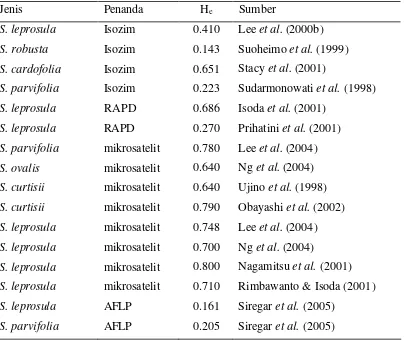
Tabel 1 N ilai keragaman genetika beberapa jenis Shorea menggunakan penanda isozim, RAPD, mikrosatelit dan AFLP.
Jenis Penanda He Sumber
S. leprosula S. robusta S. cardofolia S. parvifolia S. leprosula S. leprosula S. parvifolia S. ovalis S. curtisii S. curtisii S. leprosula S. leprosula S. leprosula S. leprosula S. leprosula S. parvifolia Isozim Isozim Isozim Isozim RAPD RAPD mikrosatelit mikrosatelit mikrosatelit mikrosatelit mikrosatelit mikrosatelit mikrosatelit mikrosatelit AFLP AFLP 0.410 0.143 0.651 0.223 0.686 0.270 0.780 0.640 0.640 0.790 0.748 0.700 0.800 0.710 0.161 0.205
Lee et al. (2000b) Suoheimo et al. (1999) Stacy et al. (2001)
Sudarmonowati et al. (1998) Isoda et al. (2001)
Prihatini et al. (2001) Lee et al. (2004) Ng et al. (2004) Ujino et al. (1998) Obayashi et al. (2002) Lee et al. (2004) Ng et al. (2004)
Nagamitsu et al. (2001) Rimbawanto & Isoda (2001) Siregar et al. (2005)
BAHAN DAN METODE
Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan di Laboratorium Silvikultur, Departemen Silvikultur, Fakultas Kehutanan IPB dan Institut Genetika Hutan dan Pemuliaan Pohon Hutan Universitas Goettingen - Jerman dari Juni sampai Oktober 2005.
Alat dan Bahan Penelitian
Alat yang digunakan dalam penelitian ini adalah mesin PCR PTC-200 Peltier Thermal Cycler MJ Research, peralatan elektroporesis, lampu UV trans iluminator, pH meter, sent rifius, pipet mikro, dan tabung mikro, sedangkan bahan yang digunakan adalah daun Shorea spp, nitrogen cair, Dneasy Plant 96 Kit
untuk isolasi DNA dari Qiagen, HotStarTaq Master Mix Kit Qiagen, primer ccmp1 sampai ccmp10 (Weising dan Gardener 1999), peralatan elektroporesis, sentripugasi, DNA MWM XIV (100-1500 bp), air destilasi, agarose, larutan TAE 1X, dan etidium bromida.
Metode Penelitian
Koleksi Sampel
Diagram alur penelitian dapat dilihat pada Gambar 3. Tiga jenis Shorea
Gambar 4 Lokasi penga mbilan sampel di lapangan.
Nanjak Makmur TNBT
Haurbentes
Berau Sumalindo
S ari Bumi Kusuma. Tering
Asialog Pasir Mayang
Sumatra Borneo
Java
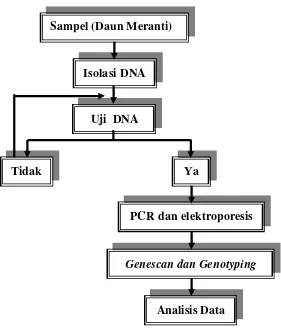
Sampel (Daun Meranti)
Isolasi DNA
Uji DNA
Ya Tidak
PCR dan elektroporesis
Genescan dan Genotyping
Analisis Data
Gambar 3 Bagan Alur Penelitian
Batu Ampar
15
Tabel 2 Koleksi sampel berdasarkan populasi dan jumlah individu per populasi.
Jenis Nama populasi* Kode populasi N Asal
Shorea acuminata Berau ACU-BR 7 Kalimantan
Pasir Mayang ACU-PM 6 Sumatra
TNBT ACU-TNBT 7 Sumatra
Nanjak Makmur ACU-NM 7 Sumatra
Shorea leprosula Haurbentes LEP-HB 6 Kalimantan
Tering LEP-TR 6 Kalimantan
Sumalindo LEP-SM 5 Kalimantan
Sari Bumi Kusuma LEP-SBK 5 Kalimantan
Pasir Mayang LEP-PM 5 Sumatra
TNBT LEP-TNBT 6 Sumatra
Asialog LEP-AS 2 Sumatra
Nanjak Makmur LEP-NM 5 Sumatra
Shorea parvifolia Batu ampar PAR-BA 5 Kalimantan
Sumalindo PAR-SM 6 Kalimantan
Sari Bumi Kusuma PAR-SBK 7 Kalimantan
Pasir Mayang PAR-PM 5 Sumatra
TNBT PAR-TNBT 6 Sumatra
Asialog PAR-AS 10 Sumatra
Nanjak Makmur PAR-NM 7 Sumatra
Total 19 113
Keterangan :
N : Jumlah individu
* : perkiraan geografis (lintang dan bujur) setiap populasi:
1. Hutan alam TNBT 01o05' - 02o06'S dan 103o15' - 103o33'E 2. Hutan alam Asialog 02o02' - 02o22'S dan 103o15' - 103o33'E 3. Hutan alam Pasir Mayang 00o08' - 03o09'S dan 101o19' - 103o20'E 4. Hutan alam Nanjak Makmur 10o22S dan 101o40'E
5. Arboretum Haurbentes 06o54' - 07o54'S dan 106o41' - 107o42'E 6. Hutan alam Sari Bumi Kusuma 01o59' - 00o36'S dan 111o19' - 114o42'E 7. Hutan alam Sumalindo 00o55' - 00o56'N dan 115o19 - 116o36'E 8. Hutan alam Berau 02o05' - 02o36'S dan 116o49' - 117o24'E 9. Hutan alam Batu ampar 00o45' - 00o50'N dan 116o48' - 117o00'E 10. Hutan alam Tering 00o00' - 00o10'N dan 115o22' - 116o38'E
Ekstraksi DNA dan PCR-cpSSR (Kloroplas Mikrosatelit)
Daun kering berukuran 2 x 1 cm diambil untuk diekstraksi DNA totalnya dengan menggunakan Dneasy 96 Plant DNA isolation Kit (Cat. No. 69181; Qiagen, Hilden). Metode ekstraksi dilakukan sesuai dengan instruksi perusahaan. Kualitas DNA hasil isolasi dielektroporesis dengan konsentrasi agarose 0.8% (w/v). Elektroporesis dilakukan menggunakan 1X larutan bufferTris-acetate
DNA diuji dengan membandingkan dengan DNA standar (Lambda DNA Marker, Cat. No 745782; Roche Mannheim) dan DNA standar Molecular Weight Standard
XIV (100 bps ladder) (Cat. No. 1721933; Roche Mannheim).
Sepuluh primer universal yang dinamakan consensus chloroplast microsatellite primer (ccmp) ccmp1 - ccmp10 (Weising & Gardener 1999) diuji untuk analisis DNA kloroplas. Sekuen DNA masing- masing primer dapat dilihat pada Table 3. Sebelum dilakukan amplifikasi, primer forward terlebih dahulu dilabel dengan pewarna fluorescence (Metabion) yaitu: 6-FAM/Biru (untuk
ccmp2, ccmp4, ccmp6 dan ccmp9), HEX/Hijau (untuk ccmp1, ccmp3, ccmp7 dan
ccmp10) dan NED/Kuning (ccmp5 dan ccmp8)
Prosedur PCR mengikuti Indrioko (2005), yaitu denaturasi awal selama 15 menit pada suhu 95oC, diikuti 39 siklus : denaturasi selama 1 menit pada suhu 94oC, annealing selama 1 menit pada suhu 50oC, extension selama 1 menit pada suhu 72oC dan final extension selama 10 menit pada suhu 72oC. Volume reaksi PCR adalah 15 µl, terdiri atas: 2.0 µl DNA template (5-10 ng), 1.8 µl primer forward (5 pmol/µl) dan primer reverse (5 pmol/µl), 1.9 µl air bebas RNAse, dan 7.5 µl HotStarTaq Master Mix Kit (Qiagen, Hilden). Hasil PCR dielektroporesis pada gel agarose dengan konsentrasi 2.0% (w/v), setelah itu gel agarose direndam dalam 1.0% (v/v) larutan etidium bromida selama 20 menit pada temperatur ruangan, pola pita diamati dibawah lampu UV transiluminator dalam ruang gelap dan didokumentasikan dengan menggunakan kamera digital.
Genescan dan Genotyping Hasil PCR
Hasil amplifikasi dipisahkan dengan elektroporesis kapiler menggunakan Sekuenser ABI PRISM 3100 (Applied Biosystems) dengan polymer 3100 POP-4 TM
(Applied Biosystem) dan standar GS500 ROX TM (Applied Biosystem). Alel-alel dianalisis menggunakan Genescan versi 3.7 (Applied Biosystem) dan
genotyping mengunakan Genotyper versi 3.7 NT (Applied Biosystem).
Bahan-bahan untuk Genescan (96 probes) terdiri atas: 1152 µl HiDi Formamide (Applied Biosystem) dan 1.5 µl standar GS 500 ROX TM (Applied Biosystems). Campuran tersebut didistribusikan secara merata ke dalam tube (12
17
[image:30.596.114.529.172.533.2]setiap tube. Sampel kemudian didenaturasi selama 2 menit pada 90oC dan disimpan dalam es sekitar 5 menit sampai analisis Genescan dilakukan.
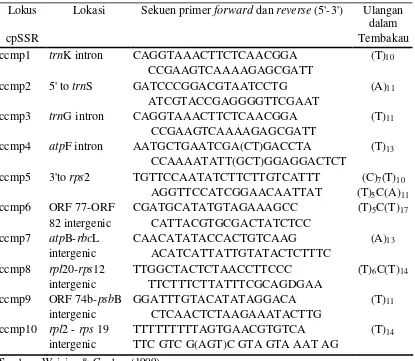
Tabel 3 Sekuen DNA pasangan primer yang diuji untuk amplifikasi cpSSR
Lokus Lokasi Sekuen primer forward dan reverse (5'-3') Ulangan dalam
cpSSR Tembakau
ccmp1 trnK intron CAGGTAAACTTCTCAACGGA (T)10 CCGAAGTCAAAAGAGCGATT
ccmp2 5' to trnS GATCCCGGACGTAATCCTG (A)11 ATCGTACCGAGGGGTTCGAAT
ccmp3 trnG intron CAGGTAAACTTCTCAACGGA (T)11 CCGAAGTCAAAAGAGCGATT
ccmp4 atpF intron AATGCTGAATCGA(CT)GACCTA (T)13 CCAAAATATT(GCT)GGAGGACTCT
ccmp5 3'to rps2 TGTTCCAATATCTTCTTGTCATTT (C)7(T)10 AGGTTCCATCGGAACAATTAT (T)5C(A)11 ccmp6 ORF 77-ORF CGATGCATATGTAGAAAGCC (T)5C(T)17
82 intergenic CATTACGTGCGACTATCTCC
ccmp7 atpB-rbcL CAACATATACCACTGTCAAG (A)13 intergenic ACATCATTATTGTATACTCTTTC
ccmp8 rpl20-rps12 TTGGCTACTCTAACCTTCCC (T)6C(T)14 intergenic TTCTTTCTTATTTCGCAGDGAA
ccmp9 ORF 74b-psbB GGATTTGTACATATAGGACA (T)11 intergenic CTCAACTCTAAGAAATACTTG
ccmp10 rpl2 - rps 19 TTTTTTTTTAGTGAACGTGTCA (T)14 intergenic TTC GTC G(AGT)C GTA GTA AAT AG
Sumber : Weising & Gardner (1999)
Analisis Data
Haplotipe cpSSR disimpulkan sebagai kombinasi dari ukuran individu alel yang ditemukan pada setiap lokus cpSSR yang dianalisis. Ukuran fragmen individu alel dalam pasang basa ditunjukkan oleh puncak gelombang tertinggi dari hasil genotyping. Pada sampel yang ditemukan fragmen cpSSR dikodekan dengan 1 dan sampel yang tidak ditemukan fragmen cpSSR dikodekan dengan 0 untuk keperluan analisis dengan program.
HASIL DAN PEMBAHASAN
Haplotipe Mikrosatelit Kloroplas
Penelitian pendahuluan dilakukan menggunakan dua sampel per populasi dan sepuluh primer mikrosatelit kloroplas (cpSSR), yaitu ccmp1 sampai ccmp10
(Weising & Gardener 1999). Dari sepuluh primer yang diuji, lima primer (ccmp4, ccmp5, ccmp7, ccmp8 dan ccmp9) tidak menghasilkan amplifikasi. Lima primer (ccmp1, ccmp2, ccmp3, ccmp6 dan ccmp10) berhasil mengamplifikasi semua sampel, seperti terlihat pada Gambar 5.
[a] [b] [c]
[image:32.596.156.461.285.640.2]
[d] [e]
Gambar 5 Lima primer yang berhasil diamplifikasi dengan PCR.
Dari lima primer yang berhasil diamplifikasi dengan PCR, tiga primer (ccmp1, ccmp2 dan ccmp10) menunjukkan pola monomorfik dengan ukuran fragmen berturut-turut adalah 113, 150 dan 101 bp. Pada primer ccmp3 dan ccmp6
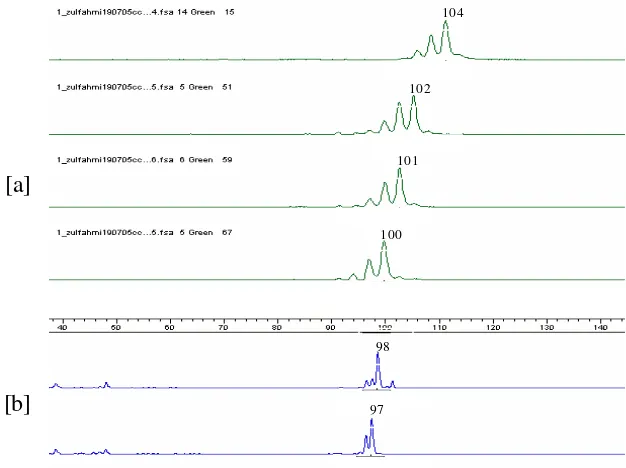
menunjukkan pola polimorfik, seperti terlihat pada Gambar 6. Hasil amplifikasi
[image:33.596.125.438.242.476.2]ccmp3 memberikan 4 tipe ukuran panjang fragmen (100, 101, 102 dan 104 bp) dan hasil ccmp6 memberikan 2 tipe ukuran panjang fragmen (97 dan 98 bp), dengan demikian didapatkan jumlah total 6 haplotipe seperti terlihat pada Table 4.
Gambar 6 Hasil GeneScan primer ccmp3 dan ccmp6 yang menunjukkan pola polimorfisme.
Keterangan :
a) Polimorfisme yang terdeteksi pada primer ccmp3, yaitu 104 bp, 102 bp, 101 bp dan 100 bp.
b) Polimorfisme yang terdeteksi pada primer ccmp6, yaitu 98 bp dan 97 bp.
Tabel 4 Definisi haplotipe dan ukuran fragmen cpSSR Ukuran amplifikasi fragmen cpSSR (bp) Haplotipe
ccmp3 ccmp6
[image:33.596.109.414.639.752.2]21 Variasi Haplotipe
[image:34.596.106.516.305.398.2]Berdasarkan hasil genescan DNA kloroplas diperoleh haplotipe umum masing- masing jenis Shorea yaitu haplotipe D untuk S. acuminata, haplotipe A untuk S. leprosula dan haplotipe C untuk S. parvifolia. Detail hasil genescan untuk S. acuminata, S. leprosula dan S. parvifolia dapat dilihat pada Lampiran 1. Ketiga jenis Shorea tersebut hanya dibedakan oleh 1-2 pasang basa seperti terlihat pada Tabel 5. Berdasarkan hasil studi ini, ada dua tipe mutasi yang menyebabkan perbedaan diantara mereka yaitu delesi dan insersi.
Tabel 5 Perbedaan antara S. acuminata, S. leprosula dan S. parvifolia berdasarkan tipe mutasi
Jenis Perbedaan Tipe mutasi
S. acuminata vs S. leprosula 1 bp pada ccmp3 (102-101) Delesi
S. acuminata vs S. parvifolia 2 bp pada ccmp3 (102-100) 1 bp pada ccmp6 (97-98)
Delesi Insersi
S. leprosula vs S. parvifolia 1 bp pada ccmp3 (101-100) 1 bp pada ccmp6 (97-98)
Delesi Insersi
Terjadinya delesi dan insersi basa pada ketiga jenis Shorea tersebut kemungkinan karena adanya seleksi alam melalui fluktuasi lingkungan yang ekstrim pada waktu lampau dan/atau waktu sekarang. Waktu terjadinya mutasi tersebut secara tepat sulit ditentukan, tetapi berdasarkan sifat DNA kloroplas yang konservatif dan kecepatan mutasi yang rendah yaitu 3.2 x 10-5 dan 7.9 x 10-5 (Provan et al. 1999) boleh jadi peristiwa mutasi terjadi pada masa lampau. Gen yang mengalami mutasi satu basa dapat mengakibatkan perubahan asam amino yang dihasilkan sehingga berdampak pada perubahan fenotipe tanaman di lapangan.
Detail frekuensi haplotipe DNA kloroplas masing- masing jenis Shorea
berdasarkan DNA kloroplas, pada S. leprosula ditemukan hanya satu haplotipe, sedangkan pada S. parvifolia ditemukan tiga haplotipe. Variasi haplotipe DNA kloroplas yang rendah juga diamati pada jenis Oak (Petit et al. 2002); Tolmiea menziensis (Soltis et al. 1989); dan Lupinus texensis (Banks & Birky 1985). Petit
et al. (2002) meneliti 12214 individu dari 2614 populasi hanya menemukan 32 haplotipe, sedangkan variasi DNA kloroplas yang cukup tinggi diamati pada common ash (Fraxinus excelsior L.) (Heuertz et al. 2001) dan Pinus pinaster
[image:35.596.109.502.304.607.2](Vendramin et al. 1998).
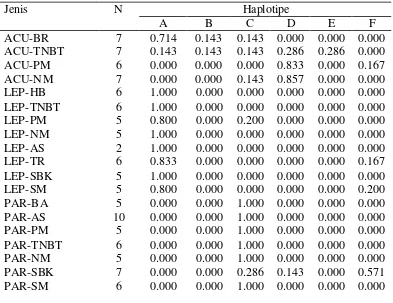
Tabel 6 Frekuensi haplotipe S. acuiminata, S. leprosula dan S. parvifolia pada setiap populasi
Haplotipe
Jenis N
A B C D E F
ACU-BR ACU-TNBT ACU-PM ACU-NM 7 7 6 7 0.714 0.143 0.000 0.000 0.143 0.143 0.000 0.000 0.143 0.143 0.000 0.143 0.000 0.286 0.833 0.857 0.000 0.286 0.000 0.000 0.000 0.000 0.167 0.000 LEP-HB LEP-TNBT LEP-PM LEP-NM LEP-AS LEP-TR LEP-SBK LEP-SM 6 6 5 5 2 6 5 5 1.000 1.000 0.800 1.000 1.000 0.833 1.000 0.800 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.200 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.167 0.000 0.200 PAR-BA PAR-AS PAR-PM PAR-TNBT PAR-NM PAR-SBK PAR-SM 5 10 5 6 5 7 6 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 1.000 1.000 1.000 1.000 1.000 0.286 1.000 0.000 0.000 0.000 0.000 0.000 0.143 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.571 0.000 Keterangan : 1
: Jenis Shorea (ACU = S. acuminata, LEP = S. leprosula, PAR = S. parvifolia).
2
: Nama populasi (BR = Berau, PM = Pasir Mayang, TNBT = Taman Nasional Bukit Tiga puluh, NM = Nanjak Makmur, AS = Asialog, HB = Haurbrntes, BA = Batu Ampar, TR = Tering, SBK, Sari Bumi Kusuma, SM = Sumalindo)
23
Shorea (yang termasuk kelo mpok angiosperma). Transfer DNA kloroplas melalui migrasi gen pada jenis Shorea hanya terjadi melalui biji. Penyebaran biji pada jenis Shorea terbatas, disebabkan oleh biji Shorea yang relatif berat, sehingga jatuh tidak jauh dari pohon induknya, yaitu sekitar 50 m untuk S. leprosula
(Ashton 1982) dan 20 m untuk S. curtisii (Obayashi et al. 2002). Hal ini kemungkinan juga akan dapat mengurangi variasi DNA kloroplas di dalam populasi.
Variasi Genetika di Dalam dan Antar Populasi
Keragaman genetika di dalam dan antar populasi ketiga jenis Shorea
ditunjukkan pada Tabel 7. Nilai diferensiasi genetika antar populasi (Gst) berdasarkan Nei (1972) yang tertinggi terdapat pada S. parvifolia, yaitu 58 % dan yang terendah terdapat pada S. leprosula, yaitu 14 %. Sedangkan nilai total diferensiasi populasi (δT) berdasarkan Gregorious (1987) yang tertinggi terdapat pada S. acuminata, yaitu 32 % dan yang terendah terdapat pada S. leprosula, yaitu 7 %.
Tabel 7 Nilai keragaman genetika di dalam dan antar populasi pada S. acuminata, S. leprosula dan S. parvifolia.
Jenis Ht* Hs* Gst* δT**
S. acuminata 0.314 0.213 0.319 0.323
S. leprosula 0.060 0.052 0.143 0.077
S. parvifolia 0.074 0.031 0.582 0.131 Keterangan :
* : Perhitungan dilakukan dengan program POPGEN versi 32 (Yeh et al. 1999). ** : Perhitungan dilakukan dengan program GSED versi 1.1k (Gillet 2005). Ht : Keragaman genetika total populasi
Hs : Keragaman genetika subpopulasi
Gst : Diferensiasi genetika antar populasi δT : Diferensiasi genetika antar populasi
Nilai Gst S. leprosula hasil studi ini lebih tinggi dibandingkan dengan studi Lee et al. (2000b) berdasarkan DNA nuklear dan penanda isozim, yaitu Gst =
0.085; Prihatini et al. (2001) berdasarkan DNA nuklear dan penanda RAPD, yaitu
penanda AFLP, yaitu Gst=031, dan studi Indrioko (2005) berdasarkan DNA kloroplas menggunakan penanda PCR-RFLP dan mikrosatelit, yaitu Gst=0.15.
Analisis keragaman molekuler (AMOVA) terhadap S. acuminata, S. leprosula dan S. parvifolia dari populasi Sumatra dan Kalimantan juga dilakukan, hasilnya disajikan pada Tabel 8. Persentase keragaman ketiga jenis Shorea antara pulau Sumatra dan Kalimantan yang tertinggi diamati pada S. Acuminata, dan yang terendah pada S. leprosula, berturut-turut nilai persentase keragamannya adalah 35% (p = 0.000) untuk S. acuminata, 13% (p = 0.048) untuk S. parvifolia
[image:37.596.108.500.438.664.2]dan 0.63% (p = 0.278) untuk S. leprosula. Persentase keragaman di dalam populasi berkontribusi secara signifikan, yaitu 55% untuk S. acuminata, 45% untuk S. parvifolia. Tingginya persentase keragaman dalam populasi pada S. acuminata disebabkan oleh bervariasinya haplotipe yang ditemukan pada tiap-tiap populasi, terutama pada populasi TNBT ditemukan lima haplotipe. Tingginya variasi haplotipe pada TNBT disebabkan oleh populasi ini adalah kawasan konservasi, yang tidak boleh dilakukan penebangan.
Tabel 8 Analisis keragaman molekuler (AMOVA)
Sumber variasi df Jumlah kuadrat Komponen variasi % variasi p S. acuminata
Antar pulau 1 2.29 0.167 35.63 0.00**
Antar populasi dalam pulau 2 1.09 0.043 9.19 0.04* Dalam populasi
S. leprosula
Antar pulau
Antar populasi dalam pulau Dalam populasi
S. parvifolia
Antar pulau
Antar populasi dalam pulau
23 1 6 32 1 5 5.97 0.06 0.32 2.43 0.71 1.83 0.259 0.0005 -0.0043 0.0760 0.015 0.049 55.18 0.63 -5.97 105.35 13.26 42.24 0.00** 0.29ns 0.12ns 0.52ns 0.04* 0.00** Dalam populasi 39 2.00 0.051 44.50 0.00** Keterangan: ns : tidak berbeda nyata, * : berbeda nyata pada taraf α 5%,
** : berbeda nyata pada taraf α = 1%
25
haplotipe di Sumatra. Pada S. parvifolia persentase keragaman dalam populasi (45%) dan antar populasi dalam pulau (42%) hampir sama, hal ini disebabkan oleh adanya haplotipe F dan D yang ditemukan pada populasi SBK dan aliran gen antar populasi di sekitar Sari Bumi Kusuma masih berjalan baik.
Pada S. leprosula, di dalam populasi, antara populasi dalam pulau dan antar pulau (Sumatra dan Kalimantan) tidak berbeda nyata secara statistik. Hal ini menunjukkan bahwa semua populasi memiliki keragaman haplotipe yang sama secara statistika meskipun pada populasi Pasir Mayang, Tering dan Sumalindo ditemukan variasi haplotipe.Nilai negatif pada keragaman antara populasi dalam pulau pada S. leprosula mengindikasikan bahwa sampel populasi pada tiap-tiap pulau tidak beragam secara statistik, hal ini tercermin dari nilai p = 0.12 yang tidak berbeda nyata. Koefisien korelasi yang negatif secara biologis menunjukkan bahwa individu sampel populasi pada level dalam pulau lebih dekat (nilai p = 0.28 tidak berbeda nyata) dibandingkan dengan level antara pulau (Schneider et al. 2000). Pada S. leprosula, persentase keragamannya dalam populasi sangat tinggi, yaitu 105 % tetapi tidak berbeda nyata secara statistik. Tingginya nilai keragaman ini disebabkan oleh tidak terpenuhi asumsi–asumsi dalam analisis AMOVA tersebut. Asumsi yang tidak terpenuhi adalah ukuran populasi yang tidak sama dengan 0.5 dan nisbah jumlah generasi terhadap ukuran populasi (T/No) yang lebih besar 0.5 (Schneider et al. 2000)
Distribusi geografis haplotipe dapat dilihat pada Gambar 7. Distribusi geografis haplotipe hasil penelitian ini tidak mencerminkan secara jelas perbedaan antar populasi. Pada S. acuminata ditemukan enam haplotipe (A, B, C, D, E, dan F). Lima haplotipe ditemukan di populasi TNBT, tiga haplotipe ditemukan di populasi Berau, dan dua haplotipe masing- masing ditemukan di populasi Pasir Mayang dan Nanjak Makmur. Pada S. leprosula, haplotipe A ditemukan pada semua populasi, sedangkan haplotipe F ditemukan pada tiga populasi yaitu populasi Tering, Sumalindo dan Sari Bumi Kus uma pada S. parvifolia. Haplotipe C pada S. parvifolia juga ditemukan pada semua populasi. Pada ketiga jenis
Gambar 7 Distribusi geografis dan frekuensi haplotipe kloroplas mikrosatelit pada Shorea spp: 2a) Shorea acuminata, 2b) Shorea leprosula, 2c) Shorea parvifolia.
Keterangan haplotipe: A B C D E F
TNBT PM
SM
SBK
BA
NM AS
TNBT
BR
PM NM
TNBT
SM
SBK
HB
TR
NM
PM AS a) Shorea acuminata
b) Shorea leprosula
27
Penggunaan metode DNA molekuler untuk mendeteksi asal geografis species telah dikembangkan oleh Deguilloux et al. (2002); (2003); (2004b) pada jenis Oak dan oleh White et al. (2000) pada red ceder (Thuja plicata). Sejarah kolonisasi dan migrasi tumbuhan dapat diamati dari distribusi haplotipe, antara lain telah diamati pada Pinus pinaster (Vendramin et al. 1998); Franxinus excelsior L (Heuertz et al. 2004); Oak (Petit et al. 2002); dan Armeria
(Plumbagiaceae) (Larena et al. 2002).
Sampai saat ini, sangat sedikit informasi atau mungkin belum ada studi tentang kolonisasi famili Dipterocarpaceae di Indonesia atau di Asia Tenggara. Dari hasil penelitian ini diduga bahwa kolonisasi Shorea spp di Indonesia berawal dari pulau Sumatra dan menyebar ke pulau Kalimantan. Hal ini tercermin dari semua haplotipe yang ditemukan di pulau Kalimantan juga ditemukan di pulau Sumatra.
Maury-Lenchon dan Curtet (1998) me nyatakan bahwa Dipterocarpaceae di Asia Tenggara berasal dari daratan Afrika kemudian menyebar ke India terus bergerak ke daratan Sunda, Malaysia, Kalimantan dan sampai ke sebagian papua New Guinea pada akhir masa Cretaceous sampai masa Tertiary (100 sampai 5.5 juta tahun yang lalu). Bukti paleogeografi menyatakan bahwa sebelum periode
glacial, pulau Kalimantan bersatu dengan daratan utama Asia Tenggara, Jawa dan Sumatra (Morley 2000 dalam Kamiya et al 2005). Kondisi ini tentunya memungkinkan kolonisasi dan aliran gen dipterokarp dari pulau Sumatra ke Kalimantan dan sebaliknya dapat terjadi. Berkaitan dengan kondisi iklim saat itu yang berfluktuasi secara drastis, mungkin telah mempengaruhi pola dan tingkat variasi interspesifik, diferensiasi jenis dan interspesifik hibridisasi yang diamati dalam jenis Shorea sekarang ini.
Jarak genetika merupakan salah satu parameter yang dapat memberikan indikasi adanya hubungan kekerabatan antar populasi. Dendogram UPGMA berdasarkan jarak genetika (Nei 1972) dan Gregorio us & Roberds (1986) dapat dilihat pada Gambar 8 dan Gambar 9. Pada Gambar 8 terlihat bahwa ketiga jenis
S. acuminata juga terdiri dari dua kelompok, yaitu S. acuminata dari populasi Pasir Mayang dan Nanjak Makmur sebagai kelompok pertama, dan S. acuminata
[image:41.596.117.505.170.398.2]dari populasi Taman Nasional Bukit Tiga Puluh sebagai kelompok kedua.
Gambar 8 Dendogram UPGMA S. acuminata, S. leprosula dan S. parvifolia
berdasarkan jarak genetik a Nei (1972)
Gambar 9 Dendogram UPGMA S. acuminata, S. leprosula dan S. parvifolia
[image:41.596.114.506.467.705.2]29
S. leprosula terbagi menjadi tiga kelompok. Kelompak pertama yaitu S. leprosula dari populasi Sari Bumi Kusuma, Nanjak Makmur, Taman Nasional Bukit Tigapuluh, Asialog dan Haurbentes, S. leprosula dari populasi Tering dan Sumalindo sebagai kelompok kedua adalah, serta S. leprosula dari populasi Pasir Mayang sebagai kelompok ketiga. S. acuminata dari populasi Berau berada dalam satu kelompok dengan S. leprosula dari populasi Pasir Mayang, hal ini disebabkan oleh banyaknya haplotipe S. acuminata dari populasi Berau yang sama dengan S. leprosula dari populasi Pasir Mayang seperti terlihat dari Gambar 7 dan Tabel 6.
Pengelompokan ketiga jenis Shorea berdasarkan Gregorious & Roberds (1986) ditemukan sedikit perbedaan dengan Nei (1972), yaitu pada S. acuminata
dari populasi Berau berada dalam satu kelompok dengan S. acuminata dari populasi Pasir Mayang, sedangkan pada dendogram berdasarkan Nei (1972), S. acuminata dari populasi Berau berada satu kelompok dengan S. leprosula dari Pasir Mayang. Perbedaan lain juga terlihat dari penempatan S. acuminata dari populasi TNBT. Pada Gambar 9, S. acuminata dari populasi TNBT ditempatkan satu kelompok dengan S. parvifolia dari populasi Sari Bumi Kusuma, sedangkan pada Gambar 8, S. acuminata dari populasi TNBT dan S. parvifolia dari populasi Sari Bumi Kusuma tidak berada dalam satu kelompok.
Dari kedua dendogram terlihat adanya populasi dari pulau Sumatra dan Kalimantan yang berada dalam satu kelompok. Hal ini menunjukkan bahwa
Shorea spp telah menyebar merata di pulau Sumatra dan Kalimantan sebelum periode glacial. Pemisahan pulau Sumatra dan Kalimantan yang terjadi setelah periode glacial, tidak membuat banyak perubahan pada DNA kloroplas Shorea
spp, sehingga variasi haplotipe antar pulau tidak berbeda.
Pembagian Haplotipe antar jenis
juga ditemukan pada S. acuminata di populasi Berau, populasi Taman Nasional Bukit Tigapuluh dan populasi Nanjak Makmur serta pada S. leprosula di populasi Pasir Mayang seperti terlihat pada Tabel 6 dan Gambar 7. Haplotipe F adalah dominan ditemukan pada S. parvifolia di populasi Sari Bumi Kusuma. Haplotipe ini juga terdapat pada S. acuminata di populasi Pasir Mayang dan pada S. leprosula di populasi Tering dan populasi Sumalindo. Terdapatnya haplotipe yang sama diantara ketiga jenis Shorea, kemungkinan disebabkan oleh terjadinya interspesifik hibridisasi diantara mereka atau dengan jenis Shorea lain.
Meskipun hibridisasi dan introgresi pada jenis Shorea adalah langka, karena tingginya derajat inkompatibilitas antar jenis, tetapi Ashton (1982); Ishiyama et al. (2003); dan Kamiya et al. (2005) telah melaporkan adanya jenis hibrid dalam genus Shorea. Ashton (1982) menemukan dua spesimen koleksi hibrid antara S. acuminata dan S. parvifolia di Sumatra Tenggara dan hibrid antara S. leprosula dan S. curtisii dari beberapa lokasi di Semenanjung Malaysia dan Singapura. Ishiyama et al. (2003) telah melakukan DNA sekuensing terhadap gen Glyceraldehide-3-phosphate dehydrogenase (GapC) empat jenis Shorea (S. acuminata, S. curtisii, S. leprosula dan S. parvifolia) dan menemukan beberapa individu mengandung tipe haplotipe lain yang tersusun atas sekuen DNA jenis berbeda.
Chan (1981) dalam Indrioko (2005) menemukan adanya pembentukan buah yang sukses dari hasil persilangan antara S. splendida dan S. stenoptera. Hal ini mengindikasikan adanya potensial hibridisasi alami antara jenis Shorea. Ada beberapa faktor yang mendukung kemungkinan terjadinya hibridisasi, yaitu ketiga jenis Shorea yang diinvestigasi tumbuh bersama dalam hutan (membentuk tegakan campuran) dan waktu pembungaan yang tumpang tindih (overlaping). Mereka tidak berbunga setiap tahun, tetapi pada interval waktu yang tidak teratur dan intensitas pembungaan massal yang bervariasi (selang 2 sampai 5 tahun) (Ashton 1982).
31
jenis. Waktu pembungaan S. acuminata tumpang tindih dengan pembungaan S. parvifolia dan S. leprosula seperti terlihat pada Gambar 10. Di tambah lagi dengan mekanisme reproduksi jenis Shorea adalah outcrossing, dengan polinator utama mereka adalah kelompok serangga (thrips) dan kumbang (Ashton 1982; Bawa 1998; Sakai et al. 1999) sehingga pertukaran gen antar jenis memungkinkan terjadi. Transfer polen pada jenis Shorea dilakukan oleh polinator.
[image:44.596.118.497.216.443.2]
Gambar 10 Waktu pembungaan pada beberapa jenis Shorea seksi Mutica famili Dipterocarpaceae di Semenanjung Malaysia (Ashton 1988).
Sakai et al. (1999) melaporkan bahwa dari 74% kumbang yang mengunjungi bunga S. parvifolia, 30% diantara mereka membawa polen, sedangkan dari 16% thrips yang mengunjungi bunga S. parvifolia, 12% diantara mereka membawa polen. Arnold (1992) mengatakan bahwa polen adalah media yang dapat menyebabkan terjadinya introgresi. Introduksi polen asing kepada bunga tanaman lain akan menghasilkan turunan (hibrid) yang membawa DNA inti bukan DNA kloroplas.
dapat diidentifikasi sebagai satu jenis tanaman tetapi tanaman tersebut mempunyai genom kloroplas jenis tanaman lain. Dari Gambar 11 terlihat bahwa setelah
backcrossing beberapa generasi (BC) diperoleh individu yang mengandung genotipe nuklear jenis A dan cytoplasm (genom kloroplas) jenis B. Jumlah kontribusi cytoplasm oleh tetua betina ha mpir konstan melalui beberapa
[image:45.596.150.450.273.571.2]backcrossing, tetapi proporsi DNA nuklear dari tetua A meningkat secara eksponensial dalam generasi berikutnya, sedangkan proporsi DNA nuklear tetua lain menurun.
Gambar 11 Mekanisme penangkapan kloroplas (Chloroplast capture) pada Eucalyptus spp (Jackson et al. 1999)
Fenomena penangkapan kloroplas telah dilaporkan antara lain pada Sunflower genus Halianthus (Rieseberg et al. 1990): Oaks (Whittemore & Schaal 1991); Eucalyptus (Jackson et al. 1999) dan Mitella (Okuyama et al. 2005). Rieseberg et al. (1990) menemukan dari 154 individu Halianthus annuus ssp.
33
Adanya haplotipe yang sama diantara ketiga jenis Shorea dapat disebabkan oleh kesalahan identifikasi di lapangan. Pada koleksi sampel di lapangan, tenaga kolektor memiliki keahlian identifikasi morfologis di lapangan yang dipakai, tetapi tidak tertutup kemungkinan adanya salah identifikasi. Untuk sampel yang meragukan secara morfologis di lapangan, dikirim ke herbarium Bogoriensis untuk diidentifikasi secara lebih teliti.
Implikasi Terhadap Konservasi Sumber Daya Genetika Shorea spp di
Indonesia.
Salah satu tujuan konservasi adalah mencegah punahnya suatu jenis. Konservasi sumber daya genetika jenis dipterokarp dapat dilakukan secara in situ
dan ex situ. Konservasi secara in situ membutuhkan ukuran populasi yang besar, sedang konservasi ex situ, individu yang dikoleksi hendaknya memiliki keragaman genetika yang mewakili konservasi in situ sehingga keragaman genetika dalam populasi tetap tersimpan dan terpelihara.
Berdasarkan nilai diferensiasi genetika S. acuminata (Gst = 0.3185), S. leprosula (Gst 0.143) dan S. parvifolia (Gst 0.5821), dan AMOVA S. acuminata
dan S. parvifolia pada semua tingkat (dalam populasi, antar populasi dalam pulau dan antar pulau) berbeda nyata secara statistik, maka strategi konservasi untuk S. acuminata dan S. parvifolia berbeda dengan S. leprosula. Untuk S. acuminata dan
S. parvifolia, konservasi dilakukan di banyak populasi dalam tiap pulau, sedang S. leprosula dilakukan di pusat-pusat keragaman (diversity hotspots) di masing-masing pulau, yaitu populasi Pasir Mayang di pulau Sumatra, populasi Tering dan Sumalindo di pulau Kalimantan.
SIMPULAN
1. Ketiga jenis Shorea ini memiliki haplotipe diagnostik, yaitu haplotipe D untuk S. acuminata, haplotipe A untuk S. leprosula dan haplotipe C untuk
S. parvifolia yang dibedakan oleh 1-2 pasang basa.
2. Jumlah total haplotipe yang ditemukan pada ketiga jenis Shorea adalah enam haplotipe, yaitu haplotipe A, B, C, D, E, dan F .
3. Variasi haplotipe dalam populasi tertinggi diamati pada S. acuminata, yaitu enam haplotipe, dan masing- masing tiga haplotipe pada S. leprosula
dan S. parvifolia.
4. Nilai diferensiasi genetika tertinggi ditemukan pada S. parvifolia (Gst = 0.58) diikuti oleh S. acuminata (Gst = 0.31) dan S. leprosula (Gst = 0.14). 5. Semua haplotipe menyebar merata pada populasi yang ada di setiap pulau,
35 DAFTAR PUSTAKA
Al Rasyid H, Marfuah, Wijayakusuma H, Hendarsyah D, 1991. Vademikum Dipterocarpaceae. Jakarta: Badan Penelitian dan Pengembangan Kehutanan Departemen Kehutanan.
Anonymous. 04 Des 2005. Kerusakan hutan 2.6 sampai 2.8 juta hektar per tahun.
www.kompas.com. [16 Maret 2006].
Ashton PS. 1982. Dipterocarpaceae, in: Van Steenis, CGGJ. (Editor). Flora Malesiana, Series 1, Spermatophyta, Vol. 9, Part 2. Martinus Nijhoff. The Hague, Boston, London. pp.237-552.
Ashton PS. 1988. Dipterocarp biology as a window to the understanding of tropical forest structure. Annu. Rev. Ecol. Syst. 19: 347-370.
Ayala FJ. 1976. Molecular genetic and evolution, in Ayala FJ. (Editor.). Molecular evolution. Sunderland Massahusetts, Sinauer Associates, Inc. pp.1-7.
Arnold ML. 1992. Natural hybridization as an evolutionary process. Ann. Rev. Ecol. Syst, 23: 237-261
Balding D. 1999. Forensic applications of microsatellite markers, in: Golstein, DB. and Schlötterer, C. (Eds.). Microsatellite: evolution and applications. Oxford University Press. pp. 198-210.
Banks, JA and Birky, CW. 1985. Chloroplast DANN diversity is low in a wild plant, Lupinus texensis. Proc. Natl. Acad. Sci USA. 82: 6950-6954
Bawa KS. 1998. Conservation of genetic resources in the Dipterocarpaceae, in: Appanah, S and Turnbull, J. M. (Eds.). A review of dipterocarps, taxonomy, ecology and silviculture. Center for International Foresty Research. Bogo r. pp.45-55.
Birky CW. 1995. Uniparental inheritance of mitochondrial and chloroplast genes : mechanism and evolution. Proc. Natl. Acad. Sci USA. 92 : 11331 - 11338. Clegg MT, Gaut BS, Learn GH, Morton BR. 1994. Rates and patters of
chloroplast DNA evolution. Proc. Natl. Acad. Sci USA. 91: 6795-6801. Cuenca A, Escalante AE, Pinero D. 2003. Long distance colonization, isolation by
Dayandan S, Ashton PS, Wilhams SM, Primack RB. 1999. Phylogenetic of tropical tree family Dipterocarpaceae based on nucleotide sequences of the chloroplast rbcL gene. Am. J. Bot. 86 (8) : 1182 - 1190.
Deguilloux MF, Pemonge MH, Petit RJ. 2002. Novel perspectives in wood certification and forensic: dry wood as a source of DNA. Proc. R. Soc. Lond. B. 269: 1039 – 1046
Deguilloux MF, Pemonge MH, Petit RJ. 2004a. Use of microsatellites to differentiate Oak populations. Ann. For. Sci. 61: 825 – 830
Deguilloux MF, Pemonge MH, Petit RJ. 2004b. DNA based control of Oak wood geographic origin in the context of cooperage industry. Ann. For. Sci. 61 : 97 – 104
Deguilloux MF, Pemonge MH, Bertel L, Kremer A, Petit RJ. 2003. Checking the geographical origin of Oak wood: molecular and statistical tools. Mol. Ecol. 12 : 1629 - 1636.
Eisen JA. 1999. Mechanistic basis for microsatellite instability, in: Golstein, D. B. and Schlötterer, C. (Eds.). Microsatellite: evolution and applications. Oxford University Press. pp. 34-48.
Estoup A, Jarne P, Cornent JM. 2002. Homoplasy and mutation model at microsatellite loci and their consequnces for population genetic analysis.
Mol. Ecol. 11: 1591-1604.
Finkelday, R. 1998. An introduction to tropical forest genetics. Institute of Forest Genetic and Forest Tree Breeding. Göttingen University.
Gillet EM. 2005. Genetic Structures from Electrophoresis Data (GSED) Version_1.1k. Institute Forest Genetic and Forest Tree Breeding . University of Goettingen. Germany
Ginot F, Bordelais I, Nguyen S, Gyapay G. 1996. Correction of source genotyping errors in automated fluorescent microsatellite analysis by enzymatic removal of one base overhangs. Nucl. Acid. Res. 24(3): 540-541.
Grant, V. 1975. Genetics of flowering plants. Columbia University Press. United States America. pp 210-227.
Grivet D, Heinze B, Vendramin GG, Petit RJ. 2001. Genome walking with consensus primers application to the Large Single Copy Region of chloroplast DNA. Mol. Ecol. 1 : 345 - 349.
37
Hancock JM. 1999. Microsatellite and other simple sequences: genomic context and mutational mechanism, in: Golstein, D. B. and Schlötterer, C. (Eds.). Microsatellite: evolution and applications. Oxford University Press. pp. 1-9. Heuertz M et al. 2004. Chloroplast DNA variation and postglacial
recolonizations of Common ash (Fraxinus excelsior L.) in Europe. Mol. Ecol. 13 : 34437 - 3452
Indrioko S. 2005. Chloroplast DNA variation in Indonesia Dipterocarpaceae- phylogenetic, taxonomic and population genetic aspects. Dissertation PhD. Cuvillier Verlag. Germany.
Ishiyama, H., Kado, T., Iwasaki, M., Matsuoka, M., Shukor, NA and Szmidt AE. 2003. Nucleotide variation in the GapC region of four species of Shorea and their putative hybrids. Tropics 13(2): 89-99
Isoda K, Yasman I, Rimbawanto A, Prihatini I. 2001. Estimation of ge netic diversity of Shorea leprosula in the Hedge-Orchad of the Inhutani I Dipterocap center East Kalimantan using DNA marker in: THIELGES,B.A., SASTRAPRADJA, S.D., RIMBAWANTO A. (Eds). In situ and ex situ
conservation of commercial tropical trees. ITTO Project PD 16/96 Rev. 4(F). Faculty of Forestry Gadjah Mada University. Yogyakarta. pp. 378-388.
Jackson HD, Steane DA, Potts BM, Vaillancourt RE. 1999. Chloroplast DNA evidence for reticulate evolution in Eucalyptus (Myrtaceae). Mol. Ecol, 8: 739-751
Kajita T et al. 998. Molecular phylogeny of Dipterocarpaceae in Southeast Asia based on nucleotide sequences of matK, trnL intron, and trnL-trnF intrgenic spacer region in chloroplast DNA. Molecular Phylogenetics and Evolution. 10. 2: 202-209.
Kamiya K, Harada K, Tachida H, Ashton PS. 2005. Phylogenetic of PgiC Gene in Shorea and its closely related genera (Dipterocarpaceae) the dominant trees in Southeast Asian tropical rain forest. Am. J. Bot. 92 (5) : 775 – 788
Larena BG, Aguilar JF, Feliner GN. 2002. Glacial- induced altitudinal migrations in Armeria (Plumbaginaceae) infered from patterns of chloroplast DNA haplotype sharing. Mol. Ecol, 11: 1965-1974.
Lee SL, Tani N, Ng KKS, Tsumura Y. 2004. Isolation and characterization of 20 microsatellite loci for an important tropical tree species Shorea leprosula
Lee SL, Wickneswari R, Mahani MC, Zakri AH. 2000a. Mating system parameters in a tropical tree species, Shorea leprosula Miq. (Dipterocarpaceae), from Malaysian lowland dipterocarp forest. Biotropica
32(4): 693-702.
Lee SL, Wickneswari R, Mahani MC, Zakri AH. 2000b. Genetic diversity of a tropical tree species, Shorea leprosula Miq. (Dipterocarpaceae), in Malaysia: implication for conservation of genetic resources and tree improvement. Biotropica. 32(2): 213-224.
Li YC, Korol AB, Fahima T, Beiles A