i
ISOLASI DAN IDENTIFIKASI KERAGAMAN GENETIKA
Cronobacter
spp. (
Enterobacter sakazakii
) YANG DIPEROLEH
DARI PRODUK PANGAN KERING
FRANSISCA WANNY HAMDANI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
ii Dengan ini saya menyatakan bahwa tesis Isolasi dan Identifikasi Keragaman Genetika Cronobacter spp. (Enterobacter sakazakii) yang Diperoleh dari Produk Pangan Kering adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, Juni 2012
Fransisca Wanny Hamdani
iii Identification of Cronobacter spp. (Enterobacter sakazakii) from Dried Foods.
Under the direction of RATIH DEWANTI HARIYADI and SRI HENDRASTUTI HIDAYAT.
Cronobacter spp. (formerly Enterobacter sakazakii) is a group of emerging pathogen that has been implicated as the causative agent of meningitis and necrotizing enterocolitis in certain groups of infants. In Indonesia several isolates have been obtained mostly from powder infant formula and weaning foods. This research aimed to obtain Cronobacter spp. isolates from other dried foods i.e. weaning foods, flours, starches and spices and evaluated the biochemical and genotypic characteristics of the isolates. Isolation was conducted according to FDA method (2002) modified with Iversen and Forsythe (2004), biochemical was conducted by API 20E test and genotypic characterization was conducted by PCR amplification of their 16S rRNA gene using 3 primer pairs respectively, 16 SUNI-L/Saka 2b (Segment 1), ESA1/16 SUNI-R (Segment 2) and 16SUNI-L/16SUNI-R (Universal segment). Results of the DNA amplification were sequenced and then analyzed for their relatedness to reference isolates of C.sakazakii from GenBank using BLAST program. In this study 62 samples of weaning foods, flours, starches and spices were screened for the presence of Cronobacter spp. Twelve typical colonies on DFI were obtained from sample of flours, starches and spices samples. Biochemical test using API 20E suggested that 11 of the 12 presumptives isolates were E. sakazakii while one was identified as E. cloacae. Genotypic analysis revealed that eleven isolates including the one identified as E.cloacae by API test were confirmed as C.sakazakii while one isolates was C.muytjensii. Relationships between strains based on segment 1 was depicted in a dendrogram using the neighbor-joining method showed that the twelve isolates can be differentiated into two groups, meanwhile when universal segment was used, the isolates can be differentiated into three groups.
iv FRANSISCA WANNY HAMDANI. Isolasi dan Identifikasi Keragaman Genetika
Cronobacter spp. (Enterobacter sakazakii) yang Diperoleh dari Produk Pangan Kering. Dibimbing oleh RATIH DEWANTI HARIYADI dan SRI HENDRASTUTI HIDAYAT.
Cronobacter spp. (Enterobacter sakazakii)adalah bakteri oportunistik yang banyak ditemukan di lingkungan kemudian dilaporkan sebagai emerging foodborne pathogen. Bakteri ini memiliki ciri-ciri umum seperti: Gram negatif, vegetatif, anaerob fakultatif, berbentuk batang pendek (0,5-1,0 x 1,0-3,0 μm) dengan pigmen warna kuning, peritrichous, dan tidak membentuk spora serta memiliki kapsul yang menyelimuti tubuhnya sebagai mekanisme pertahanan diri. Bakteri ini termasuk dalam famili Enterobacteriaceae dan genus Enterobacter, namun kemudian berdasarkan hasil analisis pendekatan polyphasic taxonomy
diklasifikasi ulang sebagai genus baru yaitu ‘Cronobacter’ (Iversen et al. 2007). Hal tersebut didasarkan pada enzim α-glukosidase, yang terekspresi pada sebagian besar Cronobacter dan hanya sedikit pada Enterobacteriaceae lain.
C. sakazakii merupakan patogen penting yang mengancam kesehatan
utamanya pada bayi kelompok tertentu yaitu: memiliki berat badan rendah, bayi lahir prematur maupun bayi yang dilahirkan oleh ibu penderita AIDS (acquired immune deficiency syndrome), sehingga mengakibatkan septicaemia, meningitis
(radang otak) dan necrotizing enterocolitis (peradangan usus). Selain itu
C.sakazakii juga dapat menyerang orang dewasa utamanya yang tergolong
memiliki daya tahan tubuh rendah.
Meskipun belum ada laporan kasus infeksi oleh Cronobacter spp. (E. sakazakii) di Indonesia, namun Estuningsih et al. (2006a) dan Meutia (2008)
melaporkan keberadaan bakteri ini di dalam makanan bayi dan susu formula. Gitapratiwi et al. (2012), melaporkan adanya E. sakazakii pada makanan bayi dan beberapa produk makanan kering seperti tepung maizena dan bubuk coklat. Selain itu Senzani (2011), menemukan adanya C. sakazakii pada sayuran segar yaitu kol yang berasal dari pasar lokal kota Bogor.
Penelitian ini bertujuan untuk mengisolasi Cronobacter spp. (Enterobacter sakazakii) dari berbagai jenis pangan kering seperti: makanan bayi, tepung, pati dan rempah-rempah kering. Isolat yang diperoleh kemudian diuji secara biokimia lalu dievaluasi keragaman genetikanya dengan metode PCR menggunakan tiga pasang primer penyandi gen 16S rRNA C. sakazakii. Hasil tersebut kemudian diidentifikasi keragaman genetikanya dengan cara mengevaluasi hubungan kekerabatan antara isolat yang diperoleh dengan isolat-isolat lokal yang diperoleh dari beberapa penelitian sebelumnya dan juga isolat-isolat dari luar Indonesia yang terdapat pada GenBank berdasarkan gen 16S rRNA.
Manfaat penelitian ini adalah menambah basis data isolat lokal C. sakazakii
v singkong (tapioka), tiga isolat dari cabai bubuk, satu isolat dari jintan, satu isolat dari ketumbar bubuk, satu isolat dari pala bubuk dan satu isolat dari merica bubuk.
Hasil analisis reaksi biokimiawi isolat-isolat yang diduga C. sakazakii dengan menggunakan program apiwebTM menunjukkan bahwa isolat FWH d2c, FWh d2u, FWH d11 dan FWH d12 terindikasi sebagai C. sakazakii dengan kemiripan masing-masing 99.9%; FWH d1 terindikasi sebagai C. sakazakii dengan kemiripan 99.1%;
FWH b2, FWH b11, FWH b15 dan FWH d16 terindikasi sebagai C. sakazakii
dengan kemiripan masing-masing 98.4%; FWH b6 dan FWH b14 terindikasi sebagai C. sakazakii dengan kemiripan masing-masing 51.9% dan hanya isolat FWH c3 yang terindikasi sebagai E. cloacae dengan kemiripan 69,1%.
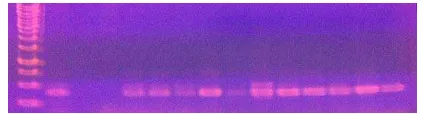
Karakterisasi genotipik berdasarkan gen 16S rRNA menyimpulkan bahwa kedua belas isolat adalah Cronobacter spp. dan menghasilkan produk PCR (amplikon) baik segmen 1 (1000 bp), segmen 2 (408 bp) dan segmen universal (1402 bp) yang sama dengan isolat ATCC 51329 sebagai kontrol positif yang tampak pada pola elektroforesisnya.
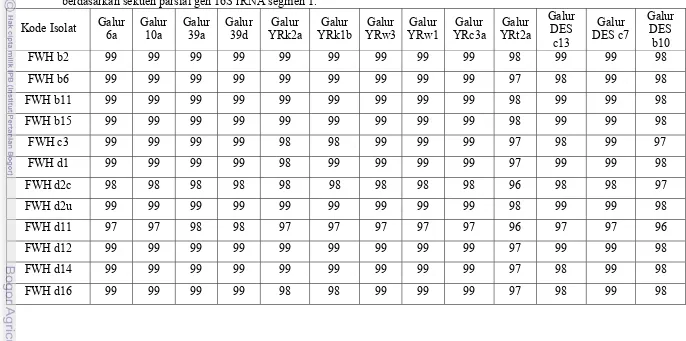
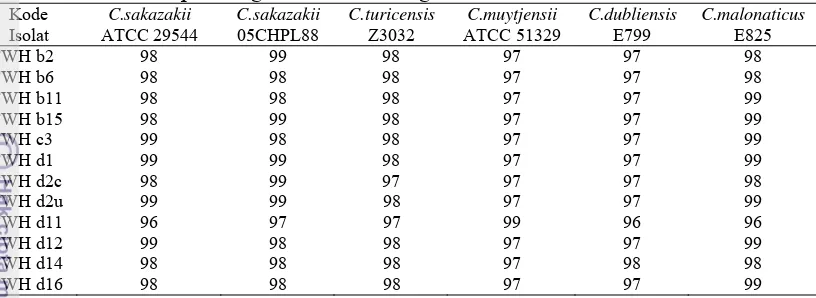
Pohon filogenetik dari isolat-isolat Cronobacter spp. berdasarkan sekuen parsial gen 16S rRNA segmen 1 menggambarkan terbentuknya 2 kelompok. Kelompok pertama menempatkan isolat-isolat lokal yaitu E. sakazakii galur 6a, 10a, 39a dan 39d, C. sakazakii galur YRw3, YRw1, YRc3a, YRt2a, YRk2a dan YRk1b, C. sakazakii galur DESb10, DESc13 dan DESc7, FWH c3, FWH d1, FWH d16, FWH d12, FWH b11, FWH b15, FWH d2c, FWH d2u, FWH b2, FWH b6 dan FWH d14 bersama dengan C. sakazakii ATCC 29544, C. sakazakii
05CHPL88 dan C. malonaticus galur E825. Kelompok kedua menempatkan isolat lokal FWH d11 bersama dengan C. muytjensii ATCC 51329.
Pohon filogenetik dari isolat-isolat Cronobacter spp. berdasarkan sekuen parsial gen 16S rRNA dengan primer universal menggambarkan terbentuknya 3 kelompok. Kelompok pertama menempatkan isolat-isolat lokal yaitu E. sakazakii
galur 6a dan 10a, C. sakazakii galur YRw3 dan YRw1 FWH b6, FWH d2c, FWH d16, FWH d11, FWH b2 dan FWH d14 bersama dengan C. sakazakii ATCC 29544 dan C. malonaticus galur E825. Kelompok kedua menempatkan isolat-isolat lokal yaitu YRc3a, YRt2a, YRk2a dan YRk1b, C. sakazakii galur DES c13,
DES b10 dan DES c7, E. sakazakii galur 39a dan 39d bersama dengan
C. sakazakii galur 05CHPL88. Kelompok ketiga menempatkan isolat lokal FWH d1 bersama dengan C. muytjensii ATCC 51329.
vi
© Hak cipta milik IPB, tahun 2012
Hak cipta dilindungi undang-undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
vii
FRANSISCA WANNY HAMDANI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Ilmu Pangan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
ix Nama : Fransisca Wanny Hamdani
NIM : F251090181
Disetujui Komisi Pembimbing
Dr. Ir. Ratih Dewanti-Hariyadi,M.Sc Dr. Ir. Sri Hendrastuti Hidayat, M.Sc Ketua Anggota
Diketahui
Ketua Program Studi Ilmu Pangan Dekan Sekolah Pascasarjana IPB
Dr. Ir. Ratih Dewanti-Hariyadi, M.Sc Dr. Ir. Dahrul Syah, M.Sc.Agr
x Puji syukur penulis panjatkan kehadirat Tuhan Yesus Kristus atas segala tuntunan dan penyertaan-Nya sehingga karya ilmiah ini dapat penulis selesaikan. Rangkaian kegiatan penelitian dan penulisan tesis ini merupakan salah satu syarat untuk memperoleh gelar Magister Sains pada Program Studi Ilmu Pangan, Sekolah Pascasarjana IPB.
Penulis mengucapkan terima kasih dan penghargaan setinggi-tingginya kepada Dr. Ir. Ratih Dewanti-Hariyadi, M.Sc. selaku ketua komisi pembimbing dan Dr. Ir. Sri Hendrastuti Hidayat, M.Sc. selaku anggota komisi pembimbing yang telah memberikan semangat, arahan, masukan dan saran selama pelaksanaan penelitian dan penulisan karya ilmiah ini. Ucapan terima kasih juga disampaikan kepada Direktorat Jenderal Pendidikan Tinggi, Kementrian Pendidikan Republik Indonesia yang telah mendanai penelitian ini melalui skema Hibah Penelitian Pascasarjana.
Ungkapan terima kasih pula kepada Ayahanda Johanis Irwan Hamdani dan ibunda dr. Sheny Sumalgo atas motivasi, semangat, doa dan kasih sayang yang selalu diberikan kepada penulis dalam suka dan duka; keluargaku khususnya: Akbar, Ika, Alky, Jasmin, Elke, Ris, Lani, Harry, Ita dan seluruh keluarga besar atas doa dan semangat yang diberikan.
Terima kasih penulis ucapakan kepada: (1) staf laboratorium SEAFAST Center IPB: Mbak Ari, Mas Yerris, Mas Taufik, Mbak Ria, Mbak Lira, (Almh) Bu Entin, Bu Sari, Pak Abah, Bu Eva atas bantuannya selama pelaksanaan penelitian, (2) Desty Gitapratiwi atas bimbingan, nasihat, motivasi dan perhatian yang diberikan selama penulis melakukan penelitian (3) sahabat-sahabatku :Indra, Meily, Pik Wie, Since, Yuni, Laura, Felix, Grace, Hong Hong; teman-teman THP UNHAS; teman-teman UOB Buana Makassar; teman-teman pelayan Pelkat PA dan Pelkat GP GPIB Bethania Makassar, yang tetap setia dalam memberikan dukungan, semangat dan doa, (4) teman-teman seperjuangan di IPN: Tina, Riyanti, Melina, Hermawan, Fenny, Mbak Wida, Rani, Rangga, Dede,Vanessa, Dian, Kiki, Mbak Lina, Mbak Tanti, Bu Indah Pak Supriyadi, Melinda, Nandi, Pak Ikhsan, Bu Tita, Bu Zita, Bu Meilan, Bu Siti, Bu Aswita, Winnie, Cicoy, Zulia, Ria, Rion dan Iza (5) keluarga besar wisma Flora: Ka Teti, Agnes, Devi, Widi, Ika, Nie, Uthe, Septi, Dimas, Angel, Halimah, Indy, Diah, Kiki, Yuyun,Vina, Abrar, Efan, Mba Fitri, Reza, Bang Rudi, Mas Yuli, (6) teman-teman lab Viro: Aceu, Mba Dwi, Herma, Pak Santoso, Mba Tuti, (7) keluarga besar Prima Copy Center dan (8) semua pihak yang tidak dapat saya sebutkan satu persatu atas dukungan dan doanya selama ini.
Semoga Tuhan Yang Maha Esa membalas budi baik Bapak/Ibu/Saudara/i semuanya. Semoga karya ilmiah ini bermanfaat.
Bogor, Juni 2012
xi Penulis dilahirkan di Ujung Pandang pada tanggal 14 Juni 1983 sebagai anak tunggal dari ayah Johanis Irwan Hamdani dan ibu dr. Sheny Sumalgo. Penulis lulus dari SMU Katolik Rajawali pada tahun 2001 dan melanjutkan pendidikan sarjana di Jurusan Teknologi Hasil Pertanian, Fakultas Pertanian dan Kehutanan, Universitas Hasanuddin Makassar, dan lulus pada tahun 2006. Pada tahun 2009, penulis mendapatkan kesempatan untuk melanjutkan pendidikan magister sains di Program Studi Ilmu Pangan, Sekolah Pascasarjana, Institut Pertanian Bogor.
xii
Halaman
DAFTAR TABEL ... xiii
DAFTAR GAMBAR ... xiv
DAFTAR LAMPIRAN ... xvi
PENDAHULUAN Latar Belakang ... 1
Tujuan Penelitian ... 3
Manfaat Penelitian ... 3
TINJAUAN PUSTAKA Karakteristik Cronobacter spp. (E. sakazakii) ... 4
Sumber Cronobacter sakazakii ... 5
Cronobacter spp. (E. sakazakii) pada Produk Pangan ... 6
Perilaku Cronobacter spp. (E.sakazakii)dalam Lingkungan Pangan ... 8
Penyakit Akibat Cronobacter sakazakii ... 9
Metode Deteksi Cronobacter spp.(E. sakazakii) ... 10
Keragaman Genetika Cronobacter sakazakii ... 16
Analisis Neighbor-Joining ... 20
BAHAN DAN METODE Tempat dan Waktu ... 22
Bakteri Uji ... 22
Bahan dan Media ... 22
Alat ... 24
Perangkat Lunak (Software) ... 24
Pelaksanaan Penelitian ... 25
Isolasi Bakteri C. sakazakii (Metode Modifikasi FDA) ... 25
Identifikasi Isolat Secara Biokimia ... 26
Analisis Keragaman Genetika Cronobacter spp. (E. sakazakii) ... 28
Isolasi DNA Genom Bakteri ... 28
Elektroforesis Gel Agarosa ... 29
Amplifikasi Gen 16S rRNA ... 30
xiii
Karakteristik Biokimia Cronobacter spp. (E. sakazakii) ... 37
Identifikasi dan Karakterisasi Genotipik Cronobacter spp. (E. sakazakii) ... 39
Isolasi DNA Genom Bakteri ... 39
Amplifikasi Gen 16S rRNA ... 40
Analisis Sekuen Parsial Gen 16S rRNA ... 43
Analisis Keragaman Genetika Cronobacter spp. (E. sakazakii) ... 49
Analisis Filogeni ... 49
Keterkaitan Antara Kekerabatan Isolat-Isolat Lokal C. sakazakii Dengan Resiko Kontaminasi pada Pangan Lokal ... 52
SIMPULAN DAN SARAN ... 53
xiv Halaman 1
2
3 4
5
6
7
8
9
10
11
12
Sifat biokimia galur-galur Cronobacter spp. ………... Pasangan primer oligonukleotida yang digunakan untuk
amplifikasi gen penyandi 16S rRNA C. sakazakii ………... Sifat biokimia spesies Enterobacter ……….. Hasil isolasi Cronobacter spp. (E. sakazakii) dari berbagai sumber berdasarkan pertumbuhan koloni pada media VRBG dan DFI ………. Isolat presumtif Cronobacter spp. (E. sakazakii) dan asal sampel yang diperoleh ..………... Hasil identifikasi takson dan tingkat kemiripan isolat presumtif
Cronobacter spp. (E. sakazakii) dengan program apiwebTM …… Sumber dan nomor akses galur acuan Cronobacter spp. yang digunakan ………... Homologi (%) antar isolat lokal yang dianalisis dengan galur-galur isolat lokal (dari Indonesia) Cronobacter spp. dari
GenBank berdasarkan sekuen parsial gen 16S rRNA segmen 1… Homologi (%) antar isolat lokal yang dianalisis dengan galur-galur isolat Cronobacter spp. luar Indonesia dari GenBank
berdasarkan sekuen parsial gen 16S rRNA segmen 1…………... Homologi (%) antar isolat lokal Cronobacter spp. hasil isolasi yang dianalisis berdasarkan sekuen parsial gen 16S rRNA segmen 1 …………... Homologi (%) antar isolat lokal yang dianalisis dengan galur-galur isolat Cronobacter spp. luar Indonesia dari GenBank
berdasarkan sekuen parsial gen 16S rRNA segmen universal ….. Homologi (%) antar isolat lokal Cronobacter spp. yang dianalisis berdasarkan sekuen parsial gen 16S rRNA segmen universal …...
5
23 28
34
36
38
44
46
47
47
48
xv
Halaman
1
2
3 4
5
6
7
8
Dendogram keragaman sekuen Neighbor-Joining berdasarkan sekuen parsial 16S rRNA segmen 1 yang menggunakan primer 16 SUNI-L dan Saka-2b berukuran 977 bp isolat C. sakazakii yang diisolasi dari makanan bayi ………... Peta amplifikasi fragmen gen 16S rRNA Cronobacter spp.
(Enterobacter sakazakii) dengan menggunakan pasangan primer 16 SUNI-L dan Saka-2b, ESA-1 dan 16 SUNI-R, 16 SUNI-L dan 16 SUNI-R ………... Diagram alir kegiatan penelitian keragaman genetika C. sakazakii.. Pertumbuhan isolat presumtif Cronobacter spp. (E. sakazakii) pada beberapa media : (a dan b) VRBG, (c) DFI, (d) TSA ……… Sel bakteri C.sakazakii yang diamati di bawah mikroskop dengan perbesaran 1000x ……….. Visualisasi DNA hasil amplifikasi fragmen 16S rRNA segmen 1, menggunakan pasangan primer 16 SUNI-L/Saka-2b bakteri
C.sakazakii pada gel agarosa 1.5% (TAE). Lajur M merupakan marker DNA ladder 1 kb. K merupakan kontrol positif yaitu ATCC 51329 dan kn merupakan kontrol negatif. Sampel terdiri dari: FWH b2 (1), FWH b6 (2),FWH b11(3), FWH c3 (4), FWH b15 (5), FWH d1 (6), FWH d2c (7), FWH d2u (8), FWH d11 (9), FWH d12 (10), FWH d14 (11), FWh d16 (12) ……… Visualisasi DNA hasil amplifikasi fragmen 16S rRNA segmen 1, menggunakan pasangan primer ESA-1/16 SUNI-R bakteri
C.sakazakii pada gel agarosa 1.5% (TAE). Lajur M merupakan marker DNA ladder 1 kb. K merupakan kontrol positif yaitu ATCC 51329 dan kn merupakan kontrol negatif. Sampel terdiri dari: FWH b2 (1), FWH b6 (2),FWH b11(3), FWH c3 (4), FWH b15 (5), FWH d1 (6), FWH d2c (7), FWH d2u (8), FWH d11 (9), FWH d12 (10), FWH d14 (11), FWh d16 (12) ……… Visualisasi DNA hasil amplifikasi fragmen 16S rRNA segmen universal, menggunakan pasangan primer 16 SUNI-L/16 SUNI-R bakteri C.sakazakii pada gel agarosa 1.5% (TAE). Lajur M merupakan marker DNA ladder 1 kb. K merupakan kontrol positif yaitu ATCC 51329 dan kn merupakan kontrol negatif. Sampel terdiri dari: FWH b2 (1), FWH b6 (2),FWH b11(3), FWH c3 (4), FWH b15 (5), FWH d1 (6), FWH d2c (7), FWH d2u (8)FWH d11 (9), FWH d12 (10), FWH d14 (11), FWh d16 (12) ……….
21
24 25
32
37
40
41
xvi
Halaman
9
10
Dendogram dari galur-galur Cronobacter spp. berdasarkan gen 16S rRNA segmen 1. Metode analisis menggunakan Neighbor joining dengan koreksi Felsenstein (1000 replikat Bootstrap). Skala menunjukkan estimasi keragaman sekuen sebesar 5% …….. Dendogram dari galur-galur Cronobacter spp. berdasarkan gen 16S rRNA segmen universal. Metode analisis menggunakan
Neighbor joining dengan koreksi Felsenstein (1000 replikat
Bootstrap). Skala menunjukkan estimasi keragaman sekuen
sebesar 5% ………
50
xvii
Halaman
1
2
3
4
5
6
7
8
9
10
11
Hasil isolasi bakteri Cronobacter spp. (E. sakazakii) dari beberapa produk pangan kering ………... Hasil identifikasi isolat diduga C. sakazakii dengan API 20E yang dianalisis dengan apiwebTM………... Hasil uji reaksi biokimiawi isolat bakteri diduga Cronobacter spp.
(E. sakazakii) dengan perangkat API 20E ……….
Konsentrasi dan kemurnian DNA genom bakteri Cronobacter spp.
(E. sakazakii) ………
Jumlah nukleotida isolat lokal Cronobacter spp. (E. sakazakii) yang berhasil disekuensing dengan primer segmen 1 ……….. Hasil identifikasi isolat hasil isolasi berdasarkan sekuen parsial gen 16S rRNA segmen 1 ……….. Homologi (%) antar isolat Cronobacter spp. dari luar Indonesia
pada GenBank ………..
Urutan basa (nukleotida) gen 16S rRNA segmen 1 isolat-isolat lokal C. sakazakii yang dianalisis setelah contig ………. Jumlah nukleotida isolat lokal Cronobacter spp. (E. sakazakii) yang berhasil disekuensing menggunakan primer universal …….. Hasil identifikasi isolat hasil isolasi berdasarkan sekuen parsial gen 16S rRNA segmen universal ………. Urutan basa (nukleotida) gen 16S rRNA segmen universal isolat-isolat lokal C. sakazakii yang dianalisis setelah contig ………
63
69
75
77
78
79
80
81
87
88
PENDAHULUAN
Latar Belakang
Enterobacter sakazakii adalah mikroba patogen emerging yang dapat mencemari pangan. Bakteri ini merupakan mikroorganisme vegetatif, fakultatif anaerob, berbentuk batang dan tidak membentuk spora. Pada mulanya bakteri ini termasuk dalam famili Enterobacteriaceae dan genus Enterobacter. Kemudian
berdasarkan hasil analisis taksonomo polifase dan keberadaan enzim α-glukosidase, yang dimiliki sebagian besar Cronobacter dan hanya sedikit pada
Enterobacteriaceae lainnya disusunlah reklasifikasi E. Sakazakii ke dalam genus baru ‘Cronobacter’ yang terbagi atas enam spesies (Iversen et al. 2007).
Beberapa penelitian menyatakan bahwa Cronobacter spp. bisa diisolasi dari
spektrum yang luas dan kemungkinan besar berasal dari tumbuhan karena disinyalir bahwa lingkungan (tanah, air, tikus dan lalat) serta makanan
(sayuran) merupakan sumber infeksi. Schmid et al. (2009), menyelidiki kemampuan Cronobacter spp. untuk berkolonisasi pada akar tanaman. Selanjutnya Walsh et al. (2010), menemukan Cronobacter spp. tumbuh lebih baik dan bertahan pada jangka waktu yang lama dalam ingridien susu formula kering terutama yang berasal dari tanaman sehingga dapat menjadi sumber kontaminasi
bagi alat-alat pengolahan dalam pabrik. Jaradat et al. (2009), menemukan
C. sakazakii lebih banyak pada produk herbal dan rempah-rempah sehingga dapat disimpulkan bahwa sumber bakteri patogen khususnya C. sakazakii adalah tanaman.
Kasus infeksi Cronobacter spp. (E. sakazakii) Indonesia belum pernah dilaporkan tetapi Estuningsih et al. (2006a; 2006b) melaporkan keberadaan bakteri ini dari susu formula lanjutan (22.73%, n=22) dan makanan bayi (46.7%, n=15); Meutia et al. (2008) melaporkan keberadaan bakteri ini pada susu formula (18.75%, n=16) dan makanan bayi (55.5%, n=9). Gitapratiwi et al. (2012), juga melaporkan keberadaan C. sakazakii pada makanan bayi (20%, n=15), pati-patian (pati jagung/maizena) (11.8%, n=17) dan produk pangan kering lainnya yaitu bubuk coklat (6.3%, n=16). Senzani (2011), mengisolasi satu isolat C.sakazakii
dari sayur kubis diantara sembilan belas sampel sayur (5.26%) yang berasal dari daerah Bogor.
Gitapratiwi et al. (2012), selanjutnya melakukan analisis keragaman genetika berdasarkan sekuen parsial gen 16S rRNA dan menyimpulkan adanya tingkat kemiripan yang bervariasi antara isolat-isolat lokal yang diperoleh dengan isolat Estuningsih et al. (2006a) dan Meutia et al. (2008), dan dengan galur-galur
C. sakazakii dari luar Indonesia berdasarkan data dari GenBank. Selain itu, disimpulkan pula bahwa isolat-isolat lokal memiliki kedekatan kekerabatan secara genetika meskipun berasal dari sumber yang berbeda (Gitapratiwi 2011).
Penelitian lebih lanjut mengenai keberadaan C. sakazakii perlu dilakukan pada beberapa produk pangan kering, kemudian dilakukan analisis secara
biokimia maupun keragaman genetika antara isolat-isolat Cronobacter spp. (E. sakazakii) yang diperoleh dengan isolat-isolat lokal dan isolat-isolat dari luar
negeri berdasarkan data dari GenBank. Informasi mengenai keragaman genetika
Cronobacter spp. yang ada di Indonesia dapat berkontribusi pada reklasifikasi spesies Cronobacter spp. (E. sakazakii).
Pengetahuan mengenai keragaman genetika bakteri patogen ini diharapkan dapat dihubungkan dengan perilakunya selama pengolahan maupun penanganan bahan pangan. Selain itu juga dapat diketahui sejauh mana bakteri Cronobacter
spp. (E. sakazakii) dapat melakukan kontaminasi silang utamanya pada
Dengan demikian diharapkan dapat diidentifikasi karakteristik penting
Cronobacter spp. (E. sakazakii) yang memberikan risiko lebih besar utamanya terhadap bayi maupun masyarakat di Indonesia serta cara pengendaliannya.
Tujuan Penelitian
1. Mendapatkan isolat Cronobacter spp. (E. sakazakii) dari beberapa bahan pangan kering di Indonesia.
2. Melakukan identifikasi secara biokimia dan genotipik isolat Cronobacter
spp. (E. sakazakii) yang diperoleh
3. Melakukan analisis keragaman genetika melalui perbandingan isolat yang diperoleh dengan isolat-isolat lokal dari beberapa penelitian sebelumnya dan juga isolat-isolat dari luar Indonesia dari data GenBank berdasarkan gen 16S rRNA.
Manfaat Penelitian
1. Mempelajari karakteristik biokimia dan genetik isolat lokal Cronobacter
spp. (E. sakazakii) di Indonesia.
TINJAUAN PUSTAKA
Karakteristik Cronobacter spp. (E. sakazakii)
Enterobacter sakazakii merupakan bakteri Gram negatif, anaerob fakultatif berbentuk batang pendek (0,5-1,0 x 1,0-3,0 μm), peritrichous, memiliki pigmen warna kuning, tidak membentuk spora dan memiliki kapsul yang menyelimuti tubuhnya sebagai mekanisme pertahanan diri. Pada media pembiakan, koloninya nampak licin berlendir namun juga terkadang kering. Karena memiliki karakteristik biokimia yang sama, bakteri ini pertama kali dikenal dengan nama
E. cloacae berpigmen kuning. Pada tahun 1980, berdasarkan perbedaan dengan
E. cloacae dalam hal kekerabatan DNA, reaksi-reaksi biokimiawi, produksi pigmen dan ketahanannya terhadap antibiotik bakteri ini dimasukkan dalam genus
Enterobacter sebagai suatu spesies baru yang diberi nama E. sakazakii sebagai
penghormatan seorang bakteriolog Jepang bernama Riichi Sakazakii (Farmer
et al. 1980).
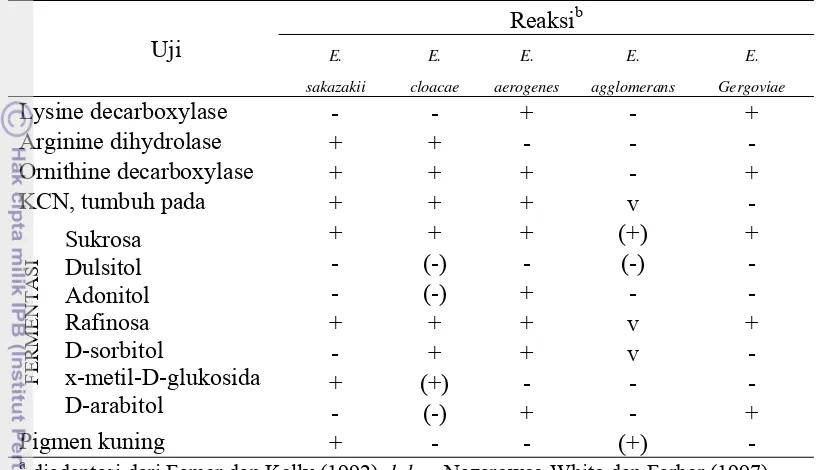
Berdasarkan profil biokimiawinya, E. sakazakii sangat menyerupai
E. cloacae, tetapi E. sakazakii selalu bersifat D-sorbitol negatif, DNase
ekstraselular positif dan membentuk koloni berpigmen kuning sedangkan
E. cloacae bersifat D-sorbitol positif, DNAse ekstraselular negatif dan tidak membentuk koloni berpigmen kuning. E. sakazakii terbagi atas 15 biogrup dengan karakteristik biokimia dulcitol positif dan α-metil-glukosida negatif yang tidak ditemukan pada karakterisiktik biokimia galur-galur Enterobacteriaceae
yang lain. Untuk biogrup 15 memiliki karakteristik yaitu indol positif dan malonat positif, dimana kombinasi karakteristik tersebut tidak ditemukan di galur-galur yang lain (Farmer et al. 1980). Pembentukan pigmen kuning lebih kuat pada suhu 25 °C dibandingkan pada suhu 36 °C. Semua galur dapat tumbuh dengan cepat pada media TSA dan membentuk koloni 1.5-3 mm setelah 1-2 hari (Nazarowec-White et al. 2003).
menjadi 16 biogrup. Spesies-spesies tersebut adalah : Cronobacter sakazakii
(biogrup 1-4, 7, 8 , 11 dan 13), Cronobacter malonaticus (biogrup 5, 9, 14),
Cronobacter dublinensis (biogrup 6, 10, 12), Cronobacter muytjensii (biogrup 15), Cronobacter turicensis (biogrup 16) dan Cronobacter genomospecies I. (Iversen et al. 2007). Cronobacter dublinensis terbagi atas beberapa subspesies yaitu : Cronobacter dublinensis subsp. dublinensis subsp. nov. (biogrup 12),
Cronobacter dublinensis subsp. lausanensis subsp. nov. (biogrup 10) dan
Cronobacter dublinensis subsp. lactaridi subsp. nov. (biogrup 6) (Iversen et al. 2008a). Karakteristik biokimia galur Cronobacter spp. (E. sakazakii) secara biokimia antara lain menunjukkan reaksi positif terhadap produksi asam dari metil-α-D-glukosida dan menunjukkan reaksi negatif terhadap produksi asam dari
dulcitol, produksi indol dan pemanfaatan malonat, untuk galur-galur Cronobacter
spp. yang lain secara keseluruhan dapat terlihat pada Tabel 1. Tabel 1 Sifat biokimia galur-galur Cronobacter spp.
Galur Cronobacter spp. Dul Ind Malo AMG
Cronobacter sakazakii subsp. sakazakii*
Cronobacter sakazakii subsp. malonaticus*
Cronobacter muytjensii*
Cronobacter dublinensis subs. dublinensis*
Cronobacter dublinensis subs. lausanensis**
Cronobacter dublinensis subs. lactaridi**
Cronobacter turicensis*
Cronobacter genomospecies I*
-
Dul : produksi asam dari dulcitol; Ind : produksi indol; Malo : pemanfaatan malonat; AMG : produksi asam dari metil-α-D-glukosida; untuk * + : 85-100% positif, - : kurang dari 15% positif, V: variable; untuk ** + : lebih besar dari 90% positif, - : kurang dari 10% positif, V : 20-80% positif.
*Iversen et al. (2007); **Iversen et al. (2008).
Sumber Cronobacter sakazakii
Cronobacter spp. merupakan bakteri patogen yang terdapat dimana-mana (Cawthorn et al. 2008). Patogen ini telah diisolasi dari berbagai sumber antara lain lingkungan, klinis, makanan maupun minuman. Salah satu sumber Cronobacter
(Mullane et al. 2008). Kontaminasi secara intrinsik pada susu formula dapat terjadi melalui penambahan bahan baku yang telah terkontaminasi atau melalui lingkungan pengolahan pada proses pengemasan. Kontaminasi ekstrinsik dapat terjadi pada saat rekonstitusi susu formula dengan peralatan yang telah terkontaminasi (Mullane et al. 2008).
Habitat alami dan cara penularan C. sakazakii hingga saat ini masih terus diselidiki. Bakteri ini biasanya dapat ditemukan di lingkungan ataupun dalam makanan. Kemampuan bakteri ini menghasilkan polisakarida ekstraseluler (gum like) dan bertahan dalam keadaan kering menunjukkan bahwa sumber utama dari lingkungan berhubungan erat dengan tanaman. Hal tersebut dibuktikan oleh penelitian Schmid et al. (2009) yang mengamati galur-galur Cronobacter spp. yang dipakai untuk melihat proses kolonisasinya pada akar tanaman tomat dan jagung, menunjukkan adanya solubilitas dari fosfat mineral dan produksi asam asetat indol. Karakteristik tersebut biasanya ditemukan pada tanaman yang terkait bakteri dan mikroorganisme rhizosfer, sehingga dapat disimpulkan bahwa habitat alami Cronobacter spp. kemungkinan berasal dari tumbuhan
Penularan C. sakazakii dapat terjadi secara eksogen, misalnya melalui kotoran manusia, dari orang ke orang, dari ibu dan bayi yang dikandung, melalui makanan, peralatan rumah sakit, dan secara endogen seperti dari usus manusia sendiri. Penularan secara pasif yang berasal dari tangan tenaga medis juga dapat menjadi modus utama penularan bakteri ini. C. sakazakii juga dapat diisolasi dari keran dan air kemasan dan dapat bertahan hidup dan berkembang biak pada atau dalam peralatan rumah sakit seperti alat hemodialisis dan maupun alat bantu pernafasan (Farmer 1999).
Cronobacter spp. (E. sakazakii) pada Produk Pangan
dari lingkungan melalui air, udara yang mengandung kotoran atau debu atau akibat kurang higienisnya personal yang terlibat dalam proses produksi (Den Aantrekker et al. 2003).
Cronobacter spp. juga dapat diisolasi dari produk pangan siap saji antara lain vanilla cream bars, produk salad, seledri, basil, lentil sprouts, onion sprouts, mung bean sprouts, sproud mixture, merica dan salad dengan campuran herbs
kering (Baumgartner et al. 2009); produk pangan segar antara lain kacang hijau (Boehme et al. 2004), produk sayuran dan produk sosis (Leclercq et al. (2002); produk pangan non kering antara lain domiatti cheese (El Sharoud et al. 2009) dan
white cheese yang diproduksi di Konya (Gökmen et al. 2010). Keberadaan
Cronobacter spp. dalam produk pangan siap saji dapat berpeluang
mengkontaminasi peralatan rumah tangga sehingga meningkatkan potensi risiko bakteri ini untuk menginfeksi orang dewasa khususnya yang memiliki daya tahan tubuh rendah (immunocompromised) (Baumgartner et al. 2009).
Keberadaan Cronobacter spp. dalam produk pangan kering telah dilaporkan oleh beberapa penelitian. Jaradat et al. (2009) mengisolasi C. sakazakii dengan total persentase tinggi dari produk rempah seperti liquorice, thyme, anise, chamomile, fennel dan sage dan campuran rempah-rempah (89.6%, n=67). Lee et al. (2010) mengisolasi C. sakazakii dan C. dublenisis dari ‘saengsik’, yaitu pangan yang terdiri atas biji-bijian, buah-buahan dan sayuran yang menjadi bubuk melalu proses pengeringan beku (freeze drying) dengan frekuensi isolasi 45% (n=41). Sementara itu Restaino et al. (2006) mengisolasi C. sakazakii dari produk tepung-tepungan, sayuran dan rempah-rempah kering dengan frekuensi isolasi 37.9% (n=83) dan Baumgartner et al. (2009) mengisolasi dari rempah-rempah dan bumbu kering dengan frekuensi isolasi 26.9% (n=26).
Berdasarkan hasil penelitian Jaradat et al. (2009), tumbuh-tumbuhan merupakan sumber utama dari bakteri patogen khususnya C. sakazakii. Tingginya persentase hasil isolasi C. sakazakii pada produk pangan kering, produk herbal dan rempah-rempah kering menunjukkan perlunya pengawasan ketat selama pengolahan, pengeringan atau pengemasan produk pangan utamanya pada susu formula dan makanan bayi. Selain itu, pada saat penyajian susu formula maupun makanan bayi perlu diperhatikan untuk menghindari terjadinya kontaminasi silang (Erkekoğlu et al. 2009)
Perilaku Cronobacter spp. (E. sakazakii)dalam Lingkungan Pangan
C. sakazakii memiliki beberapa karakteristik fisiologis khusus seperti pembentukan biofilm, resistensi terhadap antibiotik, tahan terhadap pengeringan dan tekanan osmotik (Gurtler et al. 2005; Wu et al. 2006). Bakteri C. sakazakii
tumbuh pada rentang suhu yang luas yakni 6 °C - 47 °C dengan suhu optimum pertumbuhannya 39 °C (Iversen dan Forsythe 2003). Bakteri ini memiliki kemampuan bertahan hidup dalam kondisi kering dan aw rendah sehingga
walaupun tidak tumbuh tetapi dapat bertahan dalam produk kering hingga beberapa bulan bahkan sampai dua tahun (Edelson-Mammel et al. (2005); Barron dan Forsythe 2007). Kemampuan bertahan dari Cronobacter spp dalam jangka waktu yang lama diduga karena kemampuannya mengakumulasi trehalosa dan membentuk kapsul (ekstraseluler polisakarida). Trehalosa tersebut merupakan bentuk disakarida dari glukosa mudah larut yang dapat menstabilkan protein dan membran fosfolipid sehingga melindungi bakteri dari kekeringan (Breeuwer et al.
2003). Meskipun demikian, C. sakazakii tidak dapat membentuk spora sehingga mudah diinaktivasi oleh panas.
Pasteurisasi sangat efektif dalam menghancurkan C. sakazakii (Iversen et al. 2004c). Pengasaman juga dapat mengurangi konsentrasi C. sakazakii dalam berbagai jenis susu formula dan produk makanan berbasis nabati. Dalam jus sayuran, penurunan pH setelah 48 jam berkorelasi dengan pengurangan jumlah
C. sakazakii (Kim dan Beuchat 2005).
C. sakazakii dapat hidup dalam keadaan aw rendah seperti pada produk
bubur bayi (aw 0.3-0.69) dan produk susu formula (aw 0.25-0.5). Pertumbuhan
signifikan pada suhu rendah yaitu 4 °C dengan aw 0.63-0.83 dibandingkan
penyimpanan pada suhu tinggi yaitu 21 °C dan 30 °C pada kondisi aw 0.40-0.50
dan (Gurtler dan Beuchat 2007).
C. sakazakii biasanya terdapat dalam lingkungan yang tidak bersih sehingga dapat mengkontaminasi berbagai peralatan utamanya yang digunakan dalam proses produksi (Lai 2001). Susu bubuk formula merupakan sumber infeksi yang utama bagi C. sakazakii (Drudi et al. 2006; Gurtler et al. 2005). Bakteri ini tahan terhadap pengeringan dan pH asam, panas, dan membentuk biofilm pada permukaannya.
Beberapa penelitian melaporkan bahwa spesies-spesies dari genus
Cronobacter spp. dapat membentuk biofilm dan kapsul pada berbagai jenis permukaan seperti silikon, kaca, stainless steel, lateks, polycarbonate dan
polyvinyl chloride sehingga memungkinkan bakteri ini dapat bertahan pada beberapa kondisi seperti tekanan osmotik yang tinggi, terpapar sinar UV, kondisi kering, kekurangan nutrisi, terpapar sanitisers dan antibiotik, fagosit, antibodi dan bacteriophage (Iversen et al. 2004c; Lehner et al. 2005). Produksi biofilm dipengaruhi oleh ketersediaan nutrisi dan suhu medium pertumbuhan (Kim et al.
2006).
Penyakit Akibat Cronobacter sakazakii
Penyakit akibat C. sakazakii pertama kali ditemukan pada tahun 1958 dengan 78 kasus bayi yang terkena infeksi meningitis. Beberapa publikasi menyebutkan kasus-kasus bayi baru lahir yang mengalami septicaemia,
meningitis (radang otak) dan necroitizing enterocolitis (peradangan usus) akibat
Cronobacter spp. (E. sakazakii) berkaitan dengan susu bubuk formula. Laporan mengenai infeksi akibat C. sakazakii menurut Iversen dan Forsythe (2003) dan Drudy et al. (2006) dari tahun 1958-2004 terdapat 72 kasus pada bayi dan anak-anak dimana 24 kasus diantaranya mengakibatkan kematian.
C. sakazakii dapat menginfeksi segala usia tetapi risiko terbesar adalah pada bayi usia 0-1 tahun. Pada tahun 1994 terjadi KLB (Kejadian Luar Biasa) infeksi
C. sakazakii pada bayi baru lahir di rumah bersalin di Perancis yang melibatkan 13 bayi baru lahir dan tiga diantaranya meninggal dunia (Caubilla-Barron et al.
2007). Di New Mexico pada tahun 2008, satu bayi perempuan berusia 7 minggu dikabarkan mengalami cedera otak parah dan hydrocephalus, dan bayi laki-laki berusia 7 bulan meninggal dunia akibat akibat terinfeksi oleh Cronobacter. Kasus yang melibatkan orang dewasa terjadi pada tahun 2006 berupa bacteremia yang terjadi pada seorang wanita usia 75 tahun dengan pembengkakan limpa (See et al.
2007). Pada tahun 2008 dilaporkan kasus infeksi saluran kemih akibat C. sakazakii pada seorang wanita 63 tahun dengan gagal ginjal kronis (Bhat et al. 2009).
Metode Deteksi Cronobacter spp.(E. sakazakii)
Pengujian patogen secara konvensional biasanya memerlukan 4 tahap yakni
(1) preenrichment untuk memperkaya atau mengkondisikan bakteri patogen
terutama bila bakteri dalam keadaan injured atau jumlahnya sangat rendah, (2) selective enrichment, dimana diberikan senyawa tertentu atau suatu kondisi
yang menghambat bakteri patogen lain selain yang diinginkan , (3) isolasi yakni pemupukan pada medium tertentu untuk mendapatkan koloni tipikal atau benar-benar murni bakteri yang diinginkan, dan (4) identifikasi yang dilanjutkan dengan konfirmasi yakni dengan uji-uji biokimia, serologi, immunoassay ataupun DNA untuk memastikan jenis patogen tersebut (BAM 2001).
Berbagai metode isolasi dan identifikasi Cronobacter telah dikembangkan sebagai respon atas berbagai kasus kontaminasi dan infeksi akibat bakteri ini. FDA (2002) dalam publikasinya telah mengembangkan media serta pereaksi yang digunakan dalam isolasi C. sakazakii pada susu formula. Metode ini terdiri atas beberapa tahapan yaitu :
adanya kombinasi Brilliant Green dan bile salts. EE broth terdiri atas glukosa yang mampu meningkatkan pertumbuhan bakteri Enterobacteriaceae. Isolat yang ditumbuhkan pada EE broth dapat mengubah warna media dari hijau bening menjadi keruh.
Tahapan selanjutnya adalah plating dengan cara penggoresan pada Violet Red Bile Glucose (VRBG) Agar kemudian diinkubasi selama 24 jam pada 36 ºC untuk menghambat bakteri Gram positif seperti Lactobacillus karena adanya kombinasi kristal violet dan bile salts. Pertumbuhan yang terjadi pada VRBG dapat dilihat dari pembentukan koloni yang menggumpal yang berwarna ungu bergradasi ke oranye atau kuning atau kecoklatan. Koloni yang menggumpal menandakan terjadinya presipitasi garam empedu yang merupakan bahan selektif yang ditambahkan di dalam VRBG.
Koloni positif yang tumbuh pada VRBG kemudian diseleksi kembali pada media Tryptone Soy Agar (TSA) dan diinkubasi selama 48 hingga 72 jam pada 25 ºC. Koloni positif E. sakazakii pada TSA yang berpigmen kuning kemudian dikonfirmasi dengan menggunakan sistem identifikasi biokimia, API 20E, yang memerlukan tambahan waktu selama 18 hingga 24 jam.
berfungsi sebagai penanda selektif dalam membedakan Enterobacteriaceae
(Citrobacter, Salmonella, Edwardsiella dan Proteus) penghasil H2S.
Media kromogenikselektif untuk mendeteksi keberadaan C. sakazakii pada susu formula yaitu Druggan-Forsythe-Iversen (DFI) agar kemudian dikembangkan oleh Iversen et al. (2004b). Bahan selektif yang terdapat pada media tersebut adalah suatu senyawa chromogen 4-chloro-3-indolyl-α
,D-glucopyranoside (XαGlc). Senyawa tersebut akan berikatan dengan enzim α -glukosidase pada C.sakazakii dan membentuk koloni berwarna hijau-biru. Selain itu di media ini terdapat sodium desoxycholate yang bersama-sama dengan sodium thiosulphate dan ferric ammonium citrate yang bertindak sebagai senyawa selektif. Iversen dan Forsythe (2004b) menyatakan bahwa media chromogenic
yang ditemukannya memiliki beberapa keunggulan dibandingkan metode konvensional yang dikemukakan oleh FDA antara lain dengan menggunakan media DFI maka hasil isolasi dapat diperoleh dua hari lebih awal dibandingkan dengan metode konvensional; selain itu, media DFI lebih selektif dan sensitif dalam mendeteksi keberadaan C. sakazakii dibandingkan dengan metode konvensional (metode FDA).
Media kromogenik R&F Enterobacter sakazakii Chromogenic Plating Medium (ESPM) untuk isolasi koloni presumtif C. sakazakii dari makanan maupun lingkungan dikembangkan oleh Restaino et al. (2006). Media ini terdiri dari dua substrat kromogenik yaitu 5-bromo-4-chloro-3-indoxyl-α
-D-glucopyranoside dan 5-bromo-4-chloro-3-indoxyl-β-D-cellobioside. Penggunaan medium ESPM menghasilkan empat reaksi yaitu: (1) organisme yang menghidrolisis satu atau dua substrat kromogenik (indoxyl) membentuk water-insoluble halogenated indole membentuk koloni berwarna hitam atau biru-keabu-abuan (seperti C. sakazakii), (2) organisme yang menghidrolisis satu atau dua substrat kromogenik ditambah satu atau dua karbohidrat membentuk koloni berwarna hijau (seperti Klabsiella dan galur-galur E. coli), (3) organisme yang tidak menghidrolisis substrat kromogenik tetapi mampu memfermentasi satu atau lebih karbohidrat membentuk koloni berwarna putih kekuningan (seperti
Media ESPM juga terdiri dari tiga gula (sorbitol, D-arabitol dan adonitol), indikator pH dan beberapa penghambat (bile salt, vancomycin dan cefsulodin). Restaino et al. (2006) menggunakan metode ESPM dan ESSM (Enterobacter
sakazakii Chromogenic screening medium) secara bersama-sama untuk
meningkatkan sensitifitas dan lebih spesifik.
Perbandingan dua media kromogenik yaitu DFI agar dan Enterobacter sakazakii Isolation Agar (ESIA) untuk mendeteksi C. sakazakii di lingkungan dilakukan oleh Lehner et al. (2006). Berdasarkan hasil yang diperoleh pada saat menguji 98 koloni target (positif C. sakazakii) dan non-target diperoleh media ESIA mampu menunjukkan hasil yang lebih baik jika dibandingkan media DFI agar karena dapat mengidentifikasi seluruh koloni target dengan terbentuknya warna turquoise pada media ESIA sedangkan pada media DFI ada satu koloni target yaitu galur FSM 322 memberikan hasil negatif yaitu membentuk warna putih-keabu-abuan. Perbandingan performa media ESIA dan DFI agar pada 12 isolat dari fruit powder yang telah melalui tahapan pengkayaan menggunakan mLST menunjukkan bahwa DFI agar mampu memberikan hasil positif (warna biru-hijau) lebih banyak (11 isolat) jika dibandingkan dengan media ESIA (8 isolat). Identifikasi secara molekuler kemudian dilakukan dan membuktikan bahwa baik media DFI agar maupun ESIA mampu mendeteksi C. sakazakii
karena memanfaatkan aktivitas α-glukosidase yang terdapat pada bakteri tersebut. Medium diferensiasi ‘Cronobacter’ screening broth (CSB) sebagai media agar pelengkap berdasarkan hidrolisis substrat kromogenik α-glucopyranoside
dikembangkan oleh Iversen et al. (2008b). Media CSB mampu mendeteksi seluruh galur Cronobacter dengan baik, memiliki sensitifitas dan diferensiasi lebih baik jika dibandingkan dengan media pengkayaan selektif lainnya seperti mLST. Karena sensitifitasnya 100% terhadap semua galur Cronobacter sehingga
metode ini perlu menggunakan media kromogenik yang lebih spesifik terhadap
C. sakazakii.
substrat dan bufer, dan 1 cupule berfungsi sebagai kontrol negatif. Strip tes dapat
digunakan untuk identifikasi fosfatase alkali, esterase butirat, esterase lipase-caprylate, lipase miristat, leusin arylamidase, valin arylamidase,
arylamidase sistin, tripsin, chymotrypsin, osfatase asam, phosphoamidase, α-galaktosidase, β-galaktosidase, β-glukuronidase, α-glukosidase, β-glukosidase,
N-asetil-β-glukosaminidase, α-mannosidase, dan aktivitas α-fucosidase. Setelah inkubasi dalam keadaan aerob pada suhu 36 °C selama 24 jam, adanya tingkat aktivitas enzimatik dinilai sebagai intensitas warna 0-5 sesuai dengan grafik perbandingan warna disediakan oleh pabrik (Erickson dan Kornacki 2002).
Hasil uji biokimia dan gen 16S rRNA untuk mengidentifikasi karakteristik biokimia dan sekuens nukleotida penting dari C. sakazakii dengan menggunakan
Artificial Neural Networks (ANNs) dilakukan oleh Iversen et al. (2006). Pemodelan berdasarkan ANNs dengan metode komputasi dikembangkan untuk mengidentifikasi kunci utama bagi pengujian secara biokimia dan mampu menentukan bagian penting dari sekuen DNA sehingga mampu membedakan C. sakazakii dari galur-galur lain yang memiliki kekerabatan dekat. Hasil yang diperoleh dari penelitian ini adalah perbedaan antara C. sakazakii dengan galur-galur yang lain adalah aktivitas dari α-glukosidase dengan perkiraan nilai persentase yaitu 98.7% untuk data sekuen gen 16S rRNA dan 100 % untuk data fenotipik.
Identifikasi keberadaan α-glukosidase secara genotipik dengan metode
Random Amplified Polymorphic (RAPD-PCR) dan Enterobacterial Repetitive
Intergenic Consensus (ERIC-PCR) dilakukan oleh Ye et al. (2010). Hasil yang ditunjukkan dari penelitian tersebut adalah ERIC-PCR lebih mampu mengidentifikasi C. sakazakii dibandingkan dengan RAPD-PCR karena memperlihatkan korelasi yang sama dengan pengujian API 20E maupun identifikasi berdasarkan ketahanan terhadap antibiotik.
pasang primer ini mengikat conserved region (E. coli pada posisi 88 – 107 (Esakf) dan 1017 – 998 (Esakr)) pada sekuen gen 16S rRNA dengan ukuran amplikon 929 bp. Sistem PCR yang dikembangkan oleh Lehner et al. (2004) menunjukkan adanya pohon filogenetik kedua yang berbeda di antara spesies C. sakazakii.
Beberapa isolat dari Korea diidentifikasi oleh Kim et al. (2011) ke dalam 11 biogrup berdasarkan karakterisasi biokimia dan 3 genomic group hasil analisis gen 16S rRNA. Analisis genotipik menggunakan sepasang primer yaitu P0 (AGA GTT TGA TCC TGG CTC AG) dan P6 (GTA CGG CTA CCT TGT TAC GA). Identifikasi genotipik menunjukkan bahwa biogrup Cronobacter spp. merupakan bakteri patogen.
Karakterisasi gen penyandi 16S rRNA C. sakazakii dilakukan oleh Hassan et al. (2007), kemudian mengembangkan metode PCR yang spesifik terhadap spesies C. sakazakii asal susu formula maupun ATCC 29544. Dua metode PCR untuk identifikasi E. sakazakii dikembangkan berdasarkan perbedaan sekuen pada hypervariable region V1, V2, dan V3 antara E. sakazakii dan
Enterobacteriaceae lainnya. Metode PCR1 menggunakan sepasang primer yang berlokasi pada V1/V2, sementara metode PCR2 menggunakan sepasang primer yang berlokasi pada V1/V3. PCR1 dengan amplikon berukuran 406 bp menunjukkan hasil 100 % positif terhadap E. sakazakii, dan juga mendeteksi
Citrobacter koseri/amalonaticus dan Salmonella enterica. Sebaliknya, PCR2
dengan amplikon sebesar 952 bp memberikan hasil yang positif hanya untuk
E. sakazakii; sehingga sangat memungkinkan untuk identifikasi yang spesifik terhadap E. sakazakii. Selain itu, Hassan et al. (2007) melaporkan adanya sistem PCR komersial (BAX®, Oxoid) untuk identifikasi C. sakazakii. Namun demikian, system ini belum diidentifikasi dan dideskripsikan secara detil atau telah diujikan sepenuhnya.
Tiga media kromogenik yaitu α-MUG, DFI dan ESPM dibandingkan oleh Jaradat et al. (2009). Media α-MUG (4-metil-umbelliferyl-α-D-glukosida) merupakan media nutrisi bagi Cronobacter spp. yang telah diidentifikasi secara biokimia dengan API 20E dan membentuk koloni berwarna kuning saat diamati dibawah sinar UV sedangkan ESPM merupakan media kromogenik bagi
dikemukakan oleh Restaino et al. (2006). Berdasarkan hasil penelitian Jaradat et al. (2009), media kromogenik α-MUG dan DFI menunjukkan performa yang lebih baik jika dibandingkan dengan EsPM karena media tersebut tidak dapat mendeteksi 3 koloni positif C. sakazakii dan medeteksi 7 koloni yang bukan C. sakazakii sebagai hasil positif.
Studi mengenai karakterisasi molekular Cronobacter spp. (E. sakazakii) telah banyak dipublikasikan baik untuk menjelaskan keragaman filogenetik maupun untuk identifikasi primer PCR yang tepat. Iversen et al. 2004c membandingkan metode-metode karakterisasi biokimia C. sakazakii standar (API 20E dan ID32 E) dengan hasil teknik sekuensing dari sekuen parsial gen 16S rRNA (Iversen et al. 2004a). Iversen et al. (2004d) juga mempelajari hubungan filogenetik antara galur-galur C. sakazakii berdasarkan sekuen parsial gen 16S rRNA dan hsp 60. Hasil sekuensing parsial kedua gen tersebut menunjukkan bahwa isolat yang teridentifikasi sebagai C. sakazakii dengan menggunakan kit
komersial membentuk sedikitnya empat klaster yang berbeda. Selanjutnya juga dijelaskan bahwa berdasarkan perbandingan sekuen 16S rDNA-nya, galur C. sakazakii mempunyai kemiripan yang lebih dekat dengan C. koseri (97.8% kemiripan) dibandingkan dengan spesies Enterobacter lainnya, yakni E. cloacae
(97%) dan C. freundii (96%).
Uji konfirmasi secara genetika oleh Gitapratiwi (2011), dengan menganalisis sekuen gen 16S rRNA menggunakan pasangan primer yang dikemukakan oleh Hassan et al. (2007) yaitu 16SUNI-L dan Saka-2b untuk segmen 1 dan ESA-1 dan 16SUNI-R untuk segmen 2 terhadap isolat yang diduga
C. sakazakii menyimpulkan bahwa semua isolat lokal mempunyai kemiripan
dengan C. sakazakii. Meskipun berdasarkan karakterisasi biokimia dengan API
20E hanya dua isolat yaitu DES b7a dan DES b10 yang memiliki kemiripan
C. sakazakii sementara empat isolat lainnya teridentifikasi ke dalam famili Enterobacteriaceae lainnya.
Keragaman Genetika Cronobacter sakazakii
Isolasi DNA memiliki beberapa tahapan, yaitu: (1) isolasi sel, (2) lisis
(5) presipitasi. Prinsip-prinsip dalam melakukan isolasi DNA ada 2, yaitu sentrifugasi dan presipitasi. Prinsip utama sentrifugasi adalah memisahkan substansi berdasarkan berat jenis molekul dengan cara memberikan gaya sentrifugal sehingga substansi yang lebih berat akan berada di dasar, sedangkan substansi yang lebih ringan akan terletak di atas. Prinsip dasar isolasi total DNA/RNA dari jaringan adalah dengan memecah dan mengekstraksi jaringan tersebut sehingga akan terbentuk ekstrak sel yang terdiri atas sel-sel jaringan, DNA, dan RNA. Kemudian ekstrak sel dipurifikasi sehingga dihasilkan pelet sel yang mengandung DNA/RNA total.
Prinsip yang harus dipenuhi dalam pengumpulan sampel untuk suatu penelitian molekuler adalah menjaga keadaan sampel, sehingga molekul DNA atau RNA-nya maupun enzimnya tidak mengalami kerusakan. Kerusakan-kerusakan tersebut dapat disebabkan oleh mikroorganisme ataupun oleh bahan pengawet yang digunakan selama proses penyimpanan sebelum dilakukan analisis di laboratorium. Bahan material segar merupakan bahan yang paling mudah diekstraksi karena DNA-nya belum mengalami kerusakan (Ubaidillah dan Sutrisno 2009). Kelembapan dan sinar Ultra Violet juga dilaporkan sebagai salah satu faktor yang menurunkan kualitas DNA (Roon et al. 2003).
Pendekatan yang banyak dipakai dalam analisis sekuen gen adalah 16S rRNA atau 23S rRNA. Gen penyandi 16S rRNA merupakan gen yang bersifat spesifik terhadap sel prokariotik. Bila 16S rRNA diisolasi dalam bentuk murni kemudian dicampur dalam urutan spesifik yang benar pada suhu yang sesuai, maka molekul ini akan secara spontan menyusun diri kembali membentuk subunit 30S yang identik dalam struktur dan aktivitasnya dengan subunit yang asli (Lehninger 1992).
Farmer et al. (1980) melaporkan bahwa tidak ada petunjuk generik yang jelas untuk Cronobacter spp. (E. sakazakii). Berdasarkan studi hibridisasi DNA-DNA, disimpulkan bahwa bakteri ini mempunyai 53-54% homologi dengan spesies Enterobacter dan Citrobacter. Perbandingan antara galur dari dua genera
ini menunjukkan bahwa C. sakazakii mempunyai 41% kemiripan dengan
filogeni C. sakazakii yang dilakukan berdasarkan studi heterogenitas taksonomi menggunakan 16S rRNA memberikan hasil bahwa C. sakazakii mempunyai 97.8% kemiripan dengan C. koseri dan 97% kemiripan dengan E. cloacae. Namun demikian, diperlukan studi lebih lanjut untuk memperjelas hubungan kekerabatan tersebut (Iversen et al. 2004d).
Panjang gen 16S rRNA menurut Clarridge III (2004) adalah 1500 bp yang terdiri dari bagian variable dan conserved. Gen penyandi 16S rRNA merupakan gen yang sangat bagus untuk mengindentifikasi keragaman genetika dari suatu organisme. Hal ini disebabkan oleh (i) gen 16S rRNA yang terdapat pada semua bakteri, (ii) gen 16S rRNA mempunyai fungsi yang tidak berubah sepanjang waktu, dan (iii) gen 16S rRNA (1500 bp) mempunyai ukuran dengan panjang yang mencukupi untuk tujuan informatika (Janda dan Abbot 2007). Analisis genotipik dengan menggunakan gen 16S rRNA jauh lebih mudah jika dibandingkan dengan DNA-DNA hibridisasi.
Hasil penelitian berdasarkan analsisis sekuen 16S rRNA telah menempatkan berbagai isolat E. sakazakii yang diperoleh di dunia ke dalam genus baru
Cronobacter spp. dan membagi ke-16 bio-groupCronobacter spp. ke dalam enam kelompok klaster yakni Cronobacter sakazakii, C. malonaticus, C. turicensis, C. muytjensii, C. dublinensis dan C. genomospecies 1 (Iversen et al. 2007). Saat ini Codex menggunakan nama C. sakazakii dalam panduan-panduannya bagi industri pangan (CAC 2008).
Hasil analisis keragaman genetika C. sakazakii oleh Gitapratiwi et al.
(2012) yang diisolasi dari makanan bayi dan makanan kering lainnya mempunyai tingkat kemiripan yang bervariasi dengan berbagai galur C. sakazakii yang ada di luar Indonesia maupun isolat-isolat lokal berdasarkan sekuen parsial gen 16S rRNA. Berdasarkan pohon filogenetik, semua isolat lokal yang dianalisis berada dalam satu klaster dengan C. sakazakii ATCC 29544 dan C. malonaticus galur E852. Selain itu disimpulkan bahwa antar isolat lokal memiliki kedekatan kekerabatan genetika satu sama lain meskipun berasal dari sumber isolasi yang berbeda. Hasil penelitian tersebut belum dapat menggolongkan isolat-isolat lokal
Cronobacter spp. secara tepat ke dalam lima klaster yang sesuai dengan
Beberapa program komputer dan database untuk bioinformatika yang dapat digunakan secara on line dari internet antara lain: GeneMark, NCBI (National Center for Biotechnology Information), Expasy, dan lain-lain. Pada NCBI dapat diakses program PubMed, Entrez, BLAST, Blankit, OMIM, Taxonomy, dan penelusuran struktur. Salah satu program yang umum digunakan adalah BLAST (Basic Local Alignment Search Tool) yang merupakan program untuk pencarian kesamaan yang dirancang dalam mengeksplorasi semua database sekuen yang diminta, baik berupa DNA ataupun protein. Program BLAST juga dapat digunakan untuk mendeteksi hubungan antar sekuen yang hanya berbagi daerah tertentu yang memilki kesamaan (Claverie dan Notredame 2007).
Ada beberapa variasi BLAST yang masing-masing dibedakan dari tipe sekuen (DNA atau protein) yang dicari dengan sekuen pada database. Berikut ini beberapa jenis program BLAST (Claverie dan Notredame, 2007):
BLASTP : membandingkan sekuen asam amino dengan sekuen protein dalam
database
BLASTN : membandingkan sekuen nukleotida dengan sekuen nukleotida dalam database
BLASTX : membandingkan sekuen nukleotida yang ditranslasi pada seluruh ORF (open reading frame) dengan sekuen protein database
Hasil BLAST meliputi tiga bagian yang berbeda yakni grafik yang menunjukkan bagaimana porsi similaritas sekuen yang dibandingkan, daftar hits yang berisi nama sekuen yang serupa dengan yang dicari urut berdasarkan similaritas dan penjajaran yang ada pada database (Claverie dan Notredame 2007).
Analisis Neighbor-Joining
Analisis Neighbor-Joining merupakan salah satu media pengelompokan dalam bioinformatika yang dapat membentuk suatu pohon filogeni atau dendogram. Neighbor-Joining biasanya digunakan pada pohon filogeni yang berdasarkan pada data sekuen DNA atau protein. Metode ini menggunakan algoritma yang membutuhkan pengetahuan mengenai jarak antara tiap pasang taxa (spesies atau sekuen) pada pohon filogeni.
Keunggulan metode Neighbor-Joining terhadap metode lainnya adalah dari efisiensinya secara komputasional. Neighbor-Joining merupakan algoritma yang bersifat polynomial-time. Metode ini dapat digunakan pada set data yang banyak untuk melakukan analisis filogenetik (evolusi minimum, penghematan dan kemiripan maksimum) tanpa melakukan banyak perhitungan. Metode ini juga bersifat konsisten secara statistik di bawah berbagai model evolusi. Dengan metode ini data yang diberikan pada jumlah yang cukup dapat membentuk suatu pohon filogeni dengan kekerabatan yang besar (Saitou dan Nei 1987).
YR w1 dan YR w3 yang mempunyai nilai bootstrap 97% pada cabang pohon filogenetiknya. Kedua isolat lokal tersebut diisolasi dari sampel produk makanan bayi yang berasal dari manufaktur yang sama.
METODE PENELITIAN
Tempat dan Waktu
Penelitian dilakukan di Laboratorium Mikrobiologi dan Bioteknologi Pangan Southeast Asian Food and Agricultural Science and Technology
(SEAFAST) Center, Institut Pertanian Bogor mulai bulan Mei 2011 sampai Februari 2012.
Bakteri Uji
Bakteri yang digunakan dalam penelitian ini adalah isolat-isolat bakteri
C.sakazakii hasil isolasi dari kegiatan penelitian ini. Sebagai pembanding digunakan C. muytjensii ATCC (American Type Culture Collection) 51329 (AY752937).
Bahan dan Media
Beberapa pangan kering yang digunakan untuk sumber isolasi yaitu (1) makanan bayi, (2) tepung beras, (3) tepung terigu, (4) tepung beras ketan, (5) gula halus, (6) pati jagung (maizena), (7) pati singkong (tapioka), dan (8) rempah-rempah kering. Sampel-sampel tersebut merupakan produk yang belum kadaluarsa dan diperoleh di supermarket maupun pasar tradisional.
Media untuk isolasi C.sakazakii yang akan digunakan antara lain BPW
(Bufered-Peptone Water) (CM 0509 Oxoid Ltd., UK), EE (Enterobacteria
Enrichment) broth (CM 0485 Oxoid Ltd., UK), VRBG (Violet Red Bile Glucose) Agar (CM 0485 Oxoid Ltd., UK), DFI (Druggan-Forsythe-Iversen) Agar (Oxoid, UK) danTSA (Tryptone Soy Agar) (CM 0131 Oxoid Ltd., UK).
Bahan yang digunakan untuk identifikasi fenotipik C.sakazakii yaitu NaCl 0.85%, akuabides steril, parafin cair steril dan API 20E Biochemical Strips
(bioMěrieux, Perancis).
Tris (hydroxymethyl)-amino methan (Tris) (Amersham Bioscience, Sweden) dan
Na2-EDTA (Dinatrium Ethylene Tetra Acetat) (Amersham Bioscience, Sweden),
PCI 25:24:1 (phenol, chloroform, isoamil alcohol), CI 24:1 (chloroform, isoamil alcohol), sodium asetat (Merck, Darmstadt, Germany), isopropanol (Merck, Darmstadt, Germany), etanol 70% dan HCl untuk pengaturan bufer pH. Bahan-bahan yang akan digunakan untuk analisis gel elektroforesis DNA antara lain bufer TAE (Tris-Asetat-EDTA), agarosa (SIGMA-ALDRICH CHEMIE, Steinheim, Germany), EtBr (Ethidium Bromide) (Amersham Bioscience, Sweden) dan akuabides steril.
Bahan-bahan yang digunakan untuk amplifikasi gen 16S-rRNA antara lain PCR Master Mix (Fermentas) yang terdiri atas 0.05 µ/μl Taq DNA polymerase, bufer PCR, MgCl2, dan dNTP (masing-masing 0.4 mM dATP, dCTP, dGTP dan
dTTP), akuabides steril, DNA cetakan (template) dan primer, sedangkan untuk analisis pada gel elektroforesis digunakan DNA ladder 1kb dan bufer TE pH8, bufer TAE (Tris-Asetat-EDTA), agarosa (SIGMA-ALDRICH CHEMIE, Steinheim, Germany), EtBr (Amersham Bioscience, Sweden) dan akuabides steril.
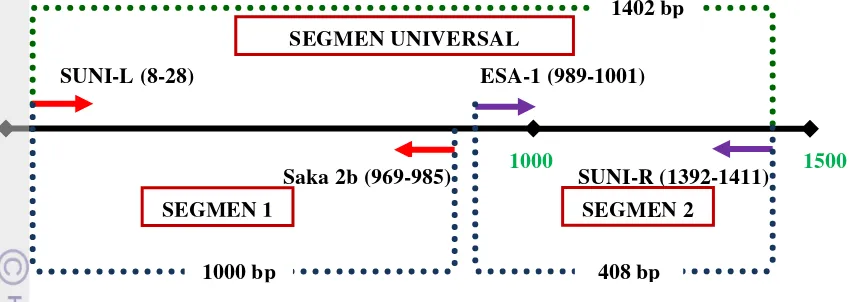
Primer yang digunakan merupakan primer gen 16S rRNA C. sakazakii
yaitu 16 SUNI-L dan Saka-2b, ESA-1 dan 16 SUNI-R dan sebagai pembanding menggunakan primer universal Escherichia coli yaitu 16 SUNI-L dan 16 SUNI-R (Tabel 1). Primer dipesan dari Alpha DNA (Notre-Dame St.W., Montreal, Quebec) dan PT Genetika Science (1st BASE Pte Ltd Singapore).
Tabel 2 Pasangan primer oligonukleotida yang digunakan untuk amplifikasi gen penyandi 16S rRNA C. sakazakii
Pasangan
Primer Sekuen Oligonukleotida
Posisi penempelan primer target
amplifikasi
Sumber 16 SUNI-L
Saka-2b
AGAGTTTGATCATGGCTCAG TCCCGCATCTCTGCAGGA
Segmen 1 (1000 bp)
Hassan et al.
2007 ESA-1
16 SUNI-R
AATCCTGCAGAGATGCG GTGTGACGGGCGGTGTGTAC
Segmen 2 (408 bp)
Hassan et al.
2007 16 SUNI-L
16 SUNI-R
AGAGTTTGATCATGGCTCAG GTGTGACGGGCGGTGTGTAC
Segmen Universal (1402bp)
Gambar 2 Peta amplifikasi fragmen gen 16S rRNA Cronobacter spp. (Enterobacter sakazakii) dengan menggunakan pasangan primer 16 SUNI-L dan Saka-2b, ESA-1 dan 16 SUNI-R, 16 SUNI-L dan 16 SUNI-R
Alat
Alat-alat yang digunakan antara lain waterbath, termometer, inkubator, pipet volumetrik berukuran 10 ml dan 25 ml, pipet mikro beserta tip berukuran 1 ml, 0.1 ml dan 2-20 μl, batang gelas pengaduk, sudip, jarum ose, labu takar berukuran 50 ml dan 1000 ml, erlenmeyer berukuran 100 ml, 125 ml, 250 ml, dan 500 ml, botol Schott Duran berukuran 100 ml, 250 ml, 500 ml dan 1000 ml, tabung reaksi bertutup, gelas ukur, cawan petri, plastik steril, pH meter, penangas air (hot plate), sentrifugasi dengan kekuatan 18000 rpm, pengaduk magnet (magnetic stirrer), oven, vortex, bunsen, timbangan elektrik, aluminium foil, perangkat elektroforesis (Bio-Rad), perangkat PCR Applied Biosystem 2720
Thermal Cycler (Foster City, California), Geldoc XR (Bio-Rad) dan
Spektrofotometer UV-Vis (Shimadzu).
Perangkat Lunak (Software)
Beberapa software yang digunakan dalam penelitian ini yaitu apiwebTM untuk menganalisis hasil uji biokimia dengan API 20E, Sequence Scanner v1.0 untuk melihat kualitas nukleotida yang dihasilkan, DNA Baser v3 dan BioEdit untuk melakukan contig sekuen, program BLAST (Basic Local Alligment Search Tool) dari situs NCBI (www.ncbi.nih.nlm.gov) untuk menganalisis hasil sekuensing serta program MEGA4 (www.megasoftware.net) untuk membentuk pohon filogeni.
Pelaksanaan Penelitian SUNI-L (8-28)
Saka 2b (969-985) SUNI-R (1392-1411)
ESA-1 (989-1001)
1000 bp 408 bp
1 1000 1500
SEGMEN 1 SEGMEN 2
SEGMEN UNIVERSAL
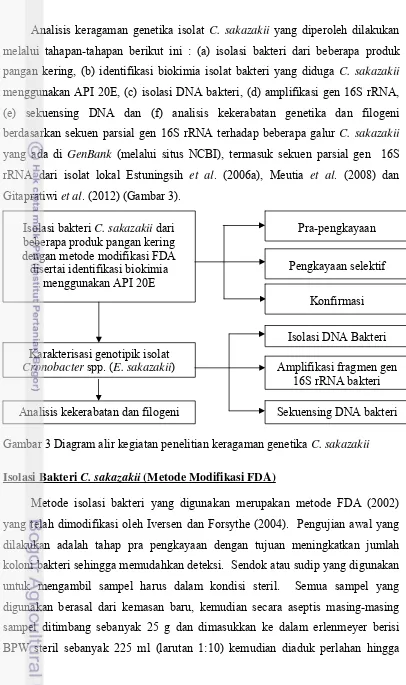
Analisis keragaman genetika isolat C. sakazakii yang diperoleh dilakukan melalui tahapan-tahapan berikut ini : (a) isolasi bakteri dari beberapa produk pangan kering, (b) identifikasi biokimia isolat bakteri yang diduga C. sakazakii
menggunakan API 20E, (c) isolasi DNA bakteri, (d) amplifikasi gen 16S rRNA, (e) sekuensing DNA dan (f) analisis kekerabatan genetika dan filogeni berdasarkan sekuen parsial gen 16S rRNA terhadap beberapa galur C. sakazakii
yang ada di GenBank (melalui situs NCBI), termasuk sekuen parsial gen 16S rRNA dari isolat lokal Estuningsih et al. (2006a), Meutia et al. (2008) dan Gitapratiwi et al. (2012) (Gambar 3).
Gambar 3 Diagram alir kegiatan penelitian keragaman genetika C. sakazakii
Isolasi Bakteri C. sakazakii (Metode Modifikasi FDA)
Metode isolasi bakteri yang digunakan merupakan metode FDA (2002) yang telah dimodifikasi oleh Iversen dan Forsythe (2004). Pengujian awal yang dilakukan adalah tahap pra pengkayaan dengan tujuan meningkatkan jumlah koloni bakteri sehingga memudahkan deteksi. Sendok atau sudip yang digunakan untuk mengambil sampel harus dalam kondisi steril. Semua sampel yang digunakan berasal dari kemasan baru, kemudian secara aseptis masing-masing sampel ditimbang sebanyak 25 g dan dimasukkan ke dalam erlenmeyer berisi BPW steril sebanyak 225 ml (larutan 1:10) kemudian diaduk perlahan hingga
Isolasi bakteri C. sakazakii dari beberapa produk pangan kering dengan metode modifikasi FDA disertai identifikasi biokimia
menggunakan API 20E
Pra-pengkayaan
Pengkayaan selektif Konfirmasi
Karakterisasi genotipik isolat
Cronobacter spp.(E. sakazakii)
Analisis kekerabatan dan filogeni
Isolasi DNA Bakteri Amplifikasi fragmen gen
bubuk tersuspensi secara seragam, lalu dipanaskan hingga 45 °C. Suspensi tersebut diinkubasi pada suhu 37 °C selama 24 jam.
Masing-masing suspensi yang telah diinkubasi diambil sebanyak 10 ml kemudian dimasukkan ke dalam 90 ml EE broth dan diinkubasi lagi pada suhu 37 °C selama 24 jam. Isolat pada EE broth yang telah diinkubasi diambil sebanyak 1 ose, lalu digoreskan sedikitnya tiga kuadran goresan dengan Direct
Streaking Method pada VRBG sehingga mendapatkan koloni terpisah.
Penggoresan untuk masing-masing isolat dilakukan secara duplo. Masing-masing cawan kemudian diinkubasikan pada 37 °C selama 24 jam. Cawan yang positif mengandung Cronobacter spp. (E. sakazakii) pada VRBG memperlihatkan koloni berbentuk gumpalan garam bile berwarna ungu.
Koloni yang muncul (tipikal dan non tipikal) pada VRBG kemudian diisolasi dengan cara digoreskan pada agar cawan DFI, dan kemudian diinkubasi pada suhu 37 °C selama 24 jam. Koloni tipikal pada DFI yang berwarna hijau kebiruan digoreskan kembali pada media TSA dan diinkubasi pada 37 °C selama 48-72 jam.
Masing-masing isolat C. sakazakii yang telah diperoleh, sebagian diuji lebih lanjut dan sebagian lainnya disimpan dalam bentuk kultur stok dengan menambahkan gliserol dan disimpan di suhu rendah -20 oC dalam bentuk manik-manik. Morfologi isolat C.sakazakii tersebut juga diamati di bawah mikroskop melalui proses pewarnaan Gram untuk memastikan bahwa isolat yang diperoleh sudah homogen. Pengamatan mikroskopik dilakukan dengan pembesaran 1000x.
Identifikasi Isolat Secara Biokimia
dapat diidentifikasi dengan menggunakan reagen. Beberapa sumur [citrate, (CIT), Voges-Proskauer (VP) dan gelatin (GEL)] diisi dengan suspensi hingga penuh, sementara beberapa sumur lainnya [arginine dihydrolase (ADH), lysine decarboxylase (LDC), ornithine decarboxylase (ODC), hydrogen peroksidase
(H2S) dan urea (URE)] ditutup dengan mineral oil, dalam hal ini digunakan
parafin cair untuk m enciptakan suasana anaerobik.
Setelah diinkubasi dalam wadah yang telah diberi suasana lembab selama 18-24 jam pada suhu 37 °C, dilakukan pembacaan terhadap reaksi-reaksi warna pada masing-masing sumur menggunakan kriteria sifat biokimia Nazarowec dan Farber (Tabel 2). Hasil pada beberapa sumur [tryptophane deaminase (TDA),
indole (IND) dan voges proskauer (VP)] dibaca setelah ditambahkan reagen terlebih dahulu. Hasil pembacaan kemudian diinterpretasikan dengan menggunakan software apiwebTM sehingga dapat dihitung persentase kemiripan isolat yang memiliki koloni tipikal pada TSA dengan C.sakazakii secara biokimiawi.
Beberapa uji biokimia pada perangkat API 20E antara lain dengan asam amino dekarboksilasi (arginine dehydrolase hingga ornithine decarboxylase) dan fermentasi karbohidrat (glukosa hingga arabinosa). Asam amino yang akan diuji antara lain arginin, lisin dan ornithin. Reaksi dekarboksilasi dapat dilihat melalui reaksi basa (warna merah pada indikator pH yang digunakan). Karbohidrat-karbohidrat yang akan diuji antara lain glukosa, mannitol, inositol, sorbitol, rhamnosa, sukrosa, melibiosa, amygladin dan arabinosa. Fermentasi ditunjukkan dengan reaksi asam (warna kuning pada indikator). Produksi hidrogen peroksidase (H2S) dan hidrolisis gelatin (GEL) terlihat dari terbentuknya warna
hitam di seluruh sumur. Reaksi positif untuk triptopan deaminase (TDA) ditunjukkan dengan warna coklat kemerahan bila ditambahkan perak klorida, dimana hasil yang positif berkorelasi dengan reaksi lisin dan fenilalanin deaminase yang merupakan karakteristik dari Proteus, Morganella dan
Providecia (Lindquist, 2001). Untuk dapat memunculkan angka persentase
kemiripan, analisis tambahan seperti uji oksidase (OX), reaksi terhadap NO2, N2,