IDENTIFIKASI KERAGAMAN GENETIK KLON KARET
DENGAN TEKNIK
AMPLIFIED FRAGMENT LENGTH
POLYMORPHISM
(AFLP)
JUNIZA FIRDHA SUPARNINGTYAS
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Identifikasi Keragaman Genetik Klon Karet dengan Teknik Amplified Fragment Length Polymorphism
(AFLP) adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Januari 2015
Juniza Firdha Suparningtyas
ABSTRAK
JUNIZA FIRDHA SUPARNINGTYAS. Identifikasi Keragaman Genetik Klon Karet dengan Teknik Amplified Fragment Length Polymorphism (AFLP).
Dibimbing oleh SURYANI dan TEUKU TAJUDDIN.
Tanaman karet di Indonesia telah banyak dikembangkan dalam bentuk klon-klon unggul. Proses pemuliaan dengan mengidentifikasi keragaman genetik secara molekuler, salah satunya dengan teknik AFLP, memiliki tingkat polimorfisme dan pengulangan yang tinggi. Tujuan penelitian ini adalah untuk mengidentifikasi keragaman genetik klon karet. Sebanyak 3 klon karet, IRR 104, RRIM 600, dan PR 300 masing-masing diisolasi DNA genomnya, kemudian direstriksi menggunakan dua enzim restriksi, diligasikan terhadap adapter, dilakukan pre-amplifikasi dan dipre-amplifikasi menggunakan 64 kombinasi pasangan primer serta analisis hasil elektroforesis dari elektroferogram gel poliakrilamid 6 %. Sebanyak 1023 pita AFLP telah dihasilkan. Keseluruhan pita polimorfis diubah menjadi dendrogram dengan kesamaan genetik ketiganya sebesar 29 %. Selanjutnya garis dendrogram terpisah menjadi dua kelompok, kelompok 1 terdiri dari klon IRR 104 dan RRIM 600 dengan kesamaan genetik sebesar 43 %. Kelompok 2 yang hanya terdiri dari klon PR 300. Informasi dendrogram yang diperoleh telah menunjukkan keragaman genetik ketiga klon karet yang bersifat polimorfis.
Kata kunci: klon karet, AFLP, polimorfisme, keragaman genetik
ABSTRACT
JUNIZA FIRDHA SUPARNINGTYAS. Genetic Diversity Identification of Rubber Tree Clones by Amplified Fragment Length Polymorphism (AFLP) Technique. Supervised by SURYANI and TEUKU TAJUDDIN.
Rubber tree in Indonesian has been developed into many clones. To make a stock breeding by identify the genetic diversity in a molecular way, the one is AFLP, have a higher level of polymorphisme and reproduciblity. The objective of this research is to identify the genetic diversity of rubber tree clones. The DNA genome of three clones contain IRR 104, RRIM 600, and PR 300 were extracted, and then restricted with two restriction enzymes, ligated into adapter, pre-amplificated, then amplificated with 64 primers combination, and analyzed the electropherogram of 6 % gel polyacrilamid. For about 1023 AFLP fragments had been detected. All of the polymorphic fragments constructed into dendrogram among the three clones reached 29 % of genetic similarity level. The dendrogram lane separated into two group, the first group contained IRR 104 and RRIM 600 showing 43 % of genetic similarity. The second group only contained PR 300. This dendrogram information had been showed the polymorphism of the genetic diversity among this three clones.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains pada
Departemen Biokimia
IDENTIFIKASI KERAGAMAN GENETIK KLON KARET DENGAN TEKNIK AMPLIFIED FRAGMENT LENGTH POLYMORPHISM (AFLP)
JUNIZA FIRDHA SUPARNINGTYAS
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas
segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Februari 2014 sampai September 2014 ini ialah penanda molekuler, dengan judul Identifikasi Keragaman Genetik Klon Karet dengan Teknik Amplified Fragment Length Polymorphism (AFLP).
Terima kasih penulis ucapkan kepada Ibu Dr Suryani, SP, MSc dan Bapak Dr Ir Teuku Tajuddin, MSc selaku pembimbing, serta Bapak Devit Purwoko yang telah banyak membantu penulis di laboratorium sehingga penelitian dan penulisan karya ilmiah ini dapat terlaksana dengan baik. Ibu Rahma, Bapak Imam C Cartealy, Mbak Leha, Ibu Anna, dan Ibu Syofi yang telah mengizinkan penulis menggunakan fasilitas di laboratorium Teknologi Gen. Ungkapan terima kasih terutama juga disampaikan kepada ayah, ibu, adik, serta seluruh keluarga besar atas segala doa, dukungan moril dan material serta kasih sayangnya. Selanjutnya kepada seluruh rekan laboratorium Kak Okky, Kak Hamisya, Kak Angel, Fazri, Oho yang telah membantu berdiskusi di perpustakaan Biotek, serta terima kasih untuk Kementerian Lingkungan Hidup BEM KM IPB 2014 serta keluarga besar BEM KM IPB juga Pinus merkusii Biokimia 47 dan Ayumas IPB yang telah
memberikan dukungandan bantuan jasa lainnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Januari 2015
DAFTAR ISI
DAFTAR TABEL viii
DAFTAR GAMBAR viii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1
METODE 2
Waktu dan Tempat 2
Bahan 2
Alat 3
Prosedur Penelitian 3
HASIL 6
DNA Karet Hasil Isolasi 6
Produk PCR AFLP DNA Karet 6
Keragaman Genetik Klon Karet 8
Pembahasan 9
DNA Karet Hasil Isolasi 9
Produk PCR AFLP DNA Karet 11
Keragaman Genetik Klon Karet 13
SIMPULAN DAN SARAN 14
Simpulan 14
Saran 14
DAFTAR PUSTAKA 14
DAFTAR TABEL
1 Konsentrasi dan kemurnian DNA karet hasil isolasi 6 2 Pasangan primer spesifik dan ukuran spesifik yang dihasilkan pada
masing-masing klon 8
DAFTAR GAMBAR
1 Hasil elektroforesis DNA isolasi 6
2 Elektroforegram DNA genom hasil isolasi terhadap pemotongan
dengan enzim restriksi EcoRI dan MseI serta ligasi adapater 7
3 Elektroforegram DNA hasil digesti/ligasi setelah dilakukan
pre-amplifikasi 7
4 Pohon filogenetik 3 klon karet berdasarkan seluruh pola pita hasil
AFLP 9
DAFTAR LAMPIRAN
1 Diagram alir penelitian 17
2 Prinsip teknik Amplified Fragment Length Polymorphism (AFLP) 18
3 Pasangan Primer AFLP 19
4 Urutan primer pada gel poliakrilamid 1. 20
5 Urutan primer pada gel poliakrilamid 2. 21
6 Elektroferogram poliakrilamid gel 1 22
PENDAHULUAN
Informasi untuk mengetahui keragaman genotipe plasma nutfah tanaman karet merupakan modal dasar untuk keberhasilan pemuliaan dalam pengembangan tanaman unggul di lapang sebagai upaya meningkatkan kapasitas produksi lateks dan hasil kayu. Teknik pemuliaan tanaman dalam perolehan informasi bibit unggul yang lebih cepat sangat diperlukan mengingat teknik pemuliaan tanaman secara tradisional memerlukan waktu seleksi yang panjang yang dapat mencapai 20 hingga 25 tahun sejak persilangan hingga menghasilkan produk dengan area penanaman yang dibutuhkan luas (Novalia 2009). Para peneliti pun beralih menggunakan teknik penelitian molekuler, salah satunya dengan mencari urutan DNA polimorfismenya untuk dipelajari perbedaan genetik dan hubungan kekerabatan antara individu (Weising et al. 2005) sehingga dapat melengkapi
pengembangan dalam pemuliaan tanaman karet yang unggul dan potensial secara cepat. Penelitian secara molekuler ini hanya membutuhkan waktu pengujian dan proses pengolahan data secara cepat, jika telah dilakukan optimasi dan bank data genom tanaman karet.
Penelitian teknik menggunakan marka molekuler yang pernah dilakukan untuk tanaman karet antara lain Restriction Fragment Length Polymorphism
(RFLP) (Lespinasse et al. 2000), Random Amplified Polymorphic DNA (RAPD)
(Zulkifli 2001; Toruan-Marthius 2002 Nurhaimi- Haris et al. 2003; Wibowo
2010), marka isozim dan mikro satelit (Lespinase et al. 2000), Simple Sequence Repeat (SSR) (Lekawipat et al., 2003; Hamzah 2014). Namun demikian, beberapa
hasil yang diperoleh tidak konsisten, misalnya pada teknik RAPD, disebabkan rendahnya akurasi dalam pengulangan hasil amplifikasi, jumlah lokus yang dihasilkan lebih sedikit, jumlah fragmen DNA yang diperoleh dari primer yang digunakan lebih sedikit, dan tingkat diferensiasi dalam analisis dendrogram klon karet yang lebih rendah sehingga hasil setiap kelompok klon karet kurang terpisah dengan baik (Toruan-Marthius et al. 2002).
Teknik Amplified Fragment Length Polymorphism (AFLP) digunakan pada
tanaman karet yang sebelumnya juga pernah dilakukan (Luo et al. 2001; Zulkifli
2001; Nurhaimi-Haris 2003; Toruan-Marthius 2002; Nurhaimi 2006) karena memiliki tingkat pengulangan dengan derajat kekonsistenan hasil yang tinggi (Mueller dan Wolfenbarger 1999). Menurut Mueller dan Wolfenbarger (1999) dalam Damayanti (2012) marka Amplified Fragment Length Polymorphism
(AFLP) dapat mendeteksi variasi dan keragaman genetik antar populasi, individu, juga spesies berdasarkan perbedaan letak basa tertentu dari marka DNA dalam genom tanaman.
2
primer dengan tiga nukleotida selektif yang divisualisasikan menggunakan gel poliakrilamid 6%. Gel poliakrilamid 6% yang digunakan dalam visualisasi akhir hasil amplifikasi AFLP akan diperoleh pita DNA sehingga dapat menunjukkan polimorfisme.
Menurut Mueller dan Wolfenbarger (1999), tingkat keragaman diperoleh berdasarkan perbedaan dan kesamaan pola pita yang muncul dari hasil pemotongan dengan dua enzim restriksi, EcoRI (G↓AATTC) dan MseI (T↓TAA)
serta primer yang digunakan. Vulysteke et al.(2007) menyatakan bahwa kedua
jenis primer dalam proses amplifikasi di teknik AFLP adalah primer dengan kelebihan satu basa –A dan –C pada proses pre-amplifikasi serta menggunakan primer dengan kelebihan tiga basa –ANN dan –CNN pada ujung 3’OH nya pada proses amplifikasi akhir. Komposisi basa yang digunakan dalam primer selektif ini tersebar luas dalam seluruh bagian genom tanaman (Mba dan Tohme 2005). Oleh sebab itu, tanpa mencari informasi urutan basa genom terlebih dahulu, teknik AFLP ini unggul dalam mendeteksi polimorfisme berdasarkan amplifikasi hasil pemotongan dengan enzim restriksi oleh primer selektif. Teknik ini juga memiliki tingkat pengulangan yang tinggi karena dapat mendeteksi secara konsisten (Invitrogen 2003).
Tujuan dari hasil penelitian ini adalah melakukan identifikasi keragaman genetik dengan AFLP dari tiga klon tanaman karet di Balai Pengkajian Bioteknologi-BPPT, Puspiptek-Serpong yaitu IRR 104, RRIM 600 dan PR 300 dengan dari penggunaan 64 kombinasi pasangan primer yang tersedia. Manfaat yang diharapkan yaitu hasil dari keragaman genetik ini bisa menjadi acuan dalam pemilihan tetua/induk unggul untuk persilangan klon selanjutnya, dan primer selektif yang diperoleh dapat digunakan di lapang untuk mendeteksi klon karet yang sama dalam penelitian ini.
METODE
Waktu dan Tempat
Penelitian dilakukan mulai bulan Februari hingga September 2014. Bertempat di Laboratorium Teknologi Gen dan Laboratorium Pertanian, Balai Pengkajian Bioteknologi-Badan Pengkajian dan Penerapan Teknologi (BPPT), di kawasan Pusat Penelitian Ilmu Pengetahuan dan Teknologi (PUSPIPTEK) Serpong, Tangerang Selatan, serta di Laboratorium Biomolekuler-Balai Besar Biogen, Cimanggu, Bogor.
Bahan
Isolasi DNA Karet. bahan yang digunakan adalah sampel daun karet dari 3 klon pohon karet yang ditanam di Balai Pengkajian Bioteknologi BPPT-Serpong (IRR 104, RRIM 600, PR 300), Poly Vinyl Poly Pirolidon (PVPP), Cethyl Trimethyl Ammonium Bromide (CTAB) 10%, kloroform, isoamilalkohol,
3 PCR AFLP. Enzim T4 DNA ligase, bufer ligasi, penanda (marker) 100 bp
DNA ladder (Fermentas), rATP, enzim MseI, enzim EcoRI, adapter EcoRI
(forward dan reverse) (1st BASE), adapter MseI (forward dan reverse) (1st BASE),
25 mM MgCl2, 2 mmol dNTP, Fast Taq DNA/Taq DNA polymerase (Fermentas)/
Dream Taq DNA polymerase, 10 pmol primer Mse-C, 10 pmol primer Eco-A, dan
10 pmol kombinasi pasangan primer terpilih (Biolabs) (Lampiran 3).
Elektroforesis Poliakrilamid. Alkohol 70%, bufer TBE 10X, 40% akrilamid/bis-akrilamid (19:1), urea, EDTA, amonium persulfat (APS) 10%, TEMED, Tris HCL, NaOH,.37% formaldehid, campuran perak nitrat, sodium
karbonat, asam asetat glasial, bind silane, sodium tiosulfat.
Analisis Data AFLP. Asam asetat glasial, nitrat perak, formaldehid, dan natrium karbonat.
Alat
Alat-alat yang digunakan adalah lumpang dan mortar, vorteks, tabung Eppendorf (1.5 mL), tabung PCR, tip, pipet mikro (1-1000 µL), spektrofotometer
Nanodrop, gel doc, perangkat UV-Vis, PCR thermocycler (Takara), perangkat
elektroforesis agarosa, dan perangkat elektroforesis poliakrialamid dan plat kaca (40 cm x 32 cm).
Prosedur Penelitian
Isolasi DNA Karet
Ekstraksi DNA (Doyle & Doyle 1988). Isolasi DNA genom tanaman karet dilakukan dengan metode Doyle & Doyle (1988) yang telah dimodifikasi. Daun karet dicuci lalu dibersihkan dari tulang daun pada air yang mengalir. Sebanyak 0.2 gr daun karet ditambahkan 300 µL PVPP kemudian digerus hingga halus dan dimasukkan ke dalam tabung Eppendorf. Selanjutnya 1000 µL buffer CTAB 10% (v/v) ditambahkan ke dalam suspensi jaringan daun yang sudah digerus, kemudian diinkubasi pada suhu 65 oC selama 30 menit. Setelah inkubasi, campuran ditambahkan 720 µL kloroform:isoamilalkohol (CIAA 24:1), campuran kemudian dikocok kuat. Sampel disentrifugasi dengan kecepatan 11000 rpm selama 10 menit. Larutan yang jernih (supernatan) diambil, kemudian dipindahkan ke tabung Eppendorf yang baru. Supernatan ditambahkan 1000 µL kloroform:isoamilalkohol (24:1), kemudian dicampur dan disentrifugasi dengan kecepatan 11000 rpm selama 10 menit.
4
natrium asetat 3 M pH 5.2 dan 270 µL alkohol absolut dingin, kemudian dihomogenkan dengan membolak-balikan tabung. Campuran diinkubasi pada lemari pendingin pada suhu -20 oC selama 30 menit kemudian disentrifugasi dengan kecepatan 14000 rpm selama 10 menit. Supernatan yang diperoleh dibuang, sedangkan peletnya dicuci dengan alkohol 70 % lalu dikeringanginkan. Pelet DNA yang sudah kering dilarutkan dengan 100 µL buffer TE.
Analisis Kuantitatif dan Kualitatif DNA. Sebanyak 2 µL DNA hasil isolasi masing-masing diuji kuantitasnya dengan menggunakan spektrofotometer
Nanodrop dan diukur pada perbandingan panjang gelombang 260 nm dan 280 nm
(Å260/Å280). Hasil yang ditampilkan berupa konsentrasi DNA hasil isolasi DNA dalam ng/µL dan rasio kemurnian masing-masing DNA hasil isolasi yang diuji terhadap kontaminasi protein.
Setiap sampel DNA hasil isolasi diambil sebanyak 4 µL untuk dilakukan elektroforesis menggunakan gel agarosa dengan konsentrasi 1.5 % dalam bufer TE 1X selama 50 menit dengan daya 50 V dan pewarna SYBR safe.
Analisis PCR AFLP (Gitzendamer 2000)
Metode AFLP yang digunakan adalah metode Gitzendamer (2000) yang telah dimodifikasi. Genom DNA dengan konsentrasi 250 ng sebanyak 1 µL dipotong dengan sepasang enzim restriksi (0.2 µL MseI dan 0.1 µL EcoRI) yang
ditambahkan adaptor 1 µL MseI dan 1 µL EcoRI, 1 µL T4 buffer, 0.2 µL T4
ligase, 1 µL rATP, dan 4.5 µL nuclease water sehingga total volume menjadi 12.5
µL. Bahan dicampur sampai homogen dan kemudian diinkubasi pada suhu 37 oC selama 2 jam dan diinaktivasi dengan suhu 65 oC selama 15 menit lalu dilanjutkan dengan suhu 65 oC selama 20 menit oleh pemotongan enzim MseI. Selanjutnya dilakukan ligasi dengan adapter EcoRI dan MseI pada suhu 37 oC selama 4 jam.
Hasil ligasi, kemudian dipakai sebagai cetakan untuk pre-amplifikasi. Untuk mengetahui berhasil atau tidaknya hasil digesti dan ligasi, maka dilakukan elektroforesis menggunakan gel agarose 1% dan buffer TAE 1X dengan tegangan 50 V selama 50 menit.
Reaksi pre-amplifikasi terdiri dari 1 µL DNA hasil ligasi, 2.5 µ L buffer PCR 10X, 2.5 µL 25 mM MgCl2, 2 µL dNTP mix, 0.2 µL Fast Taq DNA, 1 µL primer Mse-C 10 pmol, 1 µL primer Eco-A 10 pmol, dan 14.8 µL ddH2O. Proses PCR dilakukan menggunakan PCR thermocycle pada suhu 72 oC selama 2 menit
(pre-denaturasi), kemudian sebanyak 29 siklus pada 94 oC selama 30 detik (denaturasi), 56 oC selama 30 detik (penempelan primer), 72 oC selama 2 menit (ekstensi), dan suhu 60 oC selama 10 menit untuk pemanjangan akhir, serta disimpan dalam suhu 4oC. Setelah diamplifikasi, hasil yang diperoleh diamplifikasi selektif menggunakan kombinasi pasangan primer yang memiliki tambahan 3 basa pada ujung 3’OH nya (Lampiran 4).
Sebanyak 1 µL hasil pre-amplifikasi dicampur dengan 2.5 µL buffer PCR 10X, 2.5 µL 25 mM MgCl2, 2 µL dNTP campuran, 0.2 µL Taq polimerase, primer terpilih 1 µL 10 pmol MseI dan 1 µ L 10 pmol EcoRI, dan 14.8 µL nuclease water
5 dengan suhu 72 oC selama 2 menit untuk pemanjangan akhir serta penyimpanan pada suhu 4 oC.
Elektroforesis Poliakrilamid. Hasil amplifikasi PCR dielektroforesis pada 70 mL larutan PAGE 5 % (Urea, 10X TBE, 40 % akrilamid/bis-akrilamid) yang dicampurkan dengan 840 µL APS 10 % dan 35 µL TEMED. Campuran dimasukkan dalam plat kaca berukuran 30 cm x 32 cm dan didiamkan hingga memadat. Plat kaca berisi gel tersebut dilakukan pre-elektroforesis (pemanasan) dilakukan selama 30 menit (110 W) hingga suhu permukaan gel mencapai + 55 oC.
Hasil amplifikasi selektif dan marka 100 pb masing-masing diambil sebanyak 6-7 µL dicampur dengan 1 µL loading dye, kemudian didenaturasi pada
suhu 95 oC selama tiga menit dan segera didinginkan dan disimpan dalam es pada suhu minimal 4 0C. Campuran sampel sebanyak 5 µL dimasukkan pada setiap sumur dan dielektroforesis selama ± 40 menit dengan daya 80 W.
Pewarnaan Gel Poliakrilamid. Gel poliakrilamida kemudian dibilas dengan ddH2O bebas ion selama 1 menit, kemudian difiksasi dengan asam asetat glasial 10% selama 10 menit dan dibilas kembali dengan ddH2O bebas ion selama 20 detik lalu dibiarkan mengering. Gel diwarnai dengan larutan perak nitrat di atas pengocok selama 20 menit dan dicuci dengan ddH2O bebas ion selama 20 detik. Selanjutnya gel direndam dalam larutan developer (campuran 45 gram sodium
karbonat, 2.25 mL formaldehid 37 %, 300 µL sodium tiosulfat dalam 1500 mL ddH2O, yang sebelumnya disimpan pada suhu 4-10oC) dan dibiarkan hingga fragmen-fragmen dapat dilihat sesuai dengan yang diinginkan.
Setelah seluruh pita terlihat, gel direndam dalam larutan stop berupa larutan
fiksasi selama 2 menit. Gel kemudian dibilas dengan menggunakan ddH2O bebas ion dan dibiarkan mengering selama 1 hari kemudian dipindai dengan alat pemindai.
Identifikasi Data AFLP
Skoring. Pita-pita DNA dari hasil elektroferogram diubah ke dalam bentuk data biner dengan memberi nilai 1 jika terdapat pita DNA dan 0 jika tidak ada pita. Panjang pita AFLP diperhitungkan menggunakan pita standar marka DNA 10 bp [Thermoscientific] berdasarkan jarak migrasi pita AFLP dengan aplikasi
PhotoCapt MW.
Identifikasi Keragaman Genetik (Damayanti 2012). Data biner yang diperoleh dari AFLP tersebut selanjutnya digunakan dalam analisis keragaman genetik, kesamaan genetik dibuat dalam bentuk matrik dengan similarity for qualitative data (SIMQUAL). Berdasarkan nilai kesamaan genetik tersebut
dilakukan analisis pengelompokan (pengklasteran) dengan sub program SAHN dengan metode Unweighted Pair Group Method Average (UPGMA) program
6
HASIL
DNA Karet Hasil Isolasi
Isolasi DNA karet dengan menggunakan metode Doyle dan Doyle (1988) telah berhasil dilakukan, kemudian dilakukan analisis kuantitatif dan kualitatif DNA hasil isolasi. Berdasarkan analisis kuantitatif diperoleh nilai kemurnian DNA hasil isolasi berturut-turut dari klon IRR 104, RRIM 600, dan PR 300 yaitu 1.97, 2.04, dan 1.78 sedangkan konsentrasi DNA yang diperoleh secara berurutan yaitu 1024.2, 1259.5, dan 4127.3 ng µL-1 (Tabel 1). Selanjutnya hasil analisis kualitatif berupa elektroferogram (Gambar 1) menunjukkan bahwa DNA hasil isolasi memiliki ukuran DNA genom sekitar 10 kb. Hasil isolasi nampak cukup jelas menandakan bahwa DNA telah terisolasi dengan baik.
Produk PCR AFLP DNA Karet
Hasil isolasi DNA karet yang diperoleh dilanjutkan dengan pengujian pemotongan/digesti oleh enzim restriksi EcoRI dan MseI. Selanjutnya pengujian
dengan ligasi menggunakan adapter yang spesifik untuk EcoRI dan MseI tampak
berupa bayangan panjang pada gel agarosa 1.5 %. Hasil digesti-ligasi tersebut kemudian menjadi DNA cetakan untuk pre-amplifikasi menggunakan primer
EcoR-A dan Mse-C. Hasil elektroforesis dari pre-amplifikasi berupa bayangan
10 kb
Tabel 1 Konsentrasi dan kemurnian DNA karet hasil isolasi Nomor
Sampel Jenis Klon Konsentrasi DNA (ng/µL) Kemurnian (Å260/Å280)
1 Klon IRR 104 1024,2 1,97
2 Klon RRIM 600 1259,5 2,04
3 Klon PR 300 4127,3 1,78
Gambar 1 Hasil elektroforesis DNA isolasi (Marka 1 kb, 1-3: DNA klon IRR 104, klon RRIM 600, dan klon PR 300).
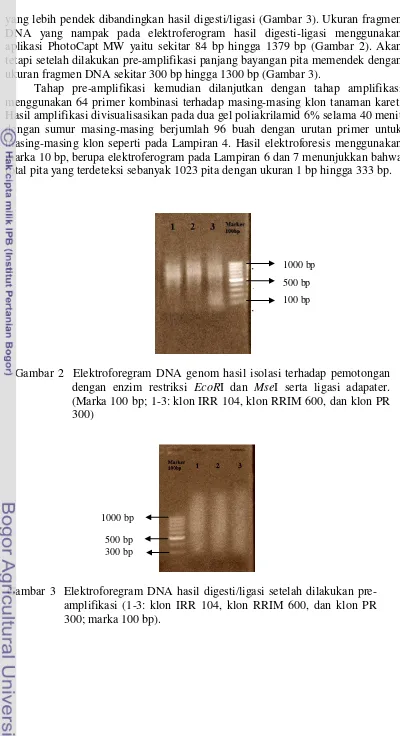
7 yang lebih pendek dibandingkan hasil digesti/ligasi (Gambar 3). Ukuran fragmen DNA yang nampak pada elektroferogram hasil digesti-ligasi menggunakan aplikasi PhotoCapt MW yaitu sekitar 84 bp hingga 1379 bp (Gambar 2). Akan tetapi setelah dilakukan pre-amplifikasi panjang bayangan pita memendek dengan ukuran fragmen DNA sekitar 300 bp hingga 1300 bp (Gambar 3).
Tahap pre-amplifikasi kemudian dilanjutkan dengan tahap amplifikasi menggunakan 64 primer kombinasi terhadap masing-masing klon tanaman karet. Hasil amplifikasi divisualisasikan pada dua gel poliakrilamid 6% selama 40 menit dengan sumur masing-masing berjumlah 96 buah dengan urutan primer untuk masing-masing klon seperti pada Lampiran 4. Hasil elektroforesis menggunakan marka 10 bp, berupa elektroferogram pada Lampiran 6 dan 7 menunjukkan bahwa total pita yang terdeteksi sebanyak 1023 pita dengan ukuran 1 bp hingga 333 bp.
Gambar 3 Elektroforegram DNA hasil digesti/ligasi setelah dilakukan pre-amplifikasi (1-3: klon IRR 104, klon RRIM 600, dan klon PR 300; marka 100 bp).
1000 bp
500 bp 300 bp
Gambar 2 Elektroforegram DNA genom hasil isolasi terhadap pemotongan dengan enzim restriksi EcoRI dan MseI serta ligasi adapater.
(Marka 100 bp; 1-3: klon IRR 104, klon RRIM 600, dan klon PR 300)
500 bp 1000 bp
8
Hasil elektroferogram AFLP (Lampiran 6 dan 7) menunjukkan jumlah pita AFLP terbanyak adalah 24 pita yang berasal dari amplifikasi MCAT/EAAG pada klon PR 300 (Lampiran 7). Klon IRR 104 hanya memiliki pita terbanyak sejumlah 10 pita oleh primer MCAC/EAGG dan MCAT/EACA. Selain itu, pita terbanyak klon RRIM 600 diperoleh sejumlah 23 pita oleh primer MCAC/EAAG. Sebaliknya, jumlah pita AFLP paling sedikit hanya menghasilkan satu pita AFLP saja pada masing-masing klon. Selanjutnya, terdapat primer yang tidak mengamplifikasi yaitu pasangan primer CTC/E-AGG, CAC/E-ACT, M-CAC/E-AAC, M-CTT/E-ACC, M-CTT/E-AAC, M-CTA/E-ACA, CTG/E-AAG, dan CTG/E-AGG untuk klon IRR 104. Primer CAC/E-ACG, M-CAT/E-AGG, dan M-CAA/E-ACG tidak mengamplifikasi pada klon RRIM 600. Serta pada klon PR 300 hanya pasangan primer M-CTC/E-AAG yang tidak menghasilkan pita AFLP.
Berdasarkan elektroferogram pada Lampiran 6 dan 7, terdapat pula primer yang menghasilkan pita-pita dengan ukuran yang unik, seperti primer MCAA/EAGC pada klon IRR 104 (Tabel 2). Pita unik merupakan pita yang memiliki satu ukuran tertentu yang hanya dihasilkan oleh satu klon saja oleh satu primer spesifik. Pita unik juga dihasilkan pada klon RRIM 600 oleh primer MCTC/EACG, MCTC/EACA, MCAC/EACG, MCTA/EAGG, dan MCTT/EACC. Berikutnya untuk klon PR 300, pita unik dihasilkan oleh pasangan primer MCTC/EAAC, MCTA/EAGG, MCAT/EACC, MCAT/EAAG, MCAC/EACC, dan MCTG/EAGG.
Keragaman Genetik Klon Karet
Elektroferogram yang diperoleh dari amplifikasi dengan teknik AFLP menghasilkan pita-pita AFLP dengan berbagai variasi ukuran. Berdasarkan variasi tersebut, dilakukan pengolahan data biner/skoring untuk pembentukan pohon Tabel 2 Pasangan primer spesifik dan ukuran unik yang dihasilkan pada
masing-masing klon
Nama Klon Pasangan Primer Spesifik Ukuran Pita Unik
9 filogenetik berupa dendrogram. Dendrogram dibuat dengan menggunakan seluruh pita hasil AFLP.
Berdasarkan hasil dendrogram dengan menggunakan program NTSYS 2.11 diperoleh koefisien kemiripan genetik antar ketiga klon adalah rendah yaitu sebesar 0.29 yang menunjukkan kekerabatan genetik sebesar 29 %. Garis filogenetik terbagi menjadi dua kelompok, kelompok 1 terdiri dari klon IRR 104 dan klon RRIM 600, sedangkan kelompok 2 hanya terdiri dari klon PR 300. Koefisien kesamaan genetik pada kelompok 1 sebesar 0.43 yang menunjukkan adanya kekekerabatan sebesar 43 % antar kedua klon tersebut. Sebaliknya, di kelompok 2 yang hanya terdiri dari klon PR 300 yang terpisah dalam kelompok tersendiri.
PEMBAHASAN
DNA Karet Hasil Isolasi
Isolasi DNA karet merupakan tahap awal dalam penelitian ini untuk mendapatkan DNA genom yang selanjutnya akan menjadi DNA cetakan dalam amplifikasi dengan teknik PCR. DNA karet diisolasi dari daun yang diambil dari daun pertama pada pucuk. Isolasi DNA memiliki empat prinsip utama, yaitu: pelisisan dinding sel, pelepasan sel-sel dari jaringan, pemisahan DNA dari makromolekul lain, dan pemurnian hasil ekstraksi DNA.
Isolasi DNA karet pada penelitian ini menggunakan metode CTAB menurut Doyle dan Doyle (1988) yang dimodifikasi menggunakan penambahan PVPP sebagai antioksidan guna mencegah reaksi oksidasi (Dewi 2008). Metode ini sering digunakan dalam proses isolasi DNA tanaman karena mudah dan dapat menghasilkan konsentrasi serta kemurnian DNA hasil isolasi yang baik. Metode isolasi DNA tersebut, menggunakan bufer ekstraksi mengandung CTAB (Cetyl Trimethyl Ammonium Bromide) yang secara kimiawi akan menghancurkan sel
(Sulandri dan Zei 2003).
10
Menurut Surzycki (2000), bufer ekstraksi CTAB banyak digunakan untuk tanaman yang mengandung banyak polisakarida untuk mengurangi aktivitas enzim nuklease yang merupakan enzim pendegradasi DNA (Yuwono 2005). Larutan EDTA yang terdapat dalam bufer ekstraksi berfungsi untuk mengkelat ion magnesium yang merupakan kofaktor enzim nuklease (Barnum 2005) sehingga melindungi asam nukleat dari degradasi (Barnum 2005; Horison et al. 2003).
Tris-HCl dalam komposisi bufer ekstraksi memiliki peran penting yaitu menjaga kondisi pH larutan tetap stabil sehingga struktur DNA terjaga selama proses isolasi (Corkill dan Rapley 2008). Penambahan 2-merkaptoetanol dalam bufer lisis digunakan untuk mereduksi ikatan disulfida yang terdapat pada sisa-sisa protein (Wilson dan Walker 2000). Selanjutnya, penggunaan kloroform:isoamilalkohol dalam tahapan proses isolasi dapat melisiskan RNA (Agustian 2008), menghilangkan protein dan polisakarida lain serta lemak (Hamzah 2014). Isopropanol dan etanol absolut akan memisahkan DNA dari garam mineral dan mengendapkannya, sekaligus akan melarutkan sisa kloroform:isoamilalkohol (Agustian 2008).
Hasil isolasi DNA karet diuji secara kuantitatif dengan menggunakan spektrofotometer Nanodrop pada panjang gelombang 260 nm dan 280 nm serta secara kualitatif menggunakan gel elektroforesis agarosa 1 % dengan pewarna
SYBR safe (Gambar 1). DNA hasil isolasi dianggap baik dan murni jika memiliki
rasio Å260/Å280 berkisar 1.8-2.0 (Walker dan Wilson 2000; Yuwono 2005).
Urutan klon karet yang diisolasi DNAnya adalah IRR 104, RRIM 600, dan PR 300. Berdasarkan hasil yang diperoleh secara kuantitatif (Tabel 1) kemurnian hasil isolasi DNA karet secara berurutan yaitu 1.97, 2.04, dan 1.78. Nilai kemurnian yang kurang dari 1.8 menandakan adanya kontaminasi protein dan polisakarida lainnya dalam DNA hasil isolasi, sedangkan kemurnian hasil isolasi DNA yang melebihi 2.0 menandakan adanya RNA yang tersisa dari hasil isolasi (Walker & Wilson 2000). Berdasarkan nilai kemurnian yang diperoleh, DNA hasil isolasi tetap dapat digunakan sebagai DNA cetakan untuk PCR dengan AFLP karena proses PCR tetap akan mengamplifikasi DNA cetakan secara spesifik oleh primer bahkan dengan kemurnian yang cukup rendah (Bintang 2010). Oleh sebab itu, dengan kemurnian yang diperoleh DNA hasil isolasi ini dapat digunakan selama proses tahapan AFLP.
Selanjutnya, konsentrasi DNA yang diperoleh secara berurutan yaitu 1024.2, 1259.5, dan 4127.3 ng µL-1. Perbedaan konsentrasi DNA hasil isolasi dapat disebabkan oleh kontaminan, terbuangnya DNA selama proses isolasi (Henry 2001) dan pemilihan sampel berupa daun muda/daun tua untuk diisolasi. Proses AFLP ini menggunakan konsentrasi DNA karet yang diencerkan sebesar 250 ng/µL (Zulkifli 2001), namun dalam penelitian yang lainnya dengan konsentrasi DNA 50 ng/µ L saja AFLP dapat dilakukan (Nurhaimi-Haris et al. 2003). Proses
pengenceran ini dimaksudkan untuk menghasilkan hasil PCR AFLP dengan ketebalan pita yang sama.
Analisis kualitatif dengan menggunakan elektroforesis gel agarosa 1% (Gambar 1) dan pewarna SYBR safe. Menurut Sambrook dan Russel (2001)
pewarna SYBR safe dapat membuat pita DNA hasil isolasi berpendar setelah
dielektroforesis di bawah sinar UV. SYBR safe memiliki nama lain metil benzen
sulfonat, yang dilarutkan dalam dimetilsulfoksida (Evenson et al 2012). Hasil
11 karet, walaupun tidak secara utuh karena adanya bayangan pada masing-masing lajur. Pita tebal yang berada di atas menunjukkan bahwa bobot molekul DNA genom yang diperoleh cukup besar, sekitar 10 kb (pasang basa). Ukuran yang cukup besar menyebabkan pada visualisasi DNA hasil isolasi tersebut menunjukkan pita DNA genom masih berada di atas karena pergerakan molekulnya yang lebih lambat (Bintang 2010). Bayangan di bawah pita DNA genom hasil isolasi menunjukkan terjadinya pemotongan/fragmentasi DNA genom yang terjadi selama proses isolasi (Hamzah 2014).
Produk PCR AFLP DNA Karet
Hasil isolasi DNA kemudian dilakukan proses PCR AFLP dengan tahapan awal berupa restriksi DNA genom hasil isolasi yang diencerkan terlebih dahulu menjadi 250 ng/µL, menggunakan dua enzim restriksi. Kedua enzim tersebut memiliki perbedaan situs pemotongan dan tingkat kemunculan hasil pemotongan DNA yang berbeda pula, selanjutnya diberi istilah enzim pemotong sering dan enzim pemotong jarang. Enzim restriksi yang digunakan dalam penelitian ini yaitu
EcoRI sebagai enzim pemotong jarang dan TruI sebagai enzim pemotong sering.
Enzim TruI yang digunakan merupakan enzim yang mengenali situs pemotongan
basa yang sama dengan enzim MseI, hanya saja suhu optimasi saat proses
pemotongan berlangsung adalah berbeda, yaitu 65oC untuk TruI dan 37oC untuk MseI. Tujuan dari pemotongan menggunakan enzim restriksi adalah menghasilkan
jumlah optimum fragmen DNA sehingga pola pita AFLP nantinya dapat diskor dengan baik karena tingkat polimorfisme yang dihasilkan lebih rinci dan tinggi.
Menurut Vulysteke et al. (2007), karena tanaman eukariot memiliki basa
adenin dan timin yang lebih tinggi dibandingkan basa guanin dan sitosin, maka digunakan enzim EcoRI dan MseI/TruI untuk menghasilkan polimorfisme yang
lebih rinci karena basa adenin dan timin yang dimiliki kedua enzim tersebut lebih tinggi. EcoRI merupakan enzim pemotong jarang yang mempunyai situs
pengenalan 6 basa (G↓AATTC), sedangkan TruI merupakan enzim pemotong
sering yang mempunyai situs pengenalan 4 basa (T↓TAA) yang dapat memotong DNA genom tanaman menjadi potongan DNA menjadi berukuran 50-2000 bp (Saunders JA et al. 2001). Enzim restriksi pemotong sering akan menghasilkan
jumlah situs potongan DNA yang sangat banyak, namun jumlah tersebut akan dikendalikan dengan situs pemotongan oleh enzim pemotong jarang (Setiawan 2001) sebelum diamplifikasi.
Tahapan AFLP selanjutnya adalah penggabungan DNA hasil pemotongan tersebut dengan adapter EcoRI dan MseI. Adapter ini berfungsi untuk membentuk
ujung tumpul dari DNA hasil pemotongan yang berujung lancip, serta memudahkan proses amplifikasi karena adapter ini memiliki situs yang komplemen terhadap primer AFLP. Vulysteke et al. (2007) menyatakan bahwa
adapter EcoRI mengandung kelebihan basa AATT pada ujung 5’ dan ujung
pemotongan TruI dengan kelebihan basa TA pada ujung 5’ nya. Keberhasilan dari
proses pemotongan dan ligasi dapat dilihat secara visual menggunakan elektroforesis seperti elektroferogram yang dihasilkan pada Gambar 2, menunjukkan ukuran bayangan/smear yang dihasilkan sekitar 84 bp hingga 1379
12
Menurut Vuylsteke et al. (2007) hasil digesti-ligasi ini menjadi cetakanbagi
primer pada proses selanjutnya, yaitu pre-amplifikasi. Tujuan dari proses ini adalah untuk meningkatkan selektifitas fragmen DNA yang telah direstriksi dan ligasi sebelumnya Proses pre-amplifikasi menggunakan primer yang berkomplemen pada fragmen DNA hasil digesti-ligasi dengan tambahan satu nukleotida selektif pada ujung 3’. Sehingga berdasarkan protokol dari Gitzendamer (2000), primer pre-amplifikasi yang digunakan adalah EcoR-A dan Mse-C. Keberhasilan dari proses pemotongan dan ligasi dapat dilihat secara visual
menggunakan elektroforesis yang ditunjukkan pada Gambar 3 memperlihatkan ukuran bayangan/smear yang dihasilkan memendek dengan ukuran sekitar 300 bp
hingga sekitar 1300 bp. Hasil tersebut sesuai dengan protokol Applied Biosystem
(2010) yang menyatakan bahwa secara visual, hasil elektroferogram pada tahap pre-amplifikasi berupa pita berbayang dengan rentang ukuran yang lebih pendek dibandingkan rantang ukuran hasil digesti/ligasi.
Tahap pre-amplifikasi kemudian dilanjutkan dengan tahap amplifikasi menggunakan 64 primer kombinasi terhadap masing-masing klon tanaman karet. Hasil amplifikasi tiga klon tanaman karet tersebut divisualisasikan dari hasil elektroforesis pada gel 6% selama 40 menit di dalam sumur yang masing-masing berjumlah 96 buah dengan urutan primer untuk masing-masing klon seperti pada Lampiran 4 dan 5. Hasil elektroforesis dengan menggunakan marka 10 bp terdapat pada Lampiran 6 dan 7 dengan total pita yang terdeteksi sebanyak 1023 pita yang berkuran variatif antara 1 bp hingga 333 bp. Ukuran yang muncul berbeda dengan ukuran hasil pre-amplifikasi sebelumnya yang memiliki rentang bayangan 300 bp – 1000 bp.
Menurut Setiawan (2001) banyaknya DNA yang terpotong sebelum mengalami digesti menyebabkan banyaknya DNA genom tidak terpotong sempurna oleh kedua enzim restriksi dan tidak terligasi dengan baik oleh kedua adapter yang menyebabkan fragmen DNA tidak teramplifikasi dengan baik karena tidak memiliki situs penempelan primer. Oleh karena itu, DNA hasil isolasi yang baik sangat penting dalam teknik AFLP ini untuk menghindari pemotongan DNA yang tidak lengkap dan dapat menyebabkan kesalahan dalam identifikasi pita polimorfis (Nurhaimi 2006). Kesalahan identifikasi pita menyebabkan tidak munculnya ukuran pita pada hasil elektroferogram di tahap digesti-ligasi maupun pre-amplifikasi sebelumnya dan menyebabkan perbedaan ukuran fragmen DNA pada elektroforegram gel poliakrilamid setelah amplifikasi. Namun, dalam penelitian ini seluruh pita yang dihasilkan tetap dianggap mewakili karakter tertentu pada masing-masing klon, maka seluruh pita tetap digunakan dalam identifikasi keragaman genetik.
13
Keragaman Genetik Klon Karet
Keragaman genetik merupakan variasi genetik yang tejadi dalam suatu spesies, khususnya, yang disebabkan oleh berbagai macam faktor. Berbagai macam variasi genetik pada tanaman khususnya, banyak dikembangkan guna menghasilkan tanaman unggul. Salah satunya di Indonesia adalah tanaman karet, khususnya dari Hevea brasillensis dengan berbagai macam klon telah dihasilkan
sejak tahun 1864 (Setiawan dan Agus 2008). Tiga klon yang digunakan dalam penelitian yaitu IRR 104, RRIM 600, dan PR 300 adalah contoh dari klon yang dikembangkan di Indonesia.
Klon pertama adalah IRR 104 (Indonesian Rubber Research) merupakan
klon karet ungul anjuran asal Indonesia berdasarkan Surat Keputusan Menteri Pertanian (2005) . Klon ini berasal dari tetua BPM 101 dengan RRIC 110 (Nancy 2007). Klon RRIC (Rubber Research Ceylon) sendiri merupakan klon asal
Srilangka, dan BPM (Balai Penelitian Medan) merupakan klon asal Indonesia. Klon kedua adalah RRIM 600 yang berasal dari Malaysia hasil persilangan TJIR 1 (Tjirandji 1) asal Indonesia dengan PB 86 (Prang Besar 86) asal Malaysia (Situmorang et al. 2012; Priyadarshan 2011). Berikutnya klon ketiga adalah klon
PR 300 asal Indonesia yang berasal dari persilangan PR 226 dan PR 228 yang keduanya juga berasal dari Indonesia. Ketiga klon tersebut merupakan klon penghasil lateks (Rachmawan et al. 2006).
DNA hasil isolasi yang telah diamplifikasi menggunakan teknik AFLP berupa hasil elektroforesis sebelumnya (Lampiran 6 dan 7) diubah dalam bentuk data biner untuk dikonstruksi menjadi pohon filogenetik, atau biasa disebut dengan tahapan skoring, sehingga diperoleh tingkat keragaman genetik antar ketiga klon yang digunakan. Penyusunan dilakukan berdasarkan kesamaan pola pita hasil AFLP, dengan bobot molekul hasil pita pada masing-masing primer digunakan sebagai parameter pembeda antar klon. Jika terdapat pita yang muncul pada posisi bobot molekul tertentu maka diberi nilai 1 dan jika tidak muncul maka diberi nilai 0 (Sari 2007 dalam Wibowo 2010). Melalui program NTSYS 2.11 dengan menggunakan metode UPGMA (Unweighted Pair Grup Method Arithmatic Mean) hasil biner ini dapat digunakan untuk konstruksi dendrogram
/pohon filogenetik tiga klon tanaman karet.
14
SIMPULAN DAN SARAN
Simpulan
Teknik AFLP berhasil mengidentifikasi keragaman genetik tiga klon karet dari perkebunan balai Biotek-BPPT, Puspiptek-Serpong berdasarkan banyaknya pita polimorfis yang muncul dan tingkat kesamaan genetik yang dihasilkan. Identifikasi keragaman genetik dari tiga klon karet yaitu, IRR 104, RRIM 600, dan PR 300 menggunakan teknik AFLP dengan 64 pasang primer menghasilkan 1023 pita AFLP dengan ukuran pita yang bervariasi antara 1 bp-333 bp yang bersifat polimorfis. Tingkat kesamaan genetik ketiga klon karet tersebut sebesar 23%, dan terbagi dua kelompok dengan tingkat kesamaan genetik pada puncak pohon filogenetik yang diperoleh antara IRR 104 dan RRIM 600 adalah 43%.
Saran
Perlu dilakukan penelitian lanjutan dengan menggunakan primer dan klon yang lain untuk hasil yang lebih spesifik dan beragam dalam penentuan keragaman antar klon tanaman karet unggulan yang ada di Indonesia. Seleksi marka lanjutan dan proses sekuensing juga bisa digunakan untuk mengkonfirmasi pita spesifik dan unik yang dapat digunakan sebagai sidik jari dari klon tanaman karet unggul setelah diperoleh keragaman genetik antar klon karet yang diuji.
DAFTAR PUSTAKA
Agustian A. 2008. Karakterisasi variasi genetik Jatropha curcas L. dengan
menggunakan marka molekuler Amplified Fragment Length Polymorphism
(AFLP) [skripsi]. Depok (ID): Universitas Indonesia.
Anwar C. 2001. Manajemen dan Budidaya Karet. Medan (ID): Pusat Penelitian
Karet
Barnum SR. 2005. Biotechnology an Introduction. 2nd Ed. US: Brooks-Cole.
Corkill G, Rapley R. 2008. The manipulation of nucleic acids: basic tools and techniques. In: Molecular Biomethods Handbook Second Edition. US: Human
Press.
Damayanti D. 2012. Analisis keragaman genetik temulawak (Curcuma xanthoriza
Roxb.) menggunakan penanda Amplified Fragment Length polymorphism
(AFLP) [tesis]. Bogor (ID): Institut Pertanian Bogor
Dewi K P.2008. Identifikasi keragaman genetik jarak pagar (Jatropha curcas L.)
berdasarkan karakter bunga dan dna menggunakan teknik Amplified Fragment Length Polymorphism (AFLP) [tesis]. Bogor (ID): Institut Pertanian Bogor.
Doyle JJ, Doyle JL. 1988. Isolation of plant DNA from fresh tissu. Focus 12:
13-15
Evenson WE, Boden LM, Muzikar KA, O’Leary DJ. 2012. 1H and 13C NMR
15 Gitzendanner M. 2000. AFLP protocols. Massachusets (US). Applied Biosystems [internet]. [diacu: 2013 Dec 21]. Tersedia pada: (Hevea brasiliensis Muell Arg.) berdasarkan metode Amplified Fragment Legth Polymorphism (AFLP). Menara Perkebunan. 71(1):1-15
Harni R. 2013. Penyakit gugur daun Odium pada tanaman karet.. Sukabumi (ID):
Balai Penelitian Tanaman Industri dan Penyegar [internet]. [diacu 2013 Des 27]. Tersedia pada: http://balittri.litbang.deptan.go.id/index.php/component/ content/article/49-infotekno/175-penyakit-gugur-daun-oidium-pada-tana man-karet
Horison C, Rustikawati, Eliyanti. 2003. Penentuan protokol yang tepat untuk menyiapkan DNA genom cabai. J. Akta Agrosia. 6(2): 38-43.
Invitrogen. 2003. AFLP® Analysis system I, AFLP® starter kit [protokol]. California (US): Life Technologies.
Saunders JA, Mitschke S, Hemetda AA. 2001. The use of AFLP techniques for DNA fingerprinting in plants [application information]. California (US): Beckman Coulter
Lespinasse D, Rodier-Goud M, Grivet L, Leconte A, Legnate H, Seguin M. 2000 A saturated genetic linkage map of rubber tree (Hevea spp) based on RFLP,
AFLP, microsatellite and isozyme markers. Ther Appl Genet 100: 127–138
Lodhi MA, Ye GN, Weeden NF, Reisch BI. 1994. A simple methode for DNA extraction from grapevine cultivars and vitis species. Plant Mol. Biol. Rep.
12:6-13.
Mathius T, Lizawati, H. Aswidinoor, I Boerhendy. 2002. Pengaruh batang bawah terhadap pola pita isoenzim dan protein batang atas pada okulasi tanaman karet (Hevea brasiliensis Muell Arg.). Menara Perkebunan 70: 20-34
Mba C, Tohme J. 2005. Use of AFLP markas in surveys of plant diversity.
Methods enzymol. 395:177-201
Mueller UG, Wolfenbarger LL. 1999. AFLP genotyping and fingerprinting. Els Sci 14:389-394
Mulsanti IW. 2010. Identifikasi dan evaluasi kemurnian genetik benih padi hibrida menggunakan mikrosatelit [tesis]. Bogor (ID): Institut Pertanian Bogor Nancy C. 2007. Klon karet unggul untuk 2006-2010. Sinar Tani Ed: 28 Maret-3
April 2007.
Nagy S, Poczai P, Cernak I, Gorji AM, Hegedus G, Taller J. 2012. PICcalc: an online program to calculate polymorphic information content for molecular genetic studies. Biochem Genet. 50:670-672
Nurhaimi-Haris, Aswidinnoor H, Toruan-Mathius N, Purwantara A. 2003. Kemiripan genetik klon karet (Hevea brasiliensis Muell. Arg.) berdasarkan
metode Amplified Fragment Length Poymorphisms (AFLP). Menara Perkebunan. 71(1):1-15.
Priyadarshan PM. 2011. Biology of Hevea rubber. India: Rubber Research Institut
16
Rachmawan A, Anas A, Woelan S. 2006. Karakteristik sifat lateks dan kayu klon-klon karet anjuran [prosiding]. Lokakarya Nasional Budidaya Tanaman Karet;
2006 Sep 4-6; Medan. Medan (ID): Balai Penelitian Sungei Putih
Sambrook J, Russel DW. 2001. Molecular clonning: a laboratory manual. 3rd ed. New York (US): CSHL Press.
Setiawan A. 2001. Pemetaan marker AFLP untuk membuat peta genetik bit gula.
Bul. Agron. 29 (2): 40-49
Setiawan DH, Agus A. 2008. Petunjuk Lengkap Budidaya Karet. Jakarta (ID): Agromedia Pustaka
Sudjarmoko B. 2013. Peran strategis industri benih dalam geakan nasional peningkatan produktivitas karet di Indonesia. Sukabumi (ID): Balai Penelitian Tanaman Industri dan Penyegar [internet]. [diacu 2013 Des 27]. Tersedia pada: http://balittri.litbang.deptan.go.id/index.php/component/content/article/49-info tekno/166-peran-strategis-industri-benih-dalam-gerakan-nasional-peningkatan-produktivitas-karet-di-indonesia
Sulandri S, Zein MSA. 2003. Panduan praktis laboratorium DNA bidang zoologi. Bogor (ID): Lembaga Ilmu Pengetahuan Indonesia.
Surzycki SJ. 2000. Basic techniques in molecular biology. Springer-Verlag ISBN
3-540-66678-8
Vulysteke M, Peleman JD, van Eijk MJT. 2007. AFLP-based transcript profiling (cDNA-AFLP) for genome-wide expression analysis. Nature Protocols 2: 1399-1413
Walker JM, Wilson K. 2000. Principles and techniwques of practica biochemistry. Cambridge (GB): Cambridge University Press.
Wibowo IY. 2010. Analisis keragaman genetik tanaman karet hasil persilangan antara RRIM 600 dan PN 1546 dengan menggunakan teknik RAPD [skripsi]. Bogor(ID): Institut Pertanian Bogor.
Woelan S, R. Tistama, Aidi D. 2007. Determinasi keragaman genetik hasil persilangan interpopulasi berdasarkan karakteristik morfologi dan teknik RAPD. J. Penelitian Karet 25(1):13-27.
Yuwono T. 2005. Teori dan Aplikasi Polymerasi Chain Reaction. Yogyakarta
(ID): Andi
Zulkifli L. 2001. Analisis pembeda klon karet tahan dan rentan penyakit gugur daun corynespora serta anaisis keragaman genetik dengan AFLP dan RAPD
17
LAMPIRAN
Lampiran 1 Diagram alir penelitian
Isolasi DNA genom 3 klon karet metode CTAB Doyle & Doyle
Uji kuantitatif dan kualitatif DNA hasil isolasi
PCR AFLP (digesti-ligasi)
PCR AFLP (pre-amplifikasi)
PCR AFLP (amplifikasi)
Elektroforesis gel poliakrilamid 6%
Skoring profil pita AFLP
18
Lampiran 2 Prinsip teknik Amplified Fragment Length Polymorphism (AFLP)
19 Lampiran 3 Pasangan Primer AFLP
Primer M-CTG M-CAA M-CTA M-CTC M-CTT M-CAT M-CAG M-GAC
E-ACT E-ACT/ M-CTG E-ACT/ M-CAA E-ACT/ M-CTA M-CTC E-ACT/ E-ACT/ M-CTT E-ACT/ M-CAT E-ACT/ M-CAG E-ACT/ M-GAC
E-ACA E-ACA/ M-CTG E-ACA/ M-CAA E-ACA/ M-CTA M-CTC E-ACA/ E-ACA/ M-CTT E-ACA/ M-CAT E-ACA/ M-CAG E-ACA/ M-GAC
E-AAC E-AAC/ M-CTG E-AAC/ M-CAA E-AAC/ M-CTA M-CTC E-AAC/ E-AAC/ M-CTT E-AAC/ M-CAT E-AAC/ M-CAG E-AAC/ M-GAC
E-ACC E-ACC/ M-CTG E-ACC/ M-CAA E-ACC/ M-CTA M-CTC E-ACC/ E-ACC/ M-CTT E-ACC/ M-CAT E-ACC/ M-CAG E-ACC/ M-GAC
E-AGC E-AGC/ M-CTG E-AGC/ M-CAA E-AGC/ M-CTA M-CTC E-AGC/ E-AGC/ M-CTT E-AGC/ M-CAT E-AGC/ M-CAG E-AGC/ M-GAC
E-AAG E-AAG/ M-CTG E-AAG/ M-CAA E-AAG/ M-CTA M-CTC E-AAG/ E-AAG/ M-CTT E-AAG/ M-CAT E-AAG/ M-CAG E-AAG/ M-GAC
E-ACG E-ACG/ M-CTG E-ACG/ M-CAA E-ACG/ M-CTA M-CTC E-ACG/ E-ACG/ M-CTT E-ACG/ M-CAT E-ACG/ M-CAG E-ACG/ M-GAC
20
Lampiran 4 Urutan primer pada gel poliakrilamid 1.
No Primer No Primer No Primer
1 M-CTC E-AAC 33 M-CTT E-ACA 65 M-CTC E-ACG
2 E-ACT 34 E-ACT 66 E-ACT
3 E-ACA 35 E-ACC 67 E-ACA
4 E-ACC 36 E-ACG 68 E-AAC
5 E-ACG 37 E-AAC 69 A-AGG
6 E-AAG 38 E-AAG 70 E-ACC
7 E-AGC 39 E-AGC 71 E-AGC
8 E-AGG 40 E-AGG 72 E-AAG
9 M-CAC E-AAC 41 M-CTA E-ACA 73 M-CAC E-ACG
10 E-ACT 42 E-ACT 74 E-ACG
11 E-ACA 43 E-ACC 75 E-ACG
12 E-ACC 44 E-ACG 76 E-ACG
13 E-ACG 45 E-AAC 77 E-ACG
14 E-AAG 46 E-AAG 78 E-ACG
15 E-AGC 47 E-AGC 79 E-ACG
16 E-AGG 48 E-AGG 80 E-ACG
17 M-CAT E-ACA 49 M-CTG E-ACA 81 M-CAT E-ACA
18 E-ACT 50 E-ACT 82 E-ACT
19 E-ACC 51 E-ACC 83 E-ACC
20 E-ACG 52 E-ACG 84 E-ACG
21 E-AAC 53 E-AAC 85 E-AAC
22 E-AGC 54 E-AAG 86 E-AAG
23 E-AGG 55 E-AGC 87 E-AGC
24 E-AAG 56 E-AGG 88 E-AGG
25 M-CAA E-ACA 57 M-CAG E-ACA 89 M-CAG E-ACA
26 E-ACT 58 E-ACT 90 E-ACT
27 E-ACC 59 E-ACC 91 E-ACC
28 E-ACG 60 E-ACG 92 E-ACG
29 E-AAC 61 E-AAC 93 E-AAC
30 E-AGC 62 E-AAG 94 E-AAG
31 E-AGG 63 E-AGC 95 E-AGC
21 Lampiran 5 Urutan primer pada gel poliakrilamid 2.
22
24
RIWAYAT HIDUP
Penulis merupakan putri sulung dari dua bersaudara yang dilahirkan pada tanggal 11 Juni 1992 di Paser, Tanah Grogot, Kalimantan Timur dari pasangan ayahanda Suparno, SH. MH dan Ibunda Budi Eko Sarwani, SPd. Tahun 2010 penulis menyelesaikan pendidikan sekolah menengah atasnya di SMA N 3 Surakarta, Jawa Tengah, dan melanjutkan pendidikan strata 1 di Institut Pertanian Bogor di Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam melalui jalur Undangan Seleksi Masuk IPB (USMI).
Selama menjalani masa studi penulis menjadi asisten praktikum Mikrobiologi Dasar dan Dasar-Dasar Biokimia pada tahun 2013. Penulis juga pernah aktif berorganisasi di Forum for Scientific Sudies (FORCES) selama 2 tahun (2010-2012) sebagai staf divisi service dan staf divisi Komunikasi dan
Informasi. Penulis juga aktif sebagai staf Kementerian Lingkungan Hidup Badan Eksekutif Mahasiswa Keluarga Mahasiswa IPB selama 2 tahun (2011-2013), dan di tahun 2014 menjadi sekretaris Kementerian Lingkungan Hidup BEM KM IPB serta tergabung dalam sahabat KS-Beriman IPB sejak tahun 2011-2014. Penulis juga aktif dalam berbagai kepanitiaan di luar organisasi dan juga tergabung dalam Organisasi Mahasiswa Daerah Surakarta-sekitarnya (Ayumas) sejak 2010-2014, UKM Panahan pada tahun 2010-2011, serta komunitas fotografi IPB (Shutter
IPB).
Penulis pernah melaksanakan magang di Balai Besar Biogen dengan penelitian awal kultur jaringan tanaman tebu Kidang Kencana, selama 2 minggu pada awal tahun 2013. Penulis juga telah melakukan praktik lapang di Laboratorium Teknologi, Balai Penelitian Bioteknologi, BPPT-Puspitek, Serpong, pada tahun 2013 dengan judul penelitian “Analisis GMO pada Makanan dan Minuman Komerisal Berbahan Dasar Kedelai” . Tahun 2014 penulis telah melaksanakan proyek penelitian di Balai Penelitian Bioteknologi, BPPT-
Puspitek, dengan judul penelitian “Identifikasi Keragaman Genetik Klon Karet dengan Teknik Amplified Fragment Length Polymorphism (AFLP)”.
Prestasi yang pernah diraih penulis selama menjadi mahasiswa adalah finalis 20 besar foto terbaik tingkat nasional dalam lomba fotografi bertema