MAJESTY EKSA PERMANA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2015
KLASIFIKASI METAGENOM PADA KASUS IMBALANCED
DATA DENGAN METODE MAHALANOBIS DISTANCE
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi Metagenom pada Kasus Imbalanced Data dengan Metode Mahalanobis Distance Based Sampling adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
MAJESTY EKSA PERMANA. Klasifikasi Metagenom pada Kasus Imbalanced Data dengan Metode Mahalanobis Distance Based Sampling. Dibimbing oleh TOTO HARYANTO.
Metagenom merupakan materi genetis yang diambil secara langsung dari lingkungan tanpa melalui proses isolasi. Pengambilan unsur genetis secara langsung dari lingkungan mengakibatkan banyak organisme yang bukan menjadi subjek penelitian ikut terambil sehingga perlu dilakukan proses klasifikasi. Namun, proses klasifikasi terkendala kasus imbalance data pada data sampel. Tujuan dari penelitian ini adalah menerapkan metode mahalanobis distance based sampling untuk mengatasi masalah imbalance data pada proses klasifikasi fragmen metagenom. Proses ekstraksi fitur dilakukan dengan metode n-mers dan pembentukan classifier dilakukan dengan metode k-nearest neighbor. Berdasarkan hasil penelitian ini dapat diketahui bahwa akurasi rata-rata pada kelas minoritas setelah dilakukan penyeimbangan data mengalami peningkatan sebesar 6.72% untuk k = 3 dan 5.79% untuk k = 5. Adapun akurasi rata-rata pada kelas minoritas untuk k = 7 setelah dilakukan penyeimbangan justru mengalami penurunan sebesar 1.11%.
Kata kunci: imbalance data, k-nearest neighbor, mahalanobis distance based sampling, metagenom, n-mers.
ABSTRACT
MAJESTY EKSA PERMANA. Metagenome Classification in Imbalanced Data with Mahalanobis Distance Based Sampling. Supervised by TOTO HARYANTO.
Metagenome is genetic material obtained from the environment without going through isolation. Genetic material obtained from the environment may contain many organisms that are not the subject of research, so it requires classification process. However, the classification process is plagued by case of imbalance data on the sample. The purpose of this research is to apply mahalanobis distance based sampling method to overcome the problem of imbalance data on metagenome fragment classification process. Feature extraction is performed using n-mers and classifier building process performed by k-nearest neighbor. The evaluation results show that the average accuracy on minority class after balancing process balancing data increased by 6.72% for k = 3 and 5.79% for k = 5. The average accuracy of the minority class for k = 7 after balancing process decreased by 1.11%.
MAJESTY EKSA PERMANA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2015 Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI METAGENOM PADA KASUS IMBALANCED
DATA DENGAN METODE MAHALANOBIS DISTANCE
Penguji:
1 Aziz Kustiyo, SSi MKom
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah yang berjudul “Klasifikasi Metagenom pada Kasus Imbalanced Data dengan Metode Mahalanobis Distance based Sampling” dapat diselesaikan. Karya tulis ini bertujuan untuk menerapkan metode mahalanobis distance based samping (MDS) untuk mengatasi masalah distribusi data yang tidak seimbang pada proses klasifikasi. Penulisan karya tulis ini tidak lepas dari bantuan berbagai pihak, yaitu:
Kedua orang tua, Bapak Suharyono dan Ibu Mujinah atas segala dukungan yang telah diberikan.
Bapak Toto Haryanto SKom, MSi selaku dosen pembimbing skripsi yang telah memberikan banyak saran, bantuan dan koreksi sehingga penulis dapat menyelesaikan karya tulis ini.
Bapak Aziz Kustiyo, SSi MKom dan Bapak Dr Eng Wisnu Ananta Kusuma, ST MT sebagai dosen penguji.
Teman-teman laboratorium riset bioinformatika atas segala saran, dukungan dan bantuan dalam proses penyusunan karya tulis ini.
Seluruh rekan-rekan dari Departemen Ilmu Komputer atas segala saran dan dukungan dalam proses penyusunan karya tulis ini.
Semoga karya tulis ini dapat memberikan manfaat bagi perkembangan teknologi informasi, khususnya dalam bidang bioinformatika.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup 2
METODE PENELITIAN 3
Pengumpulan Data 3
Praproses Data 3
Ekstraksi Fitur 4
Menghitung Jarak Mahalanobis 4
Menentukan Threshold 5
Pembagian Data 5
Membentuk Classifier 6
Evaluasi 6
Implementasi 7
HASIL DAN PEMBAHASAN 7
Pengumpulan Data 7
Praproses Data 8
Ekstraksi Fitur 8
Jarak Mahalanobis 8
Menentukan Threshold 11
Pembagian Data 11
Membentuk Classifier 12
Evaluasi 13
SIMPULAN DAN SARAN 17
Simpulan 17
Saran 18
DAFTAR TABEL
1 Confusion matrix (Chen et al. 2009) 6
2 Komposisi data penelitian 7
3 Komposisi data setelah proses thresholding 11
4 Perbandingan data latih dan data uji sebelum penyeimbangan 12 5 Perbandingan data latih dan data uji setelah penyeimbangan 12 6 Confusion matrix level genus dengan k = 3, k = 5, dan k = 7 sebelum
14 PA, NA, dan OA sebelum penyeimbangan data 16
15 Akurasi data latih seimbang 17
DAFTAR GAMBAR
1 Alur penelitian 3
2 Ilustrasi n-mers dengan n = 7 4
3 Thresholding (Chen et al. 2009) 5 4 Hasil praproses data dengan jumlah fragmen 1000 dan panjang fragmen
400 bp 8
5 Contoh hasil proses ekstraksi fitur 8
6 Grafik distribusi jarak antarorganisme pada genus Lactobacillus dan
Streptococcus 9
7 Grafik distribusi jarak antarorganisme pada ordo Lactobacillales dan
Bacillales 9
8 Grafik distribusi jarak antarorganisme pada kelas Bacilli dan Clostridia 10 9 Grafik distribusi jarak antarorganisme pada filum Firmicutes dan
1
PENDAHULUAN
Latar Belakang
Hanya sebagian kecil dari mikroorganisme dapat dikulturkan di laboratorium atau dibiakkan dalam media tumbuh buatan. Sebagian besar masih belum dapat dikulturkan dengan teknologi isolasi dan kultivasi yang ada pada saat ini. Padahal mikrob yang tidak dapat dikulturkan ini kemungkinan menyimpan gen-gen baru yang dapat diaplikasikan dalam industri ataupun bermanfaat bagi peningkatan kesejahteraan manusia. Tetapi melalui pendekatan metagenome, peneliti dimungkinkan dapat mengekstraksi DNA dari sampel yang diambil langsung dari lingkungan tanpa perlu mengidentifikasi makhluk hidup yang menjadi sumber DNA (Helianti 2008). Banyak keuntungan yang diperoleh dengan menggunakan pendekatan metagenom dalam eksplorasi gen dari DNA, misalnya mendapatkan gen dengan sifat unggul. Namun, sampel yang diperoleh langsung dari lingkungan mengandung beraneka ragam organisme sehingga harus dilakukan klasifikasi sebelum diolah lebih lanjut.
Menurut Chen et al. (2009), imbalance data merupakan kondisi ketidakseimbangan jumlah instance antara dua buah kelas. Kelas mayoritas adalah kelas yang memiliki jumlah data yang lebih besar sedangkan kelas minoritas adalah kelas yang memiliki jumlah data lebih kecil kecil. Proses klasifikasi menggunakan classifier yang berasal dari kelas mayoritas sudah memberikan akurasi yang cukup tinggi, akan tetapi untuk kelas minoritas masih memberikan nilai error yang cukup tinggi (Chen et al. 2009). Pada umumnya yang menjadi objek utama dalam sebuah penelitian adalah kelas minoritas (Su et al. 2006). Oleh karena itu, diperlukan suatu metode yang dapat mengklasifikasikan suatu objek ke dalam suatu kelas dengan tepat.
Metode yang paling dasar untuk menanggulangi masalah imbalance data adalah randomoversampling dan random undersampling. Random undersampling dilakukan dengan menghapus instance dari kelas mayoritas sementara random oversampling dilakukan dengan menduplikasi instance dari kelas minoritas. Kedua teknik tersebut mampu menengani masalah imbalancedata. Namun kedua metode tersebut memiliki beberapa kelamahan. Metode random oversampling tidak efektif untuk meningkatkan proses pengenalan pada kelas minoritas dan meningkatkan waktu proses pembentukan classifier. Metode random undersampling berpotensi membuang instance dari kelas mayoritas yang dianggap penting (He dan Ma 2013).
2
Salah satu metode klasifikasi yang umum digunakan adalah k-nearest neighbor (KNN). Metode klasifikasi KNN berusaha untuk mencari k tetangga terdekat dari suatu objek dan menggunakan mayoritas vote untuk menentukan objek dari kelas tersebut. Performa dari metode KNN sangat dipengaruhi oleh nilai k yang ditentukan. KNN biasanya menggunakan euclidean distance sebagai metode pengukuran antara data uji dengan data latih. Meskipun ini sangat sederhana dan mudah untuk diimplementasikan tetapi masih bisa memberikan hasil yang cukup baik seperti metode klasifikasi yang lain (Song et al. 2007).
Oleh sebab itu dilakukan penelitian untuk mengatasi masalah imbalance data pada proses klasifikasi menggunakan metode MDS dan menggunakan metode KNN untuk melakukan evaluasi terhadap hasil penyeimbangan data. Penelitian ini merujuk pada penelitian yang telah dilakukan oleh Chen et al. (2009) namun menggunakan KNN sebagai metode untuk mengevaluasi terhadap hasil penyeimbangan data.
Perumusan Masalah
Proses klasifikasi sudah memberikan hasil yang baik untuk classifier dari kelas mayoritas, akan tetapi memberikan hasil yang kurang memuaskan untuk classifier dari kelas minoritas. Karena pada umumnya data pada kelas minoritas menjadi objek utama dalam suatu penelitian maka muncul pertanyaan bagaimana cara meningkatkan akurasi dari classifier yang dihasilkan dari kelas minoritas.
Tujuan Penelitian
Tujuan dari penelitian ini adalah menerapkan metode MDS untuk mengatasi masalah imbalance data pada proses klasifikasi. Kemudian melakukan evaluasi hasil dari proses penyeimbangan data yang dihasilkan oleh metode MDS.
Manfaat Penelitian
Penelitian ini diharapkan dapat memberikan kontribusi dalam bidang bioinformatika terutama pada proses klasifikasi kasus imbalance data sehingga mampu meningkatkan akurasi dari kelas minoritas.
Ruang Lingkup
Lingkup dari penelitian ini, yaitu:
1 Data metagenom yang digunakan berasal dari National Centre for Biotechnology Information (NCBI).
2 Fragmen yang dihasilkan dari proses simulasi memiliki panjang yang tetap dan diasumsikan bebas sequencing error.
3
METODE PENELITIAN
Penelitian ini dilakukan melalui beberapa tahapan, yaitu pengumpulan data, praproses data, ekstraksi fitur, menghitung jarak mahalanobis, membentuk threshold, pembagian data, pembentukan classifier, evaluasi dan implementasi. Tahapan-tahapan yang dilakukan pada penelitian ini dapat dilihat pada Gambar 1.
Pengumpulan Data
Data yang digunakan pada penelitian ini adalah data metagenom yang diunduh dari situs NCBI ftp://ftp.ncbi.nlm.nih.gov/genomes/Bacteria/. NCBI merupakan suatu institusi yang fokus sebagai sumber informasi perkembangan biologi molekuler. Data metagenome yang digunakan merupakan sequence DNA organisme dengan format fasta.
Gambar 1 Alur penelitian
Praproses Data
4
terpilih. Pada saat simulasi menggunakan perangkat lunak MetaSim data dibaca sebanyak 1000 kali dengan panjang tiap dragmen 400 bp.
Ekstraksi Fitur
Metode ekstraksi fitur yang digunakan ialah n-mers. Metode ini memeriksa frekuensi kemunculan subsekuens nukleotida dari setiap fragmen DNA dengan panjang n. Fragmen DNA yang diperiksa dapat berupa basa jenis apapun, baik A, C, T, atau G. Jika n = 3, maka metode n-mers akan menghitung frekuensi kemunculan setiap subsekuens dari AAA sampai dengan GGG. Proses ekstraksi fitur menggunakan metode n-mers diilustrasikan pada Gambar 2.
Gambar 2 Ilustrasi n-mers dengan n = 7
Proses ekstraksi fitur dengan metode n-mers diawali dengan menentukan nilai n yang akan digunakan sebagai panjang subsekuens basa nukleotida yang akan dihitung frekuensinya. Nilai n akan mempengaruhi jumlah fitur yang didapatkan, semakin tinggi nilai n maka semakin banyak fitur yang diperoleh. Sebuah fragmen DNA akan dihitung frekuensi kemunculan subsekuens dari awal hingga akhir dengan metode sliding window.
Menghitung Jarak Mahalanobis
Mahalanobis distance adalah metode pengukuran jarak yang memperhatikan distribusi dari suatu objek yang ditandai dengan memperhitungkan matriks kovarian. Jarak mahalanobis digunakan dalam metode klasifikasi dengan mengukur jarak suatu objek terhadap pusat kelas (Varmuza dan Filzmoser 2009). Persamaan yang digunakan untuk menghitung jarak mahalanobis dapat dilihat pada Persamaan 1 (Varmuza dan Filzmoser 2009).
dmahalanobis= [(Za-Zb) C-1 (Za-Zb)T] .5 (1)
dengan Z merupakan vektor yang berisi nilai � yang dinormalisasi dan C− merupakan invers matriks kovarian dari fitur suatu level taksonomi.
5 variabel kontinu maka dilakukan proses normalisasi menggunakan Z-score standardization. Proses normalisasi dilakukan dengan Persamaan 2 (Larose 2005).
Z= xij-mean(xi)
std(xi) (2)
dengan
xij: fitur ke-j pada organisme ke-i.
mean(x�): rata-rata dari fitur organisme ke-i. std(x�): standar deviasi dari fitur organisme ke-i.
Menentukan Threshold
Langkah selanjutnya adalah menentukan threshold atau batasan yang berguna untuk memisahkan antara data pada kelas mayoritas dan data pada kelas minoritas. Pada tahap ini dilakukan penghapusan sampel mayoritas yang ada di luar titik threshold yang ditentukan. Proses thresholding diilustrasikan pada Gambar 3.
Gambar 3 Thresholding (Chen et al. 2009)
Titik threshold ditentukan dengan menggeser sebanyak jumlah sampel minoritas dari titik perpotongan antara sampel mayoritas dengan sampel minoritas ke arah sampel mayoritas. Proses thresholding bertujuan untuk menentukan instance dari kelas mayoritas yang akan dihapus atau dijadikan data latih baru. Pada proses ini data dari kelas mayoritas akan dikurangi berdasarkan distribusi jarak mahalanobis sedemikian sehingga jumlah instance kelas mayoritas sama dengan kelas minoritas. Proses ini diharapkan mampu memindahkan peluang kesalahan klasifikasi dari kelas minoritas ke dalam kelas mayoritas sehingga dapat meningkatkan akurasi pada kelas minoritas (Chen et al. 2009).
Pembagian Data
Total data yang digunakan terdiri atas 1088 organisme dari level genus, ordo, kelas dan filum. Proses penentuan data latih dan data uji dilakukan dengan membagi data menjadi 40% data uji dan 60% data latih. Data uji yang dipilih berasal dari level taksonomi yang sama namun dengan organisme yang berbeda dengan data latih.
6
Membentuk Classifier
Proses pembentukan classifier dilakukan dengan menggunakan metode KNN. KNN merupakan metode klasifikasi yang mengelompokkan data berdasarkan berdasarkan k tetangga terdekat dari data uji (Larose 2005). Nilai jarak antara data uji dan data latih diurutkan dari mulai yang terkecil sampai yang terbesar sejumlah nilai k yang ditentukan.
Metode KNN memiliki 3 tahapan utama (Song et al. 2007), yaitu: 1 Menentukan nilai k tetangga terdekat.
2 Menghitung jarak antara data uji dengan data latih.
3 Melakukan pengurutan data berdasarkan jarak terkecil sebanyak k.
Proses penghitungan jarak pada metode KNN dapat dilakukan dengan menggunakan euclidean distance. Jarak euclid dapat diperoleh dengan Persamaan 3 (Larose 2005).
Proses evaluasi akan dilakukan dengan menggunakan confusion matrix seperti ditunjukkan pada Tabel 1. Confusionmatrix dapat membantu dalam proses evaluasi karena menunjukkan kemampuan classifier dalam mengidentifikasi data uji.
Tabel 1 Confusion matrix (Chen et al. 2009)
Uji mayor Uji minor
Terdeteksi kelas mayor TP FN
Terdeteksi kelas minor FP TN
Performa dari classifier yang dihasilkan dievaluasi berdasarkan overall accuracy (OA), positive accuracy (PA), dan negative accuracy (NA). Possitive accuracy merupakan kemampuan classifier untuk mengklasifikasikan kelas mayoritas pada data uji. Proses penghitungan nilai PA ditunjukkan pada Persamaan 4 (Chen et al. 2009).
PA= TP+FNTP (4)
7
NA= FP+TNTN (5)
Overrall accuracy merupakan kemampuan classifier dalam mengidentifikasi keseluruhan objek dalam data uji. Proses penghitungan nilai OA ditunjukkan pada Persamaan 6 (Chen et al. 2009).
OA= TP+FP+TN+FNTP+TN (6)
Implementasi
Implementasi sistem akan dilakukan dalam lingkungan pengembangan sebagai berikut:
bahasa pemrograman : Python 2.7.
library komputasi : Biopython, Sklearn, Matplotlib, Numpy, Scipy. Sistem yang dikembangkan memiliki fungsi utama yaitu melakukan klasifikasi tingkat taksonomi pada suatu sequence DNA. Data masukan berupa sequence DNA dan keluarannya berupa klasifikasi berdasarkan tingkat taksonominya.
HASIL DAN PEMBAHASAN
Pengumpulan Data
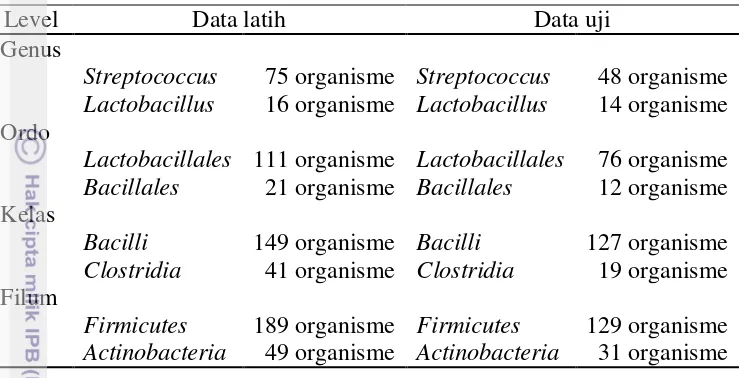
Data yang diunduh dari situs NCBI dengan alamat ftp://ftp.ncbi.nlm.nih.gov/genomes/Bacteria/ terdiri atas 1088 organisme. Komposisi data yang digunakan pada penelitian ini ditunjukkan pada Tabel 2.
Tabel 2 Komposisi data penelitian
Level Data tiap level taksonomi Jumlah Genus Streptococcus 123 organisme 153
Lactobacillus 30 organisme
Ordo Lactobacillales 187 organisme 220 Bacillades 33 organisme
Kelas Bacilli 257 organisme 317 Clostridia 60 organisme
8
Praproses Data
Pada tahap praproses data, sequence DNA metagenome akan diuraikan fragmennya menggunakan perangkat lunak MetaSim. Proses simulasi menghasilkan fragmen dengan panjang yang sama dan tidak mengandung sequencing error. Contoh hasil praproses data menggunakan perangkat lunak MetaSim dengan jumlah fragmen 1000 dan panjang fragmen 400 bp ditunjukkan pada Gambar 4.
Gambar 4 Hasil praproses data dengan jumlah fragmen 1000 dan panjang fragmen 400 bp
Ekstraksi Fitur
Proses ekstraksi fitur dilakukan dengan menggunakan metode n-mers dengan nilai n = 3 sehingga akan terdapat 64 kombinasi basa nukleotida mulai dari AAA sampai GGG. Frekuensi kemunculan basa nukleotida yang dihasilkan sudah terurut dari mulai AAA, AAC, AAG, AAT, sampai dengan GGG. Contoh hasil proses ekstraksi fitur ditunjukkan pada Gambar 5.
Gambar 5 Contoh hasil proses ekstraksi fitur
Jarak Mahalanobis
Data kelas mayoritas dan minoritas disatukan kemudian dihitung jarak antarorganisme menggunakan jarak mahalanobis. Metode ini diawali dengan menentukan nilai rata-rata dan standar deviasi dari setiap fitur organisme serta kovarian dari seluruh organisme dari level takson yang akan dihitung jaraknya. Banyak jarak yang terbentuk adalah ��2, dengan n adalah banyaknya organisme gabungan antara kelas mayoritas dan kelas minoritas.
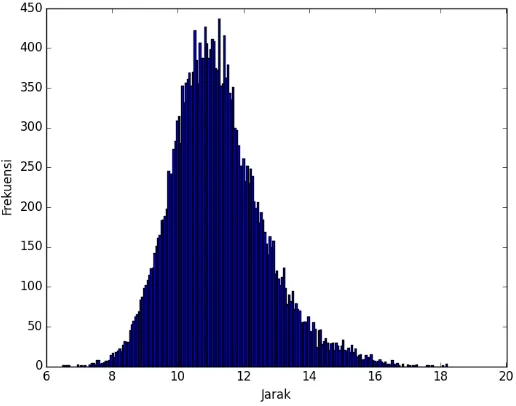
9 Pada level genus terdapat genus Lactobacillus sebagai kelas minortas dengan jumlah instance sebanyak 30 organisme. Adapun untuk kelas mayoritas terdapat genus Streptococcus dengan jumlah instance sebanyak 123. Grafik distribusi jarak antarorganisme pada genus Lactobacillus dan Streptococcus ditunjukkan pada Gambar 6.
Gambar 6 Grafik distribusi jarak antarorganisme pada genus Lactobacillus dan Streptococcus
Proses penghitungan jarak mahalanobis juga dilakukan pada level ordo. Pada level ordo terdapat ordo Lactobacillales sebagai kelas mayoritas dengan jumlah instance sebanyak 187 organisme. Adapun untuk kelas minoritas terdapat ordo Bacillales dengan jumlah instance sebanyak 33 organisme. Grafik distribusi jarak antara organisme pada ordo Lactobacillales dan Bacillales ditunjukkan pada Gambar 7.
10
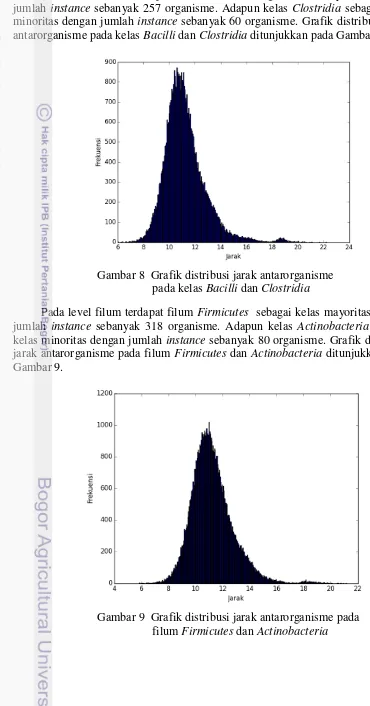
Pada level kelas terdapat kelas Bacilli sebagai kelas mayoritas dengan jumlah instance sebanyak 257 organisme. Adapun kelas Clostridia sebagai kelas minoritas dengan jumlah instance sebanyak 60 organisme. Grafik distribusi jarak antarorganisme pada kelas Bacilli dan Clostridia ditunjukkan pada Gambar 8.
Gambar 8 Grafik distribusi jarak antarorganisme pada kelas Bacilli dan Clostridia
Pada level filum terdapat filum Firmicutes sebagai kelas mayoritas dengan jumlah instance sebanyak 318 organisme. Adapun kelas Actinobacteria sebagai kelas minoritas dengan jumlah instance sebanyak 80 organisme. Grafik distribusi jarak antarorganisme pada filum Firmicutes dan Actinobacteria ditunjukkan pada Gambar 9.
11 Menentukan Threshold
Proses thresholding dilakukan untuk menghapus data yang sudah dipastikan masuk ke dalam kelas mayoritas dan menggabungkan data kelas minoritas dengan data kelas mayoritas yang masuk ke dalam threshold. Proses thresholding dilakukan berdasarkan sebaran jarak mahalanobis yang telah diketahui pada tahapan sebelumnya. Jumlah data yang diambil dari kelas mayoritas adalah sebanyak data pada kelas minoritas, sehingga didapatkan data latih yang seimbang. Namun jumlah pasangan jarak tidak sama dengan jumlah organisme yang ada, sehingga penentuan titik threshold dilakukan dengan pendekatan proporsi jumlah data kelas minoritas pada data gabuangan data antara kelas minoritas dan kelas mayoritas terhadap jumlah pasangan jarak yang terbentuk.
Data latih baru yang telah terbentuk masih berupa gabungan antara kelas mayoritas dan kelas minoritas, sehingga masih perlu dipisahkan untuk mendapatkan data latih kelas mayoritas yang baru. Perbandingan antara kelas mayor dan minor sudah sama sehingga hanya perlu membagi data tersebut menjadi dua bagian sesuai dengan grafik histogram data latih baru.
Data pasangan kelas mayor yang tersisa masih berupa pasangan jarak antarorganisme, sehingga pasangan tersebut perlu dipisahkan. Pemisahan dilakukan dengan memilih pasangan jarak sesama kelas mayoritas, kemudian dilakukan pemilihan organisme frekuensi kemunculan tertinggi sebagai data latih kelas mayoritas yang baru.
Tabel 3 menunjukkan perbandingan jumlah organisme pada tiap level taksonomi setelah dilakukan proses penyeimbangan data. Sudah tidak terdapat kesenjangan antara data pada kelas mayoritas dan kelas minoritas setelah dilakukan proses penyeimbangan data.
Tabel 3 Komposisi data setelah proses thresholding Level Data tiap level taksonomi Jumlah Genus Streptococcus 30 organisme 60
Lactobacillus 30 organisme
Ordo Lactobacillales 33 organisme 66 Bacillades 33 organisme
Kelas Bacilli 60 organisme 120 Clostridia 60 organisme
Filum Firmicutes 80 organisme 160 Actinobacteria 80 organisme
Pembagian Data
Proses pemilihan data uji dilakukan dengan mengambil 40% dari total data dalam satu level taksonomi yang sama. Adapun untuk data latih dilakukan dengan mengambil 60% dari dari data dalam satu level taksonomi yang sama. Dengan demikian, jumlah data uji antara sebelum dan sesudah proses penyeimbangan akan berbeda.
12
digunakan merupakan organisme selain data latih yang masih berada pada satu level taksonomi yang sama.
Tabel 4 Perbandingan data latih dan data uji sebelum penyeimbangan
Level Data latih Data uji
Genus
Streptococcus 75 organisme Streptococcus 48 organisme Lactobacillus 16 organisme Lactobacillus 14 organisme Ordo
Lactobacillales 111 organisme Lactobacillales 76 organisme Bacillales 21 organisme Bacillales 12 organisme Kelas
Bacilli 149 organisme Bacilli 127 organisme Clostridia 41 organisme Clostridia 19 organisme Filum
Firmicutes 189 organisme Firmicutes 129 organisme Actinobacteria 49 organisme Actinobacteria 31 organisme Pada Tabel 4 terlihat kesenjangan yang cukup besar antara jumlah instance pada kelas mayoritas dengan jumlah instance pada kelas minoritas. Adapun perbandingan antara data latih dan data uji setelah dilakukan proses penyeimbangan ditunjukkan pada Tabel 5. Pada Tabel 5 kesenjangan jumlah instance antara kelas mayoritas dan kelas minoritas sudah tidak terlalu mencolok.
Tabel 5 Perbandingan data latih dan data uji setelah penyeimbangan
Level Data latih Data uji
Genus
Streptococcus 20 organisme Streptococcus 10 organisme Lactobacillus 16 organisme Lactobacillus 14 organisme Ordo
Lactobacillales 19 organisme Lactobacillales 14 organisme Bacillales 20 organisme Bacillales 13 organisme Kelas
Bacilli 37 organisme Bacilli 23 organisme Clostridia 35 organisme Clostridia 25 organisme Filum
Firmicutes 49 organisme Firmicutes 31 organisme Actinobacteria 47 organisme Actinobacteria 33 organisme
Membentuk Classifier
13 Evaluasi
Evaluasi dilakukan dengan menghitung PA, NA, dan OA untuk setiap level taksonomi. Pada kasus ini PA dan NA merupakan kemampuan classifier untuk mengidentifikasi kelas mayoritas dan minoritas. Untuk mempermudah proses penghitungan PA, NA, dan OA digunakan confusion matrix. Confusion matrix untuk level genus dengan nilai k = 3, k = 5, dan k = 7 sebelum dilakukan penyeimbangan ditunjukkan pada Tabel 6.
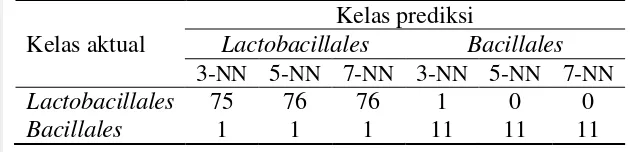
Dari Tabel 6 dapat diketahui bahwa genus Streptococcus dengan k = 3, k = 5, dan k = 7 dapat teridentifikasi dengan benar seluruhnya. Adapun untuk genus Lactobacillus dengan k = 3 dan k = 5 terdapat 9 organisme teridentifikasi dengan benar dan 5 organisme sebagai genus Streptococcus, sedangkan untuk k = 7 terdapat 8 organisme teridentifikasi dengan benar dan 8 organisme teridentifikasi sebagai genus Streptococcus.
Tabel 6 Confusionmatrix level genus dengan k = 3, k = 5, dan k = 7 sebelum dilakukan penyeimbangan
Kelas aktual diketahui bahwa keseluruhan data uji dapat diklasifikasikan dengan benar untuk genus Streptococcus untuk k = 3, k = 5, dan k = 7. Adapun untuk genus Lactobacillus terdapat 12 organisme teridentifikasi dengan benar dan 2 organisme sebagai genus Streptococcus.
Tabel 7 Confusionmatrix level genus dengan k = 3, k = 5, dan k = 7 setelah dilakukan penyeimbangan
14
Tabel 8 Confusionmatrix level ordo dengan k = 3, k = 5, dan k = 7 sebelum dilakukan penyeimbangan Kelas aktual organisme teridentifikasi dengan benar dan 1 organisme teridentifikasi sebagai ordo Bacillales. Adapun pada ordo Bacillales dengan k = 3 keseluruhan data uji dapat teridentifikasi sengan benar, k = 7 terdapat 12 organisme teridentifikasi dengan benar dan 1 organisme teridentifikasi sebagai ordo Lactobacillales sedangkan k = 7 terdapat 8 organisme teridentifikasi dengan benar.
Tabel 9 Confusionmatrix level ordo dengan k = 3, k = 5, dan k = 7 setelah dilakukan penyeimbangan
Kelas aktual dan 1 organisme teridentifikasi sebagai kelas Bacilli.
Tabel 10 Confusionmatrix level kelas dengan k = 3, k = 5, dan k = 7 sebelum dilakukan penyeimbangan
15 Tabel 11 Confusionmatrix level kelas dengan k = 3, k = 5,
dan k = 7 setelah dilakukan penyeimbangan Kelas aktual sebelum dilakukan penyeimbangan data ditunjukkan pada Tabel 12. Tabel 12 menunjukkan bahwa seluruh data uji pada filum Firmicutes dapat teridentifikasi dengan benar untuk k = 3 sedangkan untuk k = 5 dan k = 7 terdapat 1 organisme teridentifikasi sebagai filum Clostridia. Adapun pada filum Actinobacteria keseluruhan organisme dapat teridentifikasi dengan benar untuk k = 3, k = 5, dan k = 7.
Tabel 12 Confusionmatrix level filum dengan k = 3, k = 5, dan k = 7 sebelum dilakukan penyeimbangan Kelas aktual dilakukan penyeimbangan data ditunjukkan pada Tabel 13. Tabel 13 menunjukkan bahwa data uji pada filum Firmicutes dan filum Actinobacteria dapat teridentifikasi dengan benaruntuk k = 3, k = 5, dan k = 7.
16
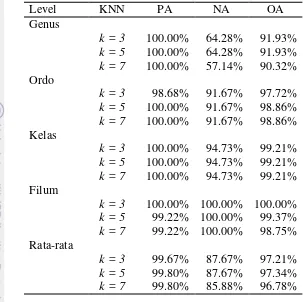
Tabel 14 PA, NA, dan OA sebelum penyeimbangan data
Level KNN PA NA OA tersebut disebabkan terdapat perbedaan yang sangat jelas pada frekuensi kemunculan kombinasi basa nukleotida antara filum Firmicutes dan Actinobacteria.
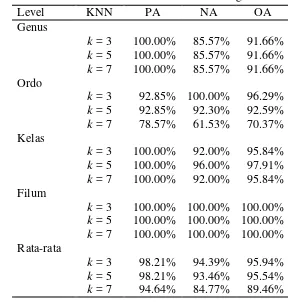
Adapun hasil penghitungan nilai PA, NA, dan OA setelah dilakukan proses penyeimbangan data ditunjukkan pada Tabel 15. Tabel 15 menunjukkan bahwa terjadi penurunan akurasi rata-rata dari kelas mayoritas dan peningkatan akurasi rata-rata kelas minoritas untuk k = 3, k = 5. Hal tersebut disebabkan oleh proses pengurangan jumlah data latih pada kelas mayoritas, sedangkan pada kelas minoritas tidak dilakukan pengurangan data latih. Adapun akurasi rata-rata untuk kelas minoritas dengan k = 7 setelah dilakukan proses penyeimbangan data justru mengalami penurunan. Hal tersebut diakibatkan karena penentuan niai k yang terlalu tinggi sehingga mengakibatkan bias pada proses klasifikasi. Kejadian serupa juga terjadi pada level ordo dan kelas, tingkat akurasi pada kelas minoritas setelah dilakukan penyeimbangan mengalami ketika nilai k = 7.
17 Tabel 15 Akurasi data latih seimbang
Level KNN PA NA OA
Berdasarkan penelitian yang telah dilakukan dapat disimpulkan bahwa: 1 Proses penanggulangan masalah imbalance data pada data latih telah berhasil
diterapkan dengan menggunakan metode MDS.
2 Pengujian menggunakan data latih yang sebelum dilakukan penyeimbangan data memberikan akurasi rata-rata kelas mayoritas lebih tinggi dibandingkan akurasi rata-rata kelas minoritas untuk nilai k. Pengujian data latih yang telah dilakukan penyeimbangan data menunjukkan bahwa akurasi rata-rata kelas mayoritas mengalami penurunan dan akurasi rata-rata kelas minoritas mengalami peningkatan untuk nilai k = 3 dan k = 5.
3 Setelah dilakukan proses penyeimbangan data pada kelas minoritas mengaami pengingkatan akurasi rata-rata sebesar 6.72% uuntuk k = 3 dan 5.79% untuk k = 5. Adapun untuk nilai k = 7 akurasi rata-rata kelas minoritas justru mengaami penurunan sebesar 1,11%.
18
5 Berdasarkan hasil pengujian proses penyeimbangan data mampu memindahkan peluang kesalahan klasifikasi pada kelas minoritas ke dalam kelas mayoritas (Chen et al. 2009).
Saran
Beberapa saran untuk penelitian selanjutnya yaitu: 1 Melakukan optimasi terhadap metode thresholding.
2 Menggunakan data latih dan data uji yang lebih besar untuk lebih mengetahui pengaruh penyeimbangan data latih.
3 Melakukan uji coba menggunakan metode klasifikasi lain misalnya SVM, logistic regression atau decision tree.
DAFTAR PUSTAKA
Chen LS, Hsu CC, Chang YS. 2009. MDS: a novel method for class imbalance learning, Di dalam: Proceedings of the 3rd International Conference on Ubiquitous Information Management and Communication; 2009 Jan 15 - 16; Suwon, Korea. New York (US): ACM. hlm 544-549.
He H, Ma Y. 2013. Imbalanced Learning: Foundations, Algorithms, and Applications. New Jersey (US): J Wiley.
Helianti. 2008. Metagenomik era baru bioteknologi [internet]. [diunduh 30 Mar 2015] Tersedia pada: http://biogen.litbang.pertanian.go.id/index.
php/2008/06/metagenomik-era-baru-bioteknologi/.
Larose DT. 2005. Discovering Knowledge in Data: An Introduction to Data Mining. 2nd ed.New Jersey (US): J Wiley.
Song Y, Huang J, Zhou D, Zha H, Giles CL. 2007. IKNN: informative k-nearest neighbor pattern classification. Di dalam: Knowledge Discovery in Databases: PKDD 2007. hlm 248-264.
Su CT, Chen LS, Yih, Y. 2006. Knowledge acquisition through information granulation for imbalanced data. Expert System with Applications. 31(3). 531-541.
19