UJI PERMUTASI EFEK SINERGIS BAHAN AKTIF
TANAMAN OBAT BERBASISKAN JEJARING DENGAN
PROTEIN TARGET
NUR HILAL A. SYAHRIR
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Uji Permutasi Efek Sinergis Bahan Aktif Tanaman Obat Berbasiskan Jejaring dengan Protein Target adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
NUR HILAL A. SYAHRIR. Uji Permutasi Efek Sinergis Bahan Aktif Tanaman Obat Berbasiskan Jejaring dengan Protein Target. Dibimbing oleh FARIT MOCHAMAD AFENDI dan BUDI SUSETYO.
Tanaman obat merupakan tanaman yang mengandung berbagai bahan aktif yang bermanfaat untuk mencegah dan menyembuhkan penyakit serta melakukan fungsi biologis tertentu. Berbeda dengan obat-obatan sintesis yang melibatkan satu senyawa kimia tunggal (one drug-one target), tanaman obat mengerahkan
efek yang menguntungkan dengan tindakan aditif atau sinergis dari beberapa senyawa kimia yang bekerja pada satu atau beberapa target ( multicomponent-network target) yang terkait dengan proses fisiologis. Suatu kombinasi bahan aktif
menunjukkan efek sinergis ketika efek bahan aktif secara bersama-sama lebih besar daripada efek masing-masing bahan aktif secara terpisah. Efek sinergis bahan aktif tanaman obat dapat diidentifikasi dengan pendekatan jejaring dengan protein target. Metode NIMS (Network target-based Identification of Multicomponent Synergy) adalah metode yang dapat digunakan untuk melihat
potensi efek sinergis dan menilai kekuatan sinergis antar kombinasi bahan aktif pada tingkatan molekular dengan mengkuantifikasi kekuatan sinergis bahan aktif yang disebut dengan skor sinergi.
Kajian efek sinergis diterapkan pada ramuan jamu yang sedang dikembangkan di Pusat Studi Biofarmaka untuk pengobatan diabetes mellitus tipe
2 yang terdiri atas 55 bahan aktif pada empat tanaman (Bratawali, Pare, Jahe, dan Sembung). Skor sinergi yang relatif tinggi ditunjukkan oleh pasangan bahan aktif antar tanaman Jahe. Efek sinergi antar tanaman yang berbeda namun mempunyai skor sinergi yang juga relatif tinggi adalah pasangan beberapa bahan aktif tanaman Jahe dan Pare, serta pasangan beberapa bahan aktif tanaman Bratawali dan Jahe. Berdasarkan hasil uji permutasi, skor sinergi tersebut signifikan.
SUMMARY
NUR HILAL A. SYAHRIR. Permutation Test Synergistic Effect of Active Ingredients of Medicinal Plants Based on Network of Protein Target. Dibimbing oleh FARIT MOCHAMAD AFENDI dan BUDI SUSETYO.
Medicinal plants contain inherently active ingredients. Such ingredients are beneficial to prevent and cure diseases, as well as to perform specific biological functions. In contrast to synthetic drugs, which is based on one single chemicals (one drug- one targrt), medicinal plants exert their beneficial effects through the additive or synergistic action of several chemical compounds. Those chemical compound act on single or multiple targets (multicomponent-network target) associated with a physiological process. Active ingredients combinations show a synergistic effect. This means that the combinational effect of several active ingredients is greater than that of individual one acting separately. A network target can be used to identify synergistic effects of plants active ingredients. The method of NIMS (Network target-based Identification of Multicomponent Synergy) is a computational approach to identify the potential synergistics effect of active ingredients. It also assessess synergistic strength of any active ingradients at the molecular level by synergy scores.
We investigate these synergistic on a Jamu formula for diabetes mellitus
type 2. The Jamu formula is composed of four medicinal plants, namely Tinospora crispa , Zingiber officinale, Momordica Charantia, and Blumea balsamivera. Our work succesfully demonstrates that the highest synergy scores
on medicinal plants synergy can be seen in pairs of several active ingredients in
Zingiber officinale. On the other hand, the synergy of pairs of active ingredients in Momordica Charantia and Zingiber officinale posseses a relatively high score.
The same occurs in Tinospora crispa and Zingiber officinale. Permutation test
sucsesfully shows significance scores.
© Hak Cipta Milik IPB, Tahun 2011
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Statistika Terapan
UJI PERMUTASI EFEK SINERGIS BAHAN AKTIF
TANAMAN OBAT BERBASISKAN JEJARING DENGAN
PROTEIN TARGET
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
Judul Tesis : Uji Permutasi Efek Sinergis Bahan Aktif Tanaman Obat Berbasiskan Jejaring dengan Protein Target
Nama : Nur Hilal A. Syahrir NIM : G152130251
Disetujui oleh Komisi Pembimbing
Dr Farit Mochamad Afendi, M.Si
Ketua Dr Ir Budi Susetyo, MS Anggota
Diketahui oleh
Ketua Program Studi Statistika Terapan
Dr Ir Indahwati, M.Si.
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
Tanggal Ujian:
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Desember 2014 ini ialah jamu informatika, dengan judul Uji Permutasi Efek Sinergis Bahan Aktif Tanaman Obat Berbasiskan Jejaring dengan Protein Target.
Terima kasih penulis ucapkan kepada Bapak Dr Farit Mochamad Afendi M.Si dan Bapak Dr Ir Budi Susetyo MS selaku pembimbing serta Bapak Dr.Eng Wisnu Ananta Kusuma selaku penguji luar komisi. Penghargaan juga penulis sampaikan kepada Bapak Rudi Heryanto M.Si dari Departemen Kimia dan Pusat Studi Biofarmaka, yang telah membantu selama pengumpulan data dan berdiskusi banyak dengan penulis. Teman-teman sejawat di Departemen Statistika terkhusus Rizal Bakri dan Nurul Qomariasih atas kerjasama tim yang baik dalam membuat tesis ini. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 1
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 5
Efek Sinergis 5
Network target-based Identification of Multicomponent Synergy (NIMS) 5
DrugCIPHER-CS 5
Topology Score (TS) 5
Agent Score (AS) 8
Phenotype Similarity (Kesamaan Fenotipe) 8
Uji Permutasi 10
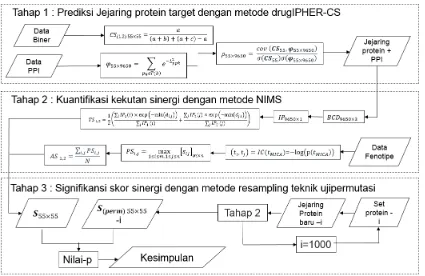
3 METODE 11
Data 11
Metode Analisis 11
Diagram Alir Penelitian 13
4 HASIL DAN PEMBAHASAN 15
Pengumpulan Data 15
Eksplorasi Data Awal 16
Prediksi Protein Target 19
Pendugaan Skor Sinergis 22
Pengujian signifikansi skor sinergis 28
5 SIMPULAN DAN SARAN 31
Simpulan 31
Saran 31
DAFTAR PUSTAKA 33
LAMPIRAN 35
DAFTAR TABEL
1 Pangkalan data, informasi yang diperoleh, dan hasi penelusuran data 15 2 Daftar bahan aktif pada tanaman, protein target, dan interaksinya 16 3 Tiga ukuran sentralitas jejaring dan skor kepentingan protein target 24
4 Peringkat skor sinergis Pare dan Bratawali 27
DAFTAR GAMBAR
1 Metode NIMS 5
2 Prinsipis dasar drugCIPHER 6
3 Contoh struktur Human Phenotype Ontology (HPO) 9 4 Ilustrasi metode resampling dengan teknik uji permutasi 10
5 Ilustrasi struktur data penelitian 11
6 Diagram alir penelitian 13
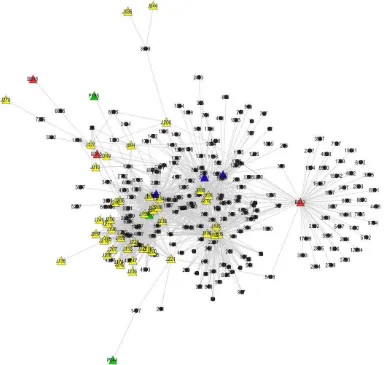
7 Jejaring bahan aktif protein target awal 17
8 Degree distribution 18
9 Jejaring Protein-Protein Interaction (PPI) 20
10 Prediksi jejaring bahan aktif dengan protein target 21
11 Degree dari protein target (drugCIPHER) 22
12 Plot betweenness,closeness, dan degree 23
13 Matriks Topology Score, Agent Score,dan Synergy Score 26 14 Skor sinergis bahan aktif pada tanaman Pare dan Bratawali 27 15 Ilustrasi sebaran hipotesis nol yang diperoleh melalui uji permutasi 28 16 P-value skor sinergis pasangan bahan aktif tanaman obat 29
DAFTAR LAMPIRAN
1 Daftar 55 bahan aktif yang terdapat pada empat tanaman 35
2 Interaksi bahan aktif dengan protein target 37
1
1
PENDAHULUAN
Latar Belakang
Pola penyakit di Indonesia bahkan di dunia telah mengalami pergeseran dari penyakit infeksi ke penyakit-penyakit metabolik degeneratif (Katno & Pramono 2008). Hal tersebut terjadi seiring dengan laju perkembangan tingkat ekonomi dan peradaban manusia serta perkembangan pola hidup yang serba praktis. Penyakit metabolik degeneratif merupakan penyakit yang disebabkan oleh gangguan metabolisme tubuh akibat konsumsi berbagai jenis makanan yang tidak terkendali serta gangguan faal tubuh sejalan dengan proses degenerasi. Beberapa contoh penyakit metabolik degeneratif antara lain: diabetes, kolesterol, hepatitis, dan rematik. Salah satu bentuk pengobatan yang diupayakan mengatasi penyakit metabolik degenaratif adalah dengan penggunaan atau konsumsi obat konvensional. Penggunaan obat konvensional dalam waktu yang lama dapat memberikan efek samping secara akumulatif dan menimbulkan dampak negatif terhadap kesehatan pasien. Berbeda dengan pengobatan berbasis tanaman obat (jamu), efek samping yang ditimbulkan relatif kecil meskipun dikonsumsi dalam waktu yang cukup lama. Selain itu, salah satu keuntungan lain dari pengobatan berbasis tanaman obat adalah kemampuannya yang memiliki lebih dari satu efek farmakologi sehingga penyakit-penyakit metabolik degenaratif yang dipicu oleh mutasi pada banyak gen lebih tepat ditangani dengan tanaman obat (Katno & Pramono 2008). Fenomena ini menjadi daya tarik beberapa peneliti untuk mengembangkan tanaman obat.
Kemampuan tanaman obat yang memiliki lebih dari satu efek farmakologi disebabkan mengandung lebih dari satu komponen bahan aktif atau senyawa aktif. Tanaman obat melibatkan banyak komponen senyawa kimia yang bekerja pada satu atau beberapa target (multicomponent-network target) berbeda dari obat konvensional yang hanya melibatkan satu senyawa kimia tunggal dengan satu target (one drug-one target) (Li & Zhang 2013). Karena terdiri dari banyak komponen senyawa kimia, maka interaksi antar komponen sangat mungkin terjadi. Interaksi antar bahan aktif pada tanaman obat dapat berupa sinergis atau antagonis. Evaluasi mengenai sinergisme bahan aktif pada tanaman obat biasanya dilakukan secara eksperimental dalam pendekatan kasus per kasus. Meskipun beberapa metode eksperimental telah dapat dilakukan untuk menyaring kombinasi bahan aktif yang menguntungkan, evaluasi sinergis secara eksperimental membutuhkan biaya dan tenaga yang besar serta waktu yang relatif lama. Identifikasi efek sinergis kombinasi bahan aktif yang banyak pada ramuan jamu tetap menjadi masalah yang belum terselesaikan. Pendekatan komputasi muncul sebagai alternatif yang mampu mengakomodasi data yang besar dengan lebih cepat serta dapat memberikan metode yang lebih menjanjikan dan diinginkan untuk studi obat multikomponen. Li et al. (2011) mengusulkan pendekatan komputasi untuk melihat potensi efek sinergis antar kombinasi bahan aktif pada tingkatan molekular dengan menggunakan metode NIMS (Network target-based Identification of Multicomponent Synergy).
2
merupakan metode yang tepat digunakan untuk melihat sinergi pasangan bahan aktif pada studi obat multikomponen. NIMS mengkuantifikasi kekuatan sinergis pasangan bahan aktif dengan mempertimbangkan keterkaitan mekanisme kerja obat yang melibatkan peran protein target serta peran penyakit. Li et al. (2011) menginisiasikan metode NIMS dan menunjukkan bahwa metode NIMS memiliki performa yang baik dalam mengkuantifikasi dan menyaring sinergisme bahan aktif pada formula TCM (Traditional Chinese Medicine). Beberapa penelitian sebelumnya menggunakan metode NIMS untuk mengungkap mekanisme kerja formula TCM pada penyakit tertentu seperti diabetes mellitus tipe 2 (Li et al. 2014) dan penyakit rematik (Li et al. 2015). Penelitian ini merupakan langkah awal evaluasi sinergis bahan aktif dengan pendekatan komputasi menggunakan metode NIMS pada ramuan Jamu.
Metode NIMS hanya menyediakan informasi mengenai kekuatan sinergi antar pasangan bahan aktif tanaman obat. Pasangan bahan aktif mana yang perlu ditelusuri lebih lanjut (signifikan) masih menjadi tantangan dalam pendekatan komputasi ini. Pendekatan statistika menjawab permasalahan tersebut dengan pengujian hipotesis. Pengujian hipotesis yang ada pada umumnya berangkat dari asumsi sebaran tertentu (pendekatan parametrik). Salah satu pendekatan non parametrik yang mampu menangani permasalahan pengujian signifikansi pada sebaran skor sinergi yang tidak diketahui memiliki sebaran tertentu ialah metode resampling. Metode resampling dengan teknik uji permutasi merupakan pendekatan yang tepat dalam menguji signifikansi hubungan yang melibatkan dua peubah. Ide dasar uji permutasi yang sederhana biasanya diterapkan pada pengujian signifikansi korelasi. Pada tulisan ini, ide dasar tersebut digunakan untuk melihat menguji signifikansi hubungan sinergis pasangan bahan aktif dengan melibatkan peubah yang diasumsikan menjadi faktor yang mempengaruhi sinergisme pasangan bahan aktif.
Perumusan Masalah
3
Tujuan Penelitian
Tujuan utama penelitian ini adalah:
1. Menduga kekuatan sinergis antar kombinasi bahan aktif melalui pendekatan komputasi berbasis jejaring dengan protein target dengan metode NIMS
2. Menguji signifikansi kekuatan sinergis antar kombinasi bahan aktif menggunakan metode resampling dengan teknik uji permutasi
Manfaat Penelitian
Penelusuran pasangan bahan aktif mana yang yang berinteraksi secara bersinergi dapat dimanfaatkan dari kajian ini. Penelitian ini merupakan inisiasi awal dalam upaya mengungkap mekanisme kerja jamu pada tingkatan molekular melalui pendekatan komputasi. Pendekatan komputasi yang digunakan pada penelitian ini mampu mengurangi biaya dan waktu penelitian dibandingkan dengan melakukan pendekatan eksperimental. Selain manfaat praktis, kajian teoritis statistika pada penelitian ini juga bermanfaat bagi kepentingan pengembangan ilmu pengetahuan dan teknologi.
Ruang Lingkup Penelitian
5
2
TINJAUAN PUSTAKA
Efek Sinergis
Efek sinergis pada bahan aktif merupakan kondisi ketika efek yang dihasilkan oleh bahan aktif secara bersama lebih besar daripada jumlah dari efek tunggal dari masing-masing bahan aktif (Spinella 2002). Evaluasi efek sinergi pada bahan aktif dapat diduga melalui pendekatan komputasi.
NIMS (Network target-based Identification of Multicomponent Synergy)
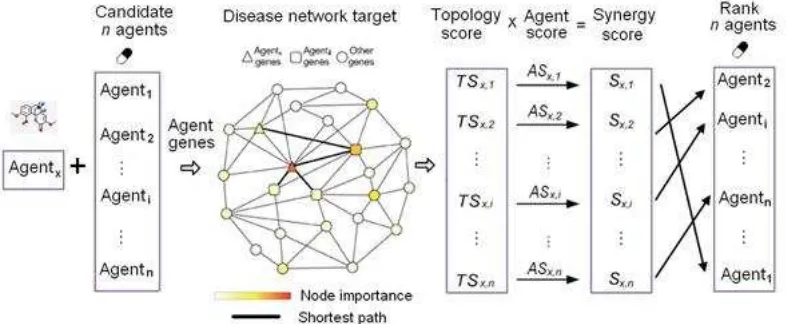
NIMS merupakan pendekatan komputasi untuk mengidentifikasi pasangan agen sinergis pada tingkat molekuler. Disini “agen” dapat mengacu pada substansi kimia seperti senyawa atau bahan aktif dengan aktifitas farmakologis atau bologis. Konsep dasar NIMS yaitu mentransfer hubungan antar bahan aktif ke dalam konteks topologi jejaring dengan memanfaatkan jejaring protein target dari bahan aktif dan jejaring PPI (Gambar 1).
NIMS mengintegrasikan dua aspek yaitu berdasarkan fitur topologi jejaring protein target bahan aktif dan kesamaan fenotipe (penyakit) dari protein target bahan aktif. Skor sinergis antar dua pasang bahan aktif diperoleh dari perkalian Topology Score( ) dan Agent Score ( ) S1,2=TS1,2 ×AS1,2.
Gambar 1. Metode NIMS (Li et al. 2011)
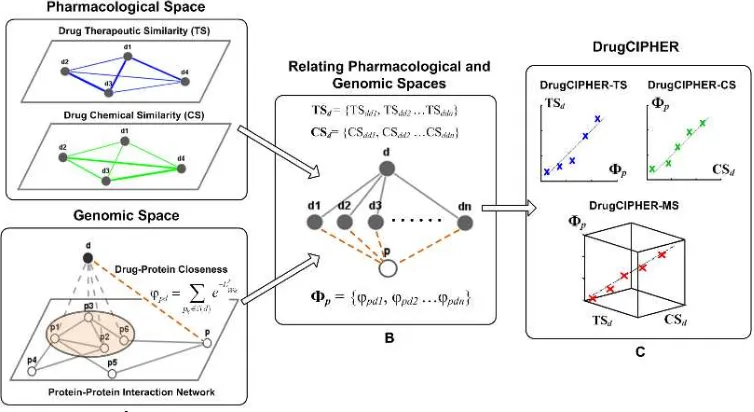
DrugCIPHER-CS
6
Gambar 2. Prinsip dasar drugCIPHER (Zhao & Li 2010)
Pada prinsipnya, drugCHIPER-CS melihat kedekatan bahan aktif terhadap protein target dalam skala genomik yang besar dengan mengkorelasikan informasi kemiripan struktur kimia bahan aktif pada ruang farmakologi dan interaksi antar protein pada tubuh manusia (PPI) pada ruang genomik. Metode ini didasarkan pada asumsi bahwa (i) obat dengan kemiripan struktur yang sama cenderung mengikat protein yang terkait (ii) hubungan fungsional antara protein dapat diukur dengan jarak pada jejaring interaksi protein. Profil protein target dari tiap bahan aktif dihitung dari skor concordance :
ρbp= cov CSb,φp σ CSb σ φp
dimana � merupakan matriks kemiripan struktur kimiawi antar bahan aktif dan � merupakan matriks kedekatan antara protein dan bahan aktif dengan basis jejaring PPI. Profil protein target untuk masing-masing bahan aktif merupakan 100 protein target dengan skor concordance tertinggi pada masing-masing bahan aktif. 100 Protein target dengan skor concordance tertinggi pada masing-masing bahan aktif diasumsikan sebagai likelihood (kemungkinan) dari bahan aktif menargetkan protein target . Profil target ini menjadi penyusun jejaring yang disebut jejaring protein target.
Kemiripan struktur kimiawi antar bahan aktif pada ruang farmakologi dihitung dengan mengubah struktur data dua dimensi bahan aktif menjadi data biner dengan bantuan 4860 fitur sidik jari molekuler unik yang merupakan substruktur berbagai senyawa melalui berbagai jenis teknik fragmentasi disebut sebagai sidik jari Kr. Data biner merupakan data yang berisikan dua nilai yaitu 0 dan 1. Nilai 0 menunjukkan ketidakhadiran substruktur pada bahan aktif dan nilai 1 menunjukkan kehadiran substruktur pada bahan aktif. Data biner antar dua bahan aktif kemudian dihitung kemiripannya berdasarkan koefisien kemiripan Tanimoto. Kemiripan dua bahan aktif berdasarkan koefisien Tanimoto:
7 dengan adalah frekuensi substruktur kedua bahan aktif bernilai 1, dan adalah frekuensi substruktur salah satu bahan aktif bernilai 1 dan lainnya bernilai 0. substruktur adalah frekuensi kedua bahan aktif bernilai 0.
Matriks kedekatan antara protein dan bahan aktif dengan basis jejaring PPI mengasosiakan ruang farmakologi dengan ruang genomik juga didefinisikan pada persamaan berikut ini: untuk mengkonversi jarak antar protein menjadi kedekatan protein. Persamaan ini menunjukkan bahwa kedekatan antara bahan aktif dan protein sama dengan penjumlahan dari kedekatan antara protein dan semua protein target dari bahan aktif . Jika dua protein tidak berhubungan, didefiniskan � = ∞.
Topology Score (TS)
berasal dari fitur topologi jejaring yang dibangun berdasarkan protein target bahan aktif dan interaksi antar protein. Ide dasar dari topology score adalah dengan melihat kedekatan bahan aktif berdasarkan kedekatan protein penting dari masing-masing bahan aktif dalam jejaring protein target. Berdasarkan perspektif jejaring target, diasumsikan bahwa semakin penting protein target sebagai node pada jejaring, maka semakin besar efek pengaruh bahan aktif terhadap protein target tersebut. Skor � dapat digunakan untuk menentukan seberapa penting protein target sebagai node dalam jejaring, dengan mengintegrasikan derajat (degree), keantaraan (betweenness), dan kedekatan (closeness).
Tiga indeks sentralitas jejaring tersebut digunakan untuk mendefinisikan sifat jejaring protein target secara terpisah atau bersama. Jika pasangan bahan aktif menghasilkan sinergi, seharusnya protein target dari bahan aktif tersebut berdekatan dalam jejaring. Dengan demikian untuk mengevaluasi skor kepentingan sebuah node � (�) dari protein target dari bahan aktif-1 , protein target dari bahan aktif -2, dan jarak jejaring antara dua set protein target tersebut,
8
Sentralitas Perantara Betweenness Centrality ( ) adalah ukuran sentralitas node dalam sebuah jejaring. Ukuran ini tidak hanya berisi informasi lokal namun mencerminkan peran sebuah node dalam struktur jejaring global. dihitung sebagai pembagian lintasan terpendek antara pasangan node yang melewati node yang diberikan. Untuk graf : = (�, ) dengan n node, dapat dituliskan sebagai
Sentralitas Kedekatan (Closeness Centrality) ( ) mengukur seberapa dekat node i terhadap semua node lain yang terdapat dalam jejaring yang didefinisikan sebagai rataan lintasan terpendek antara node i dan semua node lain yang dapat dicapai oleh node i:
Ci = nd (i, j) j∈V
di mana ( , ) adalah lintasan terpendek antara node i dan j, dan n adalah jumlah node dalam jejaring. Rataan lintasan terpendek didefinisikan sebagai rata-rata banyaknya langkah sepanjang lintasan terpendek untuk semua kemungkinan pasangan node pada jejaring. Ukuran ini menunjukkan efisiensi arus informasi dalam jejaring (Nacher & Schwartz 2008).
Agent Score (AS)
AS digunakan untuk mengukur nilai kesamaan antar pasangan bahan aktif berdasarkan fenotipe (penyakit) yang dihasilkan dari produk gen (protein target). Pasangan bahan aktif dengan mekanisme kerja yang independen namun mengobati penyakit yang sama akan lebih cenderung untuk menghasilkan efek sinergis. Jika protein target terdokumentasi memiliki eksperesi genetik berupa fenotipe dalam pangkalan data OMIM, fenotipe ini akan diidentifikasi sebagai agen fenotipe untuk agen yang diberikan. AS untuk pasangan bahan aktif-1dan bahan aktif-2
AS 1,2= i,jNPSi,j
Dimana � , adalah nilai kesamaan antara fenotipe-i dari bahan aktif-1 dan fenotipe-j dari bahan aktif-2, dan N adalah jumlah total pasangan fenotipe (Li et al. 2011).
Phenotype Similarity (Kesamaan Fenotipe)
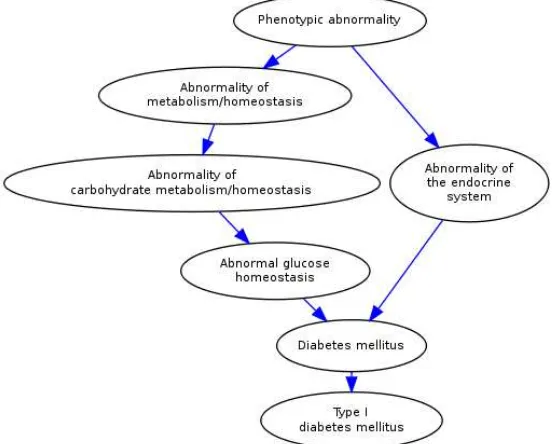
9 Ontology (HPO) menyediakan kosakata standar dan terkontrol yang disusun pada sebuah struktur HPO berbentuk directed acylic graph (DAG) atau biasa disebut multitree yang merupakan graf berarah tanpa siklus. Setiap individu dinamakan term. Struktur HPO terdiri atas lebih 10.000 terms dimana sekitar 99% dari HPO terms berada pada sub-ontologi Phenotypic Abnormality (PA). Struktur HPO memungkinkan terms untuk memiliki beberapa parent terms. Gambar contoh struktur HPO dapat dilihat pada Gambar 3.
Kesamaan antara dua fenotipe dapat dihitung berdasarkan kesamaan berpasangan dari dua set HPO term yang menjelaskan kedua fenotipe tersebut. Lord dalam Deng Y et al. 2015 mengusulkan metode untuk menggabungkan beberapa kesamaan antara dua terms menjadi satu kesamaan antara dua fenotipe dengan konsep maksimum. Misalkan fenotipe 1 dijelaskan oleh set HPO term
HPO1 = {t11, t12, . . . , t1 } dan fenotipe 2 dijelaskan oleh set HPO term HPO2 =
{t21, t22, . . . , t2 }. Maka matriks kesamaan terms T= tij m×n berisi semua skor
kesamaan berpasangan dari terms pada HPO1 dan HPO2. Kesamaan fenotipe dihitung dengan skor maksimum dari semua pasangan HPO terms pada dua set terms, didefinisikan sebagai berikut:
PSi,j= max1≤i≤m, 1≤j≤ntij
Cara yang lazim digunakan untuk mengukur kesamaan pada sebuah taxonomi ialah dengan cara mengevaluasi jarak antar node yang ingin dibandingkan. Semakin pendek lintasan dari satu node ke node yang lainnya, maka semakin besar kesamaan antar node tersebut. Resnik 2010 mengusulkan kesamaan antara dua node dengan konsep peluang serta mempertimbangkan sejauh mana kedua node tersebut memiliki informasi yang sama. Sehingga ukuran untuk mendefinisikan kesamaan antara dua terms ialah IC dari ancestors yang paling informatif untuk kedua terms.
HPO terms sim ti, tj =IC(tcommon)
10
dimana adalah ancestors yang paling informatif untuk term 1 dan term 2. IC dari terms t dalam HPO dapat didefinisikan sebagai berikut:
IC t =-log p t
dimana p adalah probabilitas pada terms yang diamati dan keturunannya di semua gen / penyakit yang terhubung dengan sub-ontologi tertentu pada HPO.
Uji Permutasi
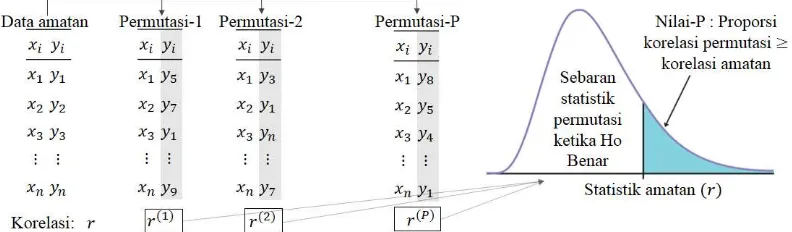
Pengujian adalah langkah penting dari statistik inferensia yang memungkinkan seseorang dalam menarik kesimpulan bahwa apakah dugaan (statistik) yang diperoleh dari data yang diamati merupakan cerminan dari populasi atau tidak. Dalam konteks pengujian skor sinergi yang diperoleh dari pendekatan komputasi, uji signifikansi dilakukan untuk melihat pasangan bahan aktif yang nyata bersinergi. Pengujian hipotesis pada skor sinergi tidak bisa dilakukan dengan pendekatan parametrik karena informasi sebaran skor sinergi yang tidak diketahui. Uji permutasi merupakan salah satu teknik untuk melakukan pengujian hipotesis dengan metode resampling pada data yang tidak memenuhi asumsi normal atau sebaran tertentu kecuali pengamatan yang menyebar independen dan identik pada sebaran hipotesis nol (Smyth dan Phipson 2010).
Uji permutasi dapat digunakan untuk menguji signifikansi hubungan yang melibatkan dua peubah (korelasi, regresi, dan hubungan lainnya yang terdifinisi dengan jelas). Ide dasar uji permutasi pada dua peubah ialah dengan mengubah urutan salah satu peubah dengan pengacakan secara konsisten pada data yang diamati. Sehingga jika diasumsikan bahwa kedua peubah mempunyai hubungan, maka diharapkan setelah dilakukan pengacakan maka hubungan antar dua peubah tersebut hilang. Hipotesis nol menyatakan tidak ada hubungan antar dua peubah. Ketika pengacakan dilakukan berulang-ulang maka diperoleh sebaran statistik uji yang merupakan kondisi sebaran hipotesis nol. Sebaran hipotesis nol sebaiknya dibangun dalam jumlah yang besar. � − � merupakan proporsi data resampling permutasi dengan nilai statistik uji ≥ statistik amatan (Hesterberg et al. 2003).
11
3
METODE
Data
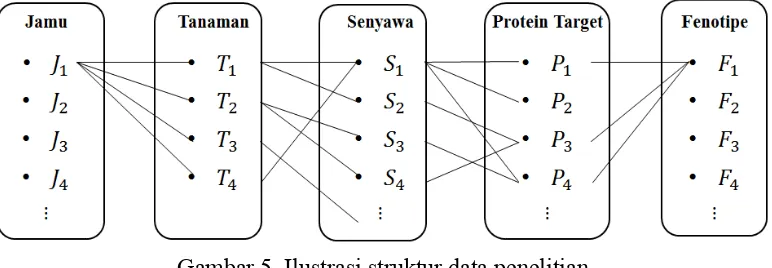
Penelitian ini berfokus pada ramuan jamu yang sedang dikembangkan di Pusat Studi Biofarmaka untuk pengobatan diabetes mellitus tipe 2. Struktur data yang digunakan secara komperehensif dapat diilustrasikan pada Gambar 5.
Gambar 5. Ilustrasi struktur data penelitian
Ramuan jamu terdiri atas empat tanaman yaitu tanaman Bratawali (Tinospora crispa), Jahe (Zingiber officinale), Pare (Momordica charantia), dan Sembung (Blumea balsamivera). Ramuan jamu tersebut merupakan hasil prediksi formula jamu baru yang berkhasiat sebagai antidiabetes oleh Nurishmaya (2014). Prediksi tersebut menggunakan model PLSDA, metode support vector machine (SVM), dan koefisien PLSDA multiways. Ramuan jamu dipilih sebagai upaya untuk mengungkap mekanisme kerja ramuan jamu yang telah dibuktikan khasiatnya sebagai ramuan jamu antidiabetes melalui uji di laboratorium. Khasiat dibuktikan menggunakan ikan zebra sebagai hewan uji.
Pengumpulan data dilakukan dengan mengumpulkan informasi dari berbagai sumber pangkalan data (Lihat 4.1 Tahapan Pengumpulan Data). Tahapan tersebut terdiri atas : 1) mengumpulkan daftar bahan aktif yang terkandung pada masing-masing tanaman formula jamu, 2) mengumpulkan daftar protein target yang terdokumentasi memiliki interaksi dengan bahan aktif, 3) mengumpulkan daftar fenotipe (penyakit) yang dihasilkan dengan terlibatnya protein target pada tahapan ke-dua, 4) mengumpulkan kosa kata terstandar mengenai fenotipe (penyakit) pada tahapan ke-tiga. Upaya ini merupakan pendekatan komputasi dalam mengukur seberapa mirip fenotipe (penyakit) satu dengan fenotipe (penyakit) lainnya (Lihat 2 Tinjauan Pustaka).
Metode Analisis
I. Prediksi protein target yang merupakan dugaan protein target dari bahan aktif menggunakan metode drugCIPHER-CS
a. Menghitung matriks kesamaan struktur kimiawi antar dua bahan aktif
� 55×55
12
sidik jari Kr. Data biner antar dua bahan aktif kemudian dihitung kemiripannya berdasarkan koefisien kemiripan Tanimoto, yaitu :
CSi,j=a+2(b+c)+da+b ;i=1,2,…,55 ;j=1,2,…,55
b. Menghitung matriks kedekatan antara protein dan bahan aktif �55×9650 φbp= e-Lppk2
pkϵT(b) c. Menghitung matriks skor concordance�55×9650
ρbp= cov CSb,φbp σ CSb σ φbp
d. 100 Protein target dengan skor concordance tertinggi pada masing-masing
bahan aktif diasumsikan sebagai likelihood dari bahan aktif menargetkan protein target .
II. Pendugaan skor sinergi 55×55 antar kombinasi bahan aktif menggunakan metode NIMS merupakan perkalian antara skor topologi (TS) dan skor agen (AS)
a. Menghitung skor topologi yang merupakan skor kedekatan protein target penting pada bahan aktif berdasarkan topologi jejaring
1. Membuat jejaring topologi yang terdiri atas jejaring protein target yang telah diprediksi pada langkah I dan jejaring PPI.
2. Menghitung tiga indeks sentralitas, 1×9650 derajat (degree), 1×9650 keantaraan (betweenness), 1×9650kedekatan (closeness).
Bi= (n-1)(n-2)1 ssts i st
s≠i≠t∈V dan Ci = nd (i, j)
j∈V dimana = 1,2,… 9650
3. Mengukur tingkat kepentingan node �1×9650 dengan menggunakan metode analisis komponen utama pada jejaring topologi berdasarkan tiga skor indeks sentralitas di atas.
IP1×9650=PCA ( B1×9650 , C1×9650 , D1×9650 )
4. Menghitung lintasan terpendek antar kombinasi bahan aktif min , dan
min ,
5. Menghitung matriks skor topologi 55×55
TS 1,2=12
b. Mengukur skor agen yang merupakan kesamaan bahan aktif berdasarkan fenotipe (penyakit) berdasarkan struktur HPO
1. Menghitung Information Content (IC) untuk setiap terms pada struktur HPO IC t =-log p t dimana = 1,2,…,1000
2. Mengukur kesamaan antar HPO terms ,
HPO terms sim ti, tj =IC(tcommon) = = 1,2,…,1000 3. Mengukur kesamaan antar fenotipe � 346×346
PSi,j= max1≤i≤m, 1≤j≤ntij
13 III. Pengujian signifikansi skor sinergi menggunakan menggunakan metode
resampling dengan teknik uji permutasi.
1. Identifikasi hipotesis. Hipotesis nol H0: Si,j=0 (bahan aktif-i dan bahan aktif-j tidak bersinergi) dan hipotesis alternatif H1: Si,j>0 (bahan aktif-i dan bahan aktif-j bersinergi).
2. Statistik uji pada dataset asli merupakan skor sinergi Saktual (55×55) yang diperoleh melalui pendekatan komputasi apada langkah II.
3. Menyusun ulang pengamatan
a. Menyusun set protein baru secara acak sehingga setiap bahan aktif memiliki pasangan set protein baru setiap kali pengacakan
b. Menghitung ulang skor sinergi
c. Mengulang langkah 3a dan 3b sebanyak 1000 kali sehingga diperoleh dataset matriks Spermutasi (55×55) sebanyak 1000
4. Menghitung proporsi setiap elemen Si,j (permutasi)≥ Si,j (aktual)
5. Menghitung p-value dengan membandingkan dataset asli dan 1000 dataset permutasi.
p-value = P Spermutasi ≥ Saktual 1000
i=1
/1000 6. Tolak 0 jika p-value < α
7. Menarik kesimpulan
Diagram Alir Penelitian
Metode analisis di atas dapat dilihat secara ringkas pada diagram alir penelitian di bawah ini:
15
4
HASIL DAN PEMBAHASAN
Pengumpulan Data
Pengumpulan data dilakukan dengan menelusuri beberapa pangkalan data yang menghimpun informasi dari publikasi ilmiah mengenai objek yang terkait. Data dikumpulkan dari berbagai pangkalan data. Tabel 1 mendeskripsikan berbagai jenis data, pangkalan data yang digunakan dalam penelusuran data tersebut, sebagian informasi yang diperoleh dari pangkalan data (khususnya informasi yang diperlukan dalam metode), dan hasil penelusaran pada pangkalan data.
Tabel 1. Pangkalan data, informasi yang diperoleh, dan hasil penelusuran data Jenis
data Pangkalan data Informasi yang diperoleh dari pangkalan data Hasil penelusuran data Senyawa KNApSAcK Bahan aktif PubChem Informasi lebih rinci
16
Jenis
data Pangkalan data Informasi yang diperoleh dari pangkalan data Hasil penelusuran data Protein target Universal
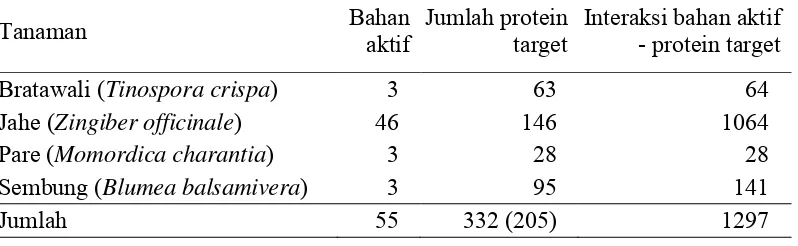
Prediksi protein target diawali dengan tahapan pengumpulan data awal yaitu daftar senyawa yang dikandung pada ramuan jamu pada pangkalan data KNApSAcK family dan The Dictionary of Natural Products (DNP). Penyaringan senyawa menghasilkan 55 senyawa aktif dari 595 senyawa yang dikumpulkan dari kedua pangkalan data tersebut. 55 bahan aktif tersebut diidentifikasi sebagai senyawa yang mempunyai aktifitas farmakologis (Lampiran 1). Masing-masing bahan aktif kemudian ditelusuri interaksinya terhadap protein target yang telah terdokumentasi pada pangkalan data PubChem. Daftar bahan aktif pada ramuan jamu, jumlah protein target yang dituju oleh bahan aktif, dan interaksi pasangan bahan aktif -protein target (Lampiran 2) secara ringkas disajikan pada tabel berikut:
Tabel 2. Daftar bahan aktif pada tanaman, protein target, dan interaksinya.
Tanaman Bahan aktif Jumlah protein target Interaksi bahan aktif - protein target
Bratawali (Tinospora crispa) 3 63 64
Jahe (Zingiber officinale) 46 146 1064
Pare (Momordica charantia) 3 28 28
Sembung (Blumea balsamivera) 3 95 141
Jumlah 55 332 (205) 1297
17 merupakan hubungan atau interaksi antar masing-masing individu yang dilambangkan dengan garis yang menghubungkan antar dua individu yang saling berhubungan. Dalam konteks farmakologi, sebuah node pada jejaring farmakologi biasanya menunjukkan: (i) gen, produk gen seperti : protein target atau entitas biologis pada jejaring biomolekuler (ii) tanaman, bahan aktif atau obat dalam jejaring ramuan; (iii) fenotipe klinis penyakit dalam jejaring fenotipe (Li & Zhang 2013). Edge pada jejaring farmakologi biasanya menunjukkan interaksi antara entitas gene, produk gen, bahan aktif, fenotipe, dsb.
Gambar 7 merupakan jejaring yang terdiri atas 55 node sebagai bahan aktif dan 205 node sebagai protein target unik. Protein target unik berasal dari 332 protein target yang tereduksi menjadi 205 protein target disebabkan oleh adanya protein yang overlapping antara protein target yang berasal dari bahan aktif pada tanaman yang berbeda. Masing-masing bahan aktif menargetkan paling sedikit sebanyak satu dan paling banyak sebanyak 57 protein target. Hubungan interaksi bahan aktif dan protein target tersebut dapat direpresentasikan sebagai edge pada jejaring. Jumlah interaksi antar bahan aktif dan protein target yang diperoleh dari dokumentasi pangkalan data PubChem sebanyak 1297.
18
Karakterisasi jejaring bahan aktif dengan protein target yang diperoleh dari bahan aktif pada empat tanaman dapat dilihat dengan degree distribution (Lihat Gambar 8). Degree distribution merupakan peluang dari sebuah node pada jumlah degree tertentu. Degree sendiri merupakan banyaknya node yang terhubung langsung dengan suatu node. Sebuah node pada jejaring dapat memiliki degree yang tinggi maupun degree yang rendah. Node yang memiliki degree yang tinggi pada umumnya jumlahnya sangat sedikit sementara node yang memiliki degree yang rendah pada umumnya jumlahnya banyak. Jejaring yang memiliki karakteristik tersebut ialah jejaring yang alamiah terjadi disebut juga scale-free network. Gambar 8 merupakan upaya dalam mengungkap karakter jejaring pada Gambar 7.
Pola scale-free network pada degree distribution dicirikan dengan power-law distribution dimana pola yang dibentuk adalah trend linier kebawah. Gambar 8(b) menunjukkan bahwa degree dari protein target mengikuti power-law distribution. Artinya, banyak protein target tertentu dituju oleh bahan aktif dalam jumlah sedikit sementara sedikit protein target yang dituju oleh bahan aktif dalam jumlah banyak. Sebaliknya, degree dari bahan aktif tidak menunjukkan pola scale-free network. Hal tersebut disebabkan oleh terbatasnya ketersediaan informasi kandungan bahan aktif tertentu dalam tanaman yang terkait dengan popularitas tanaman.
Ketersediaan bahan aktif pada tanaman seperti pada Tabel 2 terkait dengan popularitas tanaman. Jahe merupakan tanaman yang terdokumentasi mengandung banyak bahan aktif. Hal tersebut dikarenakan Jahe memiliki popularitas yang tinggi dibandingkan dengan tanaman lainnya. Penelitian ini merupakan inisiasi awal untuk menyaring pasangan bahan aktif yang bersinergi dengan pendekatan jejaring dengan protein target. Hasil yang diperoleh pada penelitian ini masih bersifat sementara seiring dengan berkembangnya penelitian lain yang memungkinkan bertambahnya jumlah daftar bahan aktif pada masing-masing tanaman tersebut.
19
Prediksi protein target dari bahan aktif
Prediksi protein target dari bahan aktif ramuan jamu diperoleh dengan menggunakan metode drugCIPHER-CS. Metode ini digagas oleh Zhao dan Li (2010) untuk memprediksi protein target dari bahan aktif dalam skala genomik yang besar dengan performa yang baik. Metode drugCHIPER-CS mengurutkan kemungkinan protein target tertentu yang menjadi target dari bahan aktif dengan mengkorelasikan kemiripan struktur kimiawi pada bahan aktif dan kedekatan antar protein target dalam jejaring PPI.
Kemiripan struktur kimiawi antar 55 bahan aktif dihitung dengan koefisien Tanimoto berdasarkan struktur 2D berupa canonical smiles yang diperoleh dari pangkalan data PubChem. Koefisien Tanimoto merupakan koefisen yang paling sering digunakan dalam mengukur kemiripan struktur kimiawi. Namun, tidak menutup kemungkinan masih terdapat koefisien lain yang memiliki performa yang lebih baik digunakan selain koefisien Tanimoto. Sidik jari molekular yang digunakan adalah sidik jari Klekota-Roth yang terdiri atas 4860 fitur. Selain sidik jari Klekota-Roth terdapat pula sidik jari PubChem yang terdiri atas 881 fitur, sidik jari estate 166 fitur, dan lainnya. Jumlah fitur sidik jari Klekota-Roth yang lebih banyak diharapkan mampu memberikan prediksi yang lebih akurat dalam mengukur indeks kemiripan struktur kimiawi.
Matriks kedekatan antara protein dan bahan aktif berbasis jarak antar protein dihitung dengan melihat jarak terpendek berdasarkan jejaring PPI (Lampiran 3). Jejaring PPI berbasis pangkalan data HPRD dapat dilihat pada Gambar 9. Jejaring PPI dibangun berdasarkan interaksi antar protein (PPI) sebanyak 37.080 dari 9.650 protein yang terdokumentasi pada Human Protein Reference Database (HPRD). Beberapa protein target hanya berinteraksi dengan sedikit protein target tertentu dan berinteraksi dengan protein yang juga tidak memiliki interaksi yang berinteraksi dengan protein target yang lain sehingga terpisahkan dari jejaring utama.
20
21
22
Degree dari bahan aktif (Lihat Gambar 11) menunjukkan pola scale free network. Artinya, banyak protein target yang dituju oleh bahan aktif yang sedikit dan sedikit protein target yang dituju oleh bahan aktif yang banyak berdasarkan hasil prediksi. Pola yang ditunjukkan oleh degree dari protein target yang telah diprediksi lebih mulus dibandingkan dengan pola degree dari protein target yang berasal dari hasil dokumentasi pada Gambar 9 (b).
Gambar 11 Degree dari protein target (drugCIPHER)
Pendugaan Skor Sinergis
Untuk melihat potensi efek sinergis dari kombinasi pasangan bahan aktif pada tingkatan molekuler digunakan metode NIMS. NIMS merupakan pendekatan baru yang digunakan untuk mengukur kekuatan sinergis dari pasangan bahan aktif tertentu dari tanaman dengan menentukan interaksi protein target dari bahan aktif tersebut pada jejaring PPI. Kajian NIMS diterapkan pada 55 bahan aktif yang dikumpulkan secara manual pada empat tanaman yang telah diprediksi menuju atau menargetkan protein target tertentu (Lihat Tabel 1) yang yang terbentuk pada NIMS adalah jejaring prediksi protein target berbasis jejaring PPI.
Topologi jejaring pada NIMS digunakan untuk menghitung skor topologi yang merupakan ukuran kedekatan protein target dari masing-masing bahan aktif berdasarkan tingkat kepentingan protein tersebut dalam jejaring. Semakin penting protein target dalam jejaring maka semakin kuat pula efek yang dihasilkan oleh bahan aktif yang menargetkan protein tersebut. Jejaring topologi NIMS dibangun dari prediksi jejaring protein target dari bahan aktif berbasis jejaring PPI namun node yang menjadi pusat perhatian adalah 1055 node yang diperoleh dari prediksi drugCIPHER-CS. Jejaring PPI dimanfaatkan untuk melihat node tersebut secara komprehensif melihat skor kepentingan suatu node.
Tiga ukuran sentralitas yang digunakan untuk melihat skor tingkat pentingnya protein target sebagai sebuah node di dalam jejaring yaitu, betweenness, closeness, dan degree. Gambar 12 menunjukkan plot antara ketiga ukuran sentralitas pada 9.673 protein target pada jejaring PPI. Ukuran degree menunjukkan fitur jejaring yang umum digunakan dan hanya mampu menangkap informasi lokal dari sebuah node dengan melihat tetangga terdekat sementara ukuran sentralitas betweenness tidak hanya berisi informasi lokal namun
23 mengidentifikasi sebuah node dalam sebuah struktur jejaring global (Nacher dan Schwartz 2008). Gambar 12 menunjukkan bahwa protein target dengan ukuran sentralitas betweenness yang tinggi juga mempunyai jumlah degree yang tinggi. Hal tersebut mengindikasikan adanya korelasi linear positif antara ukuran betweenness dan ukuran degree pada jejaring ini. Semakin banyak jumlah edge atau jumlah protein target tetangga yang terhubung dengan protein target tertentu semakin besar kecenderungan protein tersebut mempunyai pengaruh pada struktur jejaring secara global. Ukuran yang lain yaitu ukuran kedekatan (closeness) merupakan ukuran seberapa dekat sebuah node tertentu terhadap node yang lainnya. Pada beberapa konteks, ukuran ini dapat dipahami sebagai ukuran seberapa cepat arus informasi yang dapat tersebar dari sebuah node tertentu ke semua node yang jauh pada jejaring.
Gambar 12 Plot betweenness, closeness, dan degree
24
Tabel 3. Tiga ukuran sentralitas jejaring dan skor kepentingan protein target HPRD 01859 2076572.0149 4.93915E-09 270 37.3263 05639 1627436.5529 4.93912E-09 247 31.1040 01819 1044518.2163 4.93913E-09 208 22.5823 01498 1097085.1307 4.93912E-09 172 21.4240 04078 879726.5869 4.93911E-09 209 20.7595 04380 964110.2090 4.93912E-09 182 20.4014 03333 1059068.0446 4.93908E-09 159 20.3584 00589 924379.8618 4.93913E-09 188 20.2427 00277 996849.1576 4.93912E-09 168 20.0906 02534 860588.8373 4.93911E-09 198 20.0058 00579 989001.2363 4.93912E-09 161 19.6602 05670 1013615.5288 4.93908E-09 148 19.3059 03221 895070.1069 4.93912E-09 166 18.8372 00150 743737.2228 4.93910E-09 193 18.4349 02995 780247.6140 4.93911E-09 152 16.8505 01822 765853.1845 4.93908E-09 154 16.7846 02437 768921.1592 4.93913E-09 150 16.6244 03382 771028.1936 4.93909E-09 145 16.4045 00592 877210.1434 4.93908E-09 117 16.2453
⋮ ⋮ ⋮ ⋮ ⋮
18482 0 2.60348E-09 0 0
Jarak terpendek antar protein target dari semua kemungkinan pasangan bahan aktif juga diukur untuk melihat kedekatan antara protein target satu dengan yang lain. Skor topologi mengintegrasi ukuran tingkat pentingnya protein target dan skor kedekatan antar protein target. Gambar 13(a) menunjukkan skor topologi dalam bentuk matriks heat map dari kombinasi 55 bahan aktif-x dengan 55 kandidat bahan aktif. Warna merah menunjukkan skor tertinggi dan warna kuning menunjukkan skor terendah. Pasangan kombinasi dari dua bahan aktif yang berbeda sebanyak 1430. Skor topologi berada pada rentang (0.0191;1). Skor topologi ini bergantung pada jejaring yang dibentuk. Jejaring protein target berbasis jejaring PPI bukan satu-satunya jejaring yang dapat dibentuk pada metode NIMS. Li et al. (2010) memanfaatkan jejaring protein target yang berbasis jejaring penyakit tertentu untuk melihat sinergi bahan aktif dalam mengobati penyakit tertentu, namun pada penelitian tersebut jejaring protein target yang digunakan bukan jejaring protein target yang diprediksi menggunakan metode drugCIPHER. Penggunaan jejaring protein target yang diprediksi melalui drugCIPHER diharapkan mampu memberikan hasil yang lebih presisi.
25 yang berasal dari Pare. Selain dari hubungan protein target bahan aktif Pare dan Sembung di atas, kecenderungan protein target pada bahan aktif Pare memiliki kedeketan dengan protein target pada bahan aktif tanaman Jahe. Jumlah identifikasi bahan aktif Jahe yang lebih banyak dari bahan aktif lainnya menunjukkan sebagian besar pasangan bahan aktif tanaman Jahe mempunyai kedekatan protein target. Bahan aktif dari tanaman Bratawali menunjukkan kedekatan protein targetnya dengan tanaman Jahe dan antar bahan aktif Bratawali sendiri.
Selain melihat skor topologi, skor sinergi dapat ditelusuri melalui sejauh mana kemiripan pasangan bahan aktif tanaman obat dalam hal mengobati penyakit tertentu yang dikuantifikasi dengan Agent Score seperti pada Gambar 13(b). Agent Score ini diperoleh dari identifikasi keterlibatan protein pada penyakit tertentu pada pangkalan data OMIM. Agent Score diperoleh dengan menghitung kemiripan pasangan fenotipe dari bahan aktif. Beberapa metode yang dapat digunakan untuk menghitung kemiripan pasangan fenotipe dengan memanfaatkan diagram HPO dan menerapkan metode text mining. Pada penelitian ini, ukuran kemiripan yang digunakan adalah ukuran standar (default) yang tersedia pada package HPOSim pada perangkat lunak R-Languange yaitu ukuran kemiripan Resnik untuk kesamaan HPO terms dan metode funSimMax untuk skor kemiripan fenotipe. Beberapa konsep peluang yang berbeda pada ukuran kemiripan lain yaitu Ukuran Lin, Jiang-Contrath, ukuran graph IC, dan ukuran Wang serta metode kemiripan fenotipe seperti Max, Mean, funSimAvg, dan BMA dapat digunakan sebagai bahan perbandingan untuk penelitian selanjutnya.
Agent Score dari 1430 pasangan bahan aktif berada pada rentang (0.105195; 0.353001). Kemiripan pasangan bahan aktif berdasarkan penyakit yang diobati memiliki skor yang lebih rendah dibanding berdasarkan skor kedekatan protein target. Informasi dari pangkalan data mengenai gen atau produk gen memang tidak selalu menunjukkan bahwa setiap gen atau produk gen memiliki atau mengeskpresikan fenotipe (penyakit) tertentu. Kemiripan bahan aktif dalam hal melihat kesamaan fenotipe ini lebih cenderung terlihat pada bahan aktif antar tanaman Jahe.
26
Gambar 13 (a) Matriks Topology Score (b) Matriks Agent Score (c) Matriks Synergy Score
Kandidat bahan aktif
Ba
ha
n
ak
tif
-x
Kandidat bahan aktif
Ba
ha
n
ak
tif
-x
Ba
ha
n
ak
tif
-x
Kandidat bahan aktif
(a) (b)
27
Formulasi jamu pada penelitian ini terdiri atas empat tanaman. Obat tradisional berbasis tanaman di Cina yang dikenal dengan TCM (Traditional Chinese Medicine) umumnya membuat formula jamu dengan prinsip kombinasi Jun-Chen-Zuo-Si. Prinsip kombinasi tersebut digunakan dalam pembagian peran masing-masing tanaman. Tanaman Jun bertindak sebagai tanaman obat yang mengobati penyakit utama, tanaman Chen bertindak untuk meningkatkan kinerja tanaman utama, tanaman Zuo-Shi bertindak sebagai tanaman yang mengurangi efek samping dari tanaman utama (Li & Zhang 2013). Jika diasumsikan bahwa tanaman utama disini ialah tanaman Bratawali atau Pare, maka skor sinergi tanaman tersebut jika berpasangan dengan empat tanaman lainnya (Gambar 14).
Potensi skor sinergi antar bahan aktif dengan Pare dan Bratawali sebagai tanaman utama dapat lebih jelas terlihat pada Tabel 4. Terdapat 324 pasangan
Bahan aktif Utama Kandidat bahan aktif Skor Sinergi Peringkat
B013 J127 0.1270 13
28
Tabel 4 menunjukkan bahwa bahan aktif yang berasal dari tanaman Bratawali memiliki peluang yang besar untuk bersinergi dengan bahan aktif yang terdapat pada tanaman Jahe. Contoh: Bahan aktif (-)-Secoisolariciresinol (B013) dengan bahan aktif 1,7-Bis(3,4-dihydroxyphenyl)-3,5-heptanediol; (3S,5S)-form, 3',3''-Di-Me ether (J127) dan seterusnya. Daftar bahan aktif dapat dilihat pada Lampiran 1.
Pengujian Signifikansi Skor Sinergi
Metode NIMS hanya menyediakan informasi mengenai kekuatan sinergi antar pasangan bahan aktif tanaman obat. Pasangan bahan aktif mana yang perlu ditelusuri lebih lanjut (signifikan) masih menjadi tantangan dalam pendekatan komputasi ini. Uji signifikansi pada skor sinergi bertujuan untuk melihat apakah pasangan bahan aktif signifikan bersinergi atau tidak. Skor sinergi yang diperoleh melalui pendekatan komputasi menggunakan metode NIMS, selanjutnya diuji menggunakan konsep uji permutasi dengan menyusun ulang dengan mengacak kumpulan protein target yang menjadi target dari suatu bahan aktif. Penyusunan kembali dan pengacakan pada set protein target pada masing-masing bahan aktif menghasilkan statistik dari pengulangan dengan pengacakan yang kemudian membentuk sebaran statistik dari skor sinergi. Metode NIMS dilakukan dengan pengulangan sejumlah 1000 kali dengan kumpulan protein target yang berbeda untuk masing-masing 1430 pasangan aktif. Setiap pasangan bahan aktif memiliki skor sinergi permutasi sebanyak 1000 skor. 1000 skor sinergi permutasi tersebut membentuk sebaran hipotesis nol. Skor sinergi permutasi selanjutnya dibandingkan dengan skor sinergi aktual yang diperoleh pada metode NIMS dengan kumpulan protein target asli. Sebaran statistik yang dibentuk untuk setiap pasangan bahan aktif digunakan untuk menguji hipotesis dan menarik kesimpulan pasangan bahan aktif mana yang signifikan. Penarikan kesimpulan pada pengujian hipotesis ialah tolak jika p-value < �.
Gambar 15 Ilustrasi sebaran hipotesis nol yang diperoleh melalui uji permutasi.
29
B
aha
n a
kti
f x
Kandidat bahan aktif
Skor sinergi permutasi yang diperoleh dari 1000 kali ulangan pada pasangan bahan aktif N-trans-Feruloyltyramine (B018) dan Shogaols; [8]-Shogaol (J097) ditunjukkan pada Gambar 15 sebagai ilustrasi. Skor sinergi permutasi diperoleh pada rentang nilai (0.013831546 ;0.049665892) dengan sebaran skor seperti pada Gambar 15. Skor sinergi aktual yang diperoleh untuk pasangan bahan aktif tersebut ialah 0.156777867 dan proporsi sebaran skor sinergi permutasi yang lebih besar daripada skor sinergi aktual pada metode NIMS ialah 0% sehingga p-value yang diperoleh pada sebaran tersebut yaitu 0.000000. P-value yang diperoleh kemudian dijadikan sebagai dasar untuk menolak hipotesis nol yang berarti bahwa pasangan bahan aktif N-trans-Feruloyltyramine (B018) dan Shogaols; [8]-Shogaol (J097) signifikan bersinergi. Hal yang sama dilakukan untuk 1430 pasangan bahan aktif pada ramuan Jamu. P-value yang diperoleh dari uji permutasi untuk menguji signifikansi antar 1430 pasangan bahan aktif pada ramuan jamu secara ringkas dapat dilihat pada Gambar 16. Pada taraf uji sebesar 5% maka pasangan bahan aktif nyata bersinergi jika p-value < 0.005.
30
Warna gelap pada gambar heatmap (Gambar 16) menunjukkan pasangan bahan aktif signifikan bersinergi (tolak hipotesis nol), sementara warna terang menunjukkan bahwa belum cukup bukti untuk mengatakan bahwa pasangan bahan aktif signifikan bersinergi. Hasil pengujian terhadap skor sinergi untuk skor yang relatif tinggi antar beberapa bahan aktif tanaman Jahe, antar pasangan beberapa bahan aktif pada tanaman Bratawali dan tanaman Jahe, pasangan beberapa bahan aktif tanaman Sembung dan tanaman Bratawali, pasangan beberapa bahan aktif tanaman Sembung dan tanaman Jahe, serta antar pasangan bahan aktif pada tanaman Pare dan tanaman Jahe menghasilkan skor sinergi yang signifikan pada taraf uji nyata 5%.
31
5
SIMPULAN DAN SARAN
Simpulan
Metode NIMS dapat digunakan sebagai langkah awal untuk menduga efek sinergi antar pasangan bahan aktif dengan pendekatan komputasi. Metode NIMS merupakan salah satu upaya mengungkap mekanisme kerja Jamu. Validasi skor sinergis dapat didekati dengan teknik uji permutasi. Skor sinergi yang relatif tinggi dan signifikan berdasarkan hasil uji permutasi antar pasangan bahan aktif pada tanaman penyusun ramuan jamu ditemukan pada pasangan bahan aktif antar tanaman Jahe. Efek sinergi antar tanaman yang berbeda namun mempunya skor sinergi yang juga relatif tinggi adalah pasangan beberapa bahan aktif tanaman Jahe dan Pare, pasangan beberapa bahan aktif tanaman Bratawali dan Jahe, pasangan beberapa bahan aktif tanaman Bratawali dan Sembung, serta pasangan beberapa bahan aktif tanaman Sembung dan Jahe.
Saran
33
DAFTAR PUSTAKA
Afendi FM, Okada T, Yamazaki M, Hirai-Morita A, Nakamura Y , Nakamura K , Ikeda S , Takahashi H , Altaf-Ul-Amin M, Darusman LK , Saito K, Kanaya S. 2012. KNApSAcK Family databases: Integrated metabolite– plant species databases for multifaceted plant research. Plant Cell Physiol. 53(2). 1-12.doi:10.1093/pcp/pcr165
Apweiler R, Bairoch A, Wu CH, et al. 2004. UniProt: the Universal Protein knowledgebase. Nucl. Acids Res. 32 (1). D115-D119.doi: 10.1093/nar/gkh131
Deng Y, Gao L, Wang B, Guo X. 2015. HPOSim: An R package for phenotypic similarity measure and enrichment analysis based on the Human Phenotype Ontology. PLoS ONE. 10(2): e0115692. doi:10.1371/journal.pone.0115692
Goel R, Harsha HC, Pandey A, Prasad TSK. 2012. Human Protein Reference Database and Human Proteinpedia as resources for phosphoproteome analysis. Mol Biosyst. 8(2). 453-463.doi:10.1039/c1mb05340j
Hamosh A, Scott AF, Amberger JS, Bocchini CA, McKusick VA. 2005 Online Mendelian Inheriantace in Man (OMMIM), a knowladgebase of human
Katno, Pramono S. 2008. Tingkat manfaat dan keamanan tanaman obat dan obat tradisional. Balai Penelitian Tanaman Obat Tawangmangu, Fakultas Farmasi, UGM. Diakses pada tanggal 09 Juni 2015 pada perpustakaan.depkes.go.id:8180/.../BK2008-G105.pdf
Li S, Zhang B. 2013. Traditional Chinese medicine network pharmacology: theory, methodology and application. Chinese Journal of Natural Medicines. 11(2): 0110−0120.
Li S, Zhang B, Zhang NB. 2011. Network target for screening synergistic drug combinations with application to traditional Chinese medicine. BMC Systems Biology. 5(1).1-13.
Li Y, Li R, Ouyang Z , Li S. 2015. Herb Network Analysis for a Famous TCM Doctor's Prescriptions on Treatment of Rheumatoid Arthritis. Evidence-Based Complementary and Alternative Medicine.2015. 1-9.doi:10.1155/2015/451319.
Li H, Zhao L, Zhang B, Jiang Y, Wang X,Guo Y,Liu H, Li S, Tong2 X. 2014. A Network Pharmacology Approach to Determine Active Compounds and Action Mechanisms of Ge-Gen-Qin-Lian Decoction for Treatment of Type 2 Diabetes. Evidence-Based Complementary and Alternative Medicine.2014.1-12.doi:10.1155/2014/495840.
34
Nacher JC, Schwartz JM. 2008. A global view of drug-therapy interactions. BMC Pharmacology, 85-14. doi:10.1186/1471-2210-8-5
Nurishmaya MR. 2014. Pendekatan bioinformatika formulasi jamu baru berkhasiat antidiabetes dengan ikan zebra (Danio rerio) sebagai hewan model [skripsi]. Bogor (ID):Departemen Kimia, Fakultas Matematika dan Ilmu PengetahuanAlam, Institut Pertanian Bogor.
Resnik P. 1999. Semantic similarity in a taxonomy: an information-based measure and its application to problems of ambiguity in natural language. 11. 95-130.
Running WE. 1993. Chapman and Hall dictionary of natural products on CD-ROM. J Chem Info Comp Sci.33.934–935.doi:10.1021/ci00016a603
Robinson PN, Mundlos S. 2010. The Human Phenotype Ontology Clin Genet. 77: 525-534. doi:10.1111/j.1399-0004.2010.01436.x
Smyth GK, Phipson B. 2010. Permutation P-values should never be zero: calculating exact p-values when permutations are randomly drawn. Statistical Applications in Genetics and Molecular Biology 9.1-12.
Spinella M. 2002. The importance of pharmacological synergy in psychoactive herbal medicines. Alternative Medicine Review. 7(2). 130-137.
Wang Y, Xiao J, Suzek TO, Zhang J, Wang J, Bryant SH. 2009. PubChem: a public information system for analyzing bioactivities of small molecules. Nucleic Acids Res. 2(8).1-11. doi:10.1093/nar/gkp456.
35 Lampiran 1. Daftar 55 bahan aktif yang terdapat pada empat tanaman
No ID CID BAHAN AKTIF TANAMAN
1 B013 65373 (-)-Secoisolariciresinol Tinospora crispa 2 B015 5280537 N-trans-Feruloyltyramine Tinospora crispa (L.)
Hook.f.et Thoms. 3 B018 158516 N-Formylanonaine Tinospora crispa Miers 4 J010 5275727 Gingerdiols; [6]-Gingerdiol,
3-Epimer Constit. of officinale Zingiber 5 J036 3473 Gingerols; [6]-Gingerol Constit. of Zingiber
officinale 6 J044 9795270 Gingerols; [6]-Gingerol,
Demethoxy Constit. of officinale Zingiber 7 J057 10976 2-Heptanol; (ξ)-form Zingiber officinale 8 J068 7406 Isopropylbenzene Trace constit. of ginger
oil (Zingiber officinale) 9 J091 6442560 Shogaols; [8]-Shogaol Constit. of grains of
paradise (Amomum melegueta) and (Zingiber officinale) 10 J127 11068834
1,7-Bis(3,4-
dihydroxyphenyl)-3,5-12 J153 31211
4-(3,4-Dihydroxyphenyl)-2-butanone; 3-Me ether Reputed pungent principle of Zingiber officinale (ginger). Also present in cranberry, raspberry and mango
13 J191 2537 (-)-Camphor Zingiber
officinale ROSC. 14 J192 2758 1,8-Cineole Zingiber officinale
15 J193 6654 alpha-Pinene Zingiber
officinale ROSC.
16 J194 14896 beta-Pinene Zingiber
officinale ROSC. 17 J195 159055 (+)-Camphor Zingiber officinale
18 J196 26049 3-Carene Zingiber officinale
19 J198 637566 Geraniol Zingiber officinale
20 J199 643820 Nerol Zingiber officinale
21 J200 11463 Terpinolene Zingiber
36
No ID CID BAHAN AKTIF TANAMAN
22 J205 5144 Safrole Zingiber officinale
23 J206 5281794 [6]-Shogaol Zingiber officinale
24 J207 1201518 Borneol Zingiber officinale
25 J208 6616 Camphene Zingiber
officinale ROSC.
26 J211 7463 p-Cymene Zingiber officinale
27 J212 6549 (R)-linalool Zingiber officinale 28 J213 7460 alpha-Phellandrene Zingiber officinale 29 J218 5281516 (E,E)-alpha-Farnesene Zingiber
officinale ROSC. 30 J221 222284 (-)-beta-Sitosterol Zingiber officinale 31 J224 440917 (+)-S-Carvone Zingiber officinale 32 J226 6321405 (-)-Isoborneol Zingiber officinale 33 J234 5284507 (E)-Nerolidol Zingiber officinale 34 J236 11230 4-Terpineol Zingiber officinale 35 J238 17100 alpha-Terpineol Zingiber officinale
36 J240 8163 2-Undecanone Zingiber
officinale ROSC.
37 J241 8914 Nonanol Zingiber officinale
38 J242 454 Octanal Zingiber officinale
39 J247 9862 6-Methyl-5-hepten-2-one Zingiber officinale
40 J248 8141 Nonane Zingiber officinale
41 J249 6442612 10-Shogaol Zingiber
officinale ROSC. 42 J250 82227 1R,5R-(+)-alpha-Pinene Zingiber
officinale ROSC. 43 J270 5318039 Hexahydrocurcumin Zingiber officinale
44 J274 638011 Citral Zingiber officinale
45 J275 356 Octane Zingiber officinale
46 J286 11552 3-Methyl-butanal Zingiber officinale
47 J287 1031 n-Propanol Zingiber officinale
48 J288 527 Propionaldehyde Zingiber officinale 49 J291 7909 Methyl isobutyl ketone Zingiber officinale 50 P044 90677200
5,19-Epoxycucurbita-6,23-diene-3,25-diol; (3β,5β,23E)-52 P195 44445582 Karaviloside I Momordica
charantia L. 53 S002 6448 2-Bornanol; (1S,2R)-form,
Ac Blumea balsamifera
54 S030 72 3,4-Dihydroxybenzoic acid Blumea balsamifera
37 Lampiran 2. Interaksi bahan aktif dengan protein target
No IDKU Nama Protein HPRD ID
1 B013 platelet-activating factor acetylhydrolase precursor [Homo sapiens] (gi: 270133071) 3407 2 B013 Matrix metalloproteinase-9 (gi: 269849668) 387 3 B013 sentrin-specific protease 8 [Homo sapiens] (gi: 262118306) 9784 4 B013 orexin receptor type 1 [Homo sapiens] (gi: 222080095) 3863 5 B013 Neuropilin 2 [Homo sapiens] (gi: 219520294) 3643 6 B013 NLRP3 protein [Homo sapiens] (gi: 219518789) 5915 7 B013 protein-arginine deiminase type-4 [Homo sapiens] (gi: 216548487) 5635 8 B013 photoreceptor-specific nuclear receptor [Homo sapiens] (gi: 216409728) 5132 9 B013 MAS-related GPR member X1 [Homo sapiens] (gi: 195969650) 6246 10 B013 Apoptotic peptidase activating factor 1 [Homo sapiens] (gi: 187952397) 3755 11 B013 Chain A, Human Bcl2-A1 In Complex With Bim-Bh3 Peptide (gi: 167013344) 3034 12 B013 insulin-degrading enzyme isoform 1 [Homo sapiens] (gi: 155969707) 894 13 B013 Prolyl endopeptidase-like [Homo sapiens] (gi: 153217451) 17189 14 B013 beta-adrenergic receptor kinase 1 [Homo sapiens] (gi: 148539876) 182 15 B013 Alkaline phosphatase, intestinal [Homo sapiens] (gi: 124376142) 1376 16 B013 SUMO1/sentrin specific peptidase 7 [Homo sapiens] (gi: 120538355) 7154 17 B013 neurotensin receptor type 1 [Homo sapiens] (gi: 110611243) 1218 18 B013 E3 ubiquitin-protein ligase Mdm2 isoform a [Homo sapiens] (gi: 89993689) 1272 19 B013 protein Mdm4 isoform 1 [Homo sapiens] (gi: 88702791) 4082 20 B013 Myeloid cell leukemia sequence 1 (BCL2-related) [Homo sapiens] (gi: 78070770) 8870 21 B013 integrin alpha-4 precursor [Homo sapiens] (gi: 67191027) 1894
⋮ ⋮ ⋮ ⋮
⋮ ⋮ ⋮ ⋮
38
Lampiran 3. Daftar interaksi antar protein (Protein-Protein Interaction)
No Protein-1 HPRD
ID Protein-2 ID HPRD Pubmed ID
1 ITGA7 02761 CHRNA1 00007 10910772
2 PPP1R9A 16000 ACTG1 00017 9362513,12052877
3 SRGN 01513 CD44 00115 9334256
9 ERBB2IP 06090 ERBB2 01281 11278603,10878805
10
16 MUC4 11829 ERBB2 01281 12434309,11687512,11598901
17 PICK1 16176 ERBB2 01281 11278603
28 PLCG1 01398 VAV1 01284 9891995,16467851
39
RIWAYAT HIDUP
Penulis dilahirkan di Ujung Pandang pada tanggal 25 Februari 1991 dan merupakan anak ke-2 dari 7 bersaudara dari bapak A. Syahrir Badaruddin dan Ibu St Takdirah. Tahun 2008 penulis lulus dari SMAN 1 Makassar, Sulawesi Selatan dan pada tahun yang sama penulis lulus seleksi masuk Universitas Hasanuddin (Unhas) melalui Jalur Penelusuran Prestasi dan Bakat (JPPB) dan diterima di Jurusan Matematika, Program Studi Statistika, Fakultas Matematika dan Ilmu Pengetahuan Alam serta lulus pada tahun 2012. Tahun 2013 penulis menjadi mahasiswa di Sekolah Pasca Sarjana Institut Pertanian Bogor (SPS IPB) pada Program Studi Statistika Terapan.