PENGARUH MUTASI TITIK TERHADAP TRAYEKTORI
UNFOLDING
DAN
REFOLDING
PROTEIN 1GB1
DELLA TIARAPUTRI ALDRIFISIA
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Pengaruh Mutasi Titik terhadap Trayektori Unfolding dan Refolding Protein 1GB1 adalah benar karya saya dengan arahan dari dosen pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
DELLA TIARAPUTRI ALDRIFISIA. Pengaruh Mutasi Titik terhadap Trayektori Unfolding dan Refolding Protein 1GB1. Dibimbing oleh TONY IBNU SUMARYADA dan SETYANTO TRI WAHYUDI.
Mekanisme pelipatan protein merupakan hal yang sangat penting untuk memahami berbagai proses biologi. Salah satunya adalah proses unfolding dan refolding protein 1GB1 yang telah termutasi pada salah satu residunya. Penelitian ini bertujuan untuk mempelajari pengaruh mutasi titik terhadap dinamika proses unfolding dan refolding protein 1GB1 serta membandingkan trayektori unfolding dan refolding protein 1GB1 yang telah dimutasi dan tidak dimutasi. Penelitian ini dilakukan menggunakan metode simulasi dinamika molekul yang terdiri dari beberapa tahapan diantaranya preparasi dan mutasi protein, minimisasi, pemanasan atau pendinginan, ekuilibrasi, dan production run. Simulasi unfolding dilakukan pada suhu 500 K selama 2 ns dan refolding pada suhu 200 K selama 20 ns. Mutasi titik yang dilakukan mengakibatkan meningkatnya kestabilan termal protein sehingga protein mutan membutuhkan waktu lebih lama baik untuk denaturasi maupun kembali ke native state. Proses unfolding dan refolding protein memiliki trayektori yang berbeda, simulasi refolding membutuhkan waktu yang jauh lebih panjang daripada simulasi unfolding agar struktur protein dapat kembali ke keadaan awal (native state).
Kata kunci: mutasi titik, protein 1GB1, refolding, simulasi dinamika molekul, unfolding
ABSTRACT
DELLA TIARAPUTRI ALDRIFISIA. Effect of Point Mutation on Unfolding-Refolding Trajectory of Protein 1GB1. Supervised by TONY IBNU SUMARYADA and SETYANTO TRI WAHYUDI.
Protein folding mechanism is crucial for understanding various biological processes. The unfolding and refolding process of protein 1GB1 and its mutant were studied in this research. The objectives of this research are to learn the effect of point mutation on unfolding and refolding process of protein by comparing the unfolding and refolding trajectory of protein 1GB1 and its mutant. Molecular dynamics simulations method which consisted of several steps including protein preparation and mutation, minimization, heating or cooling, equilibration, and production run were used in this research. Unfolding simulation at temperature of 500 K for 2 ns and refolding simulation at 200 K for 20 ns. The result of point mutation that had been performed was increasing thermal stability of the mutant protein so that the protein took longer time for denaturation and returned to the native state. Unfolding and refolding process had a different trajectory, refolding simulation took longer time than unfolding simulation so that protein structure could return to native state.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Fisika
PENGARUH MUTASI TITIK TERHADAP TRAYEKTORI
UNFOLDING
DAN
REFOLDING
PROTEIN 1GB1
DELLA TIARAPUTRI ALDRIFISIA
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan karya tulis sebagai salah satu syarat kelulusan di Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam IPB. Judul penelitian yang dilaksanakan sejak bulan September 2014 ini ialah Pengaruh Mutasi Titik terhadap Trayektori Unfolding dan Refolding Protein 1GB1.
Selama pengerjaan penelitian hingga penulisan karya tulis ini penulis banyak mendapatkan dukungan dan bantuan dari berbagai pihak. Untuk itu penulis menyampaikan terimakasih kepada:
1. Kedua orangtua tercinta Papa Rudy Aldrin, S.Pd, Mama Aida Fitria, S.Pd, serta Adik-adik Cindyana Aldrifisia dan Muhammad Radian Alfariz atas dukungan dan kasih sayang yang tidak pernah berhenti diberikan kepada penulis.
2. Bapak Dr Tony Ibnu Sumaryada selaku dosen pembimbing pertama sekaligus dosen pembimbing akademik penulis selama tiga tahun terakhir dan Bapak Dr Setyanto Tri Wahyudi sebagai dosen pembimbing kedua yang telah banyak memberi bantuan dan saran kepada penulis selama penelitian.
3. Bapak Heriyanto Syafutra, M.Si selaku dosen penguji yang telah memberi saran kepada penulis.
4. Seluruh dosen pengajar dan staf Departemen Fisika IPB.
5. Rekan penelitian penulis Amel dan Kak Nya serta teman-teman Fisika angkatan 46, 47, 48, dan 49 atas kerja samanya selama ini.
6. Teman-teman Himaswiss, IPMM Bogor, dan Kost Harmony 2 sebagai keluarga kedua penulis yang selalu menghibur selama kuliah di IPB.
7. Semua pihak yang tidak dapat penulis sebutkan satu per satu. Penulis berharap semoga karya tulis ini bermanfaat.
Bogor, Mei 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 1
Tujuan Penelitian 1
Hipotesis 2
TINJAUAN PUSTAKA 2
Protein 1GB1 2
Mutasi Titik 3
Unfolding-Refolding Protein 3
Simulasi Dinamika Molekul 4
METODE 5
Waktu dan Tempat 5
Alat 5
Prosedur Penelitian 5
HASIL DAN PEMBAHASAN 6
Analisis Struktur Sekunder Protein 6
Analisis Root Mean Square Deviation (RMSD) 9
Analisis Jari-jari Girasi 10
Analisis Solvent Accessible Surface Area (SASA) 11
Analisis Energi Konformasi 12
SIMPULAN DAN SARAN 12
Simpulan 12
Saran 13
DAFTAR PUSTAKA 13
LAMPIRAN 15
DAFTAR TABEL
1 Residu penyusun struktur sekunder protein 2
DAFTAR GAMBAR
1 Struktur protein 1GB1 2
2 Mutasi titik 3
3 Perubahan struktur sekunder protein selama simulasi (a) unfolding WT, (b) refolding WT, (c) unfolding mutan K4A, (d) refolding mutan K4A 7 4 Perubahan konformasi protein selama simulasi (a) WT, (b) mutan K4A 8 5 Nilai RMSD protein selama simulasi (a) WT, (b) mutan K4A 9 6 Nilai jari-jari girasi protein selama simulasi (a) WT, (b) mutan K4A 10 7 Nilai SASA protein selama simulasi (a) WT, (b) mutan K4A 11 8 Energi konformasi protein selama simulasi (a) WT, (b) mutan K4A 12
DAFTAR LAMPIRAN
1 Diagram alir penelitian 15
2 Karakteristik residu penyusun protein 1GB1 16
3 Preferensi konformasi residu penyusun struktur sekunder 17 4 Residu penyusun struktur sekunder protein setelah simulasi refolding
PENDAHULUAN
Latar Belakang
Protein merupakan senyawa biokimia yang kompleks yang terdiri dari rantai asam amino yang terlipat dengan cara tertentu. Proses pelipatan protein bertujuan agar protein dapat mencapai energi konformasi yang minimum.1 Selain itu, proses pelipatan ini merupakan suatu mekanisme kontrol dalam sel hidup.2 Protein sangat berperan dalam dunia biologi. Semua protein dibentuk dari dua puluh asam amino yang sama.3 Hal yang membedakan protein yang satu dengan yang lain adalah komposisi asam aminonya.4
Dalam berbagai proses biologi, pergerakan, struktur, dan mekanisme pelipatan protein menjadi hal yang sangat penting. Protein yang telah terdenaturasi (unfolded) dapat kembali ke keadaan awal (native state) berupa lipatan (folded). Beberapa protein akan mengalami perubahan konformasi yang berpengaruh terhadap fungsinya. Pengetahuan tentang mekanisme unfolding dan refolding protein dapat memberikan pemahaman tentang beberapa penyakit yang disebabkan karena gagalnya beberapa protein kembali melipat setelah terdenaturasi sehingga protein tersebut tidak dapat menjalankan fungsi biologisnya.5
Simulasi dinamika molekul dapat digunakan untuk mempelajari proses pelipatan protein secara detail hingga skala atomik.6 Salah satu protein yang dapat digunakan dalam simulasi dinamika molekul untuk mempelajari mekanisme unfolding dan refolding adalah protein G domain B1 (indeks Protein Data Bank: 1GB1). Protein 1GB1 merupakan protein kecil karena hanya memiliki 56 rantai samping (residu) dan merupakan protein immunoglobulin yang digunakan oleh organisme untuk melawan antibodi inang (manusia).7 Berbagai mutasi terbukti dapat mengubah stabilitas protein ini tergantung pada lokasi terjadinya mutasi.
Perumusan Masalah
1. Bagaimana pengaruh mutasi titik terhadap proses unfolding pada suhu 500 K dan refolding pada suhu 200 K dari protein 1GB1?
2. Apakah proses unfolding dan refolding dari mutasi titik protein 1GB1 memiliki trayektori yang sama?
Tujuan Penelitian
1. Mempelajari pengaruh mutasi titik terhadap dinamika serta proses unfolding pada suhu 500 K dan refolding pada suhu 200 K dari protein 1GB1.
2
Hipotesis
Protein 1GB1 yang telah dimutasi akan memiliki trayektori unfolding dan refolding yang tidak persis sama.
TINJAUAN PUSTAKA
Protein 1GB1
Protein G domain B1 merupakan protein yang sangat stabil dan kecil (memiliki 56 rantai samping atau residu). Protein ini adalah salah satu protein yang paling banyak dimodelkan untuk mengetahui proses pelipatan (folding) suatu protein.9 Protein 1GB1 termasuk dalam kelompok G Streptococcus yang
Tabel 1 Residu penyusun struktur sekunder protein 1GB1 Index Residu Struktur Sekunder Kode Residu
1 Coil M
2 - 8 β-sheet 1 T, Y, K, L, I, L, N
9 - 12 Turn G, K, T, L
13 - 19 β-sheet 2 K, G, E, T, T, T, E
20 - 22 Coil A, V, D
23 - 36 α-helix A, A, T, A, E, K, V, F, K, Q, Y, A, N, D
37 - 42 Coil N, G, V, D, G, E
43 - 46 β-sheet 3 W, T, Y, D
47 - 50 Turn D, A, T, K
51 - 55 β-sheet 4 T, F, T, V, T
56 Coil E
3 membantu suatu organisme melawan pertahanan inang melalui sifat pengikatan protein yang unik.7 Protein 1GB1 memiliki residu yang terdiri atas 1 α-helix, 4β -sheet, 2 coil, dan 2 turn yang dapat dilihat di Tabel 1.8 Struktur protein 1GB1 direpresentasikan pada Gambar 1. α-helix ditunjukkan oleh warna ungu, β-sheet berwarna kuning, coil warna putih, dan turn berwarna toska.
Mutasi Titik
Mutasi titik merupakan proses menghilangkan sebuah asam amino pada protein tanpa memberikan asam amino yang baru atau penggantian sebuah asam amino dengan asam amino yang lain.10 Mutasi titik dapat mempengaruhi sifat suatu protein seperti kehilangan fungsi dan interaksi secara parsial bahkan kehilangan fungsi protein secara total. Namun di berberapa kondisi mutasi titik ini sama sekali tidak berpengaruh terhadap struktur dan fungsi suatu protein.11 Pada penelitian ini mutasi titik dilakuan karena mutasi titik tidak mengakibatkan perubahan pada struktur protein melainkan hanya mengubah sifat protein tersebut.
Penggantian sebuah asam amino pada protein akan mempengaruhi stabilitas protein tersebut. Efeknya tergantung pada lokasi mutasi dan lingkungannya dalam struktur protein. Beberapa interaksi yang muncul akibat mutasi titik seperti pembentukan ikatan hidrogen baru, interaksi hidrofobik, atau efek pada pelipatan protein.12 Ilustrasi dari mutasi titik dapat dilihat pada Gambar 2.
Gambar 2 Mutasi titik13
Unfolding dan Refolding Protein
4
Unfolding dan refolding dapat direpresentasikan melalui 4 tahap diskret dan reversible yaitu
F↔H↔S↔U
F adalah keadaan folded, H adalah keadaan protein tanpa struktur sekunder namun dengan inti hidrofobik yang terbungkus, S adalah keadaan dengan inti yang terlarut sebagian, dan U adalah keadaan unfolded sempurna.
Proses unfolding dimulai dengan merusak ujung struktur sekunder secara cepat selama beberapa pikosekon. Selama proses ini berlangsung struktur sekunder protein tetap utuh dan inti hidrofobiknya tetap terbungkus dengan baik. Proses awal ini diakhiri dengan rusaknya struktur sekunder secara sempurna. Proses selanjutnya dimulai dengan rusaknya ikatan hidrogen namun inti hidrofobik tetap utuh. Proses selanjutnya yaitu inti hidrofobik mulai rusak dan ditembus oleh beberapa molekul air dari lingkungan sekitar. Molekul air tersebut terperangkap diantara rantai samping selama beberapa pikosekon sehingga inti hidrofobik dalam keadaan terlarut sebagian. Proses terakhir yaitu rusaknya inti hidrofobik secara sempurna sehingga protein dalam keadaan tanpa lipatan (terdenaturasi). Refolding merupakan kebalikan dari proses unfolding dimana penurunan suhu diterapkan pada protein dalam keadaan terdenaturasi.14
Simulasi Dinamika Molekul
Prinsip simulasi dinamika molekul adalah menjadikan perubahan koordinat atom sebagai fungsi dari waktu dan menghitung pergerakan atom-atom yang ada di dalam molekul. Yang dijadikan sebagai titik awal dari perhitungan ini adalah struktur tunggal yang biasanya dijadikan sebagai struktur ekuilibrium yang didapatkan setelah meminimalkan energi potensial. Kecepatan pergerakan atom akan meningkat secara perlahan dari nol hingga mencapai nilai yang sesuai dengan suhu tertentu. Suhu ini disebut sebagai suhu ekuilibrasi yang dibutuhkan untuk memastikan sistem menuju ke suhu yang diinginkan.15
Fungsi energi potensial yang mendasari simulasi dinamika molekul merupakan penjumlahan antara energi potensial yang terjadi akibat interaksi ikatan (bonded interaction) dengan energi potensial akibat interaksi non-ikatan (nonbonded interaction). Interaksi ikatan pada ikatan kovalen berupa stretching, bending, dan torsion dapat dijelaskan oleh hukum Hooke
Energi stretching dari semua ikatan kovalen dalam protein direpresentasikan oleh suku penjumlahan pertama, dimana Kb adalah konstanta regangan (stretching), b adalah panjang ikatan, beq adalah nilai keseimbangan panjang ikatan. Energi bending direpresentasikan oleh suku penjumlahan kedua yang dianalogikan mirip dengan parameter pada suku pertama. Suku ketiga merepresentasikan potensial rotasi.165
Penelitian ini dilaksanakan di Laboratorium Fisika Teori dan Komputasi, Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor dimulai dari bulan September 2014 sampai bulan April 2015.
Alat
Penelitian ini menggunakan peralatan berupa alat tulis, perangkat keras dan perangkat lunak. Perangkat keras terdiri atas komputer dengan spesifikasi Quad Core Processor (Intel Core i7), RAM 12 GB, Graphic Card NVIDIA Ge Force GTS 9400, dan sistem operasi LINUX Ubuntu 12.04. Perangkat lunak yang digunakan untuk simulasi adalah NAMD (Not Just Another Molecular Dynamics Program) versi 2.9. Preparasi, analisis, dan menampilkan animasi molekul protein menggunakan VMD (Visual Molecular Dynamics Program) versi 1.9.1. NAMD dan VMD merupakan program simulasi dinamika molekuler yang dikembangkan oleh Theoretical and Computational Biophysics Group di University of Illinois, Urbana-Champaign. Perangkat lunak lain yang digunakan untuk pengolahan data antara lain CatDCD versi 4.0, Ms. Excel 2010, dan Gnuplot 4.6.4.
Prosedur Penelitian
Preparasi dan Mutasi Protein
Data koordinat protein 1GB1 yang telah diunduh dari Protein Data Bank dengan nama file 1GB1.pdb dijadikan sebagai masukan awal proses preparasi dengan mengatur jumlah framenya menjadi sama dengan satu. Selanjutnya atom hidrogen yang terdapat pada protein dihilangkan terlebih dahulu.
Tahap selanjutnya yaitu koordinat protein digeser ke (0,0,0) agar mempermudah perhitungan. Lalu protein dilarutkan pada solvent berupa air dan di tempatkan pada solvation box berdimensi (65.3×54.2×49.9) Å dengan metode Periodic Boundary Condition (PBC) agar simulasi dilakukan dengan kondisi yang mendekati eksperimen. Beberapa residu protein ada yang bersifat polar yang berarti muatannya belum tentu sama dengan nol sehingga sistem perlu dinetralkan dengan penambahan NaCl dengan konsentrasi 0.15 mol/L. Penetralan ini bertujuan agar simulasi yang dilakukan hanya dipengaruhi oleh perubahan temperatur dan tidak dipengaruhi oleh faktor lain.
6
dengan Alanine). Penggantian residu ini dilakukan karena terbukti dapat mempengaruhi kestabilan termal protein 1GB1.8 Proses mutasi dilakukan menggunakan program VMD.
Simulasi Dinamika Molekul
Simulasi dinamika molekul dilakukan menggunakan program NAMD dengan NPT Ensemble (jumlah molekul, tekanan, dan suhu dijaga konstan). Simulasi ini diawali dengan minimisasi selama 100 ps dengan masukan awal adalah protein yang telah dipreparasi. Minimisasi bertujuan agar protein berada pada keadaan stabilnya yaitu pada saat energi terendahnya.8 Untuk mengoptimalkan waktu simulasi maka minimisasi dibagi menjadi empat tahap.
Proses unfolding diawali dengan pemanasan dan ekuilibrasi protein yang telah diminimisasi. Pemanasan dilakukan dari suhu 0 K sampai 500 K selama 1 ns. Proses ekuilibrasi dilakukan selama 60 ps menggunakan protokol Langevin.9 Tahap akhir dari proses unfolding adalah production run yang dilakukan pada suhu 500 K selama 2 ns.
Proses refolding merupakan kebalikan dari proses unfolding. Protein yang telah terdenaturasi diambil koordinatnya saat sebelum terdenaturasi secara total, kemudian didinginkan dan diekuilibrasi agar kembali ke native state. Pendinginan dilakukan pada suhu 200 K selama 20 ns. Selanjutnya ekuilibrasi dilakukan selama 60 ps dengan protokol yang sama dengan proses unfolding. Kemudian dilakukan production run pada suhu 200 K selama 20 ns.
Analisis Data
Hasil production run dari proses unfolding dan refolding terlebih dahulu dihilangkan solvation box nya agar analisis tidak melibatkan solvent yang digunakan selama simulasi. Protein tersebut kemudian dianalisis menggunakan program VMD. Hasil analisis unfolding dan refolding dari struktur wild-type (tanpa mutasi) dan mutan K4A kemudian dibandingkan untuk mengetahui pengaruh mutasi titik terhadap trayektorinya.
HASIL DAN PEMBAHASAN
Analisis Struktur Sekunder Protein
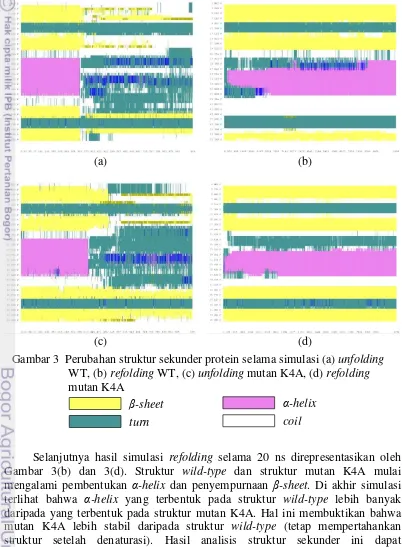
7 residu keempat (pada struktur β-sheet) memiliki karakteristik sebagai breaker dengan nilai preferensi 0.74, sedangkan alanine jika berada pada struktur β-sheet akan memiliki karakteristik sebagai weak former dengan nilai preferensi yang lebih tinggi dari pada lysine yaitu 0.97, sehingga penggantian lysine dengan alanine dapat meningkatkan kestabilan termal protein ini.
Lamanya waktu simulasi ditunjukkan oleh sumbu horizontal dari kiri ke kanan yaitu 0-2 ns. Sedangkan indeks residu protein ditunjukkan oleh sumbu vertikal dari atas ke bawah dimulai dari residu pertama sampai residu ke-56.
(a) (b)
(c) (d)
Selanjutnya hasil simulasi refolding selama 20 ns direpresentasikan oleh Gambar 3(b) dan 3(d). Struktur wild-type dan struktur mutan K4A mulai mengalami pembentukan α-helix dan penyempurnaan β-sheet. Di akhir simulasi terlihat bahwa α-helix yang terbentuk pada struktur wild-type lebih banyak daripada yang terbentuk pada struktur mutan K4A. Hal ini membuktikan bahwa mutan K4A lebih stabil daripada struktur wild-type (tetap mempertahankan struktur setelah denaturasi). Hasil analisis struktur sekunder ini dapat
Gambar 3 Perubahan struktur sekunder protein selama simulasi (a) unfolding WT, (b) refolding WT, (c) unfolding mutan K4A, (d) refolding mutan K4A
β-sheet turn
8
menunjukkan bahwa setiap residu pada protein berusaha mempertahankan struktur awalnya.8
(a)
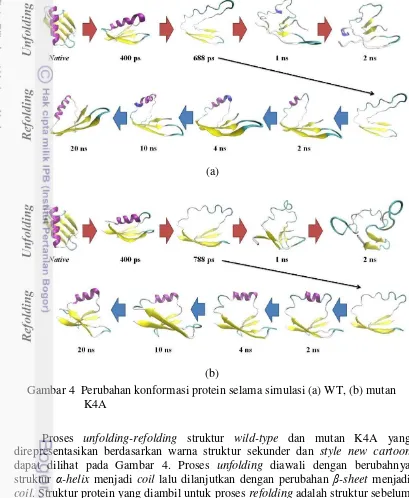
(b)
Proses unfolding-refolding struktur wild-type dan mutan K4A yang direpresentasikan berdasarkan warna struktur sekunder dan style new cartoon dapat dilihat pada Gambar 4. Proses unfolding diawali dengan berubahnya struktur α-helix menjadi coil lalu dilanjutkan dengan perubahan β-sheet menjadi coil. Struktur protein yang diambil untuk proses refolding adalah struktur sebelum protein tersebut mengalami denaturasi total karena saat suatu protein telah terdenaturasi secara total maka akan sangat susah untuk mengembalikannya menjadi seperti struktur awal (native state) berupa lipatan. Di akhir proses refolding, struktur wild-type yang terbentuk kembali memiliki kemiripan sebesar 85.7 % dengan struktur native state sedangkan struktur mutan lebih sedikit yaitu 80.4 %.
9 Analisis Root Mean Square Deviation (RMSD)
(a)
(b)
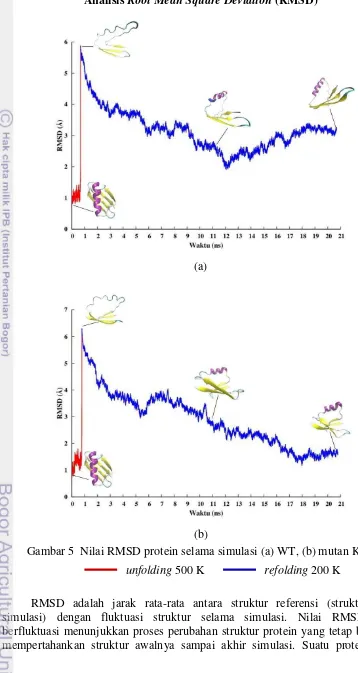
RMSD adalah jarak rata-rata antara struktur referensi (struktur awal simulasi) dengan fluktuasi struktur selama simulasi. Nilai RMSD yang berfluktuasi menunjukkan proses perubahan struktur protein yang tetap berusaha mempertahankan struktur awalnya sampai akhir simulasi. Suatu protein yang
10
berada pada native state memiliki nilai RMSD dibawah 1 Å.17 Proses unfolding ditandai dengan meningkatnya nilai RMSD. Struktur protein yang masih berada pada native state memiliki nilai RMSD kurang dari 2 Å, jika nilai RMSD berkisar antara 2 sampai 8 Å maka protein berada pada keadaan intermediate (denaturasi parsial), dan jika nilai RMSD sangat besar melebihi 8 Å maka protein telah mengalami denaturasi sempurna.18
Proses refolding struktur wild-type dimulai dengan mengambil koordinat protein pada keadaan intermediate saat nilai RMSD 5.880 Å (688 ps). Pada struktur awal ini protein wild-type yang telah terdenaturai parsial kehilangan seluruh struktur α-helix namun masih memiliki 3 β-sheet. Sedangkan proses refolding struktur mutan dimulai dengan mengambil koordinat protein saat nilai RMSD 6.278 Å (788 ps), struktur ini terdiri dari 4 β-sheet namun α-helix nya sudah berubah menjadi coil.
Gambar 5 menunjukkan bahwa proses unfolding protein pada suhu 500 K terjadi dalam selang waktu sangat cepat. Sedangkan proses refolding pada suhu 200 K memerlukan waktu yang lebih lama agar struktur protein yang telah terdenaturasi dapat kembali ke konformasi awalnya (native state). Proses unfolding mengakibatkan meningkatnya nilai RMSD secara signifikan. Sedangkan pada proses refolding, penurunan nilai RMSD terjadi secara perlahan.
Analisis Jari-jari Girasi
Jari-jari girasi merupakan indikator dari kekompakan struktur protein.19 Peningkatan jari-jari girasi menunjukkan semakin bertambahnya volume protein secara geometri yang berarti berkurangnya kerapatan dan kekompakan protein tersebut.
(a) (b)
Pada Gambar 6 terlihat bahwa mutasi titik K4A tidak mengakibatkan perubahan nilai jari-jari girasi secara signifikan. Nilai jari-jari girasi antara protein wild-type dan mutan mempunyai pola yang hampir sama. Pada proses unfolding nilai jari-jari girasi mengalami peningkatan yang sangat cepat. Hal ini merupakan indikasi terjadinya ekspansi molekul protein sehingga mengalami peningkatan volume. Berbeda dengan proses refolding yang membutuhkan waktu lebih lama
11 agar jari-jari girasi kembali turun sehingga kekompakan protein kembali meningkat dan strukturnya menjadi lebih rapat seperti native state. Dari Gambar 6 juga terlihat bahwa simulasi refolding yang dilakukan selama 20 ns masih belum cukup untuk membuat protein kembali memiliki kekompakan yang tinggi seperti native state karena nilai jari-jari girasi yang masih besar. Di akhir simulasi, nilai jari-jari girasi struktur mutan adalah 13.074 Å. Nilai ini lebih rendah daripada struktur wild-type yang bernilai 13.862 Å. Hal ini berarti protein mutan memiliki kekompakan yang lebih tinggi daripada wild-type.
Analisis Solvent Accessible Surface Area (SASA)
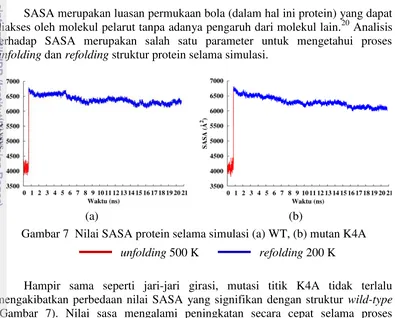
SASA merupakan luasan permukaan bola (dalam hal ini protein) yang dapat diakses oleh molekul pelarut tanpa adanya pengaruh dari molekul lain.20 Analisis terhadap SASA merupakan salah satu parameter untuk mengetahui proses unfolding dan refolding struktur protein selama simulasi.
(a) (b)
Hampir sama seperti jari-jari girasi, mutasi titik K4A tidak terlalu mengakibatkan perbedaan nilai SASA yang signifikan dengan struktur wild-type (Gambar 7). Nilai sasa mengalami peningkatan secara cepat selama proses unfolding yang berarti molekul pelarut (air) telah mengakses area dalam protein serta mulai rusaknya inti hidrofobik dari protein tersebut. Proses refolding bertujuan membentuk kembali inti hidrofobik yang utuh sehingga mengurangi luasan permukaan protein yang dapat diakses oleh molekul pelarut, namun proses ini membutuhkan waktu yang cukup lama dibandingkan dengan proses unfolding karena inti hidrofobik terbentuk secara perlahan.
Simulasi refolding selama 20 ns masih belum cukup agar inti hidrofobik protein terbentuk secara sempurna. Dibutuhkan simulasi dengan waktu yang lebih lama agar nilai SASA kembali seperti nilai SASA pada native state. Di akhir simulasi refolding terlihat bahwa mutasi K4A memiliki nilai SASA 6079.3 Å2. yang lebih rendah daripada struktur wild-type yang bernilai 6354.8 Å2.
12
Analisis Energi Konformasi
Energi konformasi merupakan energi total yang diperoleh dari penjumlahan antara energi ikatan, energi sudut, energi dihedral, dan energi akibat interaksi van der Waals antara atom-atom pada protein.21 Energi konformasi yang tinggi menandakan suatu keadaan kurangnya stabilitas suatu protein. Saat proses unfolding protein akan mengalami peningkatan nilai energi konformasi dan sebaliknya untuk proses refolding (Gambar 8(a) dan (b)). Mutasi titik K4A mengakibatkan protein memiliki energi konformasi yang lebih rendah daripada energi konformasi struktur wild-type, ini menunjukkan bahwa mutan K4A memiliki stabilitas termal lebih tinggi daripada struktur wild-type.
(a) (b)
SIMPULAN DAN SARAN
Simpulan
Pemberian suhu ekstrim terhadap protein dapat menyebabkan terjadinya proses unfolding (pada suhu tinggi) atau folding (pada suhu rendah). K4A merupakan salah satu mutasi titik yang dapat meningkatkan kestabilan termal pada protein 1GB1. Pengaruh mutasi titik ini ditinjau melalui beberapa analisis seperti struktur sekunder dan energi konformasi. Sifat stabil tersebut mengakibatkan mutan K4A terdenaturasi pada waktu yang lebih lama daripada struktur wild-type, selain itu juga struktur sekunder protein mutan yang terbentuk kembali setelah simulasi refolding mempunyai persentase yang lebih kecil daripada yang terbentuk kembali pada struktur wild-type. Kestabilan termal juga dapat dilihat dari nilai energi konformasi struktur mutan yang lebih rendah daripada struktur wild-type.
Trayektori unfolding dan refolding suatu protein tidak lah persis sama. Nilai RMSD, jari-jari girasi, SASA, dan energi konformasi mengalami peningkatan secara sangat cepat selama proses unfolding pada suhu 500 K, namun penurunannya selama proses refolding pada suhu 200 K membutuhkan waktu yang cukup lama.
13
Saran
Simulasi refolding protein pada suhu 200 K selama 20 ns belum menghasilkan kembalinya struktur protein ke native state secara sempurna. Penelitian ini perlu dilanjutkan untuk waktu simulasi refolding yang lebih lama agar didapatkan analisis yang lengkap terhadap proses refolding ini. Selain itu, variasi mutasi titik juga diperlukan untuk memahami pengaruhnya secara lebih rinci. Solvation box yang lebih besar diperlukan agar wadah pelarut tidak pecah selama simulasi unfolding pada suhu yang sangat tinggi.
DAFTAR PUSTAKA
1. Jain E, Gadia R, Mukerjee A. Probabilistic Road-Map based Protein Folding. [diunduh 2014 November 10]. Tersedia pada: www.cse.iitk.ac.in.
2. Sneppen K, Zocchi G. Physics in Molecular Biology. New York (US): Cambridge Univ Pr. 2005.
3. Tuszynski JA, Kurzynski M. Introduction to Molecular Biophysics. Basu D, editor. US: CRC Pr. 2003.
4. [PDB] Protein Data Bank. What is a Protein?. [diunduh 2014 November 9]. Tersedia pada: www.rscb.org.
5. Tapia L, Thomas S, Amato NM. A motion planning approach to studying molecular motions. Communications in Information and System. 2010; 10:53-68.
6. Wolf MG, de Leeuw SW. Fast in silico protein folding by introduction of alternating hydrogen bond potentials. Biophysics Journal. 2008; 94:3742-3747.
7. Gronenborn AM, Clore GM. Structural studies of immunoglobulin-binding domains of Streptococcal protein G. Immunomethods. 1993; 2:3-8.
8. Sawitri KN. Pengaruh mutasi terhadap kestabilan termal protein 1GB1 [tesis]. Bogor (ID): Institut Pertanian Bogor. 2014.
9. Jee J, Byeon IJL, Louis JM, Gronenborn AM. The point mutation A34F causes dimerization of GB1. Proteins: Structure, Function, and Bioinformatics. 2008; 71:1420-1431.
10. Palma R, Curmi PMG. Computational studies on mutant protein stability: The correlation between surface thermal expansion and protein stability. Protein Science. 1999; 8:913-920.
11. Eyal E, Najmanovich R, Edelman M, Sobolev V. Protein side-chain rearrangement in regions of point mutation. Proteins: Structure, Function, and Genetics. 2003; 50:272-282.
12. Johnsen K, O’Neill JW, Kim DE, Baker D, Zhang KYJ. Crystallization and
14
13. Becker OM, MacKerell AD Jr, Roux B, Watanabe M, editor. Computational Biochemistry and Biophysics. New York (US): Marcel Dekker. 2001.
14. Pande VS, Rokhsar DS. Molecular dynamics simulation of unfolding and refolding of a β-hairpin fragment of protein G. Proc. Natl. Acad. Sci. USA. 1999; 96:9062-9067.
15. Levitt M, Hirshberg M, Sharon R, Daggett V. Potential energy function and parameters for simulations of the molecular dynamics of proteins and nucleic acids in solution. Computer Physics Communication. 1995; 91:215-231.
16. Beckerman M. Molecular and Cellular Signaling. Tannessee (US): Springer Science. 2005.
17. Dokholyan NV, Li L, Ding F, Shakhnovich EI. Topological determinants of protein folding. PNAS. 2002; 99(13):8637-8641.
18. Sharma RD, Lynn AM, Sharma PK, Rajnee, Jawaid S. High Temperature unfolding of Bacillus anthracis amidase-03 by molecular dynamics simulations. Bioinformation. 2009; 3(10):430-434.
19. Lobanov MY, Bogatyreva NS, Galzitskaya OV. Radius of gyration as an indicator of protein structure compactness. Molecular Biology. 2008; 42(4):623-628.
20. Lee B, Richard FM. The interpretation of protein structure: Estimation of static accessibility. J. Mol. Biol. 1970; 55:379-400.
21. Chen Z, Lou J, Zhu C, Schulten K. Flow-indiced structural transition in the
15 Lampiran 1 Diagram alir penelitian
Mulai
Lakukan pemanasan
Ekuilibrasi
Lakukan production run
Data koordinat mutan 1GB1 setelah unfolding
Refolding
Analisis
Selesai Mutasi titik
Data koordinat 1GB1.pdb
Minimisasi Mutan 1GB1
Preparasi protein
Lakukan pendinginan
Ekuilibrasi
Lakukan production run Unfolding
Data koordinat mutan 1GB1 setelah refolding
Minimisasi Mutan 1GB1 unfolded
16
Lampiran 2 Karakteristik residu penyusun protein 1GB18
No Jenis Residu Residu Kode Nama Residu
1 Polar positif K Lysine
2 Polar negatif D Aspartic Acid
E Glutamic Acid
3 Polar netral T Threonine
N Asparagine Q Glutamine
4 Non polar G Glycine
5 Hidrophobik A Alanine
I Isoleucine L Leucine M Methionine
F Phenylalanine W Tryptophan
Y Tyrosine V Valine
6 Aromatik F Phenylalanine
17 Lampiran 3 Preferensi konformasi residu penyusun struktur sekunder8
α-helix β-sheet
Karakteristik Residu P Karakteristik Residu P Strong former A 1.45 Strong former I 1.60
E 1.53 M 1.67
L 1.34 V 1.65
Former F 1.12 Former C 1.30
H 1.24 F 1.28
M 1.20 L 1.22
Q 1.17 Q 1.23
V 1.14 T 1.20
W 1.14 W 1.19
Weak former K 1.07 Y 1.29
I 1.00 Weak former A 0.97
Indifferent C 0.77 Indifferent D 0.80
D 0.98 G 0.81
R 0.79 R 0.90
S 0.79 Strong breaker E 0.26
T 0.82 Breaker H 0.71
Strong breaker G 0.53 K 0.74
P 0.59 N 0.65
Breaker N 0.73 P 0.62
18
19
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 27 Januari 1994 di kota Padang. Penulis merupakan anak pertama dari 3 bersaudara dari ayah Rudy Aldrin, S.Pd dan ibu Aida Fitria, S.Pd. Pada tahun 2011, penulis lulus dari Sekolah Menengah Atas Negeri 1 Pulau Punjung, Dharmasraya, Sumatera Barat dan diterima di Departemen Fisika Institut Pertanian Bogor melalui jalur Seleksi Nasional Masuk Perguruan Tinggi Negeri (SNMPTN) Undangan pada tahun yang sama. Sebelumnya penulis menempuh pendidikan di Sekolah Dasar Negeri 32 Sitiung dan lulus tahun 2005, dilanjutkan di Sekolah Menengah Pertama Negeri 2 Pulau Punjung dan lulus tahun 2008.