KERAGAMAN GENETIK KELAPA SAWIT ASAL NIGERIA
DAN ASOSIASI MARKA MIKROSATELIT (SSR) DENGAN
KARAKTER
VIRESCENS
TINCHE
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
RINGKASAN
TINCHE. Keragaman Genetik Kelapa Sawit Asal Nigeria dan Asosiasi Marka Mikrosatelit (SSR) dengan Karakter Virescens. Dibimbing oleh SUDARSONO, DWI ASMONO dan DINY DINARTI.
Kelapa sawit merupakan tanaman penghasil minyak sayur utama dunia. Sempitnya sumber genetik kelapa sawit yang tersedia di Indonesia menyebabkan keterbatasan dalam pengembangan program pemuliaan. Salah satu cara untuk memperluas sumber genetik adalah menggunakan populasi introduksi. Hasil eksplorasi dan pemuliaan populasi Nigeria menunjukkan beberapa keunggulan dalam karakter komersial kelapa sawit.
Program pemuliaan kelapa sawit memiliki beberapa tujuan yaitu meningkatkan hasil dengan berbasiskan area tanam, meningkatkan kualitas minyak, memperlambat laju penambahan tinggi tanaman, dan mengembangkan varietas yang resisten terhadap berbagai hama dan penyakit. Karakter-karakter minor yang mendukung kualitas panen mulai diperhitungkan dalam perakitan varietas. Karakter warna buah seperti karakter Virescens (Vir) dapat digunakan sebagai indikator kematangan buah pada tandan kelapa sawit. Perbedaan warna buah Vir yang kontras antara buah mentah dan masak dapat memudahkan dan mempersingkat waktu yang dibutuhkan untuk panen tanpa harus memungut buah yang jatuh. Studi ini bertujuan untuk: (1) evaluasi keragaman genetik kelapa sawit famili DP-E; (2) mencari marka yang berasosiasi dengan karakter Virescens.
Beberapa marka SSR yang memiliki alel spesifik Pisifera dan Dura diperoleh dari hasil seleksi dari 105 marka SSR. Analisis keragaman genetik famili DP-E berdasarkan 25 marka SSR menunjukkan keragaman yang masih tinggi. Estimasi struktur populasi famili DP-E juga memperlihatkan adanya rekombinasi yang tinggi pada individu progeni.
Resolusi peta pautan genetik yang diperoleh dalam studi ini mencakup empat kelompok pautan (KP) dengan total cakupan peta genetik 213.1 cM. Lokus SSR mEgCIR3376 diperoleh sebagai kandidat marka yang terpaut pada karakter Virescens dengan jarak genetik 27.7 cM pada posisi Linkage Group 8. Pengujian marka pada empat populasi verifikasi memberikan hasil yang belum stabil.
Individu progeni famili DP-E yang memiliki karakter hasil tinggi, unggul dan Virescens dapat dipilih untuk dilanjutkan ke siklus pemuliaan berikutnya. Marka SSR mEgCIR3376 dapat digunakan sebagai alat bantu pada populasi tetua atau populasi pemuliaan untuk seleksi tanaman yang Virescens dengan persentase keterpautan 72.3%. Eksplorasi marka yang lebih dekat dengan karakter Virescens dapat dilakukan dengan memilih marka-marka SSR yang spesifik terdapat pada LG 8.
TINCHE. Genetic Diversity of Oil Palm Originated from Nigeria and Association Marker of Microsatellite with Virescence trait. Supervised by SUDARSONO, DWI ASMONO and DINY DINARTI.
Oil palm is one of the major oil crops of the world. The narrow genetic base of Indonesian oil palm collections limited the progress of breeding programs. One way to broaden the genetic source was to introduce other breeding populations. The exploration and breeding programs of Nigerian oil palms shows several commercial valued characters.
The breeding programs in oil palm have several purposes: to increase yield based on land expansion, to increase oil quality, to delay height increament and to develope varieties that resistant to pests and diseases. Minor traits which supported yield quality have been considered in developing improved hybrids. The contrasting color of unripe to ripe fruits Virescence (Vir) could be used as indicator of ripeness of oil palm fruit bunches. The use of this phenotypic marker could increase harvest efficiency and reduce cost, instead of counting and collecting loose fruits. The objectives of this study were to: (1) to evaluate the genetic diversity of oil palm DP-E family; (2) to identify SSR marker that associate with Virescens trait.
This study identified several SSR markers that had Pisifera and Dura alleles specific from screening of 105 markers. The genetic diversity analysis of DP-E family based on 25 SSR markers showed that the progenies still retained high genetic distance between individuals. The population estimation structure of DP-E family revealed high recombination numbers in progenies.
The resolution of genetic linkage map obtained in this study covered 213.1 cM with four Linkage Group. Loci mEgCIR3376 was identified as candidate marker linked to Virescencetrait with 27.7 cM genetic distance on LG 8. Marker verification on four other population showed that the marker was still unstable.
The progenies from DP-E family that has valued commercial characters and Virescence could be selected as candidate in the next breeding cycle. Marker mEgCIR3376 could be employed as a tool to screen Virescenceplants on parent or breeding population with 72.3% linkage percentage. An exploration of closer marker to Virescencecould be carried out by choosing SSR markers specifically linked to LG 8.
PERNYATAAN MENGENAI TESIS DAN SUMBER
INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Keragaman Genetik Kelapa Sawit Asal Nigeria dan Asosiasi Marka Mikrosatelit (SSR) dengan Karakter Virescens adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2014
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh karya tulis ini
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Pemuliaan dan Bioteknologi Tanaman
KERAGAMAN GENETIK KELAPA SAWIT ASAL NIGERIA
DAN ASOSIASI MARKA MIKROSATELIT (SSR) DENGAN
KARAKTER
VIRESCENS
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Keragaman Genetik Kelapa Sawit Asal Nigeria dan Asosiasi Marka Mikrosatelit (SSR) dengan karakter Virescens
Nama : Tinche NIM : A253100141
Disetujui oleh
Komisi Pembimbing
Diketahui oleh
Ketua Program Studi
Pemuliaan dan Bioteknologi Tanaman
Dr Ir Yudiwanti Wahyu EK, MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
Tanggal Ujian: 23 Juli 2014 Tanggal Lulus: Prof Dr Ir Sudarsono, MSc
Ketua
Dr Ir Dwi Asmono, MS, APU Anggota
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul tesis ini adalah Keragaman Genetik Kelapa Sawit Asal Nigeria dan Asosiasi Marka Mikrosatelit (SSR) dengan Karakter Virescens.
Terima kasih penulis ucapkan kepada Bapak Prof. Dr. Ir. Sudarsono, M.Sc., Bapak Dr. Ir. Dwi Asmono, MS, APU dan Ibu Dr. Ir. Diny Dinarti, M.Si. selaku pembimbing atas bimbingan, motivasi dan arahannya selama perencanaan, pelaksanaan serta penulisan tesis ini. Ungkapan terima kasih juga disampaikan kepada tim riset PT Sampoerna Agro, Tbk atas dukungan dan bantuannya selama penelitian.
Penulis juga mengucapkan terima kasih kepada teman-teman PBT angkatan 2010 atas kerjasama, semangat dan dukungannya selama studi; rekan-rekan di Plant Molecular Biology Laboratorium atas bantuannya dalam kegiatan penelitian. Ungkapan terima kasih sebesar-besarnya disampaikan kepada Ayah, Ibu, serta seluruh keluarga, atas dukungan dan doanya sehingga pendidikan ini dapat diselesaikan dengan baik.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2014
DAFTAR ISI
3! KERAGAMAN GENETIK KELAPA SAWIT (Elaeis guineensis Jacq.) POPULASI NIGERIA BERDASARKAN ANALISIS MARKA SSR (SIMPLE SEQUENCE REPEATS)
DAFTAR TABEL
2.1 Tipe dan bentuk buah kelapa sawit 6!
3.1 Genotipe terpilih berdasarkan hasil survei dan analisis segregasi karakter warna buah untuk seleksi primer 14 3.2 Primer hasil seleksi pada populasi dura dan pisifera yang terpilih
untuk genotyping 15
3.3 Data heterozigositas, jumlah alel dan Polymorphic Information Content (PIC) pada populasi DP-E dengan 25 marka SSR 16 4.1 Analisis segregasi 25 lokus SSR dan satu lokus karakter warna buah
pada famili DP-E 27
4.2 Pemetaan lokus SSR pada famili DP-E dengan kriteria LOD 0.56 – 10.56 dan fraksi rekombinan 0.25 28 4.3 Segregasi primer mEgCIR3376 pada famili G, H, K dan L berdasarkan
kelompok warna buah (Virescens dan nigrescens) 33
!
DAFTAR GAMBAR
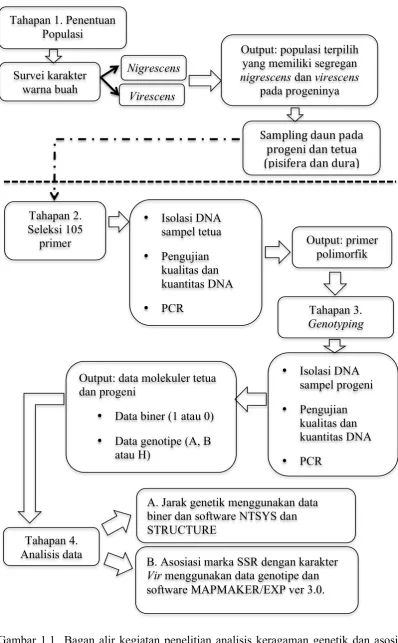
1.1 Bagan alir kegiatan penelitian analisis keragaman genetik dan asosiasi marka SSR (Simple Sequence Repeats) populasi kelapa sawit asal
Nigeria 3!
2.1 Tipe warna buah pada kelapa sawit 5!
3.1 Profil marka SSR pada genotipe pisifera (P) dan dura bulk (D1-D5)
menggunakan primer mEgCIR0588 14!
3.2 Profil marka SSR pada genotipe pisifera (P1-P10) dan dura bulk (D1-D5) menggunakan primer mEgCIR3376 15! 3.3 Dendogram analisis UPGMA populasi DP-E menggunakan 25 primer
SSR 17!
3.4 Perubahan delta K (!K) sesuai dengan perbedaan K antara (a) populasi DP-E dan (b) 47 individu progeni DP-E yang diidentifikasi oleh STRUCTURE dengan model campuran 18!!
3.5 Estimasi struktur populasi DP-E berdasarkan data genotyping 25 lokus SSR pada populasi kelapa sawit DP-E (a) dan progeni DP-E (b)
menggunakan program STRUCTURE 19!!
4.1 Separasi pita DNA pada gel akrilamid 6% dan contoh skoring 25!! 4.2 Profil marka SSR pada genotipe pisifera (P1 – P10) dan dura bulk
(D1-D5) menggunakan primer mEgCIR3376 pada tahap skrining
primer 26!!
4.3 Peta pautan genetik kelapa sawit berdasarkan famili DP-E, dikonstruksi dengan nilai LOD minimum 0.56 dan fraksi rekombinasi
maksimum 0.25 28!!
4.4 Profil marka SSR pada famili DP-E menggunakan primer
4.5 Dua tipe (A dan B) kemungkinan konfigurasi alel dan distribusi
segregasi marka mEgCIR3376 pada famili DP-E 30!
4.6 Profil marka SSR pada famili G menggunakan primer mEgCIR3376 31!!
4.7 Profil marka SSR pada famili H menggunakan primer mEgCIR3376 31 4.8 Profil marka SSR pada famili K menggunakan primer mEgCIR3376 32 4.9 Profil marka SSR pada famili L menggunakan primer mEgCIR3376 32 4.10 Konfigurasi genotipe dan distribusi segregasi marka mEgCIR3376 33
!
DAFTAR LAM
PIRAN
1 Daftar 105 primer yang digunakan dalam tahap skrining primer 44! 2 Profil marka SSR pada kelapa sawit famili DP-E 48!
3 Prosedur pembuatan larutan 55!
4 Tabel pengkodean sampel kelapa sawit untuk studi keragaman genetik dan asosiasi marka SSR dengan warna buah 58 5 Skoring pada tahap genotyping kelapa sawit famili DP-E dengan 25
marka SSR 60
6 Data skoring kelapa sawit famili DP-E untuk analisis MAPMAKER/EXP 64
1
PENDAHULUAN
Kelapa sawit (Elaeis guineensis Jacq.) merupakan salah satu komoditas perkebunan yang penting dan strategis dalam mendukung peningkatan penambahan devisa negara Indonesia. Tanaman ini merupakan tanaman penghasil minyak sayur utama dunia (39.9 %) selain kedelai (26.6 %), kanola (14.9 %), biji bunga matahari (8.8 %) dan beberapa komoditi lainnya (USDA 2014). Walaupun bukan tanaman asli Indonesia, kelapa sawit dapat tumbuh baik dan berproduksi tinggi di wilayah-wilayah Indonesia dengan curah hujan 2000-3000 mm/tahun yang menyebar merata sepanjang tahun (Hartley 1988).
Pertambahan luasan tanam dan produksi kelapa sawit saat ini belum mencukupi untuk memenuhi konsumsi dunia berdasarkan prediksi permintaan konsumsi minyak sayur dan peningkatan populasi dunia (Corley 2009). Permasalahan utama dalam meningkatkan produksi kelapa sawit yaitu pembatasan program perluasan areal tanam karena menyangkut masalah konversi dan keterbatasan luasan areal tanam yang tersedia. Hal ini menegaskan bahwa diperlukan program intensifikasi dengan cara meningkatkan potensi genetik kelapa sawit agar produktivitas per satuan hektar meningkat. Peningkatan potensi genetik dapat dilakukan melalui kegiatan pemuliaan tanaman yang memanfaatkan semua sumber daya genetik dan variasi genetik dari materi-materi yang telah diperoleh.
Pada saat ini perbaikan potensi genetik kelapa sawit tidak hanya diarahkan untuk karakter tunggal seperti peningkatan produktivitas crude palm oil (CPO) yang dilakukan pada tahun 1980-an. Asmono et al (1999) menyatakan bahwa karakter kualitas minyak menjadi perhatian utama setelah peningkatan hasil atau kuantitas. Karakter kuantitas dan kualitas minyak merupakan karakter kompleks yang tidak hanya berdiri sendiri sehingga seleksi dan perbaikan genetik kelapa sawit saat ini lebih tekankan untuk seleksi sifat berganda yang dilakukan secara simultan. Peningkatan kuantitas minyak dapat dilakukan dengan dua cara yaitu (1) meningkatkan karakter komponen produksi seperti karakter ukuran tandan, berat tandan, jumlah buah/tandan, jumlah tandan/tanaman, tebal daging buah, rasio minyak ke tandan dan (2) meminimalkan kehilangan hasil pada saat panen. Sementara itu peningkatan kualitas minyak dilakukan dengan meningkatkan karakter kandungan asam lemak tak jenuh dan kandungan metabolit sekunder penting yang bermanfaat bagi kesehatan seperti kandungan !-karoten.
Salah satu karakter penting yang dapat digunakan untuk meminimalkan kehilangan hasil pada saat panen dan penciri untuk kandungan !-karoten yaitu karakter warna buah. Pada kelapa sawit, tipe buah yang paling sering ditemui berwarna ungu gelap hingga hitam pada bagian apex dan kuning kehijauan pada bagian basal sebelum masak, yang disebut dengan nigrescens. Tipe lain yang kurang lazim ditemui berwarna hijau sebelum masak dan disebut Virescens. Tipe ini berubah warna menjadi jingga kemerahan pada saat masak, meskipun bagian apex dari eksternal buah tetap hijau (Corley & Tinker 2003). Karakter Virescens
! "!
Analisis genetik karakter Virescens belum banyak dilakukan terutama pada populasi spesifik yaitu populasi yang berasal dari Nigeria.
Populasi Pisifera Nigeria kelapa sawit yang digunakan dalam studi merupakan material introduksi dari ASD Costa Rica oleh PT Bina Sawit Makmur (anak perusahaan PT Sampoerna Agro) antara tahun 1995 hingga 1998. Studi preliminari pada material genetik ini oleh Breure (2002) menunjukkan bahwa Pisifera origin Nigeria memiliki karakter superior dalam hal hasil minyak tinggi dengan tinggi batang yang pendek dan juga beberapa karakter pendukung yang berasosiasi dengan indeks panen tinggi, seperti proporsi total bahan kering yang digunakan untuk produk ekonomi.
Marka Simple Sequence Repeats (SSR) adalah marka yang berbasis PCR
(Polymerase chain reaction) yang reprodusibel dan dapat mendeteksi lokus
multi-alelik dan kodominan. Pada kelapa sawit, marka SSR pertama kali diaplikasikan oleh Billotte et al. (2001) dan kemudian digunakan untuk konstruksi peta genetik (Billotte et al. 2005), analisis keragaman genetik, analisis parental (Thongthawee
et al. 2010), dan verifikasi hibrida (Thawaro dan Te-chato 2009).
Bulk Segregant Analysis (BSA), yang dikembangkan oleh Michelmore et
al. (1991), adalah salah satu metode untuk mengidentifikasi marka yang terpaut pada gen atau area genom tertentu secara cepat. Prinsip dasar metode ini adalah membandingkan dua kelompok sampel DNA bulk dari populasi bersegregasi hasil persilangan. Individu dalam setiap kelompok bulk memiliki kesamaan pada karakter atau gen yang diinginkan tapi memiliki perbedaan untuk karakter yang lain. Dua kelompok DNA, dalam studi ini adalah kelompok Vir dan kelompok
nigrescens (nig) dianalisis untuk mengidentifikasi marka yang dapat membedakan
warna buah.
Studi ini ditujukan untuk mempelajari struktur genetik dan keragaman genetik populasi kelapa sawit yang berasal dari Nigeria koleksi PT. Sampoerna Agro dan mempelajari asosiasi antara marka molekuler SSR dengan karakter
Virescens.
#!
B. Asosiasi marka SSR dengan karakter
2 TINJAUAN PUSTAKA
Botani Kelapa Sawit
Kelapa sawit diyakini berasal dari benua Afrika berdasarkan catatan eksplorasi Zeven (1965). Kumpulan pohon liar dan semi-liar ditemukan sepanjang garis pantai mulai dari bagian paling utara dataran Senegal ke Sierra Leone, Liberia, Pantai Gading, Ghana, Togo, Benin, Nigeria, Kamerun, Republik Rakyat Kongo, Angola hingga ke bagian paling selatan di Republik Demokratik Kongo (Corley dan Tinker 2003). Pusat asal dan keragaman kelapa sawit terkonsentrasi di hutan tropis Nigeria, Kamerun, Kongo dan Angola (Ngando-Ebongue et al.
2012).
Catatan pertama introduksi kelapa sawit di Asia Tenggara adalah adanya empat bibit yang ditanam di Kebun Raya Buitenzorg (sekarang Bogor) pada tahun 1848 di pulau Jawa. Pohon yang tumbuh dari ke empat bibit ini relatif seragam dan diperkirakan di produksi di Amsterdam, dari biji yang dibawa dari Afrika (Hartley 1988). Turunan dari empat pohon ini kemudian didistribusikan dan yang kemudian digunakan untuk pengembangan materi genetik kelapa sawit di Indonesia dan Malaysia.
Kelapa sawit (Elaeis guineensis) adalah tanaman monokotil dari ordo Arecales dan merupakan famili Palmae dan subfamili Cocosideae. Tanaman ini termasuk satu genus Cocos dengan kelapa. Kata elaeis diambil dari bahasa Yunani ‘elaion’ yang berarti minyak dan ‘elaia’ yang berarti zaitun.
Kelapa sawit memiliki satu titik tumbuh. Daun tumbuh dari meristem apikal yang terletak di bagian apex batang. Bagian ini memiliki diameter 10-12 cm dan panjang 2.5-4 cm. Tajuk kelapa sawit dewasa terdiri dari 30-50 daun. Pertumbuhan batang rata-rata per tahun sekitar 0.3-0.6 m. Pada perkebunan, penanaman ulang dilakukan jika tinggi rata-rata pohon mencapai 10 m. Diameter batang bervariasi antara 20-75 cm.
Pada kelapa sawit dewasa, akar primer dengan diameter 5-10 mm tumbuh mengarah ke bawah dari dasar tanaman atau menyebar horizontal hingga radius 3.5-4.5 m. Akar sekunder dengan diameter 1-4 mm tumbuh dari akar primer dan mengarah ke bawah, akar tersier berdiameter 0.5-1.5 mm tumbuh dari akar sekunder dan dapat mencapai panjang hingga 20 cm dengan arah tumbuh yang tidak diketahui.
Tanaman palma ini berdaun majemuk dengan pelepah daun tersusun melingkari batang berbentuk spiral. Daun kelapa sawit tersusun berselang seling
(pinnate) dan terbagi menjadi dua bagian: rachis yang menghasilkan anak daun
5
Tipe pembungaan kelapa sawit pada umumnya adalah monoecious dengan bunga jantan atau betina tumbuh terpisah dalam satu pohon. Buah yang dihasilkan berbentuk tandan yang besar dan kompak. Tipe buah kelapa sawit adalah drupe
dengan mesokarp yang mengandung banyak minyak. Biji buah (nut) terdiri dari cangkang atau endocarp, dan satu, dua atau tiga kernel. Umumnya hanya satu
kernel yang bertahan karena kernel yang lain aborsi. Tipe dan bentuk buah kelapa
sawit dibedakan menjadi beberapa jenis (Tabel 2.1).
Gambar 2.1 Tipe warna buah pada kelapa sawit. A = tipe nigrescens; B = tipe
Virescens.
Tabel 2.1 Tipe dan bentuk buah kelapa sawit (Corley dan Tinker 2003)
Karakter Terminologi Deskripsi
Tipe buah
Warna eksternal Nigrescens Mengandung antosianin di eksokarp, berwarna hitam atau coklat pada bagian apex buah
Virescens Tidak terdapat antosianin di eksokarp,
buah berwarna hijau saat masih mentah, berwarna jingga dengan ujung kehijauan saat masak
Warna mesokarp Albescens Mengandung karoten yang sangat
rendah di mesokarp, berwarna kuning pucat, bukan jingga
Bentuk buah
Ketebalan cangkang Dura Bercangkang tebal, 2-8 mm, 35-65 % mesokarp/buah, tidak ada cincin serat melingkari biji jika dibelah
Tenera Bercangkang tipis, 0.5-4 mm, 55-96% mesokarp/buah, terdapat cincin serat Pisifera Tidak bercangkang, biasanya steril
untuk bunga betina
! 6!
Pemuliaan Kelapa Sawit
Pemuliaan dan seleksi kelapa sawit berkaitan erat dengan pengembangan dura Deli berdasarkan empat pohon kelapa sawit yang diintroduksikan di Bogor tahun 1848. Kelapa sawit memiliki siklus pemuliaan yang panjang, sekitar 10 tahun, seperti: satu tahun untuk polinasi, dua hingga tiga bulan untuk persiapan dan germinasi benih, tiga tahun di lapangan sebelum panen dan empat hingga enam tahun untuk evaluasi panen. Jika ditambahkan dengan uji progeni, waktu yang dibutuhkan mendekati 20 tahun untuk mengembangkan progeni yang telah teruji. Pemuliaan kelapa sawit memiliki beberapa tujuan utama: (1) meningkatkan hasil minyak, (2) tanaman yang pendek, (3) peningkatan kualitas minyak, (4) ketahanan terhadap penyakit, (5) sifat-sifat fisiologis (indeks tandan, jumlah bobot kering dan bunch dry matter), (6) eksploitasi interaksi genotipe dan lingkungan (Rajanaidu et al. 2000).
Secara umum, ada beberapa pendekatan yang diadopsi untuk pemuliaan kelapa sawit. Pendekatan yang umum digunakan adalah Reciprocal Recurrent
Selection (RRS) dan Family and Individual Selection (FIS). RRS bertujuan untuk
mengembangkan kelompok pisifera dan dura secara terpisah dan saling melengkapi untuk sifat tertentu dimana vigor hibrida dieksploitasi ketika disilangkan. Uji lanjut dilakukan pada progeni sebelum pengembangan selanjutnya untuk memperoleh nilai pemuliaan. FIS digunakan untuk mengidentifikasi famili terbaik dan kemudian tetua terbaik dari generasi berikutnya dipilih menggunakan nilai fenotipik (Price et al. 2007).
Metode Modified Recurrent Selection (MRS) digunakan oleh sebagian besar pemulia kelapa sawit di Malaysia. Metode seleksi ini melibatkan persilangan diantara tetua terseleksi dan progeni dilanjutkan pada siklus seleksi berikutnya. Berbeda dengan RRS, introduksi bahan genetik baru dilakukan dalam program pemuliaan (Rajanaidu et al. 2000). Proses ini memungkinkan introduksi gen baru dalam program pemuliaan untuk meningkatkan variabilitas genetik (Hardon 1970).
Untuk membantu memotong siklus pemuliaan yang panjang, metode seleksi berbasis DNA digunakan untuk meningkatkan efisiensi dan presisi dalam studi gen (Collard dan Mackill 2008). Seleksi berbasis DNA lebih dapat diandalkan daripada seleksi konvensional yang berbasis fenotipe, karena fenotipe dipengaruhi oleh lingkungan dan genotipe. Penggunaan marka berbasis DNA dalam pemuliaan disebut Marker-Assisted Selection (MAS).
7
Marka Berbasis PCR
Marka berbasis PCR memiliki beberapa keuntungan sepertinya waktu yang lebih singkat untuk memperoleh hasil, jumlah DNA genom yang diperlukan lebih sedikit (5-50 ng), dan kemampuannya untuk membagi informasi sekuen primer tanpa perlu tukar menukar DNA. Marka ini dapat berdasarkan reaksi primer yang berubah-rubah, seperti RAPD (random amplified polymorphic DNA), ISSR (inter simple sequence repeats) dan AFLP (amplified fragment length
polymorphisms). Namun, banyak juga marka yang berdasarkan sekuen yang telah
diketahui, seperti mikrosatelit atau SSR (simple sequence repeats), STS (sequence
tagged sites) dan SNP (single nucleotide polymorphisms) (Godwin 2003).
SSR (simple sequence repeats) atau mikrosatelit tersebar merata di genom eukariot. Polimorfisme SSR menggambarkan variasi jumlah unit berulang di daerah tertentu dalam genom. Frekuensi pengulangan yang lebih dari 20 bp diperkirakan muncul setiap 33 kb di tanaman. Sekuen nukleotida yang mengapit pengulangan tersebut digunakan untuk mendesain primer untuk amplifikasi berbagai unit pengulangan di berbagai varietas. Primer-primer ini sangat berguna untuk deteksi cepat dan akurat lokus-lokus yang polimorfik dan informasi ini dapat digunakan untuk membangun peta fisik berdasarkan sekuen tag tersebut. Tipe polimorfisme ini sangat reprodusibel (Varshney et al. 2004).
Seleksi menggunakan marka molekuler merupakan alternatif yang menarik karena memiliki potensial untuk mengurangi waktu yang diperlukan untuk menghasilkan varietas baru dan melepasnya ke pasar. Hal ini dikarenakan kemampuan untuk menyeleksi di tahap awal (terutama pada tahap pembibitan) akan memberikan efek yang besar dalam mengurangi waktu dan sumber lain yang dibutuhkan untuk perbaikan varietas (Singh et al. 2007).
Marka SSR pada kelapa sawit pertama kali dikembangkan oleh Billote et al. (2001) dengan menskrining pustaka SSR yang kaya (GA)n, (GT)n dan (CCG)n hingga karakterisasi akhir 21 lokus SSR. Estimasi kisaran ukuran alel dan heterozigositas yang diharapkan di E. guineensis dan spesies yang berkerabat dekat E. oleifera juga dipublikasikan sekuen primer, dimana penggunaan optimal dari marka SSR dilakukan. Analisis data multivariat menunjukkan kemampuan marka SSR secara efisien mengungkapkan struktur keragaman genetik genus
Elaeis sesuai dengan asal geografis dan hubungan genetiknya berdasarkan studi
molekuler sebelumnya. Tingginya tingkat variabilitas alelik mengindikasikan bahwa SSR E. guineensis merupakan alat yang kuat untuk studi genetik genus
Elaeis, termasuk identifikasi varietas dan pemetaan genetik intra atau inter
spesifik.
Bulk Segregant Analysis (BSA)
Metode Bulk Segregant Analysis (BSA) dikembangkan oleh Michelmore
et al. (1991) untuk identifikasi cepat marka yang terpaut dengan gen atau region
! 7!
kelompok, atau bulk, individu-individu dalam kelompok tersebut identik untuk karakter atau gen yang diamati tetapi berbeda untuk gen-gen lainnya. Dua kelompok dengan karakter yang kontras berbeda (contohnya resisten dan rentan terhadap penyakit tertentu) dianalisis untuk identifikasi marka yang dapat membedakan karakter tersebut.
Keuntungan dari metode ini adalah metode ini dapat difokuskan pada
region yang diinginkan dengan marka; dan dapat dengan cepat mengetahui lokasi
gen. Metode BSA dapat digunakan untuk mencari marka-marka yang terpaut erat dengan penyakit (Devey et al. 1995; Silva et al. 2003); analisis keragaman genetik pada kultivar (Wakui et al. 2009) dan membuat peta genetik (Hong et al.
3 KERAGAMAN GENETIK KELAPA SAWIT (
Elaeis
guineensis
Jacq.) ASAL NIGERIA BERDASARKAN MARKA
SSR (
SIMPLE SEQUENCE REPEATS
)
1Abstrak
Kelapa sawit merupakan tanaman penghasil minyak sayur utama dunia. Sempitnya sumber genetik kelapa sawit yang tersedia di Indonesia menyebabkan keterbatasan dalam pengembangan program pemuliaan. Salah satu cara untuk memperluas sumber genetik adalah menggunakan populasi introduksi. Hasil eksplorasi dan pemuliaan populasi Nigeria menunjukkan beberapa keunggulan dalam karakter komersial kelapa sawit. Tujuan dari studi ini adalah untuk seleksi primer SSR yang polimorfik, mencari primer SSR yang memiliki alel spesifik untuk pisifera dan mengevaluasi keragaman genetik intrapopulasi Nigeria famili DP-E. 105 primer SSR digunakan untuk skrining primer dan 25 marka terseleksi digunakan untuk tahap evaluasi keragaman genetik. Segregasi marka yang diperoleh dari hasil seleksi 105 marka SSR cukup tinggi (91.4 % polimorfik). Marka dengan alel spesifik pisifera dapat digunakan untuk tujuan eksplorasi marka yang berasosiasi dengan karakter warna buah. Pada hasil studi ini, 25 marka SSR mampu memperlihatkan keragaman genetik dan struktur populasi famili DP-E. Analisis jarak genetik dengan pengelompokan UPGMA menghasilkan dua kelompok besar dengan koefisien kemiripan 56 % dan tiga subgrup progeni pada koefisien kemiripan 63 %. Estimasi struktur populasi dengan program STRUCTURE menunjukkan adanya rekombinasi yang tinggi pada individu progeni. Jumlah individu rekombinan yang tinggi menguntungkan untuk pemilihan individu untuk seleksi pada program pemuliaan selanjutnya.
Kata kunci: kelapa sawit, keragaman genetik, SSR, virescens, warna buah
!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!!
! 89!
Abstract
Oil palm is one of the major oil crops of the world. The narrow genetic base of Indonesian oil palm collections limited the progress of breeding programs. One way to broaden the genetic source was to introduce other breeding populations. The exploration and breeding programs of Nigerian oil palms shows several commercial valued characters. The aims of this study were to screen polymorphic SSR primers, to identify SSR primers that amplified pisifera’s specific bands and to evaluate the genetic diversity of Nigerian population DP-E family. 105 SSR markers were used in primer screening and 25 selected markers were used in genotyping. The marker segregation obtained from 105 SSR markers in this study was relatively high (91.4 % polymorphic). Markers with specific alleles for pisifera could be used to find markers that associated with fruit color trait. In this study, 25 SSR markers could reveal the genetic diversity and population structure of DP-E family. Genetic analysis with UPGMA clustering system generated two clusters with 56 % similarity coefficient and three sub-clusters of progenies DP-E with 63 % similarity coefficient. Estimation of population structure using STRUCTURE software showed high recombination numbers in progenies. The high recombination numbers in progenies would be an advantage in providing genetic materials to be selected for further breeding programs.
Keywords: fruit color, genetic diversity, oil palm, SSR, virescence
Pendahuluan
Kelapa sawit (Elaeis guineensis Jacq.) merupakan salah satu komoditas perkebunan yang penting dan strategis dalam mendukung peningkatan penambahan devisa negara Indonesia. Tanaman ini merupakan tanaman penghasil minyak sayur utama dunia (39.9 %) selain kedelai (26.6 %), kanola (14.9 %), biji bunga matahari (8.8 %) dan beberapa komoditi lainnya (USDA 2014). Suplai terbesar minyak kelapa sawit berasal dari Indonesia (33 500 MT) diikuti oleh Malaysia (20 800 MT), Thailand (2 250 MT) dan negara lainnya (6 248 MT) berdasarkan data produksi minyak sawit dunia per Juli 2014. Di Indonesia sendiri, penambahan area penanaman kelapa sawit masih terus meningkat hingga 8 % per tahun (BPS 2011). Pertambahan luasan tanam dan produksi kelapa sawit saat ini belum mencukupi untuk memenuhi konsumsi dunia berdasarkan prediksi permintaan konsumsi minyak sayur dan peningkatan populasi dunia (Corley 2009). Beberapa alternatif untuk memenuhi permintaan tersebut adalah dengan memaksimalkan pengelolaan perkebunan yang ada dan penggunaan bibit dengan karakter yang unggul.
11
yang tumbuh dari kecambah ini relatif seragam dengan tipe buah Dura dan diindikasikan berasal dari satu pohon tetua. Program pemuliaan kelapa sawit Indonesia dikembangkan dari populasi ini dan dikenal sebagai ‘kelapa sawit Deli’. Dura Deli memiliki beberapa keunggulan: buahnya besar dan mesokarp yang mengandung minyak tinggi (60 %) (Hartley 1988, Pamin 1998). Implikasi dari pengembangan dura Deli ini menyebabkan keragaman genetik kelapa sawit Indonesia menjadi relatif sempit.
Salah satu upaya untuk memperluas keragaman genetik ini adalah dengan mengintroduksikan sumber genetik baru. Populasi kelapa sawit asal Nigeria adalah salah satu sumber genetik yang digunakan. Beberapa keunggulan dari populasi Nigeria: memiliki pertumbuhan tinggi yang lambat (15-25 cm/tahun), kadar iodine tinggi (IV > 70) dan rataan kernel to bunch yang tinggi (di atas 12 %) (Rajanaidu dan Rao 2002).
Pada kelapa sawit, tipe buah yang paling sering ditemui berwarna ungu gelap hingga hitam pada bagian apex dan kuning kehijauan pada bagian basal sebelum masak, yang disebut dengan nigrescens. Tipe lain yang kurang lazim ditemui berwarna hijau sebelum masak dan disebut Virescens. Tipe ini berubah warna menjadi jingga kemerahan pada saat masak, meskipun bagian apex dari eksternal buah tetap hijau (Corley & Tinker 2003). Warna buah Virescens ini juga ditemui pada beberapa aksesi di populasi Nigeria.
Studi ini menggunakan marka SSR, yang berdasarkan pada sejumlah sekuen DNA berulang (2-5 nukleotida) yang terdapat dalam mikrosatelit. Jumlah pengulangan kopi SSR ini bervariasi antar individu dan merupakan sumber polimorfisme di tanaman. Marka SSR merupakan marka berlokus tunggal, multialelik dan kodominan (Acquaah 2007). Pada kelapa sawit, marka SSR pertama kali diaplikasikan oleh Billotte et al. (2001). Marka SSR dipilih karena jumlahnya yang melimpah dan terdistribusi merata, dihasilkan cepat melalui PCR, mudah diskoring dan informasi sekuen primernya mudah diakses melalui publikasi (Saghai-Maroof et al. 1994). Tujuan dari studi ini adalah untuk seleksi primer SSR yang polimorfik, mencari primer SSR yang memiliki alel spesifik Pisifera dan mengevaluasi keragaman genetik intrapopulasi famili DP-E asal Nigeria.
Bahan dan Metode
Pemilihan Populasi
Jumlah pohon yang digunakan dalam survei adalah 2 480 pohon, terdiri dari 53 famili hasil persilangan enam genotipe Pisifera dengan 51 genotipe Dura berdasarkan desain alpha (alpha design). Survei warna buah dikategorikan menjadi dua: Virescens (Vir) dan nigrescens (nig).
! 8"!
Ekstraksi DNA dan prosedur SSR
Sampel daun tombak digunakan untuk ekstraksi DNA. DNA diisolasi menggunakan metode CTAB berdasarkan modifikasi dari Orozco-Castillo et al.
(1994). Sampel daun tombak segar dengan ukuran ± 1 cm x 1 cm dipotong kemudian digerus hingga halus dalam mortar yang telah diisi dengan larutan buffer ekstraksi 1 000 µl dan 0.1 mg PVP. Hasil gerusan dimasukkan ke dalam tabung mikro 2 ml yang telah berisi 500 µl buffer ekstraksi (CTAB) dan 10 µl mercaptoethanol. Hasil gerusan diinkubasi selama 60 menit di waterbath dengan suhu 65 °C dan dibalik manual setiap 10 menit. Setelah inkubasi, KIAA ditambahkan kedalam campuran hingga total volume 2 ml dan divortex hingga homogen, diikuti sentrifusi dengan kecepatan 11 000 rpm selama 10 menit. Supernatan diambil dan dipindahkan ke tabung mikro 2 ml baru dan ditambahkan 1 ml KIAA, divortex hingga homogen. Campuran disentrifusi kembali dengan kecepatan 11 000 rpm selama 10 menit.
Supernatan hasil sentrifusi kedua dipindahkan ke tabung mikro 1.5 ml baru, ditambahkan Na-asetat 1/10 dari volume total dan etanol p.a hingga total volume 2 ml. Tabung dibalik berulangkali secara perlahan hingga timbul benang halus berwarna putih bening atau kekuningan. Tabung diinkubasi di suhu 4 °C selama 60 menit, setelahnya disentrifusi pada 11 000 rpm selama 10 menit. Supernatan dibuang dan pelet dikeringanginkan selama 15-30 menit. Buffer TE 1x sebanyak 300 µl ditambahkan ke dalam tabung dan dibalik berulangkali secara manual hingga larut, inkubasi di 4 °C selama 60 menit. Setelah inkubasi, RNAase 3.3 µl ditambahkan dalam tabung dan inkubasi di 37 °C selama 60 menit. Suspensi ditambahkan 1 ml etanol p.a dan disimpan pada suhu 4 °C selama 60 menit, kemudian disentrifusi pada 11 000 rpm selama 10 menit. Supernatan dibuang dan pelet dikering-anginkan selama 15 menit, kemudian ditambahkan 0.5 ml etanol 70% untuk membersihkan larutan CTAB yang tersisa. Tabung dibalik berulangkali secara perlahan, etanol dibuang dan pelet dikering-anginkan selama 15-30 menit. Pelet yang telah mengering disuspensikan kembali dengan menambahkan 200 µl buffer TE 1x, dibalik manual dan perlahan untuk menghindari terputusnya DNA. Suspensi yang dihasilkan merupakan DNA stok dan disimpan di suhu -20 °C untuk penggunaan selanjutnya.
Pengecekan kualitas dan kuantitas DNA menggunakan 0.8% gel agarose dan diwarnai dengan GelRedTM. Gel divisualisasi dengan Bio Rad Gel DocTM UV-Transluminator. Campuran Polymerase Chain Reaction (PCR) terdiri dari 9.5 µl ddH2O, 12.5 µl Promega GoTaq® Green master mix (Taq DNA polymerase, dNTPs, MgCl2 dan buffer reaksi), 1.0 µl primer forward, 1.0 µl primer reverse dan 5 ng/µl DNA template. PCR dijalankan dengan tahap denaturasi pertama pada 95 °C selama 1 menit, tahap denaturasi kedua pada 94 °C selama 30 detik, tahap
annealing sesuai dengan Ta primer selama 1 menit, tahap extension pada 72 °C
selama 2 menit dan tahap final extension pada 72 °C selama 8 menit. Proses dijalankan dengan 35 siklus sebelum suhu diturunkan ke 4 °C. Produk PCR dipisahkan menggunakan dengan Cole-Parmer® Dedicated Height Sequencer
13
Seleksi Primer dan Genotyping
Primer-primer yang dipilih dari Billotte et al. (2005) sebanyak 105 digunakan dalam proses skrining primer. Materi genetik yang digunakan dalam skoring primer adalah 10 sampel pisifera dan lima DNA bulk dari masing-masing lima genotipe dura. DNA bulk per genotipe dura diperoleh dengan cara mem-bulk
DNA dari 10 individu dura dalam genotipe yang sama. Pada tahap genotyping, populasi yang digunakan adalah famili DP-E dengan 47 sampel progeni, tetua jantan (P3) dan tetua betina (D3). Primer yang digunakan pada tahap ini adalah 25 primer SSR yang dihasilkan dari tahap seleksi primer. Kriteria seleksi primer untuk genotyping adalah primer harus polimorfik dan pita pada individu Pisifera harus heterozigot.
Analisis Data
Pita – pita yang diperoleh pada plat kaca diskoring manual sebagai data biner dengan kode (1) jika ada pita dan (0) jika tidak ada pita. Data hasil skoring digunakan untuk estimasi parameter keragaman genetik dalam populasi:
Polymorphic Information Content (PIC) menggunakan persamaan matematika:
!"#! !!! !! !!!
al. 1998) digunakan untuk estimasi heterozigositas (He) berdasarkan Nei (1972), He = 1 – "Pi2, dimana Pi adalah rataan frekuensi ke-i alel SSR. Software POPGENE (Yeh et al. 1999) digunakan untuk kalkulasi alel efektif (Ne). Analisis pengelompokan berdasarkan Unweighted pair-group with arithmetic average
(UPGMA) dengan koefisien Dice (1945) menggunakan software NTSYSpc ver. 2.02 (Rohlf 1998).
Program STRUCTURE V2.3.4 (Pritchard et al. 2000) digunakan untuk mengestimasi struktur populasi yang menunjukkan kemiripan genotipe dalam subgroup. Setiap individu dijalankan pada kisaran kluster genetik K=1 hingga K=10 dengan model admixture, dan setiap K diulang sebanyak 20 kali. Setiap run
diimplementasikan dengan burn-in period 100 000 steps diikuti dengan replikasi 250 000 Monte Carlo Markov Chain (MCMC). Plot hasil didasarkan pada consensus permutasi Q-matrix dari 20 replikasi setiap K menggunakan program CLUMPP (Jakobsson dan Rosenberg 2007) dan ditampilkan dengan bantuan program DISTRUCT (Rosenberg 2004).
Hasil dan Pembahasan
Pemilihan Populasi
! 8:!
famili memiliki segregasi warna buah Vir dan nig; 34 famili lainnya memiliki warna buah yang seluruhnya berwarna hitam (nig).
Sembilan belas famili yang memiliki segregasi warna buah diuji rasio segregasi turunannya untuk karakter tersebut. Lima populasi dura (D1, D2, D3, D4, D5) dan satu populasi DP-E terpilih untuk digunakan dalam seleksi primer
dan genotyping (Tabel 3.1). Genotipe-genotipe ini terpilih karena segregasi warna
buah Vir dan nig nya mendekati rasio 1:1 dengan uji khi kuadrat.
Polimorfisme Mikrosatelit pada Seleksi Primer
Hasil dari seleksi 105 primer spesifik SSR menujukkan lima primer (4.8 %) tidak menghasilkan pita, empat primer (3.8 %) menghasilkan pita monomorfik dan 96 primer (91.4 %) dengan pita polimorfik. Enam primer
(mEgCIR3828, mEgCIR0803, mEgCIR2423, mEgCIR0588, mEgCIR3747 dan
mEgCIR2813) hanya menghasilkan pita pada sampel dura tetapi tidak pada
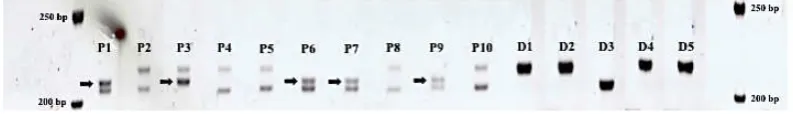
individu pisifera. Primer tersebut dapat dikategorikan sebagai primer spesifik Dura (Gambar 3.1). Dua puluh lima primer (Tabel 3.2) yang menghasilkan alel spesifik pisifera (Gambar 3.2) diperoleh dari seleksi lanjut 96 primer yang polimorfik. Primer ini digunakan selanjutnya dalam tahap genotyping.
Sampel dura (D1-D5) merupakan sampel bulk atau gabungan DNA dari 10 sampel setiap genotipe. Hasil amplifikasi pita pada kelima sampel adalah 0 (tidak ada amplikon) dan maksimal dua pita. Hal ini menunjukkan bahwa tidak ada kontaminasi pollen lain pada saat persilangan DxD dan tidak ada mislabeling pada saat pembibitan dan transportasi hingga penanaman di lapangan untuk sampel yang digunakan.
Tabel 3.1 Genotipe terpilih berdasarkan hasil survei dan analisis segregasi karakter warna buah kelapa sawit untuk seleksi primer
Genotipe Tipe Buah Jumlah Sampel
DP-E Tenera 47
Gambar 3.1 Profil marka SSR pada kelapa sawit genotipe Pisifera (P) dan Dura
bulk (D1-D5) menggunakan primer mEgCIR0588. Pada sampel Pisifera tidak ada pita yang teramplifikasi.
15
Gambar 3.2 Profil marka SSR pada kelapa sawit genotipe Pisifera (P1-P10) dan Dura bulk (D1-D5) menggunakan primer mEgCIR3376. Tanda panah pada gambar menunjukkan alel spesifik pada Pisifera.
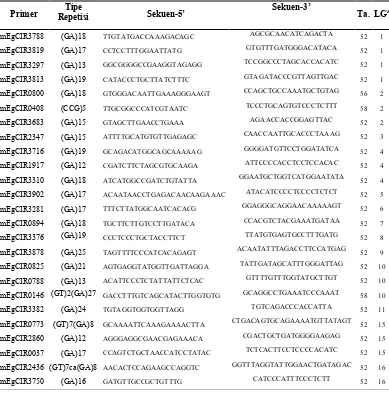
Tabel 3.2 Primer hasil seleksi pada kelapa sawit populasi dura dan pisifera yang terpilih untuk genotyping
Primer Repetisi Tipe Sekuen-5’ Sekuen-3’ Ta. LGa
mEgCIR3788 (GA)18 TTGTATGACCAAAGACAGC AGCGCAACATCAGACTA 52 1
mEgCIR3819 (GA)17 CCTCCTTTGGAATTATG GTGTTTGATGGGACATACA 52 1
mEgCIR3297 (GA)13 GGCGGGGCCGAAGGTAGAGG TCCGGCCCTAGCACCACATC 52 1
mEgCIR3813 (GA)19 CATACCCTGCTTATCTTTC GTAGATACCCGTTAGTTGAC 52 1
mEgCIR0800 (GA)18 GTGGGACAATTGAAAGGGAAGT CCAGCTGCCAAATGCTGTAG 56 2
mEgCIR0408 (CCG)5 TTGCGGCCCATCGTAATC TCCCTGCAGTGTCCCTCTTT 58 2
mEgCIR3683 (GA)15 GTAGCTTGAACCTGAAA AGAACCACCGGAGTTAC 52 2
mEgCIR2347 (GA)15 ATTTTGCATGTGTTGAGAGC CAACCAATTGCACCCTAAAG 52 3
mEgCIR3716 (GA)19 GCAGACATGGCAGCAAAAAG GGGGATGTTCCTGGATATCA 52 4
mEgCIR1917 (GA)12 CGATCTTCTAGCGTGCAAGA ATTCCCCACCTCCTCCACAC 52 4
mEgCIR3310 (GA)18 ATCATGGCCGATCTGTATTA GGAATGCTGGTCATGGAATATA 52 4
mEgCIR3902 (GA)17 ACAATAACCTGAGACAACAAGAAAC ATACATCCCCTCCCCTCTCT 52 5
mEgCIR3281 (GA)17 TTTCTTATGGCAATCACACG GGAGGGCAGGAACAAAAAGT 52 6
mEgCIR0894 (GA)18 TGCTTCTTGTCCTTGATACA CCACGTCTACGAAATGATAA 52 7
mEgCIR3376 (GA)19 CCCTCCCTGCTACCTTCT TTATGTGAGTGCCTTTGATG 52 8
mEgCIR3878 (GA)25 TAGTTTTCCCATCACAGAGT ACAATATTTAGACCTTCCATGAG 52 9
mEgCIR0825 (GA)21 AGTGAGGTATGGTTGATTAGGA TATTGATAGCATTTGGGATTAG 52 10
mEgCIR0788 (GA)13 ACATTCCCTCTATTATTCTCAC GTTTTGTTTGGTATGCTTGT 52 10
mEgCIR0146 (GT)2(GA)27 GACCTTTGTCAGCATACTTGGTGTG GCAGGCCTGAAATCCCAAAT 58 10
mEgCIR3382 (GA)24 TGTAGGTGGTGGTTAGG TGTCAGACCCACCATTA 52 11
mEgCIR0773 (GT)7(GA)8 GCAAAATTCAAAGAAAACTTA CTGACAGTGCAGAAAATGTTATAGT 52 15
mEgCIR2860 (GA)12 AGGGAGGCGAACGAGAAACA CGACTGCTGATGGGGAAGAG 52 15
mEgCIR0037 (GA)17 CCAGTCTGCTAACCATCCTATAC TCTCACTTCCTCCCCACATC 52 15
mEgCIR2436 (GT)7ca(GA)8 AACACTCCAGAAGCCAGGTC GGTTTAGGTATTGGAACTGATAGAC 52 16
mEgCIR3750 (GA)16 GATGTTGCCGCTGTTTG CATCCCATTTCCCTCTT 52 16
Keterangan: Ta. = Annealing Temperature, LG = Linkage Group, a Primer yang digunakan dikembangkan oleh Billotte et al. (2005)
! 86!
adalah 0.45 dan 0.51. Nilai ini mengindikasikan bahwa populasi yang digunakan memiliki keragaman genetik moderat (Hildebrand et al. 1992) pada set primer yang digunakan dalam seleksi. Jumlah total alel yang diperoleh adalah 327 alel dengan variasi antara satu hingga tujuh alel per lokus. Lokus mEgCIR0037 (6 alel) dan mEgCIR3382 (7 alel) menghasilkan jumlah alel tertinggi.
Analisis Keragaman Genetik pada populasi DP-E
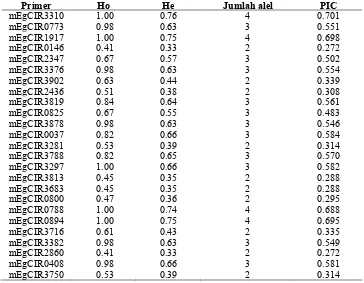
Informasi genetik pada famili DP-E disajikan dalam Tabel 3.3. Tingkat polimorfisme DP-E dievaluasi dengan mengkalkulasi nilai PIC pada 25 loci SSR. Nilai PIC maksimum diperoleh pada angka 0.701 pada primer mEgCIR3310 yang menunjukkan lokus ini yang paling informatif. Primer mEgCIR0146 dan
mEgCIR2860 adalah lokus yang paling kurang informatif (PIC = 0.272). Nilai Ho
berkisar antara 0.41 hingga 1(rataan Ho = 0.75), dan kisaran nilai He adalah 0.33-0.75 (rataan He = 0.54). Nilai rata-rata heterozigositas yang diharapkan (He) dan heterozigositas yang diamati (Ho) mengindikasikan keragaman genetik yang relatif tinggi intrapopulasi DP-E. Total alel yang dihasilkan dari 25 primer SSR adalah 69 alel dengan kisaran alel efektif 1.48-3.97 dan rerata alel efektif (Ne) 2.44.
Tabel 3.3 Data heterozigositas, jumlah alel dan Polymorphic Information Content
(PIC) pada kelapa sawit famili DP-E dengan 25 marka SSR
Primer Ho He Jumlah alel PIC
17
Hasil percobaan memperlihatkan penurunan nilai rata–rata He (0.54) dibandingkan dengan hasil yang diperoleh Putri (2010) (0.663) pada tiga populasi Nigeria dari koleksi yang sama dengan percobaan ini, dan Bakoume (2009) (0.644) pada lima populasi alami asal Nigeria. Rerata jumlah alel (Na) yang hasil percobaan ini (2.76) lebih tinggi daripada nilai yang diperoleh Abdullah et al.
(2011) (populasi elit Deli-AVROS = 2.3) tetapi lebih rendah daripada hasil penelitian Zulhermana (2009) (Nigeria = 3.3), Putri (2010) (Nigeria = 4.5), Ajambang et al. (2012) (populasi alami asal Kamerun = 4.71) dan Billotte et al.
(2001) (LaMé x Dura Deli = 5.25).
Rendahnya jumlah alel yang ditemukan dapat dijelaskan oleh jenis populasi yang digunakan dalam percobaan, dalam kasus ini material yang digunakan adalah populasi elite hasil pemuliaan dan persilangan dari beberapa individu yang mempengaruhi variabel jumlah alel dan populasi (Arias et al. 2012). Hal ini dikarenakan kecenderungan umum kehilangan keragaman genetik yang dikarenakan perbaikan genetik pada kelapa sawit (Arias et al. 2012; Bakoume 2007).
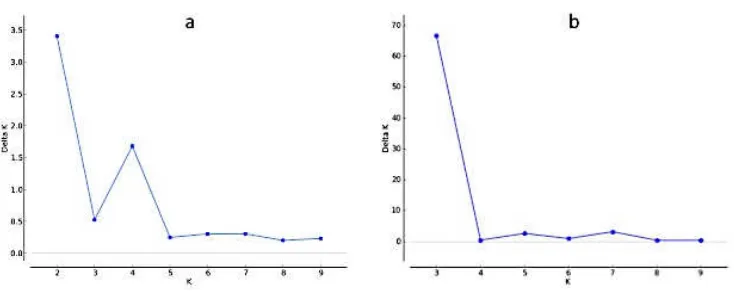
Struktur populasi DP-E ditunjukkan oleh Gambar 3.4. Jumlah subgroup populasi, K, dievaluasi menggunakan metode yang dikemukakan oleh Evanno et al.
(2005). Log likelihood hasil analisis STRUCTURE meningkat secara bertahap dari
! 87!
K= 1 hingga K=10 dan tidak menunjukkan titik optimum yang jelas (data tidak diperlihatkan). Sebaliknya, perhitungan ad hoc maksimum #K diperoleh pada K=2, yang mengindikasikan bahwa famili DP-E dapat dibagi menjadi dua subgrup (Gambar 3.4a). Analisis STRUCTURE secara terpisah juga dilakukan untuk menduga struktur populasinya progeni DP-E. #K menunjukkan nilai maksimum pada K=3, yang mengindikasikan bahwa ada tiga subgrup di progeni DP-E (Gambar 3.4b).
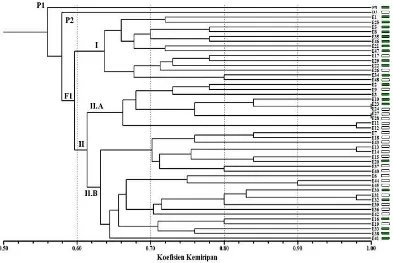
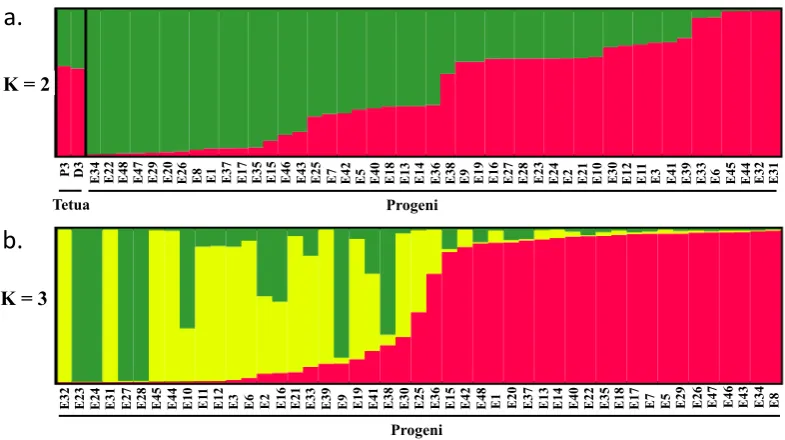
Hasil analisis pengelompokan UPGMA dan STRUCTURE sama-sama mengelompokkan famili DP-E menjadi dua grup dengan tiga subgrup, dengan komposisi grup dan subgrup yang berbeda-beda. Metode analisis seperti UPGMA hanya dapat digunakan untuk menghubungkan prosedur statistik yang memungkinkan untuk identifikasi kluster homogen antar individu (Montilla-Bascon et al. 2013). Program STRUCTURE (Pritchard et al. 2000) sendiri menggunakan pendekatan Bayesian untuk menentukan K (jumlah subpopulasi dalam suatu koleksi) dan mengestimasi proporsi genom setiap aksesi yang berasal dari setiap subpopulasi, atau disebut juga persentase campuran. Hal ini yang menyebabkan perbedaan komposisi grup hasil analisis UPGMA dan STRUCTURE.
Kemiripan genetik tetua jantan (P3) dan tetua betina (D3) yang mencapai 56% pada analisis UPGMA menunjukkan komposisi genetik kedua individu ini tidak berbeda jauh. Analisis STRUCTURE juga memperlihatkan hasil yang serupa (Gambar 3.5a). Meskipun demikian, hasil persilangan dua tetua dengan
origin yang berbeda menghasilkan variasi rekombinan yang tinggi pada
progeninya. Estimasi struktur populasi progeni (Gambar 3.5a) memperlihatkan tidak ada individu progeni yang 100% mendekati kedua tetua. Pada K=3 di populasi progeni (Gambar 3.5b), hanya individu E2 dan E16 yang mendekati pola tetuanya, sedangkan 45 progeni lainnya merupakan rekombinan.
19
Gambar 3.5 Estimasi struktur populasi kelapa sawit DP-E berdasarkan data
genotyping 25 lokus SSR pada famili DP-E (a) dan progeni DP-E
(b) menggunakan program STRUCTURE. Setiap individu direpresentasikan dengan garis vertikal tipis. Jumlah subgrup (K) ditandai dengan perbedaan warna. Tinggi setiap bar
merepresentasikan probabilitas penggolongan setiap individu dalam subgroup.
Metode pemuliaan kelapa sawit dominan menggunakan Reciprocal
Recurrent Selection (RRS) dengan salah satu poin pentingnya adalah penggunaan
dua populasi dasar: populasi ‘A’ terdiri dari dura Deli dari beberapa origin dan populasi ‘B’ terdiri dari Tenera dan Pisifera origin Afrika (Corley and Tinker 2003). Skema persilangan interpopulasi pada RRS memungkinkan ekploitasi heterosis untuk meningkatkan performa persilangan antar dua populasi (Acquaah 2007, Price et al. 2007). Persentase rekombinan yang tinggi dari hasil persilangan tetua P3 dan D3 pada famili DP-E memberikan peluang yang besar untuk eksploitasi vigor hibrida. Individu rekombinan dengan fenotipe yang unggul seperti karakter pendukung hasil, ketahanan penyakit, penambahan tinggi yang lambat dapat digunakan untuk seleksi pada siklus pemuliaan berikutnya.
Simpulan
Segregasi marka yang diperoleh dari hasil seleksi 105 marka SSR cukup tinggi. Marka dengan alel spesifik pisifera dapat digunakan untuk tujuan eksplorasi marka yang berasosiasi dengan karakter warna buah. Pada hasil studi ini, 25 marka SSR mampu memperlihatkan keragaman genetik dan struktur populasi DP-E. Analisis jarak genetik dengan pengelompokan UPGMA
! "9!
4 ASOSIASI MARKA SSR (
SIMPLE SEQUENCE REPEATS
)
DENGAN KARAKTER WARNA BUAH
VIRESCENS
PADA
POPULASI KELAPA SAWIT (
Elaeis guineensis
Jacq.) ASAL
NIGERIA
Abstrak
Program pemuliaan kelapa sawit memiliki beberapa tujuan yaitu meningkatkan hasil dengan berbasiskan area tanam, meningkatkan kualitas minyak, memperlambat laju penambahan tinggi tanaman, dan mengembangkan varietas yang resisten terhadap berbagai hama dan penyakit. Karakter-karakter minor yang mendukung kualitas panen mulai diperhitungkan dalam perakitan varietas. Karakter warna buah seperti karakter Virescens (Vir) dapat digunakan sebagai indikator kematangan buah pada tandan kelapa sawit. Perbedaan warna buah Vir yang kontras antara buah mentah dan masak dapat memudahkan dan mempersingkat waktu yang dibutuhkan untuk panen tanpa harus memungut buah yang jatuh. Konstruksi peta pautan genetik menggunakan 25 SSR menghasilkan empat Kelompok Pautan (KP) dengan total cakupan peta genetik 213.1 cM. Marka SSR mEgCIR3376 diidentifikasi sebagai kandidat marka yang terpaut pada karakter Virescens dengan jarak genetik 27.7 cM, meskipun tidak stabil pada populasi verifikasi. Marka mEgCIR3376 masih dapat digunakan sebagai alat bantu seleksi untuk merakit DxP dengan hasil tinggi dan memiliki karakter Vir
dgn menggunakan famili DP-E.
! ""!
Abstract
The breeding programs in oil palm have several purposes: to increase yield based on land expansion, to increase oil quality, to delay height increament and to develope varieties that resistant to pests and diseases. Minor traits which supported yield quality has been considered in developing improved hybrids. The contrasting color of unripe to ripe fruits virescence (Vir) could be used as indicator of ripeness of oil palm fruit bunches. The use of this phenotypic marker could improve harvest efficiency and reduce cost, instead of counting and collecting loose fruits. The linkage map of 25 SSR markers generated four Linkage Group (LG) covering 213.1 cM. Marker mEgCIR3376 has been identified as candidate marker linked to Virescens trait with 27.7 cM distance. Although the marker showed inconsistent result on verification population, mEgCIR3376 could still be used as a tool to develop DxP with have high yield traits and Virescens fruit color from DP-E family.
Keywords: association marker, linkage map, oil palm, SSR, virescence
Pendahuluan
Program pemuliaan kelapa sawit memiliki beberapa tujuan yaitu meningkatkan hasil dengan berbasiskan area tanam, meningkatkan kualitas minyak, memperlambat laju penambahan tinggi tanaman, dan mengembangkan varietas yang resisten terhadap berbagai hama dan penyakit. Selain itu, program pemuliaan kelapa sawit juga mengarah ke pengembangan materi tanaman dengan sifat-sifat pendukung lainnya, seperti percepatan masa panen, buah dengan bobot kernel yang lebih tinggi, dan kadar karotin serta vitamin E yang tinggi (Nair 2010). Karakter buah lainnya seperti warna buah belum mendapat perhatian dalam program pemuliaan karena dinilai belum memberikan keuntungan yang berarti. eksternal buah tetap hijau (Corley dan Tinker 2003).
Pada penelitian ini, karakter Virescens (Vir) menjadi fokus karena potensinya untuk digunakan sebagai indikator panen. Karakter Vir dikendalikan oleh gen tunggal dominan dan terekspresi pada kondisi dominan homozigot
(Vir/Vir) dan heterozigot (Vir/vir) (Corley dan Tinker 2003). Perbedaan warna
23
sortir di pabrik (May dan Amaran 2011, Fadilah et al. 2012). Karakter Vir ini juga dapat digunakan untuk memastikan kualitas dan kuantitas minyak dari buah yang dipanen karena jika buah dipanen terlalu muda, kandungan minyaknya akan rendah, jika terlalu tua maka banyak buah jatuh yang dipanen dan kadar asam lemak dalam minyak akan terlalu tinggi (Ying et al. 2007).
Metode seleksi berbasis marka molekuler digunakan untuk menyeleksi karakter Vir. Kelebihan dari metode ini adalah tanaman dapat diseleksi secara cepat menggunakan DNA sebelum karakter yang diinginkan terekspresi sehingga proses pemuliaan dapat disingkat. Beberapa jenis marka DNA telah digunakan untuk mencari marka yang terpaut dengan karakter Vir seperti Restriction
fragment length polymorphism (RFLP), Simple Sequence Repeat (SSR) (Singh et
al. 2006), Amplified fragment length polymorphism (AFLP) (Ying et al. 2007).
Simple Sequence Repeats (SSR) atau mikrosatelit, dengan motif pengulangan di- hingga tetra-nukleotida dan diapit oleh sekuen unik, terdapat dalam jumlah yang banyak dan tersebar diseluruh bagian genom eukariotik (Morgante et al. 2002). Dalam beberapa tahun terakhir, SSR menjadi salah satu marka molekuler yang banyak diaplikasikan pada berbagai komoditi seperti beras (Siwach et al. 2004), jagung (Vigouroux et al. 2005) dan komoditi lainnya. SSR memiliki keunggulan karena bersifat kodominan, sangat polimorfik, banyak dan tersebar diseluruh genom, hampir selalu berupa lokus tunggal, tidak ambigu, secara spesifik terpetakan di genom, dan berdasarkan pada teknologi berbasis PCR (Chen et al. 2007).
Tujuan penelitian ini adalah untuk mencari marka SSR yang berasosiasi dengan karakter warna buah Virescens menggunakan 25 marka SSR (Simple
Sequence Repeats) hasil seleksi dari studi sebelumnya.
Bahan dan Metode
Bahan tanaman
Bahan tanaman yang digunakan dalam studi adalah 47 progeni famili DP-E hasil persilangan berasal dari Pisifera asal Nigeria dan Dura MARDI. Populasi yang digunakan merupakan populasi elite pada koleksi PT Bina Sawit Makmur (anak perusahaan PT Sampoerna Agro Tbk.). Verifikasi marka dilakukan pada progeni empat famili (G, H, K, L) dengan jumlah sampel dari masing-masing famili 20 sampel. 20 sampel dari setiap famili terdiri dari 10 sampel Virescens dan 10 sampel nigrescens dan dipilih secara acak. Progeni famili G, H, K dan L merupakan half-sib dengan progeni famili DP-E.
Ekstraksi DNA dan prosedur SSR
Sampel daun tombak digunakan untuk ektraksi DNA. DNA diisolasi menggunakan metode CTAB berdasarkan modifikasi dari Orozco-Castillo et al.
! ":!
buffer ekstraksi 1 000 µl dan 0.1 mg PVP. Hasil gerusan dimasukkan ke dalam tabung mikro 2 ml yang telah berisi 500 µl buffer ekstraksi (CTAB) dan 10 µl mercaptoethanol. Hasil gerusan diinkubasi selama 60 menit di waterbath dengan suhu 65 °C dan dibalik manual setiap 10 menit. Setelah inkubasi, KIAA ditambahkan kedalam campuran hingga total volume 2 ml dan divortex hingga homogen, diikuti sentrifusi dengan kecepatan 11 000 rpm selama 10 menit. Supernatan diambil dan dipindahkan ke tabung mikro 2 ml baru dan ditambahkan 1 ml KIAA, divortex hingga homogen. Campuran disentrifusi kembali dengan kecepatan 11 000 rpm selama 10 menit.
Supernatan hasil sentrifusi kedua dipindahkan ke tabung mikro 1.5 ml baru, ditambahkan Na-asetat 1/10 dari volume total dan etanol p.a hingga total volume 2 ml. Tabung dibalik berulangkali secara perlahan hingga timbul benang halus berwarna putih bening atau kekuningan. Tabung diinkubasi di suhu 4 °C selama 60 menit, setelahnya disentrifusi pada 11 000 rpm selama 10 menit. Supernatan dibuang dan pelet dikeringanginkan selama 15-30 menit. Buffer TE 1x sebanyak 300 µl ditambahkan ke dalam tabung dan dibalik berulangkali secara manual hingga larut, inkubasi di 4 °C selama 60 menit. Setelah inkubasi, RNAase 3.3 µl ditambahkan dalam tabung dan inkubasi di 37 °C selama 60 menit. Suspensi ditambahkan 1 ml etanol p.a dan disimpan pada suhu 4 °C selama 60 menit, kemudian disentrifusi pada 11 000 rpm selama 10 menit. Supernatan dibuang dan pelet dikering-anginkan selama 15 menit, kemudian ditambahkan 0.5 ml etanol 70% untuk membersihkan larutan CTAB yang tersisa. Tabung dibalik berulangkali secara perlahan, etanol dibuang dan pelet dikering-anginkan selama 15-30 menit. Pelet yang telah mengering disuspensikan kembali dengan menambahkan 200 µl buffer TE 1x, dibalik manual dan perlahan untuk menghindari terputusnya DNA. Suspensi yang dihasilkan merupakan DNA stok dan disimpan di suhu -20 °C untuk penggunaan selanjutnya.
Pengecekan kualitas dan kuantitas DNA menggunakan 0.8 % gel agarose dan diwarnai dengan GelRedTM. Gel divisualisasi dengan Bio Rad Gel DocTM UV-Transluminator. Campuran Polymerase Chain Reaction (PCR) terdiri dari 9.5 µl ddH2O, 12.5 µl Promega GoTaq® Green master mix (Taq DNA polymerase, dNTPs, MgCl2 dan buffer reaksi), 1.0 µl primer forward, 1.0 µl primer reverse dan 5 ng/µl DNA template. PCR dijalankan dengan tahap denaturasi pertama pada 95 °C selama 1 menit, tahap denaturasi kedua pada 94 °C selama 30 detik, tahap
annealing sesuai dengan Ta primer selama 1 menit, tahap extension pada 72 °C
selama 2 menit dan tahap final extension pada 72 °C selama 8 menit. Proses dijalankan dengan 35 siklus sebelum suhu diturunkan ke 4 °C. Produk PCR dipisahkan menggunakan dengan Cole-Parmer® Dedicated Height Sequencer
pada gel akrilamid 6 % dengan buffer SB 1X (Brody dan Kern 2004) dan diwarnai dengan perak nitrat (Creste et al. 2001) untuk visualisasi.
Seleksi Primer dan Genotyping
25
Gambar 4.1 Separasi pita DNA pada gel akrilamid 6 % dan contoh skoring
DNA dari 10 individu dura dalam genotipe yang sama. Pada tahap genotyping, populasi yang digunakan adalah famili DP-E dengan 47 sampel progeni, tetua jantan (P3) dan betinanya (D3). Primer yang digunakan pada tahap ini adalah 25 primer SSR.
Verifikasi kandidat marka dilakukan pada progeni empat famili yaitu famili G, H, K dan L dengan sampel kontrol tetua dari masing-masing famili. Sampel dari setiap famili adalah 20 individu progeni yang terdiri dari 10 individu
Vir dan 10 individu nig.
Skoring
Pita-pita yang diperoleh pada plat kaca diskoring manual sebagai data biner dengan kode (1) jika ada pita dan (0) jika tidak ada pita dan data genotipe. Dalam tahap seleksi primer, primer-primer yang menghasilkan pita polimorfik akan digunakan dalam tahapan genotyping. Skoring pita DNA diilustrasikan pada gambar 4.1.
Analisis Data
Data hasil skoring ditabulasikan dalam excel dan digunakan untuk analisis asosiasi marka dengan karakter Virescens menggunakan software
! "6!
Hasil dan Pembahasan
Kriteria untuk menyeleksi primer yang akan digunakan pada studi ini adalah pita yang dihasilkan oleh primer harus polimorfik, pola pita pada individu Pisifera adalah heterozigot dan salah satu atau kedua pita tersebut memiliki ukuran basa yang berbeda dengan panjang pita pada sampel Dura. Berdasarkan kriteria di atas, 25 primer polimorfik terpilih untuk digunakan pada tahap
genotyping (Gambar 4.2).
Gambar 4.2 Profil marka SSR pada kelapa sawit genotipe Pisifera (P1-P10) dan Dura bulk (D1-D5) menggunakan primer mEgCIR3376 pada tahap skrining primer. Tanda panah menunjukkan posisi alel pada pisifera yang tidak dimiliki oleh dura.
Profil SSR pada famili DP-E dapat memberikan beberapa informasi genetik seperti membedakan tanaman homozigot dan heterozigot, PIC
(Polymorphic Information Content), heterozigositas dan jumlah alel pada lokus
yang diamplifikasi. Nilai PIC maksimum diperoleh pada angka 0.701 pada primer
mEgCIR3310 yang menunjukkan lokus ini yang paling informatif. Primer
mEgCIR0146 dan mEgCIR2860 adalah lokus yang paling kurang informatif (PIC
= 0.272). Nilai Ho berkisar antara 0.41 hingga 1(rataan Ho = 0.75), dan kisaran nilai He adalah 0.33-0.75 (rataan He = 0.54). Nilai rata-rata heterozigositas yang diharapkan (He) dan heterozigositas yang diamati (Ho) mengindikasikan keragaman genetik yang relatif tinggi intrapopulasi DP-E. Total alel yang dihasilkan dari 25 primer SSR adalah 69 alel dengan kisaran alel efektif 1.48 – 3.97 dan rerata alel efektif (Ne) 2.44.
Marker Assisted Selection menggunakan marka SSR
Segregasi 25 primer terpilih diuji menggunakan khi kuadrat dengan ekspektasi Mendel 1:1 pada taraf 0.05. Seluruh marka yang diuji memenuhi rasio tersebut sehingga dapat digunakan untuk dalam pembentukan peta genetik. Lokus warna buah dimasukkan sebagai lokus tambahan selain 25 lokus lainnya untuk mencari marka yang berasosiasi dengan warna buah.
27 beberapa lokus kemungkinan disebabkan karena jumlah sampel yang digunakan hanya 49 individu.
Resolusi peta genetik yang dihasilkan dalam studi ini jauh lebih rendah dibandingkan peta genetik yang dihasilkan Billotte et al. (2005) dengan cakupan peta genetik total 1 415 cM (densitas marka rata – rata = 5 cM); Billotte et al.
(2010) dengan cakupan 1 479 cM (densitas marka rata – rata = 6 cM); Singh et al.
! "7!
yang terbatas dalam studi ini jelas mempengaruhi cakupan peta genetik yang diperoleh dalam studi ini.
Tabel 4.2 Pemetaan lokus SSR pada kelapa sawit famili DP-E dengan kriteria LOD 0.56-10.56 dan fraksi rekombinan 0.25
KP Lokus cM LOD LGa
1 mEgCIR0773 17.7 5.56 15
mEgCIR2860 72.6 0.56 15
mEgCIR0037 15
2 mEgCIR0146 35.7 2.55 10
mEgCIR0825 14.7 6.35 10
mEgCIR0788 10
3 mEgCIR3376 27.7 3.58 8
Warna
4 mEgCIR3813 40.3 2.11 1
mEgCIR3819 4.4 10.56 1
mEgCIR3788 1
Keterangan: cM = Haldane centi morgan, LG = Linkage Group, a Berdasarkan Billotte et al. (2005)
29
Hasil analisis pautan genetik memperlihatkan bahwa karakter warna terpaut dengan lokus mEgCIR3376 di KP 3 (Gambar 4.3) atau linkage group (LG) 8 (Billotte et al. 2005) dengan jarak 27.7 cM. Jarak genetik kandidat marka yang diperoleh dalam studi ini masih lebih jauh dibandingkan dengan kandidat marka AFLP yang dihasilkan Ying et al. (2007) yaitu 6 cM. Studi Singh et al. (2014) melaporkan adanya marka RFLP MET16 yang terpaut erat dengan gen Vir. Hasil penelitian tersebut juga mengidentifikasi gen VIR. Marka MET16 dan gen Vir ini terletak pada kelompok pautan (KP) 1 (Singh et al. 2013) atau LG 8 pada peta genetik Billotte et al. (2005). Hasil penelitian ini sejalan dengan hasil studi ini dimana lokus Vir juga terpaut pada LG 8. Jarak genetik yang jauh dengan gen target akan meningkatkan resiko terjadinya rekombinasi pada saat meiosis dan mengakibatkan hilangnya marka pada generasi berikutnya.
Gambar 4.4 memperlihatkan profil marka SSR mEgCIR3376 pada famili DP-E. Sampel tetua jantan (P3) dan tetua betina (D3) digunakan sebagai kontrol alel sekaligus untuk verifikasi segregasi alel pada progeni (E1-E48). Pada lokus
mEgCIR3376, tetua jantan (pembawa sifat Vir) memiliki posisi alel 1 dan 2 yang
berbeda dengan tetua betina (nig) (alel 3).
Segregasi alel pada progeni menunjukkan 19 individu Vir dengan posisi alel 13 (satu tiga), 18 individu nig dengan posisi alel 23 (dua tiga), empat individu
Vir dengan posisi alel 23 (dua tiga) dan enam individu nig dengan posisi alel 13 (satu tiga). Individu Vir dengan alel 13 (satu tiga) dan individu nig dengan alel 23 (dua tiga) merupakan turunan tipe tetua; sedangkan individu Vir dengan alel 23 dan individu nig dengan alel 13 merupakan tipe rekombinan.
Konfigurasi alel tetua famili DP-E dapat diprediksi berdasarkan pola segregasi alel pada progeni. Gambar 4.5 memperlihatkan dua kemungkinan konfigurasi alel tetua P3 dan D3. Hasil segregasi alel pada progeni DP-E mengarah ke tipe A, dimana individu Vir alel 13 dan individu nig alel 23 jumlahnya lebih banyak dan merepresentasikan tetua, sedangkan total individu tipe rekombinan lebih sedikit.