AMPLIFIKASI GEN PENYANDI hemolysin
DARI BAKTERI Vibrio harveyi
DENGAN TEKNIK PCR(Polymerase Chain Reaction)
MUHAMMAD ARIF MULYA
DEPARTEMEN BUDIDAYA PERAIRAN
FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR
2011
AMPLIFIKASI GEN PENYANDI hemolysin
DARI BAKTERI Vibrio harveyi
DENGAN TEKNIK PCR(Polymerase Chain Reaction)
MUHAMMAD ARIF MULYA
SKRIPSI
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Perikanan pada Program Studi Teknologi dan Manajemen Perikanan Budidaya
Departemen Budidaya Perairan Fakultas Perikanan dan Ilmu Kelautan
Institut Pertanian Bogor
DEPARTEMEN BUDIDAYA PERAIRAN
FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR
2011
PERNYATAAN MENGENAI SKRIPSI
DAN SUMBER INFORMASI :
Dengan ini saya menyatakan bahwa skripsi yang berjudul :
AMPLIFIKASI GEN PENYANDI hemolysin DARI BAKTERI Vibrio harveyi DENGAN TEKNIK PCR (Polymerase Chain Reaction)
Adalah benar merupakan hasil karya yang belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Semua sumber data dan informasi yang berasal atau dikutip dari karya yang diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Bogor, Januari 2011
MUHAMMAD ARIF MULYA C14052761
LEMBAR PENGESAHAN
Judul : Amplifikasi Gen Penyandi hemolysin DariBakteri Vibrio harveyi Dengan Teknik PCR (Polymerase Chain Reaction)
Nama : Muhammad Arif Mulya
Nrp : C14052761
Program Studi : Teknologi dan Manajemen Perikanan Budidaya Departemen : Budidaya Perairan
Menyetujui,
Pembimbing I Pembimbing II
Dr. Munti Yuhana, S.Pi. M.Si Dr. Ir. Sukenda, M.Sc
NIP. 196912201994032002 NIP. 196710131993021001
Mengetahui,
Ketua Departemen Budidaya Perairan
Dr. Ir. Odang Carman, M.Sc NIP. 195912221986011001 Tanggal Pengesahan:
i
KATA PENGANTAR
Puji syukur dipanjatkan kehadirat Allah SWT, karena atas berkat rahmat serta karunia-Nya, penulis dapat menyelesaikan penyusunan skripsi hasil penelitian dengan baik. Penelitian ini berjudul “Amplifikasi Gen Penyandi
hemolysin Dari Bakteri Vibrio harveyi Dengan Teknik PCR (Polymerase Chain Reaction)”. Untuk itu penulis mengucapkan terima kasih kepada:
1. Ibu Dr. Munti Yuhana selaku pembimbing I yang telah dengan sabar memberikan bimbingan dan masukan selama penyusunan skripsi.
2. Bapak Dr. Sukendaselaku pembimbing II yang telah memberikan masukan selama penyusunan skripsi.
3. Bapak Dr.Odang Carman selaku Ketua Departemen Budidaya Perairan. 4. Bapak Dr.Alimuddin selaku Ketua Program Studi.
5. Ibu Ir. Rina Martini M.Si. selaku Kepala Laboratorium Mikrobiologi program Diploma Institut Pertanian Bogor, yang selalu membimbing dan memberikan bantuan.
6. Bapak Ranta, sebagai laboran di Laboratorium Kesehatan Ikan. Yang memberikan bimbingan dan arahan selama penelitian.
7. Keluarga yang selalu memberikan motivasi selama penelitian.
8. Teman-teman dari IPB, STIE PANDU, Tentor denrace primaatas kebersamaannya.
Penulis menyadari dalam penulisan skripsi penelitian masih terdapat kekurangan. Oleh karena itu, penulis memohon maaf apabila terdapat kesalahan dalam penyusunan skripsi ini. Penulis berharap semoga penelitian ini dapat bermanfaat.
Bogor, Januari 2011
ii
DAFTAR RIWAYAT HIDUP
Penulis dilahirkan di Bogor pada tanggal 18 Maret 1986 dari pasangan Bapak Tatang dan Ibu Aan.Penulis merupakan anak keenam dari tujuh bersaudara.
Pendidikan formal yang dilalui penulis adalah SD Negeri II Cibitung, SLTP Negeri 2 Ciampea, SMA Negeri 1 Ciampea dan lulus tahun 2005. Pada Tahun yang sama penulis lulus seleksi masuk IPB melalui jalur Undangan Seleksi Masuk Institut Pertanian Bogor (USMI) dan pada tahun 2006 memilih Program Studi Teknologi dan Manajemen Perikanan Budidaya, Fakultas Perikanan dan Ilmu Kelautan dengan kompetensi minor Manajemen Fungsional, Fakultas Ekonomi dan Manajemen.
Selama Mengikuti perkuliahan, penulis aktif menjadi pengurus Forum Keluarga Muslim C (2007-2008) dan pengurus Himpunan Mahasiswa Akuakultur (Himakua) bidang kesekretariatan (2006-2007). Penulis juga pernah menjadi asisten pada mata kuliah Dasar-Dasar Mikrobiologi Akuatik semester genap 2007/2008 dan 2008/2009, Dasar-Dasar Genetika Ikan semester ganjil 2008/2009, Penyakit Organisme Akuatik semester ganjil 2008/2009, Manajemen Kesehatan Akuakultur semester ganjil 2008/2009. Untuk menambah pengetahuan dalam budidaya perairan penulis mengikuti praktek lapangan akuakultur Pembenihan Udang Vannamei (Littopenaeus vannamei) di PT. Tirtamutiara Makmur Situbondo, Jawa Timur (Juli-Agustus, 2008).
Untuk menyelesaikan studi penulis melakukan penelitian berjudul “AMPLIFIKASI GEN PENYANDI hemolysin DARI BAKTERI Vibrio
iii
ABSTRAK
MUHAMMAD ARIF MULYA.Amplifikasi gen penyandi hemolysin dari bakteri Vibrio harveyi dengan teknik PCR (Polymerase Chain Reaction). Dibimbing oleh MUNTI YUHANA dan SUKENDA.
PenyakitVibriosis yang disebabkan oleh bakteri Vibrio harveyi berpendar,masih menjadi kendaladalam budidayaudang vannamei hingga saat ini.Pada saat wabah, kepadatan populasi bakteri ini ribuan kali sehingga menyebabkan kematian missal larva udang hingga100%. Deteksi cepat danakuratuntuk mengetahuikeberadaan bakteri pathogen pada saat populasi masih rendah mutlak dibutuhkan.Salah satu metode yang dipakai adalah dengan teknik berbasis PCR (PolymeraseChain
Reaction).Penelitianinidilakukan untuk mengevaluasi
aplikasipasanganprimerpenyandigenhemolysinuntuk mendeteksi keberadaanbakteri V.harveyiyang merupakan isolat lokal Indonesia. Sebanyak 8 isolat bakteri terpilih hasil isolasi dilakukan uji konfirmasi dengan pewarnaan Gram, penghitungan optical density (OD) bakteri, uji hemolisisdarah,ekstraksiDNAdanoptimasi PCR. Hasil uji hemolisis darahmenunjukkan bahwasemua isolat mampumelisisdarahpadamediablood
agar.GenhemodariV.harveyidengan primerMyhemo
forwardF15’-GATGGTCAGTGCCTCTCA-3’, dan
reverseR15’-CCCAGTTGTATAGCGGTA-3’berhasil teramplifikasipada isolat U5dan V-U24, menghasilkan produk berukuran sekitar 518bp. Program PCR yang digunakan yaitu ; denaturasi 94oC selama 1 menit, annealingprimer 50oC selama 1 menit dan tahap ektension DNA pada 72 oC selama 1 menit sebanyak 35 siklus. Kata kunci : luminous vibriosis, PCR, gen hemolysin.
iv
ABSTRACT
MUHAMMAD ARIF MULYA. Amplification of hemolysingene of Vibrio harveyiby Polymerase Chain Reaction(PCR) technique.Supervised by MUNTI
YUHANA and SUKENDA.
Vibriosis disease which is caused by lumiunescent Vibrio harveyi until today become a trouble in the cultivation of vannamei shrimp. When the epidemic occurs and the density of bacterial population reachs in thousands times, it causes the mass mortality of the shrimp larvae. Therefore the diagnose method that can accurately and quickly detect this bacterial pathogen is absolutely required. One of method that commonly used is PCR (polymerase chain reaction)-based method. The objective of this research was to evaluate the primer pair application targeted the hemolysin gene, so that primer pair which has been designed can successfully amplify and therefore can detect the presence of hemolysin gene in V.
harveyi. This primer pair was designed from the local vannamei strain isolated
from Indonesia. Eight vibrio isolates obtained from shrimp, were used in this study. After the cell morphologically tested with Gram staining, then the following methods were applied as follows ; optical density count, hemolysin test, DNA extraction and PCR method optimalization. The result of blood hemolysis test showed that all of V. harveyi isolates produced clear zones surround their colonies on blood agar medium. The PCR result using Myhemo primer pair of F1
5’-GATGGTCAGTGCCTCTCA-3’(forward) and R1
5’-CCCAGTTGTATAGCGGTA-3’(reverse) successfully amplified the hemolysin gene of isolates V-U5 and V-U24 in size of approximately of 518 bp. The PCR programme used in this study was at the following conditions ; DNA denaturation at 94oC for 1 min, primer annealing at 50oC for 1 min and DNA extension at 72 o
C for 1min as many as 35 cycles.
v
DAFTAR ISI
PERNYATAAN MENGENAI SKRIPSI ... 3
DAN SUMBER INFORMASI : ... 3
LEMBAR PENGESAHAN ... 4
KATA PENGANTAR ... i
DAFTAR RIWAYAT HIDUP ... ii
ABSTRAK ... iii
MUHAMMAD ARIF MULYA. Amplifikasi gen penyandi hemolysin dari bakteri Vibrio harveyi dengan teknik PCR (Polymerase Chain Reaction). Dibimbing oleh MUNTI YUHANA dan SUKENDA. ... iii
ABSTRACT ... iv
DAFTAR ISI ... v
DAFTAR GAMBAR ... vi
DAFTAR TABEL ... vii
DAFTAR LAMPIRAN ... viii
I. PENDAHULUAN ... 1
II. BAHAN DAN METODE ... 5
2.1 Isolasi Dan Karakterisasi Pewarnaan Gram Bakteri Vibrio harveyi ... 5
2.2 Karakterisasi Ekspresi Gen Hemolysin Isolat Pada Media Agar Darah. ... 5
2.3 Ekstraksi DNA ... 6
2.4 PCR ... 7
2.5 Elektroforesis ... 10
III. HASIL DAN PEMBAHASAN ... 11
3.1 HASIL ... 11
3.1.1 Isolasi Vibrio harveyi ... 11
3.1.2 Karakterisasi Ekspresi Gen Hemolysin Isolat Vibrio Pada Media Agar Darah. ... 11
3.1.4 Amplifikasi gen hemolysin Myhemo Primer ... 15
3.1.5 Amplifikasi gen hemolysin Myhemo Primer F1R1 hasil Pengenceran 17 3.2 Pembahasan ... 18
IV. KESIMPULAN DAN SARAN ... 23
vi
DAFTAR GAMBAR
Gambar
Halaman
Gambar 1 ... 8
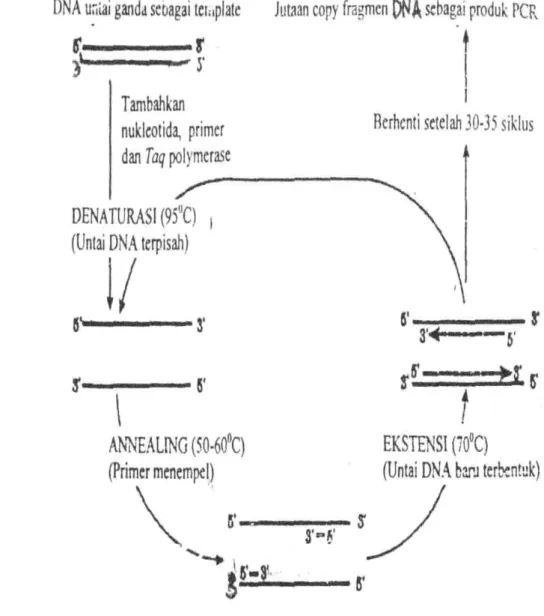
Gambar 2Siklus pembentukan molekul DNA baru dalam proses PCR.(Muladno, 2002). ... 9
Gambar 3. Morfologi koloni isolat Vibrio sp. pada media TCBS ... 11
Gambar 4. Hasil uji hemolisis pada isolat-isolat Vibrio yang ditumbuhkan pada media blood agar. ... 12
Gambar 5. Elektroforesis genom Vibrio sp... 13
Gambar 6. Grafik semi log Optical Density (OD) bakteri galur V-U5. ... 14
Gambar 7. Grafik Semi log Optical Density (OD) bakteri V-U24 ... 15
Gambar 8. Gambar gel elektroforesis hasil amplifikasi PCR menggunakan primer Myhemo F1R1 ... 16
Gambar 9.Gambar gel elektroforesis hasil amplifikasi PCR dengan menggunakan primer Myhemo F1R1 ... 16
vii
DAFTAR TABEL
Tabel Halaman
Tabel 1. ”Master mix” yang digunakan untuk PCR ... 7 Tabel 2.Karakterisasi /uji hemolisis pada blood agar dari isolat-isolat Vibrio ... 11 Tabel 3. Kuantifikasi DNA genom isolat Vibrio sp. ... 13 Tabel 4. Hasil pengukuran Optical Density (OD) bakteri galur V-U5 yang dikultur 18 -24 jam pada media LB (luria bertani). ... 14 Tabel 5. Hasil pengukuran Optical Density (OD) bakteri galur V-U24 yang dikultur 18-24 jam pada media LB (luria bertani). ... 15
viii
DAFTAR LAMPIRAN
Lampiran Halaman
Lampiran 1. Penghitungan bakteri dengan metode hitungan cawan sebar ... 27
Lampiran 2. Pewarnaan Gram... 28
Lampiran 3. Media Luria bertani (LB) ... 29
Lampiran 4. Prosedur penggunaan spektrofotometer ... 29
1
I.PENDAHULUAN
Udang vaname merupakan salah satu komoditas unggulan dalam perikanan budidaya Indonesia.Dan kini semakin populer dikalangan petambak seiring dengan adanya keputusan Menteri Kelautan dan Perikanan No. KEP. 01/MEN/2002 tentang sistem manajemen mutu terpadu hasil perikanan dan standar nasional Indonesia (SNI) no 01-7246-2006 tentang produksi udang vaname (Litopenaeus vannamei) di tambak dengan teknologi intensif untuk dapat dipergunakan oleh pembenih, pembudidaya, pelaku usaha dan instansi yang memerlukan (BSN, 2007).
Sebagai salah satu komoditas primadona untuk ekspor non migas, selama periode tahun 2003-2007 ekspor udang cenderung meningkat, yaitu dari 137.636 ton pada tahun 2003 menjadi 160.797 ton pada tahun 2007 (Poernomo, 2008). Kemudian berdasarkan data dari Kementerian Kelautan dan Perikanan (KKP) menunjukkan adanya kenaikan produksi udang nasional sebesar 2,6 % dari 338.060 ton pada 2009 menjadi 352.600 ton pada 2010. Bahkan Departemen Kelautan dan Perikanan (DKP) menargetkan dalam kurun waktu tahun 2010 – 2014 produksi udang akan ditingkatkan sampai 74,75% dari 400.000 ton menjadi 699.000 ton yang terdiri dari jenis udang vanname dan udang windu (Trobos, 2011).
Udang vaname memiliki beberapa keunggulan dibanding spesies udang lainnya, diantaranya produktivitas tinggi dapat mencapai lebih dari 13.600 kg/ha (Boyd dan Clay, 2002), masa panen lebih cepat, dan lebih resisten terhadap penyakit. Meskipun udang vaname lebih resisten terhadap penyakit, tidak dapat dipungkiri bahwa penyakit Vibriosis yang disebabkan oleh Vibrio harveyi berpendar, masih menjadi masalah dalam budidaya udang vannamei hingga saat ini.
Luminous Vibriosis atau penyakit udang berpendar disebabkan oleh bakteri Vibrio harveyi. Penyakit berpendar pada larva udang vaname termasuk penyakit berbahaya karena setiap saat dapat mengakibatkan kematian massal.
2
berbagai ikan laut dan udang di berbagai belahan dunia (Pass et al, 1987; Saeed, 1995; Zhang and Austin, 2000). Vibrio sp. berpendar juga dilaporkan sebagai agen penyebab utama pada penyakit vibriosis pada udang budidaya dan menyebabkan kerugian yang sangat besar di wilayah Asia Tenggara (Lavilla-Pitogo et al, 1990; Karuna Sagar et al, 1994; Liu et al, 1996). Vibriosis juga telah menyebabkan mortalitas pada berbagai stadia larva, pasca larva, juvenil dan dewasa (Lightner, 1983). Dampak buruk terhadap udang budidaya akibat penyakit ini, menuntut agar diperoleh informasi yang memadai mengenai cara mendeteksi keberadaan penyakit ini sejak dini. Hal ini dilakukan untuk mengambil tindakan selanjutnya yang paling tepat. Seperti dalam pengambilan keputusan untuk melakukan emergency harvesting, dan keputusan untuk menggunakan broodstock maupun larva yang akan dibudidayakan.
Deteksi dini terhadap keberadaan agen penyakit merupakan cara yang tepat untuk mengetahui penyebab serangan dan jenis penyakitnya. Jenis penyakit perlu dipastikan secepat mungkin, karena air sebagai medium hidup ikan akan memungkinkan penularan penyakit secara meluas dalam waktu relatif cepat. Pemeriksaan penyakit Vibriosis secara konvensional dengan melihat gejala klinis pada tubuh udang, mengisolasi bakteri penyebab penyakit, melakukan uji fisiologis dan biokimia membutuhkan waktu yang relatif lama. Untuk kasus penyakit Vibriosis berpendar, secara visual dapat dilihat pendaran atau luminesensinya pada malam hari, jika hal ini terjadi maka dapat dipastikan populasi sel bakteri vibrio sudah dalam kepadatan yang tinggi. Salah satu metode yang dapat digunakan untuk deteksi cepat keberadaan penyakit Vibriosis yang menyerang udang vaname adalah dengan teknik PCR (Polymerase Chain
Reaction). Dengan metode ini hanya dalam waktu hitungan jam saja hasilnya
sudah dapat diketahui.
PCR merupakan suatu metode in vitro yang digunakan untuk mensintesis sekuen DNA tertentu dengan menggunakan dua primer oligonukleotida yang menghibridisasi pita yang berlawanan dan mengapit dua target DNA (Erlich, 1989). PCR merupakan suatu teknik amplifikasi rantai DNA yang diinginkan secara in vitro. PCR dapat digunakan untuk menghasilkan sejumlah DNA yang
3
bisa dideteksi hanya dari satu molekul saja dan dapat digunakan untuk mengidentifikasi, mengkarakterisasi dan menganalisa bagian spesifik dari DNA maupun RNA (Powledge, T. M.,2003 dalam Yufrialdi, 2008).
Dalam penggunaan PCR perlu adanya gen penyandi untuk mengidentifikasi gen target secara spesifik, sehingga proses deteksi terhadap agen penyakit dapat dilakukan. Gen penyandi yang umum digunakan adalah 16S-rRNA. Namun penggunaan 16S-rRNA ini masih kurang spesifik dalam mengidentifikasi mikroorganisme sampai ke tingkat spesies. Dalam kasus
V.harveyi sangat sulit dibedakan dari jenis vibrio lainnya, dari kelompok inti (V. alginolyticus, V.campbellii, V. parahaemolyticus dan V. rotiferinus) misalnya V. harveyi, V. campbellii, dan V. rotiferinus memiliki lebih dari 99% kesamaan
untuk urutan identitas 16S rRNA (Gomezz-Gil et al. 2003 dalam Gomez et al. 2009).
Menurut Yuhana et al (2008) karakterisasi gen-gen 16S rRNA dari isolat-isolat Vibrio dengan sekuensing nukleotida, menunjukkan tingkat homologi yang sangat tinggi (homologi sebesar 99%) dengan sekuen nukleotida Vibrio harveyi. Konsekuensinya, potensi penggunaannya untuk penanda spesifik adalah kurang. Alternatif gen penyandi lain yang dapat digunakan untuk mengidentifikasi mikroorganisme melalui teknik PCR yaitu hemolysin (Yuhana et al, 2008).
Hemolysin merupakan bagian yang bertanggung jawab terhadap kerusakan
membran eritrosit (hemolisis) dan dilaporkan gen hemolysin terdapat pada beberapa genus termasuk V. harveyi (Hirono et al, 1996; Nishibuchi and Kaper, 1995; Nishibuchi et al, 1990; Zhang et al, 2001). Gen hemolysin sudah pernah diusulkan pada penelitian terdahulu (Conejero dan Hedreyda, 2004) sebagai penanda untuk deteksi Vibriosis. Sekuen gen hemolysin ini bervariasi pada spesies bakteri yang berbeda, sehingga gen ini dapat digunakan untuk mendeteksi keberadaan bakteri Vibrio sampai pada tingkat spesifik spesies.
Dengan demikian, penelitian ini dilakukan untuk mengevaluasi aplikasi pasangan primerpenyandi gen hemolysin sehingga dapat diketahui apakah sekuen primer gen penyandi hemolysin yang telah didesain mampu mengamplifikasi gen
4
genom dan amplifikasi gen penyandi hemolysin pada Vibrio harveyi digunakan sebagai langkah awal deteksi dini keberadaan bakteri V.harveyi pada udang vaname.
5
II.BAHAN DAN METODE
2.1 Isolasi Dan Karakterisasi Pewarnaan Gram Bakteri Vibrio harveyi
Media TCBS disiapkan dalam keadaan aseptik, sampel udang yang terserang vibriosis dipindahkan menggunakan ose dan digoreskan ke media TCBS kemudian diinkubasi selama 24 jam. Identifikasi bakteri Vibrio harveyi dengan menggunakan metode pewarnaan gram atau secara sederhana yaitu dengan cara media TCBS dimasukkan ke dalam ruang gelap, jika terbentuk warna hijau berpendar maka bakteri yang tumbuh tersebut merupakan bakteri V.harveyi. Bakteri V. harveyi yang diperoleh dikultur murni dan ditumbuhkan pada media TCBS dengan menggunakan agar miring. Selanjutnya, kultur dipindahkan dari media padat ke media cair (NB) yang ditambahkan dengan NaCl 1,5% dan biakan diletakkan dalam shaker selama 24 jam.
Langkah awal dalam pewarnaan Gram (Lampiran 2) yaitu bakteri V.
harveyi dioleskan diatas gelas objek. Kemudianlarutan kristal violet diteteskan
pada preparat olesan bakteri sebanyak 2-3 tetes dan dibiarkan selama 1 menit. Selanjutnya preparat dicuci dengan air mengalir dan dikeringkan dengan kertas isap. Kemudian preparat diteteskan larutan Kalium Iodida sebanyak 2-3 tetes dan dibiarkan selama 1 menit. Preparat dicuci kembali dengan air dan dikeringkan. Tahap selanjutnya preparat diteteskan alkoholdan didiamkan selama 30 detik, kemudian preparat dicuci dan dikeringkan kembali. Selanjutnya larutan safranin diteteskan dan didiamkan selama 30 detik, kemudian safranin dicuci dan dikeringkan dengan kertas isap. Kemudian preparat diamati dibawah mikroskop.
2.2 Karakterisasi Ekspresi Gen Hemolysin Isolat Pada Media Agar Darah. Media agar darah (blood agar), merupakan media differensial yang berfungsi untuk membedakan bakteri berdasarkan kemampuan mereka untuk melisiskan sel-sel darah merah. Pembuatan media agar darah dimulai dengan melarutkan TSA sebanyak 40 gram dalam akuades sebanyak 1000 ml (Lampiran 5), kemudian pH media diukur sampai mencapai 7,3. Kemudian media dipanaskan di penangas sampai larut dan diaduk sampai homogen. Setelah itu media
6
disterilkan. Ketika menunggu proses sterilisasi media, hangatkan darah kambing segar sampai suhu 500C sebanyak 5% dari volume total media atau sebanyak 50 ml yang sudah didefibrinasi dengan menggunakan larutan Na citrat. Dinginkan pula TSA steril sampai suhu mencapai 500C (Hadioeutomo, 1993).
Secara aseptik kemudian darah kambing segar dituangkan ke dalam labu berisi TSA dan dicampur dengan cara memutar-mutar labu tersebut dengan hati-hati. Kemudian media dituangkan sebanyak 12 ml ke dalam cawan petri steril. Kemudian biakan bakteri ditanam dengan menggunakan ose dan digoreskan ke media agar darah dan diinkubasikan selama 24 jam pada suhu ruang. Ekspresi dari gen hemolysin dapat diketahui dari ada tidaknya zona bening di sekitar goresan/koloni dari kultur yang ditumbuhkan yang menunjukkan adanya lisis sel darah merah.
2.3 Ekstraksi DNA
Vibrio harveyi yang dikultur selama 18-24 jam dalam media cair LB (luria bertani) (lampiran 3) diambil dengan menggunakan mikropipet sebanyak 1 ml
dan dimasukkan ke dalam eppendorf steril. Kemudian eppendorf disentrifuse selama 1 menit pada suhu 4oC dan kecepatan 12000 rpm. Selanjutnya bagian supernatan dibuang, pellet disuspensi kembali dengan menambahkan 500 µl buffer steril dan disentrifuse kembali pada kecepatan 12000 rpm selama 5 menit. Supernatan dibuang, kemudian ke dalam eppendorf diambahkan 100 µl lysozym dan diinkubasi pada suhu 37 oC selama 1 jam. Setiap 15 menit sekali eppendorf dibolak-balik perlahan. Kemudian ke dalam eppendorf ditambahkan 100 µl SDS 10% dan 10 µl Prot-K, kemudian diinkubasi kembali pada suhu 37 oC selama 1 jam.
Tahap selanjutnya ke dalam eppendorf ditambahkan 100 µl NaCl 5 M larutan dicampur dengan cara dibolak-balik perlahan. Kemudian ke dalam eppendorf ditambahkan ekstraksi buffer (2% CTAB, 100 mM Tris-HCL pH 8, 1.4 M NaCl, 20 mM EDTA) dan diinkubasi selama 20 menit pada suhu 65oC. Setelah itu ke dalam eppendorf ditambahkan 500 µl phenolchloroform-isoamyl alkohol. Larutan kemudian dihomogenkan dengan cara eppendorf dibolak-balik
7
selama 1 menit. Selanjutnya disentrifuse dengan kecepatan 10000 rpm pada suhu 4 oC selama 5 menit.
Setelah terbentuk tiga lapisan, lapisan paling atas diambil dandi pindahkan ke dalam eppendorf baru sebanyak 50-100 µl menggunakan mikropipet dan ditambahkan ethanol absolut atau isopropanol dingin dengan volume yang sama dengan supernatan yang di pindahkan.Kemudian diinkubasi dalam freezer dengan suhu -20 oC selama 20 menit. Langkah berikutnya eppendorf disentrifuse dengan kecepatan 13000 rpm selama 10 menit. Supernatan dibuang, pellet DNA dicuci dengan 1 ml ethanol 70% bersuhu -20 oC. Pellet dikeringkan di steril bench selama 15-30 menit. Kemudian aquabides ditambahkan ke dalam eppendorf sebanyak 50 µl.
2.4 PCR
Langkah awal saat PCR adalah mempersiapkan ”master-mix”. Komposisi dalam ”master-mix” dapat dilihat pada tabel berikut :
Tabel 1. ”Master mix” yang digunakan untuk PCR
Reagent Volume (µl) dH2O (steril) 18.5 Buffer (10 x) 2,5 dNTP (2,5 mM) 2.5 Forward primer (5 µM)* 1 Reverse primer (5 µM) ** 1
Taq DNA Polymerase 0,2
Primer yang digunakan ;
* Myhemo F1 5’-GATGGTCAGTGCCTCTCA-3’ ** Myhemo R1 5’-CCCAGTTGTATAGCGGTA-3’
Template DNA yang digunakan adalah, U5, U7, U8, U9, U24, U27, V-U41NL dan V-V44.
8
PrimerMyhemo dirancang dengan mengambil potongan DNA gen
hemolysin yang sebelumnya telah dilakukan penelitian oleh Yuhana et al, (2009).
yang dimulai dari urutan basa forward ke-86 dan reverse urutan basa ke-586 (Gambar 1) dengan target produk 518 bp.
>Vh_hemo gatggatcatgaataaaactattacgttacttagtgcattattactaccactaagttttgctcacgctgccgagccaacattgtct ccagagatggtcagtgcctctcaagtaagaagcgcgcaagcgaaacaaacttacacttatgtccgctgctggtaccgcacca gttattcaaaagatgaacctgcgaccgattgggaatgggcagaaaatccagacggcagttacttcacgcttgatggctactgg tggagttcggtttctttcaagaacatgttctacacagacacaccgcaaagtgttatcaagcaacgttgtgagcaaactctggac ctagcaaatgaaaacgctgacatcaccttctttgcagccgataaccgtttctcctacaaccatactatctggagcaacgaccct gtcatgcagccagaccaaatcaacaaggtcgtagcattgggtgacagcttgtctgatacaggcaacatctttaatgcatcaca atggcgattcccgaatccaaatagctggttcttgggacacttctcaaacggttttgtgtggactgagtacattgctcaagcgaaa aacttaccgctatacaactgggctgtgggtggcgcggcaggcgaaaaccaatacatcgctctgactggtgtaggtgagcaag tttcctcttacttggcatatgcgaaattagcgaaaaactacaagcctgctaataccctgtttacccttgagtttggtctaaatgac ttcatgaactacaaccgtagcgtgccagaagtgaaatcagactacgcggaagccttgattaaactgaccgatgcaggtgcga agaacttgttgttgatgacactaccagatgcaacacgtgcaccacagtttacctactcgactcaagaagaaatcaacaagatc cgcgcgaagatcgtggaaatgaatgagttcatcaaagcacaagcggcgtattacactgcacaaggctacaacgttaccttgta cgatacgcatgcactgtttgaaagcttaacagcaaatccagagcaacacggttttgtaaacgcgagccaagcttgccaagac atcaaccgctcttcatcggtagattacctataccatcactcattgcgttctgagtgtgcgtcttctggctctgataagtttgtattct gggacgtaacacacccgaccacagcaacacaccactacgtggcagaaaaaatgctagaaagtacgaatcaattgtcaaacc atcctttctaa
Gambar 1.Sekuen Primer hemolysin (Yuhana et al, 2009).
Master-mix yang telah dicampur divortex dan dispindown. Pencampuran
bahan-bahan dilakukan di atas es (on ice). Larutan master mix dimasukkan ke dalam tabung PCR sebanyak 24,5 µl untuk setiap sampel. Setelah itu, ditambahkan 0,5 µl sampel DNA. Larutan kemudian divortex dan dispindown. Kegiatan pencampuran harus dilakukan dengan cepat dan berhati-hati agar tidak terjadi kontaminasi silang antar sampel. Kemudian tabung PCR dimasukkan ke
Forward
Revers Gambar
9
dalam mesin PCR dengan program sebagai berikut; Pre start 94oC selama 2 menit, denaturasi 94oCselama 1 menit, annealing 50oCselama 1 menit dan ektension 72oC selama 1 menit yang dilakukan sebanyak 35 siklus. Untuk lebih jelasnya tahapan prosesPCR dapat dilihat pada Gambar 2 di bawah ini.
Gambar 2Siklus pembentukan molekul DNA baru dalam proses PCR.(Muladno, 2002).
10 2.5 Elektroforesis
Pembuatan gel agarosa konsentrasi 0,7% dimulai dengan melarutkan serbuk gel agarosa sebanyak 0,14 g dalam 20 ml larutkan tris boric EDTA (TBE) yang mengandung etidium bromida (0,01 g/ml). Kemudian campuran dipanaskan dalam microwave sampai agarosa larut dan larutan menjadi berwarna bening, larutan tersebut didiamkan sampai hangat lalu dituangkan ke dalam cetakan dengan ketebalan 3-5 mm. Sementara itu di dalam cetakan sudah terpasang sisir
untuk lubang. Kemudian gel dibiarkan sampai membeku. Setelah itu sisir dilepaskan dan padatan gel dimasukkan ke dalam bak elektroforesis yang berisi
buffer elektroforesis (TBE).
Kemudian sampel DNA dicampur dengan loading buffer (6X)dengan perbandingan 3 µl DNA dicampur dengan 4 µlloading dye, setelah itu DNAdimasukkan ke dalam lubang-lubang yang terdapat dalam gel dengan menggunakan mikropipet. Sementara itu marker DNA juga dimasukkan ke dalam lubang yang mengapit lubang-lubang yang berisi sampel. Setelah, itu bak elektroforesis ditutup dan listrik dialirkan dengan tegangan 200 volt dan kuat arus 70mA. DNA akan bermigrasi dari kutub negatif ke positif. Setelah DNA bermigrasi sampai tiga perempat bagian dari panjang gel, aliran listrik dihentikan. Lalu gel diangkat dan dilepaskan dari cetakannya. Kemudian keberadaan DNA dilihat dengan ultraviolet transilluminator dan didokumentasikan.
11
III. HASIL DAN PEMBAHASAN
3.1 HASIL3.1.1 Isolasi Vibrio harveyi
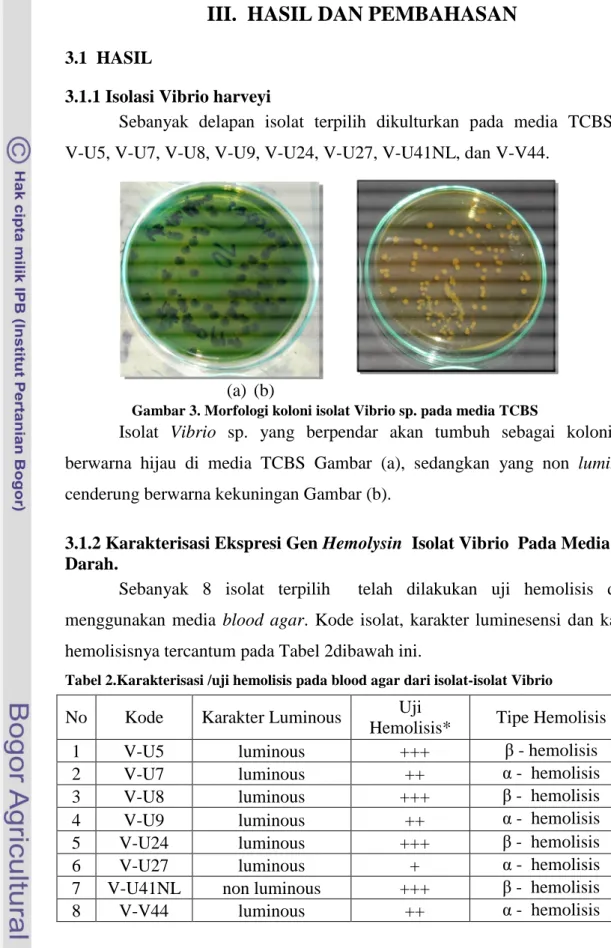
Sebanyak delapan isolat terpilih dikulturkan pada media TCBS yaitu V-U5, V-U7, V-U8, V-U9, V-U24, V-U27, V-U41NL, dan V-V44.
(a) (b)
Isolat Vibrio sp. yang berpendar akan tumbuh sebagai koloni yang berwarna hijau di media TCBS Gambar (a), sedangkan yang non luminescen cenderung berwarna kekuningan Gambar (b).
3.1.2 Karakterisasi Ekspresi Gen Hemolysin Isolat Vibrio Pada Media Agar Darah.
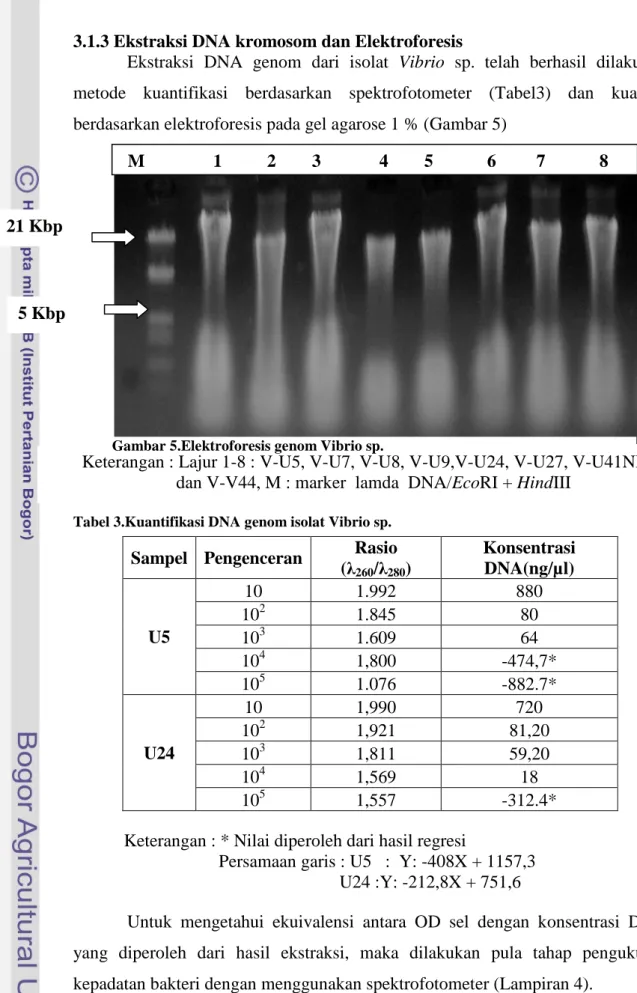
Sebanyak 8 isolat terpilih telah dilakukan uji hemolisis dengan menggunakan media blood agar. Kode isolat, karakter luminesensi dan karakter hemolisisnya tercantum pada Tabel 2dibawah ini.
Tabel 2.Karakterisasi /uji hemolisis pada blood agar dari isolat-isolat Vibrio
No Kode Karakter Luminous Uji
Hemolisis* Tipe Hemolisis
1 V-U5 luminous +++ β - hemolisis
2 V-U7 luminous ++ α - hemolisis
3 V-U8 luminous +++ β - hemolisis
4 V-U9 luminous ++ α - hemolisis
5 V-U24 luminous +++ β - hemolisis
6 V-U27 luminous + α - hemolisis
7 V-U41NL non luminous +++ β - hemolisis
8 V-V44 luminous ++ α - hemolisis
12
Berdasarkan uji hemolisis yang telah dilakukan dengan menggunakan media blood agar, isolat yang ada menunjukkan ekspresi yang berbeda-beda. Secara kualitatif bisa dikategorikan lemah/sangat lemah untuk isolat V-U27, kategori hemolisis sedang untuk isolat V-U7,V-U9 dan V-V44, sedangkan untuk kategori zona lisis kuat terlihat pada isolat V-U5, V-U8, V-U24, dan V-U41NL. Sedangkan jenis hemolisis untuk isolat V-U7, V-U9, V-U27, V-V44 adalah jenis
α - hemolisis. Sedangkan untuk isolat V-U5, V-U8, V-U24, V-U41NL memiliki
tipe β – hemolisis. Dengan melihat ekspresi isolat yang telah dikultur di media
blood agar dan menghasilkan zona lisis maka dapat diduga bahwa proses
amplifikasi gen hemolysin dari galur bakteri Vibrio dengan teknik PCR, dapat dilakukan. Hasil uji hemolisis pada media blood agar dapat dilihat pada Gambar 4 di bawah ini.
Keterangan : Isolat V-U5 (kiri) mengekspresikan kemampuan hemolisis kuat tipeβ – hemolisis, sedangkan V-U27 (kanan) memiliki kemampuan hemolisis lemah tipe
α - hemolisis.
Keterangan *: Uji hemolisis dilakukan secara kualitatif yaitu : +++ : Menunjukkan zona lisis yang kuat
++ : Menunjukkan zona lisis yang sedang
+ : Menunjukkan zona lisis yang lemah/sangat lemah
Gambar 4. Hasil uji hemolisis pada isolat-isolat Vibrio yang ditumbuhkan pada media blood agar.
13
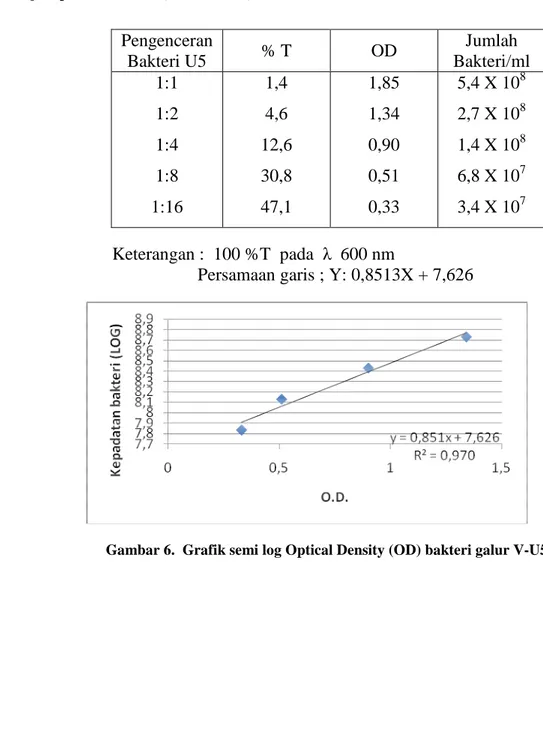
3.1.3 Ekstraksi DNA kromosom dan Elektroforesis
Ekstraksi DNA genom dari isolat Vibrio sp. telah berhasil dilakukan metode kuantifikasi berdasarkan spektrofotometer (Tabel3) dan kualitas berdasarkan elektroforesis pada gel agarose 1 % (Gambar 5)
Tabel 3.Kuantifikasi DNA genom isolat Vibrio sp.
Untuk mengetahui ekuivalensi antara OD sel dengan konsentrasi DNA yang diperoleh dari hasil ekstraksi, maka dilakukan pula tahap pengukuran kepadatan bakteri dengan menggunakan spektrofotometer (Lampiran 4).
Sampel Pengenceran Rasio (λ260/λ280) Konsentrasi DNA(ng/µl) U5 10 1.992 880 102 1.845 80 103 1.609 64 104 1,800 -474,7* 105 1.076 -882.7* U24 10 1,990 720 102 1,921 81,20 103 1,811 59,20 104 1,569 18 105 1,557 -312.4* M 1 2 3 4 5 6 7 8 21 Kbp 5 Kbp
Keterangan : Lajur 1-8 : V-U5, V-U7, V-U8, V-U9,V-U24, V-U27, V-U41NL dan V-V44, M : marker lamda DNA/EcoRI + HindIII
Keterangan : * Nilai diperoleh dari hasil regresi
Persamaan garis : U5 : Y: -408X + 1157,3
U24 :Y: -212,8X + 751,6
14
Tahap log faseoptical density (OD) diukur dari populasi sel yang dikultur selama 18-24 jam. Hasil Pengukuran dapat dilihat pada Tabel 4 dan 5 dibawah ini.
Tabel 4. Hasil pengukuran Optical Density (OD) bakteri galur V-U5 yang dikultur 18 -24 jam pada media LB (luria bertani).
Gambar 6. Grafik semi log Optical Density (OD) bakteri galur V-U5.
Pengenceran Bakteri U5 % T OD Jumlah Bakteri/ml 1:1 1:2 1:4 1:8 1:16 1,4 4,6 12,6 30,8 47,1 1,85 1,34 0,90 0,51 0,33 5,4 X 108 2,7 X 108 1,4 X 108 6,8 X 107 3,4 X 107 Keterangan : 100 %T pada λ 600 nm Persamaan garis ; Y: 0,8513X + 7,626
15
Tabel 5. Hasil pengukuran Optical Density (OD) bakteri galur V-U24 yang dikultur 18-24 jam pada media LB (luria bertani).
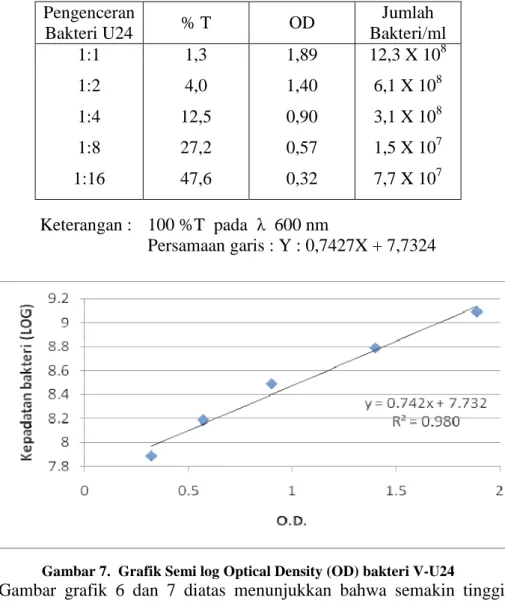
Gambar 7. Grafik Semi log Optical Density (OD) bakteri V-U24
Gambar grafik 6 dan 7 diatas menunjukkan bahwa semakin tinggi nilai
optical density (OD) maka tingkat kepadatan bakteri juga semakin tinggi. Bakteri
yang dikultur selama 18-24 jam memiliki kepadatan 12,27 X108sel/ml untuk bakteri V-U24 dan 5,4 X 108sel/ml untuk bakteri V-U5.
3.1.4 Amplifikasi gen hemolysin Myhemo Primer
Genom hasil ekstraksi selanjutnya diamplifikasi dengan menggunakan primer Myhemo F1R1 dan kemudian dilakukan visualisasi dengan melakukan elektroforesis pada gel agarose 0,7% dapat dilihat pada gambar 8 di bawah ini.
Pengenceran Bakteri U24 % T OD Jumlah Bakteri/ml 1:1 1:2 1:4 1:8 1:16 1,3 4,0 12,5 27,2 47,6 1,89 1,40 0,90 0,57 0,32 12,3 X 108 6,1 X 108 3,1 X 108 1,5 X 107 7,7 X 107 Keterangan : 100 %T pada λ 600 nm Persamaan garis : Y : 0,7427X + 7,7324
16 s
Setelah dilakukan elektroforesis. Dihasilkan dua isolat bakteri yang teramplifikasi yaitu V-U5 dan V-U24. Untuk meyakinkan hasil PCR maka dilakukan kembali PCR II dari produk PCR1 dan ternyata hasil yang didapatkan tetap hanya dua isolat yang teramplifikasi dapat dilihat pada gambar 9 dibawah ini.
M 1 2 3
518b
400b
Keterangan : Suhu annealing 500C, Lajur1 :V-U5,Laajur 2: V-U7, Lajur 3 :V-U24 Lajur 4 : V-U27, Lajur 5 : V-V44 , Lajur 6 : V-U9, Lajur 7 : V-U8, Lajur 8: V-U41NL, Lajur 9-10: kontrol positif , Lajur 11 : kontrol negatif, M : marker
Keterangan : Suhu annealing 500C, pengenceran 10X, M = marker, Lajur 1 : V-U5, Lajur 2 : V-U24, Lajur 3. Kontrol negatif
M 1 2 3 4 5 6 7 8 9 10 518b
p
400b p
Gambar 8.Gambar gel elektroforesis hasil amplifikasi PCR menggunakan primer Myhemo F1R1
Gambar 9.Gambar gel elektroforesis hasil amplifikasi PCR dengan menggunakan primer Myhemo F1R1
17
3.1.5 Amplifikasi gen hemolysin Myhemo Primer F1R1 hasil Pengenceran Untuk mengetahui kemampuan primer yang telah dirancang dalam mengamplifikasi DNA dengan konsentrasi paling minimum, dilakukan proses pengenceran pada genom kedua isolat V-U5 dan V-U24 yaitu pengenceran 102, 103, 104 dan 105 sekuen primer Myhemo F1 5-CCCAGTTGTATAGCGGTA-3, R1 5’-GATGGTCAGTGCCTCTCA-3’ dengan target sekitar 518 bp dan hasil amplifikasi dapat dilihat pada Gambar 10 di bawah ini.
Hasil visualisasi elektroforesis pada gel agarose 0,7% menunjukkan bahwa kombinasi pasangan primer Myhemo F1R1 berhasil mengamplifikasi DNA sampel sampai pada pengenceran 100X dengan konsentrasi DNA untuk V-U5 yaitu 80 ng/µl equivalent dengan 3,1 x 107 sel/ml bakteri dan V-U24 81,20 ng/µl equivalent dengan 3,1 x 107 sel/ml bakteri, dimana 2.6 fg DNA, equivalent dengan 1 sel bakteri (Lee et al, 1995b). Sedangkan untuk pengenceran 103,104, dan 105, gen target belum berhasil teramplifikasi.
M 1 2 3 4 5 6 7 89
518b pKb
400b
,
Keterangan : Suhu annealing 500C hasil pengenceran 102,103,104 , dan 105M : marker, Lajur 1 : V-U5 (102), Lajur 2 . V-U24 (102), Lajur 3-4 : V-U5 dan V-U24 (103), Lajur 5-6 : V-U5 dan V-U24 (104), Lajur 7-8: V-U5 dan V-U24 (105), Lajur 9 : kontrol negatif.
18 3.2 Pembahasan
Untuk melihat kemampuan bakteri Vibrio sp. dalam melisis sel darah merah atau untuk melihat keberadaan gen hemo pada bakteri uji yang dapat diamati secara visual, dilakukan uji dengan menggunakan media agar darah (blood agar). Terbentunya zona bening lisis, menunjukkan bahwa isolat mampu melisiskan sel darah. Proses lisis sempurna terlihat dari zona yang benar-benar jernih (β-hemolisis), proses hemolisis tidak sempurna memperlihatkan media berwarna kehijauan (α-hemolisis), proses lisis yang tidak nyata menyebabkan tidak terjadi perubahan warna media (γ-hemolisis) Suryanto et al., (2007).
Berdasarkan uji hemolisis yang telah dilakukan menunjukkan ekspresi yang berbeda-beda (Gambar 4). Jenis hemolisis untuk isolat U7, U9,
V-U27, V-V44 adalah α-hemolisis. Sedangkan untuk isolat V-U5, V-U8, V-U24, V-U41NL memiliki tipe β–hemolisis. Dan dari semua isolat yang terkumpul tidak terjadi jenis γ-hemolisis. Sehingga secara visual kita dapat menyimpulkan bahwa bakteri uji memang memiliki gen hemolysin yang berarti proses PCR untuk mendeteksi keberadaan gen hemolisin tersebut dapat dilakukan.
Berdasarkan hasil yang diperoleh, dapat diketahui bahwa penerapan prosedur PCR untuk mengetahui keberadaan bakteri Vibrio harveyi dengan sekuen primer Myhemo F1R1 telah berhasil dilakukan. Bakteri yang berhasil teramplifikasi yaitu isolat V-U5 dan V-U24 dengan panjang target hasil amplifikasi 518 bp pada Tm 500C sebanyak 35 siklus (Gambar 8). Sedangkan untuk isolat V-U7, V-U8, V-U9, V-U27, V-U41NL, dan V-V44 masih belum berhasil teramplifikasi. Sehingga masih harus dicari suhu annealing yang tepat. Suhu annealing yang tidak tepat menyebabkan tidak terjadinya penempelan primer atau primer menempel di sembarang tempat.
Pada proses annealing, keterkaitan suhu akan berhubungan dengan stabilitas molekul primer yang berikatan pada molekul DNA, dan akan sangat mempengaruhi produk PCR. Suhu yang tinggi telah diketahui akan mengganggu stabilitas ikatan hidrogen antar nukleotida primer terhadap komplemennya pada DNA cetakan. Ketidakstabilan tersebut, mungkin dapat dianalogikan dengan dinamika gerak pada tingkatan suhu. Suhu akan mempengaruhi tingkat
19
pergerakan dari untai nukleotida, dan juga berkaitan dengan kekuatan ikatan hidrogen antara nukleotida (primer dengan DNA cetakan). Apabila ditinjau dari aspek fisika, panas diartikan sebagai bentuk dari energi yang dapat dipindahkan dari satu obek pada objek lainnya karena perbedaan dalam suhu (Trefil dan Hazen, 2004).
Suhu sebenarnya adalah pengukuran dari besarnya kecepatan rata-rata atom dan molekul, termasuk vibrasinya-yaitu, energi kinetik dari partikel yang membuat zat. Pada suhu yang lebih rendah, molekul material memiliki kinetik yang lebih rendah dibandingkan pada suhu yang tinggi (suhu lebih rendah, kecepatan molekular lebih lambat, suhu lebih tinggi, kecepatan molekular lebih cepat). Sehingga penggunaan suhu 500C pada proses annealing yakni di bawah Tm yang seharusnya cukup memberikan alasan dihasilkannya produk PCR. Jika dua material berada pada suhu yang sama, maka atom dan molekulnya memiliki energi kinetik yang sama. Hal yang menjadi penting adalah bahwa semakin tinggi suhu suatu objek maka semakin cepat atom atau molekulnya bergerak (Trefil dan hazen, 2004).
DNA maupun primer, juga merupakan molekul yang terdiri dari atom-atom karbon (C), oksigen (O), nitrogen (N), fosfat (P). dan hidrogen (H). Pada suhu annealing yang optimal (primer hanya secara tepat berikatan pada komplemennya), pergerakan atom yang tinggi, mengakibatkan pasangan primer yang tidak sepenuhnya komplemen akan cenderung terlepas kembali dan gagal untuk melanjutkan proses eksetensi, sedangkan pasangan primer yang benar-benar sesuai dengan komplemennya akan tetap berkaitan pada DNA cetakan dan melanjutkan proses ekstensi. Suhu annealing yang dibutuhkan akan bergantung pada komposisi dan panjang sekuen primer. Karena pasangan G-C lebih kuat daripada A-T (pasang basa guanine:sitosin (GC) memiliki tiga ikatan hidrogen, sedangkan adenine:timin (AT) hanya memiliki dua ikatan hidrogen), semakin banyak G dan C dalam primer, primer tersebut akan berikatan lebih kuat dengan komplemennya (lebih stabil terhadap peningkatan suhu). Oleh karena itu, suhu
annealing yang digunakan menjadi lebih tinggi bila jumlah GC lebih banyak
20
Meskipun tersedia program komputer dan formula untuk mencari suhu
annealing yang optimal bagi primer yang telah dirancang, pada prakteknya
dibutuhkan sejumlah upaya trial dan error. Biasanya suhu annealing dipilih antara 40 dan 600C, walaupun untuk DNA cetakan dengan kandungan GC yang tinggi, suhu annealing setinggi 720C (sama dengan suhu ekstensi normal) dapat digunakan. Karena primer berukuran kecil, pada konsentrasi molar yang tinggi secara keseluruhan, annealing terjadi dengan cepat hanya dalam hitungan menit atau kurang (Dale dan Schantz, 2002).
Dengan melihat Gambar 8 dan 9 di atas, gen hemolysin untuk isolat V-U5 dan V-U24 telah berhasil teramplifikasi. Hasil dari pengoptimalisasian program PCR menunjukkan bahwa penurunan suhu 50C di bawah Tm yaitu 500C, gen
hemolysin berhasil teramplifikasi, menghasilkan pita tunggal dengan panjang
DNA target 518 bp. Sehingga tahap optimasi untuk kedua isolat ini telah berhasil dilakukan.
Pengoptimalisasian amplifikasi PCR dapat ditinjau dari komponen dan tahapan rekasi yang terlibat di dalamnya. Pengoptimalisasian yang menyeluruh membutuhkan beberapa upaya trial dan error. Pengoptimalan tersebut harus mencapai kondisi seperti : (1) Untai ganda DNA cetakan benar-benar terpisah pada suhu denaturasi, (2) pada suhu annealing, masing-masing primer berikatan dengan tepat hanya pada sekuen yang benar-benar komplemen (spesifik), serta (3) kondisi suhu dan durasi waktu sesuai untuk ekstensi produk dengan sempurna. Kegagalan pencapaian kondisi tersebut akan mengakibatkan kegagalan reaksi amplifikasi (Rasmussen dan Reed, 1992). Sehingga untuk isolat U7,U8, V-U9,V-U27,V-U41NL, dan V-V44 masih harus dicari suhu annealing yang tepat.
Meskipun formula dalam mencari suhu annealing optimum bagi primer telah tersedia, yaitu Tm = 4(G+C) + 2(A+T). Namun ternyata hasil optimasi menunjukkan bahwa primer Myhemo F1R1 memiliki suhu annealing 500C yaitu 50C di bawah Tm hasil perhitungan yaitu 550C. Hal ini sesuai menurut Muladno (2002) temperature penempelan primer (annealing) yang digunakan dalam proses PCR biasanya 50C di bawah Tm.
21
Selain suhu annealing yang tidak tepat menyebabkan tidak terjadinya reaksi amplifikasi, adanya variasi genetik dalam gen penyandi hemolysinpada isolat uji juga dapat mengakibatkan tidak terjadinya proses amplifikasi. Spesifisitas primer dalam bekerja memungkinkan primer tidak menemukan pasangan basa yang cocok untuk melakukan penempelan. Sehingga sifat primer yang bekerja spesifik hanya dapat mengamplifikasi DNA bakteri V. harveyi saja dan tidak memiliki kecocokan dengan DNA bakteri yang secara genetik mirip dengan V. harveyi. Selain itu menurut Saiki et al. (1990), akumulasi dari sisa sel dalam genom juga diketahui menghambat proses amplifikasi DNA.
Amplifikasi gen hemolysintelah dilaporkan sebelumnya oleh Zhang., et al (2001) yaitu dari berbagai strain bakteri Vibrio harveyi yang berasal dari lokasi geografi yang berbeda, dan menghasilkan produk amplifikasi 1,3 Kbp menggunakan primer yang disebut IFO 15634. Namun dari sebelas sampel yang diuji hanya sembilan strain yang berhasil teramplifikasi. Berdasarkan analisa oleh Zhang., et al (2001) bahwa untuk dua strain sampel yang tidak berhasil teramplifikasi diduga bakteri uji tidak memiliki gen tersebut atau kemungkinan adanya variasi genetik, sehingga proses annealing tidak berhasil dilakukan karena adanya satu atau lebih basa primer yang ada, tidak cocok dengan sekuen gen target.
Untuk melihat kemampuan sensitifitas primer dalam mengamplifikasi gen
hemolysin, maka dilakukan pengenceran serial DNA genom untuk isolat yang
telah berhasil teramplifikasi yaitu pada isolat V-U5 dan V-U24. Hasil amplifikasi menunjukkan bahwa primer Myhemo F1R1 mampu mengamplifikasi DNA sampel sampai pada pengenceran 100X dengan konsentrasi DNA untuk V-U5 yaitu 80 ng/µl equivalent dengan 3,1 x 107 sel/ml bakteri dan V-U24 81,20 ng/µl equivalent dengan 3,1 x 107 sel/ml bakteri, dimana 2.6 fg DNA, (equivalent dengan 1 sel bakteri) (Lee et al, 1995b)
Pelacak-pelacak DNA untuk identifikasi galur-galur klinis dari bakteri Vibrio terus dilakukan. Seperti pada V. parahaemoliticus telah dikembangkan lebih dari satu dekade karena juga merupakan pathogen gastroenteritis akut pada manusia yang mengkonsumsi sea food yang terkontaminasi bakteri ini (Tada et
22
al., 1992a). Pelacak DNA didesain berdasar atas faktor virulensi yang disebut the thermostable direct hemolysin gene (tdh) dan the thermostable direct hemolysin-related gene (trh). Kedua gen ini umumnya hanya dijumpai pada galur-galur yang
diisolasi dari spesimen klinis, akan tetapi jarang ditemukan pada galur-galur yang diisolasi dari lingkungan.
Metode PCR berdasarkan pada sekuen pelacak gen-gen tdh dan trh juga telah digunakan dalam pengujian non isotopik pada piringan mikrotiter yang dapat dibaca secara otomatis (Tada et al., 1992b), akan tetapi, karena proses ini melibatkan pelacak hibridisasi, pengujian tidak dapat bekerja pada galur-galur yang diisolasi dari lingkungan yang tidak mengandung kedua jenis gen tersebut. Rojrosakul et al.,(1998) telah mengembangkan fragmen pVPA7 dari pustaka genom V. parahaemoliticusuntuk mendeteksi keberadaannya pada organ hemolimfa udang. Pelacak ini menghasilkan sinyal positif untuk seluruh galur V.
parahaemoliticusyang diuji (termasuk 141 galur baik klinis maupun yang
bersumber dari lingkungan) akan tetapi memberikan hasil hibridisasi negatif terhadap spesies-spesies Vibrio yang lain dan dengan spesies lain yang mewakili dari genera non Vibrio.
Fragmen pVPA7 memiliki limit sensitifitas dari V. parahaemoliticussebesar 105 cfu/ml dengan hibridisasi dot blot. Hasil sekuensing dari fragmen pVPA7, kemudian digunakan untuk merancang primer-primer PCR yang dinamakan VPAFOR3 dan VPAREV1. Kedua primer ini menunjukkan sensitifitas sebesar 100 fg DNA dan 4x103 cfu/ml. Konsentrasi terendah dalam sel (hemolimfa udang) yang dapat dideteksi adalah sebesar 4x103 cfu/ml. Sedangkan hasil analisa PCR menggunakan pasangan primer Myhemo F1R1 pada bakteri
Vibrio harveyimemiliki limit sensitifitas sebesar 80,6 ng/µl equivalent dengan 3,1
x 107 sel/ml bakteri.
23
IV. KESIMPULAN DAN SARAN
Gen hemo dari V.harveyi berhasil teramplifikasi pada isolat V-U5 dan V-U24 dengan menggunakan primer Myhemo F1R1,menghasilkan produk berupa pita tunggal berukuran sekitar 518 bp. Program PCR menggunakan suhu
annealing 500C sebanyak 35 siklus.
Saran: Hasil dari penelitian ini dapat digunakan untuk merancang program nested PCR dengan melakukan optimasi kembali pada pasangan primer internal Myhemo F2R2.
24
DAFTAR PUSTAKA
Badan Standarisasi Nasional. 2007. Produksi Udang Vannamei (Littopenaeus.
Vannamei) di Tambak dengan Teknologi Intensif. Http: www.dostocs
.com/docs/199916721/SNI-01-7246-2006. [11 Februari 2011]
Boyd, C. E. and Clay, J. W. 2002.Evaluation of Belize aquaculture LTD, a superintensive shrimp aquaculture system.Report prepared under the World Bank, NACA, and FAO consorsium. Work in progress for public discussion.Published by the consorsium.17 pages.
Cano-Gomez, A., Bourne, D. G., Hall, M. R., Owens, L., Hoj, L. 2009. Molecular identification, typing and tracking of Vibrio harveyi in aquaculture system : current method and future prospects. Journal Aquaculture. 287 : 1-10
Conejero, M. J. U., and C. T. Hedreyda . 2004. PCR detection of hemolysin (vhh) gene in Vibrio harveyi, Appl. Microbiol., 50: 137-142.
Dale, J. W. 2002. From genes to genomes: concepts and applications of DNA technology. John Wiley and Sons Ltd, England.
Erlich, H. A. 1989. PCR technology principles and application for DNA amplification. New York: M Stockton Press.
Hadioetomo, Ratna Siri. 1993. Mikrobiologi dasar dalam praktek, teknik dan prosedur dasar laboratorium. Gramedia Pustaka Utama. Jakarta.
Haliman, R. W. dan Adijaya, D. 2007. Udang vannamei. Penebar Swadaya. Jakarta.
Hirono, I., Masuda, T., and Aoki, T. 1996. Cloning and detection of the hemolysin gene of Vibrio anguillarum. Microb. Pathog., 21, 173-182.
Karunasagar, I., Pai, R., Malathi, G. R. Karunasagar, I., 1994. Mass mortality of
Penaeus monodon larvae due to antibiotic-resistant Vibrio harveyi
infection. Aquaculture 128, 203-209.
Lavilla-Pitogo, C. R., L. L. Baticados, E. R. Cruz Lacierda, and L. D. de la Pena. 1990. Occurrence of luminous bacterial diseases of Penaeus monodon larvae in the Philippines. Aquaculture, 91: 1-13.
Lee, C. Y., Pan, S. F.,and Chen, C. H. 1995b. Sequence of a cloned pR72H fragment and its use for detection of Vibrio parahaemolyticus in shellfish with the PCR. Appl. Environ. Microbiol 61: 1311-1317
Lightner, D.V. 1983. Disease in culture penaid shrimp.p:289-320. In: J.Mc Vey (Ed.). CRC Handbook of mariculture.Vol. I. Crustacean Aquaculture. CRC Press, Boca Raton, FL.
25
Liu, P. C., K. K. Lee, K. C. YII, G. H. Kou, and S. N. Chen. 1996b. Isolation of
Vibrio harveyi from diseased kuruma prawns Penaeus japonicus.
Current Microbiology. 33: 129-132.
Muladno. 2002. Teknologi rekayasa genetika. Penebar Swadaya.Jakarta
Nishibuci, M., Khaeomanee-iam, V., Honda, T., Kaper, J. B., and Miwatani, T. 1990. Comparative analysis of the hemolysin genes of Vibrio cholera non-O1, Vibrio mimicus, and Vibrio hollisae that are similar to the tdh gene of Vibrio parahaemolyticus. FEMS Microbiol. Lett., 67, 251-256 Nishibuci, M. and Kaper, J. 1995. Thermostable direct hemolysin gene of Vibrio
parahaemolyticus: A virulence gene acquired by a marine bacterium. Infect. Immun., 63, 2093-2099
Pass, D. A. Dybdah, R., and Mannion, M. M. 1987. Investigation into the causes of mortality of the pearl oyster, Pinctada maxima (Jamson), in Western Australia. Aquaculture 65, 149-169.
Poernomo ,S.H. 2008. DKP pacu produksi udang nasional . Pusat Data, Statistik dan Informasi DKP. WWW.dkp.go.id [21 Maret 2010]
Rasmussen R, Reed G. 1992. Optimizing rapid cycle DNA amplification reaction.www.idahotechnology.com/pdfs/RapidCycler/RapidCyclist%20 V1.1992.pdf [17 April 2009]
Rojrosakul, P., V. Boonsaeng., W. Panbangred, O. Suthienkul, T. Pasharawipas, and T. W. Flagel. 1998. Detection of V. parahaemolyticus shrimp hemolymph by DNA hybridization and PCR amplification. P: 227-234.
In. T. W. Flegel (ed). Advances in shrimp biotechnology. National centre
for genetic engineering and biotechnology, Bangkok, Thailand.
Saeed, M. O. 1995. Association of Vibrio harveyi with mortalities in cultured marine fish in Kuwait. Aquaculture 136, 21-29.
Saiki, R. K. 1990. Amplification of genomic DNA. In: Innis, M. A, Gelfand, D. H, Sninsky, J. J. White (eds) PCR protocol: A guide to methode and applications. Academic press, San Diego, CA, p13-20.
Suryanto, D., Irmayanti, dan Lubis, S. 2007. Karakterisasi dan uji kepekaan antibiotik beberapa isolat Staphylococcus aureus dari Sumatera Utara. Majalah Kedokteran Nusantara Vol. 40. NO.2.Juni s2007. Departemen Mikrobiologi, Fakultas Kedokteran, Universitas Sumatera Utara.\
Tada, J., T. Ohashi, Nishimura, Y, Shirasaki, H. Ozaki, S. Fukushima, J. Takano, M. Nishibuchi, and Y. Takeda. 1992a. Detection of the thermostable direct hemolysin gene (tdh) and the thermostable direct hemolysindene (trh) of V. Parahaemolyticus by polymerase chain reaction. Mol. Cell. Probes. 6: 477-487.
Tada, J., T. Ohashi, Nishimura, H. Ozaki, S. Fukushima, J. Takano, M. Nishibuchi, and Y. Takeda. 1992b. Non-isotopik microtitre plate-based assay for detecting products of polymerase chain reaction amplification :
26
application to detection of the tdh gene of V. Parahaemolyticus by. Mol. Cell. Probes. 6: 489-494
Trefil, J, Haen, R. M. 2004. Physics Matter: An introduction to conceptual physic. John Wiley an Sons Inc, United States Of America.
Trobos. 2011. Target produksi udang meleset. Majalah Trobos Edisi Februari 2011. Http://MajalahTrobos/ Produksi vannamei [4 Februari 2011] Yufrialdi. 2008. Penggunaan PCR untuk penentuan gen CTX penghasil toksin
kolera (CTX) pada bakteri. Http://Wikipedia.org/wiki/ Penaeus_ vannamei [12 Juli 2009]
Yuhana, M., Widanarni, dan Sukenda. 2008. Desain primer, karakterisasi gen 16S rRNA dan afiliasi filogenetik dari Vibrio sp. yang di isolasi dari tambak dan pembenihan udang putih (Littopenaeus vannamei ) di Lampung, Indonesia. Laporan Penelitian Hibah Bersaing. Lembaga Penelitian dan Pengabdian Kepada Masyarakat. Institut Pertanian Bogor.
Yuhana, M.,Widyastuti, U dan Sukenda. 2009. Pengembangan penanda molekuler untuk diagnostik cepat penyakit Vibrio berpendar pada budidaya udangLittopenaeusvannamei.Laporan Penelitian Hibah Bersaing. Lembaga Penelitian dan Pengabdian Kepada Masyarakat. Institut Pertanian Bogor.
Zhang, X.-H and Austin, B. (2000) Pathogenicity of Vibrio harveyi to salmonids. J. Fish Dis., 23, 93-102
Zhang, X. -H., Meaden, P. G., and Austin, B. 2001. Duplication of hemolysin genes in a virulent isolate of Vibrio harveyi. Appl. Environ Micribiology., 67, 3161-3167
27
Lampiran 1. Penghitungan bakteri dengan metode hitungan cawan sebar
Metode hitungan cawan didasarkan pada anggapan bahwa setiap sel yang dapat hidup akan berkembang menjadi satu koloni. Untuk memenuhi persyaratan statistik, cawan yang dipilih untuk penghitungan koloni adalah yang mengandung antara 30-300 koloni. Karena jumlah mikroorganisme dalam sampel tidak diketahui sebelumnya, maka untuk memperoleh sekurang-kurangnya satu cawan yang mengandung koloni dalam jumlah yang memenuhi syarat tersebut maka harus dilakukan sederetan pengenceran dan pecawanan. Jumlah sel bakteri pada suatu sampel diketahui dengan menghitung jumlah koloni tumbuh pada media tersebut dikalikan dengan faktor pengencernya dengan satuan colony forming unit (CFU/mℓ atau CFU/g).
Metode kerja diawali dengan menyiapkan eppendorf, masing-masing berisi 0,9 mℓ fisiologis dan disusun berderet seperti tampak pada gambar di bawah ini. Sampel suspensi bakteri yang akan dihitung dikocok terlebih dahulu sampai kekeruhannya nyata. Sebanyak 0,1 mℓ suspensi bakteri diambil secara aseptik, lalu dimasukan kedalam eppendorf yang pertama (10-1), kemudian dikocok atau divortex agar homogen, lalu secara aseptik 0,1 mℓ sampel dari pengencer pertama dimasukkan ke dalam tabung pengencer kedua (10-2), dan seterusnya untuk eppendorf-eppendorf pengencer selanjutnya. Sebanyak 3 cawan petri media TSA disiapkan dan diberi kode sesuai dengan kode eppendorf pengencer yang akan disebar. Sebanyak 0,1 mℓ sampel dari tabung pengencer tiga pengencer tertinggi dipipet secara aseptik, dan masing-masing disebar pada media TSA menggunakan batang penyebar. Setelah itu diletakkan dalam posisi terbalik untuk diinkubasi pada suhu kamar selama 24 jam. Kemudian jumlah koloni yang tumbuh (30-300) dihitung dan dikalikan dengan faktor pengencernya.
Gambar. Prosedur hitungan cawan sebar
0,1mℓ 0,1mℓ 0,9mℓ Sampel 10-1 10-2 10-3 10-4 Agar TSA dalam cawan
28
Lampiran 2. Pewarnaan Gram
Pewarnaan gram digunakan untuk melihat bentuk bakteri dan sifat terhadap zat warna, cara melakukan uji ini adalah :
1.Mula-mula ambil koloni bakteri dan dioleskan pada gelas objek yang telah ditetesi akuades.
2.Preparat kemudian dihomogenkan dan dikering udarakan, jika telah dikering udara, olesan bakteri difiksasi di atas nyala api kemudian digenangi dengan kristal violet selama 1 menit.
3.Setelah itu preparat dibilas dengan akuades, olesan bakteri kemudian digenangi lagi dengan kalium iodida selama 2 menit, dibilas dengan air atau akuades.
4.Preparat kemudian dilakukan pemucatan dengan meneteskan alkohol atau ethanol 95% selama 30 detik
5.Setelah itu olesan bakteri diberi warna tandingan yaitu safranin selama 30 detik dan dibilas denga akuades.
6.Kemudian dikeringkan dan diamati di bawah mikroskop dengan menggunkan minyak imersi.
7.Bila hasil pewarnaan berwarna merah berarti Gram negatif dan bila warna ungu atau biru berarti Gram positif.
29
Lampiran 3. Media Luria bertani (LB)
Bahan-bahan yang digunakan untuk membuat LB adalah : 1. Yeast 0,125 g
2. Tripton 0,250 g 3. NaCl 0,750 g
Semua bahan dilarutkan ke dalam 25 mℓ akuades dengan menggunakan erlenmeyer, lalu ditutup dengan menggunakan alumunium foil dan dihomogenkan dengan penangas air. Setelah homogen LB diserilkan menggunakan autoclave pada suhu 1210C, tekanan 1 atm selama 15 menit. LB siap untuk digunakan.
Lampiran 4. Prosedur penggunaan spektrofotometer
1. Sebelum digunakan spektrofotometer dihidupkan terlebih dahulu 30 menit, dengan memutar power switch searah jarum jam.
2. Panjang gelombang diatur dengan memutar tombol pengatur pada 600 nm 3. Nilai transmisi harus diatur pada 0% dengan memutar tombol pengatur
kekanan/kekiri
4. Tabung blanko dibersihkan sebelum diletakkan pada ruang sampel.
5. Ruang sampel ditutup dan diatur 100% T dengan memutar tombol pengatur
6. Tabung blanko diangkat dan dimasukkan tabung yang sudah berisi cairan sampel
7. Nilai yang didapat adalah dalam bentuk %T 8. Nilai ini diolah menjadi OD = 2-Log %T
30 Lampiran 5. Media Trypticase Soy Agar (TSA)
Medium TSA ini digunakan untuk menumbuhan bakteri. Pembuatan TSA dilakukan degan cara 4 g TSA dilarutkan dengan 100 mℓ akuades dalam erlenmayer dan ditutup dengan alumunium foil. Setelah itu media dihomogenkan dengan menggunakan penangas air sambil diaduk hingga larut dan homogen. Kemudian disterilkan menggunakan autoclave pada suhu 1210C, tekanan 1 atm selama 15 menit. Setelah itu dimasukkan kedalam tabung reaksi steril atau cawan petri steril secara aseptik, setelah itu didiamkan sampai agar mengeras, selanjutnya dimasukkan ke dalam plastik yang telah disemprot dengan alkohol, lalu disimpan dalam kulkas.