III.
HASIL DAN PEMBAHASAN
A.
Kultur Murni Hasil Pengecekan Kemurnian
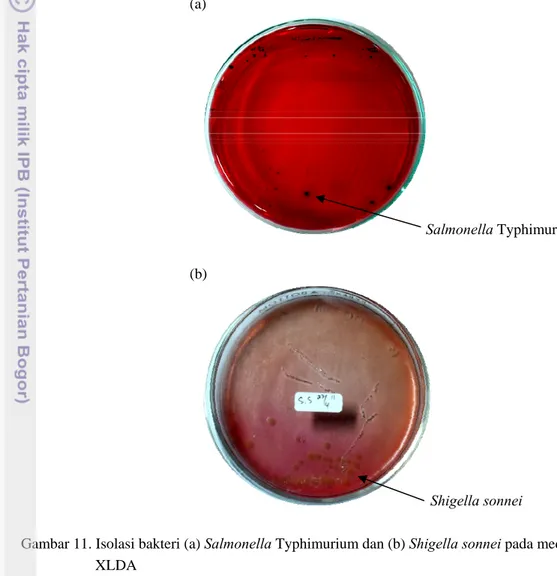
Hasil goresan kuadran kultur murni ke dalam media Xylose Lysine Deoxycholate Agar (XLDA) menghasilkan koloni Salmonella Typhimurium berwarna merah muda dengan inti hitam di tengahnya dimana hal tersebut sesuai dengan yang dijelaskan pada Bacteriological Analytical Manual mengenai
Salmonella (Andrews dan Hammack 2011). Sedangkan Shigella sonnei membentuk koloni merah muda tanpa ada inti hitam (Health Protection Agency 2007). Hasil pengisolasian Salmonella Typhimurium dan Shigella sonnei dapat dilihat pada Gambar 11. di bawah ini.
(a) Salmonella Typhimurium (b) Shigella sonnei
Gambar 11. Isolasi bakteri (a) Salmonella Typhimurium dan (b) Shigella sonnei pada media XLDA
Masing-masing satu koloni spesifik tersebut kemudian diisolasi dalam Heart Infusion Broth dan diinkubasi selama 24 jam pada suhu 37oC sebagai kultur murni mikroba spesifik Salmonella Typhimurium dan Shigella sonnei yang selanjutnya digunakan dalam proses isolasi/ekstraksi DNA dan juga diinokulasi ke dalam sampel pangan susu UHT (sampel susu UHT spike). Penelitian yang dilakukan Omiccioli et al. (2009) dan Hadjinicolaut et al. (2009) juga menggunakan XLDA dalam tahap pengayaan mengkulturkan mikroba.
B.
Inokulasi Susu dengan Kultur Murni
Salmonella
Typhimurium dan
Shigella
sonnei
(Sampel Susu UHT
spike
)
Hasil hitungan secara mikroskopi dengan menggunakan petroff-hausser terhadap suspensi
Salmonella Typhimurium pada larutan pengencer NaCl 0,85% yaitu sebesar 1,4 x 108 sel/ml
Salmonella Typhimurium untuk proses isolasi DNA dengan metode kit komersial dan 1,6 x 107 sel/ml
Salmonella Typhimurium untuk proses isolasi DNA dengan metode pendidihan, sedangkan
penghitungan terhadap Shigella sonnei yaitu sebesar 2,5 x 107 sel/ml. Ketiga penghitungan tersebut dapat dilihat pada Lampiran 5. Karena konsentrasi suspensi lebih besar dari 105 sel/ml maka dilakukan pengenceran terhadap suspensi tersebut hingga diperoleh konsentrasi suspensi sekitar 105 sel/ml. Setelah dilakukan pengenceran, jumlah Salmonella Typhimurium pada susu UHT yang akan diisolasi/diekstraksi dengan metode kit komersial yaitu sebanyak 7,0 x 104 atau 0,7 x 105 sel/ml, pada susu UHT yang akan diisolasi/diekstraksi dengan metode pendidihan sebanyak 8,0 x 104 atau 0,8 x 105 sel/ml Salmonella Typhimurium, dan jumlah Shigella sonnei pada susu UHT yang akan diisolasi/diekstraksi dengan metode kit komersial sebanyak 1,2 x 105 sel/ml.
Hasil penghitungan konsentrasi akhir Salmonella Typhimurium pada sampel susu UHT spike dan penghitungan awal mikroba natural pada susu UHT dengan metode konvensional dapat dilihat pada Lampiran 6. Hasil tersebut menunjukkan bahwa sampel susu UHT tidak mengandung mikroba
Salmonella Typhimurium dan ketika diinokulasi dengan Salmonella Typhimurium maka sampel susu UHT spike tersebut mengandung 3,9 x 104 CFU/ml (penghitungan ke-1) dan 2,6 x 104 CFU/ml (penghitungan ke-2) Salmonella Typhimurium yang tumbuh dan hidup di dalamnya. Penghitungan ke-1 merupakan tahapan inokulasi Salmonella Typhimurium ke dalam susu yang akan disolasi/ diekstraksi dengan metode kit komersial dan penghitungan ke-2 merupakan tahapan inokulasi
Salmonella Typhimurium yang akan disolasi/diekstraksi dengan metode pendidihan.
Konsentrasi Salmonella Typhimurium yang terkandung di dalam sampel susu UHT spike (104 CFU/ml) tidak sama dengan konsentrasi Salmonella Typhimurium yang ditambahkan dimana diukur dengan menggunakan pengukuran mikroskopi pada petroff-hausser (105 sel/ml). Hal tersebut dikarenakan penghitungan dengan petroff-hausser tidak dapat membedakan mikroba yang hidup dan yang telah mati sehingga Salmonella Typhimurium yang telah mati juga ikut terhitung bersama dengan sel yang hidup. Sedangkan metode konvensional pada media XLDA, Salmonella Typhimurium yang terhitung adalah mikroba yang hidup saja. Sehingga hitungan konsentrasi
Salmonella Typhimurium yang ditambahkan lebih besar dibandingkan konsentrasi akhir pada sampel susu UHT spike.
C.
Isolat/Template DNA yang Dihasilkan
Satu hal penting yang dibutuhkan untuk mendeteksi suatu mikroba dengan menggunakan
real-time PCR adalah DNA mikroba tersebut yang murni tanpa pengotor. Cara memperolehnya adalah dengan mengisolasi/mengekstraksi DNA dari dalam sel. Berbagai teknik ekstraksi DNA salah satunya metode pendidihan telah dikembangkan dari prinsip dasar tersebut, sehingga saat ini muncul berbagai teknik ekstraksi dan purifikasi DNA dalam bentuk kit, dimana prosesnya cukup mudah, cepat, dan sederhana (Sulandari dan Zein 2003).
1.
Isolasi/Ekstraksi DNA dengan Metode Pendidihan
DNA yang berasal dari bakteri Gram negatif (contohnya: Salmonella) dapat dengan mudah diisolasi/diekstraksi dengan menggunakan metode pendidihan atau mendidihkan sel bakteri di dalam air (Lee et al. 2006). Kemurnian isolat DNA yang dihasilkan dapat diketahui dengan mengukurnya pada UV-VIS spektrofotometer. Isolat DNA dikatakan murni jika rasio diantara nilai absorbansi pada panjang gelombang 260 dan 280 berada pada selang 1,8 hingga 2,0 (Nolan
et al. 2007). Di bawah ini merupakan hasil pengukuran kemurnian isolat/template DNA yang diperoleh dengan metode pendidihan (Tabel 2.).
Tabel 2. Kemurnian isolat DNA Salmonella Typhimurium dan Shigella sonnei dengan metode pendidihan
Sampel A1 (260) A2 (280) Rasio A1/A2 [DNA] ng/µl [Protein] ng/µl Spike ST1 0,068 0,097 0,6 0,367 86,964 Spike ST2 0,068 0,096 0,6 0,356 82,964 Spike SS1 0,071 0,100 0,6 0,360 85,708 Spike SS2 0,062 0,089 0,6 0,329 81,386 KM ST1 0,127 0,077 1,6 5,188 23,002 KM ST2 0,098 0,059 1,7 4,009 16,948 KM SS1 0,079 0,057 1,4 2,909 28,399 KM SS2 0,105 0,076 1,4 3,871 36,948
Ket: Spike ST1 & 2 (Sampel susu UHT spikeSalmonella Typhimurium ulangan 1 dan 2); Spike SS1 & 2 (Sampel susu UHT spikeShigella sonnei ulangan 1 dan 2); KM ST1 & 2 (Kultur Murni Salmonella Typhimurium ulangan 1 dan 2); KM SS1 & 2 (Kultur Murni Shigella sonnei ulangan 1 dan 2). A1 adalah nilai absorbansi pada panjang gelombang 260; A2 adalah nilai absorbansi pada panjang gelombang 280.
Berdasarkan hasil pengukuran spektrofotometri tersebut menunjukkan bahwa isolat DNA yang dihasilkan dari metode pendidihan belum murni yang artinya selain DNA, masih terdapat pengotor yang terkandung di dalam isolat tersebut salah satunya adalah protein. Hal ini ditunjukkan dengan nilai rasio yang tidak berada pada selang 1,8-2,0 baik pada sampel susu UHT
spike Salmonella Typhimurium dan Shigella sonnei maupun sampel kultur murni Salmonella Typhimurium dan Shigella sonnei yang diambil dari media pengayaan HIB. Adanya pengotor pada isolat tersebut ditunjukkan pula dengan tingginya nilai konsentrasi protein yang dihasilkan.
Nilai konsentrasi protein yang dihasilkan pada sampel susu UHT spike jauh lebih tinggi dibandingkan dengan konsentrasi protein yang diperoleh dari sampel kultur murni mikroba spesifik. Hal tersebut menunjukkan bahwa perlu adanya modifikasi metode pendidihan khusus untuk sampel pangan yang mengandung protein tinggi seperti susu. Selain mengandung protein, susu juga mengandung lemak, kation (kalsium/Ca2+), dan pengkelat yang tinggi dimana komponen pangan tersebut menjadi inhibitor pada pengujian dengan menggunakan real-time PCR. Inhibitor tersebut dapat mengikat dan menurunkan aktivitas enzim polimerase, menyebabkan perubahan konformasi dalam DNA target, atau bersaing dengan primer untuk menempati primer binding sites (Lee et al. 2006 dan Siebert 1999).
Hasil spektrofotometri tersebut membuktikan bahwa tahap pengisolasian DNA dari suatu matriks pangan merupakan hal yang sangat kritis dan kompleks dalam menjalankan pengujian dengan menggunakan real-time PCR. Keberadaan komponen gizi pada pangan dapat menjadi inhibitor PCR dimana dapat memberikan efek yang bervariasi, tetapi secara umum inhibitor tersebut dapat mempersulit pendeteksian DNA bakteri yang memiliki konsentrasi rendah (Lee et
al. 2006).
Hasil isolasi/ekstraksi DNA pada penelitian ini sesuai dengan penelitian yang dilakukan oleh Amagliani et al. (2006) yang membandingkan metode isolasi/ekstraksi DNA dengan cara pendidihan dan dengan kit komersial DNeasy Tissue Kit (Qiagen) berdasarkan pengukuran rasio
A260/A280 pada UV-1700 spektrofotometer di dalam sampel pangan keju mozarela yang kaya akan lemak dan kalsium sebagai inhibitor dimana inhibitor tersebut juga terkandung pada susu UHT sebagai sampel pangan yang diuji pada penelitian ini. Penelitian Amagliani et al. (2006) tersebut menunjukkan bahwa metode pendidihan menghasilkan kemurnian isolat DNA yang kurang baik dimana nilai rasio A260/A280 lebih rendah dari 1,8.
Hal tersebut terbukti pada pengujian kuantifikasi Salmonella Typhimurium dengan real-time PCR yang akan dibahas pada sub bab selanjutnya. Walaupun terdapat inhibitor di dalam isolat DNA yang diisolasi/diekstraksi dengan metode pendidihan, tetapi isolat tersebut masih dapat teramplifikasi contohnya pada pengujian penentuan spesifisitas primer InvA yang digunakan dan juga pada pengujian penentuan konsentrasi primer yang tepat dimana akan dibahas pada sub bab selanjutnya.
2.
Isolasi/Ekstraksi DNA dengan Kit Komersial
Keberadaan inhibitor baik dalam media kultur murni maupun dalam sampel pangan dapat menghambat proses amplifikasi dengan real-time PCR. Untuk mengatasinya, Chen et al. (1997) diacu dalam Lee et al. (2006) menggunakan metode kit komersial dalam proses isolasi/ekstraksi DNA Salmonella dari sampel susu nonpasteurisasi (raw milk), selain itu juga menggunakan proses pengayaan dan sentrifugasi untuk tahapan memanen/mengambil patogen. Begitu juga Omiccioli et al. (2009) dimana melakukan hal yang sama, namun menggunakan merk kit komersial yang berbeda. Isolat/template DNA yang dihasilkan diuji tingkat kemurniannya dengan menggunakan spektrofotometer dimana prinsipnya sama dengan pengukuran isolat DNA metode pendidihan. Hasil pengujiannya dapat dilihat pada Tabel 3. di bawah ini.
Tabel 3. Kemurnian isolat DNA Salmonella Typhimurium dan Shigella sonnei dengan metode kit komersial
Sampel A1 (260) A2 (280) Rasio A1/A2 [DNA] ng/µl [Protein] ng/µl KM ST 0,047 0,029 2,0 1,610 -0,034 KM SS 0,003 0,001 2,0 0,180 0,075 Spike ST 0,036 0,021 2,1 1,305 -0,162
Ket: KM ST (Kultur Murni Salmonella Typhimurium); KM SS (Kultur Murni Shigella sonnei); Spike
ST ( Sampel susu UHT spike Salmonella Typhimurium)
Hasil pengukuran tersebut menunjukkan bahwa isolat DNA ketiga sampel yaitu kultur murni
Salmonella Typhimurium, kultur murni Shigella sonnei, dan sampel susu UHT spikeSalmonella Typhimurium memiliki nilai rasio yang diharapkan yaitu berada pada selang 1,8-2,0. Hal tersebut menunjukkan bahwa kemurnian ketiga isolat DNA sangat baik, namun pada sampel susu UHT spikeSalmonella Typhimurium, nilai rasio yang dihasilkan sedikit melebihi 2,0. Jika nilai rasio yang dihasilkan melebihi 2,0 maka, hal tersebut mengindikasikan bahwa isolat DNA tidak mengandung/terkontaminasi protein tetapi masih mengandung RNA di dalamnya (Anonoim 2007). Sedangkan kultur murni Salmonella Typhimurium dan kultur murni Shigella sonnei menghasilkan nilai rasio tepat 2,0 dan konsentrasi protein yang dihasilkan pun sangat rendah jika dibandingkan dengan isolat kultur murni Shigella sonnei yang diisolasi/diekstraksi dengan metode pendidihan. Hal ini menunjukkan bahwa metode isolasi dengan menggunakan kit komersial menghasilkan isolat DNA yang jauh lebih murni dibandingkan dengan metode
pendidihan yang dilakukan. Hal tersebut dikarenakan metode kit komersial memiliki prinsip metode dengan perlakuan proteinase K yang diikuti dengan pengikatan DNA pada membran gel silika/filter sehingga kontaminan akan turun/terpisah ke dalam spin column/collection tube (Dauphin et al. 2009).
Hasil isolasi/ekstraksi DNA pada penelitian ini sesuai dengan penelitian yang dilakukan oleh Amagliani et al. (2006) yang membandingkan metode isolasi/ekstraksi DNA dengan cara pendidihan dan dengan kit komersial DNeasy Tissue Kit (Qiagen). Penelitian Amagliani et al. (2006) tersebut menunjukkan bahwa nilai rasio pada isolat DNA yang dihasilkan dengan DNeasy Tissue Kit (Qiagen) dimana memiliki tingkat kemurnian yang kurang baik bahkan lebih jelek dibanding dengan metode pendidihan, namun tidak pada penelitian yang dilakukan dimana isolat/template DNA yang dihasilkan dengan kit komersial QIAamp® DNA Blood Mini Kit (Qiagen) menghasilkan isolat/template DNA yang lebih murni dibanding dengan metode pendidihan. Hal tersebut dapat dikarenakan pengaruh modifikasi metode kit komersial yang dilakukan pada penelitian ini dari metode sesungguhnya yang berdasarkan petunjuk produser kit terkait.
Penelitian Amagliani et al. (2006) juga menunjukkan konsentrasi/yield DNA yang dihasilkan dengan metode pendidihan lebih besar dibandingkan dengan metode kit komersial. Hal tersebut juga sesuai dengan penelitian ini dimana konsentrasi DNA kultur murni Salmonella Typhimurium dan kultur murni Shigella sonnei yang diperoleh dengan metode pendidihan lebih besar dibandingkan dengan metode kit komersial, namun tidak terjadi pada sampel susu UHT spike dimana konsentrasi/yield DNA yang dihasilkan lebih besar dengan metode kit komersial dibandingkan dengan metode pendidihan. Hal tersebut dapat menunjukkan bahwa DNA
Salmonella Typhimurium lebih mudah diisolasi/diekstraksi dari sampel susu UHT dengan menggunakan metode kit komersial yang dimodifikasi.
Hal yang sama juga ditunjukkan pada penelitian Dauphin et al. (2009) dimana membandingkan kemurnian isolat/template DNA yang dihasilkan dengan berbagai macam kit salah satunya adalah QIAamp® DNA Blood Mini Kit (Qiagen) dimana kit tersebut juga digunakan pada penelitian ini. Penelitian yang dilakukan oleh Dauphin et al. (2009) menunjukkan bahwa isolat/template DNA yang dihasilkan dengan kit QIAamp® DNA Blood Mini Kit (Qiagen) memiliki kemurnian yang kurang baik dimana nilai rasio A260/A280 lebih kecil dari 1,8 sehingga menandakan bahwa isolat DNA tersebut masih mengandung inhibitor/pengotor. Hal tersebut tidak sesuai dengan kemurnian yang dihasilkan dari penelitian ini dimana menghasilkan isolat/template DNA dengan kemurnian yang baik. Hal tersebut menunjukkan bahwa modifikasi dengan penambahan CTAB terhadap metode yang berasal dari produsen berkait memperbaiki hasil kemurnian isolat DNA menjadi lebih baik.
D.
Hasil Penentuan Konsentrasi Primer
Pertama kali yang perlu dilakukan dalam pengujian real-time PCR adalah mengevaluasi dan mengkaji penggunaan konsentrasi primer yang tepat (Pestana et al. 2010). Pemilihan konsentrasi primer yang tepat dari sejumlah konsentrasi primer yang diujikan, dilakukan berdasarkan nilai Ct yang paling rendah dan fluoresen yang cukup memenuhi syarat terhadap konsentrasi primer target sehingga menghasilkan primer-dimer seminimal mungkin atau bahkan tanpa menghasilkan primer-dimer (Pestana et al., 2010). Hasil penentuan konsentrasi primer yang telah dilakukan pada berbagai macam template/isolat DNA dapat dilihat pada Tabel 4. berikut ini. Kurva amplifikasi yang dihasilkan dapat dilihat pada Lampiran 7. sampai dengan Lampiran 12.
Tabel 4. Pengaruh konsentrasi primer terhadap nilai Threshold Cycle (Ct) Template DNA [Primer] awal
(µM) [Primer] akhir (µM) Nilai Ct Kultur Murni S. Typhimurium 0,5 0,0125 19,17 1 0,0250 14,15 5 0,1250 11,39 S. Typhimurium 103 sel/ml 1 0,0250 tidak teramplifikasi 5 0,1250 31,35
Sampel susu UHT spike 1 0,0250 27,79
S. Typhimurium 5 0,1250 24,29
Konsentrasi primer akhir yang dimaksud adalah konsentrasi primer awal yang telah dicampurkan ke dalam master mix sehingga diperoleh master mix yang mengandung primer dengan konsentrasi yang lebih rendah (konsentrasi primer akhir) karena terjadi pengenceran dengan bahan-bahan lainnya seperti EvaGreen, isolat/template DNA, dan buffer TE pada master mix. Penentuan konsentrasi primer untuk sampel kultur murni Salmonella Typhimurium yang diambil dari media HIB menghasilkan nilai threshold cycle (Ct) yang berbeda pula jika diuji dengan menggunakan konsentrasi primer yang berbeda. Begitu juga pada sampel Salmonella Typhimurium 103 sel/ml dan sampel susu UHT spikeSalmonella Typhimurium.
Berdasarkan Tabel 4. di atas, konsentrasi primer yang tepat untuk pengujian kultur murni
Salmonella Typhimurium adalah 0,125 µM dibandingkan jika menggunakan konsentrasi primer 0,025 dan 0,0125 µM. Hal tersebut dikarenakan nilai Ct yang dihasilkan pada konsentrasi primer 0,125 µM paling rendah jika dibandingkan dengan nilai Ct pada konsentrasi primer lainnya.
Kemudian penentuan konsentrasi primer juga dilakukan pada suspensi kultur murni yang telah diencerkan sehingga jumlahnya menjadi lebih rendah yaitu mengandung 103 sel/ml Salmonella Typhimurium yang dihitung dengan hitungan mikroskopi pada petroff-hausser. Pengujian ini berfungsi untuk mengetahui sensitifitas metode dengan menggunakan konsentrasi primer yang berbeda. Hasil pengujian menunjukkan bahwa penggunaan konsentrasi primer 0,025 µM gen target
InvA pada Salmonella Typhimurium tidak dapat teramplifikasi sedangkan konsentrasi primer 0,125 µM masih dapat mengamplifikasi Salmonella Typhimurium sejumlah 103 sel/ml.
Penentuan konsentrasi primer ini juga dilakukan pada sampel susu UHT spike. Hasil pengujian tersebut menunjukkan bahwa konsentrasi primer 0,125 µM menghasilkan nilai Ct yang lebih rendah dibandingkan dengan konsentrasi primer 0,025 µM. Dari pengujian tersebut dapat disimpulkan bahwa penggunaan konsentrasi primer 0,125 µM cukup sesuai untuk pengujian Salmonella Typhimurium sampai konsentrasi 103 sel/ml baik berupa suspensi dari kultur murni maupun yang berada pada sampel pangan susu UHT.
Selain itu juga, indikator dalam penentuan konsentrasi primer yang tepat adalah berdasarkan melt
peak curve yang dihasilkan dimana harus mengandung satu puncak saja dan tidak menghasilkan
mispriming. Gambar melt peak curve dari pengujian kultur murni Salmonella Typhimurium, suspensi 103 sel/ml Salmonella Typhimurium, dan sampel susu UHT spikeSalmonella Typhimurium dengan menggunakan konsentrasi primer 0,125 µM adalah sebagai berikut ini (Gambar 12.).
(a) (b)
(c)
Gambar 12. Kurva puncak pelelehan/melt peak curve dengan konsentrasi primer 0,125 µM. (a) Sampel kultur murni Salmonella Typhimurium, (b) sampel 103 sel/ml
Salmonella Typhimurium, (c) Sampel susu UHT spike 105 sel/ml Salmonella Typhimurium
Ketiga kurva tersebut menghasilkan hanya satu puncak yang tinggi dan satu suhu pelelehan (Tm) yaitu sebesar 84,50 yang dapat dilihat pada tabel di Lampiran 8, 10, 12. Terbentuknya satu puncak dan satu suhu pelelehan menandakan bahwa konsentrasi primer 0,125 µM cukup tepat untuk pengujian ini, yang artinya tidak terlalu besar dimana dapat menyebabkan mispriming/primer-dimer (terbentuk lebih dari satu puncak yang tinggi) dan tidak pula terlalu rendah dimana dapat menyebabkan false-negative, namun pada kurva puncak pelelehan yang ditunjukkan pada Gambar 12c. untuk sampel susu UHT
spike terdapat/terbentuk satu puncak kecil di sebelah kiri puncak pelelehan utama yang memiliki nilai Tm yang lebih rendah, hal tersebut menandakan adanya primer-dimer (sesama primer yang saling berikatan), dimana terbentuk suatu DNA untai ganda yang sangat pendek. Terbentuknya primer-dimer tersebut sangat minimal dimana penentuan konsentrasi 0,125 µM sebagai primer yang sesuai masih memenuhi syarat dari Pestana et al. (2010).
Penelitian yang dilakukan Ahmed et al. (2010) yang melakukan optimasi konsentrasi primer mulai dari konsentrasi 0,1 hingga 0,5 µM dengan menghasilkan konsentrasi optimum sebesar 0,3 µM. Perbedaan hasil konsentrasi primer optimum yang diperoleh ini dikarenakan perbedaan label fluoresen yang digunakan. Jika menggunakan SYBR® green I sebagai label fluoresen, maka konsentrasi primer yang digunakan berada pada selang 0,05-0,3 µM sedangkan jika menggunakan TaqMan atau Molecular Beacon sebagai label fluoresen maka konsentrasi primer yang digunakan berada pada selang 0,05 hingga 0,6 µM (Pestana et al. 2010). Penelitian yang dilakukan Ahmed et al. (2010) menggunakan SYBR® Green I sebagai label fluoresen sedangkan penelitian ini menggunakan EvaGreen dye. Sehingga penggunaan konsentrasi primer yang tepat pada suatu penelitian bergantung pada bahan lain yang digunakan salah satunya adalah label fluoresen, namun berdasarkan protokol dalam kemasan EvaGreen, evagreen dye yang digunakan cara kerjanya sama dengan SYBR® Green I
dan konsentrasi primer yang tepat pada penelitian ini yaitu sebesar 0,125 µM dimana telah masuk pada selang konsentrasi primer yang sesuai jika pengujian dengan real-time PCR menggunakan SYBR® green I sebagai bahan pendar yaitu antara 0,05 hingga 0,3 µM (Pestana et al. 2010).
Penelitian Shanmugasundaram et al. (2009) dimana menggunakan reverse dan forward primer dengan urutan nukleotida yang sama, menggunakan sebanyak 0,15 µM primer pada pengujian dengan
real-time PCR. Hal tersebut tidak berbeda jauh dengan konsentrasi primer yang digunakan pada penelitian ini yaitu sebesar 0,125 µM.
E.
Hasil Penentuan Spesifisitas Primer
Reverse dan forward primer yang digunakan untuk mendeteksi gen target InvA pada Salmonella Typhimurium diperkirakan berukuran 211 bp. Kedua jenis primer tersebut telah digunakan pada penelitian yang dilakukan Shanmugasundaram et al. pada tahun 2009. Primer tersebut kemudian dianalisis dengan menggunakan Basic Local Alignment Search Tool (NCBI 2011) dan hasil analisisnya dapat dilihat pada Lampiran 13. Untuk lebih meyakinkan bahwa kedua primer tersebut spesifik terhadap Salmonella Typhimurium maka dilakukan pengujian Salmonella Typhimurium sebagai kontrol positif dan Shigella sonnei sebagai kontrol negatif dengan menggunakan real-time PCR. Hasil penentuan spesifisitas primer dengan menggunakan real-time PCR dapat dilihat pada kurva amplifikasi dan kurva puncak pelelehan pada Gambar 12. dan Gambar 13. berikut ini.
Gambar 13. Kurva amplifikasi uji spesifisitas primer
Gambar 14. Kurva puncak pelelehan (melt peak curve) uji spesifisitas primer
Salmonella Typhimurium Shigella sonnei Ct Salmonella Typhimurium Shigella sonnei
Kurva amplifikasi yang dihasilkan oleh real-time PCR menunjukkan bahwa kontrol negatif tidak teramplifikasi dan bakteri uji teramplifikasi dengan nilai Threshold Cycle (Ct) sebesar 12,37. Hal tersebut menandakan bahwa primer yang digunakan spesifik untuk Salmonella. Proses amplifikasi tersebut mencirikan bahwa forward dan reverse primer yang ditambahkan dapat berikatan dan bereplikasi dengan sejumlah sekuen nukleotida di dalam gen target InvA yang hanya terdapat pada
Salmonella khususnya serovar Typhimurium.
Selain menganalisis kurva amplifikasi (amplification curve), pengujian spesifisitas primer juga dilakukan dengan menganalisis kurva puncak pelelehan (melt peak curve) dengan melihat titik puncak pelelehan (melt peak) dan nilai Tm (Melting Temperature) yang dihasilkan pada kurva tersebut.
Kurva puncak pelelehan menunjukkan bahwa kontrol positif Salmonella Typhimurium hanya menghasilkan satu puncak. Hal tersebut menunjukkan bahwa pada pengujian tersebut tidak terjadi
mispriming yang artinya primer yang digunakan spesifik dan tidak mengamplifikasi gen lain selain gen target. Nilai Tm yang dihasilkan sebesar 84,50oC. Nilai tersebut dapat menjadi ciri khusus bagi
Salmonella Typhimurium pada pengujian selanjutnya. Selain itu kurva tersebut juga menunjukkan bahwa kultur murni Shigella sonnei tidak menghasilkan puncak dan nilai Tm yang menandakan bahwa primer tidak dapat mendeteksi bakteri lain selain Salmonella khususnya serovar Typhimurium.
Shanmugasundaram et al. (2009) dalam penelitiannya dimana menggunakan reverse dan forward primer InvA dengan urutan nukleotida yang sama menunjukkan hasil yang sama pula dengan penelitian ini dimana primer InvA tersebut spesifik Salmonella. Hasil tersebut ditunjukkan dengan dapat teramplifikasinya/menghasilkan uji positif pada berbagai subspesies dan serovar Salmonella dengan strain yang berbeda tetapi tidak dapat mengamplifikasi/menghasilkan uji negatif pada jenis mikroba selain Salmonella seperti Escherichia coli, Bacillus cereus, Yersinia intermedia, Yersinia
enterocolitica, Aeromonas hydrophila, dan Staphylococcus aureus.
F.
Kuantifikasi
Salmonella
Typhimurium pada Sampel Pangan Susu UHT
Pada pengkuantifikasian
Salmonella Typhimurium pada sampel susu UHT diperoleh dua jenis kurva standar yaitu kurva standar yang dihasilkan dari isolat/template DNA dengan metode pendidihan dan kurva standar yang diperoleh dari isolat/template DNA dengan metode kit komersial. Kedua kurva standar yang dihasilkan tersebut menghasilkan efisiensi pengujian, nilai R2 dan slope yang berbeda.1.
Kurva Standar Metode Pendidihan
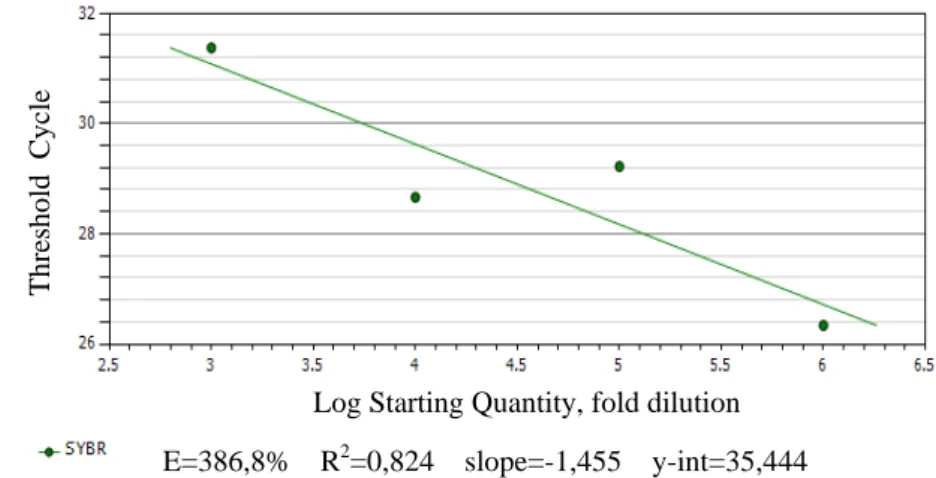
Kurva standar yang dibuat dengan menggunakan isolat/template DNA dengan metode pendidihan menghasilkan slope sebesar -1,455 dimana nilai tersebut tidak berada dalam selang
slope yang diharapkan, sehingga nilai efisiensi yang diperoleh tidak bagus yaitu sebesar 386,8% dan nilai R2 yang rendah yaitu sebesar 0,824. Kurva standar yang dibuat dengan metode pendidihan ini tidak dapat digunakan dalam pengkuantifikasian Salmonella Typhimurium dalam sampel susu UHT spike karena akan menyebabkan ketidaksesuaian dari hasil yang diperoleh.
Kurva standar yang dihasilkan dari isolat DNA dengan menggunakan metode pendidihan ditunjukkan pada Gambar 15. berikut ini dan nilai threshold cycle (Ct) serta kurva amplifikasi yang dihasilkan untuk membuat kurva standar ini dapat dilihat pada Lampiran 14a. dan Lampiran 14b. Kurva pelelehan dan kurva puncak pelelehan yang dihasilkan dalam membuat kurva standar dapat dilihat pada Lampiran 15a. dan Lampiran 15b., sedangkan tabel suhu pelelehannya dapat dilihat pada Lampiran 15c.
Gambar 15. Kurva standar metode pendidihan
Nilai efisiensi pengujian dipengaruhi oleh beberapa faktor diantaranya adalah urutan sekuen yang diamplifikasi, urutan sekuen primer yang digunakan, panjang sekuen yang diamplifikasi, dan kemurnian isolat DNA/inhibitor yang terdapat pada isolat (Siebert 1999). Kurva standar yang dihasilkan dengan metode pendidihan ini memiliki efisiensi yang buruk, hal tersebut dikarenakan tidak murninya isolat DNA yang digunakan/dihasilkan dari metode pendidihan.
Ketidakmurnian isolat DNA dapat dilihat dari hasil pengukuran dengan spektrofotometer pada Lampiran 16. dimana nilai rasio tidak berada pada selang 1,8-2,0 dan nilai konsentrasi protein yang begitu tinggi. Keberadaan inhibitor pada suatu reaksi real-time PCR dapat ditunjukkan dengan meningkatnya nilai efisiensi yang dikarenakan meningkatnya nilai Ct dan penurunan nilai absolut dari slope (Pestana et al. 2010).
Selain itu tingginya nilai persentase effisiensi yang diperoleh (>110%) menjadi indikator terjadinya pippeting error ketika dilakukannya pengenceran, terjadinya amplifikasi pada produk yang non-spesifik, dan keberadaan dari primer-dimer (Pestana et al. 2010). Primer-dimer merupakan proses saling berikatannya primer (baik itu sesama reverse primer, sesama forward primer, maupun antara reverse dan forward primer) yang teramplifikasi dan terkuantifikasi sehingga dihasilkan pengujian yang false-positive (sampel yang negatif menghasilkan uji yang positif)(Pestana et al. 2010).
Berdasarkan hasil tersebut perlu dilakukan pengembangan metode pendidihan lebih lanjut untuk mengatasi inhibitor yang terbawa pada isolat DNA salah satunya adalah dengan menambahkan Chelex-100 pada metode pendidihan seperti yang dilakukan oleh Kim et al. (2001) pada penelitiannya mengenai pengujian S. aureus pada sampel susu mastitis dimana metode pendidihan yang dilakukannya menghasilkan sensitivitas pengujian yang rendah. Chelex-100 merupakan resin pengkelat berupa kopolimer styrene divinilbenzene yang mengandung pasangan ion-ion iminodiasetat. Chelex-100 efisien diregenerasi di dalam asam encer dan dapat digunakan dalam kondisi basa, netral, dan asam lemah pada pH 4 atau lebih tinggi. Chelex-100 bertindak sebagai penukar anion pada pH yang sangat rendah. Resin chelex-100 memiliki banyak kegunaan diantaranya adalah menghilangkan logam dari reagen dan media kultur, mempurifikasi dinukleotida, menghilangkan kalsium di dalam darah, dan ekstraksi DNA untuk PCR (Bio-Rad Laboratories 2011). Kim et al. (2001) juga telah mengujikan metode pendidihan dengan menambahkan PBS dan metanol untuk menghilangkan inhibitor pada susu, namun hasilnya isolat DNA yang dihasilkan tetap mengandung inhibitor dimana mempengaruhi aktivitas Taq DNA Polimerase yang digunakan.
E=386,8% R2=0,824 slope=-1,455 y-int=35,444 Log Starting Quantity, fold dilution
Thre
shold C
y
Selain itu juga dapat menambahkan/menggantikan suatu komponen yang mengatasi inhibitor pada saat pengujian sampel susu dengan real-time PCR. Salah satunya dengan mengganti Taq DNA Polimerase dengan menggunakan Tth DNA Polimerase seperti pada penelitian yang dilakukan oleh Kim et al. (2001). Menurutnya, Tth DNA Polimerase lebih sensitif daripada Taq DNA Polimerase dalam pengujian sampel susu karena Taq DNA Polimerase diganggu/dihambat oleh inhibitor yang terdapat pada susu. Selain itu, Tth DNA Polimerase dapat menghasilkan data yang konsisten pada pengujian sebanyak tiga kali ulangan.
2.
Kurva Standar Metode Kit Komersial
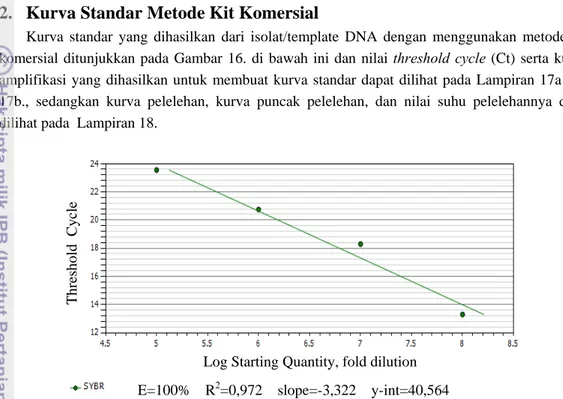
Kurva standar yang dihasilkan dari isolat/template DNA dengan menggunakan metode kit komersial ditunjukkan pada Gambar 16. di bawah ini dan nilai threshold cycle (Ct) serta kurva amplifikasi yang dihasilkan untuk membuat kurva standar dapat dilihat pada Lampiran 17a dan 17b., sedangkan kurva pelelehan, kurva puncak pelelehan, dan nilai suhu pelelehannya dalat dilihat pada Lampiran 18.
Gambar 16. Kurva standar metode kit komersial
Kurva standar yang dihasilkan dari suspensi Salmonella Typhimurium dengan konsentrasi 105 hingga 108 sel/ml yang diperoleh dengan metode kit komersial memiliki nilai slope -3,3 dimana nilai tersebut termasuk ke dalam slope yang diharapkan. Oleh karena itu, nilai efisiensi yang diperoleh sangat baik yaitu sebesar 100% dengan nilai R2 mencapai 0,972. Persamaan garis dari kurva standar tersebut yaitu y = 40,564 - 3,322x dengan y adalah nilai Ct dan x adalah log konsentrasi mikroba yang diuji. Nilai tersebut dapat dihasilkan karena isolat DNA yang digunakan cukup murni dimana dapat dilihat pada Lampiran 19. Nilai efisiensi yang mendekati dan mencapai 100% adalah indikator yang baik untuk pengujian yang reliable, reproducible, dan
robust (Pestana et al. 2010). Oleh karena itu, kurva standar ini dapat digunakan dalam proses kuantifikasi Salmonella Typhimurium pada sampel susu UHT spike.
Susu yang telah mengandung 3,9 x 104 CFU/ml Salmonella Typhimurium tersebut masuk ke dalam tahap isolasi/ekstraksi DNA dengan metode kit komersial dan diamplifikasi dengan
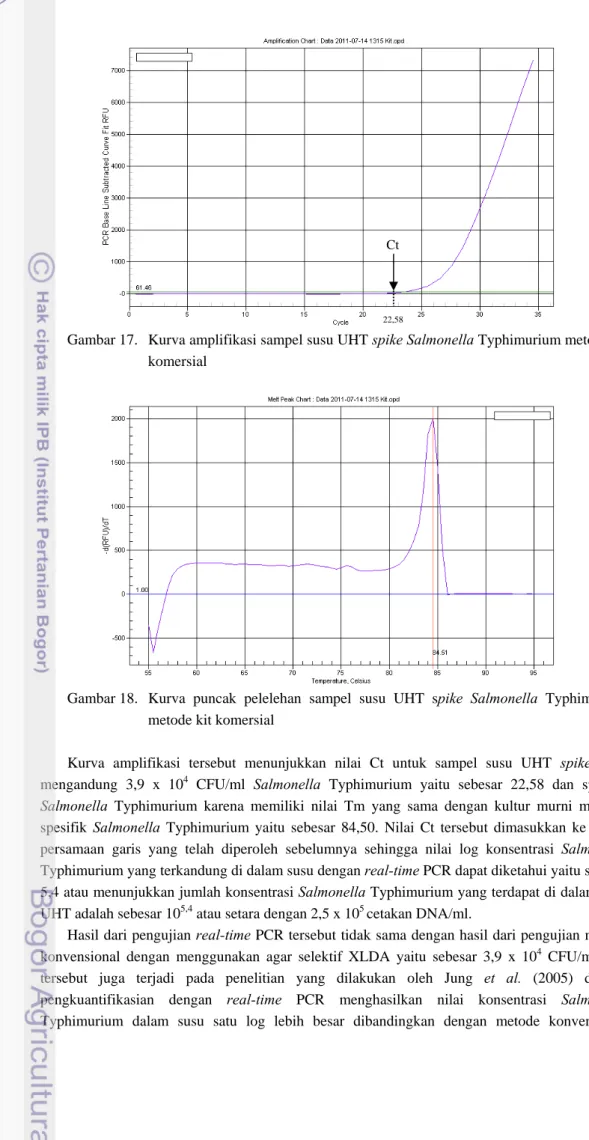
real-time PCR dengan menggunakan konsentrasi dan jenis primer yang telah optimum yang diperoleh pada pengujian sebelumnya. Kurva amplifikasi dan kurva puncak pelelehan hasil pengukuran sampel susu UHT spike dengan real-time PCR dapat dilihat pada Gambar 17. dan Gambar 18 berikut ini.
Thre
shold C
y
cle
E=100% R2=0,972 slope=-3,322 y-int=40,564 Log Starting Quantity, fold dilution
Gambar 17. Kurva amplifikasi sampel susu UHT spikeSalmonella Typhimurium metode kit komersial
Gambar 18. Kurva puncak pelelehan sampel susu UHT spike Salmonella Typhimurium metode kit komersial
Kurva amplifikasi tersebut menunjukkan nilai Ct untuk sampel susu UHT spike yang mengandung 3,9 x 104 CFU/ml Salmonella Typhimurium yaitu sebesar 22,58 dan spesifik
Salmonella Typhimurium karena memiliki nilai Tm yang sama dengan kultur murni mikroba spesifik Salmonella Typhimurium yaitu sebesar 84,50. Nilai Ct tersebut dimasukkan ke dalam persamaan garis yang telah diperoleh sebelumnya sehingga nilai log konsentrasi Salmonella Typhimurium yang terkandung di dalam susu dengan real-time PCR dapat diketahui yaitu sebesar 5,4 atau menunjukkan jumlah konsentrasi Salmonella Typhimurium yang terdapat di dalam susu UHT adalah sebesar 105,4 atau setara dengan 2,5 x 105 cetakan DNA/ml.
Hasil dari pengujian real-time PCR tersebut tidak sama dengan hasil dari pengujian metode konvensional dengan menggunakan agar selektif XLDA yaitu sebesar 3,9 x 104 CFU/ml. Hal tersebut juga terjadi pada penelitian yang dilakukan oleh Jung et al. (2005) dimana pengkuantifikasian dengan real-time PCR menghasilkan nilai konsentrasi Salmonella Typhimurium dalam susu satu log lebih besar dibandingkan dengan metode konvensional
22,58
hitungan cawan. Berdasarkan Jung et al. hal yang sama juga terjadi pada penelitian Mayer et al. (2003) dan Hein et al. (2005) dimana hasil tersebut dapat dijelaskan karena beberapa kemungkinan yaitu nilai konsentrasi yang diperoleh dengan metode konvensional hitungan cawan bergantung pada distribusi mikroba dalam sampel, dan mikroba harus hidup dan dapat dikulturkan sedangkan real-time PCR dengan mendeteksi DNA mikroba baik mikroba tersebut hidup ataupun tidak. Selain itu, pengkuantifikasian dengan real-time PCR tergantung pada keadaan fisiologis mikroba dan efisiensi metode dalam mengekstrak/mengisolasi DNA dari dalam sel. Oleh karena itu, perbedaan hasil pengkuantifikasian yang terjadi antara real-time PCR dengan metode konvensional kemungkinan besar karena kultur murni mikroba spesifik yang diinokulasi ke dalam susu berada pada fase stasioner yang banyak mengandung sel yang telah mati dan lisis (Jung et al. 2005) sehingga DNA yang berasal dari sel mati dapat terkuantifikasi oleh real-time PCR.
Tahap pengayaan (enrichment)biasanya dilakukan sebelum pengujian dengan real-time PCR untuk mencegah pengkuantifikasian sel yang sakit dan tidak dapat tumbuh/terhitung dengan metode konvensional, namun tahap pengayaan tersebut hanya dapat menyembuhkan sel mikroba yang rusak dan tidak dapat menghidupkan sel mikroba yang telah lisis/mati. Metode lain yang dapat mencegah pengkuantifikasian sel mati adalah dengan penambahan DNAse sebelum dilakukannya pelisisan sel pada tahap isolasi/ekstraksi DNA, sehingga suspensi yang mengandung DNA di luar sel akibat lisisnya sel yang telah mati akan hancur oleh DNAse (Maurer 2006).