PREDIKSI STRUKTUR SEKUNDER PROTEIN
MENGGUNAKAN HIDDEN MARKOV MODEL
PADA IMBALANCED DATA
DIAN PUSPITA SARI
DEPARTEMEN ILMU KOMPUTER
MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2014
PERNYATAAN MENGENAI SKRIPSI DAN SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Prediksi Struktur Sekunder Protein menggunakan Hidden Markov Model pada Imbalanced Data adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang telah diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2014 Dian Puspita Sari NIM G64100093
iv
ABSTRAK
DIAN PUSPITA SARI. Prediksi Struktur Sekunder Protein menggunakan Hidden Markov Model pada Imbalanced Data. Dibimbing oleh TOTO HARYANTO
Penelitian ini bertujuan untuk memprediksi struktur sekunder protein menggunakan Hidden Markov Model. Data yang digunakan sebanyak 780, dengan 600 data sebagai data latih dan 180 data sebagai data uji. Dari keseluruhan data latih yang digunakan, didapatkan sebanyak 394052 struktur sekunder protein dengan jumlah alpha-helix (H) sebanyak 152782, betha-sheet (B) sebanyak 82355, dan coil (C) sebanyak 158915. Terlihat dari hasil persentase, data yang diperoleh masih imbalanced sehingga dilakukan oversampling untuk menambah jumlah kelas yang terkecil secara acak sampai diperoleh jumlah yang sama dengan kelas yang terbesar. Hasil dari penelitian ini menunjukkan bahwa Hidden Markov Model (HMM) dapat diterapkan untuk memprediksi struktur sekunder protein dengan algoritme Viterbi. Data yang telah di oversampling menghasilkan nilai Q3 score 45.49% untuk data latih dan 43.21% untuk data uji. Adapun untuk data yang tidak dilakukan oversampling menghasilkan nilai Q3 score 43.50% untuk data latih dan 43.19% untuk data uji.
Kata kunci: Hidden Markov Model (HMM), imbalanced data, oversampling, Viterbi
ABSTRACT
DIAN PUSPITA SARI. Protein Secondary Structure Prediction using Hidden Markov Model on Imbalanced Data. Supervised by TOTO HARYANTO.
This research aimed to predict protein secondary structure using Hidden Markov Model. A total of 780 data, will be conducted with 600 training data and 180 testing data. Training data obtained protein secondary structure 394052 with 152782 alpha-helix (H), 82355 betha-sheets (B) , and 158915 coil (C). Seen from a percentage of the result, the data retrieved is still imbalanced therefore used oversampling to increase the smallest class randomly until it equal to the largest class. The result of this research show that the Hidden Markov Model (HMM) can be applied to predict the secondary structure of proteins. The data has been oversampled produced Q3 score 45.49% for training data and 43.21% for testing data. For data that was not done oversampling produced Q3 score 43.50% for training data and 43.19% for testing data.
Key words: Hidden Markov Model (HMM), imbalanced data, oversampling, Viterbi
v
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Komputer
pada
Departemen Ilmu Komputer
PREDIKSI STRUKTUR SEKUNDER PROTEIN
MENGGUNAKAN HIDDEN MARKOV MODEL
PADA IMBALANCED DATA
DIAN PUSPITA SARI
DEPARTEMEN ILMU KOMPUTER
MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2014
vi
Penguji: 1 Dr Ir Agus Buono, MSi MKom
Judul Skripsi : Prediksi Struktur Sekunder Protein menggunakan Hidden Markov Model pada Imbalanced Data
Nama : Dian Puspita Sari NIM : G64100093
Disetujui oleh
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
Tanggal Lulus:
Toto Haryanto, SKom MSi Pembimbing
viii
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan skripsi dengan judul Prediksi Struktur Sekunder Protein menggunakan Hidden Markov Model pada Imbalanced Data.
Terima kasih penulis ucapkan kepada kedua orangtua penulis, kakak-kakak penulis yaitu Muryati, Masudi, dan Wiwit, serta seluruh anggota keluarga atas segala doa dan kasih sayangnya. Bapak Toto Haryanto, SKom MSi selaku pembimbing yang telah banyak memberikan saran, ide, nasehat dan dukungan. Disamping itu, penulis juga mengucapkan terima kasih kepada teman-teman Pixels atas semangat, bantuan dan suka duka dalam kebersamaan.
Bogor, September 2014 Dian Puspita Sari
DAFTAR ISI
DAFTAR TABEL x DAFTAR GAMBAR x DAFTAR LAMPIRAN x PENDAHULUAN 1 Latar Belakang 1 Perumusan Masalah 2 Tujuan Penelitian 2 Manfaat Penelitian 2Ruang Lingkup Penelitian 2
TINJAUAN PUSTAKA 2
Imbalanced Data 5
Strategi Sampling 5
Hidden Markov Model (HMM) 5
Algoritme Viterbi 6
Precision, Recall, Akurasi, dan Q3 Score 7
METODE 7 Studi Pustaka 7 Pengambilan Data 8 Praproses Data 8 Pembagian Data 9 Strategi Sampling 9
Pembuatan Hidden Markov Model 9
Pengujian 10
HASIL DAN PEMBAHASAN 11
Praproses Data 11
Pembuatan Hidden Markov Model 13
Pengujian 14
Simpulan 16
Saran 16
DAFTAR PUSTAKA 16
x
DAFTAR TABEL
1 Codon triplet pembentuk protein (Jones dan Pevzner 2004) 3 2 Asam amino, singkatan, simbol, dan karakteristik (Polanski dan Kimmel
2007) 4
3 Confusion matrix 7
4 Visualisasi dari matriks transisi 10
5 Visualisasi dari matriks emisi 10
6 Visualisasi distribusi sebaran peluang matrik emisi pada keseluruhan data 14 7 Visualisasi distribusi sebaran peluang matrik transisi pada keseluruhan 14
8 Precision dari data uji dan data latih 15
9 Recall dari data uji dan data latih 15
10 Akurasi dari data uji dan data latih 15
11 Hasil Q3 score dari data uji dan data latih 16
DAFTAR GAMBAR
1 Proses pembentukan protein 3
2 Contoh urutan asam amino pembentuk protein 3
3 Metode Penelitian 8
4 Visualisasi format data 9
5 Ilustrasi Pemodelan Prediksi Struktur Sekunder Protein dengan Hidden
Semi Markov Model 9
6 Visualisasi praproses data 11
7 Persentase sebaran struktur sekunder protein pada data latih 12 8 Persentase sebaran struktur sekunder protein pada data uji 12
9 Ilustrasi dari duplikasi betha-sheet (B) 13
10 Persentase sebaran data struktur sekunder protein setelah dilakukan
oversampling 13
DAFTAR LAMPIRAN
1 Data asli struktur sekunder protein dengan format DSSP 18 2 Antar muka prediksi struktur sekunder protein 24
PENDAHULUAN
Latar Belakang
Protein merupakan salah satu biomakromolekul yang mempunyai peran penting dalam makhluk hidup. Secara hierarki protein dibagi menjadi tiga tingkat yaitu, struktur primer, struktur sekunder, dan struktur tersier. Struktur primer adalah urutan asam amino yang membentuk rantai polipeptida. Struktur sekunder adalah sejumlah rangkaian asam amino yang membentuk struktur tiga dimensi alpha-helix (H), betha-sheet (B), maupun coil (C) yang merupakan hasil dari sekuens asam amino yang berikatan dengan ikatan peptida (Atar et al. 2010). Struktur tersier adalah gabungan dari struktur sekunder setelah terjadi pelipatan (folding). Fungsi dari protein dapat diketahui jika sudah membentuk struktur tersier dalam bentuk 3D. Akan tetapi struktur tersier dapat ditentukan apabila struktur sebelumnya sudah diketahui.
Menurut Atar et al. (2010) struktur protein dapat diketahui dengan kristalografi sinar-X dan Nuclear Magnetic Resonance (NMR) spectroscopy. Namun kedua teknik tersebut memakan waktu dan relatif mahal. Sehingga kebanyakan yang menggunakan metode sequencing protein karena relatif lebih mudah digunakan untuk memprediksi struktur sekunder protein. Prediksi struktur sekunder protein dilakukan untuk menemukan struktur 3D protein berdasarkan struktur primer protein. Ada dua metode prediksi struktur sekunder protein, yaitu metode pemodelan komparatif dan pemodelan de novo atau ab initio. Pemodelan protein komparatif memprediksi struktur protein berdasarkan struktur protein lain yang telah diketahui, sedangkan metode ab initio struktur protein ditentukan dari sekuens primernya tanpa membandingkan dengan struktur protein lain (Martin et al. 2005).
Berbagai metode digunakan untuk memprediksi struktur sekunder protein yang berbasis komputasi seperti menggunakan Hidden Markov Model (HMM), Hidden Semi Markov Model (HSMM), BP Neural Network dan Quasi-Newton algorithm, algoritme SOM dan SOGR, dan Neural Network. Menurut Eddy (1998), Hidden Markov Model (HMM) merupakan suatu kelas dari model probabilistik yang secara umum dapat diaplikasikan untuk permasalahan deret sekuens yang bersifat linear.
Penelitian yang dilakukan oleh Martin et al. (2005) untuk memprediksi struktur sekunder protein menggunakan Hidden Markov Model dengan 2024 sekuens yang diambil secara acak dan mendapatkan tingkat akurasi 34.5% untuk data uji dan 58.3% untuk data latih. Akurasi yang didapat masih kecil karena data yang digunakan masih tidak seimbang (imbalanced). Penelitian lain dilakukan oleh He dan Edwardo (2009) yang mengusulkan metode sampling untuk menangani data yang imbalanced. Metode sampling untuk menangani imbalanced data antara lain adalah oversampling dan undersampling. Hidden Markov Model (HMM) merupakan model yang digunakan dalam penelitian ini dengan menggunakan algoritme Viterbi untuk melakukan prediksi struktur sekunder. Data yang digunakan dalam penelitian ini merupakan data yang imbalanced, sehingga dilakukan strategi sampling dengan oversampling untuk mengatasinya.
2
Perumusan Masalah
Pentingnya memprediksi struktur sekunder protein untuk mengetahui fungsi dari protein. Banyak metode yang telah digunakan untuk memprediksi struktur sekunder protein untuk meningkatkan tingkat keakurasian. Metode Hidden Markov Model cocok digunakan karena karakteristik dari sekuens asam amino cocok dengan tipe data yang digunakan dalam pembuatan model. Hidden Markov Model telah banyak digunakan dalam memprediksi struktur sekunder protein. Akan tetapi, tingkat keakurasian masih rendah yang disebabkan kondisi data yang imbalanced.
Tujuan Penelitian
Tujuan penelitian ini adalah menerapkan Hidden Markov Model untuk memprediksi struktur sekunder protein yang akan mengakomodasi imbalanced data.
Manfaat Penelitian
Penelitian ini diharapkan dapat menjadi acuan dalam pengembangan prediksi struktur sekunder protein dan selanjutnya dapat dimanfaatkan oleh berbagai kalangan khususnya di bidang kajian Bioinformatika.
Ruang Lingkup Penelitian
Ruang lingkup penelitian ini adalah sebagai berikut:
1 Pada proses pengujian struktur sekunder protein menggunakan sekuens tunggal.
2 Menggunakan Define Secondary Structure of Protein (DSSP) dan Protein Data Bank (PDB) sebagai pembatas penentuan struktur sekunder protein. 3 Data yang digunakan adalah sekuens asam amino pada enam enzim
berdasarkan enzyme commission.
TINJAUAN PUSTAKA
Struktur Protein
Protein merupakan salah satu biomakromolekul yang mempunyai peran penting dalam mahluk hidup. Proses untuk mendapatkan protein dinamakan translasi. Protein dihasilkan dari proses translasi RNA dan DNA. Proses terbentuknya protein dapat dilihat pada Gambar 1.
3
Gambar 1 Proses pembentukan protein
Satu protein terdiri atas sejumlah sekuens asam amino. Protein dibentuk mulai dari urutan sekuens DNA sampai dengan proses translasi dan mendapatkan suatu protein. Transkripsi merupakan proses pengkopian molekul DNA menjadi RNA. Translasi merupakan proses penerjemahan codon pada RNA menjadi protein. Proses translasi akan dimulai ketika bertemu dengan codon AUG. Codon AUG berfungsi sebagai start codon dan mengkodekan asam amino metionin. Proses translasi akan berhenti apabila bertemu dengan stop codon yaitu UAA, UAG, dan UGA. Pada proses transkripsi kode A (adenin) dari DNA diganti menjadi kode U (urasil) pada RNA, kode G (guanin) dari DNA diganti menjadi kode C (sitosin) pada RNA, kode C (sitosin) dari DNA diganti menjadi kode G (guanin) pada RNA, dan kode T (timin) dari DNA diganti menjadi kode A (adenin) pada RNA (Elrod dan Stansfield 2002). Ilustrasi pembentukan satu protein berdasakan sekuensnya dapat dilihat pada Gambar 2.
Gambar 2 Contoh urutan asam amino pembentuk protein
Protein merupakan elemen dasar yang terbentuk dari asam amino dasar. Terdapat 20 asam amino dengan struktur kimia yang berbeda (Polanski dan Kimmel 2007). Asam amino terbentuk dari tiga huruf (triplet) dari kombinasi Asam Deoksiribosa (DNA) yang disebut dengan codon. Codon triplet pembentuk protein dapat dilihat pada Tabel 1.
Tabel 1 Codon triplet pembentuk protein (Jones dan Pevzner 2004)
U C A G U UUU Phe UUC Phe UUA Leu UUG Leu UCU Ser UCC Ser UCA Ser UCG Ser UAU Tyr UAC Tyr UAA Stop UAG Stop UGU Cys UGC Cys UGA Stop UGG Trp C CUU Leu CUC Leu CUA Leu CUG Leu CCU Pro CCC Pro CCA Pro CCG Pro CAU His CAC His CAA Gln CAG Gln CGU Arg CGC Arg CGA Arg CGG Arg DNA : TAC CAT TGA CAG GAT ACG CCA ATC RNA : AUG GUA ACU GUC CUA UGC GGU UAG PROTEIN : Met Val Thr Val Leu Cys Arg Stop
RNA PROTEIN
DNA
4
Tabel 1 Codon triplet pembentuk protein (Jones dan Pevzner 2004)(lanjutan)
U C A G A AUU Ile AUC Ile AUA Ile AUG Met ACU Thr ACC Thr ACA Thr ACG Thr AAU Asn AAC Asn AAA Lys AAG Lys AGU Ser AGC Ser AGA Arg AGG Arg G GUU Val GUC Val GUA Val GUG Val GCU Ala GCC Ala GCA Ala GCG Ala GAU Asp GAC Asp GAA Glu GAG Glu GGU Gly GGC Gly GGA Gly GGG Gly Terdapat 64 codon yang berbeda, dengan 3 codon yang berfungsi sebagai stop codon. Dari 61 codon yang berbeda terdapat beberapa codon yang memiliki fungsi yang sama. Hal tersebut dapat memberikan keuntungan pada saat proses pembentukan protein, karena dapat menggantikan asam amino yang kemungkinan rusak (Elrod dan Stansfield 2002). Susunan asam amino pembentuk protein dapat dilihat pada Tabel 2.
Tabel 2 Asam amino, singkatan, simbol, dan karakteristik (Polanski dan Kimmel 2007)
Asam amino Singkatan Simbol Karakteristik
Alanine Ala A Nonpolar, hydrophobic
Arginine Arg R Polar, hydrophilic
Asparagine Asn N Polar, hydrophilic
Aspartic acid Asp D Polar, hydrophilic
Cystein Cys C Polar, hydrophilic
Glutamine Gln Q Polar, hydrophilic
Glutamic acid Glu E Polar, hydrophilic
Glycine Gly G Polar, hydrophilic
Histidine His H Polar, hydrophilic
Isoleucine Ile I Nonpolar, hydrophobic
Leucine Leu L Nonpolar, hydrophobic
Lysine Lys K Polar, hydrophilic
Methionine Met M Nonpolar, hydrophobic
Phenylalanine Phe F Nonpolar, hydrophobic
Proline Pro P Nonpolar, hydrophobic
Serine Ser S Polar, hydrophilic
Threonine Thr T Polar, hydrophilic
Tryptophan Trp W Nonpolar, hydrophobic
Tyrosine Tyr Y Nonpolar, hydrophobic
5
Imbalanced Data
Menurut He dan Edwardo (2009) sebuah himpunan data dikatakan imbalanced jika terdapat salah satu kelas yang direpresentasikan dalam jumlah yang tidak sebanding dengan kelas yang lain. Imbalanced data dapat diatasi dengan beberapa cara, antara lain dengan pengambilan sampel pada setiap kelas dan strategi sampling seperti oversampling dan undersampling.
Strategi Sampling
Salah satu teknik yang paling populer untuk mengatasi data yang imbalanced adalah dengan menggunakan strategi sampling. Beberapa teknik sampling antara lain adalah oversampling dan undersampling (He dan Edwardo 2009). Oversampling adalah proses menduplikasi data dari kelas minoritas, sehingga jumlah kelas minoritas mendekati kelas mayoritas. Sedangkan undersampling adalah proses membuang sebagian data dari kelas mayoritas, sehingga jumlah kelas mayoritas mendekati kelas minoritas.
Hidden Markov Model (HMM)
Hidden Markov Model (HMM) merupakan model probabilistik yang dapat diaplikasikan untuk menganalisis model deret waktu atau sekuens linear (Eddy 1998). HMM adalah salah satu pendekatan yang digunakan untuk memodelkan kumpulan sekuens tersebut. HMM telah banyak dikembangkan pada banyak permasalahan seperti speech recognition (Rabiner 1989). Menurut Rabiner (1989), aplikasi pada HMM pada akhirnya akan direduksi untuk menyelesaikan tiga jenis permasalahan, yaitu :
1 Jika diberikan suatu model λ = (A,B,π) , bagaimana menghitung peluang dari sekuens observasi O = O1,O2,...OT yang dinotasikan dengan P(O | λ).
2 Jika diberikan suatu model λ = (A,B,π) , bagaimana memilih state sekuens I = I1,I2,...IT sehingga P(O,I | λ) sebagai peluang bersama dari sekuens observasi O = O1,O2,...OT dan state sekuens tersebut memiliki nilai maksimum.
3 Mendapatkan parameter model HMM yang optimal sehingga peluang suatu observasi memiliki nilai maksimum, dengan
λ adalah model HMM
A adalah Matriks peluang transisi, B adalah Matriks peluang emisi dan
π adalah Matriks peluang awal / Matriks priority O = O1,O2,...OT adalah variabel observasi
P(O | λ) adalah peluang variabel observasi jika diberikan model
Hidden Markov Model (HMM) menggambarkan distribusi peluang dari sejumlah sekuens yang tidak terbatas (Eddy 1998). Nama "Hidden Markov Model" berawal dari fakta bahwasannya state dari sekuens merupakan orde pertama dari rantai Markov sebagai variabel yang tidak teramati. Adapun sekuens dari simbol (seperti A,C,G,T/U) merupakan variabel yang secara langsung dapat diobservasi.
6
Algoritme Viterbi
Algoritme Viterbi digunakan untuk mendapatkan state yang optimal sehingga peluang suatu observasi adalah yang paling maksimal. Untuk menemukan state terbaik, q = (q
1q2...qґ), untuk rangkaian observasi O = (o1
o
2...oґ), perlu didefinisikan kuantitas:
δ
t(i) = Maxq1,q2,..qt-1
P[q
1q2....qt-1, qt = i, o1 o2....ot | λ] (1)
Dengan menginduksi, didapat: δ
t+1(j) = [max δt(i)ij]
. b
j(o1+1 ) (2) Untuk mendapatkan kembali rangkaian state, perlu adanya penyimpanan hasil yang memaksimalkan persamaan (2), untuk tiap i dan j, dengan menggunakan tabel Aґ(j), dilakukan tahap- tahap berikut.
Inisialisasi
δ1 (i) = ᴨibi(oi) 1≤ i ≥N
ψn (1) = 0 Rekursif
δt (i) = max 1≤ i ≥N [δt-1 (i)aij]bj(ot) 2 ≤ t ≤ T , 1 ≤ j ≤ N
ψn(j) = arg max [δt-1 (i)aij] 1≤ i ≥N, 2 ≤ t ≤ T , 1 ≤ j ≤ N Terminasi
P* = max 1≤ i ≥N [δT(i)]
δT* = arg max 1≤ i ≥N [δT(i)]
dengan : δ
t(i) = rangkaian terbaik dengan kemungkinan terbesar
t = waktu perhitungan pengamatan t pertama dan berakhir pada status i. q = state
o = observasi
ψ = path terbaik pada saat sampai state ke i P = peluang
b = matriks emisi a = matriks transisi
7
Precision, Recall, Akurasi, dan Q3 Score
Pengukuran kemampuan algoritme dilakukan dengan confusion matrix yang dapat dilihat pada Tabel 3. Confusion matrix digunakan sebagai dasar dari variasi ukuran penilaian seperti precision, recall, dan akurasi karena mengandung informasi tentang data kelas aktual dan hasil prediksi.
Precision merupakan proporsi dari kelas data positif yang berhasil diprediksi dengan benar dari keseluruhan hasil prediksi kelas data positif. Recall merupakan persentase kelas data positif yang berhasil diprediksi dengan benar dari keseluruhan instance kelas positif. Akurasi merupakan jumlah dari proporsi dari kelas data positif yang berhasil diprediksi dengan benar dan proporsi dari kelas data negatif yang berhasil diprediksi dengan benar dari keseluruhan kelas data positif dan negatif. Q3 score merupakan jumlah dari proporsi dari kelas data positif yang berhasil diprediksi dengan benar dari keseluruhan data.
Tabel 3 Confusion matrix Kelas hasil prediksi Kelas aktual
Kelas positif Kelas negatif
Kelas positif TP FP
Kelas negatif FN TN
dengan :
TP = jumlah instance kelas positif yang diprediksi benar sebagai kelas positif FP = jumlah instance kelas negatif yang diprediksi salah sebagai kelas positif FN = jumlah instance kelas positif yang diprediksi salah sebagai kelas negatif TN = jumlah instance kelas negatif yang diprediksi benar sebagai kelas negatif
METODE
Metode penelitian yang digunakan dapat dilihat pada Gambar 3. Tahapan penelitian ini meliputi studi pustaka, pengambilan data, praproses data, strategi sampling dengan oversampling, model HMM dan pengujian.
Studi Pustaka
Studi pustaka dilakukan untuk mencari riset-riset yang pernah dilakukan yang terkait dengan bidang penelitian yang akan dilakukan. Dari riset yang telah dilakukan, penggunaan Hidden Markov Model (HMM) telah banyak digunakan untuk memprediksi struktur sekunder protein. Akan tetapi, masih belum banyak yang dapat menangani data yang imbalanced.
8
Pengambilan Data
Data yang diambil adalah data sekuens protein sekunder dari alamat website ftp://ftp.cmbi.ru.nl/pub/molbio/data/dssp/ yang merupakan database assignment struktur sekunder protein. Data protein yang diambil merupakan data semua protein yang ada di Protein Data Bank (PDB). Data yang diperoleh masih dalam format dengan ekstensi .dssp, oleh karena itu dilakukan proses parsing sebelum data tersebut digunakan sebagai data latih dan data uji. Hasil dari proses parsing adalah pasangan asam amino dan assigment struktur sekunder protein.
Gambar 3 Metode Penelitian
Praproses Data
Data struktur sekunder protein yang diperoleh masih dalam format dengan ekstensi .dssp dan tersegmentasi menjadi 8 struktur sehingga dilakukan praproses. Setiap satu file yang berekstensi .dssp akan diambil pasangan sekuens asam amino
Mulai Pengambilan data Pelatihan Praproses data Strategi sampling Oversampling Data latih Model HMM Pengujian Data uji Selesai Studi pustaka
9 dan struktur sekunder protein. Struktur yang kosong akan diganti dengan coil (C), dan segmen direduksi menjadi tiga, yaitu alpha-helix (H), betha-sheet (B) dan coil (C) (Wang dan Ping Li 2006). Segmen hasil reduksi adalah {I,H,G} menjadi alpha-helix (H), {E,B}menjadi betha-sheet (B), segmen {S,T,C} menjadi coil (C). Format data .dssp dapat dilihat pada Gambar 4.
Gambar 4 Visualisasi format data
Pembagian Data
Data dibagi menjadi dua, yaitu data latih dan data uji. Data yang digunakan sebagai data latih merupakan 77% dari data keseluruhan, dan 23% digunakan sebagai data uji.
Strategi Sampling
Penelitian ini menggunakan strategi sampling, karena data pada ketiga kelas imbalanced. Strategi sampling yang digunaka adalah oversampling. Pada strategi oversampling jumlah instance pada data minoritas ditambah sehingga jumlahnya mendekati data mayoritas. Strategi ini dilakukan dengan cara menduplikasi sebanyak n kali secara acak data dari kelas minoritas. Pada strategi oversampling diperoleh 1 set data hasil dari duplikasi.
Pembuatan Hidden Markov Model
Pada tahap ini dilakukan proses pembentukan model dari data latih dengan menggunakan Hidden Markov Model (HMM). Ilustrasi pemodelan prediksi struktur sekunder protein dapat dilihat pada Gambar 5.
Gambar 5 Ilustrasi Pemodelan Prediksi Struktur Sekunder Protein dengan Hidden Semi Markov Model (Martin et al. 2005)
Baris H-C menunjukkan model hidden state yang merepresentasikan alpha-helix (H), betha-sheet (B), dan coil (C). Barisan di bawah tanda panah merupakan barisan sekuens asam amino, yang merupakan sekuens observasi.
10
Hasil proses dari pemodelan adalah matriks transisi dan matriks emisi yang memiliki nilai tertentu yang dijadikan model dalam proses prediksi. Visualisasi matriks transisi dan emisi dapat dilihat pada Tabel 4 dan Tabel 5.
Tabel 4 Visualisasi dari matriks transisi
H B C
H B C
Matriks transisi merupakan matriks yang merepresentasikan kombinasi dari alpha-helix (H), betha-sheet (B), dan coil (C). Matriks transisi akan digunakan pada tahap pertama model markov. Matriks emisi merupakan matriks yang merepresentasikan kombinasi dari pasangan asam amino dengan struktur sekunder protein.
Tabel 5 Visualisasi dari matriks emisi
A R N D C Q E G H I L K M F P S T W Y V H
B C
Pengujian
Pada tahap pengujian dilakukan perhitungan precision, recall, akurasi, dan Q3 score. Persamaan dari precision, recall, akurasi, dan Q3 score score dapat dilihat pada persamaan 3, persamaan 4, persamaan 5, dan persamaan 6.
dengan :
Q3 score = Tingkat akurasi
Nh = Jumlah dari residu yang di prediksi benar pada alpha-helix (H Nb = Jumlah dari residu yang di prediksi benar pada betha-sheet (B) Nc = Jumlah dari residu yang di prediksi benar pada coil (C)
Ntot = Jumlah total dari residu yang diujikan TP TP FP 1 TP FNTP 1 TP TN N 1 Q N N N N 1 (3) (4) (5) (6)
11
HASIL DAN PEMBAHASAN
Praproses Data
Data struktur sekunder protein yang diperoleh dari database masih dalam ekstensi .dssp sehingga perlu dilakukan praproses data terlebih dahulu. Praproses data dilakukan agar memudahkan proses komputasi pada tahap berikutnya. Setiap file yang berekstensi .dssp dari semua kategori protein akan diambil asam amino dan struktur sekundernya. Kolom yang diambil sebagai pasangan data asam amino dan struktur sekunder adalah kolom ketiga dan kolom keempat yaitu {AA} yang merupakan asam amino dan {STRUCTURE} yang merupakan struktur sekunder. Visualisasi praproses data dapat dilihat pada Gambar 6. Hasil dari praproses data adalah pasangan sekuens asam amino dan struktur sekunder protein dari setiap residu asam amino. Setelah didapat struktur sekunder protein, dihitung distribusi peluang dari setiap residu asam amino, yang nantinya akan digunakan untuk perhitungan tahap selanjutnya. Format lengkap data struktur sekunder protein dapat dilihat pada Lampiran 1.
Gambar 6 Visualisasi praproses data
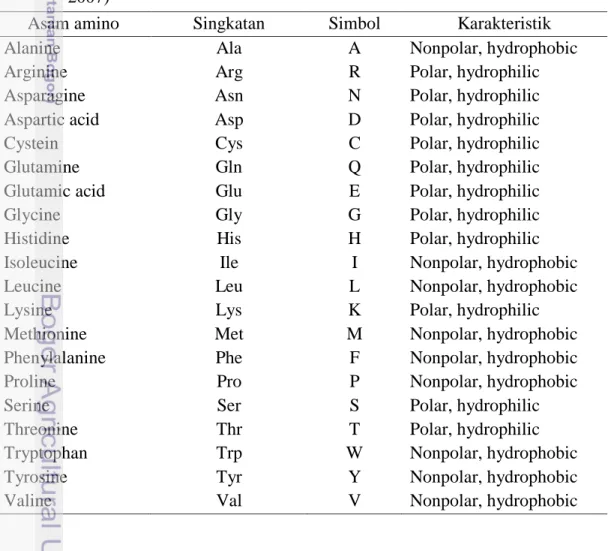
Data yang digunakan dalam penelitian ini berjumlah 780. Dengan 6 kategori berdasarkan enzyme commission yaitu, hydrolases, transferases, oxidoredutases, lyases, isomerase, dan ligases. Setiap kategori diambil 130 data sebagai sampel. Sebanyak 600 data digunakan sebagai data latih dan 180 data sebagai data uji. Dari keseluruhan data latih yang digunakan, didapatkan sebanyak 394052 struktur sekunder protein. Dengan jumlah alpha-helix (H) sebanyak 152782, betha-sheet (B) sebanyak 82355, dan coil (C) sebanyak 158915. Persentase dari sebaran data latih dapat dilihat pada Gambar 7. Terlihat dari hasil persentase, data yang diperoleh masih imbalanced sehingga dilakukan oversampling untuk menambah jumlah kelas yang terkecil secara acak sampai diperoleh jumlah yang sama dengan kelas yang terbesar.
12
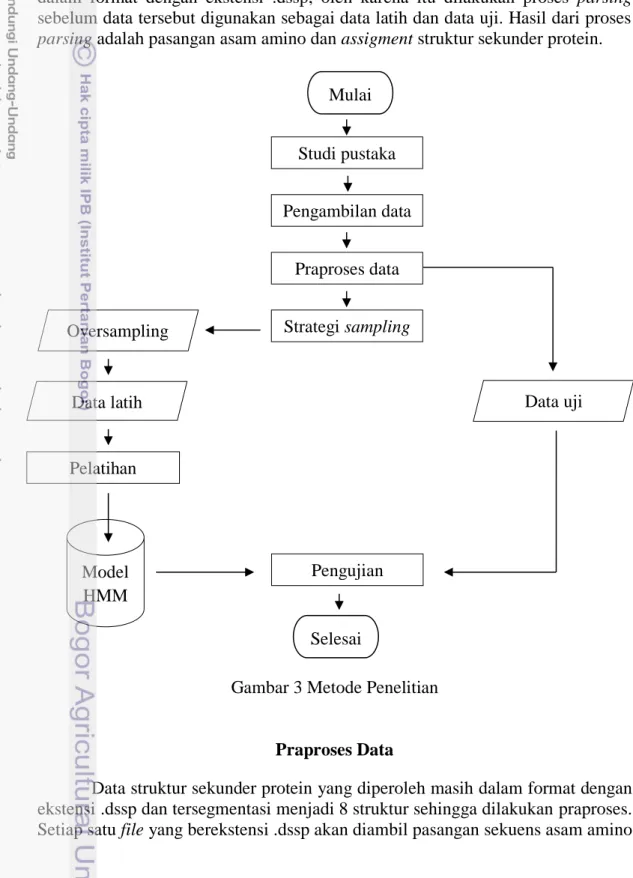
Gambar 7 Persentase sebaran struktur sekunder protein pada data latih Dari keseluruhan data uji yang digunakan, didapatkan sebanyak 115645 struktur sekunder protein dengan jumlah alpha-helix (H) sebanyak 44543, betha-sheet (B) sebanyak 20716, dan coil (C) sebanyak 50386. Persentase dari sebaran data uji dapat dilihat pada Gambar 8.
Gambar 8 Persentase sebaran struktur sekunder protein pada data uji
Strategi Sampling
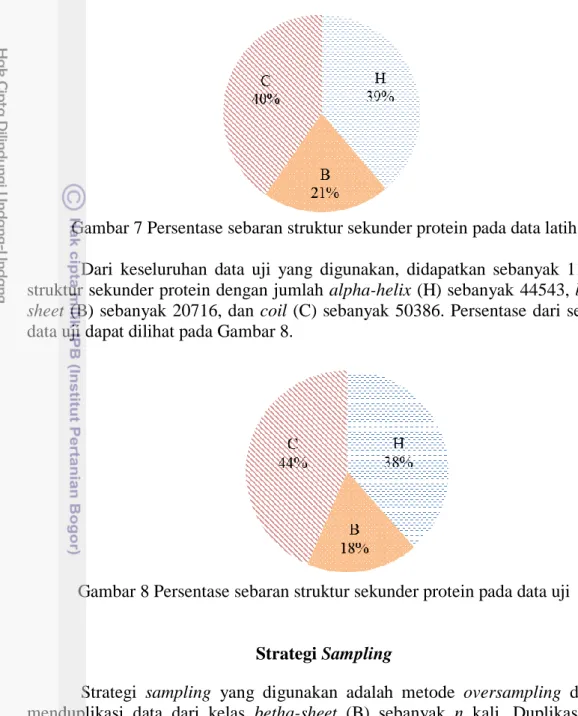
Strategi sampling yang digunakan adalah metode oversampling dengan menduplikasi data dari kelas betha-sheet (B) sebanyak n kali. Duplikasi data ditentukan dari panjang segmen betha-sheet (B). Dari setiap file data latih disetiap data dipilih segmen betha-sheet (B) yang terpanjang. Kemudian diduplikasi sebanyak n kali dengan ketentuan terdapat minimal 100 residu betha-sheet (B) hasil duplikasi disetiap data, agar jumlah dari betha-sheet (B) dapat mendekati jumlah dari alpha helix (H) dan coil (C). Hasil dari duplikasi tersebut diletakkan di barisan paling bawah pasangan asam amino dengan strukturnya. Ilustrasi dari duplikasi betha-sheet (B) dapat dilihat pada Gambar 9.
13
Gambar 9 Ilustrasi dari duplikasi betha-sheet (B)
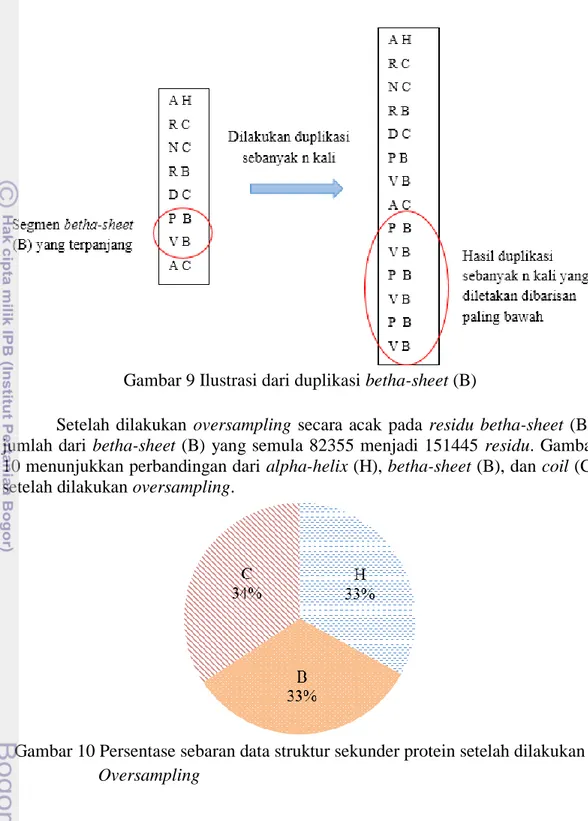
Setelah dilakukan oversampling secara acak pada residu betha-sheet (B), jumlah dari betha-sheet (B) yang semula 82355 menjadi 151445 residu. Gambar 10 menunjukkan perbandingan dari alpha-helix (H), betha-sheet (B), dan coil (C) setelah dilakukan oversampling.
Gambar 10 Persentase sebaran data struktur sekunder protein setelah dilakukan Oversampling
Pembuatan Hidden Markov Model
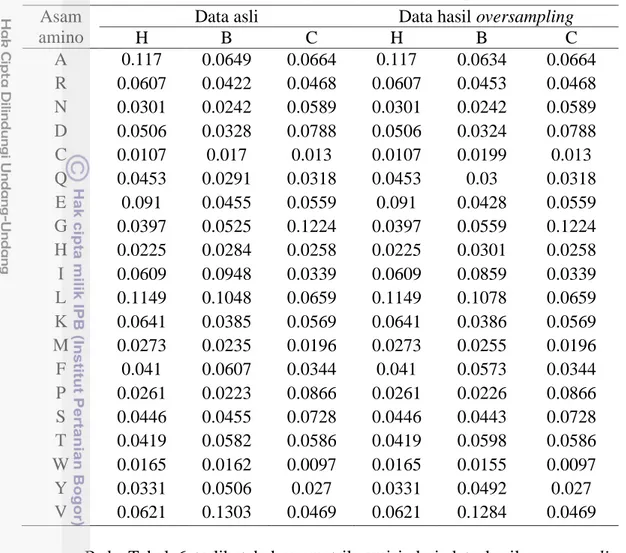
Pembuatan Hidden Markov Model dilakukan setelah didapat pasangan data asam amino dan strukturnya. Pada tahap ini setiap pasangan asam amino dan struktur protein akan direpresentasikan sebagai matrik emisi. Kombinasi dari struktur sekunder protein akan direpresentasikan sebagai matriks transisi. Pada tahapan ini dibuat matrik transisi dan matrik emisi untuk data asli dan data hasil oversampling. Visualisasi distribusi sebaran peluang dari matrik emisi dan transisi pada keseluruhan data dapat dilihat pada Tabel 6 dan Tabel 7.
14
Tabel 6 Visualisasi distribusi sebaran peluang matrik emisi pada keseluruhan data Asam
amino
Data asli Data hasil oversampling
H B C H B C A 0.117 0.0649 0.0664 0.117 0.0634 0.0664 R 0.0607 0.0422 0.0468 0.0607 0.0453 0.0468 N 0.0301 0.0242 0.0589 0.0301 0.0242 0.0589 D 0.0506 0.0328 0.0788 0.0506 0.0324 0.0788 C 0.0107 0.017 0.013 0.0107 0.0199 0.013 Q 0.0453 0.0291 0.0318 0.0453 0.03 0.0318 E 0.091 0.0455 0.0559 0.091 0.0428 0.0559 G 0.0397 0.0525 0.1224 0.0397 0.0559 0.1224 H 0.0225 0.0284 0.0258 0.0225 0.0301 0.0258 I 0.0609 0.0948 0.0339 0.0609 0.0859 0.0339 L 0.1149 0.1048 0.0659 0.1149 0.1078 0.0659 K 0.0641 0.0385 0.0569 0.0641 0.0386 0.0569 M 0.0273 0.0235 0.0196 0.0273 0.0255 0.0196 F 0.041 0.0607 0.0344 0.041 0.0573 0.0344 P 0.0261 0.0223 0.0866 0.0261 0.0226 0.0866 S 0.0446 0.0455 0.0728 0.0446 0.0443 0.0728 T 0.0419 0.0582 0.0586 0.0419 0.0598 0.0586 W 0.0165 0.0162 0.0097 0.0165 0.0155 0.0097 Y 0.0331 0.0506 0.027 0.0331 0.0492 0.027 V 0.0621 0.1303 0.0469 0.0621 0.1284 0.0469
Pada Tabel 6 terlihat bahwa matrik emisi dari data hasil oversampling dengan data asli tidak jauh berbeda. Hasil yang diperoleh untuk matrik emisi tidak jauh berbeda karena pada tahap sampling dengan oversampling tidak memperhitungkan hubungan antar pasangan sekuens. Yang diperhitungkan hanya panjang dari struktur betha-sheet (B).
Tabel 7 Visualisasi distribusi sebaran peluang matrik transisi pada keseluruhan data
Struktur sekunder protein
Data asli (%) Data hasil oversampling (%)
H B C H B C
H 0.8958 0.008 0.0962 0.8958 0.008 0.0962 B 0.0095 0.7646 0.2259 0.0052 0.8665 0.1284 C 0.0949 0.1148 0.7903 0.0945 0.116 0.7895
Pengujian
Setelah didapatkan Hidden Markov Model maka dilanjutkan dengan tahapan pengujian. Tahapan pengujian dilakukan untuk mendapatkan nilai
15 precision, recall, akurasi, dan Q3 score dari model yang diperoleh. Tahapan pengujian yang pertama dilakukan untuk data yang masih imbalanced. Untuk data latih yag diujikan didapat Q3 score sebesar 43.50 %, sedangkan untuk data uji didapat Q3 score sebesar 43.19%. Tahapan pengujian yang kedua dilakukan untuk data yang sudah disampling dan mendapatkan Q3 score sebesar 45.49% untuk data latih, 43.21% untuk data uji. Hasil dari pengujian tahapan pertama dan kedua dapat dilihat pada Tabel 11.
Tabel 8 Precision dari data uji dan data latih
Data asli (%) Data hasil oversampling (%)
H B C H B C
Data latih 41.36 62.08 63.70 39.05 77.77 57.89 Data uji 40.79 26.69 44.74 41.34 30.41 44.82
Dari tabel 8 dapat dilihat bahwa nilai precision setelah dan sebelum dilakukan oversampling tidak berubah secara signifikan. Nilai presisi yang didapat relatif masih kecil, hal itu menunjukkan bahwa masih besarnya kesalahan prediksi.
Tabel 9 Recall dari data uji dan data latih
Data asli (%) Data hasil oversampling (%)
H B C H B C
Data latih 96.66 0.74 14.75 93.31 28.73 15.82 Data uji 40.54 0.42 63.11 39.37 3.98 62.73
Berdasarkan tabel 9 hasil recall yang diperoleh untuk betha-sheet dan coil relatif kecil dibanding alpha-helix. Nilai tersebut menunjukkan bahwa data yang dikelaskan dengan benar relatif kecil.
Tabel 10 Akurasi dari data uji dan data latih
Data asli (%) Data hasil oversampling (%)
H B C H B C
Data latih 45.70 79.16 62.14 49.95 74.04 67.00 Data uji 54.43 81.98 49.97 55.13 81.17 50.12
Dari tabel 10 terlihat bahwa akurasi dari alpha-helix (H), dan coil (C) meningkat sedangkan akurasi dari betha-sheet (B) turun. Walaupun akurasi dari betha-sheet (B) menurun setelah disampling, tidak berarti bahwa teknik oversampling memberikan hasil yang kurang baik, karena jika dilihat dari precision dan recall hasil betha-sheet (B) mengalami kenaikan setelah dioversampling. Selain itu prior juga berpengaruh terhadap hasil akurasi yang didapat, karena nilai dari prior memberikan peluang awal untuk menentukan hasil prediksi.
16
Tabel 11 Hasil Q3 score dari data uji dan data latih Data Asli (%) Data hasil oversampling (%)
Data latih 43.50 45.49
Data uji 43.19 43.21
Dari hasil Q3 score yang diperoleh terlihat bahwa hasil data setelah dilakukan oversampling memiliki persentase yang lebih baik dibanding data asli. Terlihat secara keseluruhan untuk presisi, recall, dan akurasi pada alpha-helix, coil, terutama pada betha-sheet memberikan hasil yang lebih baik setelah data disampling.
SIMPULAN DAN SARAN
Simpulan
Hidden Markov Model (HMM) dapat diterapkan untuk untuk memprediksi struktur sekunder protein. Identifikasi struktur sekunder protein dengan menggunakan Hidden Markov Model (HMM) dengan data yang telah dilakukan sampling memberikan nilai Q3 score lebih baik dibandingkan dengan data yang tidak disampling. Hasil Q3 score pada data yang disampling masih rendah karena metode sampling yang digunakan adalah metode oversampling secara acak, sehingga tidak memperhitungkan hubungan kemunculan antar pasangan sekuens.
Saran
Pada penelitian selanjutnya dapat dikembangkan lebih lanjut untuk mendapatkan hasil dari precision, recall, akurasi, dan Q3 score yang lebih baik. Hal-hal yang dapat dilakukan diantaranya adalah dengan menggunakan strategi sampling yang lain seperti SMOTE untuk mengatasi imbalanced data dengan memperhitungkan hubungan kemunculan antar pasangan sekuens atau menggunakan model lain seperti BP Neural Network dan Quasi-Newton algorithm.
DAFTAR PUSTAKA
Atar E, Ersoy O, Ozyilmaz L. 2005. Prediction of protein secondary structure by SOM and SOGR algorithm. IEE. doi : 10.1109/CIMA.2005.1662358. Baldi P, Brunak S. 2001. Bioinformatics: The Machine Learning Approach.
Second Edition.Massachusetts. England (GB): MIT Press.
Eddy SR. 1998. Profile hidden markov model. Bioinformatics Review.14:755-763. Elrod S, Starnsfield W. 2002. S haum’s Outlin f Th y and P bl ms f
17 He H, Edwardo AG. 2009. Learning from imbalanced data. IEEE Transactions on
Knowledge and Data Engineering. 21(9):1263-1284.
Jones NC, Pevzner PA. 2004. An Introductions to Bioinformatics Algorithms. England (GB): MIT Press.
Martin J, Gibrat JF, Rodolphe J. 2005. Hidden markov model for protein secondary structure. Oxford University Press. 14(9): 755-763.
Polanski A, Kimmel M.2007. Bioinformatics. Germany (DE): Springer Sciene. Rabiner LR. 1989. A Tutorial on hidden markov model and selected applications
in speech recognitions. Proceedings of the IEEE. 77 (2), 257-286. Wang J, Ping Li J. 2008. Protein secondary structure prediction based on BP
neural network and quasi-newton algorithm. IEE. doi : 10.1109/CACIA.20084769988
18
Lampiran 1 Data asli struktur sekunder protein dengan format DSSP
==== Secondary Structure Definition by the program DSSP, CMBI version by M.L. Hekkelman/2010-10-21 ==== DATE=2014-03-26 . REFERENCE W. KABSCH AND C.SANDER, BIOPOLYMERS 22 (1983) 2577-2637 . HEADER OXIDOREDUCTASE 08-SEP-13 2MDA . COMPND MOL_ID: 1; MOLECULE: TYROSINE 3-MONOOXYGENASE; CHAIN: A, B; FRAGMENT: . SOURCE MOL_ID: 1; ORGANISM_SCIENTIFIC: RATTUS NORVEGICUS; ORGANISM_COMMON: BR . AUTHOR S.ZHANG,T.HUANG,A.HINCK,P.FITZPATRICK . 190 2 0 0 0 TOTAL NUMBER OF RESIDUES, NUMBER OF CHAINS, NUMBER OF SS-BRIDGES(TOTAL,INTRACHAIN,INTERCHAIN) . 11786.2 ACCESSIBLE SURFACE OF PROTEIN (ANGSTROM**2) . 108 56.8 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(J) , SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS IN PARALLEL BRIDGES, SAME NUMBER PER 100 RESIDUES . 40 21.1 TOTAL NUMBER OF HYDROGEN BONDS IN ANTIPARALLEL BRIDGES, SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-5), SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-4), SAME NUMBER PER 100 RESIDUES . 4 2.1 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-3), SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-2), SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I-1), SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+0), SAME NUMBER PER 100 RESIDUES . 0 0.0 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+1), SAME NUMBER PER 100 RESIDUES . 14 7.4 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+2), SAME NUMBER PER 100 RESIDUES . 20 10.5 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+3), SAME NUMBER PER 100 RESIDUES . 30 15.8 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+4), SAME NUMBER PER 100 RESIDUES . 2 1.1 TOTAL NUMBER OF HYDROGEN BONDS OF TYPE O(I)-->H-N(I+5), SAME NUMBER PER 100 RESIDUES . 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 *** HISTOGRAMS OF *** . 0 0 0 0 0 0 0 0 2 2 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 RESIDUES PER ALPHA HELIX . 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 PARALLEL BRIDGES PER LADDER . 2 0 3 2 2 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 ANTIPARALLEL BRIDGES PER LADDER . 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 LADDERS PER SHEET .
# RESIDUE AA STRUCTURE BP1 BP2 ACC N-H-->O O-->H-N N-H-->O O-->H-N TCO KAPPA ALPHA PHI PSI X-CA Y-CA Z-CA 1 65 A P 0 0 174 0, 0.0 2,-0.4 0, 0.0 0, 0.0 0.000 360.0 360.0 360.0 167.3 -23.1 -15.6 -7.7 2 66 A G - 0 0 71 4,-0.0 0, 0.0 0, 0.0 0, 0.0 -0.888 360.0-166.3-132.1 104.1 -24.4 -13.0 -5.3 3 67 A N > - 0 0 104 -2,-0.4 3,-0.9 1,-0.2 0, 0.0 -0.790 3.3-172.4 -92.2 111.2 -24.3 -13.6 -1.6 4 68 A P T 3 S+ 0 0 115 0, 0.0 -1,-0.2 0, 0.0 0, 0.0 0.903 88.6 48.9 -66.0 -43.9 -26.5 -11.0 0.3 5 69 A L T 3 S+ 0 0 162 2,-0.0 -2,-0.1 0, 0.0 0, 0.0 0.224 90.3 129.2 -81.9 14.5 -25.3 -12.2 3.7 6 70 A E S < S- 0 0 85 -3,-0.9 87,-0.0 2,-0.1 -4,-0.0 -0.095 70.8-115.7 -65.5 169.2 -21.8 -11.9 2.5
19 7 71 A A S S+ 0 0 46 2,-0.1 2,-0.8 87,-0.0 -1,-0.1 0.749 84.7 101.9 -79.3 -27.5 -19.0 -10.0 4.3 8 72 A V + 0 0 58 84,-0.2 2,-0.2 85,-0.1 -2,-0.1 -0.475 52.1 130.7 -67.1 105.2 -18.5 -7.4 1.6 9 73 A V - 0 0 93 -2,-0.8 11,-0.3 11,-0.2 2,-0.3 -0.775 32.7-163.5-141.5-175.7 -20.3 -4.3 2.9 10 74 A F - 0 0 104 -2,-0.2 2,-0.3 9,-0.1 9,-0.2 -0.972 7.8-141.8-164.6 172.3 -19.7 -0.6 3.5 11 75 A E E -A 18 0A 119 7,-0.9 7,-1.1 -2,-0.3 2,-0.4 -0.896 18.1-121.9-139.1 168.3 -21.0 2.5 5.2 12 76 A E E -A 17 0A 115 5,-0.3 2,-0.4 -2,-0.3 5,-0.3 -0.955 18.7-171.6-124.2 138.7 -21.4 6.2 4.4 13 77 A R E > S-A 16 0A 125 3,-2.5 3,-1.0 -2,-0.4 62,-0.1 -0.989 70.4 -19.7-128.1 129.3 -20.0 9.2 6.2 14 78 A D T 3 S- 0 0 161 -2,-0.4 3,-0.1 1,-0.3 -1,-0.1 0.697 134.3 -41.1 50.4 22.2 -20.8 12.9 5.5 15 79 A G T 3 S+ 0 0 57 1,-0.5 60,-0.5 58,-0.1 -1,-0.3 0.706 128.4 76.1 99.7 22.8 -22.1 11.9 2.1 16 80 A N E < S-AB 13 74A 13 -3,-1.0 -3,-2.5 58,-0.3 2,-0.5 -0.871 79.7-100.0-148.7 178.2 -19.5 9.4 1.1 17 81 A A E -AB 12 73A 0 56,-2.8 56,-2.3 -5,-0.3 2,-0.5 -0.942 23.2-167.1-120.2 127.4 -18.6 5.8 1.9 18 82 A V E +AB 11 72A 8 -7,-1.1 -7,-0.9 -2,-0.5 2,-0.3 -0.917 31.2 130.4-108.5 122.9 -16.0 4.7 4.3 19 83 A L E - B 0 71A 11 52,-2.4 52,-2.3 -2,-0.5 2,-0.3 -0.897 43.3-117.4-156.1-177.2 -15.1 1.0 4.2 20 84 A N E - B 0 70A 29 -11,-0.3 75,-0.5 -2,-0.3 2,-0.4 -0.950 17.3-165.2-134.1 153.9 -12.2 -1.6 4.0 21 85 A L B -H 94 0B 16 48,-1.6 2,-0.3 73,-0.3 48,-0.3 -0.972 0.5-167.8-146.6 126.8 -11.2 -4.3 1.5 22 86 A L + 0 0 42 71,-2.6 71,-0.3 -2,-0.4 2,-0.3 -0.791 11.4 168.2-110.9 155.8 -8.9 -7.2 1.7 23 87 A F - 0 0 27 44,-0.5 2,-0.3 -2,-0.3 69,-0.1 -0.927 12.6-160.7-165.2 141.4 -7.5 -9.4 -1.1 24 88 A S - 0 0 45 -2,-0.3 2,-0.3 42,-0.1 67,-0.1 -0.911 1.9-165.9-128.9 154.3 -4.8 -12.0 -1.5 25 89 A L - 0 0 22 -2,-0.3 41,-0.1 41,-0.1 40,-0.1 -0.998 10.2-143.9-144.0 136.1 -3.0 -13.5 -4.5 26 90 A R + 0 0 225 -2,-0.3 38,-0.0 39,-0.1 0, 0.0 0.050 62.9 44.9 -80.9-165.4 -0.8 -16.6 -4.8 27 91 A G S S- 0 0 59 1,-0.1 -2,-0.1 2,-0.1 0, 0.0 -0.186 87.0 -98.1 64.7-159.0 2.3 -17.1 -7.0 28 92 A T S S+ 0 0 68 110,-0.0 112,-0.2 0, 0.0 -1,-0.1 0.479 113.2 47.2-133.1 -21.7 5.0 -14.4 -7.1 29 93 A K S S+ 0 0 143 1,-0.2 2,-2.2 110,-0.1 -2,-0.1 0.882 91.4 77.5 -90.6 -47.9 4.2 -12.5 -10.2 30 94 A P + 0 0 69 0, 0.0 -1,-0.2 0, 0.0 0, 0.0 -0.408 55.9 120.7 -67.7 79.4 0.4 -11.9 -9.8 31 95 A S > + 0 0 20 -2,-2.2 2,-2.2 -3,-0.1 3,-0.6 -0.221 19.1 148.4-133.9 39.1 0.7 -9.1 -7.3 32 96 A S T > + 0 0 66 1,-0.2 3,-0.6 2,-0.1 4,-0.5 -0.132 22.4 129.6 -71.0 43.3 -1.1 -6.4 -9.2 33 97 A L T >> + 0 0 16 -2,-2.2 4,-1.1 1,-0.2 3,-0.6 0.717 61.4 74.0 -69.0 -19.8 -2.3 -4.9 -5.9 34 98 A S H X> S+ 0 0 10 -3,-0.6 4,-1.1 1,-0.3 3,-0.7 0.915 88.2 55.5 -56.9 -47.2 -0.9 -1.7 -7.4 35 99 A R H <4 S+ 0 0 156 -3,-0.6 4,-0.5 1,-0.3 -1,-0.3 0.773 100.6 63.3 -59.1 -23.4 -3.9 -1.5 -9.8 36 100 A A H X> S+ 0 0 1 -3,-0.6 3,-1.2 -4,-0.5 4,-1.0 0.898 99.8 49.2 -68.2 -41.8 -6.0 -1.6 -6.6 37 101 A V H <X S+ 0 0 11 -4,-1.1 4,-1.3 -3,-0.7 -1,-0.2 0.741 100.8 66.8 -68.9 -21.0 -4.6 1.7 -5.3 38 102 A K H 3X S+ 0 0 62 -4,-1.1 4,-1.0 2,-0.2 -1,-0.3 0.730 97.1 58.0 -68.8 -21.3 -5.4 3.1 -8.8 39 103 A V H X> S+ 0 0 0 -3,-1.2 4,-1.2 -4,-0.5 3,-1.0 0.992 107.3 39.1 -71.7 -67.3 -9.0 2.5 -7.8 40 104 A F H 3<>S+ 0 0 0 -4,-1.0 5,-2.1 1,-0.3 -2,-0.2 0.774 121.4 49.4 -55.4 -26.9 -9.4 4.6 -4.7 41 105 A E H ><5S+ 0 0 25 -4,-1.3 3,-1.1 3,-0.2 -1,-0.3 0.713 97.9 67.3 -86.1 -23.2 -7.1 7.2 -6.4 42 106 A T H <<5S+ 0 0 80 -4,-1.0 -2,-0.2 -3,-1.0 -1,-0.2 0.888 107.6 37.8 -66.9 -39.6 -9.1 7.3 -9.7
20 43 107 A F T 3<5S- 0 0 80 -4,-1.2 -1,-0.3 34,-0.1 -2,-0.1 0.118 115.4-112.7 -98.5 22.0 -12.1 8.9 -8.1 44 108 A E T < 5 + 0 0 104 -3,-1.1 -3,-0.2 80,-0.2 -2,-0.1 0.858 57.3 171.6 52.1 40.5 -10.0 11.1 -5.9 45 109 A A < - 0 0 10 -5,-2.1 2,-0.7 -6,-0.2 28,-0.2 -0.159 37.9-108.1 -71.7 173.6 -11.3 9.2 -2.8 46 110 A K E -C 72 0A 84 26,-2.7 26,-1.7 103,-0.0 2,-0.4 -0.890 30.6-149.6-110.9 104.5 -9.8 9.8 0.7 47 111 A I E -C 71 0A 6 -2,-0.7 24,-0.3 24,-0.3 3,-0.1 -0.566 14.4-174.3 -78.3 128.6 -7.8 6.8 1.8 48 112 A H E S- 0 0A 60 22,-1.7 23,-0.2 -2,-0.4 2,-0.2 0.915 74.6 -11.7 -84.3 -52.2 -7.7 6.1 5.5 49 113 A H E -C 70 0A 41 21,-2.3 21,-0.8 2,-0.0 2,-0.5 -0.657 61.4-176.2-159.5 96.3 -5.1 3.3 5.7 50 114 A L E +CD 69 148A 6 98,-2.1 98,-2.3 19,-0.3 2,-0.3 -0.821 21.5 161.9 -94.2 126.8 -3.8 1.4 2.7 51 115 A E E - D 0 147A 23 17,-2.5 2,-0.3 -2,-0.5 96,-0.2 -0.996 23.0-169.6-148.6 152.8 -1.5 -1.5 3.5 52 116 A T E + D 0 146A 9 94,-2.3 94,-2.1 -2,-0.3 15,-0.2 -0.991 16.6 153.8-143.8 131.6 -0.0 -4.6 2.1 53 117 A R - 0 0 93 13,-0.9 91,-0.1 -2,-0.3 89,-0.0 -0.617 33.6-136.4-161.9 95.3 2.0 -7.4 3.8 54 118 A P - 0 0 29 0, 0.0 3,-0.2 0, 0.0 11,-0.2 -0.152 24.9-125.2 -52.5 143.9 2.1 -11.0 2.5 55 119 A A S S+ 0 0 5 1,-0.2 2,-2.6 2,-0.1 5,-0.1 0.117 84.6 58.0 -74.2-163.3 1.7 -13.7 5.1 56 120 A Q S S+ 0 0 104 3,-1.4 -1,-0.2 1,-0.2 9,-0.0 -0.299 91.6 79.2 76.1 -55.2 4.1 -16.5 5.7 57 121 A R S S+ 0 0 117 -2,-2.6 -1,-0.2 -3,-0.2 3,-0.2 0.925 105.1 28.4 -48.0 -58.9 6.9 -14.1 6.3 58 122 A P S S- 0 0 77 0, 0.0 2,-0.3 0, 0.0 -1,-0.2 0.975 145.7 -3.3 -68.2 -54.1 5.8 -13.3 9.9 59 123 A L - 0 0 121 5,-0.0 -3,-1.4 0, 0.0 2,-0.4 -0.997 69.0-165.2-140.1 142.1 4.2 -16.7 10.5 60 124 A A + 0 0 72 -2,-0.3 4,-0.1 -3,-0.2 -3,-0.0 -0.989 64.8 45.8-133.3 137.6 3.8 -19.6 8.1 61 125 A G S S+ 0 0 78 -2,-0.4 -1,-0.1 2,-0.3 3,-0.1 0.640 116.4 39.3 106.0 23.5 1.6 -22.7 8.3 62 126 A S S S+ 0 0 114 1,-0.2 -2,-0.1 -3,-0.2 2,-0.0 0.240 106.9 47.0-161.4 -51.7 -1.5 -21.0 9.4 63 127 A P - 0 0 45 0, 0.0 -2,-0.3 0, 0.0 2,-0.3 0.044 62.2-155.9 -89.0-160.2 -2.1 -17.6 7.6 64 128 A H - 0 0 122 -3,-0.1 -8,-0.1 -4,-0.1 -38,-0.0 -0.969 39.4 -45.0-172.0 165.1 -1.8 -16.6 3.9 65 129 A L + 0 0 47 -2,-0.3 2,-0.3 -11,-0.2 -39,-0.1 0.044 63.9 170.0 -38.5 142.7 -1.3 -13.7 1.6 66 130 A E - 0 0 81 -41,-0.1 -13,-0.9 -42,-0.1 2,-0.2 -0.964 29.5-163.9-153.9 170.7 -3.2 -10.5 2.5 67 131 A Y - 0 0 4 -2,-0.3 -44,-0.5 -15,-0.2 2,-0.4 -0.701 13.9-159.9-164.6 99.8 -3.7 -6.8 1.9 68 132 A F + 0 0 60 -2,-0.2 -17,-2.5 -46,-0.2 2,-0.3 -0.726 18.6 167.8 -89.7 133.2 -5.6 -4.6 4.3 69 133 A V E - C 0 50A 0 -2,-0.4 -48,-1.6 -48,-0.3 2,-0.4 -0.999 19.4-169.6-146.7 146.7 -7.0 -1.2 3.1 70 134 A R E +BC 20 49A 109 -21,-0.8 -21,-2.3 -2,-0.3 -22,-1.7 -0.993 23.9 148.0-137.1 127.7 -9.3 1.5 4.4 71 135 A F E -BC 19 47A 0 -52,-2.3 -52,-2.4 -2,-0.4 2,-0.4 -0.973 33.0-133.3-154.3 164.6 -10.7 4.3 2.2 72 136 A E E +BC 18 46A 15 -26,-1.7 -26,-2.7 -2,-0.3 -54,-0.3 -0.981 22.3 170.2-128.9 139.9 -13.6 6.6 1.6 73 137 A V E -B 17 0A 0 -56,-2.3 -56,-2.8 -2,-0.4 5,-0.2 -0.995 49.0 -78.3-147.3 138.6 -15.4 7.5 -1.7 74 138 A P E > -B 16 0A 50 0, 0.0 4,-2.6 0, 0.0 -58,-0.3 -0.012 49.9-111.7 -35.0 132.4 -18.6 9.3 -2.5 75 139 A S T 4 S+ 0 0 54 -60,-0.5 4,-0.5 1,-0.3 -59,-0.1 0.695 120.1 42.3 -45.3 -20.3 -21.5 6.9 -1.9 76 140 A G T > S+ 0 0 51 2,-0.1 4,-0.5 1,-0.1 -1,-0.3 0.834 113.7 45.4 -96.7 -40.6 -21.9 7.1 -5.6 77 141 A D H > S+ 0 0 61 -3,-0.3 4,-2.4 1,-0.2 5,-0.3 0.631 99.9 74.7 -78.7 -15.3 -18.3 7.0 -6.9 78 142 A L H X S+ 0 0 2 -4,-2.6 4,-3.3 -5,-0.2 3,-0.3 0.992 97.4 43.6 -55.6 -64.1 -17.6 4.2 -4.5
21 79 143 A A H > S+ 0 0 68 -4,-0.5 4,-1.1 1,-0.2 -1,-0.2 0.758 111.6 62.3 -50.7 -26.0 -19.5 1.7 -6.6 80 144 A A H < S+ 0 0 38 -4,-0.5 4,-0.4 2,-0.2 -2,-0.2 0.955 114.3 26.0 -68.7 -54.0 -17.6 3.4 -9.5 81 145 A L H >X S+ 0 0 1 -4,-2.4 4,-1.6 -3,-0.3 3,-0.7 0.775 113.7 67.4 -84.3 -25.1 -14.1 2.6 -8.5 82 146 A L H 3X S+ 0 0 33 -4,-3.3 4,-1.9 -5,-0.3 5,-0.2 0.868 91.9 62.2 -60.2 -35.4 -15.1 -0.6 -6.6 83 147 A S H 3X S+ 0 0 57 -4,-1.1 4,-0.6 -5,-0.3 -1,-0.3 0.861 103.9 49.3 -57.6 -34.7 -16.1 -2.1 -9.9 84 148 A S H <4 S+ 0 0 44 -3,-0.7 4,-0.4 -4,-0.4 3,-0.3 0.877 107.2 52.8 -69.1 -41.8 -12.4 -1.7 -10.8 85 149 A V H X S+ 0 0 13 -4,-1.6 4,-2.1 1,-0.3 3,-0.3 0.763 107.1 52.5 -65.0 -25.5 -11.5 -3.3 -7.6 86 150 A R H < S+ 0 0 146 -4,-1.9 -1,-0.3 1,-0.2 -2,-0.2 0.698 103.3 58.8 -79.2 -21.4 -13.8 -6.1 -8.7 87 151 A R T < S+ 0 0 180 -4,-0.6 -2,-0.2 -3,-0.3 -1,-0.2 0.557 114.1 36.8 -80.7 -11.8 -11.8 -6.1 -11.9 88 152 A V T 4 S+ 0 0 30 -4,-0.4 2,-0.3 -3,-0.3 -2,-0.2 0.670 130.0 17.4-109.5 -30.7 -8.7 -6.8 -9.7 89 153 A S < + 0 0 23 -4,-2.1 -1,-0.3 -5,-0.1 -64,-0.0 -0.963 47.4 155.1-142.5 157.6 -10.3 -9.2 -7.1 90 154 A D S S+ 0 0 100 -2,-0.3 -1,-0.1 -3,-0.1 -4,-0.1 0.427 71.6 62.6-149.8 -37.9 -13.5 -11.3 -6.8 91 155 A D S S+ 0 0 156 -67,-0.1 2,-0.2 2,-0.0 -67,-0.1 -0.074 94.9 76.1 -91.9 34.5 -12.9 -14.1 -4.4 92 156 A V - 0 0 39 -69,-0.1 2,-0.2 -70,-0.0 -84,-0.2 -0.639 63.8-138.8-129.4-171.2 -12.2 -11.9 -1.4 93 157 A R - 0 0 149 -71,-0.3 -71,-2.6 -2,-0.2 2,-0.1 -0.793 27.5 -79.6-141.4-177.8 -14.1 -9.7 1.1 94 158 A S B H 21 0B 3 -73,-0.3 -73,-0.3 -2,-0.2 -1,-0.1 -0.343 360.0 360.0 -81.5 168.0 -14.1 -6.4 3.0 95 159 A A 0 0 66 -75,-0.5 -1,-0.2 -26,-0.3 -75,-0.1 0.423 360.0 360.0 -95.0 360.0 -12.0 -5.8 6.1 96 !* 0 0 0 0, 0.0 0, 0.0 0, 0.0 0, 0.0 0.000 360.0 360.0 360.0 360.0 0.0 0.0 0.0 97 65 B P 0 0 176 0, 0.0 2,-0.4 0, 0.0 0, 0.0 0.000 360.0 360.0 360.0 167.1 23.2 15.5 -7.8 98 66 B G - 0 0 70 4,-0.0 0, 0.0 0, 0.0 0, 0.0 -0.888 360.0-166.4-132.0 104.1 24.5 12.9 -5.4 99 67 B N > - 0 0 105 -2,-0.4 3,-0.9 1,-0.2 0, 0.0 -0.790 3.3-172.4 -92.2 111.2 24.4 13.5 -1.6 100 68 B P T 3 S+ 0 0 115 0, 0.0 -1,-0.2 0, 0.0 0, 0.0 0.903 88.6 48.9 -66.1 -43.9 26.6 11.0 0.2 101 69 B L T 3 S+ 0 0 162 2,-0.0 -2,-0.1 0, 0.0 0, 0.0 0.226 90.3 129.2 -82.0 14.5 25.4 12.1 3.7 102 70 B E S < S- 0 0 83 -3,-0.9 87,-0.0 2,-0.1 -4,-0.0 -0.095 70.8-115.7 -65.6 169.1 21.8 11.9 2.4 103 71 B A S S+ 0 0 48 2,-0.1 2,-0.8 87,-0.0 -1,-0.1 0.749 84.7 101.8 -79.3 -27.5 19.1 10.0 4.3 104 72 B V + 0 0 58 84,-0.2 2,-0.2 85,-0.1 -2,-0.1 -0.473 52.1 130.7 -66.9 105.1 18.5 7.4 1.6 105 73 B V - 0 0 93 -2,-0.8 11,-0.3 11,-0.2 2,-0.3 -0.775 32.7-163.6-141.4-175.7 20.3 4.3 2.9 106 74 B F - 0 0 107 -2,-0.2 2,-0.3 9,-0.1 9,-0.2 -0.972 7.7-141.9-164.7 172.3 19.8 0.6 3.4 107 75 B E E -E 114 0A 120 7,-0.9 7,-1.1 -2,-0.3 2,-0.4 -0.896 18.1-121.9-139.2 168.3 21.0 -2.6 5.2 108 76 B E E -E 113 0A 113 5,-0.3 2,-0.4 -2,-0.3 5,-0.3 -0.955 18.7-171.7-124.3 138.6 21.4 -6.3 4.4 109 77 B R E > S-E 112 0A 123 3,-2.5 3,-1.0 -2,-0.4 62,-0.1 -0.989 70.4 -19.7-128.0 129.3 20.0 -9.3 6.1 110 78 B D T 3 S- 0 0 160 -2,-0.4 3,-0.1 1,-0.3 -1,-0.1 0.699 134.3 -41.1 50.4 22.2 20.8 -12.9 5.5 111 79 B G T 3 S+ 0 0 58 1,-0.5 60,-0.5 58,-0.1 -1,-0.3 0.709 128.4 76.2 99.7 22.8 22.1 -12.0 2.1 112 80 B N E < S-EF 109 170A 13 -3,-1.0 -3,-2.5 58,-0.3 2,-0.5 -0.871 79.7-100.1-148.8 178.1 19.5 -9.4 1.1 113 81 B A E -EF 108 169A 0 56,-2.8 56,-2.3 -5,-0.3 2,-0.5 -0.943 23.2-167.0-120.2 127.4 18.6 -5.9 1.9 114 82 B V E +EF 107 168A 8 -7,-1.1 -7,-0.9 -2,-0.5 2,-0.3 -0.917 31.2 130.5-108.5 122.9 16.0 -4.7 4.3
22 115 83 B L E - F 0 167A 10 52,-2.4 52,-2.3 -2,-0.5 2,-0.3 -0.897 43.3-117.5-156.2-177.2 15.1 -1.0 4.2 116 84 B N E - F 0 166A 29 -11,-0.3 75,-0.5 -2,-0.3 2,-0.3 -0.950 17.3-165.2-134.1 154.0 12.3 1.6 4.0 117 85 B L B -I 190 0C 17 48,-1.6 2,-0.3 73,-0.3 48,-0.3 -0.972 0.5-167.8-146.7 126.9 11.3 4.3 1.5 118 86 B L + 0 0 41 71,-2.6 71,-0.3 -2,-0.3 2,-0.3 -0.791 11.4 168.1-111.0 155.9 8.9 7.2 1.7 119 87 B F - 0 0 29 44,-0.5 2,-0.3 -2,-0.3 69,-0.1 -0.927 12.6-160.7-165.3 141.3 7.6 9.4 -1.1 120 88 B S - 0 0 44 -2,-0.3 2,-0.3 42,-0.1 67,-0.1 -0.911 1.9-165.9-128.9 154.4 4.9 12.1 -1.5 121 89 B L - 0 0 23 -2,-0.3 41,-0.1 41,-0.1 40,-0.1 -0.998 10.2-143.8-144.0 136.0 3.0 13.6 -4.4 122 90 B R + 0 0 223 -2,-0.3 38,-0.0 39,-0.1 0, 0.0 0.049 62.9 44.9 -81.0-165.5 0.8 16.7 -4.8 123 91 B G S S- 0 0 60 1,-0.1 -2,-0.1 2,-0.1 0, 0.0 -0.186 87.0 -98.0 64.7-159.2 -2.3 17.1 -6.9 124 92 B T S S+ 0 0 69 -82,-0.0 -80,-0.2 0, 0.0 -1,-0.1 0.479 113.2 47.2-132.9 -21.6 -5.0 14.5 -7.1 125 93 B K S S+ 0 0 147 1,-0.2 2,-2.2 -81,-0.1 -2,-0.1 0.881 91.4 77.5 -90.6 -48.0 -4.2 12.6 -10.2 126 94 B P + 0 0 70 0, 0.0 -1,-0.2 0, 0.0 0, 0.0 -0.409 55.9 120.7 -67.8 79.5 -0.4 12.0 -9.8 127 95 B S > + 0 0 20 -2,-2.2 2,-2.1 -3,-0.1 3,-0.6 -0.220 19.1 148.4-134.0 39.1 -0.7 9.2 -7.2 128 96 B S T > + 0 0 68 1,-0.3 3,-0.6 2,-0.1 4,-0.5 -0.124 22.5 129.5 -70.9 42.2 1.1 6.4 -9.2 129 97 B L T >> + 0 0 15 -2,-2.1 4,-1.1 1,-0.2 3,-0.6 0.720 61.4 74.0 -67.8 -20.2 2.3 4.9 -5.9 130 98 B S H X> S+ 0 0 10 -3,-0.6 4,-1.1 1,-0.3 3,-0.7 0.916 88.3 55.6 -56.8 -47.2 0.9 1.7 -7.4 131 99 B R H <4 S+ 0 0 153 -3,-0.6 4,-0.5 1,-0.3 -1,-0.3 0.773 100.5 63.4 -59.0 -23.4 3.9 1.5 -9.7 132 100 B A H X> S+ 0 0 1 -3,-0.6 3,-1.2 -4,-0.5 4,-1.0 0.899 99.8 49.3 -68.3 -41.8 6.0 1.7 -6.6 133 101 B V H <X S+ 0 0 9 -4,-1.1 4,-1.3 -3,-0.7 -1,-0.2 0.740 100.8 66.8 -68.9 -20.9 4.6 -1.6 -5.3 134 102 B K H 3X S+ 0 0 64 -4,-1.1 4,-1.0 2,-0.2 -1,-0.3 0.729 97.1 57.9 -68.9 -21.3 5.4 -3.0 -8.7 135 103 B V H X> S+ 0 0 0 -3,-1.2 4,-1.2 -4,-0.5 3,-1.0 0.992 107.3 39.1 -71.7 -67.3 9.0 -2.5 -7.8 136 104 B F H 3<>S+ 0 0 0 -4,-1.0 5,-2.1 1,-0.3 -2,-0.2 0.774 121.4 49.4 -55.4 -26.9 9.4 -4.6 -4.7 137 105 B E H ><5S+ 0 0 24 -4,-1.3 3,-1.1 3,-0.2 -1,-0.3 0.715 97.9 67.3 -86.2 -23.2 7.1 -7.2 -6.4 138 106 B T H <<5S+ 0 0 79 -4,-1.0 -2,-0.2 -3,-1.0 -1,-0.2 0.888 107.6 37.8 -66.8 -39.5 9.0 -7.3 -9.7 139 107 B F T 3<5S- 0 0 77 -4,-1.2 -1,-0.3 34,-0.1 -2,-0.1 0.119 115.4-112.6 -98.5 21.9 12.1 -8.9 -8.1 140 108 B E T < 5 + 0 0 104 -3,-1.1 -3,-0.2 -112,-0.2 -2,-0.1 0.858 57.3 171.6 52.2 40.4 10.0 -11.1 -5.9 141 109 B A < - 0 0 9 -5,-2.1 2,-0.7 -6,-0.2 28,-0.2 -0.160 37.9-108.1 -71.6 173.6 11.3 -9.2 -2.8 142 110 B K E - G 0 168A 82 26,-2.7 26,-1.7 -89,-0.0 2,-0.4 -0.890 30.6-149.6-111.0 104.4 9.8 -9.8 0.7 143 111 B I E - G 0 167A 5 -2,-0.7 24,-0.3 24,-0.3 3,-0.1 -0.567 14.4-174.3 -78.1 128.6 7.7 -6.8 1.8 144 112 B H E S- 0 0A 57 22,-1.7 23,-0.2 -2,-0.4 2,-0.2 0.915 74.6 -11.7 -84.4 -52.3 7.7 -6.1 5.5 145 113 B H E - G 0 166A 43 21,-2.3 21,-0.8 2,-0.0 2,-0.5 -0.658 61.4-176.2-159.6 96.3 5.1 -3.3 5.7 146 114 B L E +DG 52 165A 5 -94,-2.1 -94,-2.3 19,-0.3 2,-0.3 -0.821 21.5 161.8 -94.3 126.8 3.8 -1.4 2.7 147 115 B E E -D 51 0A 25 17,-2.5 2,-0.3 -2,-0.5 -96,-0.2 -0.996 23.0-169.5-148.6 152.8 1.5 1.5 3.6 148 116 B T E +D 50 0A 9 -98,-2.3 -98,-2.1 -2,-0.3 15,-0.2 -0.991 16.6 153.8-143.9 131.7 0.1 4.7 2.1 149 117 B R - 0 0 92 13,-0.9 -101,-0.1 -2,-0.3 -103,-0.0 -0.617 33.6-136.4-162.0 95.3 -1.9 7.4 3.8 150 118 B P - 0 0 28 0, 0.0 3,-0.2 0, 0.0 11,-0.2 -0.151 24.9-125.3 -52.6 143.9 -2.0 11.0 2.5
23 151 119 B A S S+ 0 0 3 1,-0.2 2,-2.6 2,-0.1 5,-0.1 0.115 84.5 58.0 -74.1-163.2 -1.6 13.7 5.1 152 120 B Q S S+ 0 0 104 3,-1.4 -1,-0.2 1,-0.2 9,-0.0 -0.301 91.7 79.2 76.0 -55.2 -4.0 16.6 5.8 153 121 B R S S+ 0 0 121 -2,-2.6 -1,-0.2 -3,-0.2 3,-0.2 0.924 105.1 28.5 -48.0 -58.9 -6.8 14.2 6.3 154 122 B P S S- 0 0 76 0, 0.0 2,-0.3 0, 0.0 -1,-0.2 0.975 145.7 -3.3 -68.1 -54.2 -5.7 13.4 9.9 155 123 B L - 0 0 120 5,-0.0 -3,-1.4 0, 0.0 2,-0.4 -0.997 69.0-165.2-140.1 142.1 -4.1 16.8 10.5 156 124 B A + 0 0 72 -2,-0.3 4,-0.1 -3,-0.2 -3,-0.0 -0.989 64.8 45.9-133.3 137.7 -3.7 19.7 8.1 157 125 B G S S+ 0 0 76 -2,-0.4 -1,-0.1 2,-0.3 3,-0.1 0.641 116.4 39.2 106.1 23.5 -1.5 22.8 8.4 158 126 B S S S+ 0 0 117 1,-0.2 -2,-0.1 -3,-0.2 2,-0.0 0.241 106.9 47.0-161.4 -51.7 1.7 21.0 9.4 159 127 B P - 0 0 48 0, 0.0 -2,-0.3 0, 0.0 2,-0.3 0.042 62.2-155.9 -89.0-160.1 2.2 17.7 7.6 160 128 B H - 0 0 122 -3,-0.1 -8,-0.1 -4,-0.1 -38,-0.0 -0.969 39.4 -45.1-172.0 165.0 1.9 16.7 4.0 161 129 B L + 0 0 45 -2,-0.3 2,-0.3 -11,-0.2 -39,-0.1 0.043 63.9 170.1 -38.5 142.8 1.3 13.7 1.6
24 Lampiran 2 Antar muka prediksi struktur sekunder protein
25
RIWAYAT HIDUP
Penulis lahir di Pati pada tanggal 5 Mei 1993. Penulis merupakan anak ke
empat dari empat bersaudara dengan ayah bernama Pasiman dan ibu bernama Senok. Pada tahun 2010 penulis lulus dari SMA Negeri 3 Pati, dan pada tahun yang sama penulis lulus seleksi masuk Institut Pertanian Bogor melalui jalur Ujian Saringan Masuk IPB (USMI) dengan Program Studi Ilmu Komputer pada Fakultas Matematika dan Ilmu Pengetahuan Alam (MIPA).
Selama menjadi mahasiswa Institut Petanian Bogor, pada tahun 2013 penulis menjalankan praktik lapang di Kantor Komunikasi dan Informatika Kota Bogor. Penulis aktif menjadi pengurus Himpunan Mahasiswa Ilmu Komputer pada tahun 2011-2012, anggota kepanitiaan Pesta Sains Nasional 2012, anggota kepanitiaan explo sains 2012, dan anggota kepanitiaan IT Today 2012.