OPTIMISASI PARAMETER SUPPORT VECTOR MACHINE (SVM)
MENGGUNAKAN ALGORITME GENETIKA
IRAWATI
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
OPTIMISASI PARAMETER SUPPORT VECTOR MACHINE (SVM)
MENGGUNAKAN ALGORITME GENETIKA
IRAWATI
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar
Sarjana Komputer pada
Departemen Ilmu Komputer
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
ABSTRACT
IRAWATI. Optimizing Support Vector Machine Parameters using Genetic Algorithm. Supervised by MUSHTHOFA.
Support vector machine (SVM) is a popular classification method which is known to have a robust generalization capability. SVM calculates the best linear separator on the input feature space according to the training data. To classify data which are non-linearly separable, SVM uses kernel tricks to transform the data into a linearly separable data on a higher dimension feature space. Kernel trick uses various kinds of kernel functions, such as: linear kernel, polynomial, radial basis function (RBF) and sigmoid. Each function has parameters which affect the accuracy of SVM classification. In this research, we propose a combination of the SVM algorithm and genetic algorithm to search for the kernel parameters with the best accuracy. We use data from UCI repository of machine learning database: Image Letter Recognition, Diabetes, and Yeast. The results indicate that the combination of SVM and genetic algorithm is effective in improving the accuracy of classification. Genetic algorithm has been shown to be effective in systematically finding optimal kernel parameters for SVM, as a replacement for randomly choosing kernel parameters. Best accuracy for each data has been improved from previous research: 97.05% for Letter, 78.21% for Diabetes, and 58.97% for Yeast. However, for bigger data sizes, this method may become impractical due to the high time requirement.
Judul Penelitian : Optimisasi Parameter Support Vector Machine (SVM) Menggunakan Algoritme Genetika Nama : Irawati NRP : G64063524 Menyetujui: Pembimbing Mushthofa, S.Kom, M.Sc. NIP. 19820325 200912 1 003 Mengetahui:
Ketua Departemen Ilmu Komputer Institut Pertanian Bogor
Dr. Ir. Sri Nurdiati, M.Sc. NIP. 19601126 198601 2 001
PRAKATA
Alhamdulillahi Rabbil ‘alamin, puji dan syukur penulis panjatkan kepada Allah SWT atas limpahan rahmat dan hidayah-Nya sehingga tugas akhir dengan judul Optimisasi Parameter Support Vector Machine (SVM) menggunakan Algoritme Genetika dapat diselesaikan. Penelitian ini dilaksanakan mulai Maret 2010 sampai dengan Desember 2010, bertempat di Departemen Ilmu Komputer.
Penulis ingin mengucapkan terima kasih kepada semua pihak yang telah memberikan bantuan, dorongan, serta bimbingan kepada penulis selama menyelesaikan tugas akhir ini, antara lain:
1. Kedua orangtua tercinta, Ayahanda Akmal dan Ibunda Samiarti yang telah memberi inspirasi, limpahan kasih sayang, dukungan moral, serta doa selama ini.
2. Kakak-kakak tercinta, Yan Firwan, Rahmi, Hendra, Ridwan, Erninda, Fajri yang selalu memberikan semangat dan nasehat selama pengerjaan tugas akhir ini.
3. Bapak Mushthofa, S.Kom, M.Sc, selaku pembimbing atas bimbingan dan arahannya selama pengerjaan tugas akhir ini.
4. Bapak Dr.Ir.Agus Buono, M.Si, M.Kom dan Dr.Ir.Yandra A, M.Eng yang berkenan untuk menjadi penguji penelitian ini.
5. Yoga Tunseno yang selalu memberikan dukungan, doa, dan kasih sayangnya. 6. Sandy Cahya Gumilar rekan satu bimbingan atas dukungan dan kerja samanya.
7. Dian Destria, Elvina Diah S, Rahmi Hayati, dan Septiani Wesman atas semangat yang selalu diberikan.
8. Temen-teman seperjuangan Program Studi Ilmu Komputer angkatan 43 atas kebersamaan dan pengalaman berbagi ilmu selama menuntut ilmu di Ilmu Komputer IPB.
Penulis selalu berharap semoga apa yang telah dikerjakan dalam penelitian ini dapat memberikan manfaat bagi semua pihak dan dapat menjadi acuan untuk penelitian selanjutnya.
Bogor, Desember 2010
RIWAYAT HIDUP
Penulis dilahirkan di Bukittinggi, Sumatera Barat pada tanggal 13 Februari 1988 sebagai anak ke-7 dari 7 bersaudara pasangan Bapak Akmal dan Ibu Samiarti. Pada tahun 2000 penulis menyelesaikan pendidikan dasar di SDN 02 Baso. Pada tahun 2003 lulus dari SLTPN 02 Empat Angkat Candung dan pada tahun 2006 lulus dari SMUN 1 Empat Angkat.
Pada tahun 2006 penulis diterima menjadi mahasiswa Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI). Berdasarkan hasil seleksi Mayor Minor, pada tahun 2007 penulis diterima di Departemen Ilmu Komputer yang merupakan departemen pilihan pertama penulis dengan minor Riset Operasi. Pada tahun 2009, penulis melaksanakan kegiatan praktik kerja lapangan di PT Waindo Specterra selama dua bulan.
DAFTAR ISI
Halaman
DAFTAR TABEL
... vi
DAFTAR GAMBAR ... vi
DAFTAR LAMPIRAN ... vii
PENDAHULUAN Latar Belakang ... 1 Rumusan Masalah ... 1 Tujuan Penelitian ... 1 Ruang Lingkup ... 1 Manfaat ... 1 TINJAUAN PUSTAKA Klasifikasi ... 1Support Vector Machine ... 2
Multi Class SVM ... 3
K-Fold Cross Validation ... 4
Algoritme Genetika ... 4 Seleksi ... 5 Crossover ... 6 Mutasi ... 6 Elitisme ... 6 METODE PENELITIAN Pengumpulan Data ... 7 Praproses Data... 7
Data Latih dan Data Uji ... 7
SVM Training dan Testing ... 7
Optimisasi Parameter SVM... 7
Lingkup Pengembangan Sistem ... 9
HASIL DAN PEMBAHASAN Data ... 9
Praproses ... 9
Data Latih dan Data Uji ... 10
Klasifikasi ... 10
Optimisasi Parameter SVM... 10
Image Letter Recognition... 11
Pima Indians Diabetes ... 12
KESIMPULAN DAN SARAN
Kesimpulan... 15
Saran ... 15
DAFTAR PUSTAKA ... 16
DAFTAR TABEL
Halaman
1 Daftar istilah yang digunakan dalam algoritme genetika ... 4
2 Jumlah instance, attribute, class dan missing values pada data Image Letter Recognition, Pima Indians Diabetes, dan Protein Localization Sites ... 9
3 Informasi jumlah data untuk tiap kelas pada data Image Letter Recognition ... 9
4 Informasi jumlah data untuk tiap kelas pada data Pima Indians Diabetes ... 9
5 Informasi jumlah data untuk tiap kelas pada data Protein Localization Sites ... 9
6 Kode kelas pada data Yeast ... 10
7 Akurasi hasil proses pengujian dengan nilai parameter C, γ, r, dan d yang ditentukan pada data Image Letter Recognition, Pima Indians Diabetes, dan Protein Localization Sites ... 11
8 Nilai fitness terbaik data Letter secara Linear, Polynomial, RBF, dan Sigmoid ... 11
9 Nilai fitness terbaik data Diabetes secara Linear, Polynomial, RBF, dan Sigmoid ... 12
10 Nilai fitness terbaik data Yeast secara Linear, Polynomial, RBF, dan Sigmoid ... 12
11 Hasil klasifikasi tiap kelas pada data Yeast menggunakan RBF kernel... 13
12 Akurasi yang diperoleh untuk tiap kelas ... 13
13 Besarnya kenaikan nilai fitness pada optimasi parameter SVM menggunakan algoritme genetika... 13
14 Perbandingan nilai akurasi yang melakukan optimisasi parameter dengan akurasi peng-input-an nilai parameter yang ditentukan ... 15
DAFTAR GAMBAR
Halaman 1 Data dipisahkan secara linear ... 22 Soft margin hyperplane ... 3
3 Fungsi Φ memetakan data ke ruang vektor yang berdimensi lebih tinggi ... 3
4 Contoh penggunaan teknik Roulette-wheel selection ... 5
5 Proses Crossover satu titik potong (one point crossover) ... 6
6 Proses Crossover dua titik potong (two point crossover) ... 6
7 Proses Mutasi. ... 6
8 Metode Penelitian. ... 7
9 Fitness terbaik untuk setiap generasi pada data Image Letter Recognition secara Linear, Polynomial, RBF, dan Sigmoid kernel ... 12
10 Fitness terbaik untuk setiap generasi pada data Pima Indians Diabetes secara Linear, Polynomial, RBF, dan Sigmoid kernel ... 12
11 Fitness terbaik untuk setiap generasi pada data Yeast secara Linear, Polynomial, RBF, dan Sigmoid kernel... 12
12 Persebaran data Yeast menggunakan Multidimensional Scaling ... 13
13 Perbandingan nilai akurasi setiap kernel untuk data Image Letter Recognition ... 14
14 Perbandingan nilai akurasi setiap kernel untuk data Pima Indians Diabetes ... 14
DAFTAR LAMPIRAN
Halaman
1 Hasil optimisasi data Image Letter Recognition untuk Linear kernel... 18
2 Hasil optimisasi data Image Letter Recognition untuk Polynomial kernel... 19
3 Hasil optimisasi data Image Letter Recognition untuk RBF kernel ... 20
4 Hasil optimisasi data Image Letter Recognition untuk Sigmoid kernel ... 21
5 Hasil optimisasi data Pima Indians Diabetes untuk Linear kernel ... 22
6 Hasil optimisasi data Pima Indians Diabetes untuk Polynomial kernel. ... 23
7 Hasil optimisasi data Pima Indians Diabetes untuk RBF kernel. ... 24
8 Hasil optimisasi data Pima Indians Diabetes untuk Sigmoid kernel ... 25
9 Hasil optimisasi data Protein Localization Sites untuk Linear kernel ... 26
10 Hasil optimisasi data Protein Localization Sites untuk Polynomial kernel ... 27
11 Hasil optimisasi data Protein Localization Sites untuk RBF kernel ... 28
12 Hasil optimisasi data Protein Localization Sites untuk Sigmoid kernel ... 29
1
PENDAHULUAN Latar Belakang
Klasifikasi adalah pemberian kategori yang telah didefinisikan ke data. Menglasifikasikan data merupakan salah satu cara untuk mengorganisasikan data, data yang memiliki karakteristik atau ciri yang sama akan dikelompokkan ke dalam kategori yang sama. Hal ini dapat memudahkan bagi orang dalam melakukan pencarian informasi.
Meningkatnya jumlah data telah mendorong berkembangnya metode penglasifikasian secara otomatis yang mampu melakukan klasifikasi seperti penglasifikasian secara manual. Dengan metode tersebut, keuntungan yang diperoleh adalah penghematan tenaga kerja dan efektivitas yang baik.
Dari perbedaan karakteristik yang dikandung oleh setiap data memberikan tantangan tersendiri dalam melakukan klasifikasi. Suatu model klasifikasi dikatakan baik jika mencapai nilai akurasi yang paling tinggi dan model inilah yang akan digunakan untuk menentukan hasil klasifikasi akhir. Disamping itu, untuk menciptakan suatu model yang optimal dibutuhkan juga suatu nilai parameter yang mampu mengoptimalkan akurasi suatu model.
Salah satu metode klasifikasi yang cukup diperhitungkan saat ini adalah Support Vector Machine (SVM). Metode klasifikasi ini sudah diterapkan dalam beberapa bidang. Kerami dan Mufti (2004) menggunakan metode klasifikasi SVM dalam pengenalan jenis splice sites pada baris DNA. Noorniawati (2007) menggunakan metode klasifikasi SVM pada sistem temu kembali citra. Damasevicius R (2008) melakukan optimisasi parameter SVM pada kasus DNA sequences menggunakan metode Nelder-Mead sehingga akurasi meningkat sebesar 1.29%.
Pada penelitian ini dilakukan penglasifikasian terhadap data dengan menggunakan metode klasifikasi Support Vector Machine. Untuk penglasifikasian data ini dibutuhkan parameter yang mampu menghasilkan performance yang optimal untuk suatu model klasifikasi. Dengan demikian, dibutuhkan suatu algoritme yang secara sistematis mampu menentukan nilai parameter sehingga menghasilkan performance yang baik. Algoritme yang digunakan untuk optimisasi
parameter pada penelitian ini adalah algoritme genetika.
Rumusan Masalah
Rumusan masalah dalam penelitian ini adalah bagaimana melakukan pencarian parameter yang optimum dalam metode klasifikasi Support Vector Machine menggunakan algoritme genetika. Tujuan Penelitian
Tujuan dari penelitian ini adalah mengetahui efektivitas algoritme genetika dalam melakukan optimisasi parameter pada Support Vector Machine dalam melakukan klasifikasi data. Ruang Lingkup
Ruang lingkup penelitian ini difokuskan pada optimisasi parameter menggunakan algoritme genetika pada klasifikasi Support Vector Machine. Data yang digunakan pada penelitian ini adalah beberapa data koleksi dari UCI repository of machine learning database.
Manfaat
Manfaat yang dapat diperoleh dari penelitian ini adalah:
1. Mengetahui efektivitas algoritme genetika dalam pencarian parameter yang optimum pada klasifikasi Support Vector Machine. 2. Mengetahui perilaku dan kinerja Support
Vector Machine pada data dengan berbagai parameter kernel.
TINJAUAN PUSTAKA Klasifikasi
Klasifikasi adalah proses menemukan model atau fungsi yang menjelaskan dan membedakan kelas-kelas atau konsep, dengan tujuan agar model yang diperoleh dapat digunakan untuk mengetahui kelas atau objek yang memiliki label kelas yang tidak diketahui. Model yang diturunkan didasarkan pada analisis dari training data. Klasifikasi termasuk ke dalam kategori predictive data mining.
Pada proses klasifikasi terbagi menjadi dua fase yaitu, learning dan testing. Pada fase learning, sebagian data yang telah diketahui kelas datanya (training set) digunakan untuk membentuk model. Selanjutnya pada fase testing, model yang sudah dibentuk diuji dengan sebagian
2
data lainnya (test set) untuk mengetahui akurasi dari model tersebut. Jika akurasinya mencukupi maka model tersebut dapat dipakai untuk prediksi kelas data yang belum diketahui (Han & Kamber 2006).
Support Vector Machine
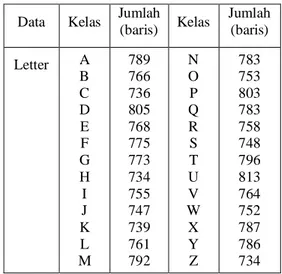
Support Vector Machine (SVM) adalah sistem pembelajaran untuk menglasifikasikan data menjadi dua kelompok data yang menggunakan ruang hipotesis berupa fungsi-fungsi linear dalam sebuah ruang fitur (feature space) berdimensi tinggi. SVM memiliki sifat yang tidak dimiliki oleh mesin pembelajaran pada umumnya yaitu dalam proses menemukan garis pemisah (hyperplane) terbaik sehingga diperoleh ukuran margin yang maksimal antara ruang input bukan-linear dengan ruang ciri menggunakan kaidah kernel (Cortes & Vapnik, 1995). Margin adalah dua kali jarak antara hyperplane dengan support vector. Titik yang terdekat dengan hyperplane disebut support vector.
Misalkan data dinotasikan sebagai xi ∊ ℜn , untuk label kelas dari data xi dinotasikan y ∊ {+1,-1} dengan i = 1,2,…,l dimana l adalah banyak data. Pemisahan data secara linear pada metode SVM dapat dilihat pada Gambar 1.
Gambar 1 Data dipisahkan secara linear Contoh di atas menunjukkan bahwa kedua kelas dapat dipisahkan oleh sepasang bidang pembatas yang sejajar. Bidang pembatas yang pertama membatasi kelas pertama sedangkan bidang pembatas yang kedua membatasi kelas kedua, sehingga diperoleh:
. + ≥ +1 = +1
. + ≤ −1 = −1 (1)
dengan
w = vektor bobot yang tegak lurus terhadap hyperplane (bidang normal)
b = posisi bidang relatif terhadap pusat koordinat
Nilai margin antara dua kelas adalah =
‖ ‖.
Margin dapat dimaksimalkan menggunakan fungsi optimisasi Lagrangian seperti berikut:
min
, ( , , ) =
1
2‖ ‖ − ( . + ) − 1 (2)
Dengan meminimumkan L terhadap w dan b, maka diperoleh:
= − = 0
= = 0
persamaan (2) dapat dimodifikasi sebagai maksimalisasi L yang hanya mengandung αi
sebagai persamaan (3).
max = −1
2 . (3)
. . = 0, ≥ 0
α yang dihasilkan digunakan untuk mencari w. Data yang memiliki nilai αi ≥ 0 merupakan support vector sedangkan sisanya memiliki nilai αi = 0.
Setelah nilai αi ditemukan, maka kelas dari
data pengujian x dapat ditentukan berdasarkan nilai fungsi keputusan:
( ) = . + (4)
dengan
xi = support vector ns = jumlah support vector
3
Jika data tidak dapat dipisahkan secara sempurna dengan pemisahan secara linear, SVM dimodifikasi dengan menambahkan variabel ξi (ξi
≥ 0, ∀I ;ξi = 0 jika xi diklasifikasikan dengan
benar) sehingga formula pencarian bidang pemisah terbaik menjadi:
min1 2 | | +
. . ( . + ) ≥ 1 − ,
≥ 0 (5)
Pencarian bidang pemisah terbaik dengan penambahan variabel ξi disebut soft margin hyperplane. C adalah parameter yang menentukan besar penalti akibat kesalahan dalam klasifikasi. Dengan demikian, dual problem yang dihasilkan pada non linear problem sama dengan dual problem yang dihasilkan dengan linear problem hanya saja rentang αi antara 0 ≥ αi ≥C.
Gambar 2 Soft margin hyperplane
Gambar 3 Fungsi Φ memetakan data ke ruang vektor yang berdimensi lebih tinggi Cara lain untuk data yang tidak dapat dipisahkan secara linear, SVM dimodifikasi dengan memasukkan fungsi ϕ(x). Dalam SVM untuk data yang tidak dapat dipisahkan secara
linear, pertama-tama data dipetakan oleh fungsi ϕ(x) ke ruang vector baru yang berdimensi lebih tinggi, seperti pada Gambar 3. Selanjutnya di ruang vector yang baru itu, SVM mencari hyperplane yang memisahkan kedua kelas secara linear. Pencarian ini hanya bergantung pada dot product dari data yang sudah dipetakan pada ruang baru yang berdimensi lebih tinggi, yaitu ϕ( )ϕ( ). Karena umumnya transformasi ( ) ini tidak diketahui dan sangat sulit untuk diketahui, maka perhitungan dot product dapat digantikan dengan fungsi Kernel yang dirumuskan sebagai berikut:
( , ) = ( ). ( ) (6)
sehingga persamaan (2) menjadi seperti berikut:
= −1
2 ( . ) (7)
Dengan demikian fungsi yang dihasilkan adalah:
( ) = ( , ) + (8)
xi = support vector dan NS adalah jumlah support vector.
Beberapa fungsi Kernel yang umum digunakan adalah:
1. Linear kernel ( , ) = . 2. Polynomial kernel
( , ) = ( . . + ) , > 0 3. Radial Basic Function
( , ) = exp(− | − | ) , > 0 4. Sigmoid kernel
( , ) = tanh ( . . + )
Dalam hal ini, γ, r, dan d merupakan parameter kernel, serta parameter C sebagai penalti akibat kesalahan dalam klasifikasi untuk masing-masing kernel.
Multi Class SVM
Untuk menglasifikasikan data yang memiliki lebih dari dua kelas, terdapat metode yang umum digunakan untuk mengimplementasikannya. Metode tersebut adalah against-all dan one-against-one. Untuk metode one-against-all,
4
dibangun k buah model SVM (k adalah jumlah kelas). Setiap model klasifikasi ke-i dilatih dengan menggunakan keseluruhan data, untuk mencari solusi permasalahan (9) (Sembiring 2007).
min 1
2( ) +
s.t ( ) ( ) + ≥ 1 − → = ,
( ) ( ) + ≥ −1 + → ≠ ,
≥ 0 (9)
Pada metode one-against-one dibangun ( ) buah model klasifikasi (k adalah jumlah kelas). Setiap model klasifikasi dilatih pada data dari dua kelas. Untuk data pelatihan dari kelas ke-i dan kelas ke-j, dilakukan pencarian solusi untuk persoalan optimasi konstrain sebagai berikut:
min 1
2( ) +
s.t ( ) ( ) + ≥ 1 − → = ,
( ) ( ) + ≥ −1 + → = ,
≥ 0 (10)
K-Fold Cross Validation
K-Fold Cross Validation adalah metode yang digunakan untuk membagi data menjadi data pelatihan dan data pengujian. K-Fold Cross Validation membagi data contoh secara acak ke dalam K subset yang saling bebas. Satu subset digunakan sebagai data pengujian dan K-1 subset sebagai data pelatihan. Proses cross validation akan diulang hingga K kali. Data awal dibagi menjadi K subset yang saling bebas secara acak yaitu, S1, S2,…,Sk, dengan ukuran setiap subset kira-kira sama. Pelatihan dan pengujian dilakukan sebanyak K kali. Pada proses ke-i, subset Si diperlakukan sebagai data pengujian dan subset lainnya diperlakukan sebagai data pelatihan. Pada proses pertama S2,…, Sk menjadi data pelatihan dan S1 menjadi data pengujian, pada proses kedua S1, S3,…, Sk menjadi data pelatihan dan S2 menjadi data pengujian, dan seterusnya (Fu 1994).
Algoritme Genetika
Dalam bukunya, DE Goldberg (1989) mendefinisikan algoritme genetika sebagai suatu
algoritme pencarian yang didasarkan pada mekanisme seleksi alam dan genetika. Algoritme ini tepat digunakan untuk penyelesaian masalah optimasi yang kompleks dan sulit diselesaikan dengan menggunakan metode optimasi yang konvensional karena memiliki beberapa keunggulan, yaitu:
1. Algoritme genetika memproses bentuk pengkodean dari parameter solusi, bukan parameter itu sendiri.
2. Algoritme genetika melakukan pencarian dalam banyak solusi yang disebut populasi solusi, bukan pada satu solusi.
3. Informasi yang digunakan dalam algoritme genetika berasal dari fungsi tujuan, bukan dari informasi tambahan.
4. Algoritme genetika menggunakan probabilistic rules, bukan deterministic rules.
Algoritme genetika banyak digunakan pada masalah praktis yang fokus pada pencarian parameter-parameter optimal. Kemunculan algoritme ini terinspirasi dari teori-teori dalam ilmu Biologi karena pada awalnya algoritme genetika digunakan oleh para ahli biologi untuk mensimulasikan biological systems, sehingga banyak istilah dan konsep biologi yang digunakan dalam algoritme genetika (Michalewicz 1996). Daftar istilah yang digunakan dalam algoritme genetika dapat dilihat pada Tabel 1.
Tabel 1 Daftar istilah yang digunakan dalam algoritme genetika
Istilah Biologi
Definisi dalam Algoritme Genetika
Gen Sifat dan karakter dari sebuah individu. Sebuah nilai yang menyatakan satuan dasar yang membentuk suatu arti tertentu. Gen bisa berupa nilai biner, float, integer maupun karakter.
Kromosom Gabungan gen-gen yang akan membentuk nilai tertentu, sering dikenal sebagai String Gen. Allele Nilai dari Gen (setting gen) Lokus Posisi gen dalam kromosom Individu Satu nilai yang menyatakan
salah satu solusi yang mungkin dari permasalahan yang diangkat.
5
Populasi Sekumpulan individu yang akan diproses bersama dalam satu siklus proses evolusi.
Generasi Menyatakan satu satuan siklus proses evolusi.
Nilai Fitness
Menyatakan seberapa baik nilai dari solusi yang didapatkan. Crossover Pertukaran string bit kromosom
antar parent.
Mutasi Perubahan nilai string bit kromosom.
Algoritme genetika dikarakterisasi berdasarkan lima komponen penting, yaitu: 1. Presentasi kromosom untuk memudahkan
penemuan solusi dalam masalah pengoptimasian.
2. Inisialisasi populasi yaitu pembentukan populasi awal.
3. Definisi nilai fitness yang merupakan ukuran baik tidaknya sebuah individu dalam proses evaluasi.
4. Proses menghasilkan populasi baru dari populasi yang ada melalui proses genetika (crossover dan mutasi ).
5. Parameter algoritme genetika seperti ukuran populasi, probabilitas proses genetika dan banyaknya generasi.
Salah satu konsep penting dalam algoritme genetika ialah hereditas (heredity), yaitu sebuah ide yang menyatakan bahwa sifat-sifat individu dapat dikodekan dengan cara tertentu sehingga sifat-sifat tersebut dapat diturunkan kepada generasi berikutnya. Konsep penting lainnya dalam teori evolusi yaitu fitness dan selection untuk proses reproduksi.
Fungsi fitness adalah fungsi yang digunakan untuk mengukur nilai kecocokan suatu kromosom. Nilai yang dihasilkan dari fungsi fitness disebut dengan nilai fitness. Nilai fitness ini akan menggambarkan seberapa baik solusi yang didapat.
Seleksi
Seleksi adalah proses memilih individu terbaik dari sebuah populasi dengan mencari kromosom dengan nilai fitness terbaik, dan kemudian dilanjutkan ke proses berikutnya yaitu crossover dan mutasi. Sementara itu, kromosom
yang memiliki nilai fitness yang rendah memiliki peluang yang kecil untuk dipilih bahkan tidak terpilih ke tahapan genetika selanjutnya (Klabankoh & Pinngern 1999).
Beberapa teknik yang dapat digunakan pada tahap seleksi adalah (Sivanandam & Deepa 2008):
1. Roulette-wheel Selection
Pada teknik Roulette-wheel selection, masing-masing kromosom menempati potongan lingkaran pada roda roulette secara proporsional sesuai dengan nilai fitness-nya. Kromosom yang memiliki nilai fitness lebih besar menempati potongan lingkaran yang lebih besar dibandingkan dengan kromosom bernilai fitness rendah (Sivanandam & Deepa 2008). Misal, terdapat empat kromosom yaitu K1, K2, K3, dan K4. Nilai fitness untuk masing-masing kromosom adalah 1, 2, 0.5, dan 0.5. Pertama, dibuat interval nilai kumulatif (dalam interval [0,1]) dari nilai fitness masing-masing kromosom dibagi total nilai fitness
dari semua kromosom. Gambar 4
mengilustrasikan teknik Roulette-wheel selection.
Gambar 4 Contoh penggunaan teknik Roulette-wheel selection
Sebuah kromosom akan terpilih jika bilangan random yang dibangkitkan berada dalam interval akumulatifnya. K2 dengan nilai fitness paling besar menempati potongan sebesar setengah lingkaran. Dengan demikian, K2 memiliki peluang sebesar 0.5 untuk terpilih sebagai orang tua.
2. Tournament Selection
Tournament selection merupakan teknik memilih individu terbaik dari suatu populasi. Berbeda dengan Roulette-wheel selection, teknik Tournament selection melibatkan beberapa turnamen diantara Nu individu yang dipilih secara
acak dari sebuah populasi. Individu terbaik yang terpilih sebagai pemenang dari sebuah turnamen adalah individu yang memiliki nilai fitness terbaik. Individu yang terpilih akan digunakan
Roulette-wheel selection
K1 [0 ; 0.25] K2 [0.25 ; 0.75] K3 [0.75 ; 0.875] K4 [0.875 ; 1]
6
untuk tahap genetika selanjutnya yaitu crossover. Tekanan seleksi mudah disesuaikan dengan mengubah ukuran turnamen. Jika ukuran turnamen lebih besar, maka individu-individu yang memiliki nilai fitness rendah memiliki peluang yang kecil untuk dipilih.
3. Rank Selection
Rank selection merupakan teknik memilih individu terbaik dari suatu populasi. Pertama, pilih dua individu terbaik secara acak. Jika suatu bilangan acak R yang dibangkitkan [0,1] kecil dari r, maka individu pertama dipilih sebagai orang tua. Jika bilangan acak R yang dibangkitkan besar sama dengan r, maka individu kedua dipilih sebagai orang tua. Langkah ini diulang hingga terpilih dua individu sebagai orang tua. Nilai r adalah sebuah parameter.
Crossover
Crossover merupakan proses
menggabungkan dua individu untuk memperoleh individu-individu baru yang diharapkan mempunyai nilai fitness yang lebih baik. Tidak semua pasangan induk akan mengalami crossover, banyaknya pasangan induk yang akan mengalami crossover akan ditentukan dengan nilai probabilitas crossover.
Teknik crossover dapat dilakukan dalam berbagai cara yaitu one point crossover atau yang disebut satu titik potong dan n-point crossover (Klabankoh & Pinngern 1999). Teknik one point crossover adalah teknik pencarian suatu titik potong yang dipilih secara acak, kemudian bagian pertama dari orang tua 1 digabungkan dengan bagian kedua dari orang tua 2. Teknik n-point crossover ialah dengan menukarkan semua gen diantara titik potong dengan induk.
Proses crossover dapat dilihat pada Gambar 5 dan Gambar 6.
Gambar 5 Proses Crossover satu titik potong (one point crossover)
Gambar 6 Proses Crossover dua titik potong (two point crossover).
Mutasi
Mutasi adalah proses penggantian gen dengan nilai inversinya. Gen 0 menjadi 1, dan gen 1 menjadi 0. Proses ini dilakukan secara acak pada posisi gen tertentu pada individu-individu yang terpilih untuk dimutasikan. Banyaknya individu yang mengalami mutasi ditentukan oleh besarnya probabilitas mutasi (Michalewicz 1996). Proses mutasi dapat dilihat pada Gambar 7.
Gambar 7 Proses Mutasi Elitisme
Elitisme adalah suatu teknik yang dilakukan untuk mempertahankan suatu individu terbaik yang memiliki nilai fitness tertinggi agar tidak mengalami kerusakan yang diakibatkan oleh proses genetika, sehingga individu tersebut dapat bertahan hidup untuk generasi selanjutnya (Michalewicz 1996).
Kromosom-kromosom yang memiliki nilai fitness yang baik akan memiliki peluang yang lebih tinggi untuk terseleksi. Setelah dilakukan beberapa kali proses generasi, algoritme genetika akan menunjukkan kromosom-kromosom terbaik, yang diharapkan merupakan solusi optimal atau mendekati optimal dari masalah yang dihadapi.
METODE PENELITIAN
Penelitian ini dikerjakan dalam beberapa tahap, yaitu: pengumpulan data, praproses, proses cross validation, klasifikasi data menggunakan metode SVM, dan optimisasi parameter SVM
7
menggunakan algoritme genetika. Tahapan yang dilakukan secara garis besar dapat dilihat pada Gambar 8.
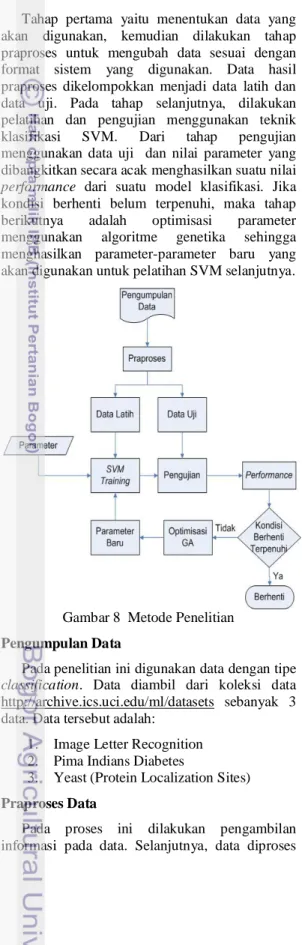
Tahap pertama yaitu menentukan data yang akan digunakan, kemudian dilakukan tahap praproses untuk mengubah data sesuai dengan format sistem yang digunakan. Data hasil praproses dikelompokkan menjadi data latih dan data uji. Pada tahap selanjutnya, dilakukan pelatihan dan pengujian menggunakan teknik klasifikasi SVM. Dari tahap pengujian menggunakan data uji dan nilai parameter yang dibangkitkan secara acak menghasilkan suatu nilai performance dari suatu model klasifikasi. Jika kondisi berhenti belum terpenuhi, maka tahap berikutnya adalah optimisasi parameter menggunakan algoritme genetika sehingga menghasilkan parameter-parameter baru yang akan digunakan untuk pelatihan SVM selanjutnya.
Gambar 8 Metode Penelitian Pengumpulan Data
Pada penelitian ini digunakan data dengan tipe classification. Data diambil dari koleksi data http://archive.ics.uci.edu/ml/datasets sebanyak 3 data. Data tersebut adalah:
1. Image Letter Recognition 2. Pima Indians Diabetes
3. Yeast (Protein Localization Sites) Praproses Data
Pada proses ini dilakukan pengambilan informasi pada data. Selanjutnya, data diproses
agar data sesuai dengan format sistem yang digunakan.
Data Latih dan Data Uji
Berdasarkan metode k-fold cross validation, untuk masing-masing data hasil tahapan praproses dibagi menjadi k subset, yaitu S1, S2,…, Sk. Pada penelitian ini ditentukan nilai k sebesar 5. Masing-masing subset memiliki ukuran yang sama. Pembagian data dilakukan secara acak dengan mempertahankan perbandingan jumlah baris data setiap kelas. Pada proses pertama S2,…, S5 dijadikan data pelatihan dan S1 sebagai data uji, pada proses kedua S1,S3,…, S5 sebagai data pelatihan dan S2 sebagai data uji, dan seterusnya sebanyak 5 kali pengulangan.
SVM Training dan Testing
Menggunakan data latih yang diperoleh dari metode k-fold cross validation, dilakukan proses pelatihan pengklasifikasian berdasarkan metode SVM menggunakan fungsi Linear Kernel, Polynomial Kernel, Radial Basis Function (RBF) Kernel dan Sigmoid Kernel. Pada proses ini akan dibentuk n model klasifikasi SVM berdasarkan banyaknya kelas pada data yang digunakan. Dari fungsi kernel yang digunakan dibutuhkan nilai parameter C untuk fungsi Linear Kernel, nilai parameter C, γ, r, dan d untuk fungsi Polynomial Kernel, dan parameter C dan γ untuk fungsi kernel RBF. Sedangkan untuk fungsi Sigmoid kernel dibutuhkan nilai parameter C, γ, dan r.
Pada tahap selanjutnya, dengan menggunakan data uji, dilakukan pengujian model klasifikasi yang telah dibentuk pada proses sebelumnya. Pada proses pengujian SVM, dihasilkan suatu performance berupa akurasi model klasifikasi pada nilai parameter kernel tertentu. Nilai performance ini merupakan fungsi objektif dari parameter SVM yang digunakan dalam pengklasifikasian data.
Optimisasi Parameter SVM
Tahapan dalam algoritme genetika dimulai dengan menginisialisasikan sebuah populasi dengan N kromosom. Gen-gen yang mengisi masing-masing kromosom dibangun secara acak. Pada penelitian ini, panjang kromosom atau jumlah gen dalam suatu kromosom ditentukan oleh banyaknya parameter kernel pada fungsi kernel SVM yang digunakan, dikalikan dengan jumlah bit yang digunakan untuk mengkodekan suatu parameter kernel. Banyaknya parameter
8
kernel SVM tergantung pada fungsi kernel yang digunakan pada pengklasifikasian data. Satu parameter SVM merupakan satu variabel pada algoritme genetika.
Populasi awal tersebut dievaluasi dengan menggunakan suatu fungsi yaitu fungsi fitness. Fungsi fitness ini bertujuan untuk menghitung nilai fitness dari suatu individu. Nilai fitness merupakan tingkat akurasi dari model klasifikasi SVM yang dipengaruhi oleh nilai parameter kernel tertentu yang dihasilkan dari proses optimisasi. Individu adalah hasil pendekodean sebuah kromosom yang berisi bilangan biner yang diubah menjadi bilangan real dalam nilai interval parameter kernel SVM dengan menggunakan persamaan berikut (Suyanto 2005):
= + ( − )
ra dan rb merepresentasikan batas atas dan batas bawah dari nilai parameter, sedangkan g merepresentasikan nilai desimal bit string. Nilai individu tersebut merupakan nilai parameter kernel. Misal terdapat tiga variabel, yaitu x1, x2, dan x3 yang dikodekan ke dalam sebuah
kromosom yang terdiri dari 9 gen. Masing-masing variabel dikodekan ke dalam 3 gen seperti berikut:
0 0 0 1 1 0 1 1 1
x1 x2 x3
Jika ketiga variabel dibatasi pada interval nilai tertentu, misalkan [-1,2], maka hasil pendekodean dari contoh di atas adalah:
= −1 + 2 − (−1) ( × 2 + × 2 + × 2 ) = −1 + 3(0 + 0 + 0) = −1 = −1 + (2 − (−1))( × 2 + × 2 + × 2 ) = −1 + 3(0,5 + 0,25 + 0) = 1,25 = −1 + 2 − (−1) ( × 2 + × 2 + × 2 ) = −1 + 3(0,5 + 0,25 + 0,125) = 1,625
Dengan demikian, diperoleh nilai desimal untuk variabel x1 -1, variabel x2 1.25, dan variabel x3
1.625. Variabel x1 yang bernilai -1 merupakan
nilai minimum suatu variabel yang nilainya sama dengan batas bawah nilai range variabel, dengan keseluruhan nilai gen sama dengan 0. Tiga gen pada variabel x3 bernilai sama dengan 1 yang
menghasilkan suatu nilai maksimum. Namun,
variabel x3 yang bernilai 1.625 tidak sama dengan
batas atas dari range yang telah ditentukan yaitu 2. Hal ini disebabkan nilai maksimum untuk tiga gen tersebut adalah kurang dari 1, sehingga nilai maksimum dari suatu variabel hanya mendekati suatu batas atas nilai range variabel. Nilai variabel yang telah diperoleh digunakan dalam tahap penglasifikasian pada metode klasifikasi SVM.
Untuk menjaga agar individu bernilai fitness tertinggi tidak hilang selama evolusi, maka perlu dilakukan prosedur elitisme, dengan cara membuat satu atau dua salinan individu terbaik dari populasi. Individu yang memiliki performance yang baik ini akan digunakan kembali pada proses genetika untuk menghasilkan populasi baru yang diharapkan menjadi populasi yang lebih baik dari sebelumnya. Proses genetika yang digunakan adalah pindah silang (crossover) dan mutasi.
Untuk proses genetika crossover, terlebih dahulu dilakukan tahapan penyeleksian terhadap seluruh kromosom dalam populasi. Dari proses seleksi menghasilkan dua buah kromosom dengan rangking tertinggi berdasarkan rangking hasil evaluasi. Dua kromosom tersebut dipilih sebagai orang tua yang akan dipindah-silangkan. Metode seleksi yang digunakan adalah metode roulette-wheel selection. Pertama, dibuat interval nilai kumulatif (dalam interval [0,1]) dari nilai fitness masing-masing kromosom dibagi total nilai fitness dari semua kromosom. Sebuah kromosom akan terpilih jika bilangan random yang dibangkitkan berada dalam interval akumulatifnya.
Proses crossover menggunakan dua kromosom induk yang diperoleh dari proses seleksi. Dari proses memindah-silangkan dua buah kromosom induk ini diharapkan memperoleh sebuah kromosom yang mengarah pada solusi yang bagus. Metode crossover yang digunakan pada penelitian ini adalah pindah silang satu titik potong (one-point crossover). Suatu titik potong dipilih secara acak, kemudian bagian pertama dari orang tua 1 digabungkan dengan bagian kedua dari orang tua 2. Pindah silang hanya bisa dilakukan jika suatu bilangan acak [0,1) yang dibangkitkan kurang dari suatu probabilitas (pc) yang ditentukan.
Proses genetika selanjutnya ialah proses mutasi. Pada proses mutasi dilakukan perubahan nilai gen yang dinilai tidak baik dalam kromosom.
9
Perubahan nilai gen tersebut berdasarkan bilangan acak yang dibangkitkan kurang dari probabilitas mutasi (pmut) yang ditentukan, sehingga gen yang bersesuaian akan diganti dengan nilai kebalikan (nilai 0 diubah menjadi 1 dan 1 diubah menjadi 0). Nilai pmut diset sebagai 1/n, dimana n adalah jumlah gen dalam kromosom.
Proses generasi crossover dan mutasi akan menghasilkan N individu baru yang akan menggantikan N individu yang lama dalam satu populasi. Penggantian populasi ini disebut generation replacement. Hasil populasi baru yang terbentuk akan dievaluasi kembali untuk mengetahui tingkat akurasi klasifikasi SVM berikutnya, dengan menggunakan nilai parameter yang baru hasil optimisasi dan diseleksi untuk mengetahui individu atau parameter kernel yang memiliki performance terbaik selanjutnya. Demikian seterusnya, hingga terpenuhi kriteria pemberhentian algoritme genetika.
Lingkungan Pengembangan Sistem
Spesifikasi perangkat keras dan perangkat lunak komputer yang digunakan dalam penelitian ini sebagai berikut :
Perangkat keras : Prosesor Intel Atom 1,66 GHz, Memori 1 GB, hard disk 80 GB, mouse. Perangkat lunak : Windows XP, Matlab 7.7.1, Notepad++ , Visual Studio 2008, Microsoft Excel.
HASIL DAN PEMBAHASAN Data
Beberapa informasi dari data yang digunakan pada penelitian disajikan dalam tabel berikut.
Tabel 2 Jumlah instance, attribute, class dan missing values pada data Image Letter Recognition, Pima Indians Diabetes, dan Yeast (Protein Localization Sites)
Data Instance
(baris) Attribute class
Missing values
Letter 20.000 17 26 None
Diabetes 768 9 2 None
Yeast 1484 9 10 None
Tabel 3 Informasi jumlah data untuk tiap kelas pada data Image Letter Recognition
Data Kelas Jumlah (baris) Kelas Jumlah (baris) Letter A B C D E F G H I J K L M 789 766 736 805 768 775 773 734 755 747 739 761 792 N O P Q R S T U V W X Y Z 783 753 803 783 758 748 796 813 764 752 787 786 734
Tabel 4 Informasi jumlah data untuk tiap kelas pada data Pima Indians Diabetes
Data Kelas Jumlah
(baris)
Diabetes 0
1
500 268 Tabel 5 Informasi jumlah data untuk tiap kelas
pada data Yeast
Data Kelas Jumlah
(baris) Yeast CYT NUC MIT ME3 ME2 ME1 EXC VAC POX ERL 463 429 244 163 51 44 37 30 20 5 Praproses
Sebelum diklasifikasikan menggunakan metode SVM, ketiga data dengan atribut bertipe kategori diubah terlebih dahulu menjadi data bertipe integer seperti, atribut kelas pada data Image Letter Recognition dan Yeast. Untuk data Letter, atribut kelas yang bertipe kategori A sampai Z diubah menjadi bertipe integer 1 sampai 26. Begitu juga dengan data Yeast, atribut kelas yang bertipe kategori diubah menjadi data bertipe integer yang diurutkan dari 1 sampai 10 sesuai
10
urutan kelas Yeast pada Tabel 5. Selain itu, juga dilakukan eliminasi terhadap kelas data yang dianggap tidak terlalu berpengaruh terhadap hasil klasifikasi. Untuk data Yeast kelas ERL yang hanya memiliki instance sebanyak 5 baris tidak digunakan dalam penelitian karena jumlah instance pada kelas tersebut yang sangat sedikit, sehingga jumlah instance data Yeast yang digunakan untuk penelitian ini adalah sebanyak 1479 baris dengan jumlah kelas data yaitu 9 kelas. Tabel 6 Kode kelas pada data Yeast
Data Kelas Kode Kelas
Yeast NUC CYT
MIT ME3 ME2 ME1 EXC VAC POX 1 2 3 4 5 6 7 8 9
Data Latih dan Data Uji
Keseluruhan data hasil tahapan praproses dibagi secara acak ke dalam 5 subset untuk masing-masing kelas pada data. Untuk data Pima Indians Diabetes yaitu 154 baris data dan data Yeast terdapat 297 baris data untuk masing-masing fold. Subset-subset tersebut digunakan sebagai data latih dan data uji sesuai dengan metode validasi silang, yaitu metode 5-fold cross validation. Namun, untuk data Image Letter Recognition dilakukan pengecualian karena jumlah baris data yang sudah cukup besar, sehingga dilakukan pemisahan data sebanyak 75% untuk data latih dan 25% untuk data uji.
Klasifikasi
Pada proses pelatihan SVM untuk fungsi Linear Kernel, Polynomial Kernel, Radial Basis Function (RBF) Kernel dan Sigmoid Kernel, dibutuhkan parameter C, γ, r, dan d. Parameter C untuk fungsi Linear Kernel, parameter C, γ, r, dan d untuk fungsi Polynomial Kernel, dan parameter C dan γ untuk fungsi kernel RBF. Sedangkan untuk fungsi Sigmoid kernel dibutuhkan nilai parameter C, γ, dan r. Proses pelatihan SVM
menggunakan library LIBSVM yang
dikembangkan oleh mahasiswa Ilmu Komputer National Taiwan University, Chih-Yuan Yang dan Chih-Huai Cheng.
Pada tahap awal, penelitian ini melakukan klasifikasi terhadap data Image Letter Recognition, Pima Indians Diabetes, dan Yeast dengan keempat fungsi Kernel dengan meng-input-kan nilai parameter yang sudah ditentukan. Dari pengklasifikasian data dengan SVM menghasilkan suatu tingkat akurasi dengan menggunakan metode 5-fold cross validation.
Pada metode 5-fold cross validation, dilakukan proses pelatihan dan proses pengujian terhadap data latih dan data uji. Proses pelatihan dan pengujian ini bertujuan untuk membangun model klasifikasi dan menghitung tingkat akurasi SVM dalam memprediksi data uji. Akurasi adalah perbandingan jumlah data yang telah diprediksi benar terhadap jumlah data uji. Rataan akurasi adalah nilai rata-rata dari akurasi di setiap nilai parameter yang digunakan. Tabel 7 menunjukkan nilai akurasi dari proses pengujian dengan nilai parameter C, γ, r, dan d yang sudah ditentukan pada data Image Letter Recognition, Pima Indians Diabetes, dan Yeast sebanyak lima kali pengujian untuk masing-masing kernel.
Optimisasi Parameter SVM
Pada tahap ini, dilakukan pengklasifikasian terhadap data Image Letter Recognition, Pima Indians Diabetes, dan Protein Localization Sites serta penghitungan tingkat akurasi SVM dengan membangkitkan nilai parameter C, γ, r, dan d secara otomatis dan acak menggunakan algoritme genetika.
Pada penelitian ini, jumlah kromosom dibatasi sebanyak 30 kromosom dan banyaknya generasi juga dibatasi tidak lebih dari 50 generasi, karena pertimbangan waktu eksekusi program. Panjang kromosom atau jumlah gen dalam suatu kromosom ditentukan oleh banyaknya parameter kernel pada fungsi kernel SVM yang digunakan, dikalikan dengan jumlah bit, dimana jumlah bit yang digunakan sebanyak 10 bit, sehingga panjang kromosom untuk Linear Kernel, Polynomial Kernel, RBF, dan Sigmoid Kernel masing-masingnya adalah 10, 40, 20, dan 30 bit.
11
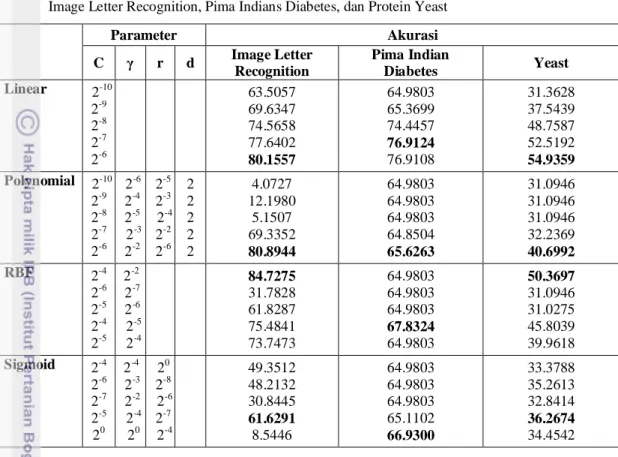
Tabel 7 Akurasi hasil proses pengujian dengan nilai parameter C, γ, r, dan d yang ditentukan pada data Image Letter Recognition, Pima Indians Diabetes, dan Protein Yeast
Parameter Akurasi C γ r d Image Letter Recognition Pima Indian Diabetes Yeast Linear 2-10 2-9 2-8 2-7 2-6 63.5057 69.6347 74.5658 77.6402 80.1557 64.9803 65.3699 74.4457 76.9124 76.9108 31.3628 37.5439 48.7587 52.5192 54.9359 Polynomial 2-10 2-9 2-8 2-7 2-6 2-6 2-4 2-5 2-3 2-2 2-5 2-3 2-4 2-2 2-6 2 2 2 2 2 4.0727 12.1980 5.1507 69.3352 80.8944 64.9803 64.9803 64.9803 64.8504 65.6263 31.0946 31.0946 31.0946 32.2369 40.6992 RBF 2-4 2-6 2-5 2-4 2-5 2-2 2-7 2-6 2-5 2-4 84.7275 31.7828 61.8287 75.4841 73.7473 64.9803 64.9803 64.9803 67.8324 64.9803 50.3697 31.0946 31.0275 45.8039 39.9618 Sigmoid 2-4 2-6 2-7 2-5 20 2-4 2-3 2-2 2-4 20 20 2-8 2-6 2-7 2-4 49.3512 48.2132 30.8445 61.6291 8.5446 64.9803 64.9803 64.9803 65.1102 66.9300 33.3788 35.2613 32.8414 36.2674 34.4542
Untuk penelitian ini ditentukan nilai probabilitas crossover sebesar 0.8, dan probabilitas mutasi juga sangat kecil, sekitar 1 dibagi dengan jumlah gen. Untuk pencarian nilai parameter C, γ, dan r dibatasi pada range 2e-10 sampai 2e0, sedangkan parameter d dibatasi pada range 2 sampai 5.
Proses penghitungan tingkat akurasi akan berhenti jika pada 10 generasi berturut-turut memiliki selisih nilai akurasi yang kecil sama dengan 0.1. Hal ini berarti, dengan kenaikan tingkat akurasi sebesar 0.1 pada suatu generasi akan dianggap sama dengan nilai akurasi pada generasi sebelumnya.
Berikut adalah nilai fitnes terbaik untuk klasifikasi SVM, yang diperoleh menggunakan algoritme genetika beserta nilai parameter-parameter kernel.
1. Image Letter recognition
Dari eksekusi program menggunakan data Image Letter Recognition, secara Linear optimum dengan akurasi 84.59% dan parameter C senilai
0.8741. Pada Polynomial kernel menghasilkan nilai parameter C, γ, r dan d masing-masing sebesar 0.4107, 0.2702, 0.7365, dan 3.6933 dengan tingkat akurasi mencapai 96.71%. Dari eksekusi program secara RBF menghasilkan nilai akurasi paling tinggi mencapai 97.05% dengan parameter C dan γ sebesar 0.9668
dan
0.3248. Dengan Sigmoid kernel menghasilkan nilai parameter C, γ, dan r masing-masing 0.9990, 0.0156, dan 0.0058 yang mengoptimumkan nilai fitness yang mencapai 78.76%. Kenaikan nilai fitness dapat dilihat pada Gambar 9 serta fitness terbaik untuk setiap kernel dapat dilihat pada Tabel 8.Tabel 8 Nilai fitness terbaik data Letter secara Linear, Polynomial, RBF, dan Sigmoid
Kernel Fitnes terbaik Linear Polynomial RBF Sigmoid 84.59% 96.71% 97.05% 78.76%
12
Gambar 9 Fitness terbaik untuk setiap generasi data Image Letter Recognition secara Linear, Polynomial, RBF, dan Sigmoid
2. Pima Indians Diabetes
Dari eksekusi program menggunakan data Pima Indians Diabetes, secara Linear menghasilkan akurasi sebesar 77.30% dengan parameter C senilai 0.0966. Eksekusi program dengan Polynomial kernel menghasilkan nilai akurasi tertinggi mencapai 78.21% dengan nilai parameter C, γ, r dan d masing-masing 0.7102, 0.0380, 0.4936, dan 2.601. Dengan RBF kernel menghasilkan akurasi sebesar 78.08% dan parameter C dan γ yang diperoleh senilai 0.5941 dan 0.0448, sedangkan hasil eksekusi dengan Sigmoid optimum pada akurasi 77.17% dengan nilai parameter C, γ, dan r masing-masing 0.224, 0.037, dan 0.006. Kenaikan nilai fitness dapat dilihat pada Gambar 10 serta fitness terbaik untuk setiap kernel dapat dilihat pada Tabel 9.
Gambar 10 Fitness terbaik untuk setiap generasi data Pima Indians Diabetes secara Linear, Polynomial, RBF, dan Sigmoid
Tabel 9 Nilai fitness terbaik data Diabetes secara Linear, Polynomial, RBF, dan Sigmoid
Kernel Fitnes terbaik Linear Polynomial RBF Sigmoid 77.30% 78.21% 78.08% 77.17%
3. Yeast (Protein Localization Sites)
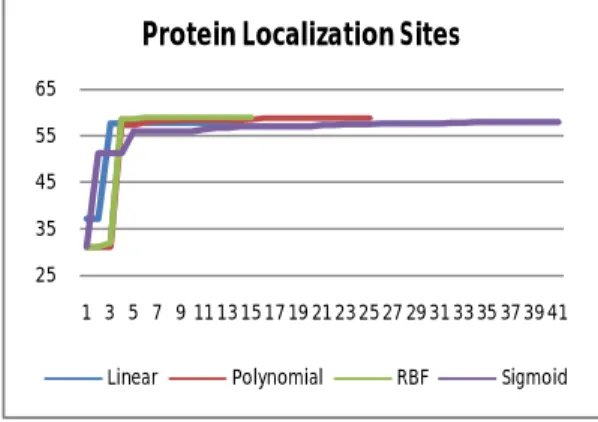
Dari eksekusi program menggunakan data Yeast, secara Linear menghasilkan akurasi sebesar 57.82% dengan parameter C senilai 0.1121. Eksekusi program dengan Polynomial kernel nilai akurasi mencapai 58.83% dengan parameter C, γ, r dan d masing-masing senilai 0.981, 0.041, 0.910, dan 3.529. Dengan RBF kernel menghasilkan nilai akurasi tertinggi mencapai 58.97% dan parameter C dan γ yang diperoleh senilai 0.6839 dan 0.0517. Hasil eksekusi dengan Sigmoid kernel, optimum pada akurasi 57.96% dengan parameter C, γ, dan r masing-masing senilai 0.8263, 0.0605, dan 0.0498. Kenaikan nilai fitness dapat dilihat pada Gambar 11 serta fitness terbaik untuk setiap kernel dapat dilihat pada Tabel 10.
Gambar 11 Fitness terbaik untuk setiap generasi data Yeast secara Linear, Polynomial, RBF, dan Sigmoid
Tabel 10 Nilai fitness terbaik data Yeast secara Linear, Polynomial, RBF, dan Sigmoid
Kernel Fitnes terbaik Linear Polynomial RBF Sigmoid 57.82% 58.83% 58.97% 57.96% 50 60 70 80 90 100 1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 Letter Recognation
Linear Polynomial RBF Sigmoid
60 65 70 75 80 1 3 5 7 9 11 13 15 17 19 21 23 25 27 Pima Indians Diabetes
Linear Polynomial RBF Sigmoid
25 35 45 55 65 1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35 37 39 41 Protein Localization Sites
13
Berdasarkan akurasi yang diperoleh pada data Yeast, dilakukan penelusuran terhadap hasil klasifikasi untuk tiap kelas data. Berikut merupakan hasil klasifikasi SVM menggunakan nilai parameter hasil optimasi algoritme genetika. Menglasifikasikan data Yeast menggunakan RBF kernel dengan nilai parameter C dan γ masing-masing adalah 0.6839 dan 0.0517.
Tabel 11 Hasil klasifikasi tiap kelas pada data Yeast menggunakan RBF kernel
Kls asal Hasil Klasifikasi 1 2 3 4 5 6 7 8 9 kls1 326 97 33 5 2 0 0 0 0 kls2 187 207 21 11 1 0 2 0 0 kls3 74 15 139 7 5 1 0 0 3 kls4 15 14 4 129 1 0 0 0 0 kls5 8 4 4 6 18 7 4 0 0 kls6 0 1 1 1 5 31 5 0 0 kls7 5 1 3 0 2 5 19 0 0 kls8 15 3 2 6 2 0 2 0 0 kls9 6 0 3 0 1 1 0 0 9
Tabel 12 Akurasi yang diperoleh untuk tiap kelas
Kelas Akurasi Kelas Akurasi
1 (CYT) 70.41% 6 (ME1) 70.45%
2 (NUC) 48.25% 7 (EXC) 54.28%
3 (MIT) 56.96% 8 (VAC) 0%
4 (ME3) 79.14% 9 (POX) 45%
5 (ME2) 35.29%
Dari nilai akurasi tiap kelas yang diperoleh, dapat dilihat bahwa kelas yang dapat diklasifikasikan dengan baik adalah kelas data ME3, ME1, dan CYT dengan akurasi masing-masing sebesar 79.14%, 70.45%, dan 70.41%, sedangkan untuk kelas data NUC, MIT, ME2, EXC, dan POX, akurasi pengklasifikasiannya hanya berkisar antara 30-60%. Untuk kelas data VAC tidak satupun data yang terklasifikasikan
dengan benar. Hal ini disebabkan karena persebaran data untuk kelas VAC yang tidak bagus sehingga sulit untuk melakukan pengelompokan.
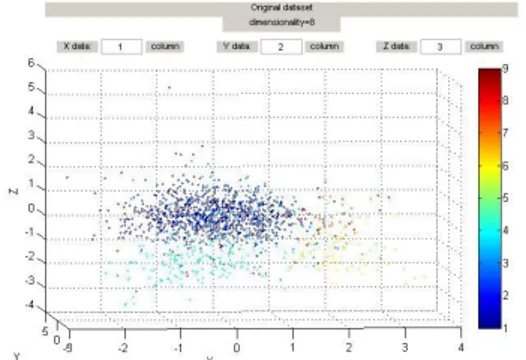
Untuk mengetahui persebaran data Yeast, pada penelitian ini digunakan analisis Multidimensional scaling.
Gambar 12 Persebaran data Yeast menggunakan Multidimensional scaling
Dari hasil multidimensional scaling dapat dilihat persebaran data untuk kelas 4 (ME3) dan data kelas 6 (ME1) yaitu mengelompok dan cukup terpisah dengan kelas data yang lain, sehingga kelas data ini dapat diklasifikasikan dengan baik dan menghasilkan nilai akurasi yang cukup bagus. Begitupun pada data kelas 1, meskipun terlihat memiliki jarak yang dekat dengan kelas yang lain, karena persebaran data kelas 1 bersifat mengelompok sehingga memungkinkan memperoleh akurasi yang bagus, sedangkan pada data kelas 8 (VAC) persebaran datanya cukup menyebar sehingga jarak antar data cukup jauh, selain itu persebaran data cukup dekat dengan data kelas yang lain, hal ini menimbulkan kesulitan dalam melakukan pengelompokan data sehingga akurasi yang dihasilkan kecil. Dari kelas data yang sulit diklasifikasikan seperti kelas 8 (VAC) inilah yang menyebabkan tingkat akurasi klasifikasi data Yeast menjadi kecil.
Berdasarkan hasil eksekusi pencarian nilai parameter SVM menggunakan algoritme genetika, terjadi peningkatan nilai akurasi pada tiga data yang digunakan. Tabel 13 menunjukkan besarnya peningkatan nilai akurasi.
Tabel 13 Besarnya kenaikan nilai fitness pada
optimisasi parameter SVM
14
Kernel Letter Diabetes YeastLinier 14.95% 11.93% 20.55%
Polynomial 36.37% 11.41% 27.74%
RBF 29.97% 13.10% 27.87%
Sigmoid 17.73% 12.19% 26.60%
Jika dibandingkan dengan pengklasifikasian ke-3 data secara SVM biasa dengan peng-input-an nilai parameter secara sembarang, maka hasil dari pencarian nilai parameter SVM yang mengoptimumkan nilai fitnes dengan menggunakan algoritme genetika menunjukkan kenaikan tingkat akurasi. Hal ini berarti, meningkatnya ketepatan dalam melakukan klasifikasi data. Tabel 14 menunjukkan perbandingan nilai akurasi yang melakukan optimisasi parameter dengan akurasi dari peng-input-an nilai parameter yang sudah ditentukan. Perbandingan nilai akurasi ini, dipetakan dalam sebuah grafik, Gambar 13 untuk data Image Letter Recognition, Gambar 14 untuk data Pima Indians Diabetes, dan Gambar 15 untuk data Yeast.
Gambar 13 Perbandingan nilai akurasi setiap kernel untuk data Image Letter Recognition
Gambar 14 Perbandingan nilai akurasi setiap kernel untuk data Pima Indians Diabetes
Gambar 15 Perbandingan nilai akurasi setiap kernel untuk data Yeast
Selain itu, juga dilakukan perbandingan nilai fitnes dengan jurnal yang juga menggunakan data Image Letter Recognition, Pima Indian Diabetes, dan Protein Localization Sites tersebut dalam proses klasifikasi. Untuk data Image Letter Recognition pada jurnal yang ditulis oleh Petter W. Frey dan David J. Slate (1991) melakukan penglasifikasian data Letter dengan menggunakan sistem klasifikasi Holland-Style. Dari penelitian tersebut, mereka menghasilkan nilai akurasi tertinggi sebesar 82.7%, sedangkan menggunakan optimisasi algoritme mencapai akurasi tertinggi sebesar 97.05% pada penglasifikasian data Letter menggunakan kernel RBF. Artinya, tingkat akurasi mengalami kenaikan sebesar 14.35%.
Pada data Pima Indians Diabetes juga dilakukan penelitian oleh Jack W. Smith, BS et al (1988). Dalam penelitian itu, mereka melakukan ramalan terhadap serangan Diabetes Mellitus dengan menggunakan algoritme pembelajaran ADAP yaitu model dari neural network. Pada penelitian tersebut diperoleh kesimpulan bahwa prediksi terhadap serangan Diabetes Mellitus dengan gejala terjadinya kelemahan katahanan tubuh terhadap penyakit influensa selama 5 tahun adalah sebesar 76%. Jika dibandingkan dengan penelitian ini, maka terjadi peningkatan nilai akurasi sebesar 2.21% menjadi 78.21%.
0 20 40 60 80 100
Linear Polynomial RBF Sigmoid Letter Recognation
Tanpa optimisasi Dengan Optimisasi
55 60 65 70 75 80
Linear Polynomial RBF Sigmoid Pima Indians Diabetes
Tanpa optimisasi Dengan Optimisasi
0 20 40 60 80
Linear Polynomial RBF Sigmoid Protein Localization Sites
15
Tabel 14 Perbandingan nilai akurasi yang melakukan optimisasi parameter dengan akurasi peng-input-an nilai parameter yang ditentukan
Kernel Letter optimasi Letter tanpa optimasi Kenaikan (%) Diabetes optimasi Diabetes tanpa optimasi Kenaikan (%) Yeast optimasi Yeast tanpa optimasi Kenaikan (%) Linear 84.5877 80.1557 4.432 77.3021 76.9124 0.3897 57.8239 34.9359 22.888 Poly 96.7059 80.8944 15.8115 78.2086 65.6263 12.5823 58.8310 40.6992 18.1318 RBF 97.0453 84.7275 12.3178 78.0796 67.8324 10.2472 58.9650 50.3697 8.5953 Sigmoid 78.7582 61.6291 17.1291 77.1713 66.9300 10.2413 57.9586 36.2674 21.6912
Paul Horton dan Kenta Nakai (1996) melakukan penelitian untuk memprediksi hubungan lokalisasi tempat terjadinya suatu pertemuan antar sel-sel protein dengan menggunakan sistem klasifikasi probabilitas. Dari data Yeast Protein diklasifikasikan ke dalam 10 kelas dengan pencapaian akurasi sebesar 55%. Nilai akurasi tersebut lebih kecil 3,97% dibandingkan dengan optimasi parameter klasifikas SVM menggunakan algoritme genetika yang menghasilkan nilai akurasi sebesar 58,97%.
Dasil perbandingan nilai akurasi yang diperoleh menunjukkan bahwa pencarian nilai parameter SVM menggunakan algoritme genetika menghasilkan nilai akurasi yang lebih besar. Hal ini, membuktikan bahwa optimisasi parameter SVM menggunakan algoritme genetika berhasil dalam pencapaian akurasi yang optimal.
KESIMPULAN DAN SARAN Kesimpulan
Hasil penelitian menunjukkan bahwa nilai akurasi dengan pencarian nilai parameter SVM secara sistematis menggunakan algoritme genetika menghasilkan suatu nilai akurasi yang lebih baik dibandingkan dengan nilai akurasi hasil eksekusi SVM tanpa optimasi dengan peningkatan akurasi terbesar yaitu 22.89%. Begitu juga dengan hasil penelitian sebelumnya yang juga melakukan klasifikasi pada data yang sama, optimisasi parameter SVM yang sistematis menggunakan algoritme genetika memperoleh nilai akurasi lebih tinggi mencapai 14.35% pada data Letter, 2.21% pada data Diabetes, dan 3.97% pada data Yeast. Dengan demikian, algoritme genetika berhasil melakukan pencarian nilai parameter SVM yang mengoptimalkan nilai akurasi pada metode klasifikasi SVM. Hal ini membuktikan bahwa
algoritme genetika efektif dalam meningkatkan nilai akurasi suatu model klasifikasi pada metode klasifikasi SVM.
Algoritme genetika sangat efektif dan efisien untuk data yang berukuran kecil. Namun, waktu eksekusi algoritme genetika akan semakin meningkat dengan meningkatnya ukuran populasi, generasi, range nilai parameter serta ukuran data. Hal ini menyebabkan lamanya proses eksekusi, sehingga kurang efisien jika dibandingkan dengan kenaikan nilai akurasi.
Saran
Pada penelitian ini masih terdapat kekurangan yang dapat diperbaiki pada penelitian selanjutnya. Untuk pengembangan selanjutnya, penulis menyarankan untuk melakukan eksekusi program secara paralel karena dapat mempercepat waktu eksekusi. Selain itu, jumlah populasi, kromosom, dan generasi juga dapat ditingkatkan untuk menghasilkan nilai akurasi yang lebih baik. Penulis juga menyarankan untuk melakukan teknik pengelompokan bertingkat untuk data yang persebarannya kurang bagus.
DAFTAR PUSTAKA
Damasevicius R. 2008. Optimization of SVM Parameters for Promoter Recognition in DNA Sequences. 20th EURO Mini Conference “Continuous Optimization and Knowledge-Based Technologies”. Neringa, Lithuania, 20-23 Mei, 2008.
Fu L. 1994. Neural Network In Computer Intelligence. Singapura : Mcgraw Hill. Frey PW, Slate DJ. 1991. Letter Recognition
16
http://www.springerlink.com/content/x83328 826p16u32u/fulltext.pdf [9 Agustus 2010]. Goldberg, David E. 1989. Genetic Algorithms in
Search, Optimization, and Machine Learning. England : Addison-Wesley Publishing Company.
Groenen P.J.F, Velden M. 2004.
Multidimensional scaling.
http://publishing.eur.nl/ir/repub/asset/1274/ei 200415.pdf [30 Oktober 2010]
Han J, Kamber M. 2006. Data Mining : Concepts and Techniques. USA : Morgan Kaufman Publishers.
Horton P, Nakai K. 1996. A Probabilistic Classification System for Predicting the Cellular Localization Sites of Proteins. http://www.aaai.org/Papers/ISMB/1996/ISM B96-012.pdf [9 Agustus 2010].
Kerami D, Murfi H. 2004. Kajian Kemampuan Generalisasi SVM dalam Pengenalan Jenis Splice Sites pada Barisan DNA. Makara, Sains. Volume 8 No. 3. Hal: 89-95.
Klabbankoh B, Pinngern O. 1999. Applied Genetic Algorithms in Information Retrieval. Bangkok: Faculty of Information Technology King Mongkut, Institute of Technology Ladkrabang.
Michalewicz Z. 1996. Genetic Algorithms + Data Structures = Evolution Programs. USA : Department of Computer Science, University of North Carolina.
Noorniawati VY. 2007. Metode Support Vector Machine untuk Klasifikasi pada Sistem Temu Kembali Citra. Skripsi. Institud Pertanian Bogor.
Sembiring K. 2007. Penerapan Teknik Support Vector Machine Untuk Mendeteksi Instruksi Pada Jaringan. Skripsi. Institut Teknologi Bandung.
Sivanandam SN, Deepa SN. 2008. Introduction to Genetic Algorithm. New York : Springer Berlin Heidelberg.
Smith JW, et al. 1988. Using the ADAP Learning Algorithm to Forecast the Onset of Diabetes Mellitus.
http://www.ncbi.nlm.nih.gov/pmc/articles/P MC2245318/pdf/procascamc00018-0276.pdf [9 Agustus 2010].
Suyanto. 2005. Algoritma Genetika dalam MATLAB. Penerbit ANDI. Yogyakarta. Vapnik V, Cortes C. 1995. Support-Vector
Networks.
http://www.springerlink.com/content/w08253 ul7m3780v8/fulltext.pdf [29 Januari 2010].
17
18
Lampiran 1 Hasil optimisasi data Image Letter Recognition untuk Linear kernel
Generasi Akurasi (%) Selisih Parameter C Waktu
1 69,63466 0 0,001946 7.12 jam 2 69,63466 0 0,001946 3 69,63466 14.93312 0,001946 4 84,56778 0 0,879025 5 84,56778 0 0,879025 6 84,56778 0 0,879025 7 84,56778 0.01996 0,879025 8 84,58774 0 0,872195 9 84,58774 0 0,872195 10 84,58774 0 0,872195 11 84,58774 0 0,85561 12 84,58774 0 0,872195 13 84,58774 0,874146
19
Lampiran 2 Hasil optimisasi data Image Letter Recognition untuk Polynomial kernel
Generasi Akurasi Selisih Parameter Waktu
C γ r d 1 60,3314 0 0,024243 0,029269 0,022144 1,429688 12.6 jam 2 60,3314 2.4955 0,024243 0,029269 0,022144 1,429688 3 62,82691 33.519 0,030481 0,029772 0,003963 1,847656 4 96,34658 0.0999 0,626342 0,170732 0,651708 4,894531 5 96,4464 0.1197 0,14244 0,430244 0,699512 3,992188 6 96,56618 0 0,144391 0,367805 0,600976 3,746094 7 96,56618 0.0998 0,144391 0,367805 0,600976 3,746094 8 96,666 0 0,144391 0,305367 0,599025 3,740234 9 96,666 0 0,144391 0,305367 0,599025 3,740234 10 96,666 0 0,144391 0,305367 0,599025 3,740234 11 96,666 0 0,144391 0,305367 0,599025 3,740234 12 96,666 0 0,144391 0,305367 0,599025 3,708008 13 96,666 0.0399 0,144391 0,305367 0,599025 3,552734 14 96,70593 0 0,410732 0,270245 0,736586 3,693359 15 96,70593 0 0,410732 0,270245 0,736586 3,693359 16 96,70593 0 0,410732 0,270245 0,736586 3,505859 17 96,70593 0,410732 0,270245 0,736586 3,693359
20
Lampiran 3 Hasil optimisasi data Image Letter Recognition untuk RBF kernel
Generasi Akurasi Selisih Parameter Waktu
C γ 1 67,07926 0 0,028382 0,028826 5.5 jam 2 67,07926 0.31942 0,028382 0,028826 3 67,39868 29.60671 0,030097 0,02853 4 97,00539 0.01996 0,993171 0,340488 5 97,02535 0 0,963902 0,324879 6 97,02535 0 0,963902 0,324879 7 97,02535 0 0,963902 0,324879 8 97,02535 0 0,963902 0,324879 9 97,02535 0.01997 0,962927 0,324879 10 97,04532 0 0,970732 0,324879 11 97,04532 0 0,966829 0,324879 12 97,04532 0 0,966829 0,324879 13 97,04532 0,966829 0,324879
21
Lampiran 4 Hasil optimisasi data Image Letter Recognition untuk Sigmoid kernel
Generasi Akurasi Selisih Parameter Waktu
C γ r 1 61,03015 0,778599 0,214635 0,030245 0,713171 33.09 jam 2 61,80874 0 0,214635 0,030245 0,630244 3 61,80874 8,963865 0,214635 0,030245 0,630244 4 70,77261 0 0,558049 0,02244 0,200976 5 70,77261 4,931124 0,558049 0,02244 0,200976 6 75,70373 0,119784 0,745366 0,014635 0,200976 7 75,82352 0 0,853659 0,00683 0,154147 8 75,82352 0 0,853659 0,00683 0,154147 9 75,82352 1,437413 0,853659 0,00683 0,154147 10 77,26093 0 0,932683 0,012684 0,137562 11 77,26093 0,698742 0,932683 0,012684 0,137562 12 77,95967 0,039928 0,932683 0,010733 0,029269 13 77,9996 0,079856 0,924878 0,010733 0,015611 14 78,07946 0 0,936585 0,010733 0,021464 15 78,07946 0,279497 0,932683 0,010733 0,013659 16 78,35895 0 0,936585 0,011708 0,015611 17 78,35895 0 0,936585 0,011708 0,015611 18 78,35895 0,09982 0,936585 0,011708 0,015611 19 78,45877 0 0,936585 0,011708 0,007806 20 78,45877 0 0,936585 0,011708 0,004879 21 78,45877 0,179677 0,936585 0,011708 0,004879 22 78,63845 0 0,998049 0,011708 0,007806 23 78,63845 0 0,998049 0,011708 0,007806 24 78,63845 0 0,999024 0,011708 0,007806 25 78,63845 0,09982 0,999024 0,011708 0,007806 26 78,73827 0 0,998049 0,015611 0,005855 27 78,73827 0,019964 0,998049 0,015611 0,005855 28 78,75824 0 0,999024 0,015611 0,005855 29 78,75824 0 0,999024 0,015611 0,005855 30 78,75824 0 0,999024 0,015611 0,005855 31 78,75824 0,999024 0,015611 0,005855
22
Lampiran 5 Hasil optimisasi data Pima Indians Diabetes untuk Linear kernel
Generasi Akurasi (%) Selisih Parameter C Waktu
1 65,36992 0 0,001884 4.85 mnt 2 65,36992 11,80059 0,001887 3 77,17051 0 0,629269 4 77,17051 0 0,290732 5 77,17051 0 0,32683 6 77,17051 0 0,639025 7 77,17051 0 0,293659 8 77,17051 0 0,300488 9 77,17051 0 0,330732 10 77,17051 0,131546 0,72 11 77,30205 0 0,096586 12 77,30205 0 0,096586 13 77,30205 0 0,096586 14 77,30205 0 0,096586 15 77,30205 0 0,096586 16 77,30205 0 0,099513 17 77,30205 0 0,099513 18 77,30205 0 0,096586 19 77,30205 0 0,096586 20 77,30205 0,096586