OPTIMISASI PARAMETER SUPPORT VECTOR MACHINE
MENGGUNAKAN ALGORITME PARTICLE SWARM OPTIMIZATION
SANDY CAHYA GUMILAR
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
OPTIMISASI PARAMETER SUPPORT VECTOR MACHINE
MENGGUNAKAN ALGORITME PARTICLE SWARM OPTIMIZATION
SANDY CAHYA GUMILAR
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar
Sarjana Komputer pada
Departemen Ilmu Komputer
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
ABSTRACT
SANDY CAHYA GUMILAR. Optimizing support vector machine parameters using particle swarm optimization. Supervised by MUSHTHOFA.
Support vector machine (SVM) is a popular classification method which is known to have a robust generalization capability. SVM calculates the best linear separator on the input feature space according to the training data. To classify data which are non-linearly separable, SVM uses kernel tricks to transform the data into a linearly separable data on a higher dimension feature space. Kernel trick uses various kinds of kernel functions, such as: linear kernel, polynomial, radial basis function (RBF) and sigmoid. Each function has parameters which affect the accuracy of SVM classification. In this research, we propose a combination of the SVM algorithm and particle swarm optimization (PSO) to search for the kernel parameters with the best accuracy. We use data from UCI repository of machine learning database: Diabetes, Image Letter Recognition and Yeast. The results indicate that the combination of SVM and PSO is effective in improving the accuracy of classification. PSO has been shown to be effective in systematically finding optimal kernel parameters for SVM, as a replacement for randomly choosing kernel parameters. Best accuracy for each data has been improved from previous research: 78.26% for Diabetes, 97.07% for Letter and 58.21% for Yeast. However, for bigger data sizes, this method may become impractical due to the high time requirement.
Keywords: support vector machine (SVM), particle swarm optimization (PSO), parameter optimization, classification.
Judul : Optimisasi Parameter Support Vector Machine Menggunakan Algoritme Particle Swarm Optimization
Nama : Sandy Cahya Gumilar NRP : G64063151 Menyetujui: Pembimbing Mushthofa, S.Kom., M.Sc. NIP. 19820325 200912 1 003 Mengetahui:
Ketua Departemen Ilmu Komputer,
Dr. Ir. Sri Nurdiati, M.Sc. NIP. 19601126 198601 2 001
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala curahan rahmat dan karunia-Nya sehingga skripsi ini dapat diselesaikan. Skripsi ini merupakan hasil penelitian yang dilakukan dari bulan Februari sampai bulan Oktober 2010 dengan bidang kajian Optimisasi Parameter Support Vector Machine Menggunakan Algoritme Particle Swarm Optimization.
Ucapan terima kasih penulis sampaikan kepada pihak yang telah membantu penyelesaian tugas akhir ini antara lain:
1 Kedua orang tua yang selalu memberikan doa dan dukungan selama melakukan penelitian. 2 Bapak Mushthofa, S.Kom., M.Sc selaku pembimbing atas bimbingan, arahan, dan nasihat yang
diberikan selama pengerjaan tugas akhir ini.
3 Dr. Yeni Herdiyeni, S.Si., M.Kom. dan Dr. Ir. Agus Buono, M.Si., M.Kom. selaku penguji atas segala waktu, masukan, dan koreksi yang telah diberikan.
4 Rekan-rekan satu bimbingan dan rekan-rekan ILKOM 43 yang telah banyak membantu penulis selama menjalani perkuliahan dan penelitian di Departemen Ilmu Komputer IPB.
5 Departemen Ilmu Komputer, staf, dan dosen yang telah banyak membantu baik selama penelitian maupun pada masa perkuliahan.
Penulis menyadari masih banyak kekurangan dalam penelitian ini. Oleh karena itu, kritik dan saran sangat penulis harapkan untuk perbaikan di masa mendatang semoga penelitian ini dapat bermanfaat, Amin.
Bogor, Desember 2010
RIWAYAT HIDUP
Penulis lahir di Jakarta pada tanggal 16 Agustus 1988. Penulis merupakan anak pertama dari dua bersaudara dengan ayah bernama Anwar Sanusi dan ibu bernama Sri Murni.
Pada tahun 2006 Penulis lulus dari SMA Negeri 1 Cileungsi Kabupaten Bogor dan pada tahun yang sama Penulis diterima di Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI). Pada tahun 2007 Penulis diterima di Departemen Ilmu Komputer Fakultas Matematika dan Ilmu Pengetahuan Alam. Pada tahun 2009 Penulis menjalankan Praktik Kerja Lapang (PKL) di Badan Pemeriksa Keuangan Republik Indonesia Jakarta selama kurang lebih 1 bulan.
DAFTAR ISI
Halaman DAFTAR ISI ... iv DAFTAR TABEL ... v DAFTAR GAMBAR ... v DAFTAR LAMPIRAN ... v PENDAHULUAN ... 1 Latar Belakang ... 1 Tujuan Penelitian ... 1 Ruang Lingkup ... 1 Manfaat ... 1 TINJAUAN PUSTAKA ... 1 Klasifikasi ... 1Support Vector Machine (SVM) ... 2
Linearly Separable Data ... 2
Soft Margin Hyperplane ... 3
Kernel Trick ... 3
Optimal Algorithm ... 3
Algoritme Particle Swarm Optimization (PSO) ... 4
Inertia Weight ... 4
K-Fold Cross Validation ... 5
METODE PENELITIAN ... 5
Data ... 5
Pembagian Data Uji dan Data Latih ... 5
Pelatihan Data Menggunakan SVM ... 5
Klasifikasi Data Menggunakan SVM ... 6
Optimisasi Parameter ... 6
Lingkup Pengembangan Sistem ... 6
HASIL DAN PEMBAHASAN... 6
Data ... 6
Praproses Data ... 7
Klasifikasi Menggunakan SVM Tanpa Optimisasi ... 8
Klasifikasi Menggunakan SVM Dengan Metode Optimisasi ... 8
Kernel dan Parameter Optimal ... 11
KESIMPULAN DAN SARAN... 12
Kesimpulan ... 12
Saran ... 12
DAFTAR PUSTAKA ... 12
DAFTAR TABEL
Halaman
1 Distribusi kelas Yeast ... 7
2 Atribut-atribut pada data Letter ... 7
3 Distibusi kelas data Letter ... 7
4 Kode kelas pada data Yeast ... 7
5 Hasil klasifikasi pembangkitan 30 parameter pada data Diabetes ... 8
6 Hasil klasifikasi pembangkitan 30 parameter pada data Letter ... 8
7 Hasil klasifikasi pembangkitan 30 parameter pada data Yeast ... 8
8 Klasifikasi data Diabetes dengan metode optimisasi ... 9
9 Klasifikasi data Letter dengan metode optimisasi ... 9
10 Klasifikasi data Yeast dengan metode optimisasi ... 9
11 Peningkatan akurasi klasifikasi SVM dengan metode optimisasi ... 9
12 Perbandingan hasil tanpa metode optimisasi dengan menggunakan metode optimisasi ... 9
13 Confusion matrix pengujian data Yeast ... 10
14 Akurasi setiap kelas data Yeast ... 11
15 Waktu proses klasifikasi dengan optimisasi ... 11
16 Waktu proses klasifikasi tanpa optimisasi ... 11
DAFTAR GAMBAR
Halaman 1 Tahap klasifikasi. ... 22 Bidang pemisah terbaik dengan margin (m) terbesar... 2
3 Metode penelitian. ... 5
4 Perbandingan hasil tanpa metode optimisasi dengan hasil menggunakan metode optimisasi pada data Diabetes. ... 10
5 Perbandingan hasil tanpa metode optimisasi dengan hasil menggunakan metode optimisasi pada data Letter. ... 10
6 Perbandingan hasil tanpa metode optimisasi dengan hasil menggunakan metode optimisasi pada data Yeast. ... 10
7 Persebaran data Yeast. ... 11
8 Perbandingan hasil setiap kernel pada setiap data. ... 11
DAFTAR LAMPIRAN
Halaman 1 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel linear... 152 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel polinomial ... 16
3 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel RBF ... 17
4 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel sigmoid ... 18
5 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel linear ... 19
6 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel polinomial ... 20
7 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel RBF ... 21
8 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel sigmoid ... 22
9 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel linear ... 23
10 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel polinomial ... 24
11 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel RBF ... 25
12 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel sigmoid ... 26
13 Hasil penggabungan SVM dan PSO menggunakan kernel linear pada data Diabetes ... 27
14 Hasil penggabungan SVM dan PSO menggunakan kernel polinomial pada data Diabetes ... 28
15 Hasil penggabungan SVM dan PSO menggunakan kernel RBF pada data Diabetes ... 29
16 Hasil penggabungan SVM dan PSO menggunakan kernel sigmoid pada data Diabetes ... 30
Halaman
18 Hasil penggabungan SVM dan PSO menggunakan kernel polinomial pada data Letter ... 32
19 Hasil penggabungan SVM dan PSO menggunakan kernel RBF pada data Letter ... 33
20 Hasil penggabungan SVM dan PSO menggunakan kernel sigmoid pada data Letter ... 34
21 Hasil penggabungan SVM dan PSO menggunakan kernel linear pada data Yeast ... 35
22 Hasil penggabungan SVM dan PSO menggunakan kernel polinomial pada data Yeast ... 36
23 Hasil penggabungan SVM dan PSO menggunakan kernel RBF pada data Yeast ... 37
24 Hasil penggabungan SVM dan PSO menggunakan kernel sigmoid pada data Yeast ... 38
25 Peningkatan akurasi setiap data pada setiap kernel ... 39
PENDAHULUAN
Latar BelakangPerkembangan teknologi yang cepat memudahkan organisasi dalam mengumpulkan dan menyimpan data berukuran besar. Diperlukan sebuah teknik yang dapat mengekstrak informasi data berukuran besar.
Klasifikasi merupakan proses
mengelompokkan data ke dalam suatu kelas. Data yang berada pada kelas yang sama relatif memiliki ciri-ciri atau informasi yang sama. Tujuan dari klasifikasi ini adalah untuk mengetahui atau memprediksi kelas dari suatu data yang belum diketahui sebelumnya berdasarkan model yang sudah diperoleh.
Salah satu teknik klasifikasi yang termutakhir adalah support vector machine (SVM). SVM sudah diterapkan dalam beberapa bidang. Kerami dan Mufti (2004) telah menggunakan SVM dalam pengenalan jenis splice sites pada baris DNA. Noorniawati (2007) menggunakan SVM untuk klasifikasi pada sistem temu kembali informasi.
Masalah dasar dari SVM adalah menentukan nilai parameter-parameter SVM yang mampu menghasilkan hyperplane atau bidang pembatas yang memisahkan data antarkelas dengan baik. Nilai parameter yang tidak tepat dapat menghasilkan akurasi yang rendah. Saat ini cara yang biasa digunakan untuk mencari nilai parameter adalah dengan cara trial and error. Jika akurasi yang didapat sudah tinggi, maka nilai parameter itu akan diambil, sedangkan jika akurasinya rendah maka akan dicoba lagi dengan nilai parameter yang lain. Cara ini tidak sistematis dan tidak dapat menjamin bahwa akurasi yang didapat sudah mencapai tingkat akurasi yang optimal.
Pada tahun 2008, Damasevicius membuat sebuah sistem pencarian nilai parameter SVM yang sistematis dengan menggabungkan antara SVM dengan metode optimisasi Nelder-Mead pada kasus DNA sequences. Ternyata akurasinya menjadi lebih optimal atau meningkat 1.29% jika dibandingkan dengan cara trial and error (Damacevicius R 2008). Pada penelitian kali ini akan dibuat juga sebuah
sistem yang sistematis dalam
mengoptimisasikan parameter sehingga mampu menghasilkan akurasi yang optimal. Metode optimisasi yang digunakan adalah algoritme particle swarm optimization (PSO). PSO itu sendiri telah diterapkan dalam berbagai permasalahan optimisasi. Sun et al. (2007) menggunakan PSO untuk Neighbor Selection
dalam Peer-to-peer Networks. Hasil yang didapat adalah PSO memberikan hasil yang lebih baik jika dibandingkan dengan metode Genetic Algorithm (GA). Tuegeh et al. (2009) melakukan optimisasi generator scheduling menggunakan PSO dan hasilnya lebih baik jika dibandingkan dengan metode Iterasi Lamda. Pada penelitian kali ini akan diuji juga efektivitas dan efisiensi dari algoritme PSO dalam menentukan parameter optimal pada SVM.
Tujuan Penelitian
Tujuan penelitian ini adalah untuk mengetahui efektivitas dan efisiensi dari algoritme PSO dalam menentukan nilai parameter-parameter SVM, sehingga dihasilkan SVM dengan akurasi terbaik.
Ruang Lingkup
Ruang lingkup pada penelitian ini meliputi: 1. Penerapan algoritme PSO dalam
menentukan nilai parameter SVM.
2. Penggunaan data yang sudah
diklasifikasikan sebelumnya ke dalam beberapa kelas. Data bersumber dari UCI repository of machine learning database (http://archive.ics.edu/ml/datasets). Data yang digunakan adalah data Diabetes, Image letter recognition, dan Yeast. 3. Kernel yang digunakan untuk klasifikasi
SVM antara lain kernel linear, polinomial, Radial Basis Function (RBF), dan sigmoid. 4. Menggunakan 5-fold cross validation untuk membagi data latih dan uji pada data Diabetes dan Yeast.
5. Populasi PSO terdiri dari 30 partikel, nilai koefisien akselerasi dan inertia weight diatur statis.
Manfaat
Manfaat dari penelitian ini adalah mengetahui efektivitas dan efisiensi dari algoritme PSO dalam menentukan parameter optimal pada SVM. Selain itu, penelitian ini dapat dijadikan rujukan arsitektur penggabungan klasifikasi menggunakan SVM dengan metode optimisasi PSO.
TINJAUAN PUSTAKA
KlasifikasiKlasifikasi adalah proses menemukan sebuah himpunan model (fungsi) yang menggambarkan dan membedakan kelas-kelas data atau berbagai konsep. Tujuannya adalah
untuk meramalkan kelas dari objek-objek yang label kelasnya belum diketahui (Han & Kamber 2006).
Gambar 1 Tahap klasifikasi.
Tahapan pada klasifikasi dapat dilihat pada Gambar 1. Klasifikasi terdiri atas dua langkah. Langkah pertama, sebuah classifier dibangun menggambarkan sekumpulan data yang telah ditetapkan kelasnya. Langkah ini biasa disebut dengan langkah pembelajaran. Pada langkah ini algoritme klasifikasi membangun classifier dengan menganalisis atau “belajar dari” data training.
Langkah kedua adalah model atau classifier tersebut digunakan untuk klasifikasi pada test set yang berisi record-record dengan label kelas belum diketahui. Akurasi sebuah classifier didasarkan pada presentase test record yang diprediksi secara benar oleh classifier.
Support Vector Machine (SVM)
SVM merupakan teknik klasifikasi yang lebih didasarkan oleh structural risk minimization daripada empirical risk minimization (Osuna et al. 1997). Proses pembelajaran pada SVM bertujuan untuk mendapatkan hipotesis berupa bidang pemisah terbaik yang tidak hanya meminimalkan empirical risk (rata-rata eror pada data pelatihan), tetapi juga memiliki generalisasi yang baik. Generalisasi adalah kemampuan sebuah hipotesis untuk menglasifikasikan data yang tidak terdapat dalam data pelatihan dengan benar (Sembiring 2007). Prinsip dasar SVM adalah linear classifier, tetapi dapat bekerja
juga pada problem non-linear dengan memasukkan konsep kernel trick pada ruang kerja berdimensi tinggi.
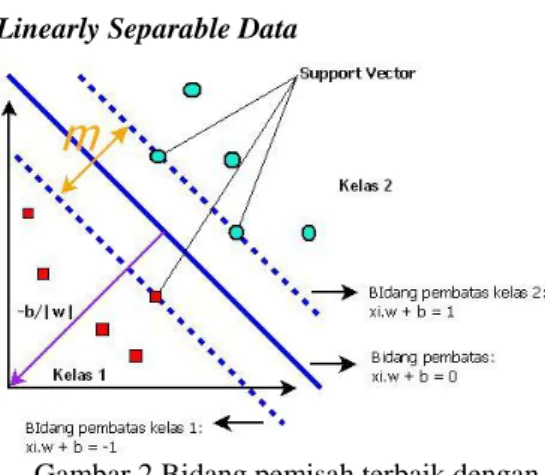
Linearly Separable Data
Gambar 2 Bidang pemisah terbaik dengan margin (m) terbesar.
Linearly separable data merupakan data yang dapat dipisahkan secara linear. Misalkan {x1,…,xn} adalah dataset dan yi є {+1,-1} adalah
label kelas dari data xi. Pada Gambar 2 terdapat
bidang pemisah yang memisahkan kedua kelas. Bidang pemisah yang terbaik adalah yang memiliki margin (m) terbesar.
Adapun data yang berada pada bidang pembatas ini adalah support vector. Untuk menentukan bidang pembatas terbaik dapat dirumuskan seperti persamaan 2.1 (Osuna et al. 1997):
(2.1)
Untuk mempermudah menyelesaikan
persamaan 2.1, maka persamaan itu diubah ke dalam dual problem menggunakan teknik pengali Lagrange menjadi seperti persamaan 2.2 (Osuna et al. 1997).
Dalam hal ini, α = adalah vektor non-negatif dari koesifien Lagrange. Dengan meminimumkan L terhadap w dan b, maka diperoleh persamaan 2.3 dan 2.4.
atau
Persamaan 2.3 dan 2.4 disubtitusikan ke persamaan 2.2 sehingga diperoleh persamaan 2.5.
Dengan demikian, dapat diperoleh nilai αi
yang nantinya digunakan untuk menemukan w. Terdapat αi untuk setiap data pelatihan. Data
pelatihan yang memiliki nilai αi ≥ 0 adalah
support vector sedangkan sisanya memiliki nilai αi = 0. Dengan demikian, fungsi keputusan yang
dihasilkan hanya dipengaruhi oleh support vector.
Formula pencarian bidang terbaik ini adalah permasalahan quadratic programming, sehingga nilai maksimum global dari αi selalu
dapat ditemukan. Setelah solusi permasalahan quadratic programming ditemukan (αi), maka
kelas dari data pengujian x dapat ditentukan berdasarkan nilai dari fungsi keputusan:
xi adalah support vector, ns = jumlah support
vector dan xd adalah data yang akan
diklasifikasikan (Osuna et al. 1997). Soft Margin Hyperplane
Untuk menglasifikasikan data yang tidak dapat dipisahkan secara sempurna, formulasi SVM harus dimodifikasi karena tidak akan ada solusi yang akan ditemukan. Pencarian bidang pemisah terbaik dengan penambahan variabel sering juga disebut soft margin hyperplane. Dengan demikian, formula pencarian bidang pemisah terbaik pada persamaan 2.1 berubah menjadi seperti persamaan 2.7.
s.t
C adalah parameter yang menentukan besar penalti akibat kesalahan dalam klasifikasi data
dan nilainya ditentukan oleh pengguna (Cortes & Vapnik 1995). Pada kasus soft margin hyperplane ini, tidak hanya meminimalkan w tetapi meminimalkan juga parameter penalti C. Kernel Trick
Pada kenyataannya tidak semua data bersifat linearly separable, sehingga sulit dicari bidang pemisah secara linear. Permasalahan ini dapat diselesaikan dengan menransformasikan data ke dalam dimensi ruang fitur (feature space) yang lebih tinggi sehingga dapat dipisahkan secara linear pada feature space yang baru. Caranya, data dipetakan dengan menggunakan fungsi pemetaan (transformasi) dalam feature space sehingga terdapat bidang pemisah yang dapat memisahkan data sesuai dengan kelasnya. Dengan menggunakan fungsi tranformasi , maka fungsi hasil pembelajaran yang dihasilkan seperti pada persamaan 2.5 (Osuna et al. 1997).
Pada umumnya transformasi tidak diketahui dan sulit untuk dipahami. Untuk mengatasi masalah ini, pada SVM digunakan “kernel trick”. Dari persamaan (2.8) dapat dilihat terdapat dot product . Jika terdapat sebuah fungsi kernel K sehingga , maka fungsi transformasi tidak perlu diketahui secara persis. Dengan demikian, fungsi yang dihasilkan seperti persamaan 2.9 (Osuna et al. 1997).
Fungsi kernel yang umum digunakan adalah sebagai berikut:
a. Linear kernel b. Polynomial kernel
c. Radial basis function (RBF)
d. Sigmoid kernel
parameter C, γ, r dan d adalah parameter-parameter pada kernel.
Optimal Algorithm
Local search algorithm bekerja menggunakan single current state dan
umumnya bergerak hanya pada tetangga dari state tersebut. Meskipun local search algorithm tidak sistematik, tetapi memiliki dua keuntungan, yaitu hanya menggunakan sedikit memori dan dapat menemukan solusi yang tepat dalam state space besar atau tidak terbatas.
Local search algorithm berguna untuk memecahkan permasalahan optimisasi. Dalam hal ini adalah local search algorithm mencari state terbaik menurut objective function. Algoritme yang optimal selalu dapat menemukan sebuah maksimum atau minimum global (Russell & Norvig 2003).
Algoritme Particle Swarm Optimization (PSO) Particle swarm optimization (PSO) adalah teknik komputasi evolusioner yang dikembangkan oleh Kennedy dan Eberhart tahun 1995. Tujuan awal PSO adalah menyimulasikan secara grafis pergerakan kawanan burung yang luwes namun tidak dapat diprediksi (Kennedy & Eberhart 1995).
Inisialisasi sistem algoritme PSO biasanya diawali pada suatu populasi yang random. Populasi tersebut biasa disebut dengan partikel. Setiap partikel merupakan suatu potensial solusi dalam suatu permasalahan optimisasi.
Partikel selalu bergerak menuju potensial solusi yang optimum. Kecepatan pergerakan tersebut dipengaruhi oleh velocity yang di-update setiap iterasinya. Perubahan velocity setiap partikel dipengaruhi oleh nilai velocity sebelumnya, posisi pbest, dan gbest. Nilai random yang berbeda dibangkitkan sebagai akselerasi pbest dan gbest (Eberhart & Shi 2001). Pbest (personal best) adalah partikel yang memberikan potensi solusi lebih baik dari partikel sebelumnya, sedangkan gbest (global best) adalah solusi terbaik yang pernah diperoleh partikel dalam suatu populasi.
Secara umum algoritme PSO adalah sebagai berikut:
1. Inisialisasi populasi partikel-partikel dengan posisi random dan velocity dalam dimensi d di problem space.
2. Untuk setiap partikel, evaluasi menggunakan fungsi fitness. Dalam hal ini, fungsi fitness adalah fungsi yang akan dioptimalkan.
3. Bandingkan partikel yang dievaluasi tadi dengan partikel pbest. Jika nilainya lebih baik dari pbest, maka set nilai pbest sama dengan nilai tersebut dan posisi pbest sama dengan lokasi partikel yang dievaluasi menggunakan fungsi fitness tersebut. 4. Identifikasi partikel terbaik dari suatu
populasi sebagai gbest.
5. Update velocity dan posisi partikel menggunakan persamaan (2.10) dan (2.11).
(2.10) (2.11) dengan:
= velocity partikel i dimensi d
= posisi partikel i dimensi d saat ini
= koefisien akselerasi personal influence
= koefisien akselerasi social influence
= pbest (personal best)
= gbest (global best)
6. Ulangi langkah kedua sampai kriteria berhenti terpenuhi, biasanya mencapai nilai optimum atau sampai pada jumlah iterasi tertentu (Eberhart & Shi 2001).
Velocity partikel setiap dimensi dibatasi oleh maksimum velocity Vmax, sehingga nilai velocity partikel tidak dapat melebihi Vmax. Jika Vmax terlalu besar, kemungkinan partikel akan melewati beberapa potensial solusi yang baik, sedangkan jika Vmax terlalu kecil, partikel tidak dapat bergerak cukup jauh untuk mencapai posisi terbaik dalam problem space, sehingga ada kemungkinan akan terjebak pada maksimum lokal. Pada awal penelitian menggunakan PSO, konstanta akselerasi dan diset sama dengan 2.0 untuk semua aplikasi. Vmax biasanya diset antara 10-20% dari range dinamik di setiap dimensi (Eberhart & Shi 2001).
Inertia Weight
Vmax bertindak sebagai pembatas kontrol kemampuan eksplorasi global dari partikel swarm. Vmax yang lebih besar memfasilitasi eksplorasi global, dan Vmax yang lebih kecil mendorong eksploitasi lokal. Pada tahun 1998 Shi dan Eberhart mencantumkan konsep inertia weight (w) ke dalam algoritme PSO sebagai kontrol eksplorasi dan eksploitasi yang lebih baik (Tuegeh et al. 2009).
Persamaan (2.12) dan (2.13) merupakan persamaan velocity dan update posisi menggunakan inertia weight. Persamaan (2.12) dan (2.13) sama dengan persamaan (2.10) dan (2.11), hanya saja ada penambahan inertia weight w sebagai faktor pengali di persamaan (2.12). Pada awal pengembangannya w diset dengan nilai antara 0.4 sampai 0.9. Pemilihan inertia weight yang sesuai akan memberikan keseimbangan antara eksplorasi global dan eksploitasi lokal (Eberhart & Shi 2001).
(2.12) (2.13)
K-Fold Cross Validation
K-fold cross validation dilakukan untuk membagi data penelitian dan data pengujian. Metode ini membagi data contoh secara acak ke dalam K subset yang saling bebas. Satu subset digunakan sebagai data pengujian dan K-1 subset sebagai data pelatihan. Proses cross validation diulang sampai K kali.
Data awal dibagi menjadi K subset yang saling bebas secara acak, yaitu S1, S2,…,Sk,
dengan ukuran setiap subset kira-kira sama. Pelatihan dan pengujian dilakukan sebanyak K kali. Pada subset ke-i, subset Si diperlakukan
sebagai data pengujian dan subset lainnya diperlakukan sebagai data pelatihan. Pada proses pertama S2,…, Sk menjadi data pelatihan
dan S1 menjadi data pengujian, pada proses
kedua S1, S3, … ,Sk menjadi data pelatihan dan
S2 menjadi data pengujian, dan seterusnya (Fu
1994, diacu dalam Noorniawati 2007)
METODE PENELITIAN
Penelitian ini dilakukan dalam beberapa tahap. Tahapan-tahapan tersebut dapat dilihat pada Gambar 3.Gambar 3 Metode penelitian.
Pada Gambar 3 dapat dilihat bahwa penelitian ini dapat dibagi menjadi tiga tahapan. Tahap pertama menentukan data yang akan digunakan, lalu data tersebut dikelompokkan menjadi dua yaitu data latih dan data uji. Tahap kedua pelatihan dan pengujian data menggunakan teknik klasifikasi SVM yang akan menghasilkan performance atau tingkat
akurasi klasifikasi data. Tahap ketiga adalah optimisasi parameter dengan menggunakan PSO, sehingga menghasilkan parameter-parameter baru yang akan digunakan untuk pelatihan SVM selanjutnya. Proses optimisasi parameter ini akan berhenti jika akurasi yang didapat sudah konvergen ke suatu nilai.
Data
Penelitian ini menggunakan data yang sudah diklasifikasikan sebelumnya ke dalam beberapa kelas. Data diambil dari UCI repository of machine learning database.
Pembagian Data Uji dan Data Latih
Penelitian ini menggunakan metode k-fold cross validation. Data akan dibagi secara acak menjadi k subset dan masing-masing subset memiliki jumlah data dan proporsi kelas yang hampir sama. Iterasi akan dilakukan sebanyak k kali. Setiap iterasi, satu subset digunakan sebagai data uji sedangkan sisanya digunakan sebagai data latih.
Pelatihan Data Menggunakan SVM
Data yang sudah terbagi menjadi k subset, kemudian dilatih menggunakan SVM. Pada iterasi pertama, subset pertama digunakan sebagai data uji. Sisa subset digunakan sebagai data latih. Pada iterasi kedua, subset kedua digunakan sebagai data uji. Begitu seterusnya sampai semua subset telah digunakan sebagai data uji.
Proses pelatihan membutuhkan parameter yang sesuai dengan jenis kernel. Sebelumnya nilai parameter tersebut telah dibangkitkan secara random oleh PSO membentuk suatu populasi pada rentang nilai tertentu. Parameter kernel yang digunakan adalah c, g, r, dan d. Setiap parameter direpresentasikan sebagai partikel dan memiliki populasi masing-masing pada layer yang berbeda. Layer pertama digunakan sebagai populasi parameter c, layer kedua digunakan oleh parameter g, layer ketiga dan keempat berturut-turut digunakan oleh parameter r dan d.
Setelah terbentuk populasi, SVM menentukan parameter mana saja yang akan digunakan pada proses pelatihan. Penentuan parameter tergantung dari jenis kernel yang digunakan. Misalnya, ketika menggunakan kernel linear, maka SVM hanya membutuhkan parameter c saja, sehingga hanya layer pertama saja yang diambil oleh SVM untuk pelatihan. Hasil dari pelatihan ini adalah model SVM yang akan digunakan pada saat pengujian.
Klasifikasi Data Menggunakan SVM
Klasifikasi menggunakan Model SVM yang dihasilkan pada tahap pelatihan dan data latih yang sudah ditentukan sebelumnya. Tahap ini akan menghasilkan performance dari objective function atau tingkat akurasi klasifikasi dalam bentuk persentase.
Optimisasi Parameter
Jika kondisi berhenti belum terpenuhi, maka partikel-partikel parameter akan dioptimisasi menggunakan PSO. Partikel akan bergerak dengan kecepatan tertentu menuju partikel yang menghasilkan akurasi tertinggi. Kecepatan pergerakan parameter dipengaruhi oleh velocity. Partikel-partikel yang telah dioptimisasi akan digunakan kembali oleh SVM dalam proses pelatihan. Proses ini berulang sampai kondisi berhenti terpenuhi.
Lingkup Pengembangan Sistem
Perangkat keras yang digunakan dalam penelitian ini berupa notebook dengan spesifikasi:
- Processor: Intel Core 2 Duo - Memory: 2.5 GB
- Hardisk: 160 GB
Perangkat lunak yang digunakan yaitu: - Sistem Operasi: Windows 7 - Microsoft Excel 2007
- Matlab R2008b sebagai bahasa pemrograman
HASIL DAN PEMBAHASAN
DataPenelitian ini menggunakan tiga data yaitu, Pima Indians Diabetes Database (Diabetes), Protein Localization Sites (Yeast), dan Letter Image Recognition Data (Letter). Instance dari setiap data sudah digolongkan ke dalam kelasnya masing-masing.
Data Diabetes merupakan data yang berisikan informasi mengenai seseorang yang positif atau negatif diabetes. Data ini memiliki 2 kelas yaitu kelas 1 dan kelas 0. Kelas 1 merepresentasikan positif diabetes, sedangkan kelas 0 merepresentasikan negatif diabetes. Data Diabetes memiliki 8 atribut dan 768 instance. 500 instance untuk kelas 0 dan 268 instance untuk kelas 1. Tidak ada missing value pada data Diabetes. Atribut-atribut pada data Diabetes antara lain number of times pregnant, plasma glucose concentration a 2 hours in an oral glucose tolerance test, diastolic blood
pressure (mmHg), triceps skin fold thickness (mm), 2-hour serum insulin (mu U/ml), body mass index (weight (kg)/height (m))^2), Diabetes pedigree function, Age (years) dan kelas (0 dan 1). Tipe-tipe atribut data Diabetes adalah integer, real dan biner. Pada tahun 1988, Smith menggunakan data ini untuk meramalkan awal dari diabetes mellitus. Metode yang digunakan Smith adalah algoritme pembelajaran ADAP. Akurasi yang diperoleh sebesar 76% dari 192 data uji (Smith 1988).
Data Yeast digunakan untuk memprediksi protein localization sites. Data ini terdiri atas 10 kelas yang merupakan representasi dari protein dari Yeast. Kesepuluh kelas tersebut antara lain CYT (cytosolic atau cytoskeletal), NUC (nuclear), MIT (mitochondrial), ME3 (membrane protein no N-terminal signal), ME2 (membrane protein uncleaved signal), ME1 (membrane protein cleaved signal), EXC (extracellular), VAC (vacuolar), POX (peroxisomal), dan ERL (endoplasmic reticulum lumen). Data Yeast memiliki 9 atribut dan 1484 instance. Distribusi instance setiap kelas pada data Yeast dapat dilihat pada Tabel 1 dan atribut-atribut pada data Yeast antara lain:
Accession number for the SWISS-PROT database (sequence name)
McGeoch's method for signal sequence recognition (mcg)
Von Heijne's method for signal sequence recognition (gvh)
Score of the ALOM membrane spanning region prediction program (alm)
Score of discriminant analysis of the amino acid content of the N-terminal region (20 residues long) of mitochondrial and non-mitochondrial proteins (Mit)
Presence of "HDEL" substring (thought to act as a signal for retention in the endoplasmic reticulum lumen). Binary attribute (erl)
Peroxisomal targeting signal in the C-terminus (pox)
Score of discriminant analysis of the amino acid content of vacuolar and extracellular proteins (vac)
Score of discriminant analysis of nuclear localization signals of nuclear and non-nuclear proteins (nuc).
Hanya atribut sequence name dan score of discriminant analysis of nuclear localization signals of nuclear and non-nuclear proteins (nuc) yang bertipe nominal. Atribut lainnya bertipe real. Tidak ada missing value pada data ini.
Tabel 1 Distribusi kelas Yeast Class N CYT 463 NUC 429 MIT 244 ME3 163 ME2 51 ME1 44 EXC 35 VAC 30 POX 20 ERL 5
Horton dan Nakai (1996) menggunakan data Yeast untuk memprediksi the cellular localization sites dari protein menggunakan sistem klasifikasi probabilistik. Akurasi yang dihasilkan sebesar 55%.
Data Letter merupakan data untuk pengenalan 26 huruf. Data ini terdiri atas 26 kelas (A sampai Z), 17 atribut dan 20.000 instance. Atribut dan distribusi kelas pada data Letter dapat dilihat pada Tabel 2 dan 3.
Tabel 2 Atribut-atribut pada data Letter Attribute
Name
Attribute Type
Description lettr Nominal Capital Letter x-box Integer horizontal position
of box
y-box Integer Vertical position of box
Width Integer Width of box High Integer Height of box Onpix Integer Total # on pixels x-bar Integer Mean x of on pixels
in box
y-bar Integer Mean y of on pixels in box
X2bar Integer Mean x variance Y2bar integer Mean y variance Xybar Integer Mean x y correlation X2ybar Integer Mean of x*x*y Xy2br Integer Mean of x*y*y x-ege Integer Mean edge count left
to right
Xegvy Integer Correlation of x-ege with y
y-ege Integer Mean edge count bottom to top Yegvx Integer Correlation of y-ege
with x
Frey dan Slate (1991) melakukan klasifikasi menggunakan data Letter dengan metode Holland-style Adaptive Classifier dan akurasi yang didapat sekitar 80%.
Tabel 3 Distibusi kelas data Letter
Kelas N Kelas N A 789 N 783 B 766 O 753 C 736 P 803 D 805 Q 783 E 768 R 758 F 775 S 748 G 773 T 796 H 734 U 813 I 755 V 764 J 747 W 752 K 739 X 787 L 761 Y 786 M 792 Z 734 Praproses Data
Sebelum diklasifikasikan dengan SVM, ketiga data tersebut diproses terlebih dahulu agar data sesuai dengan sistem atau sesuai dengan format SVM. Adapun proses yang dilakukan antara lain, mengubah atribut bertipe nominal menjadi integer, membuang atribut yang tidak berpengaruh atau tidak menjadi suatu ciri-ciri yang dapat mempengaruhi hasil klasifikasi, dan tidak mengikutsertakan kelas yang instance-nya sangat sedikit.
Semua atribut dan kelas pada data Diabetes bertipe integer dan real, sehingga data ini tidak perlu dilakukan praproses karena sudah sesuai dengan format SVM. SVM hanya bisa menerima data dengan tipe data integer dan real.
Kelas pada data Yeast bertipe nominal. Oleh karena itu, kelas-kelas tersebut diubah terlebih dahulu menjadi data bertipe integer. Setelah diubah, kelas-kelas yang ada pada data ini menjadi seperti Tabel 4.
Tabel 4 Kode kelas pada data Yeast
Kelas Kode Kelas
CYT 1 EXC 2 ME1 3 ME2 4 ME3 5 MIT 6 NUC 7 POX 8 VAC 9 ERL 10
Atribut pertama pada data Yeast merupakan sequence name, jadi tidak berpengaruh pada hasil klasifikasi, sehingga atribut ini
dihilangkan. Begitu pula dengan kelas ERL, karena kelas ERL hanya memiliki 5 instance maka kelas ini tidak diikutsertakan dalam penelitian.
Praproses pada data Letter hanya mengubah kelas yang bertipe nominal (A sampai Z) menjadi bertipe integer (1 sampai 26). Selebihnya data ini sudah sesuai dengan format SVM.
Setelah data telah sesuai dengan format SVM, lalu data tersebut dinormalisasi dengan tujuan mengubah data yang kompleks tanpa menghilangkan isi, sehingga mudah diolah. Persamaan normalisasi yang digunakan adalah
n
Klasifikasi Menggunakan SVM Tanpa Optimisasi
Setiap data diklasifikasikan menggunakan kernel linear, polinomial, RBF, sigmoid. Nilai parameter setiap kernel dibangkitkan secara acak pada range nilai tertentu. Parameter c, g, r dibangkitkan dari 2-10 sampai 25 untuk kernel linear, RBF, dan sigmoid, sedangkan pada kernel polinomial dibangkitkan antara 2-10 sampai 20. Untuk parameter d (degree) dibangkitkan secara acak antara 2 sampai 5. Penelitian ini menggunakan library LIBSVM yang dibuat oleh mahasiswa Ilmu Komputer National Taiwan University, Chih-Yuan Yang dan Chih-Huai Cheng.
Pembagian data latih dan data uji pada data Diabetes dan Yeast menggunakan metode 5-fold cross validation, sedangkan pada data Letter tanpa menggunakan cross validation karena data Letter sudah cukup banyak jadi tidak membutuhkan cross validation. Pembagiannya hanya menggunakan persentase, 75.05% menjadi data latih dan 24.95% menjadi data uji.
Hasil klasifikasi pembangkitan 30 pasangan parameter secara acak pada data Diabetes, Letter, dan Yeast pada setiap kernel berturut-turut dapat dilihat pada Tabel 5, 6, dan 7.
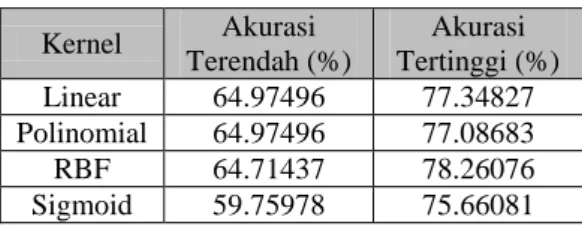
Akurasi terendah pada data Diabetes sebesar 59.75978% yang diperoleh dari kernel sigmoid, sedangkan yang tertinggi sebesar 78.26076% dari kernel RBF. Pada data Letter akurasi terendah sebesar 4.068136% yang diperoleh dari 3 kernel yaitu polinomial, RBF dan sigmoid, sedangkan yang tertinggi diperoleh dari kernel RBF sebesar 97.07415%. Pada data Yeast akurasi terendah dihasilkan oleh kernel sigmoid sebesar 27.78814%, sedangkan yang tertinggi dihasilkan oleh kernel linear dengan
tingkat akurasi sebesar 58.21553%. Untuk hasil lebih lengkap dapat dilihat pada Lampiran 1 sampai 12.
Tabel 5 Hasil klasifikasi pembangkitan 30 parameter pada data Diabetes
Kernel Akurasi Terendah (%) Akurasi Tertinggi (%) Linear 64.97496 77.34827 Polinomial 64.97496 77.08683 RBF 64.71437 78.26076 Sigmoid 59.75978 75.66081
Tabel 6 Hasil klasifikasi pembangkitan 30 parameter pada data Letter
Kernel Akurasi Terendah (%) Akurasi Tertinggi (%) Linear 66.69339 85.41082 Polinomial 4.068136 94.96994 RBF 4.068136 97.07415 Sigmoid 4.068136 72.52505
Tabel 7 Hasil klasifikasi pembangkitan 30 parameter pada data Yeast
Kernel Akurasi Terendah (%) Akurasi Tertinggi (%) Linear 33.67178 58.21553 Polinomial 31.30486 57.13353 RBF 31.30486 58.07948 Sigmoid 27.78814 57.87792
Tanpa menggunakan optimisasi, SVM mampu meningkatkan akurasi melebihi penelitian sebelumnya. Data Diabetes mampu ditingkatkan sampai 2.26076% oleh kernel RBF. Data Letter meningkat ± 17% menggunakan kernel RBF dari penelitian sebelumnya. Walaupun ada juga yang masih di bawah 80% yaitu ketika klasifikasi menggunakan kernel sigmoid. Data Yeast dapat ditingkatkan sebanyak 3.21553% dari penelitian sebelumnya. Walaupun akurasi dapat ditingkatkan, tetapi belum dapat dipastikan bahwa akurasi yang dihasilkan merupakan akurasi yang optimal.
Klasifikasi Menggunakan SVM Dengan Metode Optimisasi
Selanjutnya akan dicoba perhitungan klasifikasi SVM digabungkan dengan metode optimisasi PSO. Library SVM yang digunakan sama ketika melakukan klasifikasi tanpa optimisasi. Nilai akurasi dijadikan sebagai objective function dari PSO. Nilai parameter awal yang akan dijadikan partikel pada PSO
adalah 30 parameter yang menghasilkan akurasi terendah pada setiap data dan setiap kernel. Penelitian ini mengambil nilai akurasi yang rendah sebagai partikel karena untuk membuktikan juga bahwa PSO mampu mengoptimalkan tingkat akurasi walaupun dimulai dari nilai yang rendah. PSO akan berhenti jika peningkatan akurasinya di bawah 0.1% dalam 10 iterasi berturut-turut. Jadi, selama kondisi berhenti belum terpenuhi, PSO akan terus meng-update parameter-parameter tersebut.
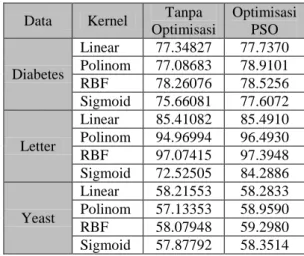
Hasil percobaan dapat dilihat pada Tabel 8 untuk data Diabetes, Tabel 9 untuk data Letter dan Tabel 10 untuk data Yeast. Grafik peningkatan akurasi setiap data berdasarkan kernel linear, polinomial, RBF, dan sigmoid berturut-turut dapat dilihat pada Lampiran 25. Tabel 8 Klasifikasi data Diabetes dengan
metode optimisasi Kernel Akurasi Awal (%) Akurasi Akhir (%) Iterasi Waktu (detik) Linear 65.1048 77.7370 14 53.3562 Poli 65.1048 78.9101 31 275.982 RBF 65.1048 78.5256 28 296.391 Sigmoid 65.1048 77.6072 18 271.362
Tabel 9 Klasifikasi data Letter dengan metode optimisasi Kernel Akurasi Awal (%) Akurasi Akhir (%) Iterasi Waktu (detik) Linear 66.4729 85.4910 21 18947.33 Poli 4.0681 96.4930 24 19818.17 RBF 7.0942 97.3948 13 31156.07 Sigmoid 7.0341 84.2886 22 43796.55
Tabel 10 Klasifikasi data Yeast dengan metode optimisasi Kernel Akurasi Awal (%) Akurasi Akhir (%) Iterasi Waktu (detik) Linear 32.5896 58.2833 19 437.242 Poli 31.3049 58.9590 17 564.992 RBF 31.3049 59.2980 23 1182.219 Sigmoid 31.3049 58.3514 18 1448.067
Pada Tabel 8, 9 dan 10 dapat dilihat bahwa PSO dapat meningkatkan akurasi walaupun dimulai dari akurasi yang kecil. Ini membuktikan bahwa PSO efektif untuk mencari nilai parameter yang mampu menghasilkan akurasi tinggi. Pada Tabel 11 dirangkum hasil peningkatan akurasi setiap data pada setiap kernel.
Tabel 11 Peningkatan akurasi klasifikasi SVM dengan metode optimisasi
Diabetes Letter Yeast
Linear 12.632% 19.018% 25.694%
Polinom 13.805% 92.425% 27.654%
RBF 13.421% 90.301% 27.993%
Sigmoid 12.502% 77.255% 27.047%
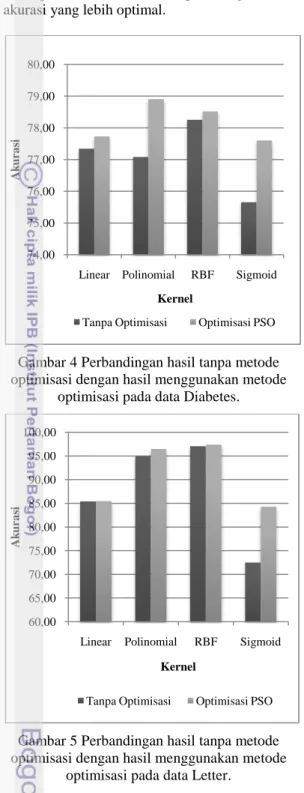
Peningkatan terbesar akurasi pada data Diabetes dan data Letter terjadi ketika menggunakan kernel polinomial. Peningkatan untuk data Diabetes dan Letter berturut-turut sebesar 13.805% dan 92.425%. Klasifikasi data Letter ketika menggunakan kernel linear hanya meningkat 19.018% karena akurasi bergerak dari 66.4729% sehingga peningkatannya tidak dapat sebesar kernel yang lainnya. Peningkatan terbesar data Yeast sebesar 27.993% ketika menggunakan kernel RBF. Tetapi, hasil tersebut jika dibandingkan dengan akurasi klasifikasi tanpa optimisasi maka peningkatannya tidak terlalu besar. Hasil perbandingan dapat dilihat pada Tabel 12 dan Gambar 4, 5, dan 6.
Tabel 12 Perbandingan hasil tanpa metode optimisasi dengan menggunakan metode optimisasi
Data Kernel Tanpa
Optimisasi Optimisasi PSO Diabetes Linear 77.34827 77.7370 Polinom 77.08683 78.9101 RBF 78.26076 78.5256 Sigmoid 75.66081 77.6072 Letter Linear 85.41082 85.4910 Polinom 94.96994 96.4930 RBF 97.07415 97.3948 Sigmoid 72.52505 84.2886 Yeast Linear 58.21553 58.2833 Polinom 57.13353 58.9590 RBF 58.07948 59.2980 Sigmoid 57.87792 58.3514 Secara garis besar peningkatan akurasi jika dibandingkan dengan hasil tanpa optimisasi masih di bawah 2%. Kecuali ketika mengklasifikasikan data Letter akurasi meningkat 11.7636%. Hasil tertinggi yang diraih tanpa optimisasi hanya 72.52505%. Tetapi ketika mengunakan metode optimisasi dalam mengklasifikasikan data Letter, akurasinya menjadi 84.2886%. Ini menunjukkan bahwa pencarian parameter menggunakan cara yang tidak sistematis memungkinkan kita terjebak pada hasil yang tidak optimal, sedangkan ketika menggunakan cara yang lebih sistematis yaitu menggunakan metode optimisasi seperti PSO dalam mencari
nilai parameter, SVM mampu menghasilkan akurasi yang lebih optimal.
Gambar 4 Perbandingan hasil tanpa metode optimisasi dengan hasil menggunakan metode
optimisasi pada data Diabetes.
Gambar 5 Perbandingan hasil tanpa metode optimisasi dengan hasil menggunakan metode
optimisasi pada data Letter.
Ketika menglasifikasikan data Letter menggunakan kernel sigmoid, hasil akurasinya tidak sebesar akurasi ketika menggunakan kernel lainnya. Ini disebabkan kernel sigmoid tidak mampu memisahkan data Letter dengan baik walaupun sudah ditransformasi ke dimensi yang lebih tinggi, sehingga terjadi banyak kesalahan prediksi ketika menggunakan kernel sigmoid.
Gambar 6 Perbandingan hasil tanpa metode optimisasi dengan hasil menggunakan metode
optimisasi pada data Yeast.
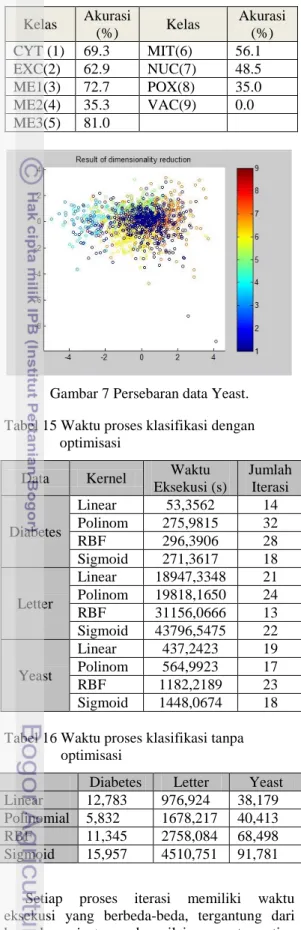
Data Yeast memiliki akurasi yang terendah dibanding dengan dua data lainnya. Ini disebabkan data terlalu menyebar. Persebaran data dapat dilihat pada Gambar 7. Pada gambar tersebut dapat dilihat bahwa data yang memiliki kelas yang sama tidak berkumpul pada suatu daerah, tetapi menyebar atau bercampuran dengan kelas yang lainnya, sehingga terjadi banyak kesalahan ketika mengklasifikasikan data Yeast. Persebaran data dalam sudut pandang yang lain dapat dilihat pada Lampiran 26.
Hasil pengujian data Yeast
direpresentasikan melalui confusion matrix pada Tabel 13. Hasil tertinggi diperoleh pada saat pengujian kelas 5 atau ME3, yaitu sebesar 81%. Sedangkan hasil terendah diperoleh saat pengujian kelas 9 atau VAC, yaitu sebesar 0%. Seluruh kelas VAC dideteksi sebagai kelas 1 sampai kelas 7. Ini disebabkan kelas VAC terlalu menyebar sehingga kelas ini tidak dapat diklasifikasikan dengan baik menggunakan SVM. Akurasi setiap kelas data Yeast dapat dilihat pada Tabel 14.
Tabel 13 Confusion matrix pengujian data Yeast
KLS TERDETEKSI 1 2 3 4 5 6 7 8 9 AK T UA L 1 321 0 0 6 5 35 96 0 0 2 4 22 4 2 0 2 1 0 0 3 0 4 32 6 1 1 0 0 0 4 7 4 9 18 5 4 4 0 0 5 13 0 0 5 132 2 11 0 0 6 75 0 1 7 7 137 14 3 0 7 183 1 0 2 15 20 208 0 0 8 6 1 1 0 0 5 0 7 0 9 14 2 1 1 7 2 3 0 0 74,00 75,00 76,00 77,00 78,00 79,00 80,00
Linear Polinomial RBF Sigmoid
A k u ra si Kernel
Tanpa Optimisasi Optimisasi PSO
60,00 65,00 70,00 75,00 80,00 85,00 90,00 95,00 100,00
Linear Polinomial RBF Sigmoid
A k u ra si Kernel
Tanpa Optimisasi Optimisasi PSO
56,00 56,50 57,00 57,50 58,00 58,50 59,00 59,50
Linear Polinomial RBF Sigmoid
A k u ra si Kernel
Tabel 14 Akurasi setiap kelas data Yeast Kelas Akurasi (%) Kelas Akurasi (%) CYT (1) 69.3 MIT(6) 56.1 EXC(2) 62.9 NUC(7) 48.5 ME1(3) 72.7 POX(8) 35.0 ME2(4) 35.3 VAC(9) 0.0 ME3(5) 81.0
Gambar 7 Persebaran data Yeast. Tabel 15 Waktu proses klasifikasi dengan
optimisasi
Data Kernel Waktu
Eksekusi (s) Jumlah Iterasi Diabetes Linear 53,3562 14 Polinom 275,9815 32 RBF 296,3906 28 Sigmoid 271,3617 18 Letter Linear 18947,3348 21 Polinom 19818,1650 24 RBF 31156,0666 13 Sigmoid 43796,5475 22 Yeast Linear 437,2423 19 Polinom 564,9923 17 RBF 1182,2189 23 Sigmoid 1448,0674 18
Tabel 16 Waktu proses klasifikasi tanpa optimisasi
Diabetes Letter Yeast
Linear 12,783 976,924 38,179
Polinomial 5,832 1678,217 40,413
RBF 11,345 2758,084 68,498
Sigmoid 15,957 4510,751 91,781
Setiap proses iterasi memiliki waktu eksekusi yang berbeda-beda, tergantung dari banyaknya instance dan nilai parameter setiap kernel. Waktu eksekusi setiap proses klasifikasi dengan optimisasi dapat dilihat pada Tabel 15,
sedangkan waktu eksekusi tanpa optimisasi pada Tabel 16. Dibandingkan dengan data yang lain, data Diabetes memiliki waktu tercepat dalam proses klasifikasi dengan optimisasi maupun tanpa optimisasi, sedangkan data Letter memiliki waktu terlama. Ini disebabkan oleh data Diabetes memiliki instance yang paling sedikit yaitu sebanyak 768 instance, sedangkan Letter memiliki instance yang terbanyak yaitu 20000 instance. Selain dipengaruhi banyaknya instance, waktu eksekusi juga dipengaruhi oleh besarnya nilai parameter. Semakin besar nilai parameter, semakin lama pula waktu eksekusinya.
Kernel dan Parameter Optimal
Hasil perbandingan setiap kernel pada setiap data dapat dilihat pada Gambar 8.
Gambar 8 Perbandingan hasil setiap kernel pada setiap data.
Data Diabetes optimal pada saat menggunakan kernel polinomial dengan akurasi sebesar 78.9101% dan nilai parameter c, g, r, dan d berturut-turut adalah 0.252101, 0.053248, 1 dan 5. Data Letter optimal ketika menggunakan kernel RBF dengan akurasi sebesar 97.3948%. Nilai parameter c dan g yang optimal untuk data Letter berturut-turut adalah 32 dan 0.209289. Data Yeast pun sama optimal ketika menggunakan kernel RBF dengan akurasi sebesar 59.2980%. Nilai parameter c dan g yang optimal untuk data Yeast berturut-turut adalah 32 dan 0.007351.
Secara umum, pada penelitian ini kernel Polinomial dan RBF mampu menghasilkan akurasi yang lebih tinggi dibandingkan kernel linear dan sigmoid. Namun tidak dapat dipastikan kernel polinomial dan RBF adalah
0 10 20 30 40 50 60 70 80 90 100
Diabetes Letter Yeast
Ak
u
ra
si
kernel terbaik karena pemilihan kernel tergantung dari data yang akan digunakan.
KESIMPULAN DAN SARAN
KesimpulanSVM merupakan salah satu metode klasifikasi. Cara kerja SVM adalah dengan cara mencari hyperplane terbaik yang memiliki margin terbesar sehingga dapat memisahkan kelas yang satu dengan kelas yang lainnya. Untuk menglasifikasikan data yang tidak dapat
dipisahkan secara linear, SVM
menranformasikan data ke dimensi yang lebih tinggi dengan menggunakan kernel trick. Ada beberapa kernel yang biasa digunakan antara lain, kernel linear, polinomial, RBF, dan sigmoid.
Setiap kernel memiliki parameter-parameter yang dapat menentukan hasil klasifikasi data. Tidak mudah dalam menentukan nilai parameter setiap kernel, sehingga pada penelitian ini SVM digabungkan dengan metode optimisasi PSO untuk mencari nilai parameter terbaik.
Penggabungan SVM dan PSO cukup efektif dalam mencari nilai parameter yang optimal. Penggabungan ini menciptakan sebuah teknik yang lebih sistematis dibandingkan dengan teknik trial and error dalam mencari nilai parameter SVM. Tetapi penggabungan ini membutuhkan waktu eksekusi yang lebih lama untuk mencapai nilai yang optimal.
Klasifikasi menggunakan SVM dan PSO mampu meningkatkan akurasi jika dibandingkan dengan penelitian sebelumnya. Data Diabetes meningkat 2.26% menjadi 78.26%, data Letter meningkat 17.07% menjadi 97.07% dan data Yeast meningkat 3.21% menjadi 58.21%.
Data Yeast memiliki akurasi yang terkecil dibandingkan dengan dua data lainnya. Ini disebabkan oleh distribusi data yang terlalu menyebar, sehingga terjadi banyak kesalahan ketika menglasifikasikannya dengan SVM. Saran
Pada penelitian ini tidak dilakukan perubahan nilai inertia weight dan correction factor. Kedua faktor tersebut mempengaruhi velocity dari partikel PSO. Pada penelitian selanjutnya diharapkan mencoba menggunakan beberapa nilai inertia weight dan koefisien akselerasi untuk mencari nilai yang terbaik, karena jika tepat dalam memilih nilai kedua faktor tersebut maka PSO dapat bekerja dengan
optimal sehingga dapat memungkinkan mendapat hasil yang lebih baik.
Selain itu, dapat juga digunakan populasi yang dinamis pada PSO. Ketika partikel terjebak pada suatu lokal optimum, partikel ini dapat dihapus atau diganti dengan partikel yang baru.
DAFTAR PUSTAKA
Cortes C, Vapnik V. 1995. Support-Vector Networks. Machine Learning. Volume 20 Hal: 273-297.
Damasevicius R. 2008. Optimization of SVM Parameters for Promoter Recognition in DNA Sequences. 20th EURO Mini Conference “Continuous Optimization and Knowledge-Based Tecnologies”. Neringa, Lithuania, 20-23 Mei, 2008.
Eberhart RC, Shi Y. 2001. Particle Swarm Optimization: Development, Applications and Resources. Congress on Evolutionary Computation, Seoul, Korea. Piscataway, NJ:IEEE Service Center.
Frey PW, Slate DJ. 1991. Letter Recognition Using Holland-Style Adaptive Classifiers. Machine Learning. Volume 6 Hal: 161-182. Han J, Kamber M. 2006. Data Mining Concepts
and Techniques. San Francisco: Morgan Kaufmann.
Horton P, Nakai K. 1996. A Probabilistic Classification System for Predicting the Cellular Localization Sites of Proteins. The fourth International Conference on Intelligent Systems for Molecular Biology. Hal: 109-115, Louis, MO, 1996.
Kennedy J, Eberhart RC. 1995. Particle Swarm Optimization. IEEE Int’l Conf. On Neural Networks. Vol. 4 1942-1948.
Kerami D, Murfi H. 2004. Kajian Kemampuan Generalisasi SVM Dalam Pengenalan Jenis Splice Sites Pada Barisan DNA. Makara, Sains. Volume 8 No. 3. Hal: 89-95.
Noorniawati VY. 2007. Metode Support Vector Machine untuk klasifikasi Pada Sistem Temu Kembali Citra. Skripsi. Institut Pertanian Bogor.
Osuna E, Freund R, Girosi F. 1997. Support Vector Machines: Training and Applications. Massachusetts Institute of Technology.
Russell S, Norvig P. 2003. Artificial Intelligence A Modern Approach Second Edition. New Jersey: Pearson Education, Inc.
Sembiring K. 2007. Penerapan Teknik Support Vector Machine Untuk Pendeteksian Instruksi Pada Jaringan. Skripsi. Institut Teknologi Bandung.
Smith JW, et al. 1988. Using the ADAP Learning Algorithm to Forecast the Onset of Diabetes Mellitus. IEEE Computer Society Press. Hal: 261-265.
Sun S, et al. 2007. A Particle Swarm Optimization for Neighbor Selection in Peer-to-peer Networks. 6th International Conference on Computer Information Systems and Industrial Management Applications (CISIM’07).
Tuegeh M, Soeprijanto, Purnomo MH. 2009. Modified Improved Particle Swarm Optimization for Optimal Generator Scheduling. Seminar Nasional Aplikasi Teknologi Informasi (SNATI). ISSN: 1907-5022.
Lampiran 1 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel linear No Akurasi c 1 64,97496 0,001446 2 65,10483 0,001323 3 67,05458 0,002376 4 69,00603 0,002725 5 74,87055 0,004039 6 76,56651 0,115242 7 76,56651 0,220223 8 76,69553 0,031914 9 76,69638 0,157032 10 76,69638 0,157505 11 76,69638 0,059653 12 76,95612 0,01496 13 76,95612 0,28759 14 76,95696 0,399946 15 77,08599 0,014865 16 77,08683 0,044549 17 77,21586 0,011512 18 77,21755 0,642114 19 77,2184 1,763464 20 77,34488 0,010851 21 77,34827 4,04242 22 77,34827 15,27195 23 77,34827 1,93853 24 77,34827 21,80109 25 77,34827 1,13842 26 77,34827 28,24121 27 77,34827 9,696877 28 77,34827 12,98976 29 77,34827 3,844399 30 77,34827 1,145454
Lampiran 2 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel polinomial No Akurasi c g r d 1 64,97496 0,375684 0,004118 0,003559 1,158958 2 64,97496 0,35443 0,022309 0,003741 2,654822 3 65,10483 0,00884 0,002469 0,003794 3,525387 4 65,10483 0,002091 0,003168 0,018482 4,977405 5 65,10483 0,00209 0,002718 0,152686 4,812157 6 65,10483 0,006341 0,026475 0,030895 2,675336 7 65,10483 0,827316 0,044865 0,011558 0,578129 8 65,10483 0,134352 0,001227 0,001623 0,257241 9 65,10483 0,008482 0,001419 0,058717 1,521745 10 65,10483 0,542041 0,013854 0,019555 4,50604 11 65,10483 0,005734 0,012199 0,001281 2,159903 12 65,10483 0,001806 0,039105 0,689322 2,713335 13 65,10483 0,056218 0,410304 0,04698 0,083374 14 65,10483 0,697898 0,009531 0,003492 4,004604 15 65,10483 0,001491 0,088461 0,030809 0,712547 16 65,10483 0,007046 0,001653 0,960881 1,284177 17 65,10483 0,00367 0,017204 0,770592 3,308825 18 65,10483 0,020982 0,008323 0,108023 0,848044 19 65,10483 0,014927 0,00608 0,016009 1,39392 20 65,75418 0,231693 0,008857 0,219372 1,981451 21 70,83185 0,179528 0,051116 0,25395 3,783154 22 70,96257 0,007363 0,25893 0,536585 2,900959 23 71,74773 0,037069 0,528884 0,266075 4,819351 24 74,87055 0,056188 0,839942 0,035365 2,392372 25 75,65317 0,022438 0,102566 0,948415 2,792795 26 76,30507 0,038303 0,300563 0,187851 4,674895 27 76,43324 0,082057 0,232683 0,17575 2,702752 28 76,43494 0,325912 0,135519 0,194351 3,562074 29 77,08599 0,302986 0,057122 0,365645 1,845458 30 77,08683 0,307749 0,106484 0,967383 2,397423
Lampiran 3 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel RBF No Akurasi c g 1 64,71437 9,369665 4,811239 2 65,10483 0,001371 12,08062 3 65,10483 0,334018 9,156561 4 65,10483 0,007073 0,471505 5 65,10483 0,04524 0,001234 6 65,10483 0,004965 0,005235 7 65,10483 0,048836 0,002601 8 65,10483 0,007109 0,492384 9 65,10483 0,146638 1,35589 10 65,10483 0,003422 1,412565 11 65,10483 0,010257 0,001385 12 65,10483 0,053263 0,001997 13 65,10483 0,013388 0,243664 14 65,10483 0,020006 0,880677 15 65,10483 0,015401 4,923616 16 65,10483 26,72191 23,0987 17 65,10483 0,034871 0,0287 18 65,10483 0,002995 0,560342 19 65,76097 5,153827 1,711646 20 67,05458 0,423678 0,002929 21 68,2302 0,448324 0,746369 22 73,43604 0,083841 0,130658 23 76,31271 1,936876 0,244857 24 76,56311 0,117513 0,081271 25 77,34742 26,43852 0,025221 26 77,47645 7,124763 0,006265 27 77,47729 0,418934 0,027092 28 77,60716 1,026426 0,014689 29 77,87115 0,796346 0,07929 30 78,26076 0,597232 0,067653
Lampiran 4 Hasil klasifikasi 30 parameter pada data Diabetes menggunakan kernel sigmoid No Akurasi c g r 1 59,75978 0,802892 1,050785 2,507338 2 61,32586 0,415735 1,360366 5,013499 3 64,06587 18,09616 1,760151 0,014907 4 64,32816 6,185702 15,92039 0,030456 5 64,5879 1,137015 0,817716 0,004645 6 64,84679 5,63241 24,10253 0,080473 7 65,10483 0,008573 0,223358 0,00155 8 65,10483 0,011386 0,009487 0,012189 9 65,10483 0,003379 0,002934 0,097128 10 65,10483 0,539449 0,003055 1,245687 11 65,10483 0,105263 0,001892 0,040905 12 65,10483 0,115094 0,065548 2,063511 13 65,10483 0,014049 0,007191 0,016222 14 65,10483 0,270011 0,00259 0,085308 15 65,10483 8,277043 0,230217 9,96289 16 65,10483 0,003373 0,151019 0,060449 17 65,10483 17,118 0,058389 4,370237 18 65,24149 0,952733 0,668243 0,09703 19 65,36542 1,557493 29,97254 2,503591 20 65,4902 0,725376 0,278991 0,432683 21 66,54019 0,142788 2,171767 0,04941 22 66,66497 0,015321 0,24235 0,057025 23 67,05458 0,57511 0,004138 0,007577 24 67,06052 0,820323 0,281409 0,036289 25 67,84568 0,752495 0,217756 0,009228 26 68,36092 0,073934 2,989089 0,001197 27 70,18589 2,936877 0,043805 1,190228 28 73,44029 0,037247 2,736951 1,474996 29 73,70427 0,952091 0,103349 0,059154 30 75,66081 0,026665 7,553248 2,901333
Lampiran 5 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel linear No Akurasi c 1 66,69339 0,001193 2 68,87776 0,001516 3 71,38277 0,002336 4 72,02405 0,002603 5 73,9479 0,00346 6 73,96794 0,003473 7 75,93186 0,004755 8 78,03607 0,006983 9 79,85972 0,01081 10 80,46092 0,014256 11 80,78156 0,015916 12 82,40481 0,030713 13 82,70541 0,036401 14 82,72545 0,038397 15 82,88577 0,041675 16 83,28657 0,06481 17 84,26854 0,104506 18 84,48898 0,15865 19 84,68938 0,183864 20 84,90982 0,434249 21 85,13026 0,617357 22 85,27054 1,091783 23 85,29058 24,15709 24 85,29058 4,491705 25 85,29058 24,62454 26 85,31062 1,004728 27 85,33066 8,911886 28 85,37074 21,90145 29 85,41082 0,852779 30 85,41082 9,595087
Lampiran 6 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel polinomial No akurasi c g r d 1 4,068136 0,003587 0,002294 0,001456 4,231864 2 4,068136 0,043757 0,00185 0,06036 3,733086 3 4,068136 0,001369 0,002721 0,769296 1,184652 4 4,068136 0,045039 0,003857 0,00354 4,786726 5 4,068136 0,005208 0,019439 0,010427 3,001311 6 4,068136 0,005268 0,120244 0,628062 0,863023 7 4,068136 0,002843 0,00579 0,014646 0,451734 8 4,068136 0,284627 0,006773 0,015315 4,555335 9 4,068136 0,003614 0,00116 0,001842 2,880267 10 4,068136 0,074457 0,001449 0,232822 0,449994 11 4,068136 0,00716 0,113943 0,05054 0,303032 12 28,37675 0,016048 0,010841 0,007032 1,394378 13 28,49699 0,011872 0,014852 0,019921 1,14943 14 31,30261 0,000985 0,102548 0,069169 4,053141 15 50,42084 0,02048 0,055349 0,021796 2,557045 16 51,38277 0,010064 0,085694 0,002184 4,855376 17 58,19639 0,006561 0,103142 0,003722 3,1013 18 58,65731 0,009095 0,032217 0,869135 2,529997 19 61,76353 0,640209 0,039087 0,0028 4,292853 20 62,50501 0,739265 0,001045 0,006489 1,276311 21 64,0481 0,042119 0,022147 0,004991 1,604705 22 69,57916 0,003384 0,173936 0,055009 4,185282 23 78,41683 0,140143 0,052942 0,013034 1,106329 24 79,71944 0,008754 0,323471 0,001054 2,019217 25 82,08417 0,031611 0,143024 0,001495 3,62844 26 86,87375 0,191506 0,088413 0,030456 2,782779 27 91,22244 0,039126 0,192654 0,150637 3,211576 28 91,24248 0,003303 0,522837 0,002423 3,625912 29 92,50501 0,124669 0,82074 0,051926 4,942196 30 94,96994 0,15205 0,688869 0,013114 3,498169
Lampiran 7 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel RBF No Akurasi c g 1 4,068136 0,008287 4,659662 2 4,068136 0,018591 19,20923 3 4,068136 0,001185 0,691173 4 4,168337 0,056344 5,571517 5 4,208417 0,093275 24,47648 6 9,739479 0,003502 0,059843 7 35,11022 0,112373 0,001103 8 36,27255 1,087976 7,411709 9 60,7014 0,105543 0,003297 10 66,19238 0,028969 0,022497 11 67,83567 0,018896 0,048542 12 68,13627 0,172964 0,003947 13 70,14028 0,01709 0,207403 14 76,65331 1,339856 0,001831 15 78,51703 0,552653 0,005368 16 80,76152 0,067441 0,061297 17 82,88577 1,75666 0,00448 18 83,46693 5,718097 0,002345 19 85,1503 9,620259 0,002632 20 86,79359 0,384501 0,028425 21 87,33467 10,8753 1,784139 22 87,49499 0,092609 0,369226 23 89,65932 2,710921 0,012634 24 94,80962 1,681847 0,909382 25 95,97194 11,27273 0,028613 26 96,33267 9,430241 0,034451 27 96,43287 0,97549 0,336529 28 96,63327 1,070578 0,286838 29 96,79359 5,273217 0,062552 30 97,07415 1,632331 0,267679
Lampiran 8 Hasil klasifikasi 30 parameter pada data Letter menggunakan kernel sigmoid No Akurasi c g r 1 4,068136 0,003002 0,011755 0,004296 2 4,068136 0,001024 0,003149 0,49424 3 4,068136 2,828878 0,098597 11,43884 4 4,068136 6,637493 0,022139 17,03815 5 5,991984 0,090098 0,894223 3,424875 6 8,797595 27,94511 5,658633 0,147631 7 9,038076 0,004768 0,095359 1,511634 8 9,338677 0,599093 6,043823 0,058649 9 11,24248 0,002784 0,041428 0,015336 10 12,00401 0,177166 0,430515 0,014598 11 12,60521 0,00942 2,719434 0,002408 12 12,74549 0,210947 0,401606 0,015856 13 14,92986 7,770586 0,309563 0,009686 14 17,03407 0,217931 0,248162 0,001012 15 17,45491 0,004326 8,018352 0,006018 16 18,01603 0,001198 17,49395 0,01229 17 18,53707 0,002697 1,149481 0,00099 18 18,77756 1,047895 0,210908 0,021543 19 18,97796 0,001748 16,89317 0,003373 20 19,23848 0,001754 0,452895 0,167938 21 19,81964 0,003479 0,948647 0,014173 22 20,96192 1,025011 0,191808 0,004198 23 31,30261 5,591836 0,10726 0,00211 24 43,96794 13,47599 0,063408 0,009737 25 48,97796 0,327725 0,067063 0,004128 26 52,28457 12,30168 0,044435 0,006981 27 53,16633 0,186468 0,064903 0,048703 28 58,53707 0,016417 0,056431 0,226362 29 63,28657 0,372222 0,002314 0,084844 30 72,52505 0,49785 0,005782 0,080933
Lampiran 9 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel linear No Akurasi c 1 33,67178 0,001438 2 36,51237 0,001811 3 45,37036 0,003057 4 45,84379 0,003138 5 48,2098 0,003594 6 48,54787 0,003779 7 52,40105 0,006858 8 55,57833 0,015546 9 55,71324 0,016678 10 56,25401 0,020243 11 57,20064 0,026045 12 57,47114 2,896627 13 57,47114 3,01223 14 57,4716 0,518711 15 57,53894 3,402083 16 57,53894 1,339118 17 57,53917 22,1056 18 57,53917 16,07111 19 57,6065 2,587375 20 57,6065 3,910773 21 57,60673 0,553961 22 57,60673 1,362725 23 57,74256 0,087612 24 57,80967 1,071345 25 57,81012 0,087756 26 57,87723 0,890366 27 57,87746 0,059958 28 58,01283 0,084992 29 58,01306 0,10703 30 58,21553 0,1554
Lampiran 10 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel polinomial No Akurasi c g r d 1 31,30486 0,003858 0,00186 0,023886 4,284482 2 31,30486 0,003775 0,024263 0,082084 0,216952 3 31,30486 0,00941 0,001042 0,563841 3,458126 4 31,30486 0,016061 0,000986 0,053563 0,668902 5 31,30486 0,003383 0,001206 0,019682 3,426398 6 31,30486 0,340299 0,002359 0,003377 4,499914 7 31,30486 0,069505 0,001037 0,078745 0,967169 8 31,30486 0,02451 0,020145 0,996387 0,778599 9 31,30486 0,494637 0,001374 0,002356 3,124618 10 31,30486 0,019135 0,001378 0,004884 3,692802 11 31,30486 0,009911 0,001836 0,00115 4,025562 12 31,30486 0,061064 0,059969 0,065804 0,336113 13 31,30486 0,506947 0,005193 0,002105 4,753952 14 31,30486 0,042047 0,004497 0,004139 0,782509 15 31,30486 0,041265 0,004684 0,020753 2,286544 16 31,30486 0,003448 0,010864 0,025518 4,175441 17 31,84608 0,727504 0,028936 0,012604 4,155648 18 31,91342 0,016454 0,022871 0,01489 3,054345 19 32,04879 0,008438 0,040327 0,738138 3,090502 20 32,72515 0,0016 0,192256 0,002307 4,660917 21 33,46908 0,806223 0,004142 0,448266 4,547273 22 36,37609 0,013287 0,150809 0,073812 3,772123 23 38,33944 0,025577 0,084052 0,139308 1,416339 24 49,89899 0,436296 0,554884 0,002993 4,894927 25 51,31929 0,426855 0,011368 0,009482 1,731304 26 52,33372 0,126803 0,333022 0,016454 2,487885 27 53,27966 0,159271 0,777073 0,043626 3,712025 28 53,34746 0,013365 0,371679 0,447751 3,77573 29 55,91594 0,275432 0,020131 0,876473 4,095003 30 57,13353 0,225082 0,218311 0,255192 2,093127
Lampiran 11 Hasil klasifikasi 30 parameter pada data Yeast menggunakan kernel RBF No Akurasi c g 1 31,30486 0,006223 0,111945 2 31,30486 0,041078 0,00281 3 31,30486 0,001761 30,5023 4 31,30486 0,006066 0,001861 5 31,30486 0,008574 0,021697 6 31,30486 0,127363 2,678431 7 31,30486 0,00288 0,002487 8 31,30486 0,336323 11,93413 9 31,30486 0,006629 0,251143 10 31,30486 0,022083 5,231167 11 31,30486 0,003938 0,032837 12 31,30486 0,008906 0,020755 13 31,30486 0,002053 0,001087 14 31,30486 0,012152 0,001616 15 31,30486 0,001708 1,013076 16 31,30486 0,001121 0,231894 17 31,30486 0,007546 1,524993 18 31,30486 0,002578 3,295862 19 31,30486 0,023856 0,0195 20 31,64269 0,032078 0,021496 21 43,81608 0,485711 0,003038 22 44,55955 2,061954 3,157118 23 44,96519 10,73407 2,288975 24 46,31699 10,9881 1,926045 25 48,88639 0,096444 0,518358 26 49,35799 12,83478 0,690661 27 54,49771 2,270785 0,002264 28 55,71393 0,221947 0,03085 29 57,81104 1,094952 0,18704 30 58,07948 11,93646 0,001581