POLIMORFISME GEN LEPTIN DAN GEN MIOSTATIN
PADA SAPI POTONG ACEH DAN MADURA
KAMALIAH
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis Polimorfisme Gen Leptin dan Gen Miostatin pada sapi potong Aceh dan Madura adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, Juli 2012
Kamaliah
ABSTRACT
KAMALIAH. Polymorphism of Leptin and Myostatin Genes in Aceh and Madura Cattles. Under direction of R.R. DYAH PERWITASARI and ACHMAD FARAJALLAH.
Aceh and Madura cattles are a native Indonesian cattle as meat production. Productivity of beef cattle in Indonesia is still low so it is not used sufficient to supply national demand. We used leptin and miostatin genes as a genetic marker to clustering, Marker-Assisted Selection (MAS), and marker for detection of permissible nature (Halal) food. The aim of this study was to determine polymorphism of Aceh and Madura cattles. This research applied sequencing method. The reconstruction of phylogenetic tree using Neighbour-Joining method with Kimura-2-Parameters model to analyse polymorphism. Aceh cattle based on leptin gene was clustered into one group with Bos indicus GCATC haplotype and
Bos taurus.Aceh cattle was assembled into one group with Bos taurus based on myostatin gene. Madura cattle had two haplotypes, one type was Bos indicus
GCATC haplotype or Bos taurus and another type was Bos indicus ATGCT haplotype or Bos frontalis.Madura cattle based on myostatin gene different from
Bos taurus group.
RINGKASAN
KAMALIAH. Polimorfisme Gen Leptin dan Gen Miostatin pada Sapi Potong Aceh dan Madura. Dibimbing oleh R.R DYAH PERWITASARI dan ACHMAD FARAJALLAH.
Sapi aceh dan sapi madura merupakan sapi lokal Indonesia penghasil daging. Produktivitas sapi potong di Indonesia saat ini masih rendah sehingga tidak memadai untuk memenuhi kebutuhan nasional. Upaya yang dilakukan oleh pemerintah melalui program seleksi secara konvensional memerlukan pencatatan produksi dan reproduksi dalam jangka waktu yang lama. Pendekatan seleksi lainnya yang lebih cepat adalah melalui penggunaan marka genetik. Gen yang digunakan sebagai marka genetik di antaranya adalah gen leptin dan gen miostatin. Penelitian ini bertujuan untuk mengetahui polimorfisme gen leptin dan gen miostatin pada sapi aceh dan sapi madura. Penelitian ini dapat digunakan sebagai informasi dasar untuk mendukung manajemen perkawinan sapi, mengetahui asal usul spesies, mengetahui hubungan kekerabatan spesies, dan identifikasi spesies di dalam produk daging campuran.
Metode yang digunakan untuk memperoleh sampel DNA dari darah dilakukan menggunakan metode ekstraksi KIT. Amplifikasi ekson 2 gen leptin dan ruas promotor gen miostatin dilakukan menggunakan metode Polymerase Chain Reaction (PCR). Amplikon diuji menggunakan Gel Poliakrilamid (PAGE 6%) dan dilanjutkan dengan pewarnaan perak. Perunutan nukleotida kedua gen dilakukan dengan metode automatic sequencing. Analisis keragaman nukleotida dilakukan menggunakan rekonstruksi pohon filogeni berdasarkan metode Neighbour-Joining (NJ) model Kimura-2-parameter.
Berdasarkan gen leptin sapi aceh mempunyai hubungan kekerabatan yang lebih dekat dengan B. indicus haplotipe GCATC dan B. taurus, berdasarkan gen miostatin sapi aceh mempunyai hubungan kekerabatan yang lebih dekat dengan B. taurus. Berdasarkan gen leptin sapi madura mempunyai hubungan kekerabatan yang lebih dekat dengan B. indicus haplotipe ATGCT, B. indicus haplotipe GCATC, dan B. taurus, sedangkan berdasarkan gen miostatin sapi madura berbeda dengan B. taurus.
Berdasarkan gen leptin sapi madura dikelompokkan ke dalam dua haplotipe kemungkinan karena sapi madura merupakan sapi hasil hibridisasi antara B. indicus dengan banteng. Bos indicus halotipe ATGCT mempunyai kesamaan karakter dengan sapi liar. Hibridisasi B. indicus dengan Banteng hingga membentuk sapi madura diperkirakan terjadi sejak masuknya kebudayaan hindu dari India ke Indonesia sekitar 1500 tahun yang lalu. Sapi B. indicus yang dibawa oleh bangsa India tersebut kemudian disilangkan dengan banteng yang ada di kepulauan Madura. Hubungan kekerabatan antara sapi aceh dengan B. taurus
diperkirakan terjadi sejak dilaksanakan program Inseminasi Buatan (IB) pertama sekali di peternakan Saree dan Indrapuri pada tahun 1972. Semen beku yang digunakan berasal dari keturunan B. taurus.
© Hak Cipta milik IPB, tahun 2012
Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
POLIMORFISME GEN LEPTIN DAN GEN MIOSTATIN
PADA SAPI POTONG ACEH DAN MADURA
KAMALIAH
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Biosains Hewan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Polimorfisme Gen Leptin dan Gen Miostatin pada Sapi Potong Aceh dan Madura
Nama : Kamaliah NIM : G352090111
Disetujui
Komisi Pembimbing
Dr. Ir. R.R. Dyah Perwitasari, M.Sc. Dr. Ir. Achmad Farajallah, M.Si. Anggota Anggota
Diketahui
Ketua Program Studi Dekan Sekolah Pascasarjana Biosains Hewan
Dr. DRS. Bambang Suryobroto Dr. Ir. Dahrul Syah, M.Sc.Agr.
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia dan izin-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Januari 2011 ini ialah mengenai polimorfisme gen, dengan judul Polimorfisme Gen Leptin dan Gen Miostatin pada Sapi Potong Aceh dan Madura.
Ucapan terima kasih penulis sampaikan kepada Ibu Dr. Ir. R.R. Dyah Perwitasari, M.Sc dan Bapak Dr. Ir. Achmad Farajallah, M.Si selaku pembimbing yang telah banyak memberikan saran. Selanjutnya, penulis mengucapkan terima kasih kepada pimpinan beserta staf Balai Pembibitan Ternak Unggul (BPTU) Indrapuri Kabupaten Aceh besar yang telah memberikan izin dan membantu selama pengambilan sampel. Disamping itu, ungkapan terima kasih penulis sampaikan kepada ayahanda, ibunda, serta seluruh keluarga atas segala doa, dukungan, bantuan, serta kasih sayangnya sehingga penulisan karya ilmiah ini dapat diselesaikan.
Semoga karya ilmiah ini bermanfaat.
Bogor, Juli 2012
RIWAYAT HIDUP
Penulis dilahirkan di Banda Aceh pada tanggal 15 Februari 1984 dari ayah Drs. Helmy Basjah dan ibu Azizan Ibrahim. Penulis merupakan putri bungsu dari tujuh bersaudara.
Tahun 2002 penulis lulus dari SMA Negeri 3 Banda Aceh dan pada tahun yang sama lulus seleksi masuk Universitas Syiah Kuala melalui jalur SPMB. Penulis memilih program studi Biologi, Fakultas Keguruan dan Ilmu Pendidikan. Selama mengikuti perkuliahan, penulis menjadi asisten mata kuliah Biologi dasar pada tahun ajaran 2005/2006, serta mata kuliah genetika pada tahun ajaran 2006/2007.
DAFTAR ISI
Sejarah Asal Usul Sapi Lokal Indonesia ... 3
Sapi Aceh ... 4
Sapi Madura ... 5
Penanda Genetik untuk Marker-Assisted Selection (MAS) ... 6
Hubungan Antara Polimorfisme Gen Leptindengan Kualitas Daging ... 7
Hubungan antara Gen Miostatindengan Otot Ganda... 9
BAHAN DAN METODE ... 11
Produk Amplifikasi dan Perunutan Nukleotida Berdasarkan Gen Leptin dan Gen Miostatin ... 16
Haplotipe Gen Leptin pada Sapi Aceh dan Sapi Madura ... 17
Keragaman Gen Leptin dan Perubahan Asam Amino ... 18
Keragaman Gen Miostatin pada Sapi Aceh dan Sapi Madura ... 19
Analisis Filogeni Berdasarkan GenLeptin ... 20
PEMBAHASAN ... 24
Keragaman Gen Leptin dan Gen Miostatin Terhadap Pengelompokkan Sapi Aceh dan Sapi Madura ... 24
DAFTAR TABEL
Halaman
1 Sampel sapi yang digunakan pada penelitian... 11
DAFTAR GAMBAR
5 Produk amplifikasi gen leptin dan gen miostatin . ... 16
6 Posisi kodon awal (ATG) pada daerah ekson 2 gen leptin ... 17
7 Posisi tiga titik mutasi menunjukkan perbedaan haplotipe. ... 18
8 Keragaman gen leptin pada sapi aceh dan sapi madura ... 19
9 Keragaman gen miostatin pada sapi aceh dan sapi madura ... 20
10 Rekonstruksi pohon filogeni gen leptin berdasarkan nukleotida menggunakan metode Neighbour-Joining (NJ), model Kimura-2-Parameter, dan butsrap pengulangan 1000x. ... 21
11 Rekonstruksi pohon filogeni gen miostatin menggunakan metode Neighbour-Joining (NJ) berdasarkan model Kimura-2-Parameter dengan butsrap pengulangan 1000x. ... 23
DAFTAR LAMPIRAN Halaman 1 Hasil pengurutan basa nukleotida gen leptin daerah ekson 2 antara sapi aceh, sapi madura, dan data GenBank. ... 32
PENDAHULUAN
Latar Belakang
Sapi potong merupakan hewan ternak utama penghasil daging setelah ayam. Produktivitas sapi potong di Indonesia saat ini masih rendah sehingga tidak memadai untuk memenuhi kebutuhan nasional. Salah satu upaya yang dapat dilakukan oleh pemerintah untuk perbaikan produktivitas sapi potong adalah melalui program pemuliaan. Program ini dapat memperbaiki mutu genetik ternak dengan menerapkan suatu pola perkawinan yang direncanakan dan dilanjutkan dengan tindakan seleksi. Program seleksi yang dilakukan secara konvensional dinilai tidak efektif. Program seleksi yang telah ditempuh memerlukan pencatatan
produksi dan reproduksi dalam jangka waktu yang relatif lama. Pendekatan seleksi lainnya yang lebih cepat adalah penggunaan marka genetik. Oleh karena itu marka genetik diperlukan sebagai informasi dasar yang berhubungan dengan ruas gen dan keragaman nukleotida berdasarkan gen yang dipilih.
Gen yang digunakan sebagai marka genetik di antaranya adalah gen leptin dan gen miostatin. Gen leptin telah dilaporkan sebagai gen utama pengontrol kualitas daging. Gen leptin terlibat di dalam pengaturan metabolisme lemak tubuh. Lemak intramuskular merupakan lemak yang dapat menyebabkan keempukan daging. Perbedaan tingkat lemak intramuskular pada bangsa sapi dipengaruhi oleh adanya perbedaan genotipe (Corva et al. 2009). Gen miostatin atau Growth Differentiation Factor (GDF-8) merupakan gen yang terlibat dalam mengatur pertumbuhan otot rangka. Mutasi pada gen miostatin menyebabkan fenomena otot ganda (Double Muscle), terutama karena pergeseran stop kodon akibat insersi, delesi, atau mekanisme ekspresi yang terganggu. Selain itu, polimorfisme gen miostatin secara gradual mempengaruhi kualitas pertumbuhan otot rangka.
Polimorfisme yang ditemukan pada gen leptin dan gen miostatin mempengaruhi deposisi lemak intramuskular dan pertumbuhan otot rangka dalam mendukung upaya peningkatan kualitas daging. Selain itu, polimorfisme gen
Sapi aceh dan sapi madura merupakan sapi lokal Indonesia yang dikenal mempunyai kualitas daging yang baik. Polimorfisme gen leptin dan gen miostatin pada sapi aceh dan sapi madura belum pernah dilaporkan. Pemanfaatan polimorfisme gen leptin dan gen miostatin diharapkan dapat menunjang seleksi
Marker-Assisted Selection (MAS) pada seleksi sapi aceh dan sapi madura.
Tujuan
Penelitian ini bertujuan untuk mengetahui polimorfisme gen leptin dan gen miostatin pada sapi aceh dan sapi madura.
Manfaat
TINJAUAN PUSTAKA
Sejarah Asal Usul Sapi Lokal Indonesia
Ternak sapi merupakan anggota famili bovidae yang muncul pada era Pleistosen. Ternak sapi berasal dari keturunan aurok liar (Bos primigenius)
(Mannen et al. 1998). Bos primigenius dibedakan menjadi tiga subtipe berdasarkan perbedaan lokasi geografis fosil ditemukan, yaitu B. primigenius primigenius ditemukan di Eropa, B. primigenius namadicus ditemukan di Asia, dan B. primigenius opisthonomus ditemukan di Afrika. Perkembangan B. primigenius membentuk 2 tipe sapi modern, yaitu B. taurus (tidak berpunuk) dan
B. indicus (berpunuk) (Loftus et al. 1994). Domestikasi B. primigenius
berkembang menjadi sapi modern seperti sapi sekarang ini. Domestikasi B. primigenius berlangsung di Asia Barat, tepatnya di antara Mediteranian dan Iran. Lokasi yang diperkirakan sebagai pusat domestikasi adalah Jordania, Lebanon, Syiria, Turkey, Irak, dan Iran. Pada saat itu B. primigenius subsp. primigenius dan
B. primigenius subsp. namadicus hidup berdampingan (Payne & Hodges 1997).
Bos primigeniusnamadicus merupakan nenek moyang dari sapi domestik Zebu (Bos indicus). Pusat domestikasi B. indicus diperkirakan di Iran kemudian berkembang menuju dua jalur utama, yaitu dari Turkmenia menuju India dan melanjutkan ke Asia bagian selatan, dan dari Bulukistan menuju Persia dan Mesopotamia selatan. Zebu dari Persia bermigrasi menuju Arab dan melanjutkan ke Afrika (Payne & Hodges 1997).
Sejarah masuknya B. indicus ke Indonesia diperkirakan bersamaan dengan masuknya kebudayaan hindu dari India sekitar 1500 tahun yang lalu (Uggla 2008). Jenis sapi B. indicus yang berkembang di Indonesia saat ini adalah sapi aceh, sapi pesisir, sapi madura, ongole, peranakan ongole, dan grati. Jenis sapi lainnya yang berkembang di Indonesia adalah sapi bali. Sapi bali (Bos javanicus)
merupakan sapi asli Indonesia. Asal usul sapi bali berbeda dari B.indicus maupun
Sapi Aceh
Sapi aceh merupakan sapi lokal Indonesia yang berkembang di daerah Nanggroe Aceh Darussalam (NAD) dan sekitarnya. Sapi aceh berasal dari
B.indicus yang dibawa oleh bangsa India sekitar 1500 tahun yang lalu (Kusdiantoro et al. 2009; Uggla 2008). Sapi aceh dikelompokkan ke dalam
B.indicus berdasarkan karakter morfologi sapi aceh berpunuk. Jenis sapi lainnya yang diintroduksi ke Aceh adalah sapi peranakan ongole. Peranakan ongole menyebar di kabupaten Aceh Besar, Pidie, Aceh Utara, dan Aceh Timur. Persebaran peranakan ongole dilakukan disilangkan dengan sapi aceh sebagai salah satu program perbaikan mutu sapi aceh yang dilakukan oleh pemerintah. Persilangan sapi aceh dengan peranakan ongole mulai dilakukan pada tahun 1968. Selanjutnya, program perbaikan mutu sapi aceh yang dilanjutkan oleh pemerintah adalah program Inseminasi Buatan. Kegiatan Inseminasi Buatan di Aceh mulai dilaksanakan pada bulan April 1972. Inseminasi Buatan dilaksanakan di Balai Peternakan Saree dan Indrapuri kabupaten Aceh Besar. Program tersebut menggunakan semen beku berasal dari bangsa sapi Brahman dan Santagertrudis. Semen beku selanjutnya yang diintroduksi ke Aceh sampai saat ini berasal dari bangsa sapi Simental, Brahman, Limousin, dan Charollais (Sari 2011).
Karakter warna tubuh sapi aceh bervariasi, yaitu merah bata, coklat, hitam,
Gambar 1 Karakter warna tubuh sapi aceh.
Sapi Madura
Sapi madura merupakan sapi lokal Indonesia yang berkembang di kepulauan Madura dan sekitarnya. Asal usul sapi madura diperkirakan dari hasil persilangan antara B. javanicus dengan B. indicus (Nijman et al. 2003). Perkawinan silang tersebut diperkirakan terjadi sejak masuknya B. indicus dari India. Bos indicus dibawa oleh bangsa India ke Indonesia bersamaan dengan masuknya kebudayaan hindu sekitar 1500 tahun yang lalu. Kemudian B. indicus
tersebut terjadi perkawinan silang dengan B. javanicus yang ada di kepulauan Madura. Jenis B. indicus lainnya yang didatangkan dari India adalah sapi ongole. Sapi ongole masuk ke Sumba pada tahun 1906. Sapi ongole disebarkan ke
wilayah Indonesia untuk disilangkan dengan sapi potong lokal termasuk sapi madura sebagai salah satu upaya meningkatkan kualitas sapi potong Indonesia.
Gambar 2 Karakter morfologi sapi madura.
Penanda Genetik untuk Marker-Assisted Selection (MAS)
Marker-Assisted Selection (MAS) merupakan proses seleksi individu yang dijadikan sebagai parental untuk generasi selanjutnya. Seleksi berdasarkan
Marker-Assisted Selection (MAS) bertujuan untuk meningkatkan kualitas genetik pada suatu bangsa ternak. DNA merupakan penanda genetik yang digunakan pada program MAS. DNA digunakan untuk menyeleksi alel yang menguntungkan. Alel yang terseleksi yaitu alel yang berpengaruh positif terhadap fenotipe yang bernilai ekonomis. Dominasi fenotipe dipengaruhi oleh dominasi alel berdasarkan hukum mendel, yaitu dominan dan resesif. Kualitas genetik dapat ditingkatkan jika
genotipe pada bangsa ternak bersifat heterozigot sehingga variasi genetik berperan penting dalam program MAS.
perkawinan silang. Teknik mendeteksi polimorfisme pada tingkat DNA diantaranya Restriction Fragment Length Polymorphism (RFLP), Random Amplified Polymorphic DNA (RAPD), AmplifiedFragment Length Polymorphism
(AFLP), Single-Strand Conformation Polymorphisms (SSCP), Mikrosatelit, dan sekuensing.
Hubungan Antara Polimorfisme Gen Leptindengan Kualitas Daging
Leptin merupakan protein hormon yang dikeluarkan dari jaringan adiposa (Frederich et al. 1995). Hormon leptin terlibat di dalam proses regulasi metabolisme dan massa lemak di dalam tubuh (Kim & Moussa 2000; Houseknecht et al. 2000; Sharifzadeh & Doosti 2010). Pada sapi massa lemak dapat digunakan sebagai pengontrol untuk menentukan kualitas daging. Lemak intramuskular merupakan lemak yang dapat menentukan kualitas daging seperti keempukan daging dan kandungan air di dalam daging (Te pas et al. 2004). Lemak intramuskular dikendalikan oleh beberapa gen utama. Salah satu gen yang mengendalikan deposisi lemak intramuskular adalah gen leptin (Hirwa et al.
2011).
Gen leptin merupakan gen obesitas yang terletak pada kromosom 4 (Friedman 2002; Pomp et al. 1997). Struktur gen leptin terdiri atas 3 ekson dan 2 intron (Taniguchi et al. 2002). Daerah ekson 1 merupakan daerah UTR (Untranslated region). Daerah ini tidak ditranskripsikan menjadi mRNA sehingga tidak ditranslasikan ke dalam asam amino. Kodon awal dimulai dari beberapa basa pada awal ekson 2, sedangkan kodon akhir diakhiri pada ekson 3 (Gambar 3).
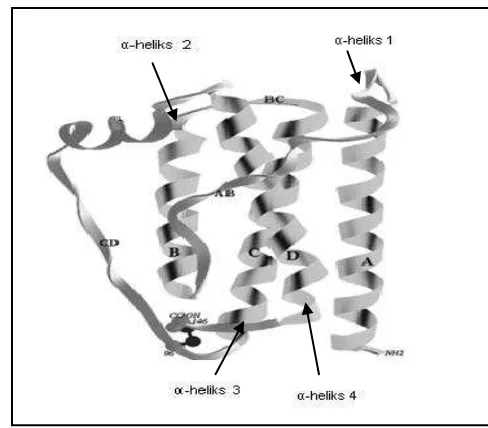
Gen leptin menyandikan 167 asam amino dengan berat molekul protein sebesar 16 kDa (Frederich et al. 1995). Protein leptin berbentuk struktur sekunder 4α-helix (Gambar 4) (Liefers 2004). Struktur α-helix terbentuk karena adanya ikatan hidrogen antara asam amino yang satu dengan asam amino lainnya di dalam satu helai yang sama (Wilmer et al. 2005).
Perbedaan genotipe pada gen leptin berhubungan dengan kandungan lemak intramuskular, berat tubuh, dan volume susu. Pada daerah ekson 2 genotipe TT menghasilkan deposisi lemak intramuskular lebih tinggi dibandingkan dengan genotipe CC pada posisi 118026 pb (Corva et al. 2009). Pada sapi Iran genotipe AB menghasilkan bobot tubuh lebih tinggi dibandingkan dengan genotipe AA (Nobari 2010). Liefer et al. (2002) menyatakan bahwa pada sapi Frisien-Heifer genotipe CT memproduksi susulebih banyak dibandingkan dengan genotipe CC.
Polimorfisme gen leptin menyebabkan terbentuknya variasi frekuensi genotipe pada setiap jenis sapi. Pada daerah ekson 2 genotipe TT tidak ditemukan pada sapi golpayegani (Nassiry et al. 2007). Genotipe CC merupakan genotipe yang paling tinggi ditemukan pada sapi hasil perkawinan silang, sedangkan genotipe CT merupakan frekuensi paling tinggi ditemukan pada Holstein Friesian
(Choundhary et al. 2005). Pada daerah ekson 3 genotipe CT merupakan genotipe yang paling tinggi di antara genotipe lainnya untuk sapi sistani dan sarabi (Aslaminejad 2010).
Polimorfisme gen leptin daerah ekson 2 terjadi akibat perubahan nukleotida C→T pada posisi 1180. Perubahan nukleotida C→T menyebabkan terjadinya perubahan asam amino arginin menjadi sistein (Javanmard et al. 2010). Pada daerah ekson 3 terjadi akibat perubahan nukleotida C→T yang menyebabkan perubahan asam amino valin menjadi alanin (Lagonigro et al. 2003). Selain itu, pada daerah intron 2 menyebabkan perubahan nukleotida G→A (Lien et al. 1997). Polimorfisme pada daerah ekson kemungkinan mengubah asam amino yang menyebabkan perubahan ekspresi protein. Polimorfisme daerah intron meskipun tidak mengubah asam amino tetapi perperan penting pada proses
Gambar 4 Struktur protein 4α-helix pada gen leptin(Zhang et al. 1997).
Hubungan antara Gen Miostatindengan Otot Ganda
Miostatin merupakan protein yang terlibat di dalam proses regulasi perkembangan massa otot (McPherron & Lee 1997). Perkembangan massa otot terjadi karena adanya peningkatan jumlah otot (Hiperplasia) dan peningkatan massa otot (Hipertropi). Massa otot merupakan salah satu faktor yang menentukan sifat ekonomis sapi potong.
Gen miostatin terletak pada kromosom 2 (Charlier et al. 1995). Struktur gen miostatin terdiri atas 3 ekson dan 2 intron (McPherron & Lee 1997). Panjang ekson 1 dan ekson 2 yaitu 373 pb dan 374 pb. Ekson 1 dan ekson 2 dipisahkan oleh intron 1 sepanjang 1849 pb. Sedangkan ekson 2 dan ekson 3 dipisahkan oleh intron sepanjang 2033 pb. Panjang ekson 3 adalah 381 pb. Daerah UTR
(Untranslated region) terletak pada akhir ekson 3 dengan ukuran yang bervariasi tergantung dari posisi poliadenilasi (Grisolia et al. 2009). Gen miostatin terdiri atas 20 haplotipe (Dunner et al. 2002).
Esmailizadeh et al. 2008). Ekspresi otot ganda pada sapi pertama sekali ditemukan pada sapi Belgian Blue dan Piedmontase. Otot ganda Belgian Blue disebabkan karena terjadi 11 delesi nukleotida sepanjang 931-936 pb. Delesi nukleotida tersebut terjadi pada daerah ekson 3. Fenomena delesi menyebabkan terjadinya kehilangan daerah aktif pada molekul protein miostatin (McPherron & Lee 1997). Ekspresi otot ganda pada Piedmontase terjadi karena adanya mutasi pada daerah ekson 3 menyebabkan perubahan nukleotida G→A pada posisi 938 pb. Perubahan nukleotida G→A menyebabkan terjadinya perubahan asam amino sistein menjadi tirosin. Selain itu, perubahan nukleotida ditemukan pada daerah ekson 1 menyebabkan perubahan transisi C→A pada posisi 282 pb. Substitusi nukleotida tersebut menyebabkan terjadinya perubahan asam amino leusin menjadi fenilalanin (McPherron & Lee 1997).
Polimorfisme gen miostatin juga ditemukan pada sapi nellore. Pada daerah ekson 1 ditemukan 3 polimorfisme, yaitu nt76(A/T), nt11(G/T), dan nt267(A/G). Pada daerah ekson 2 ditemukan 7 polimorfisme, yaitu nt414(C/T), nt433(A/T), nt420(T/G), nt445(A/T), nt527(T/A), nt641(G/A), dan nt694(G→A). Pada daerah ekson 3 ditemukan 4 polimorfisme, yaitu nt840(G→A), nt1083(C/T),
BAHAN DAN METODE
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan dari bulan Januari hingga Juli 2011 di Laboratorium Bagian Fungsi Hayati dan Perilaku Hewan Departemen Biologi FMIPA IPB.
Koleksi Sampel
Sampel yang digunakan adalah sampel darah sapi madura berasal dari koleksi Laboratorium Bagian Fungsi Hayati dan Perilaku Hewan Departemen Biologi FMIPA IPB dan sampel darah sapi aceh berasal dari Balai Pembibitan Ternak Unggul (BPTU) Indrapuri Aceh besar. Jumlah keseluruhan sampel yang digunakan 83 sampel (Tabel 1).
Tabel 1 Sampel sapi yang digunakan pada penelitian
No. Bangsa Sapi Lokasi Kode
Keterangan ; Kab = Kabupaten, Kml = Kamaliah (Kolektor), AF = Achmad Farajallah (Kolektor), Bos = Genus sapi, A=Aceh.
Ekstraksi Sampel
Ekstraksi yang digunakan untuk amplifikasi gen leptin menggunakan
Kapa Express Extract (Kapabiosystems). Ekstraksi yang digunakan untuk amplifikasi gen miostatin menggunakan Genomic DNA Mini Kit for Fresh Blood
Tahapan ekstraksi Kapa Express Extract terdiri atas proses lisis dan inaktivasi enzim protease. Endapan sel darah sebanyak 10 μl dilarutkan dalam 10x buffer Kapa Express Extract dan 1 unit enzim protease Kapa Express Extract. Larutan dilisis pada suhu 75 0C selama 10 menit. Selanjutnya, enzim protease dilakukan inaktivasi pada suhu 95 0C selama 5 menit. Reaksi diendapkan dengan sentrifugasi pada kecepatan 13000 rpm selama 1 menit. Supernatan merupakan larutan yang diambil untuk tahap amplifikasi.
Ekstraksi Genomic DNA Mini Kit for Fresh Blood (Geneaid) yang digunakan dimodifikasi pada tahap pencucian sampel darah dari alkohol dan tahap resuspensi yang menggunakan air destilata steril. Endapan sel darah sebanyak 200 μl dilisis menggunakan larutan GB buffer sebanyak 200 μl, kemudian diinkubasi
pada suhu 35 0C selama 10 menit. Selanjutnya DNA diikat pada matriks di dalam tabung. Tahap pengikatan DNA pada matriks bertujuan untuk memisahkan DNA dari makromolekul sel lainnya. DNA terikat pada matriks, sedangkan makromolekul sel lainnya mengendap pada bagian dasar tabung. DNA pada matriks dicuci menggunakan larutan 400 μl W1 buffer dan 600 μl Wash buffer. Selanjutnya DNA diencerkan menggunakan larutan Elution buffer sebanyak 100 μl.
Amplifikasi Gen Leptin
Gen leptin yang diamplifikasi yaitu ekson 2 hingga ekson 3 menggunakan mesin PCR (Polymerase Chain Reaction) ESCO Swift Maxi Thermal Cycler. Pasangan primer yang digunakan mengapit daerah ekson 2, yaitu primer forward
L5 (5’-CCATGGCAGACAGCAAATCTCGT-3’) dan primer reverse L6 (5’
elongasi pada suhu 720C selama 15 detik. Elongasi akhir pada suhu 72 0C selama 10 menit.
Amplifikasi daerah ekson 3 menggunakan pasangan primer forward L1 (5’-GTCTGGAGGCAAAGGGCAGAGT-3’)dan primer reverse L2 ( 5’ -CCACCACCTCTGTGGAGTAG-3’) (Lien et al. 1997). Panjang daerah ekson 3
yang diapit oleh sepasang primer tersebut 522 pb. Reaksi dan kondisi amplifikasi gen leptin daerah ekson 3 sama dengan reaksi dan kondisi amplifikasi pada daerah ekson 2, tetapi amplifikasi daerah ekson 3 menggunakan suhu penempelan primer pada 640C selama 15 detik.
Amplifikasi Gen Miostatin
Gen miostatin pada ruas promotor diamplifikasi menggunakan mesin PCR (PolymeraseChainReaction) ESCO Swift Maxi Thermal Cycler. Pasangan primer yang digunakan untuk amplifikasi gen miostatin ruas promotor adalah primer forward AF56 (5’-TTCAGGCTACTGAGTTGCATTTT-3’)dan reverse AF74 (5’-GCTTTCCAGCGGTAAAAGAA-3’). Urutan basa primer yang digunakan
disusun menggunakan program Primer3 berdasarkan species Bos taurus (No.
aksesAF348479.1) dari data GenBank. Sepasang primer tersebut mengapit daerah sekuen target sepanjang 580 pb. Volume reaksi total amplifikasi sebanyak 12 μl terdiri atas 1 unit KapaTaq DNA Polymerase ReadyMix (MgCl2 2mM dan dNTP 0,4 mM), masing-masing primer 0,4 μM, dan genom DNA 10-100 ng. Kondisi
pewarnaan perak berdasarkan Byun et al. (2009). Penentuan ukuran pita DNA dilakukan menggunakan rumus Fungsi Regresi Linier.
Pengurutan Nukleotida
Pengurutan nukleotida menggunakan jasa pelayanan perusahaan sekuensing. Primer yang digunakan untuk pengurutan nukleotida pada gen leptin daerah ekson 2 sama dengan primer yang digunakan pada tahap amplifikasi (L5 dan L6). Sedangkan primer yang digunakan untuk gen miostatin pada ruas promotor adalah primer forward (AF56). Gen leptin daerah ekson 3 tidak dapat dilanjutkan ke tahap sekuensing karena produk amplifikasi gen leptin daerah ekson 3 memberikan pita DNA lainnya yang bukan merupakan pita target.
Analisis Data
Data dalam bentuk urutan basa nukleotida diedit menggunakan program BioEdit versi 7.0.9.0 dan Genetyx-Win versi 4.0. Data tersebut disejajarkan menggunakan program Geneious versi 5.4.4. Kemudian data diedit kembali dan dianalisis menggunakan program Mega 5.05.
Urutan nukleotida disejajarkan dengan data dari data GenBank. Gen Leptin
disejajarkan dengan Bos indicus haplotipe ATGCT (No. akses FJ626856.1), Bos indicus haplotipe GCATC (No. akses FJ626855.1), Bos taurus (No. akses AJ512638.1), Bos frontalis (No. akses EU642566.1), dan Bos taurus Χ Bos indicus (No. akses EU921637.1). Gen Miostatin disejajarkan dengan Bos taurus
(No. aksesAF348479.1).
Persentase komposisi nukleotida pada gen Leptin dan gen Miostatin
menggunakan aplikasi yang telah tersedia pada program Mega 5.05. Urutan nukleotida yang telah diedit disejajarkan menggunakan pensejajaran Crustal W. Pada gen Leptin perubahan nukleotida menjadi asam amino menggunakan program Genetyx-Win versi 4.0.
dengan nilai bootstrap 1000x pengulangan. Filogeni berdasarkan urutan asam amino menggunakan nilai bootstrap 1000x pengulangan.
Rekonstruksi pohon filogeni pada gen Miostatin dilakukan berdasarkan urutan nukleotida menggunakan metode Neighbour-Joining (NJ), model Kimura-2-parameter, dan nilai bootstrap 1000x pengulangan.
HASIL
Produk Amplifikasi dan Perunutan Nukleotida Berdasarkan Gen Leptin dan Gen Miostatin
Hasil amplifikasi gen leptin menggunakan pasangan primer forward L5 dan reverse L6 menunjukkan ukuran 234 pb (Gambar 5a), dan amplifikasi gen miostatin menggunakan pasangan primer forward AF56 dan reverse AF74 menghasilkan produk dengan ukuran 580 pb (Gambar 5b). Sampel yang berhasil teramplifikasi berjumlah sembilan sampel, yaitu tiga sampel berasal dari populasi sapi aceh dan enam sampel berasal dari populasi sapi madura.
(a) (b)
Gambar 5 Produk amplifikasi gen leptin daerah ekson 2 (a) dan gen miostatin (b). M:Penanda, 1:sapi aceh (Kml_BosA11), 2:sapi aceh (Kml_BosA12), 3:sapi aceh (Kml_BosA13), 4:sapi madura (AF14), 5:sapi madura (AF26), 6:sapi madura (AF38), 7:sapi madura (AF32), 8:sapi madura (AF41), 9:sapi madura (AF29).
Perunutan nukleotida gen leptin setelah disejajarkan menunjukkan ukuran
sebesar 176 pb. Sisa nukleotida yang tidak berhasil disejajarkan merupakan primer reverse dan nukleotida setelahnya. Nukleotida yang tidak berhasil disejajarkan disebabkan oleh faktor efisiensi bahan kimia yang digunakan pada tahap amplifikasi sehingga menghasilkan duplikasi pada elektroferogram. Primer
reverse merupakan salah satu faktor yang menentukan keberhasilan amplifikasi. Pada posisi awal runutan nukleotida ditemukan primer forward sesuai dengan daerah yang ditargetkan. Posisi primer forward berada pada -151 nt dari kodon awal ATG. Posisi kodon awal ATG pada gen leptin terletak pada ekson 2. Posisi
kodon awal pada penelitian ini ditemukan sesuai dengan posisi pada urutan referensi dari data GenBank, yaitu B. indicus haplotipe GCATC dengan no asesi FJ626855.1 (Gambar 6). Perunutan nukleotida gen miostatin setelah disejajarkan dengan urutan referensi dari GenBank menunjukkan ukuran sebesar 523 pb. Sisa nukleotida dari hasil amplifikasi merupakan primer forward sebesar 23 pb dan nukleotida setelahnya sebesar 38 pb.
Gambar 6 Posisi kodon awal (ATG) pada daerah ekson 2 gen leptin berdasarkan
B. indicus (No. akses FJ626855.1).
Haplotipe Gen Leptin pada Sapi Aceh dan Sapi Madura
Urutan nukleotida gen leptin sepanjang 176 pb yang disejajarkan dengan urutan referensi dari data GenBank menunjukkan polimorfisme. Polimorfisme tersebut membedakan sapi aceh dan sapi madura dengan B. indicus haplotipe ATGCT, B. indicus haplotipe GCATC, B. taurus, B. frontalis, dan sapi hibridisasi
B. indicus x B. taurus dari data GenBank. Secara umum gen leptin terdiri atas dua haplotipe, yaitu haplotipe ATGCT dan haplotipe GCATC. Perbedaan haplotipe pada gen leptin didasarkan pada lima titik mutasi. Pada penelitian ini perbedaan haplotipe hanya ditemukan pada tiga titik mutasi, yaitu pada posisi 32 nt, 87 nt, dan 89 nt (Lampiran 1). Perbandingan urutan nukleotida antara sapi aceh dengan
ATGCT, sedangkan runutan nukleotida sampel sapi madura lainnya mengikuti pola haplotipe GCATC (Gambar 7b).
(a)
(b)
Gambar 7 Posisi tiga titik mutasi pada penelitian ini menunjukkan perbedaan haplotipe: (a) Haplotipe GCATC pada sapi aceh, (b) Haplotipe ATGCT dan haplotipe GCATC pada sapi madura.
Keragaman Gen Leptin dan Perubahan Asam Amino
Pada gen leptin daerah ekson 2 antara sapi aceh dan sapi madura dengan
B. indicus haplotipe ATGCT, B. indicus haplotipe GCATC, B. taurus, dan sapi hibridisasi B. indicus x B. taurus dari data GenBank ditemukan 12 varian. Tiga varian membedakan antara sapi aceh, sapi madura, dan runutan referensi dari data
hibridisasi antara B. indicus dengan B. taurus, dan satu polimorfisme ditemukan pada dua sampel sapi aceh dan satu sampel sapi madura (Gambar 8).
Perubahan satu basa nukleotida pada dua sampel sapi aceh dan satu sampel sapi madura ditemukan pada posisi - 91 nt dari kodon awal ATG. Pada posisi tersebut dua sampel sapi aceh dan satu sampel sapi madura mempunyai basa T, sedangkan satu sampel sapi aceh, lima sampel sapi madura, dan populasi lainnya dari data GenBank mempunyai basa C (Gambar 8). Tiga basa nukleotida yang membedakan antara haplotipe ATGCT dengan haplotipe GCATC ditemukan pada posisi -63 nt, -65 nt, dan -120 nt. Pada posisi -63 nt mengalami perubahan basa A menjadi basa G. Pada posisi -65 nt mengalami perubahan basa T menjadi basa C. Pada posisi -120 nt ditemukan perubahan basa G menjadi basa A. Mutasi yang terjadi pada posisi sebelum kodon awal ATG tidak ditranskripsikan ke dalam asam amino.
Gambar 8 Keragaman gen leptin daerah ekson 2 pada sapi aceh dan sapi madura
Keragaman Gen Miostatin pada Sapi Aceh dan Sapi Madura
yang membedakan antara sapi madura dengan sapi aceh dan B. taurus adalah perubahan basa T menjadi basa C pada posisi 499 nt. Selain itu, enam mutasi lainnya tidak hanya membedakan antara sapi madura dengan sapi aceh tetapi juga membedakan antar sampel sapi madura, yaitu perubahan basa C→T pada posisi 350 nt, perubahan basa G→A pada posisi 423 nt, perubahan basa A→G pada posisi 456 nt, perubahan basa C→G pada posisi 459 nt, perubahan basa A→G pada posisi 490 nt, dan satu delesi basa T ditemukan pada sampel sapi madura pada posisi 560 nt (Gambar 9).
Gambar 9 Keragaman gen miostatin daerah promotor pada sapi aceh dan sapi madura
Analisis Filogeni Berdasarkan GenLeptin
Rekonstruksi pohon filogeni gen leptin berdasarkan persamaan dan perbedaan haplotipe. Gen leptin mempunyai dua kelompok haplotipe secara umum, yaitu haplotipe ATGCT dan haplotipe GCATC. Haplotipe dibedakan berdasarkan lima titik mutasi. Pada penelitian ini perbedaan haplotipe sepanjang nukleotida yang telah diurutkan hanya ditemukan tiga titik mutasi. Pada pohon filogeni sapi aceh berada pada percabangan haplotipe GCATC, sedangkan sapi madura mempunyai dua kelompok percabangan, yaitu percabangan haplotipe AGCAT dan haplotipe ATGCT (Gambar 10). Urutan haplotipe sapi aceh
kelompok dengan B. indicus haplotipe ATGCT (No. Asesi FJ626856.1), sedangkan sapi madura mempunyai pola haplotipe GCATC berada pada kelompok B. indicus haplotipe GCATC (No. Asesi FJ626855.1). Percabangan antara sapi madura dengan B. indicus haplotipe ATGCT menunjukkan nilai bootstrap sebesar 89% berdasarkan nukleotida. Nilai bootstrap di atas (≥50%) menunjukkan kekuatan percabangan (Robust) yang mendukung pengelompokan sapi madura dengan B. indicus haplotipe ATGCT (No. Asesi FJ626856.1). Selain sapi madura, jenis sapi lainnya yang mempunyai kemiripan urutan nukleotida dengan B. indicus haplotipe ATGCT adalah B. frontalis (EU642566.1), tetapi titik mutasi posisi -64 nt pada B. frontalis berbeda dengan B. indicus haplotipe ATGCT, sehingga pada percabangan filogeni B. frontalis menunjukkan
sistergroup dari kelompok sapi madura dan B. indicus haplotipe ATGCT.
Gambar 10 Rekonstruksi pohon filogeni gen leptin berdasarkan nukleotida menggunakan metode Neighbour-Joining (NJ), model Kimura-2-Parameter, dan butsrap pengulangan 1000x.
aceh (Kml_BosA11 dan Kml_BosA13) dan sapi madura (AF38) menunjukkan percabangan khusus di dalam kelompok B. indicus haplotipe GCATC pada pohon filogeni. Percabangan tersebut menunjukkan nilai bootstrap sebesar 63%. Pengelompokan tersebut membentuk percabangan tersendiri mempunyai perbedaan pada satu nukleotida.
Pengelompokan sapi aceh dan sapi madura dengan sapi hibridisasi antara
B. indicus dengan B. taurus tidak pada satu percabangan yang sama. Sapi B. indicus X B. taurus mempunyai kesamaan haplotipe dengan B. indicus haplotipe GCATC (No. Asesi FJ626855.1), tetapi sapi hibridisasi tersebut mengalami mutasi di delapan posisi nukleotida. Perbedaan posisi nukleotida antara sapi hibrid dengan sapi aceh dan sapi madura haplotipe GCATC menyebabkan percabangan pada pohon filogeni terpisah antara kelompok sapi aceh dan sapi madura haplotipe GCATC dengan kelompok sapi B. indicus X B. taurus.
Analisis Filogeni Berdasarkan GenMiostatin
Topologi pohon filogeni berdasarkan gen miostatin memisahkan sapi aceh dengan sapi madura (Gambar 11). Pemisahan kelompok sapi aceh dengan sapi madura disebabkan oleh tujuh variasi nukleotida. Kelompok sapi aceh berada pada satu percabangan dengan B. taurus dari data GenBank (No. akses AF348479.1). Pada pohon filogeni percabangan sapi madura antar sampel yang digunakan menunjukkan perbedaan pengelompokan. Sampel sapi madura (AF38, AF14, AF26, dan AF41) mengelompok pada percabangan yang sama. Cabang pohon filogeni pada kelompok sapi madura (AF38, AF14, AF26, dan AF41) didukung dengan nilai bootstrap sebesar 94%. Sapi madura (AF29) dan sapi madura (AF32) terpisah dari percabangan pertama. Perbedaan cabang antara sapi madura (AF38, AF14, AF26, dan AF41) dengan sapi madura (AF29) dan sapi
madura (AF32) disebabkan tiga variasi nukleotida. Percabangan sapi madura (AF32) terpisah dari kelompok sapi madura lainnya. Perbedaan cabang antara sapi madura (AF32) terpisah dengan sapi madura lainnya disebabkan adanya dua variasi nukleotida. Pengelompokan sapi madura tersebut didukung dengan nilai
Pada penelitian ini Ovis aries (DQ530260.1) digunakan sebagai outgroup
untuk membedakan cabang pengelompokan sapi aceh dengan sapi madura. Ovis aries mempunyai karakter nukleotida gen Leptin yang sangat berbeda dengan sapi aceh dengan sapi madura, sehingga percabangan O. aries pada pohon filogeni tidak masuk ke dalam ingrup. Percabangan O. aries yang berbeda dari ingroup
(Sapi aceh dan sapi madura) disebabkan adanya 40 variasi nukleotida.
PEMBAHASAN
Keragaman Gen Leptin dan Gen Miostatin Terhadap Pengelompokkan Sapi Aceh dan Sapi Madura
Keragaman gen leptin dikelompokkan berdasarkan haplotipe, sedangkan keragaman gen miostatin tidak dikelompokkan berdasarkan haplotipe. Gen miostatin mempunyai 20 haplotipe (Dunner et al. 2003). Haplotipe pertama berada pada daerah ekson satu. Pada penelitian ini keragaman gen miostatin yang dianalisis adalah pada ruas promotor sehingga haplotipe gen miostatin tidak dapat terdeteksi. Gen leptin mempunyai dua haplotipe, yaitu haplotipe GCATC dan haplotipe ATGCT. Kedua haplotipe tersebut ditemukan pada sapi madura. Fenomena ini kemungkinan disebabkan oleh asal sapi madura dari dua moyang yang berbeda. Penelitian ini memperkuat simpulan Nijman et al. (2003) yang menyatakan bahwa sapi madura diperkirakan berasal dari hasil hibridisasi antara
B. indicus dengan banteng (B. javanicus).
Pengelompokan gen leptin dan gen miostatin berdasarkan analisis filogeni menunjukkan topologi yang berbeda (Gambar 10 & Gambar 11). Perbedaan topologi tersebut terletak pada pemisahan pengelompokan antara sapi aceh dengan sapi madura. Gen miostatin memisahkan kelompok sapi aceh dengan kelompok sapi madura sedangkan topologi berdasarkan gen leptin mengelompokkan sapi aceh dan sapi madura pada satu haplotipe yang sama dan sapi madura lainnya mempunyai haplotipe tersendiri. Keragaman genetik gen miostatin yang tertinggi ditemukan pada sapi madura sehingga menyebabkan pengelompokan sapi madura terpisah dari sapi aceh dan data pembanding dari GenBank. Sapi aceh mempunyai hubungan kekerabatan yang lebih dekat dengan B. taurus. Hal ini dapat diasumsikan bahwa karakter gen miostatin pada sapi aceh kemungkinan dipengaruhi oleh garis keturunan B. taurus. Pengaruh garis keturunan B. taurus
bahwa sapi madura dapat dikelompokan menjadi dua kelompok berdasarkan DNA mitokondria, yaitu kelompok B. indicus dan kelompok B. javanicus.
Potensi Keragaman Gen Leptin dan Gen Miostatin Sebagai Marka Genetik
Perubahan basa yang ditemukan pada gen leptin dan gen miostatin tidak menyebabkan terjadinya perubahan asam amino. Perubahan basa pada gen miostatin ditemukan pada ruas promotor. Nukleotida pada ruas promotor tidak ditranskripsikan ke dalam asam amino. Ruas promotor berperan sebagai tempat inisiasi enzim terhadap gen yang akan ditranskripsikan. Pada gen leptin perubahan basa C→T posisi -91 nt terjadi pada posisi sebelum kodon awal (ATG). Basa nukleotida yang diterjemahkan menjadi asam amino merupakan ruas penyandi yang diapit oleh kodon awal (ATG) dan kodon akhir (TAA, TAG, atau TGA) (Brown 2007). Perubahan basa pada daerah penyandi menentukan fenotipe yang dihasilkan. Liefers et al. (2002) mengungkapkan pada daerah penyandi genotipe AB menghasilkan produksi susu dan berat tubuh lebih tinggi dibandingkan dengan genotipe AA. Genotipe TT menghasilkan lemak karkas lebih tinggi dibandingkan dengan genotipe CC (Kononoff et al. 2005). Selain itu, Oprzadek et al. (2003) melaporkan bahwa genotipe AA merupakan genotipe yang menghasilkan berat karkas paling tinggi diantara genotipe lainnya. Bagian gen selain daerah penyandi meskipun tidak menyandikan asam amino tetapi juga menentukan fenotipe. Nobari et al. (2010) melaporkan bahwa perubahan basa pada daerah intron berpengaruh terhadap berat tubuh dan produksi susu. Titik mutasi pada gen miostatin meskipun tidak menyebabkan fenomena otot ganda tetapi juga berpengaruh terhadap deposisi lemak intramuskular. Genotipe mutan pada gen miostatin menghasilkan lemak intramuskular lebih tinggi dibandingkan dengan genotipe liar. Karkas dari genotipe heterozigot menghasilkan lemak intramuskular lebih sedikit daripada karkas genotipe homozigot (Allais et al.
2010).
Keragaman gen leptin tidak hanya berpotensi sebagai marka genetik untuk
Marker-Assisted Selection (MAS) tetapi gen leptin juga berperan sebagai marka genetik untuk mendeteksi material babi di dalam produk yang halal. Farouk et al.
SIMPULAN
Sapi Aceh berdasarkan analisis pada gen leptin dikelompokkan ke dalam haplotipe GCATC yang lebih dekat hubungan kekerabatannya dengan Bos taurus. Berdasarkan gen miostatin Sapi Aceh dikelompokkan dengan Bos taurus. Sapi Madura berdasarkan analisis gen leptin dikelompokkan ke dalam dua haplotipe yaitu haplotipe ATGCT yang lebih dekat hubungan kekerabatannya dengan sapi liar dan haplotipe GCATC yang lebih dekat hubungan kekerabatannya dengan
Bos taurus. Sapi Madura berdasarkan analisis gen miostatin berbeda kelompok dengan Bos taurus.
SARAN
DAFTAR PUSTAKA
Abdullah MAN et al. 2008. Keragaman fenotipik sapi Aceh di Nanggroe Aceh Darussalam. J Indon Trop Anim Agric 32: 1-11.
Agarwal R, Rout PK, Singh SK. 2009. Leptin a biomolecule for enhancing livestock productivity. J Biotech 8:169-176.
Allais S et al. 2010. The two mutations, Q204X and nt821, of the myostatin gene affect carcass and meat quality in young heterozygous bulls of French beef breeds. J Anim Sci 88:446-454.
Aslaminejad AS et al. 2010. Polymorphism in exon 3 of leptin gene in Iranian native cattle breeds. J Appl Anim Res 37: 159-162.
Bellinge RHS et al. 2005. Myostatin and its implications on animal breeding: a review. Anim Genet 36:1-6.
Berry C, Thomas M, Langley B, Sharma M, Kambadur R. 2002. Single cysteine to tyrosine transition inactivates the growth inhibitory function of Piedmontese myostatin. J Physiol Cell Physiol 283: 135-141.
Brown TA. 2007. Genomes 3. New York: Garland Science.
Buchanan et al. 2002. Association of a missense mutation in the bovine leptin gene with carcass fat content and leptin mRNA levels. Genet Sel Evol 34:105-116.
Byun SO, Fang Q, Zhou H, Hickford JGH. 2009. An effective method for silver-staining DNA in large numbers of polyacrilamid gels. Anal Biochem 385:174-175.
Charlier C et al. 1995. The mh gene causing double-muscling in cattle maps to bovine chromosome 2. Mammalian Genome 6:788-792.
Choundhary V et al. 2005. DNA polymorphism of leptin gene in Bos indicus and
Bos taurus cattle. Genet and Mol Biol 28:740-742.
Corva et al. 2009. Effect of leptin gene polymorphisms on growth, slaughter and meat quality traits of grazing Brangus steers. Gen and Mol Res 8: 105-116.
Dunner S et al. 2003. Haplotype diversity of the myostatin gene among beef cattle breeds. Genet Sel Evol 35:103-118.
Esmailizadeh AK et al. 2008. Effects of the myostatin F94L substitution on beef traits. J Anim Sci 86:1038-1046.
Farouk AE et al. 2006. The use of molecular technique for the detection of porcine ingredients in the Malaysian food market. Saudi Med J 27:1397-1400. Firdhausi NF. 2010. Asal usul sapi madura berdasarkan penanda molekular DNA
mitokondria [tesis]. Bogor: Program Pascasarjana, Institut Pertanian Bogor.
Frederich RC et al. 1995. Expression of ob mRNA and its Encoded Protein in rodents impact of nutrition and obesity. J Clin Invest 96:1658-1663.
Friedman JM. 2002. The function of leptin in nutrition, weight, and physiology.
Nutr Rev 60:1-15.
Grisolia AB et al. 2009. Myostatin (GDF8) single nucleotide polymorphisms in Nellore cattle. Genet Mol Res 8: 822-830.
Hirwa CDA et al. 2011. Genes related to economically important traits in beef cattle. Asian J Anim Sci 5:34-45.
Houseknecht KL et al. 2000. Growth hormone regulates leptin gene expression in bovine adipose tissue: correlation with adipose IGF-1 expression. J Endocrin
164: 51–57.
Javanmard A, Asadzadeh N, Sarhadi F. 2010. DNA polymorphism of bovine pituitary-specific transcription factor and leptin gene between Iranian Bos indicus and Bos taurus cattle. Am J Agric Biological Sci 5:282-285.
Kim S, Moussa NM. 2000. Symposium: adipocyte function, differentiation and metabolism. J Nutr 130: 3110-3115.
Kononoff PJ et al. 2005. The effect of a leptin single nucleotide polymorphism on quality grade, yield grade, and carcass weight of beef cattle. J Anim Sci
83:927-932.
Kusdiantoro M et al. 2009. On the origin of Indonesian cattle. Plosone 4:1-5.
Lagonigro R, Wiener P, Pilla F, Woolliams JA, Williams JL. 2003. A new mutation in the coding region of the bovine leptin gene associated with feed intake. Anim Genet 34: 371-374.
Liefers S. 2004. Physiology and genetics of leptin in periparturient dairy cow [disertasi]. Belanda: Animal Breeding and Genetics, Wageningen University.
Liefers SC, te Pas MFW, Veerkamp RF T. Lende van der. 2002. Associations between leptin gene polymorphisms and production, live weight, energy balance, feed intake, and fertility in holstein heifers. J Dairy Sci 85:1633-1638.
Loftus RT et al. 1994. Evidence for two independent domestications of cattle.
Proc Nadl Acad Sci 91:2757-2761.
McPherron AC, Lee SJ. 1997. Double muscling in cattle due to mutations in the myostatin gene. Proc Natl Acad Sci 94:12457–12461.
Mannen H, Tsuji S, Loftus RT, Bradly DG. 1998. Mitochondrial DNA variation and evolution of japaness black cattle (Bos taurus). Genetics 150:1169-1175.
Nassiry MR, Moussavi AH, Alashawkany AR, Ghovati S. 2007. Leptin gene polymorphism in Iranian native golpayegani and taleshi cows. Pakistan J Biological Sci 20:3738-3741.
Nei M. 1987. Molecular Evolutionary Genetics. New York: Columbia University Press.
Nijman IJ et al. 2003. Hybridization of banteng (Bos javanicus) and zebu (Bos indicus) revealed by mitochondrial DNA, satellite DNA, AFLP and microsatellites. Heredity 90:10-16.
Nobari K, Ghazanfari S, Nassiry MR, Tahmoorespur M, Jorjani E. 2010. Relationship between leptin gene polymorphism with economical traits in Iranian Sistani and Brown Swiss Cows. J Anim Vet Adv 9:2807-2810.
Oprzadek et al. 2003. Polymorphism at loci of leptin (LEP), Pit1, and STAT5A and their association with growth, feed convertion and carcass quality in black and white bulls. Anim Sci 21:135-145.
Payne WJA, Hodges J. 1997. Tropical Cattle: Origins, Breed, and Breeding Policies. Oxford: Blackwell Science Ltd.
Pomp D, Zou T, Clutter AC, Barendse W. 1997. Rapid communication mapping of leptin to bovine chromosome 4 by linkage analysis of a PCR-based polymorphism. J Anim Sci 75:1427.
Sari EM. 2011. Keragaman genetik gen hormon pertumbuhan (GH) dan hubungannya dengan kualitas karkas pada sapi Aceh [disertasi]. Bogor: Program Pascasarjana, Institut Pertanian Bogor.
Sharifzadeh A, Doosti A. 2010. Genetic polymorphism at the leptin gene in Iranian Holstein cattle by PCR-RFLP. Afr J Microbiol Res 4:1-3.
Taylor WE et al. 2001. Myostatin inhibits cell proliferation and protein synthesis in C2C12 muscle cells. J Physiol Endocrinol Metab 280: 221-228.
Taniguchi Y, Itoh T, Yamada T, Sasaki Y. 2002. Genomic structure and promoter analysis of the bovine leptin gene. IUBMBLife 53: 131-135.
Uggla CM. 2008. Investigating genetic variability within specific indigenous Indonesian cattle breeds [dissertation]. Swedish, Uppsala: Department of Animal Breeding and Genetics, University of Agricultural Sciences.
Wilmer P, Stone G, Johnston I. 2005. Environmental Physiology of Animal. USA: Blackwell Science Ltd.
Zhang F at al. 1997. Crystal structure of the obese protein leptin-E100. Nature
Lampiran 1 Hasil pengurutan basa nukleotida gen leptin daerah ekson 2 antara sapi aceh, sapi madura, dan data GenBank. Nomor di atas urutan basa menunjukkan posisi basa nukleotida dibaca secara vertikal