1
ASAL USUL SAPI MADURA BERDASARKAN PENANDA
DNA MITOKONDRIA
NIRMALA FITRIA FIRDHAUSI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
2
PERNYATAAN MENGENAI TESIS DAN SUMBER
INFORMASI
Dengan ini saya menyatakan bahwa tesis Asal Usul Sapi Madura Berdasarkan Penanda DNA Mitokondria adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir thesis ini.
Bogor, Agustus 2010
Nirmala Fitria Firdhausi
3
ABSTRACT
NIRMALA FITRIA FIRDHAUSI. The Origin of Madura Cattle Based on Mitochondrial DNA. Supervised by ACHMAD FARAJALLAH and RR. DYAH PERWITASARI
Madura cattle was one of native cattle from Indonesia. The origin of madura cattle had not known clearly. Many reports said that madura cattle came from the crosses of Bos javanicus (banteng), Bos taurus and Bos indicus. To understand the maternal inheritance in madura cattle, we analyzed the nucleotide sequence of Cyt b gen, tRNA Thr and Pro, also Dloop region from mitochondrial genom. The reconstruction of phylogenetic tree using Neighbour Joining Method based on Kimura 2 Parameter showed that madura cattle grouped into two types, that are B. javanicus type and B. indicus type. This research result supported many authors that there were a mixing of the maternal origin of madura cattle. Further investigation is needed to determine the origin madura cattle based on paternal liniages and mitochondrial diversity. This conservation investigation is needed to improve quality of madura cattle.
4
RINGKASAN
NIRMALA FITRIA FIRDHAUSI. Asal Usul Sapi Madura Berdasarkan Penanda DNA Mitokondria. Dibimbing oleh ACHMAD FARAJALLAH dan RR DYAH PERWITASARI.
Sapi madura adalah salah satu jenis sapi lokal Indonesia. Proses persilangan hingga diperoleh sapi madura selama ini tidak tercatat dengan baik dan masih terdapat perbedaan pada beberapa data hasil penelitian. Sapi madura diperkirakan berasal dari persilangan antara B. indicus dengan B. javanicus. Ada pula yang menyatakan bahwa sapi madura merupakan hasil persilangan antara pejantan B. indicus dengan betina campuran B. javanicus atau B. taurus.
Penelusuran sejarah persilangan dan kekerabatan sapi madura dapat dilakukan dengan menganalisis variasi genom mitokondria (mtDNA). Setiap individu yang memiliki indukan yang sama akan memiliki tipe mtDNA yang sama. Hal ini disebabkan karena mtDNA diturunkan melalui garis maternal.
Tujuan dari penelitian ini adalah untuk mengetahui kejelasan asal usul sapi madura berdasarkan garis keturunan maternal dengan menggunakan penanda mtDNA Cyt b, tRNA Thr, tRNA Pro, dan Dloop. Penelitian ini diharapkan dapat memberi informasi genetik dan asal-usul sapi madura, sehingga dapat digunakan untuk membantu peningkatan program pemuliaan dan konservasi sapi madura.
Sampel darah sapi madura yang digunakan berasal dari beberapa desa di Kabupaten Sampang dan Bangkalan. Sampel diekstraksi menggunakan metode yang dikembangkan oleh Sambrook et al. (1989) dengan sedikit modifikasi. Amplifikasi dilakukan dengan metode Polymerase Chain Reaction (PCR). Proses visualisasi yang dilakukan menggunakan tehnik elektroforesis gel poliakrilamid (PAGE 6%) dan dilanjutkan dengan pewarnaan sensitif perak menurut Tegelstrom (1986). Proses perunutan menggunakan metode Dideoxi Terminator dengan dNTP berlabel (big dye terminator).
Analisa data meliputi penghitungan komposisi nukleotida, laju subtitusi, jarak genetik dan rekonstruksi pohon filogeni. Penghitungan jarak genetik dilakukan berdasarkan model subtitusi Kimura 2 Parameter. Rekonstruksi pohon filogeni menggunakan metode Neighbour Joining (NJ) dengan boostrap 1000 kali.
Hasil runutan nukleotida yang didapatkan dari sampel sapi madura setelah disejajarkan, terbagi menjadi 203 bp gen Cyt b, 70 bp gen tRNA Thr, 66 bp tRNA Pro. Pada daerah Dloop terdapat hasil yang bervariasi yaitu 373 bp untuk sampel madura 14 dan 26, 374 bp untuk madura 38, 534 bp untuk sampel madura 41 dan 29, 512 bp untuk madura 32. Variasi panjang runutan pada Dloop disebabkan karena adanya ruas berulang. Ruas berulang hanya ditemukan pada sapi madura sampel 29, 32, dan 41.
5
bentuk kromosom Y diantara keduanya. Bos indicus memiliki bentuk kromosom akrosentris sedangkan B. javanicus memiliki bentuk kromosom metasentris. Perbedaan bentuk kromosom mengakibatkan gangguan pada proses spermatogenesis, sehingga terkadang F1 jantan yang dihasilkan keduanya bersifat steril.
Persilangan antara banteng dan B. indicus diperkirakan terjadi sejak masuknya kebudayaan hindu yang dibawa oleh bangsa india ke Indonesia. Awal masuknya bangsa india ke Indonesia terjadi sekitar 1800 tahun yang lalu dengan membawa sapi-sapi dari jenis B. indicus. Sapi-sapi jenis B. indicus ini kemudian disilangkan dengan banteng yang masih banyak dijumpai sebelum penggundulan hutan sekitar 150 tahun yang lalu di Pulau Madura.
6
© Hak Cipta milik IPB, tahun 2010
Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjuan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
7
ASAL USUL SAPI MADURA BERDASARKAN PENANDA
DNA MITOKONDRIA
NIRMALA FITRIA FIRDHAUSI
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Bio Sains Hewan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
8
Judul : Asal usul Sapi Madura Berdasarkan Penanda DNA Mitokondria Nama : Nirmala Fitria Firdhausi
NRP : G352080111
Tanggal Ujian: 9 Agustus 2010 Tanggal Lulus: Disetujui Oleh
Komisi Pembimbing
Dr. Ir. Achmad Farajallah, M.Si. Anggota
Ketua Program Studi Biosains Hewan
Dr. Bambang Suryobroto
Dekan Sekolah Pascasarjana
Prof. Dr. Ir. Khairil A. Notodiputro, M.S. Dr. Ir. RR. Dyah Perwitasari, M.Sc.
9
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Agustus 2009 ini ialah mengenai Asal Usul Sapi Madura Berdasarkan Penanda DNA Mitokondria.
Terimakasih penulis ucapkan kepada Bapak Dr. Ir. Achmad Farajallah, M.Si dan Ibu Dr. Ir. Dyah Perwitasari, M.Sc selaku pembimbing yang telah banyak memberikan saran. Selain itu, penulis mengucapkan terikasih kepada Loka Penelitian Sapi Potong Grati Pasuruan yang telah memberikan banyak informasi dan bantuan. Ungkapan terimakasih tak lupa penulis ucapkan kepada ayahanda, ibunda, suami, dan kakak atas segala doa, kasih sayang dan dukungannya. Penulis juga mengucapkan terimakasih kepada seluruh pihak yang telah membantu hingga terselesaikannya karya ilmiah ini.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2010
10
RIWAYAT HIDUP
Penulis dilahirkan di Jember pada tanggal 25 Juni 1985 dari ayah Suwodjo dan ibu Maryamin Nisak. Penulis merupakan putri bungsu dari tiga bersaudara. Penulis menikah dengan saudara Bahtiar Yusuf Habibi pada tahun 2010.
Tahun 2003 penulis lulus dari SMA Negeri 1 Lumajang dan pada tahun yang sama lulus seleksi masuk Universitas Jember melalui jalur SPMB. Penulis memilih jurusan Biologi Fakultas Matematika dan Ilmu Pengetahuan Alam. Selama mengikuti perkuliahan, penulis menjadi asisten untuk mata kuliah Biologi Dasar pada semester ganjil tahun ajaran 2006/2007 dan 2007/2008, serta mata kuliah Ekologi pada tahun 2007/2008. Pada tahun 2005 penulis menjadi pemakalah pada Seminar Hasil Karya Inovatif dan Penelitian Mahasiswa Kombi Jurusan Biologi. Penulis lulus dari program sarjana pada tahun 2008.
DAFTAR ISI
Mutasi Nukleotida dan Analisis Filogeni Berdasarkan Ruas Dloop.... 16
DAFTAR TABEL
Halaman 1 Komposisi basa (%) untuk mesing-masing sampel sapi madura ... 13 2 Jumlah basa sama dan basa berbeda pada keempat ruas mtDNA ... 14 3 Nilai jarak genetik (di bawah diagonal) dan nilai perbandingan kejadian
transisi dengan tranversi (di atas diagonal) berdasarkan ruas Cyt b ... 15 4 Nilai jarak genetik (di bawah diagonal) dan nilai perbandingan kejadian
DAFTAR GAMBAR
Halaman 1 Penampilan sapi bali dan banteng di Taman Nasional Alas Purwo ... 6
2 Morfologi sapi madura ... 7 3 Struktur mtDNA pada mamalia Ursus thibetanus (beruang hitam) ... 9 4 Pola pita DNA mitokondria hasil amplifikasi dalam PAGE 6% dan
produk hasil perunutan ... 14 5 Hasil rekonstruksi pohon filogeni berdasarkan runutan basa dan asam
amino dari gen Cyt b menggunakan metode NJ dengan boostrap 1000x .. 15 6 Sebagian ruas berulang sepanjang 22 nt pada Dloop. ... 17 7 Letak ruas berulang (tandem repeat) dan TAS (terminated associated
sequence) pada wilayah Dloop mitokondria sapi madura ... 17 8 Hasil rekonstruksi pohon filogeni berdasarkan ruas Dloop dengan
DAFTAR LAMPIRAN
Halaman 1 Peta Lokasi Pengambilan Sampel ... 33
2 Hasil penjajaran DNA ruas Cyt b,tRNA Thr, tRNA Pro dan Dloop
1
PENDAHULUAN
Latar Belakang
Ternak sapi merupakan hewan hasil domestikasi yang memiliki peranan penting bagi kehidupan manusia. Domestikasi sapi diperkirakan dimulai sejak 4000 hingga 5000 tahun yang lalu dari Bos prigimineus (MacHugh1997; Mannen
et al. 1998). Sapi hasil domestikasi B. prigimineus ada dua jenis, yaitu Bos taurus
dan Bos indicus. Kedua keturunan jenis sapi tersebut berkembang hingga saat ini. Jenis sapi lain yang berkembang sebagai hewan ternak adalah Bos javanicus.Bos javanicus merupakan salah satu jenis sapi asli Indonesia yang berbeda dengan B. taurus maupun B. indicus (Nijman et al. 2003; Uggla 2008).
Bos javanicus sebagai sapi asli Indonesia didomestikasi menjadi sapi bali yang kemudian tersebar di seluruh wilayah Indonesia. Selain sapi bali, beberapa sapi lokal yang berkembang di Indonesia antara lain sapi aceh, sapi pesisir, dan sapi madura. Sapi aceh dan sapi pesisir berasal dari keturunan B. indicus yang tersebar di wilayah Provinsi Aceh dan Sumatera bagian barat. Sapi madura memiliki persebaran yang terbatas di sekitar Pulau Madura dan Pulau Jawa bagian timur. Asal domestikasi sapi madura hingga saat ini belum jelas (Williamson & Payne 1965; Uggla 2008; Kusdiantoro 2009).
Sapi madura memiliki ciri-ciri morfologi yang hampir mirip dengan sapi bali. Kulit sapi madura berwarna coklat kemerahan dengan sedikit motif warna putih pada bagian pantat dan kaki. Beberapa karakter sapi madura memiliki kemiripan dengan B. indicus, antara lain lebih tahan terhadap kondisi cuaca panas, tahan terhadap kondisi makanan yang terbatas, memiliki kualitas daging yang baik, dan lebih resisten terhadap parasit (Payne & Hodges 1997).
Proses persilangan sapi hingga diperoleh sapi madura selama ini tidak tercatat dengan baik dan masih terdapat perbedaan pada beberapa data hasil penelitian. Sapi madura diperkirakan berasal dari persilangan antara B. indicus
dengan B. javanicus (Wiliamson & Payne 1965). Ada pula yang menyatakan bahwa sapi madura merupakan hasil persilangan antara pejantan B. indicus
2
Penelitian kusdiantoro (2009) berdasarkan SRY menemukan beberapa sampel sapi madura merupakan keturunan dari B. taurus.
Namikawa (1981) menduga bahwa terdapat percampuran pada sapi madura berdasarkan adanya hemoglobin beta x (Hb-β X). Munculnya Hb-β X pada darah sapi madura diperkirakan berasal dari B. javanicus. Hemoglobin β X tidak pernah dilaporkan muncul pada B. indicus maupun B. taurus.
Penelusuran sejarah persilangan dan kekerabatan sapi madura dapat dilakukan dengan menganalisis variasi genom mitokondria (mtDNA). Setiap individu yang memiliki indukan yang sama akan memiliki tipe mtDNA yang sama. Hal ini disebabkan mtDNA diturunkan melalui garis maternal. Kelebihan lain dari mtDNA adalah bersifat haploid (single copy) dan tidak mengalami rekombinasi (Tapio & Grigaliunaite 2002).
Tujuan
Tujuan penelitian ini adalah untuk mengetahui kejelasan asal usul sapi madura berdasarkan garis keturunan maternal dengan menggunakan penanda mtDNA Cyt b, tRNAThr, tRNA Pro, dan Dloop.
Manfaat
3
TINJAUAN PUSTAKA
Sejarah Domestikasi dan Persebaran Sapi
Sapi domestik termasuk dalam ordo Artiodactyla (Ungulata) dan merupakan salah satu famili dari Bovidae. Sapi domestik merupakan hewan yang paling berkembang dan dominan dalam familinya. Kerabat mereka yang paling dekat antara lain sapi liar (banteng, gaur dan kouprey), yak, bison Amerika dan Eropa, dan Kerbau (Fries & Ruvinsky 1999; Matthee & Davis 2001). Proses domestikasi berlangsung sejalan dengan mulai berkembangnya kebudayaan manusia. Sapi didomestikasi untuk membantu kehidupan manusia baik digunakan sebagai bahan pangan ataupun diambil tenaganya (Loftus et al. 1993).
Sapi domestik yang berkembang saat ini merupakan hasil domestikasi dari
B. prigimineus yang diperkirakan mulai didomestikasi sejak 5000 hingga 4000 tahun yang lalu (Mannen et al. 1998). Terdapat tiga sub spesies dari B. prigimineus yang diperkirakan merupakan asal dari sapi domestik yaitu B. prigimineus subsp prigimineus yang wilayah persebarannya di Eropa, B. prigimineus subsp namadicus yang persebarannya di Asia dan B. prigimineus
subsp ophistonomous yang memiliki persebaran di wilayah Afrika bagian utara (MacHugh et al. 1997). Bos prigimineus diperkirakan telah punah sejak 2000 tahun yang lalu (Bollongino 1997).
Sapi domestik hasil domestikasi B. prigimineus terbagi menjadi dua yaitu sapi tanpa punuk (humpless) dan sapi yang berpunuk (humped). Sapi yang tidak berpunuk tergolong dalam B. taurus. Sapi yang berpunuk disebut juga dengan zebu (B. indicus). Fungsi punuk pada B. indicus belum diketahui secara pasti. Dilihat dari komposisi penyusunnya yaitu otot dan lemak, diperkirakan punuk ini berfungsi sebagai penyimpan air (McHugh 1997).
4
Bos indicus sebagian besar tersebar di wilayah Asia bagian selatan dan Afrika. Awal masuknya B. indicus ke wilayah Asia diperkirakan dibawa oleh pengembara Verdic Aryan dari daerah Irak ke India sekitar 2200 SM hingga 1500 SM melalui jalur utara. Persebaran B. indicus kemudian berlanjut ke arah selatan menuju Srilangka dan ke arah timur menuju ke Birma, Thailand, Vietnam, Laos, Kamboja dan Malaysia (Wiliamson & Payne 1965). B. indicus diperkirakan masuk ke Indonesia bersama dengan masuknya kebudayaan Hindu (Payne & Wilson 1999)
Persebaran B. taurus sebagian besar di wilayah Eropa dan Afrika Barat. Awal masuknya B. taurus ke wilayah Asia dibawa oleh bangsa nomaden menuju ke Asia Tengah kemudian ke arah selatan dan tenggara menuju ke India. Bos taurus dibawa menuju ke arah timur yaitu Cina oleh pengembara Ural Altaic sekitar 2500 SM. Persebaran B. taurus dilanjutkan menuju ke Birma, Thailand, Vietnam, Kamboja, dan Laos. Sapi-sapi tersebut kemudian diimpor ke wilayah selatan yaitu Malaysia dan Indonesia (Wiliamson & Payne 1965; MacHugh 1997).
Persilangan (Hibridisasi) pada Sapi
Proses persilangan pada sapi dapat terjadi secara langsung (spontan) ataupun diatur oleh manusia. Proses persilangan pada sapi dilakukan untuk mendapatkan tipe-tipe baru yang lebih baik sesuai dengan kebutuhan manusia. Proses persilangan dilakukan dengan jalan mengawinkan beberapa tipe sapi yang tidak memiliki hubungan keluarga. Berbagai macam faktor dapat mempengaruhi kesuksesan dari proses persilangan diantaranya adalah kompatibilitas kromosom (Cassell 2007).
Proses persilangan umumnya membutuhkan jumlah kromosom yang sama.
5
Persilangan antara B. taurus dan B. indicus banyak dilakukan untuk mendapatkan tipe sapi baru yang unggul. Beberapa contoh hasil persilangan antara B. indicus dan B. taurus adalah sapi batangas, burmese, brangus, dan brahmousin. Persilangan antara B. javanicus dengan B. indicus sedikit mengalami hambatan dan terkadang menghasilkan F1 jantan yang steril. Hal ini diperkirakan karena adanya perbedaan bentuk kromosom Y. Perbedaan bentuk kromosom mengakibatkan gangguan pada proses spermatogenesis (Rollinson 1984; Lightner 2008).
Kromosom X pada anggota Bos dan Bison memiliki bentuk yang sama yaitu submetasentris. Hal ini yang menyebabkan betina hasil persilangan diantara Bos
maupaun antara Bos dengan Bison menghasilkan F1 betina yang fertil (Lightner 2008).
Tipe-tipe Sapi di Indonesia
Sapi domestik di Indonesia sebagian besar berasal dari B. indicus. Tidak ada data khusus yang menunjukkan awal masuknya B. indicus ke Indonesia. Bos indicus diperkirakan masuk bersamaan dengan masuknya kebudayaan Hindu sekitar 1500 tahun yang lalu (Payne & Wilson 1999)
Sapi lokal indonesia yang termasuk dalam keturunan B. indicus antara lain sapi aceh dan sapi pesisir (Uggla 2008; Kusdiantoro et al. 2009). Selain itu terdapat pula sapi bali dan sapi madura yang merupakan keturunan B. javanicus. Sapi-sapi lokal tersebut banyak berfungsi sebagai ternak potong dan ternak pekerja (Rouse 1972; Nijman 2003).
Sapi bali merupakan sapi domestik yang memiliki sejarah domestikasi yang berbeda dengan sapi-sapi yang lain. Sapi bali didomestikasi langsung dari banteng liar (B. javanicus). Domestikasi banteng telah dimulai sejak 3500 tahun yang lalu (Nijman et al. 2003; Kusdiantoro et al. 2009). Sapi bali dipertahankan keasliannya di Pulau Bali. Persebaran sapi bali sangat luas dibandingkan dengan jenis sapi lokal lainnya. Sapi bali tersebar diseluruh wilayah Indonesia, terutama Jawa, Sulawesi, Lombok, dan Pulau Timor (Vietmeyer 1983).
6
hingga ke lutut terdapat area berwarna putih. Pada telinga dan moncongnya tumbuh rambut berwarna putih (Rollinson 1984; Payne & Hodges 1997).
Gambar 1 Penampilan sapi bali (atas) (Handiwirawan et al. 2003) dan B. javanicus di Taman Nasional Alas Purwo, Jawa Timur(bawah).
Sapi pesisir merupakan keturunan dari B. indicus yang tersebar di wilayah Sumatera bagian selatan. Sapi pesisir memiliki ciri-ciri morfologi bertubuh kecil, warna badan coklat tua kehitaman, memiliki punuk, tanduk, dan gelambir. Sapi pesisir sudah menempati wilayah Kabupaten Painan (Pesisir Selatan) Padang sebelum tahun 1810 dan sudah beradaptasi dengan lingkungan lahan semak dan pesisir. Sapi lokal keturunan B. indicus lain yang terdapat di wilayah Sumatera adalah sapi aceh. Sapi aceh memiliki persebaran terbatas hanya di wilayah Provinsi Aceh dan sekitarnya (Aryogi et al. 2007).
7
warna putih ini tidak nampak terlalu jelas dibandingkan dengan sapi bali. Pada jantan tubuh bagian depan lebih teguh daripada tubuh bagian belakang. Memiliki berat sekitar 350 kg dengan tinggi sekitar 118 cm (Rouse 1972).
Sapi madura memiliki persebaran terbatas di Pulau Madura, beberapa pulau kecil disekitar Pulau Madura, dan Jawa Timur. Pulau Madura merupakan salah satu pulau kecil di sebelah utara Jawa Timur yang memiliki luas sekitar 4497 km2. Di Pulau Madura tidak terdapat padang rumput ataupun makanan ternak yang mencukupi, akan tetapi di wilayah tersebut terdapat kurang lebih 600000-700000 ekor sapi madura. Sapi-sapi madura tersebut beradaptasi dengan kondisi lingkungan yang sangat minim makanan dan kondisi lingkungan yang panas dan kering. Mereka mampu bertahan hidup walaupun peternak-peternak hanya memberikan makan daun-daun kering. Kondisi ini menyebabkan sapi madura hanya menghasilkan sedikit susu untuk anak sapi mereka dan berkembang sangat lambat (Maksum 1993; Payne & Hodges 1997).
8
Mutu sapi lokal yang ada di Indonesia masih dibawah standar. Keberadaan sapi-sapi lokal dinilai kurang memenuhi kebutuhan daging ataupun susu. Hal ini menyebabkan pemerintah mengimpor sapi-sapi jenis lain dari luar wilayah Indonesia. Sapi domestik impor yang dikembangkan di Indonesia berasal dari keturunan B. indicus, B. taurus atau persilangan keduanya. Beberapa jenis sapi impor tersebut adalah sapi ongole, peranakan ongole, brahman, simental, brangus, limousin, santa getrudis, red danis dan lain-lain (Aryogi et al. 2007).
DNA Mitokondria
Analisis genetika molekular telah diaplikasikan dalam penelitian hewan domestikasi sejak mulai digunakannya penanda Allozyme pada tahun 1960 sampai 1970. Penelitian dengan menggunakan runutan DNA dapat digunakan untuk membantu memahami proses kejadian dalam domestikasi. Hal yang dapat diamati dengan menggunakan metode analisis DNA antara lain proses evolusi, keragaman genetik dan persebaran hasil domestikasi (Astuti 2004). Berbagai macam penanda bisa digunakan untuk memahami proses tersebut, antara lain DNA mitokondria, DNA inti, dan juga kromosom Y (Bradley 1996).
Analisis genetik secara molekuler dapat dilakukan dengan lebih cepat dan akurat dengan berkembangnya tekhnologi DNA. Hal ini disebabkan karena DNA sebagai unit keturunan terkecil mempunyai sekuen yang spesifik untuk setiap spesies pada satu atau beberapa lokasi didalam kromosom. Sekuen DNA tertentu hanya dapat ditemukan pada spesies tertentu. DNA pada organisme tingkat tinggi terdapat didalam inti sel dan mitokondria (Tapio & Grigaliunaite 2003).
9
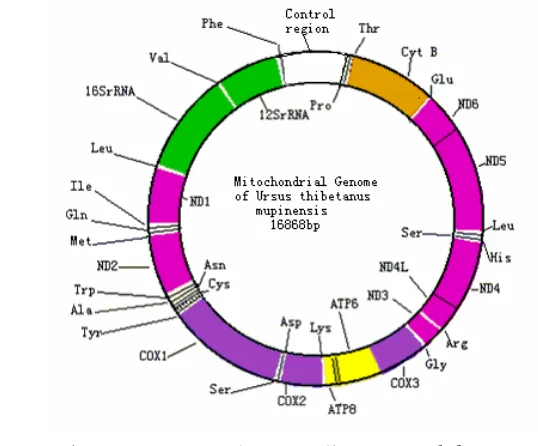
Gambar 3 Struktur mtDNA pada mamalia Ursus thibetanus (beruang hitam) (Hou et al. 2006)
Bagian-bagian dari genom mitokondria memiliki laju evolusi yang berbeda-beda, sehingga menjadi faktor penting yang menentukan penggunaan penanda DNA dalam studi sistematika dan biogeografi. Gen yang berevolusi lambat dapat dijadikan dasar penelusuran evolusi atau filogeni antar spesies. Sedangkan gen yang evolusi cepat digunakan untuk perbandingan galur-galur baru (Roderick 1996).
10
BAHAN DAN METODE
Waktu dan Tempat Penelitian
Penelitian dilaksanakan pada bulan Agustus hingga Desember 2009 bertempat di Laboratorium Bagian Fungsi Hayati dan Perilaku Hewan Departemen Biologi Institut Pertanian Bogor, Bogor.
Koleksi Darah
Sampel darah sapi madura yang digunakan adalah sampel darah yang diawetkan dalam alkohol absolut koleksi Laboratorium Fungsi Hayati dan Perilaku Hewan Departemen Biologi FMIPA IPB. Sampel darah yang digunakan berasal dari desa Polagan (sampel no.14), Tadan Tengah (sampel no. 13), desa Golbung (sampel no. 26, 29 ,30, dan 32), Paterongan (sampel no. 3 dan 7) dan desa Komis (sampel no. 38, 40, dan 41) yang berada di wilayah Kabupaten Sampang, desa Anjun (sampel no 43 dan 49), Serabi Timur (sampel no. 51 dan 52) dan Patereman (sampel no. 54) yang berada di Kabupaten Bangkalan, Pulau Madura (Lampiran 1).
Isolasi DNA Total
Sampel diekstraksi secara manual dengan menggunakan metode Sambrook
et al. (1989) dengan sedikit modifikasi. Sampel darah diambil 300 µ l dimasukkan kedalam tube 1.5 ml. Sel-sel darah dibersihkan dari alkohol dengan menambahkan
11
rpm selama 10 menit. Bagian atas sampel yang berwarna bening diambil dan dipindahkan dalam tube 1.5 ml. Molekul-molekul DNA yang mengendap dicuci menggunakan alkohol 70% sebanyak 800 µ l kemudian disentrifugasi 10000 selama 10 menit. Proses pencucian menggunakan alkohol dilakukan sebanyak dua kali. Alkohol yang tersisa diuapkan dalam wadah vakum. Molekul-molekul DNA disuspensikan dalam bufer TE (Tris-HCl 10-1 M EDTA 10-2 M pH 8.0) 60µl dan disimpan dalam freezer hingga dikerjakan lebih lanjut.
Amplifikasi mtDNA
Amplifikasi genom mitokondria menggunakan pasangan primer koleksi Dr Ir Achmad Farajallah, M.Si, yaitu AF22 (forward) 5’GCGTACGCAAT CTTACGATCA-3’ dan AF23 (reverse) 5’ ATGCAGTTAAGTCCAGCTAC-3’. Primer AF22 menempel pada basa ke 14979 dan primer AF23 menempel pada basa ke 16098 B. indicus (no akses AF492350). Ukuran mtDNA yang diharapkan dari proses amplifikasi ini adalah sebesar 1120 bp. Pasangan primer AF22 dan AF23 mengapit ruas tengah hingga akhir Cyt b, tRNA Pro, tRNA Thr dan juga bagian awal hingga tengah Dloop. Proses amplifikasi dilakukan secara in vitro
menggunakan tehnik PCR (Polymerase Chain Reaction).
Komposisi 25µ l reaksi PCR adalah sampel DNA 2 µ l (10-100 ng), RBC Bioscience taq polymerase 1,25 unit beserta sistem bufernya, dNTP 10 nmol sebanyak 1 µL, MgCl2 2µ l, primer AF22 dan AF23 masing-masing sebanyak 1µ l, dan DW steril. Seluruh bahan dicampur menjadi satu ke dalam tube PCR kemudian disentrifuse 3000 rpm selama 30 detik. Bahan yang telah disentrifuse dimasukkan kedalam mesin Thermal Cycler TAKARA MP4 untuk dilanjutkan proses amplifikasi.
Kondisi PCR yang digunakan untuk proses amplifikasi mtDNA adalah tahap denturasi awal pada suhu 940C selama 3 menit, tahap denaturasi pada suhu 940C selama 45 detik, tahap penempelan primer (annealing) pada suhu 580C selama 30 detik, dan tahap polimerasi (extension) pada suhu 720C selama 1 menit yang diulang selama 30 siklus. Reaksi PCR diakhiri dengan polimerasi (final extension) pada suhu 720C
Visualisasi produk PCR dilakukan menggunakan tehnik elektroferesis gel poliakrilamid (PAGE) 6% dalam bufer 1xTBE (Tris-HCl 10 mM, asam borat 1 M,
12
EDTA 0.1 mM). Elektroforesis dijalankan pada kondisi 200 mV selama 50 menit. Proses dilanjutkan dengan pewarnaan sensitif perak (Tegelstrom 1986) dengan sedikit modifikasi.
Perunutan DNA
Produk amplifikasi yang menunjukkan pita tunggal kemudian dimurnikan dan dijadikan cetakan dalam reaksi PCR untuk perunutan nukleotida. Primer yang digunakan dalam proses PCR sama dengan primer yang digunakan untuk amplifikasi. Reaksi PCR dilakukan dengan menggunakan metode Dideoxi Terminator dengan dNTP berlabel (big dye terminator). Perunutan nukleotida menggunakan mesin ABI Prism 3700-Avant Genetic Analyzer.
Analisis Data
Runutan nukleotida yang diperoleh kemudian diedit dengan menggunakan program bioedit versi 7.0.5. Urutan DNA yang telah diedit disejajarkan menggunakan Clustal W Vs 1.8 yang masuk dalam program MEGA 4.0 (Tamura
et al. 2007) dengan beberapa urutan DNA dari kelompok Bovidae yang
dipublikasikan dalam GenBank
sebagai pembanding yaitu B. javanicus 1 no. akses FJ556566, B. javanicus 2 no. akses EU878389, B. javanicus 3 no. akses EF693809, B. taurus 1 no. akses EU177815, B. taurus 2 (Friesian Holstein) no. akses DQ124416, B. taurus 3 (Beef Cattle) no. akses DQ124402, B. indicus no. akses AF492350, dan Bubalus bubalus no. akses AY702618.
13
HASIL
Produk Amplifikasi dan Perunutan
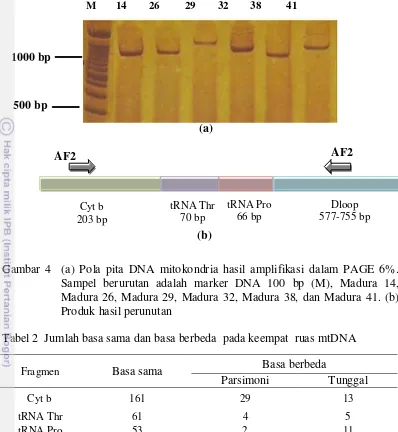
Pada sampel yang teramplifikasi dihasilkan pita tunggal berukuran sekitar 1400-1500 bp. Sampel yang berhasil dirunutkan adalah beberapa sampel dari Kabupaten Sampang (Gambar 4a). Hasil runutan nukleotida sampel sapi madura terbagi menjadi 203 bp gen Cyt b, 70 bp gen tRNA Thr, 66 bp tRNA Pro. Pada daerah Dloop terdapat hasil yang bervariasi yaitu 577 bp untuk sampel madura 14 dan 26, 578 bp untuk madura 38, 755 bp untuk sampel madura 41 dan 29, 624 bp untuk sampel 32 (Gambar 4b).
Pada proses penjajaran dengan spesies pembanding, ruas Dloop yang digunakan adalah sepanjang 373 bp untuk sampel sapi madura 14 dan 26, 374 bp untuk madura 38, 534 bp untuk sampel 41 dan 29, 405 bp untuk sampel 32. Hal ini disebabkan karena spesies pembanding yang digunakan yaitu B. javanicus
(AF693809) hanya memiliki panjang 552 bp.
Rata-rata komposisi basa pada sampel sapi madura terdiri atas 26.7% basa T, 23.8% basa C, 36.3% basa A dan 13.1% basa G. Komposisi basa terbanyak pada sampel sapi madura adalah A dan T yaitu sebesar 63.05% sedangkan G dan C sebesar 36.95% (Tabel 1).
Runutan basa dari keempat ruas terbagi menjadi basa yang sama (conserve) dan basa berbeda (variable). Basa yang berbeda terdiri dari dua jenis yaitu basa parsimoni dan basa tunggal (singleton) (Tabel 2). Basa yang bersifat parsimoni selanjutnya digunakan dalam analisis filogeni.
Tabel 1 Komposisi basa (%) untuk masing-masing sampel sapi madura
Sampel Sapi % Basa
T C A G
Madura 14 25.9 25.2 35.3 13.6
Madura 26 25.9 25.3 35.2 13.6
Madura 29 27.7 22.3 37.4 12.6
Madura 32 27.5 22.4 37.3 12.8
Madura 38 25.9 25.2 35.3 13.6
14
500 bp 1000 bp
M 14 26 29 32 38 41
Gambar 4 (a) Pola pita DNA mitokondria hasil amplifikasi dalam PAGE 6%. Sampel berurutan adalah marker DNA 100 bp (M), Madura 14, Madura 26, Madura 29, Madura 32, Madura 38, dan Madura 41. (b) Produk hasil perunutan
Tabel 2 Jumlah basa sama dan basa berbeda pada keempat ruas mtDNA
Mutasi Nukleotida dan Analisis Filogeni Berdasarkan Ruas Cyt B
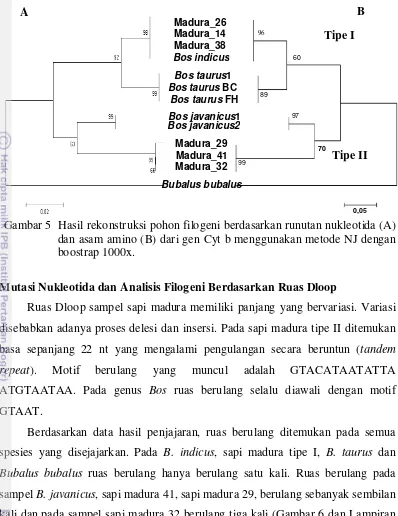
Pohon filogeni dengan menggunakan urutan basa maupun asam amino Cyt b menunjukkan topologi yang sama. Sapi madura terbagi menjadi dua kelompok. Sapi madura tipe I berkelompok dalam satu cabang dengan B. indicus dengan nilai boostrap 98% untuk pohon filogeni berdasarkan asam amino (Gambar 5a) dan 96% untuk pohon filogeni berdasarkan urutan basa (Gambar 5b). Sapi madura tipe I dan B. indicus memiliki urutan nukleotida yang sama pada ruas Cyt b. Sapi madura tipe II berkelompok dengan B. javanicus dengan nilai boostrap 63% untuk pohon filogeni berdasarkan asam amino (Gambar 5a) dan 70% untuk pohon
Fragmen Basa sama Basa berbeda
15
filogeni berdasarkan urutan nukleotida (Gambar 5b). Pengelompokan antara sapi madura tipe II dengan B. javanicus didukung dengan nilai jarak genetik sebesar 0.062 berdasarkan model Kimura 2 Parameter (Tabel 3).
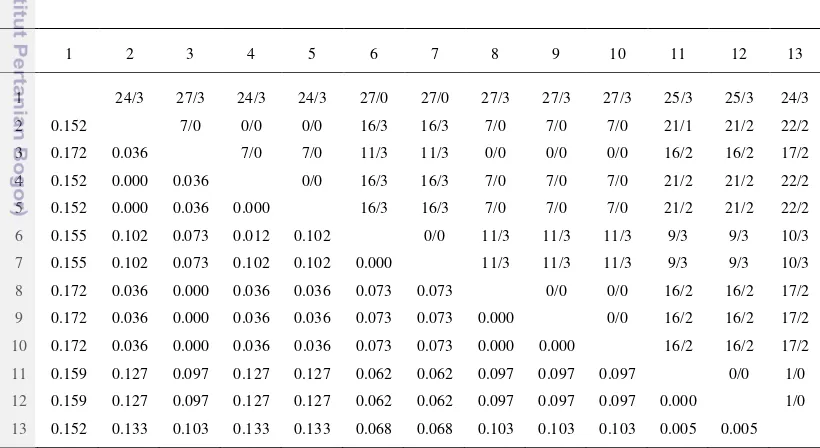
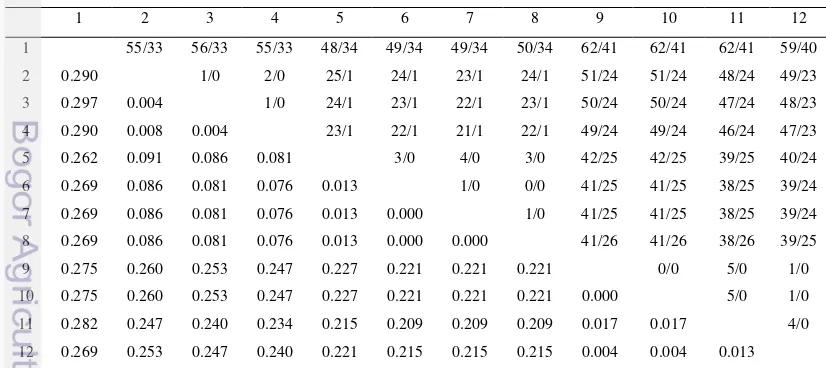
Kecilnya nilai jarak genetik menunjukkan tingkat kekerabatan yang dekat antara sapi madura tipe I dengan B. indicus dan sapi madura tipe II dengan B. javanicus. Kedua kelompok sapi madura ini dibedakan oleh 18 titik mutasi. Jarak genetik terbesar ditemukan antara Bubalus bubalus dengan B. indicus dan sapi madura tipe I yaitu 0.172. Jarak terbesar diantara anggota Bos ditemukan antara B. taurus 1, B. taurus BC, dan B. taurus FH dengan sapi madura 29 sebesar 0.133 berdasarkan model Kimura 2 Parameter (Tabel 3).
Tabel 3 Nilai jarak genetik (di bawah diagonal) dan nilai perbandingan kejadian transisi dengan tranversi (di atas diagonal) berdasarkan ruas Cyt b dengan menggunakan metode Kimura 2 Parameter (K2P).
16 Gambar 5 Hasil rekonstruksi pohon filogeni berdasarkan runutan nukleotida (A)
dan asam amino (B) dari gen Cyt b menggunakan metode NJ dengan boostrap 1000x.
Mutasi Nukleotida dan Analisis Filogeni Berdasarkan Ruas Dloop
Ruas Dloop sampel sapi madura memiliki panjang yang bervariasi. Variasi disebabkan adanya proses delesi dan insersi. Pada sapi madura tipe II ditemukan basa sepanjang 22 nt yang mengalami pengulangan secara beruntun (tandem
repeat). Motif berulang yang muncul adalah GTACATAATATTA
ATGTAATAA. Pada genus Bos ruas berulang selalu diawali dengan motif GTAAT.
Berdasarkan data hasil penjajaran, ruas berulang ditemukan pada semua spesies yang disejajarkan. Pada B. indicus, sapi madura tipe I, B. taurus dan
Bubalus bubalus ruas berulang hanya berulang satu kali. Ruas berulang pada sampel B. javanicus, sapi madura 41, sapi madura 29, berulang sebanyak sembilan kali dan pada sampel sapi madura 32 berulang tiga kali (Gambar 6 dan Lampiran 2).
Pada Bubalus bubalus terdapat mutasi pada motif GTACATAATATTA ATGAATAA dimana terjadi perubahan basa T menjadi basa G pada basa pertama (basa no. 15543 pada Gambar 6). Mutasi juga muncul pada basa ke sembilan pada motif ruas berulang dimana pada B. indicus dan B. taurus terjadi perubahan basa T menjadi C (basa no. 15549 pada Gambar 6).
A B
Tipe I
17
1111111111111111111111111111111111111111111111111111111111111111111 5555555555555555555555555555555555555555555555555555555555555555555 5555555555555555555555555555555555555555555555555555555555555555555 1122222222223333333333444444444455555555556666666666777777777788888 8901234567890123456789012345678901234567890123456789012345678901234
B.javanicus3 GATAA---GTACATAATATTAATGTAATAAGTACATAATATTAATGTAATAA Madura 29 GATAA---GTACATAATATTAATGTAATAAGTACATAATATTAATGTAATAA Madura 41 GATAA---GTACATAATATTAATGTAATAAGTACATAATATTAATGTAATAA Madura 32 GATAA---GTACATAATATTAATGTAATAAGTACATAATATTAATGTAATAA B. Taurus -CCTA---CGCAAGGGGTAATGTACATAACATTAATGTAATAA--- B. indicus -CCCA---GGCAAGAGGTAATGTACATAACATTAATGTAATAA--- Madura 14 -CCCA---GGCAAGAGGTAATGTACATAACATTAATGTAATAA--- Madura 26 -CCCA---GGCAAGAGGTAATGTACATAACATTAATGTAATAA--- Madura 38 CCCCA---GGCAAGAGGTAATGTACATAACATTAATGTAATAA--- Bub bubalus CCCTACTACTCCGAATGGGGGGGGGGACATAACATTAATGTAATAA---
Gambar 6 Bagian yang berwarna abu-abu merupakan sebagian ruas berulang sepanjang 22 nt. Keterangan: tiga baris pertama menunjukkan nomor posisi nukleotida dibaca secara vertikal, referensi dari B. indicus
(AF492350).
Ruas berulang pada sapi madura tipe II terletak di dekat tRNA Pro tepatnya pada basa ke-190 sebelum ruas Terminated Associated Sequence (TAS) (Gambar 7). Ruas TAS pada sapi madura tipe II terdeteksi dengan urutan TACATTACATTAC pada sampel sapi madura II dan B. javanicus, TACATTAAATTAT pada sapi madura tipe I, B. indicus dan B. taurus, dan TACATTATATTAT pada Bubalus bubalus. Primer AF 23 yang digunakan hanya dapat mengamplifikasi ruas awal sampai tengah Dloop sehingga ruas Conserve Sequence Block (CSB) sebagai pengatur replikasi untuk utas berat (heavy strand) tidak dapat terdeteksi.
Gambar 7 Letak ruas berulang (tandem repeat) dan TAS (terminated associated sequence) pada wilayah Dloop mitokondria sapi madura. Gambar berdasarkan Matson & Baker (2001).
18
Pada ruas Dloop nilai jarak genetik terkecil berdasarkan model Kimura 2 Parameter ditemukan antara Bos javanicus dengan Madura 41 dengan nilai sebesar 0.000. Bubalus bubalus dengan Bos taurus (Friesian Holstein) memiliki nilai jarak genetik paling besar yaitu sebesar 0.293, dengan nilai perbandingan kejadian transisi dan tranversi sebesar 56/33 (Tabel 4). Kejadian subtitusi baik transisi maupun tranversi pada ruas Dloop lebih sering terjadi daripada ruas Cyt b ataupun tRNA. Hal ini menunjukkan bahwa ruas Dloop merupakan ruas yang memiliki laju mutasi yang lebih tinggi.
Topologi filogeni berdasarkan ruas Dloop sama dengan topologi berdasarkan ruas Cyt b, yaitu terjadi dua pengelompokan sapi madura. Sapi madura tipe I berkelompok dengan B. indicus dengan boostrap 91% dan sapi madura tipe II dengan B. javanicus dengan boostrap 96% (Gambar 8).
Berdasarkan runutan nukleotida, dua kelompok sapi madura dapat dibedakan dengan munculnya insersi/delesi sepanjang 17 basa (basa ke 15430-15446) pada awal ruas Dloop. Insersi/delesi tersebut hanya ditemuka n pada sapi madura tipe II dan B. javanicus. Insersi/delesi yang membedakan dua kelompok sapi madura juga ditemukan pada basa ke 15486-15489, 15529-15541, 15809 dan 15881-15887 (Lampiran 2)
Tabel 4 Nilai jarak genetik (di bawah diagonal) dan nilai perbandingan kejadian transisi dengan tranversi (di atas diagonal) berdasarkan ruas Dloop dengan menggunakan metode Kimura 2 Parameter (K2P).
19
Gambar 8 Hasil rekonstruksi pohon filogeni berdasarkan ruas Dloop dengan menggunakan metode NJ dengan boostrap 1000x.
Mutasi pada Ruas tRNA
Ruas tRNA Thr terbagi menjadi empat belas wilayah sesuai dengan urutannya pada saat melipat membentuk struktur daun semanggi (clover leaf) seperti yang terlihat pada gambar 9. Urutan basa pada ruas tRNA Thr relatif stabil pada spesies berdekatan. Mutasi hanya ditemukan di beberapa titik antara lain pada wilayah Batang D (D stem) dimana terjadi perubahan basa C pada B. javanicus dan keturunannya (sapi madura tipe I) menjadi basa T pada B. taurus,
B. indicus dan keturunannya (sapi madura tipe II). Pemisahan antara B. javanicus
dan keturunannya dengan B. taurus, B. indicus dan keturunannya juga terlihat pada mutasi yang terjadi pada wilayah daerah beragam (variable region) yaitu basa A menjadi G dan pada wilayah Gelung T (T loop) dimana basa TC berubah menjadi CT (Gambar 9). Seluruh kejadian mutasi yang terjadi pada wilayah tRNA Pro adalah transisi, dimana perubahan terjadi hanya pada basa purin dengan purin atau pirimidin dengan pirimidin.
20
ditemukan pada wilayah Gelung T (T loop) dimana basa T berubah menjadi basa C hanya pada B. taurus. Mutasi kedua ditemukan pada wilayah batang antikodon (anticodon stem) dimana B. taurus, B. javanicus dengan keturunanya terpisahkan dari B. indicus dan keturunannya dengan adanya perubahan basa A menjadi basa G (Gambar 10).
1111111111111111111111111111111111111111111111111111111111111111111111 5555555555555555555555555555555555555555555555555555555555555555555555 2222222333333333333333333333333333333333333333333333333333333333333333 9999999000000000011111111112222222222333333333344444444445555555555666 3456789012345678901234567890123456789012345678901234567890123456789012
Bjavanicus3 GTCTTTGTAGTACATCTAATACACTGGTCTTGTAAACCAGAAAAGGAGAACAATCAACCTCCCTAAGACT Madura 29 GTCTTTGTAGTACATCTAATACACTGGTCTTGTAAACCAGAAAAGGAGAACAATCAACCTCCCTAAGACT Madura 41 GTCTTTGTAGTACATCTAATACACTGGTCTTGTAAACCAGAAAAGGAGAACAATCAACCTCCCTAAGACT Madura 32 GTCTTTGTAGTACATCTAATACACTGGTCTTGTAAACCAGAAAAGGAGAACAATCAACCTCCCTAAGACT B.taurus GTCTTTGTAGTACATCTAATATACTGGTCTTGTAAACCAGAGAAGGAGAACAACTAACCTCCCTAAGACT B.indicus GTCTTTGTAGTACATCTAATATACTGGTCTTGTAAACCAGAGAAGGAGAACAACTAACCTCCCTAAGACT Madura14 GTCTTTGTAGTACATCTAATATACTGGTCTTGTAAACCAGAGAAGGAGAACAACTAACCTCCCTAAGACT Madura26 GTCTTTGTAGTACATCTAATATACTGGTCTTGTAAACCAGAGAAGGAGAACAACTAACCTCCCTAAGACT Madura38 GTCTTTGTAGTACATCTAATATACTGGTCTTGTAAACCAGAGAAGGAGAACAACTAACCTCCCTAAGACT
Keterangan: a. Batang asam amino (acceptor stem), b. Penghubung 1 (connector 1), c. Batang D
(D stem), d. Gelung D (D loop), e. Batang D (D stem), f. Penghubung 2 (connector 2), g. Batang
anti kodon (anticodon stem), h. Gelung anti kodon, i. Batang anti kodon, j. Daerah beragam
(variable region), k. Batang T (T stem), l. Gelung T (T loop), m. Batang T (T stem), n. Batang
asam amino (Acceptor stem) (Wakita et al 1994; Helm et al 2000). Bagian yang berwarna abu-abu
adalah basa yang mengalami mutasi.
Gambar 9 Perbandingan runutan basa nukleotida tRNA Thr pada genus Bos.
111111111111111111111111111111111111111111111111111111111111111111 555555555555555555555555555555555555555555555555555555555555555555 333333333333333333333333333333333333344444444444444444444444444444 666666677777777778888888888999999999900000000001111111111222222222 345678901234567890123456789012345678901234567890123456789012345678
B.javanicus3 TCAAGGAAGAAACTGTAGTCTCACCATCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG Madura 29 TCAAGGAAGAAACTGTAGTCTCACCATCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG Madura 41 TCAAGGAAGAAACTGTAGTCTCACCATCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG Madura 32 TCAAGGAAGAAACTGTAGTCTCACCATCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG B. taurus TCAAGGAAGAAACTGCAGTCTCACCATCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG B. indicus TCAAGGAAGAAACTGTAGTCTCACCGTCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG Madura 14 TCAAGGAAGAAACTGTAGTCTCACCGTCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG Madura 26 TCAAGGAAGAAACTGTAGTCTCACCGTCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG Madura38 TCAAGGAAGAAACTGTAGTCTCACCGTCAACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTG
Keterangan: a. Batang asam amino (Acceptor stem), b. Penghubung 1 (Connector 1), c. Batang D
(D stem), d. Gelung D (D loop), e. Batang D (D stem), f. Penghubung 2 (Connector 2), g. Batang
anti kodon (anticodon stem), h. Gelung anti kodon, i. Batang anti kodon, j. Daerah beragam
(variable region), k. Batang T (T stem), l. Gelung T (T loop), m. Batang T (T stem), n. Batang
asam amino (Acceptor stem) (Wakita et al 1994; Helm et al 2000). Bagian yang berwarna abu-abu
adalah basa yang mengalami mutasi.
Gambar 10 Perbandingan runutan basa nukleotida tRNA Pro pada genus Bos.
21
PEMBAHASAN
Produk Perunutan dan Mutasi Nukleotida
Sampel sapi madura yang digunakan pada penelitian ini dipilih dari Kabupaten Bangkalan dan Sampang di Pulau Madura. Alasan pemilihan kedua kabupaten dikaitkan dengan pola persebaran sapi madura. Persebaran sapi madura berkaitan dengan proses tata niaga di Pulau Madura yang umumnya bergerak dari wilayah timur ke barat, terutama untuk dipasarkan diluar pulau (Harmadji 1993). Proses perunutan hanya berhasil dilakukan pada sampel yang berasal dari Kabupaten Sampang.
Data visualisasi dengan menggunakan gel poliakrilamid 6% menghasilkan ukuran pita tunggal yang bervariasi (Gambar 4a). Perbedaan ini disebabkan adanya perbedaan panjang pada runutan nukleotida ruas Dloop. Perbedaan panjang ini disebabkan adanya ruas berulang dan delesi. Pada sampel tipe I ukuran pita relatif sama, sebab ukuran Dloop hampir sama yaitu 578 bp untuk madura 38 dan 577 bp untuk madura 14 dan 26. Pada sampel tipe II variasi ukuran pita muncul akibat adanya ruas berulang dan delesi. Total panjang Dloop yang berhasil dirunutkan pada sampel 29 dan 41 adalah 755 bp, sedangkan sampel 32 hanya 624 bp. Hal ini menyebabkan ukuran pita pada madura 32 lebih rendah dari sampel 29 dan 41.
Berdasarkan data perunutan diketahui bahwa komposisi basa yang paling banyak menyusun ruas Cyt b, tRNA baik Thr maupun Pro, dan Dloop sapi madura adalah A dan T, yaitu sebesar 63.5% (Tabel 2). Pola komposisi basa pada sapi madura mengikuti mamalia secara umum, dimana komposisi basa terbesar pada DNA mitokondria adalah A dan T (Sbisa et al. 1997). Tapio & Grigaliunate (2002) menyatakan bahwa proporsi nukleotida A dan T pada genom mitokondria sapi kurang lebih sebesar 70%.
22
Pada runutan DNA sapi madura ditemukan beberapa kejadian mutasi baik proses delesi, insersi dan subtitusi (transisi atau tranversi). ditunjukkan dengan lebih sedikitnya jumlah basa berbeda pada Cyt b dibandingkan ruas Dloop (Tabel 2). Kestabilan ruas Cyt b juga terlihat pada kejadian jumlah subtitusi (transisi dan tranversi) nukleotida Tabel 3. Pada tabel tersebut perbandingan jumlah kejadian subtitusi (transisi dan tranversi) nukleotida pada Cyt b lebih sedikit dibandingkan dengan kejadian substitusi pada ruas Dloop. Hal tersebut menunjukkan bahwa ruas Dloop merupakan ruas yang memiliki laju mutasi tinggi, sedangkan ruas Cyt b merupakan ruas yang cenderung stabil. Kestabilan Cyt b berkaitan dengan fungsinya yang cukup penting di dalam mitokondria. Menurut Dasgupta et al. (2008), ruas Cyt b berfungsi menyandikan protein yang berperan pada proses fosforilasi oksidatif.
Perbedaan laju mutasi menyebabkan ruas Dloop lebih cocok sebagai penanda molekuler untuk intra spesies. Ruas Cyt b yang lebih stabil lebih cocok sebagai penanda molekuler antar spesies. Nei (1987) menyatakan bahwa ruas yang banyak mengalami subtitusi nukleotida kurang baik digunakan sebagai penanda molekuler antar spesies, sebab dapat meningkatkan nilai standar error.
Mutasi lain yang ditemukan pada runutan DNA sapi madura selain subtitusi adalah ruas berulang (tandem repeat). Ruas berulang menyebabkan variasi ukuran pada ruas Dloop. Ruas berulang sepanjang 22 nt dengan motif GTACAT AATATTAATGTAATAA ditemukan pada seluruh famili bovidae yang disejajarkan. Jumlah pengulangan ruas berulang berbeda-beda pada tiap spesies tersebut. Ruas berulang ditemukan berulang satu kali pada Bubalus bubalus, B. indicus, sapi madura tipe I, dan B. taurus. Pada sapi madura sampel 32 ruas berulang ditemukan berulang tiga kali dan pada B. javanicus, sapi madura 41 dan 29 berulang sembilan kali.
23
T menjadi G pada basa pertama pada motif ruas berulang (basa ke 15543 pada Gambar 6).
Pessole et al. (1999) menyatakan bahwa banyaknya pengulangan dan motif ruas berulang dapat berbeda antara individu maupun spesies. Ruas berulang tidak dimiliki oleh semua spesies mamalia. Ruas berulang dengan motif berbeda pernah ditemukan oleh Nijman et al. (2003) pada sampel sapi madura dengan urutan ATTACATTAATATTATGTACTT yang berulang sebanyak dua kali. Pada family bovidae ruas berulang pernah didokumentasikan pada Ovis aries sepanjang 75-76 nt pada ruas Dloop (Hiendleder et al. 2002).
Fungsi ruas berulang pada Dloop belum diketahui secara pasti. Keberadaan ruas berulang cenderung memperkecil ukuran genom dalam sejarah evolusinya (Avise 1994). Ruas berulang juga membentuk struktur batang gelung yang kuat (hairpain), struktur ini dapat menghambat kerja enzim polymerase secara in vitro
(Gemmel et al. 1996; Farajallah 2005). Struktur batang gelung ini diperkirakan merupakan salah satu faktor yang menyebabkan tingkat keberhasilan yang rendah selama proses amplifikasi.
Ruas berulang pada sapi madura tipe II terletak di dekat tRNA Pro tepatnya pada basa ke 15542 sebelum ruas TAS.Posisi ruas berulang pada sapi madura tipe II ini sama dengan posisi ruas berulang pada rodensia dari genus Clethrionomys
(Matson & Baker 2001), kelelawar (Wilkinson & Chapman 1991), dan ikan cod (Johansen et al. 1990). Pada genus Clethrionomys ruas berulang terletak pada basa ke 251-276 sebelum ruas TAS (Matson & Baker 2001). Menurut Gemmel et al. (1996), motif berulang (tandem repeat) pada mamalia umumnya berlokasi pada dua tempat. Lokasi pertama pada ujung 3’ berdekatan dengan ruas TAS dan yang kedua biasanya terletak diantara OH (promotor replikasi utas berat) dan promotor utas ringan yang berdekatan dengan ruas CSB.
24
Ruas TAS yang terdeteksi umumnya lebih stabil diantara spesies yang berdekatan. Berdasarkan data dapat diketahui hanya ada tiga basa nukleotida yang berbeda diantara semua spesies yang disejajarkan. Kestabilan ruas TAS ini juga diungkapkan oleh Matson dan Baker (2001), dari 57 sampel rodensia dari genus
Clethrionomys hanya ditemukan 7 (12%) nukleotida yang berbeda. Kestabilan ruas TAS berkaitan dengan fungsinya sebagai sinyal untuk terminasi pada peristiwa replikasi (Pessole et al 1999).
Kejadian mutasi juga ditemukan pada ruas tRNA baik Thr maupun Pro. Beberapa mutasi ditemukan pada wilayah batang dan gelung (Gambar 9 dan Gambar 10). Mutasi pada wilayah gelung umumnya ditemukan pada beberapa spesies yang berbeda (Kern & Kondrashov 2004). Pada Gambar 9 dapat diketahui bahwa B. javanicus (sapi madura tipe II) dan keturunannya terpisah dari B. taurus
dan B. indicus beserta keturunanya (sapi madura tipe I) berdasarkan mutasi pada wilayah gelung T. Pemisahan antara B. javanicus dengan B. indicus dan B. taurus
juga diketahui berdasarkan mutasi pada wilayah batang (Gambar 9 dan Gambar 10).
Pemisahan antara B. javanicus dengan B. indicus dan B. taurus berkaitan dengan sejarah awal domestikasi ketiga jenis sapi tersebut. B. taurus dan B. indicus memiliki nenek moyang yang sama yaitu B. prigimineus, sehingga keduanya memiliki kekerabatan yang dekat. Kedekatan diantara B. taurus dengan
B. indicus ditunjukkan dengan hasil persilangan yang fertil (Loftus et al. 1994; Verkaar et al. 2004). Lenstra & Bradley (1999) menyatakan kesuksesan persilangan antara B. indicus dan B. taurus menunjukkan bahwa kedua spesies tersebut belum sepenuhnya terspesiasi.
Analisis filogeni
25
Nijman et al. (2003) yang menyatakan bahwa terdapat dua tipe mtDNA sapi madura berdasarkan ruas Dloop yaitu tipe mtDNA B. indicus dan tipe mtDNA B. javanicus. Hal ini menunjukkan bahwa sapi madura berasal dari dua moyang betina yang berbeda.
Munculnya dua moyang betina kemungkinan kemungkinan disebabkan karena kecilnya tingkat keberhasilan persilangan antara B. javanicus dengan B. indicus. Sehingga persilangan dilakukan dengan dua cara yaitu dengan menggunakan pejantan B. javanicus dengan betina B. indicus atau sebaliknya pejantan B. indicus dengan betina B. javanicus hingga didapatkan keturunan yang fertil.
Menurut Rollinson (1984) kecilnya tingkat keberhasilan persilangan antara
B. indicus dengan B. javanicus disebabkan karena perbedaan bentuk kromosom Y diantara keduanya. Bos indicus memiliki bentuk kromosom akrosentris sedangkan
B. javanicus memiliki bentuk kromosom metasentris. Perbedaan bentuk kromosom mengakibatkan gangguan pada proses spermatogenesis, sehingga terkadang F1 jantan yang dihasilkan keduanya bersifat steril .Vietmeyer (1983) menyatakan bahwa 1 dari 4 betina dan 3 dari 4 jantan hasil persilangan B. javanicus dengan B.indicus bersifat steril. Keberhasilan persilangan antara B. indicus dengan B. javanicus adalah sekitar 70%.
Persilangan antara banteng dan B. indicus diperkirakan terjadi sejak masuknya kebudayaan Hindu yang dibawa oleh bangsa india ke Indonesia. Awal masuknya bangsa india ke Indonesia terjadi sekitar 1800 tahun yang lalu dengan membawa sapi-sapi dari jenis B. indicus. Sapi-sapi jenis B. indicus ini kemudian disilangkan dengan banteng yang banyak terdapat di wilayah dataran rendah Pulau Jawa bagian timur (Payne & Wilson 1999). Menurut Ashari dan Liem (1993), banteng masih banyak dijumpai sebelum penggundulan hutan sekitar 150 tahun yang lalu di Pulau Madura.
26
Jawa. Keturunan sapi ongole yang telah disebarkan kemudian banyak disilangkan dengan sapi potong lokal. Tujuan pemerintah mengeluarkan kebijakan ini adalah untuk menciptakan bangsa sapi potong yang berkualitas baik (Diwyanto 2008).
Rentang waktu dari awal masuknya bangsa india ke Indonesia dan program ongolisasi hingga saat ini, cukup untuk membentuk suatu bangsa sapi madura yang stabil. Pembentukan bangsa sapi membutuhkan waktu sekitar 10-20 tahun dibawah kendali manusia yang intensif, yaitu misalnya dengan metode inseminasi. Pada kondisi alami pembentukan bangsa sapi membutuhkan waktu lebih dari 100 tahun (Simm 2000). Jika waktu generasi untuk sapi adalah 4-5 tahun (Dakay et al. 2006), maka dibutuhkan lebih dari 25 generasi untuk membentuk suatu bangsa sapi baru yang stabil.
Asumsi mengenai nenek moyang sapi madura berasal dari B. taurus tidak terbukti pada penelitian ini. Berdasarkan pohon filogeni dan jarak genetik dari ruas Cyt b maupun Dloop tidak terlihat adanya kedekatan sapi madura dengan B. taurus. Hal ini juga dibukt ikan oleh penelitian Surjoatmodjo (1993) dengan cara membandingkan morfologi dari B. taurus, B. indicus, sapi bali, dan sapi madura. Morfologi yang dibandingkan antara lain tinggi gumba (punuk), panjang badan, lebar dada, tinggi panggul, lebar panggul, lebar paha, lingkar dada, lebar dahi, dan panjang daho. Berdasarkan analisis varian disimpulkan bahwa jarak kekerabatan sapi madura terdekat adalah dengan peranakan ongole (B. indicus) dan yang paling jauh dengan B. taurus. Nilai jarak kekerabatan sapi madura dengan sapi bali berada ditengah-tengah antara B. taurus dengan peranakan ongole.
27
KESIMPULAN
Hasil penelitian menunjukkan adanya percampuran pada asal usul sapi madura berdasarkan garis maternal. Berdasarkan ruas mtDNA baik yang memiliki laju mutasi yang lambat (Cyt b dan tRNA) maupun ruas yang memiliki laju mutasi tinggi (Dloop), sapi madura mengelompok menjadi dua tipe yaitu tipe B. indicus dan B. javanicus. Hasil penelitian juga tidak menunjukkan kedekatan antara sapi madura dengan B. taurus.
SARAN
28
DAFTAR PUSTAKA
Aryogi, Affandhy L, Romjali E, Hartati. 2007. Pelestarian dan pemanfaatan plasma nutfah sapi potong. Pasuruan: Loka Penelitian Sapi Potong Grati.
Astuti M. 2004. Potensi dan keragaman sumberdaya genetik sapi peranakan ongole (PO)[Catatan Penelitian]. Yogyakarta: Universitas Gadjah Mada.
Ashari T, Liem C. 1993. Karakteristik sapi madura dalam perbandingan dengan ruminansia besar lainnya dan peluang pengembangannya. Di dalam: Hasil Penelitian dan Pengembangan Sapi Madura. Prosiding Pertemuan Ilmiah; Sumenep, 11-12 Oktober 1992. Sumenep.
Avise JC. 1994. Molecular Markers, Natural Hystory and Evolution. Chapman and Hall. New York.
Bradley BG, MacHugh DE, Cunningham P, Loftus RT. 1996. Mitochondrial diversity and the origin of african and europaean cattle. Proc Natl Acad 1: 5131-5135.
Bollongino R, Edwards CJ, Alt KW, Burger J, Bradley DG. 2006. Early history of european domestic cattle as revealed by ancient DNA. Biolett 2: 155-159. Cassell B. 2007. Dairy Guidelines Dairy Crossbreeding: Why and How. Virginia:
Virginia State University.
Dasgupta S, Hoque MO, Upadhyay S, Sidransky D. 2008. Mitochondrial cytochrome b gene mutation promotes tumor growth in bladder cancer. Cancer Res 68:700-706.
Dakay I, Marton D, Bene S, Kiss B, Zsuppan Z, Szabo F. 2006. The age at first calving and the longevity of beef cows in Hungary. Arch Tierz Dummerstorf
49: 417-425
Diwyanto. 2008. Pemanfaatan sumber daya lokal dan inovasi teknologi dalam mendukung pengembangan sapi potong di Indonesia. Pengembangan Informasi Pertanian 1: 173-178
Foran DR, Hixon JE, Brown WM. 1988. Comparisons of ape and human sequences that regulate mitochondrial DNA transcription and dloop DNA synthesis. Nucleic Acids Res. 16: 5841-5861.
Farajallah A. 2005. Kajian pembentukan ruas berulang dalam daerah pengontrol genom mitokondria labi-labi [Laporan Penelitian]. Bogor: Lembaga Penelitian dan Pemberdayaan Masyarakat, Institut Pertanian Bogor.
29
Gemmel NJ, Western PS, Watson JM, Graves JAM. 1996. Evolution of the mammalian mitochondrial control region comparisons of control region sequences between monotreme and therian mammals. Mol Biol Evol 13: 798-808.
Handiwiryawan E, Noor RR, Muladno, Schuler L. 2003. The use of HEL9 and INRA035 microsatellites as specific markers for bali cattle. Arch Tierz Dummerstorf 46: 503-512.
Harmadji. 1993. Prospek pengembangan sapi madura. di dalam: hasil penelitian dan pengembangan sapi madura. Prosiding Pertemuan Ilmiah; Sumenep, 11-12 Oktober 1992. Sumenep.
Helm M, Brule H, Friede D, Giege R, Putz D, Florentz C. 2000. Search characteristic structural features of mammalian mitochondrial tRNAs. RNA 6: 1356-1379.
Hiendleder S, Kaupe B, Wassmuth R, Janke A. 2002. Molecular analysis of wild and domestic sheep questions current nomenclature and provides evidence for domestication from two different subspecies. Proc R Soc Lond 269: 893–904. Hoelzoel AR, Lopez JV, Dover GA, Brien JO. 1994. Rapid evolution of a
heteroplasmic repetitive sequences in the mitochondrial DNA control region of carnivores. J Mol Evol 39:191-199.
Hou WR, Chen Y, Wu X, Hu JC, Peng ZS, Yang J, Tang ZX, Zhou CQ, Li YM, Yang SK, Du YJ, Kong LL, Ren ZL, Zhang HY, Shuai S. 2007. A complete mitochondrial genome sequence of asian black bear sichuan subspecies (Ursus thibetanus mupinensis). Int J Biol Sci 3: 85-90.
Johansen S, Guddal PH, Johansen T. 1990. Organization of the mitochondrial genome of atlantic cod, Gadus morhoa. Biochim Biophys Acta 1218: 213-217. Kern AD, Kondrashof FA. 2004. Mechanisms and convergence of compensatory
evolution in mammalian mitochondrial tRNAs. Nature Genetics 36: 1207-1212.
Kusdiantoro M, Olsson M, Anderrson G, Purwantara B, Tol HTA, Mikko S, Martinez HR, Colenbrander B, Lenstra JA. 2007. Genetic diversity and conservation of South-East Asian cattle: from indian zebu to indonesian banteng, and then to the cambodian kouprey. Asia Link: 120-124.
Kusdiantoro M, Olsson M, Tol HTA, Mikko S, Vlamings BH, Andersson G, Martinez HR, Purwantara B, Paling, Colender B. Lenstra JA. 2009. The origin of indonesian cattle. PloS ONE 4: 1-5.
30
Lenstra JW, Bradley. DG. 1999. Systematic and phylogeny of cattle. Di dalam: Fries R dan Ruvinsky A, editor. The Genetics Of Cattle. United Kingdom. CABI Publising.
Lightner JK. 2008. Karyotipe variability within the cattle monobramin. Answer Research Journal 1: 77-78.
Loftus RT, MacHugh DE, Bradley DG, Sharp PM, Cunningham P. 1993. Evidence for two independent domestication of cattle. Proc Natl Acad 91: 2757-2761.
Maksum K. 1993. Hasil-hasil penelitian sapi madura di Sub Balai Penelitian Ternak Grati-Pasuruan. Di dalam: Hasil Penelitian dan Pengembangan Sapi Madura. Prosiding Pertemuan Ilmiah; Sumenep, 11-12 Oktober 1992. Sumenep.
Mannen H, Tsuji S, Loftus RT, Bradley DG. 1998. Mitochondrial DNA variation and evolution of japanese black cattle (Bos taurus). Genetics 150: 1169-1175. Matson CW, Baker JR. 2001. DNA sequence variation in the mitochondrial
control region of red blacked voles (Clethrionomys). Mol Biol Evol 18: 1494-1501.
Matthee CA, Davis SK. 2001. Molecular insights into the evolution of the familly bovidae: nuclear DNA perspective. bovid molecular phylogeny. Mol Biol Evol
18: 1220-1230.
McHugh DE. 1997. Molecular biogeography and genetic structure of domestic cattle [Thesis]. University of Dublin.
Nei M. 1987. Molecular Evolutionary Genetics. New York. Colombia Univ Pr. Namikawa T. 1981. Geographic distribution of bovine hemoglobin-beta (hbb)
aleles and phylogenetic analysis of the cattle in Eastern Asia. Zeitschift fur Tierzuchtung and Zuchtungsbiologie 98: 151-159.
Nijman IJ, Otsen M, Verkaar ELC, Ruitjer CD, Hanekamp E, Ochieng JW, Shamshad S, Rege JEO, Hannotte O, Barwegen MW, Sulawati T, Lenstra JA. 2003. Hybridization of banteng (Bos javanicus) and zebu (Bos indicus) revealed by mitochondrial DNA, satellite DNA, AFLP microsatelites. Heredity
90:10-16.
Pessole G, Gissi C, Chirico AD, Saccone C. 1998. Nucleotide subtitution rate of mammalian mitochondrial genomes. J Mol Evol 48:427-434.
Payne WJA, Hodges J. 1997. Tropical Cattle: Origins, Breed, and Breeding Policies. Oxford: Blackwell Science Ltd.
31
Roderick GK. 1996. geographic structure of insect population: gene flow, phylogeography and their uses. Annu Rev 41: 325-352
Rollinson DHL. 1984. Bali cattle. Di dalam: Mason IL, editor. Evolution of Domestic Cattle. New York. Longman.
Rouse JE. 1972. Cattle Of Afrika and Asia. Oklahoma. University Of Oklahoma Press.
Sambrook J, Fritsch EF, Miniatis T. 1989. Molecular Clooning: A laboratory Manual. Ed ke-8. New York: Cold Spring Harbor Laboratory Press.
Sbisa EF, Reyes A, Pesole G, Sacone C. 1997. Mammalian mitochondrial D-loop region structural analysis: identification of new conserved sequences and their functional and evolutionary implication. Gene 250:125-140.
Simm G. 2000. Genetic Improvement of Cattle and Sheep. United Kingdom: Farming Press.
Sitorus P, Soepeno, Subandriyo. 1993. Strategi, arah dan program penelitian sapi potong (sapi madura). Di dalam: Hasil Penelitian dan Pengembangan Sapi Madura. Prosiding Pertemuan Ilmiah; Sumenep, 11-12 Oktober 1992. Sumenep.
Surjoatmodjo M. 1993. Asal usul sapi madura ditinjau dari hasil pengukuran bagian-bagian tubuhnya. Di dalam: Hasil Penelitian dan Pengembangan Sapi Madura. Prosiding Pertemuan Ilmiah; Sumenep, 11-12 Oktober 1992. Sumenep.
Tamura K, Dudley J, Nei M & Kumar S (2007) MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution 10.1093/molbev/msm092.
Tapio M, Grigaliunaite I. 2002. Is there a role for mitochondrial in sheep breeding? Veterinarija Ir Zootechnika 18:108-111.
Tapio M, Grigaliunaite I. 2003. Use of mitochondrial DNA as a genetic marker in domesticated mammals. Ekologija 1: 31-33.
Tegelstrom H. 1986. Mitochondrial DNA in natural population: an improved routine for screening of genetic variation based on sensitive silver staining.
Electrophoresis 7: 226-229.
Uggla CM. 2008. Investigating genetic variability within specific indigenous Indonesia cattle breed [Disertasi]. Swedish University of Agricultural Science. Verkaar ELC, Nijman I, Beeke M, Hanekamp E, Lenstra JA. 2004. Maternal and
32
Vietmeyer ND. 1983. Little Known Asian Animals With a Promising Economic Future. Washington: National Academy Press.
Williamson G, Payne WJA. 1965. An Introduction To Animal Husbandry In The Trophic. London: Loungman Group Limited.
Wilkinson GS, Chapman AM. 1991. Length and sequence variation in evening bat dloop mtDNA. Genetics 128: 607-617.
Perbesaran Lokasi A di P. Madura
Lampiran 1. Peta Lokasi Pengambilan sampel sapi madura di Pulau Madura
Perbesaran Lokasi B di P. Madura
Keterangan:
Angka pada peta menunjukkan nomor
koleksi sampel darah sapi madura
U
B T
S
34
Lampiran 2 Hasil penjajaran DNA ruas Cyt b,tRNA Thr, tRNA Pro dan Dloop
genom mitokondria sapi madura dengan data GenBank. Keterangan:
nomor posisi nukleotida dibaca secara vertikal pada tiga baris
pertama berdasarkan referensi B. indicus (AF492350).
Ruas Cyt
Bubalusbub GAAGTATGAT GTTCCGGCCA TTCAGCCAAT GCCTATTCTG AATTCTAG BosTaurus1 ....C..A.. A...A... C... ... .GCC....
35
Bubalusbub ACCCATATAT TATCATTGGA CAACTAGCAT CTATCACATA CTTCCTCC BosTaurus1 ... C.C...C... ... ..G..CT... ...T....
36
Lampiran 2 (Lanjutan)
11111111111
55555555555
22222222222
78888888888
90123456789
37
B.javanicus3 ATTTGAACTA TTTCCTAAGC ACTATTAATA TAGTTCCATA AACGCAAA Madura 29 ... ... ... ... ...
38
Lampiran 2 (Lanjutan)
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 4444444444 4444444444 4444444444 4444444444 44444444 3333444444 4444555555 5555666666 6666777777 77778888 6789012345 6789012345 6789012345 6789012345 67890123
B.javanicus3 GAATTTGTAC CCTAACCAAA TATCACAAAC ACCACTAGCT AATAACAC Madura 29 ... ... ... ... ... Madura 41 ... ... ... ... ... Madura 32 ... ... ... ... ... B. taurus ...AC.. ... ...T... ... ..C.TA.. B. indicus ...C.. ... ...T... ... ..C.TA.. Madura 14 ...C.. ... ...T... ... ..C.TA.. Madura 26 ...C.. ... ...T... ... ..C.TA.. Madura 38 ...C.. ... ...T... ... ..C.TA.. Bub bubalus ..C...AC.. T...G..T.. C..T.G...T .A.TAC.A.C .TC...
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 4444444444 4444445555 5555555555 5555555555 55555555 8888889999 9999990000 0000001111 1111112222 22222233 4567890123 4567890123 4567890123 4567890123 45678901
39
lampiran 2 (Lanjutan)
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 5555555555 5555555555 5555555555 5555555555 55555555 3333333344 4444444455 5555555566 6666666677 77777777 2345678901 2345678901 2345678901 2345678901 23456789
B. javanicus --- GTACATAATA TTAATGTAAT AAGTACATAA TATTAATG Madura 29 --- ... ... ... ... Madura 41 --- ... ... ... ... Madura 32 --- ... ... ... ... B. taurus AAGGGGTAAT ...C. ... ..--- --- B. indicus AAGAGGTAAT ...C. ... ..--- --- Madura 14 AAGAGGTAAT ...C. ... ..--- --- Madura 26 AAGAGGTAAT ...C. ... ..--- --- Madura 38 AAGAGGTAAT ...C. ... ..--- --- Bub bubalus ATGGGGGGGG .G...C. ... ..--- ---
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 5555555555 5555555555 6666666666 6666666666 66666666 8888888888 9999999999 0000000000 1111111111 22222222 0123456789 0123456789 0123456789 0123456789 01234567
40
Lampiran 2 (Lanjutan)
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 6666666666 6666666666 6666666666 6666666666 66666666 2233333333 3344444444 4455555555 5566666666 66666666 8901234567 8901234567 8901234567 8901234567 89012345
B. javanicus3 AGTACATAAT ATTAATGTAA TAAGTACATA ATATTAATGT AATAAGTA Madura 29 ... ... ... ... ... Madura 41 ... ... ... ... ... Madura 32 --- --- --- --- --- B. taurus --- --- --- --- --- B. indicus --- --- --- --- --- Madura 14 --- --- --- --- --- Madura 26 --- --- --- --- --- Madura 38 --- --- --- --- --- Bub bubalus --- --- --- --- ---
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 6666666666 6666666666 6666777777 7777777777 77777777 7777888888 8888999999 9999000000 0000111111 11112222 6789012345 6789012345 6789012345 6789012345 67890123
41
Lampiran 2 (Lanjutan)
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 7777777777 7777777777 7777777777 7777777777 77777777 2222223333 3333334444 4444445555 5555556666 66666677 4567890123 4567890123 4567890123 4567890123 45678901
B. javanicus3 ATATTAATGT AATAAAGACA TAATATGTAT ATAGTACATT ACATTACA Madura 29 ... ... ... ... ... Madura 41 ... ... ... ... ... Madura 32 --- ---... ... ... ... B. taurus --- ---... ... ... .A....T. B. indicus --- ---... .G... ... .A....T. Madura 14 --- ---... .G... ... .A....T. Madura 26 --- ---... .G... ... .A....T. Madura 38 --- ---... .G... ... .A....T. Bub bubalus --- ---G.... ... ... .T....T.
1111111111 1111111111 1111111111 1111111111 11111111 5555555555 5555555555 5555555555 5555555555 55555555 7777777777 7777777777 7777777788 8888888888 88888888 7777777788 8888888899 9999999900 0000000011 11111111 2345678901 2345678901 2345678901 2345678901 23456789
42
B. javanicus3 GTACATTAAT CCACTAATCG TACATAGTAC ATT-ACATCA AATCTACC Madura 2 ... ... ...C.. ...-.T.... ...T.
43
Lampiran 2 (Lanjutan)
11111
55555
99999
00000
67890