ISOLASI DAN KARAKTERISASI PUSTAKA MlKROSATELlT
DENGAN METODE PENGKAYAAN
(Enrichment)
PADA TANAMAN JATl
(Tectona grandis)
ASEP SAEFULLOH
SEKOLAHPASCASARJANA
INSTITUT PERTANIAN BOGOR
SURATPERNYATAAN
Dengan ini saya menyatakan bahwa tesis yang berjudul :
ISOLASI DAN KARAKTERISASI PUSTAKA MIKROSATELIT DENGAN METODE PENGKAYAAN (Enrichment) PADA TANAMAN JATI (Tectona grandis)
Adalah karya saya sendiri dengan bimbingan dari komisi pembimbing dan belum pernah dipublikasikan dalam bentuk apapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka dibagian akhir tesis.
ABSTRAK
ASEP SAEFULLOH. lsolasi dan Karakterisasi Pustaka Mikrosatelit dengan Metode Pengkayaan (enrichment) Pada Tanaman Jati (Tectona grandis). Di bawah bimbingan Agus Purwito, Dedy Duryadi S., dan Adi Pancoro.
Jati (Tecfona grandis Linn. f.) merupakan tanaman kayu yang penting secara komersial dan dikenal luas sebagai bahan baku industri perkayuan yang berkualitas. Saat ini dipasaran lokal banyak beredar bibit jati baik yang diperbanyak dengan biji atau dengan teknik kultur jaringan (stek), bentuk morfologi yang hampir sama rnembuat kita sulit membedakan jenis mana yang merupakan bibit unggul. Untuk itu diperlukan suatu penanda rnolekuler yang dapat membedakan jenis-jenis jati tersebut. lnformasi tentang penanda molekuler dalam mengidentifikasi perbedaan genetik tanaman jati ini rnasih sangat terbatas.
Pada penelitian ini dilakukan isolasi dan karakterisasi motif mikrosatelit pada tanaman jati untuk mendapatkan primer-primer spesifik yang dapat digunakan untuk rnengkarakterisasi keragaman genetik jati. Sebanyak 16 oligonukleotida bermotif mikrosatelit telah dihibridisasi dengan fragmen DNA jati hasil pemotongan dengan enzim restriksi Alul, Rsal dan Hincll yang telah diligasikan dengan adaptor Mlul. Fragmen hasil hibridisasi kemudian dielusi dan diarnplifikasi dengan primer 21-rner untuk memperoleh produk PCR sebagai fragmen untuk diinsersikan ke vektor plasmid pGEM-T Easy sehingga diperoleh konstruksi pustaka mikrosatelit jati.
Pada penelitian ini telah diperoleh 6 jenis pustaka mikrosatelit tanaman jati yaitu: JB-I; JB-2; JB-3; JL-1; JL-2; JL-3, dan berdasarkan pengujianlverifikasi pada 30 plasmid rekombinan telah diperoleh insert yang merupakan kandidat fragmen mikrosatelit dengan ukuran sekitar 200 dan 1.200 bp.
Sekuen dilakukan terhadap empat plasmid rekombinan yang berhasil diisolasi dengan baik dari sekitar 500 koloni transforman (0.8%). Dua sampel (JB36, JB39) berupa produk PCR dengan ukuran fragmen sisipan yang disekuen berkisar 200-an pasang basa dan dua sarnpel lain (JBI-I dan JB5C) merupakan plasmid rekombinan hasil isolasi plasrnid.
Dari keempat sampel yang disekuen, hanya sampel J B I - I yang cenderung memperlihatkan adanya motif mikrosatelit. Motif yang muncul dari sampel yang dianalisis adalah [CAI,, merupakan motif campuran tidak sempurna. Diperoleh satu pasang primer mikrosatelit yang dapat mengamplifikasi suatu daerah tertentu pada genom tanaman jati. Dari uji primer mikrosatelit hasil desain dengan teknik PCR, primer tersebut dapat dibuktikan bisa mengamplifikasi lokus tertentu dari suatu genom jati.
ABSTRACT
ASEP SAEFULLOH. Isolation and Characterization of Microsattelite Library with Enrichment Method on Teak Plants (Tectona grandis). Adviser Agus Purwito, Dedy Duryadi S., and Adi Pancoro.
Teak (Tectona grandis Linn. f.) is one important commercial woody plants that used as a basic material for a high quality wood industry. Nowadays, teak plant for Indonesian local market are propagated from seed or tissue culture. Both of these plant materials have similar phenotypes. It makes difficult for teak plant breeder to select elite lines. Molecular marker is an approach to solve this problem, in which variation among teak lines can be distinguished. At this moment, molecular marker to identifying teak genetic diversity is still limited.
In this study we isolate and characterize microsatellite motif of teak to be obtained specific primers that can be use for characterization genetic diversity of teak. Teak genome has been cleaved using Alul, Rsal and Hincll restriction enzymes, then DNA fragments were ligated with Mlul adaptor. Subsequently, fragments produced were hybridised with 16 oligonucleotides which are motifs of microsatellite. The hybridised fragments were eluted and amplified with a 21- mers primer using PCR and then PCR products have been inserted into pGEM-T Easy vector.
Results showed there are 6 candidates of teak microsatellite, which were JB-1, JB-2, JB-3, JL-1, JL-2, and JL-3. Based on EcoRl restriction enzyme site on 30 tested recombinant plasmids, it may indicate that the insertion fragment with 200 and 1.200 bp in size, as one candidate of teak microsatellite region.
Four recombinant plasmids that are success isolated from 500 transformant coloni (0,8%) were sequenced. Two samples (JB36, JB 39) are PCR products with 200 bp fragment insert size and two other samples (JBI-1, JB5C) are recombinant plasmid
The results show that, only JBI-1 was tending to show microsatellite motif [CAIn, which is inperfect compound motif. One pair microsatellite primer that can amplify certain region of teak genome was obtained . From microsatellite primer assay resulted from PCR technic, the primer proved to be able to amplify certain locus of teak genome.
ISOLASI DAN KARAKTERISASI PUSTAKA MIKROSATELIT
DENGAN METODE PENGKAYAAN
(Enrichment)
PADA TANAMAN JATl
(Tectona grandis)
ASEP SAEFULLOH
Tesis
Sebagai salah satu syarat untuk rnernperoleh gelar Magister Sains pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul : lsolasi dan KaraMerisasi Pustaka Mikrosatelit dengan Metode Pengkayaan (Enrichment) Pada Tanaman Jati (Tectona grandis)
Nama : Asep Saefulloh
NIM : P28500024
Disetujui,
Komisi Pembirnbing
/%-
Dr. I . us Punvito MSc Ketua
Dr. Ir. Dedv Duryadi Solihin, DEA Anggota
/
Adi Pancoro. Phd. AnggotaDr. ld./rGluhamrnad Jusuf
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SVVT atas segala rahmat dan karunia yang telah diberikan, sehingga karya ilmiah (laporan penelitian, tesis) ini dapat diselesaikan. Penelitian ini mengambil tema mengenai Penanda Molekuler (DNA) Mikrosatelit dengan Judul " lsolasi dan Karakterisasi Pustaka
Mikrosatelit dengan Metode Pengkayaan (Enrichment) Pada Tanarnan Jati (Tectona grandis)
".
Penelitian yang dilaksanakan sejak bulan Januari 2004 sampai Desember 2005 merupakan bagian dari Penelitian DlPA yang diselenggarakan oleh Pusat Studi Regional Penelitian Biologi Tropis (SEAMEOBIOTROP).
Selama melaksanakan penelitian dan menyelesaikan laporan tesis ini penulis telah mendapatkan bimbingan, bantuan, dan dukungan dari semua pihak. Untuk itu penulis mengucapkan terima kasih yang tak terhingga kepada :
1. Dr. IT. Agus Purwito, M.Sc. dan Dr. Dedy Duryadi S., DEA. selaku dosen pembimbing yang telah banyak memberikan saran, arahan, dorongan dan bimbinaan.
2. Adi ~ i n c o r o , PhD. yang telah mengijinkan penulis menggunakan metode penelitian dan fasilitas Laboratorium Genetika ITB sekaligus bersedia menjadi
-
dosen pembimbing.3. Dr. Handoko (Direktur SEAMEO BIOTROP) yang telah mendorong penulis untuk melanjutkan studi di Program Master Bioteknologi Pascasarjana lnstitut Pertanian Bogor.
4. Ir. Arief Chandra S., M.Si. (Direktur PT. PPA Agricola) selaku atasan penulis yang selalu memberikan dorongan, dukungan, dan bantuan serta mengijinkan penulis untuk menyelesaikan studi.
5. Dr. Herman dan Dr. M. Yunus yang telah mengijinkan penulis menggunakan fasilitas Laboratorium Biologi Molekuler BB-BIOGEN.
6. Proyek DlPA 2003 dan 2005 SEAMEO BIOTROP yang telah mendanai penelitian penulis.
Ungkapan terima kasih khusus dan penghargaan penulis sampaikan kepada Bapa dan Mamah, Teh Ai, dan Eneng, serta seluruh keluarga atas segala do'a dan kasih sayang yang selalu diberikan kepada penulis. Kepada istri tercinta (Fitri Yelli, SP. M.Si.), terima kasih dan penghargaan sedalam-dalamnya penulis ucapkan atas segala dorongan dan semangat, kesabaran dan kasih sayang serta pengorbanan dan pengertiannya.
DAFTAR IS1
Halaman
DAFTAR IS1 I
DAFTAR TABEL iii
DAFTAR GAMBAR ... ... .. . ... . .. ... ... ... .. . ... . .. ..
.
...
... . .. ... ... . .. .. . ... ... ... ... ... . ivDAFTAR LAMPIRAN ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ...
... ...
...
... ... ... vPENDAHULUAN
Latar Belakang ... ...
...
... ... ... ... ... ... ... ... ... . . . 1 . .Tujuan Penel~t~an ... ... ...
...
... ... ... ... ... ... ... ...... .
. ,
. . ..
4TINJAUAN PUSTAKA
5 6 6 7
Aplikasi Mikrosate
. .
8lsolas~ Mikrosatelit ... .. . .... ... ... ... ... ... ...
... ...
... ... ... ... ... .... ... ... ... ...,... I 0Perancangan Primer ... ... .... ... ... ...
. ..
.. . ... . .. ......
... ... ... ... ....
...
... ... ... ... I 2Perancangan Primer dengan Program Internet . ..
...
...
... ... ... .... ... 13METODE PENELlTlAN
Tempat dan Waktu ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... . Bahan dan Alat
Metode Pelaksanaan
Pemotongan DNA Genom Jati ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... .... Ligasi Fragmen DNA dengan Adaptor ... ... ... ... ... ... ...
...
... ... ... ... ... ... Hibridisasi ... ... ... ... ... .. . ... ... ... ... ... .., ,,. ... ... .., ... ... ......
. .. .. . ... Persiapan membran ......
.... ......
... ... ......
... ... .., ,.. ... ... .,, ... ... ... .... . .
.
Proses h~brldlsas~ ... ... ... ...
...
... ... ......
... ......
... ... ......
... ......
... ......
Amplifikasi DNA elusi ... ... ... ... ... ... ... ... ......
... ... ... ... ... ... ... ... ... ... . Ligasi dengan Vektor Plasmid ......
... ... ......
... ... ... ... ......
... ......
... ... ... .. Transformasi . . ..
. . ..
. . .. .
. . . .. . . .......
.... ... Pembuatan sel kompeten E. Coli DH5-a ... ... ... ......
... ... ... ... ... ... Transformasi . . .. . .
. . .. .
. . ..
, . , . . . , , . . . ,.
, . . .. .
. . .. . .
. . .HASlL DAN PEMBAHASAN
Analisis Sekuensin Perancangan Primer
Posisi adaptor ... ... ... ..
.
... ... ... ... ... ......
...
... ... ... ... ... ......
. .. .. . ... ... . Motif mikrosatelit ... ... ......
... .. . ... . .. .. . .. . ....
.. .. . ... ... ... ... ... ...
...
... .... Desain primer...
... ... ......
... ... ... ... ...... ...
... ... ... ... ......
... ... ... ...SIMPULAN DAN SARAN
Sim pulan
.
. . .. . .
. . ..
.
.
. . ..
, . . .. . .
, . . .. .
. . .Saran . . . ... . . .
. . .
. . ..
. . ....
. . . , , . , . . . , . . ..
DAFTAR TABEL
Halaman
Perbandingan biaya. waktu dan hasil dari rnetode isolasi rnikrosatelit
Kornposisi reaksi pernotongan DNA genorn ...
Kornposisi reaksi ligasi DNA dengan adaptor ...
Komposisi reaksi PCR DNA terligasi adaptor ...
Kornposisi reaksi PCR DNA hasil elusi (enrichment) ...
Kornposisi reaksi ligasi DNA ke dalarn vektor
GEM"
. T Easy ...... Kornposisi reaksi pernotongan plasrnid dengan EcoRl
Pengelompokan oligonukleotida berdasarkan Melting Temperature ..
... Hasil skrining biru putih ... .... ... ...
Hasil skrining antibiotik
DAFTAR
GAMBAR
Halaman
Alur kerja pencarian motif mikrosatelit ...
Perpasangan adaptor 21-mer dan 25-mer ...
Hasil isolasi DNA genom jati
Pemotongan genom dengan enzim restriksi dan ligasi fragmen DNA dengan adaptor Mlul
Pengkayaan kandidat-kandidat fragmen DNA bermotif ...
Koloni biru-putih hasil transformasi ...
Hasil isolasi plasrnid
... Pemotongan DNA-Rekombinan dengan enzim restriksi EcoRl
... Verifikasi fragmen sisipan dengan PCR
... Urutan sekuen DNA sisipan hasil sekuensing
... Urutan nukleotida sample JBI-1
Hasil analisis primer3
DAFTAR LAMPIRAN
Halaman
1. Hasil sekuensing DNA sisipan jati (Charoen Pokphand) sarnpel J B I - I ... ...
...
43...
2. Output Primer3 44
3. Hasil sekuensing DNA sisipan jati (Charoen Pokphand) sarnpel ...
JB361, JB362, JB363, JB391 dan JB392 45
Latar Belakang
Jati (Tectona grandis Linn. f.) merupakan tanaman kayu penting secara komersial dan dikenal luas sebagai bahan baku industri perkayuan yang berkualitas. Di Indonesia, jati digolongkan sebagai kayu mewah yang termasuk kelas kayu dengan kualitas dan nilai jual yang tinggi. Penggunaan kayu jati
sudah sangat populer dan masih merupakan pilihan &ma karena keindahan
seratnya dan dikenal sebagai kayu yang tahan rayap serla jamur. Kebutuhan kayu jati baik dalam negeri maupun luar negeri selalu meningkat sedangkan populasi dan pasokannya semakin rnenipis yang disebabkan kendala siklus umur
panen jati konvensional yang relatif lama sekiir 45-60 tahun.
Dalam upaya mengantisipasi ha1 tersebut, saat ini telah dikembangkan
tanaman jati unggul yang berasal dari pohon induk terbaik setelah diseleksi dan dipilih dari tanaman jati biasa yang mempunyai sifat-sifat "lebih" dari populasi jati yang ada, kemudian diperbanyak dengan pembiakan vegetatii melalui teknik "mikropropagasi atau kukur jaringan". Tanaman induk ini berasal dari hasil
pemuliaan lokal (Indonesia) atau dari luar Indonesia, seperti Malaysia, Myanmar,
dan Thailand.
Bibi jati unggul hasil kuttur jaringan memiliki kecepatan pertumbuhan 3-4
kali dari pertumbuhan bibit jati asal biji, pertumbuhan pohon lebih seragam (>
80%). tingkat pertumbuhan per tahun lebih cepat (umur 2 tahun tinggi 4.75 m dan diameter batang 7.54 cm; Eliyani 2004), bentuk batang lebih lurus dan silindris,
serta tingkat percabangan ringan. Dengan umur panen yang dapat dicapai 15-20 tahun maka pengembalian modal lebih cepat dengan kualitas kayu layak jual, sehingga tanaman ini dapat dijadikan bidang investasi yang prospektif dimasa
yang akan datang. Beberapa "merk dagang" jati kultur jaringan yang beredar dipasaran, yaitu : Jati Biotropika, Jati Emas, Jati Super, Jati Perhutani Plus
(JPP), Jati Prima, Jati Kencana dan Jati Unggul (Tini dan Amri 2002; PPA Agricola 2003; Saefulloh 2003). Penggunaan bibit jati hasil kuktur jaringan untuk
budidaya telah dikembangkan di beberapa negara seperti India, Costa Rica, Thailand, Myanmar, Malaysia, dan Indonesia (Tini dan Amri 2002; Rimbawanto
Saat ini di pasaran lokal banyak beredar bibi jati baik yang diperbanyak dengan biji atau dengan teknik kulur jaringan, bentuk rnorfologi yang harnpir
sarna rnembuat kiia suli rnernbedakan jenis rnana yang rnerupakan bibi unggul. Jati konvensional atau jati biasa adalah tanarnan jati yang dihasilkan dari bibi asal biji (generatiif) yang rnerniliki laju perturnbuhan yang relatif larnbat dengan
tanarnan induk biasanya berasal dari hasil pemuliaan lokal.
Secara genetik sifat-sifat keunggulan jati kultur jaringan rnasih dipertanyakan. Apakah keunggulan secara rnorfologi dapat dibuktikan secara genetik? Untuk itu diperlukan suatu penanda rnolekuler yang dapat rnernbedakan
jenisjenis jati tersebut. lnforrnasi tentang penanda rnolekuler dalarn
rnengidentiikasi perbedaan genetik tanarnan jati ini rnasih sangat terbatas. Dalarn penelitian ini akan rnencoba rnengisolasi pustaka rnikrosateli dari dua
jenis jati yaitu : Jati BiotropikafJati Ernas (JB) dan Jati LokalIPerhutani (JL).
Perkembangan sejurnlah penanda rnolekuler (DNA marker) dewasa ini
telah memungkinkan untuk rnelakukan identiikasi terhadap perubahan- perubahan genetik yang terjadi dalarn suatu persilangan serta hubungannya dengan perubahan sifat kuantiiatii dan sifat kualitatif dari suatu klon (tanarnanlhewan). Selain itu, penanda rnolekuler juga dapat digunakan untuk
rnernbedakan antara suatu ras (tanarnanlhewan) dengan yang lainnya terutarna
dalarn kaitannya dengan upaya pelestarian dan rnenjaga kernumian dari ras tersebut (Philips dan Vasil2001).
Salah satu penanda rnolekuler (DNA marker) yang sangat populer dewasa ini adalah rnikrosatelit. Mikrosateli rnerupakan sekuen DNA sederhana pendek berulang, 1-6 pb berurulan mono-, di-, tri-, atau tetra- nukleotida berulang
dalarn bentuk kopi berdampingan (tandem), (CA1GT)n atau (TCG1AGCT)n (Harnada ef a/. 1982; Taulz dan Renz 1984). lstilah rnikrosateli yang sering
digunakan oleh para peneliti antara lain : shod tandem repeat (STR) (Craig ef. a/.
1988) dan simple sequences repeats (SSRs) (Jacob et. a/. 1991).
Pernetaan genorn rnenggunakan ale1 rnikrosateli dapat rnengidentifikasi
krornosom yang rnengandung gen-gen spesifik yang berpengaruh terhadap berbagai sifat kuantiiatif dan kualitatii tanarnanlhewan. Aplikasi rnikrosatelit
Pernanfaatan analisis rnikrosatelii untuk studi populasi alami dari tanarnan
tingkat tinggi belurn banyak diselidiki. Studi awal pada tanarnan hias atau tanaman budidaya rnembuktikan bahwa rnikrosatelii mempunyai marker polimotfik yang berlirnpah untuk identiikasi atau pernetaan genom (Philips dan
Vasil 2001). Penggunaan penanda mikrosatelii untuk analisis keragarnan genetik dan analisis perubahan genetik dari suatu populasi tanaman telah dilakukan pada : padi (Prasetiyono 2003; Song etal. 2003; Temnykh etal. 2000;
Chen ef a/. 1997). kedelai (Narvel et. a/. 2000), kelapa (Teulat, et. a/. 2000).
common bean (Yu ef. a/. 2000). Noway spruce (Swtti e t a/. 2002). Cannabis saliva (Gilmore, S. 2003), pinus (Provan et. a/. 2001) dan legum (Provan e t a/.
2001).
Penelitian ini mencoba mengisolasi dan mengkarakterisasi penanda rnikrosatelii yang dapat digunakan dalarn mengidentiFikasi dan verifikasi asal-usul
dan keaslian sualu klon jati unggul terkait dengan penampakan sifat kuantiiatii dan kualiiatif dari beberapa klon jati.
Penelitian pendahuluan, pembuatan pustaka rnikrosatelii telah dilakukan
baik pada klon jati unggul (jati emas) maupun pada klon jati lokal dan sampai saat ini masih berlangsung (Duryadi ef. a/. 2004; Saefulloh 2004, data belurn dipublikasi). Tahapan keja untuk rnengisolasi mikrosatelit tanaman secara urnum dimulai dengan pembuatan pustaka small-insert (200-600 pb), skrining
rnelalui hibridisasi, sekuensing, dan perancangan primer.
Metode yang digunakan dalam peneliian pendahuluan tersebut menurut
Zane e t a/. (2002) merupakan protokol konvensional. Lokus rnikrosatelii diisolasi dari sebagian pustaka genom (diseleksi ukuran insert kecil), skrining ribuan klon rnelalui hibridisasi koloni dengan probe yang mengandung repeat (Rasman eta/,
1991). Meskipun relatii sederhana, khususnya untuk genom yang kaya
mikrosatelii, pendekatan ini dapat membutuhkan waktu yang lama, biaya tinggi dan tidak efisien untuk spesies yang rnempunyai frekuensi mikrosatelii rendah. Beberapa strategi alternatii telah diemukan untuk mengurangi waktu yang
diperlukan dalam isolasi rnikrosatelit dan secara nyata dapat meningkatkan hasil
(Zane et. a/. 2002).
Zane et a/. (2002) menjelaskan pengkayaan yang dilakukan sebelum kloning dengan teknik PCR dapat meningkatkan keberhasilan dalam mengisolasi rnikrosatelii. Edwards et a/. (1996) rnengatakan penggunaan teknik pengkayaan
rnengandung motif rnikrosatelii rnencapai 50-70% dengan waktu yang relatii
singkat, dan tidak rnernbutuhkan biaya tinggi dibandingkan pendekatan konvensional.
Teknik enrichment yang dikernbangkan oleh Edwards et a/. (1996) telah terbukti berhasil rnengisolasi rnikrosatelii pada tanarnan tropis asli Indonesia
seperti rnangga (Mulyani 2003) dan durian (Kristianti 2005) di Laboratonurn Genetika Departernen Biologi Instiiut Teknologi Bandung. Dengan kin dari Dr.
Adi Pancoro (komunikasi pribadi) rnaka pada peneliiian ini akan rnenerapkan
teknik yang telah dikembangkan dan diadaptasikan pada kondisi Indonesia yang berprospek berhasil baik dan cepat.
Tujuan Penelitian
Tujuan penelitian ini adalah :
1) untuk rnengisolasi dan rnengkarakterisasi rnotii rnikrosatelii pada tanaman jati (Tectona grandis).
Jati
Tanaman jati (Tectona grandis Linn. f.) termasuk anggota famili Verbenaceae dan genus Tectona yang dibagi ke dalam tiia spesies. Genus Tectona mencakup spesies Tectona grandis Linn. f. yang dikenal di lndonesia dan dua spesies lain, yaitu T. hamiltoniana Wall. yang tumbuh di daerah kering Myanmar dan T. philippinensis Benth B1 Hooker yang tumbuh di hutan Batangas
dan Mindoro (Pulau [ling), Filipina. Dalam sistem taksonomi dan tatanama,
klasifikasi tanaman jati menurut ahli botani adalah sebagai berikut :
Divisi : Spermatophyta
Kelas : Angiospermae Sub kelas : Dicotyledoneae
Ordo : Verbenales Famili : Verbenaceae Genus : Tedona
Spesies : Teetonagrandis Linn. f.
Areal penyebaran alami tanaman jati terdapat di India, Myanmar, Thailand dan bagian baral Laos. Batas utara pada garis 25"LU di Myanmar,
batas selatan pada garis 9"LU di India. Jati tersebar pada garis 70"-100" BT. Jati bukan tanaman asli Indonesia telapi sudah tumbuh sejak beberapa abad
yang lalu di P. Kangean, Muna, Sumbawa dan Jawa (Rachrnawati ef. a/. 2002; Sumama 2001).
Di Indonesia, jati digolongkan sebagai kayu mewah termasuk kelas kayu dengan kualitas dan nilai jual yang tinggi. Kayu ini tahan rayap serta jamur
dengan angka kembang susut yang kecil. Salah satu kayu serbaguna, digunakan untuk konstruksi ringan dan berat, bahan bangunan rumah, kayu
pertukangan, ukiran dan lain-lain. Penggunaan kayu jati sudah sangat populer
dan masih merupakan pilihan utama karena keindahan seratnya.
Secara historis, nama Tectona berasal dari bahasa Portugis (teMon) artinya tumbuhan yang memiliki kualitas tinggi. Di India, tanaman jati memiliki
banyak nama daerah seperti : ching-jagu (di wilayah Asam); saigun, segun
(Sangskrit); fekkumaran, tekku (Tamil); dan adaviteeku, peddafekku,
feekuchekka (Telugu). Dalam bahasa Jerman jati dikenal dengan nama Teck,
Teakbaun, atau Java Teak, Teak (Inggris), Lyiu (Burma), Mai Sak (Thailand), Teck (Perancis), dan di Spanyol disebut Teca (Rachmawati ef. a/., 2002;
Sumarna. 2001).
Jati Unggul
Jati unggul merupakan jati yang memiliki sifat-sifat unggul yang dihasilkan dari pembiakan vegetatii melalui teknik mikropropagasi (kultur jaringan) dan
memiliki laju pertumbuhan yang cepat (fast growing). Jati konvensional atau jati biasa adalah tanaman jati yang dihasilkan dari bibi asal biji (generaw yang
memiliki laju pertumbuhan yang relatii larnbat. Tanaman induk yang diklon untuk menghasilkan jati unggul merupakan tanaman jati terbaik setelah diseleksi dan dipilih dari tanaman jati biasa yang mempunyai sifat-sifat "lebih" dari populasi jati
yang ada. Tanaman induk ini berasal dari jati hasil budidaya lokal (Indonesia)
atau jati hasil budidaya di luar Indonesia, seperti Malaysia, Myanmar, dan Thailand (Tini dan Amri 2002; PPA Agricola 2003; Saefulloh 2003).
Bibit jati hasil kultur jaringan memiliki kecepatan pertumbuhan
5-4
kalidari pertumbuhan bibit jati asal biji, pertumbuhan pohon lebih seragam (> 80 %), tinggi perlumbuhan per tahun lebih cepat (umur 2 tahun tinggi 4.75 m dan diameter batang 7.54 cm; Eliyani 2004), bentuk batang lebih lurus dan silindris,
serta tingkat percabangan ringan. Dengan umur panen 15-20 tahun pengembalian modal lebih cepat dengan kualitas kayu layak jual. Bibit jati hasil
teknologi kultur jaringan dapat lersedia dalam jumlah besar (ratusan ribu hingga jutaan bib6 per tahun) sehingga tanaman ini dapat dijadikan salah satu bidang
investasi yang prospektii di masa yang akan datang. Saaf ini, di Indonesia telah
beredar trade mark jati unggul seperti Jati Biotropika, Jati Emas, Jati Super, Jati Plus, Jati Prima, dan Jati Unggul (Tini dan Amri 2002; Saefulloh 2003). Jenis jati unggul yang digunakan dalam peneliian ini adalah Jati Biotropika (Jati Emas).
Penanda DNA (DIVA markerJ
Materi genetik (DNA) yang dijumpai dalam semua organisme sebagian
besar belum diketahui fungsinya, hanya sebagian kecil (10% dari total DNA
genom) yang diketahui beriungsi sebagai penyandi genetik yang menentukan
Segmen-segmen ini kemudian diketahui ada yang khas dalam suatu individu, jumlahnya sangat berlimpah, dan memiliki polimorfisme yang sangat tinggi sehingga dapat digunakan sebagai penciri DNA (DNA marker) dalam analisis
genom (Philips dan Vasil 2001).
Dalam banyak kasus, marker DNA sangat membantu dalam menentukan posisi gen karena marker DNA ini sering terletak pada kromosom yang sama dan
berdekatan dengan gen. Dengan demikian, semakin banyak marker DNA yang diketahui posisinya pada kromosom, maka semakin banyak pula kemungkinan
untuk mengetahui posisi gen. Marker DNA juga sangat bermanfaat dalarn studi
kekerabatan genetik dan evolusi serta aplikasi lainnya. Kenyataan ini membuat marker DNA ini menjadi sangat menentukan dalam perkembangan teknologi rekayasa DNA terutama dalam rangka pembuatan peta genetik (Philips dan Vasil
2001).
Mikrosalell
Salah satu penanda molekuler (DNA marker) yang sangat populer
dewasa ini adalah mikrosatelii. Mikrosatelit merupakan sekuen DNA sederhana pendek berulang, 1-6 pasang basa (pb) berunitan mono-, di-, tri-, atau tetra-
nukleotida berulang dalam bentuk kopi berdampingan (tandem), (CA/GT)n atau
(TCG1AGCT)n secara umum terdiri dari sekiiar 20-30 pb untai p l y GT, poly A dan poly GA (Hamada et al. 1982; Tautz and Renz 1984). lstilah mikrosatelii
yang sering digunakan oleh para penelii antara lain : short tandem repeat (STR)
(Craig et. a/. 1988); dan simple sequences repeats (SSRs) (Jacob ef. al. 1991). Mikrosatelit tersebar secara acak sepanjang genom inti pada individu tingkat tinggi (Hamada ef. al. 1982; Tautz dan Renz 1984; dan Tautz e t a/. 1986). Sekuen ini mempuyai tingkat polimorfik yang tinggi terhadap keragaman
dalam jumlah ulangan, dengan panjang <200 pb terletak disekeliling sentromer, atau berkelornpok di lokasi-lokasi heterokromatin seperti telomer (Weber dan
May 1989; Tautz 1989; dan Ellegren 1992).
Mikrosatelit dinukleotida pada mamalia memiliki jumlah ulangan antara 10 sampai kurang dari 30. Walaupun mikrosatelii yang sudah teridentikasi terdiri dari semua kombinasi nukleotida, kelompok yang sangat berlimpah dalam genom manusia terdiri dari (CA)n. Dalam genom manusia terdapat sekiar 50-
Keberhasilan mengamplifikasi mikrosatelii dengan PCR menggunakan sekuen pengapit mikrosateli sebagai primer, menjadikan lokus mikrosatelii
sebagai marker yang ideal terutama dalam analisis sidik jari DNA (DNA fingerprinting), pemetaan genom dan analisis keterpautan dalam semua spesies.
Sebuah peta dengan resolusi tinggi yang hampir semuanya terdiri dari marker
mikrosatelii sudah dikonstruksi pada tikus (Love et. a/. 1990) dan pada manusia (Guyer 1992).
Metode pengujian dan analisis lokus mikrosateli mempunyai beberapa
keuntungan dibandingkan dengan teknik molekuler lainnya. Seperti te rjadi pada
minisatelit DNA fingerprinting (pemetaan DNA), variasi muncul sebagai hasil dari variasi jumlah unit berulang. Akan ietapi, unit inti dalam mikrosatelii secara umum terdiri atas 1-6 pb panjangnya berbeda dengan karakteristik dari minisatelit yang terdiri atas 10-60 pb (Ashley dan Dow 1994). Oleh karena itu
analisis mikrosatelit lebih mempunyai keuntungan dibandingkan minisatela, berupa PCR DNA lebih baik dan lebih mudah dihibridisasi (southern bloth) dengan label pelacak. Hal ini disebabkan karena jauh lebih cepat dalam pengerjaanya, membutuhkan sedikit DNA, walaupun yang berkualias rendah.
Metode RAPD (Randomly amplified polymorphic DNA), juga
rnemanfaatkan PCR, tapi seperti DNA Fingerprinting kesulitan dalam menggambarkan perbandingan pita-pita dalam lintasan gel merupakan masalah
tersendiri. Mikrosatelit lebih tersebar secara merata pada genom dibandingkan minisatelit. Penyebaran ini tidak hanya meningkatkan kegunaannya untuk
konstruksi peta genetik tapi juga mengurangi kemungkinan masalah yang
berhubungan dengan pengujian lokus dalam studi populasi (William ef. al. 1990).
Aplikasi Mikrosatelit
Studi pendekatan molekuler untuk identiikasi terhadap perubahan-
perubahan genetik yang berhubungan dengan perubahan sifat kuantiitii dan
kualitatii pada tanaman lebih lambat perkembangannya dibandingkan pada hewan. Pemanfaatan analisis mikrosatelii untuk studi populasi alami dari tanaman tingkat tinggi belurn banyak dilakukan, studi awal pada tanaman hias
atau tanaman budidaya membuktikan bahwa mikrosatelii mempunyai marker
polimofik yang berlimpah untuk identiikasi atau pemetaan genom (Philips dan Vasil 2001). Ulangan dua pasang basa (CA/GT)n adalah tipe umum dari
sebesar 57% dari empat kemungkinan ulangan dinukleotida, sedangkan
(CG1GC)n hanya sebesar 0.7%.
Studi terakhir menunjukan bahwa (AT/TA)n dinukleotida bemlang secara
umum banyak terdapat pada tanaman tingkat tinggi yang diikuti oleh (GAKA)n
(Morgante dan Olivieri, 1993). Motif mikrosateli dinukleotida yang banyak diternukan pada tanaman mangga (Mangifera indica L.) adalah (GT)J(AG), dan
Qn I(ATGT). /(AT). /(GT), (Mulyani 2003). sedangkan hasil peneliian Kristianti
(2005) pada tanaman durian (Durio zibethinus Murr) banyak diiemukan motif
mikro~ate~t TJA,,, GJC,,, AGflG, dan GGAJCCT.. Effendi (2000) menjelaskan motii mikrosatelii yang ditemukan dalam tanaman jati didominasi oleh motii
mononukleotida (T)n dan dinukleotida (GA)n/(CT)n dan belum diiemukan adanya mot8 mikrosateli dinukleotida (AC) dan trinukleotida (GTG. AAC dan AAT).
Mikrosatelit dideteksi menggunakan PCR, dengan primer penghubung
pada sekuen unik DNA yang mengapit motif bemlang. Sekuen DNA yang menjadi primer penghubung rnuncul sekali saja dalam genom. Hal ini berarti bahwa jika suatu unit bemlang mikrosateli muncul pada banyak siklus yang
berbeda di dalam genom, maka PCR hanya akan mengamplifikasi satu situs
saja, yaitu situs yang mengapit sekuen yang berhubungan dengan sepasang sekuen primer. Pada waktu mendesain primer yang mengapit daerah sekuen sederhana untuk amplifikasi PCR, primer hams setidaknya terdiri atas 20 pasang basa untuk mencegah kesalahan pembacaan primer (tidak spesifik). Panjang
produk PCR benrariasi menunrt jumlah unit berulang pada situs tersebut. Seperti beberapa produk PCR yang diamplifikasi dari sekuen unik primer, hanya satu
atau dua pita yang dihasilkan dari satu tanaman, satu pita jika tanaman tersebut homozigot, dan dua pita jika heterozigol.
Selain banyak digunakan untuk pemetaan genom, rnikrosateli juga dapat
digunakan untuk analisis populasi. Analisis populasi dapat diiokuskan pada dua aspek yang berbeda yaitu analisis struktur genetik atau sosiobiologi. Analisis
mikrosateli dapat secara potensial diaplikasikan pada studi ekologi dan evolusi secara tidak terbatas. Dalam jangka panjang, mikrosatelit dapat menggantikan
isozim dalam genetika populasi karena polimorfisme yang tinggi. Tingkat
polimorfis yang tinggi yang diperoleh dalam suatu spesies tidak menjamin bahwa
mikrosatelit adalah keterbatasan primer untuk amplifikasi lokus spesifik (Ashley dan Dow 1994).
Penggunaan penanda DNA seperti mikrosatelit yang memiliki sifat
spesifik serta polirnorfisme yang sangat tinggi diperlukan untuk mencirikan genotipe tanaman jati yang unggul. Penanda mikrosateli adalah suatu cara
penting untuk mempelajari orisinilitas genotipe dari setiap populasi. Aplikasi
mikrosatelit selain berguna untuk rnenguji induk juga dapat menganalisis keragaman genetika, struktur populasi, sistem perkawinan dan pemetaan genetika. Bahkan dalam jangka panjang, mikrosatelii dapat menggantikan
isoenzim dalam mempelajari genetik populasi karena variabiliasnya lebih besar. Berdasarkan data tersebut, penanda mikrosateli diharapkan akan lebih
bermanfaat untuk menentukan variasi genetik pada beberapa klon tanaman jati. Oleh karena itu tahap pertama dalam penelitian ini adalah untuk mengidentiikasi dan mengisolasi lokus mikrosateli dari beberapa genom Tectona grandis yang
dapat digunakan untuk menganalisis variasi dan sbuktur genetik.
lsolasi Mikrosatelit
Teknik pernbuatan pustaka mikrosatelii menurut Zane et a/. (2002) dibagi menjadi dua strategi yaitu : a) Strategi konvensional, dengan harapan
mendapatkan sebanyak dan selengkap mungkin pustaka genom yang kemudian diseleksi kornponen DNA berulangnya; b) Strategi pengkayaan (enrichment) yaitu dibatasi hanya pada sekuen berulang target sehingga terbatas pustaka
genom yang diseleksi dan dikumpulkannya terutama hanya pada sekuen berulang target. Beberapa strategi alternatif telah ditemukan untuk mengurangi
waktu yang diperlukan dalam mengisolasi penanda mikrosateli dan secara nyata dapat meningkatkan hasil.
Masing-masing strategi yang dipakai merniliki kelebihan dan kekurangan
masing-masing berikut tahapan yang dikerjakannya. Metode konvensional, lokus mikrosateli diisolasi dari sebagian pustaka genom (diseleksi ukuran insert kecil),
skrining ribuan pustaka genomik melalui hibridisasi koloni dengan probe bermotii mikrosateli (Rasman et a/. 1991). Meskipun relatii sederhana dan pustaka genomnya lebih kaya khususnya untuk genom yang kaya mikrosatelit, metode ini
tidak efisien karena dari sekuensing koloni positif hasil skrining ribuan klon
yang mempunyai frekuensi mikrosatelit rendah. Metode enrichment, m e ~ p a k a n
metode lain yang lebih efisien karena rnetode pustaka genomnya telah dilakukan
pengkayaan (enrichment) sehingga targetnya lebih spesifik. Zane et a/. (2002) menjelaskan bahwa pengkayaan yang dilakukan sebelum kloning dengan teknik PCR dapat rneningkatkan keberhasilan dalam rnengisolasi mikrosatelit. Edwards et a/. (1996) mengatakan kemungkinan koloni rekombinan yang disekuensing
mengandung motif mikrosatelit mencapai 50-70% dengan waktu yang relati
singkat, dan tidak membutuhkan biaya tinggi dibandingkan pendekatan konvensional.
Tabel 1. Perbandingan biaya, waktu dan hasil dari metode isolasi mikrosatelit
Tahapan kerja metode konvensional adalah sebagai berikut : isolasi DNA genom, pernotongan DNA genom dengan enzim restriksi, seleksi fragmen hasil pemotongan (200-600 pb), ligasi fragmen berukuran ~ 6 0 0 pb ke dalam vektor
plasmid, transformasi plasmid rekombinan ke dalam Escherichia coli, purifikasi plasmid rekombinan, seleksi DNA rekombinan berisi motif mikrosatelit dengan cara hibridisasi, PCR, sekuensing dan desain primer mikrosatelit (Zane et a/.
2002).
Tahapan kerja rnetode pengkayaan (enrichment) adalah sebagai berikut :
pemotongan genom DNA dengan enzim restriksi blunt end; ligasi adaptor pada
kedua ujung fragrnen DNA; fragmen-fragmen yang mengandung mikrosatelit
diperkaya melalui hibridisasi ke membran nylon yang telah mengikat oligonukleotida bermotif mikrosatelit; fragmen-fragmen DNA yang terikat membran nylon kemudian diisolasi, diperkaya dan dikloning; transformasi ke dalam Escherichia colt isolasi dan purifikasi plasmid rekombinan; verifikasi
plasmid rekombinan dengan enzim restriksi atau amplifikasi PCR; analisis sekuensing dan perancangan primer (Edwards et a/. 1996; Zane eta/. 2002).
Kmvensional Pengkayaan
Surnber : Zane eta/. (2002) ~mtokol
(US Dollar) ZOO04000
1000-4000
Pustaka
(US Dollar) -400
4 0 0
1 bulan
1-2 minggu
Perancangan Primer
Perancangan primer (oligonukleotida) sangat menentukan bagi proses
polimerase chain reaction (PCR). Primer diperlukan sebagai titik awal proses sintesis DNA melalui teknik PCR. Similaritas dan ukuran primer terhadap DNA
sasaran harus diperhatikan agar diperoleh produk PCR yang dinginkan. Pada umumnya berukuran 12-24 basa dan biasanya digunakan sepasang primer yang terdiri primer forward dan reverse.
Primer universal (umum) dapat didesain dari bagian konservatif hasil
alignment dari beberapa sekuen yang diketahuildiinginkan (multiple sequence
alignment). Bagian konservatif ini merupakan bagian yang mempunyai kesamaan basa dengan panjang sekitar 12-24 basa dari beberapa sekuen yang dianalisis setelah disejajarkan. Sampai saat ini belum ada aturan yang menjamin
efektifitas suatu primer hasil perancangan, tapi beberapa syarat yang perlu diperhaiikan dalam melakukan desain primer telah dibuat. Syarat-syarat yang harus diperhatikan dalam melakukan desain primer yaitu : besar amplikon, ukuran primer, bebas dari pembentukan struktur sekunder (hairpin, dimerself-
dimer, dan cross dimer), temperature melting (titik leleh, Tm), dan komposisi
primer (Guevara-Garcia et a/. 1997; Dieffenbach dan Dveksler 1995; Old dan Primrose 1994).
Menurut Old dan Primrose (1994) primer yang baik digunakan berukuran 17-30 basa, ditentukan berdasarkan situs (urutan DNA) yang mengapit daerah target amplifikasi dan mempunyai nilai Tm sekitar 50-60°C. Nilai Tm yang terlalu
rendah mempengaruhi proses penempelan (annealing) primer pada DNA target saat proses PCR. Ukuran primer yang umum dipakai sekitar 20 basa.
Kestabilan internal dari primer ditentukan oleh perpasangan G+C dengan penyebaran basa terbaik ke arah ujung 5' dari primer (Guevara-Garcia et al.
1997). Lebih lanjut Old dan Primrose (1994) menjelaskan kandungan G+C
terbaik pada suatu primer sekitar 50%. Apabila jumlah G+C terlalu banyak pada ujung 3' peluang terjadinya struktur jepit rambut (hairpin) tinggi dan penurunan
stabilitas primer akan terjadi pada urutan 5 basa terakhir. Primer dengan kandungan G+C rendah masih dapat digunakan dengan cara menambah ukuran primer menjadi lebih panjang agar mempunyai Tm yang cukup tinggi. Hal
terpenting yang harus diperhatikan adalah mencegah terjadinya perpasangan sendiri (self-priming), dimer-duplex, dan struktur jepit rambut pada bagian akhir
Tujuan dari perancangan primer dalam penelitian ini adalah untuk menghasilkan suatu primer spesifik yang dapat digunakan untuk mengamplifikasi
fragmen unik bermoff mikrosatelit. Bagian yang menjadi kandidat situs primer
adalah bagian yang mengapit suatu motif mikrosatelit setelah situs penempelan adaptor. Beberapa alternatif situs primer forward dan reverse dapat ditentukan
dengan memperhatikan ukuran frgamen yang akan dihasilkan (ukuran produk
amplifikasi).
Perancangan primer untuk suatu keperluan tertentu dapat menggunakan urutan sekuen yang sudah tersedia di bank data DNA melalui akses internet.
Beberapa situs (website) yang menyediakan bank data DNA adalah GeneBank
www.ncbi.nlm.nih.qov
&
w.ebi.ac.uk d&bqm,
&at?s&s
.&.rlluq$ berhubungan (link). Guevara-Garcia et al. (1997) mengatakan beberapaprogram melalui akses internet yang dapat membantu dalam mendesain suatu
primer adalah Oligocalc, Primers, Primer Select (DNA Star) dan Cybergene. Primer umum biasa dirancang driri bagian konservatif hasil alignment dari beberapa sekuen yang terdapat dalam genebank dengan menggunakan program
ClusfaW
dari
vvvvuv.ebi.ac.uklclustahnrEuropean Bioinformatics
Institute(EBI)
untuk mengetahui tingkat similaritasnya. Desain primer dapat menggunakan
P C W ~
dad w.basic.nw.edu/biolools/~rimer3posisi forward dan reverse dalam suatu sekuen DNA panjang sesuai dengan kriteria yang diinginkan seperti ukuran primer (jumlah basa), kandungan G+C
dan besar Tm. Selain itu Primer3 dapat menentukan posi$i basa tempat awal primer (forward dan reverse), ukuran produk, dan beberapa kandidat primer lain
yang masih memungkinkan. Hasil program Primer3 mencakup jumlah (panjang) dan urutan basa kandidat dari suatu primer beserta posisi primer tersebut di
dalam sekuen DNA sampel tanpa memperhatikan apakah primer yang disintesis berada pada sekuen DNA target yang diinginkan. Hal ini dapat menimbulkan masalah apabila primer yang dibuat adalah primer spesifik, lebih baik sekuen dan
Perancangan primer spesifik secara prinsip sama dengan primer
universal, hanya yang lebih diperhatikan adalah bagian bukan konservatif. Secara kasat mata ditentukan kandidat primer forward dan reverse yang spesifik untuk spesies yang kita inginkan. Diharapkan hasil amplifikasi positif hanya terjadi pada sampel spesies tersebut.
Pengujian kualitas primer dapat menggunakan program Primer Design
/hnaflb
&id
wuw.c~beraene.se. Daffd KpLd& m b W
suatu primer (forward dan reverse) yang sudah dipilih baik yang ditentukan secare manual (kasat rnata) rnaupun hasil analisis Primer3. Output Cybergene
METODE PENELlTlAN
Tempat dan Waktu
Penelitian ini dilaksanakan di Laboratorium Biologi Molekuler Pusat Studi
Hayati Pusat Antar Universitas (PAU) IPB Bogor, Laboratorium Genetika Departemen Biologi lnstitut Teknologi Bandung, dan Laboratorium Biologi Molekuler BB BIOGEN Cimanggu Bogor. Waktu penelitian mulai dari Januari
2004 sampai Desember 2005.
Bahan dan Alat
Bahan
Daun jati yang digunakan sebagai bahan penelitian merupakan kultur in
vitro berumur 2 MST (Minggu Setelah Tanam), terdiri dari tanaman klon Jati Biotropika (Emas) produksi PT PPA Agricola SEAMEO BIOTROP dan klon jati
lokal yang diperoleh dari hutan jati milik Perum Perhutani di perbatasan Kabupaten Sumedang dan Kabupaten Indramayu, Jawa Barat.
Bahan untuk media tumbuh in vitro adalah media dasar MS (Murashige
and Skoog 1962), vitamin dan zat pengatur tumbuh tanaman (ZPT). Selain itu
menggunakan bahan-bahan untuk isolasi dan ekstraksi DNA, pemotongan DNA genom, ligasi adaptor, Hibridisasi, kloning vektor, transformasi, isolasi plasmid,
PCR, elektroforesis dan bahan pendukung lainnya.
Alat
Alat yang digunakan antara lain untuk mikropropagasilkultur jaringan, isolasi dan ekstraksi DNA, pemotongan DNA genorn, ligasi adaptor, Hibridisasi,
kloning vektor, transformasi, isolasi plasmid, PCR, elektroforesis dan alat
pendukung lainnya.
Metode Pelaksanaan
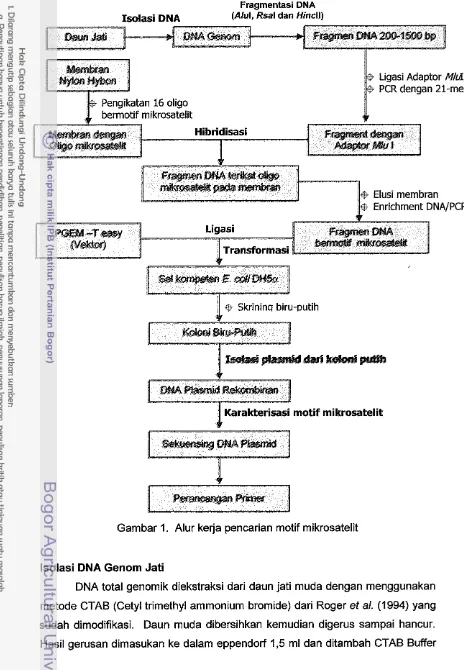
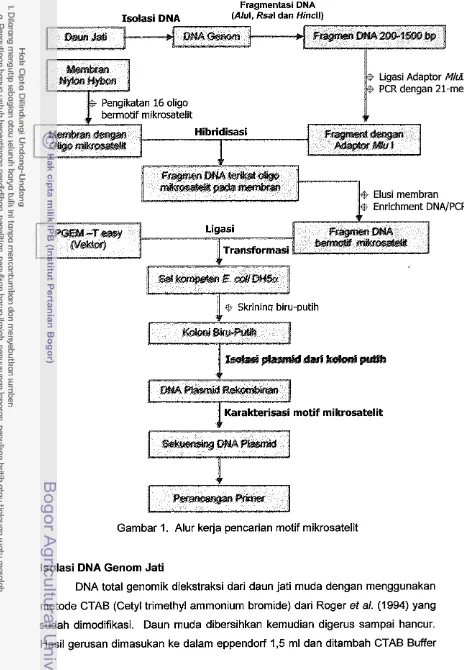
Penelitian isolasi dan karakterisasi mikrosatelit pada tanaman jati
dilakukan melalui beberapa tahap metode kerja (Gambar 1) mengikuti modifikasi Edwards et a/. (1996), yaitu : isolasi DNA genom jati, pemotongan DNA genom
Fragmentasi DNA
Isolasi DNA (Alul, Rsal dan Hindl)
DNA G e m Fragmen W 200-1 500 bp
I
Pengikatan 16 oligo krmotif mikrosatelit
Ligasi Adaptor Mlld PCR dengan 21-mer
$ Elusi membran
cD Enrichment DNAIPCR
li31Skrininq biru-~utih
iKarakterisasi motif mikrosatelit
Gambar 1. Alur kerja pencarian motif mikrosatelit
lsolasi DNA Genom Jati
DNA total genomik diekstraksi dari daun jati muda dengan menggunakan metode CTAB (Cetyl trimethyl ammonium bromide) dari Roger et a/. (1994) yang
[image:31.532.37.501.56.726.2]secukupnya kemudian diinkubasi pada 65OC selama 3 jam. Larutan ditambah Phenol dengan perbandingan 1:1, dikocok *20 menit. Campuran disentrifugasi
pada 13.000 rpm selama 3 menit. Supernatan dipindahkan ke tabung eppendorf baru, ditambahkan ClAA (Clorofom /so Amil Alkohol) kemudian dikocok i 2 0
menit. Sentrifugasi pada 13.000 rpm selama 3 menit. Fase bawah dipindahkan
ke ependorf baru, ditambahkan Sodium buffer kemudian diinkubasi pada 65 OC, selama 15 menit.
Tambahkan Sodium/Potassium asetat, kocok 5 menit. Selanjutnya ditambah Ethanol absolut dingin 2 x volume, simpan di freezer 30 menit, sentriiuse pada 13.000 rpm selama 3 menit. Ethanol absolut dibuang, kemudian
diganti dengan Ethanol 70%, sentrifuse pada 13.000 rpm selama 3 menit. DNA (endapan putih) dikering udarakan dan dilarutkan kembali dengan buffer TE.
Untuk mengetahui konsentrasi dan kemurnian DNA genom jati hasil isolasi, dilakukan analisa kuantitatii dengan menggunakan spektofotometri UV
pada panjang gelombang 260 nm dan 280 nm. Sedangkan untuk analisa kualitatif sampel DNA genom dielektroforesis dengan 1,2% gel agarose, diwarnai
dengan etidium bromida dan difoto dengan film polaroid atau disimpan dalam bentuk gambar.
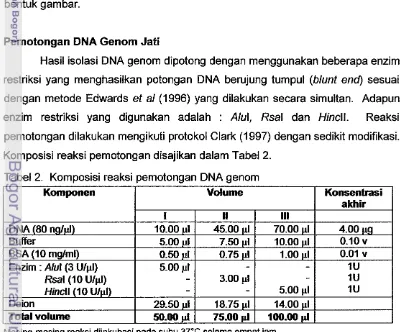
Pernotongan DNA Genorn Jati
Hasil isolasi DNA genom dipotong dengan menggunakan beberapa enzim restriksi yang menghasilkan potongan DNA berujung tumpul (blunt end) sesuai dengan metode Edwards et a1 (1996) yang dilakukan secara simultan. Adapun enzim restriksi yang digunakan adalah : Alul, Rsal dan Hincll. Reaksi
pemotongan dilakukan mengikuti protokol Clark (1997) dengan sedikit modifikasi.
Komposisi reaksi pemotongan disajikan dalam Tabel 2.
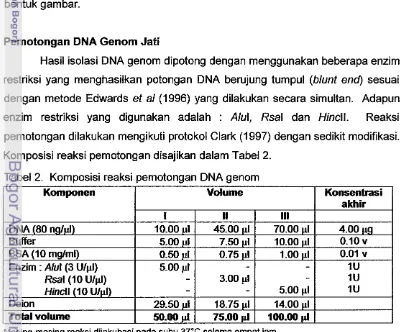
Tabel 2. Komoosisi reaksi ~emotonaan DNA aenom
[image:32.532.72.474.377.709.2]DNA genom jati mula-mula dipotong dengan enzim Alul kemudian dipotong lagi dengan enzim Rsal dan terakhir oleh Hincll. Untuk masing-masing
reaksi diinkubasi pada suhu
37°C
selama satu jam. Hasil pemotongan ketiga enzim restriksi dapat diketahui melalui elektroforesis dalam 1,2% agarose gel.Ligasi Fragmen DNA dengan Adaptor
Mengikuti protokol Edwards et a1 (1996) dengan komposisi reaksi disajikan dalam Tabel
3,
proses ligasi dilakukan pada suhu4°C
selamasemalam. Fragmen DNA hasil pemotongan enzim restriksi diligasikan dengan adaptor Mlul21-mer dan 25-mer (Produk GibcoBRL) pada sisi kanan dan kirinya.
Adaptor 21-mer (21 pb) dan 25-mer (25 pb) adalah urutan yang komplemen, dimana adaptor 25-mer mempunyai satu gugus fosfat pada ujung 5'. Pasangan
antara adaptor 21-mer dan 25-mer disajikan dalam gambar 2 berikut ini :
Tabel 3. Komposisi reaksi ligasi DNA dengan adaptor
KmpMlen
DNA 2 l m e r 25mer
I
ox
'butfef l i i -10 mM ATP
T4 DNA ligase (1 UIpI)
Keberhasilan reaksi ligasi dapat diuji dengan cara amplifikasi
menggunakan Polymerase Chain Reaction (PCR) dengan adaptor 21-mer digunakan sebagai primer. Komposisi
PCR
untuk mengetahui keberhasilanreaksi ligasi disajikan dalam Tabel
4.
Volume 1.00 pl 0.50 pl l . 0 0 ~ I 5.00 pl
5.00 111
1.00p1 1 3 U
Total volume
Gambar 2. Perpasangan adaptor 21-mer dan 25-mer
Konsentrasi akhir 200 ng
-
400 pg 2 pglpl11@d 5 pl 5 111
Air deion
1
36~50 ul1
50.00
lJ
Tabel 4. Komposisi reaksi PCR DNA terligasi adaptor
Hibridisasi
Hibridisasi merupakan pengikatan antara DNA hasil amplifikasi yang telah didenaturasi dengan membran Nylon Hybon yang telah mengandung oligonukleotida bermotif mikrosatelit. Tahapan hibridisisasi meliputi persiapan
membran, proses hibridisasi dan pengdyaan DNA bermotif mikrosdtelit melalui
amplifikasi DNA hasil elusi dengan PCR.
Persiapan membran
Sebanyak 16 macam oligonukleotida bermotif mikrosatelit produk Operon Technologies Inc., digunakan untuk mbmperkaya fragmen DNA yang diduga
mengandung mikrosatelit, yaitu : [T]zs, [ATII~, [ACI15, [AGI~s, [AATIIo, [AACIlo,
[AAGlio, [CTAlio, [TAGIio, [AGClio, [GCTlio, [GTGIio, [GGAIio, [GCClio,
W V i o
dan [AAATllo. Keenambelas oligonukleotida tersebut dibagi menjadi 3 kelompokmembran mengikuti Kusumawaty (1999) berdasarkan Melting Temperature (T,)
dari masing-masing oligonukleotida yang dihitung menggunakan rumus
Sambrook ef a/. (1989) :
T, = 81.5 + 16.6 10g [Na']
+
0.41 (%G+C)-
600111Untuk tiap kelompok membran, 10 pl dari masing-masing oligonukleotida dicampur kemudian ditambahkan 5xSSC (45 mM Natrium Sitrat pH 7,O dan 450
mM NaCI) hingga volume akhir 1 ml. Campuran oligonukleotida diteteskan ke membran Nylon Hybond N' berukuran 0,5 cm2, dibiarkan hingga meresap dan
kering baru dilakukan penetesan berikutnya sampai habis.
Membran dikering anginkan dalam suhu kamar sekitar 1 jam dan
dikeringkan dalam oven 65°C selama satu jam. Kemudian membran didedahkan pada
UV
crosslinker (Bio-Link) selama 15 detik dengan panjang gelombang 254nm energi 0,070 Joule dengan harapan oligonukleotida terikat kuat pada
membran, dilakukan pencucian 2 kali dengan Buffer Hibridisasi (50% formamida,
3 x SSC, 25 mM Na-fosfat pH 7, 2.5% SDS) pada suhu 45°C selama 2 hail dalam oven hibridisasi (ganti buffer hibridisasi setiap 24 jam). Kemudian
membran dicuci dengan 1 x SSC selama 3 jam pada suhu 45°C dan dibiarkan kering pada suhu ruang. Membran disirnpan dalam cawan petri steril pada suhu
-20°C.
Proses Hibridisasi
Proses hibridisasi mengikuti protokol Edwards et a/. (1996), sampel DNA hasil ligasi didenaturasi dengan cara memasukkan tabung eppendorf berisi
sample DNA ke dalam air mendidih selama 10 menit kemudian dituangkan ke
dalam tabung berisi 500 p1 buffer hibridisasi (3 x SSC, 25 mM Na-fosfat pH 7,
2.5% SDS) yang mengandung 1 pg primer 21-mer, dan membran yang telah mengandung motif mikrosatelit. Hibridisasi dilakukan per kelompok membran
dengan suhu hibridisasi berdasarkan rata-rata Tm, yaitu membran I dilakukan pada suhu 40°C, membran II pada suhu 55°C dan membran Ill pada suhu 72"C,
masing-masing selama 48 jam dengan menggunakan "Shake 'n Stack" oven hibridisasi. Setiap kelompok membran dicuci di dalam oven hibridisasi dengan suhu pencucian membran I 50"C, membran II 65°C dan membran Ill 82°C
sebanyak 5x (5 menit untuk setiap l x pencucian) dalam larutan 2xSSC, 0,01%
SDS dan 3x (5 menit untuk setiap l x pencucian) dalam larutan 0,5x SSC, 0,01% SDS.
Amplifikasi DNA elusi
DNA yang telah terikat pada rnembran kemudian dielusi dengan cara
merendam membran dalam 200 p1 air de-ion mendidih selama 10 menit. DNA
hasil elusi diamplifikasi (diperkaya) dengan menggunakan teknik PCR sebanyak 35 siklus dan menggunakan primer 21-mer dengan kondisi PCR sebagai berikut :
denaturasi 94°C selama 30 detik, annealing 59°C selama 1 menit, dan elongasi
72°C selama 2 menit.
Komposisi reaksi PCR disajikan dalam Tabel 5. Produk PCR dielektroforesis menggunakan 1,4% agarosa gel dalam 0,5xTBE. Pewarnaan gel
dilakukan dengan perendaman dalam ethidium bromide 2 pg1100 ml selama 15
menit kemudian gel dicuci dengan deion selama 10 menit. Gel diamati dengan
Tabel 5. Komposisi reaksi PCR DNA hasil elusi (enrichment)
Ligasi dengan Vektor Plasmid
Reaksi ligasi DNA produk PCR dilakukan mengikuti petunjuk penggunaan
kit vektor
GEM"
-
T Easy dari Promega. Proses ligasi dilakukan pada suhu 4°C selama semalam dengan komposisi reaksi ligasi disajikan dalam Tabel 6dibawah ini.
Tabel 6. Komposisi reaksi ligasi DNA ke dalam vektor
GEM"
-
T EasyTransforrnasi
Pembuatan sel kompeten E.Coli DH5-a
Pembuatan sel kompeten mengikuti protokol lnoue et a/. (1990) dengan sedikit modifikasi. Suhu m e ~ p a k a n faktor yang harus diperhatikan dalam
pembuatan sel kompeten E.Coli DH5-a, suhu harus dijaga selalu dalam kondisi
dingin selama proses perlakuan kimiawi dan dalam kondisi aseptik.
Bakteri E.Coli D H 5 a dari stok gliserol diambil 1 ose dan diinokulasi ke
dalam media SOB padat (2% Tryptone, 0,5% yeast extract, 1% (vlv) 1 M NaCI, 0,25% (vlv) 1 M KCI, 1% (vlv) 2 M MgCI,, 1,5% agar), diinkubasi pada suhu 37°C
semalam. Satu koloni tunggal E.Coli umur 16-18 jam diambil dan diinokulasi ke dalam 5-6 ml medium SOB cair pada tabung falcon 50 ml (kultur cair skala kecil)
[image:36.532.53.461.29.780.2]Dari kultur cair skala kecil, diarnbii suspensi sel sebanyak 2 rnl dan diinokulasi ke dalam 150 rnl medium SOB cair pada labu erlenrneyer 500 ml
untuk dibuat kultur cair skala besar. Diinkubasi pada suhu 18°C-22°C dishaker pada kecepatan 120 rprn dan diukur ODnya sampai diperoleh ODBoo = -0,45.
Suspensi sel diletakan di es selarna 1 jam, rnasukan t 30 ml ke dalarn tabung
Sorval, diinkubasi di es selarna 10 rnenit disentrifugasi 3.500 rprn 4°C selama 15 rnenit. Supernatan dibuang, pelet diresuspensi dalarn es dengan 16 rnl buffer TP
dingin (10 rnM PIPES, 15 mM CaCIZ.2H,0, 250 rnM KC1 diukur hingga pH 6.7
tarnbahkan 55 rnM IWnCIZ) dengan cara dijentik-jentik atau divortek beberapa detik, diinkubasi dalarn es selarna 10 menit, kemudian disentrifugasi 3.500 rpm
pada 4°C selarna 15 menit. Supernatan dibuang, pelet diresuspensi dalam es dengan 4 rnl buffer TP dingin kemudian tarnbahkan 7% DMSO (280 pl) dikocok
perlahan atau divortek beberapa detik. Suspensi sel diinkubasi dalam es selarna 10 rnenit dan siap digunakan sebagai sel kornpeten untuk proses transformasi.
Sisa suspensi sel dialiquot ke dalam rnikrotube 1,5 ml (@ 100 pl) rnasukan
secara cepat ke dalarn nitrogen cair dan disirnpan pada suhu -80°C.
Transformasi
Metode trdnsforrnasi yang digunAkAn adalah rnetode kejut panas (Heat Shock) mengikuti protokol lnoue ef a/. (1990) yang telah mengalami sedikit
rnodifikasi. Sebanyak 5 p1 DNA hasil ligasi (plasmid rekornbinan) dirnakukkan
dalam tabung Falcon 15 ml (polipropilen) dingin kernudian dicarnpur dengan 50-
100 p1 sel kom(3den. Tabung doentik-jentik secara perlatian agar bercdrnpur,
diinkubasi dalarn es selarna 20 rnenit.
Tabung di heat shock dengan cara dirnasukan ke dalam inkubator suhu 42°C selarna 50 detik dan secara cepat tabung dipindahkan ke dalam es selarna
2 rnenit. Sebagai media perturnbuhan transforman ditambahkan 950 p1 SOC
(2% Tryptone, 0,5% yeast extract, 1% (vlv) 1 M NaCI, 0,25% (vlv) 1 M KCI, 1% (vlv) 2 M MgClz, 1% (vlv) 2 M glukosa), diinkubasi pada suhu 37°C di dengan pengocokan 200 rprn selarna 1-3 jam.
Kultur sel transforman sebanyak 50-100 pi ditanarn pada medium LB
padat yang telah mengandung 100 pglml arnpisilin, 100 pI IPTG (100 rnM
Isopropythio-p-D-galactoside) dan 20 pl X-gal (50 rnglrnl), kultur diratakan
lsolasi Plasmid
lsolasi plasrnid dilakukan berdasarkan rnetode lisis alkali (Sarnbrook et
a/., 1989) dengan sedikii rnodifikasi. Koloni putih tunggal yang berasal dari kultur
transforman diiurnbuhkan dalarn 5 rnl media selektiif Tenifc Broth (1,2% tryptone, 2,4% yeast extract, 0,4% glycerol, 0,23% KH2P04, 1,25% K2HP04) yang
rnengandung 100 pglrnl arnpisilin, diinkubasi sernalarn pada suhu 37OC dengan
pengocokan 250 rpm.
Panen sel kultur bakteri dengan cara sentrifugasi pada kecepatan 14.000
rprn selarna 20 detik. Pelet yang terbentuk dilarutkan dalarn 200 p1 GTE (50 rnM
glukosa, 25 rnM Tris-CI pH 8,0, 10 rnM EDTA pH 8,O) dan dihornogenkan dengan
vorteks. Suspensi sel ditarnbahkan dengan 400 p1 buffer lisis (0,2 M NaOH, 1%
SDS), tabung dibolak-balik beberapa kali. Kernudian ditarnbahkan 150 pl 5M
Potasiurn asetat, sentrifugasi dengan kecepatan 13.000 rprn selarna 2 menit. Supernatan dipindah ke tabung baru kernudian DNA plasmid diendapkan dengan penarnbahan ethanol absolut dingin 2x volume, tabung dibolak-balik dan
dipresipitasi selarna 30 rnenit pada suhu -80°C. Selanjutnya disentrifugasi dengan kecepatan 13.000 rprn selama 2 rnenit, pelet yang terbentuk dicuci dengan ethanol 70% dingin dan dikeringkan dengan vacum. Pelet dilarutkan
kernbali dengan TE buffer yang rnengandung RNAse 10 pglrnl (Sarnbrook et a/.,
1989) dan disirnpan pada suhu -20°C.
Untuk rnengetahui kepastian bahwa fragrnen DNA insert hasil transforrnasi telah tersisip dalarn plasrnid maka dilakukan verifikasi dengan
rnelakukan pernotongan DNA rekornbinan atau hasil transforman menggunakan enzirn restriksi EcoRI.
Sekuensing
Analisis sekuensing dilakukan di PT. Charoen Pokphand Indonesia
Jakarta menggunakan ABI PRISM 3100 XL Sequenser. Sampel DNA plasrnid yang akan disekuensing disarankan rnempunyai nilai kernurnian berkisar 1,s
dengan konsentrasi 100 nglpl. Sekuensing dilakukan satu arah dengan
rnenggunakan primer T7. Sedangkan untuk produk PCR rnenggunakan primer 21-mer.
Perancangan Primer
Urutan sekuen hasil sekuensing dibuat dalarn format fasta untuk dianalisis lebih lanjut menggunakan program CIustalW dari
vvvvvv.ebi.ac.uWclustaIvv
@w
~.basic.nwu.edulbiotoolsl~rimer3 mendesain primer, dan &&@
&&
www.cvberaene.se menganalisis primer yang sudah dibuat. Sernua program analisis tersebutdiakses melalui internet.
Desain primer menggunakan Program Primer3 untuk menentukan posisi forward dan reverse dengan mernasukan kriteria yang diinginkan seperti ukuran
primer (jurnlah basa), kandungan G+C dan besar Tm. Hasil program Primer3 rnencakup jumlah (panjang) dan urutan basa kandidat dati suatu primer beserta posisi primer tenebut di dalam sekuen DNA sarnpel, posisi basa tempat awal
primer (forward dan reverse), ukuran produk, dan beberapa kandidat primer lain.
Pada perancangan primer spesifik ini apabila hasil Primer3 belum cukup baik rnaka penentuan posisi dan sekuen dari primer forward dan reverse dilakukan secara kasat rnata (manual).
Pengujian kualitas primer rnenggunakan program Primer Design Analysis
w.cvberqene.se.
E&&
-58
t;?atrrirM(fonvard dan reverse) yang sudah dipilih baik yang ditentukan secara manual
rnaupun hasil analisis Primer3. Output Cybergene meliputi ada tidaknya struktur
sekunder (haipin dan palindrom), jumlah basa primer, annealing dan melting temperature, serta rasio GIC.
Sekuen primer dari daerah pengapit motif mikrosatelit secara umurn
dirancang untuk rnenghasilkan primer yang panjangnya 18-20 nukleotida, dengan kandungan GC mendekati 50% dan T, sekitar 60°C yang dapat
digunakan untuk mengamplifikasi fragrnen unik bermotif mikrosatelit pada
HMIL
DAN
PEMBAHASANlsolasi DNA genom jati
Teknik isolasi dan karakterisasi mikrosatelit yang digunakan dalam
pene'tian ini mengacu pada teknik pengkayaan (enrichment) yang dikembangkan oleh Edwards et a/. (1996). Untuk mendapatkan hasil yang baik pada isolasi marka mikrosatelit diperlukan DNA genom yang berkualis baik
dengan kemurnian yang tinggi.
Pada penelitian ini prosedur yang digunakan untuk isolasi DNA adalah
prosedur berdasarkan cetyltrimethylammoniumbromide (CTAB). Hasil elektroforesis pada agarose 1,2% memperlihatkan bahwa hasil isolasi DNA
genom jati memiliki kemurnian yang cukup tinggi, karena tidak terlihat adanya "smeaf (Gambar 3). Data tersebut diperkuat oleh hasil pengukuran secara
kualitatii dengan speldrofotometer yang menunjukkan nilai rasio mendekati rasio murni untuk sampel DNA yaitu 1,8
-
2,O (Aquadro et a1 1992). Konsentrasi DNAyang diperoleh bervariasi antara 14.0
-
292.5 pgml dengan rasio antara 1,625-
1,992 (Absorban ?260/?280).
Gambar 3. Hasil isolasi DNA genom jati
Metode ekstraksi DNA tersebut juga telah berhasil untuk mengisolasi DNA durian (Kristianti 2005) dengan rasio rata-rata 1,778 dan DNA mangga
(Mulyani 2003) dengan rata-rata rasio 1,854
Pernolongan DNA genom jati
Pemotongan DNA genom dilakukan dengan menggunakan enzim restriksi
yang menghasilkan potongan DNA berujung tumpul (blunt end) sesuai metode Edwards et a1 (1996) yang dilakukan secara simultan, bertujuan untuk
memudahkan penempelan adaptor IMlul. DNA genom jati dipotong dengan tiga
enzim restriksi, mula-mula dipotong dengan enzim Alul, hasil pemotongan kemudian dipotong lagi dengan enzim Rsal dan terakhir hasil pemotongan Alul dan Rsal dipotong oleh Hindl. Tujuan utama pemotongan DNA genom ini
Zane et al. (2002) menjelaskan pemotongan DNA genom dengan banyak enzim restriksi dapat meningkatkan peluang mendapatkan motif-motii mikrosatelit dan mengurangi fragrnen-fragmen sisipan yang sama. Hasil pemotongan ketiga
enzim restriksi dapat diketahui melalui elektroforesis dalam 1,2% agarose gel.
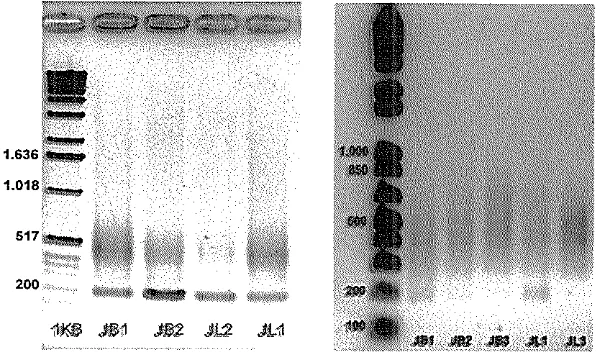
DNA genom jati yang dipotong dengan tiga enzim restriksi blunt end menghasilkan pita smear, ha1 ini menunjukan adanya variasi fragmen DNA
dengan ukuran c1.500 pb (Gambar 4). Kondisi smear yang dihasilkan
merupakan kumpulan fragrnen-fragmen DNA dengan ukuran yang berbeda-beda
hasil pemotongan tiga enzim restriksi tersebut ha1 ini disebabkan Multiple Cloning Site (titik pemotongan) tersebar pada genom DNA jati.
Pemotongan genom dengan menggunakan tiga enzim restriksi
menghasilkan variasi fragmen berukuran lebih kecil 200-1500 pb (Sumur 2) dibandingkan yang dipotong dengan dua enzim restriksi 200-2.000 pb (Sumur 3)
sehingga tidak dilakukan pernotongan fragmen DNA berukuran tertentu dari gel. Ukuran fragmen tersebut sudah cukup baik untuk proses skrining fragrnen DNA yang mengandung motif mikrosatelit. Menurut Liu & Wu (1998), Kijas et a1
(1994) dan lsagi & Suhandono (1997) fragmen DNA yang ideal untuk skrining mikrosatelit berukuran 200
-
1.500 pb.Keterangan:
M. Marker DNA 100 ob
1. Genom d~potong bilui, Rsal dan HlnCli (JB)' 2. Genom diootono Alui. Rsal dan H1nC11 IJL)'
3. Genom dkoton; ~ l u l ; dan Rsal (JB)
4. PCR hasil ligasi adaptor (JB)
5. PCR hasil ligasi adaptor (JL)
6. PCR hasil ligasi adaptor (JB)
*) JB = Jati Biotropika *) JL = Jati Lokal Perhutani
Dalam isolasi mikrosatelit apabila hasil pemotongan enzim restriksi masih menghasilkan fragmen-fragmen berukuran besar maka harus dilakukan isolasi
frgamen ukuran kecil dari gel elektroforesis. lsagi dan Suhandono (1997) mengisolasi fragmen ukuran 400-600 pb, Kijas et al. (1994) 300-1500 pb dan Liu
& Wu (1 998) 200-500 pb.
Ligasi fragmen DNA dengan adaptor
Produk PCR dari ligasi fragrnen DNA dengan adaptor Mlul pada
elektroforesis 1,2% agarose gel ditampilkan dalam Gambar 4 (sumur 4-6). Fragrnen-fragmen DNA yang terligasi adaptor berada pada ukuran sekitar 200- 1.500 pb. Hal ini menunjukan bahwa metode pengkayaan untuk isolasi
mikrosatelit lebih efisien karena fragrnen DNA target telah didapat. Dalam proses kloning, ukuran fragmen DNA yang akan disisipkan pada vektor plasmid
sangat berpengaruh terhadap efisiensi kloning dan sekuensing.
Hibridisasi
Proses hibridisasi mengacu pada protokol Edwards et a1 (1996) dengan sedikit modifikasi yaitu dengan cara membagi keenambelas oligonukleotida bermotif mikrosatelit menjadi tiga kelompok membran berdasarkan kondisi T,.
Hasil pengelompokan oligonukleotida tersebut disajikan dalam Tabel 8 berikut.
Tabel 8. Pengelompokan oligonukleotida berdasarkan Melting Temperature
Selanjutnya dilakukan elusi dari hasil hibridisasi. Elusi DNA dari membran
hibridisasi dimaksudkan untuk melepaskan fragmen-fragmen DNA yang terikat
kuat ke membran. Proses elusi ini dilakukan dengan merendam membran hasil hibridisasi dalam air mendidih selama 10 menit. Pada kondisi suhu tinggi diharapkan DNA sample yang menernpel pada membran dapat lepas. Untuk
kemudian dilakukan pengayaan DNA sampel yang dapat diduga sudah
Gambar 5. Pengayaan kandidat-kandidat fragmen DNA berrnotif
Arnplifikasi ini dilakukan untuk rnemperbanyak kandidat-kandidat fragmen
bermotif mikrosatelit hasil hibridisasi. Gambar 5 rnemperlihatkan bahwa proses hibridisasi telah berhasil mendapatkan panjang fragmen berkisar antara 200-
1.000an pb berasal dari ketiga kelompok rnembran (16 probe). Hal ini dapat
rnenunjukan pada urutan genorn DNA jati terdapat motif-motif rnikrosatelit seperti yang terdapat dalam ketiga kelompok mernbran yang digunakan.
Ligasi dan Transformasi
Produk PCR kemudian disisipkan ke vektor plasrnid pGEM-T Easy (produk Promega) untuk dijadikan pustaka rnikrosatelit (6 kelompok). Pustaka mikrosatelit tanarnan jati yang diperoleh dari penelitian ini adalah JB-I, JB-2, JB-
3, JL-1, JL-2, dan JL-3 (3 rnembran x 2 jenis jati). Reaksi ligasi DNA hasil PCR
dilakukan rnengikuti petunjuk penggunaan Kit vektor
GEM@
-
T Easy dari Prornega. Proses ligasi dilakukan pada suhu 4°C selama semalarn dengankomposisi reaksi ligasi disajikan dalarn Tabel 6 (lihat bahan dan rnetode).
Vektor yang telah membawa pustaka rnikrosatelit tanarnan jati (6
kelornpok) selanjutnya ditransforrnasi ke bakteri E. coli DH5a dalam media
selektif rnengandung 100 pglrnl ampisilin, 100 p1 IPTG (100 mM lsopropythio-P-
D-galactoside) dan 20 p1 X-gal (50 mglml). Metode transforrnasi yang digunakan adalah metode kejut panas (heat shock) rnengikuti protokol lnoue et a/. (1990)
yang telah rnengalami sedikit modifikasi (lihat bahan dan metode). Seleksi transforman dilakukan dengan skrining biru-putih, koloni berwama putih
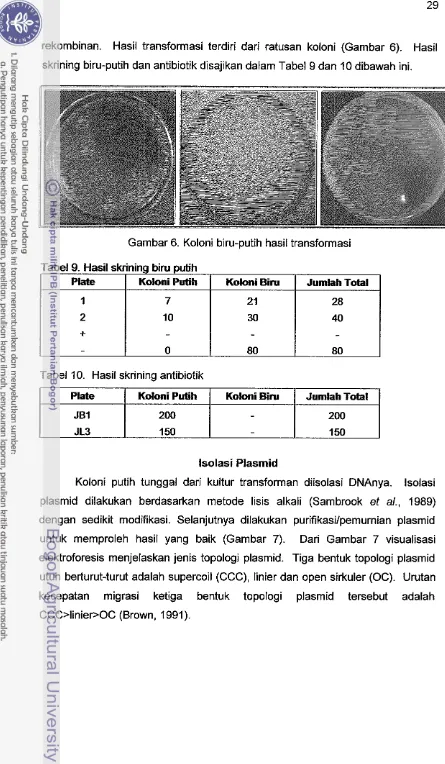
[image:43.532.118.421.56.235.2]rekombinan. Hasil transformasi terdiri dari ratusan koloni (Gambar 6). Hasil skrining biru-putih dan antibiotik disajikan dalarn Tabel 9 dan 10 dibawah ini.
Garnbar 6. Koloni biru-putih hasil transformasi
Tabel 10. Hasil skrining antibiotik
lsolasi Plasmid
Koloni putih tunggal dari kultur transforman diisolasi DNAnya. lsolasi
Plate
JB1 JL3
plasmid dilakukan berdasarkan rnetode lisis alkali (Sambrook et a/., 1989) dengan sedikit modifikasi. Selanjutnya dilakukan purifikasilpemurnian plasmid untuk rnemproleh hasil yang baik (Gambar 7). Dari Gambar 7 visualisasi
K o h i Putih
200 150
elektroforesis menjelaskan jenis topologi plasmid. Tiga bentuk topologi plasmid
utuh berturut-turut adalah supercoil (CCC), linier dan open sirkuler (OC). Urutan
kecepatan migrasi ketiga bentuk topologi plasmid tersebut adalah CCC>linier>OC (Brown, 1991).
Koloni Bim
-
-
Jumlah Total
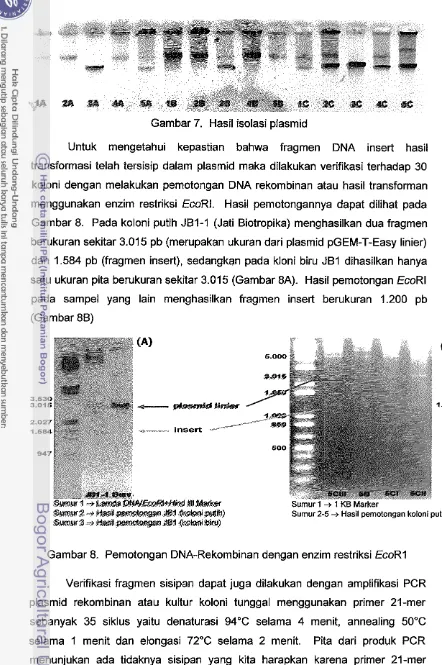
[image:44.532.34.479.9.773.2]Gambar 7. Hasil isolasi plasmid
Untuk mengetahui kepastian bahwa fragmen DNA insert hasil transformasi telah tersisip dalam plasmid maka dilakukan verifikasi terhadap 30
koloni dengan melakukan pemotongan DNA rekombinan atau hasil transforman menggunakan enzim restriksi EcoRI. Hasil pemotongannya dapat dilihat pada Gambar 8. Pada koloni putih JBI-1 (Jati Biotropika) menghasilkan dua fragmen
berukuran sekitar 3.015 pb (merupakan ukuran dari plasmid pGEM-T-Easy linier) dan 1.584 pb (fragmen insert), sedangkan pada kloni biru JB1 dihasilkan hanya
satu ukuran pita berukuran sekitar 3.015 (Gambar 8A). Hasil pemotongan EcoRl pada sampel yang lain menghasilkan fragmen insert berukuran 1.200 pb
(Gambar 86)
Gambar 8. Pemotongan DNA-Rekombinan dengan enzim restriksi EcoRl
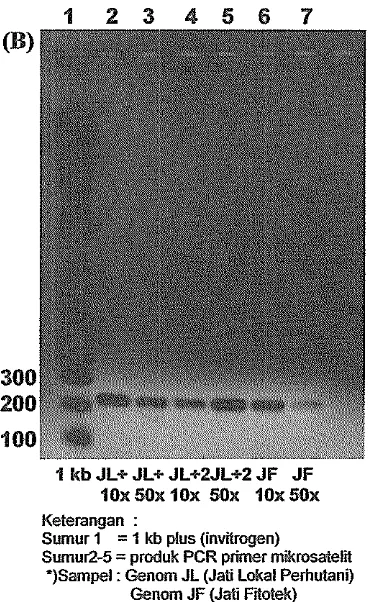
Verifikasi fragmen sisipan dapat juga dilakukan dengan amplifikasi PCR plasmid rekombinan atau kultur koloni tunggal menggunakan primer 21-mer
sebanyak 35 siklus yaitu denaturasi 94°C selama 4 menit, annealing 50°C
selama 1 menit dan elongasi 72°C selama 2 menit. Pita dari produk PCR
spesfik rnengamplilikasi adaptor yang lelah terligasi pada fragrnen DNA sisipan
tersebut. Hasil PCR dapat dilihat pada Gambar 9 dibawah ini.
Produk PCR tersebut sebagian besar be~kuran sekiar 200-300 pb yang mungkin berisi rnotiirnotii yang berbeda (Lampiran 3). Hal ini sesuai dengan
penelitian Effendi (2000) yang rnendapatkan fragrnen sisipan berkisar antara 162-334 pb pada tanarnan jati, M