i
KARAKTERISASI GEN
PARTICULATE METHANE
MONOOXYGENASE
(pMMO) BAKTERI METANOTROF
ASAL SAWAH
ANNISA RETNO FITRIANI
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
ABSTRAK
ANNISA RETNO FITRIANI. Karakterisasi Gen Particulate Methane Monooxygenase (pMMO) Bakteri Metanotrof Asal Sawah. Dibimbing oleh IMAN RUSMANA dan ALINA AKHDIYA.
Bakteri metanotrof adalah bakteri pengoksidasi metan yang memiliki enzim yang spesifik yaitu enzim metan monooksigenase (MMO). Aplikasi bakteri metanotrof dapat mengurangi emisi metan di lahan sawah. Terdapat dua jenis enzim MMO, yaitu soluble MMO (sMMO) dan particulate MMO (pMMO). Enzim pMMO adalah enzim terikat membran yang dimiliki oleh seluruh bakteri metanotrof. Isolat BGM 1 dan SKM 14 menunjukkan morfologi koloni yang berbeda ketika ditumbuhkan pada media Nitrate Mineral Salt (NMS) + 1% metanol. Amplifikasi gen pMMO dengan menggunakan primer A189f dan A682r menunjukkan pita DNA berukuran ~300 bp untuk isolat BGM 1 dan ~500 bp untuk isolat SKM 14. Sekuen yang diperoleh dianalisis menggunakan program BLAST-N dan BLAST-X. Analisis sekuen amplikon dari BGM 1 menggunakan program BLAST-N dan BLAST-X berturut-turut menunjukkan kemiripan tertinggi dengan sekuen gen (87%) dan asam amino pada protein (71%) acyl-coA dehydrogenase Xanthobacter autotrophicus Py2. Sedangkan analisis sekuen amplikon dari SKM 14 menggunakan program BLAST-N dan BLAST-X berturut-turut menunjukkan kemiripan tertinggi dengan sekuen gen (84%) dan asam amino pada protein (62%) subunit B cytochrome bc1
Sinorhizobium fredii HH103.
Kata kunci: Metan, Bakteri metanotrof, Enzim pMMO
ABSTRACT
ANNISA RETNO FITRIANI. Characterization of Particulate Methane Monooxygenase (pMMO) Gene of Methanotrophs from the Rice Field. Under direction of IMAN RUSMANA and ALINA AKHDIYA.
Metanotrophic bacteria are a methane oxidizing bacteria that have specific enzyme called methane monooxygenase (MMO). Application of metanotrophic bacteria can reduce emission of methane in rice fields. There are two types of MMO enzymes, i.e. soluble MMO (sMMO) and particulate MMO (pMMO). The pMMO enzyme is a membrane-bound enzyme that is present in all metanotrophic bacteria. BGM 1 and SKM 14 isolates showed a different colony morphology when grown on Nitrate Mineral Salt (NMS) medium + 1% methanol. The pMMO gene amplification using A189f and A682r primer showed a ~300 bp and ~500 bp of DNA band for BGM 1 and SKM 14 isolates respectively. The sequences were analyzed using BLAST-N and BLAST-X program. Analysis of amplicon sequence from BGM 1 using BLAST-N and BLAST-X program showed highest similarity with sequence of gene (87%) and amino acid in protein (71%) acyl-coA dehydrogenase Xanthobacter autotrophicus Py2 respectively. Whereas analysis of amplicon sequence from SKM 14 using BLAST-N and BLAST-X program showed highest similarity with sequence of gene (84%) and amino acid in protein (62%) subunit B cytochrome bc1 Sinorhizobium fredii HH103 respectively.
iii
KARAKTERISASI GEN
PARTICULATE METHANE
MONOOXYGENASE
(pMMO) BAKTERI METANOTROF
ASAL SAWAH
ANNISA RETNO FITRIANI
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul
: Karakterisasi Gen
Particulate Methane Monooxygenase
(pMMO)
Bakteri Metanotrof Asal Sawah
Nama
: Annisa Retno Fitriani
NIM
:
G34080114
Menyetujui:
Mengetahui:
Ketua Departemen Biologi
Fakultas Matematika dan Ilmu Pengetahuan Alam
Dr. Ir. Ence Darmo Jaya Supena, M.Sc
NIP 196410021989031002
Tanggal Lulus:
Pembimbing II,
Alina Akhdiya, M.Si
NIP 196812082001122001
Pembimbing I,
v
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala rahmat dan karunia-Nya, sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Desember 2011 sampai 2012 ini ialah Karakterisasi Gen Particulate Methane Monooxygenase (pMMO) Bakteri Metanotrof Asal Sawah.
Terima kasih penulis ucapkan kepada Bapak Dr. Ir. Iman Rusmana, M.Si dan Ibu Alina Akhdiya, M.Si atas bimbingan, bantuan pendanaan, dan pengarahan yang diberikan selama penelitian dan penyusunan skripsi ini. Terima kasih kepada Bapak Dr. Tri Atmowidi, M.Si. atas saran dan masukan yang telah diberikan sehingga tulisan ini menjadi lebih baik. Terima kasih juga kepada Papa, Mama, Teh Fifi, Kak Beny, Aziz, dan Tika atas segala doa, dukungan, dan kasih sayangnya. Penulis juga mengucapkan terima kasih kepada Hadi Rakhmanto, Tri Lugina, Retno, Ayang, Raya, Rima, Rizqi Ammar, Nida, Rini, Tata, Tya, dan Desti atas dukungan dan bantuan yang diberikan. Terima kasih juga kepada keluarga besar laboratorium Mikrobiologi, Whendi, Citra, Andri, Azizah, Ai, Dita, Issanto, Aida, Prima, Putri, Kak Sipri, Mbak Lena, Pak Jaka, Bu Heni, Mas Aldian, serta teman-teman Biologi 45 atas segala doa, dukungan, dan perhatiannya. Serta pihak-pihak yang secara tidak langsung telah membantu dalam pengumpulan data karya ilmiah ini.
Semoga karya ilmiah ini bermanfaat.
Bogor, November 2012
RIWAYAT HIDUP
Penulis dilahirkan di Pekalongan pada tanggal 15 April 1991 dari ayah Joni Aliando dan Ibu Henny. Penulis merupakan anak kedua dari empat bersaudara.
Tahun 2008 penulis lulus dari SMA Negeri 5 Bogor dan pada tahun yang sama lulus seleksi masuk IPB melalui Seleksi Masuk Perguruan Tinggi Negeri. Penulis memilih program studi Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam.
Selama mengikuti perkuliahan, penulis aktif menjadi anggota Divisi Pengembangan Sumber Daya Manusia (PSDM) Himpunan Mahasiswa Biologi (Himabio) pada tahun 2009-2010, Ketua Divisi Kesekretariatan Grand Biodiversity Biologi IPB tahun 2010, Sekretaris Masa Pengenalan Departemen (MPD) Biologi tahun 2010, Divisi Acara Workshop dan Diskusi Artikel Ilmiah Populer Biologi tahun 2010. Penulis menjadi peserta lomba Pekan Ilmiah Mahasiswa FMIPA tahun 2010 dengan judul “Ragam Cendawan Entomopatogen di Wahana Wisata Cangkuang”.
vii
DAFTAR ISI
Halaman
DAFTAR TABEL ... iv
DAFTAR GAMBAR ... iv
DAFTAR LAMPIRAN ... iv
PENDAHULUAN ... 1
Latar belakang ... 1
Tujuan ... 1
BAHAN DAN METODE ... 1
Waktu dan Tempat ... 1
Bahan... ... 1
Metode Penelitian ... 1
Peremajaan dan Pengkulturan Bakteri Metanotrof... ... 1
Isolasi Genom, Amplifikasi Gen pMMO, dan Visualisasi Amplikon ... 1
Perunutan DNA dan Analisis Bioinformatika ... 2
HASIL ... 2
Peremajaan dan Pengkulturan Bakteri Metanotrof ... 2
Amplifikasi Gen pMMO dan Visualisasi Amplikon ... 2
Perunutan DNA dan Analisis Bioinformatika ... 3
PEMBAHASAN ... 3
SIMPULAN ... 4
SARAN ... 4
DAFTAR PUSTAKA ... 4
LAMPIRAN ... 6
DAFTAR TABEL
Halaman
1. Morfologi koloni isolat bakteri metanotrof BGM 1 dan SKM 14 pada media NMS ... 2 2. Hasil analisis sekuen amplikon gen pMMO dengan menggunakan program BLAST-N... . 3 3. Hasil analisis sekuen amplikon gen pMMO dengan menggunakan program BLAST-X ... 3
DAFTAR GAMBAR
Halaman
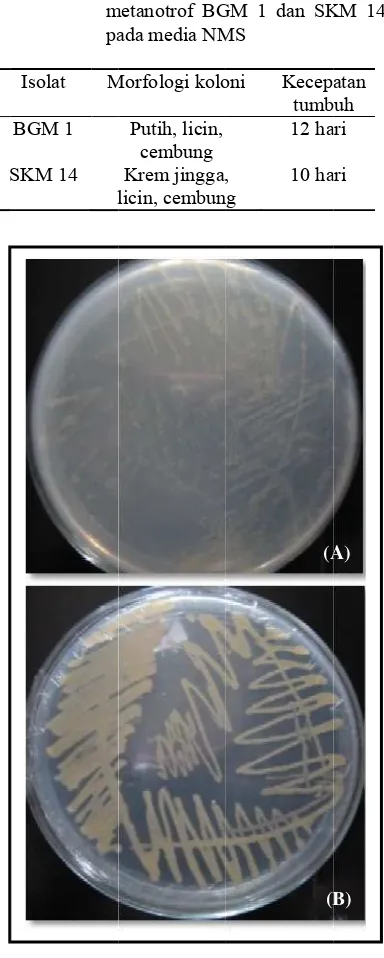
1. Koloni isolat bakteri metanotrof yang ditumbuhkan pada media NMS + 1% metanol. (A) Koloni BGM 1 berwarna putih. (B) Koloni SKM 14 berwarna krem jingga ... 2 2. Pita DNA hasil amplifikasi gen pMMO dari isolat bakteri metanotrof BGM 1 dan
SKM 14... ... 3
DAFTAR LAMPIRAN
Halaman
PENDAHULUAN
Latar Belakang
Suasana anaerob pada bagian bawah sedimen sawah merupakan habitat yang sesuai untuk bakteri penghasil metan (metanogen). Bakteri metanogen menggunakan CO2, metil,
dan asetat sebagai sumber karbon yang kemudian diubah menjadi metan melalui proses metanogenesis. Sebaliknya pada permukaan sedimen terdapat oksigen terlarut sehingga suasananya menjadi aerob dan sangat cocok untuk pertumbuhan bakteri metanotrof. Bakteri metanotrof adalah bakteri Gram negatif yang menggunakan metan sebagai sumber karbon untuk menghasilkan energinya. Whittenburry et al. (1970) menggolongkan bakteri metanotrof ke dalam lima genus berdasarkan perbedaan morfologi, tipe bentuk fase istirahat, struktur membran intrasitoplasma, dan beberapa karakter fisiologis. Kelima genus tersebut ialah
Methylomonas, Methylobacter, Methylococcus, Methylosistis, dan Methylosinus. Sedangkan berdasarkan perbedaan jalur biosintesisnya (jalur RuMP dan Serin), bakteri metanotrof digolongkan menjadi tiga tipe, yaitu metanotrof tipe I (Methylomonas dan
Methylobacter), tipe II (Methylosistis dan
Methylosinus), dan tipe X (Methylococcus capsulatus) (Hanson & Hanson 1996).
Karakteristik penting dari metanotrof ialah memiliki enzim metan monooksigenase (MMO) yang dapat mengkatalisis metan menjadi metanol. Oksidasi metan oleh bakteri metanotrof di lahan sawah dapat mencapai 80% dari total metan yang diproduksi oleh bakteri metanogen (Conrad & Rothfus 1991). Selain dapat mengoksidasi metan, enzim MMO juga berperan dalam degradasi berbagai senyawa polutan seperti trikloroetilen, isomer-isomer dari dikloroetilen, vinil klorida, dan kloroform (Graham et al. 1992). Terdapat dua tipe MMO, yaitu soluble MMO (sMMO) dan
particulate MMO (pMMO) (Hanson & Hanson 1996). Enzim pMMO merupakan enzim terikat membran yang dimiliki oleh seluruh bakteri metanotrof dan dapat terekspresi dalam media dengan kandungan tembaga yang tinggi. Sedangkan enzim sMMO hanya dimiliki oleh beberapa bakteri metanotrof dan dapat terekspresi dalam media dengan kandungan tembaga rendah (Stanley et al. 1983).
Laboratorium Mikrobiologi FMIPA IPB, telah mengoleksi beberapa isolat bakteri metanotrof yang berasal dari lumpur sawah di Bogor dan Sukabumi. Beberapa isolat telah
diidentifikasi secara molekular dan dikarakterisasi secara terbatas reaksi fisiologis dan biokimianya. BGM 1 merupakan isolat yang mampu mengakumulasi amonium lebih tinggi dibandingkan isolat lainnya, sedangkan SKM 14 merupakan isolat yang memiliki nilai oksidasi metan tertinggi (Hapsari 2008). Hasil analisis gen 16S rRNA yang dilakukan oleh Astuti (2009) memperlihatkan bahwa BGM 1 memiliki tingkat kemiripan 74% dengan
Methylocystis rosea strain SV97T dan SKM 14 memiliki tingkat kemiripan 65% dengan
Methylobacter sp. Clone GASP-OKA-565-E11. Kedua isolat tersebut memiliki enzim pMMO namun belum dilakukan karakterisasi gennya. Informasi mengenai karakterisasi gen
pMMO dari isolat BGM 1 dan SKM 14 dapat digunakan landasan ilmiah untuk pemanfaatan bakteri tersebut di lahan pertanian.
Tujuan
Penelitian ini bertujuan untuk mengkarakterisasi gen particulate methane monooxygenase (pMMO) pada metanotrof asal sawah Bogor dan Sukabumi.
BAHAN DAN METODE
Waktu dan Tempat
Penelitian ini dilaksanakan dari bulan Desember 2011 sampai Agustus 2012 di Laboratorium Mikrobiologi, Departemen Biologi, FMIPA, IPB.
Bahan
Bakteri yang digunakan dalam penelitian ini adalah isolat bakteri metanotrof BGM 1 dan SKM 14. Kedua bakteri ini diisolasi dari lumpur sawah asal Bogor dan Sukabumi (Hapsari 2008).
Metode
Peremajaan dan Pengkulturan Bakteri Metanotrof. Kultur bakteri metanotrof diremajakan dan dikulturkan pada media agar NMS + 1% metanol (Lampiran 1) dengan teknik gores kuadran. Inkubasi dilakukan pada suhu ruang selama 3-14 hari.
Isolasi DNA Genom, Amplifikasi Gen
pMMO, dan Visualisasi Amplikon. DNA genom diisolasi dari kultur padat bakteri metanotrof yang ditumbuhkan dengan cara sebagaimana disebutkan diatas. Isolasi DNA genom dilakukan menurutmetode Lazo (Lazo
A682r (Bourne et
reaksi PCR sebagai ber Buffer KOD µl dNTPs, 1 DNA templa Polymerase
selama 30 (Perkin El kondisi rea denaturasi ( menit), ann
(72ºC, 1 m menit). Am agarosa kem
Bromide ( dilakukan Translumina Perunu Bioinforma DNA hasil memanfaatk Base di Si dibandingka terdapatpad Blast-N dan Center for mengetahui isolat yang menggunaka Alignment E al. 2011).
Peremajaan Metanotrof
Isolat SKM 14 ya + 1% meta morfologi k Gambar 1). putih dan ko jingga. Kec isolat juga lebih cepat d
(GAASGC
al. 2001). To sebanyak 50 rikut: 31,5 μ D Hot Start, 3 1,5 μl
masing-ate, dan 1 μl (1U/μl). A siklus mengg lmer Biosyst aksi PCR se 94ºC, 5 menit
nealing (54ºC menit), dan po
mplikon dilar mudian diwar (EtBr). Visu menggunaka ator. utan DNA atika. Perunu
l amplifikasi kan jasa perus ingapura. Sek an dengan da GenBank m n Blast-X dari
Biotechnology
tingkat kemir dianalisa. An an software Editor dan M
HA
n dan Pen f
bakteri meta ang ditumbuhk anol selama 1 koloni yang b Koloni isol oloni isolat SK cepatan pertum
berbeda. Iso dibandingkan
CNGAGAAGA otal volume ca μl dengan ko
μl ddH2O, 5
3 μl 25mM M
-masing prime KOD Hot Sta Amplifikasi d
gunakan mes tem, USA) ebagai beriku t), denaturasi C, 1 menit), ost-elongasi ( rikan pada rnai dengan E
ualisasi pita an perangka A dan utan nukleotid i dilakukan sahaan sekuen kuen yang d
data sekuen menggunakan
situs NCBI (N gy Information
ripan gen pMM
nalisis bioinfo
Bioedit S
MEGA 5.0 (Ta
SIL
ngkulturan
anotrof BGM kan pada med 14 hari menu berbeda (Tabe at BGM 1 b KM 14 berwar mbuhan kolon olat BGM 1 isolat SKM 1
AASGC) ampuran omposisi µl 10x MgSO4, 5
er, 1,5 μl art DNA dilakukan sin PCR dengan ut: pra-(94ºC, 1 elongasi (75ºC, 7 1% gel Ethidium a DNA at UV-Analisis da pada dengan nsing 1st diperoleh
n yang program
National n) untuk
MO dari formatika
Sequence
amura et
Bakteri
[image:10.595.317.509.97.576.2]M 1 dan dia NMS unjukkan el 1 dan berwarna rna krem ni kedua tumbuh 4. T S G A A m m u S
Tabel 1 M m pa Isolat M BGM 1 SKM 14 l
Gambar 1 K ya N B K jin Amplifikasi Amplikon Visualisasi menggunakan menunjukkan untuk isolat B SKM 14 (Gam
Morfologi ko metanotrof BG
ada media NM
Morfologi kolo Putih, licin, cembung Krem jingga, licin, cembun Koloni isolat ang ditumbu NMS + 1% m
GM 1 berw Koloni SKM
ngga.
Gen pMMO
i DNA n primer A
pita DNA b BGM 1 dan ~ mbar 2).
loni isolat b GM 1 dan SK MS oni Kecep tumb 12 ha , ng 10 ha bakteri meta uhkan pada metanol. (A) K
rwarna putih 14 berwarna
O dan Visu
Gambar 2
Perunutan Bioinforma
Hasil (Lampiran 2 BLAST-N 87% (245/2 sekuen gen
Xanthobacte
sekuen a menunjukka nukleotida o
B cytochrom HH103 (Ta menggunaka sekuen amp kemiripan dehydrogena BG8. Sedan menunjukka Cytochrome (Tabel 3 dan
M
Pita DNA h
pMMO da metanotrof B (Keterangan DNA ladder,
Sumur 2 = BG
DNA atika
analisis seku 2) BGM 1 m menunjukkan 281 nukleoti n acyl-coA
er autotrophi
amplikon p
an tingkat kem
overlap) denga me bc1 pada abel 2 dan L
an program likon BGM 1
71% d ase pada Meth
ngkan sekuen an tingkat ke e b pada M
n Lampiran 3)
1
hasil amplifik ari isolat BGM 1 dan S
M = mark
, Sumur 1 = S GM 1).
dan
uen amplikon menggunakan
n tingkat ke ida overlap)
dehydrogenas
icus Py2. Se
pMMO SKM miripan 84% (
an sekuen gen
Sinorhizobiu
Lampiran 3). BLAST-X menunjukkan dengan A
hylomicrobium
n amplikon S miripan 62%
Methylocystis s ).
~ 5
~ 30
2
kasi gen bakteri SKM 14. ker 1 kbSKM 14,
Analisis pMMO program emiripan dengan se pada edangkan M 14 (135/160 n subunit um fredii Analisis terhadap n tingkat Acyl-CoA m album SKM 14 % dengan sp. SC2 T I B S a b T I B S m d p j d B m t s S p ( F d d
500 bp
00 bp
Tabel 2 Has
pMM pro Isolat Sekue yang BGM 1 Gen a dehyd p Xanth autot SKM 14 Gen s cyto bc Sinor fredi a 245/281 nukl b 135/160 nuk
Table 3 Has
pMM pro Isolat Sekue yang BGM 1 Acy dehyd p Methy bium B SKM 14 Cytoc p Meth sp
Isolat b memiliki mo dengan SKM putih sedang
ingga (Tabe dipengaruhi o Bakteri metan macam pigme terlarut sepert sering ditemu Selain itu, produksi pig (Hanson & Ha Frenzel 2001) dan karotenoi dipengaruhi o
sil analisis se
MMO denga gram BLAST en bakteri homolog i acyl-coA drogenase pada hobacter trophicus Py2 subunit B ochrome 1 pada rhizobium i HH103 leotida overlap
kleotida overla
sil analisis sek
MMO denga gram BLAST en bakteri homolog i yl-CoA drogenase pada ylomicro m album BG8 chrome b pada hylocystis p. SC2
PEMBAH
bakteri met orfologi kolo14. Koloni kan SKM 1 el 1). Warn oleh pigmen y notrof mengh en terlarut dan ti putih, krem ukan (Murrell
beberapa pe gmen kuning anson 1996), , serta pigmen id (Holt et al
oleh faktor ge
ekuen ampliko an menggu T-N % identitas N ak 87%a C
07
84%b H 68 ap ap kuen ampliko an menggu T-X % dentitas N ak 71% ZP 98 56 62% YP 06 02
HASAN
tanotrof BG oni yang b i BGM 1 ber 14 berwarnabiakan, dan tahapan fisiologis bakteri (Murrell & Dalton 1992).
Oksidasi metan pada bakteri metanotrof diinisiasi oleh enzimmethane monooxygenase
(MMO) yang mampu menambahkan satu atom oksigen ke dalam molekul metan untuk membentuk senyawa metanol. Selain mengkatalisis reaksi oksidasi metan, enzim MMO terikat membran (pMMO) juga berperan dalam proses degradasi senyawa hidrokarbon terhalogenasi seperti trikloroetilen (Dispirito et al. 1992). Enzim pMMO tersusun atas tiga subunit yaitu α, , dan yang masing-masing disandikan oleh gen pmoA, pmoB, dan pmoC
(Semrau et al. 1995).
Amplifikasi gen pMMO menggunakan primer A189f dan A682r menghasilkan pita DNA dengan ukuran ~500 bp dan ~300 bp berturut-turut untuk isolat BGM 1 dan SKM 14. Analisis sekuen amplikon dari BGM 1 menggunakan program BLAST-N dan BLAST-X berturut-turut menunjukkan kemiripan tertinggi dengan sekuen gen (87%) dan asam amino pada protein (71%) acyl-coA dehydrogenase Xanthobacter autotrophicus
Py2. Sedangkan analisis sekuen amplikon dari SKM 14 menggunakan program BLAST-N dan BLAST-X berturut-turut menunjukkan kemiripan tertinggi dengan sekuen gen (84%) dan asam amino pada protein (62%) subunit B cytochrome bc1 Sinorhizobium fredii HH103.
Hasil analisis kedua sekuen amplikon tersebut tidak sesuai dengan gen target (pMMO
pada bakteri metanotrof). Ketidaksesuaian hasil analisis tersebut diduga disebabkan antara lain oleh spesifitas primer yang rendah, sekuen yang dianalisis terlalu pendek, atau keterbatasan data base pada GeneBank.
Primer A189F/A682r diduga kurang spesifik untuk mengamplifikasi gen pMMO
bakteri tropis walaupun primer ini berhasil diaplikasikan untuk mendeteksi serta menganalisis keragaman gen pMMOA metanotrof di padang rumput dan oaks di Denmark (Bourne et al. 2001). Holmes et al. (1995) melaporkan bahwa primer A189F/A682r juga dapat mengamplifikasi gen
Ammonium Mono-oxygenase (AMO) yang terdapat pada bakteri pengoksidasi amonium. Selain primer tersebut, gen pMMO juga dapat diamplifikasi menggunakan primer A189f/mb661 yang lebih spesifik untuk mendeteksi gen pMMO bakteri metanotrof (Bourne et al. 2001).
Ketepatan dan nilai tingkat kemiripan pada hasil analisis kesejajaran sekuen menggunakan program BLAST-N dan BLAST-X juga sangat dipengaruhi oleh
panjang sekuen DNA yang dianalisis dan kelengkapan data base yang terdapat pada GeneBank. Semakin panjang sekuen yang dianalisis dan semakin banyak data base yang terdapat pada GeneBank, maka semakin tinggi tingkat ketepatan dan nilai tingkat kemiripan yang akan diperoleh. Sebagian besar data yang terdapat pada GenBank berasal dari sampel daerah temperate. Sedangkan sekuen DNA yang dianalisis pada penelitian ini berasal dari bakteri tropis. Oleh karena itu, hasil analisis sekuen amplikon gen pMMO yang diperoleh dalam penelitian ini tidak sesuai dengan gen target.
SIMPULAN
Morfologi isolat BGM 1 dan SKM 14 memperlihatkan perbedaan warna koloni, tepian, dan elevasi. BGM 1 memperlihatkan warna koloni putih, tepian licin, dan elevasi cembung, sedangkan SKM 14 memperlihatkan warna koloni krem jingga, tepian licin, dan elevasi cembung. Analisis sekuen amplikon dari BGM 1 menggunakan program BLAST-N dan BLAST-X berturut-turut menunjukkan kemiripan tertinggi dengan sekuen gen (87%) dan asam amino pada protein (71%) acyl-coA dehydrogenase Xanthobacter autotrophicus
Py2. Sedangkan analisis sekuen amplikon dari SKM 14 menggunakan program BLAST-N dan BLAST-X berturut-turut menunjukkan kemiripan tertinggi dengan sekuen gen (84%) dan asam amino pada protein (62%) subunit B cytochrome bc1 Sinorhizobium fredii HH103. Ketidaksesuaian hasil analisis tersebut diduga disebabkan antara lain oleh spesifitas primer yang rendah, sekuen yang dianalisis terlalu pendek, atau keterbatasan data base pada GeneBank.
SARAN
Perlu dilakukan lebih lanjut penelitian terhadap gen pMMO BGM 1 dan SKM 14 dengan menggunakan primer yang lebih spesifik.
DAFTAR PUSTAKA
5
Bourne DG, McDonald IR, Murrell JC. 2001. Comparison of pmoA PCR primer sets as tools for investigating methanotroph diversity in three Danish soils. Appl Environ Microbiol 67:3802-3809.
Conrad R and Rothfus F. 1991. Methane oxidation in the soil surface layer of aflooded rice field and the effect of ammonium. Biol Fertil Soil 12:28-32. Dispirito AA et al. 1992. Trichloroethylene
oxidation by the membrane associated methane monooxygenase in type I, type II and type X methanotrophs.
Biodegradation 2:151–164.
Eller G, Frenzel P. 2001. Changes in activity and community structure of methane-oxidizing bacteria over the growth period of rice. Appl Environ Microbiol 67:2395-2403.
Graham DW, Korich DG, Leblanc RP, Sinclair NA, Arnold RG. 1992. Application of a colorimetric plate assay for soluble methane monooxygenase activity. Appl Environ Microbiol 58:2231-2236.
Hanson R, Hanson TE. 1996. Metanotropic bacteria. J Microbial Rev 60:439-471. Hapsari W. 2008. Isolasi dan karakterisasi
bakteri metanotrof asal sawah di Bogor dan Sukabumi [skripsi]. Bogor : Institut Pertanian Bogor.
Holmes AJ, Costello A, Lidstrom ME, Murrell JC. 1995. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily
related. FEMS Microbiol Lett 132:203– 208.
Holt JG, Krieg NR, Sneath PHA, Staley JT, Williams ST. 1994. Bergey’s Manual of Determinative Bacteriology. Ed ke-9. Lippincott William & Wilkins: a Wolter Kluwer Co.
Lazo GR, Roffey R, Gabriel DW. 1987. Conservation of plasmid DNA sequences and pathovar identification of strain
Xantholomonas campestris. Pytopathology 77: 1461-1467.
Murrell JC, Dalton H. 1992. Methane and Methanol Utilizers. New York: Springer-Verlag.
Semrau et al. 1995. Particulate methane monooxygenase genes in methanotrophs.
J Bacteriol 177:3071-3079.
Stanley SH, Prior SD, Leak DJ, Dalton H. 1983. Copper stress underlies the fundamental change in intracellular location of methane monooxygenase in methane-oxidizing organisms: studies in batch and continuous culture. Biotechnol Lett 5:487-492.
Tamura K et al. 2011. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol 28:2731-2739. Whittenburry RKC, Phillips, Wilkinson JG.
7
Lampiran 1 Komposisi media Nitrate Mineral Salt (NMS) 300 ml + 1% metanol 1. 120 µl NH4Cl
2. 1200 µl CaCl2
3. 0.3 g MgSO4
4. 150 µl Trace Element
5. 1632 µl KH2PO4
6. 0.3 gr NaNO3
7. 2151 µl Na2HPO4
Lampiran 2 Sekuen pita DNA isolat BGM 1 dan SKM 14 BGM 1
Lampiran 3 Hasil sekuen gen pMMO, BLAST-N, dan BLAST-X isolat BGM 1 dan SKM 14 A. Urutan nukleotida gen pMMO isolat BGM 1
ACGACACCGTGAGGAAGAAGGGGTCCACCACCTCGTCGTTGGACTTGTCG CCGTCGACGATGGGGCCCACGAGAGCCTCCTTGGACATCACCTCCTTTTC CACCACGATGGGCCCCGACTGGTTGCCGCGGAGGCCCAGGCCGTCCCACT CGGAGGGGTTGGCCTTCACCTCGTCCTTGGTGACGAGGAAGGGGAAAGGT CGGAATAGTCACCAGAGAATCCGGGGCTGGTGGTCTGCACTATGTACCAG TCTCAAAAGCCTCCGGAGGTGGTCCAGGAGGCCTTCTTCTCCGCCT B. Hasil BLAST-N sekuen gen pMMO isolat BGM 1 dengan data GenBank
11
D. Urutan nukleotida gen pMMO isolat SKM 14