FOSFAT DARI Pseudomonas sp.AZM-1MELALUI MUTAGENESIS DENGAN TRANSPOSON
ALI ZUM MASHAR
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Dengan ini saya menyatakan bahwa tesis yang berjudul:
Isolasi Gen Yang Terlibat Dalam Pelarutan Fosfat Dari Pseudomonas sp. AZM-1 Melalui Mutagenesis Dengan Transposon
adalah karya saya sendiri dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber data dan informasi yang dikutip dalam tesis ini telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka di bagian akhir tesis ini. Oleh karena itu, semua isi tesis ini dapat dipertanggung jawabkan kebenarannya.
Bogor, April 2009
SUHARSONO, ARIS TRIWAHYUDI.
Pseudomonas sp. AZM-1 is soil microbe which have phosphate-solubilizing from enzyme metabolite activities mechanism so this bacterium have a gene related to phosphate-solubilizing. The aim of this reseach was to isolate, clone and characterize gene involved in phosphate-solubilizing. To isolate a gene involved in phosphate-solubilizing, the mutant was contructed using transposon mini Tn5Km1 to yield become Non-Phosphate-Solubilizing (NPS) Pseudomonas sp. Transposon delivery was carried out through conjugation between Pseudomonas sp. AZM-1 and E. coli S17 ( pir) carrying pUTmini-Tn4Km1. The frequency of transconjugation was abaut 3.0 x 10-9 cells per recipient. These NPS bacteria did not solubilize phosphate contained in the pykovskaya medium. DNA fragment interrupted by transposon (called flanking DNA) was isolated by Inverse Polymerase Chain Reaction (Inverse PCR) and cloned into pGEM-T Easy. DNA sequence analysis showed that the flanking DNA is 1006 bp and contains an Open Reading Frame (ORF). This ORF contains 215 amino acids allignment analysis of amino acid sequence reduced from this ORF showed that this ORF is identic with DNAse1 from Pseudomonas fluorescens strain Pf-5. This gene might function indirectly in the phosphate solubilization in Pseudomonas sp. AZM-1.
Keyword: Pseudomonas sp. AZM-1, phosphate-solubilizing, Transposon mini Tn5, DNAse1, endA gene.
Pseudomonas sp. AZM-1 Melalui Mutagenesis Dengan Transposon. Dibimbing oleh: SUHARSONO, ARIS TRIWAHYUDI
Pseudomonas sp.AZM-1 adalah mikroba tanah yang memiliki kemampuan dalam pelarutan fosfat melalui mekanisme enzimatik sehingga bakteri ini memiliki gen yang berhubungan dengan pelarutan fosfat. Mikroba gram negatif ini memiliki respon positif terhadap transposon mini Tn5Km1. Tujuan penelitian ini adalah untuk mengisolasi, mengklon dan mengkarakterisasi gen yang terlibat dalam pelarutan fosfat.
Penelitian ini dimulai dengan mengisolasi bakteri pelarut fosfat, isolasi fragmen DNA genom dari mutan negatif pelarut, amplifikasi fragmen DNA pengapit menggunakan inverse PCR, pengklonan fragmen hasil PCR pada pGEM®-T Easy, introduksi plasmid ke dalam bakteri E. coli galur DH5α. Fragmen DNA vektor rekombinan dari mutan negatif pelarut fosfat yang didapat di sekuen dengan menggunakan primer T7 dan SP6. Untuk mengisolasi gen yang terlibat dalam pelarutan fosfat, bakteri ini dimutasikan dengan transposon mini Tn5Km1 sehingga menjadi bakteri mutan yang tidak mampu melarutkan fosfat. Introduksi transposon dilakukan melalui konjugasi antara Pseudomonas sp.AZM-1 dengan E. coli S17 ( pir) yang mengandung pUT mini–Tn5Km1. Frekuensi transkonjugasi sel bakteri mutan negatif pelarut fosfat didapatkan dari media sebar adalah 3.00 x 10-9. Bakteri mutan ini tidak dapat melarutkan fosfat di dalam media pykovskaya.
Fragmen DNA yang tersisipi transposon diisolasi dengan cara Polymerase Chain Reaction terbalik (inverse PCR) dan diklon ke dalam pGEM®-T Easy. Analisis mutan menunjukkan bahwa DNA pengapit transposon berukuran 1006 pb dan mengandung kerangka baca (Open Reading Frame). Kerangka baca ini mengandung 215 asam amino. Analisis penyejajaran urutan nukleotida dan asam amino yang menggunakan program BLAST X, menunjukkan bahwa fragmen tersebut adalah endonuklease 1 yang memiliki kesamaan genom dengan Pseudomonas fluorescens Pf-5. Analisis kesejajaran dari urutan asam amino yang dideduksi dari ORF ini menunjukkan bahwa ORF ini memiliki kesamaan dengan DNAse1 yang disandikan oleh gen endA dari Pseudomonas fluorescens galur Pf-5. Gen ini mungkin secara tidak langsung dalam pelarutan fosfat di dalam Pseudomonas sp. AZM-1.
@ Hak Cipta milik IPB, tahun 2009
Hak Cipta dilindungi Undang-undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebut sumber
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik atau tinjauan suatu masalah
b. Pengutipan tidak merugikan kepentingan yang wajar IPB
ISOLASI GEN YANG TERLIBAT DALAM PELARUTAN FOSFAT DARI Pseudomonas sp.AZM-1 MELALUI
MUTAGENESIS DENGAN TRANSPOSON
ALI ZUM MASHAR
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Pseudomonas sp.AZM-1 Melalui Mutagenesis Dengan Transposon
Nama Mahasiswa : Ali Zum Mashar
Nomor Pokok : P055040071
Program Studi : Bioteknologi
Menyetujui Komisi Pembimbing
Dr. Ir. Suharsono, DEA, Ketua
Dr. Aris Triwahyudi, M.Si.
Anggota
Diketahui Ketua Program
Studi Bioteknologi
Dr. Ir. Muhammad Jusuf, DEA.
Dekan Sekolah Pascasarjana
Prof. Dr. Ir. Khairil Anwar Notodiputro, MS.
Bismillairrahmanirrahiim,
Segala puji penulis panjatkan kepada Allah Subhanaahu Wa Ta’alaa atas limpahan berkat dan rahmat-Nya yang tiada terhingga sehingga penulis dapat menyelesaikan penelitian dan penulisan tesis ini yang berjudul ” Isolasi Gen Yang Terlibat Dalam Pelarutan Fosfat Dari Pseudomonas aeroginosa Melalui Mutagenesis Dengan Transposon”.
Terima kasih penulis sampaikan kepada pihak-pihak yang telah membimbing dan membantu dengan penuh perhatian dan kecintaan selama proses penelitian ini baik secara langsung maupun tidak langsung. Terimakasih disampaikan kepada Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB yang telah menyediakan fasilitas penelitian ini. Untuk itu penulis menyampaikan terima kasih kepada:
1. Dr. Ir. Suharsono, DEA selaku pembimbing yang telah meluangkan waktu didalam memberikan pembimbingan dan wawasan berfikir yang sangat berharga bagi penulis.
2. Dr. Aris Triwahyudi, M.Si selaku pembimbing yang telah memberi bimbingan, nasehat dan bahan selama proses penelitian bagi penulis.
3. Dr. Ir.Muhammad Jusuf, DEA selaku ketua Program Studi Bioteknologi SPs IPB.
4. Anak dan Istriku tercinta atas segala pengorbanannya yang memberikan dukungan moril dan materiil.
5. Teman-temanku: Firdaus (Btk), mbak Pepy (Biorin), mbak Arie (Seafast), Rika Indri Astuti (Biologi), Syahnada Jaya, Pak Mulya, Efrizal, dan teman-teman se-angkatan bioteknologi 2004 yang banyak membantu.
Akhirnya berharap Allah SWT memberikan balasan rahmat dan kebaikan kepada kita semua. Semoga karya ilmiah ini dapat bermanfaat bagi kita semua.
Bogor, April 2009
Penulis dilahirkan di Bakung, Kec. Mijen, Kabupaten Demak, Propinsi Jawa Tengah pada tanggal 19 Mei 1972. Penulis adalah anak keenam dari tujuh bersaudara dari pasangan yang berbahagia H. M. Ali Mukti dan Hj. Sri Tasriah. Saat ini penulis telah menikah dengan Sonni Anna, SPt. dan dikaruniai tiga orang anak : Zumarizka Akbar Ibrahim, Kalyana Daeva Ali dan Mughni Vada Ali.
Penulis lulus dari SMU Negeri I Kudus tahun 1991 dan melanjutkan di Fakultas Pertanian UNSOED Purwokerto. Penulis pada bulan September 2004 melanjutkan pendidikan Sekolah Pascasarjana di Institut Pertanian Bogor, Program Studi Bioteknologi. Setelah lulus S1 (1997) bekerja sebagai PNS Deptrans dan PPH Kalteng dan saat ini menjadi peneliti di Puslitbangtrans Depnakertrans serta tenaga ahli di beberapa perusahaan swasta Nasional.
Selama mahasiswa penulis aktif di organisasi kegiatan kampus seperti ketua Badan Pelaksana Harian SMPT Unsoed, ketua Unit Kerohanian Islam (UKI) Faperta, ketua pembinaan UKKI Unsoed, ketua Perhimpunan Mahasiswa Agroindustri Jateng, ketua KSIMP Faperta, dan lain.lain. Penulis aktif di dalam kegiatan kompetisi keilmiahan antara lain mendapatkan Juara I LCTQ Unsoed (1993), Juara I LP2P4 Unsoed dan antar perguruan tinggi se-Jawa Tengah (1994), finalis Lomba Karya Inovatif Produktif Nasional LKIP (1994), Juara I LKTI Mapres Unsoed (1995), Juara I LP2PA (1995), Juara II LKTI Nasional di UGM (1996), juara harapan I pameran ilmiah PIMNAS IX dan menjadi mahasiswa berprestasi utama I Faperta Unsoed dan Mahasiswa berprestasi utama dan teladan Unsoed (1995), menerima beasiswa PERTAMINA, Supersemar dan PPA Dikti.
DAFTAR GAMBAR ...………….……..…… iv
DAFTAR LAMPIRAN...……..…… vi
PENDAHULUAN Latar Belakang...…………. 1
Tujuan Penelitian ...…………. 3
TINJAUAN PUSTAKA Mobilitas Unsur Phosfat ...…………. 4
Mikroba Pelarut Phosfat ... 5
Peran Transposom dalam Mutagenesis... 7
aBAHAN DAN METODE Waktu dan Tempat Penelitian...…………. 9
Bahan...…………. 9
Metode Penelitian...…………. 10
Isolasi bakteri pelarut fosfat...…………. 11
Mutagenesis dengan Transposon ... 12
Seleksi mutan ...…………. 13
Isolasi DNA genom mutan P. aeroginosa ... 14
Pemotongan dan ligasi DNA yang mengandung mini-Tn5Km1 ... 15
Amplifikasi DNA yang diapit mini Tn5Km1 ... 16
Pengklonan hasil amplifikasi fragmen DNA pengapit mini Tn5... 17
Transformasi E. coli DH5α... ... 18
Seleksi transforman rekombinan...…………. 19
Isolasi Plasmid ... 19
Pengurutan nukleotida dan analisis bioinformatika ... 19
HASIL DAN PEMBAHASAN Skrining Pseudomonas sp. mikroba pelarut fosfat ... 21
Mutagenesis dengan Transposon dan seleksi mutan ... 21
Isolasi dan pemotongan DNA genom total mutan negatif pelarut fosfat -P. aereus ..... ... 22
Amplifikasi Fragmen DNA Pengapit MiniTn5 Menggunakan Inverse PCR... 23
Pengklonan dan Hasil Amplifikasi Fragmen DNA Pengapit mini Tn5... 24
Pengurutan fragmen DNA pengapit miniTn5... 26
Analisis Kesamaan Gen Sisipan Dengan Data Gen di Genbank... 28
Peta Enzim Restriksi Endonuklease fragmen ... 31
Urutan asam amino deduksi dari DNA yang disisipi Tn5………... ... 31
Analisis Domain Fragmen DNA Pengapit Tn5………... 33
SIMPULAN DAN SARAN Simpulan ... 35
Saran ... 35
DAFTAR PUSTAKA ... 36
Halaman 1. 2. 3. 4. 4. 5. 6. 7. 8. 9. 10.
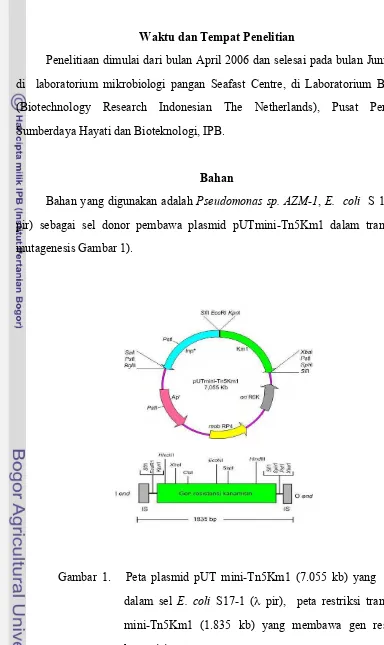
Peta plasmid pUT mini-Tn5Km1 (7.055 kb) yang berada dalam sel E. coli S17-1 (λ pir), peta restriksi transposon mini-Tn5KmS17-1 (S17-1.835 kb) yang membawa gen resistensi ...
Peta plasmid pGEMT-Easy ...
Tahapan isolasi, pengklonan dan karakterisasi fragmen gen yang terlibat dalam pelarutan fosfat dari P. aeroginosa....
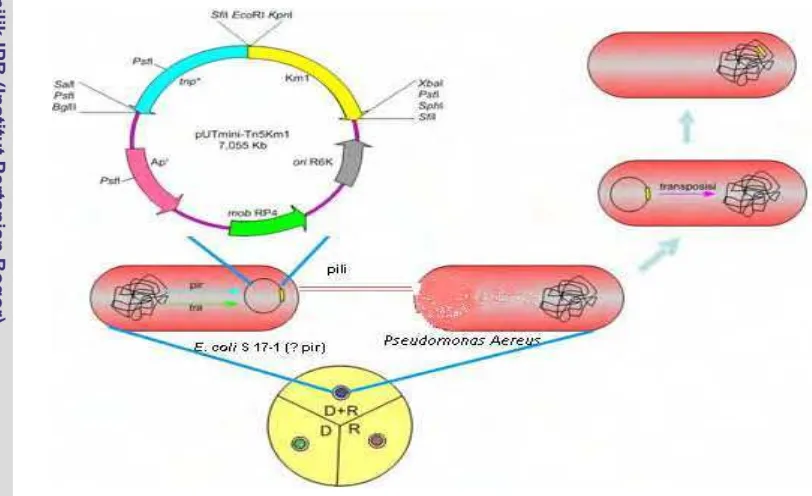
Diagram skematis proses mutagenesis dengan transposon. Konjugasi dilakukan di atas filter milipore (0.45 µm) pada media LB. Pada suspensi campuran donor (D) dengan resipien (R) akan terjadi serangkaian proses transfer dan penyisipan transposon ...
Proses mutagenesis dengan transposon. Konjugasi dilakukan di atas filter milipore (0.45 µm) pada media LA modifikasi. Pada suspensi campuran donor (D) dengan resipien (R) akan terjadi serangkaian proses transfer dan penyisipan transposon ...
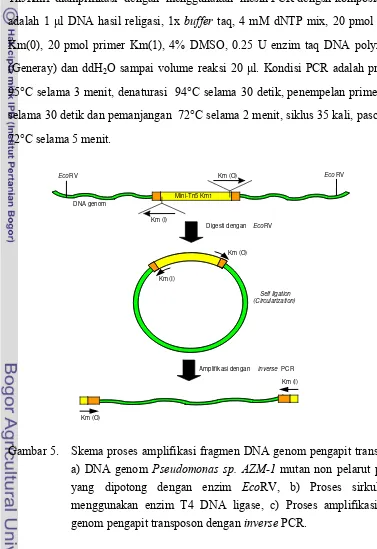
Skema proses amplifikasi fragmen DNA genom pengapit transposon. a) DNA genom Pseudomonas Aereus mutan non pelarut phosfat yang dipotong dengan enzim EcoRV, b) Proses sirkularisasi menggunakan enzim T4 DNA ligase, c) Proses amplifikasi DNA genom pengapit transposon dengan inverse PCR ...
Penampilan koloni P. aeroginosa pembawa gen pelarut fosfat pada media phykovskaya: a. Peremajaan dari sel tunggal P. aeroginosa hasil isolasi, b. Pertumbuhan fase logaritmatik, c. Penampilan pertumbuhan murni pada media pykovskaya menunjukkan zona bening...
Mutan P. aeroginosa padapikovskaya agar + Chl 50 µg/mL + Km 50 µg/mL yang diinkubasi selama 24 jam ...
Penapisan terhadap mutan untuk mendapatkan mutan negatif pelarut fosfat: a. koloni P. aeroginosa tipe liar pelarut fosfat, b. mutan P. aeroginosa negatif pelarut fosfat...
Elektroforesis gel agarose DNA genom mutan P. aeroginosa yang bukan pelarut fosfat...
DNA hasil Inverse PCR (1. sampel; 2. penanda ukuran 1 kb plus). ...
12. 13. 14. 15. 16. 17. 18. 19. 20. 21. .
yang mengandung 100 μg/ml ampisilin, 10 l 100 mM isopropiltio-β -galaktosida (IPTG) (Promega) dan 50 l 2%(b/v) X-gal ( 5-bromo-4-choloro-3-indolyl-β-D-galactoside) (Promega) hasil inkubasi pada suhu 37°C semalam...…………....
Hasil PCR dengan koloni putih sebagai cetakan. 1. penanda 1 kb plus; 2. koloni 1; 3. koloni 2; 4. koloni 3; 5. koloni 4. ……….………
Verifikasi palsmid rekombinan dengan pemotongan enzim restriksi EcoRI (1. penanda 1 kb plus; 2. plasmid tidak dipotong; 3. plasmid dipotong dengan EcoRI)...
Urutan nukleotida dari DNA sisipan di dalam pGEM®-T Easy ….……….
Urutan gen sesungguhnya yang diinaktivasi oleh mini-Tn5 ...
Kesejajaran endonuklease 1 yang disandikan oleh gen endA dari Pseudomonas Fluorescen Pf-5……….………..
Dendogram filogenetik berdasarkan urutan nukleotida menunjukkan kesejajaran endonuklease P. aeroginosa dengan endonuklease I pada P. Fluorescens Pf-5. Filogenetik berdasarkan urutan nukleotida………...……..
Peta restriksi endonuklease1 (DNAse1) dari P. aeroginosa ………..……….
Deduksi asam amino penyusun enzim DNAse1 berdasarkan kodon-kodon pada daerah ORF fragmen gen pada P. aeroginosa ….………..
Kesejajaran asam amino lokal fragmen EcoRV dari P. aeroginosa dengan endonuclease I (P. fluorescens Pf-5) YP_259205……….
Halaman 1. Hasil pengurutan nukleotida dengan primer T7 - SP6... 40
2. Komposisi berbagai media pertumbuhan Pseudomonas sp. AZM-1……...... 42
3. Multiple aligment Pseudomonas sp. AZM-1dengan berbagai strain mikroba
kekerabat... ... ... ... 43
4. Filogenetik tree nukleotida model real branch dan model cladogram... 48
5. Jarak genetik berdasarkan pada urut-urutan enzim DNAse I di GenBank dengan gen Pseudomonas spp...... 49
6. Kesejajaran jarak genetik Pseudomonas sp. sesuai urutan enzim DNAseI………….. 50
7. Hasil analisis penyejajaran sesuai urutan nukleotida fragmen gen yang diperoleh dengan transposon Tn5Km1 dari Pseudomonas sp.AZM-1 dengan yang dimiliki Pseudomonas spp lain pada data Gene Bank dengan menggunakan program Blast-n dan Blast-p... 51
Halaman 9. Hasil pengurutan nukleotida dengan primer T7... 40
10.Komposisi berbagai media pertumbuhan Pseudomonas sp.AZM-1..……...... 42
11.Multiple aligment Pseudomonas sp.AZM-1dengan berbagai strain mikroba
sekerabat... ... 43
12.Filogenetik tree nukleotida model real branch dan model cladogram... 48
13.Jarak genetik berdasarkan pada urut-urutan enzim DNAse I di GenBank dengan gen Pseudomonas sp Jarak Genetik berdasarkan pada urut-urutan nukleotida gen
DNAse I di GenBank dengan gen Pseudomonas sp.AZM-1 ...... 49
14.Kesejajaran jarak genetik Pseudomonas sp.AZM-1 sesuai urutan enzim DNAseI.... 50
15.Hasil analisis penyejajaran sesuai urutan nukleotida fragmen gen yang diperoleh dengan transposon Tn5Km1 dari Pseudomonas sp.AZM-1 dengan yang dimiliki Pseudomonas spp lain pada data Gene Bank dengan menggunakan program Blast-n dan Blast-p... 51
Salah satu penyebab sulitnya melakukan peningkatan produksi pangan yang
cepat dan mencapai swasembada nasional adalah karena banyak hambatan dalam
pengembangan lahan pertanian pangan yang baru. Pembukaan lahan subur
pertanian pangan baru masih terbatas dan tidak berimbang dengan konversi lahan
pertanian produktif menjadi fungsi lain seperti pemukiman di pulau Jawa. Konversi
lahan pertanian ke industri dan pemukiman di Jawa mencapai 62.000 ha/tahun,
sementara itu pemerintah melalui Deptan RI hanya menggantinya dengan membuka
sawah baru seluas 18.000 ha/th. Padahal untuk mencapai swasembada pangan di
Indonesia saat ini, sekurangnya diperlukan tambahan luas tanam 1 juta hektar yang
berarti harus membuka lahan baru dan meningkatkan produktivitasnya. Sangat
ironis jika melihat potensi lahan di Indonesia yang tersedia untuk pertanian masih
besar dan belum dimanfaatkan secara optimal.
Membuka dan mencetak lahan baru untuk pertanian pangan di luar pulau Jawa
melalui pengembangan lahan yang potensial untuk pertanian adalah solusi
kecukupan pangan jangka panjang. Indonesia mempunyai lahan tidur (alang-alang)
seluas 1,08 juta ha, lahan pasang surut potensial seluas 9,5 juta ha dan lahan
marginal kering 11 juta ha. Bahkan dilaporkan oleh Suhartanto (2006), bahwa di
Indonesia sebenarnya terdapat 45,79 juta ha tanah Podzolik Merah Kuning
(PMK/Ultisol), 14,11 juta Ha tanah Oxisol dan 27,06 juta ha Gambut (histosol)
yang merupakan potensi lahan yang bisa diupayakan untuk pertanian dan potensi
lahan yang sesuai untuk sawah di Indonesia ada sekitar 23 juta hektar. Jenis tanah
masam menempati 29,7 % dari luas total daratan Indonesia atau sekitar 90 juta
hektar dengan tanah PMK menempati urutan teratas dalam hal luasannya.
Jutaan hektar lahan di atas kurang menarik untuk diupayakan sebagai lahan
pertanian oleh karena banyaknya faktor pembatas produktivitas fisik dan kimia
cara-cara konvensional cukup mahal, seperti pemberian kapur/dolomit untuk
menetralisir pH. Penambahan pupuk fosfat (P) secara berkelebihan harus dilakukan
pada lokasi ini agar unsur tersebut tersedia dan dapat diambil oleh tanaman.
Disamping itu, kekurangan hara dan jasad renik penyubur di dalam tanah juga
menjadi faktor pembatas produksi petani sehingga produktivitas tanaman budidaya
yang dicapai rendah, seperti kedelai: 0,5 - 0,8 ton/ha, padi: 1,5 – 3,0 ton/ha, dan
jagung: 2 – 3 ton/ha yang masih jauh di bawah potensi genetis komoditas ini.
Kekurangtersediaan hara fosfat (P) menurunkan hasil panen biji-bijian sampai
35%, bahkan lebih mirip gejala cekaman fisik seperti kekurangan air. Permasalahan
fisiologis yang ditunjukkan oleh tanaman jika kekurangan unsur fosfat ini adalah
terhambatnya metabolisme sel tanaman karena energi aktivasi sel yang terbatas
sehingga ekspresinya akan menghambat tumbuhnya tunas secara normal, tanaman
kerdil, daun tumbuh tidak normal, hijau ungu tua dan cepat kuning yang pada
gilirannya menurunkan mutu dan jumlah/bobot hasil panen (Panday 1997)
Melalui Bioteknologi dengan rekayasa genetik yang diterapkan pada mikroba
tanah atau tanaman, cekaman fisik di atas dapat diatasi sehingga tanaman dapat
normal memanfaatkan unsur fosfat di alam secara cukup meskipun dalam jumlah
ketersediaan yang terbatas (Mashar 2006). Beberapa mikroba penghasil enzim
pelarut fosfat yang telah dikenal seperti Pseudomonas sp., Alcaligenes sp., Bacillus
pumillus, Mycorhiza glamous, Aspergillus niger, dll. telah mulai banyak
dimanfaatkan peranannya baik dalam pertanian, industri pupuk (Prihatini et al.
1966) bahkan industri pengolahan pangan dan pakan ternak. Fosfat yang dilarutkan
menjadi tersedia untuk dapat diserap sel tanaman atau hewan. Enzim pelarut fosfat
disandikan oleh suatu gen tertentu. Guna lebih memanfaatkan gen mikroba di atas
maka isolasi gen penyandi pelarut fosfat khususnya dari bakteri Pseudomonas
dilakukan dalam penelitian ini.
Penelitian ini bertujuan untuk mengisolasi gen yang terlibat dalam pelarutan
fosfat dari bakteri Pseudomonas sp. AZM-1 melalui teknik transposon. Transposon
penanda resistensi terhadap kanamisin. Mini-Tn5Km1 merupakan salah satu tipe
transposon turunan dari transposon Tn5 (de Lorenzo et al. 1990). Transposon ini
dapat menyisip secara acak pada genom bakteri dan penyisipannya bersifat stabil
(Herrero et al. 1990). Penyisipan mini-Tn5Km1 ke dalam genom akan menyebabkan
bakteri tersebut memiliki sifat resisten terhadap kanamisin. Transposon mini Tn5
Km1 dalam penelitian ini digunakan sebagai pelacak dan penginaktivasi gen yang
terlibat dalam pelarutan fosfat pada bakteri Pseudomonas sp. AZM-1.
Tujuan Penelitian
Tujuan penelitian ini adalah :
1. Melakukan pengklonan fragmen DNA genom yang terlibat dalam proses
pelarutan fosfat melalui teknik mutagenesis dengan transposon.
Mobilitas Unsur Fosfat
Tanah asam adalah pembatas bagi pertumbuhan tanaman karena
menyebabkan ion Fe, Al, Mn, dan Ca akan mengikat kuat ion fosfat (P). pH tanah
berpengaruh terhadap bentuk ion P, jumlah dan tingkat dekomposisi bahan organik,
serta kegiatan jasad mikro (Brady 1974, Ismunadji et al. 1991). Toksik H+ lebih
mendominasi tanah lapisan atas dan ion hidrogen dari fosfat inorganic asam fosfat
(H3PO4) mengalami ionisasi membentuk ion H2PO4- , HPO42-, PO43- akan di ikat
menjadi Al-P, Fe-P dan Mn-P. Dalam keadaaan netral atau alkalin akan diikat
menjadi Ca-P (Marschner 1995; Ismunadji et al. 1991).
Alumunium (Al) di dalam tanah larut dan bersifat racun bagi tanaman.
Kelarutan Al menjadi tinggi pada kondisi asam. Pada kondisi asam Al lebih
mengikat ion fosfat dari H2PO4-, (HPO4)2- dan PO43-. Mula-mula ikatan tersebut
bersifat koloidal dan lambat, selanjutnya menjadi kristal varisit AlPO42H2- dan
strengit FePO42- (Nyakpa et al. 1988; Havlin et al. 1999). Alumunium
mendominasi ikatan dengan –P pada lapisan sub soil dan pada kondisi asam (pH
4.0) bentuk ikatan Al3+ adalah Al(H2O2)63+. Johnson dan Wood (1990)
menemukan kation Al3+ dapat mengikat PO43+ pada DNA sehingga menghambat
proses replikasi DNA bakteri bintil akar. Ketika pH meningkat, Al berada dalam
bentuk Al(OH)2+ dan Al(OH)2+ akan reaktif mengikat ion H2PO4-, HPO42- sebagai
unsur fosfat yang dapat diserap oleh tanaman. Ketika mendekati pH netral dalam
bentuk Al(OH)3, pada kondisi basa dalam bentuk Al(OH)4- dan akan berikatan
dengan PO43- membentuk komplek (Al(OH)4) 3 PO4 (Marschner 1995; Delhaize dan
Ryan 1995). Ion Al ini memiliki afinitas yang tinggi dan sangat menganggu
terhadap biomolekul anionik seperti asam lemak, karbohidrat, protein dan asam
nukleat (Aniol 1984; de Lima & Copeland 1994; Kochian 1995; Sivaguru et al.
1999). Al juga dapat menimbulkan cekaman oksidatif dengan terbentuknya oksigen
Dalam tanah mineral, P terdiri atas dua bentuk yaitu P-organik (3,75% dari P
total) seperti fitin, fosfolipid, asam nukleat dan koenzim, dan P-inorganik (25 –
90%) dari P total) seperti Ca3(PO4)2 dan Al(OH)4)2H2PO4 (Sutedjo 1994;
Cosgrove 1967). Bahan-bahan penyumbang fosfat di tanah berasal dari pupuk
kandang, sisa tanaman, pupuk buatan dan mineral/batuan tanah. Asam fitat dan
fitin merupakan fraksi terbesar dari P-organik. Selain itu menurut Alexander
(1977) bahwa sel-sel mikroba sangat kaya dengan asam nukleat dan jika mikroba
itu mati maka asam nukleatnya siap untuk dimineralisasi.
Mikroba Pelarut Fosfat
Mikroba pelarut fosfat yang pertama dikomersialisasi adalah Fosfobacterin
dari bakteri Bacillus megaterium var.phosphaticum (Smith et al., 1965). Beberapa
jenis cendawan dilaporkan mampu melarutkan Al-P dan Fe-P seperti Aspergillus sp
dan Pinicillium (Das 1963), Sclerotium dan Fusarium (Alexander, 1978).
Banyak jenis bakteri pelarut P antara lain genus Pseudomonas, Bacillus,
Mycobacterium, Micrococcus, Flavobacterium, Bacterium, Citrobacter, dan
enterobacter (Premono 1994; Illmer et al. 1995). Bakteri Pseudomonas putida,
Citrobacter intermedium dan Serratia mesenteroides mampu meningkatkan
kelarutan batuan fosfat 6 – 19 kali lipat (Premono et al. 1991) sedangkan
Pseudomonas fluorescens dan P. putida mampu meningkatkan P terlarut pada
tanah asam sampai 50 % ( Premono, 1994). P. aeroginosa salah satu bakteri
pelarut fosfat, yang bersifat gram negatif. Hasil penelitian terhadap Pseudomonas
sp oleh Surya (2006) menunjukkan bahwa pertumbuhan optimal mikroba ini pada
pH 6 pada media modifikasi HKsuk dimana dalam 24 jam diperoleh populasi
maksimum 1.97 x 109 sel dengan laju pertumbuhan spesifik maksimum 0,5807 sel
per jam yang dicapai sampai pada jam ke-4 dalam kultur dan melambat setelah jam
ke-8.
Mekanisme pelarutan unsur P terikat oleh bakteri pelarut fosfat terjadi
trifosfomonoesterase dan fosfoamidase) yang terdapat di dalam tanah.
Enzim-enzim tersebut bertanggung jawab dalam hidrolisis bahan organik yang
mengandung fosfor menjadi fosfat organik (HPO4-2 dan HPO4-1) yang tersedia
bagi tanaman (Tabatabai 1982). Menurut Pikovskaya dalam Bora dan Bezbaruah
(1999) bahwa bakteri pelarut fosfat menghasilkan zona bening disekitar koloni
yang mengandung 20 gram batuan fosfat sebanding dengan 5 gram tricalcium
fosfat. Zona bening mengindikasikan fosfat terlarut yang disebabkan oleh aktivitas
enzim Fosfomonoesterase (FMEase). Fosfomono esterase merupakan enzim
hidrolase yang berperan dalam Penambahan H2O pada ikatan ester fosfat
(Lehninger 1995). Enzim FMEase dihasilkan secara dominan dibawah kondisi
ketersediaan fosfat rendah.
Enzim FMEase dapat dibagi menjadi FMEase asam dan FMEase alkali
tergantung jenis subtrat dan pH optimumnya. Dalam kisaran basa FMEase
intraseluler pada umumnya tinggi, sedangkan FMEase ekstraseluler tinggi pada
kondisi kisaran asam (Schinner et al., 1996). Sumber fosfat yang mampu
menginduksi produksi enzim FMEase ini adalah fenilfosfat dan p-nitrofenilfosfat
yang mengindikasikan kemampuan hidrolisis bentuk p-organik (Ruiz et al. 2000).
FMEase mengubah subtrat menjadi gugus alkohol (ROH) dan asam fosfat. Reaksi
hidrololisis p-nitrofosfat oleh FMEase sebagai berikut:
p-nitrofenilfosfat + H2O -- FMEaseÆ p-nitrofenol + H3PO4.
Selain oleh reaksi di atas, sumber fosfat organik adalah phytat (myo-inositol
1,2,3,4,5,6-hexakisphosphat) yang banyak tersimpan dalam jaringan tanaman
seperti sereal dan biji-bijian. Fitat diuraikan oleh phytase
(Myo-inositol-heksakisphosphate phosphohydrolase (EC 3.1.3.8)) menjadi inositol dan asam
orthophosphoric pertama ditemukan oleh Suzuki et al., 1907 (Wang, X., et al.,
pneumoniae XY-5. Beberapa jenis Bacillus memproduksi phytase yang
melarutkan fosfat dari Myo-inositol-heksakisphosphate.
Ekspresi gen promotor PhyC tergantung pada ketersediaan unsur fosfat dan
regulator PhoP yang penting dalam transkripsi gen PhyC. Ikatan PhoP~P pada
kotak PhoP mendorong transkripsi PhyC dan menandakan meningkatnya
konsentrasi PhoP~P. Letak posisi PhoP berada pada sekuen -35 dan – 30 sebelum
promotor PhyC. DNAse1 yang berada pada sekuen –80 dari lokasi PhyC bekerja
menguraikan ikatan PhoP~P menjadi PhoP yang kemudian berinteraksi dengan
promotor PhyC sehingga menjadi aktif melakukan transkripsi (Makarewicz et al.
2006).
Peran Transposon dalam Mutagenesis
Metode transposon merupakan salah satu metode yang sering digunakan
untuk analisis genetika molekuler pada berbagai macam bakteri (Voelker dan
Dybvig 1998) dan fungi (Firon et al. 2003). Transposon merupakan elemen DNA
yang dapat meloncat atau menyisip dari satu tempat ke tempat lainnya di dalam
DNA (Snyder & Champness 2003). Beberapa contoh penggunaan transposon
dalam teknik mutagenesis adalah seperti untuk mengidentifikasi gen yang terlibat
dalam virulensi bakteri Listeria monocytogenes pathogen pada manusia, analisis
penanda molukuler pada Bradyrhyzobium japonicum (Wahyudi, et al. 1998),
isolasi gen yang berperan dalam pembentukan magnet pada Magnetospirillum
magneticum (Wahyudi et al., 2001), pengklonan fragmen DNA genom yang
terlibat dalam toleransi terhadap asam dan alumunium pada B. Japonicum (Astuti
et al. 2006).
Transposon seperti mini Tn5 telah digunakan dalam mutagenesis dan
pengklonan DNA bakteri gram negatif (Bruijn & Lupski 1984) seperti pada
Eubacteria (de Lorenzo et al. 1990). Penggunaan Mini Tn5 transposon lebih
disukai karena frekuensi transposisinya tinggi, spesififas DNA target yang relatif
genom beberapa spesies bakteri (Berg & Ber 1983). Mini Tn5Km1 adalah salah
satu tipe turunan dari transposon Tn5 yang membawa gen penanda resistensi
kanamisin (de Lorenzo et al. 1990) sehingga penyisipannya ke dalam genom
bakteri menyebabkan bakteri tersebut menjadi resisten terhadap kanamisin.
Penyisipan transposon ini bersifat acak dan penyisipannya bersifat stabil pada
genom bakteri (Herrero et al. 1990).
Reiznikoff and Wiliam (2002) menyatakan bahwa mekanisme transposisi
transposon terjadi melalui beberapa tahap dengan pemotongan dan penyambungan.
Pertama ikatan Tnp 19 pb secara dimerisasi membentuk Tnp-DNA struktur
komplek synaptic, kemudian melengkung (flanking) kedua ujungnya membentuk
ikatan cincin dan lepas dari utas DNA sebagai donor. Selanjutnya cincin DNA
yang mengandung transposon menempel pada untai DNA target. Dengan kehadiran
ion Mg 2+ cincin DNA transposon mengudar dan masuk tersambung dengan DNA
target sehingga membentuk untaian DNA mutagenesis.
Bakteri tanah gram negatif seperti Pseudomonas dapat dimutasikan dengan
transposon. Mini transposons Tn5 memiliki situs gen resisten kanamisin,
kloramfenikol, streptomycin-spectinomycin, dan tetrasiklin yang digunakan sebagai
penanda seleksi pengklonan spesifik NotI yang diapit oleh terminal 19 pb urutan
Waktu dan Tempat Penelitian
Penelitiaan dimulai dari bulan April 2006 dan selesai pada bulan Juni 2007
di laboratorium mikrobiologi pangan Seafast Centre, di Laboratorium BIORIN
(Biotechnology Research Indonesian The Netherlands), Pusat Penelitian
Sumberdaya Hayati dan Bioteknologi, IPB.
Bahan
Bahan yang digunakan adalah Pseudomonas sp. AZM-1, E. coli S 17-1 (
pir) sebagai sel donor pembawa plasmid pUTmini-Tn5Km1 dalam transposon
mutagenesis Gambar 1).
Gambar 1. Peta plasmid pUT mini-Tn5Km1 (7.055 kb) yang berada
dalam sel E. coli S17-1 (λ pir), peta restriksi transposon
mini-Tn5Km1 (1.835 kb) yang membawa gen resistensi
Media Pikovskaya (Bora dan Bezbaruah, 1999) digunakan sebagai media
dasar seleksi Pseudomonas sp. AZM-1 yang resisten terhadap antibiotik. Media LB
(Tripton 10 g/L, NaCl 10 g/L, ekstrak khamir 5 g/L, agar-agar 15 g/L yang
ditambahkan kanamisin 50 μg/ml) digunakan untuk menumbuhkan E. coli.
Primer hulu KmO (5’-ACACTGATGAATGTTCCGTTG-3’) dan primer
hilir KmI (5’-ACCTGCAGGCATGCAAGCTTC-3’) digunakan untuk inverse
PCR, E. coli DH5α dan plasmid pGEMT-Easy digunakan sebagai vektor
pengklonan (Gambar 2.)
Gambar 2. Peta plasmid pGEMT-Easy
Metode Penelitian
Penelitian ini dilakukan secara bertahap yaitu isolasi bakteri pelarut fosfat
dari pupuk hayati Bio P 2000 Z, seleksi dan pemurnian bakteri pelaruf fosfat,
mutasi bakteri pelarut fosfat, seleksi yang tidak dapat melarutkan fosfat,
pengklonan gen yang disisipi transposon, isolasi gen sasaran dan karakterisasi gen
E. coli
S 17-1 (λ pir)
Mutan Bakteri
Pengklonan Gen MUTAGENESIS diparental matting
Gen Sasaran
Karakterisasi gen Sasaran
Gambar 3. Tahapan isolasi, pengklonan dan karakterisasi fragmen gen yang terlibat dalam pelarutan fosfat dari Pseudomonas sp. AZM-1.
Isolasi bakteri pelarut fosfat. Mikroba yang membawa sifat pelarut fosfat
dari pupuk hayati dengan media seleksi pikovskaya (Bora dan Bezbaruah, 1999;
Hesham, 2004), kemudian mikroba diidentifikasi. Hasil identifikasi mikroba Seleksi bakteri
pelarut fosfat
pelarut phosfat yang didapat adalah Pseudomonas sp. AZM-1. Kemudian
dimurnikan dan diperbanyak untuk diliofilisasi agar terjaga kemurniannya.
Sebelum bahan mikroba digunakan untuk penelitian, dilakukan peremajaan
kembali dengan menginokulasikan koloni yang ditumbuhkan dari ampul liofilisasi
ke dalam plate agar pikovskaya. Kondisi inkubasi dilakukan pada suhu ruang
selama 24 jam, kemudian bakteri ditambahkan pada cawan Pikovskaya selama 4-5
jam untuk mendapatkan fase logaritmatiknya (Surya, 2006). Selanjutnya
diinokulasikan pada media (1) Pikovskaya + Chl 50 µg/ml, (2) Pikovskaya +
kanamisin (Km) 50 μg/ml, (3) Pikovskaya + ampisilin (Ap) 50 μg/ml, dan (4)
Pikovskaya saja sebagai kontrol positif terhadap resistensi antibiotik.
Mutagenesis dengan Transposon. Mutasi Pseudomonas sp.AZM-1 menjadi tidak dapat melarutkan fosfat dilakukan dengan menyisipkan transposon
mini-Tn5Km1, yang terdapat di dalam plasmid pUTmini-Tn5Km1 (Gambar 2) yang
dibawa E. coli S 17-1 (λ pir) ke dalam genom Pseudomonas sp. AZM-1 melalui
metode diparental mating. E. coli S 17-1 (λ pir) disebut sebagai donor (D) dan
Pseudomonas sp. AZM-1 disebut sebagai resipien (R). R ditumbuhkan pada 50 ml Pikovskaya dalam erlenmeyer 250 ml di dalam inkubator bergoyang dengan
kecepatan 140 rpm, pada suhu ruang selama 12 jam. D ditumbuhkan pada media
50 ml LB yang mengandung kanamisin 50 μg/ml dalam erlenmeyer 250 ml di
dalam inkubator bergoyang dengan kecepatan 140 rpm, suhu 370C selama 18-20
jam.
Sel R dicuci dan diresupensi dalam garam fisiologis (NaCl 0.85%) sebanyak
3 kali dengan cara disentrifugasi selama 10 menit pada 17500 g. Supernatan yang
didapat dibuang, dan pelet dicuci dengan garam fisiologis, demikian pula bagi sel
D, namun sel D disentrifugasi pada 800 g selama 5 menit.
Pelet sel R yang telah dipanen ditambahkan LB sebanyak 40µl. Suspensi sel
R kemudian dipindahkan ke dalam tabung mikro 1.5 ml yang berisi pelet sel D.
Suspensi campuran D dan R kemudian diresuspensi perlahan dan dipindahkan ke
dilakukan selama 12, 18, dan 24 jam. Pelet sel donor maupun resipien juga
diletakkan di atas membran milipore sebagai kontrol negatif. Pada masing-masing
jam inkubasi yang telah ditentukan, membran milipore diangkat dan dimasukkan
ke dalam tabung mikro 1.5 ml yang berisi 1 ml garam fisiologis, kemudian dikocok
untuk melepaskan sel dari membran. Masing-masing 100 µl suspensi disebarkan di
atas media Pikovskaya (+ Chl 50 µg/ml + Km 50 µg/ml) dan diinkubasi selama
3-4 hari. Tahapan proses mutagenesis seperti ditunjukkan pada Gambar 3-4.
Gambar 4. Diagram skematis proses mutagenesis dengan transposon. Konjugasi dilakukan di atas filter milipore (0.45 µm) pada media LB. Pada suspensi campuran donor (D) dengan resipien (R) akan terjadi
serangkaian proses transfer dan penyisipan transposon.
Seleksi Mutan. Sel transkonjugan yang tumbuh pada masing-masing
cawan Pikovskaya + Chl 50 μg/ml + Km 50 μg/ml dibuat replikanya pada media
yang sama. Inkubasi dilakukan pada suhu 320C selama 12 jam. Koloni konjugan
yang tumbuh dengan tidak menunjukkan zona bening merupakan koloni
Pseudomonas sp. AZM-1 yang termutasi (mikroba tidak mampu melarutkan fosfat) sebagai mutan negatif pelarut fosfat. Selanjutnya setiap koloni mutan
dengan menggoreskan satu lup penuh koloni mutan dan diverifikasi juga melalui
pengamatan terhadap bentuk sel secara mikroskopis. Jika tidak terdapat
pertumbuhan zona bening dan verifikasi morfologi secara mikroskop memiliki
bentuk sel yang sama maka koloni Pseudomonas sp. AZM-1 tersebut digunakan untuk analisa lebih lanjut.
Isolasi DNA genom mutan Pseudomonas sp. AZM-1. Isolasi DNA genom dilakukan dengan menggunakan metode CTAB. Satu lup Pseudomonas sp.
AZM-1 dibiakkan di dalam 50 ml media King’s B (Lampiran 2.), pada suhu ruang
di inkubator bergoyang dengan kecepatan 60 rpm selama 24 jam. Sebanyak 50 ml
kultur sel mutan Pseudomonas sp. AZM-1 diambil dan dimasukkan ke dalam 2 tabung sentrifugasi 50 ml steril masing-masing 25 ml, kemudian disentrifugasi
selama 10 menit pada kecepatan 8500xg. Pelet yang didapat kemudian diresuspensi
dengan 250 µl bufer TE 1x (10 mM Tris HCL pH 8.0, 1mM EDTA), dan
dipindahkan ke dalam tabung mikro 1.5 ml steril. Kemudian disentrifugasi
kembali pada kecepatan 9000xg selama 10 menit, proses ini diulang 2-3 kali.
Suspensi kemudian ditambahkan 5 µl lisozim, tabung mikro 1.5 ml dibolak-balik
untuk mempercepat lisis sel. Selanjutnya, suspensi diinkubasi pada suhu 370C
selama 30 menit. Proses lisis sel dilanjutkan dengan menambahkan 500 µl SDS
10% dan proteinase K sebanyak 10 µl, tabung mikro 1.5 ml kemudian
dibolak-balik. Suspensi diinkubasi pada suhu 370C selama 60 menit. Sebanyak 80 µl NaCl
dan 100 µl CTAB 10% ditambahkan ke dalam suspensi, kemudian diinkubasi pada
suhu 650C selama 20 menit, tabung kembali dibolak-balik.
Purifikasi DNA dilakukan dengan menambahkan 650 µl fenol :
kloroform: isoamilalkohol (25:24:1). DNA dipisahkan dari debris sel dengan cara
disentrifugasi pada 13000xg selama 10 menit. Supernatan yang mengandung DNA
dipurifikasi dengan menambahkan 650 µl kloroform:isoamil alkohol (24:1) dan
selanjutnya disentrifugasi pada 13000xg selama 10 menit. Untuk pengendapan
DNA, supernatan yang didapat ditambahkan etanol absolut sebanyak 2 kali volume
supernatan dan sodium asetat 3M 10 % volume, pengendapan dibantu dengan
inkubasi di dalam mesin pembeku (-200C) selama 30 menit dan kemudian
dilakukan sentrifugasi pada 13000xg selama 15 menit. Pelet yang didapatkan
disentrifugasi (13000xg; 15 menit), fase supernatan dibuang sedangkan pelet
dikeringudarakan dengan cara membuka tutup tabung mikro 1.5 ml dan dibiarkan
selama beberapa jam (2-3 jam). Kemudian pelet DNA dilarutkan dalam 20 µl
ddH2O steril.
Pemotongan dan ligasi DNA yang mengandung mni-Tn5Km1. DNA
genom Pseudomonas sp. AZM-1 dipotong dengan EcoRV kemudian
potongan-potongan ini diligasikan kembali sehingga membentuk DNA linier seperti Gambar
6. Pemotongan DNA dilakukan dengan mencampurkan berturut-turut bufer
restriksi 2 µl, BSA 1 µl, dan enzim EcoRV 0.1 µl, kemudian campuran ditera
dengan ddH2O hingga volume akhir 20 µl. Campuran diinkubasi pada suhu 370C
selama 3 jam. DNA yang telah dipotong dengan enzim EcoRV, kemudian
diekstraksi dengan menggunakan metode ekstraksi fenol/kloroform. Proses
ekstraksi dilakukan dengan menambahkan 80 µl ddH2O pada tabung mikro 1.5 ml
yang berisi DNA hasil pemotongan dengan enzim EcoRV, kemudian ditambahkan
larutan fenol: kloroform: isoamilalkohol (25:24:1) satu kali volume DNA (100 µl).
Suspensi tersebut kemudian disentrifugasi pada 13000 xg selama 15 menit, fase
cair (aquosa) kemudian dipindahkan pada tabung mikro 1.5 ml baru dan steril.
DNA dipresipitasi dengan etanol absolut dan 0.1 volume sodium asetat 3 M, untuk
kemudian diinkubasi pada -200C selama 30 menit. DNA yang terpotong kemudian
dicuci dua kali dengan menggunakan etanol 70% dan disentrifugasi pada 13000 xg
selama 15 menit. Pelet DNA kemudian dikeringudarakan selama 2-3 jam. Pelet
DNA dilarutkan dalam 5 μL ddH2O steril.
DNA yang telah dipotong kemudian diligasi dengan menggunakan enzim
T4 DNA ligase sehingga membentuk molekul DNA sirkuler yang mengandung
mini Tn5Km1 yang diapit oleh primer Km O dan Km I. Ligasi dilakukan dengan
mencampurkan bufer ligase sebanyak 2 µl, DNA sebanyak 5 µl, enzim T4 DNA
ligase sebanyak 1 µl, campuran dilarutkan dengan ddH2O hingga volume reaksi 20
Amplifikasi DNA yang diapit mini-Tn5Km1. Fragmen DNA yang
diapit mini-tn5 diamplifikasi menggunakan primer Km(O) dan primer Km(I)
dengan menggunakan cetakan DNA yang telah diligasikan pada Eco RV sehingga
menjadi bentuk sirkuler. Fragmen sirkuler DNA yang mengapit transposon
mini-Tn5Km1 diamplifikasi dengan menggunakan mesin PCR dengsn komposisi PCR
adalah 1 l DNA hasil religasi, 1x buffer taq, 4 mM dNTP mix, 20 pmol primer
Km(0), 20 pmol primer Km(1), 4% DMSO, 0.25 U enzim taq DNA polymerase
(Generay) dan ddH2O sampai volume reaksi 20 l. Kondisi PCR adalah pra-PCR
95°C selama 3 menit, denaturasi 94°C selama 30 detik, penempelan primer 56°C
selama 30 detik dan pemanjangan 72°C selama 2 menit, siklus 35 kali, pasca-PCR
[image:32.612.100.477.184.733.2]72°C selama 5 menit.
Gambar 5. Skema proses amplifikasi fragmen DNA genom pengapit transposon.
a) DNA genom Pseudomonas sp. AZM-1 mutan non pelarut phosfat yang dipotong dengan enzim EcoRV, b) Proses sirkularisasi menggunakan enzim T4 DNA ligase, c) Proses amplifikasi DNA
genom pengapit transposon dengan inverse PCR. Km (I)
Km (O) Digesti dengan EcoRV
Self ligation (Circularization)
Pengklonan hasil amplifikasi fragmen DNA pengapit mini Tn5.
Pengklonan fragmen hasil PCR dilakukan dengan cara menyisipkan fragmen hasil
PCR yang telah dimurnikan pada pGEM®-T Easy. Hasil PCR diligasikan ke dalam
pGEM®-T Easy mengikuti petunjuk Promega. Sebelum diligasikan hasil PCR
dipurifikasi menggunakan Wizard® SV Gel & PCR Clean-up System (Promega,
USA). Metode purifikasi ini berperan dalam mengisolasi DNA dari gel agarosa
hasil elektroforesis. Setelah elektroforesis, gel yang berisi pita DNA dipotong
dan hasil cacahan dari gel tersebut dimasukkan ke dalam tabung mikro 1.5 ml. Ke
dalam tabung tersebut ditambahkan 10 l membrane binding solution per 10 mg
cacahan gel, lalu dikocok dan diinkubasi pada suhu 50-65oC sampai gel
benar-benar larut. Setelah larut, larutan dimasukkan ke dalam tabung penampung yang
dilengkapi dengan minikolom dan diinkubasi pada suhu ruang selama 1 menit.
Selanjutnya tabung penampung tersebut disentrifugasi pada kecepatan 10000 xg
selama 1 menit, cairan yang melewati minikolom dibuang dan mini kolom
dimasukkan kembali pada tabung penampung. Proses pencucian dilakukan
dengan menambahkan 700 l membrane wash solution yang mengandung etanol
dan disentifugasi pada kecepatan 10.000 xg selama 1 menit. Cairan dibuang dan
minikolom dimasukkan kembali pada tabung penampung.
Tahap pencucian diulang dengan menambahkan 500 l membrane wash
solution dan disentrifugasi kembali pada kecepatan yang sama selama 5 menit.
Untuk proses elusi, minikolom dipindahkan dengan hati-hati pada tabung mikro 1.5
ml yang baru dan ditambahkan 50 l air bebas nuklease pada minikolom. Lalu
diinkubasi pada suhu ruang selama 1 menit dan disentrifugasi pada kecepatan
10000 xg selama 1 menit. Minikolom dibuang dan DNA yang tertampung pada
tabung mikro 1.5 ml disimpan pada suhu 4oC atau –20oC.
DNA hasil purifikasi kemudian diligasikan dengan vektor plasmid
pGEM®-T Easy. Ligasi dilakukan dengan mencampurkan berturut-turut 3 l hasil
PCR, 1 l (10 ng) pGEM®-T Easy, 1 l (1U) T4 DNA ligase, 1 l 10x rapid buffer
30°C selama semalam. Plasmid rekombinan yang terbentuk kemudian
dintroduksikan ke dalam sel E. coli DH5α.
Transformasi E. coli DH5α. Hasil penyisipan produk PCR pada vektor
pGMT-Easy diintroduksikan ke dalam E. coli galur DH5α mengikuti metode yang
dipublikasikan Suharsono (2002) yang meliputi dua tahap yaitu pembuatan sel
kompeten dan transformasi genetik bakteri. (1) Pembuatan bakteri kompeten,
koloni tunggal E. coli DH5α dibiakkan dalam 2 ml media LB [1% bakto-tryptona,
0,5% bakto-ekstrak khamir, 1% (b/v) NaCl] pada suhu 37°C semalam dan
bergoyang 250 rpm. Sebanyak 100 l E. coli DH5α dibiakkan kembali dalam 10
ml LB pada suhu 37°C di inkubator bergoyang sampai densitas bakteri 4.107 –
7.107 sel/ml (OD600nm sama dengan 0.4 sampai 0.5). Sebanyak 1.5 ml kultur bakteri
disentrifugasi 3000 rpm pada suhu 4°C selama 10 menit, endapan bakteri
disuspensikan dengan larutan penyangga transformasi (TB) (10 mM PIPES, 15
mM CaCl2.2H2O, 250 mM KCl, 55 mM MnCl2.4H2O, pH 6.7) sebanyak 0.33μl
volume kultur bakteri dan diinkubasi di es selama 10 menit. Selanjutnya
disentrifugasi dengan kecepatan 3000 rpm pada suhu 4°C selama 10 menit,
endapan bakteri diresuspensi dengan TB sebanyak 1/12 volume TB awal, ditambah
3.3 μl DMSO dan diinkubasi di es selama 10 menit. (2) Transformasi bakteri,
sebanyak 50 μl bakteri kompeten dicampur dengan 10 μl hasil ligasi dan
diinkubasikan di es selama 20 menit. Selanjutnya diberikan kejutan panas 42 °C
selama 45 detik, ditambahkan 100 l media YT (16 g/l tripton, 10 g/l
bakto-ekstrak khamir, 5 g/l NaCl, pH 7.0) dan diinkubasi pada suhu 37 °C selama 20
menit. Bakteri disebar pada media Luria Bertani Agar (LA) yang mengandung 100
μg/ml ampisilin, 10 l 100 mM isopropiltio-β-galaktosida (IPTG) (Promega) dan
50 l 2%(b/v) X-gal (5-bromo-4-choloro-3-indolyl-β-D-galactoside) (Promega)
dan diinkubasi pada suhu 37°C semalam.
Seleksi transforman rekombinan. Bakteri E. coli DH5α yang mengandung
vektor rekombinan diseleksi menggunakan seleksi resistensi terhadap antibiotik
plasmid rekombinan) diverifikasi menggunakan koloni PCR dan pemotongan
plasmid rekombinan dengan enzim restriksi EcoRI. Komposisi koloni PCR adalah
1x buffer taq, 2 mM dNTP mix, 10 pmol primer Km(0), 10 pmol primer Km(1),
4% DMSO, 0.125 U enzim Taq DNA polymerase (Generay) dan ddH2O sampai
volume reaksi 10 l. Koloni PCR dilakukan dengan mengambil koloni putih
dengan tusuk gigi dan ditotolkan pada ddH2O pada tabung PCR dan dipanaskan
95°C selama 10 menit, ditambahkan komponen PCR lainnya dan dilakukan PCR
dengan rincian tahapan denaturasi 94°C selama 30 detik, penempelan primer 56°C
selama 30 detik dan pemanjangan 72°C selama 2 menit, siklus 30 kali dan
pasca-PCR 72°C selama 5 menit.
Isolasi plasmid. Plasmid rekombinan dipotong dengan mencampur 50 ng
plasmid rekombinan, 1x buffer EcoRI, 20 U enzim EcoRI (New England Bio Labs
Inc.) dan ddH2O sampai volume total 10 l, selanjutnya diinkubasi 37°C selama 3
jam.
Pengurutan nukleotida dan analisis bioinformatika. Pengurutan fragmen
DNA dilakukan dengan menggunakan DNA sequencer ABI Model 3100/3130
MERCIAN. Pengurutan DNA menggunakan primer T7 dan primer SP6. Primer
T7 terletak pada jarak sekitar 55 pb sebelum sisipan dan SP6 pada 140 pb setelah
sisipan. Hasil kedua sekuensing tersebut digabungkan untuk mencari urut-urutan
utuh sisipan dengan cara mencari daerah yang tumpang tindih. Situs EcoRV
dicari dari hasil pengurutan dan dijadikan sebagai ujung-ujung potongan sesuai
dengan letaknya di dalam genom Pseudomonas sp. AZM-1. Identifikasi urutan
basa nukleotida dan deduksi asam amino hasil pengurutan fragmen DNA
dilakukan dengan beberapa analisis. Analisis kesejajaran lokal (local alignment)
berdasarkan urutan nukleotida dan asam amino dilakukan dengan program
BLAST (Basic Local Alignment Search Tools) yang diakses dari NCBI (National
Center for Biotechnology Information) (http://www.ncbi.nlm.nih.gov/ BLAST/).
Urutan asam amino yang dideduksi dari urutan DNA menggunakan program
nukleotida untuk mencari open reading frame (ORF) menggunakan program
BESTORF (http://www.softberry/bestorf/htm). Analisis situs restriksi (restriction
site) fragmen DNA dilakukan dengan menggunakan program NEB Cutter
(http://www.firstmarket.com/ cutter/cut2.html). Analisis similaritas dan filogenetik
dari urutan nukleotida dan deduksi asam amino dari berbagai strain/species juga
dilakukan dengan menggunakan program BLAST (http://www.ncbi.nlm.nih.gov/
Skrining Pseudomonas sp. AZM-1 mikroba pelarut fosfat
Mikroba yang membawa gen pelarut fosfat adalah Pseudomonas sp. AZM-1.
Peremajaan kembali mikroba hasil isolasi dilakukan dengan menyebarkan ke dalam
plate agar pikovskaya pada inkubasi suhu ruang selama 24 jam (Gambar 6a),
kemudian digoreskan pada media pikovskaya di tabung reaksi selama 4-5 jam untuk
mendapatkan fase logaritmatiknya (Gambar 6b). Selanjutnya digoreskan pada media
Pikovskaya + Chl 100 µg/mL dan yang menunjukkan zona bening dipilih dan
dipindahsemaikan dalam cawan agar dan didapatkan koloni-koloni yang
menunjukkan zona bening (Gambar 6c). Hanya Pseudomonas sp. AZM-1 yang dapat
melarutkan fosfat dan resisten terhadap chloramfenikol yang digunakan untuk bahan
penelitian.
a b c
[image:37.612.101.520.95.788.2]
Gambar 6. Penampilan koloni Pseudomonas sp. AZM-1 pembawa gen pelarut
fosfat pada media phykovskaya: a. Peremajaan dari sel tunggal
Pseudomonas sp. AZM-1 hasil isolasi, b. Pertumbuhan fase
logaritmatik, c. Penampilan pertumbuhan murni pada media
pykovskaya menunjukkan zona bening.
Mutagenesis dengan Transposon dan Seleksi Mutan
Mutagenesis dengan transposon mini Tn5Km1 pada Pseudomonas sp. AZM-1
didapatkan dari hasil inkubasi selama 24 jam. Hasil suspensi sel transkonjugan yang
diinkubasi selama 24 jam menghasilkan kepadatan sel pada kisaran 106
- 108.
Frekuensi transkonjugasi yang terjadi tampak tinggi yaitu 1.4 x 10-4 dimana mutan
Pseudomonas sp. AZM-1 mampu hidup pada media pykovskaya agar yang telah
ditambahkan antibiotik. Namun, mutan non pelarut fosfat dari pertumbuhan hasil
sebaran tersebut masih belum menampakkan perbedaan antara koloni mutan
transkonjugan pelarut fosfat dengan mutan non pelarut fosfat karena koloni yang
terbentuk masih berhimpit antara Pseudomonas sp. AZM-1 mutan non pelarut
fosfat dengan dan mutan pelarut fosfat. Frekuensi transkonjugasi mutan negatif
pelarut fosfat yaitu 3 x 10-9 yang didapatkan dari pengenceran 10-9 yaitu diperoleh
tiga buah sel tunggal mutan non pelarut fosfat.
Gambar 7. Mutan Pseudomonas sp. AZM-1 padapikovskaya agar + Chl 50 µg/mL + Km 50 µg/mL yang diinkubasi selama 24 jam.
Koloni mutan transkonjugan hasil konjugasi ditapis ulang untuk mendapatkan
mutan negatif pelarutan fosfat yang diharapkan. Penapisan terhadap mutan
menunjukkan bahwa frekuensi mutan negatif pelarut fosfat adalah kecil. Bila mutan
ini tumbuh berhimpitan dengan tipe liar pelarut fosfat maka penampilan dalam
media pykovskaya lebih dominan membentuk zona bening. Untuk mendapatkan
koloni dari sel tunggal mutan negatif pelarut fosfat maka koloni mutan negatif
pelarut fosfat yang tidak menampakkan membentuk zona bening diencerkan dengan
faktor x 10-9. Dari penapisan ulang ini beberapa mutan negatif pelarutan fosfat
[image:39.612.101.503.58.792.2]
a b
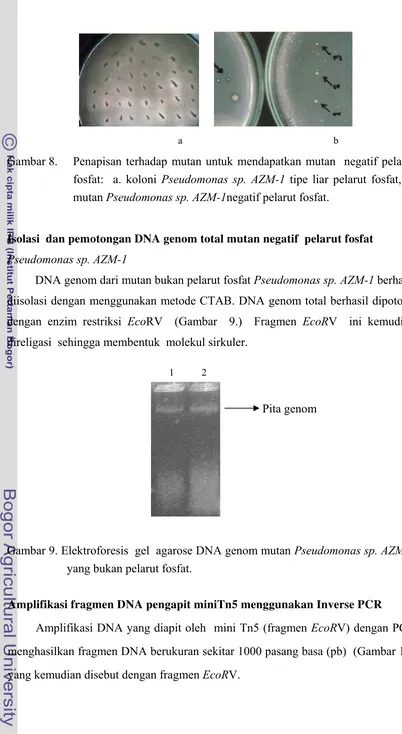
Gambar 8. Penapisan terhadap mutan untuk mendapatkan mutan negatif pelarut
fosfat: a. koloni Pseudomonas sp. AZM-1 tipe liar pelarut fosfat, b. mutan Pseudomonas sp. AZM-1negatif pelarut fosfat.
Isolasi dan pemotongan DNA genom total mutan negatif pelarut fosfat
Pseudomonas sp. AZM-1
DNA genom dari mutan bukan pelarut fosfat Pseudomonas sp. AZM-1 berhasil diisolasi dengan menggunakan metode CTAB. DNA genom total berhasil dipotong
dengan enzim restriksi EcoRV (Gambar 9.) Fragmen EcoRV ini kemudian direligasi sehingga membentuk molekul sirkuler.
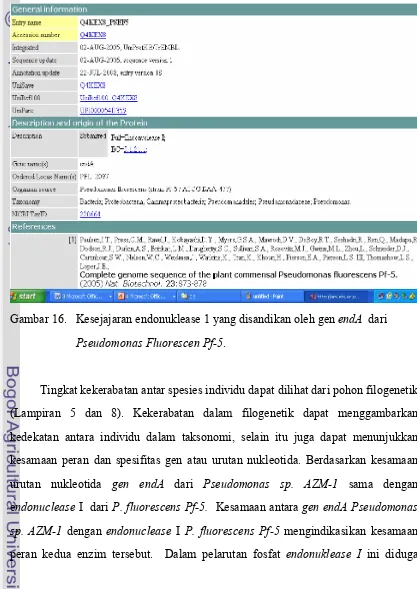
Gambar 9. Elektroforesis gel agarose DNA genom mutan Pseudomonas sp. AZM-1
yang bukan pelarut fosfat.
Amplifikasi fragmen DNA pengapit miniTn5 menggunakan Inverse PCR
Amplifikasi DNA yang diapit oleh mini Tn5 (fragmen EcoRV) dengan PCR
menghasilkan fragmen DNA berukuran sekitar 1000 pasang basa (pb) (Gambar 10)
yang kemudian disebut dengan fragmen EcoRV.
Gambar 10. DNA hasil PCR yang diapit oleh mini TN5 (1=sampel; 2=penanda
ukuran 1 kb plus).
Pengklonan dan hasil amplifikasi fragmen DNA pengapit miniTn5
Amplikon fragment yang diapit miniTn5 disisipkan pada pGEM®-T Easy dan kemudian diintroduksikan pada E. coli DH5α. Keberhasilan penyisipan (ligasi) dan
transformasi diketahui melalui seleksi biru putih dan resistensi bakteri terhadap
antibiotika ampisilin pada media tumbuh bakteri. E. coli yang dapat bertahan hidup
di media seleksi yang mengandung antibiotik ampisilin, X-gal dan IPTG berarti
mengandung plasmid, dan koloni E. coli yang berwarna putih di media seleksi
adalah E. coli yang mengandung plasmid rekombinan, sedangkan yang berwarna
biru adalah E. coli yang mengandung plasmid non-rekombinan (Gambar 11).
Gambar 11. Koloni-koloni E. coli DH5α berwarna biru (diduga tidak membawa plasmid rekombinan dan putih diduga membawa palsmid
rekombinan) pada media LA yang mengandung 100 μg/ml ampisilin,
10 l 100 mM isopropiltio-β-galaktosida (IPTG) (Promega) dan 50 l
2%(b/v) X-gal (5-bromo-4-choloro-3-indolyl-β-D-galactoside) (Promega) hasil inkubasi pada suhu 37°C semalam.
1 2
3000 pb 2000 pb 1500 pb 1000 pb 700 pb 500 pb
Koloni biru
1 2 3 4 5
1000 pb
Gen lacZ menyandi β-galactosidase (β-gal) yang mengubah substrat X-gal
yang tidak berwarna menjadi berwarna biru bila ada IPTG yang berfungsi
menginduksi promotor lac Z. Bila amplikon DNA yang diapit mini Tn5 menyisip
pada gen lacZ, maka gen lacZ tidak dapat diekspresikan sehingga koloni E. coli
berwarna putih. Bila tidak ada penyisipan pada lacZ, maka koloni yang terbentuk
akan berwarna biru.
Verifikasi koloni putih dilakukan menggunakan PCR koloni. Verifikasi
dilakukan untuk memastikan fragmen yang menyisip pada pGEM®-T Easy adalah fragmen yang diharapkan yaitu ukurannya sesuai dengan ukuran hasil amplifikasi
fragmen DNA pengapit miniTn5. Hasil verifikasi menunjukkan bahwa 4 koloni
yang berwarna putih yang diPCR menghasilkan pita yang ukurannya sesuai dengan
DNA sisipan (Gambar 12). Hasil ini menunjukkan adanya konsistensi hasil. Selain
itu terdapat satu koloni putih yang tidak membawa sisipan sebagai koloni putih
palsu. Adanya koloni putih palsu ini disebabkan oleh kemungkinan X-gal tidak
tersebar secara merata di media.
Gambar 12. Hasil PCR dengan koloni putih sebagai cetakan.
1=. penanda 1 kb plus; 2= koloni 1; 3= koloni 2;
4= koloni 3; 5= koloni 4
PCR koloni yang menghasilkan fragmen DNA yang sesuai dengan ukuran
yang diharapkan belum tentu hasil dari amplifikasi fragmen yang menyisip pada
650pb 350pb 3000pb
1 2 3
2000pb 1500 pb
1000pb 700 pb
yang memiliki kesamaan dengan DNA yang disisipkan. Oleh karena itu plasmid dari
koloni putih ini telah diisolasi, dan dipotong dengan EcoRI yang mengapit DNA
sisipan di situs pengklonan pGEM®-T Easy. Pemotongan dengan EcoRI
menghasilkan 3 fragmen DNA yang berukuran sekitar 3000 pb, 650 pb dan 350 pb.
Fragmen berukuran 3000 pb merupakan fragmen vektor pGEM®-T Easy, sedangkan gabungan fragmen 650 pb dan 350 pb merupakan fragmen sisipan (Gambar 13).
Gambar 13. Verifikasi palsmid rekombinan dengan pemotongan enzim restriksi
EcoRI (1= penanda 1 kb plus; 2= plasmid tidak dipotong; 3= plasmid dipotong dengan EcoRI).
Terpotongnya sisipan menjadi dua fragmen menunjukkan adanya tempat
pemotongan EcoR I pada sisipan tersebut. Jumlah ukuran fragmen sisipan sesuai
dengan amplikon fragmen DNA pengapit miniTn5 yaitu sekitar 1000 pb.
Pengurutan fragmen DNA pengapit miniTn5
Pengurutan DNA dengan primer T7 dan SP6 (Lampiran 1.) menghasilkan
urutan yang didalamnya terdapat primer Km O, Km I, EcoRI dan EcoRV. Primer
Km O terdapat pada urutan ke 47 sampai dengan ke 67 dan komplemen dari Km I
pada urutan ke 1032 sampai dengan urutan ke 1052 dengan pengurutan basa primer
terbalik, EcoRI pada urutan nukleotida ke 39, 707 dan ke 1065 serta tempat
DNA sisipan dimulai dari awal primer Km O sampai dengan akhir komplemen
dari primer Km I, sehingga DNA sisipan berukuran 1006 pb (Gambar 14). Adanya
tiga situs EcoRI menunjang hasil pemotongan plasmid rekombinan dengan EcoRI
yang menghasilkan tiga fragmen, fragmen 3000 pb yang merupakan vektor pGEM®
-T Easy dan dua fragmen sisipan yang masing-masing berukuran 668 pb dan 358 pb.
Analisis Kesamaan Gen Sisipan Dengan Data Gen di Genbank
Urutan gen yang diinaktifkan oleh penyisipan transposon pada mutan
Pseudomonas sp. AZM-1 adalah nukleotida setelah primer Km O dan sebelum
primer komplemen Km I dan Eco RV terletak di kedua sisi mini Tn5. Adanya
ligasi pada situs Eco RV dan amplifikasi dengan Km O dan Km I menyebabkan
situs Eco RV terletak diantara Km O dan Km I (Gambar 17). Olah sebab itu urutan
yang sesungguhnya di dalam Pseudomonas sp. AZM-1 EcoRV terletak di kedua
ujung Km O dan Km I diapit oleh Eco RV. Karena EcoRV memotong GATATC
diantara T dan A dan primer Km O dan Km I adalah bagian mini Tn5 , maka
urutan nukleotida yang diinaktifasi oleh Tn5 adalah ATC - sebelum Km I – setelah
Km O – GAT yang berukuran 964 pb (Gambar 15).
Gambar 15. Urutan gen sesungguhnya yang diinaktivasi oleh mini-Tn5
Analisis kesejajaran lokal (Lampiran 3, 4, 6 dan 7.) menunjukkan bahwa
fragmen EcoRV ini memiliki tingkat kesamaan lokal yang sangat tinggi yaitu 100
% dengan nukleotida pada genom Pseudomonas fluorescens Pf-5 (UNIPROT:
1 atcaccctggacagcgcgatcagcccagtgttctgggtaaaggcgttgta
51 gggaaagctgttgaacaggccgccttttgccctgctgttgctgtttctcg 101 cctgttccgcccaagccgatgcgccccgcaccttcagcgaagccaagaaa 151 gtcgcctggaagctctacgccccccaatccaccgagttctattgcggctg 201 caaatacaacggcaaccgggtgaacctggcggcctgcggctatgttccgc 251 gcaagaacgccaagcgcgccgaacgcattgaatgggagcatatagtgccg 301 gcctggcagatcggccatcagcgccagtgctggcaacaaggcggtcgcaa 351 gaactgcacacgcaccgattcggtctatcagcgcgccgaggccgacctgc 401 acaatctggtaccgagcattggcgaagtgaatggtgaccgcagcaatttc 451 agcttcggctggctgccggaacaatcgggccagtacggttcgtgcctgac 501 ccaggtcgacttcaaggccaagaaggtcatgcctcgcccttcgattcgcg 551 ggatgatcgcccgcacctacttctacatgagcaagcagtacggactgcgc 601 ctttccaaacaggatcggcagctcttcgaagcctggaacaagacctatcc 651 agtacaggcctgggagcgccagcgcaatcagagcgtggcttgcgtgatgg 701 ggcgcggcaatgaattcgtcggcgcagtcaacctcaaggcttgtggctga 751 acgcagatcgcccgcgggcggcgggcgatcattgagcctatggaaacggg 801 cttactgcgcggcttgatgctcgatgaccgtgacttcgatgtcacgcttc 851 ttggccttgaccagcttttcggtcacttcattcttcttcgcctcggcctc 901 ggcctgggaagcaaacggaccgaccacgaccagacgcttgccgtccacgg
Q4KEX8 PSEPF), 87 % dengan Pseudomonas entomophila (UNIPROT: Q1IC29
PSEE4) dan 86% dengan Pseudomonas fluorescens PfO-1 (UNIPROT: Q3KEZ8
PSEF5). Pf-5 adalah gen endA (penyandi enzim endonuklease) sejenis gen endX,
[image:45.612.98.515.181.770.2]gen yang menyandikan enzim endonuklease ekstraseluler (DNAse1) (Gambar 16) .
Gambar 16. Kesejajaran endonuklease 1 yang disandikan oleh gen endA dari
Pseudomonas Fluorescen Pf-5.
Tingkat kekerabatan antar spesies individu dapat dilihat dari pohon filogenetik
(Lampiran 5 dan 8). Kekerabatan dalam filogenetik dapat menggambarkan
kedekatan antara individu dalam taksonomi, selain itu juga dapat menunjukkan
kesamaan peran dan spesifitas gen atau urutan nukleotida. Berdasarkan kesamaan
urutan nukleotida gen endA dari Pseudomonas sp. AZM-1 sama dengan
endonuclease I dari P. fluorescens Pf-5. Kesamaan antara gen endA Pseudomonas
sp. AZM-1 dengan endonuclease I P. fluorescens Pf-5 mengindikasikan kesamaan
memiliki peran yang sama dengan DNAseI yang berperan dalam ekspresi gen
promotor PhyC. DNAse1 berada pada sekuen – 80 dari lokasi PhyC bekerja
menguraikan ikatan PhoP~P menjadi regulator PhoP yang berada pada sekuen -35
dan -30 sebelum promotor PhyC, kemudian berinteraksi dengan promotor PhyC
sehingga menjadi aktif melakukan transkripsi (Makarewicz et al. 2006). Gambaran
filogenetik berdasarkan pada urutan nukleotida gen endA Pseudomonas sp. AZM-1.
[image:46.612.100.520.229.691.2]dari berbagai spesies ditunjukkan pada Gambar 17.
Gambar 17. Dendogram filogenetik berdasarkan urutan nukleotida menunjukkan
kesejajaran endonuklease Pseudomonas sp. AZM-1 dengan
Peta Enzim Restriksi Endonuklease Fragmen
Analisis situs pemotongan untuk enzim restriksi pada suatu fragmen DNA
penting untuk rekayasa genetika. Analisis situs restriksi terhadap fragmen EcoRV
menunjukkan bahwa fragmen tersebut tidak mengandung situs yang terdapat pada
[image:47.612.101.518.229.634.2]situs pengklonan (MCS= multi cloning sites) dari pGEM-T Easy kecuali EcoR1
(Gambar 18). Situs EcoRI terdapat di dalam fragmen DNA pengapit Tn5 sehingga
tidak dapat digunakan untuk mengeluarkan sisipan fragmen tersebut dari pGEM-T
Easy.
Gambar 18. Peta restriksi endonuklease1 (DNAse1) dari Pseudomonas sp. AZM-1
Urutan asam amino yang dideduksi dari DNA yang disisipi miniTn5
Deduksi urut-urutan asam amino dari DNA yang disipi oleh Tn5 yang
1 atcaccctggacagcgcgatcagcccagtgttctgggtaaaggcgttgta
51 gggaaagctgttgaacaggccgccgttttgccctgctgttgctgtttctc 101 gcctgttccgcccaagccgatgcgccccgcaccttcagcgaagccaagaa
S A Q A D A P R T F S E A K K 151 agtcgcctggaagctctacgccccccaatccaccgagttctattgcggct
V A W K L Y A P Q S T E F Y C G C 201 gcaaatacaacggcaaccgggtgaacctggcggcctgcggctatgttccg
K Y N G N R V N L A A C G Y V P 251 cgcaagaacgccaagcgcgccgaacgcattgaatgggagcatatagtgcc R K N A K R A E R I E W E H I V 301 ggcctggcagatcggccatcagcgccagtgctggcaacaaggcggtcgca P A W Q I G H Q R Q C W Q Q G G R 351 agaactgcacacgcaccgattcggtctatcagcgcgccgaggccgacctg K N C T R T D S V Y Q R A E A D L 401 cacaatctggtaccgagcattggcgaagtgaatggtgaccgcagcaattt H N L V P S I G E V N G D R S N 451 cagcttcggctggctgccggaacaatcgggccagtacggttcgtgcctga F S F G W L P E Q S G Q Y G S C L 501 cccaggtcgacttcaaggccaagaaggtcatgcctcgcccttcgattcgc T Q V D F K A K K V M P R P S I R 551 gggatgatcgcccgcacctacttctacatgagcaagcagtacggactgcg G M I A R T Y F Y M S K Q Y G L 601 cctttccaaacaggatcggcagctcttcgaagcctggaacaagacctatc R L S K K Q D R Q L F E A W N K A 651 cagtacaggcctgggagcgccagcgcaatcagagcgtggcttgcgtgatg R T Y F Y M S K Q Y G L R L S K Q 701 gggcgcggcaatgaattcgtcggcgcagtcaacctcaaggcttgtggctg D R Q L F E A W N K T Y P V Q A W 751 aacgcagatcgcccgcgggcggcgggcgatcattgagcctatggaaacgg E R Q R N Q S V A C V M G R G N 801 gcttactgcgcggcttgatgctcgatgaccgtgacttcgatgtcacgctt E F V G A V N L K A C G - - - - - - 851 cttggccttgaccagcttttcggtcacttcattcttcttcgcctcggcct 901 cggcctgggaagcaaacggaccgaccacgaccagacgcttgccgtccacg
[image:48.612.102.498.77.760.2]951 gtcaccacattggat
Gambar 19. Deduksi asam amino penyusun enzim DNAse1 berdasarkan kodon-kodon pada daerah ORF fragmen gen pada
Pseudomonas sp. AZM-1.
Asam amino yang menyusun endonuklease1Pseudomonas sp. AZM-1 adalah : Ala (A) 20 9.3%
Arg (R) 18 8.4% Asn (N) 12 5.6% Asp (D) 6 2.8% Cys (C) 9 4.2% Gln (Q) 17 7.9%
Glu (E) 11 5.1% Gly (G) 17 7.9% His (H) 3 1.4% Ile (I) 6 2.8%
Thr (T) 7 3.3% Trp (W) 7 3.3% Tyr (Y) 10 4.7% Val (V) 14 6.5%
Analisis Domain Fragmen DNA Pengapit Tn5
Analisis kesejajaran lokal asam amino dari fragmem EcoRV dari
Pseudomonas sp. AZM-1 dengan Endonuclease I dari P. fluorescens Pf-5
menunjukkan adanya kesamaan 100 % dimana sebanyak 215 urutan susunan asam
[image:49.612.95.523.68.790.2]amino yang terdapat pada fragmen EcoRV Pseudomonas sp. AZM-1 memiliki kesamaan urutan asam amino dari P. fluorescens Pf-5 yang diketahui bahwa urutan asam amino 1 - 229 adalah endonuclease I (P. fluorescens Pf-5 (YP_259205)) (Gambar 20).
Daerah konservatif yang memiliki nilai kesamaan tinggi tersebut terbentang
dari asam amino ke 35 (104:3) sampai 245 (748:3) sedangkan daerah terjaga yang
menunjukkan domain endonuclease 1 pada P. fluorescens Pf-5 ada pada asam
amino ke 15 – 229 (Gambar 21).
Gambar 21. Daerah terjaga yang menunjukkan domain endonuclease 1 pada P.
SIMPULAN
Fragmen DNA yangdiduga terlibat dalam pelarutan fosfat telah berhasil
diisolasi dan diklonkan ke dalam PGMT-Easy. Fragmen ini berukuran 1006 pb yang
mengandung kerangka baca (open reading frame) untuk 215 aa. Analisis kesejajaran
lokal berdasarkan nukleotida dan asam amino menunjukkan bahwa fragmen ini
mempunyai kesamaan 100 % dengan gen end A dari Pseudomonas fluorescens pf-5.
SARAN
Isolasi fragmen gen endA yang diperoleh dari penelitian ini merupakan langkah
awal untuk penelitian lebih lanjut pada keterlibatan endonuklease 1 (DNAse1) dalam
pelarutan fosfat pada bakteri Pseudomonas sp.AZM-1. Untuk mengkonfirmasi hasil
penelitian ini gen endA harus diekspresikan ke dalam bakteri bukan pelarut fosfat
sehingga dapat melarutkan fosfat. Selain itu, ekspresi gen endA dari bakteri pelarut
SIMPULAN
Fragmen DNA EcoRV dari P. aereus yang berukuran 1006 bp telah dianalisis.
Analisis kesejajaran lokal menunjukan bahwa fragmen ini mempunyai kesamaan 100%
dari bagian P. fluorescens Pf-5 yang merupakan gen endA.. Analisis kesejajaran dari aa
yang dideduksi dari fragmen EcoRV ini menunjukan adanya 215 asam amino dari
fragmen EcoRV yang memiliki kesamaan lokal sebesar 100% dengan bagian protein
yang dimiliki P. fluorescens PF-5.
SARAN
Isolasi gen endA yang diperoleh dari penelitian ini merupakan langkah awal
untuk penelitian lebih lanjut pada tingkat ekspresi. Bukti terjadinya inaktivasi pelarutan
fosfat pada P. aereus karena endonuklease1 DNAse1 diinaktivasi transposon mini
Tn5Km1 sebagai fenomena baru yang perlu pengkajian lebih lanjut terhadap mekanisme
DAFTAR PUSTAKA
Alexander. 1977. Introduction to Soil Mycrobiology. Cornel university, USA.
Aniol, A. 1984. Induction of aluminum tolerance in wheat seedlings by low doses of aluminum in the nutrien solution. Plant Physiol 123:223-227.
Astuti, R. I., Wahyudi, A.T., Mubarik, N.R. 2006 Clonning of Genomic DNA Fragment involved in Acic-Alimunium Tolerance in Bradyrhyzobium japonicum 38 Through Transposon Mutagenesis. J Mikrobiol Indones 1 : 35-39.
Berg DE, Ber CM. 1983. The prokaryotic transposable element Tn5. Biotechnology 1: 417-435.
Bora, I.P. and Bezbaruah. 1999. Rock Phospate Solubilizing Bacteria from Tea (Camellia sinensis) soil and Their Response to Certain Organophosphorus pesticides. Trop Ecol 40: 175-161.
Brady. 1974. The Natural and Properties of Soil. 10th Editions. Mc. Milan, New York.
Bruijn FJ, Lupski JR. 1984. The use of transposon Tn 5mutagenesis in the rapid generation of correlated physical and gebeticalmaps of DNA segments cloned into multicopy plasmids. A review. Gene 27: 131-149.
Co