HASIL DAN PEMBAHASAN
Analisis Mutasi Gen KRAS Menggunakan Metode HRM dan RFLP pada DNA Standar Sel Kultur
Analisis mutasi gen KRAS menggunakan metode HRM telah dilakukan terhadap DNA standar untuk mengetahui bentuk standar kurva DNA yang tidak mengalami mutasi/wild type dan DNA mutan. DNA wild type diisolasi dari sel kultur BT549 (Breast Tumor), DNA standar mutan diperoleh dari sel kultur A549 (Human Lung Carcinoma) yang diketahui memiliki mutasi homozigot pada kodon 12 (GGT>AGT), dan sel kultur HCT 116 (Human Colon Tumor) yang diketahui memiliki mutasi heterozigot pada kodon 13 (GGC>GAC) (Krypuy et al. 2006).
(a)
Gambar 10 Hasil analisis mutasi gen KRAS DNA standar menggunakan metode HRM. Ket (a) normalized graph dan (b) differential graph
BT549: Kontrol wild type
A549: Kontrol mutan homozigot kodon 12 HCT116: Kontrol mutan heterozigot kodon 13
Hasil analisis HRM DNA standar menunjukkan perbedaan bentuk kurva DNA wild type dan DNA mutan dalam bentuk normalized graph (Gambar 10a) dan differential graph (Gambar 10b). Bentuk kurva yang diperoleh pada analisis HRM menurut (Reed et al. 2007) berasal dari kurva penurunan intensitas fluoresen yang terjadi seiring dengan kenaikan suhu, akibat adanya perubahan dari bentuk DNA utas ganda menjadi DNA utas tunggal. Nilai fluoresen dalam kurva
0 20 40 60 80 100 78 80 82 84 N o rm a liz ed F lu o re sc en Temperatur (0C) -20 -15 -10 -5 0 5 78 80 82 84 N o rm a liz ed m in u s w ild t yp e Temperatur (0C) (b)
tersebut kemudian dinormalisasi dalam skala 0 – 100 menjadi bentuk kurva
normalized graph. Krypuy et al. (2006) menggunakan kurva differential graph
untuk menunjukkan perbedaan bentuk kurva yang lebih jelas antara DNA yang mengalami mutasi dengan DNA wild type. Kurva differential graph diperoleh dengan cara menjadikan kurva standar wild type sebagai kurva acuan yang bernilai 0. Nilai kurva standar mutan diperoleh dari hasil pengurangan nilai-nilai
normalized fluorescence yang diperoleh terhadap normalized fluorescence kurva
standar DNA wild type. Transformasi nilai fluoresen menjadi bentuk normalized
graph dan differential graph dilakukan secara terintegrasi di dalam software
HRM, sehingga bisa diperoleh data secara langsung dalam bentuk grafik. Untuk memudahkan interpretasi hasil analisis HRM selanjutnya hanya digunakan bentuk kurva differential graph.
Analisis HRM gen KRAS dilakukan terhadap produk hasil amplifikasi yang memiliki ukuran sekitar 92 pb. Ukuran panjang basa ini dipilih sesuai dengan hasil penelitian Krypuy et al. (2006) yang telah melakukan analisa HRM terhadap gen KRAS dengan menggunakan produk PCR sebesar 189 pb dan 92 pb. Hasil analisis HRM yang terbaik dengan tingkat sensitivitas data yang lebih tinggi diperoleh pada produk PCR dengan ukuran 92 pb. Hal ini diduga karena adanya perbedaan jumlah basa Guanin dan Sitosin pada kedua produk PCR tersebut. Produk PCR yang memiliki ukuran yang lebih besar, memiliki jumlah basa Guanin dan Sitosin yang lebih banyak, sehingga menyebabkan tingginya energi yang dibutuhkan untuk merubah DNA utas ganda menjadi utas tunggal akibat banyaknya ikatan rangkap tiga yang dimilikinya. Hal ini didukung pula oleh Liew
et al. (2004) yang menyatakan bahwa analisis genotyping untuk melihat
perbedaan pola mutasi akibat terjadinya perubahan satu basa menggunakan metode HRM diperoleh dengan menggunakan produk PCR yang berukuran kecil. Namun demikian, ukuran amplikon untuk mendapatkan bentuk kurva yang optimal sebaiknya diperoleh berdasarkan hasil optimasi.
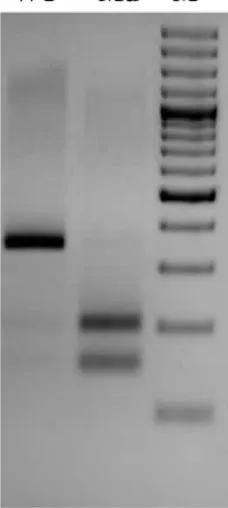
Hasil analisis mutasi gen KRAS menggunakan metode RFLP pada DNA standar menunjukkan perbedaan pita yang jelas (Gambar 11). Hasil analisis mutasi kodon 12 (Gambar 11a) menunjukkan bahwa DNA mutan kodon 12 (A549) memiliki ukuran sekitar 157 pb, sedangkan DNA wild type BT549 dan DNA mutan kodon 13 memiliki ukuran sekitar 128 pb. Hasil analisis mutasi kodon 13 (Gambar 11b) menunjukkan bahwa DNA mutan kodon 13 (HCT116) memiliki ukuran sekitar 157 pb, sedangkan DNA wild type BT549 memiliki ukuran sekitar 125 pb dan DNA mutan kodon 12 memiliki ukuran yang beragam.
(a) (b)
Gambar 11 Hasil analisis mutasi gen KRAS menggunakan metode RFLP. (a) kodon 12; (b) kodon 13 M A549 BT HCT M A549 BT HCT 1000 pb 500 pb 200 pb 100 pb 3000 pb 1000 pb 500 pb 200 pb 100 pb Mutan Wild type Mutan Wild type
Analisis RFLP gen KRAS kodon 12 dan 13 menggunakan enzim yang memiliki daerah pemotongan pada DNA wild type, sehingga DNA yang mengalami mutasi tidak terpotong. Analisis RFLP pada kodon 13 menggunakan primer forward yang dimodifikasi agar terjadi perubahan basa pada kodon 10 dan 11 (GGA GCA > GGC CCA) sehingga diperoleh sekuen yang dikenali oleh enzim restriksi BglI pada tipe wild type (Kimura et al. 2007). DNA yang mengalami mutasi pada kodon 13 tidak akan terpotong oleh enzim tersebut, sedangkan DNA yang mengalami mutasi pada kodon 12 terpotong pada beberapa titik oleh enzim BglI, sehingga menghasilkan beberapa pita pada hasil analisis gel elektroforesis.
Uji Sensitivitas Metode HRM dan RFLP dalam Analisis Mutasi Gen KRAS
Hasil uji sensitivitas metode HRM terhadap analisis mutasi gen KRAS pada kodon 12 dan 13 (Gambar 12) menunjukkan bahwa pada komposisi DNA mutan sebesar 6,25% kurva DNA mutan sudah mendekati garis kurva wild type. Sehingga batas sensitivitas yang menunjukkan kurva DNA mutan yang masih dapat diamati dengan jelas diperoleh pada konsentrasi DNA mutan sebesar 12,5%.
(a)
(b)
Gambar 12 Hasil uji sensitivitas analisis mutasi gen KRAS menggunakan metode HRM. (a) kodon 12; (b) kodon 13.
-12 -10 -8 -6 -4 -2 0 2 78 80 82 84 normali ze d m inus w il d ty pe Temperatur (0C) 100% mutan 50% mutan 25% mutan 12,5% mutan 6,25% mutan 3,125% mutan 1,625% mutan 0% mutan -14 -12 -10 -8 -6 -4 -2 0 2 78 80 82 84 Nor m al iz e d m inus w il d ty pe Temperatur (0C) 100% mutan 50% mutan 25% mutan 12,5% mutan 6,25% mutan 3,125% mutan 1,625% mutan 0% mutan
Hasi uji sensitivitas mutasi gen KRAS kodon 12 menunjukkan bahwa pada konsentrasi 100% DNA mutan nampak secara jelas kurva yang terbentuk mencapai suhu melting DNA optimum sekitar 82 0C. Ketika komposisi DNA mutan diturunkan dengan penambahan DNA wild type sebesar 50%, kurva
melting DNA bergeser ke sebelah kiri yang mirip dengan jenis mutasi heterozigot
pada analisis mutasi gen KRAS kodon 13. Hal ini menunjukkan bahwa dalam analisis mutasi gen menggunakan metode HRM keberadaan DNA wild type akan membentuk pola hasil analisa seperti bentuk mutasi jenis heterozigot. Oleh karena itu, hasil analisis HRM hanya dapat menunjukkan kemungkinan terjadinya variasi/mutasi pada sekuen DNA, tanpa diketahui letak terjadinya mutasi. Metode lain yang digunakan untuk mengetahui terjadinya mutasi umumnya adalah metode RFLP dan sekuensing DNA.
Hasil uji sensitivitas teknik RFLP menunjukkan bahwa terdapat perbedaan pita yang jelas antara DNA mutan dan DNA wild type. Hasil analisis gel elektroforesis pada produk pemotongan menggunakan enzim restriksi menunjukkan bahwa adanya perbedaan antara pita DNA mutan dan DNA wild
type masih nampak jelas pada komposisi DNA mutan sebesar 3,125%. Pita DNA
yang tipis dan cenderung tidak jelas nampak pada komposisi DNA mutan dibawah 3,125% (Gambar 13). Dengan demikian, dapat diketahui bahwa komposisi DNA mutan minimal yang terdapat dalam sampel sebesar 3,125%.
(a)
(b)
Gambar 13 Hasil uji sensitivitas metode RFLP dalam analisis mutasi gen KRAS. (a) kodon 12 dan (b) kodon 13. Ket A= 100% mutan, B= 50%:50%, C= 25%:75%, D= 12,5%:87,5%, E= 6,25%:93,75%, F= 3,125%: 96,875%, G= 1,625%: 98,375% dan H= 100% wild type.
300 pb 200 pb 100 pb 300 pb 200 pb 100 pb A B C D E F G H A B C D E F G H
Analisis Mutasi Gen EGFR menggunakan Metode HRM dan RFLP pada DNA Standar
Analisis mutasi gen EGFR dilakukan terhadap ekson 19 dan ekson 21. Mutasi ekson 19 terjadi akibat adanya delesi pada posisi kodon 746 sampai kodon 752. Jumlah kodon yang mengalami mutasi bervariasi pada setiap sampel, sehingga ukuran panjang basa pada produk amplifikasi akan bervariasi (Kawada
et al. 2008). Analisis HRM EGFR exon 19 menghasilkan data perbedaan pola
kurva yang nampak jelas antara DNA mutan dengan DNA wild type (Gambar 14a). Bentuk kurva DNA mutan menunjukkan adanya satu puncak dan satu lembah, sebagai akibat dari keberadaan alel mutan yang mempengaruhi produk hasil amplifikasi.
(a)
(b)
Gambar 14 Hasil analisis mutasi gen EGFR menggunakan metode HRM. (a) ekson 19 dan (b) ekson 21
HCT116: DNA standar wild type 55b : DNA standar mutan ekson 19
DNA standar 100% mutan ekson 21 L858R DNA standar 50% mutan ekson 21 L858R
Mutasi yang terjadi pada ekson 21 adalah susbtitusi satu pasang basa. Perubahan jenis basa pada sekuen DNA wild type menyebabkan terjadinya pergeseran kurva yang dihasilkan sesuai dengan jenis perubahan basa. DNA standar mutan yang digunakan memiliki mutasi kodon 858 T>G, sehingga akan menghasilkan melting DNA pada suhu yang lebih besar dibandingkan dengan
-8 -6 -4 -2 0 2 4 6 8 84 85 86 87 Nor m al ize d m inus nor m al Temperatur (0C) -10 -5 0 5 10 15 86,5 87,5 88,5 89,5 Nor m al ize d m inus nor m al Temperatur (0C)
DNA wild type (Gambar 14b). Kurva yang muncul pada DNA mutan 100% berbentuk satu puncak, sedangkan pada DNA mutan yang dicampur dengan 50% DNA wild type nampak terdapat kurva yang berbentuk satu lembah dan satu puncak. Hal ini terjadi karena pada DNA standar 100% mutan produk PCR yang terbentuk adalah 100% DNA yang mengalami mutasi, sedangkan pada komposisi DNA mutan 50% terdapat DNA wild type ikut teramplifikasi dan memepengaruhi kurva yang terbentuk.
Hasil analisis RFLP gen EGFR ekson 21 menunjukkan adanya pola yang jelas antara DNA mutan dengan DNA wild type. Mutasi kodon 858 ditandai dengan adanya pita yang berukuran sekitar 348 pb (Gambar 15b), sedangkan mutasi pada kodon 861 ditandai dengan adanya dua pita berukuran sekitar 200 pb dan 148 pb (Gambar 15c). Hasil ini sesuai dengan penelitian Kawada, et al. (2008).
(a) (b) (c)
Gambar 15 Hasil analisis mutasi gen EGFR menggunakan metode RFLP. (a) ekson 19, (b) ekson 21 kodon 858 dan (c) ekson 21 kodon 861. Ket: WT = wild type, Mu = mutan , M = marker
Analisis RFLP untuk mengetahui mutasi pada ekson 19 tidak dilakukan karena analisis elektroforesis gel terhadap produk hasil amplifikasi menghasilkan dua pita yang berukuran sekitar 254 pb dan 239 pb (Gambar 15a), sehingga DNA
wild type dan DNA yang mengalami mutasi dapat dibedakan dengan jelas.
Menurut Kawada et al. (2008) delesi yang terjadi pada gen EGFR ekson 19 dapat bervariasi sesuai dengan jumlah basa yang hilang, sehingga ukuran dua pita yang terbentuk akan bervariasi.
Uji Sensitivitas Metode HRM dan RFLP dalam Analisis Mutasi Gen EGFR
Hasil uji sensitivitas analisis mutasi gen EGFR menunjukkan bahwa pada komposisi DNA mutan sebesar 6,25%, bentuk kurva yang dihasilkan sudah mendekati bentuk kurva DNA wild type. Bentuk kurva DNA mutan yang masih dapat jelas berbeda dengan DNA wild type diperoleh pada komposisi DNA mutan sebesar 12,5%, sehingga batas sensitivitas metode HRM dalam analisis mutasi gen EGFR adalah 12,5% DNA mutan (Gambar 16). Bentuk kurva pada DNA
1000 pb 500 pb 300 pb 200 pb 100 pb WT Mu M WT Mu M 3000 pb 1000 pb 500 pb 300 pb 200 pb 100 pb WT Mu M 3000 pb 1000 pb 500 pb 300 pb 200 pb 100 pb
cetakan yang memiliki komposisi 100% DNA mutan berupa satu puncak dominan yang berbeda dengan DNA wild type. Bentuk kurva yang muncul ketika komposisi DNA mutan dikombinasikan dengan DNA wild type berubah menjadi berbentuk satu lembah dan satu puncak yang sama dengan bentuk hasil HRM exon 19.
Gambar 16 Hasil uji sensitivitas analisis mutasi gen EGFR menggunakan metode HRM.
Hasil uji sensitivitas teknik RFLP terhadap produk nested PCR exon 21 menggunakan enzim MscI menunjukkan adanya perbedaan pita yang jelas antara DNA mutan dan DNA wild type (Gambar 17). Pita DNA dengan komposisi 3,125% mutan masih menunjukkan pita berukuran sekitar 348 pb yang merupakan penanda adanya mutasi. DNA yang memiliki komposisi DNA mutan lebih rendah dari 3,125% DNA mutan sudah tidak menghasilkan pita yang berukuran sekitar 348 pb. Dengan demikian, batas sensitivitas metode RFLP dalam menganalisis mutasi gen EGFR adalah 3,125% DNA mutan.
Gambar 17 Hasil uji sensitivitas metode RFLP pada analisis mutasi gen EGFR. Ket: perbandingan DNA mutan dengan DNA wild type A= 100% mutan, B= 50%:50%, C= 25%:75%, D= 12,5%:87,5%, E= 6,25%: 93,75%, F= 3,125%:96,875%, dan H= 100% wild type. M= DNA marker.
Hasil uji sensitivitas metode HRM dan RFLP terhadap gen KRAS dan EGFR menunjukkan nilai sensitivitas yang sama. Metode HRM memiliki nilai
-8 -4 0 4 8 12 86,5 87,5 88,5 89,5 Nor m al iz e d m inuw w il d ty pe Temperatur (0C) 100% mutan 50% mutan 25% mutan 12,50% mutan 6,25% mutan 3,125% mutan 1,625% mutan 0% mutan A B C D E F G H M A B C D E F G H M A B C D E F G H 500 pb 300 pb 200 pb 100 pb Mutan Wild type
sensitivitas sebesar 12,5% dan metode RFLP memiliki nilai sensitivitas sebesar 3,125%. Batas sensitivitas kedua metode ini lebih tinggi dibandingkan dengan metode DNA sekuensing. DNA sekuensing membutuhkan minimal 30% DNA mutan di dalam sampel agar dapat menghasilkan data analisis yang jelas (Bosari
et al. 1995). Metode HRM dan RFLP dapat digunakan sebagai metode alternatif
yang lebih sensitif untuk menganalisis mutasi gen pada sampel yang memiliki banyak pengotor dari DNA wild type, DNA sel normal dan serat jaringan sel.
Isolasi DNA dari Sampel yang Disimpan dalam Kertas Saring
Hasil isolasi DNA dari setiap sampel kertas saring menunjukkan variasi yang sangat beragam (Lampiran 1). Konsentrasi DNA yang diperoleh berkisar antara 5,7 ng/µL - 369,2 ng/µL. Konsentrasi ini relatif sedikit, namun cukup untuk digunakan sebagai DNA cetakan dalam proses PCR. Semua sampel kertas saring menghasilkan DNA yang dapat diamplifikasi dengan primer gen KRAS dan gen
EGFR sehingga dapat diketahui termasuk jenis mutan atau wild type. Menurut
Hong Do et al. (2008) jumlah sampel DNA yang diperlukan sebagai DNA cetakan untuk terjadinya amplifikasi menggunakan metode HRM tidak harus besar. Konsentrasi DNA sebesar 1 ng/µL masih dapat menghasilkan pola analisis HRM yang masih bisa terbaca dengan baik. Hal ini didukung pula oleh hasil penelitian Pang et al. (2012) yang menyatakan bahwa kuantitas DNA tidak memiliki pengaruh yang signifikan terhadap hasil analisis genotyping. Hasil penelitian ini membuktikan bahwa kertas saring dapat dijadikan sebagai media penyimpanan sementara sampel cairan pleura sebelum digunakan dalam analisis molekuler.
Analisis HRM dan RFLP Cairan Pleura Pasien Kanker Paru yang Disimpan dalam Kertas Saring
Analisis mutasi gen KRAS
Hasil analisis mutasi gen KRAS terhadap 63 sampel pasien kanker paru menunjukkan hasil yang cukup signifikan. Satu pasien mengalami mutasi pada kodon 12. Sebanyak 62 sampel lainnya memiliki profil mutasi gen KRAS wild
type. Mutasi gen KRAS pada kodon 13 tidak ditemukan. Hasil analisis ini
diperoleh berdasarkan kemiripan bentuk kurva pada DNA sampel dengan bentuk kurva DNA standar pada analisis HRM dan keberadaan pita penanda adanya mutasi pada analisis RFLP.
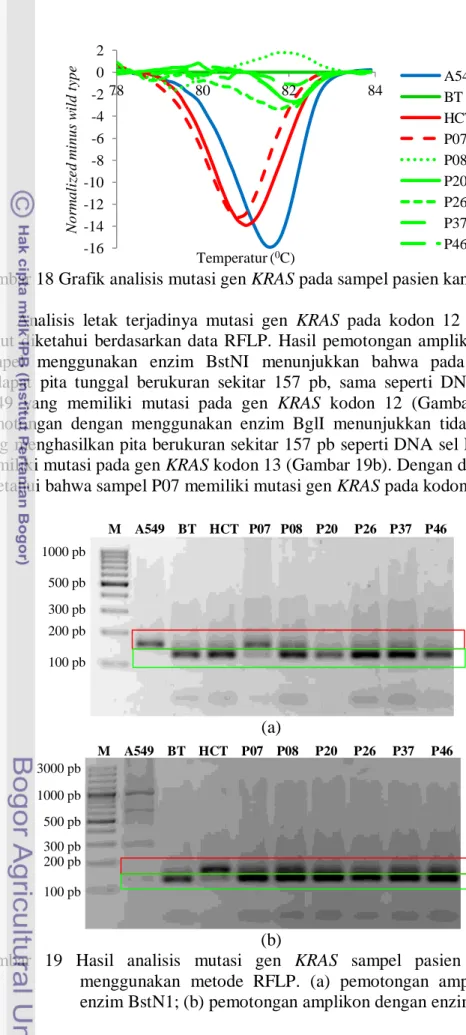
Hasil analisis HRM menunjukkan terdapat kemiripan bentuk kurva pada pasien P07 dengan bentuk kurva standar mutan (Gambar 18). Bentuk kurva P07 memiliki pola yang sama dengan kurva DNA sel mutan HCT116. Bentuk kurva ini berbeda dengan beberapa sampel lain seperti sampel P08, P20, P26, P37, dan P46 yang menunjukkan kemiripan dengan DNA wild type. Berdasarkan hasil analisis tersebut dapat disimpulkan bahwa sampel P07 memiliki mutasi gen KRAS kodon 13. Kesimpulan ini belum bisa digunakan, karena analisis HRM hanya bisa digunakan untuk melihat adanya variasi atau mutasi tanpa mengetahui posisi terjadinya mutasi. Kurva yang terbentuk disebabkan adanya perubahan satu pasang basa pada untai DNA P07 yang mirip dengan DNA mutan (Wittwer 2009).
Gambar 18 Grafik analisis mutasi gen KRAS pada sampel pasien kanker paru
Analisis letak terjadinya mutasi gen KRAS pada kodon 12 atau 13 lebih lanjut diketahui berdasarkan data RFLP. Hasil pemotongan amplikon gen KRAS sampel menggunakan enzim BstNI menunjukkan bahwa pada sampel P07 terdapat pita tunggal berukuran sekitar 157 pb, sama seperti DNA standar sel A549 yang memiliki mutasi pada gen KRAS kodon 12 (Gambar 19a). Hasil pemotongan dengan menggunakan enzim BglI menunjukkan tidak ada sampel yang menghasilkan pita berukuran sekitar 157 pb seperti DNA sel HCT116 yang memiliki mutasi pada gen KRAS kodon 13 (Gambar 19b). Dengan demikian dapat diketahui bahwa sampel P07 memiliki mutasi gen KRAS pada kodon 12.
(a)
(b)
Gambar 19 Hasil analisis mutasi gen KRAS sampel pasien kanker paru menggunakan metode RFLP. (a) pemotongan amplikon dengan enzim BstN1; (b) pemotongan amplikon dengan enzim BglI.
-16 -14 -12 -10 -8 -6 -4 -2 0 2 78 80 82 84 Nor m al iz e d m inus w il d ty pe Temperatur (0C) A549 BT HCT P07 P08 P20 P26 P37 P46 M A549 BT HCT P07 P08 P20 P26 P37 P46 M A549 BT HCT P07 P08 P20 P26 P37 P46 3000 pb 1000 pb 500 pb 300 pb 200 pb 100 pb 1000 pb 500 pb 300 pb 200 pb 100 pb Mutan Wild type Wild type Mutan
Analisis mutasi gen EGFR
Hasil analisis mutasi gen EGFR terhadap 63 sampel pasien kanker paru menunjukkan sebanyak lima sampel mengalami mutasi pada ekson 19 yaitu sampel P16, P18, P19, P42 dan P52, dan sebanyak lima sampel yaitu sampel P13, P37, P43, P48, dan P53 mengalami mutasi pada ekson 21 kodon 858. Mutasi gen
EGFR ekson 21 pada kodon 13 tidak ditemukan. Sebanyak 53 sampel lainnya
memiliki profil gen EGFR wild type (Lampiran 2).
Hasil analisis HRM mutasi EGFR ekson 19 menunjukkan adanya kemiripan bentuk kurva DNA sampel yang mengalami mutasi dengan bentuk kurva DNA standar mutan yang dapat diamati dengan jelas (Gambar 20). Sampel yang mengalami mutasi memiliki bentuk kurva yang sama dengan bentuk kurva DNA standar mutan (55b), sedangkan pada sampel yang wild type selain bentuk kurva tidak menunjukkan satu lembah dan satu puncak seperti bentuk kurva DNA standar mutan, letak kurva juga hampir mendekati DNA standar wild type.
Gambar 20 Grafik analisis mutasi gen EGFR ekson 19 pada sampel pasien kanker paru.
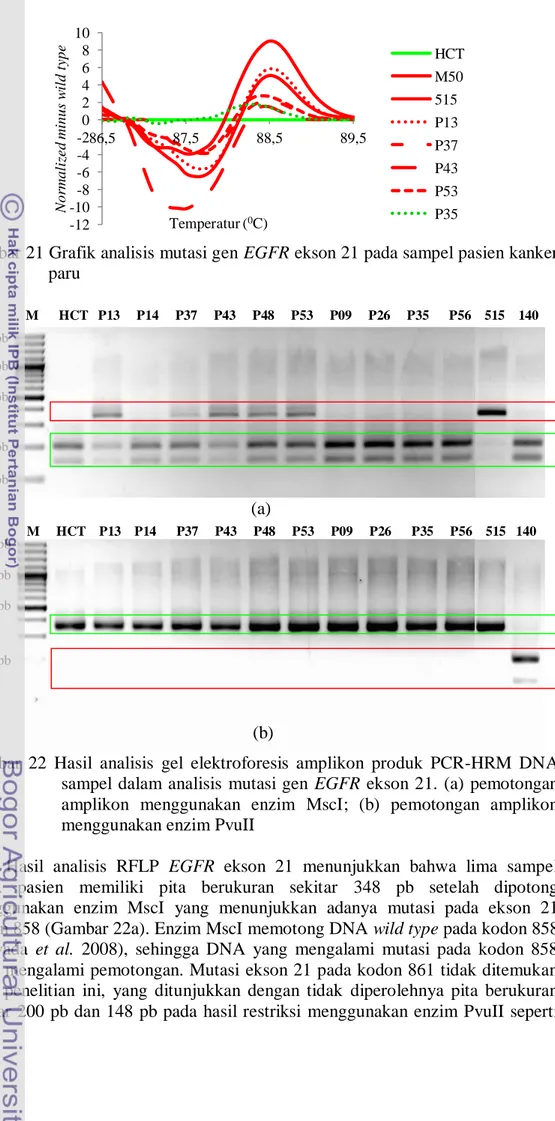
Hasil analisis mutasi gen EGFR ekson 21 menggunakan metode HRM menunjukkan adanya kemiripan bentuk kurva DNA sampel yang mengalami mutasi dengan bentuk kurva DNA standar mutan (Gambar 21). Sampel yang mengalami mutasi memiliki bentuk kurva yang mirip dengan kurva DNA standar mutan dan sampel yang wild type memiliki bentuk kurva yang mirip DNA standar
wild type serta letaknya hampir berimpitan. DNA standar mutan yang digunakan
adalah DNA sintetik dengan konsentrasi 50% mutan (M50) dan sampel sitologi pleura (515) yang telah diketahui memiliki mutasi pada kodon 858 berdasarkan hasil sekuensing. -8 -6 -4 -2 0 2 4 6 8 10 84 85 86 87 Nor m al iz e d m inus w il d ty pe Temperatur (0C) HCT 55b P16 P42 P10 P38 P58
Gambar 21 Grafik analisis mutasi gen EGFR ekson 21 pada sampel pasien kanker paru
(a)
Gambar 22 Hasil analisis gel elektroforesis amplikon produk PCR-HRM DNA sampel dalam analisis mutasi gen EGFR ekson 21. (a) pemotongan amplikon menggunakan enzim MscI; (b) pemotongan amplikon menggunakan enzim PvuII
Hasil analisis RFLP EGFR ekson 21 menunjukkan bahwa lima sampel DNA pasien memiliki pita berukuran sekitar 348 pb setelah dipotong menggunakan enzim MscI yang menunjukkan adanya mutasi pada ekson 21 kodon 858 (Gambar 22a). Enzim MscI memotong DNA wild type pada kodon 858 (Kawada et al. 2008), sehingga DNA yang mengalami mutasi pada kodon 858 tidak mengalami pemotongan. Mutasi ekson 21 pada kodon 861 tidak ditemukan pada penelitian ini, yang ditunjukkan dengan tidak diperolehnya pita berukuran sekitar 200 pb dan 148 pb pada hasil restriksi menggunakan enzim PvuII seperti
-12 -10 -8 -6 -4 -2 0 2 4 6 8 10 86,5 87,5 88,5 89,5 Nor m al iz e d m inus w il d ty pe Temperatur (0C) HCT M50 515 P13 P37 P43 P53 P35 M HCT P13 P14 P37 P43 P48 P53 P09 P26 P35 P56 515 140 3000 pb 1000 pb 500 pb 200 pb 100 pb (b) M HCT P13 P14 P37 P43 P48 P53 P09 P26 P35 P56 515 140 3000 pb 1000 pb 500 pb 200 pb Wild type Wild type Mutan Mutan
pada pita standar mutan 140 (Gambar 22b). Enzim PvuII memotong gen yang mengalami mutasi pada daerah kodon 861, gen yang tidak mengalami mutasi tidak terpotong (Kawada et al. 2008).
Prevalensi mutasi gen KRAS dan EGFR
Berdasarkan hasil analisis menggunakan metode HRM dan RFLP dari 63 pasien kanker paru diketahui satu pasien (1,6%) mengalami mutasi gen KRAS, dan 10 pasien (15,9%) memiliki mutasi gen EGFR, sedangkan 52 pasien (82,5%) menunjukkan profil gen KRAS dan EGFR wild type (Gambar 23).
Gambar 23 Prevalensi mutasi gen KRAS dan EGFR pada pasien kanker paru Nilai prevalensi mutasi gen KRAS dan EGFR yang diperoleh menunjukkan nilai yang lebih rendah jika dibandingkan dengan hasil penelitian yang telah dilaporkan. Beberapa hasil penelitian analisis mutasi gen KRAS dan EGFR dengan menggunakan sampel standar (dahak, sitologi pleura dan FFPE) menunjukkan prevalensi mutasi gen KRAS pasien kanker paru sekitar 18% - 33% (Lu et al. 2004; Ramirez et al. 2003; Krypuy et al. 2006), dan prevalensi mutasi gen EGFR sekitar 12% – 50% (Cortes-Funes et al. 2005; Huang et al. 2004; Tokumo et al. 2005; Zhang et al. 2005; Han et al. 2005). Di Indonesia data mengenai prevalensi mutasi gen KRAS dan EGFR pada kasus kanker paru belum pernah dilaporkan. Data hasil analisis Laboratorium KalGen (Kalbe Genomic)-SCI PT Kalbe Farma, Tbk (KalGen Lab, 2012) menunjukkan jumlah prevalensi mutasi gen EGFR pasien kanker paru sebesar 28% (46/164).
Perbedaan nilai prevalensi mutasi gen sangat ditentukan oleh perbedaan karakteristik faktor genetik pasien di setiap daerah. Beberapa hasil analisis mutasi gen EGFR misalnya, di beberapa daerah menunjukkan nilai yang bervariasi (Tabel 2). Selain itu ras dan keturunan pasien juga mempengaruhi profil mutasi gen pada pasien kanker paru (Tabel 2). Mutasi gen EGFR banyak ditemukan pada pasien yang berasal dari keturunan asia, wanita, tidak memiliki riwayat perokok dan jenis sel kanker adenokarsinoma (Shigematsu et al. 2005; Bell et al. 2005), sedangkan mutasi gen KRAS lebih banyak ditemukan pada pasien yang berasal dari keturunan non-asia dibandingkan dengan pasien asia timur (Lung et al. 1992; Wang et al. 1998), memiliki riwayat perokok, wanita dan jenis sel kanker adenokarsinoma (Graziano et al. 1999).
1,60% 15,90%
82,50%
Tabel 2 Data prevalensi mutasi gen EGFR pada pasien kanker paru di beberapa negara
Penelitian Asal pasien Ras Prevalensi
mutasi
Huang et al. (2004) Cina Asia 8/16 (50%)
Tokumo et al. (2005) Jepang Asia 10/21 (48%)
Zhang et al. (2005) Cina Asia 12/30 (40%)
Han et al. (2005) Korea Asia 17/90 (19%)
Kim et al. (2005) Korea Asia 6/27 (22%)
Cortes-Funes et al. (2005) Spanyol Kaukasia 10/83 (12%) Cappuzo et al. (2005) Italia Kaukasia 15/89 (17%) Taron et al. (2005) Jepang, USA,
Eropa
Asia, kaukasia
17/68 (25%)
Jika dilihat berdasarkan jenis kelamin, hasil penelitian ini menunjukkan bahwa prevalensi mutasi gen EGFR ditemukan 21,43% pada pria dan 15,3% pada wanita (Tabel 3). Hasil penelitian ini menunjukkan bahwa mutasi gen EGFR banyak ditemukan pada pasien pria dibandingkan dengan wanita. Hasil ini berbeda dengan hasil penelitian yang telah dilakukan sebelumnya yang menyebutkan bahwa mutasi gen EGFR lebih banyak ditemukan pada pasien wanita. Hal ini menjadi sebuah penemuan yang menarik. Serangkaian penelitian lebih lanjut perlu dilakukan untuk memperoleh data pasti mengenai prevalensi mutasi gen EGFR pada pasien kanker paru Indonesia. Kondisi lingkungan, hormonal, genetik maupun kemungkinan adanya faktor lain yang berpengaruh perlu dianalisis.
Tabel 3. Prevalensi mutasi gen EGFR dan KRAS berdasarkan jenis kelamin
Jenis kelamin Jumlah Prevalensi
Mutasi EGFR Mutasi KRAS
Pria 28 6/28 (21,43%) -
Wanita 26 4/26 (15,3%) -
Tidak diketahui 9 - 1
Total 63 10/63 (15,9%) 1/63 (1,6%)
Sumber sampel sebagai bahan analisis molekuler juga memiliki pengaruh terhadap hasil analisis. Sampel histologi seperti hasil biopsi dan mikrodiseksi menunjukkan akurasi yang lebih tinggi dibandingkan dengan sampel sitologi. Namun demikian, sampel yang berasal dari sitologi lebih banyak dipilih dibandingkan dengan sampel histologi karena pengambilan sampel yang relatif lebih mudah tanpa operasi, dan dapat tersedia dalam jumlah banyak sehingga tidak harus dilakukan pengambilan sampel ulang jika sampel tidak memenuhi standar untuk dilakukan analisa (Pang et al. 2012).
Faktor berikutnya yang berpengaruh terhadap data hasil analisis mutasi gen adalah tingkat sensitivitas metode analisis yang dipilih. Beberapa hasil penelitian yang telah dilakukan umumnya menunjukkan data prevalensi mutasi berdasarkan hasil analisis menggunakan metode sekuensing. Pada penelitian ini digunakan metode HRM dan RFLP yang menunjukkan nilai sensitivitas yang tinggi. Kedua metode ini mampu mendeteksi adanya DNA mutan dalam campuran DNA wild
type dan senyawa lain yang pada umumnya menjadi pengotor di dalam sampel,
dalam batasan deteksi 12,5% dan 3,125% DNA mutan. Nilai ini jauh lebih tinggi jika dibandingkan dengan metode sekuensing yang memiliki batasan deteksi 30% DNA mutan. Berdasarkan hal tersebut, aplikasi metode HRM dan RFLP ini dapat dilakukan dalam analisis sampel yang berasal dari cairan pleura yang banyak memiliki pengotor. Pada cairan pleura selain sel-sel tumor banyak ditemukan sel leukosit, protein dengan kadar tinggi, glukosa dengan kadar rendah dan DNA sel epitel normal yang turut terbawa (Arbaningsih 2010). Dengan demikian, pemilihan metode HRM dan RFLP bukan menjadi faktor penyebab rendahnya nilai prevalensi mutasi yang diperoleh pada penelitian ini.
Hasil penelitian ini menunjukkan bahwa mutasi gen KRAS dan EGFR terjadi pada pasien yang berbeda. Pasien yang menunjukkan hasil analisis mengalami mutasi gen KRAS memiliki profil gen EGFR wild type, demikian pula sebaliknya. Menurut Marchetti et al. (2005) mutasi gen KRAS dan EGFR bersifat
mutually exclusive. Kedua jenis mutasi gen ini sangat jarang terjadi secara
bersamaan dalam satu pasien. Kasus terjadinya mutasi gen KRAS dan EGFR secara bersamaan dalam satu pasien hanya pernah dilaporkan terdapat pada dua pasien oleh Han et al. (2005). Mutasi gen EGFR umumnya terjadi dalam satu lokasi. Sesuai dengan hasil peneliian tersebut, hasil penelitian ini menunjukkan bahwa mutasi gen EGFR dan KRAS tidak terjadi secara bersamaan pada satu pasien. Demikian pula, mutasi EGFR ekson 19 tidak ditemukan bersamaan dengan mutasi pada ekson 21. Pang et al. (2012) melaporkan bahwa mutasi gen
EGFR dapat terjadi pada dua ekson secara bersamaan. Hasil penelitiannya
menunjukkan mutasi ganda terjadi sebanyak 2,7% dan umumnya ditemukan bersamaan dengan mutasi pada ekson 18.
Hasil penelitian ini terbatas hanya diarahkan untuk mengetahui prevalensi mutasi pada gen EGFR yang difokuskan pada ekson 19 dan 21, analisis lebih lanjut dapat dilakukan untuk mengetahui profil mutasi gen EGFR pada ekson 18, sehingga prevalensi mutasi gen EGFR secara keseluruhan kemungkinan besar bisa bertambah. Analisis mutasi gen EGFR ekson 18 meskipun secara prevalensi jarang ditemukan dibandingkan mutasi pada ekson 19 dan 21 namun banyak dilakukan seiring dengan hasil penelitian yang menunjukkan adanya resistensi obat terapi target gefitinib dan erlotinib pada pasien yang memiliki mutasi gen
EGFR ekson 18.
Hasil penelitian ini menunjukkan bahwa masih ada 82,5% pasien yang masih belum diketahui profil mutasi gen yang menyebabkan terjadinya kanker. Dengan demikian penelitian lebih lanjut dapat dilakukan dengan melakukan analisis molekuler pasien kanker paru menggunakan jenis biomarker gen lain seperti PIK3CA, RAF, EML4-ALK, dan lain-lain.
Potensi Pengembangan Kertas Saring sebagai Media Pengiriman Sampel
Pada penelitian ini telah berhasil membuktikan bahwa DNA dapat diisolasi dari kertas saring yang berisi sampel cairan pleura dengan konsentrasi yang beragam mulai dari 5,7 ng/µL hingga 369,2 ng/µL. DNA yang diperoleh dari kertas saring tersebut dapat digunakan sebagai DNA cetakan dalam proses PCR sehingga adanya mutasi gen KRAS dan EGFR dapat dianalisis. Meskipun konsentrasi DNA yang diperoleh dalam jumlah sedikit namun data hasil analisis
HRM dan RFLP masih dapat diperoleh dengan jelas. Menurut Pang et al. (2012) konsentrasi DNA bukan merupakan syarat utama keberhasilan analisis mutasi gen menggunakan metode berbasis PCR dibandingkan dengan faktor kualitas DNA. Meskipun jumlah DNA yang berhasil diisolasi sedikit namun kualitasnya baik maka teknik tersebut dapat digunakan.
Penggunaan kertas saring sebagai media peyimpanan dan pengiriman sampel medis seperti darah, virus dan protein telah banyak dilakukan (Dobbs et
al. 2002; Chaisomchit et al. 2005). Chaisomchit et al.(2005) telah melakukan uji
kestabilan penyimpanan DNA dalam bentuk kertas saring selama kurun waktu 11 tahun. DNA berhasil diisolasi dari sampel darah yang disimpan dalam kertas saring pada suhu ruang selama kurun waktu 11 tahun. DNA tersebut masih bisa digunakan sebagai DNA cetakan dalam proses PCR. Hasil penelitiannya telah membuktikan bahwa kertas saring dapat digunakan untuk menyimpan sampel darah selama lebih dari 10 tahun. Dobbs et al. (2002) telah menggunakan kertas saring sebagai media penyimpanan dan pengiriman sampel sel tumor hasil operasi. Berdasarkan hasil penelitiannya, kualitas DNA yang diisolasi dari kertas saring memiliki kualitas yang sama dengan DNA yang diisolasi dari sel tumor secara langsung.
Metode analisis mutasi gen KRAS dan EGFR menggunakan cairan pleura sebagai sumber sampel umumnya disimpan dalam bentuk bentuk pulasan sitologi (Krypuy et al. 2006; Namoto et al. 2006; Hong Do et al. 2008). Sel tumor yang terdapat dalam cairan pleura terlebih dahulu dilihat menggunakan mikroskop sehingga dalam analisis molekuler dapat dipilih hanya sel-sel tumor yang dijadikan sebagai sumber isolasi DNA sampel. Berbeda halnya dengan sampel yang disimpan dalam kertas saring. DNA yang diisolasi berasal dari DNA bebas yang terdapat dalam cairan dan beberapa sel yang tidak bisa diketahui kanker atau
wild type. Jumlah DNA dari sel normal yang terserap dalam kertas saring dalam
jumlah yang relatif besar kemungkinan besar dapat menyebabkan hasil analisis negatif data mutasi yang diperoleh.
Kertas saring memiliki potensi yang besar untuk diaplikasikan sebagai media penyimpanan sampel yang tahan lama dan menjadi model pengiriman sampel yang efisien jika dibandingkan dengan sampel dalam bentuk cairan. Teknik ini juga lebih sederhana, tidak membutuhkan peralatan khusus. Teknik ini memiliki potensi manfaat lebih besar sebagai media pengiriman sampel dari rumah sakit daerah yang tidak memiliki fasilitas analisis molekuler ke Laboratorium analisis molekuler yang jaraknya cukup jauh, seperti dari rumah sakit daerah di Nusa Tenggara Barat dan Papua ke Laboratorium di Jakarta. Serangkaian penelitian lebih lanjut untuk melakukan validasi dan stadarisasi aplikasi metode penyimpanan sampel cairan pleura dalam kertas saring perlu dilakukan sebelum dapat diaplikasikan secara rutin dalam aplikasi medis.