Keragaman Genetik Dua Puluh Aksesi Plasma Nutfah
Jatropha
spp. Menggunakan Marka
Simple Sequence Repeat
(Genetic Diversity of Twenty Accessions of
Jatropha
spp.
Germplasms based on Simple Sequence Repeat Markers)
Kristianto Nugroho*, Rerenstradika T. Terryana, Reflinur, Puji Lestari, Karden Mulya, dan I Made Tasma Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Jl. Tentara Pelajar 3A, Bogor 16111 Indonesia
Telp. (0251) 8337975; Faks. (0251) 8338820; *E-mail: [email protected]
Diajukan: 10 Januari 2017; Direvisi: 7 Maret 2017; Diterima: 20 April 2017
ABSTRACT
Physic nut (Jatropha curcas L.) is a potential plant which produces alternative energy to fossil fuels. The information about genetic diversity of the genus Jatropha is important to determine the direction of future breeding activities. The purpose of this study was to analyze the genetic diversity among 20 accessions of Jatropha from Indonesia and Thailand by using 20 SSR markers. A total of 129 alleles were detected with a range between 4 alleles and 9 alleles per locus and an average of 6.5 alleles. The value of gene diversity ranged from 0.53 to 0.86 with an average of 0.75 and the value of PIC ranged from 0.49 to 0.84 with an average of 0.71. As many as twelve markers had PIC values >0.70 and those markers were informative to discriminate Jatropha members. The average of major allele frequency was 37% with a range from 18% to 55%. There were seven SSR markers which were able to discriminate heterozygous genotype with heterozygosity values ranged from 0.05 to 0.11 with an average of 0.03. The results of phylogenetic analysis showed that the twenty Jatropha accessions were separated into two main clusters at the similiarity coefficient of 0.70. The first cluster consisted of 17 J. curcas accessions, while the second cluster consisted of 3 accessions, namely J. podagrica, J. gossypifolia, and J. multifida. The genetic diversity data from this study will be used as the basic information for parental selection in breeding program in order to produce new varieties with high oil yield character.
Keywords: Jatropha spp., genetic diversity, SSR.
ABSTRAK
Jarak pagar (Jatropha curcas L.) merupakan salah satu tanaman yang potensial sebagai penghasil energi alternatif bahan bakar fosil. Informasi mengenai keragaman genetik genus Jatropha spp. sangat penting untuk menentukan arah kegiatan pemuliaan ke depan. Tujuan penelitian ini adalah menganalisis keragaman genetik 20 aksesi plasma nutfah Jatropha spp. asal Indonesia dan Thailand menggunakan 20 marka SSR. Sebanyak 129 alel berhasil dideteksi dengan rentang 4 9 alel per lokus dan rerata 6,5 alel. Nilai diversitas gen sebesar 0,53 hingga 0,86 dengan rerata 0,75, sedangkan nilai PIC sebesar 0,49 hingga 0,84 dengan rerata 0,71. Sebanyak 12 marka memiliki nilai PIC > 0,70 dan bersifat informatif untuk membedakan individu jarak. Rerata frekuensi alel utama yang diperoleh sebesar 37% dengan rentang 18–55%. Sebanyak 7 marka SSR mampu membedakan genotipe heterozigot dengan nilai heterozigositas sebesar 0,05 hingga 0,11 dengan rerata 0,03. Hasil analisis filogenetik menunjukkan bahwa 20 aksesi Jatropha spp. memisah menjadi dua klaster utama pada koefisien kesamaan 0,70. Klaster pertama terdiri atas 17 aksesi J. curcas,sedangkan klaster kedua terdiri atas 3 aksesi, yaitu J. podagrica, J. gossypifolia,
dan J. multifida. Data keragaman genetik yang diperoleh pada penelitian ini dapat dimanfaatkan sebagai dasar pemilihan
tetua persilangan dalam rangka menghasilkan varietas unggul baru dengan karakter kadar minyak tinggi sesuai yang diharapkan.
Kata kunci: Jatropha spp., keragaman genetik, SSR.
PENDAHULUAN
Bahan bakar fosil selama ini menjadi sumber energi utama yang banyak dimanfaatkan oleh masya-rakat. Ketersediaan bahan bakar fosil yang semakin berkurang dan sifatnya yang tidak dapat diperbarui membuat isu mengenai krisis energi global semakin merebak (Djumali dan Nurnasari 2014). Selain itu, sisa buangan bahan bakar fosil yang mencemari lingkungan dan diduga ikut berkontribusi terhadap efek rumah kaca membuat kebutuhan untuk mencari energi alternatif yang ramah lingkungan semakin meningkat (Sun et al. 2008).
Jarak pagar (Jatropha curcas L.) merupakan salah satu tanaman yang potensial sebagai penghasil bahan bakar alternatif. Biji jarak pagar menghasilkan minyak yang dapat dikonversi menjadi biodiesel yang performanya tidak jauh berbeda dengan bahan bakar fosil (Mastan et al. 2013). Pemanfaatan minyak jarak pagar sebagai bahan bakar alternatif telah dilakukan semasa Perang Dunia II (Kumar dan Sharma 2008). Tanaman jarak pagar memiliki keunggulan, yaitu adaptif pada lahan marjinal, toleran kekeringan dan suhu tinggi, memiliki periode gestasi yang singkat, dan tidak dapat dimakan (non-edible plant) sehingga pemanfaatannya sebagai bahan bakar tidak akan me-nimbulkan persaingan kebutuhan, seperti pada pem-buatan bioetanol dari jagung atau singkong (Mastan et al. 2013).
Selain jarak pagar, terdapat anggota genus Jatropha lain yang tersebar di seluruh dunia dengan total mencapai 170 spesies (Heller 1996). Di antara-nya, terdapat jarak bali atau bottleshrub plant (J. podagrica Hook.) yang selain dimanfaatkan sebagai tanaman hias, juga memiliki potensi sebagai donor karakter sifat minyak tinggi karena kandungan minyak pada bijinya lebih dari 50% (Khrisnan dan Paramathma 2009). Selain itu, ada pula bellyache bush (J. gossypifolia L.) yang toleran terhadap cekam-an kekeringcekam-an dcekam-an salinitas tinggi serta jarak cina atau coral plant (J. multifida L.) yang memiliki ukuran buah yang besar. Anggota genus Jatropha tersebut memiliki potensi sebagai sumber gen tertentu yang dapat dikombinasikan dengan jarak pagar untuk menghasilkan varietas unggul baru dengan kadar minyak yang tinggi.
Dalam kegiatan pemuliaan jarak pagar, karak-terisasi genetik merupakan titik awal dalam proses seleksi yang dapat dilakukan, baik secara morfologi, biokimia maupun molekuler (Jingura dan Kamusoko, 2015; Zulfahmi 2013). Karakterisasi secara molekuler mempunyai banyak keuntungan, yaitu bersifat stabil, tidak dipengaruhi oleh lingkungan, serta dapat dilaku-kan pada semua fase pertumbuhan tanaman (Hidayatun et al. 2011). Selain itu, data hasil
karak-terisasi secara molekuler dapat meningkatkan presisi data karakterisasi morfologis (Santoso et al. 2006).
Simple sequence repeats (SSR) atau mikrosatelit merupakan penanda molekuler yang saat ini banyak diaplikasikan dalam kegiatan karakterisasi secara molekuler. SSR merupakan sekuen DNA bermotif pendek yang berulang secara berpasangan 2–5 unit nukleotida yang tersebar di dalam genom, terutama pada organisme eukariotik (Azrai 2005). Menurut Lightfoot dan Iqbal (2013), marka ini bersifat kodomi-nan, memiliki polimorfisme tinggi, serta berbasis PCR sehingga secara teknis mudah diaplikasikan. Marka SSR banyak diterapkan dalam bidang pertanian, ter-utama pada kegiatan karakterisasi material genetik, seleksi tanaman, analisis sidik jari DNA, dan pemeta-an genetik terkait karakter kupemeta-antitatif tertentu (Madesis et al. 2013).
Saat ini, marka SSR yang telah dikembangkan untuk menganalisis keragaman genetik genus Jatropha adalah yang berbasis fast isolation by AFLP of sequences containing repeats (FIASCO) (Sun et al. 2008), genomic (G)-SSR (Wen et al. 2010), dan expressed sequence tagged (EST)-SSR (Laosatit et al. 2013). Metode FIASCO yang digunakan oleh Sun et al. (2008) memiliki kelebihan, yaitu tidak memerlukan sekuen genom total tanaman jarak sehingga biaya pe-nelitian menjadi lebih murah, namun tingkat polimor-fisme marka SSR yang dihasilkan lebih rendah. Sementara, pengembangan G-SSR ataupun EST-SSR terkendala biaya yang lebih mahal karena memerlu-kan sekuensing genom total, namun marka yang di-hasilkan memiliki polimorfisme lebih tinggi serta ber-kaitan langsung dengan karakter agronomis tertentu (Laosatit et al.2013; Wen et al. 2010).
Di Indonesia, pemanfaatan marka SSR untuk ke-giatan pemuliaan berbasis marka pada genus Jatropha belum banyak dilakukan. Penelitian sejenis telah dilakukan oleh Saptadi et al. (2011), namun materi genetik yang digunakan baru sebatas J. curcas dan J. multifida. Tujuan penelitian ini adalah meng-analisis keragaman genetik 20 aksesi Jatropha spp. asal Indonesia dan Thailand menggunakan 20 marka SSR. Informasi hubungan genetik antarindividu Jatropha memberikan manfaat penting bagi perbaik-an tperbaik-anamperbaik-an dalam rperbaik-angka memperoleh sifat-sifat yang menguntungkan (Tasma et al. 2013).
BAHAN DAN METODE Materi Genetik
Penelitian ini dilaksanakan di Laboratorium Biologi Molekuler, BB Biogen mulai bulan November 2015 hingga Maret 2016. Bahan tanaman yang
diguna-kan, yaitu 20 aksesi Jatropha spp. yang terdiri atas 16 aksesi J. curcas asal Indonesia dan 4 aksesi (J. curcas, J. podagrica, J. gossypifolia, dan J. multifida) asal Thailand (Kasetsart University). Daftar aksesi tanam-an jarak ytanam-ang digunaktanam-an beserta karakter morfologis-nya tercantum pada Tabel 1, sedangkan keragaman karakter morfologis bunga jarak dapat dilihat pada Gambar 1. Kedua puluh individu Jatropha tersebut ditanam di dalam pot di Rumah Kaca Cikeumeuh, BB Biogen, Bogor.
Dua puluh pasang marka SSR yang digunakan pada penelitian ini (Tabel 2) didesain oleh Seoul National University (SNU) dan Kasetsart University. Marka tersebut didesain berbasis genom jarak pagar menggunakan J. curcas varietas ‘Cn’ asal Thailand se-bagai referensinya (Lee et al., belum dipublikasikan). Varietas ‘Cn’ merupakan varietas lokal asal Provinsi Chai Nat, Thailand dengan karakter batang yang tinggi dan bunga berwarna hijau cerah (Muakrong et al. 2014).
Ekstraksi DNA
DNA diekstraksi menggunakan metode Doyle dan Doyle (1990) yang dimodifikasi. Sebanyak 0,5 g potongan daun jarak digerus di dalam mortar steril dengan menggunakan nitrogen cair. Bubuk hasil penggerusan kemudian dimasukkan ke dalam tabung mikro 2 ml diikuti dengan penambahan sebanyak 800 µl bufer ekstraksi (Tris-HCl 100 mM [pH 8,0], NaCl 1,4 M, EDTA 20 mM [pH 8,0], cetyltrimethylammonium bromide [CTAB] 2% [w/v], polyvinylpyrrolidone [PVP] 2% [w/v], natrium disulfit 0,38% [w/v], dan asam askorbat 1% [w/v]). Sampel diekstrak dua kali meng-gunakan larutan kloroform : isoamil alkohol (24 : 1) lalu dipresipitasi menggunakan isopropanol. Pelet DNA yang terbentuk lalu dicuci dengan larutan etanol 70% dan dikeringkan menggunakan alat SpeedVac™ Concentrator (Thermo Fisher Scientific, USA) untuk menghilangkan sisa-sisa etanol. Pelet yang telah kering dilarutkan dalam 100 µl larutan TE (Tris 10 mM [pH 8,0] dan EDTA 1 mM) yang ditambah RNAse A (10 mg/ml), diikuti inkubasi pada suhu 37°C selama 1 jam.
Tabel 1. Daftar sampel Jatropha spp. yang digunakan pada penelitian ini beserta beberapa karakter morfologinya.
Individu Asal koleksi
Warna daun dewasa Bentuk dan susunan tulang daun Warna tangkai
daun Warna bunga Tipe bunga
Warna buah masak
Kadar lemak kasar (%) J. curcas Majene #1 Majene, Sulawesi Barata Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning 29,6 J. curcas Majene #2 Majene, Sulawesi Barata Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning * J. curcas IP3-P #1 Pakuwon, Jawa Barat Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning 28,2 J. curcas IP3-P #2 Pakuwon, Jawa Barat Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning * J. curcas IP3-P #3 Pakuwon, Jawa Barat Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning * J. curcas Bacan #1 P. Bacan, Maluku Utara Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning 27,5 J. curcas Bacan #2 P. Bacan, Maluku Utara Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas Bacan #3 P. Bacan, Maluku Utara Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas Bacan #4 P. Bacan, Maluku Utara Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas NP#1 Nusa Penida, Bali Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning * J. curcas NP #2 Nusa Penida, Bali Hijau Bulat menjari Hijau Hijau muda Monoecious Kuning * J. curcas IP3-M #1 Malang, Jawa Timur Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas IP3-M #2 Malang, Jawa Timur Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas IP3-M #3 Malang, Jawa Timur Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas IP3-M #4 Malang, Jawa Timur Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas IP3-M #5 Malang, Jawa Timur Hijau Bulat menjari Hijau Hijau muda Hermafrodit Kuning * J. curcas Thailand Kassetsart University, Thailand Hijau tua Bulat menjari Hijau Hijau muda Monoecious Kuning 27,3 J. podagrica Thailand Kassetsart University, Thailand Hijau tua Bulat menjari Merah kehijauan Merah Monoecious Hijau tua 34,6 J. gossypifolia Thailand Kassetsart University, Thailand Hijau tua
kemerahan
Bulat menjari Ungu Merah keunguan
Monoecious Hijau tua 15,8 J. multifida Thailand Kassetsart University, Thailand Hijau tua Bulat menjari Hijau kemerahan Merah Monoecious Kuning 26,1 Sumber: Terryana et al. (2016), belum dipublikasikan.
* tidak dilakukan pengukuran.
Gambar 1. Karakter morfologis warna bunga Jatropha spp. A = J. curcas Majene #1, B = J. gossypifolia Thailand, C = J. podagrica
Thailand, D = J. multifida Thailand. Sumber: Terryana et al. (2016), belum dipublikasikan.
Amplifikasi DNA
Setiap sampel diamplifikasi dalam total reaksi 10 µl yang mengandung template DNA 20 ng sebanyak 2 µl; Kapa2G Fast ReadyMix (Kapa Biosystems, USA) sebanyak 5 µl; primer forward dan reverse dengan konsentrasi 10 µM masing-masing sebanyak 0,5 µl, dan ddH2O steril. Reaksi PCR dilakukan dalam mesin
PCR T1 Thermocycler (Biometra, Germany) dengan profil PCR yang telah digunakan oleh Saptadi et al. (2011) sebagai berikut: denaturasi awal dilakukan pada suhu 95°C selama 5 menit, diikuti sebanyak 35 siklus proses denaturasi pada suhu 94°C selama 30 detik, tahap penempelan primer (annealing) pada suhu 56°C selama 1 menit, dan tahap perpanjangan basa (elongation) pada suhu 72°C selama 1 menit. Reaksi PCR diakhiri dengan tahap akhir perpanjangan basa (final extension) pada suhu 72°C selama 5 menit. Hasil PCR kemudian dielektroforesis pada gel
agarosa 4% pada tangki berisi bufer TAE 1× dengan tegangan 100 volt selama 3,5 jam.
Analisis Data
Hasil elektroforesis pada gel agarose 4% seperti disajikan pada Gambar 2 diskor sebagai data biner. Setiap pita yang terlihat pada gel dianggap sebagai satu alel. Pita-pita DNA yang memiliki laju migrasi yang sama dianggap memiliki lokus yang sama. Pita yang terlihat diberi skor 1, pita yang tidak terlihat di-beri skor 0, sedangkan sampel yang tidak menghasil-kan amplikon diberi skor 9 dan dianggap sebagai data yang hilang. Hasil skoring berupa data biner. Penen-tuan posisi pita dilakukan dengan perangkat lunak Gel Analyzer (Lazar 2010). Data skor biner selanjutnya dianalisis dengan menggunakan program Sequential Agglomerative Hierarchical and Nested-Unweighted Pair-Group Method with Arithmetic (SAHN-UPGMA) pada perangkat lunak NTSYS versi 2.1. (Rohlf 2000). Hasil analisis disajikan dalam bentuk dendrogram Tabel 2. Daftar 20 primer SSR yang digunakan dalam penelitian ini (Lee et al., belum dipublikasikan).
Nama primer Forward (5’-3’) Reverse (5’-3’)
Scaffold 2–9 ACTGCTCTGCTGTGCTGTGT TGTTTGGGAGCTAAAGGGAA Scaffold 3–2 AGAACGCATGAGTGCATTATT GGGTAAATATGTGTTTAGCGGC Scaffold 5–1 TGGTGCTTGTAAATTGAGTGC AGGCGCAGTAAGCTGTTGTT Scaffold 6–1 ATACCCCATTCCTCTGGTCC AATACGGGTCCAGGCCTATC Scaffold 6–2 TTAGCCCCAGTCAAAGGATTT TTGGTTGGTTGGTTCTCAAA Scaffold 7–1 TGCTGTTCATCCTTCAATGG TCGAAGAGATGCACCAGATG Scaffold 8–1 TCTTATGCGCATCGACTGAC TTAACTCCACCTGGCTTTGG Scaffold 9–2 GGGTTTTGGAAAAACGTTGA GTATGTCTGGCAGCGTTCCT Scaffold 10–2 TTCCTAGATTTCAGCCCCAA AGGACATCCCACTAATCCCC Scaffold 13–1 AGGCCCATAACGAACAACAC GCCGAGTCAGAGGATTTGAG Scaffold 13–2 TCCAGATTCCTAGCTGATTATTGA AATCATAATGCCCGACCTTG Scaffold 16–1 GGCTCCTCTCTCTGTTTGGG AGCAAACAGATTCCGATTGAG Scaffold 19–1 GGGTTTGAATTTGATGCAGG CAGGTGCACTAACAAAACCG Scaffold 19–2 GTAGGGTTGCCTTCAAACCA TGATTCCGACCCTCTCATTT Scaffold 21–1 TTCCTTCAGCCCAAGAAAAA TGAGTTGGAATGGTGTGGAA Scaffold 21–2 AAAAAGGAATAATGGCTTGATTT TGGGAGGACATACTTCGATCA Scaffold 27–1 TGCACAATTGGACATTCATATTT TAGAGGAGGCTCCGTCAAAA Scaffold 31–2 TCACGAGGCAAACACTGAAG ATCAAAGCTGGTCTCCCTCC Scaffold 46–2 ATAGGAAATCGAAAACCGCC TGATTCTGTGGTGATCCGAA Scaffold 49–2 GGTGGTGTGTGGCTTATGTG AAAAATGGTGCACTACCAAAAA
Gambar 2. Hasil elektroforesis amplikon DNA Jatropha spp. pada pada gel agarosa 4%. A = Scaffold 13-1, B = Scaffold 5-1, M = 100 bp DNA
ladder, 1 = J. curcas Thailand, 2 = J. gossypifolia Thailand, 3 = J. podagrica Thailand, 4 = J. multifida Thailand, 5 = J. curcas
Majene #1, 6 = J. curcas Majene #2, 7 = J. curcas P. Bacan #1, 8 = J. curcas P. Bacan #2, 9 = J. curcas P. Bacan #3, 10 = J. curcas P. Bacan #4, 11 = J. curcas IP3-P#3, 12 = J. curcas IP3-P#1, 13 = J. curcas IP3-P#2, 14 = J. curcas NP #1, 15 = J. curcas
NP #2, 16 = J. curcas IP3-M#1, 17 = J. curcas IP3-M#2, 18 = J. curcas IP3-M#3, 19 = J. curcas IP3-M#4, 20 = J. curcas IP3-M#5.
500 bp 100 bp 500 bp 100 bp 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 M A A
dan matriks kesamaan genetik. Analisis statistik di-lakukan menggunakan perangkat lunak PowerMarker 3.2 (Liu dan Muse 2005) untuk mengetahui nilai frekuensi alel utama, diversitas gen, heterozigositas, dan Polymorphic Information Content (PIC) marka-marka SSR.
HASIL DAN PEMBAHASAN Polimorfisme Marka SSR
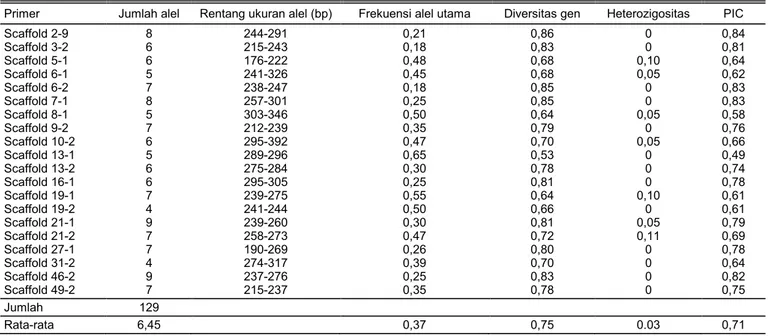
Sebanyak 129 alel berhasil dideteksi pada pene-litian ini menggunakan 20 marka SSR (Tabel 3). Rerata jumlah alel per marka yang terdeteksi seba-nyak 6,45 dengan kisaran 4 9 alel per lokus. Kisaran alel yang diperoleh pada penelitian ini lebih banyak dibanding dengan penelitian Basha et al. (2009) yang menggunakan 17 marka dengan kisaran 2 4 alel per lokus, Mastan et al. (2013) yang menggunakan 7 marka terseleksi polimorfik dengan kisaran 2 5 alel per lokus, dan Vischi et al. (2013) yang juga menggu-nakan 7 marka terseleksi polimorfik dengan kisaran 2 3 alel per lokus. Menurut Tasliah et al. (2013), jumlah alel yang dihasilkan pada suatu penelitian ke-ragaman genetik dipengaruhi oleh jumlah marka, jumlah sampel, dan variasi sampel yang digunakan. Jumlah alel yang semakin banyak terdeteksi akan bermanfaat terutama bila alel-alel tersebut merupa-kan koleksi dari plasma nutfah yang masih belum dimanfaatkan dan memiliki kaitan dengan gen-gen penting yang berhubungan dengan karakter unggul tertentu (Santoso et al. 2006).
Rerata frekuensi alel utama yang diperoleh sebe-sar 37% dengan nilai terendah sebesebe-sar 18% pada mar-ka Scaffold 3-2 dan Scaffold 6-2 dan nilai tertinggi 55%
pada marka Scaffold 19-1. Yadav et al. (2011) mem-peroleh rentang frekuensi alel utama yang lebih tinggi sebesar 37 98% dengan rerata 77%. Nilai diversitas gen atau heterozigositas yang diharapkan (He), yang menurut Chaerani et al. (2011) menunjukkan peluang terambilnya dua alel secara acak dari suatu populasi, sebesar 0,53 (Scaffold 13-1) hingga 0,86 (Scaffold 2-9) dengan rerata 0,75. Sementara itu, nilai heterozigosi-tas yang diamati (Ho) pada penelitian ini sebesar 0,05 hingga 0,11 dengan rerata 0,03. Nilai Ho pada peneliti-an ini lebih kecil dibpeneliti-anding dengpeneliti-an nilai He dan hanya terdeteksi pada lima lokus saja, yaitu Scaffold 6-1, Scaffold 8-1, Scaffold 10-2, Scaffold 21-1, dan Scaffold 21-2. Jumlah ini lebih sedikit dibanding dengan hasil yang diperoleh Yadav et al. (2011) yang berhasil memperoleh 17 marka yang mampu men-deteksi alel heterozigot dari 21 marka yang diguna-kan dengan rentang nilai 0,04 1,00 dengan rerata 0,30.
Rendahnya heterozigositas pada genus Jatropha kemungkinan disebabkan oleh kegiatan introduksi yang jarang dilakukan, perbanyakan tanaman yang lebih banyak dilakukan secara vegetatif, dan terjadi-nya inbreeding (Ambrosi et al. 2010; Rosado et al. 2010). Menurut Ariyanto dan Utami (2006), nilai heterozigositas berkaitan erat dengan tingkat ke-ragaman genetik, semakin besar proporsi alel heterozigot, semakin tinggi keragaman genetik suatu populasi, begitu pula sebaliknya. Populasi dengan tingkat keragaman genetik tinggi akan memiliki pe-luang hidup lebih besar dan lebih mampu merespons setiap perubahan lingkungan yang terjadi, sebaliknya populasi dengan tingkat homozigositas tinggi akan lebih rentan terhadap perubahan lingkungan dan Tabel 3. Keragaan dua puluh marka SSR yang digunakan pada penelitian ini.
Primer Jumlah alel Rentang ukuran alel (bp) Frekuensi alel utama Diversitas gen Heterozigositas PIC
Scaffold 2-9 8 244-291 0,21 0,86 0 0,84 Scaffold 3-2 6 215-243 0,18 0,83 0 0,81 Scaffold 5-1 6 176-222 0,48 0,68 0,10 0,64 Scaffold 6-1 5 241-326 0,45 0,68 0,05 0,62 Scaffold 6-2 7 238-247 0,18 0,85 0 0,83 Scaffold 7-1 8 257-301 0,25 0,85 0 0,83 Scaffold 8-1 5 303-346 0,50 0,64 0,05 0,58 Scaffold 9-2 7 212-239 0,35 0,79 0 0,76 Scaffold 10-2 6 295-392 0,47 0,70 0,05 0,66 Scaffold 13-1 5 289-296 0,65 0,53 0 0,49 Scaffold 13-2 6 275-284 0,30 0,78 0 0,74 Scaffold 16-1 6 295-305 0,25 0,81 0 0,78 Scaffold 19-1 7 239-275 0,55 0,64 0,10 0,61 Scaffold 19-2 4 241-244 0,50 0,66 0 0,61 Scaffold 21-1 9 239-260 0,30 0,81 0,05 0,79 Scaffold 21-2 7 258-273 0,47 0,72 0,11 0,69 Scaffold 27-1 7 190-269 0,26 0,80 0 0,78 Scaffold 31-2 4 274-317 0,39 0,70 0 0,64 Scaffold 46-2 9 237-276 0,25 0,83 0 0,82 Scaffold 49-2 7 215-237 0,35 0,78 0 0,75 Jumlah 129 Rata-rata 6,45 0,37 0,75 0.03 0,71
serangan hama dan penyakit (Wigati 2003; Yusron 2005). Oleh karena itu, peningkatan keragaman genetik Jatropha perlu diupayakan, baik melalui ke-giatan persilangan, mutasi maupun introduksi dari luar (Hapshoh et al. 2016).
Nilai PIC yang menunjukkan tinggi rendahnya tingkat polimorfisme diperoleh sebesar 0,49 (Scaffold 13-1) hingga 0,84 (Scaffold 2-9) dengan rerata 0,71. Nilai PIC yang diperoleh pada penelitian ini lebih tinggi dibanding dengan hasil penelitian Yadav et al. (2011) yang memperoleh rentang nilai PIC 0,04 0,61 dengan rerata 0,25, namun lebih rendah daripada ha-sil penelitian Mastan et al. (2013) yang memperoleh rentang nilai PIC 0,78 1,00 dengan rerata 0,91.
Menurut Hildebrand et al. (1992), marka yang sa-ngat informatif memiliki nilai PIC >0,7. Berdasarkan kriteria Hildebrand, terdapat 12 marka yang sangat informatif dari penelitian ini yang bermanfaat untuk membedakan individu-individu Jatropha spp. ke depannya (Tabel 3). Marka-marka tersebut adalah Scaffold 2-9, Scaffold 3-2, Scaffold 6-2, Scaffold 7-1, Scaffold 9-2, Scaffold 13-2, Scaffold 16-1, Scaffold 21-1, Scaffold 21-2, Scaffold 27-1, Scaffold 46-2, dan Scaffold 49-2.
Analisis Filogenetik
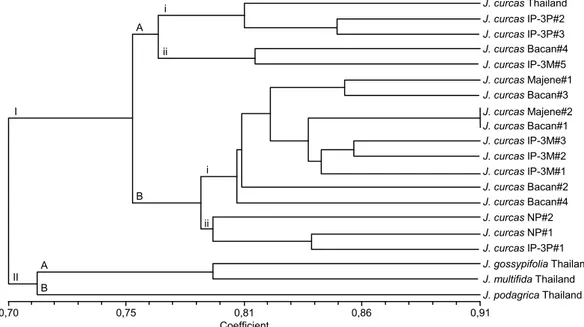
Analisis filogenetik menunjukkan bahwa kedua puluh individu Jatropha mengelompok menjadi dua klaster utama pada koefisien kesamaan 0,70 (Gambar 3). Klaster pertama terdiri atas 17 aksesi yang seluruhnya merupakan spesies J. curcas, se-dangkan klaster kedua terdiri atas 3 aksesi yang
ter-diri atas spesies J. podagrica, J. multifida, dan J. gossypifolia asal Thailand. Selanjutnya, klaster per-tama terbagi kembali menjadi dua subklaster, yaitu subklaster IA dan subklaster IB. Pada subklaster IA, hampir semua aksesi J. curcas IP-3M mengelompok di dalam subklaster ini, kecuali IP-3M #1 yang berada di subklaster IB(ii) bersama J. curcas asal Nusa Penida. Hal ini menunjukkan bahwa di dalam aksesi J. curcas yang sama sekalipun masih terdapat variasi genetik di dalamnya. Sementara itu, kedua aksesi J. curcas asal Majene mengelompok pada subklaster IB(i) bersama dengan J. curcas asal Pulau Bacan dan Pakuwon.
Variasi genetik di dalam aksesi (intragroup) J. curcas sebelumnya telah dilaporkan pada penelitian Wen et al. (2010). Adanya variasi genetik intragroup tersebut membuat pemilihan tetua persilangan harus dilakukan lebih teliti karena individu dengan nama aksesi yang sama belum tentu memiliki karakter molekuler yang sama. Di sinilah marka SSR berperan penting dalam membedakan individu-individu dengan nama aksesi yang sama. Meskipun demikian, menurut Wen et al. (2010) besarnya variasi genetik intragroup pada Jatropha lebih kecil dibanding dengan variasi genetik intergroup-nya.
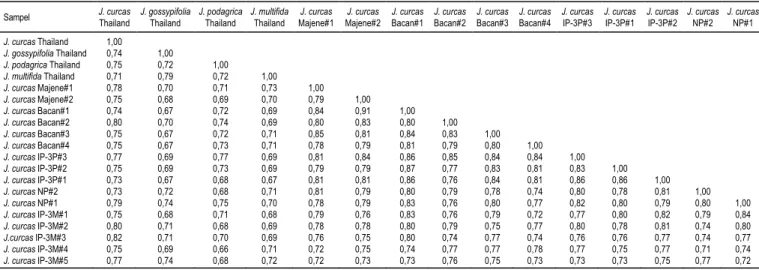
Pada subklaster IB(i) terdapat dua aksesi jarak yang memiliki kekerabatan paling dekat, yaitu J. curcas Majene #2 dengan P. Bacan #1. Meskipun berasal dari wilayah yang berbeda dan memiliki karakter morfologis yang berbeda (Tabel 1), kedua aksesi tersebut memiliki nilai kesamaan genetik sebesar 91% berdasarkan matriks kesamaan genetik
Gambar 3. Dendrogram 20 aksesi plasma nutfah Jatropha spp. berdasarkan 20 marka SSR menggunakan programUPGMA dengan perangkat lunak NTSYS.
i ii A I II A B B i ii 0,70 0,75 0,81 0,86 0,91 Coefficient J. curcas Thailand J. curcas IP-3P#2 J. curcas IP-3P#3 J. curcas Bacan#4 J. curcas IP-3M#5 J. curcas Majene#1 J. curcas Bacan#3 J. curcas Majene#2 J. curcas Bacan#1 J. curcas IP-3M#3 J. curcas IP-3M#2 J. curcas IP-3M#1 J. curcas Bacan#2 J. curcas Bacan#4 J. curcas NP#2 J. curcas NP#1 J. curcas IP-3P#1 J. gossypifolia Thailand J. multifida Thailand J. podagrica Thailand
(Tabel 4). Hal ini menunjukkan bahwa marka SSR yang digunakan pada penelitian ini membedakan aksesi Jatropha spp. bukan berdasarkan wilayah asal ataupun karakter morfologisnya, namun lebih ber-dasarkan hubungan filogeniknya. Hasil serupa diper-oleh pada penelitian Satyawan dan Tasma (2011) me-ngenai keragaman genetik jarak pagar di KP Pakuwon, Sukabumi, yaitu pengelompokan berdasar-kan marka RAPD tidak berkorelasi dengan daerah asal tiap-tiap aksesi. Hasil serupa juga diperoleh pada pengelompokan Jatropha spp. asal beberapa daerah di India dengan menggunakan marka RAPD dan AFLP (Sudheer et al. 2009) serta EST (Yadav et al. 2011). Menurut Satyawan dan Tasma (2011), hal ter-sebut mendukung hipotesis bahwa penyebaran ta-naman jarak pagar lebih banyak difasilitasi oleh manusia. Kedua individu jarak pagar dengan jarak genetik sangat dekat tersebut tidak potensial untuk dijadikan sebagai tetua persilangan ke depannya karena akan memperbesar peluang terjadinya inbreeding.
Selain itu, terdapat pula dua aksesi Jatropha spp. dengan kekerabatan paling jauh, yaitu antara J. podagrica asal Thailand dan J. curcas IP3-M #4 dengan nilai kesamaan genetik sebesar 66% (Tabel 4). Secara morfologis, kedua aksesi tersebut memiliki tampilan fenotipik yang berbeda, yaitu J. podagrica asal Thailand memiliki warna bunga merah, sedang-kan J. curcas IP3-M#4 memiliki warna bunga hijau muda (Tabel 1, Gambar 1). Selain memiliki jarak ge-netik yang cukup jauh, kedua aksesi tersebut po-tensial untuk dijadikan sebagai tetua persilangan ka-rena adanya peluang donor karakter kadar minyak tinggi pada spesies J. podagrica yang menurut hasil pengukuran memiliki kadar lemak kasar sebesar 34,6% (Tabel 1). Potensi tersebut memungkinkan ter-jadinya perbaikan karakter kadar minyak pada
spe-sies J. curcas yang menurut hasil pengukuran memi-liki kadar lemak kasar rerata di bawah 30% (Tabel 1). Sementara itu, pada klaster kedua aksesi J. gossypifolia asal Thailand memiliki kekerabatan lebih dekat dengan J. multifida (Gambar 3). Menurut Satyawan dan Tasma (2011), J. gossypifolia dan J. multifida memiliki konfigurasi kromosom yang mirip dengan J. curcas selama pembelahan meiosis se-hingga diduga hubungan di antara ketiga spesies ter-sebut lebih dekat.
KESIMPULAN
Analisis keragaman genetik 20 aksesi plasma nutfah Jatropha spp. berdasarkan 20 marka SSR menghasilkan dua klaster utama pada koefisien ke-miripan genetik 0,70. Pengelompokan tersebut tidak berkaitan dengan wilayah asal, namun berdasarkan hubungan filogenik. Sebanyak dua belas marka me-nunjukkan tingkat polimorfisme yang tinggi dan dapat digunakan untuk membedakan aksesi Jatropha spp. untuk tujuan seleksi. Aksesi-aksesi Jatropha spp. dengan jarak genetik yang jauh direkomendasikan untuk dijadikan sebagai tetua dalam persilangan untuk memperkecil peluang terjadinya inbreeding. Selain itu, karakter kadar minyak yang tinggi juga dapat dijadikan sebagai acuan dalam pemilihan tetua persilangan agar ke depannya dapat diperoleh varie-tas unggul jarak pagar dengan potensi hasil kadar minyak yang lebih tinggi.
Penelitian ini didanai DIPA BB Biogen TA 2015. Penulis menyampaikan terima kasih kepada Prof. Peerasak Srinives dan Prof. Patcharin Tanya dari Kasetsart University, Thailand atas bantuan materi genetik beberapa spesies Jatropha spp. serta Prof. Suk-Ha Lee dari SNU atas bantuan marka molekuler yang digunakan pada penelitian ini.
Tabel 4. Matriks kesamaan genetik 20 aksesi plasma nutfah Jatropha spp. berdasarkan 20 Marka SSR.
Sampel J. curcas Thailand J. gossypifolia Thailand J. podagrica Thailand J. multifida Thailand J. curcas Majene#1 J. curcas Majene#2 J. curcas Bacan#1 J. curcas Bacan#2 J. curcas Bacan#3 J. curcas Bacan#4 J. curcas IP-3P#3 J. curcas IP-3P#1 J. curcas IP-3P#2 J. curcas NP#2 J. curcas NP#1 J. curcas Thailand 1,00 J. gossypifolia Thailand 0,74 1,00 J. podagrica Thailand 0,75 0,72 1,00 J. multifida Thailand 0,71 0,79 0,72 1,00 J. curcas Majene#1 0,78 0,70 0,71 0,73 1,00 J. curcas Majene#2 0,75 0,68 0,69 0,70 0,79 1,00 J. curcas Bacan#1 0,74 0,67 0,72 0,69 0,84 0,91 1,00 J. curcas Bacan#2 0,80 0,70 0,74 0,69 0,80 0,83 0,80 1,00 J. curcas Bacan#3 0,75 0,67 0,72 0,71 0,85 0,81 0,84 0,83 1,00 J. curcas Bacan#4 0,75 0,67 0,73 0,71 0,78 0,79 0,81 0,79 0,80 1,00 J. curcas IP-3P#3 0,77 0,69 0,77 0,69 0,81 0,84 0,86 0,85 0,84 0,84 1,00 J. curcas IP-3P#2 0,75 0,69 0,73 0,69 0,79 0,79 0,87 0,77 0,83 0,81 0,83 1,00 J. curcas IP-3P#1 0,73 0,67 0,68 0,67 0,81 0,81 0,86 0,76 0,84 0,81 0,86 0,86 1,00 J. curcas NP#2 0,73 0,72 0,68 0,71 0,81 0,79 0,80 0,79 0,78 0,74 0,80 0,78 0,81 1,00 J. curcas NP#1 0,79 0,74 0,75 0,70 0,78 0,79 0,83 0,76 0,80 0,77 0,82 0,80 0,79 0,80 1,00 J. curcas IP-3M#1 0,75 0,68 0,71 0,68 0,79 0,76 0,83 0,76 0,79 0,72 0,77 0,80 0,82 0,79 0,84 J. curcas IP-3M#2 0,80 0,71 0,68 0,69 0,78 0,78 0,80 0,79 0,75 0,77 0,80 0,78 0,81 0,74 0,80 J.curcas IP-3M#3 0,82 0,71 0,70 0,69 0,76 0,75 0,80 0,74 0,77 0,74 0,76 0,76 0,77 0,74 0,77 J. curcas IP-3M#4 0,75 0,69 0,66 0,71 0,72 0,75 0,74 0,77 0,77 0,78 0,77 0,75 0,77 0,71 0,74 J. curcas IP-3M#5 0,77 0,74 0,68 0,72 0,72 0,73 0,73 0,76 0,75 0,73 0,73 0,73 0,75 0,77 0,72
DAFTAR PUSTAKA
Ambrosi, D.G., Galla, G., Purelli, M., Barbi, T., Fabbri, A., Lucretti, S., Sharbel, T.F & Barcaccia, G. (2010) DNA Markers and FCSS analyses shed light on the genetic diversity and reproductive strategy of Jatropha curcas L. Diversity, 2, 810 836.
Ariyanto, D. & Utami, R. (2006) Evaluasi laju pertumbuhan, ke-ragaman genetik, dan estimasi heterosis pada persilangan antarspesies ikan patin (Pangasius sp.). Jurnal Perikanan, 8 (1), 81 86.
Azrai, M. (2005) Pemanfaatan markah molekuler dalam proses seleksi pemuliaan tanaman. Jurnal AgroBiogen, 1 (1), 26 37. Basha, S.D., Francis, G., Makkar, H.P.S., Becker, K. & Sujatha, M.
(2009) A comparative study of biochemical traits and molecular markers for assessment of genetic relationships between
Jatropha curcas L. germplasm from different countries. Plant Science, 176, 812 823.
Chaerani, Hidayatun, N. & Utami, D.W. (2011) Keragaman genetik 50 aksesi plasma nutfah kedelai berdasarkan sepuluh penanda mikrosatelit. Jurnal AgroBiogen, 7 (2), 96 105.
Djumali & Nurnasari, E. (2014) Karakter tanaman yang mem-pengaruhi hasil tanaman jarak pagar (Jatropha curcas L.).
Jurnal Agronomi Indonesia, 42 (1), 66 73.
Doyle, J.J. & Doyle, J.L. (1990) Isolation of plant DNA from fresh tissue. Focus, 12, 13 15.
Hapshoh, S., Syukur, M., Wahyu, Y. & Widodo. (2016) Pewarisan karakter kualitatif cabai hias hasil persilangan cabai besar dan cabai rawit. Jurnal Agronomi Indonesia, 44 (3), 286 291. Heller, J. (1996) Physic nut (Jatropha curcas L.). Promoting the
Conservation and Use of Underutilized and Neglected Crops 1. [Online] Available from: https://www.bioversityinternational. org/uploads/tx_news/Physic_nut_Jatropha_curcas_L._161.pdf. [Accessed 12 October 2015].
Hidayatun, N., Chaerani & Utami, D.W. (2011) Sidik jari DNA 88 plasma nutfah ubi jalar di Indonesia berdasarkan delapan penanda SSR. Jurnal AgroBiogen, 7 (2), 119 127.
Hildebrand, C.E., David, C., Torney, C. & Wagner, P. (1992) Informativeness of polymorphic DNA markers. Los Alamos Science, 20, 100 102.
Jingura, R.M. & Kamusoko, R. (2015) Utility of markers for determination of genetic diversity in Jatropha: A review. The Open Renewable Energy Journal, 8, 1 6.
Khrisnan, P.R. & Paramathma, M. (2009) Potentials and Jatropha
species wealth of India. Current Science, 97, 1000 1004. Kumar, A. & Sharma, S. (2008) An evaluation of multipurpose oil
seed crop for industrial uses (Jatropha curcas L.): A review.
Industrial Crops and Products, 1, 1 10.
Laosatit, K., Tanya, P., Saensuk, C. & Srinives, P. (2013) Development and characterization of EST-SSR markers from
Jatropha curcas EST database and their transferability across
Jatropha-related species/genus. Biologia, 68 (1), 41 47. Lazar, I. (2010) Gel Analyzer 2010 User’s Manual 2010. [Online]
Available from: http://www.gelanalyzer.com/downloads/ users_manual_2010.pdf. [Accessed 10 October 2016].
Lightfoot, D.A. & Iqbal, M.J. (2013) Molecular mapping and breeding with microsatellite markers. In: Kantartzi, S.K. (ed.)
Microsatellites Methods and Protocols. Illinois, Humana Press. pp. 297 317.
Liu, K. & Muse, S.V. (2005) PowerMarker: An Integrated Analysis Environment for Genetic Marker Analysis. North Carolina State University, Bioinformatics Research Center.
Madesis, P., Ganopoulos, I. & Tsaftaris, A. (2013) Microsatellites: evolution and contribution. In: Kantartzi, S.K. (ed.) Microsatellites Methods and Protocols. Illinois, Humana Press. pp. 1 13.
Mastan, S.G., Sudheer, P.D.V.N., Rahman, H., Ghosh, A., Rathore, M.S., Prakash, C.R. & Chikara, J. (2013) Molecular characterization of intra-population variability of Jatropha curcas
L. using DNA based molecular markers. Molecular Biology Reports, 39 (4), 4383 4390.
Muakrong, N., Tanya, P. & Srinives, P. (2014) Kamphaeng Saen 1, Kamphaeng Saen 2, and Kamphaeng Saen 3: New ornamental
Jatropha cultivars derived through an interspecific cross.
HortScience, 49 (8),1083 1085.
Rohlf, F.J. (2000) NTSYSpc: Numerical Taxonomy and Multivariate Analysis Sistem. Version 2.1. New York, Exeter Software. Rosado, T.B., Laviola, B.G., Faria, D.A., Pappas, M.R., Bhering,
L.L., Quirino, B. & Grattapaglia, D. (2010) Diversity in a large germplasm collection of the biofuel crop Jatropha curcas L. in Brazil. Crop Science, 50, 2372 2382.
Santoso, T.J., Utami, D.W. & Septiningsih, E.M. (2006) Analisis sidik jari DNA plasma nutfah kedelai menggunakan markah SSR. Jurnal AgroBiogen, 2 (1), 1 7.
Saptadi, D., Hartati, R.R.S., Setiawan, A., Heliyanto, B. & Sudarsono (2011) Pengembangan marka simple sequence repeat untuk Jatropha spp. Jurnal Penelitian Tanaman Industri, 17 (4), 140 49.
Satyawan, D. & Tasma, I.M. (2011) Genetic diversity analysis of
Jatropha curcas provenances using randomly amplified polymorphic DNA markers. Jurnal AgroBiogen, 7 (1), 47 55. Sudheer, D.V.N.P, Sinha, R., Kothari, P. & Reddy, M.P. (2009)
Isolation of novel microsatellites from Jatropha curcas L. and their crossspecies amplification. Molecular Ecology Resources, 9 (1), 431 33.
Sun, Q.B., Li, L.F., Li, Y., Wu, G.J. & Ge, X.J. (2008) SSR and AFLP markers reveal low genetic SSR and AFLP markers reveal low genetic diversity in the biofuel plant Jatropha curcas
in China. Crop Science, 48, 1865 1871.
Tasliah, Rijzaani, H., Hariyadi, T.Z.P., Yuriyah, S., Rebin, Masumah & Silitonga, T.S. (2013) Analisis keragaman genetik 161 aksesi mangga Indonesia menggunakan marka mikrosatelit. Jurnal AgroBiogen, 9 (3), 125 134.
Tasma, I.M., Warsun, A., Satyawan, D. & Martono, B. (2013) Analisis kekerabatan 50 aksesi kelapa sawit (Elaeis guineensis
Jacq.) asal Kamerun berdasarkan marka mikrosatelit. Jurnal AgroBiogen, 9 (1), 19 27.
Vischi, M., Raranciuc, S., Baldini, M., Agrarie, S. & Udine, U. (2013) Evaluation of genetic diversity between toxic and non-toxic
Jatropha curcas L. accessions using a set of simple sequence repeat (SSR) markers. African Journal of Biotechnology, 12 (3), 265 274.
Wen, M., Wang, H., Xia, Z., Zou, M., Lu, C. & Wang, W. (2010) Development of EST-SSR and genomic-SSR markers to assess genetic diversity in Jatropha curcas L. BMC Research Notes, 3, 42.
Wigati, E. (2003) Variasi genetik ikan anggoli (Pristipomoides multidens) berdasarkan pola pita allozim. Biodiversitas, 4 (2), 73 79.
Yadav, H.K., Ranjan, A., Asif, M.H., Mantri, S., Sawant, S.V. & Tuli, R. (2011) EST-derived SSR markers in Jatropha curcas L.: Development, characterization, polymorphism, and transferability across the species/genera. Tree Genetics and Genomes 7, 207 219.
Yusron, E. (2005) Pemanfaatan keragaman genetik dalam pe-ngelolaan sumber daya hayati laut. Oseana, 30 (2), 29 34. Zulfahmi. (2013) Penanda DNA untuk analisis genetik tanaman.