Karakteristik Molekuler dan Patogenesitas Virus H5N1 clade 2.3.2 asal
Indonesia
Dharmayanti NLPI1,HartawanR1,Hewajuli DA1,Hardiman1,Wibawa H2Pudjiatmoko3
1Balai Besar Penelitian Veteriner, Jl. RE Martadinata 30 Bogor 16114
E-mail: [email protected]
2Balai Besar Veteriner Wates Jogjakarta, Jl. Raya Yogya Wates Km. 27 PO Box 18 Wates 55602 3Direktorat Jendral Peternakan, Kementerian Pertanian
(Diterima 4 Maret 2013; disetujui 23 April 2013)
ABSTRACT
Dharmayanti NLPI, Hartawan R, Hewajuli DA, Hardiman, Wibawa H, Pudjiatmoko. 2013. Molecular characteristic and pathogenicity of Indonesian H5N1 clade 2.3.2 viruses. JITV 18(2): 99-113.
The outbreak of disease in late 2012 in Indonesia caused high duck mortality. The agent of the disease was identified as H5N1 clade 2.3.2. The disease caused economic loss to the Indonesian duck farmer. The clade 2.3.2 of H5N1 virus has not previously been identified, so this study was conducted to characterize 4 of H5N1 clade 2.3.2 viruses by DNA sequencing in eight genes segment virus namely HA, NA, NS, M, PB1, PB2, PA and NP. The pathogenicity test of clade 2.3.2 viruses in ducks was compared to clade 2.1.3 viruses which predominat circulating in Indonesia. Results of phylogenetic tree analysis showed that the four of clade 2.3.2 viruses isolated in 2012 was the new introduced virus from abroad. Further analysis showed eight genes were in one group with the clade 2.3.2 viruses, especially those from VietNam and did not belong to Indonesia viruses group. The pathogenicity test in ducks showed that virus H5N1 clade 2.3.2 and clade 2.1.3 have similar clinical symptoms and pathogenicity and cause death in 75% of ducks on days 3-6 after infection.
Key Words: H5N1 Virus, Clade 2.3.2, Clade 2.1.3, Phylogenetic Tree, Pathogenecity
ABSTRAK
Dharmayanti NLPI, Hartawan R, Hewajuli DA, Hardiman, Wibawa H, Pudjiatmoko. 2013. Karakteristik Molekuler dan Patogenesitas Virus H5N1 clade 2.3.2 asal Indonesia. JITV 18(2): 99-113
Wabah kematian pada itik yang disebabkan oleh virus H5N1 clade 2.3.2 pada akhir tahun 2012 di Indonesia, menyebabkan kerugian ekonomis yang berarti bagi peternak itik. Virus H5N1 clade 2.3.2 ini diduga belum pernah diidentifikasi sebelumnya, sehingga pada penelitian ini akan dilakukan karakterisasi genetika dari empat virus H5N1 clade 2.3.2 dengan melakukan sekuensing DNA pada delapan gen yaitu HA, NA, NS, M, PB1, PB2, PA dan NP milik virus tersebut dan dilakukan uji patogenesitas virus pada itik di laboratorium berkeamanan tingkat tinggi Biosafety Level 3 (BSL-3) untuk mengetahui keganasan virus clade 2.3.2 dibandingkan dengan virus clade 2.1.3 yang predominan bersirkulasi di Indonesia. Hasil analisis pohon filogenetika memperlihatkan bahwa empat virus clade 2.3.2 yang diisolasi pada tahun 2012 merupakan introduksi virus dari luar negeri. Analisis selanjutnya memperlihatkan pada delapan gen berada dalam satu kelompok dengan virus kelompok clade 2.3.2 terutama yang berasal dari VietNam dan tidak berada dalam kelompok virus Indonesia. Uji patogenesitas pada itik menunjukkan bahwa virus H5N1 clade 2.3.2 dan clade 2.1.3 meimbulkan gejala klinis dan patogenesitas yang sama dan menimbulkan kematian pada 75% itik pada hari ke 3-6 setelah infeksi.
Kata Kunci: Virus H5N1, Clade 2.3.2, Clade 2.1.3, Pohon Filogenetika, Patogenesitas
PENDAHULUAN
Unggas air diketahui sebagai inang dari semua subtipe virus Influenza A, sehingga memberikan suatu resiko kesehatan yang sangat serius terhadap spesies hewan lain secara luas dan memungkinkan terjadinya
genetic mixing. Meskipun beberapa subtipe virus Influenza A yang terdapat dalam tubuh inang alami bersifat tidak patogen atau tidak virulen tetapi keberadaannya menyebabkan inang alami tersebut
menjadi reservoir AI (Webby et al. 2007; Hewajuli dan
Dharmayanti 2012). Selain itu, unggas air liar yang juga merupakan reservoir alami virus influenza tipe A dan berperan penting terhadap ekologi dan propagasi virus. Dari reservoir ini, virus influenza tipe A biasanya dapat ditularkan ke unggas lain, mamalia termasuk manusia dan dapat menyebabkan wabah penyakit yang sangat parah atau mematikan. Reservoir alami dan inang virus influenza A yang paling heterogen adalah
unggas air terutama Anseriformes (itik, entok dan
angsa) dan Charadriiformes (burung camar (laut),
di dunia (Webster et al. 1992; Hewajuli dan Dharmayanti. 2012).
Di Indonesia, pada bulan September-Nopember 2012 dilaporkan terjadinya kematian tinggi pada itik di daerah Jawa Tengah, Jawa Timur dan DI Yogyakarta dan penyebab wabah diidentifikasi sebagai virus AI subtipe H5N1 clade 2.3.2 (Wibawa et al. 2012). Virus ini sebelumnya belum pernah dilaporkan bersirkulasi di Indonesia, karena pre dominan virus H5N1 di Indonesia adalah virus H5N1 clade 2.1 yang terbagi menjadi clade 2.1.1; 2.1.2; 2.1.3 (WHO/OIE/FAO 2008). Belum banyak diketahui apakah virus ini berasal dari mutasi virus sebelumnya ataukah merupakan introduksi virus dari luar Indonesia. Di Indonesia bersirkulasinya virus AI H5N1 clade 2.3.2 masih menciptakan tanda tanya besar karena belum banyak diketahui karakter genetik dan fenotipik virus ini. Virus clade 2.3.2 telah dilaporkan bersirkulasi di beberapa negara seperti Vietnam, Bulgaria, China, India, Hongkong, Jepang, Korea, Laos, Nepal, Bangladesh, Nepal, Mongolia (Marinova-Petkova et al. 2012; Nagarajan et al. 2012; WHO 2012).
Penelitian ini bertujuan untuk mengetahui karakter genetik pada genom virus yaitu pada delapan gen virus dan biologi virus clade 2.3.2 yang bersirkulasi di Indonesia dibandingkan dengan clade 2.1.3. Dengan diketahuinya karakter virus tersebut, diharapkan dapat memberikan informasi karakter virus AI clade 2.3.2 Indonesia guna mengetahui kemungkinan terjadinya
reassortant virus AI clade 2.3.2 dengan virus clade 2.1 atau virus influenza lainnya yang belum diketahui tingkat patogenisitasnya pada unggas ataupun manusia. Informasi ini diharapkan dapat untuk memprediksi dalam persiapan menghadapi pandemi flu burung yang
kemungkinan dapat terjadi serta up date vaksin sesuai
dengan virus yang bersirkulasi di lapang.
MATERI DAN METODE
Virus
Empat virus yang digunakan pada penelitian ini yaitu virus yang diperoleh dari monitoring sirkulasi virus AI/H5N1 pada bulan November 2012 di pasar
tradisional di Kabupaten Lamongan yaitu
A/Environment/East Java/LBM-LM13/2012 yang
diisolasi dari mesin bubut ayam yang telah diperoleh
pada penelitian sebelumnya (Dharmayanti dan
Hartawan, 2012 data tidak diterbitkan) dan tiga virus AI H5N1 yang dikirim oleh Direktur Kesehatan Hewan – Direktorat Jendral Peternakan ke Bbalitvet pada bulan November 2012 untuk diidentifikasi lebih lanjut. Tiga virus tersebut adalah
A/duck/Sukoharjo/BBVW-1428-9/2012, A/duck/Bantul/BBVW-1443-9/2012, dan
A/duck/Sleman/BBVW-1463-10/2012.
Propagasi virus AI telur pada telur embrio bertunas
spesific pathogen free
Virus AI yang terdapat dalam cairan alantois diekstraksi RNAnya dengan menggunakan QIAmp RNA mini Kit (Qiagen). Hasil ekstraksi berupa RNA dijadikan templat untuk reaksi RT-PCR. Hasil RT-PCR yang positif teridentifikasi virus AI/H5N1 akan dilanjutkan dengan menanam spesimen tersebut pada
telur embrio bertunas specific pathogen free umur 9-11
hari. Hasil positif adanya pertumbuhan virus pada telur SPF sel MDCK akan dilakukan identifikasi ulang dengan RT-PCR menggunakan primer spesifik H5 dan N1 (Lee et al. 2001; Wright et al. 1995). Jika hasil identifikasi ulang menunjukkan positif virus AI/H5N1 maka akan dilakukan sekuensing DNA untuk mengetahui karakter molekuler dari virus-virus tersebut.
Sekuensing DNA dan analisisnya
Isolat AI/H5N1 dari koleksi lapang ataupun isolat kiriman dari Direktur Kesehatan Hewan dilakukan sekuensing DNA. Sekuensing DNA akan dilakukan pada delapan gen virus AI/H5N1. Empat gen akan dilakukan sekensing lengkap sedangkan empat gen lainnya akan dilakukan sekuensing partial sesuai dengan target mutasi. Sekuensing dan strategi sekuensing DNA untuk gen HA, NS dan M dilakukan sesuai dengan Dharmayanti (2009); Hoffmann et al. (2001) dan Senne et al. (1996), untuk gen NA sesuai dengan Komadina (2008, komunikasi pribadi). Primer sekeunsing untuk gen PB1, PB2, NP dan PA didisain oleh Dharmayanti (2012, data tidak dipublikasi). Sekuensing DNA dilakukan dengan mesin Genetyx Analyzer 3130 (Applied Biosystems, USA). Hasil sekuensing akan diverifikasi dan dianalisis dengan
software BioEdit, version 7
(http://www.mbio.ncsu.edu/BioEdit). Pohon
filogenetika dikontruksi dengan neighbor-joining
bootstrap analysis (1000 replicates) menggunakan the Kimura two-parameter model dalam MEGA, versi 4 (http://www.megasoftware.net).
Uji patogenisitas pada Itik
Itik sebelumya diperiksa terhadap adanya antibodi dan keberadaan virus AI/H5N1 sebelum digunakan. Jika diketahui secara antibodi dan PCR negatif maka hewan (ayam/itik) akan diadaptasi selama 3 hari di isolator lab BSL-3. Perlakuan itik dibagi menjadi empat kelompok yaitu (A) diinfeksi virus A/H5N1 clade 2.3.2., (b) clade 2.3.2. dan (c) clade 2.1.3 serta satu kelompok sebagai kelompok kontrol (Naive group).
Semua persiapan inokulasi virus di laboratorium BSL-3. Setiap kelompok terdiri dari 4 ekor itik (3 ekor diinfeksi virus, 1 ekor sebagai infeksi kontak untuk mengetahui apakah virus dapat ditularkan ke itik lainnya) yang dipelihara di isolator BSL-3. Infeksi virus AI/H5N1 pada ayam disesuaikan dengan dosis
106EID50 intra nasal. Setiap hari dilakukan pengamatan
terhadap gejala klinis dan dilakukan pengambilan trakea dan kloaka serta jika ada hewan yang mati akan dilakukan pengambilan organ (trakea, paru-paru, otak, dan usus) untuk direisolasi virus yan berada di organ itik yang diinfeksi virus H5N1. Pengambilan darah dilakukan pada 5 hari sebelum infeksi, dan hari ke- 2, 4, 6, 9, 11, 13, 14 setelah infeksi.
Perlakuan hewan coba:
Itik diberi pakan dan minum ad libitum, diberikan istirahat cukup, suhu nyaman untuk mencegah stress dan pada akhir percobaan jika ada yang belum mati, dibunuh sesuai kaidah/etika pada unggas. Penelitian ini dilakukan di Laboratorium moduler Biosafety Level -3 (BSL 3) milik Balai Besar Penelitian Veteriner, Badan Pengembangan dan Pertanian, kementerian Pertanian pada awal bulan sampai dengan akhir bulan Desember 2012.
HASIL PENELITIAN
Identifikasi dengan RT-PCR subtipe H5N1
Hasil identifikasi dengan RT-PCR menggunakan primer H5 dan N1 memperlihatkan bahwa ke empat virus adalah virus H5N1. Selanjutnya dilakukan sekuensing DNA pada delapan gen yaitu HA, NA, NS1, M, PA, NP, PB1 dan PB2 empat virus yaitu A/Environment/East Java/LBM-LM13/2012, A/duck/
Sukoharjo/BBVW-1428-9/2012, A/duck/Bantul/
BBVW-1443-9/2012), dan A/duck/Sleman/BBVW-1463-10/2012.
Karakter molekuler virus clade 2.3.2
Protein hemagglutinin (HA)
Empat virus yang digunakan dalam penelitian ini
pada analisis multiple sequence alignment asam amino
protein hemagglutinin menunjukkan rangkaian multiple
basic amino acid pada cleavage site gen HA yang sesuai dengan gambaran virus HPAI (Tabel 1). Selain itu, empat virus yang dianalisis pada penelitian ini
masih mengenal avian receptor (α2,3) karena
mempunyai residu asam amino glutamat pada posisi 222 (pada H3 pada posisi 226) dan glisin pada posisi 224 (posisi 228 pada virus H3) (Stevens et al. 2006).
Substitusi sebuah asam amino pada protein
hemaglutinin dapat mengubah spesifisitas pengikatan
sialyl-linkage Neu5Ac2-3Gal (Ac2-3) menjadi Neu5Ac2-3Gal (Ac2-6) atau sebaliknya. Substitusi Ser205Tyr yang terletak pada antigenik D dari
hemaglutinin virus H3 (jaraknya jauh dari reseptor
binding site), menghasilkan perubahan spesifisitas pengikatan reseptor dari 2-3 ke 2-6. Perubahan
Leu226Gln pada pocket receptor binding site juga
mengubah spesifisitas pengikatan reseptor 2-6 ke 2-3. Perubahan ini sangat penting karena berpengaruh pada kemampuan infeksi virus pada inangnya.
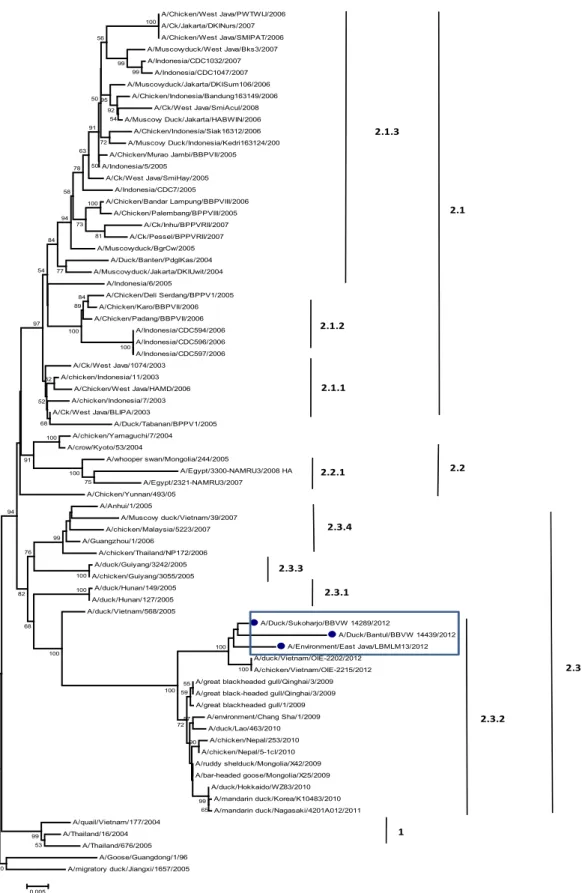
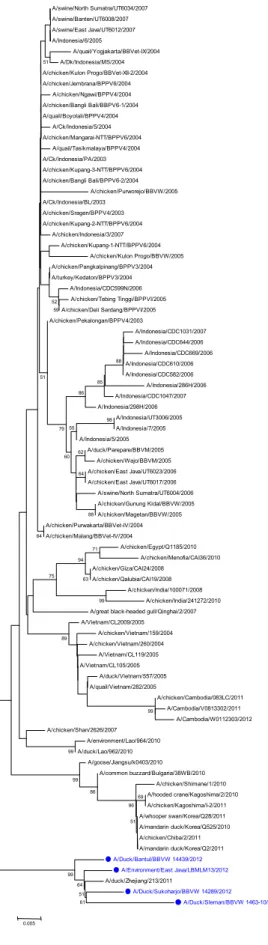
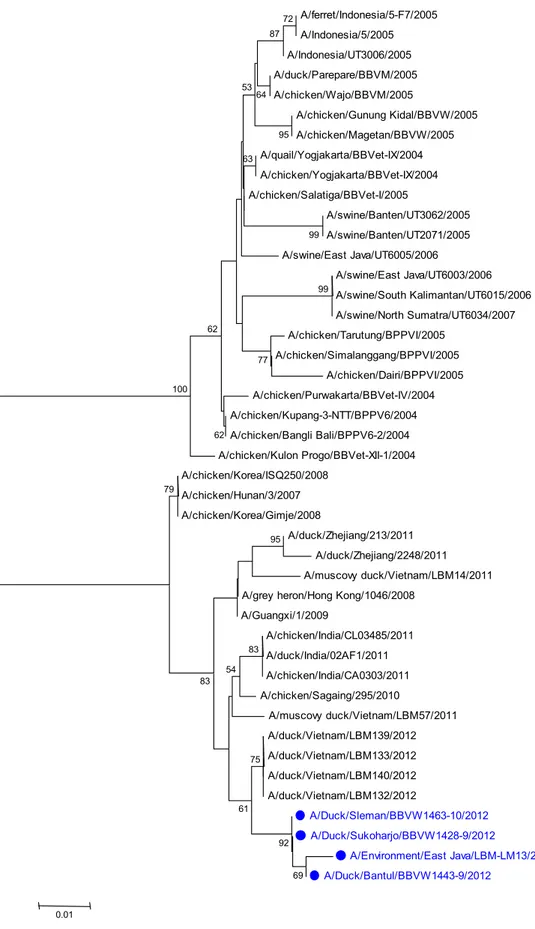
Virus clade 2.3.2 Indonesia mempunyai kesamaan nukleotida 97-98% dengan virus clade 2.3.2 VietNam dan 91-94% dengan virus Indonesia clade 2.1. Analisis phylogenetic gen HA (Gambar 1) memperlihatkan bahwa empat virus tidak berada dalam kelompok virus clade 2.1 namun dalam kelompok virus calde 2.3.2 yang mempunyai kedekatan genetik yang tinggi dengan
virus clade 2.3.2 asal Viet Nam. Virus
A/Environment/East Java/LBM-LM13/2012
memperlihatkan satu kelompok dengan virus clade 2.3.2 lainnya termasuk yang berasal dari Indonesia. Jumlah tempat glikosilasi pada protein HA sebanyak 8 (data tidak ditunjukkan).
Neuraminidase
Seperti halnya virus H5N1 asal Indonesia lainnya, empat virus yang digunakan dalam penelitian
mempunyai 20-residue deletion pada stalk region
(residu 48-68) (Tabel 1). Tempat glikosilasi pada regio
stalk dari protein neuraminidase berperan dalam menjaga struktur tetramer dari protein (Luo et al. 1993). Semua virus pada penelitian ini tidak mempunyai
tempat glikosilasi pada stalk protein neuraminidase
karena delesi di daerah ini. Delesi pada daerah ini akan meningkatkan retensi virion pada membran plasma (Matrosovich et al. 1999).
Non struktural protein
Empat virus ini mempunyai 230 asam amino pada protein NS1 dan terdapat delesi pada posisi 80-84 (Tabel 1). PDZ binding motif mempunyai sekuen ESEV yang menunjukkan avian origin. C-terminal dari virus avian influenza protein NS1 memiliki urutan konsensus dari PDZ domain ligand (Obenauer et al. 2006). Motif yang dapat terikat ke PDZ mengandung protein yang terlibat dalam jalur sinyal selular inang. Virus influenza manusia memiliki berbagai motif seperti RSKV atau RSEV. Motif PDZ pada NS1 merupakan faktor virulensi baru dari virus influenza A (Jackson et al. 2008). Pada penelitian ini, empat dari enam virus yang digunakan mempunyai motif ESEV yang menunjukkan bahwa virus berasal dari spesies unggas.
10 20 30 40 50 60 70 . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . . . . | . .
A/Ck/East Java/Bl-Ipa/2003 EIIFLKICRPTRNEWECRCSDSSDPLVVAASIIGILHLILWILDRLFFKCIYRRLKYGLKRGPSTAGVPESMREEYR A/Indonesia/5/2005 ...KT... A/Indonesia/CDC597/2006 ...N...F... A/duck/Zhejiang/2248/2011 ...E... A/grey heron/Hong Kong/1046/20 ...E... A/duck/Vietnam/LBM140/2012 ...K...E... A/duck/Vietnam/LBM139/2012 ...K...E... A/duck/Vietnam/LBM136/2012 ...K...E... A/Indonesia/CDC610/2006 ...Y...K.I...A...F...M... A/Indonesia/CDC1031/2007 ...Y...K.I...A...F...D... A/Indonesia/CDC1047/2007 ...Y...I...A...N...F... A/duck/Vietnam/OIE-2202/2012 ...I...E... A/chicken/Vietnam/OIE-2215/201 ...I...E... A/duck/Vietnam/27354/2009 ...I...N...E... A/Environment/East Java/LBMLM1 ...I...S...E... A/Duck/Sukoharjo/BBVW 14289/20 ...I...E... A/Duck/Bantul/BBVW 14439/2012 ...I...T...E... A/Duck/Sleman/BBVW 1463-10/201 ...I...E...
Gambar 1. Asam amino yang bertanggung jawab pada sensitivitas amantadin pada posisi 27 dan 31 (dalam kotak)
Tabel 1. Karakter genetik virus H5N1 clade 2.3.2 asal Indononesia
Virus
Asam amino
HA NA NS1 M2 PB2
222 224 20 stalk di 48-68 Delesi di 80-84 PDZ binding motif 27 31 274
Env/East Java/LBM-LM13/12 Q G Ya Ya ESEV I S E
Duck/Sukoharjo/BBVW 14289/12 Q G Ya Ya ESEV I S E Duck/Bantul/BBVW 14439/12 Q G nd Ya ESEV I S E Duck/Sleman/BBVW/ 1463-10/12 Q G Ya Ya ESEV I S E nd: tidak dikerjakan M2 Protein
Protein M2 bertanggung jawab terhadap sensitivitas amantadin. Substitusi 1 asam amino dari 5 asam amino (26, 27, 30, 31 dan 34) pada domain domain transmembran M2 menghasilkan hilangnya sensitivitas protein M2 terhadap amantadin (Hay et al.1986; Pinto et al. 1992). Virus yang digunakan pada penelitian ini menunjukkan residu pada posisi 26L, 27I, 30A dan 31S pada protein M2 sehingga kemungkinan virus baru masih sensitif terhadap amantadin. Sekuen asam amino ini serupa dengan sekuen asam amino virus H5N1 asal Viet Nam (Gambar 1 dan Tabel 1)
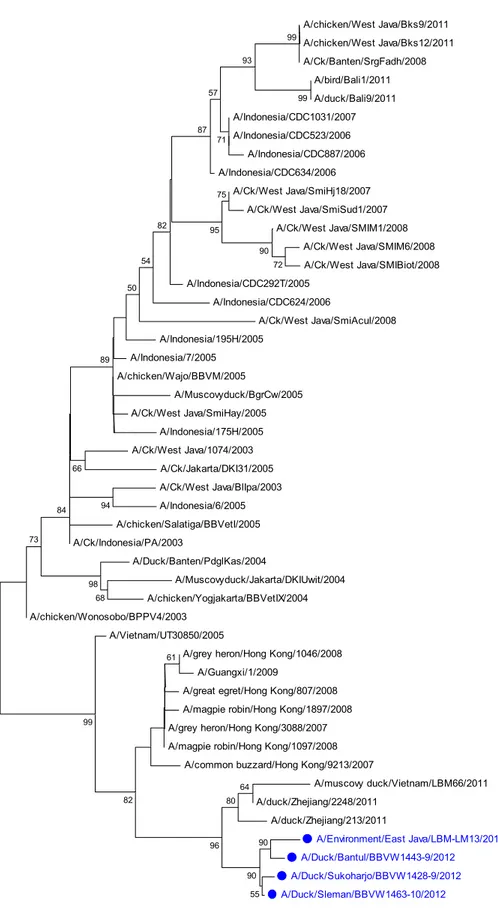
Analisis filogenetika
Untuk mengetahui kedekatan genetik virus baru dengan virus H5N1 lainnya, dilakukan analisis filogenetika pada gen HA, NA, NS1, M1, PA, PB1,
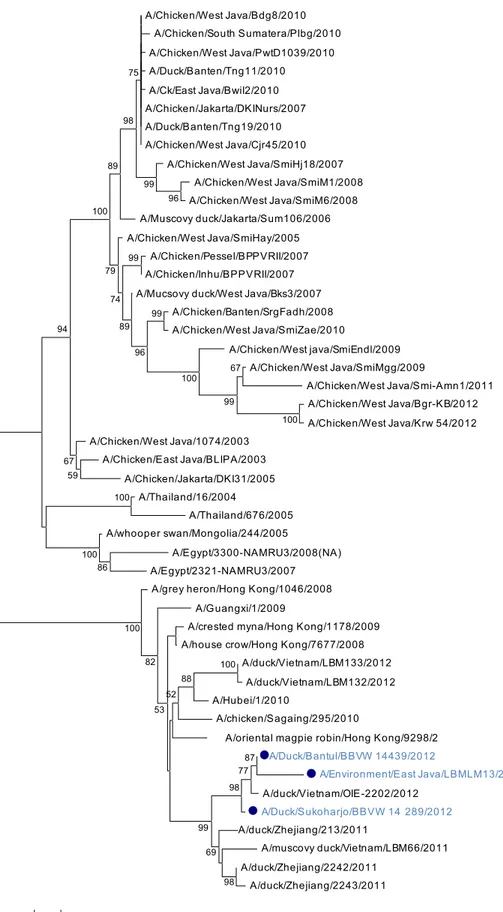
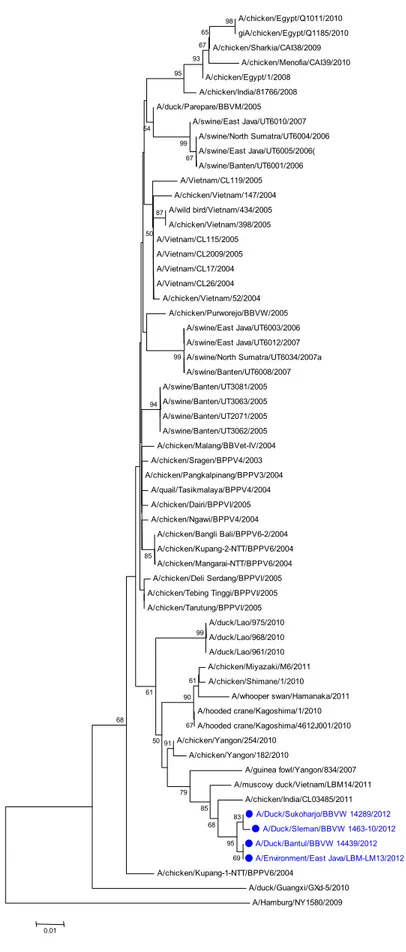
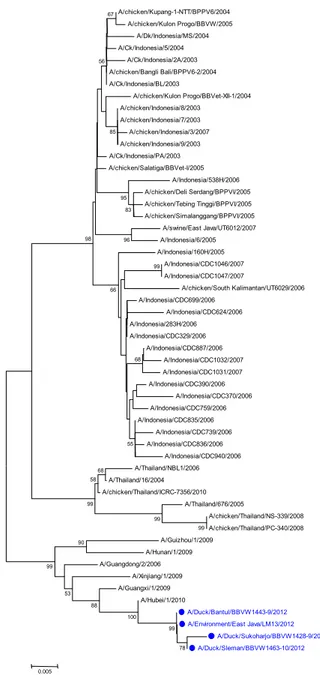
PB2 dan NP (Gambar 2-9). Analisis filogenetika memperlihatkan bahwa virus baru tidak berada dalam satu grup dengan virus H5N1 Indonesia dan virus baru dalam phylogenetic HA berada dalam kelompok clade 2.3.2. (Gambar 2). Hal yang sama terjadi pada pohon filogenetika gen lainnya, semua gen berada dalam satu kelompok dan berdekatan dengan virus asal VietNam, sehingga introduksi virus diduga berasal dari luar Indonesia.
Gambar 2-9. Pohon filogenetika gen Hemagglutinin (HA) gene (Gambar 2), NA (Gambar 3), M1 (Gambar 4), NS1 (Gambar 5), NP (Gambar 6), PA (Gambar 7), PB1 (Gambar 8) dan PB2 (Gambar 9). Warna biru menunjukkan empat virus yang dikarakterisasi pada penelitian ini. Pohon filogenetika dikonstruksi dengan
software MEGA version 4, menggunakan
neighbor-joining bootstrap analysis (1,000 replicates) Tamura- 2 Parameter model.
A/Chicken/West Java/PWTWIJ/2006 A/Ck/Jakarta/DKINurs/2007 A/Chicken/West Java/SMIPAT/2006 A/Muscovyduck/West Java/Bks3/2007 A/Indonesia/CDC1032/2007 A/Indonesia/CDC1047/2007 A/Muscovyduck/Jakarta/DKISum106/2006 A/Chicken/Indonesia/Bandung163149/2006 A/Ck/West Java/SmiAcul/2008 A/Muscovy Duck/Jakarta/HABWIN/2006 A/Chicken/Indonesia/Siak16312/2006 A/Muscovy Duck/Indonesia/Kedri163124/200 A/Chicken/Murao Jambi/BBPVII/2005 A/Indonesia/5/2005 A/Ck/West Java/SmiHay/2005 A/Indonesia/CDC7/2005 A/Chicken/Bandar Lampung/BBPVIII/2006 A/Chicken/Palembang/BPPVIII/2005 A/Ck/Inhu/BPPVRII/2007 A/Ck/Pessel/BPPVRII/2007 A/Muscovyduck/BgrCw/2005 A/Duck/Banten/PdglKas/2004 A/Muscovyduck/Jakarta/DKIUwit/2004 A/Indonesia/6/2005 A/Chicken/Deli Serdang/BPPV1/2005 A/Chicken/Karo/BBPVII/2006 A/Chicken/Padang/BBPVII/2006 A/Indonesia/CDC594/2006 A/Indonesia/CDC596/2006 A/Indonesia/CDC597/2006 A/Ck/West Java/1074/2003 A/chicken/Indonesia/11/2003 A/Chicken/West Java/HAMD/2006 A/chicken/Indonesia/7/2003 A/Ck/West Java/BLIPA/2003 A/Duck/Tabanan/BPPV1/2005 A/chicken/Yamaguchi/7/2004 A/crow/Kyoto/53/2004 A/whooper swan/Mongolia/244/2005 A/Egypt/3300-NAMRU3/2008 HA A/Egypt/2321-NAMRU3/2007 A/Chicken/Yunnan/493/05 A/Anhui/1/2005 A/Muscovy duck/Vietnam/39/2007 A/chicken/Malaysia/5223/2007 A/Guangzhou/1/2006 A/chicken/Thailand/NP172/2006 A/duck/Guiyang/3242/2005 A/chicken/Guiyang/3055/2005 A/duck/Hunan/149/2005 A/duck/Hunan/127/2005 A/duck/Vietnam/568/2005 A/Duck/Sukoharjo/BBVW 14289/2012 A/Duck/Bantul/BBVW 14439/2012 A/Environment/East Java/LBMLM13/2012 A/duck/Vietnam/OIE-2202/2012 A/chicken/Vietnam/OIE-2215/2012 A/great blackheaded gull/Qinghai/3/2009 A/great black-headed gull/Qinghai/3/2009 A/great blackheaded gull/1/2009
A/environment/Chang Sha/1/2009 A/duck/Lao/463/2010 A/chicken/Nepal/253/2010 A/chicken/Nepal/5-1cl/2010 A/ruddy shelduck/Mongolia/X42/2009 A/bar-headed goose/Mongolia/X25/2009 A/duck/Hokkaido/WZ83/2010 A/mandarin duck/Korea/K10483/2010 A/mandarin duck/Nagasaki/4201A012/2011 A/quail/Vietnam/177/2004 A/Thailand/16/2004 A/Thailand/676/2005 A/Goose/Guangdong/1/96 A/migratory duck/Jiangxi/1657/2005 100 100 65 99 90 57 55 59 72 100 100 100 75 100 100 100 53 99 99 99 100 100 99 70 84 89 100 76 68 82 91 94 100 81 54 92 95 72 56 50 50 91 63 73 78 58 94 77 84 82 54 97 68 52 0.005 2.1.2 2.1.1 2.2.1 2.3.3 2.3.4 2.3.1 2.3.2 2.3 2.1 2.1.3 2.2 1
Gambar 2. Pohon filogenetika gen HA, root dengan menggunakan A/Goose/Guangdong/1/96. Isolat yang digunakan pada penelitian ini ditunjukkan dalam kotak
A/Chicken/West Java/Bdg8/2010 A/Chicken/South Sumatera/Plbg/2010 A/Chicken/West Java/PwtD1039/2010 A/Duck/Banten/Tng11/2010 A/Ck/East Java/BwiI2/2010 A/Chicken/Jakarta/DKINurs/2007 A/Duck/Banten/Tng19/2010 A/Chicken/West Java/Cjr45/2010 A/Chicken/West Java/SmiHj18/2007 A/Chicken/West Java/SmiM1/2008 A/Chicken/West Java/SmiM6/2008 A/Muscovy duck/Jakarta/Sum106/2006 A/Chicken/West Java/SmiHay/2005 A/Chicken/Pessel/BPPVRII/2007 A/Chicken/Inhu/BPPVRII/2007 A/Mucsovy duck/West Java/Bks3/2007
A/Chicken/Banten/SrgFadh/2008 A/Chicken/West Java/SmiZae/2010 A/Chicken/West java/SmiEndl/2009 A/Chicken/West Java/SmiMgg/2009 A/Chicken/West Java/Smi-Amn1/2011 A/Chicken/West Java/Bgr-KB/2012 A/Chicken/West Java/Krw 54/2012 A/Chicken/West Java/1074/2003 A/Chicken/East Java/BLIPA/2003 A/Chicken/Jakarta/DKI31/2005 A/Thailand/16/2004 A/Thailand/676/2005 A/whooper swan/Mongolia/244/2005 A/Egypt/3300-NAMRU3/2008(NA) A/Egypt/2321-NAMRU3/2007 A/grey heron/Hong Kong/1046/2008
A/G uangxi/1/2009
A/crested myna/Hong Kong/1178/2009 A/house crow/Hong Kong/7677/2008
A/duck/Vietnam/LBM133/2012 A/duck/Vietnam/LBM132/2012 A/Hubei/1/2010
A/chicken/Sagaing/295/2010
A/oriental magpie robin/Hong Kong/9298/2 A/Duck/Bantul/BBVW 14439/2012 A/Environment/East Java/LBMLM13/2012 A/duck/Vietnam/OIE-2202/2012 A/Duck/Sukoharjo/BBVW 14 289/2012 A/duck/Zhejiang/213/2011 A/muscovy duck/Vietnam/LBM66/2011 A/duck/Zhejiang/2242/2011 A/duck/Zhejiang/2243/2011 100 87 77 100 98 98 69 99 88 52 53 82 100 100 86 100 67 99 100 99 96 59 67 94 96 99 89 74 79 100 99 89 98 75 0.005
A/Ck/Banten/Srg-Fadh/2008 A/chicken/West Java/Bks12/2011 A/chicken/West Java/Bks9/2011 A/Ck/West Java/Smi-Acul/2008 A/bird/Bali1/2011 A/duck/Bali9/2011 A/Chicken/West Java/Krw54/2012 A/Chicken/Wesj Java/BgrKB/2012 A/Indonesia/CDC1031/2007 A/Indonesia/CDC938/2006 A/Indonesia/CDC887/2006 A/Indonesia/CDC1032/2007 A/Ck/West Java/Bks2/2007 A/Muscovyduck/West Java/Bks3/2007 A/Ck/Pessel/BPPVRII/2007 A/Ck/Inhu/BPPVRII/2007 A/Indonesia/245H/2005 A/Indonesia/292H/2006 A/Indonesia/CDC669/2006 A/Indonesia/CDC836/2006 A/Indonesia/542H/2006 A/Duck/Jakarta/DKI-Slmt306/2006 A/Muscovyduck/Jakarta/DKI-Sum106/2006 A/Ck/West Java/Smi-Hj18/2007 A/Ck/Jakarta/DKI-Nurs/2007 A/Bird cucak wilis/Jakarta/Walkot 1/2007 A/Indonesia/CDC1047/2007 A/Ck/Bengkulu/Bkl/2010 A/Indonesia/CDC1046/2007 A/Ck/South Sumatera/PlbgO/2010 A/Ck/West Java/Smi-Sud1/2007 A/Ck/West Java/SMI-M1/2008 A/Ck/West Java/SMI-M6/2008 A/Ck/West Java/SMI-Biot/2008 A/Muscovyduck/Bgr-Cw/2005 A/Ck/West Java/1074/2003 A/chicken/Yogjakarta/BBVet-IX/2004 A/quail/Yogjakarta/BBVet-IX/2004 A/chicken/Purworejo/BBVW/2005 A/Muscovyduck/Jakarta/DKI-Uwit/2004 A/Duck/Banten/Pdgl-Kas/2004 A/Ck/West Java/Smi-Hay/2005 A/Indonesia/CDC7/2005 A/Indonesia/160H/2005 A/Indonesia/CDC184/2005 A/Indonesia/7/2005 A/chicken/Magetan/BBVW/2005 A/chicken/Wajo/BBVM/2005 A/chicken/Gunung Kidal/BBVW/2005 A/duck/Parepare/BBVM/2005 A/Indonesia/569H/2006 A/Indonesia/CDC624/2006 A/Indonesia/CDC623/2006 A/Indonesia/CDC194P/2005 A/Indonesia/175H/2005 A/Indonesia/321H/2006 A/Indonesia/CDC370/2006 A/Indonesia/298H/2006 A/Indonesia/5/2005 A/Indonesia/341H/2006 A/Indonesia/CDC390/2006 A/turkey/Kedaton/BPPV3/2004 A/chicken/Pangkalpinang/BPPV3/2004 A/chicken/Kupang-3-NTT/BPPV6/2004 A/chicken/Indonesia/7/2003 A/quail/Tasikmalaya/BPPV4/2004 A/Ck/Indonesia/BL/2003 A/chicken/Kupang-2-NTT/BPPV6/2004 A/Ck/Indonesia/PA/2003 A/Ck/Indonesia/5/2004 A/Ck/Indonesia/2A/2003 A/Dk/Indonesia/MS/2004 A/chicken/Sragen/BPPV4/2003 A/chicken/Mangarai-NTT/BPPV6/2004 A/chicken/Wonosobo/BPPV4/2003 A/chicken/Pekalongan/BPPV4/2003 A/chicken/Ngawi/BPPV4/2004 A/Ck/East Java/Bl-IPA/2003 A/chicken/Indonesia/11/2003 A/Ck/Jakarta/DKI31/2005 A/chicken/Kupang-1-NTT/BPPV6/2004 A/chicken/Purwakarta/BBVet-IV/2004 A/chicken/Malang/BBVet-IV/2004 A/chicken/Salatiga/BBVet-I/2005 A/Indonesia/CDC596/2006 A/Indonesia/CDC625/2006 A/Indonesia/CDC594/2006 A/chicken/Deli Serdang/BPPVI/2005 A/chicken/Tarutung/BPPVI/2005 A/chicken/Simalanggang/BPPVI/2005 A/chicken/Dairi/BPPVI/2005 A/chicken/Tebing Tinggi/BPPVI/2005 A/Ck/Indonesia/4/2004 A/Thailand/1(KAN-1)/2004 A/dog/Thailand-Suphanburi/KU-08/04 A/chicken/Thailand/ICRC-213/2007 A/mallard/Vietnam/3/2003 A/chicken/Vietnam/4/2003 A/duck/Vietnam/317/2005 A/wild bird/Vietnam/434/2005 A/Vietnam/CL36/2004 A/quail/Vietnam/177/2004 A/goose/Vietnam/264/2004 A/chicken/Hebei/326/2005 A/Duck/Sukoharjo/BBVW1428-9/2012 A/Duck/Sleman/BBVW1463-10/2012 A/Environment/East Java/LBM-LM13/2012 A/Duck/Bantul/BBVW1443-9/2012 A/chicken/Hebei/718/2001 A/chicken/Hebei/108/02 A/Goose/Guangdong/1/96 A/HongKong/483/97 99 57 100 98 99 85 95 84 64 97 82 94 97 73 61 67 53 54 90 60 81 63 97 72 54 85 66 68 87 64 56 53 69 62 59 51 82 64 73 65 66 53 0.01
A/chicken/West Java/Bks9/2011 A/chicken/West Java/Bks12/2011 A/Ck/Banten/SrgFadh/2008 A/bird/Bali1/2011 A/duck/Bali9/2011 A/Indonesia/CDC1031/2007 A/Indonesia/CDC523/2006 A/Indonesia/CDC887/2006 A/Indonesia/CDC634/2006 A/Ck/West Java/SmiHj18/2007 A/Ck/West Java/SmiSud1/2007 A/Ck/West Java/SMIM1/2008 A/Ck/West Java/SMIM6/2008 A/Ck/West Java/SMIBiot/2008 A/Indonesia/CDC292T/2005 A/Indonesia/CDC624/2006 A/Ck/West Java/SmiAcul/2008 A/Indonesia/195H/2005 A/Indonesia/7/2005 A/chicken/Wajo/BBVM/2005 A/Muscovyduck/BgrCw/2005 A/Ck/West Java/SmiHay/2005 A/Indonesia/175H/2005 A/Ck/West Java/1074/2003 A/Ck/Jakarta/DKI31/2005 A/Ck/West Java/BlIpa/2003 A/Indonesia/6/2005 A/chicken/Salatiga/BBVetI/2005 A/Ck/Indonesia/PA/2003 A/Duck/Banten/PdglKas/2004 A/Muscovyduck/Jakarta/DKIUwit/2004 A/chicken/Yogjakarta/BBVetIX/2004 A/chicken/Wonosobo/BPPV4/2003 A/Vietnam/UT30850/2005
A/grey heron/Hong Kong/1046/2008 A/Guangxi/1/2009
A/great egret/Hong Kong/807/2008 A/magpie robin/Hong Kong/1897/2008 A/grey heron/Hong Kong/3088/2007 A/magpie robin/Hong Kong/1097/2008
A/common buzzard/Hong Kong/9213/2007
A/muscovy duck/Vietnam/LBM66/2011 A/duck/Zhejiang/2248/2011 A/duck/Zhejiang/213/2011 A/Environment/East Java/LBM-LM13/2012 A/Duck/Bantul/BBVW1443-9/2012 A/Duck/Sukoharjo/BBVW1428-9/2012 A/Duck/Sleman/BBVW1463-10/2012 90 55 90 64 80 96 61 82 99 99 72 99 90 68 98 93 75 95 71 57 87 94 73 82 54 50 66 84 89 0.005
A/swine/North Sumatra/UT6034/2007 A/swine/Banten/UT6008/2007 A/swine/East Java/UT6012/2007 A/Indonesia/6/2005 A/quail/Yogjakarta/BBVet-IX/2004 A/Dk/Indonesia/MS/2004 A/chicken/Kulon Progo/BBVet-XII-2/2004 A/chicken/Jembrana/BPPV6/2004 A/chicken/Ngawi/BPPV4/2004 A/chicken/Bangli Bali/BBPV6-1/2004 A/quail/Boyolali/BPPV4/2004 A/Ck/Indonesia/5/2004 A/chicken/Mangarai-NTT/BPPV6/2004 A/quail/Tasikmalaya/BPPV4/2004 A/Ck/Indonesia/PA/2003 A/chicken/Kupang-3-NTT/BPPV6/2004 A/chicken/Bangli Bali/BPPV6-2/2004 A/chicken/Purworejo/BBVW/2005 A/Ck/Indonesia/BL/2003 A/chicken/Sragen/BPPV4/2003 A/chicken/Kupang-2-NTT/BPPV6/2004 A/chicken/Indonesia/3/2007 A/chicken/Kupang-1-NTT/BPPV6/2004 A/chicken/Kulon Progo/BBVW/2005 A/chicken/Pangkalpinang/BPPV3/2004 A/turkey/Kedaton/BPPV3/2004 A/Indonesia/CDC599N/2006 A/chicken/Tebing Tinggi/BPPVI/2005 A/chicken/Deli Serdang/BPPVI/2005 A/chicken/Pekalongan/BPPV4/2003 A/Indonesia/CDC1031/2007 A/Indonesia/CDC644/2006 A/Indonesia/CDC669/2006 A/Indonesia/CDC610/2006 A/Indonesia/CDC582/2006 A/Indonesia/286H/2006 A/Indonesia/CDC1047/2007 A/Indonesia/298H/2006 A/Indonesia/UT3006/2005 A/Indonesia/7/2005 A/Indonesia/5/2005 A/duck/Parepare/BBVM/2005 A/chicken/Wajo/BBVM/2005 A/chicken/East Java/UT6023/2006 A/chicken/East Java/UT6017/2006 A/swine/North Sumatra/UT6004/2006 A/chicken/Gunung Kidal/BBVW/2005 A/chicken/Magetan/BBVW/2005 A/chicken/Purwakarta/BBVet-IV/2004 A/chicken/Malang/BBVet-IV/2004 A/chicken/Egypt/Q1185/2010 A/chicken/Menofia/CAI36/2010 A/chicken/Giza/CAI24/2008 A/chicken/Qalubia/CAI19/2008 A/chicken/India/100071/2008 A/chicken/India/241272/2010 A/great black-headed gull/Qinghai/2/2007 A/Vietnam/CL2009/2005 A/chicken/Vietnam/159/2004 A/chicken/Vietnam/260/2004 A/Vietnam/CL119/2005 A/Vietnam/CL105/2005 A/duck/Vietnam/557/2005 A/quail/Vietnam/282/2005 A/chicken/Cambodia/083LC/2011 A/Cambodia/V0813302/2011 A/Cambodia/W0112303/2012 A/chicken/Shan/2626/2007 A/environment/Lao/964/2010 A/duck/Lao/962/2010 A/goose/Jiangsu/k0403/2010 A/common buzzard/Bulgaria/38WB/2010 A/chicken/Shimane/1/2010 A/hooded crane/Kagoshima/2/2010 A/chicken/Kagoshima/I-2/2011 A/whooper swan/Korea/Q28/2011 A/mandarin duck/Korea/Q525/2010 A/chicken/Chiba/2/2011 A/mandarin duck/Korea/Q2/2011 A/Duck/Bantul/BBVW 14439/2012 A/Environment/East Java/LBMLM13/2012 A/duck/Zhejiang/213/2011 A/Duck/Sukoharjo/BBVW 14289/2012 A/Duck/Sleman/BBVW 1463-10/2012 69 51 96 99 61 51 64 99 86 99 99 71 99 63 94 88 98 89 85 88 75 86 64 62 55 60 79 59 52 51 64 51 0.005
A/ferret/Indonesia/5-F7/2005 A/Indonesia/5/2005 A/Indonesia/UT3006/2005 A/duck/Parepare/BBVM/2005 A/chicken/Wajo/BBVM/2005 A/chicken/Gunung Kidal/BBVW/2005 A/chicken/Magetan/BBVW/2005 A/quail/Yogjakarta/BBVet-IX/2004 A/chicken/Yogjakarta/BBVet-IX/2004 A/chicken/Salatiga/BBVet-I/2005 A/swine/Banten/UT3062/2005 A/swine/Banten/UT2071/2005 A/swine/East Java/UT6005/2006 A/swine/East Java/UT6003/2006 A/swine/South Kalimantan/UT6015/2006 A/swine/North Sumatra/UT6034/2007 A/chicken/Tarutung/BPPVI/2005 A/chicken/Simalanggang/BPPVI/2005 A/chicken/Dairi/BPPVI/2005 A/chicken/Purwakarta/BBVet-IV/2004 A/chicken/Kupang-3-NTT/BPPV6/2004 A/chicken/Bangli Bali/BPPV6-2/2004 A/chicken/Kulon Progo/BBVet-XII-1/2004 A/chicken/Korea/ISQ250/2008 A/chicken/Hunan/3/2007 A/chicken/Korea/Gimje/2008 A/duck/Zhejiang/213/2011 A/duck/Zhejiang/2248/2011 A/muscovy duck/Vietnam/LBM14/2011 A/grey heron/Hong Kong/1046/2008
A/Guangxi/1/2009 A/chicken/India/CL03485/2011 A/duck/India/02AF1/2011 A/chicken/India/CA0303/2011 A/chicken/Sagaing/295/2010 A/muscovy duck/Vietnam/LBM57/2011 A/duck/Vietnam/LBM139/2012 A/duck/Vietnam/LBM133/2012 A/duck/Vietnam/LBM140/2012 A/duck/Vietnam/LBM132/2012 A/Duck/Sleman/BBVW1463-10/2012 A/Duck/Sukoharjo/BBVW1428-9/2012 A/Environment/East Java/LBM-LM13/2012 A/Duck/Bantul/BBVW1443-9/2012 69 95 92 83 75 61 54 83 79 99 99 100 95 72 87 77 64 62 62 63 53 0.01
A/chicken/Egypt/Q1011/2010 giA/chicken/Egypt/Q1185/2010 A/chicken/Sharkia/CAI38/2009 A/chicken/Menofia/CAI39/2010 A/chicken/Egypt/1/2008 A/chicken/India/81766/2008 A/duck/Parepare/BBVM/2005 A/swine/East Java/UT6010/2007 A/swine/North Sumatra/UT6004/2006 A/swine/East Java/UT6005/2006( A/swine/Banten/UT6001/2006 A/Vietnam/CL119/2005 A/chicken/Vietnam/147/2004 A/wild bird/Vietnam/434/2005 A/chicken/Vietnam/398/2005 A/Vietnam/CL115/2005 A/Vietnam/CL2009/2005 A/Vietnam/CL17/2004 A/Vietnam/CL26/2004 A/chicken/Vietnam/52/2004 A/chicken/Purworejo/BBVW/2005 A/swine/East Java/UT6003/2006 A/swine/East Java/UT6012/2007 A/swine/North Sumatra/UT6034/2007a A/swine/Banten/UT6008/2007 A/swine/Banten/UT3081/2005 A/swine/Banten/UT3063/2005 A/swine/Banten/UT2071/2005 A/swine/Banten/UT3062/2005 A/chicken/Malang/BBVet-IV/2004 A/chicken/Sragen/BPPV4/2003 A/chicken/Pangkalpinang/BPPV3/2004 A/quail/Tasikmalaya/BPPV4/2004 A/chicken/Dairi/BPPVI/2005 A/chicken/Ngawi/BPPV4/2004 A/chicken/Bangli Bali/BPPV6-2/2004 A/chicken/Kupang-2-NTT/BPPV6/2004 A/chicken/Mangarai-NTT/BPPV6/2004 A/chicken/Deli Serdang/BPPVI/2005 A/chicken/Tebing Tinggi/BPPVI/2005 A/chicken/Tarutung/BPPVI/2005 A/duck/Lao/975/2010 A/duck/Lao/968/2010 A/duck/Lao/961/2010 A/chicken/Miyazaki/M6/2011 A/chicken/Shimane/1/2010 A/whooper swan/Hamanaka/2011 A/hooded crane/Kagoshima/1/2010 A/hooded crane/Kagoshima/4612J001/2010 A/chicken/Yangon/254/2010 A/chicken/Yangon/182/2010 A/guinea fowl/Yangon/834/2007 A/muscovy duck/Vietnam/LBM14/2011 A/chicken/India/CL03485/2011 A/Duck/Sukoharjo/BBVW 14289/2012 A/Duck/Sleman/BBVW 1463-10/2012 A/Duck/Bantul/BBVW 14439/2012 A/Environment/East Java/LBM-LM13/2012 A/chicken/Kupang-1-NTT/BPPV6/2004 A/duck/Guangxi/GXd-5/2010 A/Hamburg/NY1580/2009 98 83 69 95 68 99 85 65 67 93 61 67 90 79 67 99 95 99 91 50 61 87 94 68 85 54 50 0.01
A/chicken/Kupang-1-NTT/BPPV6/2004 A/chicken/Kulon Progo/BBVW/2005 A/Dk/Indonesia/MS/2004 A/Ck/Indonesia/5/2004 A/Ck/Indonesia/2A/2003 A/chicken/Bangli Bali/BPPV6-2/2004 A/Ck/Indonesia/BL/2003 A/chicken/Kulon Progo/BBVet-XII-1/2004 A/chicken/Indonesia/8/2003 A/chicken/Indonesia/7/2003 A/chicken/Indonesia/3/2007 A/chicken/Indonesia/9/2003 A/Ck/Indonesia/PA/2003 A/chicken/Salatiga/BBVet-I/2005 A/Indonesia/538H/2006 A/chicken/Deli Serdang/BPPVI/2005 A/chicken/Tebing Tinggi/BPPVI/2005 A/chicken/Simalanggang/BPPVI/2005 A/swine/East Java/UT6012/2007 A/Indonesia/6/2005 A/Indonesia/160H/2005 A/Indonesia/CDC1046/2007 A/Indonesia/CDC1047/2007 A/chicken/South Kalimantan/UT6029/2006 A/Indonesia/CDC699/2006 A/Indonesia/CDC624/2006 A/Indonesia/283H/2006 A/Indonesia/CDC329/2006 A/Indonesia/CDC887/2006 A/Indonesia/CDC1032/2007 A/Indonesia/CDC1031/2007 A/Indonesia/CDC390/2006 A/Indonesia/CDC370/2006 A/Indonesia/CDC759/2006 A/Indonesia/CDC835/2006 A/Indonesia/CDC739/2006 A/Indonesia/CDC836/2006 A/Indonesia/CDC940/2006 A/Thailand/NBL1/2006 A/Thailand/16/2004 A/chicken/Thailand/ICRC-7356/2010 A/Thailand/676/2005 A/chicken/Thailand/NS-339/2008 A/chicken/Thailand/PC-340/2008 A/Guizhou/1/2009 A/Hunan/1/2009 A/Guangdong/2/2006 A/Xinjiang/1/2009 A/Guangxi/1/2009 A/Hubei/1/2010 A/Duck/Bantul/BBVW1443-9/2012 A/Environment/East Java/LM13/2012 A/Duck/Sukoharjo/BBVW1428-9/2012 A/Duck/Sleman/BBVW1463-10/2012 78 99 99 100 99 88 90 53 99 68 58 99 99 96 83 95 68 85 67 56 98 66 55 0.005
Gambar 9. Pohon filogenetika gen PB2
Uji patogenesitas virus AI subtipe H5N1 pada itik
Uji ini dilakukan untuk mengetahui apakah terdapat perbedaan keganasan/pathogenisitas virus clade 2.3.2 dan 2.1.3 pada itik. Hasil penelitian menunjukkan bahwa kelompok itik yang diinfeksi oleh virus
A/Duck/Sukoharjo/BBVW1428-9/2012 dan A/
Environment/LBM-LM13/2012, (clade 2.3.2) dan virus
A/Chicken/West Java/Krw54/2012 (clade 2.1.3)
menunjukkan gejala klinis yang sama yaitu lesu, lemah, muka bengkak (Gambar 10), perdarahan subkutan dan gejala tortikolis tidak terjadi pada semua itik. Virus
membunuh 50-75% itik. Kematian rata-rata terjadi pada hari ke-3 sampai ke-6 (Gambar 11 dan 12). Shedding virus tidak terjadi pada semua itik (Tabel 2), dari berbagai organ berhasil dideteksi adanya virus H5N1 ketika ditumbuhkan di telur embrio bertunas dan di RT-PCR ulang (data tidak ditunjukkan). Titer antibodi terbentuk pada itik yang hidup sampai akhir penelitian yaitu berkisar antara 4-6 log 2 (Data tidak ditunjukkan). Sampai hari keempat belas sebagian itik masih tetap hidup baik yang diinfeksi oleh virus clade 2.3.2 dan virus clade 2.1.3 meskipun masih tampak sakit (Gambar 13). Sehingga patogenisitas virus clade 2.3.2 dan
Tabel 2.Shedding virus kelompok itik yang diinfeksi virus H5N1
Kelompok itik diinfeksi virus
Shedding virus di kloaka (itik shedding/jumlah itik)
h 1 h 2 h 3 h 4 h 5 h 6 h 7 h 8 h 9 h 10 h 11 h 12 h13 h 17 Sukoharjo/BBVW-1428-9/1 0/4 0/4 0/4 0/4 2/3 2/3 0/3 0/1 0/1 0/1 0/1 0/1 0/1 0/1 LBM-LM13/12 0/4 2/2 2/2 0/2 0/2 0/2 0/2 0/2 0/2 0/2 0/2 0/2 0/2 0/2 Krw54/12 0/4 0/4 0/4 0/4 2/4 2/4 0/2 0/2 0/2 0/2 0/2 0/2 0/2 0/2 Kontrol 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 0/4 h1-h14: hari setelah infeksi
virus clade 2.1.3 dalam penelitian ini tidak
menunjukkan perbedaan. Kedua virus bersifat patogen dan dapat menimbulkan kematian pada itik. Sehingga gejala klinis berupa tortikolis, lemah, lesu, muka bengkak dan terkadang tortikolis pada itik kemungkinan tidak selalu disebabkan oleh virus clade 2.3.2 karena clade 2.1.3 juga dapat menyebabkan hal yang sama. Meskipun virus clade 2.3.2 sekarang sedang aktif
Gambar 10. Gejala muka bengkak terjadi pada itik 2 hari setelah infeksi
Gambar 11. Itik yang diinfeksi virus clade 2.3.2 mengalami tortikolis dan mati pada hari kelima setelah infeks
Gambar 12. Itik yang diinfeksi virus clade 2.1.3 mengalami tortikolis mati pada hari keenam setelah infeksi
Gambar 13. Itik yang hidup pada hari ke-14 setelah infeksi
bersirkulasi di Indonesia, kewaspadaan terhadap virus clade 2.1.3 harus tetap ditingkatkan karena virus ini terbukti telah banyak menimbulkan kematian pada unggas dan manusia, sehingga dengan diidentifikasinya dua clade virus H5N1 di Indonesia memputuhkan perhatian yang serius karena virus ini bersifat patogen dan zoonosis.
PEMBAHASAN
Sejak diidentifikasi di Indonesia pada akhir tahun 2012, virus H5N1 clade 2.3.2 banyak menimbulkan pertanyaan, bagaimanakan patogenisitasnya terhadap itik yang umumnya tahan dengan virus H5N1 dapat mati dan bagaimana cara penanggulangannya serta apakah vaksin yang beredar saat ini dapat digunakan untuk memvaksinasi itik. Pada penelitian ini telah dilakukan karakterisasi genetik virus clade 2.3.2 secara lengkap pada delapan gen dari virus AI. Delapan gen yang dianalisis memperlihatkan bahwa virus clade 2.3.2 merupakan introduksi dari luar Indonesia, sedangkan analisis protein memperlihatkan virus ini masih sensitif terhadap amantadin, berbeda dengan virus clade 2.1.3 yang telah lebih dari 60% resisten terhadap amantadin (Dharmayanti et al. 2010). Namun demikian pada penelitian Lee et al. (2008) clade 2.3.2 di Vietnam
menunjukkan penurunan sensitivitas terhadap
oseltamivir, meskipun tidak resisten terhadap
amantadin. Jumlah tempat glikosilasi pada protein HA dan NA tidak mengalami kenaikan atapun penurunan, sama seperti sebagian besar profil virus H5N1 clade 2.1.3 asal Indonesia. PDZ binding motif pada protein NS1 memperlihatkan virus adalah avian origin karena mempunyai motif ESEV. Selain itu, pada protein NS1 juga mempunyai delesi di 80-84. Dari analisis tersebut, karakter virus clade 2.3.2 masih menunjukkan virus genotipe Z sama dengan virus clade 2.1 sebelumnya.
Pada penelitian ini juga memperlihatkan
patogensitas virus clade 2.3.2 dan clade 2.1.3 adalah sama karena dapat menyebabkan kematian itik pada 3-6 hari post infeksi, gejala klinis dan shedding virus juga tidak terdapat perbedaan antara virus AI clade 2.3.2 dengan clade 2.1.3. Namun yang menarik adalah itik yang hidup tidak ditemukan shedding virus dan ketika shedding tidak terdeteksi, titer antibodi mulai muncul, sehingga data ini kemungkinan dapat digunakan sebagai salah satu dasar program vaksinasi pada itik.
Unggas air termasuk itik dilaporkan sebagian besar menunjukkan resistensi terhadap virus AI sehingga unggas air hanya sebagai reservoir AI tanpa menunjukkan gejala klinis penyakit AI yang jelas atau parah apabila terinfeksi AI. Pada wabah itik yang terjadi di Indonesia pada akhir tahun 2012 dan hasil uji patogenesitas pada penelitian ini memperlihatkan bahwa itik menunjukkan gejala klinis yang jelas dan menimbulkan kematian. Hal ini juga pernah dilaporkan oleh Capua et al (2001) bahwa virus HPAI dapat menimbulkan penyakit yang parah dengan gangguan syaraf sampai dengan kematian pada unggas air. Selain itu, penelitian yang dilakukan oleh Saito et al (2009) juga menunjukkan bahwa virus-virus H5N1 yang
diisolasi dari kasus outbreak di Thailand tidak hanya
mampu menyebabkan kematian pada ayam dan burung
puyuh tetapi juga pada Anseriformes (itik) dengan
gejala klinis perdarahan pada paruh, depresi dan tortikolis sebelum kematian terjadi.
KESIMPULAN
Analisis filogenetika pada delapan gen genom virus memperlihatkan bahwa virus AI subtipe H5N1 clade 2.3.2 asal Indonesia pada bukan berasal dari kelompok virus Indonesia sehingga merupakan introduksi virus dari luar Indonesia. Virus H5N1 clade 2.3.2 mempunyai patogenesitas yang sama dengan virus H5N1 clade 2.1.3 karena mempunyai gejala klinis dan dapat membunuh itik dalam 3-6 hari setelah infeksi. Shedding virus pada itik yang diinfeksi virus clade 2.3.2 dan clade 2.1.3 ditemukan disemua organ namun shedding virus di kloaka hanya terdeteksi pada hari ke 6-7 setelah infeksi.
UCAPAN TERIMA KASIH
Ucapan terima kasih kepada Kepala Badan Litbang Pertanian, Kementerian Pertanian atas dukungannya pada penelitian ini. Terima kasih kepada Drh Rahendra,
Dinas Peternakan Kabupaten Lamongan atas
bantuannya dilapangan dan kepada Bapak Nana Suryana, Teguh Suyatno dan Ace Endang atas bantuan teknisnya. Penelitian ini didanai oleh Badan Litbang Pertanian, Kementerian Pertanian.
DAFTAR PUSTAKA
Capua I, Mutinelli F. 2001. Mortality in Muscovy ducks (Cairina moschata) and domestic geese (Anser anservar domestica) associated with natural infection with a highly pathogenic avian influenza virus of H7N1 subtype. Avian Pathol. 30:179-183.
Dharmayanti NLPI. 2009. Perubahan genom dan karakter virus avian influenza subtipe H5N1 pada unggas di Indonesia (disertasi S3). [Jakarta (Indones)]: Universitas Indonesia.
Dharmayanti NLPI, Ibrahim F, Soebandrio A. 2010. Amantadine resistant of Indonesian influenza H5N1 subtype virus during 2003-2008. Microbiol Indones. 5:11-16. Hay AJ, Zambon MC, Wolstenholme AJ, Skehel JJ, Smith
MH.1986. Molecular basis of resistance of influenza A viruses to amantadine. J Antimicrob Chemother; (8 (suppl B): 19-29.
Hewajuli DA, Dharmayanti NLPI. 2012. Hubungan AI dan unggas air dalam menciptakan keragaman genetik serta peran unggas air sebagai reservoir pada penyebaran virus AI. Wartazoa. 22:12-23.
Hoffmann E, Stech J, Guan Y, Webster, RG, Perez DR. 2001. Universal primer set for the full-length amplification of all influenza A viruses. Arch Virol. 146:2275-2289.
Jackson D, Hossain MJ, Hickman D, Perez DR, Lamb RA. 2008. A new influenza virus virulence determinant: the NS1 protein four C-terminal residues modulate pathogenicity. Proc Natl Acad Sci. 105:4381-4386. Le MTQ, Wertheim HFL, Nguyen HD, Taylor W, Hoang
PVM, Vuong CD, Nguyen HL, Nguyen K, Nguyen HH, Nguyen TQ, TV, Van TD, Ngoc BT, Bui TN, Nguyen, BG, Nguyen LT, Luong S.T., Phan PH, Pham HV, Nguyen, T, Fox A, Nguyen CV, Do HQ, Crusat M, Farrar J, Nguyen HT, de Jong MD, Horb P. 2008. Influenza A H5N1 Clade 2.3.4 virus with a different antiviral susceptibility profile replaced Clade 1 Virus in Humans in Northern Vietnam PLoS ONE. Vol. 3. Issue 10. http://www.ncbi.nlm.nih.gov/pubmed/?term=Influenza+ A+H5N1+Clade+2.3.4+virus+with+a+different+antivir al+susceptibility+profile+replaced+Clade+1+.
Lee MS, Chang PC, Shien JH, Cheng MC, Shieh HP. 2001. Identification and subtyping of avian influenza viruses by reverse transcription-PCR. J Virol Methods. 97:13-22. (Tidak ada dalam teks)
Luo G, Chung J, Palese P. 1993. Alterations of the stalk of the influenza virus neuraminidase: Deletions and insertions. Virus Res. 29. 141-153.
Nagarajan S, Tosh C, Smith DK, Peiris JSM, Murugkar HV, Sridevi R, Kumar M, Katare M, Jain R, Syed Z, Behera P, Cheung CL, Khandia R, Tripathi S, Guan Y, Dubey S.C. 2012. Avian influenza (H5N1) virus of clade 2.3.2 in domestic poultry in India. PLoS ONE. Vol. 7 Issue 2. http://www.ncbi.nlm.nih.gov/pubmed/ 22363750. Marinova-Petkova A, Georgiev G, Seiler P, Darnell S, Franks
J, Krauss S, Ewbby RJ, Webster RG. 2012. Spread of influenza virus A (H5N1) clade 2.3.2.1 to Bulgaria in common Buzzards. Emerg Infect Dis. 18. 10:1596-1602.
Matrosovich M, Zhou N, Kawaoka Y, Webster R. 1999. The surface glycoprotein of H5 influenza viruses isolated from human, chickens and wild aquatic birds have distinguishable properties. J Virol. 73:1146-115. Obenauer JC, Denson J, Mehta PK, Su X, Mukatira S,
Finkelstein DB, Xu X, Wang J, Ma J, Fan Y, Rakestraw KM, Webster RG, Hoffmann E, Krauss S, Zheng J, Zhang Z, Naeve CW. 2006. Large-scale sequence analysis of avian influenza isolates. Science. 311:1576-1580.
Pinto LH, Holsinger LJ, Lamb RA. 1992. Influenza virus M2 protein has ion chennel activity. Cell. 69.517-28. Saito T, Watanabe C, Takemae N, Chaisingh A, Uchida Y,
Buranathai C, Suzuki H, Okamatsu M, Imada T, Parchariyanon S, Traiwanatam N,YamaguchiS. 2009. Pathogenicity of highly pathogenic avian influenza viruses of H5N1 subtype isolated in Thailand for different poultry species. Vet Microbiol. 133:65-74. Senne DA, Panigrahy B, Kawaoka Y, Pearson JE, Suss J,
Lipkind M, Kida H, Webster RG. 1996. Survey of the hemagglutinin (HA) cleavage site sequence of H5 and H7 avian influenza viruses : amino acid sequence at the HA cleavage site as a marker of pathogenicity potential. Avian Dis. 40. 425-437.
Stevens A, Blixt O, Tumpey TM, Taubenberger JK, Paulson JC, Wilson IA. 2006. Structure and receptor specificity of the hemagglutinin from an H5N1 influenza virus. Science. 312:404-410.
Webby RJ, Webster RG, Richt JA. 2007. Influenza viruses in animal wildlife Populations. Curr Top Microbiol Immunol. 315:67-83.
Webster RG, Bean WJ, Gorman OT, Chambers TM, Kawaoka, Y. 1992. Evolution and ecology of influenza A viruses. Microbiol Rev. 56:152-179.
Wibawa H, Prijono WB, Dharmayanti NLPI, Irianingsih SH, Miswati Y, Rohmah A, Andesyha E, Romlah, Daulay RSD, Safitria K. 2012. Investigasi wabah penyakit pada itik di Jawa Tengah, Yogyakarta, dan Jawa Timur: Identifikasi sebuah clade baru virus avian influenza subtipe H5N1 di Indonesia. Bul Lab Vet. 12:2-9. Wright KE, Wilson GAR, Novosad D, Dimock C, Tan D,
Weber JM. 1995. Typing and subtyping of influenza viruses in clinical samples by PCR. J Clinical Microbiology. 33:1180-1184.
WHO/OIE/FAO H5N1 Evolution Working Group. 2008. Toward a unified nomenclature system for highly pathogenic avian influenza virus (H5N1) [conference summary].http://www.who.int/influenza/gisrs_laborator y/h5n1_nomenclature/en.
WHO 2012. Influenza at the human-animal interface. Summary and assessment as of 4 June 2012. http://www.who.int/influenza/human_animal_interface/I nfluenza_Summary_IRA_HA_interface_04June12.pdf.