KLASIFIKASI FRAGMEN METAGENOME MENGGUNAKAN
OBLIQUE DECISION TREE DENGAN OPTIMASI
ALGORITME GENETIKA
ALFAT SAPUTRA HARUN
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2014
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi Fragmen Metagenome Menggunakan Oblique Decision Tree dengan Optimasi Algoritme Genetika adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2014 Alfat Saputra Harun NIM G64100046
ABSTRAK
ALFAT SAPUTRA HARUN. Klasifikasi Fragmen Metagenome Menggunakan Oblique Decision Tree dengan Optimasi Algoritme Genetika. Dibimbing oleh WISNU ANANTA KUSUMA.
Analisis metagenome merupakan salah satu bidang kajian bioinformatika yang penting. Bidang ini terkait dengan analisis sampel genom yang diambil langsung dari lingkungan. Tujuan penelitian ini adalah melakukan klasifikasi fragmen metagenom ke dalam tingkat taksonomi genus menggunakan oblique decision tree yang dioptimasi dengan algoritme genetika. Proses ekstraksi ciri dilakukan dengan menggunakan frekuensi 2-mers, 3-mers, dan 4-mers. Proses klasifikasi diawali dengan membuat model berupa pohon keputusan menggunakan data latih dari 10 organisme yang termasuk ke dalam 3 genus. Dari penelitan yang dilakukan diperoleh pohon keputusan yang dapat digunakan untuk mengklasifikasikan fragmen metagenome baru ke dalam genus yang relevan. Evaluasi dilakukan dengan mengukur akurasi dari model klasifikasi. Model klasifikasi yang dihasilkan dengan metode ini kemudian dibandingkan dengan model yang dihasilkan dengan metode Naïve Bayes dan didapatkan bahwa model yang dihasilkan oleh metode oblique decision tree menghasilkan tingkat akurasi yang lebih baik dibandingkan metode Naïve Bayes, yaitu untuk fitur 3-mers dengan panjang fragmen 0.5 Kbp sampai dengan 10 Kbp metode oblique decision tree menghasilkan rata-rata akurasi 93.06%, sedangkan metode Naïve Bayes memperoleh rata-rata akurasi 89.11%. Dari percobaan menggunakan beberapa jenis fitur didapatkan bahwa semakin tinggi nilai k untuk k-mers maka semakin tinggi rata akurasi yang dihasilkan, dengan fitur 4-mers menghasilkan rata-rata akurasi tertinggi dan fitur 2-mers menghasilkan rata-rata-rata-rata akurasi terendah. Dari penggunaan beberapa jenis panjang fragmen didapatkan bahwa semakin panjang fragmen yang digunakan maka semakin tinggi hasil akurasi yang didapatkan.
Kata kunci: algoritme genetika, klasifikasi, metagenom, oblique decision tree
ABSTRACT
ALFAT SAPUTRA HARUN. Metagenome Fragment Classification Using Oblique Decision Tree with Genetic Algorithm Optimization. Supervised by WISNU ANANTA KUSUMA.
Metagenome analysis is one of the most important bioinformatics field. This field is related to genome sample which is taken directly from environment. The purpose of this research is to classify metagenome fragment into genus taxonomic level using oblique decision tree that optimized by genetic algorithm. Feature extraction is performed using 2-mers, 3-mers, and 4-mers frequencies. Classification process is conducted by creating model as a decision tree using the training data of 10 organisms that belong to 3 different genus. From the research conducted, obtained decision tree that could be used to classify new metagenome fragments to the relevant genus. Evaluation is conducted by measuring the
accuracy of the classification model. Classification model that generated by this method were then compared to the classification model by Naïve Bayes method and from the experiment, model by oblique decision tree obtained higher accuracy than the model of Naïve Bayes, there for the 3-mers feature with 0.5 Kbp to 10 Kbp fragment length oblique decision tree acquired 93.06% accuracy, and for Naïve Bayes method acquired 89.11% accuracy. From the experiment that using several types of features it was acquired that higher k value for k-mers will be resulted in better mean accuracy, with the 4-mers feature acquired highest mean accuracy and 2-mers feature acquired lowest mean accuracy. From using different fragment length it was acquired that the longer the fragment that used resulted in higher accuracy.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI FRAGMEN METAGENOME MENGGUNAKAN
OBLIQUE DECISION TREE DENGAN OPTIMASI
ALGORITME GENETIKA
ALFAT SAPUTRA HARUN
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2014
Penguji: 1 Dr Ir Agus Buono, MSi MKom
PRAKATA
Puji dan syukur ke hadirat Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis bisa menyelesaikan tugas akhir dengan judul Klasifikasi Fragmen Metagenome Menggunakan Oblique Decision Tree dengan Optimasi Algoritme Genetika. Penelitian ini dilaksanakan sejak bulan November 2013 sampai dengan Juni 2014.
Terima kasih penulis ucapkan kepada Bapak Dr Wisnu Ananta Kusuma, selaku pembimbing tugas akhir atas bimbingan dan arahan beliau selama pengerjaan tugas akhir ini. Terima kasih pula kepada kedua dosen penguji, yaitu Bapak Dr Agus Buono dan Bapak Muhammad Asyhar Agmalaro, MKom untuk segala saran dan masukannya. Tidak lupa ucapan terima kasih saya haturkan kepada kedua orang tua dan seluruh keluarga, atas segala doa dan dukungannya, serta kepada teman-teman Ilmu Komputer 47 dan semua pihak yang terkait secara langsung maupun tidak atas dukungannya selama penelitian ini dilakukan.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2014 Alfat Saputra Harun
DAFTAR ISI
DAFTAR TABEL vi DAFTAR GAMBAR vi DAFTAR LAMPIRAN vi PENDAHULUAN 1 Latar Belakang 1 Perumusan Masalah 2 Tujuan Penelitian 2 Manfaat Penelitian 2Ruang Lingkup Penelitian 2
METODE 2
Pengumpulan Data 2
Praproses Data 3
Ekstraksi Fitur 3
Pembagian Data 4
Oblique Decision Tree 4
Pengujian ODT-GA 7
Analisis 8
HASIL DAN PEMBAHASAN 8
Praproses Data 8
Ekstraksi Fitur 8
Pembagian Data 9
Pelatihan ODT-GA 9
Analisis 9
Panjang Fragmen dan Jenis Fitur 10
Perbandingan dengan Metode Naïve Bayes 11
Perhitungan Sensitivity dan Specificity 12
SIMPULAN DAN SARAN 14
Simpulan 14
Saran 15
DAFTAR PUSTAKA 15
DAFTAR LAMPIRAN 16
DAFTAR GAMBAR
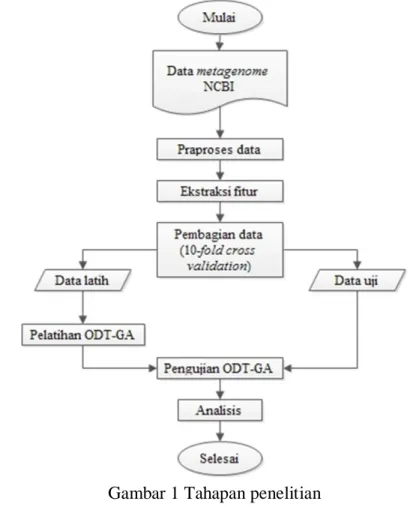
1 Tahapan penelitian 3
2 Contoh ekstraksi fitur menggunakan 3-mers 4
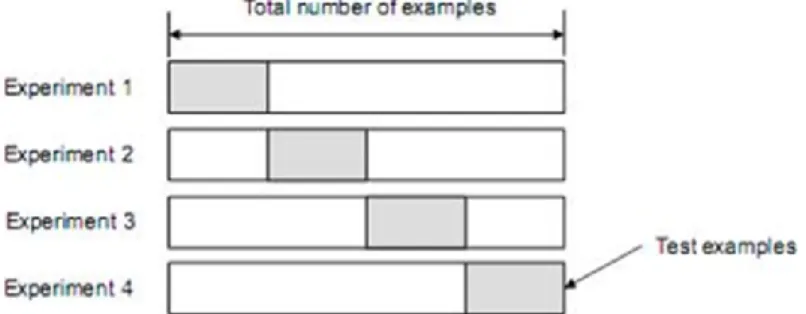
3 Ilustrasi k-fold cross validation 4
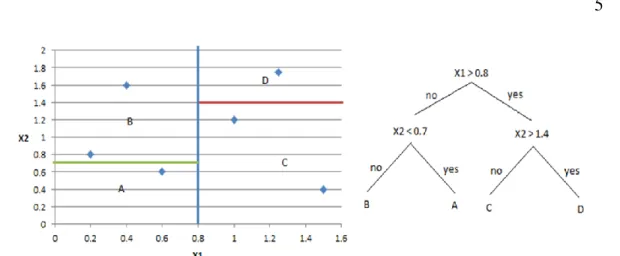
4 Contoh kelompok data yang dipisahkan oleh axis-parallel decision
tree 5
5 Contoh operasi uniform crossover 6
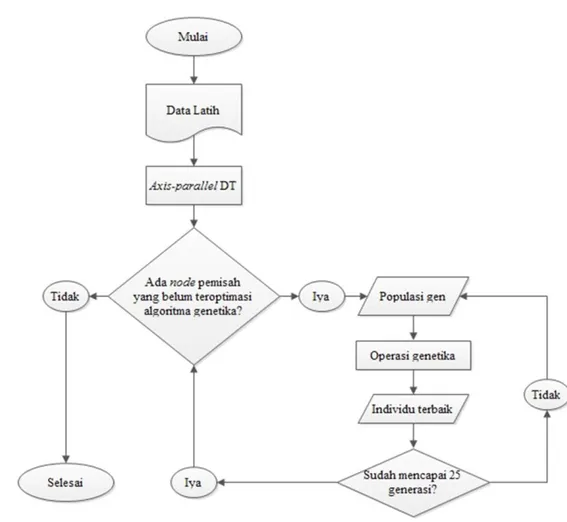
6 Diagram alir proses pembentukan oblique DT 7
7 Contoh isi fail CSV fitur 3-mers untuk panjang fragmen 200 bp 9 8 Perbandingan akurasi fitur 3-mers metode ODT-GA dan Naïve Bayes 11
9 Sensitivity fitur 2-mers 12
10 Sensitivity fitur 3-mers 12
11 Sensitivity fitur 4-mers 12
12 Specificity fitur 2-mers 13
13 Specificity fitur 3-mers 13
14 Specificity fitur 4-mers 13
DAFTAR LAMPIRAN
1 Daftar organisme yang digunakan pada penelitian 16
2 Akurasi metode ODT-GA sebelum penskalaan 16
PENDAHULUAN
Latar Belakang
Analisis metagenome merupakan salah satu bidang kajian bioinformatika yang penting dan terus berkembang. Menurut Handelsman et al. (1998) metagenome adalah kumpulan data genome dari suatu komunitas mikroba di alam dan data tersebut bisa digunakan untuk mengetahui fungsi biologis mikroba-mikroba tersebut. Studi yang mempelajari tentang metagenome disebut metagenomika. Berbeda dengan studi yang mempelajari genom (genomika), metagenomika tidak memerlukan pure clonal cultures dari sequencing individu tertentu. DNA yang berasal dari berbagai organisme dalam suatu komunitas mikrob dapat diperoleh melalui proses sequencing secara langsung (McHardy dan Rigoutsos 2007).
Proses DNA sequencing yang dilakukan secara langsung menghasilkan fragmen-fragmen dari berbagai organisme bercampur. Kondisi ini memungkinkan fragmen dari suatu organisme memiliki overlap dengan fragmen dari organisme lain sehingga dapat mengakibatkan kesalahan perakitan fragmen-fragmen yang terkandung di dalam komunitas tersebut (Wooley et al. 2010). Untuk meminimalkan kesalahan perakitan maka salah satu langkah yang dilakukan adalah binning. Proses binning bisa dilakukan dengan metode supervised learning atau unsupervised learning. Pada metode supervised learning, fragmen-fragmen diklasifikasikan berdasarkan level taksonomi tertentu, misalnya genus.
Beberapa peneliti telah melakukan penelitian terkait dengan pengklasifikasian fragmen metagenome. McHardy et al. (2007) telah membangun aplikasi yang dinamai PhyloPythia. Aplikasi ini mengklasifikasikan fragmen metagenome menggunakan data latih 340 organisme dengan metode support vector machine (SVM) dan frekuensi 5-mers sebagai fiturnya. Hasil akurasi yang diperoleh cukup bervariasi, yaitu untuk panjang fragmen ≥ 5 Kilo base pair (Kbp) antara 60%-90%, untuk panjang fragmen ≤ 3 Kbp sebesar 40%, dan untuk panjang fragmen 1 Kbp diperoleh akurasi < 10%. Penelitian ini kemudian diteliti lebih lanjut oleh Ariny (2013) dengan menggunakan fitur spaced k-mers.
Rosen et al. (2008) kemudian melakukan pengklasifikasian fragmen metagenome yang mengandung 635 organisme. Metode yang digunakan adalah naïve Bayes classifier (NBC) dengan fitur k-mers. Penelitian ini menghasilkan akurasi 38% untuk fragmen dengan panjang 500 bp dengan k = 3, serta akurasi tertinggi 88.8% setelah menggunakan k = 15. Rahmawati (2013) kemudian melakukan penelitian lebih lanjut dengan menambahkan fitur spaced k-mers.
Didasari oleh penelitian-penelitian klasifikasi fragmen metagenome sebelumnya, penelitian ini menggunakan metode lain, yaitu oblique decision tree dengan optimasi algoritme genetika (ODT-GA) dengan fitur 2-mers, 3-mers, dan 4-mers. Penggunaan metode ini diharapkan bisa menghasilkan decision tree yang memberikan akurasi tinggi, sedangkan penggunaan beberapa jenis ekstraksi fitur yang berbeda akan dibandingkan sehingga fitur yang meningkatkan akurasi hasil klasifikasi fragmen metagenome jika menggunakan metode ini dapat diketahui.
2
Perumusan Masalah
Rumusan masalah dalam penelitian ini yaitu besar akurasi yang diperoleh dengan menggunakan metode ODT-GA dan perbandingannya dengan metode Naïve Bayes. Rumusan masalah penelitian ini juga adalah pengaruh panjang fragmen dan jenis fitur yang digunakan terhadap hasil akurasi pengklasifikasian ODT-GA.
Tujuan Penelitian
Tujuan penelitian ini adalah untuk mengklasifikasikan fragmen metagenome ke dalam tingkat taksonomi genus dengan menggunakan metode ODT-GA kemudian membandingkan dengan metode klasifikasi Naïve Bayes. Tujuan lainnya adalah mengetahui pengaruh panjang fragmen dan jenis fitur yang digunakan terhadap hasil akurasi dari klasifikasi ODT-GA.
Manfaat Penelitian
Penelitian diharapkan dapat memberikan kontribusi untuk mendukung proses analisis metagenome sequence pada penelitian-penelitian yang akan datang.
Ruang Lingkup Penelitian
Penelitian yang dilakukan terbatas pada ruang lingkup tertentu, yaitu data yang digunakan hanya terdiri dari 10 organisme yang termasuk ke dalam 3 genus, fragmen yang digunakan mempunyai panjang tetap dan bebas dari sequencing error, serta tingkat taksonomi yang digunakan untuk klasifikasi hanya terbatas pada tingkat genus. Fitur k-mers yang digunakan terbatas pada nilai k = 2, k = 3, dan k = 4. Untuk panjang fragmen yang digunakan hanya dibatasi pada 200 base pair (bp), 500 bp, 1 Kilo bp (Kbp), 5 Kbp, dan 10 Kbp.
METODE
Penelitian dilakukan dalam beberapa tahapan yang diilustrasikan pada Gambar 1.
Pengumpulan Data
Data yang digunakan pada penelitian ini adalah data metagenome yang diunduh dari situs National Centre for Biotechnology Information (NCBI). NCBI merupakan suatu institusi yang fokus di bidang biologi molekuler dan menjadi
3 sumber informasi untuk perkembangan bidang tersebut. Data metagenome ini merupakan sequence DNA organisme dengan format FASTA.
Praproses Data
Sekuens DNA metagenome yang ada diuraikan fragmennya menggunakan perangkat lunak MetaSim (Richter et al. 2008), yaitu suatu perangkat lunak untuk mensimulasikan kerja sequencer. Data yang diproses dibaca berulang kali sesuai dengan kebutuhan penelitian. Pada penelitian ini data yang dipersiapkan berjumlah 3 ribu fragmen dengan rincian jumlah fragmen organisme-organisme yang berada pada genus yang sama adalah 1000 fragmen. Keluaran dari pengolahan MetaSim ini adalah fail FASTA yang berisi sekuens DNA yang sudah terfragmen dengan panjang yang telah ditentukan.
Ekstraksi Fitur
Ekstraksi fitur pada penelitian ini adalah dengan melakukan pembacaan frekuensi nukleotida dengan k-mers pada sekuens DNA yang telah dibangkitkan dengan menggunakan MetaSim. Sebagai contoh, untuk k-mers dengan k = 3 maka dihitung frekuensi kemunculan dari kombinasi nukleotida yang memiliki panjang
4
3, yaitu AAA, AAC, AAT dan seterusnya hingga GGG. Nukleotida terdiri atas empat basa utama , yaitu Adenin, Cytosine, Thymine, dan Guanine yang disingkat menjadi A, C, T, G sehingga dimensi fitur dari k-mers dapat dihitung dengan 4k. Contoh untuk ekstraksi fitur menggunakan fitur 3-mers dapat dilihat pada Gambar 2.
Pembagian Data
Data yang digunakan pada penelitian ini dibagi menjadi dua bagian, yaitu data latih dan data uji. Data yang digunakan terdiri atas 10 organisme yang termasuk pada 3 kelompok genus berbeda. Data latih dan data uji dibagi menggunakan k-fold cross validation dengan k = 10. Metode k-fold cross validation melakukan perulangan sebanyak k kali untuk membagi sebuah himpunan contoh secara acak menjadi k-subset yang saling bebas. Setiap ulangan terdapat satu subset yang digunakan untuk pengujian sedangkan sisanya digunakan untuk pelatihan.
Oblique Decision Tree
Decision tree (DT) merupakan salah satu metode yang populer digunakan untuk klasifikasi. Sebagian besar algoritme pembentukan tree menguji nilai dari suatu atribut tunggal untuk menjadi node pemisah. Pembentukan node seperti hal di atas bisa digambarkan seperti hyperplane yang paralel dengan salah satu sumbu pada dimensi atribut, oleh karena itu tree yang terbentuk disebut juga dengan axis-parallel DT. Bentuk axis-axis-parallel DT mudah digunakan karena model yang dihasilkan bisa lebih mudah dibaca oleh pengguna, namun juga axis-parallel DT dimungkinkan untuk menghasilkan akurasi rendah dikarenakan banyak kelompok data yang tidak bisa dipisahkan dengan hyperplane yang axis-parallel.
Gambar 2 Contoh ekstraksi fitur menggunakan 3-mers
5
Oblique DT menggunakan pengujian nilai dari banyak atribut yang kemudian membentuk suatu hyperplane yang tidak selalu axis-parallel untuk menjadi node pemisah, dan dalam beberapa kasus bisa menghasilkan ukuran tree yang lebih kecil serta akurasi yang lebih baik dibanding axis-parallel DT. Namun sekalipun dengan beberapa kelebihan tersebut, oblique DT masih jarang digunakan dikarenakan kerumitan untuk pembacaan model yang dihasilkan dan juga kebutuhan komputasi yang lebih besar dibanding dengan kebutuhan komputasi untuk membentuk axis-parallel DT.
Oblique DT sendiri menggunakan hyperplane yang tidak selalu axis-parallel untuk memisahkan kelompok data. Jika dicontohkan d adalah banyaknya atribut dari fitur yang digunakan, maka sebuah hyperplane bisa dituliskan sebagai
∑
atau
dengan w adalah vektor suatu koefisien, x adalah vektor nilai atribut fitur, dan b adalah koefisien. Hyperplane dikatakan bisa memisahkan sekelompok data jika dapat ditemukan nilai dari w dan b sedemikian sehingga
∑ ( ) terpenuhi untuk data di atas hyperplane ∑ ( ) terpenuhi untuk data di bawah hyperplane dengan nilai xji adalah nilai fitur atribut ke-i dari data ke-j.
Dalam membentuk oblique DT terlebih dahulu perlu dicari node pemisah yang axis-parallel. Dalam menentukan node pemisah yang paling baik maka harus dipilih atribut yang bisa menjadi pemisah paling murni, yaitu atribut yang dapat memisahkan suatu kelas tanpa tercampur dengan kelas lainnya. Ukuran yang digunakan untuk melihat besaran kemurnian atau purity ini disebut informasi. Untuk mengukur baik tidaknya suatu pemisah maka perlu diketahui nilai selisih antara entropi sebelum dilakukan pemisahan dengan entropi setelah dilakukan pemisahan. Jika k adalah banyak kelas, entropi bisa dihitung dengan cara
6
∑
dengan P(di) adalah peluang dari kelas i. Jika dicontohkan terdapat 2 jenis kelas
dalam suatu kelompok data, yaitu kelas X dan kelas Y. Data kemudian dipisahkan menurut nilai dari suatu atribut sehingga terbagi menjadi dua kriteria. Jika n adalah jumlah data keseluruhan, k1 adalah jumlah data pada kriteria pertama, k2
adalah jumlah data pada kriteria kedua, x adalah jumlah data yang termasuk dalam kelas X, xi adalah jumlah data kelas X pada kriteria ke-i, y adalah jumlah data
yang termasuk dalam kelas Y, yi adalah jumlah data kelas Y pada kriteria ke-i,
maka peningkatan informasi untuk pembagian menurut kriteria tersebut bisa diketahui menurut perhitungan di bawah.
( ) ( )
( )
Setelah ditemukan axis-parallel DT maka dilakukan optimasi dengan algoritme genetika. Gen yang digunakan adalah nilai w dan b yang merupakan koefisien dari hyperplane dan panjang gennya adalah d+1 dengan d adalah jumlah atribut fitur, dalam hal ini fitur yang digunakan adalah frekuensi k-mers, dengan jumlah fitur atau dimensinya 4k. Sebagai contoh, 2-mers akan memberikan jumlah fitur 42 atau sama dengan 16. Pada library yang digunakan algoritme genetika menggunakan prinsip pairwise tournament selection, operator genetika uniform
crossover dengan probabilitas 1 dan tanpa mutasi. Contoh operasi uniform crossover bisa dilihat pada Gambar 5. Ukuran populasi yang digunakan adalah 20 . Populasi awal dibentuk dari axis-parallel hyperplane terbaik yang mengisi 10% dari populasi awal dan sisanya diinisialisasi secara random dengan koefisien wi, b [-200,200]. Algoritme genetika berhenti setelah mencapai 25 generasi.
Sebelum crossover Individu 1 w1 w2 w3 w4 w5 w6 w7 w8 … Individu 2 w1 w2 w3 w4 w5 w6 w7 w8 … Setelah crossover Individu 1 w1 w2 w3 w4 w5 w6 w7 w8 … Individu 2 w1 w2 w3 w4 w5 w6 w7 w8 …
7
Untuk lebih jelasnya langkah pembentukan oblique DT bisa dilihat pada Gambar 6.
Untuk menerapkan oblique DT pada penelitian ini digunakan library DT_oblique yang didapatkan dari perangkat lunak KEEL (Alcalá-Fdezet al. 2009). KEEL merupakan perangkat lunak untuk data mining yang sebagian besar algoritmenya didasarkan pada prinsip evolusi, sedangkan library DT_oblique merupakan implementasi pada KEEL dari penelitian Cantú-Paz dan Kamath (2003) tentang algoritme oblique classifier yang dioptimasi menggunakan algoritme genetika.
Pengujian ODT-GA
Model decision tree yang dihasilkan dari penelitian ini digunakan untuk mengklasifikasikan data uji yang terdiri dari 300 fragmen untuk setiap fold atau perulangan. Untuk setiap perulangan didapatkan akurasi dari fragmen data uji yang ditempatkan pada genus yang sesuai. Hasil akurasi dari tiap perulangan kemudian dirata-ratakan sehingga didapatkan akurasi model yang dihasilkan oleh ODT-GA secara keseluruhan.
8
Analisis
Berdasarkan hasil pengujian diperoleh keakuratan metode yang digunakan dalam pengklasifikasian fragmen metagenome dengan mengukur akurasi dari pengujian data uji dengan rumus:
∑
∑
Selain akurasi, dihitung pula sensitivity dan specificity dari tiap genus dari hasil pengujian. Rumus yang digunakan untuk menghitung nilai sensitivity dan specificity, yaitu:
∑ ∑ ∑
∑ ∑ ∑
HASIL DAN PEMBAHASAN
Praproses Data
Data yang diunduh dari NCBI masih berupa database dari sekuens DNA sehingga perlu diuraikan menjadi fragmen-fragmen menggunakan perangkat lunak MetaSim. Terdapat 10 organisme yang berasal dari 3 genus berbeda yang digunakan pada penelitian ini. Daftar organisme yang digunakan pada penelitian ini dapat dilihat di Lampiran 1.
Pada penelitian ini terdapat beberapa panjang fragmen yang diteliti, yaitu 200 bp, 500 bp, 1 Kbp, 5 Kbp, dan 10 Kbp. Untuk tiap percobaan panjang fragmen, digunakan 3 ribu fragmen dengan rincian 1000 fragmen untuk masing-masing genus.
Ekstraksi Fitur
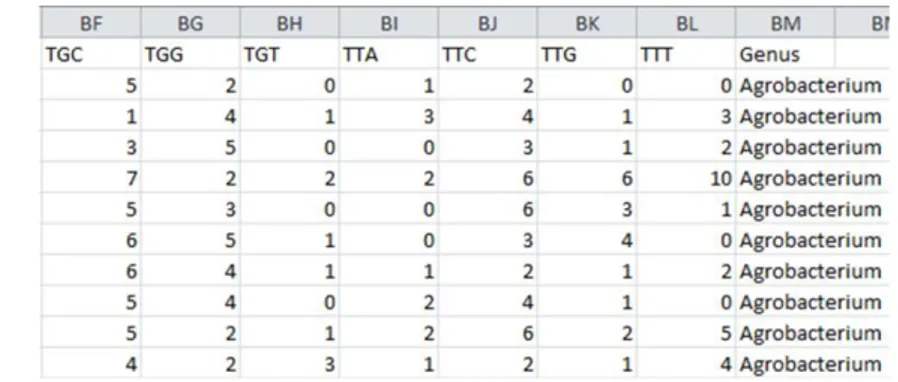
Dari fragmen-fragmen yang telah dihasilkan sebelumnya dilakukan ekstraksi fitur sehingga diperoleh atribut dari fragmen-fragmen tersebut yang dapat digunakan untuk proses klasifikasi. Untuk tiap kelompok panjang fragmen dilakukan 3 jenis ekstraksi fitur, yaitu menggunakan 2-mers, 3-mers, dan 4-mers. Jadi untuk percobaan dengan panjang fragmen 200 bp dihasilkan 3 buah fail berformat comma separated value (CSV) yang masing-masing dibagi menjadi data latih dan data uji kemudian digunakan untuk klasifikasi. Begitu pula untuk panjang fragmen 500 bp, 1 Kbp, 5 Kbp, dan 10 Kbp sehingga total fail CSV yang diperoleh adalah 15. Untuk contoh isi dari fail CSV yang dihasilkan dari ekstraksi fitur, misalnya untuk jenis fitur 3-mers, bisa dilihat pada Gambar 7.
9
Pembagian Data
Dari 3 ribu fragmen yang terbagi 1000 fragmen untuk setiap genus, dilakukan pembagian menjadi data latih dan data uji menggunakan 10-fold cross validation. Jadi terdapat 10 kali perulangan untuk tiap kelompok data dan di setiap perulangan 300 fragmen menjadi data uji sedangkan 2700 lainnya menjadi data latih. Jumlah fragmen yang menjadi data uji sama banyak dari setiap genus, yaitu 100.
Pelatihan ODT-GA
Proses pelatihan ODT-GA diawali dengan menskalakan data latih dan data uji dengan range nilai 0 hingga 1 terlebih dahulu sebelum melakukan pelatihan maupun pengujian. Proses penskalaan ini dilakukan untuk mencegah fitur atau atribut bernilai besar yang bisa mendominasi atribut lain yang bernilai kecil. Proses penskalaan terbukti dapat meningkatkan nilai akurasi dari penelitian ini. Untuk hasil akurasi sebelum penskalaan dapat dilihat pada Lampiran 2. Selanjutnya data latih digunakan untuk menghasilkan model klasifikasi yang mengklasifikasikan data uji.
Analisis
Analisis dilakukan terhadap hasil akurasi yang diperoleh dengan memvariasikan panjang fragmen dan jenis fitur. Kemudian akurasi dari model yang dihasilkan oleh metode ODT-GA dibandingkan dengan akurasi dari model yang dihasilkan oleh metode Naïve Bayes. Dilakukan analisis juga terhadap sensitivity untuk masing-masing genus.
Gambar 7 Contoh isi fail CSV fitur 3-mers untuk panjang fragmen 200 bp
10
Panjang Fragmen dan Jenis Fitur
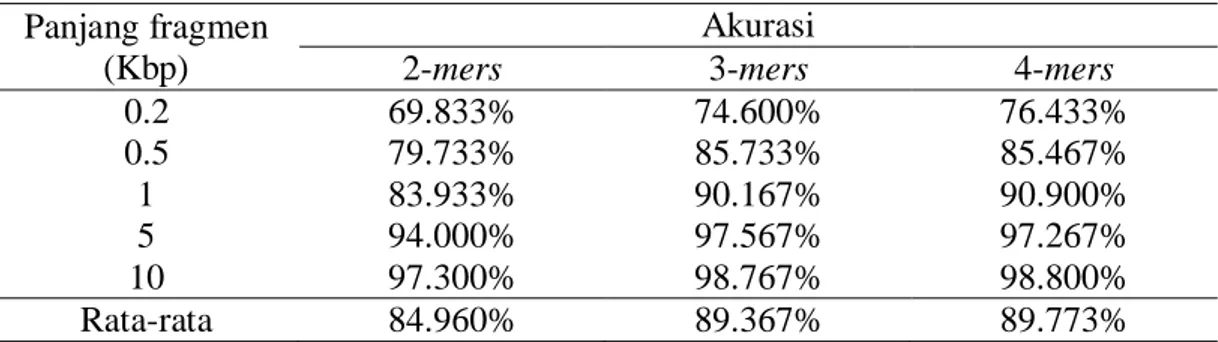
Untuk hasil akurasi berdasarkan jenis fitur, nilai akurasi yang dianalisis merupakan nilai akurasi dari setiap panjang fragmen yang diuji cobakan, yaitu 200 bp, 500 bp, 1 Kbp, 5 Kbp, dan 10 Kbp. Oleh karena itu didapatkan 5 akurasi untuk masing-masing fitur yang digunakan. Hasil akurasi ini ditunjukkan pada Tabel 2.
Panjang fragmen terpendek yang digunakan adalah 0.2 Kbp dan memperoleh akurasi yang cukup baik, yaitu 69.833% untuk fitur 2-mers, 74.6% untuk fitur 3-mers, dan 76.433% untuk fitur 4-mers. Untuk fragmen terpanjang yaitu 10 Kbp didapatkan akurasi tinggi sebesar 97.3% untuk fitur 2-mers, 98.767% untuk fitur 3-mers, dan 98.8% untuk fitur 4-mers. Dilihat dari data yang ada penggunaan fitur 4-mers secara keseluruhan memberikan rata-rata akurasi yang lebih tinggi dibanding jenis fitur lainnya, dengan fitur 4-mers memiliki rata-rata akurasi 89.773%, 3-mers 89.367%, dan 2-mers 84.96%.
Terjadinya peningkatan akurasi dari penggunaan fitur 2-mers ke 4-mers disebabkan oleh jumlah atribut informasi yang tersedia, yaitu pada fitur 2-mers hanya memberikan informasi frekuensi dari kombinasi basa dengan panjang 2, sedangkan fitur 3-mers dan 4-mers memberikan informasi yang lebih baik dengan menghitung frekuensi dari kombinasi basa dengan panjang 3 dan 4. Pada beberapa kasus bisa dilihat bahwa fitur 3-mers memberikan akurasi yang lebih baik dibanding fitur 4-mers. Hal ini diduga disebabkan oleh parameter dari klasifikasi ODT-GA yang belum optimal, seperti peluang crossover atau populasi awal yang digunakan. Keterbatasan library yang digunakan pada penelitian ini belum memberikan keleluasaan bagi pengguna untuk memodifikasi nilai-nilai parameter tersebut.
Tabel 1 Hasil akurasi klasifikasi ODT-GA berdasarkan jenis fitur dan panjang fragmen
Panjang fragmen (Kbp)
Akurasi
2-mers 3-mers 4-mers
0.2 69.833% 74.600% 76.433% 0.5 79.733% 85.733% 85.467% 1 83.933% 90.167% 90.900% 5 94.000% 97.567% 97.267% 10 97.300% 98.767% 98.800% Rata-rata 84.960% 89.367% 89.773%
Berdasarkan data pada Tabel 2, semakin panjang fragmen yang digunakan maka semakin tinggi pula akurasi yang didapatkan. Ekstraksi fitur yang dilakukan pada penelitian ini adalah menghitung frekuensi kemunculan kombinasi basa dengan panjang tertentu, sehingga semakin panjang fragmen yang digunakan, semakin terlihat jelas perbedaan frekuensi kombinasi basa di setiap fragmen dan mempermudah untuk mengklasifikasikan ke dalam suatu genus. Apabila fragmen yang digunakan pendek maka frekuensi dari kombinasi basa banyak terdapat kemiripan.
11
Perbandingan dengan Metode Naïve Bayes
Akurasi dari model klasifikasi yang dihasilkan oleh metode ODT-GA dibandingkan dengan model yang dihasilkan oleh Naïve Bayes. Pemilihan metode Naïve Bayes sebagai pembanding adalah untuk membandingkan dengan metode sebelumnya yang pernah dilakukan oleh Rahmawati (2013) dengan penyesuaian data yang digunakan, dengan jumlah fragmen yang digunakan untuk penelitian ini lebih sedikit dan atribut fitur tidak diproses menggunakan Principal Component Analysis. Hasil yang digunakan untuk perbandingan disesuaikan dengan percobaan sebelumnya, yaitu hasil akurasi dari jenis fitur 3-mers untuk panjang fragmen 500 bp, 1 Kbp, 5 Kbp, dan 10 Kbp.
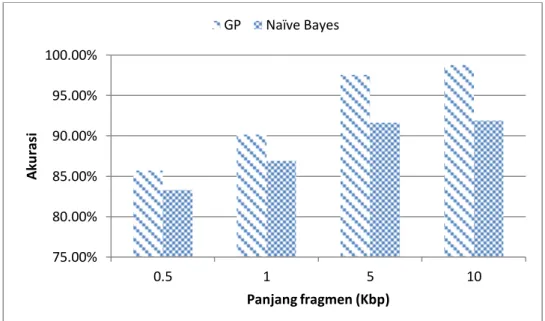
Gambar 8 Perbandingan akurasi fitur 3-mers metode ODT-GA dan Naive Bayes Dari Tabel 3 dapat dilihat bahwa secara keseluruhan hasil akurasi dari model yang dihasilkan oleh ODT-GA lebih tinggi dibandingkan dengan akurasi yang dihasilkan dengan metode Naïve Bayes. Dari rata-rata akurasi, metode ODT-GA memberikan nilai rata-rata akurasi sebesar 89.367% sedangkan metode Naïve Bayes menghasilkan rata-rata akurasi 86.333%. Terlihat pada grafik di atas bahwa selisih akurasi antara metode ODT-GA dengan Naïve Bayes semakin besar seiring dengan bertambahnya panjang fragmen. Nilai selisih akurasi terkecil antara kedua metode berada pada panjang fragmen 500 bp dengan metode ODT-GA mempunyai akurasi > 2.4%. Sementara itu, selisih akurasi terbesar terdapat pada panjang fragmen 10 Kbp dengan metode ODT-GA menghasilkan akurasi > 6.8%. Dari hasil ini dapat disimpulkan bahwa seiring dengan bertambahnya panjang fragmen, metode ODT-GA memberikan hasil klasifikasi yang semakin baik dibandingkan dengan metode Naïve. Nilai akurasi dari metode Naïve Bayes dapat dilihat lebih jelas pada Lampiran 3.
75.00% 80.00% 85.00% 90.00% 95.00% 100.00% 0.5 1 5 10 A ku ra si Panjang fragmen (Kbp) GP Naïve Bayes
12
Perhitungan Sensitivity dan Specificity
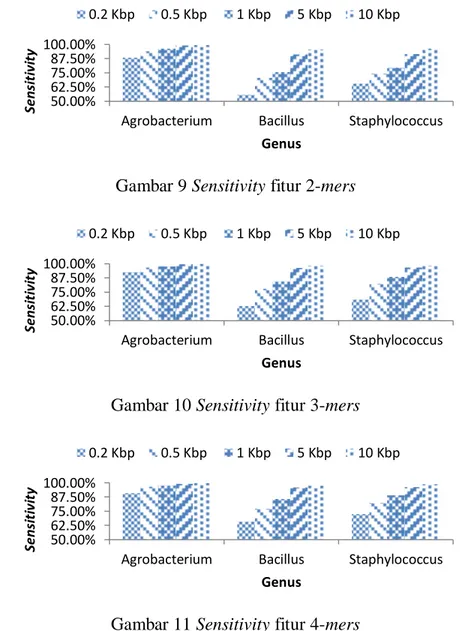
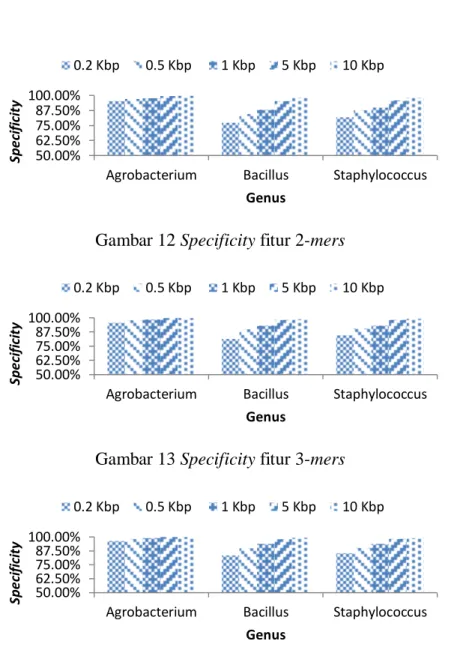
Perhitungan sensitivity dan specificity dilakukan terhadap masing-masing genus untuk melihat akurasi dari model yang dihasilkan terhadap tiap genus. Untuk contoh perhitungan sensitivity, dari 1000 fragmen yang masuk ke dalam genus Bacillus, 973 fragmen berhasil diklasifikasikan ke dalam genus Bacillus, sedangkan 27 lainnya diklasifikasikan ke genus lainnya, maka sensitivity untuk genus Bacillus adalah 973 fragmen yang benar dibagi dengan 1000 fragmen yang seharusnya diklasifikasikan ke dalam genus Bacillus kemudian dikalikan 100%, yang hasilnya 97.3%. Hasil perhitungan sensitivity dapat dilihat pada Gambar 9, 10, 11 dan perhitungan specificity bisa dilihat di Gambar 12, 13, 14.
Gambar 9 Sensitivity fitur 2-mers
Gambar 10 Sensitivity fitur 3-mers
Gambar 11 Sensitivity fitur 4-mers
50.00% 62.50% 75.00% 87.50% 100.00%
Agrobacterium Bacillus Staphylococcus
Se n si ti vi ty Genus 0.2 Kbp 0.5 Kbp 1 Kbp 5 Kbp 10 Kbp 50.00% 62.50% 75.00% 87.50% 100.00%
Agrobacterium Bacillus Staphylococcus
Se n si ti vi ty Genus 0.2 Kbp 0.5 Kbp 1 Kbp 5 Kbp 10 Kbp 50.00% 62.50% 75.00% 87.50% 100.00%
Agrobacterium Bacillus Staphylococcus
Se n si ti vi ty Genus 0.2 Kbp 0.5 Kbp 1 Kbp 5 Kbp 10 Kbp
13
Gambar 12 Specificity fitur 2-mers
Gambar 13 Specificity fitur 3-mers
Gambar 14 Specificity fitur 4-mers
Genus Agrobacterium memperoleh nilai rata-rata sensitivity tertinggi untuk semua jenis fitur, yaitu untuk fitur 2-mers sebesar 95.58%, untuk fitur 3-mers sebesar 97.28%, dan untuk fitur 4-mers sebesar 96.92%. Nilai rata-rata sensitivity terendah diperoleh oleh genus Bacillus, yaitu untuk fitur 2-mers didapat nilai sebesar 77.74%, untuk fitur 3-mers sebesar 83.98%, dan untuk 4-mers sebesar 84.58%. Untuk nilai specificity, genus Agrobacterium memperoleh nilai rata-rata specificity tertinggi dengan nilai untuk fitur 2-mers sebesar 97.93%, untuk fitur 3-mers sebesar 98.44%, dan untuk fitur 4-3-mers sebesar 98.61%. Nilai rata-rata terendah untuk specificity diperoleh genus Bacillus, dengan nilai untuk fitur 2-mers yaitu 88.78%, untuk fitur 3-2-mers sebesar 92.37%, dan untuk fitur 4-2-mers sebesar 92.78%.
Dari percobaan yang telah dilakukan genus Agrobacterium memiliki nilai akurasi yang tinggi pada fragmen pendek dibandingkan dengan genus Bacillus maupun Staphylococcus. Jika dilihat dari taksonominya, Bacillus dan Staphylococcus memiliki kedekatan dalam klasifikasi ilmiahnya, yaitu kedua
50.00% 62.50% 75.00% 87.50% 100.00%
Agrobacterium Bacillus Staphylococcus
Sp ec if ic it y Genus 0.2 Kbp 0.5 Kbp 1 Kbp 5 Kbp 10 Kbp 50.00% 62.50% 75.00% 87.50% 100.00%
Agrobacterium Bacillus Staphylococcus
Sp ec if ic it y Genus 0.2 Kbp 0.5 Kbp 1 Kbp 5 Kbp 10 Kbp 50.00% 62.50% 75.00% 87.50% 100.00%
Agrobacterium Bacillus Staphylococcus
Sp ec if ic it y Genus 0.2 Kbp 0.5 Kbp 1 Kbp 5 Kbp 10 Kbp
14
genus tersebut termasuk pada ordo yang sama yaitu Bacillales, sedangkan Agrobacterium memiliki kesamaan dengan dua genus tersebut hanya pada tingkat kingdom Bacteria. Hal ini menyebabkan genus Bacillus dan Staphylococcus cenderung memiliki kemiripan sehingga dalam klasifikasi yang berfragmen pendek seringkali kedua genus tersebut tertukar dalam penempatannya. Seiring dengan bertambahnya panjang fragmen, akurasi kedua genus tersebut membaik dikarenakan informasi yang didapatkan pada fragmen yang panjang lebih lengkap sehingga bisa lebih mudah dalam mengklasifikasikan antara keduanya
SIMPULAN DAN SARAN
Simpulan
Berdasarkan hasil yang diperoleh dari penelitian yang telah dilakukan, dapat disimpulkan bahwa metode klasifikasi dengan menggunakan ODT-GA dan ekstraksi fitur 2-mers, 3-mers, dan 4-mers berhasil mengklasifikasikan fragmen metagenome ke dalam level taksonomi genus dan memiliki akurasi yang cukup baik, yaitu untuk fragmen 200 bp mendapatkan akurasi > 69.83% dan fragmen 10 Kbp mendapatkan akurasi > 97%. Dari penelitian juga diketahui bahwa penggunaan fragmen panjang memberikan akurasi yang lebih tinggi dibanding penggunaan fragmen pendek.
Metode ODT-GA juga memberikan akurasi yang lebih baik dibanding metode Naïve Bayes. Rata-rata akurasi untuk semua fragmen dengan jenis fragmen 3-mers pada metode ODT-GA lebih tinggi dibandingkan rata-rata akurasi metode Naïve Bayes.
Berdasarkan perhitungan dari nilai sensitivity dan specificity yang telah dilakukan, nilai yang diperoleh untuk sensitivity berkisar dari 55.6% sampai dengan 99.95% sedangkan nilai untuk specificity beriksar dari 77.5% sampai dengan 99.95%. Semakin tinggi nilai sensitivity bermakna bahwa model yang dihasilkan semakin baik mendeteksi bahwa suatu fragmen termasuk dalam genus tertentu, sedangkan semakin tinggi nilai specificity bermakna bahwa model yang dihasilkan semakin baik mendeteksi bahwa suatu fragmen tidak termasuk dalam anggota genus tertentu. Dari nilai sensitivity dan specificity yang diperoleh, dengan nilai specificity lebih tinggi dibandingkan nilai sensitivity, bisa dikatakan bahwa model yang dihasilkan lebih baik dalam hal mengenali bahwa suatu fragmen bukan termasuk dalam suatu genus (true negative) dibanding mengenali bahwa suatu fragmen termasuk dalam kelas genus tertentu (true positive). Untuk contohnya, model yang dihasilkan lebih baik dalam mengenali bahwa suatu fragmen bukanlah termasuk dalam genus Bacillus sehingga fragmen tersebut dikelaskan ke genus selain Bacillus daripada mendeteksi bahwa suatu fragmen termasuk ke dalam genus Bacillus sehingga harus dikelaskan ke dalam genus Bacillus.
15 Saran
Penelitian berikutnya bisa meningkatkan jumlah genus pada data yang digunakan sehingga model yang dihasilkan dapat mengklasifikasikan fragmen-fragmen dari genus lainnya. Saran lainnya adalah mencari jenis fitur lain yang lebih representatif sehingga bisa memberikan akurasi yang lebih baik apabila dilakukan klasifikasi menggunakan ODT-GA, kemudian melakukan tuning dari segi optimasi algoritme genetikanya seperti mengubah nilai probabilitas untuk crossover dan menambahkan operasi mutasi dalam prosesnya.
DAFTAR PUSTAKA
Alcalá-Fdez J, Sánchez L, García S, Jesus MJ, Ventura S, Garrell JM, Otero J, Romero C, Bacardit J, Rivas VM, et al. 2009. KEEL: A software tool to assess evolutionary algorithms to data mining problems. Soft Computing 13(3): 307-318. doi: 10.1007/s00500-008-0323-y.
Ariny. 2013. Klasifikasi fragmen metagenome menggunakan metode Support Vector Machine [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Cantú-Paz E, Kamath C. 2003. Inducing oblique decision trees with evolutionary algorithms. IEEE Transactions on Evolutionary Computation 7(1):54-68. Handelsman J, Rondon MR, Brady SF, Clardy J, Goodman RM. 1998. Molecular
biological access to the chemistry of unknown soil microbes: A new frontier for natural products. Chemistry & Biology 5(10): 245-249.
McHardy AC, Martin HG, Tsirigos A, Hugenholtz P, Rigoutsos I. 2007. Accurate phylogenetic classification of variable-length DNA fragments. Nature Methods. 4(1): 63-72. doi: 10.1038/nmeth976.
McHardy AC, Rigoutsos I. 2007. What’s in the mix: phylogenetic classification of metagenome sequence samples. Current opinion in Microbiology. 10(5): 499-503. doi: 10.1016/j.mib.2007.08.004.
Rahmawati V. 2013. Perbandingan ekstraksi ciri k-mers dan spaced k-mers pada klasifikasi fragmen metagenome dengan Naïve Bayes Classifier [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Richter DC, Ott F, Auch AF, Schmid R, Huson DH. 2008. MetaSim: a sequencing simulator for genomics and metagenomics. PLoS ONE. 3(10): 1-12. doi: 10.1371/journal.pone.0003373.
Rosen G, Garbarine E, Caseiro D, Polikar R, Sokhansanj. 2008. Metagenome fragment classification using n-mer frequency profiles. Advances in Bioinformatics. doi: 10.1155/2008/205969.
Wooley JC, Godzik A, Friedberg I. 2010. A primer on metagenomics. PLoS Computational Biology. 6(2): 1-13. doi: 10.1371/journal.pcbi.1000667.
16
Lampiran 1 Daftar organisme yang digunakan pada penelitian
Spesies Genus Agrobacterium fabrum str. C58 chromosome circular Agrobacterium Agrobacterium radiobacter K84 chromosome 2
Agrobacterium vitis S4 chromosome 1
Bacillus amyloliquefaciens FZB42
Bacillus Bacillus anthracis str. ‘Ames
Ancestor’ chromosome Bacillus cereus 03BB102
Bacillus pseudofirmus OF4 chromosome
Staphylococcus aureus subsp aureus JH1 chromosome
Staphylococcus Staphylococcus epidermis ATCC
12228 chromosome
Staphylococcus haemolyticus JCSC1435 chromosome
Lampiran 2 Akurasi metode ODT-GA sebelum penskalaan Panjang fragmen
(Kbp)
Akurasi
3-mers 4-mers Spaced 3-mers
0.2 74.167% 74.633% 75.133% 0.5 83.800% 84.833% 84.200% 1 89.767% 90.633% 90.400% 5 95.833% 96.867% 97.033% 10 97.267% 98.133% 97.667% Rata-rata 88.167% 89.019% 88.887%
Lampiran 3 Akurasi metode Naïve Bayes
Panjang fragmen (Kbp) Akurasi
3-mers 0.5 84.233% 1 89.800% 5 97.933% 10 98.333% Rata-rata 89.113%
17
RIWAYAT HIDUP
Alfat Saputra Harun lahir pada tanggal 3 Februari 1993 bertempat di Gorontalo, dari pasangan Riansyah Harun dan Trisnawaty Bowta. Pada tahun 2010, penulis lulus dari MAN Insan Cendekia Gorontalo dan diterima sebagai mahasiswa di Departemen Ilmu Komputer, Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB.
Selama menjadi mahasiswa, penulis menjadi asisten praktikum Algoritme Pemrograman (2012) di Departemen Ilmu Komputer. Pada tahun 2013, penulis melaksanakan kegiatan Praktik Kerja Lapangan (PKL) di DataOn (PT. Indodev Niaga Internet) selama 35 hari kerja. Selama masa perkuliahan, penulis juga pernah beberapa kali terlibat dalam proyek baik di dalam maupun di luar kampus, diantaranya adalah pembuatan Sistem E-voting Untuk Pemilihan Raya Fakultas Pertanian Institut Pertanian Bogor dan penerapan Sistem Rekomendasi Pengembangan Layanan Perbankan Untuk Daerah Bogor.