DENGAN MENGGUNAKAN TEKNIK RAPD
IMAM YOGI WIBOWO
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
ABSTRAK
IMAM YOGI WIBOWO. Analisis Keragaman Genetik Tanaman Karet Hasil
Persilangan antara RRIM 600 dan PN 1546 dengan Menggunakan Teknik RAPD.
Dibimbing oleh SURYANI dan TETTY CHAIDAMSARI.
Rendahnya produktivitas karet menjadi permasalahan utama pada
perkebunan karet rakyat di Indonesia. Perakitan genotip unggul baru tanaman
karet memerlukan waktu yang cukup lama yaitu antara 25-30 tahun. Cara yang
tepat dan akurat diperlukan untuk menentukan kualitas klon karet baru secara dini.
Penelitian ini bertujuan untuk melihat keragaman genetik tanaman karet hasil
persilangan antara RRIM 600 dan PN 1546 menggunakan teknik RAPD. Dua
sampel DNA tetua karet dan 25 sampel DNA karet hasil persilangan diisolasi dan
diamplifikasi dengan menggunakan 16 primer kombinasi mTcCIR dan 60 primer
RAPD. Hasil amplifikasi dengan 16 primer kombinasi mTcCIR menghasilkan
kandidat marka seleksi yang spesifik, sedangkan hasil amplifikasi dengan 60
primer RAPD menghasilkan pola pita yang polimorfisme pada gel agarose. Pohon
filogenetik disusun untuk mempelajari kelompok dan jarak kekerabatan tanaman
karet hasil persilangan. Hasil penelitian menunjukkan primer kombinasi ‟d‟ dan
‟i‟ dapat dijadikan kandidat marka yang spesifik untuk RRIM 600. Pohon
filogenetik menunjukkan bahwa 25 tanaman karet hasil persilangan memiliki 10
kelompok kekerabatan yang tinggi dengan koefisien 89%. Kelompok kekerabatan
yang terbentuk memperlihatkan 25 tanaman karet hasil persilangan memiliki
keragaman yang tinggi.
Product between RRIM 600 and PN 1546 using RAPD Technique. Under the
direction of SURYANI and TETTY CHAIDAMSARI.
Low productivity of rubber product had been the main problem in public
rubber plantation in Indonesia. New assembly of excellent genotipe rubber plant
requires 25-30 years. The more appropriate and accurate method is needed to
early determine the new rubber plant clone. This research aimed to shows genetic
variation of rubber plants-crossed product between RRIM 600 and PN 1546 with
RAPD technique. Two DNA samples from rubber plants parent and 25 DNA
samples from rubber plants-crossed products were isolated and amplified by using
16 mTcCIR combination primers and 60 RAPD combination primers.
Amplification by using 16 mTcCIR combination primers produced a specific
selected marker candidates, while amplification by using 60 RAPD primers
produced polymorphism ribbon pattern in agarose gel. Phylogenetic tree was
created based on the ribbon to identify the group and family distances of rubber
plants-crossed product. Result showed that combination primer ‟d‟ and ‟i‟can be
developed into specific marker candidates for RRIM 600. Phylogenetic tree
showed that 25 rubber plants-crossed product have 10 high family groups with
coeficient 89%. The family group showed that 25 rubber plant-crossed product
had high diversity.
ANALISIS KERAGAMAN GENETIK TANAMAN KARET
HASIL PERSILANGAN ANTARA RRIM 600 DAN PN 1546
DENGAN MENGGUNAKAN TEKNIK RAPD
IMAM YOGI WIBOWO
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biokimia IPB
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
NIM
: G84061363
Disetujui,
Komisi Pembimbing
Dr. Suryani, SP, M.Sc
Dr. Tetty Chaidamsari, M.Si
Ketua
Anggota
Diketahui,
Dr. I Made Artika, M.App.Sc
Ketua Departemen Biokimia
PRAKATA
Puji dan syukur penulis panjatkan kehadirat Allah SWT atas rahmat,
berkah, dan karunia-Nya sehingga penulis dapat menyelesaikan penelitian dan
penulisan skripsi ini dengan baik. Sholawat dan salam semoga selalu tercurah
kepada Rasulullah Muhammad SAW beserta keluarga, sahabat, dan para
pengikutnya hingga akhir zaman. Penelitian yang dilakukan penulis mengambil
judul Analisis Keragaman Genetik Tanaman Karet Hasil Persilangan antara RRIM
600 dan PN 1546 dengan Menggunakan Teknik RAPD. Kegiatan penelitian ini
dilakukan dari bulan Januari hingga Juli 2010, bertempat di Laboratorium Biologi
Molekuler dan Rekayasa Genetik, Balai Penelitian Bioteknologi Perkebunan
Indonesia, Jalan Taman Kencana No.1 Bogor.
Penulis mengucapkan terima kasih kepada semua pihak yang telah
membantu dalam penyelesaian penelitian dan penulisan skripsi, terutama kepada
Dr. Suryani, SP. M.Sc selaku pembimbing utama dan Dr. Tetty Chaidamsari,
M.Si selaku pembimbing lapangan yang telah memberikan saran, kritik, dan
bimbingannya. Serta Mba Nina Yuniar dan Mba Herti Sugiarti atas peran dan
kerjasamanya. Selain itu, penulis juga mengucapkan terimakasih kepada kedua
orang tua untuk semua do'a, dukungan, semangat, dan kasih sayangnya yang
sangat berarti bagi penulis. Serta kepada Haya, Sapto, Bayu, Arief Purwo,
Rahmat, Naila, Dhita, Ria, Ainun, Zessy, Iman, Satrio, Akbar, Haryadi, Mas Diki
dan teman-teman yang lainnya di DPM KM IPB 2009-2010, Biokimia „43, dan
Pondok Al-Izzah yang tidak dapat disebutkan satu persatu atas segala bantuan,
saran, dan motivasi yang diberikan. Penulis berharap semoga hasil penelitian ini
dapat bermanfaat bagi semua orang yang memerlukannya.
Bogor, November 2010
Penulis dilahirkan di kabupaten kecil di perbatasan Jawa Barat dan Jawa
Tengah tepatnya di Kabupaten Cilacap pada tanggal 17 Desember 1987 dari Ayah
bernama Hadi Sumaryo dan Ibu bernama Sayidah. Penulis merupakan anak
pertama dari tiga bersaudara.
Tahun 2006 penulis menyelesaikan pendidikan Sekolah Menengah Atas di
SMA Negeri 1 Cilacap dan pada tahun yang sama melanjutkan studi di Institut
Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI). Di IPB
penulis mengambil Mayor Biokimia dari Departemen Biokimia, Fakultas
Matematika dan Ilmu Pengetahuan Alam dengan Minor Manajemen Fungsional
dari Departemen Manajemen, Fakultas Ekonomi dan Manajemen.
Selama mengikuti perkuliahan penulis pernah menjadi asisten mata kuliah
Pendidikan Agama Islam untuk mahasiswa Tingkat Persiapan Bersama tahun
ajaran 2007/2008 dan asisten praktikum Struktur dan Fungsi Subseluler untuk
mahasiswa S1 Biokimia tahun ajaran 2009/2010. Penulis juga aktif dalam
kegiatan organisasi kemahasiswaan di IPB, antara lain: penulis pernah menjadi
Ketua Komisi E Dewan Perwakilan Mahasiswa TPB IPB periode 2006/2007, Staf
Divisi Hubungan Luar LDK DKM Al-Hurriyyah IPB, Ketua Dewan Perwakilan
Mahasiswa FMIPA IPB periode 2007/2008, Wakil Ketua Dewan Perwakilan
Mahasiswa KM IPB periode 2008/2009, dan Ketua Dewan Perwakilan
Mahasiswa KM IPB periode 2009/2010. Selain itu, penulis juga pernah aktif
dalam beberapa kepanitiaan seperti panitia Masa Perkenalan Kampus Mahasiswa
Baru tahun 2007, panitia pelatihan jurnalistik with Metro TV tahun 2006, panitia
Open House IPB tahun 2007, dan beberapa kepanitiaan lainnya. Penulis
melakukan Praktik Lapang di Laboratorium Biologi Molekuler dan Rekayasa
Genetik, Balai Penelitian Bioteknologi Perkebunan Indonesia, Jalan Taman
Kencana No.1 Bogor dengan judul Teknik Random Amplified Polymorphic DNA
untuk Seleksi Dini Penyakit Layu pada Tanaman Jahe.
DAFTAR ISI
Halaman
DAFTAR GAMBAR …………...……...………..
ix
DAFTAR LAMPIRAN ……….
x
PENDAHULUAN .………...………
1
TINJAUAN PUSTAKA
Karet ………..
1
Klon Tanaman Karet ……….
2
Keragaman Genetik ………...
3
Marka DNA ………...
4
Polymerase Chain Reaction ...
4
Mikrosatelit ...
5
Random Amplified Polymorphic DNA ………...………
5
BAHAN DAN METODE
Alat dan Bahan ………..
6
Metode ………...
6
HASIL DAN PEMBAHASAN
Hasil Isolasi DNA Karet …...……….
8
Produk PCR DNA Karet dengan Primer Kombinasi (Semi
Mikrosatelit)….……...………...
8
Hasil Amplifikasi 25 Progeni Hasil Persilangan dengan
Primer Kombinasi Terpilih ………
10
Hasil RAPD Karet ..………...
13
Analisis Pola Konstruksi Filogenetik ….………...
13
SIMPULAN DAN SARAN
Simpulan ………
17
Saran ………..
17
DAFTAR PUSTAKA ...
17
Halaman
1
Tanaman karet (Hevea barasiliensis Muell Arg.) ...
2
2
Elektroforegram DNA 25 progeni karet hasil isolasi ...
9
3
Elektroforegram hasil amplifikasi 16 primer kombinasi terhadap
(a) PN1546; (b) RRIM 600………..
10
4
Elektroforegram hasil amplifikasi primer kombinasi c
terhadap 25 progeni ...………..
11
5
Elektroforegram hasil amplifikasi primer kombinasi d
terhadap 25 progeni ...………..
11
6
Elektroforegram hasil amplifikasi primer kombinasi i
terhadap 25 progeni ...………..
11
7
Elektroforegram hasil amplifikasi primer kombinasi m
terhadap 25 progeni ...………..
12
8
Elektroforegram hasil amplifikasi primer kombinasi n
terhadap 25 progeni ...………..
12
9
Hasil RAPD pada RRIM 600 dengan menggunakan primer OPD,
OPM, dan OPN ………
14
10
Pohon filogenetik 25 progeni hasil persilangan dengan 2 tetua tanaman
karet berdasarkan pola pita hasil RAPD menggunakan 60 jenis primer..
16
DAFTAR LAMPIRAN
Halaman
1
Tahap penelitian ...
21
2
Tahap amplifikasi DNA dengan marka RAPD ……….
22
3
Tahap amplifikasi DNA dengan primer kombinasi ………..
23
4
Daftar sekuen primer yang digunakan dalam penelitian ……….
24
5
Hasil pengukuran kuantitatif DNA dengan spektrofotometer ………..
25
6
Matriks kemiripan genetik antara 27 genotip tanaman karet
berdasarkan proporsi fragmen yang dimiliki secara bersama ...………
26
7
Elektroforegram hasil RAPD Karet dengan Primer OPA ……….
27
8
Elektroforegram hasil RAPD Karet dengan Primer OPB ……….
29
PENDAHULUAN
Karet (Hevea brasiliensis Muell Arg.) merupakan tanaman perkebunan yang sangat penting dan bernilai ekonomis tinggi bagi Indonesia. Akan tetapi, permasalahan utama yang dihadapi oleh perkebunan karet alam Indonesia adalah rendahnya produktivitas dan kualitas karet. Rendahnya produktivitas karet alam Indonesia disebabkan sebagian besar atau lebih dari 84% perkebunan karet yang ada merupakan perkebunan karet rakyat yang tidak dikelola secara profesional. Sisanya sekitar 16% merupakan perkebunan milik negara atau perkebunan besar yang dikelola secara profesional (Setiawan & Agus 2008).
Produktivitas perkebunan karet rakyat hanya sekitar 610 kg/ha/tahun, padahal produktivitas perkebunan besar negara atau swasta masing-masing mencapai 1107 kg dan 1190 kg/ha/tahun (Direktorat Jenderal Bina Produksi Perkebunan 2004). Menurut Setiawan dan Agus (2008), produktivitas karet yang dikelola oleh rakyat jauh lebih rendah dari pada produktivitas karet milik negara atau perusahaan besar karena pengelolaannya sangat berbeda. Salah satu faktor penyebabnya adalah bibit yang ditanam di perkebunan rakyat umumnya bukan berasal dari klon karet unggul, sehingga produktivitasnya rendah. Nilai ekonomis karet terletak pada kemampuannya dalam menghasilkan lateks, sedangkan produk nonlateks seperti kayu dianggap sebagai hasil samping terutama untuk kayu bakar (Boerhendhy & Dwi 2006). Oleh karena itu perlu dilakukan kegiatan pemuliaan tanaman karet untuk memperoleh karasteristik klon tanaman karet dengan produksi lateks yang tinggi.
Tantangan yang dihadapi dalam proses seleksi pada tanaman karet dengan potensi produksi lateks tinggi melalui perakitan genotipe unggul baru memerlukan waktu yang cukup lama yaitu antara 25-30 tahun. Oleh karena itu diperlukan cara yang tepat dan akurat untuk menentukan kualitas klon karet baru secara dini. Penggunaan parameter seleksi marka molekuler yaitu marka DNA dengan teknik Random
Amplified Polimorphic DNA (RAPD)
diharapkan dapat menjawab tantangan tersebut. Penelitian ini bertujuan untuk melihat keragaman genetik tanaman karet hasil persilangan klon karet antara RRIM 600 dengan PN 1546 dengan menggunakan teknik RAPD. Berdasarkan teknik RAPD akan didapatkan pola pita yang bersifat polimorfik
yang kemudian direkonstruksi menjadi pohon filogenetik untuk melihat tingkat kekerabatan dari hasil persilangan tanaman karet antara RRIM 600 yang memiliki produksi lateks yang tinggi dengan PN 1546 yang memiliki jenis kayu yang baik. Penelitian ini diharapkan dapat memberikan informasi ilmiah mengenai tingkat keragaman genetik hasil persilangan klon karet antara RRIM 600 (tetua betina) dengan PN 1546 (tetua jantan) sehingga dapat diketahui progeni (anakan) yang unggul dan progeni tersebut dapat digunakan sebagai klon karet anjuran baru. Hipotesis dari penelitian ini adalah teknik RAPD dapat digunakan untuk melihat keragaman genetik tanaman karet hasil persilangan klon karet antara RRIM 600 dan PN 1546.
TINJAUAN PUSTAKA
Karet
Karet (Hevea brasiliensis Muell Arg.) merupakan tanaman yang berasal dari Amerika Latin, khususnya Brasil. Menurut sistem klasifikasi, tanaman karet berada pada
divisi Spermatophyta, subdivisi
Angiospermae, kelas Dycotyledonae, ordo Euphorbiales, family Euphorbiaceae, genus Hevea, dan spesies Hevea brasiliensis
(Setiawan & Agus 2008). Tanaman karet merupakan pohon yang tingginya dapat mencapai 25 meter dengan diameter batang cukup besar. Batang karet pada umumnya tumbuh lurus ke atas dengan percabangan di bagian atas. Daun karet terdiri dari tangkai utama sepanjang 3-20 cm dan tangkai anak daun sepanjang 3-10 cm dengan kelenjar di ujungnya. Setiap daun karet biasanya terdiri dari tiga anak daun yang berbentuk elips memanjang dengan ujung runcing. Daun karet berwarna hijau dan menjadi kuning atau merah menjelang rontok. Karet memiliki buah dengan diameter 3-5 cm dan memiliki pembagian ruangan yang jelas, biasanya 3-6 ruang (Gambar 1) (Setiawan & Agus 2008).
Produk utama yang dipanen dari tanaman karet adalah lateks. Tanaman karet menghasilkan lateks dengan kuantitas dan kualitas yang terbaik sehingga merupakan spesies tanaman karet yang sangat menguntungkan secara komersial (Astuti 2008). Menurut Sumarmadji (2001), tanaman karet yang produktif menghasilkan karet, normalnya melakukan regenerasi lateks 3-4 hari setelah penyadapan. Siswanto (1994) menyebutkan bahwa lateks dibentuk dan
2
terakumulasi di dalam sel-sel jaringan pembuluh lateks yang tersusun pada setiap bagian tanaman. Produksi lateks yang didapat dari penyadapan bergantung pada lamanya aliran dan kecepatan biosintesis lateks. Akhir-akhir ini berbagai usaha telah banyak dilakukan oleh para pemulia tanaman karet untuk meningkatkan produksi lateks tanaman karet, misalnya dengan pengembangan klon karet, penggunaan stimulan, maupun pemberian nutrisi dan perawatan khusus yang mendukung pertumbuhan optimual tanaman (Nurhaimi-Haris et al. 2003).
Tanaman karet yang digunakan dalam penelitian ini adalah tanaman karet hasil persilangan antara RRIM 600 dan PN 1546. Persilangan tersebut merupakan persilangan jauh (interpopulasi) karena berasal dari populasi yang berbeda dengan latar belakang genetik yang jauh. RRIM 600 berasal dari populasi Wickham 1876 sedangkan PN 1546 berasal dari populasi Plasma Nutfah IRRDB 1981 (Woelan et al. 2007). Hasil penelitian yang dilakukan oleh Tistama et al. (2006) menyatakan bahwa jarak genetik antara klon yang berasal dari populasi Wickham 1876 dan plasma nutfah IRRDB 1981 cukup jauh, yaitu sebesar 63%. Menurut Woelan et al. (2007) persilangan antar dua tetua dengan perbedaan jarak genetik yang jauh akan menghasilkan klon unggul baru yang lebih baik. Hal tersebut juga dikemukaan oleh Chaidamsari et al. (1993) dan Nurhaimi et al. (1998) bahwa persilangan lebih baik bila
menggunakan tetua yang tingkat
kemiripannya sangat rendah.
Gambar 1 Tanaman karet (Hevea brasiliensis Muell Arg.) (Lorenzi 2008).
Klon Tanaman Karet
Sejak pertama kali ditemukan sebagai tanaman yang tumbuh liar, hingga kini telah dikembangkan beberapa klon dari tanaman
karet. Klon adalah tanaman hasil perbanyakan bukan secara generatif (biji) melainkan secara vegetatif atau aseksual yang diambil dari mata tunas pada batang (Tarigan 2008). Klon tanaman karet memiliki kelebihan dibandingkan dengan tanaman yang dikembangkan melalui biji. Kelebihan klon antara lain, produksi lateks yang tinggi dan konsisten selama umur produktifnya, tahan terhadap penyakit, kuat dan kokoh sehingga tidak mudah roboh oleh tiupan angin, pohon tumbuh ke atas, cabang menyebar merata di sekeliling batang, dan memiliki kulit murni, halus, tebal, serta lekas pulih setelah disadap. Selain kelebihannya, klon tanaman karet juga memiliki kekurangan seperti perbedaan daya tahan pada setiap klon terhadap hama penyakit dan lingkungan yang dapat mempengaruhi pertumbuhan klon (Astuti 2008).
Klon tanaman karet dilakukan pertama kali pada tahun 1910 oleh seorang ahli hortikultura bernama Helten. Sejak saat itu, kegiatan klon tanaman karet semakin berkembang dan hingga saat ini di setiap negara produsen karet telah memiliki klon-klon unggulan. Klon karet yang sudah dirilis dapat dibedakan menjadi tiga jenis, yaitu klon primer, sekunder, dan tersier. Klon sekunder merupakan persilangan dari klon-klon primer, dan keturunan klon sekunder disebut klon tersier. Klon sekunder dan tersier umumnya lebih modern dan cara pemuliaannya lebih maju, yaitu dengan menggunakan teknologi terbaru (Setiawan & Agus 2008). Teknologi terbaru yang sering digunakan adalah dengan menggunakan teknik molecular breeding.
Beberapa contoh klon yang dihasilkan oleh lembaga penelitian di Indonesia adalah serial klon AVROS (AVROS 33, AVROS 49, dan AVROS 80) dan serial klon TM (TM 2, TM 6, dan TM 9) yang dihasilkan oleh lembaga penelitian di pulau Sumatera. Beberapa klon yang dihasilkan oleh lembaga penelitian di Jawa antara lain BD 5, GT 1, WAR 4, TJIR1, LCB 479, dan LCB 1320 (Tarigan 2008).
Menurut Nancy (2007) klon-klon unggul karet yang direkomendasikan Pusat Penelitian Karet Balai Penelitian Sembawa untuk periode 2006-2010 terdiri dari dua kelompok. Pertama, kelompok klon anjuran komersial yang merupakan sekelompok klon dengan data yang lebih lengkap dan sudah dapat dikembangkan oleh pengguna. Klon-klon ini sudah berupa benih bina, kecuali klon IRR 42 dan IRR 112 masih dalam proses pengajuan untuk pelepasannya sebagai
benih bina. Kedua, Klon harapan, merupakan kelompok klon yang mempunyai potensi pertumbuhan dan produksi tinggi tetapi belum berupa benih bina.
Menurut Nancy (2007) klon-klon anjuran komersial terdiri dari tiga katagori. Pertama klon penghasil lateks: BPM 24, BPM 107, BPM 109, IRR 104, PB 217 dan PB 260. Kedua, klon penghasil lateks dan kayu: BPM 1, PB 330, PB 340, RRIC 100, AVROS 2037, I RR 5, IRR 32, IRR 39, IRR 42, IRR 112 dan IRR 118. Ketiga, Klon penghasil kayu (cocok dikembangkan di kawasan hutan): IRR 70, IRR 71, IRR 72 dan IRR 78. Sedangkan klon harapan: IRR 24, IRR 33, IRR 41, IRR 54, IRR 64, IRR 105, IRR 107, IRR 111, IRR 119, IRR 141, IRR 208, IRR 211 dan IRR 220.
Setiawan dan Agus (2008) menyebutkan bahwa klon-klon yang tepat untuk perkebunan skala besar diantaranya AVROS, PBM 1, PBM 24, GT 1, LCB 1320, PR 255, PR 261, PR 300, RRIM 600, dan RRIM 712. Untuk perkebunan rakyat, Setiawan dan Agus (2008) menganjurkan untuk memilih klon AVROS 2037, BPM 1, BPM 24, GT 1, PR 261, PR 300, dan PR 303. Sementara itu, untuk perkebunan-perkebunan kecil klon yang dianjurkan sebagai pilihan adalah BPM 107, BPM 109, IAN 710, IAN 717, PB 217, PB 235, PB 260, PPN 2001, PPN 2002, PPN 2005, PPN 2049, PR 302, PR 307, PR 309, PR 311, PE 314, RRIC 100, RRIC 101, RRIC 102, RRIC 110, RRIM 717, RRIM 728, TM 2, TM 4, TM 5, TM 6, TM 8, dan TM 9.
Produktivitas lateks dari klon-klon yang dianjurkan tersebut umumnya akan semakin meningkat sesuai dengan semakin bertambahnya umur tanaman. Pada tahun-tahun pertama, produksinya biasanya hanya 100-1000 kg karet kering/hektar/tahun. Produksi tersebut akan terus meningkat hingga mencapai puncaknya pada tahun ke-8,
yaitu sebesar 2000 kg karet
kering/hektar/tahun. Setelah itu, produksinya akan terus menurun hingga dilakukan proses peremajaan kembali. Rata-rata produksi lateks dari klon unggulan pada lima tahun pertama adalah 1200-1500 kg/hektar/tahun. Setelah mencapai umur sepuluh tahun, produksi rata-ratanya adalah 1600-1800 kg/hektar/tahun (Setiawan & Agus 2008).
Pembuatan batang bawah untuk bibit pada pembuatan klon karet dianjurkan menggunakan biji berasal dari klon AVROS 2037, BPM 24, GT 1, PB 260 dan RRIC 100. Agar diperoleh mutu biji yang baik maka tanaman yang dapat diambil bijinya adalah
tanaman yang berumur lebih dari 10 tahun dan dipelihara sesuai standar. Pada umumnya biji yang dapat dimanfaatkan berasal dari perkebunan besar atau proyek-proyek peremajaan karet rakyat dengan hamparan yang cukup luas (Nancy 2007).
Keragaman Genetik
Indonesia merupakan suatu negara dengan keanekaragaman hayati sangat tinggi (megabiodiversity). Kekayaan ini semakin penting artinya sekarang setelah ilmu pengetahuan dan teknologi mencapai derajat dengan akselerasi yang belum pernah terjadi sebelumnya. Kemajuan ilmu dan teknologi di bidang bioteknologi telah membuka khasanah baru dalam memanfaatkan sumber daya hayati ini. Sumber-sumber gen dari berbagai organisme dapat dilacak dan dipindahkan ke organisme lain untuk tujuan perbaikan penampilan organisme tersebut (genetically
modified organisms) (Suryanto 2003). Menurut Damayanti (2007) keragaman genetik merupakan variasi genetik di dalam setiap spesies yang mencakup aspek biokimia, struktur, dan sifat organisme yang diturunkan secara fisik dari induknya dan dibentuk dari DNA.
Menurut Suryanto (2003)
keanekaragaman genetika dapat terjadi karena adanya perubahan nukleotida penyusun DNA. Perubahan ini mungkin dapat mempengaruhi fenotipe suatu organisme yang dapat dipantau dengan mata telanjang, atau mempengaruhi reaksi individu terhadap lingkungan tertentu. Secara umum keanekaragaman genetik dari suatu populasi dapat terjadi karena adanya mutasi, rekombinasi, atau migrasi gen dari satu tempat ke tempat lain. Perkembangan ilmu dan pengetahuan dalam biologi molekuler, khususnya pada pengkajian karakter bahan genetik telah menghasilkan kemajuan yang sangat pesat bagi perkembangan penelaahan suatu organisme dan pemanfaatannya bagi kesejahteraan manusia. Di bidang taksonomi, sebagai contoh Avise & Lansman (1983) dan Brown (1983) mengungkapkan peran DNA mitokondria (mtDNA) dalam studi keanekaragaman genetika dan biologi populasi pada hewan. mtDNA dapat digunakan sebagai penanda genetika karena ukurannya relatif kecil. Secara umum penggunaan teknik molekuler untuk tujuan identifikasi suatu organisme mempunyai keunggulan seperti lebih akurat, lebih cepat, dan untuk mikroba dapat mencakup
4
keseluruhan mikroba termasuk yang “viable
but not yet culturable”.
Penelitian mengenai keragaman genetik pada tanaman merupakan salah satu kegiatan penting untuk mendukung pemuliaan tanaman (Ardiana 2009). Perbedaan tanaman dapat dideteksi melalui beberapa penanda, antara lain dengan pola pita DNA (Lamadji 1998), yang sering disebut sebagai penanda molekuler. Penanda molekuler berperan penting dalam konservasi dan pengelolaan sumber daya genetik tanaman (Karp et al. 1997).
Poerba dan Martanti (2008)
menyebutkan bahwa penanda molekuler banyak digunakan dalam analisis keragaman genetik tumbuhan, salah satunya adalah
random amplified polymorphic DNA
(RAPD). Teknik ini digunakan untuk mengidentifikasi genotipe tumbuhan, karena memiliki kelebihan dalam pelaksanaan dan analisisnya. Dibandingkan dengan penanda DNA yang lain, seperti restriction fragment
length polymorphisms (RFLP) dan simple sequence repeats (SSR), teknik RAPD lebih
murah, mudah dilakukan, cepat memberikan hasil, menghasilkan polimorfisme pita DNA dalam jumlah banyak, tidak memerlukan pengetahuan tentang latar belakang genom yang dianalisis dan mudah memperoleh primer acak yang diperlukan untuk menganalisis genom semua jenis organisme (Tingey et al., 1994).
Marka DNA
Marka DNA adalah suatu sekuen pendek DNA yang menunjukkan adanya polimorfis antara individu berbeda dalam satu spesies.
Marka DNA mempunyai tingkat
polimorfisme yang sangat tinggi, jumlahnya tidak terbatas, tidak dipengaruhi oleh lingkungan, dan tingkat heritabilitasnya hampir 100%. Pada dekade terakhir marka DNA lebih dikembangkan penggunaannya dalam pemetaan. Marka yang diperoleh dari DNA disebut juga marka molekular. Suatu marka akan efektif jika marka dapat membedakan antara dua tetua yang berbeda genotipenya dan marka yang digunakan juga harus diwariskan pada keturunannya. Marka juga akan efektif jika dapat dideteksi dengan mudah dalam populasi yang diuji (Wirnas 2005).
Peta genetik dibuat berdasarkan rekombinasi homolog yang terjadi selama meiosis sehingga disebut juga peta meiosis. Jika dua atau lebih marka berdekatan dalam
kromosom maka alel-alelnya cenderung untuk diwariskan secara bersama-sama. Frekuensi rekombinan yang terjadi antara marka atau lokus yang dijelaskan oleh marka digunakan untuk menentukan jarak keterpautan antara dua lokus atau marka. Beberapa faktor yang menentukan kepadatan marka dalam peta adalah panjang genom, jumlah marka yang digunakan, distribusi marka yang polimorfis, distribusi marka dalam genom, distribusi pindah silang, jenis dan ukuran populasi serta strategi pemetaan yang dipilih (Wirnas 2005).
Polymerase Chain Reaction
Reaksi berantai polimerase (PCR) adalah metode amplifikasi suatu sekuen DNA tertentu. PCR merupakan cara yang sensitif, selektif, dan sangat cepat untuk
memperbanyak sekuen DNA yang
diinginkan. Spesifisitas didasarkan pada pemakaian dua oligonukleotida primer yang terhibridisasi ke sekuen komplementer di untai DNA yang berlawanan dan mengapit sekuen target. Sampel DNA mula-mula dipanaskan untuk memisahkan kedua untai;
primer dibiarkan berikatan dengan DNA; dan
masing-masing untai disalin oleh suatu DNA polimerase yang dimulai di tempat primer. Kedua untai DNA masing-masing berfungsi sebagai cetakan untuk síntesis DNA baru dari dua primer. Siklus berulang denaturasi panas,
penyatuan primer dengan sekuen
komplementernya, dan pemanjangan primer yang telah menyatu tersebut dengan DNA polimerase menyebabkan perbanyakan eksponensial segmen DNA dengan panjang tertentu (Murray et al. 2009).
Komponen-komponen utama yang terlibat dalam proses PCR terdiri atas DNA target yang akan diamplifikasi (cetakan DNA),
primer, deoksinukleotida trifosfat (dNTPs),
dan enzim termostabil DNA polimerase. Molekul DNA target berfungsi sebagai cetakan untuk menghasilkan perbanyakan sekuens DNA yang diinginkan. Primer merupakan satu fragmen DNA pendek yang berukuran 10-20 basa (b) yang berperan dalam inisiasi sintesis untaian DNA. Semakin panjang primer, maka semakin spesifik daerah yang akan diamplifikasi. DNA polimerase yang digunakan dalam proses PCR adalah Taq polimerase DNA yang diisolasi dari mikroorganisme Thermus aquaticus. Enzim ini bersifat tahan terhadap
suhu panas (94˚C) dan memiliki kecepatan amplifikasi 2-4 kilobasa per menit atau 35-70
basa per detik. Enzim taq polimerase berfungsi sebagai biokatalis dalam síntesis untaian DNA baru, sedangkan molekul dNTPs digunakan untuk membentuk kompleks rantai baru (Bangun 2002).
Proses PCR pada prinsipnya melibatkan tiga langkah yang diulangi dalam beberapa siklus, yaitu (1) Denaturasi termal dengan meningkatkan suhu pada tabung reaksi sampai 95°C; (2) Primer annealing
merupakan tahap saat primer akan berpasangan dengan sekuens DNA cetakan (template) yang sudah dalam bentuk ss-DNA pada 35-60°C; (3) Ekstensi primer, pada tahap ini suhu ditingkatkan kembali sampai 75°C yang merupakan suhu optimum untuk kerja Taq DNA polimerase yang akan memulai reaksi pada ujung 3‟-hidroksil dari
primer. Selain DNA cetakan dalam reaksi
PCR juga diperlukan primer oligonukleotida, DNA polimerase termostabil (misalnya Taq
DNA polimerase), empat macam
deoksiribonukleotida (dNTPs), dan buffer PCR pH tertentu (Surahman et al. 2007).
Konsentrasi DNA dan MgCl2
mempengaruhi hasil amplifikasi PCR. Konsentrasi DNA yang lebih tinggi atau lebih rendah dari konsentrasi optimum menghasilkan pola pita DNA yang lebih sedikit dan kurang baik. Konsentrasi MgCl2 yang lebih rendah atau lebih tinggi dari konsentrasi optimum menghasilkan jumlah pita yang lebih sedikit. Konsentrasi Taq DNA polimerase berpengaruh pada ketajaman pita DNA. Konsentrasi emzim Taq DNA polimerase yang tinggi menghasilkan intensitas pita DNA yang lebih tajam dibandingkan hasil amplifikasi dengan konsentrasi enzim yang lebih rendah (Lengkong et al. 2001).
Untuk mencapai amplifikasi DNA yang optimum diperlukan kondisi reaksi yang optimum yang ditentukan misalnya oleh konsentrasi DNA cetakan, konsentrasi
magnesium, konsentrasi Taq DNA
polimerase, konsentrasi primer dan dNTPs, suhu annealing, waktu dan jumlah siklus tertentu. Oleh sebab itu, perlu ada optimasi PCR. Disamping itu, keberhasilan juga ditentukan oleh primer yang baik. Persyaratan primer yang baik antara lain adalah bebas dari kemungkinan terjadinya
primer-dimer dan formasi
self-complementarity, serta stabilitas internal yang
tepat (Surahman et al. 2007).
Mikrosatelit
Mikrosatelit, juga dikenal dengan simple
sequence repeats (SSR) adalah kelas terkecil
dari sekuen berulang. Sekuen yang berulang terdiri dari dua, tiga atau empat nukleotida (di-, tri-, dan tetranukleotida berulang). Salah satu contoh umum mikrosatelit adalah dinukleotida berulang (CA)n, dimana n menunjukkan jumlah total nukleotida berulang/repeats yang berada pada kisaran 10 dan 100. Marker ini sering menunjukkan polimorfisme inter dan intra spesifik dengan level tinggi (Semagn et al. 2006).
Reaksi PCR untuk SSR dilakukan dengan primer forward dan reverse yang berikatan pada ujung 5` dan 3` dari DNA cetakan. Fragmen produk PCR biasanya dipisahkan pada gel poliakrilamid dengan pewarnaan AgNO3, dengan autoradiografi atau dengan sistem deteksi menggunakan fluorescents. Gel agarosa (biasanya 3%) dengan pewarnaan EtBr dapat digunakan saat perbedaan dalam ukuran alel antar sampel lebih besar dari 10bp (Semagn et al. 2006).
Pengembangan mikrosatelit melibatkan beberapa tahap dimulai dari pembentukan pustaka untuk pengembangan set primer yang dapat mengamplifikasi lokus mikrosatelit yang polimorfik, meliputi: konstruksi pustaka mikrosatelit, identifikasi lokus mikrosatelit yang unik, identifikasi area yang sesuai untuk disain primer, mendapatkan produk PCR, evaluasi dan interpretasi pola banding, dan penilaian produk PCR yang polimorfik.
SSR sekarang merupakan marker yang dipilih pada banyak area genetika molekuler karena mikrosatelit sangat polimorfik (bahkan untuk spesies atau galur yang berkerabat dekat), memerlukan DNA dalam jumlah kecil, dan dapat diautomasi (Semagn
et al. 2006).
Random Amplified Polymorphic DNA Random amplified polymorphic DNA
(RAPD) merupakan salah satu teknik molekuler berupa penggunaan penanda tertentu untuk mempelajari keanekaragaman genetika. Dasar analisis RAPD adalah penggunaan mesin PCR yang mampu mengamplifikasi sekuen DNA secara in vitro.
Penggunaan teknik RAPD memang
memungkinkan untuk mendeteksi
polimorfisme fragmen DNA yang diseleksi dengan menggunakan satu primer arbitrasi (primer acak), terutama karena amplifikasi DNA secara in vitro dapat dilakukan dengan baik dan cepat dengan adanya PCR.
6
Polimorfisme diartikan sebagai terbentuknya
lokus-lokus yang berbeda pada
elektroforegram hasil amplifikasi yang dapat membedakan antara satu sampel dengan sampel yang lain (Suryanto 2003). Teknik RAPD dipilih untuk analisis genetik dengan berbagai alasan, antara lain tidak membutuhkan latar belakang pengetahuan tentang genom yang akan dianalisis, bisa
menggunakan primer-primer untuk
organisme prokariotik maupun eukariotik, mampu menghasilkan polimorfisme sampai mendekati jumlah yang tak terhingga, menggunakan bahan-bahan yang relatif murah kecuali enzim Taq polimerase, cocok untuk membuat diagnosis silsilah (filogeni) suatu spesies dan amplifikasinya tidak bergantung pada radioaktif (Surahman et al. 2007). Keunggulan praktis dari teknik RAPD terletak pada kesederhanaan dan kecepatannya. RAPD lebih efisien 4 – 6 kali dibandingkan RFLP bila digunakan untuk pemetaan polimorfisme yang terpaut dengan resistensi penyakit dan 10 kali lebih efisien dalam waktu dan tenaga kerja (Surahman et
al. 2007). Selain itu, umumnya teknik RAPD
mempunyai lebih banyak lokus yang dianalisis secara simultan tiap percobaan (nisbah multipleks) daripada teknik RFLP (Powell et al. 1996).
Marka RAPD diperoleh berdasarkan kemungkinan adanya suatu sekuen DNA homolog dengan suatu sekuen primer oligonukleotida. Primer oligonukleotida acak akan menempel di dua tempat yang komplementer terhadap sekuens cetakan DNA genomik dalam orientasi yang berlawanan. Apabila kedua tempat penempelan primer berada dalam jarak yang berdekatan (< 4000 pasang basa), maka primer tunggal oligonukleotida akan mengawali terjadinya amplifikasi DNA secara eksponensial pada suatu reaksi PCR (Surahman et al. 2007).
Marka RAPD biasanya berperilaku sebagai marka genetik dominan (Surahman et
al. 2007). Dominasi dalam hal ini tidak
menunjuk pada pengertian klasik tentang interaksi antar alel intralokus, tetapi lebih semata-mata pada sudut pandang hubungan fenotipe dan genotipe. Bila pita RAPD teramati pada gel, fragmen yang berasal dari amplifikasi pada individu diploid homozigot dan heterozigot tidak dapat dibedakan. Dalam hal ini pada individu diploid homozigot (AA) untuk lokus RAPD, amplifikasi dilakukan dari dua kopi alel RAPD (A). Pada individu heterozigot (Aa) untuk lokus yang sama, alel
(A) diamplifikasi dan alel (a) tidak diamplifikasi. Deteksi fragmen RAPD tidak cukup memiliki kepekaan kuantitatif untuk membedakan dua kondisi tersebut. Pada dua kondisi tersebut akan teramati intensitas pita yang identik. Sementara itu, genotipe homozigot resesif (aa) dikenali dengan tidak munculnya pita atau genotipe nol.
BAHAN DAN METODE
Alat dan Bahan
Alat-alat yang digunakan meliputi tabung sentrifus, neraca analitik, gelas ukur, gelas piala, mortar, mesin inkubator, penangas air, mesin autoklaf, pipet Mohr, bulp, tabung Erlenmeyer, pipet mikro, dan tabung mikro. Selain itu juga digunakan perangkat elektroforesis, sentrifus (Backman Centrifuge & Eppendorf Centrifuge 5417R), mesin PCR (PCR Biometra Tpersonal), mesin transiluminator UV (T 2201 Sigma), dan mesin DNA speed vac (Savant DNA Speed
Vac 110).
Bahan-bahan yang digunakan untuk amplifikasi meliputi sampel DNA tetua {PN 1546 (tetua jantan) dan RRIM 600 (tetua betina)}, sampel DNA 25 progeni (anakan) hasil persilangan klon karet antara PN 1546 dan RRIM 600, Taq DNA polimerase, buffer
complete PCR, primer OPA (1, 2, 3, 4, 8, 10,
11, 12, 13, 14, 15, 17, 18, 19, dan 20), primer OPB (4, 11, 17, 19, dan 20), primer OPC (2, 3, 5, 7, 8, 9, 12, 13, 14, 19, dan 20), primer OPD (1, 2, 3, 4, 5, 7, 8, 10, 11, 14, 15, 18, dan 20), primer OPM (5, 7, 8, 9, 12, 16, 20), primer OPN (3, 5, 9, 10, 15, 16, dan 18), primer OPY (9 & 14), primer mTcCIR (15, 26, 37, 229), molecular water (MW), dan larutan dNTPs. Bahan-bahan yang digunakan untuk elektroforesis meliputi larutan TBE 0.5x, bubuk agarosa, larutan EtBr, loading dye buffer, dan marker 1 kb DNA Ladder plus.
Metode
Isolasi DNA Daun Karet (Khanuja 1999)
Sebanyak 1 gram bobot basah daun karet digerus dengan menggunakan N2 cair. Setelah halus, sampel tersebut dimasukkan ke dalam tabung sentrifus. Tambahkan sebanyak 3 mL
buffer extraction (BE) hangat ke dalam
tabung yang berisi sampel lalu dikocok hingga homogen. Setelah itu dinkubasi pada penangas air pada suhu 60°C selama 1 – 2
jam dengan setiap lima belas menit sekali dilakukan pengocokan secara perlahan. Setelah satu jam, ditambahkan 3 mL larutan kloroform:isoamilalkohol (24:1) lalu disentrifus dengan kecepatan 7840 g selama 10 menit pada 25°C. Supernatan yang dihasilkan kemudian diambil dan ditambahkan sebanyak 0.5 volume NaCl 5 M, setelah dikocok kemudian ditambahkan sebanyak 0.6 volume larutan isopropanol dan diinkubasi pada suhu 25°C selama 1 jam. Setelah diinkubasi, sampel kemudian di sentrifus dengan kecepatan 7840 g selama 10 menit pada 25°C. Pelet yang dihasilkan kemudian dicuci dengan etanol 80%. Selanjutnya dilakukan sentrifus kembali pada kecepatan dan waktu yang sama seperti sebelumnya. Etanol tersebut kemudian dibuang dan pelet yang terbentuk kemudian dikeringanginkan. Pelet tersebut kemudian ditambahkan dengan 0.5 mL larutan bufer Tris-EDTA (TE), larutan kemudian dihomogenkan dan dipindahkan ke tabung mkro. Sampel kemudian diekstrak dengan larutan kloroform:isoamilalkohol (24:1) sebanyak 1 volume lalu disentrifus dengan kecepatan 7840 g selama 10 menit pada 25°C. Supernatan yang terbentuk kemudian diambil lalu ditambahkan sebanyak 2 volume larutan etanol absolut dingin. Sentrifus kembali pada 7840 g selama 10 menit (25°C), pelet yang terbentuk kemudian dicuci dengan larutan etanol 80% dan disentrifuse kembali pada kecepatan yang sama selama 5 menit. Etanol kemudian dibuang dan pelet dikering anginkan. Pelet kemudian dilarutkan dengan 30 µL ddH20/MW.
PCR DNA Tetua Karet dengan Primer Kombinasi (Semi Mikrosatelit)
Sampel DNA yang digunakan pada tahap ini adalah sampel DNA tetua, yaitu RRIM 600 dan PN 1546. Pembuatan campuran PCR dilakukan pada tabung mikro dengan komposisi antara lain 1 µL sampel DNA tetua (30 ng), 2.5 µL buffer complete, 1 µL dNTPs, 1 µL primer forward mTcCIR (15, 26, 37, atau 229), 1 µL primer reverse mTcCIR (15, 26, 37, atau 229), 0.5 µL Taq DNA polimerase, 18 µL MW (Molecular
Water) sehingga volume total menjadi 25 µL.
Campuran tersebut kemudian dihomogenisasi dengan menggunakan speed vac selama beberapa saat (±1 menit), kemudian dimasukkan ke dalam mesin PCR.
Program suhu yang digunakan pada optimasi PCR adalah 94°C selama 3.30 menit
untuk denaturasi DNA, 35°C selama 1 menit untuk penempelan primer, 72°C selama 2 menit untuk tahapan perpanjangan rantai. Untuk siklus berikutnya program suhu yang digunakan 94°C selama 1 menit untuk denaturasi DNA, 45°C selama 1 menit untuk penempelan primer, 72°C selama 2 menit untuk tahapan perpanjangan rantai hingga sebanyak 35 kali siklus, serta 72°C selama 4 menit terakhir untuk memastikan DNA yang diamplifikasi terenaturasi seluruhnya. Hasil PCR kemudian dilihat dan dipisahkan dengan menggunakan elektroforesis gel agarosa.
RAPD Karet
Sampel DNA yang digunakan pada tahap ini merupakan sampel DNA tetua (RRIM 600 & PN 1546) dan 25 progeni (anakan) hasil persilangan tanaman karet antara RRIM 600 dengan PN 1546. Sebanyak 1 µL sampel DNA progeni (30 ng), 2.5 µL buffer complete PCR, 1 µL dNTPs, 1 µL primer (primer yang digunakan OPA, OPB, OPC, OPD, OPM, OPN, dan OPY dengan jumlah total 60 primer), 0.5 µL Taq DNA polimerase, 19 µL MW (Molecular Water) dimasukkan ke dalam tabung mikro sehingga volume total menjadi 25 µL. Campuran tersebut kemudian dihomogenisasikan dengan menggunakan mesin DNA speed vac selama beberapa saat (±1 menit), lalu dimasukkan ke dalam mesin PCR dengan program suhu yang sama seperti pada proses sebelumnya dengan suhu
annealing 35°C dengan jumlah siklus
sebanyak 44 siklus. Hasil PCR kemudian dilihat dan dipisahkan dengan menggunakan elektroforesis gel agarosa.
Amplifikasi 25 Progeni Hasil Persilangan dengan Semi Mikrosatelit
Sebanyak 1 µL sampel DNA (30 ng), 2.5 µL buffer complete, 1 µL dNTPs, 1 µL primer forward mTcCIR (15, 26, 37, atau 229), 1 µL primer reverse mTcCIR (15, 26, 37, atau 229), 0.5 µL Taq DNA polimerase, 18 µL MW (Molecular Water) dimasukkan ke dalam tabung mikro sehingga volume total menjadi 25 µL. Kemudian dilakukan proses homogenisasi dengan menggunakan alat
speed vac selama beberapa saat (±1 menit),
kemudian dimasukkan ke dalam mesin PCR dengan program suhu yang sama seperti pada proses sebelumnya di atas, dengan suhu
annealing 35°C sebanyak 44 siklus. Hasil
amplifikasi kemudian dipisahkan dengan menggunakan elektroforesis gel agarosa.
8
Elektroforesis DNA
Pembuatan Larutan TBE 0.5x.
Sebanyak 100 mL larutan TBE 5x dilarutkan ke dalam 900 ml akuades. Komposisi larutan stok TBE 5x per 500 mL terdiri dari
Tris-base 27 gram, a 13.75 gram, dan EDTA 0.5
M pH 8.0 sebanyak 10 mL.
Pembuatan Gel Agarosa. Sebanyak 0.6
gram bubuk agarosa ditimbang dan ditambahkan dengan larutan TBE 0.5x sebanyak 60 mL. Larutan kemudian dipanaskan di dalam microwave sampai semua agarosa larut. Setelah suhu larutan hangat, kemudian ditambahkan dengan 3 µL EtBr dan kocok hingga homogen. Setelah itu tuang larutan ke dalam cetakan yang telah disiapkan.
Elektroforesis DNA. Seperangkat bak
elektroforesis disiapkan, kemudian larutan TBE 0.5x yang telah disiapkan dituangkan ke dalam bak. Selanjutnya gel agarosa yang telah memadat dimasukkan ke dalam bak hingga gel terendam penuh. Setelah itu, di tempat terpisah diteteskan sebanyak 1 µL larutan loading dye pada microfilm. Sebanyak 1 µL sampel DNA dicampurkan dengan
loading dye. Sampel kemudian dimasukkan
ke dalam sumur gel yeng tersedia. Sampel tersebut kemudian dielektroforesis pada 75 volt hingga semua sampel DNA bermigrasi dari arah kutub negatif ke kutub positif. Hasil elektroforesis dilihat di atas lampu UV transiluminator dan dipotret.
Analisis segregasi marka RAPD
Fragmen hasil amplifikasi terhadap DNA tetua karet (RRIM 600 dan PN 1546) dan DNA 25 progeni (anakan) hasil persilangan merupakan lokus DNA yang bersifat dominan. Evaluasi dari pita-pita yang dihasilkan dilihat dari fragmen DNA yang mempunyai berat molekul tertentu. Ada atau tidaknya marka RAPD diskor secara manual. Kriteria penskoran berdasarkan muncul tidaknya lokus, lokus yang muncul diberi skor 1 dan yang tidak muncul diberi skor 0. Data biner yang diperoleh selanjutnya diolah menjadi dendrogram atau disebut dengan pohon filogenetik dengan menggunakan program statistik khusus NTSYS versi 2.02, dengan menggunakan metode UPGMA (Unweighted Pair Group Method Arithmatic
Mean).
HASIL DAN PEMBAHASAN
Hasil Isolasi DNA Karet
DNA karet pada penelitian ini diisolasi dari daun tanaman karet. Hasil isolasi DNA dari 25 progeni hasil persilangan, secara kualitatif DNA yang dihasilkan terlihat memiliki konsentrasi yang sangat tinggi dan memiliki ukuran sekitar 12000 bp, hal tersebut diperlihatkan pada Gambar 2. Secara kuantitatif, tingginya konsentrasi DNA juga dapat terlihat dari nilai konsentrasi yang dihasilkan berkisar antara 715 - 3035 ng/μL. Tingkat kemurnian sampel DNA hasil isolasi terhadap kontaminan protein dapat dilihat melalui analisis rasio antara nilai OD260 dengan nilai OD280. Hasil analisis menunjukkan bahwa rasio antara nilai OD260 dengan nilai OD280 berada pada kisaran 1.3 - 2.1. Menurut Muladno (2010), rasio nilai
OD260 dengan nilai OD280 yang menunjukkan kemurnian DNA terhadap protein berada pada kisaran 1.8-2.0, sehingga dapat diketahui bahwa masih terdapat kontaminan protein pada DNA yang telah berhasil diisolasi tersebut. DNA hasil isolasi tersebut telah mengalami perlakuan dengan pemberian RNase, sehingga kemungkinan adanya kontaminan RNA sangat kecil (tidak ada). Oleh karena itu, dengan melihat elektroforegram DNA secara kualitatif, maka DNA dapat digunakan untuk proses PCR-RAPD karena memiliki konsentrasi yang tinggi.
Perbandingan nilai OD260 dengan nilai
OD280 yang tidak tepat berada pada kisaran 1.8-2.0 kemungkinan dipengaruhi oleh beberapa hal seperti tingkat spesifitas alat yang menurun akibat penggunaan yang terus-menerus atau DNA yang tidak terlarut secara sempurna sehingga akan mempengaruhi pembacaan pada alat spektrofotometer. Konsentrasi DNA yang digunakan untuk proses PCR-RAPD pada penelitian ini hanya
30 ng/μL yang dihitung dengan
memperhatikan faktor pengenceran.
Menurut Nurhaimi-Haris et. al. (2003), tinggi rendahnya konsentrasi DNA yang berhasil diisolasi akan berdampak pada kualitas fragmen hasil amplifikasi terhadap DNA tersebut. Konsentrasi DNA terlalu rendah akan dapat menghasilkan fragmen DNA sebagai pita yang sangat tipis pada gel atau bahkan pita tidak terlihat secara visual, sebaliknya konsentrasi DNA terlalu tinggi akan menyebabkan fragmen DNA terlihat tebal sehingga sulit dibedakan antara satu pita
Gambar 2 Elektroforegram DNA 25 progeni karet hasil isolasi. dengan pita lainnya. Oleh karena itu, untuk
menghasilkan hasil yang optimal, perlu dilakukan pengenceran sehingga sampel DNA yang akan diamplifikasi memiliki konsentrasi yang sama dan tidak berpengaruh pada pita hasil amplifikasi.
Produk PCR DNA Tetua Karet dengan Primer Kombinasi (Semi Mikrosatelit).
Pada penelitian ini digunakan empat pasang primer mTcCIR yang dirancang untuk semi mikrosatelit, yaitu mTcCIR 15, mTcCIR 26, mTcCIR 37, dan mTcCIR 229. Empat pasang primer tersebut kemudian dibuat menjadi 16 pasang primer kombinasi yang selanjutnya disebut primer kombinasi a, b, c, d, e, f, g, h, i, j, k, l, m, n, o, dan p seperti yang tercantum pada Tabel 1.
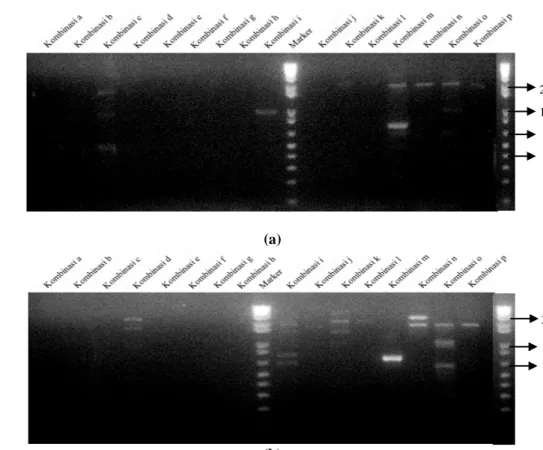
Produk PCR pada penilitian ini ternyata dapat menghasilkan beberapa kandidat primer kombinasi yang dapat digunakan sebagai pembeda antara PN 1546 (tetua jantan) dan RRIM 600 (tetua betina). Beberapa kandidat tersebut antara lain primer kombinasi c, d, i, m, dan n yang secara lengkap dapat dilihat pada Gambar 3. Alasan dipilihnya beberapa kandidat tersebut antara lain pada primer kombinasi c, pita DNA hanya muncul pada PN 1546, yaitu pada 400 bp, 850 bp, dan 2000 bp, sedangkan pada RRIM 600 tidak muncul. Sehingga primer kombinasi c dapat digunakan untuk membedakan antar dua tetua.
Kandidat primer kombinasi berikutnya adalah primer kombinasi d, pada kombinasi ini pita hanya muncul pada RRIM 600 yaitu pada 2000 bp dan 3000 bp, namun tidak muncul pada PN 1546. Selanjutnya adalah primer kombinasi i, pada primer kombinasi ini terdapat perbedaan yang cukup mendasar mengenai munculnya pita pada kedua tetua, pada PN 1546 muncul pada 1000 bp
sedangkan pada RRIM 600 muncul pada 650 bp, 850 bp, 1650 bp, dan 2000 bp, sehingga dapat digunakan sebagai pembeda pada analisis 25 DNA progeni hasil persilangan. Tabel 1 Primer kombinasi yang digunakan untuk amplifikasi.
Nama Primer Kombinasi
Primer Kombinasi
A mTcCIR 15 forward + mTcCIR 15
reverse
B mTcCIR 26 forward + mTcCIR 15
reverse
C mTcCIR 37 forward + mTcCIR 15
reverse
D mTcCIR 229 forward + mTcCIR 15
reverse
E mTcCIR 15 forward + mTcCIR 26
reverse
F mTcCIR 26 forward + mTcCIR 26
reverse
G mTcCIR 37 forward + mTcCIR 26
reverse
H mTcCIR 229 forward + mTcCIR 26
reverse
I mTcCIR 15 forward + mTcCIR 37
reverse
J mTcCIR 26 forward + mTcCIR 37
reverse
K mTcCIR 37 forward + mTcCIR 37
reverse
L mTcCIR 229 forward + mTcCIR 37
reverse
M mTcCIR 15 forward + mTcCIR 229
reverse
N mTcCIR 26 forward + mTcCIR 229
reverse
O mTcCIR 37 forward + mTcCIR 229
reverse
P mTcCIR 229 forward + mTcCIR 229
reverse
Primer kombinasi m memunculkan pita
pada PN 1546 dikisaran 700 bp dan 2000 bp, sedangkan di RRIM 600 muncul dikisaran 700 bp (Gambar 3). Sama halnya pada primer kombinasi m, pada primer kombinasi n juga memunculkan pita di PN 1546 pada kisaran 2000 bp sedangkan di RRIM 600 pada
kisaran 2000 bp dan 3000 bp.
10
(a)
(b)
Gambar 3 Elektroforegram hasil amplifikasi 16 primer kombinasi terhadap (a) PN 1546; (b) RRIM 600.
Sehingga kedua primer kombinasi tersebut juga merupakan kandidat yang akan digunakan sebagai pembeda pada analisis DNA 25 progeni hasil persilangan. Kandidat primer kombinasi terpilih tersebut kemudian diamplifikasikan terhadap 25 progeni (anakan) ) hasil persilangan antara RRIM 600 dan PN 1546. Hasil amplifikasi tersebut diharapkan dapat memperlihatkan karakter 25 progeni terhadap kedua tetuanya berdasarkan primer kombinasi terpilih tersebut, sehingga dapat diambil beberapa marka yang diduga spesifik untuk dapat membedakan antara tetua jantan (PN 1546) dan tetua betina (RRIM 600).
Hasil Amplifikasi 25 Progeni Hasil Persilangan dengan Primer
Kombinasi Terpilih
Amplifikasi terhadap DNA tetua karet, yaitu RRIM 600 dan PN 1546 dengan menggunakan 16 primer kombinasi menghasilkan lima primer kombinasi terpilih sebagai kandidat marka seleksi terhadap dua tetua karet tersebut yang selanjutnya diamplifikasikan terhadap 25 DNA progeninya. Hasil amplifikasi 25 DNA
progeni dengan lima primer kombinasi terpilih (kombinasi c, d, i, m, dan n) menghasilkan beberapa kesimpulan yang menunjukkan kedekatan masing-masing progeni dengan tetua jantan (PN 1546) maupun tetua betinanya (RRIM 600).
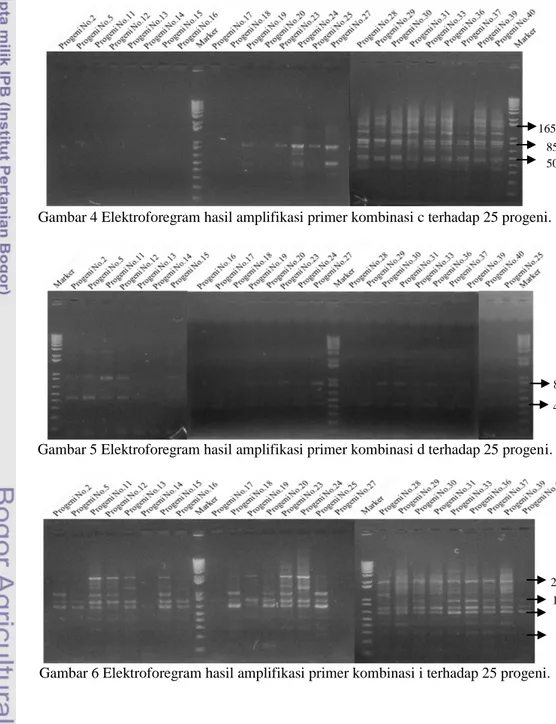
Pada kombinasi c, dari 25 progeni yang diamplifikasi, progeni no.19, 23, 24, 25, 27, 28, 29, 30, 31, 33, 36, 37, 39, dan 40 dapat diprediksi memiliki sifat lebih dekat pada PN 1546 karena memunculkan pita baik pada 400 bp, 850 bp, maupun 2000 bp. Sedangkan pada progeni no.2, 5, 11,12, 13, 14, 15, 16 ,17, 18, dan 20 karena tidak memunculkan pita, maka dapat diprediksi memiliki sifat lebih dekat pada RRIM 600 (Gambar 4).
Hasil amplifikasi primer kombinasi d terhadap 25 progeni menunjukkan bahwa pada progeni no.14, 15, dan 28 diprediksi memiliki sifat lebih dekat dengan PN 1546 karena pada elektroforegram tidak memunculkan pita. Sedangkan pada progeni no.2, 5, 11, 12, 13, 16, 17, 18, 19, 20, 23, 24, 25, 27, 29, 30, 31, 33, 36, 37, 39, dan 40 diprediksi memiliki sifat lebih dekat dengan
RRIM 600 karena memperlihatkan
munculnya pita pada kisaran 450 bp dan 850
1000 bp 650 bp 3000 bp 400 bp 650 bp 1000 bp 2000 bp
bp (Gambar 5). Pita yang muncul pada hasil amplifikasi tersebut berbeda dengan pita yang muncul pada tetua. Hasil amplifikasi primer kombinasi d terhadap kedua tetua hanya muncul pada RRIM 600, yaitu pada 2000 dan 3000 bp, sementara pada PN 1546 tidak muncul. Oleh karena itu, pada hasil amplifikasi primer kombinasi d terhadap 25 progeni, progeni yang tidak memunculkan pita dianggap lebih dekat dengan PN 1546 sedangkan yang memunculkan pita dianggap lebih dekat dengan RRIM 600 walaupun secara ukuran pita yang muncul berbeda. Pita yang muncul tersebut kemungkinan
merupakan suatu karakter yang muncul akibat adanya persilangan antar dua tetua karet, yaitu PN 1546 dan RRIM 600. Pada hasil amplifikasi 25 progeni dengan
menggunakan primer kombinasi i
memperlihatkan bahwa progeni no.5, 13, 16, 19, 24, 25, 28, dan 29 diprediksi lebih dekat dengan RRIM 600 karena memunculkan pita pada 650, 850, dan 2000, namun pada progeni no.14, 17, dan 27 pita tidak muncul sehingga sulit diprediksi apakah lebih dekat ke tetua jantan (PN 1546) atau tetua betina (RRIM 600).
Gambar 4 Elektroforegram hasil amplifikasi primer kombinasi c terhadap 25 progeni.
Gambar 5 Elektroforegram hasil amplifikasi primer kombinasi d terhadap 25 progeni.
Gambar 6 Elektroforegram hasil amplifikasi primer kombinasi i terhadap 25 progeni.
2000bp 1000bp 650bp 300bp 1650bp 850bp 400bp 850bp 500bp
12
Gambar 7 Elektroforegram hasil amplifikasi primer kombinasi m terhadap 25 progeni.
Gambar 8 Elektroforegram hasil amplifikasi primer kombinasi n terhadap 25 progeni.
Hal yang cukup menarik pada elektroforegram hasil amplifikasi primer kombinasi i terhadap 25 progeni adalah munculnya progeni yang diprediksi memiliki sifat intermediet (mengandung sifat dari dua tetuanya, yaitu PN 1546 dan RRIM 600). Sifat intermediet tersebut pada hasil amplifikasi dengan primer kombinasi i diprediksi muncul pada progeni no.2, 11, 12, 15, 18, 20, 23, 30, 31, 33, 36, 37, 39, dan 40. Hal itu terlihat dari munculnya pita yang sama baik pada PN 1546 (1000 bp) maupun RRIM 600 (650 bp, 850 bp, 1650 bp, dan 2000 bp (Gambar 6).
Analsis hasil amplifikasi primer kombinasi m terhadap 25 progeni menunjukkan bahwa pada progeni no. 2, 5, 11, 12, 15, 16, 17, 18, 19, 23, 24, dan 31 diprediksi memiliki sifat lebih dekat dengan PN 1546 karena dapat memunculkan pita pada kisaran 700 bp dan 2000 bp, sedangkan pada progeni no.14, 20, 27, 28, 29, 33, dan 36 diprediksi memiliki sifat lebih dekat dengan RRIM 600 karena hanya memunculkan pola pita yang sama dengan tetua pada 700 bp sedangkan pada 2000 bp tidak muncul (Gambar 7). Selain itu, pada progeni no.13, 25, 30, 37, 39, dan 40 tidak memunculkan
pita sehingga tidak dapat diprediksi kedekatan sifatnya terhadap tetuanya.
Hasil amplifikasi primer kombinasi n terhadap 25 progeni menunjukkan progeni no.2, 5, 11, 12, 15, 17, 23, 24, 25, 27, 28, 29, 30, 31, 33, 36, 37, dan 40 memiliki sifat intermediet karena memunculkan pita pada kisaran 2000 bp dan 3000 bp. Selain itu, pada progeni no.13 dan 39 membentuk pola pita yang berbeda dengan kedua tetuanya, serta pada progeni no.14, 16, 18, 19, dan 20 tidak memunculkan pita sehingga tidak dapat diprediksi kedekatan sifatnya terhadap tetuanya (Gambar 8).
Berdasarkan hasil analisis tersebut, terlihat bahwa primer kombinasi d, i, dan n memiliki tingkat kekonsistenan yang hampir sama. Hal itu terlihat terutama pada kombinasi d bahwa berdasarkan hasil analisis terhadap 25 progeni sebagian besar cenderung mengarah pada RRIM 600, hanya pada beberapa progeni saja yang berbeda, yaitu lebih cenderung mengarah pada PN 1546. Akan tetapi, yang menarik adalah pada primer kombinasi i dan n sebagian besar tidak ada yang mengarah kesalah satu sifat tetua, yaitu RRIM 600 atau PN 1546, tetapi kombinasi antara keduanya atau disebut dengan istilah intermediet (mengandung sifat
650bp 400bp 200bp 2000bp 300bp 650bp 3000bp 1650bp
dari kedua tetuanya). Namun hal itu tetap dianggap konsisten terhadap primer kombinasi d karena dalam intermediet mengandung sifat dari salah satu tetuanya yaitu RRIM 600.
Primer kombinasi c dan m menunjukkan hasil yang berbeda dari primer kombinasi yang lain, yaitu lebih banyak mengarah pada PN 1546 sehingga dianggap tidak dapat disarankan sebagai marka seleksi yang spesifik terhadap RRIM 600. Bila dibandingkan dengan primer kombinasi i dan n, primer kombinasi c dan m memiliki tingkat kekonsistenan yang hampir sama, karena primer kombinasi i dan n sebagian besar menunjukkan hasil analisis yang bersifat intermediet. Oleh karena itu, dari kelima primer kombinasi terpilih yang ada, untuk sementara primer kombinasi d dapat disarankan sebagai marka seleksi yang cukup spesifik terhadap RRIM 600 yang memiliki tingkat produksi lateks yang tinggi. Selanjutnya hasil analisis tersebut akan dikonfirmasi dengan data pada hasil analisis RAPD.
Hasil RAPD Karet
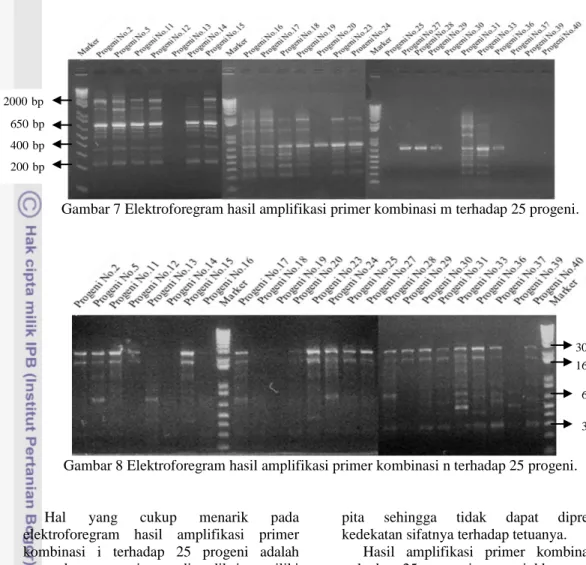
RAPD karet dilakukan dengan
menggunakan 60 jenis primer terpilih terhadap kedua tetua karet dan 25 progeni hasil persilangan untuk melihat tingkat kekerabatannya. Enam puluh primer terpilih yang digunakan dalam teknik RAPD terhadap sampel DNA karet adalah OPA (1, 2, 3, 4, 8, 10, 11, 12, 13, 14, 15, 17, 18, 19, dan 20), OPB (4, 11, 17, 19, dan 20), OPC (2, 3, 5, 7, 8, 9, 12, 13, 14, 19, dan 20), OPD (1, 2, 3, 4, 5, 7, 8, 10, 11, 14, 15, 18, dan 20), OPM (5, 7, 8, 9, 12, 16, 20), OPN (3, 5, 9, 10, 15, 16, dan 18), dan OPY (9 & 14). Hasil analisis RAPD terhadap 2 sampel DNA tetua karet dan 25 sampel DNA progeni hasil persilangan sebagian besar memperlihatkan polimorfisme. Hal itu menunjukkan sebagian besar primer yang digunakan memiliki komplemen pada setiap sampel DNA yang dianalisis baik pada tetua maupun progeninya, sehingga dapat memunculkan polimorfisme. Namun, ada beberapa sampel DNA yang tidak memunculkan polimorfisme atau bahkan tidak memunculkan pita hasil amplifikasi sama sekali. Hal tersebut menurut Purwanta (2010) kemungkinan dapat disebabkan oleh beberapa hal seperti tidak adanya situs penempelan primer yang komplemen pada DNA cetakan, tingkat
kemurnian DNA cetakan yang kurang baik, atau terfragmentasinya DNA cetakan.
Menurut Purwanta (2010), prinsip teknik RAPD didasarkan pada kemampuan primer menempel pada cetakan DNA. Primer yang didesain berupa primer tunggal pendek dimaksudkan agar primer tersebut dapat menempel secara acak pada DNA genom organisme. Oleh karena itu, akan terdapat banyak pola fragmen DNA yang terbentuk. Pola fragmen DNA tersebut dapat dilihat dengan adanya pola pita pada gel agarosa. Selain ditentukan oleh ada tidaknya situs penempelan primer, Purwanta (2010) menyebutkan bahwa keberhasilan teknik ini juga ditentukan oleh kemurnian dan keutuhan DNA cetakan. DNA cetakan yang tidak murni akan mengganggu penempelan primer pada situsnya dan akan menghambat aktifitas enzim polymerase DNA. Enzim ini berfungsi untuk melakukan polimerisasi DNA. Sedangkan DNA cetakan yang banyak mengalami fragmentasi dapat menghilangkan situs penempelan primer.
Pola pita yang dihasilkan dari hasil analisis RAPD terhadap sampel DNA karet menunjukkan polimorfisme yang cukup bagus. Hal itu terlihat dari elektroforegram hasil elektroforesis DNA seperti yang terlihat pada Gambar 9 yang memperlihatkan pola pita yang berbeda-beda pada setiap sampel DNA yang dianalisis (polimorfisme). Pita-pita DNA hasil RAPD terlihat muncul pada ukuran DNA yang bervariatif. Berdasarkan pengukuran dengan menggunakan DNA marker (1 kb DNA Ladder) terlihat bahwa pita-pita tersebut muncul pada kisaran 250 bp–5000 bp. Hal yang sama juga terlihat pada elektroforegram DNA 25 progeni (anakan) hasil persilangan (gambar terlampir). Hal itu menunjukkan bahwa antara tetua karet dengan 25 progeninya memiliki tingkat
keragaman yang tinggi. Untuk
memperlihatkan hal tersebut, pita-pita DNA yang polimorfik tersebut selanjutnya dirubah menjadi data biner untuk dikonstruksi menjadi pohon filogenetik, sehingga akan terlihat tingkat keragaman antara tetua karet dengan 25 progeni hasil persilangannya. Melalui tingkat keragaman tersebut, maka akan diketahui kelompok dan jarak genetik masing-masing progeni dengan kedua tetuanya.
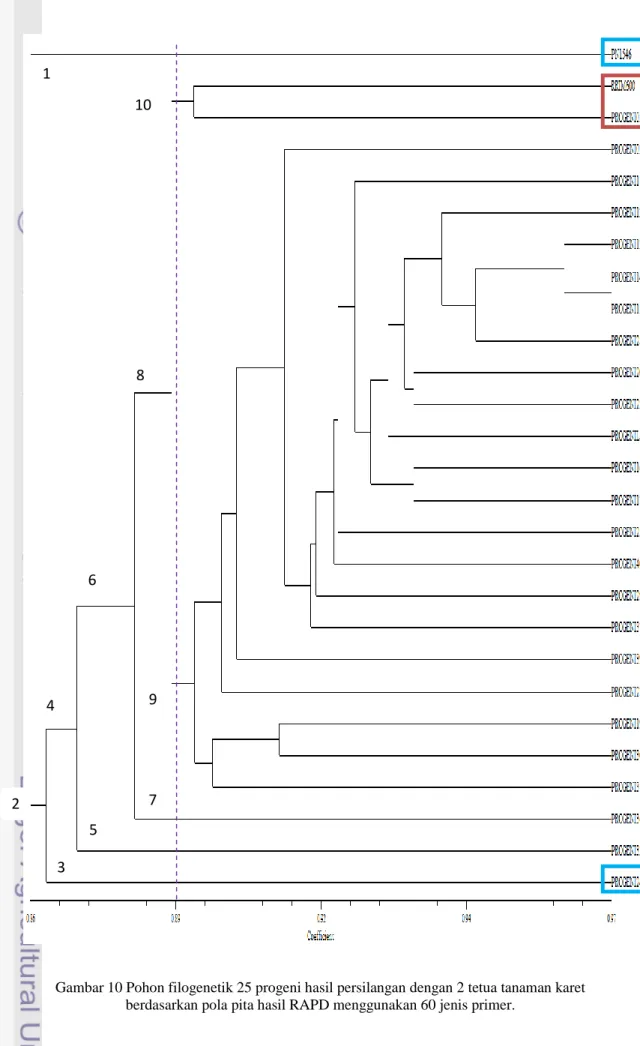
Analisis Pola Konstruksi Filogenetik
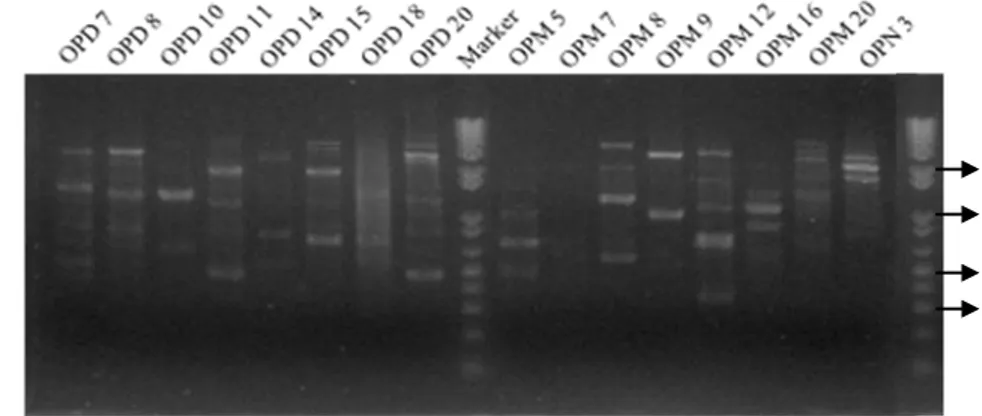
Penyusunan dendogram pohon filogenetik dilakukan berdasarkan kesamaan pola pita
14
Gambar 9 Hasil RAPD pada RRIM 600 dengan menggunakan Primer OPD, OPM, dan OPN. hasil RAPD. Bobot-bobot molekul pada pita
hasil RAPD dianggap sebagai variabel yang digunakan untuk membedakan antar progeni hasil persilangan tanaman karet. Sehingga, melalui cara ini dapat dibuat kesepakatan biner, jika terdapat pita yang muncul pada posisi berat molekul tertentu (jumlah pasang basa tertentu) maka diberi nilai 1 dan jika tidak ada maka bernilai 0 (Sari 2007). Melalui program statistik khusus NTSYS versi 2.02, dengan menggunakan metode UPGMA (Unweighted Pair Group Method
Arithmatic Mean) hasil data biner tersebut
dapat digunakan untuk membuat dendogram pohon filogenetik tanaman karet hasil persilangan dengan tetuanya. Dendogram yang terbentuk menunjukkan kelompok kekerabatan pada 25 tanaman karet hasil persilangan terhadap kedua tetuanya, berdasarkan kelompok kekerabatan tersebut maka akan diketahui jarak kekerabatan antara 25 tanaman karet hasil persilangan dengan kedua tetuanya.
Konstruksi pohon filogenetik terhadap 25 progeni hasil persilangan dengan 2 tetua tanaman karet melalui perbandingan hasil pola RAPD menunjukkan bahwa ada tingkat kekerabatan yang sangat jauh antara PN 1546 dengan RRIM 600. Hal itu terlihat pada
koefisien similaritas 86% yang
memperlihatkan dua kelompok kekerabatan, yaitu kelompok 1 dan kelompok 2 (Gambar 10). Kelompok 1 hanya terdiri dari PN 1546 sedangkan kelompok 2 terdiri dari RRIM 600 dan 25 progeni hasil persilangan. Pada kelompok 2 terdapat dua kelompok, yaitu kelompok 3 dan kelompok 4. Kelompok 3 terdiri dari progeni No.24 sedangkan kelompok 4 terdiri dari RRIM 600 dan 24 progeni hasil persilangan. Berdasarkan hal tersebut terlihat bahwa progeni No.24 memiliki tingkat kekerabatan yang cukup dekat dengan PN 1546 (Gambar 10). Pada
kelompok 4 terbagi menjadi dua kelompok, yaitu kelompok 5 dan kelompok 6. Kelompok 5 hanya terdiri dari progeni No.33 sedangkan kelompok 6 terdiri dari RRIM 600 beserta 23 progeni hasil persilangan. Pada kelompok 6 terbagi menjadi dua kelompok, yaitu kelompok 7 dan kelompok 8, yang kemudian pada koefisien similaritas 89% kelompok 8 terbagi menjadi dua kelompok, yaitu kelompok 9 dan kelompok 10. Pada kelompok 10 terlihat bahwa progeni No.5 berada satu kelompok dengan RRIM 600 dengan jarak genetik yang sama, sehingga dapat diprediksi bahwa progeni No.5 berdasarkan analisis filogenetik hasil RAPD memiliki karakter yang sama dengan RRIM 600 (Gambar 10). Hal tersebut diperkuat oleh Sari (2007) yang menyatakan bahwa suatu sampel yang berada pada kelompok kekerabatan yang sama menandakan bahwa spesies (sampel) tersebut memiliki jarak kekerabatan yang dekat. Pohon filogenetik pada Gambar 10 juga memperlihatkan bahwa pada tingkat similaritas 97% atau pada puncak pohon filogenetik, progeni No.14 dengan progeni No.15 diduga memiliki kesamaan sifat genetik. Beberapa progeni yang diduga memiliki karakter yang sama juga terlihat pada progeni No.16 dan 17, progeni No.20 dan 23, progeni No.19 dan 30 yang masing-masing memiliki jarak genetik yang sama.
Pola konstruksi pohon filogenetik juga memperlihatkan bahwa 25 progeni hasil persilangan tanaman karet berdasarkan pola pita hasil RAPD dengan menggunakan 60 jenis primer memiliki kecenderungan lebih dekat dengan tetua betina, yaitu RRIM 600 yang secara fisiologi memiliki kemampuan produksi lateks yang tinggi. Hal itu terlihat pada kelompok 2 pohon filogenetik (Gambar 10). Namun, data filogenetik tersebut belum dapat digunakan untuk menyimpulkan
500bp
300bp
2000bp
apakah 25 progeni hasil persilangan tersebut memiliki tingkat produksi lateks yang tinggi seperti halnya RRIM 600 sebagai tetuanya. Karena, tingkat produksi lateks pada suatu tanaman karet tidak dipengaruhi oleh satu gen saja melainkan sangat dipengaruhi oleh banyak gen atau poligenik. Sehingga perlu dilakukan analisis lanjutan untuk mengetahui lebih rinci terkait tingkat pautan gen yang mempengaruhi kesamaan tersebut.
Setelah melihat hasil analisis filogenetik, kemudian dilakukan konfirmasi pada hasil analisis PCR dengan primer kombinasi terpilih terhadap 25 progeni hasil persilangan. Untuk melakukan konfirmasi tersebut, dipilih beberapa progeni yang secara filogenetik memperlihatkan tingkat kekerabatan yang tinggi terhadap tetuanya, dalam hal ini dipilih progeni No.5 yang terlihat memiliki tingkat kekerabatan yang tinggi dengan RRIM 600 dan progeni No. 24 yang memiliki tingkat kekerabatan yang tinggi dengan PN 1546. Pada progeni No.5 secara filogenetik memperlihatkan tingkat kedekatannya dengan RRIM 600. Setelah dikonfirmasi dengan hasil analisis PCR dengan primer kombinasi terpilih memperlihatkan bahwa pada primer kombinasi c, d, dan i memperlihatkan hasil yang sama pada progeni No.5, yaitu diprediksi cenderung lebih dekat dengan RRIM 600. Pada progeni No.24 secara filogenetik memperlihatkan tingkat kecenderungannya lebih dekat dengan PN 1546.
Berdasarkan konfirmasi terhadap hasil analisis PCR dengan primer kombinasi terpilih pada 25 progeni hasil persilangan, menunjukkan bahwa pada primer kombinasi c dan m memperlihatkan hasil yang sama untuk progeni No.24, yaitu memiliki kecenderungan untuk lebih dekat dengan PN 1546. Pada primer kombinasi n, untuk progeni No.5 maupun progeni No.24 memperlihatkan hasil yang bersifat intermediet (memiliki sifat kedua tetuanya) sehingga tidak dapat disarankan untuk dijadikan marka pembeda. Sebelum dilakukan konfirmasi terhadap hasil analisis filogenetik, hasil PCR menggunakan primer kombinasi (semi mikrosatelit) terhadap 25 progeni hasil persilangan menunjukkan bahwa primer kombinasi d dapat disarankan sebagai marka seleksi untuk RRIM 600, sedangkan primer kombinasi c dan m sebagai marka seleksi untuk PN 1546. Berdasarkan hasil konfirmasi terhadap hasil analisis filogenetik, dapat disarankan bahwa primer kombinasi c dan m dapat digunakan sebagai
marka seleksi untuk PN 1546, sedangkan primer kombinasi d dan i dapat disarankan sebagai marka seleksi untuk RRIM 600. Marka DNA yang muncul pada setiap primer kombinasi selanjutnya dapat dianalisis lebih lanjut dengan sequensing DNA sehingga dapat diketahui secara spesifik karakter pembeda pada masing-masing tetua. Sehingga secara umum, berdasarkan pembahasan di atas, diketahui bahwa teknik RAPD dapat digunakan untuk memprediksi sifat/karakter progeni hasil persilangan tanaman karet dari sudut molekuler (bidang molekuler). Selain itu, teknik RAPD juga cukup untuk memperlihatkan tingkat kecenderungan produksi tanaman karet hasil
persilangan berdasarkan tingkat
kekerabatannya, namun untuk menunjukkan tingkat produksi lateks pada tanaman karet masih diperlukan data pendukung lainnya.
16
Gambar 10 Pohon filogenetik 25 progeni hasil persilangan dengan 2 tetua tanaman karet berdasarkan pola pita hasil RAPD menggunakan 60 jenis primer.
1 2 3 4 5 6 7 8 9 10