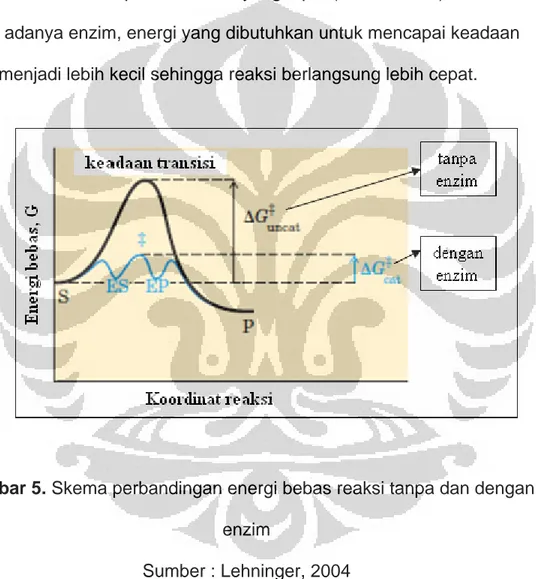
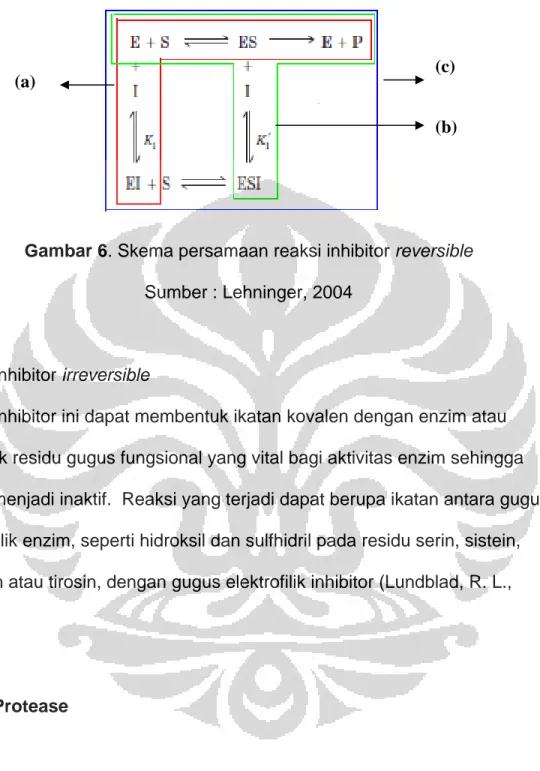
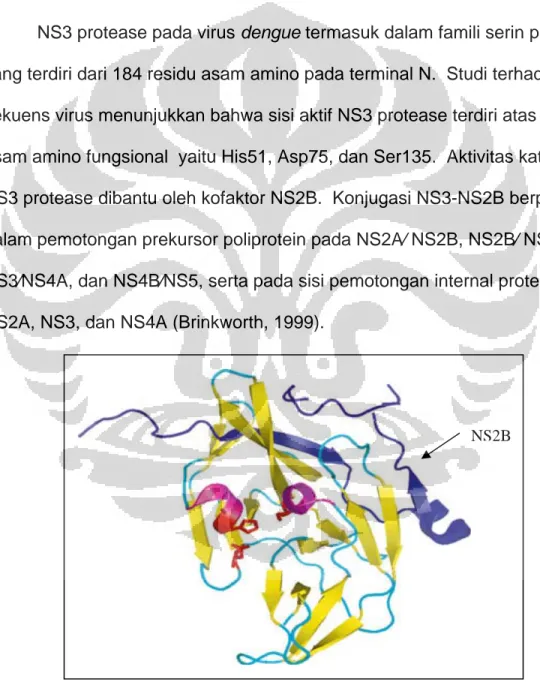
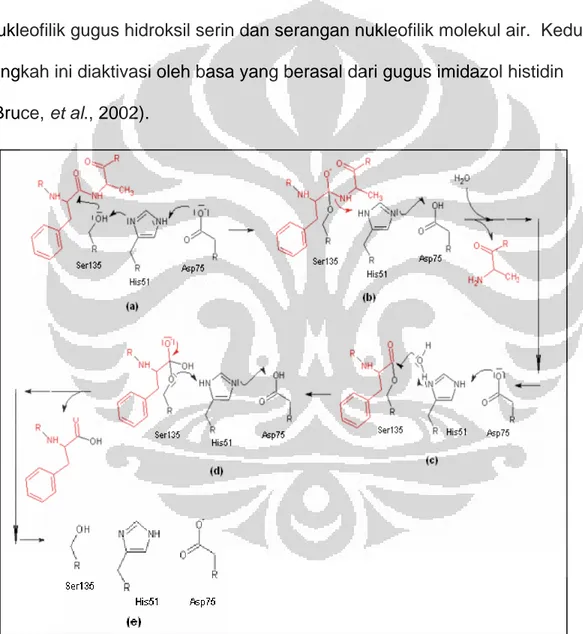
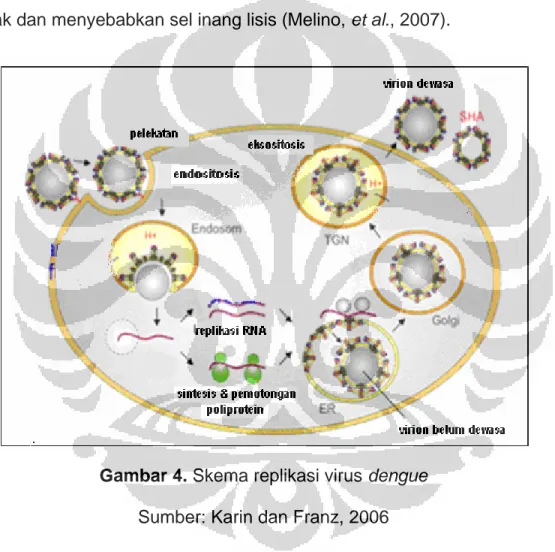
PERANCANGAN PEPTIDA SIKLIS SEBAGAI INHIBITOR POTENSIAL UNTUK ENZIM NS3-NS2B PROTEASE VIRUS DENGUE SECARA IN SILICO MELALUI MOLECULAR DOCKING
Teks penuh
Gambar
Dokumen terkait
Selanjutnya hasil yang dicapai dari kegiatan yang dilaksanakan pada tahun anggaran 2014 ini secara garis besar telah dapat dilaksanakan, dimana dari kegiatan
2. Barang bekas tersebut ada dan milik penjual sendiri, penjual juga sebenarnya tahu kondisi dari barang tersebut, namun tidak mau menjelaskan secara lengkap kepada
Pakaian dan Aksesori Pakaian terbuat dari tekstil berupa kain jadi berbentuk lembaran (tenunan maupun rajutan atau kaitan) dengan cara memotong dan menjahit
Blok ini berisi kelompok kelainan jiwa akibat penyakit otak, kerusakan otak, atau keadaan lain yang merusak fungsi otak.. Kerusakan fungsi ini bisa primer
Perhitungan neraca massa dan neraca panas diperlukan untuk menentukan kebutuhan bahan baku dan aspek penunjang lain yang diperlukan sesuai dengan kapasitas pabrik yang akan
seakan-akan itu adalah hari terakhirmu, suatu hari akan benar." ("If you live each day as if it was your last, someday you`ll most certainly be right.") Kutipan
• Untuk mendownload data formulir klik tombol Ralat di salah satu baris data pada tabel Daftar Pesawat maka sistem akan menampilkan halaman baru yang dapat dilihat
Inti dari revitalisasi mitos sungai Ninifala adalah menghidupkan kembali nilai-nilai yang terkandung dalam cerita tersebut sehingga bermuara pada kesadaran koletif masyarakat