PENENTUAN POHON FILOGENETIK BAKTERI
XILANOLITIK SISTEM ABDOMINAL RAYAP TANAH
BERDASARKAN 16S
r
RNA
SKRIPSI
SEPTHIA DWI SUKARTININGRUM
DEPARTEMEN KIMIA
FAKULTAS SAINS DAN TEKNOLOGI UNIVERSITAS AIRLANGGA
PENENTUAN POHON FILOGENETIK BAKTERI XILANOLITIK
SISTEM ABDOMINAL RAYAP TANAH BERDASARKAN
16S
r
RNA
SKRIPSI
Sebagai Salah Satu Syarat untuk Memperoleh Gelar Sarjana Sains Bidang Kimia pada Fakultas Sains dan
Teknologi Universitas Airlangga
Disetujui Oleh :
Pembimbing I,
Prof. Dr. Ni Nyoman Tri Puspaningsih, M.Si NIP. 19630615 198701 2 001
Pembimbing II,
LEMBAR PENGESAHAN SKRIPSI
Judul : Penentuan Pohon Filogenetik Bakteri Xilanolitik Sistem Abdominal Rayap Tanah berdasarkan 16S rRNA
Penyusun : Septhia Dwi Sukartiningrum
NIM : 080810518
Pembimbing I : Prof. Dr. Ni Nyoman Tri Puspaningsih, M.Si Pembimbing II : Dr. Ni’matuzahroh
Tanggal seminar : 23 Juli 2012
Disetujui Oleh : Pembimbing I,
Prof. Dr. Ni Nyoman Tri Puspaningsih, M.Si NIP. 19630615 198701 2 001
Pembimbing II,
Dr. Ni’matuzahroh NIP. 19680105 199203 2 003
Mengetahui,
Ketua Program Studi S1 Kimia Departemen Kimia Fakultas Sains dan Teknologi
Universitas Airlangga
PEDOMAN PENGGUNAAN SKRIPSI
Skripsi ini tidak dipublikasikan, namun tersedia di perpustakaan dalam lingkungan Universitas Airlangga. Diperkenankan untuk dipakai sebagai referensi kepustakaan, tetapi pengutipan seijin penulis dan harus menyebutkan sumbernya sesuai kebiasaan ilmiah.
KATA PENGANTAR
Puji syukur kehadirat Allah S.W.T yang telah melimpahkan rahmat dan hidayah-Nya sehingga penulis dapat menyelesaikan penulisan skripsi yang berjudul “Penentuan Pohon Filogenetik Bakteri Xilanolitik Sistem Abdominal Rayap Tanah berdasarkan 16S rRNA”
Skripsi ini dibuat untuk memenuhi persyaratan akademis pendidikan
sarjana sains dalam bidang kimia Fakultas Sains dan Teknologi Universitas
Airlangga.
Pada kesempatan ini, penulis ingin mengucapkan terima kasih yang sebesar-besarnya kepada :
1. Prof. Dr. Ni Nyoman Tri Puspaningsih, M.Si selaku dosen pembimbing I atas bimbingan dan nasehatnya selama penyusunan dan penyelesaian skripsi ini, 2. Dr. Ni’matuzahroh selaku dosen pembimbing II atas bantuan dan kesabaran
dalam memberikan bimbingan kepada penulis,
3. Dr. Sri Sumarsih, M.Si selaku penguji I atas nasehat dan sarannya kepada penulis,
4. Drs. Hamami, M.Si selaku penguji II atas saran dan masukannya kepada penulis,
6. Dr. Alfinda Novi Kristanti, DEA selaku Ketua Departemen Kimia yang telah memberikan fasilitas serta arahan selama penyusun belajar di Departemen Kimia,
7. Bapak dan ibu dosen Departemen Kimia Universitas Airlangga yang telah senantiasa membagikan ilmu dan nasehat kepada penulis,
8. Bapak dan ibu selaku orang tua yang memberikan kasih sayang, doa, kepercayaan, dan dukungan baik secara moril maupun materi,
9. Ibu A.A. Istri Ratna Dewi yang telah menemani dan memberi saran saat penelitian,
10. Seluruh keluarga besar Departemen Kimia dan FSAINTEK yang telah memberikan banyak ilmu, nasehat, dan dukungan,
11. Teman-teman satu penelitian (Amaliah dan Previta) yang telah banyak membantu dalam mengerjakan penelitian, memberi saran dan dukungan yang sangat berharga, memberi keceriaan serta hiburan ketika penulis bersedih, 12. Sahabat-sahabat tercinta (Laudita, Faya, Yudistia, Yudha, Dyah Respati,
Mella, Mala) yang telah sabar menampung dan mendengarkan segala keluh kesah dan isak tangis, memberi saran dan dukungan, serta memberikan tempat berteduh ketika penulis lelah,
14. Teman–teman S1 Kimia angkatan 2008 yang senantiasa menemani dalam menuntut ilmu, memberikan banyak dukungan serta semangat selama penulis menjalankan masa pendidikan S1 di Universitas Airlangga,
15. Kakak-kakak di laboratorium Proteomik, TDC (mbak Nita, mbak One, mbak Laura, mas Ivan, mbak Titin) atas kesabaran dalam membagikan ilmu dan menuntun penulis dari awal sampai akhir penelitian,
16. Serta pihak–pihak yang tidak dapat disebutkan satu persatu yang banyak memberikan saran, masukan, dan pengalamannya,
Penulis menyadari bahwa masih terdapat banyak kekurangan dalam penyusunan skripsi ini, oleh karena itu kritik dan saran yang besifat membangun untuk kesempurnaan penulisan skripsi ini sangat diperlukan. Semoga skripsi ini dapat bermanfaat bagi semua pihak.
Surabaya, Juli 2012 Penulis,
Sukartiningrum, S.D., 2012, Penentuan Pohon Filogenetik Bakteri Xilanolitik Sistem Abdominal Rayap Tanah berdasarkan 16S rRNA. Skripsi ini di
bawah bimbingan Prof. Dr. Ni Nyoman Tri Puspaningsih, M.Si., dan Dr. Ni’matuzahroh, Departemen Kimia, Fakultas Sains dan Teknologi, Universitas Airlangga, Surabaya
ABSTRAK
Penelitian bertujuan untuk mengetahui hubungan kekerabatan bakteri xilanolitik isolat B dan 7 penghasil endo-β-1,4-xilanase hasil isolasi dari sistem abdominal rayap tanah Macrotermes sp. Identifikasi dilakukan dengan cara mendesain pohon filogenetik berdasarkan sekuen gen penyandi 16S rRNA. Sekuen gen penyandi 16S rRNA didapatkan dengan cara mengamplifikasi gen penyandi 16S rRNA menggunakan teknik PCR. Amplifikasi gen penyandi 16S rRNA menggunakan primer forward B27F dan primer reverse U1492R. Dari proses amplifikasi gen penyandi 16S rRNA dengan teknik PCR, didapatkan sekuen gen penyandi 16S
rRNA milik bakteri xilanolitik isolat B dan isolat 7 sebesar 1460 bp dan 1423 bp. Desain pohon filogenetik menunjukkan bahwa bakteri xilanolitik sistem abdominal rayap tanah isolat B mempunyai hubungan kekerabatan terdekat dengan Bacillus anthracis dan bakteri xilanolitik sistem abdominal rayap tanah isolate 7 mempunyai hubungan kekerabatan terdekat dengan Escherichia fergusonii.
Sukartiningrum, S.D., 2012, Determination of Phylogenetic Tree of Xylanolitic Bacteria Abdominal System Soil Termite based on 16S rRNA.
This script is supervised by Prof. Dr. Ni Nyoman Tri Puspaningsih, M.Si., and Dr. Ni’matuzahroh, Department of Chemistry, Faculty of Science and Technology, Airlangga University, Surabaya.
ABSTRACT
The purpose of research is to determine the relationship of isolates B and 7 from endo-β-1,4-xylanase-producing xylanolitic bacteria. Identification was done by designed a phylogenetic tree based on molecular identification of genes encoding 16S rRNA. Sequences of genes encoding 16S rRNA can be obtained by amplification of genes encoding 16S rRNA using PCR technique. Amplification for 16S rRNA gene encoding using B27F forward primer and U1492R reverse primer. From gene coding for 16S rRNA amplification by PCR technique, it has been found the sequences of gene encoding 16S rRNA sequences belonging xylanolitic isolates B and isolates 7 of 1460 bp and 1423 bp. Design of a phylogenetic tree showed that the xylanolitic bacteria abdominal system soil termite isolate B has a closest relathionship with Bacillus anthracis and the xylanolitic bacteria abdominal system soil termite isolate 7 has a closest relathionship with Escherichia fergusonii.
DAFTAR ISI 2.1 Keberadaan Mikroorganisme Penghasil Xilanase pada Rayap ... 6
2.1.1 Protozoa pada rayap... 6
3.2 Sampel dan Bahan Penelitian ... 20
3.5.3 Peremajaan bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7... 25
3.5.4 Perbanyakan sel dan isolasi DNA kromosom bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7 ... 25
3.5.5 Penentuan konsentrasi DNA kromosom ... 26
3.5.6 Elektroforesis gel agarosa ... 27
3.5.7 Proses amplifikasi gen penyandi 16S rRNA dengan teknik PCR ... 27
3.5.8 Sekuensing gen penyandi 16S rRNA ... 28
3.5.9 Desain pohon filogenetik ... 28
BAB IV HASIL DAN PEMBAHASAN 4.1 Isolasi DNA Kromosom Bakteri Xilanolitik Sistem Abdominal Rayap Tanah Isolat B dan Isolat 7 ... 30
4.2 Amplifikasi Gen Penyandi 16S rRNA dengan Teknik PCR ... 31
DAFTAR TABEL
Nomor Judul Tabel Halaman
2.1 Komposisi ribosom pada prokaryot dan eukaryot... 9 2.2 Primer universal untuk amplifikasi 16S rRNA... 12 2.3 Macam-macam bufer elektroforesis... .. 18 4.1 Hasil BLAST bakteri xilanolitik sistem abdominal rayap tanah
isolat B……….. 38 4.2 Hasil BLAST bakteri xilanolitik sistem abdominal rayap tanah
DAFTAR GAMBAR
Nomor Judul Gambar Halaman
2.1
DAFTAR LAMPIRAN
Nomor Judul
1 Perhitungan Pembuatan Media Luria-Bertani Padat dan Cair 2 Perhitungan Pembuatan Larutan untuk Isolasi DNA Kromosom 3 Perhitungan Pembuatan Larutan dan Bahan untuk Elektroforesis 4 Perhitungan Larutan Kerja untuk Reaksi PCR
5 Proses Alignment Sekuen Gen Penyandi 16S rRNA Bakteri Xilanolitik Menggunakan Program Clone Manager
6 Proses Pelacakan Sekuen Gen Penyandi 16S rRNA Bakteri Xilanolitik dengan Sekuen Gen Penyandi 16S rRNA Bakteri Lain di Genbank
7 Proses Multiple Sequence Alignmet dari Data BLAST yang Diperoleh dengan Menggunakan Program ClustalW dan MEGA5 8 Proses Pendesainan Pohon Filognetik Menggunakan Program
DAFTAR LAMPIRAN
Nomor Judul
1 Perhitungan Pembuatan Media Luria-Bertani Padat dan Cair 2 Perhitungan Pembuatan Larutan untuk Isolasi DNA Kromosom 3 Perhitungan Pembuatan Larutan dan Bahan untuk Elektroforesis 4 Perhitungan Larutan Kerja untuk Reaksi PCR
5 Proses Alignment Sekuen Gen Penyandi 16S rRNA Bakteri Xilanolitik Menggunakan Program Clone Manager
6 Proses Pelacakan Sekuen Gen Penyandi 16S rRNA Bakteri Xilanolitik dengan Sekuen Gen Penyandi 16S rRNA Bakteri Lain di Genbank
7 Proses Multiple Sequence Alignmet dari Data BLAST yang Diperoleh dengan Menggunakan Program ClustalW dan MEGA5 8 Proses Pendesainan Pohon Filognetik Menggunakan Program
BAB II
TINJAUAN PUSTAKA
2.1 Keberadaan Mikroorganisme Penghasil Xilanase pada Rayap
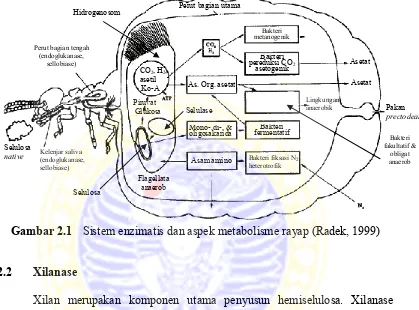
Rayap termasuk serangga perusak kayu yang sangat potensial karena rayap dapat mencerna material-material yang terkandung di dalam kayu. Material-material tersebut berupa selulosa, hemiselulosa, dan lignin. Adanya kemampuan pencernaan khusus tersebut dikarenakan di dalam sistem pencernaan rayap terdapat suatu mikroorganisme khusus yang membantu mencerna dan merombak material-material tersebut (Setford, 2005) (Gambar 2.1). Mikroorganisme tersebut antara lain metazoa, protozoa atau protista, bakteri, dan fungi (Purwadaria dkk., 2004). Enzim yang berfungsi untuk mencerna dan merombak hemiselulosa adalah enzim xilanolitik. Mikroorganisme penghasil enzim xilanolitik tersebut dapat juga ditemukan di dalam sistem pencernaan rayap.
2.1.1 Protozoa pada rayap
Protozoa dapat ditemukan di dalam sistem pencernaan rayap jenis
Mastotermitidae, Kalotermitidae, dan Rhinotermitidae. Protozoa tersebut berperan dalam melumatkan selulosa sehingga dapat dicerna dan diserap oleh rayap.
2.1.2 Bakteri pada rayap
Pada rayap famili Termitidae (Macrotermes, Odontotermes, dan
yang berperan sebagai penghasil enzim xilanolitik, yaitu Bacillus sp. (Shimizu et al., 1998) dan Bacillus pumilus (Purwadaria dkk., 2004).
Gambar 2.1 Sistem enzimatis dan aspek metabolisme rayap (Radek, 1999)
2.2 Xilanase
Xilan merupakan komponen utama penyusun hemiselulosa. Xilanase merupakan enzim ekstraseluler yang menghidrolisis polisakarida β-1,4-xilan yang merupakan komponen utama hemiselulosa pada tumbuhan. Berdasarkan substrat yang dihidrolisis, xilanase dapat diklasifikasikan sebagai β-1,4-xilosidase, eksoxilanase, dan endo-β-1,4-xilanase (endoxilanase) (Richana, 2002). Gambar 2.2 menunjukkan sisi pemotongan rantai xilan dari enzim-enzim xilanolitik. 2.2.1 Endo-β-1,4-xilanase
Endo-β-1,4-xilanase merupakan salah satu kelompok xilanase yang mampu menguraikan rantai utama xilan menjadi xilooligosakarida rantai pendek dan xilosa dan juga mampu memutus ikatan β-1,4 pada bagian dalam rantai xilan
Selulosa
secara teratur (Bravman et al, 2001). Ikatan yang diputus ditentukan berdasarkan panjang rantai substrat, derajat percabangan, ada tidaknya gugus substitusi, dan pola pemutusan dari enzim hidrolase (Richana, 2002).
Gambar 2.2 Enzim xilanolitik pada xilan tumbuhan (Collins et al., 2005) 2.3 Identifikasi Bakteri
Proses identifikasi bakteri mengalami perkembangan dari tahun ke tahun. Pada awal perkembangannya, pengklasifikasian bakteri hanya didasarkan pada morfologinya. Dari identifikasi morfologi inilah muncul nama-nama bakteri seperti Bacillus, Coccus, Streptococcus, dan lain-lain. Pada tahap perkembangan selanjutnya, pengklasifikasian bakteri menggunakan metode pendekatan fisiologi. Sistem penamaan bakteri yang didasarkan pada penggabungan sifat morfologi dan fisiologi menjadi semakin sulit dan kompleks, sehingga dilakukan perkembangan pengklasifikasian lebih lanjut.
(Murray and Holt in Boone et al., 2001). Metode ini digunakan untuk menentukan kebaharuan isolat bakteri yang ditemukan dari alam.
2.3.1 Ribosom RNA
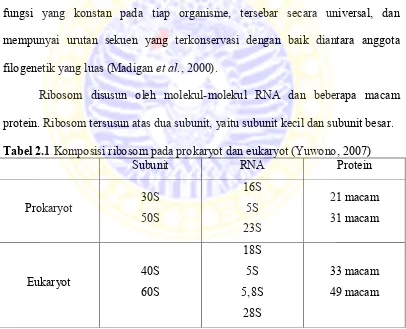
Ribosom merupakan organel kecil dan padat dalam sel yang terdiri atas protein dan molekul RNA (ribonucleic acid). Ribosom berfungsi dalam proses translasi (sintesis protein). Suatu sel dapat mengandung sampai 10.000 ribosom sehingga massa selnya dapat mencapai 40% dari massa total sel bakteri (Yuwono, 2007). Ribosom RNA merupakan molekul yang sempurna karena mempunyai fungsi yang konstan pada tiap organisme, tersebar secara universal, dan mempunyai urutan sekuen yang terkonservasi dengan baik diantara anggota filogenetik yang luas (Madigan et al., 2000).
Ribosom disusun oleh molekul-molekul RNA dan beberapa macam protein. Ribosom tersusun atas dua subunit, yaitu subunit kecil dan subunit besar. Tabel 2.1 Komposisi ribosom pada prokaryot dan eukaryot (Yuwono, 2007)
Carl Woese (1967) menampilkan tiga domain dari “theory of life”
berdasarkan gen yang mengkode ribosom RNA, meliputi eubacteria, archaea, dan eukariot. Gen yang mengkode ribosom RNA bersifat conserved (dipertahankan) oleh suatu spesies dan tersebar di seluruh organisme. Urutan DNA pengkode
rRNA, yaitu rDNA, digunakan untuk merekonstruksi filogenetik, mengidentifikasi golongan taksonomi suatu organisme, memperkirakan hubungan suatu golongan dengan golongan lainnya, serta mengestimasi tingkat perbedaan suatu spesies dengan spesies lainnya. Ribosom DNA (rDNA) ini penting dalam pembuatan pohon filogenetik yang berkaitan dengan evolusi.
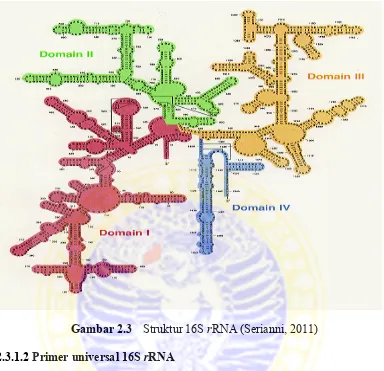
2.3.1.1 16S rRNA
16S RNA ribosom atau 16S rRNA adalah komponen dari subunit kecil 30S pada ribosom prokariot. Sekuen dari 16S rRNA mencapai 1500 bp. Sekuen basa tersebut digunakan untuk merekonstruksi filogeni. 16S rRNA mempunyai fungsi, antara lain menerjemahkan posisi protein dari ribosom, berinteraksi dengan 23S dan membantu dalam pengikatan dua subunit ribosom yaitu unit 50S dan 30S, serta menstabilkan pasangan kodon-antikodon melalui pembentukan ikatan hidrogen antara atom N1 dari adenine dengan 'OH pada mRNA.
Gambar 2.3 Struktur 16S rRNA (Serianni, 2011) 2.3.1.2 Primer universal 16S rRNA
Analisis 16S rRNA dilakukan dengan bantuan primer universal. Primer universal menargetkan dan memperkuat wilayah lestari gen 16S rRNA agar dapat mengamplifikasi seluruh urutan 16S rRNA secara lengkap. Primer universal
terdiri dari primer forward (mengamplifikasi bagian awal) dan primer reverse
(mengamplifikasi bagian akhir) (Huber et al., 2002). Pasangan primer universal
Tabel 2.2 Primer universal untuk amplifikasi 16S rRNA (Weidner et al., 1996)
Dalam ilmu biologi, filogeni atau filogenesis adalah kajian mengenai hubungan antara kelompok-kelompok organisme yang dikaitkan dengan proses evolusi. Filogenetik merupakan ilmu yang mempelajari hubungan evolusi beberapa kelompok organisme yang berbeda (contohnya spesies atau populasi). Dalam studi filogenetik, cara yang paling tepat untuk menghubungkan beberapa kelompok organisme adalah dengan membuat atau mendesain pohon filogenetik. Pohon filogenetik digunakan untuk membatasi taksa masing-masing kelompok individu yang saling terhubung.
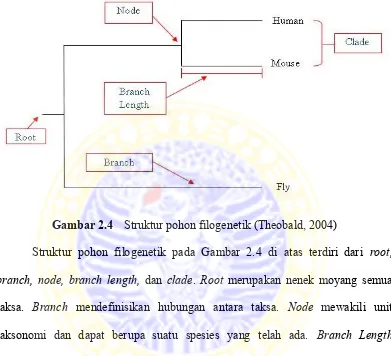
2.4.1 Struktur pohon filogenetik
Sebuah pohon filogenetik terdiri dari node dan cabang. Masing-masing
pohon filogenetik dapat menghubungkan dua node yang mempunyai kekerabatan. Pola percabangan pohon ini disebut topologi.
Gambar 2.4 Struktur pohon filogenetik (Theobald, 2004)
Struktur pohon filogenetik pada Gambar 2.4 di atas terdiri dari root, branch, node, branch length, dan clade. Root merupakan nenek moyang semua taksa. Branch mendefinisikan hubungan antara taksa. Node mewakili unit taksonomi dan dapat berupa suatu spesies yang telah ada. Branch Length
mewakili jumlah perubahan yang telah terjadi. Clade berupa kelompok dua taksa atau lebih.
2.5 Polymerase Chain Reaction (PCR)
Polymerase chain reaction atau reaksi rantai polimerase adalah teknik ilmiah dalam biologi molekular yang digunakan untuk mengamplifikasi beberapa basa DNA menjadi ribuan sampai jutaan kopi basa. PCR dikembangkan oleh Kary Mullis pada 1983 (Bartlett et al., 2003). Reaksi PCR merupakan reaksi replikasi DNA yang terjadi di luar tubuh makhluk hidup.
2.5.1 Komponen PCR
Reaksi polymerase chain reaction membutuhkan beberapa komponen. Komponen-komponen tersebut meliputi primer, dNTP, bufer, kation divalen, DNA cetakan, dan DNA polimerase.
Primer adalah sepasang DNA untai tunggal atau oligonukleotida rantai pendek yang menginisiasi gen DNA target. dNTP alias building blocks berfungsi sebagai ‘batu bata’ penyusun DNA yang baru. dNTP terdiri atas 4 macam sesuai dengan basa penyusun DNA, yaitu dATP, dCTP, dGTP, dan dTTP. Bufer berfungsi untuk mengkondisikan reaksi dan menyediakan lingkungan kimia yang cocok agar PCR berjalan optimum dan menstabilkan DNA polimerase. Kation divalen yang umum digunakan dalam reaksi PCR adalah magnesium (Mg2+). Kation divalen berfungsi sebagai kofaktor DNA polimerase. DNA cetakan merupakan sumber gen DNA target. DNA polimerase berfungsi sebagai enzim. DNA polimerase mempunyai suhu optimal sekitar 700C (Sambrook et al., 2001). 2.5.2 Tahapan PCR
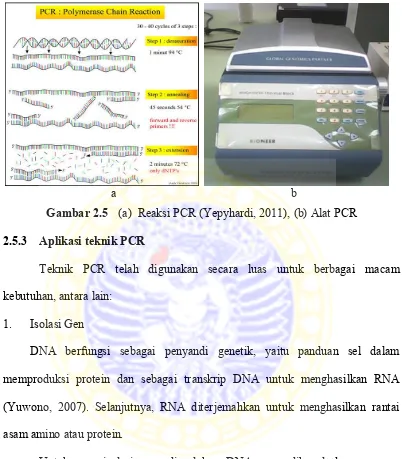
2.5(b)). Reaksi PCR mempunyai siklus optimal antara 20 - 40 siklus, dimana masing-masing siklus terdiri dari 2 - 3 suhu berbeda (Rychlik et al., 1990). Reaksi PCR mempunyai beberapa tahapan, antara lain inisiasi, denaturasi, annealing,
extension, dan elongasi akhir (Gambar 2.5(a)).
Tahap inisiasi dilakukan pada suhu 94- 960 C selama untuk 1 - 9 menit. Tahap denaturasi dilakukan pada suhu 94- 980 C selama 30 - 60 detik. Pada suhu ini, DNA untai ganda akan memisah menjadi DNA untai tunggal. Tahap
annealing merupakan tahap yang memberikan kesempatan bagi primer untuk menempel pada DNA cetakan di tempat yang komplemen dengan sekuen primer. Suhu reaksi yang dipakai pada saat annealing berkisar antara 50- 650 C selama 20 - 40 detik. Biasanya suhu annealing diturunkan tiga sampai lima derajad celcius di bawah Tm dari primer yang digunakan. Perpanjangan atau extension adalah tahapan dimana DNA polimerase akan mensintesis untai DNA baru untuk melengkapi untai DNA cetakan (DNA template) dengan menambahkan dNTP dalam arah 5' ke 3'. Suhu yang umum dipakai pada tahap extension adlah 720C.
a b
Gambar 2.5 (a) Reaksi PCR (Yepyhardi, 2011), (b) Alat PCR 2.5.3 Aplikasi teknik PCR
Teknik PCR telah digunakan secara luas untuk berbagai macam kebutuhan, antara lain:
1. Isolasi Gen
DNA berfungsi sebagai penyandi genetik, yaitu panduan sel dalam memproduksi protein dan sebagai transkrip DNA untuk menghasilkan RNA (Yuwono, 2007). Selanjutnya, RNA diterjemahkan untuk menghasilkan rantai asam amino atau protein.
Untuk mengisolasi gen, diperlukan DNA yang dikenal dengan nama ‘probe’. DNA probe memiliki urutan basa nukleotida yang sama dengan gen target yang akan diisolasi. Probe dibuat dengan teknik PCR dengan menggunakan primer yang sesuai dengan gen tersebut.
2. Sekuensing DNA
termination method) yang sudah dimodifikasi menggunakan dye-dideoxy terminator, dimana pada proses awal reaksi PCR hanya menggunakan satu primer dan tambahan dideoxynucleotide yang diberi label fluorescent. Warna fluorescent
setiap basa berbeda. Perbedaan warna tersebut digunakan untuk membedakan dan menentukan urutan basa suatu DNA yang tidak diketahui.
2.6 Elektroforesis Gel Agarosa
Menurut Yuwono (2007), elektroforesis adalah suatu teknik pemisahan molekul sel berdasarkan massa dan bentuk molekulnya dengan menggunakan medan listrik yang dialirkan pada suatu medium yang mengandung sampel yang akan dipisahkan. Elektroforesis memanfaatkan muatan listrik yang ada pada makromolekul, yaitu DNA yang bermuatan negatif. Jika molekul yang bermuatan negatif dilewatkan melalui suatu medium, kemudian dialiri arus listrik dari kutub positif ke kutub negatif, maka molekul tersebut akan bergerak dari kutub negatif ke kutub positif.
Teknik elektroforesis dapat digunakan untuk menganalisis DNA, RNA, maupun protein. Elektroforesis DNA digunakan untuk menganalisis fragmen-fragmen DNA hasil pemotongan enzim restriksi, hasil isolasi DNA kromosom, produk PCR, dan lain sebagainya. Elektroforesis DNA memerlukan gel agarosa (Gambar 2.6(a)). Agarosa merupakan suatu bahan semi-padat berupa polisakarida yang diekstraksi dari rumput laut.
beberapa macam. Macam-macam bufer yang digunakan untuk elektroforesis dan melarutkan agarosa dapat dilihat pada Tabel 2.3.
Tabel 2.3 Macam-macam bufer elektroforesis (Sambrook, 2001)
Buffer Working solution Concentrated stok solution (per
liter)
BAB III
METODE PENELITIAN
3.1 Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan pada bulan Februari 2012 sampai dengan Juni 2012 di Laboratorium Proteomik, ITD, Universitas Airlangga.
3.2 Sampel dan Bahan Penelitian 3.2.1 Sampel penelitian
Sampel penelitian ini adalah isolat bakteri xilanolitik sistem abdominal rayap tanah (isolat A, isolat B, dan isolat 7) yang merupakan koleksi bakteri xilanolitik milik Ratnadewi dkk (2007).
3.2.2 Bahan penelitian
3.3 Alat Penelitian
Peralatan yang digunakan dalam penelitian ini adalah autoklaf (TOMY
3.4 Diagram Alir Penelitian
Isolasi DNA kromosom bakteri xilanolitik sistem abdominal rayap tanah isolat A, B, dan 7
Penentuan konsentrasi dan kemurnian DNA kromosom
Amplifikasi gen 16S rRNA dengan teknik PCR
Elektroforesis gel agarosa
Sequensing produk PCR
Desain pohon filogenetik
3.5 Prosedur Penelitian 3.5.1 Pembuatan larutan
Pembuatan larutan meliputi pembuatan 0,05 M bufer TE (bufer Tris-Cl, EDTA), 3 M Na-asetat, bufer Loading Dye, bufer TAE (Tris-asetat EDTA). 3.5.1.1 Pembuatan 50 mM bufer TE
0,05 M bufer TE dibuat dari larutan stok 0,5 M tris-Cl pH 8 dan 0,5 M EDTA pH 8.
Larutan stok 0,5 M tris-Cl pH 8 dibuat sebanyak 50 mL. Ditimbang 3,0285 gr tris-base kemudian dilarutkan dalam 25 mL akuades. Selanjutnya ditambahkan HCl 1 M dan diukur pH-nya sampai mencapai pH 8. Lalu ditambahkan akuades hingga mencapai volume 50 mL.
Larutan stok 0,5 M EDTA dibuat sebanyak 50 mL. Ditimbang 9,306 EDTA kemudian dilarutkan dalam 50 mL akuades. Lalu ditambahkan NaOH 1 N sampai mencapai pH 8.
0,05 M bufer TE dibuat sebanyak 50 mL. Dicampur 5 mL 0,5 M tris-Cl pH 8 dan 5 mL 0,5 M EDTA pH 8, kemudian diencerkan dengan akuades hingga mencapai volume 50 mL.
Ketiga larutan di atas disterilkan menggunakan autoklaf pada suhu 1210C selama 15 menit.
3.5.1.2 Pembuatan 3 M Na-asetat
3.5.1.3 Pembuatan bufer Loading Dye
Bufer Loading Dye dibuat dari campuran 0,25% Bromophenol Blue dan 40% sukrosa. Ditimbang 0,0125 gr Bromophenol Blue dan 2 gr sukrosa, kemudian dilarutkan dalam 5 mL akuades. Bufer Loading Dye disimpan di dalam lemari pendingin -200C.
3.5.1.4 Pembuatan bufer TAE
Bufer TAE dibuat dari campuran tris-base, asam asetat glasial, dan EDTA pH 8. Ditimbang 48,4 gr tris-base, 11,42 gr asam asetat glasial, dan 20 mL 0,5 M EDTA pH 8, kemudian dilarutkan dalam 200 mL akuades.
3.5.2 Pembuatan media Luria-Bertani padat dan cair
Media Luria-Bertani padat dibuat dari campuran 1% Tryptone, 1% NaCl, 2% Bacto Agar, dan 0,5% Yeast Exstract. Ditimbang 0,5 gr Tryptone, 0,5 gr NaCl, 0,25 gr Yeast Exstract, dan 1 gr Bacto Agar. Semua bahan dilarutkan dalam 50 mL akuades. Selanjutnya media tersebut dipindahkan ke dalam Erlenmeyer dan disterilkan menggunakan autoklaf pada suhu 1210C selama 15 menit. Media steril yang masih hangat-hangat kuku dihomogenkan kemudian dituang ke dalam cawan Petri steril. Media yang telah memadat disimpan dalam lemari pendingin 40C.
Media Luria-Bertani cair dibuat dari campuran 1% Tryptone, 1% NaCl, dan 0,5% Yeast Exstract. Ditimbang 0,6 gr Tryptone, 0,6 gr NaCl, dan 0,3 gr
3.5.3 Peremajaan bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7
Proses peremajaan bakteri dimulai dari mengambil biakan bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7 dari kultur sebelumnya dengan menggunakan kawat ose. Kawat yang mengandung biakan bakteri digoreskan pada media Luria-Bertani padat menggunakan metode streak. Selanjutnya biakan diinkubasi pada oven dengan suhu 370C selama 18 jam.
3.5.4 Perbanyakan sel dan isolasi DNA kromosom bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7
Proses perbanyakan sel bakteri xilanolitik dilakukan dengan menumbuhkan bakteri pada media Luria-Bertani cair. Kultur bakteri di-shaker
dengan kecepatan 150 rpm selama 18 jam pada suhu 370C.
volume total campuran, kemudian dicampur secara perlahan, dan diinkubasi selama 12 jam pada suhu -200C. Setelah diinkubasi, campuran disentrifugasi pada kecepatan 12.000 rpm selama 5 menit suhu 40C. Supernatan yang terbentuk dibuang, pelet yang ada di dasar tabung dicuci dengan 500 µL etanol 70%, lalu disentrifugasi kembali dengan kecepatan 12.000 rpm selama 10 menit suhu 40C. Supernatan dibuang dan pelet yang terbentuk dikeringkan selama beberapa menit pada temperatur ruang. Pelet yang telah kering dilarutkan dalam 30 µL ddH2O.
Larutan lisozim berfungsi untuk proses lisis dinding sel bakteri. Penambahan larutan STEP (SDS, Tris-cl, EDTA, Proteinase-K) berfungsi untuk penyempurnaan kerusakan dinding dan membran sel bakteri secara kimiawi dan enzimatis. Penambahan fenol jenuh berfungsi untuk memisahkan protein dari DNA. Pada tahap ini akan terbentuk tiga lapisan, yaitu lapisan atas merupakan larutan DNA dan RNA yang larut dalam fasa air, lapisan tengah merupakan larutan protein, dan lapisan bawah merupakan sisa fenol. Natrium asetat dan etanol absolut dingin berfungsi untuk presipitasi DNA (Brown, 2001).
3.5.5 Penentuan konsentrasi DNA kromosom
3.5.6 Elektroforesis gel agarosa
Elektroforesis DNA menggunakan gel agarosa 1%. Gel agarosa 1% dibuat dari 0,4 gr agarosa yang dilarutkan dalam 40 mL bufer TAE 0,5x. Sampel DNA kromosom dicampur dengan bufer Loading Dye dengan perbandingan 3:1, kemudian dielektroforesis pada tegangan 60 - 70 Volt sampai warna biru bermigrasi sepanjang 3
4 gel. Gel agarosa kemudian direndam dalam larutan EtBr 0,5 g/mL dalam bufer TAE 0,5x selama 5 - 10 menit. Pita-pita DNA diamati dengan sinar UV dan didokumentasi dengan kamera digital.
3.5.7 Proses amplifikasi gen penyandi 16S rRNA dengan teknik PCR
Bahan yang digunakan untuk proses amplifikasi gen penyandi 16S rRNA dengan teknik PCR adalah 2,5 µL bufer PCR 10x (KCl 0,5 M, Tris-HCl 0,1 M pH 8,0, MgCl2 0,015 M), 1 µL dNTP 0,01 M, 2 µL MgSO4, 2 µL primer B27F (5’AGAGTTTGATCCTGGCTCAG 3’), 2 µL primer U1492R (5’ -GGTTACCTTGTTACGACTT- 3’), 6 µL DNA cetakan, 1 µL Taq DNA polimerase 5 units/ µL, dan 5 µL ddH2O.
3.5.8 Sekuensing gen penyandi 16S rRNA
Sekuensing gen penyandi 16S rRNA dilakukan di laboratorium Macrogen, Singapura dengan menggunakan primer forward B27F dan primer reverse
U1492R.
3.5.9 Desain pohon filogenetik
Pohon filogenetik didesain menggunakan perbandingan sekuen 16S rRNA dari bakteri lain pada program pelacakan database Basic Local Alignment Search Tool (BLAST) dengan alamat situs http://blast.ncbi.nlm.nih.gov/Blast.cgi.
Alignment divisulisasikan menggunakan program ClustalW. Pembentukan pohon filogenetik dilakukan dengan menggunakan program MEGA5. Langkah-langkah pembuatan pohon filogenetik adalah sebagai berikut:
1. Sekuen 16S rRNA milik bakteri xilanolitik didesain menggunakan program
Clone Manager dengan langkah-langkah: a. Buka program Clone Manager.
b. Pilih menu Align Align multiple sequences.
c. Pilih tipe alignment Multi-Way dan align sequences as DNAnext. d. Masukkan data basa nukleotida hasil sekuen primer forward dan reverse
finish.
e. Gabungkan basa-basa nukleotida hasil sekuen 16S rRNA dan simpan dalam format text document (.txt).
a. Buka program BLAST pada alamat situs http://blast.ncbi.nlm.nih.gov/.
b. Klik pilihan nucleotide blast.
c. Masukkan data 16S rRNA pada kolom upload file, klik pilihan Others
pada menu Database, ganti dengan 16S ribosomal RNA sequences.
d. Klik BLAST.
e. Pilih beberapa data sekuen bakteri yang diperlukan get selected sequences.
f. klik Send tofile pilih format FASTA ok.
3. Multiple sequence alignmet dari data BLAST yang diperoleh dibuat dengan menggunakan program ClustalW dan MEGA5 dengan langkah sebagai berikut:
a. Klik kanan pada data hasil BLAST format FASTA open with MEGA5. b. Pada tampilan layar M5: Alignment Explorer, pilih menu Alignment
Align by Clustal W ok, tunggu sampai proses berhenti. c. Pilih menu Data Export alignment MEGA format. 4. Pohon filognetik didesain menggunakan program MEGA5.
a. Buka program MEGA5.
b. Pada toolbar, pilih PhylogenyConstruct/Test Neighbor-Joining Tree. c. Pilih data hasil multiple sequence alignmet dalam format MEGA open
compute.
BAB IV
HASIL DAN PEMBAHASAN
4.1 Isolasi DNA Kromosom Bakteri Xilanolitik Sistem Abdominal Rayap Tanah Isolat B dan Isolat 7
Isolasi DNA kromosom bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7 menggunakan metode Sambrook et al (1989). Hasil isolasi DNA kromosom dicek dengan elektroforesis gel agarosa 1%. Bufer yang digunakan adalah bufer TAE (Tris-asetat EDTA). Hasil elektroforesis isolasi DNA kromosom isolat B dan 7 dapat dilihat pada Gambar 4.1.
Gambar 4.1 Elektroforesis hasil isolasi DNA kromosom (B) isolat B, (7) isolat 7 Hasil isolasi DNA kromosom dari kedua isolat diukur konsentrasi dan kemurniannya menggunakan alat Nanodrop Spectrophotometer ND-1000. Tujuan pengukuran adalah untuk memastikan DNA yang akan digunakan untuk proses amplifikasi 16S rRNA dengan teknik PCR mempunyai konsentrasi dan kemurnian yang sesuai untuk amplifikasi. Hasil pengukuran menunjukkan bahwa isolat B mempunyai konsentrasi sebesar 500,5 ng/µL dengan nilai kemurnian 1,89 dan isolat 7 mempunyai konsentrasi sebesar 183,4 ng/µL dengan nilai kemurnian
DNA kromosom dianggap murni apabila rasio kemurniannya berada diantara nilai 1,8 – 2,0 (Brown, 2001). DNA kromosom yang mempunyai nilai kemurnian < 1,8 menandakan bahwa DNA kromosom tersebut belum murni karena terkontaminasi dengan protein. Adanya kontaminasi protein tersebut disebabkan oleh beberapa faktor, salah satunya adalah human error. Pada tahap penambahan larutan fenol proses isolasi DNA kromosom akan terbentuk tiga lapisan, dimana fasa DNA terdapat pada lapisan atas dan fasa protein terdapat pada lapisan tengah. Ketika melakukan pemisahan fasa DNA, ketidak hati-hatian akan menyebabkan fasa protein ikut terambil bersamaan dengan fasa DNA. Fasa protein yang terbawa akan ikut mengalami presipitasi bersama dengan DNA pada tahap penambahan natrium asetat dan etanol absolut sehingga menyebabkan DNA kromosom tidak murni.
DNA kromosom dapat digunakan untuk proses amplifikasi dengan teknik PCR apabila mempunyai konsentrasi antara 100 – 500 ng/µL (Grunenwald, 2003). Nilai konsentrasi tersebut ditentukan dengan cara mengukur nilai absorbansi DNA pada panjang gelombang 260 nm. Setiap satu satuan A260nm sebanding dengan 50 µg.ml-1 DNA untai ganda. Nilai kemurnian dan konsentrasi yang tidak sesuai akan menyebabkan DNA cetakan mengalami kerukasakan sehingga gen target tidak dapat teramplifikasi (Lindahl and Nyberg, 1972).
4.2 Amplifikasi Gen Penyandi 16S rRNA dengan Teknik PCR
(Bartlett, 2003). Bahan-bahan yang diperlukan dalam proses amplifikasi gen penyandi 16S rRNA dengan teknik PCR adalah DNA cetakan, sepasang primer, Mg2+, dNTPs, bufer PCR, dan Taq DNA polymerase. Untuk amplikasi 16S rRNA bakteri xilanolitik sistem abdominal rayap tanah isolat B dan isolat 7, digunakan pasangan primer B27F dan U1492R dengan urutan basa nukleotida sebagai berikut:
Primer forward B27F : 5’ -AGAGTTTGATCCTGGCTCAG- 3’ Primer reverse U1492R : 5’ -GGTTACCTTGTTACGACTT- 3’.
DNA cetakan bertindak sebagai sumber gen 16S rRNA yang akan diamplifikasi. dNTPs mengandung kelompok-kelompok basa, yaitu dATP, dCTP, dGTP dan dTTP yang berfungsi sebagai sumber basa nukleotida ketika DNA polimerase mensintesis untai DNA target yang baru. Primer merupakan oligonukleotida yang akan mengenali dan menempel pada wilayah DNA yang ditargetkan sehingga memungkinkan terjadinya proses amplifikasi. Primer hanya akan mengamplifikasi wilayah DNA tertentu yang dibatasi oleh primer forward
dan reverse (Brown, 2001). Bufer berfungsi menstabilkan DNA polimerase. Mg2+ berfungsi sebagai kofaktor dari DNA polimerase (Sambrook and Russel, 2001). DNA polimerase berfungsi sebagai enzim yang mensintesis untai baru DNA. Untuk proses PCR, diperlukan DNA polimerase yang termostabil seperti Taq
polimerase yang sudah umum digunakan dalam reaksi PCR.
mengikat untai-untai DNA sehingga DNA mengalami denaturasi dari rantai ganda menjadi rantai tunggal (Brown, 2001). Proses annealing memberikan kesempatan bagi primer untuk menempel pada DNA cetakan yang mempunyai pasangan basa yang komplemen dengan primer. Suhu annealing yang optimal untuk suatu primer didasarkan pada komposisi basa, sekuen nukleotida, serta panjang dan konsentrasi primer. Suhu annealing yang disarankan adalah 50C dibawah Tm primer (Grunenwald, 2003). Annealing harus dilakukan pada suhu yang tepat. Apabila suhu terlalu tinggi, primer tidak akan menempel pada sisi DNA target. Bila suhu terlalu rendah, dapat menyebabkan primer menempel pada daerah yang tidak spesifik dan menghasilkan lebih dari satu gen yang diamplifikasi. Proses extension merupakan proses perpanjangan rantai DNA oleh DNA polimerase. Perpanjangan rantai ini telah dibatasi oleh primer yang menempel pada saat
annealing. DNA polimerase berfungsi sebagai enzim yang bertugas mensintesis untai DNA baru untuk melengkapi untai DNA cetakan dengan menambahkan dNTP dalam arah 5' ke 3'. Umumnya pada proses extension digunakan suhu 720 C, yaitu suhu optimum dari Taq polimerase.
ditemukan pita DNA hasil amplifikasi 16S rRNA. Hal tersebut menunjukkan bahwa suhu 550C terlalu tinggi untuk proses annealing sehingga primer tidak menempel pada sisi DNA target dan tidak terjadi amplifikasi.
Gambar 4.2 Elektroforesis hasil amplifikasi gen penyandi 16S rRNA bakteri xilanolitik sistem abdominal rayap tanah dengan primer B27F dan U1492R suhu 530 C. Lajur 1 adalah pita DNA gen penyandi 16S
rRNA isolat B; 2 adalah GeneRulerTM 1 kb DNA ladder; 3 adalah pita DNA gen penyandi 16S rRNA isolat 7.
basa nukleotida penyandi gen 16S rRNA isolat B dan isolat 7 didapatkan dari hasil sekuensing yang dilakukan di laboratorium Macrogen, Singapura dengan menggunakan primer forward B27F dan primer reverse U1492R.
4.3 Desain Pohon Filogenetik Bakteri Xilanolitik Sistem Abdominal Rayap Tanah Isolat B dan Isolat 7
Pohon filogenetik didesain dengan menggunakan sekuen gen penyandi 16S rRNA milik bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7. Sekuen gen penyandi 16S rRNA merupakan molekul yang sempurna karena memiliki daerah conserved (dipertahankan) dan fungsi yang konstan pada tiap organisme, tersebar secara universal, dan mempunyai urutan sekuen yang terkonservasi dengan baik diantara anggota filogenetik yang luas (Madigan et al., 2000). Selain itu, pada uji konvensional sebelumnya diketahui bahwa kedua isolat ini termasuk dalam jenis prokariot, sehingga untuk mendapatkan sekuen yang universal, digunakan molekul 16S rRNA.
Primer forward B27F dan primer reverse U1492R digunakan untuk proses sekuensing basa nukleotida. Proses sekuensing dengan kedua primer akan menghasilkan urutan basa nukleotida secara menyeluruh. Sekuen lengkap gen penyandi 16S rRNA diperlukan untuk melakukan proses BLAST. Hasil sekuen yang telah lengkap diproses dengan menggunakan program Clone Manager.
1. Gen penyandi 16S rRNA bakteri xilanolitik sistem abdominal rayap tanah isolat B mempunyai sekuen 1460 bp
2. Gen penyandi 16S rRNA bakteri xilanolitik sistem abdominal rayap tanah isolat 7 mempunyai sekuen 1423 bp
Hasil sekuen gen penyandi 16S rRNA dari isolat B dan isolat 7 dilacak homologinya terhadap sekuen 16S rRNA milik bakteri lainnya yang ada di dalam
GenBank melalui program BLAST dengan alamat situs
http://blast.ncbi.nlm.nih.gov/Blast.cgi. sekuen-sekuen 16S rRNA yang didapat dari program BLAST disimpan dalam format FASTA dan diolah kembali menggunakan program ClustalW dan MEGA5 untuk mendapatkan multiple sequence alignmet (MSA). Hasil MSA disimpan dalam format MEGA.
Tabel 4.1 Hasil BLAST bakteri xilanolitik sistem abdominal rayap tanah isolat B
Kode NCBI Nama Spesies Panjang
sekuen (bp)
Persentase Homologi
- Isolat B 1460 100%
NR_041248.1 Bacillus anthracis 1306 94%
NR_043403.1 Bacillus thuringiensis 1486 93%
NR_024697.1 Bacillus weihenstephanensis 1531 92%
NR_036880.1 Bacillus mycoides 1513 92%
NR_025240.1 Bacillus marisflavi 1506 89%
NR_041942.1 Bacillus acidicola 1548 89%
NR_025511.1 Bacillus luciferensis 1502 89%
NR_026144.1 Bacillus halmapalus 1504 89%
NR_025373.1 Bacillus shackletonii 1503 89%
Tabel 4.2 Hasil BLAST bakteri xilanolitik sistem abdominal rayap tanah isolat 7
Kode NCBI Nama Spesies Panjang
sekuen (bp)
Persentase Homologi
- Isolat 7 1423 100%
NR_027549.1 Escherichia fergusonii 1473 99%
NR_024570.1 Escherichia coli 1450 99%
NR_025569.1 Escherichia albertii 1494 99%
NR_026332.1 Shigella dysenteriae 1487 99%
NR_041527.1 Citrobacter youngae 1490 97%
NR_044371.1 Salmonella enterica subsp. houtenae 1437 97% NR_044370.1 Salmonella enterica subsp. indica 1414 97% NR_041696.1 Salmonella enterica subsp. arizonae 1491 97% NR_044373.1 Salmonella enterica subsp. diarizonae 1368 97%
NR_024640.1 Enterobacter asburiae 1422 96%
Kedua tabel hasil BLAST menunjukkan kedekatan antara bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7 dengan bakteri lainnya yang ada di dalam GenBank. Dari Tabel 4.1 diketahui bahwa bakteri xilanolitik sistem abdominal rayap tanah isolat B mempunyai homologi terdekat sebesar 94% dengan Bacillus anthracis (kode NCBI NR_041248.1). Dari Tabel 4.2 diketahui bahwa bakteri xilanolitik sistem abdominal rayap tanah isolat 7 mempunyai homologi terdekat sebesar 99% dengan Escherichia fergusonii (kode NCBI NR_027549.1).
Pada Gambar 4.3 menunjukkan bahwa bakteri xilanolitik sistem abdominal rayap tanah isolat B tidak berada pada cabang maupun node yang sama dengan bakteri-bakteri yang ada di dalam pohon filogenetik. Bakteri xilanolitik isolat B mempunyai cabang tersendiri yang terpisah dari kesepuluh bakteri yang ada dalam pohon filogenetik. Tetapi di dalam tabel homologi BLAST (Tabel 4.1), diketahui bahwa isolat B mempunyai homologi sekuen 16S rRNA paling dekat dengan Bacillus anthracis. Hal tersebut menunjukkan bahwa secara filogenetik, bakteri xilanolitik isolat B tidak mempunyai kesamaan spesies dengan kesepuluh spesies bakteri di dalam pohon filogenetik, tetapi mempunyai kemiripan urutan basa sebesar 94% dengan Bacillus anthracis.
Pada Gambar 4.4 menunjukkan bahwa bakteri xilanolitik sistem abdominal rayap tanah isolat 7 berada pada cabang yang sama dengan Escherichia fergusonii. Dua isolat yang berada pada cabang yang sama menandakan kesamaan spesies (Ludwig and Klenk, 2001). Pada tabel homologi BLAST (Tabel 4.2) juga
ditunjukkan bahwa bakteri xilanolitik sistem abdominal rayap tanah isolat 7 mempunyai homologi sekuen 16S rRNA paling dekat dengan Escherichia fergusonii. Dari hasil pohon filogenetik dan homologi BLAST, diketahui bahwa bakteri xilanolitik isolat 7 mempunyai kesamaan spesies dengan Escherichia fergusonii dan mempunyai homologi sekuen 16S rRNA sebesar 99%.
dan 7 tidak mempunyai kemiripan urutan basa nukleotida maupun kedekatan filogenetik satu sama lain. Kedua isolat mempunyai root (nenek moyang) yang sama tetapi mengalami perubahan yang berbeda satu sama lain ketika berevolusi. Selain itu, kedua isolat bakteri xilanolitik (isolat B dan 7) bukan merupakan spesies bakteri baru karena nilai homologi kedua isolat bakteri xilanolitik berada diantara 94 – 99 %. Bakteri bisa dikatakan spesies baru apabila memiliki kemiripan homologi basa nukleotida < 70% (Wayne, 1987). Hal ini dikemukan oleh Wayne, dimana suatu spesies dapat dikatakan memiliki hubungan dengan salah satu kelompok spesies yang telah ada apabila mempunyai nilai homologi gen lebih besar dari 70% bila dibandingkan dengan seluruh gen yang mengalami hibridisasi DNA-DNA. Nilai total gen yang mengalami hibidrisasi DNA-DNA merupakan kunci utama dari penentuan dan pembatasan hubungan kekerabatan spesies baru tersebut dengan spesies yang telah ada.
Belum ada rekomendasi cara yang pasti untuk menentukan pembatasan homologi genus bakteri maupun untuk menentukan level tertinggi kemiripan suatu genus bakteri. Menurut Stackebrandt and Goebel (1994), suatu isolat bakteri yang baru ditemukan dapat dikatakan berada dalam satu kelompok genus dengan bakteri yang telah ada di data GenBank apabila memiliki homologi sekuen gen 16S rRNA dengan nilai antara 97 – 99 %. Jika nilai homologi sekuen gen 16S
BAB V
KESIMPULAN DAN SARAN 5.1 Kesimpulan
Dari penelitian ini dapat disimpulkan bahwa :
1. Identifikasi molekular berdasarkan 16S rRNA hanya dapat dilakukan pada bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7 yang menghasilkan sekuen gen penyandi 16S rRNA sebesar 1460 bp dan 1423 bp.
2. Hubungan kekerabatan bakteri xilanolitik sistem abdominal rayap tanah isolat B dengan Bacillus anthracis sebesar 94% dan hubungan kekerabatan bakteri xilanolitik sistem abdominal rayap tanah isolat 7 dengan Escherichia fergusonii sebesar 99% pada program pelacakan database Basic Local Alignment Search Tool (BLAST) dapat diketahui berdasarkan desain pohon filogenetik yang telah dibuat.
5.2 Saran
Diharapkan dapat dilakukan penelitian lebih lanjut untuk mengetahui identifikasi dan karakteristik dari bakteri xilanolitik sistem abdominal rayap tanah isolat B dan 7 secara lengkap. Serta dapat dilakukan penelitian lanjutan untuk bakteri xilanolitik sistem abdominal rayap tanah isolat A agar dapat diketahui hubungan kekerabatannya dengan bakteri lain yang ada pada program pelacakan database
DAFTAR PUSTAKA
Bartlett, J.M., Stirling, D., 2003, A short history of the polymerase chain reaction,
Methods Mol Biol., 226: 3-6
Bravman, T., Mechaly, A., Shulami, S., Belakhov, V., Baasov, T., Shoham, G., Shoham, Y., 2001, Glutamic acid 160 is the acid-base catalyst of β-xylosidase from Bacillus stearothermophilus T-6: a family 39 glycoside hydrolase, FEBS Letters, 495 : 115–119
Brown, T.A, 2001, Gene Cloning and DNA Analysis An Introduction, Fourth Edition, Blackwell Publishings, United Kingdom
Coenye T., Vandamme P., 2003, Intragenomic heterogeneity between multiple 16S ribosomal RNA operons in sequenced bacterial genomes, FEMS Microbiol. Lett., 228 (1): 45–49
Collins, T., Gerday, C., and Feller, G., 2005, Xylanases, xylanase families and extremophilic xylanases, FEMS Microbiology Reviews, 29 (2005) : 3-23 Gillis, M., Vandamme, P., Vos, P.D., Swings, J., Kersters, K., 2001, Polyphasic
Taxonomy, In Boone, Castenholz and Garrity (Editors), Bergey’s Manual of Systematic Bacteriology, 2nd Edition, Volume 1, The archaea and the deeply branching and phototrophic bacteria, Springer, New York, 43-48 Grunenwald, H., 2003, Optimization of polymerase chain reactions, In Bartlett
and Stirling (Editors), PCR Protocols, Methods in Molecular Biology., 226: 89-98
Huber, H., Hohn, M.J., Rachel, R., Fuchs, T., Wimmer, V.C., and Stetter, K.O., 2002, A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont, Nature, 417 (6884): 63–7
Lindahl, T., Nyberg, B., 1972, Rate of depurination of native deoxyribonucleic acid, Biochemistry 11, 3610–3618
Ludwig, W., Klenk, H.P., 2001, Overview: A phylogenetic backbone and taxonomic framework for procaryotic systematic, In Boone, Castenholz and Garrity (Editors), Bergey’s Manual of Systematic Bacteriology, 2nd Edition, Volume 1, The archaea and the deeply branching and phototrophic bacteria, Springer, New York, 49–65
Madigan, M.T., Martinko, J.M., and Parker, J., 2000, Biology of Microorganisms
Murray, R.G.E., Holt, John G., 2001, The history of bergey’s manual, In Boone, Castenholz and Garrity (Editors), Bergey’s Manual of Systematic Bacteriology, 2nd Edition, Volume 1, The archaea and the deeply branching and phototrophic bacteria, Springer, New York, 1-13
Pradhika, E.I., Mikrobiologi Dasar, http://ekmon-saurus.blogspot.com, 15 November 2011
Purwadaria, T., Ardiningsih, P., Ketaren, P.P., dan Sinurat, A.P., 2004, Isolasi dan penapisan bakteri xilanolitik mesofil dari rayap, Jurnal Mikrobiology Indonesia, Vol 9, No 2 (2004)
Radek, R., 1999, Flagellates, bacteria, and fungi associated with termites : diversity and function in nutrition-A review, Ecotropica, 5 (2) : 183-196 Ratnadewi, A.A.I., Handayani, W., Hadi, A. F., Sa’diyah, H., Budi, L., 2007,
Isolasi, Pemurnian dan Karakterisasi Enzim Xilanolitik Asal Mikrob dalam Sistem Intestin Rayap untuk Memproduksi Xilooligosakarida sebagai Pereduksi Resiko Kanker, Universitas Jember, Jember
Richana, N., 2002, Produksi dan prospek enzim xilanase dalam pengembangan bioindustri di Indonesia, Buletin AgroBio, 5 (1) : 29-36
Rychlik, W., Spencer, W.J., Rhoads, R.E., 1990, Optimization of the annealing temperature for DNA amplification in vitro, Nucl Acids Res, 18 (21): 6409–6412
Sambrook, J., Fritsch, E.F., Maniatis, T., 1989, Molecular Cloning, A Laboratory Manual, Second Edition, Volume 1, Cold Spring Harbor Laboratory Pres, New York
Sambrook, J., Russell, D. W., 2001, Molecular Cloning: A Laboratory Manual
3rd edition, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, USA
Serianni, A.S., Principles of Biochemistry, http://www.nd.edu, 13 November 2011
Setford, S., 2005, Intisari Ilmu Hewan Merayap, Erlangga, Jakarta
Shimizu, H., Ohkuma, M., Moriya, K., Akiba, T., and Kudo, T., 1998,
Purification and Characterization of Xylanase Produced by Bacillus sp. from Termite Guts. In Ohmiya, K., Hayashi, K., Sakka, K., Kobayashi, Y., Karita, S., and Kimura, T. (Eds.), Genetics, Biochemistry, and Ecology of Cellulose Degradation, Uni Publishers Co. Ltd, Tokyo
Stackebrandt, E., Goebel, B.M., 1994, Taxonomic note: A place for DNA–DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology, 1994, Int. J. Syst. Bacteriol, 44: 846-849
Tarumingkeng, R. C., 2001, Biologi dan Perilaku Rayap, Program Studi Ilmu Hayati Institut Pertanian Bogor, Bogor, diakses online dari url http : //tumoutou.net/biologi_&_perilaku_rayap.htm/, 12 November 2011
Theobald, D., 2004, Phylogenetic Trees Represent Evolutionary Relationships, http://www.talkreason.org, 12 November 2012
Wayne, L.G., Brenner, D.J., Colwell, R.R., Grimont, P.A.D., Kandler, O. , Krichevsky, M.I., Moore, L.H., Moore, W.E.C., Murray, R.G.E., Stackebrandt, E., Starr, M.P., Truper, H.G., 1987, Report of the ad hoc committee on reconciliation of approaches to bacterial systematic, Int. J. Syst. Bacteriol, 37: 463–464
Weidner, S., Arnold, W., Pühler, A., 1996, Diversity of uncultured microorganisms associated with the seagrass Halophila stipulacea
estimated by restriction fragment length polymorphism analysis of PCR-amplified 16S rRNA genes,Appl Env Microbiol, 62 (3): 766–71
Weisburg, W.G., Barns, S.M., Pelletier, D.A., and Lane, D.J., 1991, 16S ribosomal DNA amplification for phylogenetic study, Journal of Bacteriology, 173 (2): 697-703
Woese, C.R., 1967, The Genetic Code: The Molecular Basis for Genetic Expression, Erlangga, Jakarta
Yepyhardi, Mengenal PCR, http://sciencebiotech.net, 13 November 2011 Yuwono, T., 2007, Biologi Molekular, Penerbit Erlangga, Jakarta
LAMPIRAN 1
Perhitungan Pembuatan Media Luria-Bertani Padat dan Cair
1. Media Luria-Bertani padat
50 mL media LB padat: - 1% tripton = 1 𝑔𝑟
100 𝑚𝐿 x 50 mL = 0,5 gr - 1% NaCl = 1 𝑔𝑟
100 𝑚𝐿 x 50 mL = 0,5 gr - 0,5% yeast = 0,5 𝑔𝑟
100 𝑚𝐿 x 50 mL = 0,25 gr - 2% bacto agar = 2 𝑔𝑟
100 𝑚𝐿 x 50 mL = 1 gr 2. Media Luria-Bertani cair
60 mL media LB cair: - 1% tripton = 1 𝑔𝑟
100 𝑚𝐿 x 60 mL = 0,6 gr - 1% NaCl = 1 𝑔𝑟
100 𝑚𝐿 x 60 mL = 0,6 gr - 0,5% yeast = 0,5 𝑔𝑟
LAMPIRAN 2
LAMPIRAN 3
Perhitungan Pembuatan Larutan dan Bahan untuk Elektroforesis 1. Bufer TAE
Stok 50x bufer TAE 200 mL:
- tris-base (H2NC(CH2OH)3) = 200 mL
1000 mL x 242 gr = 48,4 gr
- asam asetat glacial = 200 mL
1000 mL x 57,1 gr = 11,42 gr
- 0,5 M EDTA pH 8 = 200 mL
1000 mL x 20 mL = 20 mL
Larutan kerja = 0,5x TAE dalam 500 mL V1 x M1 = V2 x M2
V1 x 50x = 500 mL x 0,5x
V1 = 5 mL 2. Gel agarosa
1% gel agarosa 40 mL = 1 𝑔𝑟
3. Bufer loading dye
Bufer loading dye 5 mL
- 0,25% Bromophenol Blue (C19H10Br4O5S, BPB) = 0,25 𝑔𝑟
100 𝑚𝐿 x 5 mL = 0,0125 gr
- 40% sukrosa (C12H22O12) = 40 𝑔𝑟
LAMPIRAN 4
Perhitungan Larutan Kerja untuk Reaksi PCR
Stok 0,1 M primer forward B27F dan 0,1 M primer reverse U1492R - Larutan kerja = 10 mM primer forward B27F 20 µL
V1 x M1 = V2 x M2
V1 x 0,1 M = 20 µL x 0,01 M V1 = 2 µL
- Larutan kerja = 0,01 M primer reverse U1492R 20 µL V1 x M1 = V2 x M2
LAMPIRAN 5
Proses Alignment Sekuen Gen Penyandi 16S rRNA Bakteri Xilanolitik
Menggunakan Program Clone Manager
1. Tampilan menu awal program Clone Manager
LAMPIRAN 6
Proses Pelacakan Sekuen Gen Penyandi 16S rRNA Bakteri Xilanolitik
dengan Sekuen Gen Penyandi 16S rRNA Bakteri Lain di Genbank
1. Menu pada program BLAST
LAMPIRAN 7
Proses Multiple Sequence Alignmet dari Data BLAST yang Diperoleh dengan
Menggunakan Program ClustalW dan MEGA5 1. Proses menuju multiple sequence alignmet
3. Proses multiple sequence alignmet
LAMPIRAN 8
Proses Pendesainan Pohon Filognetik Menggunakan Program MEGA5 1. Menu awal program MEGA5
3. Proses pendesainan pohon filogenetik