KERAGAMAN GENETIK AREN
ASAL SULAWESI TENGGARA BERDASARKAN
MARKA RANDOM AMPLIFIED POLYMORPHIC DNA
TESIS
Oleh :
ARIANI SYAHFITRI HARAHAP
127001015/ MAET
PROGRAM STUDI AGROEKOTEKNOLOGI
FAKULTAS PERTANIAN
UNIVERSITAS SUMATERA UTARA
MEDAN
KERAGAMAN GENETIK AREN
ASAL SULAWESI TENGGARA BERDASARKAN
MARKA RANDOM AMPLIFIED POLYMORPHIC DNA
TESIS
Diajukan Sebagai Salah Satu Syarat untuk Memperoleh Gelar Magister dalam Program Magister Agroekoteknologi pada Fakultas Pertanian
Universitas Sumatera Utara
Oleh :
ARIANI SYAHFITRI HARAHAP
127001015/MAET
PROGRAM STUDI AGROEKOTEKNOLOGI
FAKULTAS PERTANIAN
UNIVERSITAS SUMATERA UTARA
MEDAN
Judul Tesis : Keragaman Genetik Aren Asal Sulawesi Tenggara Berdasarkan Marka Random Amplified Polymorphic DNA
Nama : Ariani Syahfitri Harahap
NIM : 127001015
Program Studi : Magister Agroekoteknologi
Disetujui oleh, Komisi Pembimbing
Dr.Ir.Lollie Agustina P.Putri, M.Si
Ketua Anggota
Mohammad Basyuni, S.Hut, M.Si, Ph.D
Diketahui oleh,
Ketua Program Studi Dekan Fakultas Pertanian
Prof. Dr. Ir. Abdul Rauf, MP Prof. Dr. Ir. Darma Bakti, MS
Telah diuji pada
Tanggal : 29 Desember 2014
PANITIA PENGUJI TESIS
Ketua : Dr. Ir. Lollie Agustina P.Putri, M.Si
Anggota : Mohammad Basyuni, S. Hut, M.Si, Ph.D
Dr. Diana Sofia, SP, MP
ABSTRAK
ARIANI SYAHFITRI HARAHAP : Keragaman Genetik Aren Asal Sulawesi Tenggara Berdasarkan Marka Random Amplified Polymorphic DNA, dibimbing
oleh Lollie Agustina P. Putri dan Mohammad Basyuni.
Penelitian ini bertujuan untuk mengetahui keragaman genetik aren asal Sulawesi Tenggara berdasarkan marka Random Amplified Polymorphic DNA. Penelitian ini telah dilakukan di Laboratorium Terpadu Fakultas Kedokteran USU pada April - November 2014. Populasi yang diamati meliputi 27 aksesi tanaman aren yang berasal dari Sulawesi Tenggara terdiri dari tiga lokasi yaitu Konawe Selatan, Kendari dan Konawe. Penelitian ini menggunakan 10 primer yaitu OPN 03, OPC 12, OPD 03, OPD 13, OPD 16, OPH 09, OPB 07, OPH 12, OPH 13 dan SB 19. Perhitungan dan analisis deskriptif dari penelitian ini menggunakan software Darwin 5.05. Hasil penelitian menunjukkan bahwa dari 10 primer acak yang dipergunakan dalam penelitian ini telah diperoleh total 41 pola pita DNA. Ukuran pita DNA yang dihasilkan bervariasi berkisar antara 170 bp sampai 3376 bp. Persentase pita yang polimorfik bervariasi berkisar antara 66.7% sampai 100%. Tingkat keinformatifan dari setiap primer bervariasi dari 0.35 sampai 0.50. Dari 27 aksesi yang dianalisis, 21 aksesi yang bisa diproses oleh software, karena ada beberapa aksesi yang tidak teramplifikasi sehingga tidak memenuhi persentase yang distandarkan. Analisis kluster dari 21 aksesi aren asal Sulawesi Tenggara menunjukkan tingkat keragaman genetik yang tinggi dan memiliki hubungan kekerabatan yang dekat dengan membentuk 3 kluster utama dan 6 subkluster. Sebagian mengelompok berdasarkan populasinya dan
sebagian lainnya mengelompok secara acak. Aksesi yang berasal dari Konawe
Selatan menyebar pada 2 kluster, aksesi yang berasal dari Kendari menyebar pada 3 kluster dan ada yang mengelompok sendiri, sedangkan aksesi yang berasal dari Konawe menyebar pada 2 kluster.
ABSTRACT
ARIANI SYAHFITRI HARAHAP : Genetic Diversity of Sugar Palm from South East Sulawesi Based on Random Amplified Polymorphic DNA Markers, supervised by Lollie Agustina P. Putri and Mohammad Basyuni.
The purpose of the research was to find out genetic diversity of sugar palm from
South East Sulawesi based on Random Amplified Polymorphic DNA Markers. The
research was conducted at Integrated Laboratory of Faculty of Medicine, University of Sumatra Utara, April-November 2014. Ten RAPD primers, OPN 03, OPC 12, OPD 03, OPD 13, OPD 16, OPH 09, OPB 07, OPH 12, OPH 13 and SB 19 was used to study genetic relationship and genetic diversity among 27 accessions of sugar palm from South East Sulawesi consisting of three locations namely South Konawe, Kendari and Konawe. Darwin 5.05 software was used to calculate and describe the results. The results showed that the amplification of 27 accessions of sugar palm using 10 random primers obtained a total of 41 DNA band numbers. The size of DNA bands were varied ranging from 170 bp to 3376 bp. The percentage of polymorphic bands were also varied between 66.7% to 100%. Level of polymorphic information content of each primer showed variation from 0.35 to 0.50. From Twenty one of 27 accessions were processed by software due to miss-amplified as well as standard of percentage was excluded. Cluster analysis from 21 accessions of sugar palm from South East Sulawesi showed high genetic diversity and had near genetic relationship among them to form 3 main clusters and 6 subclusters. Some of them were grouped based on their population and some others were randomly. Accessions from South East Konawe spread into 2 clusters, accessions from Kendari spread into 3 clusters and were grouped itself, while accessions from Konawe extended in 2 clusters.
RIWAYAT HIDUP
Penulis dilahirkan di Sosopan pada tanggal 28 Juni 1987 dari ayah Mester
Harahap dan ibu Ratna Sari Daulay. Penulis merupakan putri pertama dari empat
bersaudara.
Tahun 2005 penulis lulus dari SMA Negeri 18 Medan dan pada tahun 2006 masuk
ke Fakultas Pertanian USU melalui jalur ujian tertulis Seleksi Penerimaan
Mahasiswa Baru. Penulis memilih Program Studi Pemuliaan Tanaman,
Departemen Budidaya Pertanian Fakultas Pertanian Universitas Sumatera Utara.
Tahun 2010 penulis menyelesaikan pendidikan Strata-1 pada Program Studi
Pemuliaan Tanaman, Departemen Budidaya Pertanian Fakultas Pertanian
Universitas Sumatera Utara. Penulis sempat bekerja sebagai Asisten Pribadi
Dosen Fakultas Ekonomi Universitas Sumatera Utara dan membantu Program
Studi PWD dan PW Sekolah Pascasarjana Universitas Sumatera Utara dalam hal
KATA PENGANTAR
Puji dan syukur penulis ucapkan kepada Allah SWT atas segala rahmat
dan karunia-Nya sehingga penulis dapat menyelesaikan tesis yang berjudul
”Keragaman Genetik Aren Asal Sulawesi Tenggara Berdasarkan Marka Random
Amplified Polymorphic DNA”.
Pada kesempatan ini penulis ingin mengucapkan terima kasih
sebesar-besarnya kepada kedua orang tua penulis yang telah membesarkan dan mendidik
penulis selama ini. Penulis menyampaikan ucapan terima kasih kepada Ibu
Dr.Ir.Lollie Agustina P.Putri, M.Si (Ketua) dan Bapak Mohammad Basyuni,
S.Hut, M.Si, Ph.D (Anggota) yang telah membimbing dan memberikan berbagai
masukan berharga kepada penulis dari mulai menetapkan judul, melakukan
penelitian sampai pada ujian akhir serta Ibu Dr. Diana Sofia, SP, M.Si (Penguji),
Bapak Prof. Dr. Drs. Dwi Suryanto, M.Si (Penguji) dan Bapak Luthfi A.M.
Siregar, SP, M.Agr.Sc, Ph.D (Penguji) yang memberikan berbagai masukan
berharga kepada penulis.
Disamping itu, penulis juga mengucapkan terima kasih kepada Dinas
Peternakan Provinsi Sumatera Utara, Bapak Ahmad Suryadin, Bapak Efendi
Manik, Ryan Iskandar SP, Srinidyanti Misrun SP, Giovanna Bastini Siagian SP,
Mariana Romina Manik, Ahmad Kamal S.Pt, Marianus Sitepu SP, semua staf
pengajar dan pegawai di Program Studi Agroekoteknologi. Semoga tesis ini
bermanfaat.
Medan, Desember 2014
DAFTAR ISI
ABSTRAK ... i
ABSTRACT ... ii
DAFTAR RIWAYAT HIDUP ... iii
KATA PENGANTAR ... iv
Polymerase Chain Reaction (PCR) ... 13
RAPD (Random Amplified Polymorphic DNA) ... 14
BAHAN DAN METODE Tempat dan Waktu Penelitian ... 17
Bahan dan Alat ... 17
Metode Isolasi DNA Pohon Aren ... 18
Uji Kualitas DNA ... 20
Uji Kuantitas DNA ... 20
RAPD DNA Pohon Aren ... 21
Elektroforesis Hasil Amplifikasi ... 21
Analisis Data ... 22
HASIL DAN PEMBAHASAN Analisis Profil Pita Hasil Isolasi DNA Tanaman Aren ... 23
Analisis Profil Pita Hasil Amplifikasi PCR Tanaman Aren ... 27
Analisis Kluster Tanaman Aren ... 40
KESIMPULAN DAN SARAN Kesimpulan ... 48
Saran ... 48
DAFTAR PUSTAKA ... 49
DAFTAR TABEL
No. Hal
1. Daftar koleksi aren yang digunakan untuk analisis RAPD ... 17
2. Urutan basa primer dari sepuluh primer yang digunakan ... 18
3. Hasil amplifikasi sepuluh primer yang digunakan ... 28
4. Polymorphic Information Content (PIC) pada sepuluh primer ... 30
5. Perbedaan persentase pita polimorfik (%) antara aren asal Sulawesi Tenggara dan Sumatera Utara dengan primer yang sama ... 40
DAFTAR GAMBAR
No. Hal
1. Bagian Tanaman Aren... 7 2. Elektroforegram uji kualitatif 27 DNA tanaman aren... 23 3. Elektroforegram amplifikasi 27 DNA aren dengan primer OPD 03.... 31
4. Elektroforegram amplifikasi 27 DNA aren dengan primer OPH 12.... 32 5. Elektroforegram amplifikasi 27 DNA aren dengan primer OPB 07 .... 32 6. Elektroforegram amplifikasi 27 DNA aren dengan primer OPD 16.... 33 7. Elektroforegram amplifikasi 27 DNA aren dengan primer OPN 03.... 34 8. Elektroforegram amplifikasi 27 DNA aren dengan primer OPC 12 .... 34 9. Elektroforegram amplifikasi 27 DNA aren dengan primer SB 19 ... 35 10.Elektroforegram amplifikasi 27 DNA aren dengan primer OPD 13.... 35 11.Elektroforegram amplifikasi 27 DNA aren dengan primer OPH 09.... 36 12.Elektroforegram amplifikasi 27 DNA aren dengan primer OPH 13.... 37 13.Dendogram 21 aksesi tanaman aren asal Sulawesi Tenggara yang
dianalisis berdasarkan matrix dissimilarity simple matching ... 42
14.Profil radial Neighbour-Joining Tree (NJtree) dari 21 aksesi aren di
Sulawesi Tenggara yang dianalisis berdasarkan matrix dissimilarity simple matching ... 43
15.Analisis faktor Principal Coordinates Analysis (PCoA) aksis I (horizontal)
DAFTAR LAMPIRAN
No. Hal
1. Alur penelitian ... 53
2. Koleksi 27 aksesi tanaman aren di Sulawesi Tenggara ... 54
3. Peta lokasi sampel tanaman aren di Sulawesi Tenggara ... 55
4. Pembuatan larutan stok dan bufer ... 56
5. Proses isolasi DNA ... 58
6. Proses uji kualitatif ... 59
7. Proses PCR-RAPD ... 60
8. Proses elektroforesis hasil PCR-RAPD ... 61
9. Hasil uji kuantitatif 27 aksesi DNA tanaman aren ... 62
ABSTRAK
ARIANI SYAHFITRI HARAHAP : Keragaman Genetik Aren Asal Sulawesi Tenggara Berdasarkan Marka Random Amplified Polymorphic DNA, dibimbing
oleh Lollie Agustina P. Putri dan Mohammad Basyuni.
Penelitian ini bertujuan untuk mengetahui keragaman genetik aren asal Sulawesi Tenggara berdasarkan marka Random Amplified Polymorphic DNA. Penelitian ini telah dilakukan di Laboratorium Terpadu Fakultas Kedokteran USU pada April - November 2014. Populasi yang diamati meliputi 27 aksesi tanaman aren yang berasal dari Sulawesi Tenggara terdiri dari tiga lokasi yaitu Konawe Selatan, Kendari dan Konawe. Penelitian ini menggunakan 10 primer yaitu OPN 03, OPC 12, OPD 03, OPD 13, OPD 16, OPH 09, OPB 07, OPH 12, OPH 13 dan SB 19. Perhitungan dan analisis deskriptif dari penelitian ini menggunakan software Darwin 5.05. Hasil penelitian menunjukkan bahwa dari 10 primer acak yang dipergunakan dalam penelitian ini telah diperoleh total 41 pola pita DNA. Ukuran pita DNA yang dihasilkan bervariasi berkisar antara 170 bp sampai 3376 bp. Persentase pita yang polimorfik bervariasi berkisar antara 66.7% sampai 100%. Tingkat keinformatifan dari setiap primer bervariasi dari 0.35 sampai 0.50. Dari 27 aksesi yang dianalisis, 21 aksesi yang bisa diproses oleh software, karena ada beberapa aksesi yang tidak teramplifikasi sehingga tidak memenuhi persentase yang distandarkan. Analisis kluster dari 21 aksesi aren asal Sulawesi Tenggara menunjukkan tingkat keragaman genetik yang tinggi dan memiliki hubungan kekerabatan yang dekat dengan membentuk 3 kluster utama dan 6 subkluster. Sebagian mengelompok berdasarkan populasinya dan
sebagian lainnya mengelompok secara acak. Aksesi yang berasal dari Konawe
Selatan menyebar pada 2 kluster, aksesi yang berasal dari Kendari menyebar pada 3 kluster dan ada yang mengelompok sendiri, sedangkan aksesi yang berasal dari Konawe menyebar pada 2 kluster.
ABSTRACT
ARIANI SYAHFITRI HARAHAP : Genetic Diversity of Sugar Palm from South East Sulawesi Based on Random Amplified Polymorphic DNA Markers, supervised by Lollie Agustina P. Putri and Mohammad Basyuni.
The purpose of the research was to find out genetic diversity of sugar palm from
South East Sulawesi based on Random Amplified Polymorphic DNA Markers. The
research was conducted at Integrated Laboratory of Faculty of Medicine, University of Sumatra Utara, April-November 2014. Ten RAPD primers, OPN 03, OPC 12, OPD 03, OPD 13, OPD 16, OPH 09, OPB 07, OPH 12, OPH 13 and SB 19 was used to study genetic relationship and genetic diversity among 27 accessions of sugar palm from South East Sulawesi consisting of three locations namely South Konawe, Kendari and Konawe. Darwin 5.05 software was used to calculate and describe the results. The results showed that the amplification of 27 accessions of sugar palm using 10 random primers obtained a total of 41 DNA band numbers. The size of DNA bands were varied ranging from 170 bp to 3376 bp. The percentage of polymorphic bands were also varied between 66.7% to 100%. Level of polymorphic information content of each primer showed variation from 0.35 to 0.50. From Twenty one of 27 accessions were processed by software due to miss-amplified as well as standard of percentage was excluded. Cluster analysis from 21 accessions of sugar palm from South East Sulawesi showed high genetic diversity and had near genetic relationship among them to form 3 main clusters and 6 subclusters. Some of them were grouped based on their population and some others were randomly. Accessions from South East Konawe spread into 2 clusters, accessions from Kendari spread into 3 clusters and were grouped itself, while accessions from Konawe extended in 2 clusters.
PENDAHULUAN
Latar Belakang
Tanaman aren sebelumnya dikenal dengan nama botani Arenga
saccharifera Labill, tetapi sekarang sudah direvisi dengan nama jenis Arenga
pinnata Merr. Tanaman aren bisa dijumpai dari pantai barat India sampai ke
sebelah selatan Cina dan juga kepulauan Guam. Habitat aren juga banyak terdapat
di Philipina, Malaysia, dataran Assam di India, Laos, Kamboja, Vietnam, Birma
(Myanmar), Srilanka dan Thailand. Namun, tanaman yang termasuk dalam
keluarga Arecaceae ini berasal dari Indonesia (Lempang, 2012).
Aren (A. pinnata) termasuk salah satu jenis tanaman palma, yang tersebar
hampir di seluruh wilayah Indonesia, terutama di 14 provinsi, yaitu Papua,
Maluku, Maluku Utara, Sumatera Utara, Sumatera Barat, Jawa Barat, Jawa
Tengah, Banten, Sulawesi Utara, Sulawesi Selatan, Sulawesi Tenggara, Bengkulu,
Kalimantan Selatan dan Aceh (Permentan, 2014).
Data dari Ditjenbun (2010), pada tahun 2010 luas tanaman aren sekitar
59.388 ha dengan produksi sekitar 33.181 ton gula aren. Tanaman Aren
(A. pinnata) mempunyai banyak manfaat, diantaranya sebagai penghasil nira
(bahan utama gula aren, minuman, cuka, dan alkohol), sumber energi terbarukan
(bioetanol), sumber karbohidrat (tepung), bahan campuran minuman
(kolang-kaling), bahan bangunan (batang) dan sebagai tanaman konservasi dan reklamasi
untuk lahan-lahan kritis. Pada masa sekarang masyarakat hanya memanfaatkan
tanaman aren yang berasal dari alam, sehingga bukan tidak mungkin suatu saat
juga belum dilakukan sehingga populasi jenis palm ini kurang diketahui
(Lempang, 2012).
Informasi keragaman genetik sangat diperlukan untuk mendukung
kegiatan konservasi dan pemuliaan tanaman. Untuk kegiatan konservasi, besarnya
keragaman genetik mencerminkan sumber genetik yang diperlukan untuk adaptasi
ekologi dalam jangka waktu pendek dan evolusi dalam jangka panjang, sedangkan
untuk pemuliaan, keragaman genetik yang luas diperlukan dalam kegiatan seleksi.
Program pemuliaan jangka panjang yang memanfaatkan plasma nutfah untuk
memperbaiki sifat-sifat agronomi dari aksesi/jenis terpilih harus didasarkan pada
perkiraan determinasi genetik yang lebih akurat, sehingga penentuan individu
tanaman sebagai bahan dalam perbaikan genetik dapat dilakukan dengan tepat
(Rahayu dan Handayani, 2010).
Keanekaragaman genetik dapat terjadi karena adanya perubahan
nukleotida penyusun DNA. Perubahan ini mungkin dapat mempengaruhi fenotipe
suatu organisme yang dapat dilihat secara langsung atau mempengaruhi reaksi
individu terhadap lingkungan tertentu. Secara umum keanekaragaman genetik dari
suatu populasi dapat terjadi karena adanya mutasi, rekombinasi, atau migrasi gen
dari satu tempat ke tempat lain (Suryanto, 2003).
Penanda molekuler banyak digunakan dalam analisis keragaman genetik
tumbuhan, salah satunya adalah Random Amplified Polymorphic DNA (RAPD).
Teknik ini digunakan untuk mengidentifikasi genotipe tumbuhan, karena memiliki
kelebihan dalam pelaksanaan dan analisisnya. Dibandingkan dengan penanda
DNA yang lain, seperti Restriction Fragment Length Polymorphisms (RFLP) dan
cepat memberikan hasil, menghasilkan polimorfisme pita DNA dalam jumlah
banyak, tidak memerlukan pengetahuan tentang latar belakang genom yang
dianalisis dan mudah memperoleh primer acak yang diperlukan untuk
menganalisis genom semua jenis organisme. Walaupun metode ini kurang
sempurna dan memiliki kelemahan dalam konsistensi produk amplifikasi
(Jones et al., 1997), tetapi kelemahan ini dapat diatasi dengan mengoptimalkan
ekstraksi, kondisi PCR dan pemilihan primer yang tepat.
Analisis RAPD menggunakan primer sepuluh basa sering digunakan untuk
studi kekerabatan dan identifikasi varietas (CIMMYT, 1998). Liu dan Furnier
(1993) melaporkan penggunaan RAPD selalu memperlihatkan keragaman lebih
tinggi dari pada alozim dan RFLP, sehingga sangat mendukung upaya analisis
keragaman genetik jika latar belakang genomnya belum diketahui. Teknik RAPD
telah digunakan untuk meningkatkan efisiensi seleksi dini pada tanaman tahunan
(Grattapaglia et al., 1992).
Penelitian tentang kekerabatan pada tanaman aren telah dilakukan oleh
Harahap (2013) menggunakan penanda RAPD bertujuan untuk mengetahui
keragaman genetik tanaman aren populasi alam di daerah Tapanuli Selatan
berdasarkan marka RAPD.
Perumusan Masalah
Informasi mengenai keragaman genetik tanaman sangat diperlukan untuk
mendukung program pemuliaan dan konservasi. Perbaikan varietas tanaman dapat
dilaksanakan jika ada sumber plasma nutfah yang memadai. Selain itu, masih
tanaman aren dengan menggunakan penanda RAPD dan penelitian genetik yang
dapat mendukung upaya konservasinya.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengetahui keragaman genetik aren asal
Sulawesi Tenggara berdasarkan marka RAPD.
Hipotesis Penelitian
Hipotesis penelitian ini adalah adanya keragaman genetik aren asal
Sulawesi Tenggara berdasarkan marka RAPD.
Manfaat Penelitian
Manfaat dari penelitian ini adalah diharapkan dapat memberikan manfaat
dalam informasi mengenai keragaman genetik, inventarisasi plasma nutfah, dan
TINJAUAN PUSTAKA
Deskripsi Tanaman Aren
Menurut Steenis (1950), klasifikasi tanaman aren sebagai berikut ini:
Kingdom : Plantae
Filum : Spermatophyta
Sub Filum : Angiospermae
Kelas : Monocotyledonae
Ordo : Spadicitlorae
Famili : Palmae
Genus : Arenga
Spesies : Arenga pinnataMerr.
Aren memiliki akar yang dapat tumbuh dalam sampai 10 m dengan akar
serabut berwarna putih kekuningan dan mengandung saponin, flavonoida dan
polifenol. Perakaran pohon aren meyebar dan cukup dalam, sehingga tanaman ini
dapat diandalkan sebagai vegetasi pencegah erosi, terutama untuk daerah yang
tanahnya mempunyai kemiringan lebih dari 20% (Harahap, 2013).
Diameter batang sampai dengan 70 cm dengan tinggi mencapai 5-15 m,
kadang-kadang tinggi mencapai 20 m (Lasut, 2012). Waktu pohon masih muda
batang aren belum kelihatan karena tertutup oleh pangkal pelepah daun, ketika
daun paling bawahnya sudah gugur, batangnya mulai kelihatan. Permukaan
batang ditutupi oleh serat ijuk berwarna hitam yang berasal dari dasar tangkai
Daun majemuk dengan panjang sampai dengan 5.5 m; anak daun panjang
130-150 cm dengan lebar 5-8 cm; bagian bawah pangkal pelepah daun ditumbuhi
ijuk , berwarna hitam. Perbungaan berupa tandan bunga bercabang, menggantung
dengan panjang mencapai 60 cm atau lebih. Tandan bunga tumbuh pada daerah
bekas pelepah daun (Lasut, 2012). Pohon aren mempunyai tajuk (kumpulan daun)
yang rimbun. Daun aren muda selalu berdiri tegak di pucuk batang, daun muda
yang masih tergulung lunak seperti kertas. Pelepah daun melebar di bagian
pangkal dan menyempit ke arah pucuk. Susunan anak daun pada pelepah seperti
duri-duri sirip ikan, sehingga daun aren disebut bersirip. Oleh karena pada
ujungnya tidak berpasangan lagi daun aren disebut bersirip ganjil. Pada bagian
pangkal pelepah daun diselimuti oleh ijuk yang berwarna hitam kelam dan
dibagian atasnya berkumpul suatu massa yang mirip kapas yang berwarna cokelat,
sangat halus dan mudah terbakar. Massa yang menempel pada pangkal pelepah
daun aren tersebut dikenal dengan nama kawul (Jawa barat), baruk (Tana Toraja)
dan beru (Bugis) (Lempang, 1996).
Perbungaan berupa tandan bunga bercabang, menggantung dengan
panjang mencapai 60 cm atau lebih. Tandan bunga tumbuh pada daerah bekas
pelepah daun. Perbungaan dimulai dari pucuk, selanjutnya secara berturut-turut
menyusul pada bagian bawah. Biasanya 2-5 bunga pertama betina, sedangkan
rangkaian bunga pada bagian bawah adalah bunga jantan. Bunga jantan berwarna
kecoklatan, berbentuk bulat telur memanjang, daun bunga tiga, dan kelopak bunga
tiga helai, bunga betina warna kehijauan dengan mahkota bunga segitiga
beruas-ruas, bakal buah memiliki ruang tiga dan putik tiga. Tandan bunga betina aren
tumbuh dan membentuk buah (Lasut, 2012). Aren mulai berbunga, kira-kira
setelah tanaman berumur 7 – 10 tahun. Tangkai malai bunga dapat disadap setiap
hari selama 2-3 bulan menghasilkan 10-30 liter nira tiap hari (Haris, 1994).
Buah aren terbentuk akibat dari penyerbukan secara alami, pelaksanaannya
dengan bantuan angin. Buah aren berbentuk lonjong (dengan panjang sampai 5
cm dengan diameter sampai 3 cm) dan beruang tiga. Biji pada buah aren muda
mengandung kristal Ca-oksalat, yang bila menyentuh kulit dapat menyebabkan
iritasi dan menimbulkan rasa gatal (Lasut, 2012).
Gambar 1. Bagian Tanaman Aren
(a) Pohon Aren, (b) Daun Aren, (c) Buah Aren, (d) Biji Aren (Lasut, 2012)
Buah dan biji aren berkembang sangat lambat, membutuhkan tiga (3)
tahun untuk matang, dan biji masak fisiologis pada saat umur 36 bulan setelah
periode antesis dimana bunga telah berkembang sempurna dan fungsional. Berat
embrio maksimum dicapai pada umur 30 bulan. Selama proses pematangan,
penebalan dinding sel endosperm terjadi secara progresif sampai semua rongga
endosperm terisi pada 36 bulan setelah antesis, karena itulah struktur endosperm
a
b
tanaman aren sangat keras, hal ini menjadi karakteristik keluarga palem-paleman
(Haris, 1994).
Keragaman Genetik
Keragaman tingkat genetik merupakan tingkat keragaman yang paling
rendah dalam organisasi biologi. Keragaman genetik sangat penting bagi tanaman
untuk beradaptasi terhadap perubahan lingkungan yang terjadi disekitarnya.
Informasi keragaman genetik tanaman pada tingkat, individu, spesies maupun
populasi perlu diketahui, sebagai dasar pertimbangan dalam menyusun strategi
konservasi, pemuliaan, pengelolaan dan pemanfaatan sumberdaya genetik
tanaman secara berkelanjutan. Penilaian keragaman genetik tanaman dapat
dilakukan dengan menggunakan penanda morfologi, biokimia dan molekuler
DNA (Zulfahmi, 2013).
Keragaman genetik yang tinggi merupakan salah satu faktor penting untuk
merakit varietas unggul baru. Peningkatan keragaman genetik dapat dilakukan
dengan memanfaatkan plasma nutfah yang tersedia di alam dan dapat pula dengan
melakukan persilangan. Sifat-sifat tertentu sering tidak ditemukan pada sumber
gen yang ada sehingga teknologi lainnya perlu diterapkan (Hutami et al, 2005).
Keragaman yang tinggi didalam populasi memberikan dasar yang luas
untuk program pengembangan. Dasar untuk seleksi dalam proses ini sama seperti
konservasi ex-situ tetapi lebih difokuskan pada tingkat tertinggi dari
heterozigositas. Untuk menghasilkan program seleksi yang efektif, seleksi dengan
individu yang jumlahnya lebih banyak dilakukan di dalam populasi sehingga
Informasi keragaman genetik juga diperlukan untuk mendukung kegiatan
konservasi. Besarnya keragaman genetik mencerminkan sumber genetik yang
diperlukan untuk adaptasi ekologi dalam jangka waktu pendek dan evolusi dalam
jangka panjang. Menurut Nuryani et al (2002) pengujian secara molekuler akan
mampu mengungkapkan tidak saja keragaman genetik juga mengungkap tingkat
kekerabatan tanaman.
Isolasi DNA
Isolasi DNA tanaman diawali dengan penghancuran dinding sel tanaman.
Kegagalan dalam memecah dinding sel akan mempengaruhi hasil akhir isolasi.
Proses inilah yang membuat isolasi DNA tanaman lebih sulit dibandingkan isolasi
DNA bakteri karena tanaman memiliki dinding sel yang kuat dan tebal.
Penghancuran dinding sel dapat dilakukan secara kimiawi dan mekanik. Secara
mekanik dapat dilakukan dengan cara penggerusan menggunakan mortar dingin
dan bantuan nitrogen cair. Penggunaan nitrogen cair membuat daun menjadi
kering dan mudah untuk dihancurkan. Nitrogen cair juga menjaga suhu tetap
dingin sehingga DNA tidak rusak. Nitrogen cair memiliki suhu minus 196°C.
Selain itu, dengan menggunakan nitrogen cair maka hasil penggerusan berupa
serbuk sehingga mengurangi peluang berkurangnya sampel dibandingkan bila
hasilnya berupa ekstrak cair yang mudah lengket pada mortar. Selain nitrogen
cair, penggerusan sampel daun ditambahkan juga Polivynilpolipirolidon (PVPP).
PVPP berfungsi sebagai antioksidan untuk mencegah terbentuknya warna coklat
(browning) pada DNA. PVPP menghambat enzim polifenol oksidase yang dapat
mendegradasi rantai DNA dan menyebabkan teroksidasinya senyawa fenol
Bahan lain yang yang digunakan selama isolasi antara lain larutan bufer,
larutan Tris-HCl, larutan ethylenediamine tetraacetic acid (EDTA), larutan cetyl
trimethyl ammonium bromide (CTAB) 10%, larutan kloroform:isoamilalkohol
(24:1), larutan NaCl, isopropanol, alkohol absolut, alkohol 70%, dan bufer TE
(Tris-HCl:EDTA). Larutan bufer adalah suatu sistem dalam larutan yang terdiri
dari campuran basa lemah dan asam konjugatnya atau asam lemah dan basa
konjugatnya, yang berfungsi untuk mempertahankan perubahan pH larutan
walaupun ditambahkan sedikit asam kuat atau basa kuat. Larutan bufer yang
digunakan pada isolasi DNA terdiri atas beberapa senyawa yang memiliki fungsi
berbeda. Larutan Tris-HCl digunakan untuk memberikan kondisi pH yang
optimum dan menjaga kestabilan pH. EDTA digunakan untuk melemahkan
kekuatan dinding sel, karena dapat mengkelat ion magnesium yang merupakan
kofaktor enzim nuklease (Herison et al., 2003). Larutan CTAB 10% dalam bufer
ekstraksi berfungsi untuk mengurangi senyawa polisakarida dan menghilangkan
polifenol yang juga merupakan kontaminan saat isolasi DNA. Kontaminan
tersebut akan mengendap bersama CTAB sedangkan DNA tidak mengendap.
Larutan kloroform:isoamilalkohol (24:1) untuk menghilangkan lemak,
protein, polisakarida, dan pengotor lainnya karena keberadaan senyawa-senyawa
tersebut dapat mempengaruhi kualitas dan kuantitas DNA yang diisolasi. Larutan
tersebut juga berfungsi memisahkan DNA dari membran sel yang memiliki bobot
molekul lebih besar. Kloroform:isoamilalkohol yang memiliki densitas paling
tinggi akan berada di dasar tabung sentrifus. Larutan yang berada di bagian tengah
merupakan protein yang telah larut dalam kloroform:isoamilalkohol. Supernatan
Selain itu, penambahan isoamilalkohol mengurangi busa yang muncul saat
ekstraksi DNA.
Penggunaan larutan NaCl pada konsentrasi tinggi untuk mengatasi
keberadaan polisakarida pada konsentrasi yang tinggi (Khanuja et al., 1999).
Penambahan isopropanol bertujuan mengendapkan DNA. Penambahan alkohol
absolut bertujuan memekatkan larutan DNA dan menghilangkan residu kloroform
yang digunakan pada proses deproteinase (Ausubel et al., 1990). DNA yang
diperoleh dicuci dengan alkohol 70% untuk menghilangkan sisa-sisa pengotor.
DNA yang diperoleh dilarutkan dengan bufer TE sehingga dapat disimpan dan
digunakan untuk analisis lebih lanjut.
Penanda Molekuler
Penanda genetik, biasa juga disebut dengan 'marka', merupakan ekspresi
pada individu yang terlihat oleh mata atau terdeteksi dengan alat tertentu, yang
menunjukkan dengan pasti genotipe suatu individu. Beberapa penanda genetik
sangat terpercaya karena bersifat lembam, tidak mudah berubah karena pengaruh
lingkungan. Penanda genetik sangat penting dalam penyelidikan philogeni suatu
organisme (Tao et al., 2009).
Penanda molekuler atau penanda DNA adalah suatu sekuen pendek DNA
yang menunjukkan adanya polimorfisme antara individu berbeda dalam satu
spesies. Penanda molekuler mempunyai tingkat polimorfisme yang sangat tinggi,
jumlahnya tidak terbatas, tidak dipengaruhi oleh lingkungan, dan tingkat
heritabilitasnya hampir 100%. Suatu penanda akan efektif jika dapat membedakan
antara dua tetua yang berbeda genotipenya dan dapat dideteksi dengan mudah
Penanda genetik hanya berguna apabila polimorfik dan terpaut dengan
sifat yang akan diamati atau dengan penanda genetik lain. Syarat polimorfik
diperlukan karena penanda genetik harus bisa membedakan individu-individu
dalam populasi yang diteliti. Suatu penanda genetik paling tidak harus bisa
mengelompokkan individu dalam dua kelompok. Syarat terpaut dengan penanda,
gen atau sifat lain diperlukan karena fungsi penanda genetik adalah sebagai tanda
pengenal yang harus melekat pada sifat yang diteliti (Sharma et al., 2008).
Penanda molekuler (molecular marker) menawarkan sejumlah keuntungan
dibandingkan dengan penanda fenotipik konvensional, yaitu:
1. Penanda molekuler bersifat stabil dan dapat terdeteksi pada semua jaringan,
tanpa terpengaruh oleh pertumbuhan, differensiasi, perkembangan, atau status
pertahanan sel-sel tanaman.
2. Penanda molekuler tidak dipengaruhi oleh faktor lingkungan, dan
3. Umumnya tidak memiliki efek pleiotropi atau epistasis
(Nasir, 2002).
Teknik molekuler telah memberikan peluang pengembangan dan
identifikasi peta genetik spesies tanaman. Pendekatan genetika molekuler
menggunakan penciri DNA telah berhasil membentuk penanda molekuler yang
mampu mendeteksi gen dan sifat-sifat tertentu, evaluasi keragaman, kekerabatan,
serta adanya evolusi pada tingkat genetik (Maftuchah dan Zainuddin, 2013).
Teknologi penanda molekuler pada tanaman berkembang sejalan dengan
semakin banyaknya pilihan penanda molekuler. Penanda pertama berdasarkan
pada hibridisasi DNA seperti RFLP. Penanda kedua berdasarkan pada reaksi
sekuen-sekuen nukleotida sebagai primer, seperti RAPD dan AFLP. Penanda
ketiga berdasarkan pada PCR dengan menggunakan primer yang menggabungkan
sekuen komplementer spesifik dalam DNA target, seperti Sequence Tagged Sites
(STS), Sequence Characterized Amplified Regions (SCARs), SSRs atau
mikrosatelit, dan Single NucleotidePolymorphisms (SNPs) (Azrai, 2005).
Polymerase Chain Reaction (PCR)
PCR merupakan fasilitas dalam mempelajari genetik tanaman maupun
hewan. Sidik DNA, analisis forensik, pemetaan genetik dan filogenetik dapat
dipelajari dengan PCR. Beberapa teknik analisis keanekaragaman genetik,
membutuhkan amplifikasi daerah genom tertentu dari suatu organisme (Demeke
dan Adams. 1994).
Kemajuan teknologi telah memungkinkan para ilmuan untuk meniru
urutan nukleotida suatu gen dengan cara melakukan amplifikasi DNA dengan
teknik reaksi berantai polimerase (PCR). Amplifikasi DNA dilakukan secara in
vitro (di dalam tabung) dengan menggunakan: (1) enzim DNA polymerase; (2)
dNTP (dinukleotida triphosphat; (3) oligonukleotida primer; dan (4) molekul
DNA cetakan (DNA template)
Primer biasanya terdiri dari 10-20 nukleotida dan dirancang berdasarkan
daerah konservatif dalam genom tersebut. Makin panjang primer, makin spesifik
daerah yang diamplifikasi. Jika suatu kelompok organisme memang berkerabat
dekat, maka primer dapat digunakan untuk mengamplifikasi daerah tertentu yang
sama dalam genom kelompok tersebut. Beberapa faktor seperti konsentrasi DNA,
hibridisasi primer harus dikontrol dengan hati-hati agar dapat diperoleh pita-pita
DNA yang utuh dan baik (Suryanto, 2003).
Proses PCR meliputi sejumlah siklus untuk amplifikasi suatu sikuen DNA
tertentu. Setiap siklus amplifikasi terdiri atas tiga tahap berurutan.
1. Denaturasi. Tahap pertama pada system amplifikasi PCR adalah denaturasi
DNA sampel dengan menaikkan suhu dalam tabung reaksi sampai 950
2. Penempelan primer. Suhu campuran diturunkan antara 37
C.
tabung reaksi ini berisi DNA target, dua primer oligonukleotida dalam jumlah
berlebihan, polymerase Taq yang tahan panas, keempat deoksiribonukleotida
dan bufer yang mengandung Mg.
0 C - 600
3. Polimerasi. Pada tahap ini, suhu dinaikkan sampai 72
C. pada
tahap ini, primer menempel pada sikuen komplementernya pada DNA target. 0
(Sudjadi, 2008).
C, yang merupakan
suhu optimum polymerase Taq. Sintesis DNA diinisiasi pada ujung
3’-hidroksil pada setiap primer.
Random Amplified Polymorphic DNA (RAPD)
Teknik PCR-RAPD merupakan salah satu teknik molekuler untuk
mempelajari keanekaragaman genetika. Dasar analisis RAPD adalah
menggunakan mesin PCR yang mampu mengamplifikasi sekuen DNA secara
acak. Teknik ini melibatkan penempelan primer yang dirancang secara khusus
sepuluh oligonukleotida pada cetakan DNA yang komplementer, selanjutnya akan
dibentuk menjadi utas DNA baru. Proses selanjutnya sama dengan proses dasar
orientasi sekuen yang komplementer terhadap primer di dalam genom tanaman
(Azrai, 2005).
Teknik RAPD hanya digunakan pada satu primer arbitrasi yang dapat
menempel pada kedua utas DNA setelah didenaturasi pada situs tertentu yang
homolog dengan spesifitas penempelan yang tinggi. Potongan DNA yang
teramplifikasi berdasarkan pilihan penempelan yang bersifat acak dan tidak harus
berkaitan dengan gen tertentu. Penggunaan penanda RAPD relatif sederhana dan
mudah dalam hal preparasi. Teknik RAPD memberikan hasil yang lebih cepat
dibandingkan dengan teknik molekuler lainnya (Bardakci, 2001).
Penanda RAPD bersifat dominan, fragmen DNA yang dihasilkan tidak
dapat membedakan individu yang memiliki genotipe homozigot (AA) dengan
heterozigot (Aa), sedangkan yang tidak ada pita secara jelas menunjukkan
genotipe resesif (aa). Fragmen DNA hasil amplifikasi RAPD diskoring dengan
ketentuan “1” untuk ada pita dan “0” untuk tidak ada pita, data tersebut kemudian
digunakan untuk menghasilkan matrik biner untuk analisis statistik selanjutnya.
Keuntungan utama penanda RAPD adalah secara teknik lebih sederhana dan cepat
dalam pengujiannya, tidak memerlukan informasi sekuen DNA sehingga penanda
ini dapat digunakan secara luas, jumlah sampel DNA yang dibutuhkan sedikit,
primer tersedia secara komersial, dan tidak menggunakan senyawa radioaktif
(Zulfahmi, 2013).
Purwanta (2010) menyebutkan bahwa keberhasilan teknik RAPD
ditentukan oleh kemurnian dan keutuhan DNA cetakan. DNA cetakan yang tidak
murni akan mengganggu penempelan primer pada situsnya dan akan menghambat
polimerasi DNA, sedangkan DNA cetakan yang banyak mengalami fragmentasi
dapat menghilangkan situs penempelan primer.
Penggunaan penanda RAPD relatif sederhana dan mudah dalam hal
preparasi. Teknik RAPD memberikan hasil yang lebih cepat dibandingkan dengan
teknik molekuler lainnya. Teknik ini juga mampu menghasilkan jumlah karakter
yang relatif tidak terbatas, sehingga sangat membantu untuk keperluan analisis
keanekaragaman organisme yang tidak diketahui latar belakang genomnya. Pada
tanaman tahunan RAPD dapat digunakan untuk meningkatkan efisiensi seleksi
awal. Teknik RAPD sering digunakan untuk membedakan organisme tingkat
tinggi (eucaryote). Namun demikian beberapa peneliti menggunakan teknik ini
untuk membedakan organisme tingkat rendah (procaryote) atau melihat
perbedaan organisme tingkat rendah melalui piranti organel sel seperti
BAHAN DAN METODE
Tempat dan Waktu
Penelitian ini dilaksanakan di Laboratorium Terpadu Fakultas Kedokteran
Universitas Sumatera Utara, Medan yang dimulai pada bulan April sampai dengan
November 2014.
Bahan dan Alat
Bahan yang digunakan adalah materi genetik DNA dari Siagian (2014)
pada tiga populasi alami tanaman aren hasil koleksi dari berbagai lokasi di
Sulawesi Tenggara, dapat dilihat pada Tabel 1.
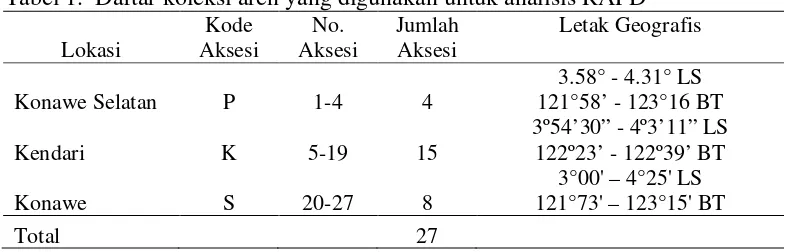
Tabel 1. Daftar koleksi aren yang digunakan untuk analisis RAPD
Lokasi Aksesi Kode Aksesi No. Jumlah Aksesi Letak Geografis
Konawe Selatan P 1-4 4 121°58’ - 123°16 BT 3.58° - 4.31° LS
Kendari K 5-19 15 3º54’30” - 4º3’11” LS 122º23’ - 122º39’
Konawe
BT
S 20-27 8 121°73' – 123°15' 3°00' – 4°25' LS Total
BT
27
Bahan kimia yang digunakan adalah CTAB 5%, NaCl, Tris, HCl, NaOH,
isopropanol, EDTA, Asam Asetat Glasial, agarose, Ethidium Bromide (EtBr),
kloroform, isoamilalkohol, β-Mercaptoethanol, PVPP, etanol 70%, etanol absolut,
DNA Marker Ladder I Kb, Go Taq ® Green Master Mix, nitrogen cair, loading
dye, aquades, aquabidestila dan 10 primer (OPN 03, OPC 12, OPD 03, OPD 13,
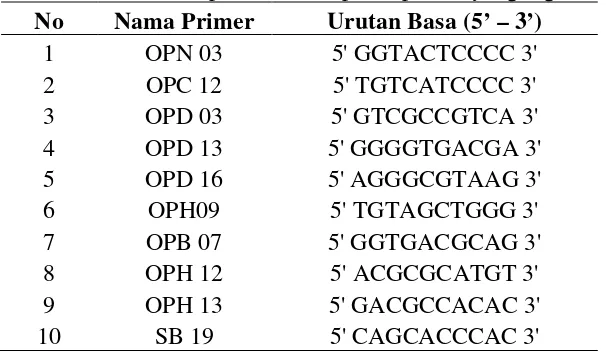
OPD 16, OPH 09, OPB 07, OPH 12, OPH 13 dan SB 19). Urutan basa dari
Tabel 2. Urutan basa primer dari sepuluh primer yang digunakan
Peralatan yang digunakan antara lain adalah Global Positioning System
(GPS), gunting, mesin PCR, pipet mikro (1 – 10 μl, 2 – 200 μl, 100 – 1000 μl),
tips (1 ml, 200 µl, 10 µl), tube (2 ml, 1,5 ml, 100 µl), vortex, waterbath, pH meter
elektrik, perangkat elektroforesis, timbangan analitik, oven, nanophotometer, UV
transluminator, lemari es, unit gel dokumentasi, sentrifus, erlenmeyer, botol
scout, masker, sarung tangan, alat tulis dan sebagainya.
Metode
Isolasi DNA Tanaman Aren
Isolasi DNA pada penelitian ini berdasarkan pada metode isolasi berbasis
CTAB menurut prosedur Orozco-Castillo et al (1994) yang dimodifikasi dengan
penambahan β-Mercaptoethanol dan PVPP (Toruan dan Hutabarat, 1997).
Isolasi DNA dilakukan terhadap daun tanaman aren yang telah diambil
dari beberapa lokasi di Sulawesi Tenggara. Tahapan isolasi yang dilakukan adalah
daun aren dibuang tulang daunnya lalu dicuci dan dikeringkan dengan tisu.
Sebanyak 0.2 gram daun aren digerus menggunakan mortar sambil ditambahkan
tabung dikocok menggunakan vortex lalu tabung diinkubasi dalam waterbath
pada suhu 650
Tabung disentrifus dengan kecepatan 13.000 rpm pada suhu ruang selama
10 menit. Supernatan yang diperoleh dipindahkan pada tabung sentrifus lain, lalu
1 ml kloroform:isoamilalkohol (24:1) ditambahkan ke dalam tabung lalu tabung
dikocok menggunakan vortex dan tabung disentrifus lagi dengan kecepatan
13.000 rpm pada suhu ruang rpm selama 10 menit. Supernatan yang diperoleh
dipindahkan lalu 1 ml isopropanol dingin ditambahkan ke dalam tabung.
Supernatan dihomogenkan dengan membolak-balik tabung lalu tabung disimpan
dalam lemari es (4
C selama 30 menit. Setiap 10 menit sekali tabung dibolak balik
dengan perlahan-lahan. Setelah itu, tabung diinkubasi pada suhu ruang selama 4-5
menit lalu 1 ml kloroform:isoamilalkohol (24:1) ditambahkan ke dalam tabung.
0C) selama satu malam kemudian tabung disentrifus kembali
dengan kecepatan 13.000 rpm pada suhu 40
Kemudian etanol absolut dingin ditambahkan lalu dibolak-balik hingga
homogen. Setelah itu, supernatan diinkubasi dalam freezer (-20
C selama 10 menit. Supernatan yang
diperoleh dibuang kemudian pelet dikeringanginkan. Pelet yang sudah kering
dilarutkan dengan bufer TE sebanyak 100 µl kemudian tabung dispin manual
hingga homogen.
0C) selama 30
menit kemudian supernatan disentrifus lagi dengan kecepatan 13.000 rpm pada
suhu 40C selama 10 menit. Supernatan dibuang sedangkan pelet dicuci
menggunakan etanol 70% dan pelet dikeringanginkan. Pelet DNA yang sudah
Uji Kualitas DNA
Gel agaros 0.8% dibuat dari 0.28 gram agaros dan 35 ml larutan bufer
TAE 1x. kemudian dipanaskan hingga larut dengan menggunakan hot plate dan
didinginkan pada suhu kamar hingga hangat. Selanjutnya, ditambahkan 0.5 µl
EtBr dan dituang ke dalam cetakan gel elektroforesis yang telah dipasang sisir
(cetakan sumur) hingga gel memadat. Gel yang sudah padat dipindahkan ke dalam
bak elektroforesis, kemudian tuang TAE 1x ke dalam bak elektroforesis. Sampel
yang akan dielektroforesis dicampur dengan loading dye dengan perbandingan 5:1
(DNA: loading dye). Setelah tercampur maka diinjeksi ke dalam sumur gel agaros
menggunakan pipet mikro. Setelah semua sampel selesai diinjeksi maka alat
elektroforesis dihubungkan pada power supply yang dialiri tegangan listrik 80 volt
selama 45 menit. Hasil elektroforesis diamati dengan bantuan lampu UV
transilluminator dan didokumentasikan menggunakan gel documentation.
Uji Kuantitas DNA
Pengujian dilakukan dengan menggunakan alat spektrofotometri. Larutan
stok DNA diambil sebanyak 2 µl, kemudian alat dijalankan. Absorbansi (A)
diukur pada panjang gelombang 260 nm dan 280 nm. Tingkat kemurnian DNA
ditentukan dengan nilai perbandingn A260/A280. Menurut Wilson dan Walker
(2010), sampel DNA murni akan menghasilkan rasio A260/A280 berkisar 1.8-2.0.
Nilai kemurnian yang lebih dari 2.0 menunjukkan bahwa sampel mengandung
kontaminan RNA, sedangkan nilai kemurnian yang kurang dari 18 menunjukkan
RAPD DNA Pohon Aren
Pembuatan master mix PCR dilakukan dalam tabung mikro dengan
komposisi untuk satu kali reaksi dengan total volume 25 µl antara lain Go Taq
PCR 12.5 µl, nuclease free water 9.5 µl, primer 1 µl dan DNA sampel 2 µl
dengan konsentrasi DNA sebesar 10 µg/ml. Proses amplifikasi dilakukan
menggunakan mesin PCR. Program running PCR sebanyak 45 siklus dengan
reaksi: predenaturasi 940C selama 2 menit, denaturasi 940C selama 1 menit,
annealing 360C selama 1 menit, extension 720C selama 2 menit, post extension
720C selama 10 menit, dan kondisi akhir PCR 40C (Setiyo, 2001).
Elektroforesis Hasil Amplifikasi
Elektroforesis hasil amplifikasi dilakukan menggunakan gel agaros 1%.
Gel dibuat dengan melarutkan 1.3 gram agaros pada 130 ml bufer TAE 1x,
kemudian dipanaskan hingga larut dengan menggunakan hot plate dan
didinginkan pada suhu kamar hingga hangat. Selanjutnya, ditambahkan 1.5 µl
EtBr dan dituang ke dalam cetakan gel elektroforesis yang telah dipasang sisir
(cetakan sumur) hingga gel memadat. Gel yang sudah padat dipindahkan ke dalam
bak elektroforesis, kemudian tuang bufer TAE 1x ke dalam bak elektroforesis.
Marker yang digunakan adalah 1 kb DNA ladder sebanyak 5 µl dicampur dengan
loading dye sebanyak 2 µl. Sampel hasil PCR sebanyak 8 µl dan marker yang
telah dicampur dengan loading dye diinjeksi ke dalam sumur gel agaros
menggunakan pipet mikro. Setelah semua sampel selesai diinjeksi maka alat
elektroforesis dihubungkan pada power supply yang dialiri tegangan listrik 100
volt selama 65 menit. Hasil elektroforesis diamati dengan bantuan lampu UV
Analisis Data
Pola pita yang muncul pada gel diterjemahkan ke dalam data biner dengan
skoring manual. Setiap pita mewakili satu karakter dan diberi nilai berdasarkan
ada tidaknya pita. Angka satu “1” untuk pita yang terbentuk dan angka nol “0”
untuk pita yang tidak terbentuk.
Polymorphic Information Content (PIC) untuk beberapa marker dihitung
dengan menggunakan rumus:
= Polymorphic Information Content (PIC) pada marker
i
(1-f
= frekuensi dari pita primer yang muncul
i
Nilai PIC untuk dominan marker seperti RAPD memiliki nilai maksimum
yaitu 0.5 untuk f
) = frekuensi dari pita yang tidak muncul
i
Untuk melihat persentase pita polimorfik menggunakan rumus berikut ini: = 0.5 (Ma et al, 2013).
% Pita Polimor�ik=∑ lokusyangpolimor�ik ∑ ������������� x 100
Matriks jarak atau ketidaksamaan genetik untuk semua kombinasi
pasangan individu dapat dilakukan dengan dua tipe analisis deskriptif dari
keragaman : (1) Principal Coordinates Analysis (PCoA), suatu jenis analisis
faktorial pada tabel ketidaksamaan untuk mendapatkan group origin utama dan
(ii) Neighbour-Joining Tree (NJtree) berdasarkan Saitou dan Nei (1978) untuk
memperoleh gambaran dari kekerabatan diantara individu-individu. Perhitungan
dan analisis deskriptif ini menggunakan software DARwin 5.05 (Perrier dan
HASIL DAN PEMBAHASAN
Analisis Profil Pita Hasil Isolasi DNA Tanaman Aren
Proses isolasi DNA tanaman aren menggunakan metode Orozco-Castillo
et al (1994) yang dimodifikasi dengan penambahan β-Mercaptoethanol dan PVPP
(Toruan dan Hutabarat, 1997) karena metode ini lebih praktis dan dapat
menghasilkan DNA yang baik dari tanaman aren dibandingkan dengan metode
lainnya yang telah dicobakan. Uji kualitatif terhadap 27 sampel DNA dilakukan
dengan elektroforesis gel agaros 0.8%. Uji ini dilakukan untuk mengetahui
kualitas DNA yang diperoleh. Hasil yang diperoleh dari 27 sampel DNA tanaman
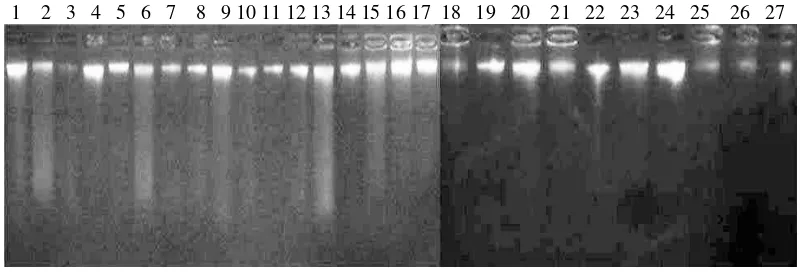
aren dapat dilihat pada Gambar 2.
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 2. Elektroforegram uji kualitatif 27 DNA tanaman aren
Ket: Konawe Selatan (1-4), Kendari (5-19), dan Konawe (20-27)
Elektroforegram menunjukkan isolasi DNA telah berhasil, dapat dilihat
dari fragmen DNA yang tampak pada gel. Fragmen DNA yang menunjukkan
bahwa DNA memiliki pita yang terang dan tebal terdapat pada aksesi aren nomor
1, 2, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 19, 20, 21, 22, 23 dan 24
sedangkan pita yang agak tipis dan kurang terang terdapat pada aksesi aren
nomor 3, 18, 25, 26 dan 27 dengan demikian bisa digunakan dalam analisis
DNA genom dapat diisolasi dengan berbagai macam teknik. Pada
prinsipnya, sel harus dipecah terlebih dahulu menggunakan beberapa agensia,
baik secara fisik maupun kimiawi. Senyawa yang sering digunakan untuk
memecah sel pada isolasi DNA genom adalah CTAB. Senyawa CTAB biasanya
digunakan untuk isolasi DNA dari jaringan tanaman. Setelah sel dipecah
selanjutnya dilakukan isolasi dan pemurnian DNA (Yuwono, 2008).
Sudjadi (2008) teknik pemecahan sel dapat dibagi dalam metode fisik,
metode mekanik, dan metode kimiawi. Sel diperlakukan dengan pemaparan
senyawa kimiawi yang mempengaruhi dinding sel. Metode kimiawi lebih banyak
digunakan untuk preparasi DNA.
Dalam suatu teknik isolasi DNA masih diperlukan suatu tahapan untuk
meminimalkan senyawa-senyawa kontaminan yang dapat mengganggu reaksi
PCR seperti polisakarida dan metabolit sekunder. Hal ini disebabkan keberadaan
polisakarida dan metabolit sekunder dalam sel tanaman sering menyulitkan dalam
isolasi asam nukleat (Maftuchah dan Zainuddin, 2013).
Kandungan senyawa sekunder dalam sel tanaman berbeda-beda, maka
setiap tanaman membutuhkan prosedur isolasi yang optimum agar diperoleh DNA
genom yang dapat digunakan sebagai bahan dalam analisis molekuler. Optimasi
prosedur tersebut dapat dilakukan terhadap komposisi larutan bufer lisisnya
ataupun teknik penanganan fisik dalam pemisahan DNA genom dari senyawa
lain. Pada prinsipnya optimasi prosedur ini bertujuan melindungi DNA genom
dari degradasi akibat senyawa sekunder yang dilepaskan ketika sel dihancurkan
Hasil penelitian sebelumnya yang dilakukan oleh Harahap (2013),
mengenai keragaman genetik pada populasi aren Sumatera Utara menghasilkan
pita DNA yang cukup jelas dan tebal. Bila dibandingkan dengan hasil uji
kualitatif pada aren asal Sulawesi Tenggara maka menunjukkan hasil yang sama
(Gambar 2).
Uji kuantitatif DNA dilakukan secara spektrofotometri pada panjang
gelombang 260 nm dan 280 nm sehingga diperoleh nilai kemurnian dan
konsentrasi DNA hasil isolasi. Panjang gelombang 260 nm merupakan serapan
maksimum untuk asam nukleat, sedangkan panjang gelombang 280 nm
merupakan serapan maksimum untuk protein. Hasil pengukuran dapat dilihat pada
Lampiran 9.
Kemurnian DNA yang diperoleh pada penelitian ini berkisar antara
0.500-1.949. Dari 27 sampel DNA tanaman aren, sebanyak 20 tanaman aren memiliki
nilai kemurnian 1.8-2.0 yang menunjukkan DNA yang diisolasi telah murni
(Wilson dan Walker, 2010). Aksesi tersebut yaitu aksesi nomor 1, 3, 4,6,7, 9, 10,
12, 13, 14, 15, 18, 19, 20, 21, 22, 23, 24, 25 dan 27. Namun, ada juga nilai
kemurnian sampel DNA di bawah 1.8. Aksesi tersebut yaitu aksesi nomor 2, 5, 8,
11, 16, 17 dan 26.
Konsentrasi DNA yang dihasilkan berkisar antara 0.05-55.90 µg/ml.
Konsentrasi paling rendah diperoleh pada aksesi nomor 11 sebesar 0.05 µg/ml
sedangkan konsentrasi paling tinggi diperoleh pada aksesi nomor 22 sebesar 55.90
µg/ml. Konsentrasi yang digunakan untuk proses PCR-RAPD adalah 0.5-50 ng/µl
Prinsip dasar pada spektrofotometri adalah sampel harus jernih dan larut
sempurna. Tidak ada partikel koloid apalagi suspensi. DNA yang mengandung
basa-basa purin dan pirimidin dapat menyerap cahaya UV. Pita ganda DNA dapat
menyerap cahaya UV pada 260 nm, sedangkan kontaminan protein atau phenol
dapat menyerap cahaya pada 280 nm. Dengan adanya perbedaan penyerapan
cahaya UV ini, sehingga kemurnian DNA dapat diukur dengan menghitung nilai
absorbansi 260 nm dibagi dengan nilai absorbansi 280 (Å260/Å280) dan nilai
kemurnian DNA berkisar antara 1.8-2.0 (Fatchiyah, 2011).
Menurut Haris et al (2003), konsentrasi DNA akan berdampak pada
kualitas fragmen hasil amplifikasi. Konsentrasi DNA yang terlalu rendah akan
menghasilkan fragmen yang sangat tipis pada gel atau bahkan tidak terlihat secara
visual, sebaliknya konsentrasi DNA yang terlalu tinggi akan menyebabkan
fragmen terlihat tebal sehingga sulit dibedakan antara satu fragmen dengan
fragmen lainnya.
Salah satu keuntungan pemakaian analisis keragaman genetik tanaman
dengan menggunakan teknik molekuler yang memanfaatkan teknologi amplifikasi
PCR adalah kuantitas DNA yang diperlukan hanya sedikit. Di samping itu, dalam
pelaksanaan teknik RAPD tingkat kemurnian DNA yang dibutuhkan tidak perlu
terlalu tinggi, atau dengan kata lain teknik amplifikasi PCR relatif toleran
terhadap tingkat kemurnian DNA (Maftuchah dan Zainuddin, 2013).
Hasil penelitian yang sebelumnya dilakukan oleh Harahap (2013),
mengenai keragaman genetik pada tanaman aren Sumatera Utara menunjukkan
bahwa kemurnian DNA yang diperoleh berkisar antara 1.39 - 2.23 dan konsentrasi
kuantitas pada aren asal Sulawesi Tenggara maka menunjukkan hasil yang lebih
baik (Lampiran 9).
Analisis Profil Pita Hasil Amplifikasi PCR Tanaman Aren
Hasil amplifikasi menggunakan 10 primer yang digunakan yaitu OPN 03,
OPC 12, OPD 03, OPD 13, OPD 16, OPH 09, OPB 07, OPH 12, OPH 13 dan SB
19 pada 27 aksesi tanaman aren menghasilkan produk PCR yang dapat dibaca dan
diskor, sehingga hasilnya dapat dianalisis. Namun, tidak semua primer
mengamplifikasi DNA pada 27 aksesi tanaman aren. Hasil PCR dapat dilihat pada
Gambar 3-12.
Lima dari sepuluh primer yang digunakan mengamplifikasi DNA pada 27
aksesi yaitu primer OPC 12, OPD 03, OPD 13, OPH 13 dan SB 19 sedangkan
lima primer lagi yaitu OPN 03, OPD 16, OPH 09, OPB 07 dan OPH 12 tidak
mengamplifikasi DNA pada 27 aksesi. Jumlah DNA yang paling banyak tidak
teramplifikasi terdapat pada OPD 16 dan OPH 12 yaitu masing-masing 5 aksesi
pada aksesi nomor 16, 17, 20, 26, dan 27 sedangkan yang paling sedikit terdapat
pada OPN 03 yaitu 3 aksesi pada aksesi nomor 20, 26, dan 27. Sementara OPH 09
dan OPB 07 tidak mengamplifikasi DNA yaitu masing-masing 4 aksesi pada
aksesi nomor 16, 17, 20, dan 26 (Tabel 3).
Pada penelitian ini, primer yang tidak sesuai dengan sekuen DNA tanaman
aren tidak menghasilkan produk amplifikasi karena tidak terdapat situs yang
komplementer pada DNA tanaman aren dengan sekuen primer tersebut dan bisa
juga tidak adanya pita. Selain itu, amplifikasi DNA tergantung dari kecocokan
Tabel 3. Hasil amplifikasi sepuluh primer yang digunakan
Keberhasilan suatu primer dalam mengamplifikasi DNA cetakan
ditentukan oleh ada tidaknya homologi sekuen nukleotida primer dengan sekuen
nukleotida DNA cetakan. Selain itu juga dipengaruhi oleh kualitas dan kuantitas
DNA, konsentrasi MgCl2
Kesesuaian primer, efisiensi dan optimasi proses PCR menentukan
keberhasilan dalam teknik ini. Primer yang tidak spesifik dapat menyebabkan
teramplifikasinya daerah lain dalam genom yang tidak dijadikan sasaran atau
sebaliknya tidak ada daerah genom yang teramplifikasi. Optimasi PCR juga
diperlukan untuk menghasilkan karakter yang diinginkan. Optimasi ini
menyangkut suhu denaturasi dan annealing DNA dalam mesin PCR. Suhu
denaturasi yang rendah dapat menyebabkan belum terbukanya DNA utas ganda
sehingga tidak dimungkinkan terjadinya polimerisasi DNA baru. Proses
penempelan primer pada utas DNA yang sudah terbuka memerlukan suhu
optimum, sebab suhu yang terlalu tinggi dapat menyebabkan amplifikasi tidak
terjadi atau sebaliknya suhu yang terlalu rendah menyebabkan primer menempel , enzim Taq DNA polimerase, dan suhu pelekatan
pada sisi lain genom yang bukan sisi homolognya; akibatnya dapat teramplifikasi
banyak daerah tidak spesifik dalam genom tersebut. Suhu penempelan (annealing)
ini ditentukan berdasarkan primer yang digunakan yang dipengaruhi oleh panjang
dan komposisi primer (Suryanto, 2003).
Keberhasilan teknik ini ditentukan oleh ada tidaknya situs penempelan
primer, kemurnian DNA dan keutuhan DNA cetakan (Bardakci, 2001).
Konsentrasi DNA genom merupakan faktor terpenting dalam reaksi amplifikasi.
Konsentrasi DNA yang terlalu tinggi dapat meningkatkan kontaminan yang
menggangu reaksi amplifikasi (Chen, 2000).
Konsentrasi primer berpengaruh terhadap intensitas produk PCR-RAPD.
Menurut Padmalatha dan Prasad (2006) konsentrasi primer yang terlalu rendah
atau yang terlalu tinggi menyebabkan tidak terjadinya amplifikasi. Rasio yang
rendah antara primer dan DNA cetakan dapat menyebabkan produk RAPD yang
dihasilkan tidak konsisten. Magnesium merupakan komponen yang penting dalam
reaksi PCR dan mempengaruhi kualitas profil RAPD yang dihasilkan
(Pharmawati, 2009). Magnesium mempengaruhi penempelan primer serta aktifitas
enzim (Padmalatha dan Prasad, 2006). Konsentrasi MgCl2
Secara umum, hasil penelitian ini menunjukkan bahwa pola pita yang
dihasilkan oleh sepuluh primer yang digunakan memperlihatkan pola pita yang
berbeda dan ada juga yang serupa pada aksesi Konawe Selatan, Kendari dan
Konawe. Dari kesepuluh primer yang digunakan menghasilkan jumlah pola pita
sebanyak 3-6 pita DNA per primer. Ukuran pita-pita DNA yang dihasilkan yang tinggi juga
mempengaruhi jumlah pita yang dihasilkan dan mengakibatkan penurunan
bervariasi antara 170 bp sampai dengan 3376 bp. Total pola pita dari kesepuluh
primer yang tampak sebanyak 41 dengan rata-rata 4.1 pita per primer dengan pita
polimorfik sebanyak 39 pita dan pita yang monomorfik sebanyak 2 pita.
Persentase pita yang polimorfik bervariasi sebesar 66.7% sampai 100% dengan
rata-rata 93.3% untuk seluruh primer. Tingkat keinformatifan dari setiap primer
bervariasi dari 0.35 sampai 0.5 dengan rata-rata 0.47 yang menunjukkan
kesamaan rasio tingkat keinformatifan primer (Tabel 4).
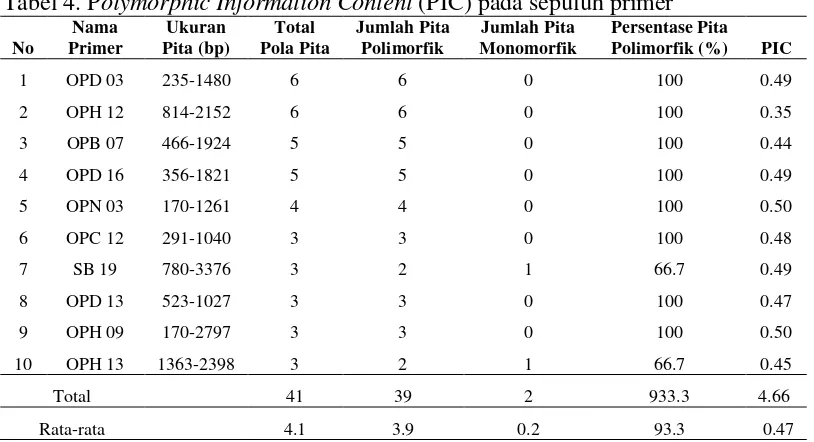
Tabel 4. Polymorphic Information Content (PIC) pada sepuluh primer
No
Jumlah pola pita tertinggi terdapat pada primer OPD 03 dan OPH 12 yang
berjumlah 6 pola pita sedangkan jumlah pola pita terendah terdapat pada primer
OPC 12, SB19, OPD 13, OPH 09 dan OPH 13 yang berjumlah 3 pola pita.
Ukuran pita tertinggi terdapat pada primer SB 19 sebesar 3376 bp sedangkan
ukuran pita terendah terdapat pada primer OPN 03 dan OPH 09 sebesar 170 bp.
Jumlah pita polimorfik tertinggi terdapat pada primer OPD 13 dan OPH 12
yaitu 6 pita polimorfik sedangkan jumlah pita polimorfik yang terendah terdapat
OPD 16, OPN 03, OPC 12, OPD 13, dan OPH 09 sedangkan SB 19 dan OPH 13
memiliki persentase polimorfik sebesar 66.7%.
Nilai PIC tertinggi terdapat pada OPN 03 dan OPH 09 yaitu sebesar 0.5
sedangkan yang terendah terdapat pada primer OPH 12 yaitu sebesar 0.35. Dari
hasil diatas dapat diketahui bahwa primer yang paling diskriminatif dilihat dari
total pola pita adalah primer OPD 03 dan OPH 12 yaitu sebesar 6 pola pita
sedangkan dilihat dari nilai PIC adalah primer OPN 03 dan OPH 09 yaitu sebesar
0.50.
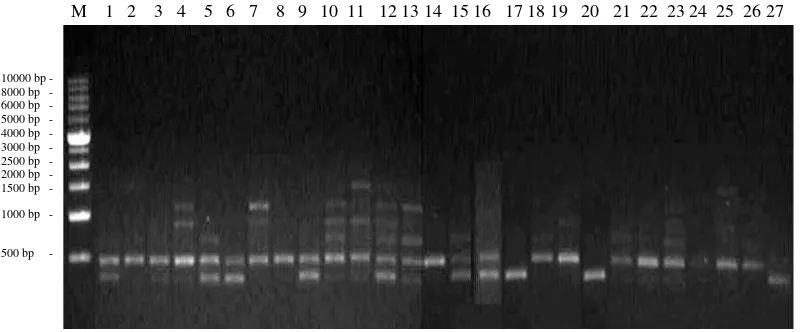
Primer OPD 03 menunjukkan pola pita yang berbeda antara aksesi
Konawe Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan
sebanyak 6 pola pita dengan ukuran pita berkisar antara 235 bp – 1480 bp.
Persentase pita yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini
sebesar 0.49 (Gambar 3).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 3. Elektroforegram amplifikasi 27 DNA aren dengan primer OPD 03 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
Primer OPH 12 menunjukkan pola pita yang berbeda antara aksesi
Konawe Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan
Persentase pita yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini
sebesar 0.35 (Gambar 4).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 4. Elektroforegram amplifikasi 27 DNA aren dengan primer OPH 12 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
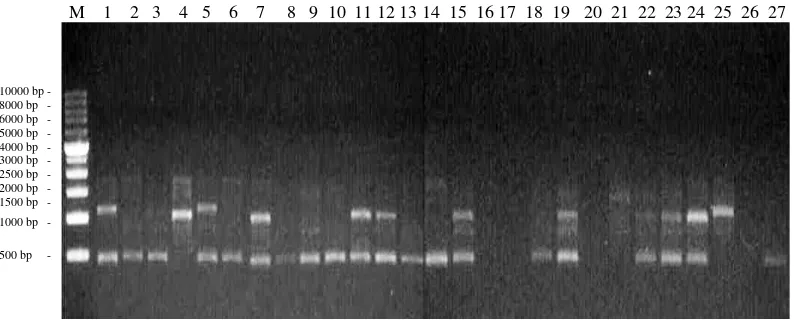
Primer OPB 07 menunjukkan pola pita yang berbeda antara aksesi
Konawe Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan
sebanyak 5 pola pita dengan ukuran pita berkisar antara 466 bp – 1924 bp.
Persentase pita yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini
sebesar 0.44 (Gambar 5).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Primer OPD 16 menunjukkan pola pita yang serupa antara aksesi Konawe
Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan sebanyak 5
pola pita dengan ukuran pita berkisar antara 356 bp – 1821 bp. Persentase pita
yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini sebesar 0.49
(Gambar 6).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 6. Elektroforegram amplifikasi 27 DNA aren dengan primer OPD 16 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
Primer OPN 03 menunjukkan pola pita yang berbeda antara aksesi
Konawe Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan
sebanyak 4 pola pita dengan ukuran pita berkisar antara 170 bp – 1261 bp.
Persentase pita yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini
sebesar 0.50 (Gambar 7). 10000 bp -
8000 bp - 6000 bp - 5000 bp - 4000 bp - 3000 bp - 2500 bp - 2000 bp - 1500 bp - 1000 bp -
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 7. Elektroforegram amplifikasi 27 DNA aren dengan primer OPN 03 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
Primer OPC 12 menunjukkan pola pita yang serupa antara aksesi Konawe
Selatan dan Konawe sedangkan pada aksesi Kendari menunjukkan pola pita yang
berbeda dengan total pola pita yang dihasilkan sebanyak 3 pola pita dengan
ukuran pita berkisar antara 291 bp – 1040 bp. Persentase pita yang polimorfik
sebesar 100%. Tingkat keinformatifan primer ini sebesar 0.48 (Gambar 8).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 8. Elektroforegram amplifikasi 27 DNA aren dengan primer OPC 12 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
Primer SB 19 menunjukkan pola pita yang serupa antara aksesi Konawe
Selatan dan Konawe sedangkan pada aksesi Kendari menunjukkan pola pita yang
ukuran pita berkisar antara 780 bp – 3376 bp. Persentase pita yang polimorfik
sebesar 66.7%. Tingkat keinformatifan primer ini sebesar 0.49 (Gambar 9).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 9. Elektroforegram amplifikasi 27 DNA aren dengan primer SB 19 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
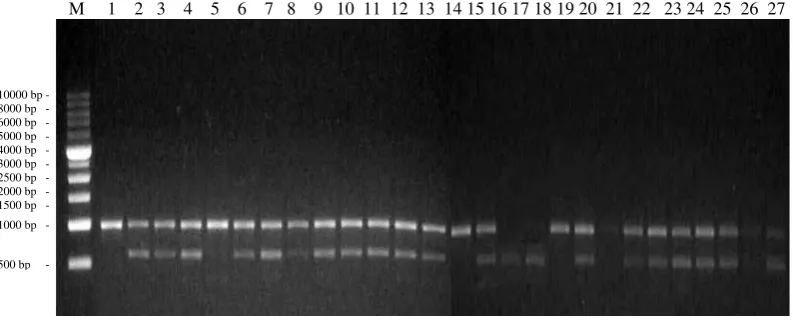
Primer OPD 13 menunjukkan pola pita yang serupa antara aksesi Konawe
Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan sebanyak 3
pola pita dengan ukuran pita berkisar antara 523 bp – 1027 bp. Persentase pita
yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini sebesar 0.47
(Gambar 10).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Primer OPH 09 menunjukkan pola pita yang berbeda antara aksesi
Konawe Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan
sebanyak 3 pola pita dengan ukuran pita berkisar antara 170 bp – 2797 bp.
Persentase pita yang polimorfik sebesar 100%. Tingkat keinformatifan primer ini
sebesar 0.50 (Gambar 11).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 11. Elektroforegram amplifikasi 27 DNA aren dengan primer OPH 09 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
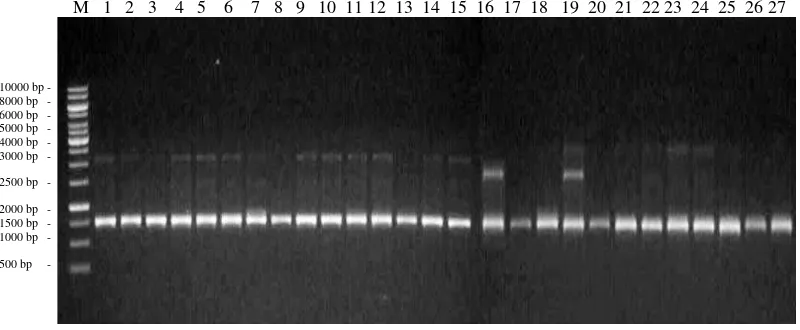
Primer OPH 13 menunjukkan pola pita yang serupa antara aksesi Konawe
Selatan, Kendari dan Konawe dengan total pola pita yang dihasilkan sebanyak 3
pola pita dengan ukuran pita berkisar antara 1363 bp – 2398 bp. Persentase pita
yang polimorfik sebesar 66.7%. Tingkat keinformatifan primer ini sebesar 0.45
(Gambar 12). 10000 bp - 8000 bp - 6000 bp - 5000 bp - 4000 bp - 3000 bp - 2500 bp - 2000 bp - 1500 bp -
1000 bp -
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Gambar 12. Elektroforegram amplifikasi 27 DNA aren dengan primer OPH 13 Ket : M = marker ladder 1 kb, Konawe Selatan (1-4), Kendari (5-19),
dan Konawe (20-27)
Jumlah dan intensitas pita DNA yang dihasilkan setelah amplifikasi DNA
dengan PCR sangat tergantung bagaimana primer mengenal urutan DNA
komplementernya pada cetakan DNA (DNA template) yang digunakan. Hasil
amplifikasi DNA tidak selalu memperoleh pita dan intensitas yang sama.
Intensitas pita DNA hasil amplifikasi pada setiap primer sangat dipengaruhi oleh
kemurnian dan konsentrasi cetakan DNA. Cetakan DNA yang mengandung
senyawa-senyawa seperti polisakarida dan senyawa fenolik, serta konsentrasi
DNA yang terlalu kecil sering menghasilkan pita DNA amplifikasi yang redup
atau tidak jelas (Weeden et al., 1992). Sebaran situs penempelan primer pada
cetakan DNA dan adanya kompetisi tempat penempelan primer pada cetakan
DNA menyebabkan satu fragmen diamplifikasi dalam jumlah banyak dan
fragmen lainnya sedikit. Proses amplifikasi mungkin saja diinisiasi pada beberapa
tempat, namun hanya beberapa set yang dapat dideteksi sebagai pita sesudah
diamplifikasi.
Pola pita DNA yang dihasilkan dari hasil amplifikasi menunjukkan
adanya polimorfisme. Jumlah pita polimorfik hasil amplifikasi berbeda-beda.
mengamati adanya variasi. Polimorfisme pita DNA dapat dipengaruhi oleh
banyaknya variasi individu dalam suatu populasi. Tingginya polimorfisme pita
pada penelitian ini menunjukkan tingginya keragaman genetik pada tanaman aren
asal Sulawesi Tenggara. Adanya perbedaan pola pita yaitu berdasarkan jumlah
dan ukuran pita menggambarkan adanya genom tanaman yang sangat kompleks
(Grattapaglia et al., 1992).
Menurut Demeke dan Adams (1994), amplifikasi DNA dengan primer
acak pada analisis RAPD biasanya menghasilkan 5-20 fragmen untuk setiap
primer. Jumlah fragmen hasil amplifikasi dengan RAPD memang lebih rendah
dibandingkan dengan hasil amplifikasi menggunakan AFLP (Haris et al., 2003).
Kelemahan RAPD adalah pemunculan pita DNA kadang-kadang tidak
konsisten. Hal ini lebih sering terjadi jika suhu annealing yang digunakan terlalu
tinggi. Dalam analisis kekerabatan, hal ini dapat diatasi dengan menggunakan
primer yang lebih banyak. Ruas DNA yang berulang sering berlipat ganda,
homologi urutan nukleotida pada pita-pita DNA dengan mobilitas yang sama pada
gel tidak diketahui, penanda RAPD bersifat dominan dan tingkat keberulangannya
(reproducibility) rendah (Demeke dan Adams, 1994).
Selain itu, untuk melihat tingkat keinformatifan dari primer yang
digunakan maka perlu diketahui nilai PIC dari primer tersebut. PIC mengacu pada
nilai suatu penanda untuk mendeteksi polymorphisme di dalam suatu populasi.
PIC tergantung pada banyaknya dapat ditemukan alel dan distribusi dari
frekuensinya (Anderson et al., 1993). Semakin besar nilai PIC suatu primer maka