PENAPISAN
Streptomyces
spp PENGHASIL
MICROBIAL
TRANSGLUTAMINASE
DAN PENGKLONAN DNA PENYANDINYA
MELALUI KONSTRUKSI PUSTAKA GENOM
SEPRIANTO
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul “Penapisan Streptomyces spp Penghasil Microbial Transglutaminase dan Pengklonan DNA Penyandinya Melalui Konstruksi Pustaka Genom” adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
SEPRIANTO. Penapisan Streptomyces spp Penghasil Microbial Transglutaminase dan Pengklonan DNA Penyandinya melalui Konstruksi Pustaka Genom. Dibimbing oleh SUHARSONO dan DEWI SESWITA ZILDA.
Enzim transglutaminase (TGase, E.C. 2.3.2.13) merupakan enzim yang mengkatalisis reaksi perpindahan gugus asil antara residu glutamin (Gln) yang berfungsi sebagai donor asil dan residu lisin (Lys) sebagai aseptor yang membentuk ikatan silang (crosslinking) ε-( -glutamyl) lisin isopeptida yang menghasilkan ikatan kovalen inter atau intramolekuler yang berikatan silang dengan protein makanan. Produksi enzim Microbial Transglutaminase (MTGase) dari strain liar mempunyai beberapa kelemahan yaitu selain pertumbuhan selnya lambat, produk yang dihasilkan sedikit dan protein yang dihasilkan juga bercampur dengan protein lainnya. Selain itu proses pemurnian cukup sulit karena memerlukan beberapa tahap pemurnian. Pendekatan teknologi rekayasa genetika sangat diperlukan untuk mendapatkan hasil yang lebih baik sehingga dapat dikembangkan dalam skala industri.
Penelitian ini bertujuan untuk mendapatkan bakteri yang menghasilkan MTGase dan mengklon fragmen DNA penyandinya. Untuk itu, penelitian ini dilakukan melalui beberapa tahapan yaitu, penapisan bakteri Streptomyces spp penghasil MTGase, identifikasi isolat penghasil MTGase dan pembuatan pustaka genom dengan mengkloning fragmen DNA 40 kb yang mengandung gen penyandi MTGase.
Hasil penelitian menunjukkan bahwa penapisan terhadap bakteri Streptomyces spp penghasil MTGase yang diisolasi dari berbagai jenis tanah di Indonesia, mendapatkan satu isolat yang potensial yaitu TTA 02 SDS 14. Analisis molekuler dengan PCR membuktikan bahwa isolat tersebut mengandung gen MTGase. Sekuen parsial gen MTGase dari isolat tersebut memiliki homologi 93 % dengan MTGase dari Streptomyces cinnamoneus. Berdasarkan sekuen gen 16S rRNA, isolat TTA 02 SDS 14 memiliki kekerabatan terdekat dengan Streptomyces thioleteus dengan homologi 99%. Pustaka genom dengan sisipan fragmen ~40 Kb yang membawa gen MTGase telah berhasil dikonstruksi dengan menggunakan fosmid pCC1FOSTM di dalam E.coli EPI300-TIR.
SUMMARY
SEPRIANTO. Screening of Streptomyces spp Producing Microbial Transglutaminase and Cloning of Its Transglutaminase Gene Through Genomic Library Construction. Supervised by SUHARSONO and DEWI SESWITA ZILDA.
Transglutaminase (TGase, E.C. 2.3.2.13) is an enzyme that catalyzes acyl group exchange between the -carboxyamide groups of peptide-bond glutamine residues (Gln) act as the acyl donors and the ε -amino groups of lysine residues (Lys) serve as acyl acceptors that crooslinking ε-( -glutamyl) lysine bonds are formed, resulting covalent bond intermolecular or intramolecular wiht food protein. Production of Microbial Transglutaminase (MTGase) based on wild bacterial strains have several drawbacks such as slow cell growing, low yield and a large amount of non-target proteins interfered with target proteins, and the difficulty to purify native proteins that requires multiple purification steps. DNA recombinant technology is an emerging and well established approach to provide bacterial proteins in sustainable way at the industrial scale.
This research aimed to obtain bacterial isolates which capable MTGase production and to clone ~40 kb DNA fragment containing MTGase gene. Therefore, it was conducted several stages that screening of Streptomyces spp MTGase production, Indentification of MTGase producing isolate and construction fosmid-based of a genomic library from the isolate and cloning of a ~40 kb fosmid harboring a MTGase gene.
These result showed that screening of MTGase producing Streptomyces spp isolated from Indonesian soils was obtained one potential isolate, designated as TTA 02 SDS 14. The partial MTGase-encoding gene was amplified by PCR and sequenced. The sequence result indicate its similarity of 93 % with that of Streptomyces cinnamoneus. Based on 16S rRNA gene sequencing of isolate TTA 02 SDS 14 was identified as neighbor of Streptomyces thioleteus with the similarity at 99%. Fosmid-based construction of a genomic library from the isolate and subsequent screening led to the isolation of a ~40 kb fosmid harboring a MTGase gene was successfully to constructed with used fosmid library pCC1FOSTM into E.coli EPI300-TIR
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Bioteknologi
PENAPISAN
Streptomyces spp
PENGHASIL
MICROBIAL
TRANSGLUTAMINASE
DAN PENGKLONAN DNA PENYANDINYA
MELALUI KONSTRUKSI PUSTAKA GENOM
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
Judul Tesis : Penapisan Streptomyces spp Penghasil Microbial Transglutaminase dan Pengklonan DNA Penyandinya Melalui Konstruksi Pustaka Genom
Nama : Seprianto NIM : P0530121
Disetujui oleh Komisi Pembimbing
Prof. Dr. Ir. Suharsono, DEA Ketua
Dr. Dewi Seswita Zilda M.Si Anggota
Diketahui oleh
Ketua Program Studi Bioteknologi
Prof. Dr. Ir. Suharsono, DEA
Dekan Sekolah Pascasarjana
Dr. Ir. Dahrul Syah, M.Sc Agr
PRAKATA
Puji dan syukur penulis sampaikan kepada Allah Subhanahu Wa Ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan penelitian yang dilaksanakan sejak Agustus β014 dengan judul “Penapisan Streptomyces spp Penghasil Microbial Transglutaminase dan Pengklonan DNA Penyandinya Melalui Konstruksi Pustaka Genom”. Tesis ini merupakan hasil penelitian yang dilakukan di Laboratorium Bioteknologi, Pusat Penelitian dan Pengembangan Daya Saing Produk dan Bioteknologi Kelautan dan Perikanan, P3DSBKP-KKP, Jakarta.
Terima kasih penulis ucapkan kepada Bapak Prof. Dr. Ir. Suharsono, DEA dan Ibu Dr. Dewi Seswita Zilda M.Si selaku pembimbing yang telah memberikan bimbingan dan arahan selama penelitian hingga penyusunan tesis ini. Kepada Bapak Prof. Dr. Aris Tri Wahyudi, M.Si selaku penguji luar komisi yang telah banyak memberikan masukan dalam sidang tesis serta Ibu Dr. Utut Widiastuti M.Si atas saran untuk perbiakan dalam kesempurnaan tesis ini. Penulis juga mengucapkan terima kasih kepada Bapak Dr. rer. nat Agustinus Robert Uria M.Sc selaku pembimbing lapangan yang telah banyak memberikan ilmu serta bimbingan selama penelitian berlangsung. Ucapan terima kasih juga penulis sampaikan kepada Pusat Penelitian dan Pengembangan Daya Saing Produk dan Bioteknologi Kelautan Perikanan (P3DSPBKP) yang telah mendanai dan memfasilitasi penelitian ini dan DIKTI atas Beasiswa BPPDN (Beasiswa Pendidikan Pascasarjana Dalam Negeri) selama menempuh pendidikan di pascasarjana IPB
Di samping itu, penghargaan penulis sampaikan kepada Ibu Dr. Ekowati Chasanah M.Sc dan Ibu Ir. Yusro Nuri Fawzya M.Si yang telah banyak memberikan bimbingan dan arahan selama penelitian. Kepada seluruh staf dan peneliti laboratorium Bioteknologi, laboratorium Kimia, laboratorium Mikrobiologi, teman-teman mahasiswa seperjuangan di laboratorium Bioteknologi (P3DSPBKP) dari berbagai universitas, serta rekan–rekan Pascasarjana Bioteknologi 2013-2014 IPB. Penulis mengucapkan terima kasih atas segala bantuan dan kerjasamanya dalam menyelesaikan penelitian ini. Karya kecil ini penulis persembahan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya sehingga penulis dapat menyelesaikan destinasi ini dengan baik.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
2 TINJAUAN PUSTAKA 3
Enzim Microbial Transglutaminase 3
Karakteristik Streptomyces sp 5
Teknologi Rekombinan DNA 5
Pustaka Genom 6
3 METODE 8
Kerangka Penelitian 8
Waktu dan Tempat 9
Bahan 9
Prosedur Penelitian 10
4 HASIL DAN PEMBAHASAN 14
Penapisan Streptomyces spp Penghasil MTGase 14 Identifikasi Isolat TTA 02 SDS 14 Berdasarkan Sekuen 16S rRNA 16
Konstruksi Pustaka Genom Menggunakan Plasmid 18
Konstruksi Pustaka 40 kb Menggunakan Fosmid 20
Penapisan Klon Rekombinan 21
Analisis Fosmid pCC1FOSTM Rekombinan 23
Analisis Sekuensing 24
5 KESIMPULAN DAN SARAN 26
Kesimpulan 26
Saran 26
DAFTAR PUSTAKA 27
LAMPIRAN 31
DAFTAR TABEL
1 Beberapa primer yang digunakan dalam penelitian ini 10 2 Uji aktivitas enzim transglutaminase dari isolat terpilih 14
DAFTAR GAMBAR
1 Mekanisme reaksi enzim TGase 3
2 Struktur kristal 3 dimensi MTGase 4
3 Diagram alur penelitian 8
4 Peta CopyControl fosmid pCC1FOSTM vektor 9
5 Peta plasmid pJET1.2/blunt dan pGEMT-Easy 10
6 Hasil amplifikasi gen parsial MTGase isolat terpilih 15 7 Pensejajaran sekuen asam amino MTGase isolat TTA 02 SDS 14 dengan
MTGase dari kelompok bakteri lain 16
8 Hasil Amplifikasi gen 16S rRNA isolat TTA 02 SDS 14 16 9 Konstruksi pohon filogenetik isolat TTA 02 SDS 14 dengan beberapa
bakteri lain sebagai outgroup 18
10 Konstruksi pustaka genom dengan plasmid pJET1.2/blunt 19 11 Konstruksi pustaka genom dengan DNA sisipan pada fosmid pCC1FOSTM 20
12 Hasil seleksi fragmen ~40 kb 21
13 Hasil transformasi E. coli EPI300-T1 dengan hasil ligasi antara
fragmen ~40 kb dan pCC1FOSTM 22
14 Penapisan pustaka genom per pool 96-well microplate dengan
primer SDS 14F/14R 23
15 Hasil penapisan klon yang mengandung gen MTGase pada pool F
dan pool H 23
16 Hasil pemotongan fosmid pCC1FOSTM rekombinan 24 17 Analisis sekuen gen parsial MTGase pada vektor pGEMT-MTG1 26
DAFTAR LAMPIRAN
1 PENDAHULUAN
Latar Belakang
Peran mikroorganisme dalam menghasilkan berbagai enzim yang bermanfaat dalam kehidupan manusia telah dikenal cukup luas dan pengembangannya terus berlangsung sampai saat ini. Penggalian potensi mikroorganisme yang mampu menghasilkan enzim yang bermanfaat dalam kehidupan manusia (enzyme bioprospecting) merupakan salah satu kegiatan yang penting dalam penelitian dibidang mikrobiologi dan enzimologi (Oetomo 2012). Salah satunya adalah bioprospecting terhadap mikroorganisme penghasil enzim transglutaminase terutama untuk produk pangan. MTGase telah banyak digunakan untuk meningkatkan kualitas tekstur beberapa makanan, seperti kamaboko, ham, sosis, tahu, dan mie (Motoki dan Seguro 1998).
Enzim transglutaminase (TGase, E.C. 2.3.2.13) merupakan enzim yang mengkatalisis reaksi perpindahan gugus asil antara residu glutamin (Gln) yang berfungsi sebagai donor asil dan residu lisin (Lys) sebagai aseptor yang membentuk ikatan silang (crosslinking) ε-( -glutamyl) lisin isopeptida yang menghasilkan ikatan kovalen inter atau intramolekuler yang berikatan silang dengan protein makanan (Griffin et al. 2002). TGase tersebar luas di alam dan telah dipelajari secara ekstensif pada hewan. Aktivitas TGase pada hewan tergantung Ca+2 dan terlibat dalam berbagai fungsi biologis mulai dari pembekuan darah hingga diferensiasi sel (Aeshclimann et al. 1994). TGase dari mikroba dikenal dengan nama MTGase.. Sumbernya yang langka dan rumit dalam proses pemisahan serta pemurniannya menjadikan enzim ini sangat mahal dengan harga sekitar US$ 80/unit (Zhu et al. 1995). Salah satu kelebihan MTGase dibandingkan dengan enzim transglutaminase yang berasal dari hewan adalah aktivitas enzim MTGase tidak bergantung pada ion Ca+2 dan biaya produksi lebih rendah karena kemudahan dalam produksi dan purifikasinya (Langston et al. 2006).
Aktivitas MTGase banyak dihasilkan oleh genus Streptoverticillium spp (Wu et al. 1996), serta beberapa spesies bakteri seperti Candida albicans (Ruis et al. 1995), Bacillus subtilis (Kobayashi et al. 1998) Streptomyces platensis dan Streptomyces lividans. (Lin et al. 2006). Beberapa penelitian lain melaporkan Streptomyces yang menghasilkan MTGase dengan aktivitas yang tinggi seperti Streptoverticilliummobaraense (Kikuchi et al. 2003), Streptomyces cinnamoneum (Duran et al. 1998), Streptomyces ladakanum (Ashie dan Lanier 2000), Streptomyces fradiae (Liu et al. 2007), Streptomyces hygroscopicus (Cui et al. 2007). Meskipun beberapa genus Pseudomonas juga dilaporkan menghasilkan MTGase (Bech et al. 2002), namun sampai saat ini genus Streptomyces masih merupakan yang dominan.
2
kloning dan ekspresi gen penyandi MTGase sangat diperlukan. Pendekatan rekayasa genetika memberikan kelebihan pada produksi skala industri yaitu diantaranya dapat dilakukan overekspresi dengan menginduksi dengan induser, menambahkan fusi protein untuk proses pemurnian dan dapat menghindari bakteri patogen sebagai sumber enzim dengan mengkloning gen penyandi protein target ke dalam sel inang. Lin et al. (2004, 2006) telah berhasil melakukan kloning dan mengekspresikan gen MTGase yang berasal dari Streptomyces platensis dan Streptoverticillium ladakanum ke dalam sel inang Streptomyces lividans menggunakan vektor pIJ702 dengan promoter melC dengan berat molekul 38 kDa. Akan tetapi hasil yang dilaporkan oleh Lin et al. (2006) menunjukkan bahwa ekspresi gen MTGase diregulasi oleh promoter indigenus dari gen MTGase Streptomyces lividans serta berhasil memproduksi enzim MTGase rekombinan dalam skala pabrik percontohan (pilot plant) dan proses pemurnian yang dilakukan secara efisien.
Meskipun enzim ini telah diproduksi secara komersial, namun penelitian yang berkaitan dengan penapisan mikroba penghasil MTGase, medium produksi dan aplikasi MTGase masih banyak dilakukan guna mendapatkan keunggulan tertentu. Medium produksi enzim MTGase umumnya mengandung ekstrak khamir, pepton, dan sodium kaseinat sebagai nutrisi bagi mikroorganisme penghasil. Modifikasi telah banyak dilakukan untuk mendapatkan komponen media yang murah serta aktivitas enzim yang baik seperti pemanfaatan hidrolisat sorgum jerami (Tellez-Luiz et al. 2004), limbah cair tahu dan hidrolisat tepung tapioka (Fawzya et al. 2011) namun belum memberikan hasil yang maksimal. Untuk itu pendekatan teknik molekular dengan metode PCR merupakan suatu alternatif penapisan mikroba penghasil MTGase dengan mencari gen penyandi enzim MTGase dari bakteri tanah khususnya dari spesies Streptomyces spp yang kemudian dikloning dan diekspresikan ke dalam Escherichia coli sehingga menghasilkan MTGase rekombinan yang dapat diproduksi dalam skala industri.
Tujuan Penelitian
Penelitian ini bertujuan mendapatkan Streptomyces spp yang menghasilkan enzim Microbial Transglutaminase (MTGase) dan mengklon fragmen DNA yang mengandung gen penyandi MTGase melalui pendekatan konstruksi pustaka genom.
Manfaat Penelitian
3
2 TINJAUAN PUSTAKA
Enzim Microbial Transglutaminase (MTGase)
Transglutaminase (TGase) merupakan enzim yang termasuk ke dalam kelompok transferase, memiliki peran yang sangat penting dalam pembentukan gel dan elastisitas produk berbahan dasar daging lumat surimi. Enzim ini mengkatalis polimerisasi dan membentuk ikatan silang (crosslinking) ε-( glutamyl) lisin antara residu glutamin (Gln) dan lisin (Lys) pada protein bahan pangan (Motoki dan Seguro 1998). Mekanisme kerja enzim transglutaminase adalah mengkatalisis reaksi pengikatan asam amino-asam amino yang ditambahkan pada molekul protein. Hal ini ditunjukkan oleh terikatnya metionin pada berbagai protein pangan. Kandungan metionin pada kasein, protein-protein kedelai dan gluten meningkat setelah dilakukan pengikatan silang menggunakan TGase (Nielsen et al. 1995). Salah satu sumber penghasil enzim TGase adalah mikroorganisme, sehingga enzim yang dihasilkan biasanya disebut dengan Microbial Transglutaminase (MTGase). Pada umumnya MTGase berasal dari kelompok Actinomycetes seperti dari genus Streptoverticillium dan Streptomyces (Fawzya et al. 2011).
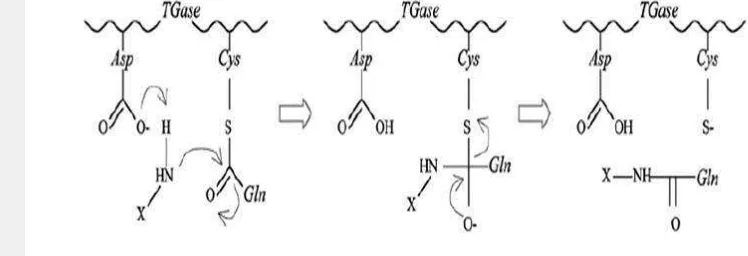
Enzim TGase mempunyai nama sistematika yaitu amin -glutamiltransferase yang termasuk dalam kelas enzim transferase (E.C.2), asiltransferase (E.C.2.3), aminoasiltransferase (E.C.2.3.2), protein glutamin -glutamiltransferase dan mempunyai nama alternatif yaitu fibrinoligase. Mekanisme reaksi TGase diawali dengan amina nukleofil (X-NH2) menyerang -C Gln yang terasilasi -Cys diikuti proses transfer H+ dari S-C(O)’’NH2-X yang diduga Asp sebagai asam atau basa. Oksianion yang dihasilkan mengalami penataan ulang dan menyebabkan deasilasi dari residu Cys. Pelepasan produk isopeptida atau Gln -teramidasi dan pembentukan kembali Cys thiolat disajikan pada Gambar 1 (Motoki dan Seguro 1998).
4
A B
Gambar 2 Struktur kristal 3 dimensi MTGase. (A) Struktur kuartener (B) sisi aktif enzim MTGase (Kashiwagi et al. 2002)
Transglutaminase tersebar secara luas pada mikroorganisme eukariot dan prokariot. Berdasarkan sumbernya, menurut Greenberg et al. (1991) enzim transglutaminase dibagi kedalam dua golongan. Golongan pertama yaitu mamalia transglutaminase (TGase), aktivitas enzim ini ditemukan tersebar secara luas dalam plasma, jaringan dan cairan ekstraseluler. Enzim transglutaminase tersebut digolongkan ke dalam 5 golongan, yaitu :
a) Faktor XIII (plasma dan plasenta) yang tersusun dari rantai a2 dan rantai b2 (a2b2) berasosiasi secara kovalen. Berbeda dengan yang lain enzim ini diaktifkan oleh trombin selama koagulasi darah dan memiliki berat molekul 360 kDa.
b) Transglutaminase jaringan (TGc) ditemukan pada hati babi guinea, makrofag tikus, dan sel endothelia manusia, enzim ini memiliki berat molekul 77 kDa.
c) Transglutaminase keratinosit (TGk) ditemukan pada kulit, kuku dan rambut, enzim ini mengandung ester atau tioester palmitat dan miristat yang bertanggung jawab untuk melekatnya membran dan berat molekul dari enzim ini 90 kDa
d) Transglutaminase epidermis yang struktur subunitnya bersifat monomer dan memiliki aktivitas protease dengan berat molekul 80 kDa
e) Transglutaminase prostata yang struktur subunitnya bersifat homodimetrik dengan berat molekul 150 kDa, namun enzim ini tidak memiliki aktivitas protease.
5
Karakteristik Streptomyces sp
Sekitar 90 % Actinomycetes yang disolasi dari tanah merupakan genus dari Streptomyces. Bakteri ini merupakan kelompok bakteri gram positif, yang secara taksonomi merupakan salah satu genus dari filum Actinobacteria, ordo Actinomycetales, dan famili Streptomycetaceae. Streptomyces bersifat toleran yang dapat hidup pada suhu 45 – 55 °C. Hampir semua Streptomyces bersifat aerobik dan memiliki kemampuan untuk mendegradasi material yang sulit diuraikan seperti lignin, kitin, pektin dan komplek aromatik (Paul dan Clark 1996). Streptomyces dapat memproduksi senyawa volatil yaitu geosmin yang memiliki bau khas pada tanah. Bakteri ini juga memiliki kemampuan menghasilkan senyawa–senyawa metabolit seperti enzim, inhibitor, biopigmen dan imunomodifer (Hayawaka 2003)
Streptomyces memiliki ukuran dengan diameter antara 0.5 – 1.0 µm dengan ciri-ciri ialah pleomorfisme sel-selnya dan kecendrungan membentuk filamen (hifa) bercabang. Pada beberapa spesies, hifa-hifa itu bersatu membentuk miselium. Streptomyces bersifat aerobik yaitu tumbuh pada kondisi lingkungan yang banyak oksigen atau konsentrasi karbondioksida sedikit, serta mampu tumbuh optimum pada temperatur 25 °C dan pH 6.5 - 8.5. Bakteri ini juga bersifat kemoorganotrof dan mesofilik. Streptomyces kebanyakan hidup saprofit pada tanah, dan juga ditemukan pada tumbuhan yang membusuk. Reproduksi bakteri ini adalah dengan miselium aerial atau germinasi spora yang memiliki permukaan halus sampai sedikit kasar (Madigan et al. 2012)
Streptomyces sp mempunyai ukuran genom 8 mb dengan kandungan basa G+C yang cukup tinggi yaitu 73 % (Kieser et al. 1992). Bakteri ini telah banyak dipelajari dan digunakan untuk industri terutama kerena kemampuannya memproduksi sejumlah metabolit sekunder seperti antibiotik dan antikanker.
Teknologi DNA Rekombinan
Transformasi adalah memasukkan molekul DNA rekombinan ke dalam sel hidup, salah satunya bakteri yang kemudian tumbuh dan membelah untuk memperbanyak klon. Secara umum, terdapat dua tujuan dari transformasi DNA rekombinan pada bakteri. Tujuan yang pertama adalah memproduksi DNA rekombinan dalam jumlah yang besar dari material awal yang sedikit. Tujuan kedua adalah agar jumlah produksi DNA rekombinan memenuhi untuk tahap pemurnian. Pemurnian dari DNA rekombinan penting sekali dilakukan, karena dalam hasil ligasi selain terdapat vektor plasmid dan DNA sisipan yang telah terligasi terdapat pula molekul vektor tidak terligasi, molekul DNA tidak terligasi, molekul vektor yang mengalami resirkularisasi tanpa DNA sisipan dan DNA rekombinan yang membawa fragmen DNA yang salah (Brown 2006).
6
temperatur dingin (-80ºC). Penyimpanan pada suhu ruang akan menurunkan kualitas sel kompeten (Moelhard 2007)
Salah satu vektor kloning yang sering digunakan dalam konstruksi pustaka genom adalah pJET1.2/blunt. Vektor ini merupakan plasmid yang bersifat linear yang dapat menerima sisipan dari 6 kb sampai 10 kb. Pada ujung 5’ vektor mengandung gugus fosforil sehingga forforilasi primer tidak diperlukan. Ujung γ’dA-overhangs dihasilkan dengan menggunakan Taq DNA polimerase dengan menggunakan blunt repairing enzymes. Produk PCR blunt –end dihasilkan oleh pembacaan DNA polimerase yang dapat langsung diligasikan ke dalam vektor dalam waktu 5 menit. Vektor pJET1.2/blunt mengandung lethal gene (gen mematikan) yang dapat membunuh host jika tersisipkan dalam host bakteri, tetapi apabila vektor ini tersisipkan oleh fragment DNA (gen) maka lethal gen tersebut menjadi tidak aktif sehingga dalam proses seleksi sel transforman yang mengandung fragmen sisipan saja yang dapat hidup. (Thermoscientific, Cat.No. #K1231)
CopyControlTM Fosmid Library pCC1FOSTM Vektor (Epicentre) merupakan salah satu vektor yang digunakan untuk pustaka genom. Vektor ini memiliki ukuran 8.1 kb yang bersifat linear yang dapat menerima sisipan 40 kb fragmen DNA. pCC1FOSTM membawa gen resisten kloramfenikol sebagai seleksi antibiotik dan gen LacZ sebagai seleksi biru putih. Keunggulan menggunakan vektor pCC1FOSTM adalah DNA yang diperoleh dapat diinduksi untuk mendapatkan high copy number dalam penggandaan klon rekombinan dari single copy menjadi 10–200 copies per selnya, sehingga meningkat DNA fosmid untuk sekuensing, sidik jari (fingerprint), subkloning, in-vitro transkripsi, dan aplikasi lainnya. (Epicentre.Cat.No.CCFOS110).
Pustaka Genom
Pustaka genom (genomic library) adalah kumpulan klon rekombinan dalam bentuk plasmid, phage atau kosmid yang membawa fragmen molekul DNA tertentu secara independen, berukuran tertentu dan merupakan kumpulan keseluruhan informasi genetik suatu organisme (Winnaker et al. 1987). Suatu gen spesifik dalam kumpulan klon rekombinan dipengaruhi oleh panjang fragmen DNA dan tingkat kompleksitas genom. Semakin tinggi tingkat suatu spesies, maka semakin kompleks genomnya. Klon rekombinan yang membawa pustaka genom dapat diukur menjadi fragmen kecil, biasanya 500 bp sampai 1000 bp untuk memudahkan dalam proses sekuensing (Muse dan Gibson 2004). Beberapa tahap penting yang dapat menentukan keberhasilan pembuatan pustaka genom adalah penyiapan fragmen DNA, penyiapan vektor yang sesuai, reaksi ligasi, dan perbanyakan DNA dalam sel inang (Losick et al. 2008).
7 dikarenakan keberadaan dari vektor yang membawa gen resisten antiobiotik. Kebanyakan vektor memiliki sekuen origin of replication (ORI), sehingga vektor dapat memperbanyak diri dalam sel secara independen. Plasmid sebagai vektor kloning memiliki ukuran kurang dari 10 kb. Sebagai vektor kloning, plasmid harus memiliki daerah penyisipan bagi DNA. Daerah ini disebut dengan situs enzim restriksi yang biasanya membentuk multiple cloning site. Hal yang penting pula bagi plasmid sebagai vektor kloning adalah angka penggandaan atau copy number (Brown 2006).
8
3 METODE PENELITIAN
Kerangka Penelitian
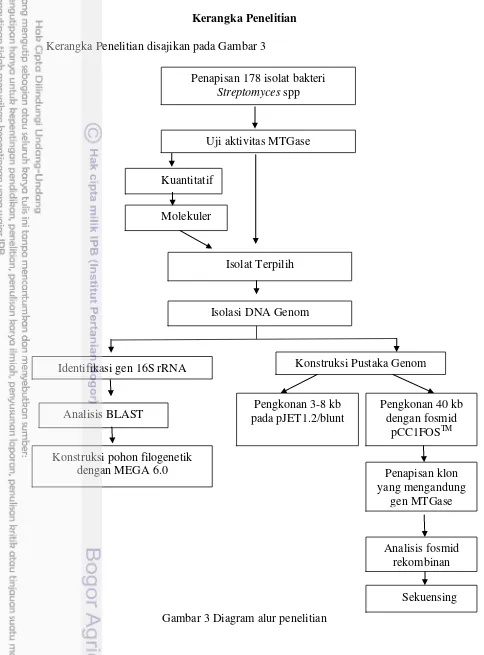
Kerangka Penelitian disajikan pada Gambar 3
Gambar 3 Diagram alur penelitian Penapisan 178 isolat bakteri
Streptomyces spp
Uji aktivitas MTGase
Isolat Terpilih Molekuler
Kuantitatif
Identifikasi gen 16S rRNA Konstruksi Pustaka Genom
Konstruksi pohon filogenetik dengan MEGA 6.0
Pengkonan 3-8 kb pada pJET1.2/blunt Isolasi DNA Genom
Pengkonan 40 kb dengan fosmid
pCC1FOSTM Analisis BLAST
Penapisan klon yang mengandung
gen MTGase
Analisis fosmid rekombinan
9
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan pada bulan Oktober 2014 – Desember 2015 di Laboratorium Bioteknologi Pusat Penelitian dan Pengembangan Daya Saing Produk dan Bioteknologi Kelautan dan Perikanan (P3DSBKP– KKP) Jakarta.
Bahan
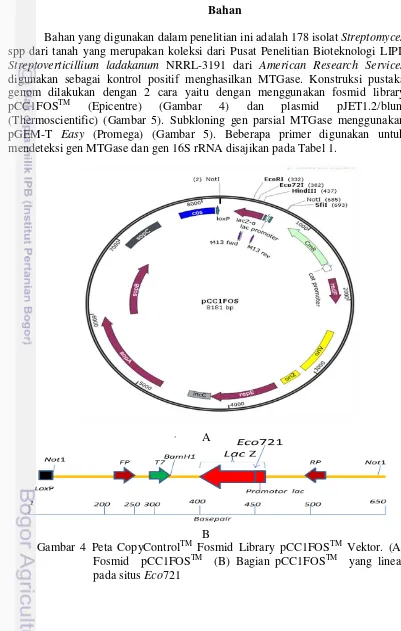
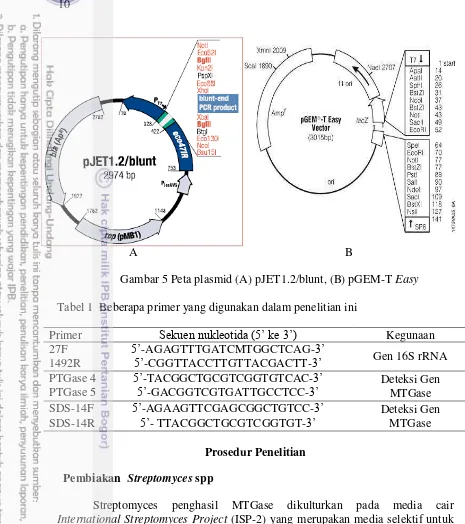
Bahan yang digunakan dalam penelitian ini adalah 178 isolat Streptomyces spp dari tanah yang merupakan koleksi dari Pusat Penelitian Bioteknologi LIPI, Streptoverticillium ladakanum NRRL-3191 dari American Research Services digunakan sebagai kontrol positif menghasilkan MTGase. Konstruksi pustaka genom dilakukan dengan 2 cara yaitu dengan menggunakan fosmid library pCC1FOSTM (Epicentre) (Gambar 4) dan plasmid pJET1.2/blunt (Thermoscientific) (Gambar 5). Subkloning gen parsial MTGase menggunakan pGEM-T Easy (Promega) (Gambar 5). Beberapa primer digunakan untuk mendeteksi gen MTGase dan gen 16S rRNA disajikan pada Tabel 1.
Gambar 4 Peta CopyControlTM Fosmid Library pCC1FOSTM Vektor. (A) Fosmid pCC1FOSTM (B) Bagian pCC1FOSTM yang linear pada situs Eco721
A
10
A B
Gambar 5 Peta plasmid (A) pJET1.2/blunt, (B) pGEM-T Easy Tabel 1 Beberapa primer yang digunakan dalam penelitian ini
Primer Sekuen nukleotida (5’ ke γ’) Kegunaan
27F 5’-AGAGTTTGATCMTGGCTCAG-γ’
Gen 16S rRNA
1492R 5’-CGGTTACCTTGTTACGACTT-γ’
PTGase 4 5’-TACGGCTGCGTCGGTGTCAC-γ’ Deteksi Gen
MTGase PTGase 5 5’-GACGGTCGTGATTGCCTCC-γ’
SDS-14F 5’-AGAAGTTCGAGCGGCTGTCC-γ’ Deteksi Gen
MTGase SDS-14R 5’- TTACGGCTGCGTCGGTGT-γ’
Prosedur Penelitian
Pembiakan Streptomyces spp
Streptomyces penghasil MTGase dikulturkan pada media cair International Streptomyces Project (ISP-2) yang merupakan media selektif untuk Streptomyces dengan komposisi yaitu malt ekstrak 1%, yeast ekstrak 0.4% dan glukosa 0.4%. Kultivasi bakteri dilakukan dengan menginokulasikan 1 koloni bakteri Streptomyces spp dari media padat ke dalam 15 mL media cair ISP, kemudian biakan diinkubasi pada suhu 30 0C di dalam inkubator bergoyang dengan kecepatan 125 rpm selama 5–7 hari.
Penapisan Streptomyces spp Penghasil MTGase
11 L–glutaminilglycine (CBZ). Satu unit aktivitas didefinisikan sebagai jumlah pembentukan 1 µmol hidroksamat dalam waktu 1 menit pada suhu 37 °C. Sebanyak 1500 µL kultur bakteri dimasukan ke dalam tabung mikro 2 ml dan disentrifugasi dengan kecepatan 8000 rpm. Supernatan diambil 100 µL kemudian direaksikan dengan reagen A (200 µL 0.1 M bufer sitrat pH 6, 25 µL 2.0 M hydroxylamine, 25 µL glutathione dan 75 µL 0.1 M carbobenzoxyl-L-glutaminylglycine/CBZ). Selanjutnya larutan diinkubasi pada suhu 37 °C selama 60 menit dan dihentikan dengan penambahan 425 µ L reagen B (15% TCA yang mengandung 5% FeCl3). Selanjutnya hasil reaksi disentrifugasi (4500 rpm selama 15 menit), kemudian diinkubasi pada suhu 37 °C selama 30 menit dan supernatan dibaca absorbansinya dengan spektrofotometer pada = 5β5 nm. Untuk kontrol negatif, supernatan dari reagen A tanpa larutan dari bakteri ditambahkan setelah larutan direaksikan dengan reagen B. Kontrol positif menggunakan bakteri Streptoverticillium ladakanum NRRL 3191. Kurva standar dibuat menggunakan L-glutamic acid- -monohydroxamat acid sebagai standar.
Amplifikasi Gen MTGase dengan Metode PCR
Isolat yang menghasilkan MTGase berdasarkan uji aktivitas, kemudian ditapis dengan teknik Polymerase Chain Reaction (PCR). Untuk itu, bakteri dibiakan dalam media ISP-2 pada inkubator bergoyang dengan 125 rpmselama 24 jam pada suhu 37°C. Sebanyak 1.5 mL biakan bakteri ditransfer ke dalam tabung mikro 1,5 mL dan disentrifugasi pada kecepatan 8000 rpm selama 10 menit. Isolasi DNA dilakukan dengan mengikuti prosedur standar Presto mini gDNA Bacteria Kit (Geneaid). Amplifikasi gen MTGase menggunakan primer PTGase-4 dan PTGase-5 dengan 25 µL reaksi PCR yang mengandung 1 µL DNA genom 30 µg/mL , 12.5 µL PCR Taq Green Master Mix 2x (Thermo Scientific), 1 µL selama 40 menit. Setelah itu gel didiamkan dalam buffer TAE 1x yang telah diberi Cyber Gold 1x selama 30 menit. DNA di dalam gel divisualisasikan dengan UV transiluminator BIORAD dan didokumentasikan dengan Gel –Doc apparatus (Biometra).
Identifikasi Isolat Terpilih dengan 16S rRNA
12
dan pemanjangan 72 0C selama 1 menit. Siklus PCR dilakukan sebanyak 30 siklus, dan pasca-PCR dilakukan pada suhu 72 0C selama 7 menit dan diisimpan pada suhu 4 0C. Hasil PCR disekuensing di tempat Genetika Science, Malaysia menggunakan Genetic Analyzer dan hasil sekuen dianalisis menggunakan software bioedit, kemudian disejajarkan dengan sekuen 16S rRNA data GeneBank pada situs (http://www.ncbi.nlm.nih.gov) dengan menggunakan perangkat lunak BLASTn (Basic Local Alignmet Search Tools for nucleotide) (Altschul et al. 1990). Analisis kekerabatan terdekat dengan pohon filogenetik menggunakan MEGA6.0 (Tamura et al. 2013).
Konstruksi Pustaka Genom dengan Plasmid pJET1.2/blunt
DNA genom dipotong secara acak dengan mengunakan enzim restriksi BglII (NEB). Ke dalam tabung mikro secara berurutan ditambahkan 68 µL Extraction Kit (Thermo Scientific). DNA kemudian ditambahkan dengan DNA blunting enzymes (NEB) untuk mendapatkan kedua ujung genom secara tumpul (blunt).
Fragmen DNA disisipkan ke dalam vektor pJET1.2/blunt (Thermo scientific) menggunakan enzim T4 ligase (Thermo scientific), pada suhu 220C selama 12 jam. Hasil ligasi dimasukan ke dalam sel kompeten E. coli TOP10 (Invitrogen) dengan metode elektroporasi berdasarkan Sambrook dan Rusell (2001). Hasil transformasi ditumbuhkan di media agar LB yang mengandung ampisilin (100 g/mL) selama semalam pada suhu 37 0C. Koloni yang tumbuh dipindahkan pada media LB cair yang mengandung ampisilin (50 g/mL) pada 96-well microplate untuk dipilih yang mengandung gen MTGase. Masing–masing koloni yang tumbuh dengan baik dalam 96-well microplate diamplifikasi dengan PCR menggunakan primer SDS 14-F/SDS 14-R. Koloni yang positif membawa gen MTGase disekuen untuk mendapatkan fragmen DNA yang mengandung gen utuh hinggamemperolehdaerah open reading frame (ORF).
Konstruksi dan Penapisan Pustaka Genom dengan CopyControlTM Fosmid Library KIT (pCC1FOSTM Vektor)
Isolasi Fragmen ~40 kb yang Berujung Tumpul
13 Ukuran ~40 kb dipilih berdasarkan marker fosmid control DNA yang ditandai pita tunggal dengan ukuran 36 kb. Elektroforesis dilakukan dengan voltase 70 volt selama 15 menit pertama kemudian voltase diturunkan menjadi 40 volt selama 6 jam. Gel yang mengandung fragmen ~40 kb DNA dipotong dan DNA di gel dipurifikasi dengan menggunakan Epicentre’s GELaseTM agarose (Epicentre)dan peqGOLD Gel Exstraction Kit (peqLab).
Konstruksi Pustaka Genom ~40 kb
Fragmen ~40 kb diligasikan ke dalam vektor pCC1FOSTM dengan total volume reaksi 10 µL dengan mencampurkan 1 µL 10x Fast-Link Ligation Buffer, 1 µL 10 mM ATP, 1 µL pCC1FOSTM 0.5 µg/µL , 6 µL DNA ~40 kb 30 µg/mL dan 1 µL Fast-Link DNA Ligase 2 U/µL. Reaksi diinkubasi selama 4 jam pada temperatur ruang kemudian diinaktifkan pada suhu 70 0C selama 10 menit. Transformasi dilakukan dengan elektroforasi sesuai metode Sambrook dan Rusell (2001) dengan menggunakan 2 µL hasil ligasi dan 75 µL sel E. coli EPI300-T1R. Hasil elektroforasi ditambah dengan 1 mL media LB cair dan dikultur selama 1 jam pada inkubator bergoyang dengan suhu 37 0C. Kemudian bakteri disebar dalam media LB padat yang mengandung kloramfenikol (Cm) 12.5 µg/mL, Xgal 20 mg/mL dan IPTG 0.1 M dan diinkubasi selama 24 jam pada suhu 37 0C.
Penapisan Klon yang Mengandung Gen MTGase dengan Metode 96-well microplate
Semua koloni yang berwarna putih ditumbuhkan di dalam media cair LB+Cm (12.5 g/mL) pada 96-well microplate selama 16 jam pada suhu 37 0C. Sebanyak 10 L kultur pada masing–masing sumuran (well) digabungkan berdasarkan baris pada sumuran (pool). Kemudian gen MTGase yang terdapat di dalam biakan bakteri diamplifikasi dengan PCR menggunakan primer SDS-14F dan SDS-14R dengan 25 µL reaksi PCR yang mengandung 1 µL kultur sel E coli EPI300-T1R transforman, 12,5 µL KAPA Taq Hot Start Master Mix 2x (KAPA Biosystem), 1 µL masing–masing primer 10 µM, 9,5 µL ddH2O.
Analisis Fosmid pCC1FOSTM Rekombian
14
4 HASIL DAN PEMBAHASAN
Penapisan Streptomyces spp penghasil MTGase
Streptomyces spp merupakan kelompok bakteri yang dominan menghasilkan enzim MTGase (Wu et al. 1996). Berdasarkan pengukuran aktivitas, dari 178 isolat Streptomyces spp (Lampiran 1), terdapat 10 isolat berpotensi menghasilkan enzim transglutaminase dengan ditandai aktivitas enzim yang cukup tinggi. Eshra et al (2015) melaporkan penapisan Actinomycetes yang menghasilkan MTGase dari 21 jenis tanah yang berbeda diperoleh 15 isolat yang potensial menghasilkan MTGase berdasarkan pengujian aktivitasnya. Bakteri Streptoverticillium ladakanum NRRL-3191 dari American Research Services digunakan sebagai kontrol penghasil MTGase (Tabel 2). Aktivitas MTGase ditunjukkan dengan adanya perubahan warna kuning pada larutan menjadi warna merah kecoklatan dengan penambahan indikator reagen B (15% TCA yang mengandung 5% FeCl3). Hal ini dikarenakan terbentuknya komplek ion logam hasil reaksi antara asam hidroxamat dengan TCA sehingga terjadi perubahan warna dari kuning menjadi merah muda (Bourneouw et al. 2012). Aktivitas enzim didefinisikan sebagai satu unit enzim yang diperlukan untuk menghasilkan produk 1 µmol asam hidroxamat per menit pada kondisi tertentu. Penentuan aktivitas MTGase dilakukan dengan menggunakan metode kolorimetri prosedur hidroxamat dengan menggunakan kurva standar asam L-glutamat -monohidroxamat (Lampiran 2)
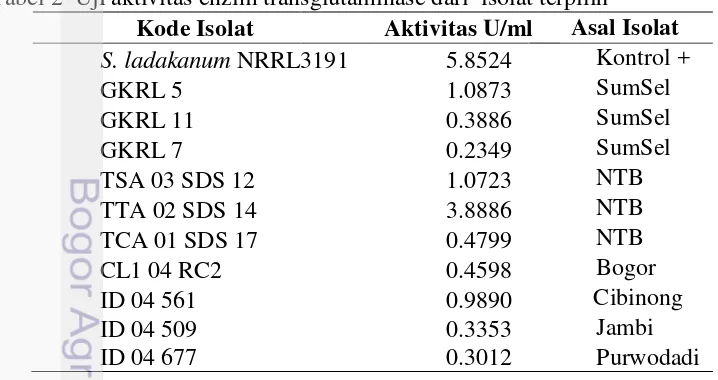
Isolat TTA 02 SDS 14 mempunyai aktivitas transglutaminase yang lebih tinggi dibanding dengan isolat lainnya yaitu sebesar 3.3886 U/mL yang tidak jauh berbeda dengan S. ladakanum NRRL-3191 (Tabel 2). Isolat TTA 02 SDS 14 menghasilkan enzim transglutaminase secara ektraseluler pada suhu 25 0C dengan pH media 6.0 selama 7 hari. Pengukuran aktivitas enzim MTGase menggunakan buffer sitrat pada pH 6. Kondisi pH rendah atau pH tinggi dapat menyebabkan terjadinya proses denaturasi enzim dan ini akan mengakibatkan menurunnya aktivitas enzim (Hames dan Hoper 2005). Isolat TTA 02 SDS 14 ini diisolasi dari savana Gunung Tambora, Dompu Nusa Tenggara Barat (NTB).
Tabel 2 Uji aktivitas enzim transglutaminase dari isolat terpilih
Kode Isolat Aktivitas U/ml Asal Isolat S. ladakanum NRRL3191 5.8524 Kontrol +
15 Menurunnya aktivitas enzim karena perubahan pH larutan yang disebabkan oleh berubahnya keadaan ion enzim dan ion substrat. Perubahan ini dapat terjadi pada residu asam amino yang berfungsi sebagai katalitik pengikat substrat maupun mempertahankan struktur tersier dan kuartetener enzim yang aktif. Macedo et al. (2011) melaporkan bahwa pH optimum untuk aktivitas MTGase dari Streptomyces sp adalah pH 6 – 6.5 dengan menggunakan buffer sitrat. pH optimum tersebut merupakan titik optimum enzim MTGase untuk bekerja secara optimal melakukan reaksi katalisis CBZ-gln-gly dan hidrosilamin membentuk hidrosamat. Karakteristik pH optimum setiap spesies berbeda-beda dalam menghasilkan MTGase. Menurut Suzuki et al. (2000) pH optimum pada Bacillus subtilis adalah pH 7.5 – 8.2 dengan buffer Tris-HCl serta Warratao dan Yongsawatdigul (2005) melaporkan bahwa pH optimum aktivitas transglutaminase pada mamalia dan ikan nila (Oreochromis niloticus) adalah pH 7–7.5 dengan buffer Tris-HCl.
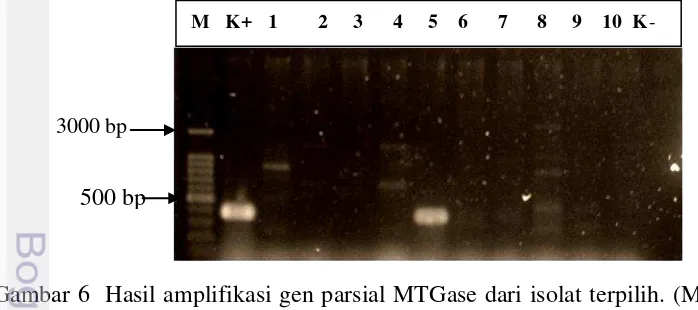
Kondisi aktivitas transglutaminase ini menjadi acuan untuk pengujian secara molekuler untuk memastikan bahwa kesepuluh isolat tersebut benar-benar memiliki gen MTGase. Konfirmasi bahwa 10 isolat tersebut mengandung gen penyandi MTGase dilakukan dengan PCR (Polymerase Chain Reaction) menggunakan primer PTGase 4 dan PTGase 5. Hasil PCR menunjukkan bahwa isolat TTA 02 SDS 14 mengandung pita DNA berukuran 470 bp (Gambar 6). Ukuran ini sesuai dengan sebagian gen MTGase yang diamplifikasi dengan primer tersebut. Gen MTGase memiliki panjang open reading frame (ORF) 1254 bp yang menyandi 418 asam amino (Lin et al. 2006). Hal ini juga diperkuat dengan hasil sekuen gen MTGase (Lampiran 3) dari isolat TTA 02 SDS 14 yang memiliki kemiripan dengan gen MTGase dari Streptomyces cinnamoneus dengan homologi sebesar 93 % (Lampiran 4). Analisis pensejajaran sekuens protein dari sebagian gen MTGase dari isolat TTA 02 SDS 14 dengan beberapa gen MTGase dari beberapa kelompok Streptomyces dan bakteri lain juga menunjukkan adanya daerah konservatif yang menandakan adanya persamaan sekuens asam amino yang dimilikinya (Gambar 7). Homologi sekuen asam amino dari beberapa organisme prokariot dan eukariot menunjukkan adanya sekuens yang konservatif pada bagian sisi aktif yang bersifat triad katalitik (Li et al. 2009)
16
conserve region conserve region Gambar 7 Pensejajaran sekuens asam amino MTGase isolat TTA 02 SDS 14
dengan MTGase dari beberapa kelompok bakteri
Identifikasi Isolat TTA 02 SDS 14 Berdasarkan Sekuen 16S rRNA
Isolat TTA 02 SDS 14 diindentifikasi berdasarkan sekuen DNA dari gen penyandi 16S rRNA dengan menggunakan primer 27F dan 1492R. Produk PCR yang dihasilkan adalah fragmen DNA berukuran 1400 bp (Gambar 8). Besarnya ukuran pita yang muncul sesuai dengan ukuran yang diharapkan dari gen 16S rRNA bakteri yaitu antara 1300 – 1500 bp (Marchesi et al. 1998).
Gambar 8. Hasil amplifikasi gen 16S rRNA isolat TTA 02 SDS 14. (1) DNA isolat TTA 02 SDS 14 (M) Marker 100 bp DNA Ledder
Analisis BLAST bertujuan untuk mensejajarkan dan mencocokan hasil sekuensing yang diperoleh dari sampel penelitian dengan data di GenBank. Analisis hasil BLAST tersebut memberikan informasi mengenai bakteri apa yang mempunyai kesamaan dengan urutan DNA sampel sehingga dapat digunakan untuk identifikasi bakteri. Informasi dari hasil BLAST tersebut berupa Score, Query Coverage, E-value dan Maximum identity. Score adalah jumlah keselarasan semua segmen dari urutan database yang cocok dengan urutan nukleotida. Nilai skor menunjukkan keakuratan nilai penjajaran sekuens berupa nukleotida yang tidak diketahui dengan sekuens nukleotida yang terdapat di dalam GenBank. Semakin tinggi nilai skor yang diperoleh maka semakin tinggi tingkat homologi
M 1
1500 bp 10000 bp
1400 bp
17 kedua sekuens. Query coverage adalah persentasi dari panjang nukleotida yang selaras dengan database yang terdapat pada BLAST. Max identity adalah nilai tertinggi dari persentasi identitas atau kecocokan antara sekuen query dengan sekuen database yang tersejajarkan (Altschul et al, 1990).
Berdasarkan hasil sekuen gen 16S rRNA (Lampiran 5) dan hasil identitas pada tingkat genus tetapi berbeda pada tingkat spesies. Nilai E-value merupakan nilai dugaan yang memberikan ukuran statistic yang signifikan terhadap kedua sekuen. Nilai E-value yang semakin tinggi menunjukkan tingkat homologi antara sekuen semakin rendah, sedangkan nilai E-value yang semakin rendah menunjukkan tingkat homologi antar sekuens semakin tinggi. Nilai E-value bernilai 0 (nol) menunjukkan bahwa kedua sekuen tersebut identik (Claverie dan Notredame 2003).
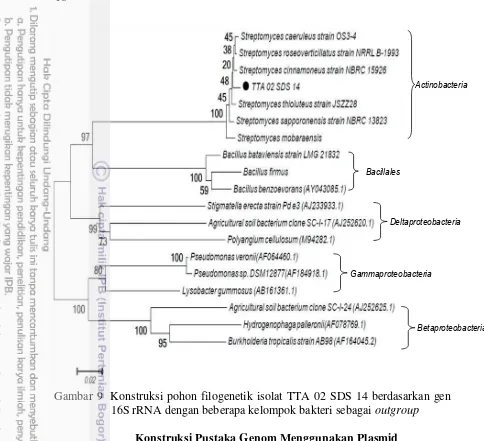
Homologi sekuen 16S rRNA dari masing-masing isolat bakteri dengan sekuens 16S rRNA dari database GenBank dapat diketahui bahwa tidak ada sekuens 16S rRNA bakteri yang identik. Untuk melihat hubungan kekerabatan yang mirip antara isolat TTA 02 SDS 14 dengan beberapa spesies Steptomyces dari hasil BLAST dan beberapa kelompok bakteri lain sebagai kelompok luar dapat disajikan dalam bentuk pohon filogenetik. Hasil analisis pohon filogenetik juga menunjukkan bahwa Isolat TTA 02 SDS 14 memiliki kekerabatan terdekat dengan Steptomyces thioleteus yangditunjukan pada Gambar 9.
Analisis filogenetik menggunakan metode Neighbour Joining yaitu pasangan nukleotida yang mengalami perubahan terkecil diantara sekuen yang telah dibandingkan. Nilai jarak dilambangkan oleh garis skala yang menunjukkan jumlah subtitusi nekleotida untuk tiap posisi sekuen. Nilai jarak sebesar 0.02 pada hasil konstruksi pohon filogenetik menunjukkan rendahnya subtitusi nukleotida pada sekuen 16S rRNA pada masing- masing pengelompokan berdasarkan tingkat devisio dari kelompok Actinobacteria, Bacillales, Deltaproteobacteria, Gammaproteobacteria dan Betaproteobacteria. Pengelompokan ini untuk melihat seberapa jauh dan dekatnya hubungan kekerabatan masing–masing kelompok. Isolat TTA 02 SDS 14 masih tergolong kedalam Actinobacteria. Metode Bootstrap digunakan untuk menguji keakuratan suatu titik cabang pohon filogenetik. Stabilitas pengelompokkan (robustness) diperhitungkan menggunakan bootstrap dengan 1000 kali ulangan. Nilai bootstrap sebesar 48 dari 100 kali untuk titik percabangan antara isolat TTA 02 SDS 14 dengan Streptomyces thioleteous menunjukkan tingkat kepercayaan yang cukup rendah berdasarkan atas kemiripan kedua sekuen gen 16S rRNA pada bakteri tersebut. Rendahnya nilai tersebut kemungkinan disebabkan kurang banyak spesies acuan dari strain yang ada di GenBank.
18
Gambar 9 Konstruksi pohon filogenetik isolat TTA 02 SDS 14 berdasarkan gen 16S rRNA dengan beberapa kelompok bakteri sebagai outgroup
Konstruksi Pustaka Genom Menggunakan Plasmid
Informasi tentang gen MTGase yang lengkap (full-length) sangat diperlukan untuk ekspresi gen tersebut. Tahapan awal adalah membuat konstruksi pustaka genom dengan menyisipkan fragmen DNA 3-8 kb (Gambar 10). Konstruksi pustaka genom pertama kali dilakukan dengan menggunakan plasmid pJET1.2/blunt yang mampu menerima sisipan dengan kisaran ukuran 6-10 kb. Produk PCR fragmen DNA ujung tumpul (blunt –end) dapat langsung diligasikan ke dalam vector pJET1.2/blunt dalam waktu 5 menit. Vektor ini mengandung gen mematikan (lethal gene) yang terletak pada daerah multiple cloning site (MCS). Apabila fragmen DNA asing tidak tersisipkan dalam daerah MCS, gen mematikan akan diekspresikan sehingga menyebabkan koloni E. coli tidak dapat tumbuh. Akan tetapi, apabila vektor ini disisipi oleh fragmen DNA maka lethal gen tersebut menjadi tidak diekspresikan sehingga dalam proses seleksi sel transforman, hanya sel yang mengandung plasmid yang membawa sisipan saja yang dapat hidup. Penggabungan fragmen DNA atau amplikon akan mengganggu gen mematikan, sehingga membuat klon rekombinan hidup (Thermoscientific.Cat.Not.#K1231).
Actinobacteria
Bacillales
Deltaproteobacteria
Gammaproteobacteria
19 Dalam penelitian ini, fragmen DNA dari 3-8 kb dihasilkan dari pemotongan secara acak DNA genom dengan menggunakan enzim restriksi BglII (Gambar 10A). Setelah dimigrasikan di gel agarose, gel yang mengandung fragmen DNA yang berukuran 3-8 kb diisolasi dari gel. DNA diekstraksi dari gel agarose 1% dan kemudian diperbaiki dikedua ujung terminal fragmen DNA dengan menggunakan DNA blunting enzyme agar dapat ligasikan langsung ke dalam pJET1.2/blunt. Inkubasi pada suhu 70 0C selama 10 menit bertujuan untuk menginaktivasi kerja enzim yang dapat menghindari kemungkinan terjadinya ligasi antar molekul sendiri (self ligation). Hasil ligasi diintroduksikan ke dalam E. coli TOP10 dan disebar pada media LB+Amp (100 g/mL), kemudian diinkubasi selama 24 jam pada suhu 37 0C. Faktor efisiensi ligasi juga turut mempengaruhi keberhasilan insersi gen ke dalam plasmid. Efisiensi ligasi dapat dipengaruhi oleh berbagai faktor seperti posisi situs restriksi, jenis situs restriksi dan urutannya serta jenis enzim yang memotong situs restriksi (Dhesi 2005).
Hasil transformasi menunjukkan koloni dapat tumbuh pada media ampicilin namun dalam jumlah klon yang sedikit yaitu hanya 58 klon rekombinan saja (Gambar 10B). Sisipan fragmen DNA 3-8 kb yang terlalu besar membuat efisiensi transformasi menjadi rendah, walaupun vektor pJET1.2/blunt mampu menerima sisipan maksimal 10 kb. Efisiensi transformasi yang rendah biasanya terjadi karena situs integrasi yang tidak efisien, DNA sisipan yang sulit dan kondisi transformasi tidak optimal (Wu dan Letchworth, 2004). Hal ini tentu terlalu sedikit untuk mewakili seluruh genom. Semua koloni kemudian dipindahkan ke medium LB+Amp (100 g/mL) cair dalam 96-well microplate untuk proses skrining menggunakan primer SDS 14F/ SDS 14R. Hasil PCR koloni menunjukkan tidak ada koloni yang positif. Hal ini menunjukkan bahwa DNA sisipan tidak mengandung gen MTGase. Ukuran pustaka plasmid (plasmid library) terlalu kecil, sehingga tidak cukup untuk menemukan klon positif yang membawa gen MTGase. Oleh sebab itu pembuatan pustaka genom dengan menggunakan fosmid pCC1FOSTM yang dapat menerima sisipan ~40 kb perlu dilakukan.
A B
Gambar 10 Konstruksi pustaka genom dengan plasmid pJET1.2/blunt. (A) Hasil elusi pemotongan DNA genom 3 – 8 kb, (B) E. coli TOP10 transforman dalam media seleksi LB+Amp (100 g/mL)
20 Kb 8 Kb 3 Kb
500 bp
20
Konstruksi Pustaka Genom Menggunakan Fosmid
Pembuatan pustaka genom membutuhkan DNA dalam skala besar, sehingga diperlukan modifikasi dalam tahapan proses ekstraksi DNA. DNA genom diisolasi menggunakan metode CTAB ((Piel et al. 2004). Gen MTGase dari mikroba merupakan kelompok gen yang berukuran besar. Lin et al. (2006) mengisolasi fragmen 2,9 kb yang membawa gen MTGase dengan panjang open reading frame (ORF) 1254 bp yang menyandi 418 asam amino yang diekpresikan di dalam S. lividans. Pembuatan pustaka genom ini bertujuan untuk mendapatkan gen MTGase secara lengkap (full-lenght). Konstruksi pustaka genom dibuat dengan menggunakan fosmid pCC1FOSTMvektor yang memiliki panjang 8.1 Kb
dengan besar sisipan 40 kb yang dikemas dalam phage lambda yang ditransfeksikan ke dalam E coli (Gurgui dan Piel, 2010). Vektor pCC1FOSTM menerima sisipan DNA dengan kedua ujung tumpul. Hal ini dikarenakan vektor pCC1FOSTM telah mengalami linearisasi dengan pemotongan menggunakan enzim restriksi Eco721 yang tumpul (blunt) yang telah terfosforilasi (Choi et al. 2012). Situs penyisipan bagi DNA sisipan terletak pada gen LacZ pada sekuen ke-450 yang terdapat pada vektor (Gambar 11).
21 dipurifikasi dengan enzimatis tampak lebih baik dibandingkan dengan yang diperoleh menggunakan kit kolom karena hampir tidak ada smear DNA yang diamati berdasarkan marker fosmid DNA 36 kb dari hasil visualisasi gel agarose 1% (Gambar 12).
Gambar 12 Hasil seleksi fragmen ~40 kb. (1) fragmen ~40 kb ekstrak GELaseTM agarose, (2) fragmen ~40 kb peqGOLD Gel Exstraction Kit (3) seleksi DNA genom dengan LMPagarose 1%, (M) marker fosmid 36 kb
Penapisan Klon Rekombinan
Proses penyisipan fragmen ~40 kb ke dalam fosmid pCC1FOSTM (Epicentre) menggunakan enzim Fast-link DNA ligase pada suhu ruang selama 4 jam. Hasil ligasi dikemas dalam protein pengemas (bakteriofage) dan ditransfeksikan ke dalam E. coli EPI300-T1R.. Hasil transfeksi menunjukkan tidak ada koloni E. coli EPI300-T1R yang tumbuh pada media seleksi. Kemungkinan protein pengemas bakteriofage yang digunakan dalam pekerjaan ini rusak dikarenakan penanganan yang kurang baik selama pengiriman sehingga kondisinya sudah mencair. Dikarenakan pengemasan pCC1FOSTM rekombinan ke dalam fage tidak berhasil, maka pCC1FOSTM rekombinan diintroduksikan langsung ke dalam E. coli EPI300-TIR dengan metode elektroporasi. Berkat adanya outoinduksi dengan induction solution, pCC1FOSTM rekombinan dapat memperbanyak diri menjadi kopi tinggi (high copy number) yang ditandai dengan adanya koloni biru dan putih (Gambar 13). Hasil transformasi menghasilkan ± 700 koloni putih yang membawa sisipan (Gambar 13A). Jumlah koloni ini tergolong sedikit untuk pustaka genom dengan sisipan yang besar, namun sudah cukup mewakili untuk penapisan terhadap klon yang mengandung gen MTGase. Koloni yang mengandung vektor yang membawa sisipan berwarna putih sedangkan yang tidak membawa sisipan berwarna biru. Koloni berwarna biru timbul karena pada vektor terdapat gen lacZ yang menyandi enzim -galaktosidase. Protein ini mengenal X-gal (5-bromo-4-chloro-indolxyl-β -D-galactoside) menjadi 5-bromo-4-chloroindoxyl yang mudah teroksidasi menjadi 5,5'-dibromo-4,4'-dichloro-indigo dengan pigmen warna biru yang tidak larut sedangkan isopropyl β-D-1-thiogalactopyranoside (IPTG) berfungsi sebagai induser yang akan mengaktifkan operon lac sehingga dapat mengekspresikan gen lacZ (Sriram et al. 2011). Bila fragmen DNA sisipan menyisip pada gen lacZ, maka gen lacZ tidak dapat diekspresikan untuk menghasilkan enzim
M 1 2
3 M
36 Kb 36 Kb
22
galaktosidase sehingga koloni E. coli EPI300-T1R berwarna putih dan positif membawa DNA sisipan ~40 kb.
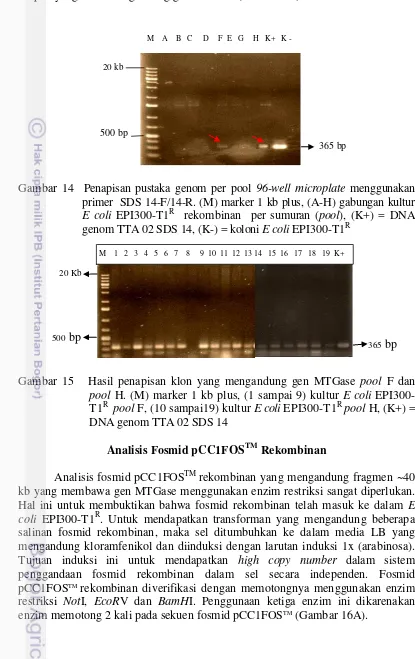
Koloni yang positif membawa sisipan ~40 kb (koloni putih) ditumbuhkan dalam media LB+Cm+IPTG+Xgal sebagai duplikat dalam proses skrining (Gambar 13B). Selanjutnya, koloni ditumbuhkan kembali ke dalam media cair LB+Cm (12.5 g/mL) pada 96-well microplate (Gambar 13C). Sebanyak 10 L kultur pada masing–masing sumuran (well) dan digabungkan berdasarkan baris pada sumuran. Dalam 1 mikroplate terdapat 8 pool (pool A sampai pool H) dan 1 pool dapat mewakili 12 sumuran yang diamplifikasi dengan PCR menggunakan primer SDS 14F/SDS 14R.
A B C
Gambar 13 Hasil transformasi E. coli EPI300-T1R dengan hasil ligasi antara fragmen ~40 kb dan pCC1FOSTM. (A) Koloni transforman dalam media seleksi LB+C+IPTG+Xgal, (B) Duplikat koloni transforman, (C) Kultur koloni dalam media cair LB+Cm untuk penapisan pustaka genom dalam 96-well microplate
Koloni yang tumbuh juga memiliki ketahanan terhadap antibiotik kloramfenikol karena fosmid pCC1FOSTM terdapat gen CAT. Gen ini menyandi enzim kloramfenikol asetil transferase yang menginaktivasi kloramfenikol lewat ikatan secara kovalen dengan satu atau dua grup asetil yang berasal dari asetil-S-koenzim A dengan grup hidroksil pada molekul kloramfenikol. Beberapa peneliti pernah mengungkapkan resistensi terhadap chloramfenikol (Cm) diperantarai oleh enzim yang terletak pada plasmid yang disebut asetil transferase kloramfenikol (CAT). Enzim CAT disandi oleh famili gen CAT yang terdapat dalam bakteri Gram negatif (Nogrady et al. 2005). Aktivitas antimikroba dari kloramfenikol disebabkan oleh adanya gugus nitrofenil dan gugus propandiol pada strukturnya. Perubahan yang terjadi pada kedua gugus ini dapat mempengaruhi aktivitas antimikroba tersebut (Kadam 2008).
Hasil PCR dengan primer SDS 14F/ SDS 14R terhadap semua koloni yang ditumbuhkan di dalam media cair LB+Cm (12.5 g/ml) pada 96-well microplate pada tingkat gabungan sumuran (pool) menunjukkan ada pita yang teramplifikasi dengan ukuran ± 365 bp pada pool F dan H (Gambar 14). Ukuran ini sesuai dengan ukuran kontrol positif yang digunakan yaitu DNA dari isolat TTA 02 SDS 14. Hal ini menjadi acuan untuk penapisan pada masing–masing sumuran dengan menggunakan primer yang sama dan kondisi PCR yang sama. Penapisan pada tingkat sumuran juga menunjukkan bahwa dari semua sumuran yang diamplifikasi dengan PCR juga memiliki ukuran pita yang sama dengan kontrol positif sebesar
23 365 bp kecuali pada klon 8F yang mengindikasikan bahwa klon 8F membawa sisipan yang tidak mengandung gen MTGase (Gambar 15).
Gambar 14 Penapisan pustaka genom per pool 96-well microplate menggunakan primer SDS 14-F/14-R. (M) marker 1 kb plus, (A-H) gabungan kultur E coli EPI300-T1R rekombinan per sumuran (pool), (K+) = DNA genom TTA 02 SDS 14, (K-) = koloni E coli EPI300-T1R
Gambar 15 Hasil penapisan klon yang mengandung gen MTGase pool F dan pool H. (M) marker 1 kb plus, (1 sampai 9) kultur E coli EPI300-T1R pool F, (10 sampai19) kultur E coli EPI300-T1R pool H, (K+) = DNA genom TTA 02 SDS 14
Analisis Fosmid pCC1FOSTM Rekombinan
Analisis fosmid pCC1FOSTM rekombinan yang mengandung fragmen ~40 kb yang membawa gen MTGase menggunakan enzim restriksi sangat diperlukan. Hal ini untuk membuktikan bahwa fosmid rekombinan telah masuk ke dalam E coli EPI300-T1R. Untuk mendapatkan transforman yang mengandung beberapa salinan fosmid rekombinan, maka sel ditumbuhkan ke dalam media LB yang mengandung kloramfenikol dan diinduksi dengan larutan induksi 1x (arabinosa). Tujuan induksi ini untuk mendapatkan high copy number dalam sistem penggandaan fosmid rekombinan dalam sel secara independen. Fosmid pCC1FOSTM rekombinan diverifikasi dengan memotongnya menggunakan enzim
restriksi NotI, EcoRV dan BamHI. Penggunaan ketiga enzim ini dikarenakan enzim memotong 2 kali pada sekuen fosmid pCC1FOSTM (Gambar 16A).
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 K+
20 Kb
365 bp 500 bp
A)
M A B C D F E G H K+ K -
500 bp
365 bp
24
Hasil pemotongan enzim restriksi NotI terhadap pCC1FOSTM rekombinan membentuk tiga pita dengan ukuran yang berbeda. Enzim NotI memotong pada sekuen ke-2 dan sekuen ke-643 yang mengapit daerah multiple cloning site (MCS) pada vektor. Pita pertama berukuran ~40,6 kb merupakan fragmen sisipan dan membawa sebagian fragmen dari vektor sebesar 600 bp. Enzim restriksi Not1 memotong pada sekuen fosmid rekombinan menghasilkan potongan lebih besar dari ~40 kb (Choi et al, 2012). Sedangkan pita yang kedua berukuran ~7,5 kb yang merupakan ukuran dari fosmid pCC1FOSTM yang digunakan. Namun pita
yang ketiga berukuran ~3 kb merupakan bagian dari sisipan ~40 kb yang dipotong oleh Not1. Hasil potongan ~3 kb ini kemudian dipurifikasi dari gel dan diamplifikasi PCR menggunakan primer PTGase 4/ PTGase 5 dan SDS 14F/ SDS 14R (Gambar 16B). PCR dengan masing–masing pasangan primer dapat mengamplifikasi gen MTGase parsial dengan ukuran yang sesuai yaitu 470 bp dan 365 bp. Hasil amplifikasi produk PCR menggunakan primer PTGase 4/PTGase 5 disubkloning ke dalam vektor kloning pGEM-T Easy. Selanjutnya dilakukan sekuensing untuk melihat keberadaan sisipan pada vektor.
A B
Gambar 16 Hasil pemotongan fosmid pCC1FOSTMrekombinan. A. (M1) marker
fosmid 36 kb, (1) fragmen ~40 kb tanpa potong, (2) fragmen ~40 kb dipotong dengan NotI, (3) fosmid rekombinan dipotong dengan NotI, (4) fosmid rekombinan dipotong dengan EcoRV, (5) fosmid rekombinan dipotong dengan BamHI (M2): marker 1 kb plus, B.
Hasil amplifikasi gen parsial MTGase dari fragmen ~3 kb (a) menggunakan primer PTGase4/5 (b) menggunakan primer SDS 14F/R
Analisis Sekuensing
25 menunjukkan bahwa berdasarkan dedukasi urutan nukleotida fragmen sisipan yang mempunyai kemiripan dengan gen MTGase yang dihasilkan oleh Streptomyces cinnamoneus dengan homologi sebesar 92% (Lampiran 8) walaupun terdapat kodon stop (TGA) pada nukleotida ke 369 – 371. Hasil penelusuran ini hampir sama dengan analisis BLAST dari gen parsial MTGase dari isolat TTA 02 SDS 14 yang pernah dilakukan sebelumnya.
T7_promoter NcoI
1 TAATACGACTCACTATAGGGGGCCAATCCATGCTCCGGCCGCCATGGCGGCCGCGG EcoRI
59 GAATTCGATTTACGGCTGCGTCGGTGTCACCTGGGTCAACTCGGGTCCCTACCCGAC 1 L R L R R C H L G Q L G S L P D
117 GAACAAACTGGCGTTCGCGTTCTTCGACGAGAACAGATACAAGAACGACCTCAAGAA 20 E Q T G V R V L R R E Q I Q E R P Q E
174 CACCAGCCCCCGCCCCGACGAAACGCGGGCGGAGTTCGAGGGTCGCATCGCCAAGGA 40 H Q P P P R R N A G G V R G S H R Q G
235 CAGTTTCGACGAGGGGAAGGGGTTCAAGCGGGCGCGCGATGTGGCGTCCCTCCTCAA 59 Q F R R G E G V Q A G A R C G V P P Q
293 CAAGGCCCTGGAAGGTGCCCACGACGAGGGGACGTACATCGACAACCTCAAGACGGA 78 Q G P G R C P R R G D V H R Q P Q D G
351 GCTCACGAACAGCAATGACGCTCTGCTCCTCGGGGACAGCCGCTCGAACTTCTACTC 97 A H E Q Q - R S A P R G Q P L E L L L
408 GGCGCTGAGGAACACACCGTCCTTCAAGGAAAGAGACGGAGGCAATCACGACCGTCA 116 G A E E H T V L Q G K R R R Q S R P
NotI
467 CAATCACTAGTGAATTCGCGGCCGCCTGCAGGTCGACCATATGGGAGAGCTCCCAAC
524 GCGTTGGATGCATAGCTTGAGTATTCTATAGTGTCACCTAAATAGCTTGGCGTAATC
581 ATGGTCATAGCTGTTTCCTGTGTGAAATTGTTATCCGCTCACAATTCCAC
Gambar 17. Analisis sekuen gen parsial MTGase pada vektor pGEMT-MTG1 (tanda ): daerah sisipan, (cetak miring): kodon stop, (blok kuning): sebagian sekuen gen MTGase, (tidak di blok): sebagian dari sekuen vektor pGEMT-Easy.
26
5 SIMPULAN DAN SARAN
Simpulan
Penapisan terhadap 178 isolat Streptomyces spp yang disolasi dari tanah menghasilkan satu isolat yang potensial menghasilkan enzim Microbial Transglutaminase (MTGase) dengan kode TTA 02 SDS 14 dengan aktivitas 3.3886 U/ml. Analisis molekuler dengan PCR membuktikan bahwa isolat ini juga mengandung gen MTGase. Berdasarkan Identifikasi gen parsial 16S rDNA, TTA 02 SDS 14 memiliki kekerabatan dekat dengan Streptomyces thioleteus dengan homologi 99%. Konstruksi DNA genom ~40 kb dari isolat ini telah berhasil disisipkan ke dalam fosmid pCC1FOSTM dan fosmid rekombinan telah berhasil
diintroduksikan ke dalam sel E coli EPI300-T1R. Hasil amplifikasi PCR dengan primer spesifik menunjukkan bahwa beberapa klon rekombinan mengandung sebagian gen MTGase.
Saran
27
DAFTAR PUSTAKA
Aeschlimann D, Paulsson M. 1994. Transglutaminase: protein cross-linking enzymes in tissues and body fluids. Thrombosis Haemostasis 71: 402–15. Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. 1990. Basic local
alignment search tool. J Mol Biol 215:403 – 410.
Ashie IN, Lanier TC. 2000. Transglutaminases in seafood processing. Food Science and Technology. New York (US). Marcel Dekker 147-166.
Bech L, Rasmussen G, Halkier T, Chiba OM, Anderson NL, Kauppinen MS, Sandal TI, Novozymes, Assignee. 2002. Transglutaminase from oomycetes. New York (US) Patent 6.428993.
Bourneow C, Benjakul S, Sumpavapol P, Kittikun H. 2012. Isolation and cultivation of transglutaminaseproducing bacteria from seafood processing
factories. Innovative Romanian Food Biotechnology. dunărea de jos”
university –galaţi 10: 28 – 39.
Brown TA. 2006. Gene Cloning and DNA Analysis an Introduction, 4nd ed. Australia: Blackwell Publishing Asia Pty Ltd.
Choi S, Chang MS, Stuecker T, Chung C, Newcombe DA, Venkateswaran K. 2012. A fosmid cloning strategy for detecting the widest possible spectrum
of microbes from the international space station drinking water system. Gen
Inform J 10(4):249-255.
Classen D, Lee-son NM, Lin C, Menzies JM, Yew DS. 2002. Ability of Eschericia coli to distinguish between self and foreign DNA as demonstrated by trends in transformation efficiency. J Exper Microbiol Immuno 2: 201-206.
Claverie J, Notredame C. 2003. Bioinformatics for Dummies. New York (US). Wiley Publishing Inc
Cui L, Du G, Zhang D, Liu H, Chen J. 2007. Purification and characterization of transglutaminase from a newly isolated Streptomyces hygroscopicus. Food chemJ 105(2): 612-618.
Chung CT, Niemela SL, Miller RH. 1989. One step preparation of competent Escherichia coli: transformation and storage of bacterial cells in the same solution. Proc Natl Acad SciUSA 86: 2172-2175.
Dhesi S. 2005. Investigation of the relationship between terminal sequence variation and the ligation efficiency of T4 DNA ligase on blunt-ended DNA. J Exper Microbiol Immuno 8:14-20.
Duran R, Junqua M, Schimitter JM, Gancet C, Goulas P. 1998. Purification, charaterization, and gene cloning of transglutaminase from Streptoverticilium cinnamoneum CBS 683.68. Biocimie 80:313-319.
Eshra DH, El-Iraki SM, Abo BTM. 2015. Isolation and indentification of actinomycetes transglutaminase producing strains. Int J Curr Sci 18: E76-88 Fawzya YN, Zilda DS, Poernomo A, Kristina I, Nursyam H. 2011. Karakterisasi dan aplikasi enzim transglutaminase dari Streptoverticillium ladakanum pada daging lumat ikan mata goyang. J Pasca Panen dan Bioteknologi Kelautan dan Perikanan 6(2): 157–166.
28
Greenberg CS, Birckbicher, Rice RH. 1991. Transglutaminase: Multifuntional cross-linking enzymes that stabilize tissue. J Biol chem 299:825-829.
Griffin M, Casadio R, Bergamini CM. 2002. Transglutaminasesμ Nature’s biological glues. J Biol Chem368: 377–396.
Gurgui C, Piel J. 2010. Metagenomic approaches to identify and isolate bioactive natural products from microbiota of marine sponges. Methods in Molecular Biology. Germany (DE): Springer Science + Business Media LLC
Haard NF, Simpson BK. 2000. Seafood Enzyme. New York (US): Marcel Dekker, Inc. 147-161.
Hagstrom AJF, Pinhassi, Zweiefel UL. 2000. Biogeoghraphycal diversity among marine bacterioplankton. Aqua Microbiol Tecnhnol 21: 231-244.
Hames NF, Simpson BK. 2000. Biochemistry: Bios Instant Notes. 3nd ed. New York (US): Taylor and Francis Group.
Hartwell, Leland .2008. Genetics: from genes to genomes. Boston (GB): McGraw-Hill Higher Education. ISBN 0-07-284846-4.
Hayawaka M. 2003. Selective isolation of rare Actinomycetes genera using pretreatment techniques. Ipek kurboke (Editor). Queensland (AU): University of The Sunshine Coast, Faculty of Science.
Ho ML, Leu SZ, Hseih JK, and Jiang ST. 2000. Technical approach to simplify the purification method and characterization og microbial transglutaminase produced from Streptoverticillium ladakanum. J.JFS: Food Chem Toxic 65 (1):76-80
Jawetz EJL. Melnick EA, Adelberg GF, Brooks JS, Butel LN, Ornston. 1995. Medical Microbiology. 2nd ed. San Francisco (US) University of California. Kadam S. 2008. Principle of Medicinal Chemistry. Pragati Books Pvt. Pune. 93 Kanaji T, Ozaki H, Takao T, Kawajiri K, Ide H, Motoki M, Simonishi Y. 1993
Primary structure of microbial transglutaminase from Streptoverticillium sp. strain s-8112. J Biol Chem 268: 11565–11572.
Kashiwagi T, Yokoyama K, Ishikawa K, Ono K, Ejima D, Matsui H, Suzuki E. 2012. Crystal structure of microbial transglutaminase from Streptoverticillium mobaraense. J Biol Chem 277(46): 44252-44260. Kieser HM, Kieser T, Hopwood A. 1992. A combined genetic and physical map
of Streptomyces coelicolor A3(2) cromosome. J Bacteriol 174: 5496-5507. Kikuchi Y, Date M, Yokoyama K, Umezawa Y, Matsui H. 2003. Secretion of
active-form Streptoverticillium mobaraense transglutaminase by Corynebacterium glutamicum : processing of the pro-domain by a co-secreted subtilisin-like protease from Streptomyces albogriseolus . Appl Environ Microbiol 69: 358 –366.
Kobayashi K, Suzuki SI, Izawa Y, Miwa K, Yamanaka S. 1998. Transglutaminase in sporulating cells of Bacillus subtilis. J Gen Appl Microbiol 44: 85–91. Lane DJ, Pace B, Olsen GJ, Stahl DA, Sogin ML, Pace NR. 1985. Rapid
determination of 16S ribosomal RNA sequences for phylogenetic analysis. P Nat Aca Sci 82: 6λ55−6λ5λ.