IDENTIFIKASI KERAGAMAN GEN
PITUITARY SPECIFIC
POSITIVE TRANSCRIPTION FACTOR-1
(
Pit
1|
Hinf
1) PADA
SAPI
FRIESIAN HOLSTEIN
DI BIB LEMBANG,
BBIB SINGOSARI, DAN BET CIPELANG
SKRIPSI
TIFANNY SUKMAWATI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
TIFANNY SUKMAWATI. D14070244. 2011. Identifikasi Keragaman Gen Pituitary Specific Positive Transcription Factor-1 (Pit1|Hinf1) pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang. Skripsi. Mayor Ilmu Produksi dan Teknologi Peternakan. Institut Pertanian Bogor.
Pembimbing utama : Prof Dr Ir Cece Sumantri M Agr Sc Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D
Identifikasi keragaman gen perlu dilakukan pada kegiatan seleksi dengan metode genetika molekuler terkait dengan tingkat produksi ternak. Salah satu gen yang diduga memiliki pengaruh terhadap sifat pertumbuhan, dan karkas serta produksi susu dan kualitas susu adalah gen Pit1. Penelitian ini bertujuan untuk mengidentifikasi keragaman fragmen gen Pit1|Hinf1 pada sapi perah Friesian Holstein (FH) dari BIB Lembang, BBIB Singosari, dan BET Cipelang; serta pada sapi pedaging dari BET Cipelang sebagai pembanding. Sampel darah yang digunakan dalam penelitian ini berjumlah 89 ekor, meliputi: sapi FH jantan yang berasal dari BIB Lembang (17 ekor), dan dari BBIB Singosari (32 ekor), serta sapi FH betina dari BET Cipelang (40 ekor). Adapun sebagai pembanding dipakai sapi pedaging betina yang berasal dari BET Cipelang berjumlah 36 ekor, meliputi: sapi Simmental (12 ekor), Limousin (14 ekor), Angus (5 ekor), dan Brahman (5 ekor). Amplifikasi gen Pit1 dilakukan dengan teknik PCR, sedangkan untuk menentukan genotipnya dilakukan dengan teknik Restriction Fragmen Length Polymorphism
(RFLP) dengan enzim restriksi Hinf1.
Gen Pit1 pada semua sapi FH dari ketiga lokasi tersebut dan kelima bangsa sapi pedaging dari BET Cipelang menghasilkan tiga variasi genotipe, yaitu AA (611 pb), AB (611 pb, 367 pb, 244 pb) dan BB (367 pb, 244 pb); serta dua variasi alel, yaitu alel A dan alel B. Sapi FH dari ketiga lokasi tersebut mempunyai frekuensi genotipe AA (0,062-0,118), AB (0,176-0,550), dan BB (0,350-0,706); dengan frekuensi alel B (0,625-0,794) lebih tinggi dibandingkan alel A (0,206-0,375). Proporsi frekuensi alel A dan B pada sapi pedaging didapatkan berbeda-beda pada setiap bangsa. Berdasarkan hasil analisis Chi-square gen Pit1|Hinf1 menunjukkan sapi FH dari ketiga lokasi serta sapi pedaging bangsa Simmental dan Limousin berada dalam keseimbangan Hardy-Weinberg (χ2 hitung < χ2tabel), sedangkan untuk sapi pedaging bangsa Angus dan Brahman tidak dapat dianalisis. Nilai heterozigositas gen Pit1|Hinf1 pada sapi FH menghasilkan heterozigositas pengamatan (Ho=0,416) lebih rendah dibandingkan heterozigositas harapan (He=0,418). Pada sapi pedaging, nilai heterozigositas pengamatan (Ho) lebih tinggi dari nilai heterozigositas harapan (He). Kekecualian pada sapi Angus yang mempunyai nilai heterozigositas pengamatan (Ho) lebih rendah dari nilai heterozigositas harapan (He). Kesimpulan yang dapat diambil adalah gen
Pit1|Hinf1 sapi FH dari BIB Lembang, BBIB Singosari dan BET Cipelang serta sapi pedaging dari BET Cipelang bersifat polimorfik (beragam).
ABSTRACT
Identification Polymorphism of Pituitary Specific Positive Transcription Factor-1
(Pit1|Hinf1) Gene in Friesian Holstein Cows in BIB Lembang, BBIB Singosari, and BET Cipelang
Sukmawati, T., C. Sumantri, and A. Anggraeni
Gene Pit-1 or POU1F1 or GHF1 is identified as the pituitary specific transcription factor that regulates the expression of the growth hormone (GH) and Prolactin (PRL) genes in the anterior pituitary. This study was aimed to identify polymorphism of the
Pit1|Hinf1 gene from the collected DNA extraction by using Polymerase Chain Reaction – Restriction Fragment Length Polymorphism (PCR-RFLP) methods. A total of 89 dairy cattles, were used from BIB Lembang (17 hds), BBIB Singosari (32 hds), and BET Cipelang (40 hds). A totally number of 37 beef cattle that were used for a comparison, namely Simmental (12 hds), Limousin (14 hds), Angus (5 hds), and Brahman (5 hds) from BET Cipelang. Three genotypes were indentified, namely AA (611 bp), AB (611 bp, 367 bp, 244 bp) and BB (367 bp, 244bp), genotypes resulting two alleles namely A and B alleles. The frequency of the B allele was higher than that of A allele (B=0.625-0.794 vs A=0.206-0.375); meanwhile in beef cattle the frequencies of these two alleles were diverse by breeds. The Pit1 gene in dairy cattle and beef cattle of Simmental and Limousin was in an equilibrium (χ2calculation<χ2table), while those for Angus and Brahman could not be analized. The highest heterozygosity value of dairy cattle was found in population BET Cipelang, while in beef cattle the highest one was in Brahman. The conclusion was that Pit1|Hinf1 gene in HF cattle from BIB Lembang, BBIB Singosari, and BET Cipelang; as well as beef cattle from BET Cipelang were polymorphic.
IDENTIFIKASI KERAGAMAN GEN
PITUITARY SPECIFIC
POSITIVE TRANSCRIPTION FACTOR-1
(
Pit
1|
Hinf
1)
PADA
SAPI
FRIESIAN HOLSTEIN
DI BIB LEMBANG,
BBIB SINGOSARI, DAN BET CIPELANG
TIFANNY SUKMAWATI D14070244
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman Gen Pituitary Specific Positive Transcription Factor1 (Pit1|Hinf1) pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang
Nama : Tifanny Sukmawati NIM : D14070244
Menyetujui,
Pembimbing Utama, Pembimbing Anggota,
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc) (Ir. Anneke Anggraeni, M.Si, Ph.D) NIP.19591212 198603 1 004 NIP. 19630924 199803 2 001
Mengetahui, Ketua Departemen,
Ilmu Produksi dan Teknologi Peternakan
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc) NIP.19591212 198603 1 004
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 11 Desember 1988 di Jakarta. Penulis adalah
anak pertama dari tiga bersaudara dari pasangan Bapak Bambang Sukmohutomo dan
Ibu Hafizoh.
Pada tahun 2001 penulis menyelesaikan pendidikan dasar di SD Negeri 1
Ciputat. Pendidikan lanjutan menengah pertama diselesaikan pada tahun 2004 di
SLTP Negeri 85 Jakarta, dan pendidikan lanjutan menengah atas diselesaikan pada
tahun 2007 di SMA Negeri 87 Jakarta.
Penulis diterima sebagai mahasiswa pada Departemen Ilmu Produksi dan
Teknologi Peternakan, Fakultas Peternakan Institut Pertanian Bogor melalui jalur
Seleksi Penerimaan Mahasiswa Baru (SPMB) pada tahun 2007. Selama mengikuti
pendidikan, penulis aktif sebagai anggota Koperasi Mahasiswa IPB (KOPMA IPB)
periode 2008/2009, dan Badan Eksekutif Mahasiswa (BEM) Fakultas Peternakan
IPB periode 2008/2009 dan 2009/2010. Penulis juga aktif di berbagai kepanitian di
KATA PENGANTAR
Puji syukur senantiasa dipanjatkan kepada Allah SWT, karena berkat rahmat
dan karunia-Nya penelitian dan penulisan skripsi ini dapat diselesaikan dengan baik.
Skripsi dengan judul Identifikasi Keragaman Gen Pituitary Specific Positive Transcription Factor-1 (Pit1|Hinf1) Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang ditulis untuk memenuhi persyaratan guna memperoleh gelar sarjana pada Departemen Ilmu Produksi dan Teknologi
Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Konsumsi susu dan daging sapi menunjukkan peningkatan yang signifikan
dari tahun-tahun. Peningkatan permintaan konsumsi ini sejatinya perlu diiringi
dengan peningkatan produktifitas dan populasi ternak sapi perah dan sapi pedaging.
Salah satu upaya yang bisa dilakukan untuk meningkatkan produktivitas ternak sapi
adalah dengan melakukan seleksi dan persilangan. Seleksi genetik seiring dengan
kemajuan teknologi terutama di bidang biologi molekuler upaya seleksi dapat
dilakukan pada tingkat DNA dengan cara mencari keragaman gen yang mengontrol
sifat yang penting seperti sifat pertumbuhan dan produksi susu.
Gen Pit1 merupakan salah satu gen yang dapat menghasilkan protein yang
diduga mengendalikan ekspresi gen penyandi hormon pertumbuhan dan hormon
prolaktin. Setelah proses kelahiran, gen Pit1 diduga memiliki pengaruh besar
terhadap pertumbuhan karkas serta produksi dan kualitas susu. Oleh karena itu
adanya keragaman gen Pit1 pada sapi perah dan sapi pedaging perlu diidentifikasi
yang diharapkan bisa meningkatkan produksi dan kualitas susu sapi perah dan
pertumbuhan dan kualitas karkas sapi pedaging.
Akhir kata, menyadur sebuah pepatah, tiada gading yang tak retak. Penulis
menyadari bahwa skripsi ini masih jauh dari sempurna. Meskipun demikian, Penulis
berharap semoga skripsi ini dapat bermanfaat dan berkontribusi positif terhadap
kemajuan dunia peternakan, khususnya di Indonesia.
Bogor, Mei 2011
DAFTAR ISI
Pituitary-Specific Positive Transcription factor 1 ... 5
Bahan dan Alat Pengambilan Sampel ... 10
Bahan dan Alat untuk Ekstraksi DNA ... 10
Primer ... 11
Bahan dan Alat untuk Reaksi PCR ... 11
Bahan dan Alat untuk Elektroforesis Produk PCR ... 11
Bahan dan Alat untuk Genotyping ... 11
Prosedur ... 12
Pengambilan Sampel ... 12
Ekstraksi DNA ... 12
Amplifikasi DNA ... 13
Elektroforesis Produk PCR ... 13
Genotyping ... 14
Analisis Data ... 15
HASIL DAN PEMBAHASAN ... 17
Amplifikasi Gen Pit1 ... 17
Pendeteksian Keragaman Gen Pit1 dengan Teknik PCR-RFLP ... 18
Frekuensi Gen Pit1 ... 20
Keseimbangan Hardy-Weinberg ... 22
Heterozigositas ... 23
KESIMPULAN DAN SARAN ... 25
Kesimpulan ... 25
Saran ... 25
UCAPAN TERIMA KASIH ... 26
DAFTAR TABEL
Nomor Halaman
1. Sampel Darah Sapi Friesian Holstein dan Sapi Pedaging ... 10 2. Frekuensi Genotipe dan Alel Gen Pit1|Hinf1 pada
DAFTAR GAMBAR
Nomor Halaman
1. Sapi Friesian Holstein ... 3 2. Bangsa-bangsa Sapi Pedaging ... 4 3. Rekonstruksi Struktur Gen Pit1 pada Ovis aries ... 6 4. Visualisasi Hasil Amplifikasi Gen Pit1 Sapi FH dan Sapi Pedaging
DAFTAR LAMPIRAN
Nomor Halaman
PENDAHULUAN Latar Belakang
Protein hewani berupa daging dan susu yang berasal dari sapi pedaging dan
sapi perah memiliki peranan yang sangat penting dalam memenuhi kebutuhan gizi
masyarakat Indonesia. Konsumsi susu di masyarakat terus mengalami peningkatan
dan diperkirakan akan terus meningkat secara signifikan pada tahun-tahun
selanjutnya. Begitupula untuk konsumsi daging sapi yang terus meningkat
kebutuhannya, sehingga memerlukan jumlah pemotongan sapi semakin banyak
(Dirjen Peternakan, 2009). Adanya peranan penting tersebut, maka produktifitas dan
populasi ternak sapi perah dan sapi pedaging menjadi bagian yang perlu mendapat
perhatian dalam dunia peternakan di Indonesia.
Aspek genetik dan lingkungan maupun interaksi keduanya merupakan
faktor-faktor yang dapat mempengaruhi produktifitas sapi perah dan sapi pedaging. Salah
satu upaya yang bisa dilakukan untuk meningkatkan produktivitas ternak sapi adalah
dengan melakukan seleksi dan persilangan. Perbaikan genetik melalui seleksi pada
sifat kuantitatif yang banyak dilakukan adalah secara konvensional. Namun, cara ini
belum memberikan hasil yang optimal karena disamping membutuhkan waktu yang
lama juga memerlukan biaya yang cukup besar. Seleksi keunggulan genetik pada
ternak sapi perah dan sapi pedaging dapat juga dilakukan melakukan aplikasi teknik
DNA yang memerlukan identifikasi keragaman gen yang terkait dengan sejumlah
sifat nilai ekonomis.
Kemajuan dalam bidang biologi molekuler memungkinkan upaya seleksi
dapat dilakukan pada tingkat DNA, yaitu dengan cara mencari keragaman gen yang
mengontrol sifat yang ingin diperbaiki seperti sifat pertumbuhan dan produksi susu.
Salah satu gen pada sapi yang berkaitan dengan sifat tersebut adalah gen Pit1. Gen
Pit1 merupakan salah satu gen yang dapat menghasilkan protein yang diduga
mengendalikan ekspresi gen penyandi hormon pertumbuhan dan hormon prolaktin.
Setelah proses kelahiran, gen Pit1 diduga memiliki pengaruh besar terhadap
pertumbuhan karkas serta produksi dan kualitas susu. Oleh karena itu adanya
keragaman gen Pit1 pada sapi perah dan sapi pedaging perlu diidentifikasi yang
diharapkan mampu meningkatkan produksi dan kualitas susu sapi perah dan
yang digunakan dalam mengindentifikasi keragaman suatu fragmen gen adalah
dengan menggunakan teknik Polymerase Chain Reaction (PCR), sedangkan untuk
menentukan genotipnya dilakukan dengan teknik Restriction Fragmen Length
Polymorpism (RFLP) dengan enzim restriksi Hinf1. Aplikasi teknik ini diharapkan
dapat dipakai untuk mengidentifikasi keragaman genetik dari gen Pit1|Hinf1 pada sapi perah dan sapi pedaging di dalam ketiga lokasi tersebut.
Tujuan
Penelitian ini bertujuan untuk mengidentifikasi keragaman fragmen gen
Pit1|Hinf1 pada sapi perah Friesian Holstein (FH) dari BIB Lembang, BBIB Singosari, dan BET Cipelang; serta sapi pedaging dari BET Cipelang sebagai
TINJAUAN PUSTAKA Sapi Friesian Holstein
Sapi Friesian Holstein merupakan bangsa sapi perah yang banyak terdapat di
Amerika Serikat dengan jumlah sekitar 80-90% dari seluruh sapi perah yang ada.
Sapi ini berasal dari Negeri Belanda yaitu dari propinsi North Holand dan West
Friesland, kedua daerah yang memiliki padang rumput yang bagus. Sapi FH
menyebar ke seluruh dunia, baik negara subtropis ataupun di negara tropis. Ciri-ciri
yang paling menonjol pada sapi perah Friesian Holstein (FH) yaitu warna tubuhnya
memiliki dua warna, hitam dan putih (black Holstein) atau merah dan putih (red
Holstein).
Sumber : www.wapedia.mobi/id
Gambar 1. Sapi Friesian Holstein (FH)
Bangsa sapi FH merupakan bangsa sapi perah yang memiliki tingkat produksi
susu tertinggi jika dibandingkan dengan bangsa sapi perah lainnya. Dengan tingkat
produksi susun rata-rata setiap satu masa laktasi (10 bulan) adalah sekitar 3.050 liter
atau sekitar 10 liter per ekor per hari. Pada daerah asalnya produksi susu per masa
laktasi rata-rata sebanyak 7.245 liter atau sekitar 20 liter per hari (Putranto, 2006).
Bangsa-bangsa Sapi Pedaging
Sapi pedaging merupakan sapi yang dipelihara untuk menghasilkan
pertumbuhan dengan karkas yang optimal. Bangsa-bangsa sapi pedaging yang sudah
dikenal cukup baik di Indonesia adalah sapi Peranakan Ongole (PO), Simmental,
Brahman, Limousin, dan Angus. Setiap bangsa tersebut memiliki keungulan dan
Sapi Simmental
Blakely & Bade (1991) menyatakan sapi Simmental berasal dari Lembah
Simme di Swiss (Gambar 2a.). Sapi Simmental memiliki karakteristik warna bulu
krem kecoklatan hingga sedikit merah, bulu muka berwarna putih dan bagian ekor
dan lutut kebawah berwarna putih dengan ukuran tanduk tidak begitu besar. Ternak
sapi ini berukuran besar, pertumbuhan ototnya sangat baik dan tidak banyak
penimbunan lemak dibawah kulit (Pane, 1986). Sapi ini terkenal karena
pertumbuhannya cepat, badannya panjang dan padat (Blakely & Bade, 1991).
2a. Sapi Simmental 2b. Sapi Brahman
2c. Sapi Limosin 2d. Sapi Angus
Sumber : www.infoternak.com
Gambar 2. Bangsa-bangsa Sapi Pedaging
Sapi Brahman
Sapi Brahman (Gambar 2b.) berasal dari keturunan kelompok sapi Zebu dari
India memiliki campuran Bos taurus dari Inggris yang telah dikembangkan di
Amerika serikat. Sapi ini memiliki kaki yang panjang, punuk besar, telinga panjang
menjulai kebawah dan bergelambir; serta warna bulu umumnya berwarna putih atau
kelabu muda, akan tetapi ada juga yang berwarna kemerah-merahan (Pane, 1986).
Menurut Williamson dan Payne (1993) sapi Brahman merupakan sapi pedaging yang
tumbuh baik di padang pengembalaan yang buruk dan kering, tahan panas,
Sapi Limousin
Sapi Limousin (Gambar 2c.) berasal dari Perancis Tengah di bagian selatan.
Sapi Limousin merupakan tipe sapi pedaging dengan warna bulu merah keemasan,
kaki dari lutut kebawah memiliki warna merah agak muda, dan umumnya terdapat
lingkaran berwarna merah agak muda disekeliling mata (Pane, 1986). Bobot lahir
sapi ini tergolong kecil sampai medium yang berkembang menjadi golongan besar
pada saat dewasa. Betina dewasa dapat mencapai 575 kg, sedangkan pejantan dewasa
mencapai berat 1100 kg (Blakely & Bade, 1991). Sapi peranakan Limousin sudah
banyak tersebar dan memiliki produktivitas yang bagus di sejumlah wilayah di
Indonesia.
Sapi Angus
Sapi Angus (Gambar 2d.) berasal dari Skotlandia dan disebut juga
Aberdeen-Angus. Sapi ini merupakan hasil persilangan antara sapi Buchman Bumlies dengan
Angus Dodies. Sapi Angus memiliki warna bulu hitam, keriting, dan halus. Jantan
dan betina tidak memiliki tanduk; tubuhnya panjang dan kompak, serta kualitas
karkasnya tinggi (Pane, 1986). Sifat-sifat yang menonjol dan mempunyai arti penting
adalah ketahanan terhadap hawa dingin, kemampuan yang baik dalam memelihara
anak dan menyusui, masak dini, fertilitas tinggi, tidak banyak kesulitan dalam
melahirkan, kualitas karkas yang istimewa dengan tulang-tulang yang kecil (Blakely
& Bade, 1991).
Gen Pit1 ( Pituitary-Specific Positive Transcription Factor 1)
Gen Pit1 (Pituitary-Specific Positive Transcription Factor 1) dikenal juga
dengan nama POU1F1; atau growth hormone factor 1 (GHF1) merupakan faktor
transkripsi spesifik pituitari yang berperan untuk perkembangan pituitari dan ekspresi
hormon pada mamalia. Pit1 merupakan anggota dominan POU yang mengandung
protein, yaitu kelompok regulator transkripsi yang mempunyai peran kunci dalam
diferensiasi dan pembelahan sel (Mangalam et al., 1989). Secara in vivo, kebanyakan
dari protein POU berperan penting dalam proses perkembangan yang terkait dengan
sistem saraf. Gen Pit1 adalah faktor transkripsi khusus untuk ekspresi gen penyandi
hormon pertumbuhan, hormon prolaktin, dan hormone tirotropin β- subunit (Brunsch
mengendalikan ekspresi gen penyandi growth hormone releasing hormone gene
(GHRH). Faktor transkripsi dibutuhkan untuk penempelan enzym RNA polymerase
pada bagian promotor suatu gen (Yuwono et al., 2005).
Rekonstruksi struktur gen Pit1 yang ditampilkan berdasarkan pada struktur
gen Pit1 pada Ovis aries. Hal ini dapat dilakukan karena keduanya tergolong
mamalia.
Keterangan : Panjang : 5787 pb
Ekson 1 (Ex1) : 142 pb Ekson 4 (Ex4) : 165 pb
Ekson 2 (Ex2) : 150 pb Ekson 5 (Ex5) : 61 pb
Ekson 3 (Ex3) : 225 pb Ekosn 6 (Ex6) : 211 pb
Gambar 3. Rekonstruksi Struktur Gen Pit1 pada Ovis aries (Bastos et al., 2006)
Rekonstruksi struktur gen Pit1 pada Ovis aries mempunyai 6 ekson dan 5
intron, dimana panjang fragmen ekson 1 (Ex1) 142 pb, ekson 2 (Ex2) 150 pb, ekson 3
(Ex3) 225 pb, ekson 4 (Ex4) 165 pb, ekson 5 (Ex5) 61 pb, dan ekson 6 (Ex6) 211 pb.
Ekson 2 pada Ovis aries terbagi menjadi dua yaitu ekson 2A (78 pb) dan ekson 2
(72 pb).
Beberapa hasil penelitian keragaman gen Pit1 yang sudah dilakukan antara
lain oleh Dybuss et al. (2004) pada sapi Polish hitam putih dengan menggunakan
metode RFLP dan menggunakan enzim Hinf1 ditemukan dua alel yaitu A (0,243)
dan B (0,757). Hal ini serupa dengan hasil yang didapatkan Edriss et al. (2008) pada
sapi Holstein di Ishafan yang ditemukannya tiga variasi genotipe yaitu AA
(0,018-0,050), AB (0,350-0,564), dan BB (0,418-0,600) dengan frekuensi alel A
(0,225-0,300) dan alel B (0,700-0,775); serta dilaporkan pula oleh Jawasreh et al. (2009)
pada sapi Friesian dan sapi lokal di Jordania bahwa frekuensi alel A (0,333) dan alel
B (0,733).
Renaville et al. (1997) menyatakan alel A pada lokus Pit-1|Hinf1 dapat diasosiasikan dengan produksi susu, kadar protein dan persentase lemak pada sapi
perah. Sehubungan dengan itu, Zwierzchowski et al. (2002) menunjukkan bahwa alel
A pada lokus Pit-1 berdampak positif pada sifat produksi susu. Berdasarkan hasil
yang potensial dan berpengaruh signifikan terhadap sejumlah sifat-sifat kuantitatif
(QTL) seperti produksi susu, protein, dan lemak susu khususnya pada ternak sapi.
Polymerase Chain Reaction – Restriction Fragment Length Polymorphism (PCR – RFLP)
Metode PCR merupakan suatu metode yang dapat digunakan untuk
memperbanyak segmen DNA secara in vitro (Ausbel, 1995). Segmen DNA tersebut
kemudian dapat diketahui runutan nukleotidanya, salah satunya dengan
menggunakan enzim restriksi. Enzim restriksi dapat memotong DNA secara spesifik
dan terbatas pada situs yang dikenalinya (Lewin, 1994). Williams (2005)
menambahkan bahwa PCR merupakan suatu teknik untuk menggandakan jumlah
molekul DNA pada ruas-ruas tertentu dan monomer-monomer nukleotida yang
dilakukan secara in vitro. Proses ini berjalan dengan bantuan primer dan enzym
polymerase. Primer merupakan oligonukleotida spesifik yang menempel pada bagian
DNA yang akan diperbanyak. Enzim Polymerase merupakan enzim yang dapat
mencetak urutan DNA baru. Hasil dari proses PCR dapat divisualisasikan dengan
elektroforesis.
Proses PCR terdiri dari 3 tahapan, yaitu (1) Denaturasi, yaitu struktur DNA
utas ganda mengudar menjadi utas tunggal, (2) Anealing, yaitu penempelan primer
pada sekuens DNA komplementer yang akan diperbanyak, (3). Ekstensi, yaitu
pemanjangan primer oleh DNA polymerase (Muladno, 2002). Menurut
Viljoen et al. (2005), reaksi yang terjadi dalam mesin thermalcycler secara umum
dapat dibagi menjadi tiga tahap yaitu tahap denaturasi DNA cetakan, tahap annealing
atau penempelan primer dan tahap extension, yaitu pemanjangan primer atau
polimerase. Reaksi ini umumnya terjadi dalam 25-30 siklus. Pada tahap denaturasi,
DNA dipanaskan hingga 94 oC sehingga DNA untai ganda berpisah menjadi DNA
untai tunggal. Tahapan yang paling menentukan dalam proses PCR adalah tahap
penempelan primer, karena tiap pasangan primer memiliki suhu penempelan primer
yang spesifik. Tahap pemanjangan primer terjadi pada suhu 27 oC. Pada tahapan ini
enzim taq polymerase, buffer PCR, dNTP, dan Mg2+ memulai aktifitasnya
memperpanjang primer.
PCR-RFLP merupakan salah satu metode analisis lanjutan dari produk PCR.
menvisualisasikan perbedaan level DNA yang didasarkan pada penggunaan enzim
pemotong (restriction enzyme) yang dapat memotong DNA pada tempat sekuens
nukleotida spesifik. Metode PCR memanfaatkan runutan nukleotida yang bisa
dikenali oleh enzim rerstriksi yang disebut sebagai situs restriksi. Jika situs restriksi
mengalami mutasi (meskipun pada satu basa) maka enzim restriksi tidak mampu
mengenalinya. Ada tidaknya situs restriksi kemudian dapat digunakan untuk
mengetahui ada tidaknya mutasi. Analisis RFLP biasa digunakan untuk mendeteksi
adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti
produksi dan kualitas susu (Sumantri et al., 2007).
Keragaman Genetik
Keragaman genetik dalam suatu populasi digunakan untuk mengetahui dan
melestarikan bangsa-bangsa dalam populasi terkait dengan penciri suatu sifat khusus.
Menurut Frankham et al. (2002), beragamnya sumber daya genetik, maka akan
semakin taan populasi tersebut untuk hidup dalam jangka waktu yang lama; serta
semakin tinggi daya adaptasi populasi terhadap perubahan lingkungan. Estimasi
perhitungan keragaman genetik dalam populasi secara kuantitatif dapat diperoleh
melalui dua ukuran keragaman variasi populasi yaitu proporsi lokus polimorfisme
dalam populasi dan rata-rata proporsi individu heterozigot dalam setiap lokus (Nei,
1987). Polimorfisme genetik dalam suatu populasi dapat digunakan dalam
menentukan hubungan antar subpopulasi yang terfragmentasi dalam suatu spesies
(Hartl dan Clark, 1997). Keragaman genetik dalam antara subpopulasi dapat
diketahui dengan melihat persamaan dan perbedaan frekuensi alel di antara
subpopulasi (Li et al., 2000). Menurut Falconer dan Mackay (1996), suatu alel
dikatakan polimorfik jika memiliki frekuensi alel sama dengan atau kurang dari 0,99.
Hukum Hardy-Weinberg menyatakan frekuensi genotipe suatu populasi yang
cukup besar akan selalu dalam keadaan seimbang bila tidak ada seleksi, migrasi,
mutasi, dan genetic drift; selain itu, silang dalam dan silang luar juga dapat
mempengaruhi frekuensi genotipe (Noor, 2000). Derajat heterozigositas merupakan
rataan persentase lokus heterozigositas tipe individu atau rataan persentase individu
heterozigot dalam populasi (Nei, 1987). Avise (1994) menyatakan bahwa semakin
tinggi derajat heterozigositas suatu populasi maka daya tahan hidup populasi tersebut
Beberapa penelitian dilakukan untuk melihat keragaman genetik sebagai
salah satu contohnya adalah keragaman genetik sapi FH Indonesia.
Rahmani et al. (2004) melaporkan keragaman genetik sapi FH berdasarkan gen
hormon pertumbuhan di BPTU Baturraden dan ditemukan 4 alel A, B, C, dan D
dengan lima tipe genotipe melaporkan bahwa frekuensi gen A dan gen B pada
peternakan sapi FH yang ada di Baturraden memiliki nilai yang hampir sama
METODE Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler Ternak
bagian Pemuliaan Genetika, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian
ini dilakukan selama 5 bulan, yaitu dari bulan Juli sampai dengan November 2010.
Materi Sampel Darah
Sampel DNA yang digunakan adalah berasal dari sampel darah koleksi
Laboratorium Genetika Molekuler Ternak bagian Pemuliaan Genetika, Fakultas
Peternakan, Institut Pertanian Bogor. Sampel darah yang digunakan berjumlah 89
ekor sapi perah jenis Friesian Holstein dan sapi pedaging yang berasal dari BET
Cipelang sebanyak 36 ekor (Tabel 1).
Tabel 1. Sampel Darah Sapi Friesian Holstein (FH) dan Sapi Pedaging
No. Bangsa Sapi Jenis
5 Limousin Betina Pedaging BET Cipelang 14
6 Angus Betina Pedaging BET Cipelang 5
7 Brahman Betina Pedaging BET Cipelang 5
Bahan dan Alat Pengambilan Sampel
Bahan yang digunakan adalah alkohol 70%, es, dan kapas. Alat yang
digunakan antara lain jarum venoject, vaccutainer 10 ml, antikoagulan heparin dan
termos.
Bahan dan Alat Untuk Ekstraksi DNA
Bahan yang digunakan adalah sampel darah 200µl, EDTA (Ethylinediamine
tetraacetic), destilation water, 40 µl SDS 10% (Sodium Dodecyl Sulfat), 10 µl enzim
Proteinase K 5mg/ml, 400 µl phenol, 400 µl CIAA, 800 µl etanol absolute, etanol
Buffer, dan 100 µl TE 80% (Tris EDTA). Peralatan yang digunakan adalah tabung
eppendorf 1,5 ml, satu set mikro pipet, tip, vortexmixer, autoclave, mikrosentrifuge,
rotary mixer, inkubator, refrigerator, dan freezer.
Primer
Sekuen primer yang digunakan dalam penelitian ini menggunakan primer
yaitu berdasarkan Javanmard et al. (2005), yaitu dengan primer forward adalah
5’-GAG CCT ACA TGA GAC AAG CAT C-3’ dan primer reverse adalah 5’-AAA
TGT ACA ATG TGC CTT CTG A-3’ dengan produk PCR 611 pb dan berada di
daerah ekson 6.
Bahan dan Alat Untuk Reaksi PCR
Bahan-bahan yang digunakan dalam reaksi Polymerase Chain Reaction
(PCR) adalah sampel DNA, enzyme taq polymerase, 5×buffer, MgCl2, dNTPs, dan
pasangan primer AF94 dan AF95 (Forward dan Reverse). Alat yang digunakan
adalah alat sentrifugasi, vortex, pipet mikro, tabung PCR, mesin PCR (Thermal
Cycler), lemari es, deep freezer dan inkubator.
Bahan dan Alat Untuk Elektroforesis Produk PCR
Bahan-bahan yang digunakan yaitu loading dye (bromthymol blue 0,01%,
Xylene cyanol 0,01% dan gliserol 50%) dan untuk membuat 1 lembar gel agarose
1,5% adalah sebagai berikut: agarose 0,45 g, 0,5 TBE 30 ml, dan 2,5 µl ethidium
bromide (EtBr). Alat-alat yang digunakan antara lain adalah microwive, stearer,
magnetic stearer, gelas ukur, tabung kimia, gel tray, pencetak untuk sumur (comb),
power supply electrophoresis 100 Volt, tip, pipetmikro, alat foto UV trans
illuminator dan sarung tangan.
Bahan dan Alat Untuk Genotyping
Bahan-bahan yang digunakan yaitu enzim restriksi HinfI dengan buffernya,
loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%) dan
untuk membuat 1 lembar gel agarose 2% adalah sebagai berikut: agarose 0,6 g, 0,5
TBE 30 ml, dan 2,5 µl EtBr. Alat-alat yang digunakan antara lain adalah microwive,
stearer, magnet stearer, gelas ukur, tabung kimia, gel tray, pencetak untuk sumur
(comb), power supply electrophoresis 100 Volt, tip, pipetmikro, alat foto UV trans
Prosedur
Pengambilan Sampel Darah
Sampel darah sapi telah dikoleksi dari sapi FH jantan dan betina dari BIB
Lembang, BET Cipelang, dan BBIB Singosari; dan darah sapi pedaging betina
berasal dari BET Cipelang sebagai pembanding. Sampel darah diambil melalui vena
jugularis menggunakan jarum venoject dan tabung vaccutainer yang mengandung
antikoagulan heparin. Sampel darah kemudian ditambahkan ethanol absolute dengan
perbandingan 1:2 dan disimpan pada suhu ruang sampai kemudian dilakukan
analisis.
Ekstraksi DNA
Ekstraksi DNA dilakukan dari sampel darah dengan menggunakan metode
Sambrook et al. (1989), sampel darah yang disimpan dalam alkohol 70% diambil
sebanyak 200 μl ditambahkan 1000 μl DW (destilation water) kemudian dikocok atau di vortex dan didiamkan selama lima menit, setelah itu disentrifugasi dengan
kecepatan 8000 rpm selama lima menit. Bagian supernatannya dibuang, tambahkan
1000 μl DW (destilation water) kemudian dikocok atau di vortex dan didiamkan selama lima menit, setelah itu disentrifugasi dengan kecepatan 8000 rpm selama lima
menit. Kemudian ditambahkan 10 μl proteinase K yang berfungsi untuk menghancurkan protein, 350 μl 1xSTE (sodium tris-EDTA) dan 40 μl 10% SDS (sodium dodesil sulfat) yang berfungsi untuk melisiskan membran sel. Campuran
tersebut kemudian diinkubasi pada suhu 55 oC selama 2 jam sambil dikocok pelan
menggunakan alat pemutar (tilting).
Molekul DNA kemudian dimurnikan dengan metode fenol-chloroform, yaitu
dengan menambahkan 40 μl 5M NaCl, 400 μl larutan fenol dan 400 μl CIAA (chloroform iso amil alcohol), lalu dikocok pelan (tilting) pada suhu ruang selama
satu jam. Molekul DNA yang larut dalam fase air dipisahkan dari fase phenol dengan
alat sentrifugasi pada kecepatan 12.000 rpm selama 5 menit. Setelah terbentuk fase
Molekul DNA kemudian dipisahkan dari etanol absolut dengan cara
sentrifugasi pada kecepatan 12.000 rpm selama lima menit, kemudian supernatan
yang diperoleh dibuang. Endapan yang terbentuk kemudian dicuci dengan
menambahkan 70% etanol sebanyak 800 μl dan disentrifugasi kecepatan 12.000 rpm selama lima menit. Supernatan yang diperoleh kemudian dibuang sehingga
didapatkan endapan molekul DNA. Endapan tersebut didiamkan sampai kering. Lalu
endapan DNA disuspensikan dalam 100 μl 80% buffer TE (tris EDTA).
Amplifikasi DNA
Perbanyakan gen Pit1 yang diapit oleh primer forward dan reverse secara
in-vitro dilakukan menggunakan mesin PCR Thermal Cycler. Primer tersebut
merupakan primer hasil desain untuk mengamplifikasi gen Pit1 pada ekson 6.
Produk PCR yang diharapkan adalah 611 pb (Pit1 ekson 6). Campuran untuk
mengamplifikasi gen Pit1 ini terdiri dari sampel DNA, pasangan primer (forward
dan reverse), taq polymerase dan buffernya, MgCl2, dan dNTPs.
Proses amplifikasi yang terjadi dalam mesin PCR Thermal Cycler ini
berlangsung dalam tiga tahapan. Tahap pertama adalah denaturasi awal pada suhu
94 oC selama lima menit. Tahap kedua merupakan 30 siklus amplifikasi yang terdiri
dari denaturasi pada suhu 94 oC selama 45 detik, penempelan primer pada suhu 60 oC
selama 45 detik, dan pemanjangan molekul DNA pada suhu 72 oC selama satu menit.
Tahap ketiga adalah pemanjangan akhir molekul DNA pada suhu 72 oC selama lima
menit. Terakhir adalah inkubasi pada suhu 4 oC hingga digunakan untuk analisis
lebih lanjut.
Elektroforesis Produk PCR
Elektroforesis produk PCR dilakukan menggunakan 5 μl produk PCR pada
gel agarose 1,5% dengan tegangan 100 volt selama 30 menit. Gel dibuat dengan cara
memanaskan agarose 0,45 g yang dilarutkan dalam larutan 0,5 TBE 30 ml serta
menambahkan 2,5 µl EtBr pada saat distearer sampai didapatkan larutan jernih.
Larutan yang masih cair dituangkan ke dalam pencetak gel serta menempatkan sisir
di dekat tepian gel dan gel dibiarkan mengeras. Apabila gel sudah mengeras, sisir
dicabut sehingga akan terbentuk sumur-sumur yang digunakan untuk menempatkan
sebanyak 5 µl produk PCR dicampur dengan loading dye (bromthymol blue 0,01%,
elektroforesis yang sudah terisi larutan buffer dan dialiri listrik, molekul DNA yang
bermuatan negatif pada pH netral akan bergerak (migrasi) ke arah positif (anode).
Setelah elektroforesis selesai gel agarose diambil untuk dilihat panjang pita DNA
dengan menggunakan sinar Ultraviolet yaitu dengan menarik garis lurus antara posisi
pita dari masing-masing sampel DNA yang ingin diukur dengan posisi pita DNA
marker, kita dapat mengestimasi ukuran sampel DNA karena ukuran DNA pengukur
telah diketahui.
Genotyping
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism
(PCR-RFLP) dilakukan menggunakan 6 μl produk PCR dipindahkan kedalam tabung
0,5 ml yang ditambahkan 0,2 µl enzim restriksi Hinf1 yang mengenali situs
pemotongan lima basa (G|AnTC). Campuran tersebut diinkubasikan dalam inkubator
pada suhu 37 °C selama 16 jam.
Sampel DNA yang telah dipotong dengan enzim restriksi di tambahkan
loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%)
sebanyak 1 µl, dielektroforesis pada gel agarose 2% dengan tegangan 100 volt
selama 30 menit. Setelah elektroforesis selesai gel agarose diambil untuk dilihat
panjang pita DNA dengan menggunakan sinar Ultraviolet dan dibandingkan dengan
marker untuk mengetahui panjangnya. Setiap pita DNA dari setiap sampel di
perbandingkan untuk menentukan genotipe pita DNA. Satu posisi migrasi yang sama
dianggap sebagai satu tipe atau alel. DNA yang tampak seperti pita difoto untuk
diabadikan susunan pita-pitanya.
Penentuan tipe genotipe menurut Javanmard et al. (2005), yaitu sapi
dikatakan mempunyai genotipe AA apabila terdapat satu fragmen DNA yaitu 611 pb.
Genotipe AB ditunjukan dengan tiga fragmen DNA yaitu 611, 367, dan 244 pb.
Analisis Data Frekuensi Alel dan Genotipe
Keragaman genotipe tiap-tiap individu dapat ditentukan dari migrasi pita-pita
DNA hasil pemotongan dengan enzim restriksi. Masing-masing sampel
dibandingkan berdasarkan ukuran (marker) yang sama dan dihitung frekuensi
genotipe dan alelnya (Nei dan Kumar, 2000):
Keterangan :
Xii = Frekuensi genotipe ke-ii
Xi = Frekuensi alel ke-i
nii = Jumlah individu yang bergenotipe ii
nij = Jumlah individu yang bergenotipe ij
N = Jumlah individu sampel
Keseimbangan Hardy-Weinberg
Keseimbangan Hardy-Weinberg diuji dengan menggunakan perhitungan
Chi-Kuadrat (Nei dan Kumar, 2000) :
Keterangan:
χ2
= uji Chi-kuadrat
O = Jumlah pengamatan genotipe ke-i
E = Jumlah harapan genotipe ke-i
Heterozigositas
Keragaman genetik dapat diketahui melalui estimasi frekuensi heterozigositas
pengamatan yang diperoleh dari masing-masing populasi dengan menggunakan
rumus Weir (1996) sebagai berikut :
Keterangan:
Ho = Heterozigositas pengamatan (populasi)
N = Jumlah individu yang diamati
Nilai heterozigositas berdasarkan frekuensi alel dapat dihitung menggunakan
rumus berikut (Nei dan Kumar, 2000):
Keterangan:
He = Nilai heterozigositas harapan
Xi = Frekuensi alel
HASIL DAN PEMBAHASAN Amplifikasi Gen Pit1
Gen Pit1 ekson 6 pada sapi Friesian Holstein (FH) dari lokasi BIB
Lembang, BBIB singosari dan BET Cipelang; sapi pedaging (Simmental, Limousin,
Angus, dan Brahman) yang berasal dari BET Cipelang telah berhasil di amplifikasi
dengan menggunakan teknik Polymerase Chain Reaction (PCR). Persentase
keberhasilan amplifikasi gen Pit1 dalam penelitian ini adalah 100%. Hasil
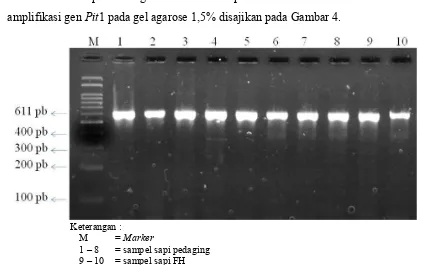
amplifikasi gen Pit1 pada gel agarose 1,5% disajikan pada Gambar 4.
Keterangan :
M = Marker
1 – 8 = sampel sapi pedaging 9 – 10 = sampel sapi FH
Gambar 4. Visualisasi Hasil Amplifikasi Gen Pit1 Sapi FH dan Sapi Pedaging pada Gel Agarose 1,5%
Amplifikasi gen Pit1 menggunakan mesin Termal Cycler pada penelitian ini
menghasilkan fragmen dengan panjang 611 pasang basa (pb). Perkiraan panjang
fragmen yang diamplifikasi dapat diketahui dengan menyesuaikan sekuens gen Pit1
yang diperoleh dari GeneBank no.akses Y15995 dan AM490263 dengan pasangan
primer berdasarkan literatur Javanmard et al. (2005) disajikan pada Gambar 5.
Menurut Viljoen et al. (2005), yang harus diperhatikan dalam optimasi PCR
diantaranya adalah suhu anneling, konsentrasi Mg2+, konsentrasi primer, konsentrasi
DNA target; selain itu Muladno (2002) menambahkan komponen lainnya yang
dibutuhkan adalah ensim Taq DNA polymerase, deoxynucleoside triphosphat
(dNTP), dan larutan penyangga (buffer). Pada gen Pit1 untuk penelitian ini kondisi
digunakan oleh Javanmard et al. (2005). Suhu anneling adalah suhu yang
memungkinkan terjadinya penempelan primer pada DNA cetakan dan perpanjangan
DNA dimulai dari primer selama proses PCR. Setiap gen memiliki suhu anneling
yang berbeda untuk dapat menghasilkan panjang fragmen yang diinginkan untuk
diamati.
Pendeteksian Keragaman Gen Pit1 dengan Teknik PCR-RFLP
Metode Restriction Fragment Length Polymorphism (RFLP) digunakan untuk
mengindentifikasi keragaman gen Pit1 pada sapi FH dan sapi pedaging dari produk
PCR dengan memanfaatkan runutan nukleotida yang bisa dikenali oleh enzim
rerstriksi yang disebut sebagai situs restriksi. Sumantri et al. (2007), analisis RFLP
sering digunakan untuk mendeteksi adanya keragman gen yang terkait dengan
sejumlah sifat nilai ekonomis, seperti produksi dan kualitas susu; selain itu analisis
RFLP digunakan untuk mendeteksi lokasi genetik dalam kromosom yang
menyandikan penyakit yang diturunkan(Orita et al., 1989).
Ada tidaknya situs restriksi kemudian dapat digunakan untuk mengetahui ada
tidaknya mutasi. Proses pemotongan gen Pit1 dilakukan dengan menggunakan enzim
restriksi Hinf1 yang mengenali situs pemotongan pada posisi G|AnTC.
781 tgcatacaga attattttct tctcagtaag tcagtgccct cttgtggcag aaagtggata
841 aacaatgtcg gggttccctc cttaatttct tcctgtgact ctggtaaaag gagcctacat
901 gagacaagca tctaaatgtt caaaaaaact tcacatttat tattgttgaa aagctttgaa
961 ggtgttttca gcgtctttag gtttcctttt tacgttaatg ttagtactaa tatttaggaa
1021 atgtaaccta acttgatttt gatgggccta aaccatcatc tcccttcttt cctgccaact
1081 ccccacctcc cagtattgct gctaaagacg ccctggagag acactttgga gaacagaata
1141 agccttcctc tcaggagatc ctgcggatgg ctgaagaact aaacctggag aaagaagtgg
1201 tgagggtttg gttttgtaac cgaaggcaga gagaaaaacg ggtgaagaca agcctgaatc
1261 agagtttatt tactatttct aaggagcatc tcgaatgcag ataggctctc ctattgtgta
1321 atagcgagtg tttctacttt tcattccttt ctcttctcca gccaaaatag aaattagtta
1381 tttggttagc ttcaaaaaat cacatcagta atttttgcag aagtgtttct tttctacttt
1441 aaaaataaat acaatttaaa ttatgttgat gaattattct cagaaggcac attgtacatt
1501 t
Keterangan : = Primer
= Situs Pemotongan
Alel A : 5’ --- GACAAGCCTAAATCAGAGTTTAT ---3’ Alel B : 5’ --- GACAAGCCTGAATCAGAGTTTAT ---3’
Keragaman gen Pit1 yang terjadi pada penelitian ini disebabkan adanya
mutasi titik yang terjadi pada basa ke 1256. Mutasi pada gen Pit1 ialah mutasi
subsitusi basa (transisi) dari Guanin (G) menjadi Adenin (A) (Gambar 5). Menurut
Toland (2008), keragaman DNA dapat terjadi karena akibat dari mutasi. Mutasi
adalah suatu perubahan struktur kimia gen yang menyebabkan berubahnya fungsi
gen. Mutasi titik atau Point Mutation adalah mutasi yang terjadi hanya pada satu titik
nukleotida. Menurut Paolella (1998), mutasi titik dapat dibedakan berdasarkan tipe
perubahan runutan nukleotida yaitu penghilangan (delesi), penyisipan (insersi),
subtitusi (transisi dan transversi), kesalahan pembacaan (profreading errors), dan
perubahan struktur kimia pada basa.
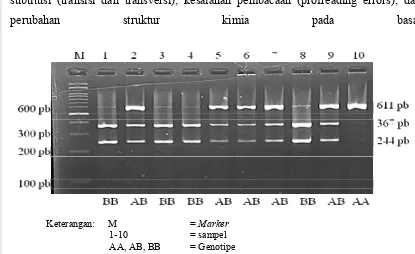
Keterangan: M = Marker
1-10 = sampel AA, AB, BB = Genotipe
Gambar 6. Pola Pita Gen Pit1|Hinf1 pada Gel Agarose 2%
Pendeteksian keragaman gen Pit1 pada sampel dengan panjang fragmen 611
pb. Posisi pemotongan dengan menggunakan Hinf1, menghasilkan panjang fragmen
367 pb dan 244 pb. Produk PCR fragmen gen Pit1 yang telah dipotong dengan enzim
restriksi Hinf1 menghasilkan tiga macam fragmen, yaitu fragmen yang tidak
terpotong menghasilkan satu pita yang dikenal dengan genotipe AA dengan panjang
fragmen 611 pb, menghasilkan dua pita 367 pb dan 244 pb yang dikenal dengan
genotipe BB, dan fragmen gabungan yang menghasilkan tiga pita dengan panjang
fragmen 611 pb, 367 pb, dan 244 pb yang dikenal dengan genotipe AB (Gambar 6).
Penelitian ini sesuai dengan Jawasreh et al. (2009) pendeteksian keragaman
Friesian di Jordania menghasilkan tiga macam fragmen, yaitu fragmen yang tidak
terpotong menghasilkan satu pita yang dikenal dengan genotipe AA (611 pb),
menghasilkan dua pita yang dikenal dengan genotipe BB (367 pb dan 244 pb), dan
fragmen gabungan yang menghasilkan tiga pita dengan panjang fragmen yang
dikenal dengan genotipe AB (611 pb, 367 pb, dan 244 pb).
Frekuensi Genotipe dan Alel Gen Pit1
Hasil analisis frekuensi genotipe dan alel sapi FH dari lokasi BIB lembang,
BBIB Singosari dan BET Cipelang yang berjumlah 89 ekor; serta sapi pedaging dari
BET Cipelang yang berjumlah 36 ekor disajkan pada Tabel.2.
Tabel 2. Frekuensi Genotipe dan Alel Gen Pit1|Hinf1 pada Sapi Friesian Holstein
dan Sapi Pedaging
Gen Pituitary-Specific Positive Transcription Factor 1 (Pit1) merupakan
faktor transkripsi spesifik pituitari yang berperan untuk perkembangan pituitari dan
mengandung protein, yaitu kelompok regulator transkripsi yang mempunyai peran
kunci dalam diferensiasi dan pembelahan sel (Mangalam et al., 1989).
Frekuensi Genotipe sapi FH di lokasi BIB Lembang frekuensi genotipe yang
tertinggi adalah genotipe BB (0,706), begitu pula dengan yang terdapat di lokasi
BBIB Singosari yang tertinggi adalah genotipe BB (0,563); sedangkan di lokasi BET
Cipelang frekuensi genotipe yang tertinggi adalah genotipe AB (0,550) (Tabel 2).
Frekuensi alel adalah frekuensi relatif dari suatu alel dalam populasi atau jumlah
suatu alel terhadap jumlah total yang terdapat dalam suatu populasi
(Nei dan Kumar, 2000).
Frekuensi alel sapi FH dari ketiga lokasi tersebut memperoleh alel B (0,702)
lebih tinggi dari alel A (0,298). Sapi pedaging yang berasal dari BET Cipelang terdiri
bangsa Simmental, Limousin, Angus, dan Brahman. Frekuensi genotipe yang
terdapat pada sapi Simmental yang tertinggi genotipe BB (0,500), sapi Limousin
yang tertinggi genotipe AB, sapi Angus yang tertinggi genotipe AA (0,800); serta
sapi Brahman yang tertinggi genotipe AB (1), hal ini dikarenakan dari seluruh
sampel sapi Brahman yang digunakan bergenotipe AB.
Frekuensi alel yang terdapat pada sapi pedaging berbeda disetiap bangsa.
Sapi Simmental mempunyai frekuensi alel B (0,708) yang lebih tinggi dari alel A
(0,292), begitu pula yang terjadi pada sapi Limousin yang mempunyai frekuensi alel
B (0,607) yang lebih tinggi dari alel A (0,393), sapi Angus mempunyai frekuensi alel
A (0,800) yang lebih tinggi dari alel B (0,200), sedangkan pada sapi Brahman antara
alel A dan alel B mempunyai frekuensi yang sama (0,500). Menurut Nei (1987)
menyatakan bahwa suatu alel dapat dikatan polimorfik jika memiliki frekuensi alel
sama dengan atau kurang dari 0,99. Oleh karena itu, gen Pit1 sapi FH dari ketiga
lokasi yang diamati dan sapi pedaging yang berasal dari BET Cipelang bersifat
polimorfik (beragam).
Keragaman genotipe gen Pit1 sapi FH dari ketiga lokasi tersebut dapat
dibandingkan dengan beberapa penelitian sebelumnya. Penelitian ini sesuai dengan
Edriss et al. (2008) bahwa sapi Holstein di Isafahan memiliki tiga variasi genotipe
yaitu AA (0,018-0,050), AB (0,350-0,450), dan BB (0,500-0,600) dan yang tertinggi
adalah genoptipe BB (0,500-0,600). Frekuensi alel yang didapatkan yaitu alel B
melaporkan hasil penelitiannya terhadap sapi Friesian di Jordania yaitu mempunyai
frekuensi alel B (0,8255) lebih tinggi dari alel A (0,1744).
Gen Pit1 merupakan salah satu gen kandidat yang perlu dicari hubungan atau
keterkaitannya dengan performa pertumbuhan, kualitas karkas dan juga performa
laktasi pada beberapa bangsa sapi seperti yang telah dilaporkan oleh beberapa
penelitian sebelumnya (Woollard et al. 1994; Moody et al. 1995; Zwierzhowski et al.
2001; Dybus et al. 2003; Oprzadek et al. 2003; Zhao et al. 2004; Viorica et al. 2007).
Hasil penelitian yang dilakukan oleh Renaville et al. (1997) dapat diasosiasikan
bahwa alel A pada lokus Pit1|HinfI dengan produksi susu dan protein superior, dan inferior untuk persentase lemak pada sapi perah. Sehubungan dengan itu,
Zwierzchowski et al. (2002) menunjukkan bahwa alel A pada lokus Pit-1 berdampak
positif pada sifat produksi susu. Selain itu menurut Zhao et al. (2000) melaporkan
bahwa polimorfisme Pit1|HinfI menampakkan efek pada sifat pertumbuhan sapi Angus dan menjadi kandidat gen untuk digunakan dalam Marker-Assisted Selection
(MAS). Polimorfisme gen Pit-1 yang terjadi pada ekson 6, selain bisa berasosiasi
dengan sifat karkas dan produksi susu, juga bisa dengan sifat seleksi yang lain,
seperti berat badan umur 1 tahun (Zhao et al., 2004). Berdasarkan laporan
Mattos et al. (2004), keragaman genetik gen Pit1 yang berinteraksi dengan
bGH/MspI berpengaruh teradap variasi persentase lemak pada ternak perah.
Keseimbangan Hardy-Weinberg
Keseimbangan Hardy-Weinberg merupakan suatu keadaan dimana dalam
suatu populasi yang terdapat faktor penghambat (mutasi, seleksi, migrasi, dan genetic
drift), maka frekuensi gen dominan dan resesif dalam suatu populasi tidak akan
berubah dari satu generasi ke generasi lainnya. Keseimbangan variasi genotipe
penelitian ini dengan menggunakan uji Chi-square (χ2). Hasil uji Chi-square tersebut dapat disajikan pada Tabel 3.
Berdasarkan hasil analisis Chi-square diperoleh χ2 hitung < χ2tabelterdapat padasapi FH dari lokasi BIB Lembang, BBIB Singosari dan BET Cipelang berada
dalam keseimbangan Hardy-Weinberg, begitu pula yang terjadi pada sapi Simmental,
gen dalam populasi yang cukup besar terjadi jika tidak ada seleksi, mutasi, migrasi
Keterangan : tn = tidak berbeda nyata Td = tidak dapat dianalisis
Keseimbangan yang terjadi pada sapi FH dari ketiga lokasi yang diamati, sapi
Simmental dan sapi Limousin terjadi bukan dikarenakan tidak adanya seleksi, karena
seluruh sapi yang berada pada lokasi tersebut merupakan hasil seleksi. Hal tersebut
dapat terjadi diduga karena sifat yang diseleksi tidak terkait langsung dengan gen
Pit1, sehingga dalam keadaan keseimbangan. Pada lokasi BET Cipelang untuk sapi
pedaging bangsa Angus tidak dapat dianalisis karena memiliki derajat bebas χ2 adalah 0, disebabkan pada bangsa tersebut hanya terdapat 2 macam genotipe dan 2
macam alel; sedangkan untuk bangsa Brahman tidak dapat dianalisis karena
memiliki derajat bebas χ2 adalah -1, disebabkan pada bangsa tersebut hanya terdapat 1 macam genotipe dan 2 macam alel. Derajat bebas χ2 merupakan hasil pengurangan antara jumlah genotipe dengan jumlah alel (Allendorf dan Luikart, 2007).
Heterozigositas
Heterozigositas disebut juga sebagai rataan keragaman genetik. Pendugaan
nilai heterozigositas memiliki arti penting untuk diketahui, yaitu untuk mendapatkan
gambaran variabilitas genetik (Marson et al., 2005). Berdasarkan hasil analisis nilai
heterozigositas sapi FH dari tiga lokasi, nilai heterozigositas yang tertinggi adalah
sapi FH dari BET Cipelang 0,550; sedangkan pada sapi pedaging nilai
heterozigositas yang tetinggi adalah bangsa Brahman yaitu 1. Menurut Javanmard et
al. (2005) nilai heterozigositas kurang dari 0,5 mengindikasi rendahnya variasi gen
heterozigositas suatu populasi maka daya tahan hidup populasi tersebut akan
semakin tinggi. Hasil analisis heterozigositas pada tiga populasi sapi FH dan pada
populasi BET Cipelang untuk sapi Simmental, Limousin, Angus dan Brahman tersaji
pada Tabel 4.
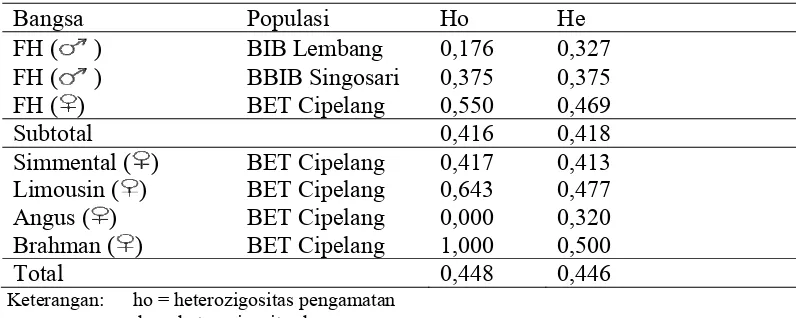
Tabel 4. Heterozigositas Gen Pit1|Hinf1
Bangsa Populasi Ho He
FH ( ) BIB Lembang 0,176 0,327
BBIB Singosari 0,375 0,375 FH ( )
FH ( ) BET Cipelang 0,550 0,469
Subtotal 0,416 0,418
Simmental ( ) BET Cipelang 0,417 0,413
Limousin ( ) BET Cipelang 0,643 0,477
Angus ( ) BET Cipelang 0,000 0,320
Brahman ( ) BET Cipelang 1,000 0,500
Total 0,448 0,446
Keterangan: ho = heterozigositas pengamatan he = heterozigositas harapan
Sapi FH dari BIB Lembang mempunyai nilai heterozigositas pengamatan (Ho)
lebih rendah dari nilai heterozigositas harapan (He), Sapi FH dari BBIB Singosari
mempunyai nilai heterozigositas pengamatan (Ho) sama dengan nilai heterozigositas
harapan (He); sedangkan Sapi FH dari BET Cipelang mempunyai nilai
heterozigositas pengamatan (Ho) lebih tinggi dari nilai heterozigositas harapan (He).
Sapi pedaging bangsa Simmental, Limousin, dan Brahman mempunyai nilai
heterozigositas pengamatan (Ho) lebih tinggi dari nilai heterozigositas harapan (He);
sedangkan sapi pedaging bangsa Angus nilai heterozigositas pengamatan (Ho) lebih
rendah dari nilai heterozigositas harapan (He). Tingginya nilai heterozigositas (Ho)
KESIMPULAN DAN SARAN Kesimpulan
Berdasarkan hasil penelitian dapat disimpulkan bahwa hasil analisis gen
Pit1|Hinf1 pada sapi FH dari lokasi BIB Lembang, BBIB Singosari dan BET Cipelang dan sapi pedaging dari lokasi BET Cipelang bersifat polimorfik. Terdapat
dua alel yaitu alel A dan alel B, sehingga diperoleh tiga variasi genotipe, yaitu AA,
AB dan BB. Berdasarkan hasil analisis Chi-square sapi FH dari ketiga lokasi dan
juga sapi pedaging bangsa Simmental dan Limousin dari BET Cipelang berada
dalam keseimbangan Hardy-Weinberg (χ2 hitung < χ2tabel), sedangkan sapi pedaging bangsa Angus dan bangsa Brahman tidak dapat dianalisis. Nilai
heterozigositas tertinggi terdapat pada sapi Limousin dan terendah pada sapi Angus.
Saran
Perlu dilakukan pendeteksian lebih lanjut untuk mendeteksi hubungan antara
alel yang dimiliki oleh sapi FH pejantan dan sapi FH betina dengan hasil dari
produksi susu dari keturunannya. Jumlah sampel yang lebih banyak dari lokasi,
seperti balai-balai penelitian, industri dan peternakan rakyat untuk lebih menambah
UCAPAN TERIMA KASIH
Alhamdulillah, puji dan syukur penulis panjatkan kehadirat Allah SWT
karena atas rahmat dan karunia-Nya penulis dapat menyelesaikan skripsi ini dengan
baik walaupun masih jauh dari kesempurnaan. Penelitian dan skripsi ini dapat
diselesaikan berkat bantuan dari berbagai pihak. Untuk itu, pada kesempatan ini
penulis ingin mengucapkan terima kasih yang tulus dan ikhlas kepada :”Papa dan
Mama” tercinta, yang selalu melimpahkan cinta dan kasih sayang yang tulus, serta
doa dan dukungan baik secara moril maupun materil dengan tiada putusnya; serta
untuk kedua adikku Melinda Utami dan Devi Amalia (terima kasih atas doa dan
dukungannya).
Terima kasih kepada Prof. Dr. Ir. Cece Sumantri, M.Agr. Sc dan Ir. Anneke
Anggraeni, M.Si, Ph.D selaku dosen pembimbing dalam membimbing,
mengarahkan, dan menasehati penulis. Terima kasih kepada Ir. Sri Darwati, M.Si,
Ir. Anita Sardiana Tjakradijaja, M.Rur.Sc, M. Baihaqi, S.Pt. M.Sc sebagai dosen
penguji. Keluarga besar LGMT FAPET IPB : Kak Erick, Kak Restu, Kak Surya, Kak
Dina, Annisa O.R., Diny W., Wike, Irine, Paulina, Ferdy, Lenny, Revy, Rahmah,
Erwin, dan Bapak Ichsan. Sahabat-sahabat tercinta: Rayjansof Chairi, Hans Budi
Findranov, M. Fachri Jamasyari dan terutama Asia Muflihah yang telah memberikan
motivasi, dukungan, dan bantuannya disaat susah maupun senang; serta Fadlullah
Abdurrachman yang telah memberikan perhatian, pengertian, motivasi, dukungan,
dan bantuannya. Teman-teman IPTP 44: Nur’adhadinia, Andhika W. J.,dan Gabby E.
M. (terima kasih untuk kebersamaan dan persahabatan yang indah). Untuk
teman-teman “Pondok Bidadari”: Winda P., Indah P., Sherly A., Listika M., Tika S.R.,
Nurul I., Fatma S., dan Selvi. Semua pihak yang tidak dapat penulis sebutkan, yang
telah memberikan bantuan baik sebelum, selama maupun sesudah penelitian.
Tiada kata yang dapat penulis ucapkan selain doa untuk kalian semoga Allah
membalas semua kebaikan kalian dengan kebaikan yang lebih baik dan lebih besar.
Sesungguhnya tiada balasan untuk suatu kebaikan kecuali dengan kebaikan pula.
Bogor, 14 Mei 2011
DAFTAR PUSTAKA
Allendorf, f. W. & G. Luikart. 2007. Conservation and The Genetics of Populations. Blackwell Publishing. USA.
Ausubel. 1995. Short Protocol in Moleculer Biology.3rd Ed. New York.
Avise, J. C. 1994. Molecular Markers. Natural History Evolution. Chapman and Hall, Inc., Washington.
Bastos, E., I. Santos, I. Pasmentier, J. L. Castrillo, A. Cravador, H. Guedes-Pinto, & R. Renaville. 2006. Ovis aries POU1F1 gene: cloning, characterization and polymorphism analysis. Springer Netherland. 126: 303-314.
Blakely, J., & D. H. Bade. 1991. Ilmu Peternakan. Edisi Ke-4. Terjemahan: Bambang Srihandono. Gadjah Mada University Press, Yogyakarta.
Brunsch, C., I. Strenstein, P. Reinecke, & J. Bieniek. 2002. Analysis of association of Pit-1 genotypes with growth, meat quality and carcass composition traits in pigs. J. Appl. Genet. 43:85-91.
Curi, R. A., D. A. Palmieri, L. Suguisawa, H. N. Olievera, A. C. Silviera, & C. R. Lopes. 2006. Growth and carcass traits associated with GH|Alu1 I and POU1F1|Hinf 1 gene polymorphism in Zebu and crossbred beef cattle. Genet and Mol Biotech. 29:56-61.
De Mattos, K. K., D. L. S. Nassif, L. M. Mario, & F. F. Ary. 2004. Association of bGH and Pit-1 gene variants with milk production traits in dairy Gyr bulls.
Pesq. Agropec. Bras. Brasillia, 39:147 – 150.
[Ditjennak] Direktorat Jenderal Peternakan. 2009. Statistik Peternakan 2009. Jakarta: Departemen Pertanian.
Dybus A., M. Kmiec, Z. Sobek, W. Pietrzyk, & B. Wisniewski. 2003. Associations between polymorphisms of growth hormone releasing hormone (GHRH) and pituitary transcription factor 1 (PIT1) genes and production traits of Limousine cattle. Arch. Tierz., Dummerstorf 46:527-534.
Edriss, V., M. A. Edriss, H. R. Rahmani, & B. E. Sayed. 2008. Pit-1 gene polymorphism of Holstein Cows in Ishafan Province. Tabatabaei college of Agriculture. Biotecnol. 7:209-212.
Falconer, D. S. & T. F. C. Mackay. 1996. Introduction to Quantitative Geneticss. 4th Ed. Longman, New York.
Frankham, R., J. D. Ballou & D. A. Briscoe. 2002. Introduction to Consevation Genetics, Cambridge University Press.
Hartl, D. L., & A. G. Clark . 1997. Principle of Population Genetic. Sinauer Associates, Sunderland, MA.
Jawasreh, K. I. Z., F. Awawdeh, I. Rawashdeh, F. Hejazeen, & M. Al-Thalib. 2009. The allele and genotype frequencies of bovine pituitary specific transcription factor and leptin genes in Jordanian cattle population by using PCR-RFLP. Austr. J. of Basic and Appl. Sci., 3:1601-1606.
Lewin, B. 1994. Genes V. Oxford University Press. New York.
Li, X., K. Li, B. fan, Y. Gong, S. Zhao, Z. Peng, & B. Liu. 2000. The genetic diversity of seven pigs breeds in china, estimated by means of microsatellites. J. Anim. Sci 9:1193-1195.
Mangalam, H. J., V. R. Albert, H. A. Ingraham, M. Kapiloff, L. Wilson, C. Nelson, H. Elsholtz, & M. G. Rosenfeld. 1989. A pituitary POU-domain protein, Pit -1, activities both growth hormone and prolactin promoters transcriptionally. Genes Dev., 3:946-958.
Marson, E. P., B. Jose, S. F. Flavio, M. Vieira, C. D. C. B. Julio, P. E. Joanir, G. G. F. Luis, & B. M. Geron. 2005. Genetic characterization of European Zebu composite bivine using RFLP markers. Genet Mol Res 4:496-505.
Montaldo, H. H., & C. A. M. Herrera. 1998. Use of molecular markers and major genes in te genetic improvement of livestock. J. of. Biotechnol. 1 No 2.
Moody D.E., D. Pomp, & W. Barendse. 1995. Restriction fragment length polymorphism amplification products of the bovine Pit-1 gene assignmentof Pit-1 to bovine chromosome 1. Anim. Genet. 26:45-47.
Muladno. 2002. Seputar Teknologi Rekayasa Genetik. Pustaka Wirausaha Muda dan USESE Foundation. Bogor.
Nei, M. 1987. Molecular Evalutionery Genetics. Columbia University Press. New York.
Nei, M., & S. Kumar. 2000. Molecular Evaluation and Phylogenetics. Oxford University Press. New York.
Noor, R. R. 2000. Genetika Ternak. Penebar Swadaya, Depok.
Oprzadek J, K. Flisikowski, L. Zweirzchowski, & E. Dymnicki. 2003. Polymorphisms at loci of leptin (LEP), Pit1 and STA-T5A and their association with growth, feed conversion and carcass quality in Black-and-White bulls. Anim. Sci. Pap. and Rep. 21:223-231.
Orita, M., H. Iwahana, H. Kanazawa, K. Hayashi, & T. Sekia. 1989. Detection of polymorphism of human DNA by gel electrophoresis as single-strand conformation polymorphisms. Proc. Natl. Acad. Sci. 86:2766-2770.
Pane, I. 1986. Pemuliabiakan Ternak Sapi. Gramedia. Jakarta.
Rahmani, N., Muladno, & C. Sumantri. 2004. Analisis polimorfisme gen bovine growth hormone (bGH) pada sapi perah Friesian-Holstein di BPTU Baturraden. Pros. Seminar Nasional Teknologi Peternakan dan Veteriner. Puslitbang Peternakan hlm 183-194.
polymorphism, milk and conformation traits for Italian Holstein-Friesian Bulls. J. Dairy Sci. 80:3431-3438.
Sambrook, J., E. F. Fritsch, & J. F. Medrano. 1989. Molecular Cloning : A Laboratory Manual. Second Edition. Cold Spring Harbor Laboratory Press, New York.
Sumantri, C., A. Anggraeni, A. Farajallah, & D. Perwitasari. 2007. Keragaman mikrosatelit DNA sapi perah FH di Balai Pembibitan Ternak Unggul Baturraden. J. I T V 12:124-133.
Tambasco, D. D., C. C. P. Paz, M. Tambasco-Studart, A. P. Pereira, M. M. Alencor, A. R. Freitas, L. L. Coutinho, I. U. Packer, & L. C. A. regitano. 2003. Candidate genes for growth traits in beef cattle crosses Bos Taurus x Bos Indicus. J. Anim Breed Genet 120:51-60.
Viljoen, G. J., L. H. Nel, & J. R. Crowther. 2005. Molecular Diagnostic PCR Handbook. Springer, Dordrecht, Netherland.
Viorica, C., A. Vlaic, & I. Gaboreanu. 2007. Hinf-1 polymorphism of k-casein and Pit-1 genes in Romanian Simmental cattle. Zootehnie şi Biotehnologii, 40:59-64.
Williams, J. L. 2005. The Use of Marker-assisted selection in animal breeding and biotechnology. Rev. sci. tech. Off. int. Epiz., 24 : 379-391.
Williamson, G., & W. J. A. Payne. 1993. Pengantar Peternakan Daerah Tropis. Terjemahan: S. G. N. Djiwa Darmadja. Gajah Mada University Press, Yogyakarta.
Woollard, J., C. B. Schmitz, A. E. Freeman, & C. K. Tuggle. 1994. HinfI polymorphismsat the bovine PIT1 locus. J. Anim. Sci, 72:3267.
Yuwono, T. 2005. Biologi Molekuler. Penerbit Erlangga, Yogyakarta.
Zhao, Q., M. E. Davis, & H. C. Hines. 2000. Association of two Pit-1 gene polymorphisms with growth rate in beef cattle. J. Anim. Sci.78:77
Zhao, Q., M. E. Davis, & H. C. Hines. 2004. Associations of polymorphism in the Pit-1 gene with growth and carcass traits in Angus beef cattle. J. Anim. Sci. 82:2229-2233.
Zwierzchowski, L., J. Kryzzweski, N. Strazalkowska, E, Siadkowska, & A. Ryniewicz. 2002. Effect of polymorphisms of growth hormone (GH), Pit-1, and leptin (LEP) genes, cow’s age, lactation stage and somatic cell count milk yield and composition of Polish Black and white cows. Animal Science Papers Reports, Inst. Genet. Anim. Breed,. Jastrzebiec, Poland 20:213-227.
Zwierzchowski, L., J. Oprzadek, E. Dymnicki, & P. Dzierzbicki. 2001. An association of growth hormone, kappa-casein, beta-lactoglobulin, leptin and
Lampiran 1. Sekuens Gen Pit1 pada Sapi Diakses di GenBank (Kode Akses Y115995 dan AM490263)
LOCUS Y15995 1301 bp DNA linear MAM
14-NOV-2006
DEFINITION Bos taurus PIT-1 gene, partial. ACCESSION Y15995
VERSION Y15995.1 GI:3766420
KEYWORDS growth hormone factor I; PIT1 gene. SOURCE Bos taurus (cattle)
AUTHORS Bodner,M., Castrillo,J.L., Theill,L.E., Deerinck,T., Ellisman,M.
and Karin,M.
TITLE The pituitary-specific transcription factor GHF-1 is a homeobox-containing protein
JOURNAL Cell 55 (3), 505-518 (1988) PUBMED 2902927
REFERENCE 2
AUTHORS Moody,D.E., Pomp,D. and Barendse,W.
TITLE Restriction fragment length polymorphism in amplification products
AUTHORS Dierkes,B., Kriegesmann,B., Baumgartner,B.G. and Brenig,B.
/gene="PIT-1"
1 caatgagaaa gttggtgcaa atgaaagaaa aaggaaacgg agaacaacaa tcaggtatac
61 ttttgagata ttaagagtta gtggagaaga aaatgatatt ttacaaatgg aatgaacatt
121 tgagtataat atagtttcaa tataacataa aaatgaatag agccaattga gaaaataggt
181 gaaaaagcac aacattcaat aaattacttc tgagaaacag ctggaaattt aaaatttgat
241 ggaaaaatat gtattgtttg attcaagaac agttttgctc tgcaagtttt ggataaaaca
301 gaagctgtac aatcacagct aaaaagaatg actgtttcta ctgtgtcata atgtgttgat
361 ttatgtttag acataaatct tgctccggga aagagcccat ggactgtagc ctacaggttc
421 ctctgtccat gggattttcc aggcaagaat aatggagtgg gttgccattt ccttctccag
481 gagatcttcc cgacccaggg attgaacccg gatctcctac attgtaggca gatgctttac
541 catctgagcc acaagggaag tcacctatct atattatttc aaattaacaa aactggtcac
601 tagtatttta gttgcttaaa gttcaaaatg acttctagca tttcaagcca gattgttcat
721 tggttggtta gttgtacact aaacatctca ataacctgag ttctggggga catttagaaa
781 tgcatacaga attattttct tctcagtaag tcagtgccct cttgtggcag aaagtggata
841 aacaatgtcg gggttccctc cttaatttct tcctgtgact ctggtaaaag gagcctacat
901 gagacaagca tctaaatgtt caaaaaaact tcacatttat tattgttgaa aagctttgaa
961 ggtgttttca gcgtctttag gtttcctttt tacgttaatg ttagtactaa tatttaggaa
1021 atgtaaccta acttgatttt gatgggccta aaccatcatc tcccttcttt cctgccaact
1081 ccccacctcc cagtattgct gctaaagacg ccctggagag acactttgga gaacagaata
1141 agccttcctc tcaggagatc ctgcggatgg ctgaagaact aaacctggag aaagaagtgg
1201 tgagggtttg gttttgtaac cgaaggcaga gagaaaaacg ggtgaagaca agcctgaatc
1261 agagtttatt tactatttct aaggagcatc tcgaatgcag a
LOCUS AM490263 451 bp DNA linear MAM
10-FEB-2007
DEFINITION Bos taurus partial PIT-1 gene for growth hormone factor I, exon 6,
individual 3. ACCESSION AM490263
VERSION AM490263.1 GI:125629405
KEYWORDS growth hormone factor I; PIT-1 gene. SOURCE Bos taurus (cattle)
AUTHORS Jamli,S. and Heslop-Harrison,J.S.
TITLE Diversity in growth-related genes in Brakmas, a Malaysian cattle
breed
JOURNAL Unpublished
REFERENCE 2 (bases 1 to 451) AUTHORS Heslop-Harrison,P.J. TITLE Direct Submission
/PCR_primers="fwd_name: PIT1f, fwd_seq:
1 aaaccatcat ctcccttctt tcctgccaac tccccacctc ccagtattgc tgctaaagac
61 gccctggaga gacactttgg agaacagaat aagccttcct ctcaggagat cctgcggatg
121 gctgaagaac taaacctgga gaaagaagtg gtgagggttt ggttttgtaa ccgaaggcag
181 agagaaaaac gggtgaagac aagcctaaat cagagtttat ttactatttc taaggagcat
241 ctcgaatgca gataggctct cctattgtgt aatagcgagt gtttctactt ttcattcctt
301 tctcttctcc agccaaaata gaaattagtt atttggttag cttcaaaaaa tcacatcagt
361 aatttttgca gaagtgtttc tttcctactt taaaaataaa tacaatttaa attatgttga
Lampiran 2. Modifikasi Metode Ekstraksi DNA Menggunakan Metode
Phenolcloroform (Sambrook et al., 1989)
200 μl sampel dara dalam ETO + DW 1000 μl
Vortex, didiamkan selama 5 menit
Sentrifugasi 8000 rpm, selama 5 menit
Supernatan dibuang
+DW 1000 μl
Vortex, didiamkan selama 5 menit
Sentrifugasi 8000 rpm, selama 5 menit
Supernatan dibuang
+ 5 mg/ml Proteinase K 10 μl + SDS 10% 40 μl + 1x STE sampai volume 400 μl
Kocok pelan pada suhu 55 °C, selama 2 jam
+ 400 μl phenol + 400 μl CIAA + 40 μl NaCl 5 M
Kocok pelan pada suhu ruang, selama 1 jam
Sentrifugasi 1200 rpm, selama 5 menit
Pindahkan bagian DNA (bening) dalam tabung 1,5 ml, 200 μl
+ 800 μl ETOH absolute + 40 μl NaCl 5 M
Freezing over night
Sentrifugasi 12000 rpm, selama 5 menit
Supernatan dibuang
+ 800 μl ETOH 70%
Sentrifugasi 12000 rpm, selama 5 menit
Supernatan dibuang, keringkan
+ 100 μl TE 80%
IDENTIFIKASI KERAGAMAN GEN
PITUITARY SPECIFIC
POSITIVE TRANSCRIPTION FACTOR-1
(
Pit
1|
Hinf
1) PADA
SAPI
FRIESIAN HOLSTEIN
DI BIB LEMBANG,
BBIB SINGOSARI, DAN BET CIPELANG
SKRIPSI
TIFANNY SUKMAWATI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN