ANALISIS KERAGAMAN GENETIK LABI-LABI
(Amyda cartilaginea) BERDASARKAN GENOM MITOKONDRIA
EVI RIZKY AMELIA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
ABSTRAK
EVI RIZKY AMELIA. Analisis Keragaman Genetik Labi-labi (Amyda cartilaginea) Berdasarkan Genom Mitokondria. Dibimbing oleh ACHMAD FARAJALLAH dan R.R. DYAH PERWITASARI.
Amyda cartilaginea termasuk ke dalam Ordo Testudinata, Subordo Cryptodira dan Famili Trionychidae. Amyda cartilaginea dilaporkan telah mengalami penurunan populasi yang diakibatkan oleh pengaruh manusia dan lingkungan. Oleh sebab itu, perlu dilakukan penelitian untuk mengetahui keragaman genetik spesies tersebut dengan menggunakan penanda DNA melalui pendekatan teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphisms (PCR-RFLP). Teknik tersebut dilakukan untuk mengetahui keragaman genetik mtDNA dengan menggunakan sepasang primer 577 dan 578. Daerah yang diamplifikasi ialah daerah pengontrol yang diapit oleh gen tRNA Pro dan tRNA Phe berdasarkan runutan acuan Dogania subplana. Produk amplifikasi yang dihasilkan sebesar 500 pasang basa dan 710 pasang basa. Produk tersebut dipotong dengan menggunakan lima macam enzim restriksi yaitu, HhaI (GCG• C), HaeIII(GG• CC), AluI (AG• CT ), MboI (• GAT C), dan DraI (TTT• AAA). Dari 39 sampel yang dianalisis ditemukan 12 haplotipe. Haplotipe 2 menyebar pada beberapa wilayah sedangkan haplotipe lain menyebar pada sampel dengan wilayah tertentu saja. Dari 12 haplotipe diperoleh keragaman nukleotida total A. cartilaginea sebesar 5.4%.
ABSTRACT
EVI RIZKY AMELIA. Genetic Diversity of soft-shelled turtle (Amyda cartilaginea). Under supervision of ACHMAD FARAJALLAH and R.R. DYAH PERWITASARI.
Amyda cartilaginea belongs to the Order Testudinata, Suborder Cryptodira and Family Trionychidae. Recently, the population of A. cartilaginea has been decreasing because of human exploitation and environmental destruction. Therefore, it is important to carry on the research in genetic diversity among this spesies. The research was conducted using the mitochondrial DNA marker by Polymerase Chain Reaction-Restriction Fragment Length Polymorphisms (PCR–RFLP) technique. PCR technique was conducted to observe genetic diversity of mtDNA using a pair of primer 577 and 578. The amplified region was control region which flanked tRNA Pro and tRNA Phe based on reference sequence of Dogania subplana. The amplification resulted in the product of 500 base pairs and 710 base pairs. The PCR product was then cut by restriction endonuclease, namely HhaI (GCG• C), HaeIII(GG• CC), AluI (AG• CT ), MboI (• GAT C) and DraI (TTT• AAA). Twelve haplotypes was found among 39 samples.. Haplotype 2 showed wide distribution compared to other haplotypes. Nucleotide diversity among haplotypes is 5.4 %.
ANALISIS KERAGAMAN GENETIK LABI-LABI
(Amyda cartilaginea) BERDASARKAN GENOM MITOKONDRIA
EVI RIZKY AMELIA
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi : ANALISIS KERAGAMAN GENETIK LABI-LABI (Amyda cartilaginea) BERDASARKANGENOM MITOKONDRIA
Nama : Evi Rizky Amelia NIM : G34101008
Menyetujui,
Pembimbing 1 Pembimbing 2
Dr. Ir. Achmad Farajallah, M.Si. Dr. Ir. R.R. Dyah Perwitasari, M.Sc.
NIP : 131878947 NIP : 131916787
Mengetahui,
Dejan Fakultas Matematika dan Ilmu Pengetahuan Alam
Dr. Ir. Yonny Koesmaryono, M.S
NIP : 131473999
RIWAYAT HIDUP
Penulis dilahirkan di Bogor pada tanggal 20 Mei 1983. Penulis merupakan anak kedua
dari enam bersaudara dari pasangan Asep M Yusuf dan Euis Siti Saelah.
Penulis lulus dari SMU Negeri I Leuwiliang pada tahun 2001 dan pada tahun yang sama
penulis lulus seleksi masuk IPB melalui jalur Undangan Seleksi Masuk IPB (USMI). Penulis
memilih jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam.
Selama mengikuti perkuliahan, penulis pernah menjadi asisten praktikum mata kuliah
Vertebrata pada tahun ajaran 2004/2005. Penulis juga pernah mengikuti praktek lapang dengan
judul Manajemen Pemeliharaan Sapi Perah di Kawasan Usaha Peternakan Sapi Perah
PRAKATA
Puji dan syukur penulis panjatkan ke Hadirat Allah SWT atas segala rahmat dan
hidayahNya sehingga karya ilmiah ini berhasil diselesaikan. Judul yang dipilih dalam penelitian ini
adalah Analisis Keragaman Genetik Labi-labi Amyda cartilaginea Berdasarkan Genom
Mitokondria.
Pada kesempatan ini penulis ingin menyampaikan penghargaan dan terima kasih kepada
Bapak Dr. Ir. Achmad Farajallah, M.Si dan Ibu Dr. Ir. RR. Dyah Perwitasari, M.Sc, selaku
pembimbing yang telah memberikan fasilitas, pengarahan, kritik, dan saran selama pelaksanaan
penelitian dan penulisan laporan. Terima kasih penulis sampaikan kepada Ibu Nisa Rachmania
Mubarik sebagai penguji yang telah banyak memberikan kritik dan masukan kepada penulis. Di
samping itu, ucapan terima kasih juga penulis tujukan kepada Bapak Bambang Suryobroto, Ibu
Taruni Sri Prawasti, Bapak Tri Heru, Bapak Tri Atmowidi, Ibu Rika Raffiudin, Bapak Adi.
Ungkapan terima kasih penulis sampaikan kepada Bapak, Mama, Kakek, Nenek, Teh
Rini, Mas Gundul Ignas, Iyank, Leli, dan Adit serta yang terkasih ‘Aa Jembar’ yang telah
memberikan dukungan serta doa dan kasih sayangnya kepada penulis. Tak lupa kepada
teman-teman Zoologi Anne Susanti, Cynthia, Ae, Hijrah, Yudith, Ati, Duti, Fitri, Anne Nelistia dan
teman-teman Biologi 38 terima kasih atas persahabatannya selama ini.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2005
Evi Rizky Amelia
DAFTAR ISI
Halaman
DAFTAR TABEL ... vi
DAFTAR GAMBAR ... vi
DAFTAR LAMPIRAN ... vi
PENDAHULUAN Latar Belakang ... 1
Tujuan ... 1
Waktu dan Tempat ... 1
BAHAN DAN METODE Bahan. ... 2
Metode ... 2
Ekstraksi dan Isolasi DNA... 2
Amplifikasi Ruas DNA Target dengan PCR ... 2
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi ... 2
Analisis Keragaman ... 3
HASIL DAN PEMBAHASAN Hasil... 3
Amplifikasi Ruas DNA Target dengan PCR... 3
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi... 3
Analisis Keragaman... 4
Pembahasan... 4
KESIMPULAN DAN SARAN Kesimpulan... 5
DAFTAR PUSTAKA ... 5
DAFTAR TABEL
Halaman
1 Pola pemotongan enzim restriksi masing-masing haplotipe... 3
2 Keragaman nukleotida antar haplotipe mtDNA A. cartilaginea (di atas diagonal), situs restriksi yang terdapat pada kedua haplotipe (di bawah diagonal) dan jumlah situs restriksi setiap haplotipe (diagona)l... 5
DAFTAR GAMBAR Halaman 1 Labi-labi Amyda cartilaginea... 1
2 Posisi primer 577 dan 578 berdasarkan peta genom labi-labi Dogania subplana (Farajallah 2002) ... 2
DAFTAR LAMPIRAN Halaman 1 Peta situs restriksi dari 12 haplotipe mtDNA A. cartilaginea... 8
2 Peta penyebaran sampel A. cartilaginea yang dianalisis... 9
3 Pola pemotongan Enzim restriksi DraI, MboI, HhaI, AluI dan HaeIII... 9
4 Komposisi larutan gel poliakrilamid 5% larutan pewarnaan perak ... 10
PENDAHULUAN
Latar Belakang
Amyda cartilaginea merupakan kura-kura air tawar bercangkang lunak yang termasuk ke dalam Ordo Testudinata, Subordo Cryptodira, FamiliTrionychidae (Iverson 1992). Famili Trionychidae secara umum disebut dengan labi-labi. Anggota famili ini mempunyai karapas (perisai dorsal) dan plastron (perisai ventral) yang sebagian besar terdiri atas tulang rawan (soft-shelled turtle). Amyda cartilaginea (Gambar 1) mempunyai ciri-ciri kepala berwarna hitam atau abu-abu, kepala hewan muda pada umumnya dijumpai bintik-bintik berwarna kuning; leher relatif panjang, kepala hampir dapat mencapai bagian belakang badan; mata berukuran relatif kecil; lubang hidung terletak di ujung belalai; mulut mempunyai bibir yang tebal; perisai relatif bulat; kaki mempunyai selaput penuh dan jari-jarinya mempunyai cakar yang relatif kuat dan berujung lancip (Iskandar 2000).
Gambar 1 Labi-labi Amyda cartilaginea.
Amyda cartilaginea dapat dijumpai di daerah perairan yang tenang, berarus lambat, kadang-kadang membenam di dalam lumpur atau pasir dan dasar perairan. Daerah penyebaran A. cartilaginea meliputi Myanmar bagian timur dan selatan, Thailand, Kamboja, Laos,Vietnam, Malaysia, Singapura (Lim & Das 1999), sedangkan di Indonesia terdapat di Sumatera, Jawa, Kalimantan (Iskandar 2000). Lim dan Das (1999) melaporkan populasi A. cartilaginea dan spesies kura-kura air tawar lainnya mengalami penurunan karena eksploitasi besar-besaran, khususnya penggunaan spesies ini sebagai makanan eksotis, obat tradisional Cina, dan lemaknya digunakan dalam industri kosmetik. Turtle jelly yang berasal dari perisainya dapat digunakan dalam pengobatan kanker dan juga sebagai obat kuat. Labi-labi kecil umumnya lebih disukai dibanding yang berukuran besar, karena mempunyai proporsi tulang rawan dan gelatinous skin yang tinggi (Behler 1997)..
Genom mitokondria (mtDNA) sering digunakan sebagai penanda genetik dalam mempelajari keragaman genetik hewan dan hubungan sistematik dalam beberapa tingkat hierarki (Lamb et al. 1994). Molekul mtDNA berbentuk sirkular, bersifat haploid karena diturunkan secara maternal, berukuran kecil (15-20 kb), lebih sederhana dibanding DNA inti dan dapat melakukan replikasi sendiri. Genom mitokondria pada sebagian besar hewan terdiri atas 22 gen yang menyandikan tRNA, 2 gen yang menyandikan rRNA (12S dan 16S), 13 gen yang menyandikan protein dan satu ruas DNA berukuran besar yang tidak menyandikan yang disebut sebagai daerah pengontrol atau DLoop (Avise 1994). Daerah pengontrol telah dibuktikan merupakan bagian yang paling bervariasi pada genom mitokondria dan tempat dimulainya replikasi (Avise 1994).
Teknik Polymerase Chain Reaction (PCR) dapat digunakan untuk memperbanyak DNA secara in vitro dengan menggunakan sepasang primer. Variasi antar individu dari produk hasil PCR dapat dikarakterisasi menggunakan berbagai teknik, antara lain Restriction Fragment Length Polymorphisms (RFLP). Dengan teknik ini, setiap enzim restriksi memotong molekul DNA pada runutan nukleotida secara spesifik yang disebut dengan situs restriksi atau situs pemotongan sehingga menghasilkan sejumlah fragmen DNA. Teknik tersebut telah banyak digunakan dalam mempelajari keragaman dalam populasi, filogeografi intraspesifik (Walker et al. 1995), sejarah biogeografi, hibridisasi, pola dan tingkat evolusi dan konservasi (Lawton & Lkiens 2000). Analisis dengan menggunakan teknik RFLP adalah cara yang cepat, relatif murah sehingga dapat digunakan dalam jumlah sampel yang besar.
Tujuan
Waktu dan Tempat
Penelitian ini dilaksanakan dari bulan Februari sampai dengan Juli 2005 di Laboratorium Zoologi Departemen Biologi, FMIPA IPB.
BAHAN DAN METODE
Bahan
Sampel DNA total labi-labi A. cartilaginea yang merupakan koleksi Laboratorium Zoologi Departemen Biologi FMIPA IPB, digunakan dalam penelitian ini. Sampel tersebut berasal dari Pangkalan Bun (sembilan ekor), Senggau (tiga ekor), Banjar Baru (11 ekor), dan Bengkulu (15 ekor). Sampel dalam bentuk DNA total sebanyak 38, sedangkan satu sampel dari Senggau dalam bentuk darah segar (Lampiran 2).
Metode
Ekstraksi dan Isolasi DNA
Sampel darah segar labi-labi asal Senggau diambil dari jantung menggunakan siring berheparin dalam kondisi labi-labi deep anaesthetized. Sel-sel darah dipisahkan dari plasma, dilisis menggunakan NaCl 0.2% yang mengandung 1 mM EDTA (Etilen Diamin Tetraacetic Acid) sebanyak 1x volume, kemudian diendapkan pada kecepatan 5000 g selama 10 menit. Hal ini dimaksudkan untuk membuang sebanyak mungkin molekul hemoglobin. Endapan sel-sel darah sebanyak 200 ìl disuspensikan dalam bufer 1x STE (NaCl 10 mM, Tris HCl 20 mM, EDTA 0.1mM, pH 8.00) 2 ml dan ditambahkan SDS (Sodium Dodesil Sulfat) 10% 200 ìl dan Proteinase-K 50 mg/ml 30 ìl. Campuran ini diinkubasi 550 C selama 2 jam sambil dikocok pelan. Ekstraksi dilanjutkan dengan menambahkan NaCl 5 M 200 ìl, larutan fenol 2 ml dan kloroform : isoamil alkohol (24:1) 2 ml dikocok pelan selama 2 jam dalam suhu ruang pada kecepatan 3500 rpm selama 10 menit. Fase DNA dipisahkan dari fase fenol kemudian dipindahkan ke dalam kantung dialisis DNA dan dilanjutkan dengan dialisis dalam bufer TE (Tris HCl 2 mM dan EDTA 2 mM) dan diinkubasi dalam refrigerator selama tiga malam. Setiap hari bufer TE diganti dengan yang baru. Molekul DNA murni dari kantung dialisis dipindahkan ke tabung penyimpan dan tabung aliquot. Sampel DNA dalam tabung aliquot kemudian yang digunakan dalam penelitian ini.
Amplifikasi Ruas DNA Target dengan PCR
Ruas daerah pengontrol mtDNA diamplifikasi secara in vitro menggunakan PCR ialah daerah pengontrol yang diapit oleh gen tRNA Pro dan tRNA Phe berdasarkan runutan acuan (Gambar 2). Penggandaan DNA ini meng-gunakan pasangan primer 577 (5’-TTC ACG AGA GAT AAG CAA CC-3’) dan primer 578 (5’-CTC GGA TTT TGG GGT TTG AC-3’). Proses PCR menggunakan mesin TaKaRa PCR Thermal Cycler MP4 dengan tahap denaturasi pada suhu 940 C selama 60 detik, penempelan primer pada suhu 520 C selama 2 menit dan sintesis DNA pada suhu 720C selama 2 menit, dan dilakukan sebanyak 30 siklus. Sebelum penggandaan, dilakukan denaturasi awal pada suhu 940 C selama 5 menit. Di akhir penggandaan dilakukan proses sintesis akhir pada suhu 720 C selama 7 menit dan penyimpanan pada suhu 100 C sampai mesin dimatikan.
Gambar 2 Posisi primer 577 dan 578 berdasarkan peta genom labi-labi Dogania subplana (Farajallah 2002)
Komposisi pereaksi PCR sebanyak 50 ì l terdiri atas sampel DNA 2 ì l (10-100 ng), 10x bufer Polimerase 5 ì l, MgCl2 2 mM 4 ì l, dNT P 2.5 mM 6 ì l, Taq Gold Polymerase 1 ì l, primer 577
teknik elektroforesis gel poliakrilamid 5% dan dilanjutkan dengan pewarnaan perak (Tegelstrom 1986 yang dimodifikasi) (Lampiran 4).
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi
Untuk mengetahui keragaman daerah pe-ngontrol, produk PCR dipotong mengguna-kan lima macam enzim restriksi, yaitu HhaI (GCG•C), HaeIII(GG•CC), AluI (AG•CT ), MboI (•GAT C) dan DraI (TTT•AAA). Komposisi pereaksi terdiri atas produk PCR seba-nyak 4 ìl, enzim restriksi sebanyak 2 unit, bufer enzim (sesuai petunjuk produsen) dan air steril sampai volume reaksi 5 ìl. Campuran diinkubasi pada suhu 370 C yang diinapkan semalam. Hasil pemotongan dimigrasikan pada gel poliakrilamid 5 % dengan menggunakan bufer 1x TBE (Tris 0.5 M, Asam Borat 0.65 M, EDTA 0.02 M) 165 mV selama 100 menit. Dalam setiap gel ikut juga dimigrasikan DNA ladder 100 pb (Promega). Visualisasi DNA dalam gel poliakrilamid dilakukan dengan pewarnaan perak (Tegelstrom 1986 yang dimodifikasi).
Analisis Keragaman
Fragmen-fragmen mtDNA hasil pemotongan oleh enzim restriksi kemudian diterjemahkan ke dalam haplotipe. Ukuran fragmen dapat bervariasi diantara individu berdasarkan ada atau tidaknya situs restriksi (Lawton dan Lkiens 2000). Analisis keragaman dihitung dengan metode Nei dan Kumar (2000) berdasarkan ada atau tidaknya situs restriksi. nij : jumlah situs restriksi pada kedua haplotipe i dan j
ni : jumlah situs restriksi pada haplotipe i
nj : jumlah situs restriksi pada haplotipe j
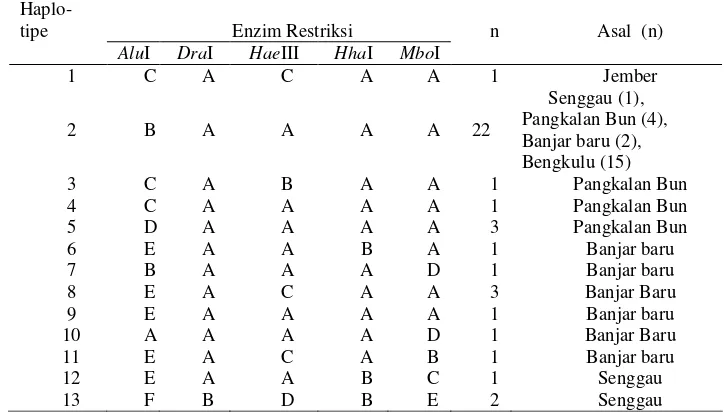
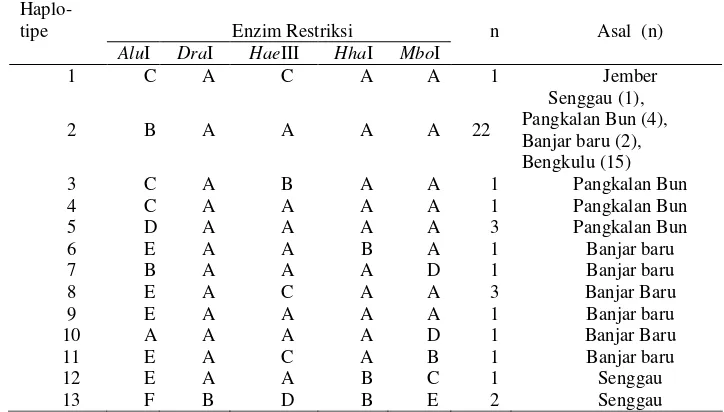
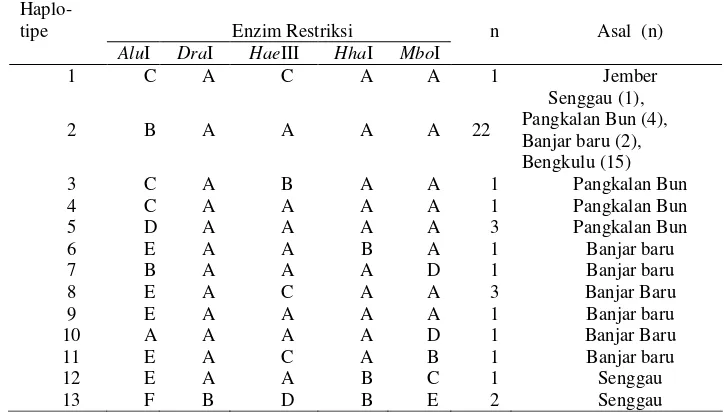
Tabel 1 Pola pemotongan enzim restriksi masing-masing haplotipe
Keterangan : Huruf A-F menunjukkan pola pemotongan enzim restriksi, AluI (A: 220+190+90, B: 215+285, C: 80+90+190+140, D: 420+80, E: 500, F: 80+(300+repeat) ), DraI (A: 145+355, B: 710), HaeIII (A: 52+97+100+251, B: 150+100+250, C: 500, D: 110+600), HhaI (A: 80+430, B: 200+300), MboI (A: 90+410+, B: 220+90+(190+repeat), C: 190+90+220, D: 500, E: 90+620 ).
HASIL DAN PEMBAHASAN Hasil
Haplo-Amplifikasi Ruas DNA Target dengan PCR Amplifikasi DNA terhadap 39 sampel A. cartilaginea menggunakan sepasang primer 577 dan 578 menghasilkan produk PCR sebesar 500 pb untuk sampel yang berasal dari Jember, Senggau, Pangkalan Bun, Banjar Baru. Perbedaan ditemukan pada satu
sampelyang berasal dari Senggau yang mempunyai produk PCR berukuran 710 pb.
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi
Produk PCR yang dipotong dengan enzim AluI, DraI, HaeIII, HhaI dan MboI menghasilkan pola potongan beragam (polimorfik).
Penelitian ini menemukan bahwa setiap haplotipe diwakili oleh satu daerah sampel atau dengan kata lain memiliki pola penyebaran lokal. Haplotipe 1 hanya ditemukan pada sampel yang berasal dari Jawa yang diwakili oleh Jember. Sampel dari Kalimantan mempunyai beberapa haplotipe..
Haplotipe 3, 4 dan 5 untuk sampel dari Pangkalan Bun, haplotipe 6, 7, 8, 9, 10 dan 11 untuk sampel dari Banjar Baru, dan haplotipe 12 dan 13 untuk sampel dari Senggau.
Haplotipe 2 sebagian besar ditemukan pada sampel yang berasal dari Sumatera yang diwakili oleh Bengkulu, namun juga ditemukan pada sampel yang berasal dari Kalimantan yang diwakili oleh Pangkalan Bun, Banjar Baru, dan Senggau.
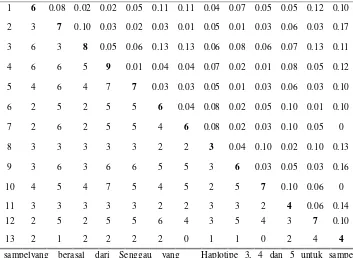
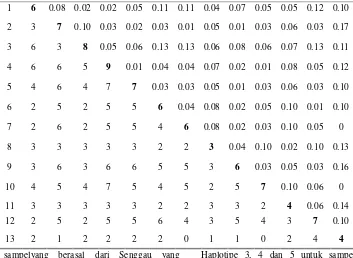
Tabel 2 Keragaman nukleotida antar haplotipe mtDNA A. cartilaginea (di atas diagonal), situs restriksi yang terdapat pada kedua haplotipe (di bawah diagonal) dan jumlah situs restriksi setiap haplotipe (diagonal)
Analisis Keragaman
Nilai keragaman setiap haplotipe dihitung berdasarkan ada atau tidaknya situs restriksi (Tabel 2). Dari ke-13 haplotipe yang ditemukan diperoleh nilai keragaman nukleotida total sebesar 6.1 %
Pembahasan
Perbedaan produk PCR berukuran 710 pb tersebut kemungkinan disebabkan adanya insersi atau sampel tersebut bukan A. cartilaginea.
pb. (Sekarsari 2000). Produk PCR menggunakan primer 577-578 akan memperbanyak domain tengah dari daerah pengontrol beberapa spesies Testudinata berkisar antara 479-501 bp (Farajallah 2002). Perbedaan panjang produk PCR yang berbeda-beda antar spesies bisa disebabkan oleh adanya insersi atau delesi pada ruas DNA. Hal ini mengacu pada hasil PCR dengan primer yang sama untuk labi-labi D. subplana yang juga beragam (Farajallah 2002).
Pemotongan produk PCR dengan enzim restriksi menghasilkan sejumlah fragmen DNA. Penjumlahan panjang fragmen DNA pada gel poliakrilamid akan sama dengan panjang ruas DNA sebelum dipotong. Enzim DraI mempunyai situs pengenalan enam nukleotida, sehingga peluang menemukan satu situs restriksi setiap 46 nukleotida (4096 nukleotida). Enzim-enzim tersebut mempunyai situs pengenalan empat nukleotida, sehingga setiap 44
nukleotida (256
nukleotidaakan ditemukan satu situs pemotongan. Enzim pemotong empat basa akan memotong DNA menjadi potongan yang lebih kecil dibanding enzim pemotong enam basa, sehingga mempunyai peluang yang lebih besar untuk mendapatkan fragmen yang diinginkan dibanding enzim pemotong enam basa.Posisi situs restriksi (Lampiran 1) untuk setiap enzim restriksi bervariasi yang disebabkan adanya perbedaan pada runutan DNA (Snyder dan Champness 1997). Situs restriksi umumnya terdiri atas 4-6 nukleotida atau lebih yang mempunyai urutan basa yang spesifik (Ausubel 1990). Setiap pola situs restriksi disebut haplotipe. Penelitian ini menemukan 13 haplotipe (Tabel 1). Adanya 13 haplotipe yang ditemukan menunjukkan tingkat keragaman yang cukup tinggi pada A. cartilaginea.
Hal ini menunjukkan bahwa keragaman dalam setiap populasi relatif besar yang kemungkinan disebabkan adanya pencampuran dari populasi lain (Farajallah 2002) dan beradaptasi dengan lingkungan sekitar. Selain itu, pada sampel dari Kalimantan didukung oleh keadaan ekologi sungai di Kalimantan yang berubah-ubah di sepanjang alirannya, serta didalamnya terdapat jenis-jenis hewan yang dapat beradaptasi pada kondisi kimia, fisika dan biologi habitatnya yang khas (MacKinnon et al. 2000). Sungai-sungai di Kalimantan terdiri atas sungai besar dan anak sungai meliputi Sungai Kapuas yang membelah Kalimantan
Barat dan sebagian Kalimantan tengah, Sungai Barito yang mengaliri Kalimantan selatan. Sungai-sungai tersebut semakin lebar menuju ke laut, karena ada tambahan air dari anak sungai yang membentuk sungai utama. Kecepatan arus, kedalaman air dan komposisi substrat bervariasi menurut panjang aliran dan lebar sungai sehingga mempengaruhi biota yang dapat hidup didalamnya (MacKinnon et al. 2000).
Haplotipe 2 sebagian besar ditemukan pada sampel yang berasal dari Sumatera yang diwakili oleh Bengkulu, namun juga ditemukan pada sampel yang berasal dari Kalimantan yang diwakili oleh Pangkalan Bun, Banjar Baru, dan Senggau. Haplotipe 2 mempunyai penyebaran yang luas. Haplotipe 2 diperkirakan merupakan haplotipe awal sebelum terjadinya variasi.
Penyebaran haplotipe dalam suatu daerah dapat dihubungkan dengan sejarah geologi dan perubahan iklim pada masa glasial dan interglasial pada jutaan tahun yang lalu (Encalada et al. 1996). Weber dalam Das dan Bauer tahun 1998 berhipotesis bahwa Sungai Musi dan pada pantai timur Sumatera pernah bersambung dengan Sungai Kapuas di Kalimantan bagian barat yang merupakan bagian dari sistem sungai Sunda utara. Hal tersebut dibuktikan dengan adanya kemiripan ikan air tawar antara kedua daerah. Menurut Walker et al. (1995), kura-kura mempunyai model penyebaran yang sama dengan ikan.
Nilai keragaman nukleotida total diperoleh dari nilai rata-rata keragaman nukleotida keseluruhan. Nilai ini lebih rendah dibandingkan dengan nilai keragaman nukleotida mtDNA labi-labi D. subplana sebesar 6,9 % (Sekarsari 2000), Cuora amboinensis (Lestary 2001), dan Cyclemys dentata sebesar 10,6 %. (Indrasari 2002).
KESIMPULAN
DAFTAR PUSTAKA
Ausuble et al. 1990. Current Protocols in Molecular Biology. Volume ke-1. London : Greene Publishing Associates.
Avise JC. 1994. Molecular Markers, Natural History and Evolution. New York : Chapman and Hall.
Behler JL. 1997. Troubles Times for Turtles. New York : Wildlife Conservation Society. Das I, Bauer AM. 1998. Systematics and
biogeography of Borneon Geckos of the
genus Cnemaspis strauch, 1887 (Sauria : Geckonidae), with the description of a new spesies. The Raffl Bull of Zool 46 (1) : 11-28.
Encalada SE, Lahanas PN, Bjorndal KA, Bolten AB, Miyamoto MM. 1996. Phylogeography and population structure of the Atlantic and Mediterranian green turtle Chelonia mydas : a mitochondrial DNA control region sequence assessment. Mol Ecol 5, 473-483.
Farajallah A. 2002. Karakterisasi genom mitokondria labi-labi, Dogania subplana (Trionychidae, Testudines, Reptilia) [disertasi]. Bogor : Program Pascasarjana, Institut Pertanian Bogor.
Indrasari N. 2002. Analisis keragaman genetik kura-kura daun (Cyclemys dentata) dengan teknik PCR-RFLP [skripsi]. Bogor : Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Iskandar DT. 2000. Kura-kura dan Buaya Indonesia dan Papua Nugini. Bandung : PALMedia Citra.
Iverson JB. 1992. A Revised Checklist with Distribution maps of The Turtle. New York : Wildlife Conservation Society.
Lamb T, Lydeard C, Walker RB, Gibbon JW. 1994. Molecular systematics of map turtles (Graptemys) : a comparison of mitochondrial restriction site versus sequences data. Syst Biol. 43 (4):543-559. Lawton JH, Lkiens GE. 2000. Molecular
Methods in Ecology. Oxford : Blackwell Science.
Lestary ID. 2001. Analisis keragaman genetik kura-kura air tawar (Cuora amboinensis) berdasarkan haplotipe mtDNA [skripsi]. Bogor : Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Lim BL, Das I. 1999. Turtle of Borneo and Peninsular Malaysia. Kota Kinabalu : Natural History.
MacKinnon K, Hatta G, Halim H, Mangalik A. 2000. Ekologi Kalimantan. Kartikasari et al, penerjemah. Jakarta : Prenhallindo. Terjemahan dari : The Ecology of Kalimantan.
Nei M, Kumar S. 2000. Molecular Evolution and Phylogenetics. New York : Oxford Univ Pr.
Sekarsari LI. 2000. Analisis keragaman genetik labi-labi, Dogania subplana (Testudines : Trionychidae) melalui teknik PCR-RFLP [skripsi]. Bogor : Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Snyder L, Champness W. 1997. Molecular Genetics of the Bacteria. Washington DC : ASM Press.
Tegelstrom H. 1986. Mitochondrial DNA in natural population : an improved routine for screening of genetic variation based on sensitive silver staining. Electrophoresis 7: 226-229.
52
200
220
80 90 145 280 410
Lampiran 1 Peta situs restriksi dari 13 haplotipe mtDNA A. cartilaginea.
HaeIII HaeIII AluI HaeIII
149 215 249 2
5
HhaI
6
7
8
9
MboI 11
12
HaeIII
110 200 410 420
13 4
10
1
HhaI AluI DraI AluI MboI
420 Alu I Haplotipe
3
250 150
Lampiran 4 Komposisi larutan gel Poliakrilamid 5% dan larutan pewarnaan perak (dalam 200 ml akuades)
1. Akrilamid 30% 3.3 ml 2. 5x TBE (Tris 0.5 M, Asam Borat 0.65 M, EDTA 0.02 M) 4 ml 3. Akuades steril 12.5 ml 4. APS (Ammonium PerSulfat) 10% 150 ìl 5. TEMED (Tetramethylendiamine) 15 ìl
1. CTAB (Cetyl Trimethyl Ammonium Bromide) 0.2 gram 2. Akuades 200 ml 3. Amoniak (NH4OH) 2.4 ml
4. AgNO3 0.32 gram
10 N NaOH 80 ìl NH4OH 0.8 ml
5. Akuades 200 ml 6. Na2CO3 4 gram
Formaldehyde 100 ìl 7. Asam asetat 1% 200 ìl
ANALISIS KERAGAMAN GENETIK LABI-LABI
(Amyda cartilaginea) BERDASARKAN GENOM MITOKONDRIA
EVI RIZKY AMELIA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
ABSTRAK
EVI RIZKY AMELIA. Analisis Keragaman Genetik Labi-labi (Amyda cartilaginea)
Berdasarkan Genom Mitokondria. Dibimbing olehACHMAD FARAJALLAH danR.R. DYAH
PERWITASARI.
Amyda cartilagineatermasuk ke dalam Ordo Testudinata, Subordo Cryptodira dan Famili
Trionychidae. Amyda cartilaginea dilaporkan telah mengalami penurunan populasi yang
diakibatkan oleh pengaruh manusia dan lingkungan. Oleh sebab itu, perlu dilakukan penelitian untuk mengetahui keragaman genetik spesies tersebut dengan menggunakan penanda DNA
melalui pendekatan teknik Polymerase Chain Reaction-Restriction Fragment Length
Polymorphisms (PCR-RFLP). Teknik tersebut dilakukan untuk mengetahui keragaman genetik mtDNA dengan menggunakan sepasang primer 577 dan 578. Daerah yang diamplifikasi ialah
daerah pengontrol yang diapit oleh gen tRNA Pro dan tRNA Phe berdasarkan runutan acuan
Dogania subplana.Produk amplifikasi yang dihasilkan sebesar 500 pasang basa dan 710 pasang
basa. Produk tersebut dipotong dengan menggunakan lima macam enzim restriksi yaitu, HhaI
(GCG• C), HaeIII(GG• CC), AluI (AG• CT ), MboI (• GAT C), danDraI (TTT• AAA). Dari 39 sampel yang dianalisis ditemukan 12 haplotipe. Haplotipe 2 menyebar pada beberapa wilayah sedangkan haplotipe lain menyebar pada sampel dengan wilayah tertentu saja. Dari 12 haplotipe diperoleh keragaman nukleotida totalA. cartilagineasebesar 5.4%.
ABSTRACT
EVI RIZKY AMELIA. Genetic Diversity of soft-shelled turtle (Amyda cartilaginea). Under
supervision ofACHMAD FARAJALLAHand R.R. DYAH PERWITASARI.
PENDAHULUAN
Latar Belakang
Amyda cartilaginea merupakan kura-kura air tawar bercangkang lunak yang termasuk ke dalam Ordo Testudinata, Subordo Cryptodira, FamiliTrionychidae (Iverson 1992). Famili Trionychidae secara umum disebut dengan labi-labi. Anggota famili ini mempunyai karapas (perisai dorsal) dan plastron (perisai ventral) yang sebagian besar terdiri atas tulang rawan (soft-shelled turtle). Amyda cartilaginea (Gambar 1) mempunyai ciri-ciri kepala berwarna hitam atau abu-abu, kepala hewan muda pada umumnya dijumpai bintik-bintik berwarna kuning; leher relatif panjang, kepala hampir dapat mencapai bagian belakang badan; mata berukuran relatif kecil; lubang hidung terletak di ujung belalai; mulut mempunyai bibir yang tebal; perisai relatif bulat; kaki mempunyai selaput penuh dan jari-jarinya mempunyai cakar yang relatif kuat dan berujung lancip (Iskandar 2000).
Gambar 1 Labi-labi Amyda cartilaginea.
Amyda cartilaginea dapat dijumpai di daerah perairan yang tenang, berarus lambat, kadang-kadang membenam di dalam lumpur atau pasir dan dasar perairan. Daerah penyebaran A. cartilaginea meliputi Myanmar bagian timur dan selatan, Thailand, Kamboja, Laos,Vietnam, Malaysia, Singapura (Lim & Das 1999), sedangkan di Indonesia terdapat di Sumatera, Jawa, Kalimantan (Iskandar 2000). Lim dan Das (1999) melaporkan populasi A. cartilaginea dan spesies kura-kura air tawar lainnya mengalami penurunan karena eksploitasi besar-besaran, khususnya penggunaan spesies ini sebagai makanan eksotis, obat tradisional Cina, dan lemaknya digunakan dalam industri kosmetik. Turtle jelly yang berasal dari perisainya dapat digunakan dalam pengobatan kanker dan juga sebagai obat kuat. Labi-labi kecil umumnya lebih disukai dibanding yang berukuran besar, karena mempunyai proporsi tulang rawan dan gelatinous skin yang tinggi (Behler 1997)..
Genom mitokondria (mtDNA) sering digunakan sebagai penanda genetik dalam mempelajari keragaman genetik hewan dan hubungan sistematik dalam beberapa tingkat hierarki (Lamb et al. 1994). Molekul mtDNA berbentuk sirkular, bersifat haploid karena diturunkan secara maternal, berukuran kecil (15-20 kb), lebih sederhana dibanding DNA inti dan dapat melakukan replikasi sendiri. Genom mitokondria pada sebagian besar hewan terdiri atas 22 gen yang menyandikan tRNA, 2 gen yang menyandikan rRNA (12S dan 16S), 13 gen yang menyandikan protein dan satu ruas DNA berukuran besar yang tidak menyandikan yang disebut sebagai daerah pengontrol atau DLoop (Avise 1994). Daerah pengontrol telah dibuktikan merupakan bagian yang paling bervariasi pada genom mitokondria dan tempat dimulainya replikasi (Avise 1994).
Teknik Polymerase Chain Reaction (PCR) dapat digunakan untuk memperbanyak DNA secara in vitro dengan menggunakan sepasang primer. Variasi antar individu dari produk hasil PCR dapat dikarakterisasi menggunakan berbagai teknik, antara lain Restriction Fragment Length Polymorphisms (RFLP). Dengan teknik ini, setiap enzim restriksi memotong molekul DNA pada runutan nukleotida secara spesifik yang disebut dengan situs restriksi atau situs pemotongan sehingga menghasilkan sejumlah fragmen DNA. Teknik tersebut telah banyak digunakan dalam mempelajari keragaman dalam populasi, filogeografi intraspesifik (Walker et al. 1995), sejarah biogeografi, hibridisasi, pola dan tingkat evolusi dan konservasi (Lawton & Lkiens 2000). Analisis dengan menggunakan teknik RFLP adalah cara yang cepat, relatif murah sehingga dapat digunakan dalam jumlah sampel yang besar.
Tujuan
Waktu dan Tempat
Penelitian ini dilaksanakan dari bulan Februari sampai dengan Juli 2005 di Laboratorium Zoologi Departemen Biologi, FMIPA IPB.
BAHAN DAN METODE
Bahan
Sampel DNA total labi-labi A. cartilaginea yang merupakan koleksi Laboratorium Zoologi Departemen Biologi FMIPA IPB, digunakan dalam penelitian ini. Sampel tersebut berasal dari Pangkalan Bun (sembilan ekor), Senggau (tiga ekor), Banjar Baru (11 ekor), dan Bengkulu (15 ekor). Sampel dalam bentuk DNA total sebanyak 38, sedangkan satu sampel dari Senggau dalam bentuk darah segar (Lampiran 2).
Metode
Ekstraksi dan Isolasi DNA
Sampel darah segar labi-labi asal Senggau diambil dari jantung menggunakan siring berheparin dalam kondisi labi-labi deep anaesthetized. Sel-sel darah dipisahkan dari plasma, dilisis menggunakan NaCl 0.2% yang mengandung 1 mM EDTA (Etilen Diamin Tetraacetic Acid) sebanyak 1x volume, kemudian diendapkan pada kecepatan 5000 g selama 10 menit. Hal ini dimaksudkan untuk membuang sebanyak mungkin molekul hemoglobin. Endapan sel-sel darah sebanyak 200 ìl disuspensikan dalam bufer 1x STE (NaCl 10 mM, Tris HCl 20 mM, EDTA 0.1mM, pH 8.00) 2 ml dan ditambahkan SDS (Sodium Dodesil Sulfat) 10% 200 ìl dan Proteinase-K 50 mg/ml 30 ìl. Campuran ini diinkubasi 550 C selama 2 jam sambil dikocok pelan. Ekstraksi dilanjutkan dengan menambahkan NaCl 5 M 200 ìl, larutan fenol 2 ml dan kloroform : isoamil alkohol (24:1) 2 ml dikocok pelan selama 2 jam dalam suhu ruang pada kecepatan 3500 rpm selama 10 menit. Fase DNA dipisahkan dari fase fenol kemudian dipindahkan ke dalam kantung dialisis DNA dan dilanjutkan dengan dialisis dalam bufer TE (Tris HCl 2 mM dan EDTA 2 mM) dan diinkubasi dalam refrigerator selama tiga malam. Setiap hari bufer TE diganti dengan yang baru. Molekul DNA murni dari kantung dialisis dipindahkan ke tabung penyimpan dan tabung aliquot. Sampel DNA dalam tabung aliquot kemudian yang digunakan dalam penelitian ini.
Amplifikasi Ruas DNA Target dengan PCR
Ruas daerah pengontrol mtDNA diamplifikasi secara in vitro menggunakan PCR ialah daerah pengontrol yang diapit oleh gen tRNA Pro dan tRNA Phe berdasarkan runutan acuan (Gambar 2). Penggandaan DNA ini meng-gunakan pasangan primer 577 (5’-TTC ACG AGA GAT AAG CAA CC-3’) dan primer 578 (5’-CTC GGA TTT TGG GGT TTG AC-3’). Proses PCR menggunakan mesin TaKaRa PCR Thermal Cycler MP4 dengan tahap denaturasi pada suhu 940 C selama 60 detik, penempelan primer pada suhu 520 C selama 2 menit dan sintesis DNA pada suhu 720C selama 2 menit, dan dilakukan sebanyak 30 siklus. Sebelum penggandaan, dilakukan denaturasi awal pada suhu 940 C selama 5 menit. Di akhir penggandaan dilakukan proses sintesis akhir pada suhu 720 C selama 7 menit dan penyimpanan pada suhu 100 C sampai mesin dimatikan.
Gambar 2 Posisi primer 577 dan 578 berdasarkan peta genom labi-labi Dogania subplana (Farajallah 2002)
Komposisi pereaksi PCR sebanyak 50 ì l terdiri atas sampel DNA 2 ì l (10-100 ng), 10x bufer Polimerase 5 ì l, MgCl2 2 mM 4 ì l, dNT P 2.5 mM 6 ì l, Taq Gold Polymerase 1 ì l, primer 577
Waktu dan Tempat
Penelitian ini dilaksanakan dari bulan Februari sampai dengan Juli 2005 di Laboratorium Zoologi Departemen Biologi, FMIPA IPB.
BAHAN DAN METODE
Bahan
Sampel DNA total labi-labi A. cartilaginea yang merupakan koleksi Laboratorium Zoologi Departemen Biologi FMIPA IPB, digunakan dalam penelitian ini. Sampel tersebut berasal dari Pangkalan Bun (sembilan ekor), Senggau (tiga ekor), Banjar Baru (11 ekor), dan Bengkulu (15 ekor). Sampel dalam bentuk DNA total sebanyak 38, sedangkan satu sampel dari Senggau dalam bentuk darah segar (Lampiran 2).
Metode
Ekstraksi dan Isolasi DNA
Sampel darah segar labi-labi asal Senggau diambil dari jantung menggunakan siring berheparin dalam kondisi labi-labi deep anaesthetized. Sel-sel darah dipisahkan dari plasma, dilisis menggunakan NaCl 0.2% yang mengandung 1 mM EDTA (Etilen Diamin Tetraacetic Acid) sebanyak 1x volume, kemudian diendapkan pada kecepatan 5000 g selama 10 menit. Hal ini dimaksudkan untuk membuang sebanyak mungkin molekul hemoglobin. Endapan sel-sel darah sebanyak 200 ìl disuspensikan dalam bufer 1x STE (NaCl 10 mM, Tris HCl 20 mM, EDTA 0.1mM, pH 8.00) 2 ml dan ditambahkan SDS (Sodium Dodesil Sulfat) 10% 200 ìl dan Proteinase-K 50 mg/ml 30 ìl. Campuran ini diinkubasi 550 C selama 2 jam sambil dikocok pelan. Ekstraksi dilanjutkan dengan menambahkan NaCl 5 M 200 ìl, larutan fenol 2 ml dan kloroform : isoamil alkohol (24:1) 2 ml dikocok pelan selama 2 jam dalam suhu ruang pada kecepatan 3500 rpm selama 10 menit. Fase DNA dipisahkan dari fase fenol kemudian dipindahkan ke dalam kantung dialisis DNA dan dilanjutkan dengan dialisis dalam bufer TE (Tris HCl 2 mM dan EDTA 2 mM) dan diinkubasi dalam refrigerator selama tiga malam. Setiap hari bufer TE diganti dengan yang baru. Molekul DNA murni dari kantung dialisis dipindahkan ke tabung penyimpan dan tabung aliquot. Sampel DNA dalam tabung aliquot kemudian yang digunakan dalam penelitian ini.
Amplifikasi Ruas DNA Target dengan PCR
Ruas daerah pengontrol mtDNA diamplifikasi secara in vitro menggunakan PCR ialah daerah pengontrol yang diapit oleh gen tRNA Pro dan tRNA Phe berdasarkan runutan acuan (Gambar 2). Penggandaan DNA ini meng-gunakan pasangan primer 577 (5’-TTC ACG AGA GAT AAG CAA CC-3’) dan primer 578 (5’-CTC GGA TTT TGG GGT TTG AC-3’). Proses PCR menggunakan mesin TaKaRa PCR Thermal Cycler MP4 dengan tahap denaturasi pada suhu 940 C selama 60 detik, penempelan primer pada suhu 520 C selama 2 menit dan sintesis DNA pada suhu 720C selama 2 menit, dan dilakukan sebanyak 30 siklus. Sebelum penggandaan, dilakukan denaturasi awal pada suhu 940 C selama 5 menit. Di akhir penggandaan dilakukan proses sintesis akhir pada suhu 720 C selama 7 menit dan penyimpanan pada suhu 100 C sampai mesin dimatikan.
Gambar 2 Posisi primer 577 dan 578 berdasarkan peta genom labi-labi Dogania subplana (Farajallah 2002)
Komposisi pereaksi PCR sebanyak 50 ì l terdiri atas sampel DNA 2 ì l (10-100 ng), 10x bufer Polimerase 5 ì l, MgCl2 2 mM 4 ì l, dNT P 2.5 mM 6 ì l, Taq Gold Polymerase 1 ì l, primer 577
teknik elektroforesis gel poliakrilamid 5% dan dilanjutkan dengan pewarnaan perak (Tegelstrom 1986 yang dimodifikasi) (Lampiran 4).
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi
Untuk mengetahui keragaman daerah pe-ngontrol, produk PCR dipotong mengguna-kan lima macam enzim restriksi, yaitu HhaI (GCG•C), HaeIII(GG•CC), AluI (AG•CT ), MboI (•GAT C) dan DraI (TTT•AAA). Komposisi pereaksi terdiri atas produk PCR seba-nyak 4 ìl, enzim restriksi sebanyak 2 unit, bufer enzim (sesuai petunjuk produsen) dan air steril sampai volume reaksi 5 ìl. Campuran diinkubasi pada suhu 370 C yang diinapkan semalam. Hasil pemotongan dimigrasikan pada gel poliakrilamid 5 % dengan menggunakan bufer 1x TBE (Tris 0.5 M, Asam Borat 0.65 M, EDTA 0.02 M) 165 mV selama 100 menit. Dalam setiap gel ikut juga dimigrasikan DNA ladder 100 pb (Promega). Visualisasi DNA dalam gel poliakrilamid dilakukan dengan pewarnaan perak (Tegelstrom 1986 yang dimodifikasi).
Analisis Keragaman
Fragmen-fragmen mtDNA hasil pemotongan oleh enzim restriksi kemudian diterjemahkan ke dalam haplotipe. Ukuran fragmen dapat bervariasi diantara individu berdasarkan ada atau tidaknya situs restriksi (Lawton dan Lkiens 2000). Analisis keragaman dihitung dengan metode Nei dan Kumar (2000) berdasarkan ada atau tidaknya situs restriksi. nij : jumlah situs restriksi pada kedua haplotipe i dan j
ni : jumlah situs restriksi pada haplotipe i
nj : jumlah situs restriksi pada haplotipe j
Tabel 1 Pola pemotongan enzim restriksi masing-masing haplotipe
Keterangan : Huruf A-F menunjukkan pola pemotongan enzim restriksi, AluI (A: 220+190+90, B: 215+285, C: 80+90+190+140, D: 420+80, E: 500, F: 80+(300+repeat) ), DraI (A: 145+355, B: 710), HaeIII (A: 52+97+100+251, B: 150+100+250, C: 500, D: 110+600), HhaI (A: 80+430, B: 200+300), MboI (A: 90+410+, B: 220+90+(190+repeat), C: 190+90+220, D: 500, E: 90+620 ).
HASIL DAN PEMBAHASAN Hasil
Haplo-teknik elektroforesis gel poliakrilamid 5% dan dilanjutkan dengan pewarnaan perak (Tegelstrom 1986 yang dimodifikasi) (Lampiran 4).
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi
Untuk mengetahui keragaman daerah pe-ngontrol, produk PCR dipotong mengguna-kan lima macam enzim restriksi, yaitu HhaI (GCG•C), HaeIII(GG•CC), AluI (AG•CT ), MboI (•GAT C) dan DraI (TTT•AAA). Komposisi pereaksi terdiri atas produk PCR seba-nyak 4 ìl, enzim restriksi sebanyak 2 unit, bufer enzim (sesuai petunjuk produsen) dan air steril sampai volume reaksi 5 ìl. Campuran diinkubasi pada suhu 370 C yang diinapkan semalam. Hasil pemotongan dimigrasikan pada gel poliakrilamid 5 % dengan menggunakan bufer 1x TBE (Tris 0.5 M, Asam Borat 0.65 M, EDTA 0.02 M) 165 mV selama 100 menit. Dalam setiap gel ikut juga dimigrasikan DNA ladder 100 pb (Promega). Visualisasi DNA dalam gel poliakrilamid dilakukan dengan pewarnaan perak (Tegelstrom 1986 yang dimodifikasi).
Analisis Keragaman
Fragmen-fragmen mtDNA hasil pemotongan oleh enzim restriksi kemudian diterjemahkan ke dalam haplotipe. Ukuran fragmen dapat bervariasi diantara individu berdasarkan ada atau tidaknya situs restriksi (Lawton dan Lkiens 2000). Analisis keragaman dihitung dengan metode Nei dan Kumar (2000) berdasarkan ada atau tidaknya situs restriksi. nij : jumlah situs restriksi pada kedua haplotipe i dan j
ni : jumlah situs restriksi pada haplotipe i
nj : jumlah situs restriksi pada haplotipe j
Tabel 1 Pola pemotongan enzim restriksi masing-masing haplotipe
Keterangan : Huruf A-F menunjukkan pola pemotongan enzim restriksi, AluI (A: 220+190+90, B: 215+285, C: 80+90+190+140, D: 420+80, E: 500, F: 80+(300+repeat) ), DraI (A: 145+355, B: 710), HaeIII (A: 52+97+100+251, B: 150+100+250, C: 500, D: 110+600), HhaI (A: 80+430, B: 200+300), MboI (A: 90+410+, B: 220+90+(190+repeat), C: 190+90+220, D: 500, E: 90+620 ).
HASIL DAN PEMBAHASAN Hasil
Haplo-Amplifikasi Ruas DNA Target dengan PCR Amplifikasi DNA terhadap 39 sampel A. cartilaginea menggunakan sepasang primer 577 dan 578 menghasilkan produk PCR sebesar 500 pb untuk sampel yang berasal dari Jember, Senggau, Pangkalan Bun, Banjar Baru. Perbedaan ditemukan pada satu
sampelyang berasal dari Senggau yang mempunyai produk PCR berukuran 710 pb.
Pemotongan Hasil Amplifikasi dengan Enzim Restriksi
Produk PCR yang dipotong dengan enzim AluI, DraI, HaeIII, HhaI dan MboI menghasilkan pola potongan beragam (polimorfik).
Penelitian ini menemukan bahwa setiap haplotipe diwakili oleh satu daerah sampel atau dengan kata lain memiliki pola penyebaran lokal. Haplotipe 1 hanya ditemukan pada sampel yang berasal dari Jawa yang diwakili oleh Jember. Sampel dari Kalimantan mempunyai beberapa haplotipe..
Haplotipe 3, 4 dan 5 untuk sampel dari Pangkalan Bun, haplotipe 6, 7, 8, 9, 10 dan 11 untuk sampel dari Banjar Baru, dan haplotipe 12 dan 13 untuk sampel dari Senggau.
Haplotipe 2 sebagian besar ditemukan pada sampel yang berasal dari Sumatera yang diwakili oleh Bengkulu, namun juga ditemukan pada sampel yang berasal dari Kalimantan yang diwakili oleh Pangkalan Bun, Banjar Baru, dan Senggau.
Tabel 2 Keragaman nukleotida antar haplotipe mtDNA A. cartilaginea (di atas diagonal), situs restriksi yang terdapat pada kedua haplotipe (di bawah diagonal) dan jumlah situs restriksi setiap haplotipe (diagonal)
Analisis Keragaman
Nilai keragaman setiap haplotipe dihitung berdasarkan ada atau tidaknya situs restriksi (Tabel 2). Dari ke-13 haplotipe yang ditemukan diperoleh nilai keragaman nukleotida total sebesar 6.1 %
Pembahasan
Perbedaan produk PCR berukuran 710 pb tersebut kemungkinan disebabkan adanya insersi atau sampel tersebut bukan A. cartilaginea.
pb. (Sekarsari 2000). Produk PCR menggunakan primer 577-578 akan memperbanyak domain tengah dari daerah pengontrol beberapa spesies Testudinata berkisar antara 479-501 bp (Farajallah 2002). Perbedaan panjang produk PCR yang berbeda-beda antar spesies bisa disebabkan oleh adanya insersi atau delesi pada ruas DNA. Hal ini mengacu pada hasil PCR dengan primer yang sama untuk labi-labi D. subplana yang juga beragam (Farajallah 2002).
Pemotongan produk PCR dengan enzim restriksi menghasilkan sejumlah fragmen DNA. Penjumlahan panjang fragmen DNA pada gel poliakrilamid akan sama dengan panjang ruas DNA sebelum dipotong. Enzim DraI mempunyai situs pengenalan enam nukleotida, sehingga peluang menemukan satu situs restriksi setiap 46 nukleotida (4096 nukleotida). Enzim-enzim tersebut mempunyai situs pengenalan empat nukleotida, sehingga setiap 44
nukleotida (256
nukleotidaakan ditemukan satu situs pemotongan. Enzim pemotong empat basa akan memotong DNA menjadi potongan yang lebih kecil dibanding enzim pemotong enam basa, sehingga mempunyai peluang yang lebih besar untuk mendapatkan fragmen yang diinginkan dibanding enzim pemotong enam basa.Posisi situs restriksi (Lampiran 1) untuk setiap enzim restriksi bervariasi yang disebabkan adanya perbedaan pada runutan DNA (Snyder dan Champness 1997). Situs restriksi umumnya terdiri atas 4-6 nukleotida atau lebih yang mempunyai urutan basa yang spesifik (Ausubel 1990). Setiap pola situs restriksi disebut haplotipe. Penelitian ini menemukan 13 haplotipe (Tabel 1). Adanya 13 haplotipe yang ditemukan menunjukkan tingkat keragaman yang cukup tinggi pada A. cartilaginea.
Hal ini menunjukkan bahwa keragaman dalam setiap populasi relatif besar yang kemungkinan disebabkan adanya pencampuran dari populasi lain (Farajallah 2002) dan beradaptasi dengan lingkungan sekitar. Selain itu, pada sampel dari Kalimantan didukung oleh keadaan ekologi sungai di Kalimantan yang berubah-ubah di sepanjang alirannya, serta didalamnya terdapat jenis-jenis hewan yang dapat beradaptasi pada kondisi kimia, fisika dan biologi habitatnya yang khas (MacKinnon et al. 2000). Sungai-sungai di Kalimantan terdiri atas sungai besar dan anak sungai meliputi Sungai Kapuas yang membelah Kalimantan
Barat dan sebagian Kalimantan tengah, Sungai Barito yang mengaliri Kalimantan selatan. Sungai-sungai tersebut semakin lebar menuju ke laut, karena ada tambahan air dari anak sungai yang membentuk sungai utama. Kecepatan arus, kedalaman air dan komposisi substrat bervariasi menurut panjang aliran dan lebar sungai sehingga mempengaruhi biota yang dapat hidup didalamnya (MacKinnon et al. 2000).
Haplotipe 2 sebagian besar ditemukan pada sampel yang berasal dari Sumatera yang diwakili oleh Bengkulu, namun juga ditemukan pada sampel yang berasal dari Kalimantan yang diwakili oleh Pangkalan Bun, Banjar Baru, dan Senggau. Haplotipe 2 mempunyai penyebaran yang luas. Haplotipe 2 diperkirakan merupakan haplotipe awal sebelum terjadinya variasi.
Penyebaran haplotipe dalam suatu daerah dapat dihubungkan dengan sejarah geologi dan perubahan iklim pada masa glasial dan interglasial pada jutaan tahun yang lalu (Encalada et al. 1996). Weber dalam Das dan Bauer tahun 1998 berhipotesis bahwa Sungai Musi dan pada pantai timur Sumatera pernah bersambung dengan Sungai Kapuas di Kalimantan bagian barat yang merupakan bagian dari sistem sungai Sunda utara. Hal tersebut dibuktikan dengan adanya kemiripan ikan air tawar antara kedua daerah. Menurut Walker et al. (1995), kura-kura mempunyai model penyebaran yang sama dengan ikan.
Nilai keragaman nukleotida total diperoleh dari nilai rata-rata keragaman nukleotida keseluruhan. Nilai ini lebih rendah dibandingkan dengan nilai keragaman nukleotida mtDNA labi-labi D. subplana sebesar 6,9 % (Sekarsari 2000), Cuora amboinensis (Lestary 2001), dan Cyclemys dentata sebesar 10,6 %. (Indrasari 2002).
KESIMPULAN
pb. (Sekarsari 2000). Produk PCR menggunakan primer 577-578 akan memperbanyak domain tengah dari daerah pengontrol beberapa spesies Testudinata berkisar antara 479-501 bp (Farajallah 2002). Perbedaan panjang produk PCR yang berbeda-beda antar spesies bisa disebabkan oleh adanya insersi atau delesi pada ruas DNA. Hal ini mengacu pada hasil PCR dengan primer yang sama untuk labi-labi D. subplana yang juga beragam (Farajallah 2002).
Pemotongan produk PCR dengan enzim restriksi menghasilkan sejumlah fragmen DNA. Penjumlahan panjang fragmen DNA pada gel poliakrilamid akan sama dengan panjang ruas DNA sebelum dipotong. Enzim DraI mempunyai situs pengenalan enam nukleotida, sehingga peluang menemukan satu situs restriksi setiap 46 nukleotida (4096 nukleotida). Enzim-enzim tersebut mempunyai situs pengenalan empat nukleotida, sehingga setiap 44
nukleotida (256
nukleotidaakan ditemukan satu situs pemotongan. Enzim pemotong empat basa akan memotong DNA menjadi potongan yang lebih kecil dibanding enzim pemotong enam basa, sehingga mempunyai peluang yang lebih besar untuk mendapatkan fragmen yang diinginkan dibanding enzim pemotong enam basa.Posisi situs restriksi (Lampiran 1) untuk setiap enzim restriksi bervariasi yang disebabkan adanya perbedaan pada runutan DNA (Snyder dan Champness 1997). Situs restriksi umumnya terdiri atas 4-6 nukleotida atau lebih yang mempunyai urutan basa yang spesifik (Ausubel 1990). Setiap pola situs restriksi disebut haplotipe. Penelitian ini menemukan 13 haplotipe (Tabel 1). Adanya 13 haplotipe yang ditemukan menunjukkan tingkat keragaman yang cukup tinggi pada A. cartilaginea.
Hal ini menunjukkan bahwa keragaman dalam setiap populasi relatif besar yang kemungkinan disebabkan adanya pencampuran dari populasi lain (Farajallah 2002) dan beradaptasi dengan lingkungan sekitar. Selain itu, pada sampel dari Kalimantan didukung oleh keadaan ekologi sungai di Kalimantan yang berubah-ubah di sepanjang alirannya, serta didalamnya terdapat jenis-jenis hewan yang dapat beradaptasi pada kondisi kimia, fisika dan biologi habitatnya yang khas (MacKinnon et al. 2000). Sungai-sungai di Kalimantan terdiri atas sungai besar dan anak sungai meliputi Sungai Kapuas yang membelah Kalimantan
Barat dan sebagian Kalimantan tengah, Sungai Barito yang mengaliri Kalimantan selatan. Sungai-sungai tersebut semakin lebar menuju ke laut, karena ada tambahan air dari anak sungai yang membentuk sungai utama. Kecepatan arus, kedalaman air dan komposisi substrat bervariasi menurut panjang aliran dan lebar sungai sehingga mempengaruhi biota yang dapat hidup didalamnya (MacKinnon et al. 2000).
Haplotipe 2 sebagian besar ditemukan pada sampel yang berasal dari Sumatera yang diwakili oleh Bengkulu, namun juga ditemukan pada sampel yang berasal dari Kalimantan yang diwakili oleh Pangkalan Bun, Banjar Baru, dan Senggau. Haplotipe 2 mempunyai penyebaran yang luas. Haplotipe 2 diperkirakan merupakan haplotipe awal sebelum terjadinya variasi.
Penyebaran haplotipe dalam suatu daerah dapat dihubungkan dengan sejarah geologi dan perubahan iklim pada masa glasial dan interglasial pada jutaan tahun yang lalu (Encalada et al. 1996). Weber dalam Das dan Bauer tahun 1998 berhipotesis bahwa Sungai Musi dan pada pantai timur Sumatera pernah bersambung dengan Sungai Kapuas di Kalimantan bagian barat yang merupakan bagian dari sistem sungai Sunda utara. Hal tersebut dibuktikan dengan adanya kemiripan ikan air tawar antara kedua daerah. Menurut Walker et al. (1995), kura-kura mempunyai model penyebaran yang sama dengan ikan.
Nilai keragaman nukleotida total diperoleh dari nilai rata-rata keragaman nukleotida keseluruhan. Nilai ini lebih rendah dibandingkan dengan nilai keragaman nukleotida mtDNA labi-labi D. subplana sebesar 6,9 % (Sekarsari 2000), Cuora amboinensis (Lestary 2001), dan Cyclemys dentata sebesar 10,6 %. (Indrasari 2002).
KESIMPULAN
DAFTAR PUSTAKA
Ausuble et al. 1990. Current Protocols in Molecular Biology. Volume ke-1. London : Greene Publishing Associates.
Avise JC. 1994. Molecular Markers, Natural History and Evolution. New York : Chapman and Hall.
Behler JL. 1997. Troubles Times for Turtles. New York : Wildlife Conservation Society. Das I, Bauer AM. 1998. Systematics and
biogeography of Borneon Geckos of the
genus Cnemaspis strauch, 1887 (Sauria : Geckonidae), with the description of a new spesies. The Raffl Bull of Zool 46 (1) : 11-28.
Encalada SE, Lahanas PN, Bjorndal KA, Bolten AB, Miyamoto MM. 1996. Phylogeography and population structure of the Atlantic and Mediterranian green turtle Chelonia mydas : a mitochondrial DNA control region sequence assessment. Mol Ecol 5, 473-483.
Farajallah A. 2002. Karakterisasi genom mitokondria labi-labi, Dogania subplana (Trionychidae, Testudines, Reptilia) [disertasi]. Bogor : Program Pascasarjana, Institut Pertanian Bogor.
Indrasari N. 2002. Analisis keragaman genetik kura-kura daun (Cyclemys dentata) dengan teknik PCR-RFLP [skripsi]. Bogor : Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Iskandar DT. 2000. Kura-kura dan Buaya Indonesia dan Papua Nugini. Bandung : PALMedia Citra.
Iverson JB. 1992. A Revised Checklist with Distribution maps of The Turtle. New York : Wildlife Conservation Society.
Lamb T, Lydeard C, Walker RB, Gibbon JW. 1994. Molecular systematics of map turtles (Graptemys) : a comparison of mitochondrial restriction site versus sequences data. Syst Biol. 43 (4):543-559. Lawton JH, Lkiens GE. 2000. Molecular
Methods in Ecology. Oxford : Blackwell Science.
Lestary ID. 2001. Analisis keragaman genetik kura-kura air tawar (Cuora amboinensis) berdasarkan haplotipe mtDNA [skripsi]. Bogor : Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Lim BL, Das I. 1999. Turtle of Borneo and Peninsular Malaysia. Kota Kinabalu : Natural History.
MacKinnon K, Hatta G, Halim H, Mangalik A. 2000. Ekologi Kalimantan. Kartikasari et al, penerjemah. Jakarta : Prenhallindo. Terjemahan dari : The Ecology of Kalimantan.
Nei M, Kumar S. 2000. Molecular Evolution and Phylogenetics. New York : Oxford Univ Pr.
Sekarsari LI. 2000. Analisis keragaman genetik labi-labi, Dogania subplana (Testudines : Trionychidae) melalui teknik PCR-RFLP [skripsi]. Bogor : Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Snyder L, Champness W. 1997. Molecular Genetics of the Bacteria. Washington DC : ASM Press.
Tegelstrom H. 1986. Mitochondrial DNA in natural population : an improved routine for screening of genetic variation based on sensitive silver staining. Electrophoresis 7: 226-229.
52
200
220
80 90 145 280 410
Lampiran 1 Peta situs restriksi dari 13 haplotipe mtDNA A. cartilaginea.
HaeIII HaeIII AluI HaeIII
149 215 249 2
5
HhaI
6
7
8
9
MboI 11
12
HaeIII
110 200 410 420
13 4
10
1
HhaI AluI DraI AluI MboI
420 Alu I Haplotipe
3
250 150
Lampiran 4 Komposisi larutan gel Poliakrilamid 5% dan larutan pewarnaan perak (dalam 200 ml akuades)
1. Akrilamid 30% 3.3 ml 2. 5x TBE (Tris 0.5 M, Asam Borat 0.65 M, EDTA 0.02 M) 4 ml 3. Akuades steril 12.5 ml 4. APS (Ammonium PerSulfat) 10% 150 ìl 5. TEMED (Tetramethylendiamine) 15 ìl
1. CTAB (Cetyl Trimethyl Ammonium Bromide) 0.2 gram 2. Akuades 200 ml 3. Amoniak (NH4OH) 2.4 ml
4. AgNO3 0.32 gram
10 N NaOH 80 ìl NH4OH 0.8 ml
5. Akuades 200 ml 6. Na2CO3 4 gram
Formaldehyde 100 ìl 7. Asam asetat 1% 200 ìl