POLA DISTRIBUSI PENYU LEKANG (
Lepidochelys olivacea
)
BERDASARKAN STRUKTUR GENETIK DI TELUK
CENDRAWASIH, PULAU KAPOPOSANG, DAN KUPANG
NUZHA ZATU SHABRINA
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa Skripsi yang berjudul Pola Distribusi Penyu Lekang (Lepidochelys olivacea) Berdasarkan Struktur Genetik di Teluk Cendrawasih, Pulau Kapoposang, dan Kupang adalah benar merupakan hasil karya sendiri dengan arahan dari Komisi Pembimbing dan belum diajukan dalam bentuk apapun kepada Perguruan Tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan tercantum dalam Daftar Pustaka di bagian akhir Skripsi ini.
Dengan ini saya melimpahkan hak cipta dan karya tulis ini kepada Institut Pertanian Bogor
Bogor, Agustus 2015
Nuzha Zatu Shabrina
ABSTRAK
NUZHA ZATU SHABRINA. Pola Distribusi Penyu Lekang (Lepidochelys olivacea) Berdasarkan Struktur Genetik di Teluk Cendrawasih, Pulau Kapoposang, dan Kupang. Dibimbing oleh HAWIS MADDUPPA dan NYOMAN M.N. NATIH.
Penyu lekang (Lepidochelys olivacea) adalah salah satu jenis penyu yang dapat ditemukan di Indonesia. Pola distribusi penyu lekang (Lepidochelys olivacea) berdasarkan struktur genetik kurang dikenal. Oleh karena itu, penelitian ini bertujuan untuk mengetahui pola distribusi penyu lekang (Lepidochelys olivacea) berdasarkan struktur genetik mitokondria. DNA sitokrom oksidase 1 (CO1) digunakan sebagai penanda. Pola distribusi menunjukkan bahwa tidak adanya struktur genetik antar populasi penyu lekang (Lepidochelys olivacea) antara tiga populasi Teluk Cendrawasih, Pulau Kapoposang, dan Kupang (FST = -0,023; P-value = 0,462). Keragaman genetik yang tinggi diamati pada tiga populasi (h = 1.00 +/- 0,04). Aliran gen tinggi tampaknya dipengaruhi oleh perilaku migrasi penyu. Pola migrasi penyu lekang bergerak dalam perairan Indonesia.
Kata kunci: Lepidochelys olivacea, DNA Barcoding, struktur genetik, migrasi
ABSTRACT
NUZHA ZATU SHABRINA. The Distribution Pattern of Olive Ridley Sea Turtle (Lepidochelys olivacea) Based on Its Genetic Structure in Cendrawasih Bay, Kapoposang Island, and Kupang. Supervised by HAWIS MADDUPPA and NYOMAN M.N. NATIH.
The olive ridley sea turtle (Lepidochelys olivacea) is one species of sea turtles that can be found in Indonesia. The distribution pattern of olive ridley turtle (Lepidochelys olivacea) based on the genetic structure is poorly known. Therefore, this study aimed to determine the distribution pattern of olive ridley sea turtle (Lepidochelys olivacea) based on the genetic structure Mitochondrial. DNA Cytochrome Oxidase 1 (CO1) was used as marker. The distribution pattern indicated that the absence of inter-population genetic structure of olive ridley turtle (Lepidochelys olivacea) between three populations of Cendrawasih Bay, Kapoposang Island, and Kupang (FST=-0.023;P-value=0.462). High genetic diversity was observed in three populations (h=1.00+/-0.04). High gene flow seems influenced by the behavior of turtle migration. The olive ridley turtle migration patterns moving within the Indonesian waters.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan
pada
Departemen Ilmu dan Teknologi Kelautan
POLA DISTRIBUSI PENYU LEKANG (
Lepidochelys olivacea
)
BERDASARKAN STRUKTUR GENETIK DI TELUK
CENDRAWASIH, PULAU KAPOPOSANG, DAN KUPANG
NUZHA ZATU SHABRINA
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PRAKATA
Puji Syukur penulis panjatkan kepada Allah SWT atas rahmat dan anugerah-Nya sehingga penulis dapat menyelesaikan penyusunan skripsi berjudul “Pola Distribusi Penyu Lekang (Lepidochelys olivacea) Berdasarkan Struktur Genetik di Teluk Cendrawasih, Pulau Kapoposang, dan Kupang”. Penelitian ini merupakan tugas akhir sebagai salah satu syarat untuk memperoleh gelar kesarjanaan di Departemen Ilmu dan Teknologi Kelautan yaitu Sarjana Ilmu Kelautan.
Terima kasih penulis ucapkan kepada Bapak Dr. Hawis H. Madduppa, S.Pi, M.Si dan Bapak Dr. Ir. Nyoman M.N. Natih, M.Si selaku dosen pembimbing yang telah memberikan arahan dan bimbingan. Terima kasih juga penulis ucapkan kepada teman-teman “Genetic Team”, Keluarga Besar ITK 48, dan Riandi Ernanda atas dukungannya selama ini. Ungkapan terima kasih juga disampaikan kepada Ayah Ossi Tanushy, Ibu Asih Kurniasih, Adik Hoggy Hartawan, dan serta seluruh keluarga atas do’a dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
METODE 2
Bahan 2
Alat 3
Ekstraksi 3
Amplifikasi DNA (PCR) 3
Elektroforesis Hasil PCR 4
Sekuensing DNA 4
Prosedur Analisis Data 4
Rekontruksi Filogenetik 4
Analisis Pola Distribusi Berdasarkan Struktur Genetika Populasi 5
HASIL DAN PEMBAHASAN 5
Rekontruksi Filogenetik 5
Pola Distribusi Berdasarkan Struktur Genetik Lepidochelys olivacea 6
SIMPULAN DAN SARAN 8
Simpulan 8
Saran 9
DAFTAR PUSTAKA 9
LAMPIRAN 11
DAFTAR TABEL
1 Keragaman genetik dan keragaman nukleotida L. olivacea 6
2 Matriks Pasangan Populasi (Fst-s) 6
3 Analisis AMOVA antar populasi penyu lekang (Lepidochelys olivacea) 7
DAFTAR GAMBAR
1 Lokasi pengambilan sampel penyu lekang (L. olivacea). 2
2 Pohon filogenetik 5
3 Pola migrasi penyu lekang di perairan Indonesia 8
DAFTAR LAMPIRAN
1 Hasil edit sekuen penyu lekang (Lepidochelys olivacea) menggunakan
perangkat lunak MEGA 6. 12
2 Hasil analisis IBD (Isolation by Distance) 19
3 AMOVA 19
PENDAHULUAN
Latar Belakang
Penyu merupakan biota yang hidup di laut lepas dan bermigrasi hingga ribuan kilometer. Penyu lekang (Lepidochelys olivacea) banyak ditemukan di Samudera Atlantik, Samudera Pasifik, dan Samudera Hindia (Whiting et.al.
2007). Penyu lekang merupakan salah satu jenis dari enam jenis penyu yang ada di perairan Indonesia. Keberadaan populasi L. olivacea di Indonesia mengalami perubahan habitat karena kegiatan antropogenik dan pemanasan global (Maulany
et.al. 2012). Kegiatan antropogenik yang mengancam populasi penyu lekang (Lepidochelys olivacea), yaitu pemanfaatan penyu lekang yaitu konsumsi penyu dan perdagangan penyu serta adanya reklamasi habitat penyu yaitu alih fungsi pantai sebagai tempat pariwisata. Akibat penurunan populasi L. olivacea tersebut telah masuk pada daftar merah IUCN (International Union for Conservation of Nature) katagori Vulnerable (IUCN 2014).
Penyu memiliki dua famili yaitu, Cheloniidae dan Dermochelyidae. Penyu lekang (Lepidochelys olivacea) termasuk kedalam famili Cheloniidae. Salah satu ciri dari L. olivacea yakni memiliki ukuran tubuh yang relative lebih kecil dibandingkan dengan kelompok penyu lainnya. Karapas L. olivacea memiliki warna abu – abu, sedangkan pada bagian bawah tubuh (plastron) L. olivacea
memiliki warna krem keabuan. Bobot Lepidochelys olivacea berkisar antara 50 kg hingga 75 kg dengan panjang tubuh 150 sentimeter. Sementara bobot tukik
Lepidochelys olivacea 16 gram hingga 19 gram dengan panjang rata-rata karapas tukik hingga 44 mm. Penyu lekang bermigrasi disepanjang perairan dengan kedalaman menyelam hingga 150 meter (Polovina et.al. 2004). Arribadda merupakan sifat unik penyu lekang yang secara serentak mendarat untuk bertelur (Rodriguez-Zarate et.al. 2013). Jarak migrasi penyu dapat mencapai ribuan kilometer dari habitat pakan hingga habitat peneluran (Akira et.al. 2012). Diduga
L. olivacea memiliki pola migrasi yang sempit untuk berkembangbiak (Jensen
et.al. 2013). Pantai pasir dengan kemiringan landai merupakan tempat bertelur penyu yang dapat ditemukan di pantai-pantai di Indonesia (Purnama et.al. 2013).
2
Tujuan Penelitian
Penelitian ini bertujuan mengetahui pola distribusi penyu lekang (Lepidochelys olivacea) berdasarkan struktur genetik menggunakan penanda DNA Mitokondria (mtDNA) sitokrom oksidase 1 (CO1).
METODE
Waktu dan Tempat
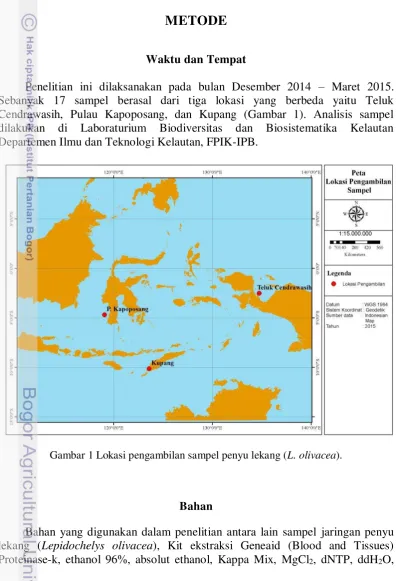
Penelitian ini dilaksanakan pada bulan Desember 2014 – Maret 2015. Sebanyak 17 sampel berasal dari tiga lokasi yang berbeda yaitu Teluk Cendrawasih, Pulau Kapoposang, dan Kupang (Gambar 1). Analisis sampel dilakukan di Laboraturium Biodiversitas dan Biosistematika Kelautan Departemen Ilmu dan Teknologi Kelautan, FPIK-IPB.
Gambar 1 Lokasi pengambilan sampel penyu lekang (L. olivacea).
Bahan
3 PCR buffer, forward primer LTEi9 (GGG AAT AAT CAA AAG AGA AGG), reverse primer H950 (GTC TCG GAT TTA GGG GTT TG), amplitaq, agarose, TAE buffer, EtBr, loading dye, dan low mass ladder.
Alat
Alat yang digunakan dalam penelitian ini antara lain alat tulis, bunsen, cawan petri, pisau ekstraksi, pinset, tray, sarung tagan karet, vortex, heating block, alat spin kecil, alat sentrifuge, microtube, micropippet, pippet tips, spin filter, Collection Tube, thermo cycler, kalkulator, gelas ukur, labu erlenmeyer,
microwave, alat elektroforesis, dan UV transluminator.
Ekstraksi
Ekstraksi DNA bertujuan untuk menghancurkan sel dan mengambil jaringan pada sampel, membuat ekstraksi secara murni serta melindungi DNA dari degradasi. Kegiatan ekstraksi pada penelitian ini menggunakan kit Geneaid. Pada ekstraksi ini, hal yang pertama dilakukan adalah mencacah sampel penyu lekang (Lepidochelys olivacea) dengan menggunakan alat ekstraksi. Setelah didapat hasil gerusan berupa cacahan halus dari sampel penyu lekang tersebut, dimasukkan kedalam tube berukuran 1.5 ml dan GST Buffer sebanyak 200 μl dan memasukkan Proteinase K sebanyak 20 μl dan dilanjutkan dengan kegiatan
heating pada suhu 60 oC selama 2 hingga 4 hari hingga sampel lisis. Centifuge selama 2 menit pada 14-16,000xg, transfer supernatan dan tempatkan pada tube 1.5 ml yang baru. Tambahkan 200 μl GSB Buffer dan vortex selamam 10 detik. Tambahkan absolut ethanol sebanyak 200 μl, kocok selama 10 detik. Selanjutnya tempatkan GD Colum dalam 2 ml Collection Tube, dan transfer semua campuan ke dalam GD Colum kemudian centrifuge selama 1 menit pada 14-16,000xg. Buang 2 ml Collection Tube yang mengandung campuran tersebut. Tempatkan GD Colum ke 2 ml Collection Tube yang baru. Tambahkan 400 μl W1 Buffer ke GD Colum. Centrifuge selama 30 detik pada 14-16,000xg. Buang campurannya dan tempatkan kembali pada 2 ml Collection Tube tersebut. Tambah Wash Buffer sebanyak 600 μl ke GD Colum. Centrifuge selama 30 detik pada 14-16,000xg. Buang campurannya dan tempatkan kembali pada 2 ml Collection Tube tersebut. Centrifuge selama 3 menit pada 14-16,000xg untuk mengringkan. Pindahkan GD Colum kering ke tube berukuran 1.5 ml. Tambah secara berurutan Elution Buffer dan ddH2O sebanyak 100 μl, diamkan selama 3 menit dan centrifuge selama 30
detik pada 14-16,000xg untuk mengelusi DNA.
Amplifikasi DNA (PCR)
4
CAA AAG AGA AGG), rev_primer H950 (GTC TCG GAT TTA GGG GTT TG) merupakan primer yang cocok untuk analisis DNA mitokondria penyu laut (Abreu-Grobois et.al. 2006). Komponen utama dalam PCR adalah DNA template, Kapa Mix, dNTPs, buffer PCR, MgCl2, primer LTEi9 (GGG AAT AAT CAA AAG AGA AGG) (forward), primer H950 (GTC TCG GAT TTA GGG GTT TG) (reverse), dan amplitaq. PCR dilakukan dengan menggunakan mesin PCR (thermo cycler) dan diatur siklusnya sesuai untuk penyu laut yaitu 38 siklus. Setiap siklusnya terdiri dari denaturation pada suhu 94°C selama 30 detik, annealling
pada suhu 50°C selama 30 detik, dan extention pada suhu 72°C selama 45 detik. Kemudian dilanjutkan dengan final elongated pada suhu 72°C selama 5 detik.
Elektroforesis Hasil PCR
Elektroforesis merupakan metode pemisahan senyawa kimia yang didasarkan pada laju pergerakan molekul dalam aliran listrik (Madduppa 2014). Elektroforesis ini bertujuan untuk mengetahui ada tidaknya DNA dalam produk PCR kita. Ukuran dan integritas DNA ditentukan berdasarkan elektroforesis gel agarose 1%. Komposisi agar dalam tahap elektroforesis terdiri atas agarose 0,5 gram, TAE Buffer sebanyak 50 ml, dan EtBr sebanyak 4 μL. Pada tahap elektroforesis produk hasil PCR dicampur dengan loading dye sebanyak 1 μL. Sebanyak 4 μL Low mass ladder dimasukan kedalam sumur pertama dan selanjutanya diikuti dengan campuran produk hasil PCR. Elektroforesis menggunakan tegangan sebesar 220 V dan arus 400 mA dengan lama elektroforesis 25 menit. Hasil elektroforesis dilihat menggunakan UV transluminator.
Sekuensing DNA
Sekuensing DNA merupakan pengurutan basa nukleotida terdapat dalam suatu sampel DNA. Produk PCR dikirimkan ke sequencing facilty UC Berkeley, Dept. of Moleculler and Cell Biology Sequencing Facility, USA dengan menggunakan mesin sequencer ABI 1377.
Prosedur Analisis Data
Rekontruksi Filogenetik
Rekontruksi filogenetik dilakukan dengan menggunakan software MEGA 6.0 (Molecular Evolutionary Genetic Analysis). Pohon filogenik dibuat dengan cara mengedit data hasil sekuensing, menganalisis statistik pensejajaran DNA dengan menggunakan tools alignment CrustalW, dan menggunakan metode
5 pembanding dari luar spesies yang diteliti. Dalam penelitian digunakan Tridacna corecea sebagai outgroup.
Analisis Pola Distribusi Berdasarkan Struktur Genetika Populasi
Analisis untuk struktur genetika populasi dilakukan dengan menggunakan perangkat lunak Arlequin 3.5. Struktur genetik untuk data DNA Mitokondria dapat dilihat dengan menggunakan nilai keragaman haplotipe (h) dan keragaman nukleotida (π). Analisis hubungan antara jarak genetic dan jarak geografi menggunakan metode perbandingan Wright F-statistic (Fst) dan Isolation by Distance (IBD). Uji pola distribusi berdasarkan struktur genetik menggunakan analisis statistik yaitu AMOVA (Analysis Mulecular Variance) yang juga menggunakan perangkat lunak yang sama.
Isolation by Distance (IBD) dilakukan pada Isloation by Distance Web Service (http://ibdws.sdsu.edu/) pada menu Genetic Distance / Similarties dengan memasukan format Geographic Genetic Matrix yang telah dibuat pada perangkat lunak Microsoft Excel sebagai input data jarak genetik dan jarak geografi. Jarak geografi ditentukan dengan menggunakan perangkat lunak Google Earth, ditentukan degan nilai jarak geografi terdekat antara populasi tersebut.
HASIL DAN PEMBAHASAN
Rekontruksi Filogenetik
Hasil sequence DNA berupa urutan nukleotida dengan nilai rata-rata 500 bp hingga 800 bp. Tingkat homologi yang didapat dari BLAST sebesar 99%.
6
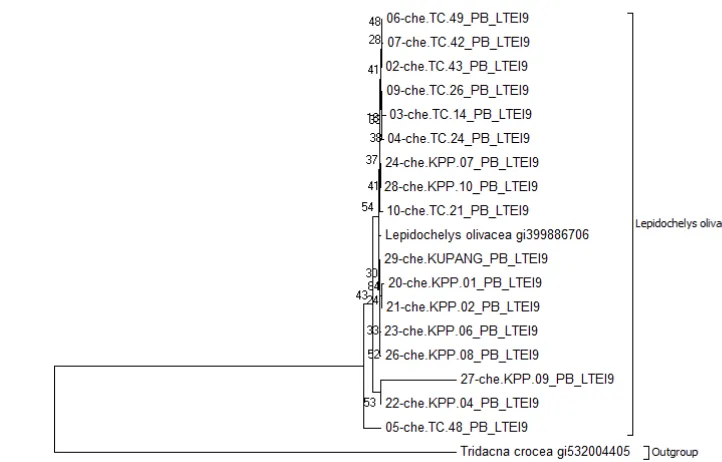
Gambar 2 merupakan rekontruksi filogenetik yang membentuk dua clade
besar, antara Lepidochelys olivacea dengan outgrup dari spesies Tridacna crocea.
Pada clade Lepidochelys olivacea, terbentuk cabang kecil yang membuktikan bahwa adanya perbedaan interspesies. Perbedaan interspesies dipengaruhi oleh perbedaan komposisi basa pada urutan nukleotida antar individu namun tidak mempengaruhi kesamaan spesies antar populasi. Perbedaan interspesies dapat dipengaruhi oleh marka gen CO1 yang cukup handal untuk mendiskriminasi antarspesies (Susanto et.al. 2011). Pada Gambar 2, perbedaan antarspesies yang kecil menunjukan asumsi bahwa spesies Lepidochelys olivacea masih tergolong masih dalam 1 spesies yang sama. Nilai keragaman genetik tiap populasi menunjukan ukuran suatu populasi (Nuryanto 2009). Nilai keragaman genetik pada masing-masing populasi adalah 1,00 ± 0,06, 1,00 ± 0,06, dan 1,00 ± 0,00. Nilai keragaman genetik tiap populasi termasuk kategori tinggi yakni 0,8 – 0,1 (Nei 1987) Nilai keragaman nukleotida untuk populasi Teluk Cendrawsih 0.20 ± 0,11, Kapoposang 0.40 ± 0,22, dan Kupang 0.00 ± 0,00 (Tabel 1). Nilai keragaman nukleotida pada populasi Kupang 0.00 ± 0,00 karena jumlah individu di Kupang hanya terdiri dari satu individu.
Tabel 1 Keragaman genetik dan keragaman nukleotida L. olivacea
No Sample Site n Nhp h π
1 Teluk Cendrawasih 8 8 1.00 +/- 0.06 0.20 +/- 0.11 2 Pulau Kapoposang 8 8 1.00 +/- 0.06 0.40 +/- 0.22 3 Kupang 1 1 1.00 +/- 0.00 0.00 +/- 0.00
Keterangan: jumlah sekuen (n), nomor genetik (Nhp), keragaman genetik (h), keragaman nukleotida (π)
Pola Distribusi Berdasarkan Struktur Genetik Lepidochelys olivacea
Pola ditribusi diverifikasi menggunakan analisis Fixation Index (Fst),
Isolation by Distance (IBD), dan AMOVA. Fixation Index (Fst) digunakan untuk menentukan nilai perbedaan dan menentukan struktur genetika populasi (Kusuma 2014). Nilai fst antar populasi sebesar 0.462, maka perbedaan antar populasi hampir mendekati tidak ada atau semua sampel homogen. Hal ini juga diperkuat oleh matriks fst (Tabel 2), bahwa nilai minus (-) pada matriks juga menunjukkan signifikan tidak ada yang berbeda.
Tabel 2 Matriks Pasangan Populasi (Fst-s)
Keterangan: S (Significance), NS (Not Significance)
7 (Lepidochelys olivacea) dari tiap populasi tidak memiliki korelasi antara jarak genetik dan jarak geografis serta tidak memiliki struktur populasi yang diteliti.
Analysis Molecular Variance (AMOVA) didasarkan pada perbedaan tingkat mutasi antara pasangan haplotipe yang berbeda dan hasil analog dari variasi komponen dan nilai Fst serta dapat mencerminkan keragaman antar populasi yang diteliti (Roewer et.al. 1996).
Tabel 3 Analisis AMOVA antar populasi penyu lekang (Lepidochelys olivacea) Sumber Variasi d.f. Persentase variasi (%) Fstap α
Antar populasi 2 -2.34 -0.023 0.05
Dalam populasi 14 102.34
Total 16
Keterangan: Significance level (α)
Analisis AMOVA menunjukan nilai persentase variasi antar populasi sebesar -2.34 % (Tabel 3) yang menunjukan bahwa keragaman antar populasi yang sangat kecil atau tidak ada perbedaan tiap populasi yang diteliti. Nilai persentase variasi dalam tiap populasi sebesar 102% (Tabel 3) menunjukan bahwa keragaman sangat tinggi terjadi di dalam tiap populasi.
8
Gambar 3 Pola migrasi penyu lekang di perairan Indonesia
Aliran gen dipengaruhi oleh perilaku migrasi dan individual fertile antar populasi (Henuhili 2008). Pola migrasi penyu lekang (Lepidochelys olivacea) di Indonesia (Gambar 3) menyatakan bahwa penyu lekang bergerak dari wilayah peneluran atau berkembangbiak bergerak dari atas wilayah Papua Barat kemudian melewati Laut Arafura, Laut Banda, Selat Ombai, Selat Makasar, Laut Flores, Laut Sawu selanjutnya bergerak menuju Samudera Hindia (Ardiansyah 2004). Analisis sebelumnya menyatakan bahwa L. olivacea melakukan migrasi setelah berkembangbiak dan menyebar untuk mencari makan (Limpus 2008). Pola distribusi penyu lekang (Lepidochelys olivacea) secara global diduga banyak terdapat di Asia Tenggara hingga dan Pasifik Barat (Limpus 2008) (Lampiran 4).
SIMPULAN DAN SARAN
Simpulan
Populasi Teluk Cendrawasih, Pulau Kapoposang, dan Kupang memiliki keragaman genetik (haplotype) yang tinggi. Nilai keragaman nukleotida yang tinggi hanya terdapat pada populasi Teluk Cendrawasih dan Pulau Kapoposang. Pola distribusi berdasarkan struktur genetik menggunakan analisis Fixation Index
9 lekang (Lepidochelys olivacea) bergerak dari Samudera Pasifik melalui perairan Indonesia menuju Samudera Hindia yang menyebabkan distribusi global penyu lekang banyak terdapat di Pasifik Barat hingga Asia Tenggara.
Saran
Perlu dilakukan penelitian lebih lanjut menggunakan sampel yang berasal dari seluruh populasi penyu di Indonesia agar dapat memberikan informasi mengenai pola distribusi struktur penyu lekang (Lepidochelys olivacea) berdasarkan struktur genetik di Indonesia.
DAFTAR PUSTAKA
Akira R, I Nengah W, IB Windia A. 2012. Komposisi genetik penyu hijau (Chelonia mydas) hasil tangkapan liar dari Nusa Tenggara Barat (Bima dan Teluk Cempi). J Med Vet Indones. 1(1):22-36.
Ardiansyah F. 2004. Marine Turtle in Indonesia. Indonesian Sea Turtle Conservation [Internet]. [diunduh 2015 Mei 6]. Tersedia pada: https:// assets.panda.org/downloads/brochureturtlecop7indonesiacbd.pdf
Bohonak, AJ. 2001. IBD (Isolation by Distance): A Program for Analyses of Isolation by Distance. San Diego (CA): San Diego State University.
Handoyo D, Rudiretna A. 2001. Prinsip umum dan pelaksanaan polymerase chain reaction (PCR). Unitas. 9(1): 17-29.
Lepidochelys olivacea management units in Australia and assessing the potential impact of mortality in ghost nets. Endang Species Res. 21: 241-253 Kumar S, Nei M, Dudley J, Tamura K. 2008. MEGA: a biologistcentric software for evolutionary analysis of DNA and protein sequences. Brief Bioinform. 9: 299–306.
Kusuma AB. 2014. Konenktifitas dan Keragaman Genetik pada Karang Lunak serta Implikasinya Terhadap Kawasan Konservasi Laut Sarcophyton trocheliophorum [tesis]. Bogor (ID): Institut Pertanian Bogor.
Limpus CJ. 2008. A Review of Australian Marine Turtle: 4. Olive Ridley Turtle (Lepidochelys olivacea). Brisbane: Queensland Environment Protection Agency.
Madduppa H. 2014. Bioekologi dan Biosistematika Ikan Terumbu. Bogor (ID): IPB Press.
10
Purwo National Park, East Java, Indonesia: implications for hatchery management. Mar Biol. 159: 2651–266.
Nei M. 1987. Moleculer evolutionary genetics. Columbia University. Press. New York. 512 hal.
Nuryanto A & Kochzius M. 2009. Highly restricted gene flow and deep evolutionary linieages in the giant clam Tridacna maxima. Coral Reefs, 28: 607-619.
Polovina JJ, Balazs GH, Howell EA, Parker DM, Seki PS, Dutton PH. 2004. Forage and migration habitat of loggerhead (Caretta caretta) and olive ridley (Lepidochelys olivacea) sea turtles in the central North Pacific Ocean.
Fisheries Oceanography.13(1): 36-51.
Purnama D, Zamani NP, Farajallah A. 2013. Microsatellite DNA analysis on the polyandry ofgreen sea turtle Chelonia mydas. Hayati J Biosci. 20(4): 182-186.
Rodhiguez-Zarate CJ, Rocha-Olivares A, Beheregaray LB. 2013. Genetic signature of a recent metapopulation bottleneck in the olive ridley turtle (Lepidochelys olivacea) after intensive commercial exploitation in Mexico.
Biological Conservation. 168: 10–18.
Roewer L, Kayser M, Dieltjies P, Nagy M, Bakker E, Krawczak, de Knijft P. 1996. Analysis of molecular variance (AMOVA) of Y-chromosome-specific microsatellites in two closely related human populations. Human Molecular Genetic. 5(7): 1029-1033.
Supartha DAPAS, Wandia IN, Adnyana IBW. 2013. Struktur genetik penyu hijau Di Kepulauan Derawan, Kalimantan Timur, dengan marker molekul D-Loop Dna mitokondria. J Med Vet Indones. 2(3): 347 – 359.
Susanto AH, Nuryanto A, Soedibja PHT. 2011. Phylogeography and genetic diversity of humpback grouper Cromileptes altivelis based on cytochrome c oxidase 1. Jurnal Natur Indonesia. 14(1): 47-51.
11
12
Lampiran 1 Hasil edit sekuen penyu lekang (Lepidochelys olivacea) menggunakan perangkat lunak MEGA 6.
19
Lampiran 2 Hasil analisis IBD (Isolation by Distance)
Analysis 1: MANTEL TEST for matrix correlation between genetic distance and
geographic distance [user-entered genetic distances].
Correlation of genetics and geographic distance: Z = 5.1260, r = 0.9154
Null hypothesis that r>=0 (typical Ho if genetic similarity such as M is used): one-sided p = 1.0000. 842 randomizations below, 158 tied, 0 above.
Null hypothesis that r<=0 (typical Ho if genetic distance such as F-st is used): one sided p = 0.1580
Lampiran 3 AMOVA
AMOVA design and results : ---
20
Va and FST : P(rand. value > obs. value) = 0.46139 P(rand. value = obs. value) = 0.00098
P-value = 0.46237+-0.01419
21
RIWAYAT HIDUP
Penulis dilahirkan di Bekasi pada tanggal 25 Januari 1993 dari Ayah Ossi Tanushy dan Ibu Asih Kurniasih. Penulis adalah putri pertama dari 2 bersaudara. Tahun 2011 penulis lulus dari SMA PGRI 1 Bekasi dan pada tahun yang sama penulis lulus seleksi masuk Institut Pertanian Bogor (IPB) melalui SNMPTN jalur undangan dan diterima di Departemen Ilmu dan Teknologi Kelautan, Fakultas Perikanan dan Ilmu Kelautan. Penulis memperoleh beasiswa BBM pada tahun 2013 dan tahun 2015, serta beasiswa BUMN pada tahun 2013. Penulis aktif dalam organisasi Himpunan Mahasiswa Ilmu dan Teknologi Kelautan (HIMITEKA-IPB) sebagai sekretaris divisi Pemanfaatan Sumber Daya Manusia (PSDM) periode 2013-2014.