KOMUNITAS ARKAEA METANOGEN DAN EMISI GAS
METANA (CH
4) DI LAHAN SAWAH YANG DIBERI
PUPUK HAYATI BAKTERI METANOTROF
ASRIANTO
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul komunitas arkaea metanogen dan emisi gas metana (CH4) di lahan sawah yang diberi pupuk hayati
bakteri metanotrof adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Januari 2015 Asrianto
RINGKASAN
ASRIANTO. Komunitas arkaea metanogen dan emisi gas metana (CH4) di lahan
sawah yang diberi pupuk hayati bakteri metanotrof. Dibimbing oleh IMAN RUSMANA dan NISA RACHMANIA MUBARIK.
Gas metana menjadi salah satu gas rumah kaca (GRK) di lahan sawah yang berkontribusi dalam pemanasan global. Metana memiliki daya absorbsi yang jauh lebih besar dari karbon dioksida. Lahan sawah merupakan lingkungan khas bagi dua komunitas prokariotik yaitu arkaea metanogen di daerah yang anerob dan bakteri metanotrof di daerah aerob. Arkaea metanogen merupakan mikroorganisme yang memiliki kemampuan metanogenesis. Metabolisme arkaea metanogen dapat merubah karbon dioksida, asam format, asetat, metanol, metilamin dan karbon monoksida menjadi metana sedangkan bakteri metanotrof dapat menggunakan metana sebagai sumber karbon dan energi. Analisis kelimpahan arkaea metanogen di lahan sawah menjadi penting guna menghubungkan dengan emisi metana yang dilepaskan. Metode kultur tidak efektif untuk menginvestigasi mikroorganisme sehingga diperlukan pendekatan studi metagenomik. Salah satu studi metagenomik yang digunakan adalah Polymerase Chain Reaction Denaturing gradient Gel Electrophoresis (PCR-DGGE). PCR-DGGE dapat memperlihatkan struktur dan suksesi komunitas di suatu lingkungan.
Penelitian diawali dengan pengambilan tanah sawah yang telah diberi perlakuan yaitu metode celup dan sebar (200 kg NPK/ha) serta kontrol (300 kg NPK/ha). Sebelum penanaman metode celup didahului dengan perendaman benih dalam kultur bakteri metanotrof selama ± 15 menit. Pengukuran gas dilakukan untuk mengestimasi fluks metana di lahan petak penelitian. Tanah yang sudah diambil kemudian dilakukan ekstraksi DNA menggunakan PowerSoil®DNA Isolation Kit. Hasil ekstraksi diamplifikasi dengan primer gen 16S rRNA 0357F-GC Clamp dan 0915aR. Produk PCR di migrasi pada gel poliakrilamida 6% dengan gradien 40-56%. Pita yang muncul pada gel poliakrilamid dipotong dan diamplifikasi ulang dengan primer tanpa GC - clamp. Produk PCR dikirim untuk disekuensing.
Hasil Penelitian menunjukkan bahwa fluks emisi metana pada metode celup bernilai negatif yang berarti ada serapan metana oleh aktivitas bakteri metanotrof. Pengunaan bakteri metanotrof efektif mereduksi metana di lahan sawah. Keragaman arkaea metanogen menggunakan primer 0357F dan 0915aR cenderung tidak memperlihatkan variasi pita yang banyak. Profil DGGE hanya menampakkan dua pita setiap petak perlakuan dan waktu pengamatan. Pita hasil DGGE MIR 1 memiliki tingkat kemiripan 82% dengan Ignisphaera aggregans galur AQI.S1 sedangkan MIR 2 82% dengan Methanosarcina barkeri galur MS.
SUMMARY
ASRIANTO. Methanogenic archaea community and methane emissions in rice fields applied by methanotrophic biofertilizer. Supervised by IMAN RUSMANA and NISA RACHMANIA MUBARIK.
Methane is one of the green house gases emited from rice fields that contribute to global warming. In fact, methane has greater global warming potency than that of carbon dioxide. Rice fields are a typical environment for two procaryote communities related to methane methabolism i.e. methanogenic archaea in anaerobic area and methanotrophic bacteria in aerobic area. Methanogenic archaea have ability to perform methanogenesis. Methanogenesis metabolic can transform carbon dioxide, formic acid, acetate, methanol, methylamine and carbon monoxide into methane whereas methanotrophic bacteria can use methane as carbon dan energy sources. Analysis the abundance of methanogenics archaea in rice fields is important related to methane emissions. Culture method can not investigated all microb in the environment due to unculture microbes so it is necessary to use metagenomic study to figure out microbial diversity in the enviroment. One method for metagenomic study is Polimerase Chain reaction Denaturing gradient Gel Electrophoresis (PCR-DGGE). PCR-DGGE can determine structure and succession of microbial community in the environment.
The study was began by sampling soil samples that had been treated i.e soaking and spreading method (200 kg NPK/ha), control (300 kg NPK/ha). Soak ing method was preceded by a process soaking the seeds in bacterial cultured of methanotrophs for 15 minutes. Gas measurements was conducted to estimate the methane flux in rice fields. Soil samples were extracted of their DNA using PowerSoil® DNA Isolation Kit. Amplification the 16S rRNA gene using primers 0357F-GC and 0915aR. PCR products were migrated on 6% polyacrylamide gel with a gradient denaturant of 40 – 56%. DNA bands appeared on polyacrylamide gel were cut using scalpel and re-amplified using the primers without GC - clamp. PCR products were then sequenced.
The results showed that fluxes of methane emissions were negative, it means that methane was utilized by methanotrophic activities. Methanotrophic bacteria reduced methane emission efectively in rice fields. The diversity of methanogenic archaea determined using primers 0357F-GC and 0915aR did not show many bands. DGGE profiles revealed two bands for each plot treatment and observation time. MIR 1 band had 82% similarity to Ignisphaera aggregans strain AQI.S1 16S while MIR 2 band was 82% similar with Methanosarcina barkeri strain MS.
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Mikrobiologi
KOMUNITAS ARKAEA METANOGEN DAN EMISI GAS
METANA (CH
4) DI LAHAN SAWAH YANG DIBERI PUPUK
HAYATI BAKTERI METANOTROF
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
Mikrobiologi
Judul Tesis : Komunitas arkaea metanogen dan emisi gas metana (CH4) di lahan
sawah yang diberi pupuk hayati bakteri metanotrof Nama : Asrianto
NIM : G351120071
Disetujui oleh Komisi Pembimbing
Dr Ir Iman Rusmana, MSi Ketua
Dr Nisa Rachmania M, MSi Anggota
Diketahui oleh
Ketua Program Studi
Prof Dr Anja Meryandini, MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Segala puji penulis panjatkan kepada Allah atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul yang dipilih dalam penelitian ini ialah komunitas arkaea metanogen dan emisi gas metana (CH4) di
lahan sawah yang diberi pupuk hayati bakteri metanotrof. Penelitian ini didanai dari DIKTI melalui Hibah Kompetensi tahun 2014 kepada ketua pembimbing.
Terima kasih penulis ucapkan kepada Bapak Dr Ir Iman Rusmana MSi dan Ibu Dr Nisa Rachmania Mubarik MSi selaku pembimbing, serta Dr Ir Aris Tjahjoleksono, DEA selaku penguji yang telah banyak memberi saran. Penulis sampaikan terimah kasih kepada seluruh dosen program studi mikrobiologi atas dedikasi, inspirasi dan ilmunya. Ungkapan terima kasih juga penulis sampaikan kepada orangtua tercinta (La Udi Lopa dan Wa Heto) dan Istriku tersayang Rahmatia Sahila, S.Pd serta seluruh keluarga (Alimanisa, Herdianti, Alwi Misran, Citra Lestari, Muhammad Sahidin S.KM, Rahmaniar, A.Md, Rahmawati, S.Pi, Hartini, S.Pd) atas segala doa dan dukungannya.
Terima kasih juga penulis ucapkan kepada rekan–rekan seperjuangan: Fachruddin, Abu Ayyub Dustan, Garuda, Ikbal, Mustamin, La Ode Huli dan Jumadin, rekan rekan Mikrobiologi 2012 terkhusus Hari Kapli, Muh. Habibullah, Arif Rahman Jabal, Randi, Mahyarudin, Nur Antriana, Wulan Fitri Safari, Nezharia Nurza Harca, Anja Asmarani, Vita Aggun Cahyani, Dina, Mona Primanita), Laboran Lab. Bakteriologi (Pa’ Jaka), rekan-rekan yang tergabung dalam IR CREW (Iman Rusmana Crew) dan rekan-rekan Forum Wacana Sultra atas dukungan, saran dan bantuan sehingga saya dapat menyelesaikan karya ilmiah ini.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
TINJAUAN PUSTAKA 2
Emisi Metana di Lahan Sawah 2
Komunitas Metanogen dan Metanotrof 2
Metagenomik 6
METODE 7
Alat dan Bahan 7
Prosedur Penelitian 8
Sampling Tanah dan Pengukuran Gas Metana 8
Ekstraksi Total DNA 8
Amplifikasi Fragmen gen 16S rRNA 8
Komposisi Denaturing Gradient Gel Electrophoresis (DGGE) 9 Determinasi dan Pembuatan Pohon Filogeni 9
HASIL DAN PEMBAHASAN 9
Hasil 9
Pembahasan 13
SIMPULAN DAN SARAN 17
Simpulan 17
Saran 17
DAFTAR PUSTAKA 17
DAFTAR TABEL
1 Perbedaan arkaea metanogen dan bakteri metanotrof 4 2 Karakteristik beberapa arkaea metanogen 5
3 Runutan sekuen primer gen 16S rRNA 8
4 Konsentrasi dan purifikasi DNA 10
5 BLAST sekuen hasil PCR 12
DAFTAR GAMBAR
1 Skema produksi, oksidasi dan emisi metana di lahan sawah 3 2 Fluks metana tanaman padi pada 26 HST, 47 HST, dan 96 HST 10 3 Hasil amplifikasi menggunakan gen 16S rRNA arkaea metanogen 11
4 Hasil profil DGGE gen 16S rRNA 11
5 Hasil amplifikasi ulang pita hasil DGGE 12
6 Pohon filogeni arkaea metanogen di lahan sawah Sukabumi 13
DAFTAR LAMPIRAN
1 Media tumbuh bakteri metanotrof 23
2 Hasil uji tanah 24
3 Sistem klasifikasi jenis tanah 25
4 Runutan basa BLAST-N pita MIR 1 26
PENDAHULUAN
Latar Belakang
Lahan Sawah merupakan kegiatan antropogenik di sektor pertanian yang menjadi salah satu sumber terjadinya pemanasan global yang mengemisikan metana (CH4), dan dinitrooksida (N2O). Lahan sawah melepaskan emisi metana
ke atmosfer kira–kira sebesar 15% dari total emisi global (IPCC 1994). IPCC (2007) melaporkan kontribusi metana terhadap pemanasan global menempati urutan kedua setelah karbondioksida. Kemampuan metana untuk meningkatkan suhu bumi sangat tinggi, hal ini karena kapasitas absorbsi sinar infra merah per molekul 25 kali lebih besar dibandingkan karbondioksida (Lelieveld et al. 1993; Hanson dan Hanson 1996).
Tanah sawah merupakan salah satu daerah khas, lingkungan aerobik dihuni oleh komunitas bakteri metanotrof dan lingkungan anerobik, umumnya didominasi oleh komunitas arkaea yang memiliki kemampuan metanogenesis (Adachi 2011). Proses perubahan metana terdapat dua mekanisme alamiah yaitu, pertama, reaksi metana dengan radikal OH di lapisan troposfera. Kedua, oksidasi oleh bakteri metanotrof di daerah rhizosfer. Sehubungan dengan proses oksidasi metana, Conrad dan Rothfus (1991) melaporkan oksidasi metana oleh bakteri metanotrof di lahan sawah dapat mencapai 80%. Hal ini menjadi sebuah pendekatan baru yang strategis sebagai upaya mitigasi emisi metana di lahan sawah dengan memanfaatkan bakteri metanotrof yang secara alami mampu menggunakan metana sebagai sumber karbon juga mampu melakukan fiksasi nitrogen karena memiliki gen nifH dan nifD (Dedysh et al . 2004). Pingak (2013) melaporkan bahwa aplikasi pupuk hayati yang terdiri atas konsorsium bakteri metanotrof, efektif menurunkan emisi metana sekaligus meningkatkan produksi gabah.
2
emisi metana melalui pemberian konsorsium bakteri metanotof keragaman arkaea metanogen melalui pendekatan PCR-DGGE.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengkaji efektivitas kombinasi bakteri metanotrof, sebagai agen pereduksi metana (CH4) dan mengamati keragaman
komunitas arkaea metanogen melalui analisis PCR-DGGE.
Manfaat Penelitian
Penelitian ini diharapkan mendapatkan informasi mengenai efektifitas reduksi gas metana (CH4) dan keragaman komunitas arkaea metanogen di lahan
sawah, Desa Cidahu, Kecamatan Cicurug, Sukabumi.
Ruang Lingkup Penelitian
Penelitian ini meliputi pengambilan sampel tanah sawah, pengukuran emisi metana, ekstraksi DNA asal tanah sawah, amplifikasi DNA, analisis DGGE, sekuensing, dan pembuatan pohon filogeni untuk melihat kekerabatan.
TINJAUAN PUSTAKA
Emisi Metana di Lahan Sawah
Fenomena peningkatan temperatur global dari tahun ke tahun terjadi kerena efek rumah kaca yang disebabkan oleh meningkatnya emisi gas karbondioksida (CO2), metana (CH4), dinitrooksida (N2O) dan kloroflouro karbon
(CFC) sehingga energi matahari terperangkap dalam atmosfer bumi.
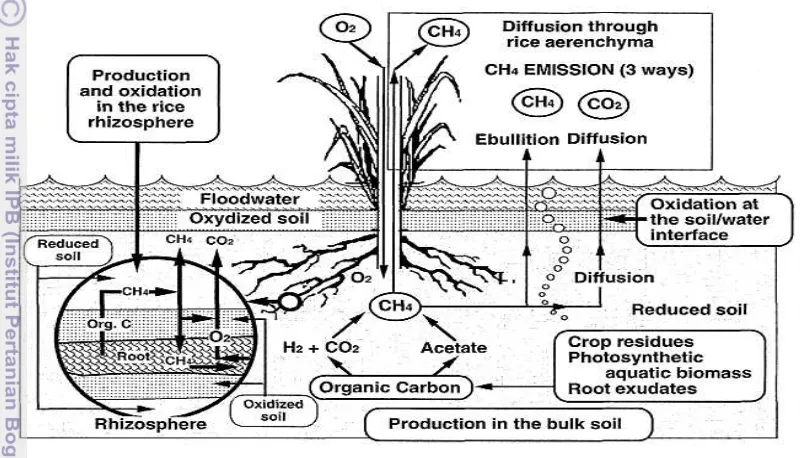
3 (Couwenberg 2009 di dalam Muna 2011) keberadaan aerenkim digunakan untuk mendistribusikan oksigen ke zona akar sebagai bentuk adaptasi sistem perakaran di tanah yang lembab. Keberadaan oksigen di zona perakaran memungkinkan terjadinya oksidasi metana di zona tersebut. Pada saat yang sama, metana yang ada di zona perakaran ditrasportasikan menuju atmosfer dengan melewati zona aerob melalui jaringan aerenkim. Setyanto (2006) tanaman padi berperan aktif sebagai media pengangkut metana dari lahan sawah ke atmosfer lebih dari 90% diemisikan melalui jaringan aerenkim.
Gambar 1 Skema produksi, oksidasi dan emisi metan di lahan sawah (Mer dan Roger 2001)
Komunitas Metanogen dan Metanotrof
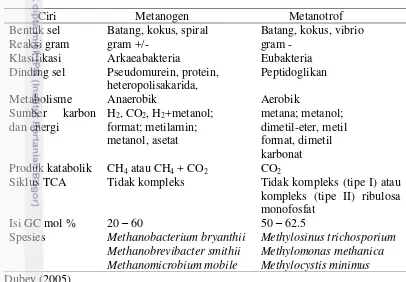
Dilingkungan akuatik seperti tanah sawah, emisi metana ditentukan oleh dua proses mikroorganisme yang berbeda, yaitu produksi metana oleh arkaea metanogen dan konsumsi metana oleh bakteri metanotrof (Rudd dan Taylor 1980). Kedua komunitas mikroorganisme ini memiliki ciri dan karakter yang berbeda (Tabel 1).
Arkaea metanogen adalah mikroorganisme prokariotik filum Euryarchaeota yang menghasilkan metana sebagai produk akhir metabolisme Proses metabolism yang terjadi pada arkaea metanogen dapat mengubah karbon dioksida, asam format, asetat, metanol, metilamin dan karbon monoksida menjadi metana (Tabel 2) (Ciceron dan Oremland 1988). Arkaea metanogen menggunakan asetat sebagai sumber karbon utama menghasilkan kira-kira 80% produksi metana, sedangkan susbtrat lainnya seperti CO2 dan format berkontribusi 10-30%
4
salinitas, dan pH tinggi (Liu et al. 2008; Jones et al. 1987). Sebagian besar arkaea metanogen bersifat termofilik, dapat berfungsi pada rentang suhu 20 – 40oC bahkan pada suhu 98oC (Topp dan Pattey 1997).
Arkaea metanogen secara filogeni dibagi lima ordo, yaitu Methanobacteriales, Methanococcales, Methanosarcinales, Methanophyrales, dan Methanomicrobiales (Boone et al. 1993). Menurut Garcia (1990) kategori arkaea metanogen berdasarkan gen 16S rRNA menjadi tiga kelompok : kelompok I yaitu Methanobacterium and Methanobrevibacter, kelompok II yaitu Methanococcus, serta kelompok III yaitu Methanospirillum dan Methanosarcina.
Tabel 1 Perbedaan arkaea metanogen dan bakteri metanotrof
Ciri Metanogen Metanotrof
Bentuk sel Batang, kokus, spiral Batang, kokus, vibrio
Reaksi gram gram +/- gram -
Siklus TCA Tidak kompleks Tidak kompleks (tipe I) atau kompleks (tipe II) ribulosa
Bakteri metanotrof adalah mikroorganisme aerob yang dapat tumbuh dan berkembang menggunakan metana sebagai satu-satunya sumber energi. Oleh karena itu, oksidasi metana hanya dapat terjadi pada lingkungan yang bersifat aerob pada zona perakaran dan pada bagian yang bersifat aerob pada lapisan permukaan tanah.
5 analisis gen 16S rRNA menunjukkan bahwa isolat BGM 1 dan BGM 3 memiliki kemiripan dengan Methylocystis rosea (Metanotrof tipe II), BGM 2 dan BGM 9 memiliki kemiripan dengan Methylococcus capsulatus (Metanotrof tipe X), dan SKM 14 memiliki kemiripan dengan Methylobacter sp. (Metanotrof tipe I).
Tabel 2 Karakteristik beberapa arkaea metanogen
6
Metagenomik
Metagenomik merupakan pendekatan yang dapat diajukan sebagai solusi untuk mengatasi keterbatasan pemetaan genom yang berbasis kultur. Metagenomik dikembangkan dengan pendekatan pengumpulan informasi genetik dari total DNA mikroorganisme yang berasal dari sampel lingkungan, termasuk dari lingkungan yang ekstrem, tanpa melalui tahap kultur terlebih dahulu. Menurut Patrick dan Handelsman (2005) metagenomik merupakan analisis tanpa pengkulturan dari campuran genom mikroorganisme. Upaya analisis metagenomik memerlukan penanda molekuler baik universal maupun penanda yang spesifik. Beberapa gen telah digunakan sebagai penanda molekuler bagi kelompok prokariot dan yang paling banyak digunakan ialah gen penyandi 16S rRNA. Stackebrandt dan Goebel (1995) melaporkan gen 16s rRNA bersifat ubikuitus dengan fungsi yang identik pada seluruh organisme sehingga dapat digunakan sebagai penanda molekuler. Gen ini mengkode RNA ribosomal pada subunit kecil ribosom dan memiliki urutan nukleotida yang khas. Perkembangan teknologi molekuler dalam menganalisis ekologi bakteri, dengan menggunakan sekuen gen 16S rRNA sebagai marker molekular untuk mengidentifikasi mikroorganisme telah mengubah persepsi tentang keragaman komunitas bakteri. Gen 16S rRNA memiliki sembilan region (V1–V9) yang memiliki ukuran panjang basa yang berbeda-beda.
Beberapa teknik berbasis metagenomik yang digunakan untuk menganalisis keanekaragaman mikroorganisme ialah Low Molecular Weight (LMW), Denaturing Gradient Gel Electrophoresis (DGGE), Temperature Gradient Gel Electrophoresis (TGGE), Fluorescent in-situ Hybridization (FISH), Terminal Restriction Fragment Length Polymorphism (TRFLP) dan Amplified Ribosomal DNA Restriction Analysis (ARDRA), dan Single Stranded Conformation Polymorphism (SSCP) (Höfle 1998; Muyzer et al. 1993; Amann et al. 1990; Liu et al. 1997; Smit et al. 1997; Schwieger dan Tebbe 1998). DGGE pertama kali dilakukan untuk menganalisis sampel air danau (Ovreas et al. 1997) kemudian dikembangkan untuk meneliti keragaman komunitas mikroorganisme. Variasi sekuen di antara gen 16S rRNA dari bakteri yang berbeda-beda memberikan
7
METODE
Bahan
Bahan yang digunakan dalam penelitian ini antara lain benih padi Varietas Ciherang, pupuk NPK dan pupuk hayati dengan kombinasi isolat bakteri metanotrof yaitu BGM 1, BGM 5, BGM 9, dan SKM 14 (Hapsari 2008).
Prosedur Penelitian
Penelitian dilakukan dengan menggunakan tiga perlakuan yaitu: Kontrol (300 kg/ha NPK), Celup (Pupuk hayati + 200 kg/ha NPK) dan Sebar (Pupuk hayati + 200 kg/ha NPK). Metode celup didahului dengan proses perendaman benih dalam kultur konsorsium bakteri metanotrof selama ± 15 menit sedangkan metode sebar hanya menyebarkan atau menyiramkan kultur bakteri di sekitar petak. Peremajaan isolat bakteri dilakukan dengan menumbuhkan kembali isolat bakteri sesuai dengan media tumbuhnya. Isolat bakteri metanotrof diremajakan pada media agar – agar Nitrate Mineral Salt (NMS) + 1% metanol (Lampiran 1). Masing-masing biakan kemudian diinkubasi pada suhu ruang selama 3–7 hari. Bakteri-bakteri hasil peremajaan dikulturkan dalam media cair sesuai jenisnya. Kultur kemudian diinkubasi selama 5–7 hari pada penangas bergoyang dalam suhu ruang (± 37oC).
Sampling Tanah
Sampel tanah dikoleksi pada bulan Juni – September 2013 di sawah Desa Cidahu, Kecamatan Cicurug, Kabupaten Sukabumi, Jawa Barat. Analisis molekuler dilakukan di Laboratorium Mikrobiologi, Departemen Biologi, FMIPA dan Labaratorium Terpadu, Institut Pertanian Bogor (IPB). Tanah untuk keperluan ekstraksi diambil menggunakan syringe ukuran 10 mL pada 26 HST, 47 HST dan 96 HST. Analisis karakteristik fisik dan kimiawi tanah diambil dari setiap petak lokasi petak perlakuan, sebanyak ±1000 gram kemudian dikirim ke Balai Penelitian Tanah, Bogor.
Pengukuran Emisi Gas Metana
Gas dikumpulkan dengan menggunakan sungkup statis tertutup pada tanaman padi. Sampel gas sebanyak 100 mL diambil dengan menggunakan syringe dan dimasukkan ke dalam tabung kosong. Sampel gas diambil pada saat setelah penyungkupan (t0), dan 3 jam (t3) setelah penyungkupan. Sampel gas
8
Ekstraksi Total DNA
Ekstraksi DNA dilakukan dengan menggunakan PowerSoil® DNA Isolation Kit (Mobio Laboratories, Carlsbad,CA, USA). Prosedur ekstraksi mengikuti protokol yang direkomendasikan oleh produsen. DNA hasil ekstraksi diukur konsentrasi dan kemurniannya dengan teknik spektrofotometri menggunakan Nano drop 2000 (Thermo Scientific,Wilmington, DE, USA). Di samping itu, DNA hasil ekstraksi dan amplifikasi dicek dengan cara elektroforesis pada gel agarosa dalam Tris-Acetic acid-EDTA (TAE) 1X (1% agarosa untuk DNA genom; 1.5% agarosa untuk DNA amplikon). Elektroforesis dilakukan pada tegangan 80 volt selama 1 jam. Pita DNA hasil elektroforesis diwarnai menggunakan etidium bromide selama ± 15 menit. Selanjutnya gel diamati UV Transiluminator dan Gel Doc.Untuk keperluan dokumentasi dengan menggunakan G : BOX (Syngene, Frederick, MD, USA).
Amplifikasi Fragamen Gen 16s rRNA
Amplifikasi gen 16S rRNA dilakukan menggunakan GoTaq Green Master Mix (Promega, Madison, WI, USA). Komponen reaksi PCR dilakukan dengan total volume 25 µL terdiri atas masing–masing 1.5 µL primer forward dan reverse (10 pmol µ L), 13 µ L GoTaq® DNA Polymerase, 4 µ L DNA sebagai cetakan, dan Nuclease-Free Water. Kondisi PCR yang digunakan yaitu denaturasi awal pada suhu 95 oC selama 2 menit, diikuti denaturasi pada suhu 95 oC selama 1 menit, penempelan primer pada suhu 63 oC selama 1 menit, perpanjangan primer pada suhu 72 oC selama 1 menit, dan pemanjangan akhir pada suhu 72 oC selama 5 menit. Amplifikasi dilakukan sebanyak 30 siklus. Amplifikasi menggunakan Perkin Elmer (Gene PCR System 2400). Runutan primer yang digunakan untuk amplifikasi gen 16S rRNA arkaea metanogen tersaji pada Tabel 3.
Tabel 3 Runutan sekuen primer gen 16S rRNA
Primer Sekuen
0357F* 5’-CCCTACGGGGCGCAG CAG -3’
0915aR 5’-GTGCTCCCCCGCCAATTCCT-3’
*Sekuen GC-Clamps CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGG (Watanabe 2004) dihibridisasi pada ujung 5’ primer forward.
Komposisi Denaturing Gradient Gel Electrophoresis (DGGE)
9 Probes, Carlsbad, CA, USA) selama ± 1 jam. Visualisasi gel dilakukan dengan menggunakan G:BOX (Syngene, Frederick, MD, USA). Pita DNA pada gel DGGE dipotong menggunakan scalpel steril. Potongan gel direndam dalam tabung mikro berisi 100 µL aqua bidestillata steril, dilanjutkan dengan inkubasi pada suhu 37oC selama ± 2 jam, kemudian disimpan pada suhu 4oC (Bodelier et al. 2005). Sebanyak 10 µL DNA hasil elusi digunakan sebagai cetakan untuk reaksi nested PCR. Amplifikasi dilakukan menggunakan primer dan kondisi PCR yang sama dengan sebelumnya tetapi tanpa GC-clamp. DNA amplikon kemudian dikirim ke perusahaan jasa sekuensing.
Determinasi dan Pembuatan Pohon Filogeni
Sekuen yang diperoleh dibandingkan dengan sekuen yang tersedia di NCBI (http://www.ncbi.nlm.nih.gov). Database sekuen gen 16s rRNA yang diperoleh dari penelitian dideterminasi menggunakan program Basic Local Alignment Search Tool Nucleotide (BLAST- N). Aligment sekuen dan konstruksi pohon filogeni menggunakan piranti lunak program Mega 5.2.
HASIL DAN PEMBAHASAN
Hasil
Karakteristik tanah
Tanah pada petak perlakuan kontrol pada 26 HST waktu pengambilan sampel tersusun dari 10% pasir, 43% debu dan 47% liat. Tanah pada petak perlakuan celup pada 26 HST tersusun dari 16% pasir, 33% debu dan 51% liat. Tanah pada petak perlakuan sebar pada 26 HST tersusun 18% pasir, 38% debu dan 48% liat. Petak kontrol, celup, dan sebar memiliki kisaran pH yang relatif sama, yaitu masing-masing 5.3, 5.3 dan 5.1. Kandungan karbon dari petak kontrol, celup, dan sebar secara berurutan ialah 1.97%, 1.81%, dan 1.91%. Kandungan nitrogen dari petak kontrol, celup, dan sebar secara berurutan ialah 0.17%, 0.17% dan 0.14%. Rasio C/N dari tiap petak ialah 12, 11 dan 14. (Lampiran 2).
Emisi metana
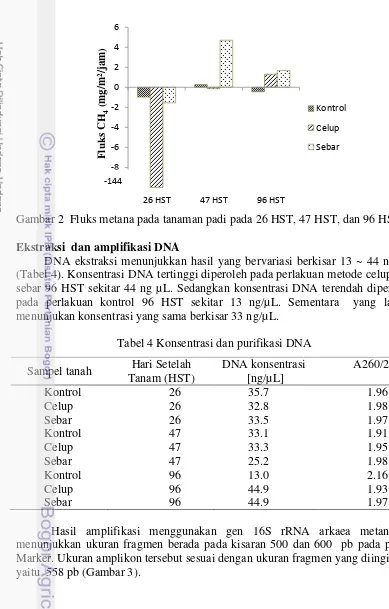
Hasil analisis gas menunjukkan terdapat perbedaan antara tiga perlakuan kontrol, metode celup dan metode sebar (Gambar 2). Berdasarkan analisis fluks metana (CH4) tertinggi secara berurutan terdapat pada perlakuan sebar pada 47
10
Gambar 2 Fluks metana pada tanaman padi pada 26 HST, 47 HST, dan 96 HST Ekstraksi dan amplifikasi DNA
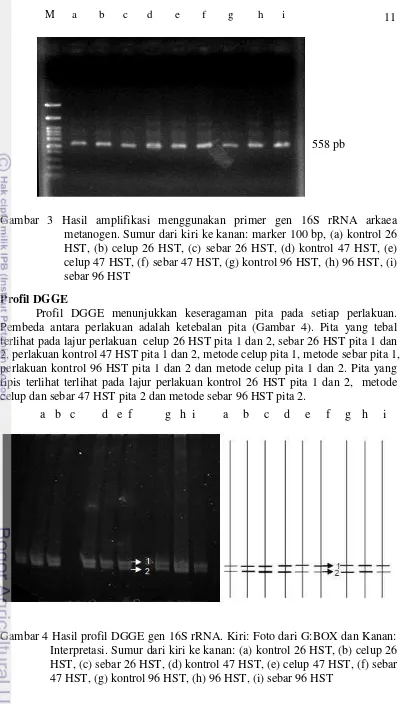
DNA ekstraksi menunjukkan hasil yang bervariasi berkisar 13 ~ 44 ng/µ L (Tabel 4). Konsentrasi DNA tertinggi diperoleh pada perlakuan metode celup dan sebar 96 HST sekitar 44 ng µL. Sedangkan konsentrasi DNA terendah diperoleh pada perlakuan kontrol 96 HST sekitar 13 ng/µL. Sementara yang lainya menunjukan konsentrasi yang sama berkisar 33 ng/µL.
Tabel 4 Konsentrasi dan purifikasi DNA
11
Gambar 3 Hasil amplifikasi menggunakan primer gen 16S rRNA arkaea metanogen. Sumur dari kiri ke kanan: marker 100 bp, (a) kontrol 26 HST, (b) celup 26 HST, (c) sebar 26 HST, (d) kontrol 47 HST, (e) celup 47 HST, (f) sebar 47 HST, (g) kontrol 96 HST, (h) 96 HST, (i) sebar 96 HST
Profil DGGE
Profil DGGE menunjukkan keseragaman pita pada setiap perlakuan. Pembeda antara perlakuan adalah ketebalan pita (Gambar 4). Pita yang tebal terlihat pada lajur perlakuan celup 26 HST pita 1 dan 2, sebar 26 HST pita 1 dan 2, perlakuan kontrol 47 HST pita 1 dan 2, metode celup pita 1, metode sebar pita 1, perlakuan kontrol 96 HST pita 1 dan 2 dan metode celup pita 1 dan 2. Pita yang tipis terlihat terlihat pada lajur perlakuan kontrol 26 HST pita 1 dan 2, metode celup dan sebar 47 HST pita 2 dan metode sebar 96 HST pita 2.
Gambar 4 Hasil profil DGGE gen 16S rRNA. Kiri: Foto dari G:BOX dan Kanan: Interpretasi. Sumur dari kiri ke kanan: (a) kontrol 26 HST, (b) celup 26 HST, (c) sebar 26 HST, (d) kontrol 47 HST, (e) celup 47 HST, (f) sebar 47 HST, (g) kontrol 96 HST, (h) 96 HST, (i) sebar 96 HST
M a b c d e f g h i
558 pb
12
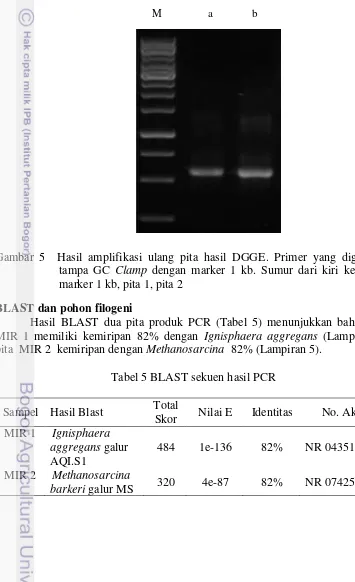
Posisi pita yang sama pada profil DGGE dicukupkan dengan mengelusi 2 pita yang tebal. Hasil elusi digunakan sebagai cetakan untuk melakukan amplifikasi ulang dengan primer yang sama tanpa GC-clamp. Dua pita DGGE yang dipotong berhasil diamplifikasi ulang dan menunjukkan fragmen yang diharapkan memiliki ukuran target 558 pb (Gambar 5).
Gambar 5 Hasil amplifikasi ulang pita hasil DGGE. Primer yang digunakan tampa GC Clamp dengan marker 1 kb. Sumur dari kiri ke kanan: marker 1 kb, pita 1, pita 2
BLAST dan pohon filogeni
Hasil BLAST dua pita produk PCR (Tabel 5) menunjukkan bahwa pita MIR 1 memiliki kemiripan 82% dengan Ignisphaera aggregans (Lampiran 4), pita MIR 2 kemiripan dengan Methanosarcina 82% (Lampiran 5).
Tabel 5 BLAST sekuen hasil PCR Sampel Hasil Blast Total
Skor Nilai E Identitas No. Akses MIR 1 Ignisphaera
aggregans galur AQI.S1
484 1e-136 82% NR 043512.1 MIR 2 Methanosarcina
barkeri galur MS 320 4e-87 82% NR 074253.1
13 Konstruksi pohon filogeni menggunakan metode Neighbour Joining dengan best model Tamura 3 Parameter dan nilai bootstrap 1000 kali.
Gambar 6 Pohon filogenetik arkaea metanogen dari DGGE di lahan sawah Sukabumi. Sekuen yang diperoleh ditandai segitiga (▲)
Pembahasan
Kondisi lingkungan sangat mempengaruhi keberadaan komunitas arkaea metanogen dan bakteri metanotrof di dalam tanah. Dubey (2005) menjelaskan emisi metana dari sawah dikendalikan oleh parameter yang kompleks dan berhubungan dengan karakteristik fisik dan biologis tanah dengan praktek budidaya yang spesifik. Berdasarkan kriteria dari USDA (1987), tanah perlakuan kontrol termasuk kategori tanah lempung berdebu (Silty Clay) sedangkan perlakuan celup dan sebar tergolong tanah liat (Clay) (Lampiran 3). Sass et al. (1999) melaporkan emisi metana di tanah liat (Clay) sangat rendah. Menurut Sulaeman et al. (2005) bahwa pH tanah pada semua perlakuan kontrol, celup dan sebar tergolong masam. Kondisi tanah masam mempengaruhi aktivitas metabolik arkaea metanogen.
Emisi metana
Emisi metana (CH4) pada masing-masing perlakuan relatif rendah, emisi
14
metabolik arkaea metanogen. Isolat bakteri metanotrof sebelumnya telah diuji di laboratorium. Rusmana dan Akhdiya (2009) serta Hapsari (2008) menunjukkan bahwa isolat BGM 9 memiliki aktivitas mengurangi CH4 yang paling baik.
Selanjutnya Hanif (2010) melaporkan isolat SKM 14 memiliki aktivitas oksidasi metan tertinggi. Kedua, rendahnya emisi metana ke atmosfer diduga aktivitas arkaea metanogen yang kurang optimal. Fenomena ini didasarkan pada hasil analisis pH tanah yang rendah yaitu rata-rata pH 5. Kondisi pH tanah <5.75 menyebabkan bakteri metanogen tidak mampu hidup dengan baik sehingga berpengaruh terhadap fluks metana yang diemisikan (Isminingsih 2009). Wang et al. (1993) melaporkan bahwa kisaran pembentukan metana yang ideal terjadi pada pH 6.9 – 7.1. Penurunan pH tanah juga mempengaruhi sejumlah reaksi biokimia yang berdampak pada biomas mikroorganisme dan kandungan karbon organik tanah. Secara umum pemberian konsorsium bakteri metanotrof melalui metode celup cukup efektif mereduksi keberadaan metana di sekitar perakaran padi. Nilai fluks negatif yang cukup besar pada 26 HST dan 47 HST merupakan indikasi optimalnya aktivitas metanotrof. Efektivitas metode celup dalam mereduksi metana di sekitar perakaran karena asosiasi yang cukup efektif antara benih padi yang mengandung berbagai senyawa-senyawa organik eksudat akar sehingga bakteri metanotrof dapat lebih mudah untuk berafiliasi dan beradaptasi.
Pada pengamatan 96 HST secara teoritis nilai fluks yang menuju atmosfer lebih rendah, hal ini akibat dari proses pengeringan petak sawah sehingga sawah tanah akan bersifat oksik yang menyebabkan arkaea metanogen tidak mampu secara optimal untuk melangsungkan metabolisme. Sementara sifat oksik tanah akan menguntungkan bakteri metanotrof yang secara alamiah hidup pada lingkungan yang aerob. Nilai fluks negatif pada perlakuan kontrol diduga akibat sangat rendahnya metana yang terbentuk akibat kurang optimalnya pertumbuhan arkaea metanogen.
Ekstraksi dan amplifikasi DNA
Ekstraksi DNA dari lingkungan alami biasanya serupa dengan ekstraksi DNA kultur murni. Langkah dasar yang harus dilakukan meliputi penghancuran matriks tanah, lisis sel, pemisahan DNA dari serpihan sel, dan pemurnian DNA (Meyer 2006). Hasil ekstraksi menunjukkan kemurnian DNA cukup baik dengan parameter rasio absorbansi A260/A280 rata-rata berkisar 1.9. Nilai rasio absorbansi A260/A280 yang dianjurkan berkisar 1.8 - 2.0 (Sambrook dan Russell 2001). Menurut Santella (2006) rasio absorbansi pada panjang gelombang A260/A280 yang berada di atas kisaran nilai DNA murni menunjukkan terdapat kontaminasi RNA sedangkan rasio 1.8 menunjukan masih terdapat kontaminasi protein. Kemurnian DNA menjadi salah satu faktor penentu keberhasilan proses amplifikasi DNA.
15 (2008) menggunakan primer Ar109F/Ar915R untuk mengidentifikasi 16S rRNA arkaea metanogen di lahan sawah. Penggunaan primer untuk mendeteksi kelimpahan atau identifikasi arkaea metanogen di suatu lingkungan dapat menggunakan sekuen yang dapat kompatibel dengan sekuen region V1-V9 gen 16S rRNA. Lebih lanjut Lane et al. (1985) melaporkan region gen 16S rRNA (rDNAs) V1, V2, V3, V4, V5, V6, V7, V8, dan V9 dapat digunakan untuk identifikasi. Ukuran gen 16S rRNA memiliki daerah lestari (conserved) dan region hypervariable (V1-V9). Region gen 16S rRNA memiliki panjang basa yang berbeda pada setiap ruas region. Primer 0357F/0915aR memiliki ukuran target 558 pb, sekuen primer ini berada pada region V3-V5. Secara umum produk PCR region V3 menghasilkan informasi DGGE yang banyak (Yu et al. 2007). Selain region V3 yang seringkali menjadi target untuk keperluan identifikasi, region (V1-V3 atau V3-V5) cukup baik digunakan untuk mendeteksi kelimpahan mikroorganisme di lingkungan (Wahl et al. 2013). Lebih lanjut Yu dan Morrison (2004) melaporkan bahwa profil DGGE region V3 jauh lebih baik daripada region lainnya.
Keragaman arkaea metanogen
Profil DGGE menunjukkan di setiap perlakuan kontrol, metode celup dan metode sebar terdapat dua pita yang sama di setiap petak dan waktu perlakuan. Adanya dua pita mengasumsikan spesies yang berbeda. Hal yang menjadi pembeda profil pita DGGE ialah ketebalan pita yang terbentuk. Pita yang tampak tebal mendeskripsikan dominansi populasi mikroorganisme yang mendiami suatu petak perlakuan. Tingginya tingkat dominansi populasi bergantung pada kandungan dan kualitas karbon organik tanah, tekstur tanah, Eh, pH, pengairan, dan penggunaan pupuk.
Rendahnya jumlah pita yang muncul pada profil DGGE mengunakan primer 0357F/0915aR berdasarkan pada dua asumsi. Pertama, pengunaan denaturan yang tidak sesuai dan waktu separasi yang kurang efektif (Watanabe et al. 2004). Canche et al. (2013) melaporkan bahwa gen 16S rRNA konsorsium bakteri, ukuran 580 pb memperlihatkan keragaman pita yang banyak, DGGE dilakukan pada gradien 30-70% dengan lama migrasi 18 jam. Kedua, penggunaan primer yang digunakan yang mengamplifikasi DNA pada ukuran cukup besar 558 pb terletak pada region V3-V5. Ercolini (2004) melaporkan bahwa produk PCR kurang dari 500 pb memberikan hasil yang lebih baik untuk profil DGGE. Primer dengan produk PCR 600 pb menghasilkan profil DGGE yang kurang baik (Cheng et al. 2009; Thies 2007).
16
Kelimpahan Methanosarcina di lahan sawah telah banyak dilaporkan oleh peneliti di berbagai tempat. Rajgopal et al. (1988) berhasil mengisolasi dan mengkarakterisasi arkaea metanogen di lahan sawah, yaitu Methanosarcina dan Methanobacterium. Fetzer et al. (1993) mengisolasi empat genus di lahan sawah Italia salah satunya ialah Methanosarcina. Di tempat yang sama Groβkopf et al. (1998) mengisolasi Methanosarcina, Methanosaeta, dan Methanobacterium. Kudo et al. (1997) melaporkan bahwa Methanosarcina sangat dominan. Watanabe et al. (2006) dan Sakai et al. (2008) mengidentifikasi Methanomicrobiales, Methanosarcinales, Methanosaotaceae dan Rice cluster I. Wang et al. (2010) mendeteksi Methanosaetaceae, Methanomicrobiales, Rice cluster I, Methanocellales, dan Methanosarcinaceae. Ma dan Lu (2011) melaporkan berdasarkan analisis gen 16S rRNA terdeteksi keberadaan struktur komunitas arkaea metanogen di antaranya Methanosarcinaceae, Methanosaetaceae (ordo Methanosarcinales). Daebeler et al. (2013) melaporkan analisis gen mcrA menemukan komunitas Methanosarcinaceae sangat melimpah dibandingkan arkaea metanogen lainnya. Weber et al. (2001) melaporkan arkaea metanogen di lahan sawah yang ditambahkan jerami sebagai sumber organik, terdeteksi Methanobacterium spp dan Methanosarcina spp.
Keberadaan Methanosarcina dapat juga ditemukan di dalam rumen berbagai hewan ternak. Conway de Macario et al. (1987) mengidentifikasi Methanosarcina sp pada burung Angsa. Mukhopadhyay et al. (1991) mengisolasi Methanosarcina pada feses Kambing. Sharp et al. (1998) berhasil mengidentifikasi Methanosarcinales pada rumen Sapi muda. Jarvis et al. (1999) mengisolasi arkaea pada ternak pemakan rumput (Grazing cattle) yang secara morfologi mirip dengan Methanosarcina, selanjutnya dianalisis gen 16S rRNA yang menunjukkan kemiripan dengan Methanosarcina barkeri. Singh et al. (2011) berhasil mengidentifikasi secara molekuler arkaea yang memiliki kekerabatan dengan Methanosarcina barkeri. Sundset et al. (2009) mengidentifikasi arkaea metanogen yaitu Methanobacteriaceae, Methanosarcinaceae dan Methanobrevibacter pada rumen Kijang. Laporan lain Methanosarcinales (Methanosarcina) juga ditemukan di berbagai tempat, yaitu di danau (Simankova et al. 2001), sungai (Buriánková 2013), laut baltik kedalaman 241 meter (Von Klein et al. 2002), hutan (Li et al. 2012).
Kelimpahan Methanosarcina yang begitu banyak di berbagai tempat disebabkan oleh kemampuan menggunakan substrat yang beragam dalam metabolismenya. Genus Methanosarcina mampu menggunakan H2+CO2, metanol,
metilamin dan asetat (Madigan et al. 2000). Hal ini dipertegas sebelumnya oleh Boone et al. (1993) bahwa Methanosarcina dan Methanosaeta (Ordo Methanosarcinales) dapat menggunakan asetat untuk memproduksi metana. Methanosarcina barkeri dapat menggunakan H2 + CO2, metanol, beberapa
17 SIMPULAN DAN SARAN
Simpulan
Pengunaan kombinasi pupuk NPK 200 kg/ha dan konsorsium isolat bakteri metanotrof metode celup mampu mengurangi emisi metana di lahan sawah Desa Cidahu, Sukabumi. Keragaman komunitas metanogen menggunakan primer gen 16S rRNA 0357F dan 0915aR di setiap petak menunjukkan dua pita amplikon. Pita amplikon MIR 1 memiliki tingkat kemiripan 82% dengan Ignisphaera aggregans galur AQI.S1 sedangkan pita amplikon MIR 2 82% dengan Methanosarcina barkeri galur MS.
Saran
Perlu dilakukan pengukuran gas di malam hari guna mengetahui total fluks aktual selama 24 jam. Analisis metagenom berdasarkan mRNA gen fungsional perlu dilakukan untuk mengetahui arkea metanogen yang aktif dalam menghasilkan gas metana.
DAFTAR PUSTAKA
Adachi K. 2001. Methanogenic archaea and methanotrophic bacteria in a subtropical paddy field and their interaction: controling methane emission paddy fields. Microbes Environ. 16 : 197 – 205.
Achtrich C, Schuhmann A, Wind T, Conrad R. 1995. Role of interspecies H2
transfer to sulfate and ferric iron reducing bacteria in anoxic paddy soil. J Microbiol Ecol. 16 : 61 – 70.
Amann RI, Krumholz L, Stahl DA. 1990. Fluorescent-oligo-nucleotide probing of whole cells for determinative phylogenetic, and environmental studies in microbiology. J Bacteriol. 172 : 762 – 770.
Amann RI, Ludwig W, Schleifer KH. 1995. Phylogenetic identification and in situ detection of individiual microbial cells without cultivation. Microbiol Rev. 59 : 143 – 169.
Astuti DA. 2009. Karakterisasi Fisiologi dan identifikasi molekuler isolat-isolat bakteri metanotrof asal sawah wilayah Bogor dan Sukabumi [skripsi]. Bogor (ID) : Institut Pertanian Bogor.
Atlas RM. 2005. Handbook Media for Environmental Microbiology. London (ENG) : CRC Press.
Bock AK, Schonheit P. 1995. Growth of Methanosarcina barkeri (Fusaro) under non methanogenic condition by the fermentation of pyruvate to acetat : ATP syntesis via the mechanism of substrate level phosphorylation. J Bacteriol. 177 : 2002 – 2007.
18
Boone D, Whitman W, Rouvier P.1993. Diversity and Taxonomy of methanogens In:Methanogenesis. Ferry JG, Editor. New York (US) : Chapman and Hall.
Buriánková I, Brablcová L, Mach V, Dvořák P, Chaudhary PP, Rulik M. 2013. Identification of Methanogenic archaea in the hyporheic sediment of Sitka Stream. PLoS ONE. 8 (11): e80804. doi:10.1371/journal.pone.0080804. Canché BC, Sima MT, Loria JIV, Alvares HE, Manzanero BHC, Herrera RR,
Ojeda RV, Gaviria LA. 2013. Simple and inexpensive DNA extraction protocol for studying the bacterial composition of sludges used in microbial fuel cells. Gen Mol Res. 12 (1) : 282 – 292.
Ciceron RJ, Oremland RS. 1988. Biogeochemical aspects of atmospheric methane. J Global Biogeochem Cycle. 2 : 299 – 327.
Chen K, Pachter L. 2005. Bioinformatics for whole genome shotgun sequencing of microbial communities. Plos Comp Biol. 1 : 24.
Cheng YF, Mao SY, Liu JX, Zhu WY. 2009. Molecular diversity analysis of rumen methanogenic archaea from goat in eastern China by DGGE methods using different primer pairs. App Microbiol. 48 : 585 – 592.
Chin KJ, Conrad R. 1995. Intermediary metabolism in methanogenic paddy soils and the influence of temperature. FEMS Microbiol Ecol. 18 : 85 – 102. Conrad R, Rothfus F. 1991. Methane oxidation in the soil surface layer of a
flooded rice field and the effect of ammonium. Biol Fertil Soil. 12 : 28 – 32. Conway de Macario E, Macario AJL, Miller TL, Wolin MJ. 1987. Antigenic
diversity of methanogenic bacteria from interstinal tracts of animal. Syst Appl Microbiol. 9 : 210 – 213.
Cowan DA. 2008. Microbial genomes - the untapped resource. Trends Biotechnol. 18 : 14 – 16.
Daebeler A, Gansen M, Frenzel P. 2013. Methyl fluoride affects methanogenesis rather than community composition of methanogenic archaea in a rice field soil. PLOS ONE. 8 (1) : e53656.doi:10.1371/journal.pone.0053656.
Dalal RC, Wang W, Robertson GP, Parton WJ. 2003. Nitrous oxide emission from australian agricultural lands and mitigation Options: a review. Aust J Soil Res. 41 : 165 – 195.
Dedysh SN, Rickle P, Liesack W. 2004. NifH and NifD phylogenies: an evolutionary basis for understanding nitrogen fixation capabilities of methanotrophic bacteria. Microbiol. 150 : 1301 – 1313.
Dubey SK. 2005. Microbial ecology of methane emission in rice agroecosystem : A Review. Appl Ecol Environ Res. 3 (2) : 1 – 27.
Ercolini D. 2004. PCR-DGGE fingerprinting : novel strategies for detection of microbes in food. J Microbiol. 56 : 297 – 314.
Ernawanto QD, Saeni MS, Sastiono A, Partohardjono S. 2003. Dinamika metana pada lahan sawah tadah hujan dengan pengolahan tanah varietas dan bahan organik yang berbeda. Forum Pascasarjana IPB. 26 (3) : 241 – 255
Fetzer S, Bak F, Conrad R. 1993. Sensivity of methanogenic bacteria from paddy soil to oxygen and dessication. FEMS Microbiol Ecol. 12 : 107 – 115. Garcia IL.1990. Taxonomy and ecology of methanogens. FEMS Microbiol Rev.
87 : 297 – 308.
19 Groβkopf R, janssen PH, Liesack W. 1998. Diversity and structure of the methanogenic community in anoxic rice paddy soil microcosms as examined by cultivation and direct 16S rRNA gene sequence retrieval. App Environ Microbiol. 64 (3) : 960.
Hanif SN. 2010. Aktivitas fiksasi nitrogen dan oksidasi metan kultur campuran
bakteri metanotrof pada lumpur sawah [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Hanson R, Hanson TE. 1996. Methanotrophic bacteria. J Microbiol Rev. 60 (2) : 439 – 471.
Hapsari W. 2008. Isolasi dan karakterisasi bakteri metanotrof dari lahan persawahan di daerah Bogor dan Sukabumi [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Höfle MG.1998. Genotyping of bacterial isolates from the environment using low molecular weight RNA fingerprints. Mol Microb Ecol Manual. 3.3.7: 1 – 23. [IPCC] Intergovernmental Panel on Climate Change 1994. Climate change
1994.Cambridge (US) : Cambridge University Press.
[IPCC] Intergovernmental Panel on Climate Change. 2007. The physical science basis. Cambridge (US) : Cambridge University Press.
Isminingsih. 2009. Studi kecenderungan emisi gas rumah kaca (GRK) dan neraca karbon pada berbagai sistem pengelolaan tanaman padi [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Jarvis GN, Strompl C, Burgess DM, Skillman LC, Moore ERB, Joblm KN. 1999. Isolation dan identification of ruminal methanogen from Grazing Cattle. Curr Microbiol. 40 : 327 – 332.
Jumadi A, Inubushi H. 2012. Methane and nitrous oxide and community stucture of methanogenic archaea in paddy soil of south Sulawesi. Microbiol Indones. 6 (3) : 98 – 106.
Jones WJ, Nagle JDP, Whitman WB.1987. Methanogen sand the diversity of archaebacteria. Microbiol Rev. 51 : 135 – 177.
Kellenberger E. 2001. Exploring the unknown: The silent revolution of microbiology. Embo Rep. 2 (1) : 5 – 7.
Kudo Y, Nakajima T, Miyaki, Oyaizu H. 1997. Methanogen flora of paddy soil in Japan. FEMS Microbiol Ecol. 22 : 39 – 48.
Lane D, Pace B. Olsen GJ, Stahl DA, Sogn ML. 1985. Rapid determination of 16S ribosomal RNA sequences for filogenetic analyses. Proc Natl Acad Sco. 82 : 6955 – 6959.
Lelieveld J, Crutzen PJ, Bruhl C. 1993. Climate effects of atmospheric methane. Chemosphere. 26 : 739 – 768.
Li Ya, Lan SK, Rong YP, Cheng QW, Huang CJ, Wu SH, Yi Y. 2012. Isolation and characterization of a new Methanosarcina mazei strain GFJ07 from a mountain forest pond. Malay J Microbiol. 8(1) : 6 – 10.
Liu WT, Marsh TL, Cheng H, Forney LJ. 1997. Characterization of Microbial diversity by determining terminal restriction fragment Length polymorphisms of gene sencoding 16S rRNA. Appl Environ Microbiol. 63 : 4516 – 4522
20
Ma K, Lu Y. 2011. Regulation of microbial methane production and oxidation by intermittent drainage in rice feld soil. FEMS Microbiol Ecol. 75 : 446 – 456. Madigan MT, Martinko JM, Parker J. 2000. Brock Biology of Microorganisms.
Ed Ke 49. New Jersey (US) : Prentice Hall.
Mer JL, Roger P. 2001. Production, oxidation,emission and comsumption of methane by soil: A review. Eur J Soil Biol. 37 : 25 – 50.
Meyer QC. 2006. Metagenomic approaches to gene discovery [Thesis]. Bellville (ZA) : University of Western Cape.
Mukhopayay B, Purwantini E, Conway de Macario E, Daniel L. 1991. Characterization of methanosarcina strain isolate from goat feces and that grow on H2-CO2 only after adaptation. Curr Microbiol. 23 : 165 – 173.
Muna N. 2011. Aplikasi kompos bungkil jarak pagar untuk mereduksi emisi gas rumah kaca dan karbodioksida (CO2), metana (CH4) dan nitrooksida
(N2O)dari perkebunan jarak pagar [skripsi]. Bogor (ID): Institut Pertanian
Bogor.
Muyzer G, De Wall EC, Luttelinden AG. 1993. Profiling of complex microbial population by Denaturing Gradient Gel Electrphoresis analysis of Polymerase Chain reaction amplifield genes coding for 16S rRNA. App Environ Microbiol. 59 (3) : 695 – 700.
Muyzer G, Smalla K. 1998. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology. Antonie van Leeuwenhoek. 73 (1) : 127 – 141.
Niederberger TD, Gotz DK, MC Donald IR, Ronimus RS, Morgan HW. 2006. Ignisphaera aggregans gen. nov.,sp. Nov., a novel hyperttermophilic crenarchaeota isolated from hot springs in Rotuna and Tokaru, New Zealand. Int J Sys Evol Microbiol. 56 : 965 ‒ 971.
Ovreas L, Forney L, Daae FL, Torsvik V. 1997. Distribution of bacterioplankton in meromictic lake Salenvannet as determined by denaturing gradient gel electrophoresis of PCR-amplified gene fragments coding for 16S rRNA. Appl Environ Microbiol. 63 : 3367 – 3373.
Patrick DS, Handelsman J. 2005. Metagenomics for studying unculturable microorganism: cutting the Gordian knot. Genome Biol. 6 : 229.
Peng J, Zhe L, Junpeng R, Yahai L. 2008. Dynamics of the methanogenic archaeal community during plant residue decomposition in an anoxic rice field soil. Appl Environ Microbiol. 2894 – 2901.
Pingak GMF. 2013. Efektivitas bakteri metanotrof dan Ochrobactrum anthropic sebagai pupuk hayati dan pereduksi emisi gas CH4 serta N2O di sawah
anorganik [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Rajgopal BS, Belay N, Daniel L. 1998. Isolation and characterization of methanogenic bacteria from paddies. FEMS Microbiol Ecol. 53 : 153 – 158. Rudd JWN, Taylor CD. 1980. Methane cycling in aquatic environment. Adv Aq
Microbiol. 2 : 77 – 150.
Rusmana I, Akhdiya A. 2009. Isolation and characterization of methanotrophic bacteria from rice fields. Biotropia 16: 71 – 78.
Sakai S, Imachi H, Hanada S, Ohashi A, Harada H. and Kamagata Y. 2008. Methanocella paludicola gen. nov., sp. nov., a methane-producing archaeon,
the frst isolate of the lineage ‘Rice Cluster I’, and proposal of the new
21 Sambrook JF, Russell DW. 2001. Molecular Cloning: A Laboratory Manual. 3rd
Edition. New York (US): Cold Spring Harbor Laboratory Press.
Santella RM. 2006. Approaches to DNA extraction and whole genome amplification. Cancer Epidemiol Biomarkers Prev. 15 (9) : 1585 – 1587. Sass RL, Fisher FMJ, Ding A, Huang Y. 1999. Exchange of methane from rice
fields. J Geophys Res Atm.104 : 26943 – 26951.
Schloss PD, Handelsman J. 2003. Biotechnological prospects metagenomics. Environ Biotechmol. 14 : 303 – 310.
Schwieger F, Tebbe CC.1998. A new approach to utilize PCR-Single-Strand Conformation Polymorphism for 16S rRNA-based microbial community analysis. Appl Environ Microbiol. 64 : 4870 – 4876.
Singh KM, Pandya PR, Parnerkar S, Tripathi AK, Rank DN, Kathari RK, Joshi CG. 2011. Molecular identifikation of methanogenic archaea from Surti Buffaloes ( Bubalus bubalis) reveal more dehidrogenotrophic methanogen phylotypes. Braz J Microbiol. 42 : 132 – 139.
Simankova MV, Parshina SN, Tourova TP, Kolganova TV, Zehnder AJ, Nozhevnikova AN. 2001. Methanosarcina lacustris sp. nov., a new psychrotolerant methanogenic archaeon from anoxic lake sediments. Syst Appl Microbiol. 24(3) : 362 – 367.
Sekiguchi Y. 2006. Yet-to-be cultural microorganism relevant to methane fermentation processes. Microbes Environ. 21 : 1 – 15.
Setyanto P. 2006. Varietas padi rendah emisi gas rumah kaca. Warta Penel Pengemb Penel. 28 (4) : 1 – 13.
Sharp R, Ziemer CJ, Stern MD, Stahl DA. 1998. Taxon spesific association between protozoal and methanogen population in the rumen and a model rumen system. FEMS Microbial Ecol. 26 : 71 – 78.
Smit E, Leeflang P, Wernars K. 1997. Detection of shifts in microbial community structure and diversity in soil caused by copper contamination using amplified ribosomal DNA restriction analysis. FEMS Microbiol Ecol. 23 : 249 – 261.
Stackebrandt E, Goebel BM. 1995. A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology. Int J Syst Bacteriol. 44 : 846 – 849.
Sugano A, Tsuchimoto H, Cho cho T, Kimura M, Asakawa S. 2005. Succession of methanogenic archaea in rice straw incorporated into a Japanese rice dan karakteristik tanah pada beberapa macam sistim budidaya. Bul agron. 34 (3) : 181 – 187.
Sundset MA, Edward JE, Cheng YF, Senosiain RS, Fraile MN, Nothwood KS, Praesteng KE, Glad T, Mathiesen SD, Wright AD. 2009. Molecular diversity of rumen microbiome of Norwegian reindeer on natural summer pasteure. Microb Ecol. 57 : 335 – 348.
22
Topp E, Pattey E. 1997. Soil as a source and sinks for atmospheric methane. Can J Soil Sci. 77 : 167 – 178.
[USDA] United States Department of Agriculture. 1987. USDA Soil Texture Classification [internet]. [diunduh 2014]. Tersediapada [http://www.ggtern. com/2014/01/15/usda-textural-soil-classification].
Von Klein D, Arab H, Völker H, Thomm M. 2002. Methanosarcina baltica sp. nov., a novel methanogen isolated from the Gotland Deep of the Baltic Sea. Extremophiles. 6 (2) :103 – 10.
Wahl B, Ernst F, Kumar Y, Müller B, Stangier K , Paprotka T. 2013. Defining the microbial composition of environmental samples using next generation sequencing. GATC Biotech [internet].[diunduh 20 September 2014]. Tersedia pada: www.gatc-biotech.com/pdf.
Wang ZP, Delaune RD, Masscheleyn, Patrick WH. 1993. Soil redox and pH effects on methane production in flooded rice soil. J Soil Sci Soc Am. 57 : 382 – 385.
Wang Y, Hu C, Zhu B, Xiang H, He X. 2010. Effects of wheat straw application on methane and nitrous oxide emissions from purplish paddy fields. Plant Soil Environ. 56 (1) : 16 – 22.
Watanabe T, Susumu A, Asumi N, Kazunari N, Makoto K. 2004. DGGE method for analyzing 16S rDNA of methanogenic archaeal community in paddy field soil. FEMS Microbiol Let. 232 (2) : 153 – 163.
Watanabe T, Kimura M. Asakawa S. 2006. Community structure of methanogenic archaea in paddy feld soil under double cropping (rice–wheat). Soil Biol Biochem. 38 : 1264 – 1274.
Weber S, Lueders T, Friedrich MW, Conrad R. 2001. Methanogenic population involved in the degradation of rice straw in anoxic paddy soil. FEMS Microbiol Ecol. 38 : 11 – 20.
Whittenbury R, Dalton H. 1981. The methylotrophic bacteria. Starr MP, Stolph H, Truper HG, Balows A, Schlegel HG, Editor. Berlin (DE): Springer Berlin Heidelberg.
Whittenbury R, Krieg NG. 1984. Methylococcacea fam. Nov. Krieg NR, Holt JG, Editor. Philadelhia (US) : Williams & Wilkins Co.
Yu Z, Gonzales RG, Schanbacher FL, Morrison M. 2007. Electrophoresis-specific PCR and denaturing gradient gel archaea profiling of methanogens by regions of archaeal 16S rRNA genes in evaluations of different hypervariable.Appl Environ Microbiol. 74 (3) : 340 – 889.
Yu Z, Morrison M. 2004. Electrophoresis PCR-Denaturing Gradient Gel by fingerprinting of microbial communities genes for use in RRS regions of comparisons of different hypervariable. Appl Environ Microbiol. 70 (8) : 480.
23
LAMPIRAN
Lampiran 1 Media tumbuh bakteri metanotrof
Nama Media Komposisi Jumlah (g) per 1 liter dH2O
Nitrate Mineral Salt (NMS)
Agar-Agar MgSO4•7H2O
12.50 1.000
CaCl2•6H2O 0.200
KNO3 atau NaNO3 1.000
KH2PO4 0.272
Na2HPO4•12H2O 0.717
NH4Cl
Metanol
4.000
1%
Na2EDTA 0.500
Trace Element (TE) FeSO4•7H2O 0.200
H3BO4 0.030
CoCl2•6H2O 0.020
ZnSO4•7H2O 0.010
MnCl2•4H2O 3.000
Na2MoO4•2H2O 3.000
NiCl2•6H2O 2.000
CaCl2•2H2O 1.000
Lampiran 2 Hasil uji analisis tanah
No Kode
Tekstur % pH Materi Organik HCl 25%
Bray1 P2O5
ppm
Nilai Tukar kation ( NH4-Acetat IN, pH7 KClIN
Pasir Debu liat H2O KCl
Walkley & Black
C %
Kjeildahl N% C/N
P2O 5 K2O Ca Mg K Na Total KTK
KB
%
Al3+ H+
1 A 10 43 47 5.3 4.3 1.97 0.17 12 145 58 69.7 11.85 2.55 1.15 0.77 16.32 21.25 77 0.2 0.2 2 B 16 33 51 5.3 4.3 1.81 0.17 11 76 57 44.6 12.37 2.00 1.11 0.92 16.40 22.19 74 0.16 0.21 3 C 18 38 44 5.1 3.9 1.90 0.14 14 65 46 3.2 10.02 1.98 0.89 0.87 13.76 20.11 68 0.71 0.39 4 D 18 35 47 5.1 3.9 2.12 0.15 14 56 63 2.1 10.34 2.12 1.18 0.89 14.53 20.46 71 0.44 0.29 5 E 16 42 42 4.9 3.6 1.33 0.13 10 45 31 2.2 9.10 1.92 0.58 0.99 12.59 21.95 57 1.21 0.56 6 F 20 29 51 5.0 3.8 1.50 0.12 13 65 43 1.7 9.10 1.80 0.82 0.72 12.44 21.27 58 0.9 0.48
Keterangan : Analisis dilakukan di Balai Penelitian Tanah Bogor tahun 2013
24
mg/100 g Cmolc/kg Cmolc/kg
Lampiran 3 Sistem klasifikasi jenis tanah
http://www.ggtern.com/2014/01/15/usda-textural-soil-classification/
Code Tekstur Kriteria
Pasir (Sand) Debu (Silt) Liat (Clay)
A : Kontrol 10 43 47 Lempumg berdebu
(Silty Clay)
B : Celup 16 33 51 Liat (Clay)
C : Sebar 18 38 44 Liat (Clay)
D : Kontrol 18 35 47 Liat (Clay)
E : Celup 16 42 42 Lempumg berdebu
(Silty Clay)
F : Cebar 20 29 51 Liat (Clay)
Ket : Kode A, B dan C pengamatan pada 26 HST; Kode D,E dan F pengamatan pada 96 HST
2
Lampiran 4 Runutan basa, BLAST- N pita MIR 1
> Ignisphaera aggregans strain AQ1.S1
GGTTGATCCTGCCGGACCCGACCGCTATCGGGGTGGGGCTAAGCCATGGAAGTCGTACGCCCACCA
AGTGGTGGGCGTGGCGGACGGCTGAGTAACACGTGGCTAACCTACCCTCGGGACGGGGATAGCCCC
GGGAAACTGGGGCTAATCCCCGATAGGTGGAGGGGCCTGGAATGGTCCTCCACCGAAAGGGTCAGA
CCGCGATGAAGGTCTGACCGCCCGAGGATGGGGCTGCGGCCCATCATGGTTGTTGGCGGGGTAACG
GCCCGCCAAGCCGATAACGGGTAGGGGCCGTGAGAGCGGGAGCCCCCAGATGGGCACTGAGACAAG
GGCCCAGGCCCTACGGGGTGCACCAGGCGCGAAACCTCCGCAATGCGGGAAACCTGTGACGGGGTC
ACCCCGAGTGCCCCGAAGAGGGGCTTTTCCCCGCTGTAAGTAGGCGGGGGAATAAGCGGGGGGCAA
GTCTGGTGTCAGCCGCCGCGGTAATACCAGCCCCGCGAGTGGTCGGGACGTTTACTGGGCCTAAAG
CGCCCGTAGCCGGCCCAGTAAGTCCCCACATAAATCCTCGGGCTCAACCCGAGGGCTTGTGGGGAT
ACTGCTGGGCTAGGGGGCGGGAGAGGCCGAGGGTACTCCCGGGGTAGGGGCGAAATCCGATAATCC
CGGGAGGACCACCAGTGGCGAAGGCGCTCGGCTGGAACGCGCCCGACGGTGAGGGGCGAAAGCCGG
GGGAGCGAACCGGATTAGATACCCGGGTAGTCCCGGCTGTAAACGATGCGGGCTAGGTGTTGGACG
GGCTTTGAGCCCGTCCAGTGCCGCAGGGAAGCCGTTAAGCCCGCCGCCTGGGGAGTACGGCCGCAA
GGCTGAAACTTAAAGGAATTGGCGGGGGAGCACCACAAGGGGTGGAGCCTGCGGCTTAATTGGAGT
CAACGCCGGGAATCTTACCGGGGGCGACAGCAGGATGACGGCCAGGCTAACGACCTTGCCCGACAC
GCTGAGAGGAGGTGCATGGCCGTCGCCAGCTCGTGCCGTGAGGCGTCCGGTTAAGTCCGGCAACGA
GCGAGATCCCCGCCCCCAGTTGCTACCCCGACCTCCGGGTCGGGGGCACACTGGGGGGACTGCCGC
CGATAAGGCGGAGGAAGGAGGGGGCCACGGCAGGTCAGCATGCCCCGAATCCCCCGGGCTGCACGC
GGGCTACAATGGCGGGGACAGCGGGTACCGACCCCGAAAGGGGGAGGTAATCCCTGAAACCCCGCC
GTAGTTGGGATCGAGGGTTGCAACTCACCCTCGTGAACGTGGAATCCCTAGTAACCGCGCGTCAAC
ATCGCGCGGTGTACGTCCCTGCTCCTTGCACACACCGCCCGTCGCTCCACCCGAGCGGGGGAGAGG
TGAGGCCTGCTCCATCCGGGGTTATCCCGGGTGGGGTGGGTCGAACCTCTCCTCCGCAAGGGGGGA
GAAGTCGTAACAAGGTAGCCGTACCGGAAGGTGCGGC
3
Lampiran 5 Runutan basa, BLAST - N, pita MIR 2
4
> Methanosarcina barkeri strain MS
GCTCAGTAACACGTGGATAACCTGCCCTTGGGTCTGGCATAACCCCGGGAAACTGGGGATAATTCC
GGATAACGCATATATGCTGGAATGCTTTATGCGTAAAATGGATTCGTCCGCCCAAGGATGGGTCTG
CGGCCTATCAGGTAGTAGTGGGTGTAATGTACCTACTAGCCTACAACGGGTACGGGTTGTGAGAGC AAGAGCCCGGAGATGGATTCTGAGACATGAATCCAGGCCCTACGGGGCGCAGCAGGCGCGAAAACT
TTACAATGCGGGAAACCGTGATAAGGGGACACCGAGTGCTAGCATCATATGCTGGCTGTCCAGGTG
TGTAAACTACACCTGTTAGCAAGGGCCGGGCAAGACCGGTGCCAGCCGCCGCGGTAACACCGGCGG
CCCGAGTGGTGATCGTGATTATTGGGTCTAAAGGGTCCGTAGCCGGTTTGGTCAGTCCTCCGGGAA
ATCTGATAGCTCAACTATTAGGCTTTCGGGGGATACTGCCAGACTTGGAACCGGGAGAGGTAAGAG
GTACTACAGGGGTAGGAGTGAAATCTTGTAATCCCTGTGGGACCACCTGTGGCGAAGGCGTCTTAC
CAGAACGGGTTCGACGGTGAGGGACGAAAGCTGGGGGCACGAACCGGATTAGATACCCGGGTAGTC
CCAGCCGTAAACGATGCTCGCTAGGTGTCAGGCATGGCGCGACCGTGTCTGGTGCCGCAGGGAAGC
CGTGAAGCGAGCCACCTGGGAAGTACGGCCGCAAGGCTGAAACTTAAAGGAATTGGCGGGGGAGCA
CAACAACGGGTGGAGCCTGCGGTTTAATTGGACTCAACGCCGGACAACTCACCGGGGACGACAGCA
ATATGTAGGTCAGGCTGAAGACCTTACCTGAATCGCTGAGAGGAGGTGCATGGCCGTCGCCAGTTC
GTACTGTGAAGCATCCTGTTAAGTCAGGCAACGAGCGAGACCCGTGCCCACTGTTACCAGCATATC
CTCCGGGATGATGGGTACTCTGTGGGGACCGCCGGTGTTAAATCGGAGGAAGGTGCGGGCCACGGT
AGGTCAGTATGCCCCGAATTTCCCGGGCTACACGCGGGCTACAATGAATGGGACAATGGGTCCCTC
CCCTGAAAAGGGCTGGTAATCTCACAAACCCATCCTTAGTTCGGATCGAGGGCTGTAACTCGCCCT
CGTGAAGCTGGAATCCGTAGTAATCGCGTTTCAATATAGCGCGGTGAATACGTCCCTGCTCCTTGC
ACACACCGCCCGTC
5
RIWAYAT HIDUP
Penulis lahir di Tombula pada tanggal 01 Desember 1989 sebagai anak pertama dari lima bersaudara, dengan ayah bernama Bapak La Udi Lopa dan Ibu bernama Wa Heto binti La Rindo. Penulis lulus dari SMU Negeri 1 Tongkuno pada tahun 2007 dan pada tahun yang sama diterima di Program studi Biologi, Fakultas Keguruan dan Ilmu Pendidikan, Universitas Haluoleo. Penulis menyelesaikan program sarjana akhir tahun 2011.
Tahun 2012 penulis melanjutkan studi program Magister di Institut Pertanian Bogor mayor Mikrobiologi Fakultas Matematika dan Ilmu Pengetahuan Alam melalui program Beasiswa Unggulan 2012 (sekarang BPP-DN). Hasil penelitian tesis diajukan untuk publikasi di Makara Journal of Science dengan judul ″Methanogenic archaea community in rice fields by PCR-DGGE analysis″.