ANALISIS KERAGAMAN GENETIK IKAN BAUNG
(
Hemibagrus nemurus
) DENGAN MENGGUNAKAN
TEKNIK PCR-RFLP PADA DNA MITOKONDRIA
NANDA PADRIA
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis dengan judul “Analisis Keragaman Genetik Ikan Baung (Hemibagrus nemurus) dengan Menggunakan Teknik PCR-RFLP pada DNA Mitokondria” adalah karya saya beserta komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, September 2012
ABSTRACT
NANDA PADRIA. Analysis of Genetic variability of yellow chatfish (Hemibarus nemurus) by using PCR-RFLP technique on mitochondrial DNA. Under direction of SUHARSONO dan ESTU NUGROHO.
This research has an objective to analyze the genetic variation among four Hemibagrus nemurus population by using PCR-RFLP of D-loop region of mitochondrial DNA. H. nemurus were collected from Cirata, Serayu, Jambi and Bogor. Four restriction enzymes were used to digest D-loop region mt-DNA, i.e. MboI, HaeIII, AluI and RsaI. Based on this analysis, these four population have seven composite haplotypes. The highest haplotype diversity was found at the population from Jambi. Population from Serayu has just one composite haplotype indicating that this population has the same ancestor. Population from Cirata and from Bogor has the same composite haplotype indicating that there was probably migration between these two populations.
RINGKASAN
Nanda Padria. P051100151. Analisis Keragaman Genetik Ikan Baung (Hemibagrus nemurus) dengan Menggunakan Teknik PCR-RFLP pada DNA Mitokondria. Dibimbing oleh SUHARSONO dan ESTU NUGROHO.
Ikan baung sebagai salah satu komoditi perikanan air tawar masih mempunyai kendala dalam upaya budidaya sehingga buididaya ikan ini tidak seintensif komoditi perikanan air tawar lainnya. Permasalahan yang masih ada yaitu ketidakmampuan untuk berkembang biak di penangkaran (Muflikhah et al. 1993). Penyediaan benih ikan baungpun masih mengandalkan tangkapan dari alam, karena pemijahan buatan yang menghasilkan daya tetas telur yang masih rendah (Muflikhah et al. 1993; Sukendi. 2005). Salah satu kajian yang perlu dilakukan untuk memperbaiki secara genetik ikan baung adalah kajian keragaman genetik. Informasi dari kajian ini dapat digunakan sebagai informasi dalam melakukan pemuliaan pada ikan ini sehingga depresi silang dalam (inbreeding depression) dapat dicegah. DNA mitokondria diwariskan secara maternal sehingga asal-usul tetua dapat diketahui.
Sampel ikan baung diambil dari 4 populasi, yaitu dari Cirata, Bogor, Serayu dan Jambi. Setiap populasi diambil 10 ikan secara acak, kemudian untuk ekstraksi DNA diambil sampel pada bagian sungut ikan sebanyak 5-10 mg. Ekstraksi DNA dilakukan dengan metode phenol chloroform (Nugroho 2001). Hasil ektraksi DNA kemudian di ampifikasi untuk memperbanyak daerah D-loop mtDNA dengan PCR. Hasil PCR kemudian dipotong dengan 4 enzim restriksi yaitu MboI, HaeIII, AluI dan RsaI secara terpisah. DNA hasil pemotongan dimigrasikan di gel agarose. Analisis data dilakukan dengan menggunakan software TFPGA (tool for population genetic analysis).
Pada penelitian ini, pemotongan dengan 4 enzim restriksi pada daerah D-loop mtDNA ikan baung dari 4 populasi menghasilkan 7 haplotipe komposit. Pemotongan daerah D-loop 4 populasi ikan baung dengan MboI, AluI dan RsaI menghasilkan pola pemotongan yang beragam. Sementara enzim restriksi HaeIII tidak menghasilkan keragaman pada daerah D-loop mtDNA 4 populasi ikan baung. Populasi ikan baung dari Jambi mempunyai nilai heterozigositas tertinggi yaitu sebesar 0,58, dan populasi Serayu mempunyai nilai heterozigositas terendah yang hanya menghasilkan 1 haplotipe komposit dengan nilai heterozigositas 0,00. Populasi ikan baung dari Cirata dan Bogor mempunyai pola pemotongan daerah D-loop mtDNA yang sama, yang menghasilkan 2 haplotipe komposit. Bila dilihat dari hasil penghitungan jarak genetik Wright, populasi Cirata mempunyai jarak yang dekat dengan populasi Bogor yaitu sebesar 0,1. Hasil uji Fst berpasangan menunjukkan bahwa populasi Cirata dan Bogor tidak berbeda secara signifikan dan berbeda dengan populasi Jambi dan Serayu.
© Hak cipta milik Institut Pertanian Bogor, tahun 2012
Hak cipta dilindungi undang-undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
ANALISIS KERAGAMAN GENETIK IKAN BAUNG
(
Hemibagrus nemurus
) DENGAN MENGGUNAKAN
TEKNIK PCR-RFLP PADA DNA MITOKONDRIA
NANDA PADRIA
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
HALAMAN PENGESAHAN
Judul : Analisis Keragaman Genetik Ikan Baung (Hemibagrus nemurus) dengan Menggunakan Teknik PCR-RFLP pada DNA Mitokondria
Nama : Nanda Padria
NIM : P051100151
Program Studi : Bioteknologi
Disetujui,
Komisi Pembimbing
Prof. Dr. Ir. Suharsono, DEA Dr. Ir. Estu Nugroho, M.Sc
Ketua Anggota
Diketahui,
Ketua Program Studi Dekan Sekolah Pascasarjana Bioteknologi
Prof. Dr. Ir. Suharsono, DEA Dr. Ir. Dahrul Syah, M.Sc. Agr
PRAKATA
Puji dan Syukur penulis ucapkan kehadirat Allah SWT karena berkat Rahmat dan Hidayah-Nya penulis dapat menyelesaikan penelitian dengan judul ” Analisis Keragaman Genetik Ikan Baung (Hemibagrus nemurus) dengan Menggunakan Teknik PCR-RFLP pada DNA Mitokondria”.
Ucapan terima kasih penulis sampaikan kepada:
1. Prof. Dr. Ir. Suharsono, DEA. sebagai ketua komisi pembimbing dan Dr. Ir. Estu Nugroho, M.Sc sebagai anggota komisi yang telah mencurahkan waktu dan perhatian untuk membimbing dan memotivasi penulis.
2. Dr. Ir. Agus Oman Sudrajat, M.Sc. sebagai dosen penguji luar komisi yang telah memberi masukan dan saran yang sangat berguna bagi kesempurnaan tesis ini.
3. Balai Riset Perikanan Budidaya Air Tawar (BRPBAT) Sempur-Bogor yang telah membiayai Penelitian ini, terutama kepada Bapak Ir. Jojo Subagya selaku penanggung jawab proyek penelitian ini yang telah mempercayakan penelitian ini kepada saya.
4. Mbak Luna dan Mbak Sri yang telah membantu saya selama melakukan penelitian ini.
5. Kedua orang tua saya Bapak Jasrul Padri dan Ibu Sahriati atas semua dukungan dan perhatian serta kasih sayang yang diberikan, adikku Dinda Juwita Padria. 6. Rekan-rekan seperjuangan S2 Bioteknologi 2010 dan rekan-rekan sesama
alumni Universitas Riau yang melanjutkan studi di IPB.
7. Segenap karyawan serta staff Pusat Pemuliaan Sumberdaya hayati dan Bioteknologi IPB yang telah membantu penyelesaian studi penulis.
Serta semua pihak yang tidak bisa penulis sebutkan satu persatu.
Bogor, September 2012
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 8 Agustus 1987 di Rokan Kecamatan Rokan IV Koto Kabupaten Rokan Hulu Provinsi Riau, merupakan anak pertama dari dua orang bersaudara dari pasangan Bapak Jasrul Padri dan Ibu Sahriati.
DAFTAR ISI
2.3 Pengukuran Keragaman Genetik ... 7
2.4 DNA Mitokondria ... 8
3.5 Pemotongan dengan Enzim Restriksi ... 12
3.6 Elektroforesis dan Visualisasi ... 12
3.7 Analisis Data ... 12
4 HASIL DAN PEMBAHASAN 4.1 Hasil ... 15
4.2 Pembahasan ... 21
4.2.1 Keragaman Genetik dan Keperluan dalam Budidaya ... 24
5 KESIMPULAN DAN SARAN 5.1 Kesimpulan ... 29
5.2 Saran ... 29
DAFTAR GAMBAR
Halaman
1 Hasil amplifikasi D-loop region mtDNA ... 15
2 Pola pemotongan D-loop region mtDNA dengan enzim restriksi MboI 18 3 Pola pemotongan D-loop region mtDNA dengan enzim restriksi
HaeIII ... 18 4 Pola pemotongan D-loop region mtDNA dengan enzim restriksi AluI .. 19 5 Dendogram (wright genetic distance)/ ketidakmiripan empat populasi
DAFTAR TABEL
Halaman
1. Pola potongan D-loop mtDNA dengan 4 enzim restriksi ... 16
2. Haplotipe Komposit pada 4 populasi ikan baung ... 17
3. Variasi genetik empat populasi ikan baung berdasarkan D-loop
mtDNA yang dipotong dengan 4 enzim restriksi yaitu MboI, HaeIII, AluI dan RsaI ... 19 4. Jarak genetik empat populasi ikan baung berdasarkan D-loop mtDNA
yang dipotong dengan 4 enzim restriksi yaitu MboI, HaeIII, AluI dan RsaI ... 20 5. Uji Fst berpasangan empat populasi ikan baung berdasarkan D-loop
mtDNA yang dipotong dengan 4 enzim restriksi yaitu MboI, HaeIII, AluI dan RsaI ... 21 6. Kondisi jumlah calon induk ikan baung pasca angkut sampai dengan
proses adaptasi di lingkungan kolam instalasi penelitian, Cibalagung .. 25
DAFTAR LAMPIRAN
Halaman
1. Lokasi penyamplingan populasi ikan baung (Hemibagrus nemurus) .. 37 2. Hasil analisis menggunakan software TFPGA untuk nilai variasi
genetik empat populasi ikan baung berdasarkan D-loop mtDNA yang
dipotong dengan 4 enzim restriksi yaitu MboI, HaeIII, AluI dan RsaI. 1). Cirata; 2). Serayu; 3). Jambi; 4). Bogor ... 38
3. Hasil analisis menggunakan software TFPGA untuk nilai jarak genetik
empat populasi ikan baung berdasarkan D-loop mtDNA yang
dipotong dengan 4 enzim restriksi yaitu MboI, HaeIII, AluI dan RsaI. 1). Cirata; 2). Serayu; 3). Jambi; 4). Bogor ... 43
4. Hasil analisis menggunakan software TFPGA untuk nilai uji Fst
berpasangan empat populasi ikan baung berdasarkan D-loop mtDNA
1
I.
PENDAHULUAN
1.1. Latar Belakang
Hemibagrus nemurus yang di Indonesia dikenal dengan nama ikan baung merupakan komoditi perikanan air tawar yang baru mendapat perhatian dalam
sektor budidaya.H. nemurus termasuk ke dalam ordo Siluriformes dan famili Bagridae (Nelson 2006).Daerah penyebaran ikan baung adalah di kawasan tropika
yang meliputi Afrika, Asia Tenggara dan Asia Timur (Kottelat et al. 1993).
Sebagai salah satu komoditi perikanan air tawar, saat ini budidaya ikan
baung masih belum seintensif komoditi budidaya ikan air tawar lainnya, karena
ketidakmampuan untuk berkembang biak di penangkaran (Muflikhah et al. 1993). Penyediaan benih ikan baungpun masih mengandalkan hasil tangkapan dari alam,
karena pemijahan buatan masih menghasilkan daya tetas telur ikan baung yang
masih rendah (Muflikhah et al. 1993; Sukendi 2005).Dengan demikian banyak kajian yang perlu dilakukan dalam upaya mencari informasi yang diperlukan
dalam pembudidayaan komoditi ini.
Kajian genetik merupakan salah satu usaha dalam mendapatkan informasi
yang diharapkan dapat meningkatkan efisiensi budidaya ikan baung ini.Salah
satunya adalah dengan mengkaji variasi genetik populasi ikan baung. Menurut
Dunham (2004), variasi genetik/genotipe penting untuk sintasan jangka panjang
suatu spesies atau populasi tersebut terutama untuk beradaptasi pada perubahan
lingkungan. Variasi genetik sangat penting dalam suatu populasi ikan untuk
mendapatkan varietas yang unggul.Keragaman genetik sangat berguna dalam
melakukan seleksi, baik seleksi alam maupun seleksi buatan yang dilakukan oleh
manusia.Keragaman genetik menjadi penting karena perbaikan mutu genetik
didasari oleh keragaman genetik yang dimiliki oleh suatu populasi (Rina 2001).
Penentuan variasi genetik pada ikan dapat dilakukan berdasarkan karakter
morfologi dan genotipenya. Variasi genotipe dapat dilakukan melalui pendekatan
molekuler dengan berbagai metode.Salah satunya adalah dengan teknik
PCR-RFLP dari mtDNA.Analisis mtDNA untuk mengetahui keragaman genetik pada
2 memperlihatkan adanya keragaman genetik berdasarkan mtDNA dari populasi
ikan.
Sedikitnya informasi mengenai keragaman genetik merupakan kendala
yang ditemui dalam usaha menghasilkan kualitas genetik yang unggul.Penurunan
kualitas genetik terkadang disebabkan oleh tingginya tingkat inbreeding (silang dalam).Untuk mencegah terjadinya inbreeding, kajian asal-usul yang berdasarkan pada kesamaan genetik dari suatu populasi harus diketahui.Untuk itu kajian
tentang keragaman genetik ikan baung merupakan kajian yang penting dalam
perbaikan genetik ikan ini.DNA mitokondria diwariskan melalui induk betina,
sehingga asal-usul tetua suatu individu dapat diketahui melalui DNA mitokondria.
Untuk menghindari terjadinya inbreeding dari populasi awal yang sama analisis DNA mitokondria sangat diperlukan.
Selain berguna untuk memberikan informasi dalam melakukan perbaikan
genetik terutama untuk mencegah terjadinya inbreeding.Penelitian ini juga diharapkan mampu memberikan data tentang keragaman genetik ikan baung yang
ada. Sebelumnya kajian keragaman genetik pada dareah mtDNAikan baung telah
dilakukan oleh Nugroho et al. (2005) pada populasi yang berasal dari Jambi, Wonogiri dan Jatiluhur dan Hany (2003) pada populasi yang berasal dari Cirata,
Wonogiri dan Jatiluhur yang menunjukkan adanya keragaman pada populasi
tersebut. Kajian keragaman genetik yang telah dilakukan ini sangat berguna
untuk melakukan konservasi sumberdaya genetik.Penelitian ini juga sangat
bermanfaat untuk perbaikan budidaya ikan baung.
1.2. Tujuan dan Manfaat
Tujuan dari penelitian ini adalah untuk mengetahui keragaman genetik
ikan baung dari beberapa populasi, sehingga data ini dapat dipergunakan sebagai
3
1.3. Hipotesis Penelitian
Hipotesis pada penelitian ini adalah ikan baung dari populasi Cirata,
5
II.
TINJAUAN PUSTAKA
2.1. Ikan Baung
Ikan baung (Hemibagrus nemurus) merupakan salah satu komoditas perairan umum yang mempunyai prospek untuk dibudidayakan.Jenis ikan ini
mudah dipelihara di kolam atau keramba jaring apung (KJA), dan dapat cepat
menyesuaikan diri terhadap pemberian pakan buatan (Hardjamulia dan Suhenda
2000).Distribusi ikan baung relatif luas di pulau Jawa, Sumatra dan Kalimantan
(Kottelat et al. 1993). Jenis ikan ini digemari masyarakat, dan mempunyai arti penting khususnya Sumatra, seperti Jambi (Nasution et al. 1993).
Untuk mengatasi keterbatasan penyediaan benih yang bermutu baik, maka
berbagai penelitian telah dilakukan, mulai dari sumber dayanya di alam (Alawi et al. 1992; Samuel et al. 1995; Sukendi 2000), pembenihannya (Gaffar dan Muflikhah 1992; Muflikhah et al. 1993; Hardjamulia dan Suhenda 2000; Sukendi 2000) sampai pada pembesarannya (Muflikhah dan Gaffar 1992; Muflikhah dan
Aida 1996).
Peluang pengembangan ikan baung semakin terbuka setelah berhasil
dilakukannya pemijahan secara buatan dengan teknik kawin rangsang (induce breeding) (Gaffar dan Muflikhah 1992). Namun sukses tersebut belum diimbangi dengan keberhasilan dalam produksi benih secara masal sehingga pasokan benih
untuk usaha budidaya skala komersil masih sangat terbatas (Sutrisno et al. 2005). Namun pemijahan buatan pada ikan baung masih menghasilkan daya tetas telur
ikan baung yang masih rendah (Muflikhah et al. 1993; Sukendi 2005).
Banyak kajian yang harus dilakukan untuk mendapatkan benih dengan
kualitas genetik yang unggul.Salah satunya dengan melakukan kajian keragaman
genetik pada ikan baung, untuk menyesuaikan penurunan performa yang
6
Variasi genetik merupakan hal yang penting dalam respon seleksi, dan
breedingpada populasi cukup besar dan secara genetik beragam. Karena itu, keragaman genetikmerupakan hal yang penting saat membuat dan menjalankan
program breeding.Desain breeding dengan strukturfamili yang tepat sangat penting untuk mempertahankan effective population size yang besar dan mendapatkan respon seleksi jangka panjang dengan tingkat inbreeding yang rendah (Olesen et al. 2003).
2.2. Keragaman Genetik
Luasnya perairan yang terbentuk dipermukaan bumi baik merupakan
perairan tawar, payau ataupun laut menjadi tempat berbagai macam biota baik
yang berukuran mikro ataupun makro.Salah satu jenis biota yang banyak terdapat
diperairan adalah ikan.Habitat ikan menempati perairan tawar yang teridentifikasi
berjumlah 1100 spesies, sedangkan habitat ikan yang menempati perairan laut
yang teridentifikasi berjumlah 8500 spesies (Kottelat et al. 1993).
Secara umum keragaman genetik menjelaskan (biodiversity) yang meliputi keragaman struktural maupun fungsional dari kehidupan komunitas dengan
ekosistemnya, populasi dan spesies (Soewardi2007).Keragaman hayati, memiliki
peranan penting untuk keseimbangan ekosistem, demikian pula keragaman
genetik.Semakin tinggi keragaman genetik, maka semakin tinggi populasi yang
mampu bertahan hidupdalam jangka waktu yang lebih lama, karena semakin
tinggi daya adaptasi populasi terhadap perubahan lingkungan.Dengan demkian,
keragaman genetik merupakan kunci penting dalam memelihara keberlanjutan dan
kinerja produksi dari suatu spesies (Slamat 2009).
Struktur genetika dan tingkat keanekaragaman suatu populasi dipengaruhi
oleh mekanisme genetik secara internal maupun eksternal. Faktor yang dapat
menyebabkan peningkatan keragaman genetik antara lain adalah faktor mutasi dan
migrasi. Sedangkan faktor-faktor yang menurunkan keragaman genetik
diantaranya adalah seleksi alam, silang dalam (inbreeding) serta penghanyutan gen (genetic drift) (Gardner et al. 1991).
Depresi inbreeding mungkin dihasilkan dari peningkatan homozigositas
7
2008).Peningkatan homozigositas menimbulkan ekspresi sejumlah alel yang
bersifat merugikan pada individu inbred, sehingga menurunkan kebugarannya. Pengurangan heterozigositas mengurangi kebugaran individu inbred pada lokus
dimana heterozigot mempunyai keuntungan dibandingkan dengan homozigot
(Allendorf dan Luikart 2008).
2.3. Pengukuran Keragaman Genetik
Keragaman genetik dapat diukur dan dikuantifikasikan pada beberapa
tingkat.Pertama, perbedaan ukuran fragmen DNA.Kedua, perbedaan urutan DNA
di dalam DNA, yang menyebabkan perbedaan protein.Kadang-kadang perbedaan
fenotipe dari variasi produk genetik ditentukan oleh satu atau dua lokus
(Beaumont dan Haore 2003).
Berbagai macam metode dapat digunakan untuk mendeteksi adanya
polimorfisme molekuler, tetapi yang paling banyak digunakan saat ini adalah
metode elektroforesis.Penanda DNA memiliki keuntungan yang lebih dari pada
menggunakan penanda morfometrik, meristik ataupun protein (Allozyme) untuk mengkaji struktur stok dan variasi genetik ikan (Carvalho danPitcher1994).
Polimorfisme yang dideteksi berdasarkan elektroforesis DNA memiliki beberapa
kelebihan dibandingkan dengan karakter kuantitatif dan jumlah kromosom karena
alasan berikut:
1. Sangat sedikit atau tidak ada sama sekali pengaruh lingkungan dan
memungkinkan membandingkan secara langsung sampel yang dikoleksi
dari lapangan.
2. Di bawah kontrol dari beberapa loci dan dapat secara mudah
diinterpretasikan dalam frekuensi genotipe maupun alel. Dari frekuensi ini
kemudian dapat dihitung tingkat heterozigositas (keragaman dalam
populasi) dan jarak genetik (keragaman genetik antar populasi (Soewardi
2007).
Banyak metode analisis keragaman genetik yang menggunakan teknik
elektroforesis dalam visualisasi hasilnya, misalnya, elektroforesis protein atau
8
length polymorphism), AFLP (Amplified fragment length polymorphism) dan mikrosatelit.
Deteksi keragaman DNA mitokondria sangat umum menggunakan
polymerase chain reaction (PCR), yang diikuti dengan analisis enzim restriksi atau sekuensing secara langsung produk PCR (Allendorf dan Luikart
2008).Dengan teknik RFLP, potongan DNA dapat digunakan sebagai penanda
genetik tanpa harus melakukan pengurutan DNA.Jumlah copy yang tinggi dari
fragmen DNA yang dihasilkan dari isolasi DNA tertentu atau dari hasil PCR dapat
dipotong dengan sejumlah enzim restriksi endonuklease sehingga menghasilkan
potongan-potongan DNA yang ukurannya bervariasi. Cara ini merupakan cara
dalam mengidentifkasi variasi genetik yang disebabkan oleh mutasi (Beaumont
dan Haore 2003). Keragaman situs restriksi biasanya dihasilkan dari pergantian
nukleotida pada situs restriksi. Oleh karena itu keragaman situs restriksi dapat
dideteksi dengan menggunakan RFLP dan elektroforesis gel (Allendrof dan
Luikart 2008).
Teknik PCR-RFLP telah banyak digunakan dalam kajian keragaman
genetik pada ikan.Berikut beberapa penelitian analisis keragaman genetik yang
menggunakan teknik PCR-RFLP: Hasil penelitian Imsiridou et al. (1997) pada greek chub (Leuciscus cephalus),Rina (2001) pada ikan Pangasius, Arifin dan Kurniasih (2007) pada ikan nila (Oreochromis niloticus), Slamat (2009) pada ikan betok (Anabas testudineus),Mulyasari et al. (2010) pada ikan kelabau (Osteochilus kelabau),telah memperlihatkan adanya keragaman genetik berdasarkan pemotongan daerah mitokondria dengan beberapa enzim restriksi.
2.4. DNA Mitokondria
DNA mitokondria berbeda dengan DNA inti walaupun keduanya berada
dalam satu sel.Pewarisan DNA mitokondria kepada generasi berikutnya terjadi
secara maternal dan tidak ada rekombinasi. Pada DNA ini terjadi laju mutasi
yang relatif tinggi.Karena itu, DNA mitokondria dapat dimanfaatkan untuk
menentukan keragaman genetik antar individu dalam satu populasi, hubungan
9
DNA mitokondria umumnya berbentuk sirkular (siklik) rantai ganda dan
merupakan molekul superkoil kecuali DNA mitokondria di dalam Tetrahymena dan Paramecium yang berbentuk linier.Ukuran DNA mitokondria bervariasi antar spesies tetapi tetapsama antara mitokondria di dalam spesies tertentu. Semua
DNA mitokondria tanpa memandang ukurannya, mempunyai komplemen gen
yang sama, walaupun ada sedikit variasi. Perbedaan ukuran diantaraspesies
disebabkan oleh banyaknya DNA pemisah diantara gen-gen mitokondria (Sumadi
dan Marianti 2007).Jumlah mitokondria tiap-tiap sel sangat beragam, ada sel yang
tidak mempunyai mitokondria, tetapi ada pula yang mempunyai mitokondria
banyak sekali bahkan sampai ratusan ribu.Spermatozoa dan Flagellata seperti Chromulina hanya mempunyai satu mitokondria pada tiap-tiap selnya.Sel-sel hati rata-rata mempunyai 800 mitokondria tiap selnya, sedangkan landak laut dan
Amoeba raksasa Chaos-chaos mempunyai hampir 500.000 mitokondria.
DNA mitokondria mempunyai peranan yang penting dalam menyandi
beberapa tRNA, rRNA dan protein yang digunakan dalam sintesis protein di
mitokondria (Griffiths et al. 2000).Ukuran mtDNA kurang dari 20 kb dan diwariskan secara maternal.mtDNA mempunyai gen yang menyandi enzim dan
protein dalam respirasi seperti: cytochrome b, NADH 1-7, NADH-41, cytochrome
c oxidase (COX-1-3), ATP 6;8;9, 12s RNA, 16s RNA, dan tRNA (Lemire 2005).
Perbedaan antara gen DNA mitokondria dan gen yang terdapat pada inti
sel mencakupi 2 faktor. Pertama, DNA mitokondria diwariskan secara maternal,
dan tidak mengikuti hukum Mandel.Kedua, DNA mitokondria adalah haploid dan
mempunyai laju mutasi 5-10 kali lebih cepat dari pada gen pada inti sel.Rata-rata
laju keragaman DNA mitokondria pada vertebrata adalah 1-2% per juta tahun
(Randi 2000).Mutasi pada DNA mitokondria disebabkan faktor lingkungan atau
mutasi pada gen inti sel yang berdampak pada DNA mitokondria (Lemire 2005).
Menurut Dunham (2004), analisis variasi genetik DNA mitokondria
merupakan alternatif untuk mempelajari genetika populasi ikan, sebab laju mutasi
DNA mitokondria lebih cepat dibandingkan dengan DNA genom. Daerah control
region yang hipervariabel, memungkinkan penelusuran evolusi yang cepat.Hasil
analisisini sering kali dipergunakan untuk mengungkapkan keragaman genetik
11
III.
METODOLOGI PENELITIAN
3.1. Waktu dan Tempat
Penelitian ini dilakukan pada bulan Oktober 2011 sampai dengan bulan
April 2012, di Laboratorium Genetik Balai Riset Budidaya Air Tawar Bogor.
3.2. Bahan Penelitian
Bahan yang digunakan dalam penelitian ini adalah ikan Baung yang
merupakan hasil penyamplingan dari beberapa populasi yaitu, Cirata (Jawa Barat)
yang disampling dari Waduk Cirata, Bogor (Jawa Barat) yang disampling dari
sungai Cisadane, Serayu (Jawa Tengah) yang disampling dari sungai Serayu dan
Jambi yang merupakan hasil domestikasi BBAT Jambi. Masing-masing populasi
diambil 10 ekor ikan dengan bobot tubuh 500-700 gram.Lokasi pengambilan
sampel ikan baung disajikan pada Lampiran 1. Primer forward LH 1509 (5’-
DNA ikan diekstraksi dari potongan sungut ikan dengan menggunakan
metode phenol chloroform (Nugroho 2001). Potongan sungut sebanyak 5-10 mg
dimasukkan dalam mikrotube 1,5 ml yang telah diisi dengan 500 ml TNES Urea
(10 mM Tris-HCL pH 7,5, 125 mM NaCl, 10 mM EDTA pH 7,5, 0,5% SDS dan
4 mM Urea), kemudian ditambah 10 µl/ml proteinase K. Setelah itu tabung
divortex selama 1 menit kemudian sampel diinkubasi selama 24 jam pada suhu
370C. Selanjutnya larutan phenol chloroform ditambahkan pada campuran
tersebut sebanyak 1000 µl dan divotex selama 1 menit. Tabung yang berisi
campuran kemudian disentrifuse dengan kecepatan 10.000 rpm selama 10 menit.
12
dengan 1000 µl ethanol 90% dan 10 µl CH3COONa. Sampel lalu divortex selama
1 menit.Kemudian disentrifuse dengan kecepatan 10.000 rpm selama 10 menit,
lalu supernatannya dibuang dan dikeringkan pada suhu kamar. Endapan
dilarutkan di dalam 100 µl TE (Tris-HCL 1M pH7,5, EDTA 500 mM pH 8) dan
disimpan pada suhu 40C sebelum digunakan pada tahap selanjutnya.
3.4. Amplifikasi DNA
Amplifikasi daerah D-loop mtDNA menggunakan primer forward LH
1509 dan primer reverse FH 1202. Komposisi bahan untuk amplifikasi DNA
adalah 3 µl DNA, primer forward 10 µM, primer reverse 10µM, dan 18 µl H2O
yang dicampurkan pada 1 unit dry taq (GE Healthcare) sehingga volume totalnya
25 µl. PCR dilakukan sebanyak 35 siklus.
Untuk proses PCR, denaturasi dilakukan pada suhu 940C selama 2 menit,
penempelan primer pada suhu 500C selama 1 menit, dan ekstensi pada suhu 720C
selama 2 menit.
3.5. Pemotongan dengan Enzim Restriksi
Hasil amplifikasi DNA dengan PCR dipotong menggunakan 4 jenis enzim
restriksi secara terpisah, yaitu: MboI, HaeIII,AluI dan RsaI. Pemotongan mtDNA dilakukan dengan mencampurkan 5 U enzim restriksi, 1 µl buffer enzim, 3 µl
DNA hasil PCR dan 5 µl H2O, kemudian diinkubasi pada suhu 370C selama 24
jam.
3.6. Elektroforesis dan Visualisasi
Elektroforesis hasil PCR dan hasil pemotongan DNA dengan enzim
restriksi dilakukan dalam gel agarose 1-2% dengan 1 X buffer TBE (89 mM tris,
89 mM boric acid, 2 mM EDTA) dan ditambahkan 0,5 µg/ml ethidium bromide
dan diamati dibawah UV transluminator.
3.7. Analisis Data
Pola pita dari hasil pemotongan DNA dari setiap individu dari setiap
13
pemotongan enzim restriksi).Hasil pengelompokan haplotipe tersebut diolah
menggunakan software TFPGA (Tools for PopulationGenetic Analysis).
Adapun nilai-nilai yang diamati untuk melihat perbedaan antar populasi
dari hasil pemotongan dengan 4 enzim restriksi adalah:
1. Frekuensi haplotipe dan heterozigositas populasi
Frekuensi alel=
ni= frekuensi alel ke i
N= total sampel pada satu populasi
Heterozigositas: He = 1- ∑ pi= frekuensi alel ke-i
2. Analisa Jarak genetik (Genetic distance)
Jai = ∑ ai2
Ij = identitas genetik yang dinormalkan untuk lokus ke-j
3. Uji Fst berpasangan (Indeks fiksasi)
Fst = –
Dimana: HT = total heterozigositas dalam populasi total
15
IV.
HASIL DAN PEMBAHASAN
4.1. Hasil
Hasil amplifikasi PCR menunjukkan bahwa daerah D- loop mtDNA ikan
baung memiliki panjang sekitar 1800 pasang basa (Gambar 1). Hal ini sesuai
dengan penelitian Nugroho et al. (2005) yang menyebutkan bahwa daerah D-loop mtDNA ikan baung mempunyai panjang sekitar 1800-2000 pasang basa (pb).
Estimasi ukuran (pasang basa) hasil pemotongan daerah D-loop mtDNA
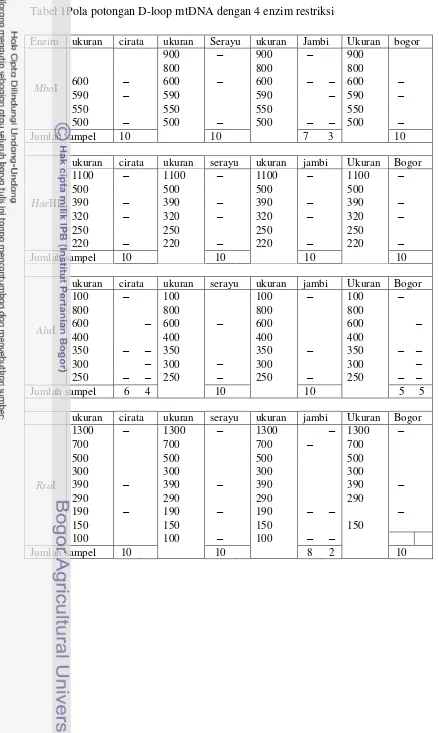
ikan baung dengan menggunakan 4 enzim restriksi tercantum pada Tabel 1.Hasil
pemotongan daerah D-loop mtDNA ikan baung dengan 4 enzim restriksi
dikelompokkan berdasarkan pola pemotongan yang dihasilkan yang disajikan
pada Tabel 2. Contoh hasil pemotongan daerah D-loop mtDNA ikan baung
dengan 4 enzim restriksi tercantum pada Gambar 2 sampai dengan 4.
Gambar 1 Hasil Amplifikasi D-loop region mtDNA CIRATA SARAYU JAMBI BOGOR M
16
Tabel 1Pola potongan D-loop mtDNA dengan 4 enzim restriksi
17
Secara umum, hasil pemotongan dengan 4 enzim restriksi menghasilkan 7
kelompok haplotipe yang disebut haplotipe komposit, seperti yang tertera pada
Tabel 2.
Tabel 2 Haplotipe komposit pada 4 populasi ikan baung.
Enzim
Pemotongan dengan enzim restriksi MboI menghasilkan 2 jenis pemotongan, pola A dengan estimasi ukuran potongan adalah 650, 590 dan 500
pasang basa (pb)dan pola B dengan estimasi ukuran potongan adalah 900, 650 dan
18
monomorfik pada setiap populasi, dengan estimasi ukuran potongannya adalah
1100, 390, 320 dan 220 (pb). Pemotongan dengan enzim restriksi AluI menghasilkan 2 pola pemotongan dengan estimasi ukuran potongan untuk pola A
adalah 1000, 350 dan 250 (pb) dan untuk pola B adalah 600, 350, 300 dan 250
(pb).
Pemotongan dengan enzim restriksi RsaI menghasilkan 3 pola pemotongan (Tabel 2). Estimasi ukuran potongan untuk pola A adalah 1300,
300, dan 190 (pb), estimasi ukuran untuk pola B adalah 1300, 300 dan 100 (pb)
dan estimasi ukuran untuk pola C adalah 1300, 290 dan 100 (pb).
Gambar 2Pola pemotongan D-loop region mtDNA dengan enzim restriksiMboI
Gambar 3Pola pemotongan D-loop region mtDNA dengan enzim restriksiHaeIII CIRATA JAMBI SERAYU BOGOR M
CIRATA JAMBI SERAYU BOGOR M
900 600 550 500
1100
19
Gambar 4.Pola pemotongan D-loop region mtDNA dengan enzim restriksi AluI Uji descriptive dengan program TFPGA menunjukkan bahwa ikan baung
yang berasal dari populasi Jambi mempunyai nilai heterozigositas yang paling
tinggi yaitu 0,5800 diikuti oleh ikan baung dari populasi Bogor dengan nilai
heterozigositas 0,5000, populasi ikan baung dari Cirata 0,4800. Dan ikan baung
dari populasi Serayu mempunyai nilai heterozigositas yang paling rendah yaitu
0,0000 (Tabel 3). Penghitungan nilai heterozigositas dengan software TFPGA
disajikan pada Lampiran 2.
Tabel 3 Variasi genetikempat populasi ikan baung berdasarkan D-loop mtDNA yang dipotong dengan 4 enzim yaitu MboI, HaeIII AluI dan RsaI.
Tingginya nilai heterozigositas pada populasi ikan baung dapat dilihat juga
20
dalam situs pemotongan enzim restriksi.Keragaman ini mengindikasikan bahwa
ikan tersebut mempunyai tetua yang beragam. Ikan baung dari populasi jambi
mempunyai 4 haplotipe komposit yaitu BAAC, AAAC, AAAB dan BAAB, dan
ikan baung dari populasi Bogor dan Cirata yang mempunyai 2 haplotipekomposit
yaitu AAAA dan AABA. Sedangkan ikan baung yang berasal dari populasi
Serayu hanya mempunyai satu haplotipe komposite yaitu tipe BABB.
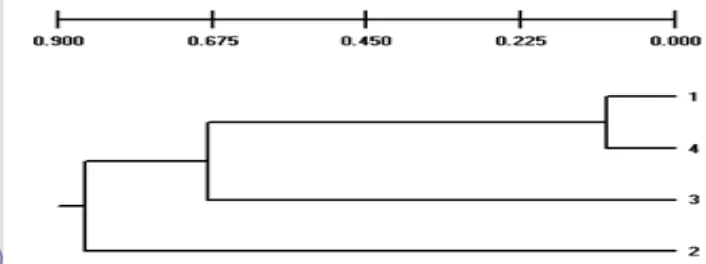
Jarak genetik atau ketidakmiripan antara empat populasi ikan baung
disajikan pada Tabel 4. Ikan baung yang berasal dari populasi Cirata mempunyai
nilai jarak genetik atau ketidakmiripan yang dekat dengan populasi Bogor yaitu
sebesar 0,100. Penghitungan nilai Jarak genetik dengan menggunakan software
TFPGA disajikan pada Lampiran 3.Berdasarkan penghitungan jarak genetik
tersebut, hubungan keempat populasi ikan baung disajikan dengan dendogram
(Gambar 5).
Tabel 4 Jarak genetikempatpopulasi ikan baung berdasarkan D-loop mtDNA yang dipotong dengan 4 enzim yaitu MboI, HaeIII AluI dan RsaI.
Populasi Cirata Serayu Jambi Bogor
Cirata 0.8718 0.6856 0.1000
baung yang berasal dari Cirata, dan terpisah dari populasi ikan baung yang berasal
dari Jambi dan Serayu. Dan ikan baung dari populasi Jambi dan Serayu juga
21
Gambar 5Dendogram (wright genetic distance)/ ketidakmiripan genetik empat populasi ikan baung berdasarkan D-loop mtDNA yang dipotong dengan 4 enzim yaitu MboI, HaeIII AluI dan RsaI. 1). Cirata; 2).
Penghitungan nilai uji Fst berpasangan menggunakan software TFPGA disajikan
pada Lampiran 4.
Tabel 5 Uji Fst berpasangan 4 populasi ikan baung berdsarkan mtDNA yang direstriksi dengan 4 enzim yaitu MboI, HaeIII AluI dan RsaI.
Populasi Cirata Serayu Jambi Bogor
Cirata 0.0000 * 0.0000* 0.7535 menghasilkan pola pemotongan, yakni 3 pola, kemudian diikuti oleh enzim
22
pemotongan mengindikasikan bahwa urutan nukleotida pada daerah D- loop
mtDNA pada ikan baung dari empat populasi adalah beragam.
Pemotongan dengan enzim restriksi HaeIII yang hanya menghasilkan 1 pola pemotongan pada 4 populasi ikan baung, menunjukkan bahwa empat
populasi ikan baung tersebut tidak mempunyai keragaman pada situs pengenalan
enzim restriksi HaeIII. Nugroho et al. (2005) telah menggunakan enzim restriksi HaeIII untuk melihat keragaman genetik pada ikan baung dari populasi Jambi, Wonogiri dan Jatiluhur yang juga menghasilkan 1 pola pemotongan.Hal ini
menunjukkan bahwa dari 6 populasi ikan baung yaitu ikan baung dari populasi
Cirata, Serayu, Jambi, Bogor, Wonogiri dan Jatiluhur tidak mempunyai
keragaman di daerah D-loop pada situs pengenalan enzim restriksi HaeIII.
Pada penelitian ini, pemotongan daerah D-loop mtDNA ikan baung dari 4
populasi (Cirata, Serayu, Jambi dan Bogor) dengan 4 enzim restriksi
menghasilkan 7 jenis haplotipekomposit. Hal ini menunjukkan adanya keragaman
antar populasi tersebut. Dan bila melihat hasil heterozigositas pada tiap-tiap
populasi, ikan baung yang berasal dari Jambi merupakan populasi yang
mempunyai nilai heterozigositas paling tinggi yaitu 0,5800, yang telah mengalami
peningkatan dibandingkan dengan hasil pengamatan Nugroho et al. (2005) pada populasi ini yang memperoleh nilai diversitas haplotipe sebesar 0,111 dan hanya
dua jenis haplotipekomposit. Peningkatan keragaman genetik ini, karena populasi
yang berasal dari BBAT (Balai Budidaya Air Tawar) Jambi, didugamengalami
penambahan populasi dari luar dalam rangka untuk program seleksi.Penelitian
Leesa-Nga et al. (2000) juga memperlihatkan bahwa heterozigositas tertinggi juga ditemukan pada populasi ikan baung yang berasal dari pembenihan (hatchery) (0,111 ± 0,036) bila dibandingkan dengan populasi liar yang berasal dari
Sukhothai (0,041 ± 0,023).
Populasi Serayu yang hanya mempunyai 1 haplotipe dengan diversitas
genetik sebesar 0,000, menunjukkan keragaman populasi ini sangat rendah.
Rendahnya keragaman genetik kemungkinan disebabkan oleh penurunan
keragaman genetik walaupun tidak ada data dari penelitian lain yang dapat
dijadikan tolok ukur untuk menyatakan penurunan keragaman genetik ikan baung
23
Untuk populasi Cirata dapat dikatakan telah terjadi penurunan keragaman
genetik karena pada penelitian sebelumnya yang dilakukan oleh Hanny (2003)
populasi ini mempunyai nilai diversitas haplotipe sebesar 0,8000 dengan 5 jenis
haplotipekomposit, sedangkan pada penelitian ini hanya menghasilkan 2 haplotipe
komposit dengan diversitas haplotipe sebesar 0,4800.
Bila dilihat dari data pemotongan dengan 4 enzim restriksi pada populasi
ikan baung yang berasal dari populasi Bogor dan Cirata, terdapat kemiripan dalam
pola pemotongan. Setelah uji Fst berpasangan ternyata tidak ada perbedaan yang
nyata antara populasi Bogor dan populasi Cirata, dan bila dilihat data jarak
genetik dan dendogramnya terlihat bahwa populasi Bogor dan Cirata berada
dalam satu cluster dengan jarak genetik 0,1000. Bila dilihat dari kondisi
geografisnya sumber populasi Bogor dan Cirata memang berdekatan, tetapi
merupakan dua populasi yang terpisah.Populasi Bogor berasal dari sungai
Cisadane dan populasi Cirata berasal dari waduk Cirata.Jadi tidak bisa
disimpulkan bahwa terjadi migrasi atau geneflowsecara alami yang dapat membuat kekerabatan kedua populasi tersebut dekat secara genetik. Sebagaimana
yang dikatakan oleh Simmon et al. (2006), gene flow atau migrasi merupakan suatu proses perpindahan gen antar populasi. Jika ada dua populasi dengan
struktur genetik yang berbeda dan kemudian terjadi perpindahan gen antar
populasi dalam kurun waktu tertentu, maka akan menjadi mirip atau serupa secara
genetik.
Dugaan yang lain yang bisa digunakan untuk menjawab fenomena ini,
adalah kedua populasi ini memang berasal dari satu tetua. Kesamaan tetua ini
dapat disebabkan oleh perpindahan individu atau populasi dari satu habitat ke
habitat lainnya yang dilakukan oleh manusia, atau kedua populasi tersebut
merupakan hasil introduksi dari populasi yang sama. Kesamaan yang tinggi juga
ditemukan Nugroho et al. (2005) pada populasi ikan baung yang berasal dari populasi Wonogiri dan Jatiluhur, dimana kedua populasi tersebut mempunyai
24
4.2.1. Keragaman Genetik dan Keperluan dalam Budidaya
Keragaman genetik yang diamati pada 4 populasi ikan baung ini tergolong
rendah bila dibandingkan beberapa spesies air tawar lainnya, misalnya ikan patin
(Pangasius) berkisar antara 0,568-0,934 (Rina 2001), ikan nila (Oreocrhromis niloticus) yang mempunyai keragaman haplotipe berkisar antara 0,533-0,7579 (Arifin dan Kurniasih 2007),ikan betok (Anbas testudineus) yang berkisar antara 0,6-0,9384 (Slamat 2009). Tetapi keragaman genetik ikan baung lebih tinggi dari
pada beberapa spesies air tawar lainnya sepertiikan kancra (Tor soro) yang mempunyai keragaman haplotipe 0 (Nugroho et al. 2006), dan ikan kelabau (Osteochilus kelabau) yang berkisar antara 0-0,189 (Mulyasari et al. 2010). Keragaman genetik ikan baung pada penelitian ini relatif lebih tinggi bila
dibandingkan dengan hasil penelitian Nugrohoet al. (2005) pada 3 populasi ikan baung yang berasal dari populasi Jambi, Wonogiri dan Jatiluhur yang mempunyai
keragaman haplotipe secara berturut 0,111, 0,508 dan 0,173. Keragaman genetik
ikan air tawar memang lebih rendah bila dibandingkan dengan dengan ikan air
laut yang mempunyai jumlah haplotipe berkisar antara 6-17 dengan nilai
keragaman haplotipe 0,6-0,9 (Nugroho 2002). Penyebab rendahnya tingkat
variasi genetik pada ikan air tawar karena ikan air tawar mempunyai tingkat
migrasi yang lebih rendah sehingga peluang adanya persilangan dengan jenis ras
lainnya semakin kecil pula (Arifin dan Kurniasih 2007).
Informasi nilai keragaman genetik ini akan sangat berguna dalam
melakukan perbaikan budidaya pada ikan baung. Dari informasi keragaman
genetik yang ada, pemilihan strategi perbaikan kualitas genetik ikan baung dapat
dilakukan.Misalnya,untuk mencegah depresi silang dalam, makaikan baung dari
populasi Serayu yang mempunyai keragaman genetik yang rendah tidak
dikawinkan sesamanya. Begitu juga dengan populasi ikan baung yang lain.
Adanya keragaman genetik memungkinkan teknik hibridisasi atau teknik seleksi
untuk memperbaiki kualitas genetik ikan baung dapat dilakukan.Chambers (1983)
menyatakan bahwa seleksi dapat mengubah konstribusi frekuensi gen yang
ditentukan oleh peluang satu atau lebih genotipe yang divariasikan pada generasi
25
Perubahan genetis pada sifat-sifat yang dihasilkan program breeding selektif bisa dimanfaatkan untuk: 1). peningkatan produksi dan pendapatan; 2)
mengurangi waktu dan biaya produksi; 3) meningkatkan efisiensi sumberdaya
faktor-faktor produksi; 4) meningkatkan standar etika dalam produksi 5).
mengurangi penggunaan obat-obatan dan bahan kimia untuk menangani penyakit
dan parasit; dan 6). meningkatkan kualitas produk dan nilai ekonomis dan nilai
non ekonomis produk (Olese et al. 2003).
Daya adaptasi ikan baung tergantung dari asal populasi ikan tersebut.
Keragaan ikan baung yang berasal dari 4 populasi yaitu ikan baung berasal dari
populasi Cirata (waduk Cirata), Serayu (sungai Serayu), Jambi (BBAT Jambi) dan
Bogor (sungai Cisadane) yang diperolah dari hasil penelitian Subagya et al. (2011) disajikan pada Tabel 6. Kelangsungan hidup (survival rate) ikan baung di kolam Instalasi Penelitian Cibalagung tergantung dari populasi asal.
Tabel 6Kondisi jumlah calon induk ikan baung pasca angkut sampai dengan proses adaptasi di lingkungan kolam Instalasi Penelitian, Cibalagung.
Waktu pengamatan,SR
Dari data diatas dapat dilihat sampai bulan ke-3 masa adaptasi ikan baung
yang berasal dari populasi Bogor memiliki nilai adaptasi yang tertinggi yaitu
92,50% diikuti oleh populasi Cirata (90,97%), Serayu (81,25%) Cisadane
(80,00%) dan populasi Jambi mempunyai nilai kelangsungan hidup terendah yaitu
26
Setelah induk diaklimatisasi di kolam instalasi penelitian Cibalagung,
kemudian dilanjutkan dengan melihat performa bioreproduksi ikan baung dari 4
populasi, yaitu populasi Cirata, Serayu, Jambi dan Bogor. Derajat pembuahan
dan daya tetas induk ikan baung dari 4 populasi disajikan pada Tabel 7.
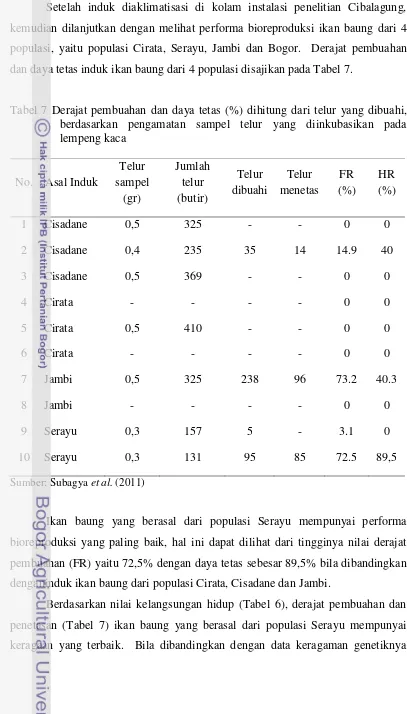
Tabel 7 Derajat pembuahan dan daya tetas (%) dihitung dari telur yang dibuahi, berdasarkan pengamatan sampel telur yang diinkubasikan pada lempeng kaca
Ikan baung yang berasal dari populasi Serayu mempunyai performa
bioreproduksi yang paling baik, hal ini dapat dilihat dari tingginya nilai derajat
pembuahan (FR) yaitu 72,5% dengan daya tetas sebesar 89,5% bila dibandingkan
denganinduk ikan baung dari populasi Cirata, Cisadane dan Jambi.
Berdasarkan nilai kelangsungan hidup (Tabel 6), derajat pembuahan dan
penetasan (Tabel 7) ikan baung yang berasal dari populasi Serayu mempunyai
27
yang rendah berdasarkan hasil pemotongan dengan 4 enzim restriksi pada daerah
D-loop mtDNA yang mengindikasikan bahwa pada populasi Serayu terjadi
tingkat inbreeding yang tinggi yang mengakibatkan nilai heterozigositasnya yang rendah. Pada populasi Serayu ini inbreeding yang tinggi tidak menyebabkan depresi inbreeding yang berakibat pada menurunnya kualitas keragaan bioreproduksi.Hal ini berbeda dengan hasil penelitian Fromen et al.(2007) yang menunjukkanbahwa penurunan nilai tingkat kelulushidupan, daya tetas telur dan
laju pembuahan pada ikan threespine stickleback akibat dari depresi
inbreeding.Untuk sifat-sifat yang lain seperti ketahanan terhadap penyakit dan cekaman lingkungan, laju pertumbuhan dan tingkat kelulushidupan pada ikan
baung yang berasal dari populasi Serayu ini belum ada data. Untuk itu tingkat
29
V.
KESIMPULAN DAN SARAN
5.1. Kesimpulan
Berdasarkan analisis PCR-RFLP dari daerah D-loop mtDNA 4 populasi
ikan baung dari Cirata, Serayu, Jambi dan Bogor mempunyai keragaman genetik.
Dari 4 jenis enzim restriksi yang digunakan untuk memotong daerah D-loop
mtDNAkeempat populasi tersebut mempunyai7 haplotipekomposit. Populasi
ikan baung yang berasal dari Jambi mempunyainilai heterozigositas tertinggi
dengan 4 komposit haplotipe dan populasi ikan baung yang berasal dari Serayu
mempunyai nilai heterozigositas terendah yaitu hanya mempunyai1
haplotipekomposit.Populasi ikan baung yang berasal dari Cirata dan Bogor
menunjukkan bahwa kedua populasi tersebut tidak bebeda secara signifikan.
5.2. Saran
Untuk memastikan keragaman genetik mtDNA yang rendah pada populasi
Serayu, maka perlu dilakukan analisis keragaman mtDNA dengan melakukan
31
DAFTAR PUSTAKA
Alawi HM, Ahmad, Rusliadi, Pardiman. 1992. Some biological aspect of micrones catfish (Macrones nemurus C.V) from Kampar River. Terubuk XVII 52:32-47.
Allendorf FW, Luikart G. 2008. Conservation and the Genetic of Populations. Blacwell Publishing.
Arifin OZ, Kurniasih T. 2007. Variasi genetik tiga populasi ikan nila (Oreochromis niloticus) berdasarkan polimorfisme mtDNA. J. Riset Akuakultur 2 (1):67-75.
Baker AJ, Birt P. 2000. Polymerase Chain Reaction Molecular Methods In Ecology. Blacwell Science Ltd. Oxford.
Beaumont AR, Hoare K. 2003. Biotechnology and Genetics in Fisheries and Aquaculture. Blacwell Publishing.
Carvalho GR, Pitcher TJ. 1994. Molecular Genetics and Fisheries. Chapman and Hall. London.
Chambers SM. 1983. Genetic Principle for Manager.Bringlame. New York. Dunham RA. 2004. Aquaculture and Fisheries Biotechnology: Genetic
Approaches. UK Cabi Publishing.
Frommen JG, Luz C, Mazzi D, Bakker TCM. 2007. Inbreeeding depression affects fertilization success and survival but not breeding coleration in threespine stickleback. Behavior145 (425-441). Also available online-www.brill.nl/beh [12 Mei 2012].
Gaffar AK, Muflikhah N. 1992. Pemijahan Buatan dan Pemeliharaan Larva Ikan Baung.Prosiding Seminar Hasil Penelitian Perikanan Air Tawar 1991/1992. Balitkanwar Bogor.
Gardner EJ, Simmon MJ, Snustad PD. 1991. Population and Evolutionary Genetics. Chichester Brisbane, New York.
Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, Gelbart WM. 2000. An Introduction Genetic Analysis. Seventh Edition. W. H. Freeman. New York.
32
Hardjamulia A, Suhenda N. 2000. Evaluasi sifat reproduksi dan sifat gelondong generasi pertama empat strain ikan baung (Mystsu nemurus) di Jaring apung. JPPI6 (3-4):24-35.
Imsiridou A . 1997. Genetic differentiation and phylogenetic relationship among greek chub Leuciscus cephalus L. (Pisces, Cyprinidae) population as revealed by RFLP analysis of mitochondrial DNA. Bioch Syst Ecol. 26:415-429.
Kottelat M, Whittenm AJ, Kartikasari SN, Wirjoatmodjo S. 1993. Freshwater Fishes of Western Indonesia and Sulawesi: Ikan Air Tawar Indonesia bagian barat dan Sulawesi. Jakarta: Periplus Edition (HK) ltd.
Lemire B. 2005. Mitochondrial genetics. Wormbook, ed. The C. elegans Research Community, Wormbook, doi/10.1895/wormbook.1.25.1, http://wormbook.org/pdf [12Mei 2012].
Leesa-Nga SN et al. 2000. Biochemical polymorphism in yellow chatfish, Mystus nemurus (C&V), from Thailand. Bioch genet 38:77-85.
Muflikhah N, Yosmaniar, Zahri M. 1993. Pematangan gonad dan pemijahan buatan ikan baung (Mystus nemurus). Prosiding Seminar Hasil Penelitian Perikanan Air Tawar 1992/1993. Balitkanwar, Puslitbangkan, Badan Litbang Pertanian. hlm243-247.
Muflikhah N, Aida SN. 1996. Pengaruh frekuensi pemberian pakan yang berbeda terhadap pertumbuhan dan kelangsungan hidup benih ikan baung (Mystus nemurus). Prociding Lolitkanwar. No. 2/1996.hlm 108-111.
Mulyasari, Iskandariah, Anang HK, Gleni HH. 2010. Analisis variasi genotipe ikan kelabau (Osteochilus kelabau) dengan metode mitokondria-restriction fragment length polymorphism (RFLP). J Riset Akuakultur 5(1):43-45.
Nasution Z, Utomo AD, Prasetyo D dan Yusuf S. 1993. Kajian ekonomi pada sumber daya perikanan baung di DAS Batang Hari, propinsi Jambi. Rakernis Balitkanwar Sukamandi 24-26 Mei 1993.
Nelson JS. 2006. Fishes of The World. Fouth Edition.J Willey. New Jersey. Nugroho E. 2001. Genetic divergence of kingfish from Japan, Australia and New
Zealand inferred by microsatellite and mitochondrial DNA control region marker. Fish Sci. 67.
Nugroho E. 2002. Rapid fluctuation of genetic variability in artificially propogated population of red sea bream. Indonesian Jurnal Agriculture Biotechnology. Indonesian Agency for Agriculture Research and Devolopment 7(1): 1-7.
33
2005.Depertemen kelautan dan perikanan Badan riset kelautan dan perikanan, pusat riset perikanan budidaya, balai riset balai riset perikan budidaya air tawar tahun 2005.
Nugroho E, Subagya J, Asih S, Kurniasih T. 2006. Evaluasi keragaman genetik ikan kancra dengan menggunakan marker mtDNA D-loop dan random amplified polymorpishm DNA (RAPD). J. riset akuakultur 1 (2):211-217.
Olesen I, Gjadrem T, Bentsen HB, Gjerde B, Rye M. 2003. Breeding program for sustainable aquaculture. Di dalam: B. B Jana and Carl D. Webster, editor. Sustainable Aquaculture.Global Perspective. Food Product Press an Imprint of The Haworth Press, Inc.hlm 179-204.
Park LK, Moran P. 1995. Development in molecular genetic techniques in fisheries. Di dalam: Gary R. Carvalho and T. T. Pitcher, editor. Molecular Genetics in Fisheries. Padstow: Chapman and Hall, TJ Press Ltd.hlm 1-28.
Randi E. Mitochondrial DNA di dalam: Baker, Alan J, editor Molecular Method in Ecology. Oxford: Blackwell Science. hlm 136-167.
Rina. 2001. Keragaman genetik ikan Pangasius Indonesia berdasarkan analisis DNA mitokondria dengan teknik PCR-RFLP [tesis]. Bogor: Program Pascasarjana, Institut Pertanian Bogor.
Samuel S, Adjie, Akrimi. 1995. Beberapa aspek biologi ikan baung (Mystus nemurus) di daerah aliran sungai Batang Hari, Propinsi Jambi. Oceanologi dan Limnologi di Indonesia 28: 1-13.
Simmons M, Mickett K, Kucuktas H, Li P, Dunham R, Luiz Z. 2006. Comparison of domestic and wild channel catfish (Ictalurus puncatus) population provides no evidence for genetic impact. Fish Molecular Genetic and Biotechnology Laboratory, 203 Swingle Hall, Department of Fisheries and Allied Aquacultures and Program of Cell and Molecular Biosciences, Aquatic Genomic Unit, Auburn University, Auburn .AL 36894, USA. Aquaculture 252:133-146.
Slamat. 2009. Keanekaragaman genetik ikan betok (Anabas testudineus Bloch) pada tiga ekosistem perairan rawa di Provinsi Kalimantan Selatan [tesis]. Bogor: Sekolah Pascasarjana, Intitut Pertanian Bogor.
Soewardi K. 2007. Pengelolaan Keragaman Genetik Sumberdaya Perikanan dan Kelautan.Departemen Manajemen Sumberdaya Perikanan Fakultas Perikanan dan Kelautan IPB.153 hal.
34
Sukendi. 2001. Biologi reproduksi dan pengendaliannya dalam upaya pembenihan ikan baung (Mystus nemurus) di perairan sungai Kampar Riau. Disertasi.Program pasca sarjana ipb.
Sukendi. 2005. Pengaruh kombinasi penyuntikan hCG dan ekstrak kelenjar hipofisa ikan mas (Cyprinus carpio) terhadap daya rangsang ovulasi dan kualitas telur ikan baung (Mystus nemurus C.V). J. Perikanan dan Kelautan 102: 75-81.
Sumadi, Marianti A. 2007. Biologi Sel. Graha Ilmu. Yogyakarta.
35
37
Lampiran 1 Lokasi penyamplingan populasi ikan baung (Hemibagrus nemurus)
no Populasi Provinsi Lokasi
1 Cirata Jawa Barat Waduk Cirata
2 Serayu Jawa Tengah Sungai Serayu perbatsan dengan Cilacap
3 Jambi Jambi Hasil domestikasi BBAT Jambi
4 Bogor Jawa Barat Sungai Cisadane perbatasan dengan
38
Lampiran 2 Hasil analisis menggunakan software TFPGA untuk nilai Variasi genetik empat populasi ikan baung berdasarkan D-loop mtDNA yang dipotong dengan 4 enzim yaitu MboI, HaeIII,AluI dan RsaI. 1). Cirata; 2). Serayu; 3).Jambi; 4). Bogor.
Data set contains genotypes of individuals sampled from populations.
Organism Type: Diploid
Marker Type: Codominant
DESCRIPTIVE STATISTICS
RESULTS FOR EACH POPULATION.
POPULATION 1
Locus 1 # obs. at locus= 10
allele: # obs: allele freq: # hets: het freq:
1 12 0.6000 0.0000 0.0000
2 8 0.4000 0.0000 0.0000
3 0 0.0000 0.0000 0.0000
4 0 0.0000 0.0000 0.0000
5 0 0.0000 0.0000 0.0000
6 0 0.0000 0.0000 0.0000
7 0 0.0000 0.0000 0.0000
Heterozygosity: 0.4800
Heterozygosity (unbiased): 0.5053
39
% polymorphic loci (no criterion): 100.0000
% polymorphic loci (99% criterion): 100.0000
% polymorphic loci (95% criterion): 100.0000
40
% polymorphic loci (no criterion): 0.0000
% polymorphic loci (99% criterion): 0.0000
% polymorphic loci (95% criterion): 0.0000
41
% polymorphic loci (no criterion): 100.0000
% polymorphic loci (99% criterion): 100.0000
% polymorphic loci (95% criterion): 100.0000
42
Ave. sample size: 10.0000
Ave. heterozygosity: 0.5000
Ave. heterozygosity (unbiased): 0.5263
Ave. heterozygosity (direct count): 0.0000
% polymorphic loci (no criterion): 100.0000
% polymorphic loci (99% criterion): 100.0000
43
Lampiran 3Hasil analisis menggunakan software TFPGA untuk nilai jarak genetikempat populasi ikan baung berdasarkan D-loop mtDNA yang dipotong dengan 4 enzim yaitu MboI, HaeIII,AluI dan RsaI. 1). Cirata; 2). Serayu; 3).Jambi; 4). Bogor.
Data set contains genotypes of individuals sampled from populations.
Organism Type: Diploid
Marker Type: Codominant
GENETIC DISTANCES
Roger's (1972) distance and Wright's (1978) modification.
Populations Roger's Wright's
compared distance modification
1 vs. 2 0.8718 0.8718
1 vs. 3 0.6856 0.6856
1 vs. 4 0.1000 0.1000
2 vs. 3 0.8426 0.8426
2 vs. 4 0.8660 0.8660
44
Lampiran 4Hasil analisis menggunakan software TFPGA untuk nilai uji Fst berpasanganempat populasi ikan baung berdasarkan mtDNA yang dipotong dengan 4 enzim yaitu MboI, HaeIII,AluI dan RsaI. 1). Cirata; 2). Serayu; 3).Jambi; 4). Bogor.
Data set contains genotypes of individuals sampled from populations.
Organism Type: Diploid
Marker Type: Codominant
Exact tests for population differentiation (Raymond and Rousset 1995)
# of dememorization steps: 1000
# of batches: 10
# of permutations per batch: 2000
Pairwise analysis of all populations
Groups compared: 1 vs. 2
locus 1 : p = 0.0000 S.E. :0.0000
Results over loci not calculated
Groups compared: 1 vs. 3
locus 1 : p = 0.0000 S.E. :0.0000
Results over loci not calculated
Groups compared: 1 vs. 4
locus 1 : p = 0.7535 S.E. :0.0061
45
Groups compared: 2 vs. 3
locus 1 : p = 0.0000 S.E. :0.0000
Results over loci not calculated
Groups compared: 2 vs. 4
locus 1 : p = 0.0000 S.E. :0.0000
Results over loci not calculated
Groups compared: 3 vs. 4
locus 1 : p = 0.0000 S.E. :0.0000