ANALISIS METAGENOM UNTUK PENCIRIAN KOMUNITAS BAKTERI DAN FUNGI PADA TEMPE
CECILIA ANNA SEUMAHU
SEKOLAH PASCA SARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI DISERTASI DAN SUMBER INFORMASI
Dengan ini saya menyatakan bahwa disertasi berjudul ‘Analisis Metagenom
untuk Pencirian Komunitas Bakteri dan Fungi pada Tempe’ adalah karya saya sendiri dan belum diajukan dalam bentuk apapun kepada perguruan tinggi dimanapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka dalam disertasi ini.
Bogor, September 2012
ABSTRACT
CECILIA ANNA SEUMAHU, Metagenome Analysis to fingerprint Bacterial and Fungi Communities in Tempe. Supervised by ANTONIUS SUWANTO (Major Advisor), IMAN RUSMANA, DEDY DURYADI SOLIHIN (Co advisor).
Tempe is an Indonesian traditional fermented food produced using Rhizopus as a starter culture. In practice, however, the starter culture as well as fermentation processes would involved a polymicrobial fermentation, which generated a unique tempe flavor and texture. This condition makes Indonesian tempe as one of the most complex fermented food, while at the same time would make it difficult to scale up tempe production with uniform quality and consistency. Because of complex microbial communities involved, culture-independent methods or metagenomic analysis would be an essential tool to unravel this complex microbial fermentation. The aim of this study was to compare a number of tempe microbial communities based on Amplified Ribosomal Intergenic Sequence Analysis (ARISA). Fresh tempe samples were obtained from tempe producers in Java and Moluccas. 16S rRNA gene libraries and DNA sequencing were employed to analyze further the nature of microbial diversity in two selected tempe samples. The results of our study showed that different tempe producer possessed different Bacterial ARISA (BARISA) or fungi ARISA (FARISA) profiles. However, BARISA profiles were found to be more discriminative than FARISA, and therefore BARISA would be more useful for tempe genetic fingerprint or barcoding. Bacterial communities in two tempe samples (EMP and SDJD) were further investigated employing DNA sequence analysis of their 16S rRNA gene libraries. Sequences closely related toKlebsiella sp were found to be the majority among bacterial communities present in SDJD sample, while sequences closely related to Acetobacter sp. were found as predominant isolates in EMP samples. These significant differences in bacterial communities might reflect differences in tempe flavor or texture associated with their unique production or locality.
RINGKASAN
Sebagai makanan fungsional tradisional Indonesia, tempe dikonsumsi dalam jumlah yang relatif tinggi dan dapat ditemukan dalam berbagai variasi jenis dan cara pengolahan. Variasi ini perlu dijaga karena menjadi ciri khas makanan Indonesia yang dapat dikembangkan dalam menghadapi program perdagangan bebas ASEAN. Walau demikian, database mikroorganisme yang terlibat dalam proses fermentasi tempe belum pernah dibuat dan ditelaah peluang adanya hubungan antara rasa tempe yang berbeda pada daerah sentra produksi terhadap komposisi mikroorganisme dan jaminan keamanan pangannya
DNA serta kemurnian DNA dengan rasio 260/280, amplifikasi gen 16S rRNA, visualisasi hasil amplifikasi sekuen ITS 16S-23S rRNA serta analisis ARISA untuk komunitas bakteri dan fungi di tempe.
© Hak Cipta milik IPB, tahun 2012 Hak Cipta dilindungi Undang-Undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber.
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karyailmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah
b. Pengutipan tidak merugikan kepentingan yang wajar IPB.
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan atas segala hikmat dan kebijaksanaan yang telah dilimpahkan bagi penulis sehingga disertasi dengan
judul “Analisis Metagenom Untuk Pencirian Komunitas Bakteri dan Fungi pada
Tempe” ini dapat diselesaikan dengan baik. Bagian dari disertasi ini juga telah dipublikasikan padaHayati Journal of BioscienceVol 19 No 2. Juni 2012 dengan
judul : “Comparison of DNA Extraction Methods for Microbial Community
Analysis in Indonesian Tempe Employing Amplified Ribosomal Intergenic
Spacer Analysis”.
Penelitian ini juga terlaksana atas bantuan berbagai pihak. Untuk itu penulis sangat berterima kasih kepada berbagai pihak:
1. Para pembimbing: Prof. Dr. Antonius Suwanto, M.Sc., Dr, Iman Rusmana, M.Si dan Dr. Dedy Duryadi Solihin, DEA atas segala bimbingan dan arahan yang diberikan selama penelitian dan penulisan disertasi ini dilakukan.
2. Direktorat Jenderal Pendidikan Tinggi Kementerian Pendidikan dan Kebudayaan atas beasiswa pendidikan BPPS yang penulis terima.
3. Direktorat Penelitian dan Pengabdian kepada Masyarakat Direktorat Jenderal Pendidikan Tinggi Kementerian Pendidikan dan Kebudayaan atas dana penelitian yang diberikan melalui penelitian Hibah Strategies Nasional dengan nomor kontrak 524/SP2H/PP/DP2M/VII/2010, Tanggal 24 Juli 2010 dan 406/SP2H/PL/Dit.Litabmas/IV/2011, Tanggal 14 April 2011.
4. Dekan dan Civitas Akademika Fakultas Teknobiologi Atmajaya Indonesia di Jakarta atas semua fasilitas laboratorium dan ijin penggunan isolat Klebsiella pneumoniae ATCC35657 koleksi Fakultas Teknobiologi UNIKA Atmajaya yang mendukung kelancaran penelitian ini.
5. Para Penguji Luar Komisi: Prof. Dr. Anja Meryandini, Prof. Dr. Suyanto Pawiroharsono, Prof. Dr. Aris Tri Wahyudi dan Dr. Yuri Sutanto yang berkenan meluangkan waktu untuk menguji dan memberi masukan demi perbaikan disertasi ini.
7. Arhad Kamahayanikam Vratyastoma dan Alvonsus Alvin atas semua bantuan dan kebersamaan selama penelitian ini berlangsung di Lab Riset FTB UNIKA Atmajaya.
8. Ibu Tati, Ibu Yunning, Pak Bambang, Pak Ridwan, Pak Dahrul, Pak Dahni dan Pak Nurdin atas semua bantuan selama penelitian ini dilakukan.
9. Dr. Nikmans Hatu yang telah membantu pengurusan dana penelitian. Dra. Cynthia Wattimena, M.Sc. yang telah meluangkan waktu untuk mencari literatur yang penulis butuhkan. Sanita Suryani, M.Si dan Ibu Lisa Hitalessy-Telussa atas bantuannya selama penulis melakukan pengambilan sampel. Handy E.P. Leimena, S.Si., M.Si. atas semua arahannya dalam bidang Ekologi. Ir. Delly Matruti, M.Si yang selalu bersedia dititipkan sampel sebelum dibawa ke lab.
10. Teman-teman seangkatan MIK 2008 dan 2009 atas kebersamaannya selama kuliah.
11. Papa dan Mama (alm), Papa dan Mama mertua (alm) dan semua saudara-saudara: John, Odie, Ella, Ain, Anes, Ici dan Eten yang selalu memberi dukungan Doa dan bantuan yang tidak terhingga.
12. Corinus Titihalawa dan Cecilia Daniela Titihalawa, suami dan anak tercinta atas semua pengorbanan dan dukungannya selama penulis menempuh pendidikan dan penelitian.
13. Semua pihak yang tidak dapat disebutkan namanya satu persatu yang telah banyak memberikan kontribusi selama penelitian ini berjalan.
Atas semua kebaikan yang diberikan, penulis mengucapkan terima kasih yang sebesar-besarnya. Penulis tidak dapat membalas semua kebaikan ini namun biarlah Tuhan yang empunya segala berkat melimpahkan berkat atas semua kebaikan yang diberikan.
Bogor, September 2012
RIWAYAT HIDUP
Penulis dilahirkan di Malang pada tanggal 19 April 1972 dari ayah Drs. Johannis Gerzon Seumahu, M.Pd. M.AppSc, dan Ibu Dra. Cecilia Cornelia Haullussy. Penulis menyelesaikan pendidikan S1 di Jurusan Biologi FMIPA IPB pada tahun 1997. Pendidikan S2 diselesaikan penulis pada tahun 2005 di Program studi Bioteknologi Sekolah Pasca Sarjana IPB Bogor dan pada tahun 2008 tercatat sebagai mahasiswa program S3 di Mayor Mikrobiologi Departemen Biologi FMIPA IPB.
DAFTAR ISI
Halaman
DAFTAR TABEL………... vii
DAFTAR GAMBAR……….. ix
DAFTAR LAMPIRAN……… xi
PENDAHULUAN Latar Belakang………... 1
Perumusan Masalah………... 2
Tujuan Penelitian……….. 3
Manfaat Penelitian………. 3
Kebaruan (Novelty)……… 3
Hipotesis Penelitian……… 3
Bagan Alir Penelitian……… 4
TINJAUAN PUSTAKA Proses Fermentasi yang Melibatkan Konsorsium Mikroba………..……. 5
Pentingnya Fingerprinting Komunitas Sebagai Sistem Barcoding pada Proses Fermentasi…….……….…… 6
Teknik Culture-Independent untuk Mempelajari Mikroba yang Terlibat dalam Suatu Proses Fermentasi Makanan..……… 7
Automated Intergenic Spacer Analysis(ARISA)….………. 8
Pustaka Gen 16S rRNA………. 11
Tempe Sebagai Makanan Tradisional yang Kaya Nutrisi………. 12
METODE PENELITIAN Tempat dan Waktu Penelitian……… 17
Bahan Penelitian……… 17
Pengambilan Sampel……… 17
Penyiapan Sampel……….. 17
Ekstraksi dengan Metode Fermentas DNA Purification Kit (FDEK)………... 18
Ekstraksi dengan Metode Power Food Microbial DNA Isolation Kit MOBIO (PFMDIK-MOBIO)………. 19
Analisis Kuantitas dan Kualitas DNA Hasil Ekstraksi……….. 19
Analisis Inhibitor PCR……….. 20
Analisis Keragaman Komposisi Mikroorganisme………. 20
Kloning dan Sequencing Gen 16S rRNA……….. 21
Analisis Isolat Tunggal dengan ARISA……….... 22
HASIL Optimasi Metode Ekstraksi DNA Mikroba di Tempe………..…………. 23
Analisis Gen 16s rRNA yang Berasal dari Komunitas Bakteri pada
Tempe………..………..…… 39
Analisis profil ARISA Isolat Tunggal Bakteri, Suatu Upaya Analisis Keamanan Pangan………..…. 40
PEMBAHASAN Optimasi Metode Ekstraksi DNA Mikroba di Tempe………….……. 45
Analisis Profil ARISA Type Tempe dari Sejumlah Produsen Tempe………. 50
Analisis Gen 16S rRNA yang Berasal dari Komunitas Bakteri pada Tempe ………...……….… 57
Analisis Profil ARISA Isolat Tunggal Bakteri, Suatu Upaya Analisis Keamanan Pangan……….. 63
SIMPULAN DAN SARAN Simpulan……….….. 65
Saran……….…… 65
DAFTAR PUSTAKA……….. 67
DAFTAR TABEL
Halaman
1. Bakteri dan khamir yang dilaporkan berasosiasi dengan proses fermentasi
tempe……….. 14
2. Konsentrasi DNA dan rasio A260/280 dan A260/230 untuk hasil ekstraksi
dengan menggunakan metode FDEK dan PFMDIK………..………. 23
3. Perbandingan profil OTU BARISAtypedan FARISAtypeyang diperoleh dari
DNA hasil ekstraksi dengan metode FDEK dan PFMDIK……….……… 25
4. Profil perbedaan perlakuan proses pembuatan tempe pada berbagai produsen.. 30
5. Keragaman OTU pada BARISA type dan FARISA type dari Berbagai Produsentempe………..…….……….
36
6. Ukuran Profil BARISA Isolat TunggalBacillus……… 41
DAFTAR GAMBAR
Halaman
1. Prinsip dasarAutomated Ribosomal Intergenic Spacer Analysis…….. 9
2. Kedudukan primer ITSF dan ITSReub……… 10
3. Kedudukan dan ukuran amplikon yang dihasilkan dari primer 2234C
dan 3126T……….. 11
4. Amplifikasi PCR gen 16S rRNA dari sampel tempe……… 24
5. Profil BARISAtypedariintergenic spaceryang diamplifikasi dengan
dua metode ekstraksi DNA……….. 25
6. Profil FARISA typedari daerah intergenic spacer yang diamplifikasi
dengan dua metode ekstraksi DNA……….. 26
7. Profil BARISA type (tiga ulangan) dari intergenic spacer yang
diamplifikasi dari DNA hasil ekstraksi dengan metode PFMDIK…… 27
8. Profil FARISA type (tiga ulangan) dari intergenic spacer yang
diamplifikasi dari DNA hasil ekstraksi dengan metode PFMDIK…… 28
9. Profil BARISA dari intergenic spacer yang diamplifikasi dari DNA
hasil ekstraksi dari beberapatempe……… 32
10. Profil BARISA dari intergenic spacer yang diamplifikasi dari DNA
hasil ekstraksi dari beberapatempe……… 33
11. Profil FARISA dari intergenic spacer yang diamplifikasi dari DNA
hasil ekstraksi dari beberapatempe..………. 34
12 Profil FARISA dari intergenic spacer yang diamplifikasi dari DNA
hasil ekstraksi dari beberapatempe..………. 35
13. Pohon filogenetik hubungan keterkaitan delapan komunitas bakteri
pada delapan tempe berdasarkanSorensen’s Similarity coefficient…... 37
14 Pohon filogenetik hubungan keterkaitan delapan komunitas fungi
pada delapan tempe berdasarkanSorensen’s Similarity coefficient... 38
15. Persentase spesies bakteri yang dominan ditemukan pada tempe
SDJD……….. 39
16. Persentase spesies bakteri yang dominan ditemukan pada tempe
EMP……….
17. Perbandingan profil BARISA isolat tunggal Bacillus yang terkultur
dan diisolasi dari tempe dibandingkan terhadap isolat ATCC…….. 41
18. Perbandingan profil BARISA isolat tunggal Klebsiella yang terkultur
DAFTAR LAMPIRAN
Halaman
1. Proses pengolahan pada delapan produsen tempe yang diamati….…… 79
2. BARISA type yang ditemukan dalam jumlah yang melimpah dan
berulang pada tempe……….……….………….….………. 80
3. FARISA type yang ditemukan dalam jumlah yang melimpah dan
berulang (dishading) pada tempe……….…………. 81
4. MatriksSorensen’sSimilarity coefficientkomunitas OTU BARISA….. 82
5. MatriksSorensen’sSimilarity coefficientkomunitas OTU FARISA….. 82
6. Identitas bakteri hasil analisis sequencing dari gen 16S rRNA yang
ANALISIS METAGENOM UNTUK PENCIRIAN KOMUNITAS BAKTERI DAN FUNGI PADA TEMPE
CECILIA ANNA SEUMAHU
Disertasi
Sebagai salah satu syarat memperoleh gelar Doktor pada
Mayor Mikrobiologi
SEKOLAH PASCA SARJANA INSTITUT PERTANIAN BOGOR
Penguji Luar Komisi Pembimbing:
Penguji Luar Komisi Ujian Tertutup (18 Oktober 2012)
1. Prof. Dr. Anja Meryandini (Guru Besar Departemen Biologi FMIPA IPB) 2. Prof. Dr. Suyanto Pawiroharsono (Deputi Bidang Teknologi Agroindustri dan
Bioteknologi-Badan Pengkajian dan Penerapan Teknologi,BPPT)
Penguji Luar Komisi Ujian Terbuka (14 November 2012)
1. Prof. Dr. Aris Tri Wahyudi (Guru Besar Departemen Biologi FMIPA IPB) 2. Dr. Yuri Sutanto (Senior Scientist-Aquaculture Technology Division pada PT.
LEMBAR PENGESAHAN
Judul Disertasi : Analisis Metagenom untuk Pencirian Komunitas Bakteri dan Fungi pada Tempe
Nama : Cecilia A. Seumahu
NRP : G361080011
Disetujui, Komisi Pembimbing
Prof. Dr. Antonius Suwanto, M.Sc. Ketua
Dr. Iman Rusmana, M.Si . Dr. Dedy Duryadi Solihin, DEA
Anggota Anggota
Diketahui,
Ketua Mayor Mikrobiologi Dekan Sekolah Pascasarjana
Prof. Dr. Anja Meryandini Dr. Ir. Dahrul Syah, M.Sc.Agr.
PENDAHULUAN
Latar Belakang
Sebagai makanan tradisional Indonesia, tempe telah banyak diketahui sebagai makanan yang dibuat dengan memanfaatkan fungi dari genus Rhizopus. Perkembangan penelitian tempe mencatat bahwa proses fermentasi tempe tidak hanya melibatkan Rhizopus saja tetapi juga melibatkan sejumlah bakteri dan khamir yang berperan dalam peningkatan nilai gizi tempe (Liem et al. 1977; Klus et al. 1993; Keuth & Bisping 1994; Denter et al. 1998; Klus & Bars 1998; Morenoet al.2002; Kimet al.2006; Baruset al. 2008; Sari 2009; Stefania 2009). Sampai saat ini penelitian yang dilakukan lebih diarahkan pada peran masing-masing mikroba secara terpisah-pisah padahal dalam suatu proses fermentasi mikroba akan bekerja dalam bentuk suatu konsorsium (Ampe et al. 1999; Randazo et al. 2002; Ogier et al. 2004; Scheirlinck et al. 2007, 2008). Konsorsium mikroba serta kondisi proses fermentasinya perlu dilihat secara menyeluruh karena sangat berpengaruh terhadap cita rasa, tekstur, kandungan nutrisi serta jaminan keamanan tempe (Watanabe et al. 2007; Hubertet al. 2008; Hugenholtz 2008; Rohmet al.2010).
fermentasi untuk memperoleh tempe dengan kandungan nutrisi serta tingkat keamanan pangan yang tinggi.
Perumusan Masalah
Tahapan penting dalam pemuliaan tempe sebagai makanan tradisional bernilai gizi penting adalah perlunya dilakukan identifikasi mikroba yang berperan dalam proses fermentasinya. Hal ini menjadi penting karena mikroba yang terlibat selama proses fermentasi berperan dalam pembentukan cita rasa, tekstur serta produksi komponen nutrisi pada makanan fermentasi. Informasi tentang mikroba yang terlibat, dapat digunakan sebagai acuan dalam merancang kultur starter dan dapat menjamin pengontrolan proses terkendali serta memiliki jaminan keamanan pangan. Teknik yang digunakan dalam mempelajari bakteri pada tempe sampai saat ini masih terbatas pada teknikculture-dependentsaja.
Dalam suatu proses fermentasi dapat dilibatkan berbagai macam jenis mikroba baik terkultur maupun tidak terkultur. Saat ini teknik yang berbasis culture-independent telah dikembangkan dengan baik untuk mengontrol produksi dan jaminan kualitas makanan. Teknik ini dapat memberi gambaran utuh tentang mikroba yang terlibat dalam proses fermentasi tempe. Upaya pemuliaan tempe dapat didasari dengan melakukan karakterisasi peran mikroba dalam memberi kontribusi tertentu untuk kandungan gizi serta aspek keamanan pangannya. Pengetahuan ini dapat menjadi aset penting dalam pemuliaan tempe sebagai makanan tradisional. Berdasarkan hal yang dikemukakan di atas, dapat dirumuskan masalah dasar penelitian ini:
1. Perlunya metode ekstraksi DNA yang bisa memberikan gambaran yang menyeluruh tentang komunitas mikroba dalam proses fermentasi.
2. Bagaimana pengaruh variasi pola produksi tempe terhadap komunitas mikroba pada produk akhir fermentasi Tempe?
3. Apakah profil komunitas bakteri atau fungi pada tempe dapat digunakan sebagai penciri ataubarcode?
Tujuan Penelitian
Menganalisis keragaman komunitas mikroorganisme (bakteri dan cendawan) dan mencari sistem diagnostik keragaman komunitas terbaik yang dapat digunakan sebagai penciri genetik (barcode) atau “fingerprinting” pada tempe.
Manfaat Penelitian
Penelitian ini merupakan penelitian dasar yang memberikan informasi tentang variasi keragaman mikroba yang terdapat dalam tempe Indonesia. Penelitian ini diharapkan dapat membuka jalan untuk identifikasi dan inventarisasi mikroba pada tempe agar dapat membantu pengembangan proses fermentasi yang lebih andal. Informasi ini juga menjadi batu loncatan untuk pengembangan kontrol kualitas tempe.
Kebaruan (Novelty)
Penelitian ini merupakan penelitian pertama untuk pencirian tempe dan melihat pengaruh keragaman mikroba terhadap perbedaan karakteristik tempe di Indonesia. Penelitian ini tidak hanya dilakukan untuk melihat keberadaan mikroba yang terkultur tetapi juga melihat keberadaan mikroba tidak terkultur. Penelitian ini juga memberikan informasi tentang adanya plasma nutfah mikroba baru yang masih dapat dieksplorasi lebih lanjut.
Hipotesis Penelitian
Bagan Alir Penelitian
Sampel Tempe
Isolasi DNA Metode PFMDIK Isolasi DNA Metode FDEK
Pengukuran Kuantitas DNA dan Uji Penghambatan PCR
Profil DNA Metode FDEK
Metode Standar untuk Isolasi Mikroba dari Tempe
A R I S A
Proil DNA Metode PFMDIK
Sampel Tempe Bogor Sampel Tempe Ambon Sampel Tempe Malang
Profil ARISA Mikroba dalam Proses Fermentasi Tempe
Pembuatan Pustaka Gen 16S rRNA dari Dua Tempe Profil ARISA Dua Isolat Bakteri dari Tempe dengan dua Isolat ATCC
Gambaran Struktur Komunitas Bakteri pada Tempe yang Berbeda
Perbandingan Kesamaan Isolat secara Genetik Sampel Tempe Sidoarjo
TINJAUAN PUSTAKA
Proses Fermentasi yang Melibatkan Konsorsium Mikroba
Makanan fermentasi adalah substrat makanan yang ditumbuhi oleh mikroba, dapat dimakan (edible) dan mengandung berbagai macam enzim yaitu: amilase, protease, lipase dan enzim penghidrolisis polisakarida. Selain itu pada makanan fermentasi dapat ditemukan pula protein dan lipid sebagai produk non toksik serta cita rasa, aroma serta tekstur yang menyenangkan dan menarik untuk dikonsumsi oleh manusia (Steinkraus 1997). Proses fermentasi makanan dapat dikategorikan menjadi proses fermentasi spontan yang tidak menggunakan kultur starter (NKS) dan proses fermentasi menggunakan kultur starter (KS). Kultur starter yang digunakan dapat berupa isolat tunggal atau kultur campuran. Proses fermentasi spontan (NKS) maupun fermentasi KS, masih memungkinkan masuknya berbagai mikroba lain yang akhirnya terlibat dalam proses fermentasi tersebut (Holzapfel 2002). Proses tersebut dapat pula melibatkan berbagai jenis bakteri terkultur maupun tidak terkultur (Ampeet al. 1999).
Komposisi mikroba yang terlibat dalam proses fermentasi makanan sangat penting karena berkontribusi terhadap cita rasa, tekstur dan nilai gizi dari makanan. Produksi komponen aroma juga dapat dihasilkan atau dimodifikasi dari eksopolisakarida dan protein yang ada pada bahan makanan. Komponen nutrisi seperti vitamin juga dapat dihasilkan karena peranan mikroba dalam proses fermentasi (Holzaffel 2002; Hugenholtz 2008). Secara tradisional komposisi mikroba dalam proses fermentasi yang menghasilkan produk tertentu dapat digunakan kembali untuk mempercepat kondisi fermentasi pada proses fermentasi selanjutnya. Hal ini dilakukan dengan menggunakan kultur backslopping atau dengan menggunakan residu fermentasi kering (Holzaffel 2002; Kourkoucaset al. 2006).
PentingnyaFingerprinting komunitas Sebagai SistemBarcoding Pada Proses Fermentasi
Produk fermentasi yang beragam dapat dihasilkan dari bahan baku yang sama. Mikroba merupakan parameter paling sulit untuk dikontrol selama proses fermentasi. Mikroba dalam proses fermentasi keju, dapat berasal dari kultur bakteri asam laktat yang ditambahkan maupun kultur bakteri asam laktat lain dalam starter. Bakteri lain, khamir dan fungi dapat juga timbul secara spontan dari lingkungan. Populasi mikroba yang tumbuh selama proses fermentasi sangat sulit dikontrol karena kompleksnya dinamika dan interaksi antar mikroba. Pengetahuan tentang struktur dan dinamika dari komunitas mikroba yang terlibat menjadi penting karena dapat memberi pemahaman lebih baik tentang variasi karakteristik produk fermentasinya. Hal ini disebabkan adanya pertumbuhan dan metabolisme mikroba yang ada. Kontrol komposisi mikroba akan memudahkan pemilihan karakteristik tertentu dan mencegah penurunan kualitas produk fermentasi (Jani & Barbier 2008).
mikroba dalam proses fermentasinya secara menyeluruh (Ogieret al. 2004). Studi tentang fingerprinting komunitas kultur starter akan membantu memberi pemahaman hubungan antara keragaman mikroba dan karakteristik spesifik yang dihasilkan. Analisis ini dapat menjadi dasar perancangan suatu kultur starter dan pengontrolan proses fermentasi (Lvet al.2012)
Teknik Culture-Independent untuk Mempelajari Mikroba yang Terlibat dalam Suatu Proses Fermentasi Makanan
Upaya untuk mempelajari komunitas mikroba dalam suatu proses fermentasi makanan secara tradisional perlu dilakukan dan tidak hanya terbatas pada teknik culture-dependent saja tetapi juga penting untuk melihat keragaman komunitas dengan menggunakan teknik berbasisculture-independent(Ampeet al. 1999). Keterbatasan metode culture-dependent dalam kurun waktu dua dekade menyebabkan adanya pengembangan teknik culture-independent yang berdasarkan pada analisa asam nukleat. Teknik ini juga telah banyak digunakan dalam mempelajari komunitas mikroba pada ekosistem makanan untuk mengontrol produksi dan jaminan kualitas makanan yang baik (Justeet al. 2008).
Teknik culture-independent merupakan teknik yang mampu memberikan gambaran komunitas dalam suatu sistem makanan. Keterbatasan teknik culture-independent sangat dipengaruhi oleh kemampuan metode ekstraksi DNA yang digunakan. Ekstraksi DNA dari makanan dan ada tidaknya nuklease maupun inhibitor PCR yang dapat terekstraksi dari bahan makanan tersebut sangat mempengaruhi kualitas DNA hasil ekstraksi (Metwallyet al. 2008). Metode yang digunakan haruslah metode terbaik untuk sampel spesifik, kriteria utamanya didasarkan pada kualitas dan kuantitas DNA hasil ekstraksi (Jara et al. 2008). Kuantitas dan kualitas DNA bersifat sangat kritis untuk interpretasi analisis komunitas mikroba (Thakuria et al. 2008). Ekstraksi DNA dari tempe belum banyak dipelajari padahal metode ekstraksi sangat bersifat spesifik untuk matriks atau bahan dasar makanan yang berbeda (Abrioruelet al. 2006; Jaraet al. 2008).
membedakan variasi organisme secara luas. Sekuen gen yang dipilih harus memiliki sekuen variabel dan konservatif dimana domain yang variabel memungkinkan diskriminasi dalam selang yang luas dari suatu tingkatan taksonomi. Sekuen target ini harus diapit oleh daerah yang koservatif. Gen yang dapat digunakan sebagai penanda (marker) dapat dikelompokkan menjadi dua yaituubiquitously conserved genesdanfunctional genes(Justeet al. 2008).
Automated Intergenic Spacer Analysis(ARISA)
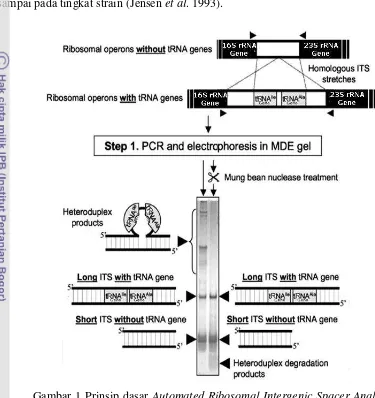
Metode rRNA Intergenic Spacer Analysis (RISA) merupakan salah satu metode culture-independent yang melibatkan amplifikasi PCR daerah intergenik antara gen 16S dan 23S rRNA. Daerah ini menunjukkan heterogenitas tinggi yang bergantung pada spesies baik dalam hal panjang amplikon maupun sekuen yang dihasilkan (Gambar 1). Kedua tipe variasi pada teknik ARISA ini telah banyak digunakan untuk membedakan strain-strain maupun spesies yang berkerabat secara dekat (Jensen et al. 1993; Maes et al. 1997) dan daerah ini dapat pula digolongkan sebagai ubiquitously conserved genes. Fisher dan Triplett (1999) menggunakan teknik ini dengan memodifikasi teknik RISA dan menggunakan primer oligonukleotida terlabel fluoresens pada proses PCR. Teknik ini selanjutnya disebut Automated RISA (ARISA). Metode ARISA sangat memudahkan analisis data yang dihasilkan.
terdeteksi secara tepat (Ranjard et al. 2001). Profil ARISA (Gambar 1) sangat bergantung pada jumlah kopi operon gen rRNA yang ada pada sel (Dafonchio et al. 2003) dan bersifat spesifik karena mampu memilah keragaman mikroba sampai pada tingkat strain (Jensenet al. 1993).
Gambar 1 Prinsip dasar Automated Ribosomal Intergenic Spacer Analysis (ARISA-Dafonchioet al, 2003).
primer tersebut mampu mengeksplorasi keragaman bakteri dengan memberikan tingkat keragaman yang tinggi, tidak menyebabkan bias dan menghasilkan fingerprinting molekuler yang mudah dianalisis (Gambar 2). Pasangan primer ini telah diuji terhadap berbagai macam sumber sampel dan mampu mereduksi bias dalam analisa ARISA yang memungkinkan diperolehnya gambaran secara global tentang komunitas suatu sistem ekologi. Pada analisis keragaman fungi, Ranjard et al. (2001) telah merancang pasangan primer 2234C dan 3126T yang mampu mendeteksi variasi fungi dan dapat digunakan untuk melihat keragaman fungi dalam kelompok taksonomi utama fungi (Gambar 3). Analisis keragaman dilakukan dengan melihat fragmen DNA yang muncul dengan ukuran berbeda sebagai peak dalam ARISA type. Fragmen DNA ini didefinisikan sebagai OTU (Operational Taxonomic Unit) (Hewson & Fuhrman 2004; Ramete 2009).
Profil ARISA yang baik sangat bergantung pada kualitas DNA total hasil isolasi. Metode ekstraksi DNA sangat mempengaruhi kuantitas dan kualitas DNA untuk berhasilnya tahapan amplifikasi (Simon et al. 1996; Abriouel et al. 2006; Thakuria et al. 2008). Metode yang digunakan dalam ekstraksi DNA total perlu dioptimasi untuk mendapatkan metode terbaik untuk memberi gambaran menyeluruh tentang total komunitas mikroba dalam suatu sistem ekologi. Bahan makanan dapat mengandung bahan pengganggu PCR dan metode yang digunakan harus mampu mengekstrak semua DNA dari sampel. Hal ini diperlukan agar dapat memberikan profil yang sebenarnya tentang semua mikroba yang ada (Abriouelet al. 2006; Ranjard et al. 2001). Faktor yang perlu diperhatikan dalam proses tersebut adalah korelasi nyata antara jumlah ribotipe reprodusibel yang dihasilkan dan ekstrak DNA hasil ekstraksi. Metode yang digunakan dalam ekstraksi DNA
total juga perlu dioptimasi untuk mendapatkan metode terbaik (Ranjard et al. 2001).
Pustaka Gen 16S rRNA
Gen 16S rRNA merupakan gen yang selalu ada pada semua prokariot dan menjalankan fungsi sama. Perubahan sekuen pada gen ini akan menandai jarak evolusi antara mikroba yang berkerabat. Intensitas perubahan dapat terjadi dengan kecepatan dan lokasi dalam gen 16S rRNA yang tidak seragam antara setiap mikroba. Panjang gen 16S rRNA adalah sekitar 1550 bp dan tersusun atas daerah variable dan konservatif. Gen ini cukup besar dan memiliki sifat polimorfisme interspesifik yang dapat digunakan sebagai pembeda nyata secara statistik. Primer universal dirancang sebagai fragmen komplemen pada awal dan ujung gen. Daerah gen yang variable diapit oleh primer dan dapat digunakan dalam analisis komparatif taksonomi (Clarridge 2004).
Weisburg et al. (1991) telah menemukan bahwa amplifikasi gen 16S-rRNA, kloning dan sekuensing merupakan metode untuk mengidentifikasi mikroorganime tanpa harus dikulturkan. Pendekatan pustaka gen 16S rRNA dapat dimanfaatkan dalam mengidentifikasi spesies spesifik yang berpotensi sebagai indikator dalam mempelajari struktur suatu komunitas (Hartman & Widmer, 2006). Marchesi et al. (1998) telah merancang sepasang primer yang dapat digunakan untuk mengamplifikasi gen 16S rRNA dari kelompok bakteri yang disebut primer 63F dan 1387R.
Tempe Sebagai Makanan Tradisional yang Kaya Nutrisi
Kedelai merupakan bahan makanan yang unik karena kaya akan nutrisi. Kedelai mengandung karbohidrat kompleks, protein nabati, lipid, serat, oligosakarida, senyawa fitokimia (khususnya isoflavon) dan mineral. Protein kedelai dikenal sebagai protein yang lengkap dan setara dengan albumin telur. Substitusi protein hewani dengan protein kedelai akan mereduksi ekskresi kalsium urin dan mereduksi osteoporosis. Kandungan serat yang larut dan tidak larut juga memberi keuntungan bagi kesehatan, sedangkan oligosakaridanya berperan sebagai komponen prebiotik di kolon. Isoflavon genistein dan daidzein dari kedelai memiliki sejumlah fungsi biologis dan mineralnya berada dalam bentuk yang tersedia sehingga menjadi penting karena mereduksi osteoporosis dan hipertensi (Andersonet al. 1999). Kedelai juga mengandung asam lemak gliserid seperti asam palmitat, stearat, oleat, linoleat dan linoleat (de Reu et al. 1994). Zamora & Veum (1979) menyatakan bahwa nutrisi kacang kedelai yang difermentasi dengan adanya fungi lebih signifikan meningkatkan average daily gain (ADG) dan rasio gain:feed (G:F) dari tikus. Hal ini menunjukkan bahwa proses fermentasi berperan penting dalam menyediakan nutrisi yang lebih siap untuk diserap.
besi dan kalsium yang terbaik bagi bayi karena kandungan mineralnya relatif tinggi dan kandunganphytat-nya rendah. Tempe yang berbahan dasar kedelai dan diproses melalui fermentasi tradisional diproses melalui tahapan proses soaking (perendaman dalam kondisi asam), dehulling(pengelupasan mantel biji), cooking (perebusan) dan fermentasi. Karena kedelai sebagai bahan baku tempe mengandung sejumlah senyawa antinutrisi dan senyawa yang bersifat toksik, Egounlety & Aworh (2003) melaporkan bahwa tahapan preparasi sebelum proses fermentasi perlu dilakukan sebagai tahapan persiapan substrat untuk selanjutnya mengalami pemecahan secara enzimatik oleh fungi. Pada tahapan preparasi ini juga, proses cooking secara signifikan mereduksi inhibitor trypsin pada kacang-kacangan, dehulling mengeliminasi tannin dan fermentasi dengan Rhizopus oligosporus mereduksi asam phytat sebanyak 30,7% pada kedelai. Proses Soaking-dehulling-cooking dan fermentasi dengan Rhizopus oligosporus mengeliminasistachyoseyang merupakan oligosakarida yang sangat flatulent.
GalurRhizopusyang digunakan sebagaistarter sangat beragam. Baumann & Bisping (1995) pernah melaporkan adanya 36 strain Rhizopus asal Indonesia yang biasa digunakan untuk membuat tempe. Rhizopus yang digunakan dalam pembuatan tempe mampu membentuk karotenoid dan menurunkan jumlah tocopherol dengan jumlah vitamin E yang tetap, menurunkan asam lemak gliserid (de Reu et al. 1994), mampu melakukan proteolisis, meningkatkan jumlah asam amino bebas dalam suatu proses fermentasi tempe (Baumann & Bisping 1995), serta memproduksi ergosterol (Denter et al. 1998). Proses fermentasi ini dapat memberikan hasil yang berbeda bila galur fungi dikombinasikan dengan bakteri dalam proses fermentasi (de Reuet al. 1994; Baumann & Bisping 1995; Denteret al. 1998). Kemampuan proteolisis R. stolonifer, R. oryzae dan R oligosporus dalam melakukan proteolisis sangat beragam, bahkan sangat bergantung pada galur dan bukan spesies (Baumann & Bisping 1995). Rhizopus juga telah
dilaporkan mampu mensintesis β-carotene namun kemampuan ini tidak dimiliki
oleh semua spesies ini (Denter et al. 1998). Jenis asam lemak yang didegradasi dan dihasilkan juga sangat beragam (de Reuet al. 1994).
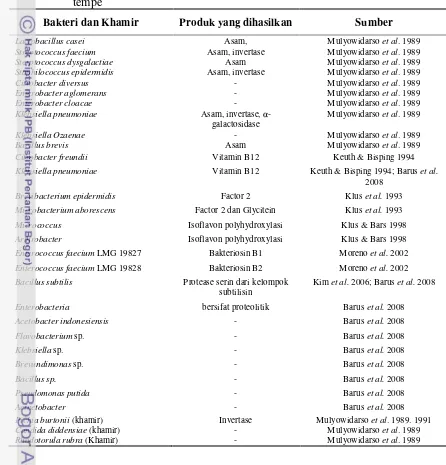
melaporkan adanya keterlibatan mikroba lain seperti bakteri dan kapang dalam proses ini. Liem et al. (1977) pernah membuktikan peran bakteri dalam membentuk vitamin B12 pada tempe (Tabel 1).
Tabel 1 Bakteri dan khamir yang dilaporkan berasosiasi dengan proses fermentasi tempe
Bakteri dan Khamir Produk yang dihasilkan Sumber
Lactobacillus casei Asam, Mulyowidarsoet al. 1989
Streptococcus faecium Asam, invertase Mulyowidarsoet al. 1989
Sterptococcus dysgalactiae Asam Mulyowidarsoet al. 1989
Staphilococcus epidermidis Asam, invertase Mulyowidarsoet al. 1989
Citrobacter diversus - Mulyowidarsoet al. 1989
Enterobacter aglomerans - Mulyowidarsoet al. 1989
Enterobacter cloacae - Mulyowidarsoet al. 1989
Klebsiella pneumoniae Asam, invertase, α
-galactosidase
Mulyowidarsoet al. 1989
Klebsiella Ozaenae - Mulyowidarsoet al. 1989
Bacillus brevis Asam Mulyowidarsoet al. 1989
Citrobacter freundii Vitamin B12 Keuth & Bisping 1994
Klebsiella pneumoniae Vitamin B12 Keuth & Bisping 1994; Baruset al.
2008
Brevibacterium epidermidis Factor 2 Kluset al.1993
Microbacterium aborescens Factor 2 dan Glycitein Kluset al.1993
Micrococcus Isoflavon polyhydroxylasi Klus & Bars 1998
Arthrobacter Isoflavon polyhydroxylasi Klus & Bars 1998
Enterococcus faeciumLMG 19827 Bakteriosin B1 Morenoet al.2002
Enterococcus faeciumLMG 19828 Bakteriosin B2 Morenoet al.2002
Bacillus subtilis Protease serin dari kelompok
subtilisin
Kimet al.2006; Baruset al.2008
Enterobacteria bersifat proteolitik Baruset al.2008
Acetobacter indonesiensis - Baruset al.2008
Flavobacteriumsp. - Baruset al.2008
Klebsiellasp. - Baruset al.2008
Brevundimonassp. - Baruset al.2008
Bacillus sp. - Baruset al.2008
Pseudomonas putida - Baruset al.2008
Acinetobacter - Baruset al.2008
Pichia burtonii(khamir) Invertase Mulyowidarsoet al. 1989. 1991
Candida diddensiae(khamir) - Mulyowidarsoet al. 1989
Rhodotorula rubra(Khamir) - Mulyowidarsoet al. 1989
tingginya keragaman bakteri pada tempe. Keterlibatan khamir dalam fermentasi tempe di Malaysia juga telah dilaporkan oleh Morenoet al. (2002).
Bakteri asam laktat juga telah dilaporkan berpengaruh terhadap komposisi beberapa produk makanan dengan bahan baku kedelai. Bakteri ini dilaporkan berpengaruh terhadap komposisi senyawa fitokimia dan sifat antioksidan dari kecambah kedelai sebagai hasil proses fermentasinya (Hubert et al. 2008). Pada produk pasta kedelai (doenjang), bakteri asam laktat dan bakteri-bakteri lain seperti Bacillus dilaporkan berperan dalam membentuk komponen-komponen pembentuk cita rasa, protein dan bakteriosin pada makanan ini (Kimet al. 2009). Kondisi lingkungan yang berbeda antara daerah penghasil tempe juga dapat berpengaruh terhadap hasil fermentasi. Baumann dan Bisping (1995) menyatakan bahwa suhu yang rendah akan menurunkan kecepatan fermentasi, namun justru dapat meningkatkan kandungan asam amino bebas pada tempe.
Mengacu pada penelitian yang dikemukakan di atas terlihat bahwa dalam proses fermentasi tempe di Indonesia melibatkan sangat banyak spesies Rhizopus sebagai kulturstarter pada daerah produsen tempe yang berbeda-beda. Selain itu karena kondisi lingkungan produsen tempe yang berbeda-beda, dapat terjadi bahwa proses fermentasi melibatkan banyak sekali mikroba lain selain kultur starter. Varietas biji kedelai yang digunakan produsen tempe juga berbeda pada daerah penghasil tempe yang berbeda. Hal ini dapat berpengaruh pada hasil akhir fermentasi. Peran kapang Rhizopus, selain menyediakan beberapa komponen penting juga berperan untuk menyediakan substrat bagi bakteri dalam mensintesis komponen cita rasa dan komponen nutrisi lain.
menunjukkan bahwa sampel sourdough yang berasal dari depositor yang sama akan dikelompokkan bersama. Korelasi antara tipe tepung, propinsi penghasil atau karakteristik teknologi (pH, waktu fermentasi dan suhu) tidak ditemukan demikian pula asosiasi signifikan antara spesies bakteri asam laktat tertentu dengan salah satu dari variabel yang diamati. Keragaman variasi komunitas bakteri asam laktat ini secara signifikan sangat dipengaruhi oleh lingkungan bakerytempat terjadinya fermentasisourdough.
Walaupun berkontribusi secara positif, ternyata mikroba yang terlibat dalam proses fermentasi tempe dapat juga memberi dampak yang merugikan. Rhizonin adalah hepatotoksik siklopeptida dari kultur fungiRhizopus microspores yang tumbuh pada kacang-kacangan di Mozambique. Penelitian menunjukkan bahwa toksin ini sebenarnya tidak diproduksi oleh fungi tetapi oleh bakteri dari genus Burkhoderia yang merupakan endosimbion dalam sitosol fungi (Partida-Martinez et al. 2007a, 2007b.). Rohm et al. (2010) menunjukkan bahwa bakteri toksigenik ini juga ditemukan pada kulturstarterdalam produksi tempe atau sufu. Profil metabolik dari fungi selama kondisi standar untuk produksi tempe atau sufu, menunjukkan bahwa toxin dan rhizonin diproduksi dalam jumlah cukup kritis.
METODE PENELITIAN
Tempat dan Waktu Penelitian
Metode yang digunakan dalam penelitian ini adalah metode eksperimen laboratorium. Penelitian ini dilakukan di Laboratorium Riset Fakultas Teknobiologi UNIKA Atmajaya Jakarta mulai bulan Agustus 2010 hingga bulan April 2012.
Bahan Penelitian
Bahan-bahan yang digunakan dalam penelitian ini adalah sampel Tempe yang berasal dari beberapa daerah (Jawa Barat, Jawa Timur dan luar Jawa-Ambon), Isolat Klebsiella 135 (Hanjaya 2001), Bacillus GR9 (Wati 2011), Klebsiella pneumoniae ATCC 35657 (Koleksi Fakultas Teknobiologi-UNIKA Atmajaya Jakarta) danBacillus cereusATCC10876 (Koleksi IPBCC, Bogor).
Pengambilan Sampel
Sampel diambil dari delapan pengrajin dari empat daerah yaitu di Jawa Barat (Bogor), Jawa Timur (Malang dan Sidoarjo) dan Maluku (Ambon). Sampel tempe diambil dari dua produsen di tiap-tiap daerah. Proses pembuatan tempe pada setiap produsen dicatat dan dirangkum sebagai data penunjang dalam penelitian ini.
Penyiapan Sampel
selanjutnya disentrifugasi dengan kecepatan 800xg (Sorval Legend Micro 17R Centrifuge) selama 1 menit dan supernatan diambil untuk selanjutnya disentrifugasi lagi dengan kecepatan 13,000xg untuk diambil peletnya. Pelet yang diperoleh selanjutnya dicuci dengan larutanTris-EDTA (TE) pH 8.0 dan disentrifugasi lagi pada 13,000xg. Supernatan dibuang dan selanjutnya pelet siap diekstraksi dengan menggunakan kit ekstraksi komersil sesuai prosedur yang dijelaskan oleh perusahaan (Fermentas DNA purification Kit (FDEK) dan Power Food Microbial DNA Isolation Kit MOBIO (PFMDIK)).
Ekstraksi dengan MetodeFermentas DNA Purification Kit(FDEK)
Tahapan ini dilakukan dengan melakukan ekstraksi dan purifikasi DNA dari sampel tempe yang diambil dengan menggunakan metode Fermetas DNA purification kit. Pelet hasil pencucian dengan TE selanjutnya diresuspensikan
dengan 200 μ l TE dan selanjutnya ditambahkan 400 μ l lysis solution dan
diinkubasi pada 650C selama 5 menit. Kloroform selanjutnya ditambahkan
sebanyak 600 μ l, dibolak-balik sebentar dan selanjutnya disentrifugasi dengan
kecepatan 10,000xg (Sorval Legend Micro 17R Centrifuge) selama 2 menit. Fase bagian atas selanjutnya dipindahkan ke tabung eppendorf baru dan ditambahkan
800 μ l precipitation solution 1x dan dibiarkan pada suhu ruang selama 1-2 menit.
Sentrifugasi selanjutnya dilakukan dengan kecepatan 10,000xg selama 2 menit.
Supernatan selanjutnya dibuang dan pellet dilarutkan dalam 100 μ l NaCl.
Ekstraksi dengan Metode Power Food Microbial DNA Isolation Kit MOBIO (PFMDIK).
Tahapan ini diawali dengan melakukan ekstraksi dan purifikasi DNA dari sampel tempe yang diambil dengan menggunakan metode PFMDIK. Ekstraksi dengan kit ini dimulai dengan meresuspensikan pelet pada 450 μ l solution PF1 yang telah dihangatkan pada suhu 550C. Suspensi kemudian dipindahkan ke tabung MicroBead dan diletakkan secara horizontal pada vortex serta dilakukan pengadukkan selama 10 menit dengan kecepatan maksimum (2500 rpm-Heidolp REAX control). Sentrifugasi selanjutnya dilakukan pada kecepatan 13,000xg selama 1 menit, supernatan yang diambil dan dipindahkan pada tabung baru serta
diberi 100 μ llarutan PF3 yang dicampur dengan cara divortex. Inkubasi dilakukan
pada suhu 40C selama 5 menit dan selanjutnya disentrifugasi lagi dengan kecepatan 13,000xg selama 1 menit. Supernatan diambil dan ditambah dengan
900 μ l larutan PF3 serta divortex. Campuran selanjutnya dipipet dan dimasukkan
ke tabung spin filter untuk selanjutnya dilakukan sentrifugasi selama 1 menit. Tahapan ini diulang sampai semua campuran habis. Spin filter selanjutnya dipindahkan pada tabung yang baru dan diteteskan larutan PF4 sebanyak 650 μ l dan disentrifugasi lagi pada 13,000xg selama 1 menit. Larutan yang terbuang
melalui filter dibuang dan filter kembali ditetesi 650 μ l larutan PF5 dan dilakukan
sentrifugai lagi pada kecepatan 13,000xg selama 1 menit. Larutan yang terbuang melalui filter selanjutnya dibuang dan dilakukan sentrifugasi lagi selama 2 menit pada kecepatan 13,000xg. Spin filter selanjutnya dipindahkan pada tabung baru
yang bersih dan ditetesi 100 μ l larutan PF6. Sentrifugasi dilakukan lagi dengan
kecepatan 13,000xg selama 1 menit. DNA yang diperoleh selanjutnya disimpan pada suhu -200C.
Analisis Kuantitas dan Kualitas DNA Hasil Ekstraksi
kualitas yang baik harus menunjukkan rasio absorbansi 260/230 nm dan 260/280 nm lebih besar atau sama besar dengan 1.80 (Sambrooket al. 1998)
Analisis Inhibitor PCR
Pada analisis inhibitor PCR, amplifikasi gen 16S rRNA dilakukan dengan menggunakan primer 1387r dan 63f (Marchesi et al. 1998) dengan kondisi PCR: pradenaturasi 940C, 5 menit; denaturasi 940C, 30 detik; pelekatan primer 550C, 30 detik pemanjangan 720C, 1 menit dan tahap akhir 720C, 20 menit dengan 35 siklus PCR (C1000TMThermal Cycler-BIORAD). Amplifikasi dilakukan dengan menggunakan GoTaq Green Master Mix (Promega) dan hasil amplifikasi dilarikan pada gel agarose dengan konsentrasi 1%.
Analisis Keragaman Komposisi Mikroorganisme
Analisa ARISA dilakukan dengan menggunakan metode yang dijelaskan oleh Cardinale et al. (2004) dengan menggunakan primer ITSF dan ITSFReub untuk kelompok bakteri (B-ARISA) dan primer 2234C dan 3126T untuk kelompok fungi (F-ARISA) (Ranjardet al.2001) sampai ditemukan profil ARISA yang maksimum dan konsisten
Untuk primer ITSF/ITSFReub (B-ARISA) dan primer 2234C dan 3126T (F-ARISA), diamplifikasi dalam reaksi yang mengandung 1 x buffer PCR, 1.5 U Taq DNA polymerase, 0.2 mM (masing-masing) dNTP dan 0.25 μ M (masing
-masing) primer dalam volume akhir 25 μ l. Campuran direaksikan pada 940
C selama 3 menit, diikuti 30 siklus amplifikasi dengan kondisi pradenaturasi 940C selama 45 detik, annealing 56.80C selama 1 menit Untuk B-ARISA dan 60.70C untuk F-ARISA, dan pemanjangan 720C selama 2 menit dengan reaksi akhir dilakukan pada 720C selama 7 menit.
Jumlah standar dari produk PCR (antara 0.5 dan 1 μ l) bersama dengan 0.8 μ l
internal size standard, 1000 atau 2500 ROX ditambahkan ke 13 μ l formamide
tabung kapiler yang diisi dengan electrophoresis polymer POP-4 (Applied Biosystem). Sampel dilarikan pada kondisi ABI 310 denaturing elektrophoresis selama 1 jam untuk masing-masing sampel, dan data dianalisa menggunakan GeneScan 3.1 software program (Perkin-Elmer). Keluaran program berupa sejumlah puncak (electropherogram), ukurannya diestimasi melalui perbandingan terhadap internal size standard. Software GeneScan akan menghitung tinggi dan area puncak yang proporsional dengan kuantitas DNA dalam fragment. Semua perlakuan ARISA di ulang sebanyak tiga kali. Reprodusibilitas masing-masing sampel DNA makanan diukur sebagai rata-rata dan standar deviasi relatif dari jumlah puncak yang dikalkulasi dari 3 ulangan percobaan yang tidak saling terkait. Data profil ARISA selanjutnya diedit dan diolah untuk melakukan perhitungan karakter ekologi yaitu nilai keragaman. Fragmen DNA dengan ukuran berbeda yang muncul sebagai peak dalam ARISA type didefinisikan sebagai OTU (Operational Taxonomic Unit) (Hewson & Fuhrman 2004; Ramete 2009). Nilai indeks Shannon-Wiener dan Index Simpson selanjutnya dihitung dengan rumus yang digunakan oleh Hewson & Fuhrman (2004). Karakter OTU yang ada pada profil komunitas bakteri dan fungi selanjutnya dianalisis dengan
menghitung Sorensen’s Index (Lampiran 4 dan 5). Analisis pengelompokan
dilakukan dengan metode unweighted-pair-group mean-average (UPGMA) dan software MEGA5.1 (Hewson & Fuhrman 2004; Nimnoiet al. 2010)
Kloning dan Sequencing Gen 16S rRNA
dilakukan dengan mencari kesamaan sekuen pada data base National Center for Biotechnology Information(NCBI).
Analisis Isolat Tunggal dengan ARISA
Analisis ini dikerjakan dengan melakukan ekstraksi genom isolat tunggal beberapa bakteri yang diperoleh langsung dari tempe:Klebsiella135 ( Hanjaya, 2001) danBacillusGR9 (Wati, 2011) maupun yang merupakan isolat ATCC: Klebsiella pneumoniaeATCC 35657 (Koleksi FTB-UNIKA Atmajaya Jakarta) danBacillus cereusATCC10876 (Koleksi IPBCC, Bogor). Amplifikasi daerah intergenik gen 16S-23S rRNA dilakukan dengan menggunakan primer
ITSF/ITSFReub dan analisis ARISA dilakukan sesuai kondisi yang dikemukakan oleh Cardinaleet al.(2004). Analisis ini dilakukan di PT. Wilmar Benih
HASIL
Optimasi Metode Ekstraksi DNA Mikroba di Tempe
Kuantitas dan Kualitas DNA. Kuantitas dan kualitas DNA yang baik perlu diperoleh sebelum analisis metagenomik komunitas mikroba dilakukan. Dua metode ekstraksi DNA mikroba digunakan untuk memperoleh metode terbaik dalam mempelajari komposisi mikroba yang ada pada tempe. Hasil ekstraksi DNA mikroba dari dua metode tersebut selanjutnya diukur konsentrasi dan kualitasnya. Tabel 2 menunjukkan bahwa konsentrasi DNA yang tinggi diperoleh dari hasil ekstraksi dengan menggunakan metode PFMDIK. Hasil ekstraksi DNA genom dengan metode PFMDIK tervisualisasi sebagai pita yang smear dan sangat tipis pada gel agarose. DNA genom hasil ekstraksi dengan metode FDEK sama sekali tidak tervisualisasi walaupun keduanya divisualisasi dengan konsentrasi DNA yang sama sebesar 100 ng/μ l (data tidak ditampilkan). Konsentrasi DNA hasil ekstraksi juga menunjukkan hasil yang tidak berbias antar ulangan dibandingkan dengan metode FDEK. Kualitas DNA yang baik juga diperoleh dengan menggunakan metode PFMDIK. Rasio A260/280 menunjukkan nilai yang lebih tinggi dibandingkan dengan pada metode FDEK (Tabel 2). Kontaminasi protein lebih direduksi dengan melakukan ekstraksi metode PFMDIK. Nilai A260/230 yang diperoleh dari kedua metode ekstraksi tergolong sangat kecil (Tabel 2). Walaupun nilai ini pada hasil ekstraksi dengan metode PFMDIK sangat kecil dibandingkan dari metode FDEK, kontaminasi bahan organik tidak banyak berkontribusi sebagai inhibitor proses PCR (Gambar 4).
Tabel 2 Konsentrasi DNA dan rasio A260/280 dan A260/230 untuk hasil ekstraksi dengan menggunakan metode FDEK dan PFMDIK.
Sampel Rata-rata Kons DNA(ng/μ l) ± SD (n=3)
Rata-rata Rasio A260/280 ± SD (n=3)
Rata-rata Rasio A260/230 ± SD
(n=3)
TU-FDEK 3.47 ± 1.55 1.05 ± 0.20 0.95 ± 0.14
Uji Penghambatan PCR. Kualitas DNA hasil ekstraksi perlu dianalisis lebih lanjut untuk melihat kemungkinan terbawanya inhibitor PCR. Inhibitor PCR akan mengganggu proses amplifikasi sehingga gambaran komunitas mikroba yang ada pada suatu lingkungan menjadi terbatas. Amplifikasi DNA dilakukan terhadap gen 16S rRNA. Metode PFMDIK lebih memungkinkan diperolehnya hasil amplifikasi gen 16S rRNA yang lebih baik (Gambar 4). Hal ini menunjukkan bahwa inhibitor PCR lebih banyak tereduksi dengan menggunakan metode ini dibandingkan metode FDEK. Walaupun nilai A260/230 dari hasil ekstraksi dengan metode PFMDIK rendah (Tabel 2), tetapi inhibitor proses PCR asal makanan telah direduksi selama proses ekstraksi DNA. Kontaminasi bahan organik yang ada tidak berpengaruh terhadap proses PCR karena inhibitor PCR yang berasal dari makanan telah dihilangkan. Hasil amplifikasi gen 16S rRNA dari DNA hasil ekstraksi dengan metode FDEK sebaliknya akan semakin menipis bila templat DNA yang digunakan disimpan dalam waktu lama.
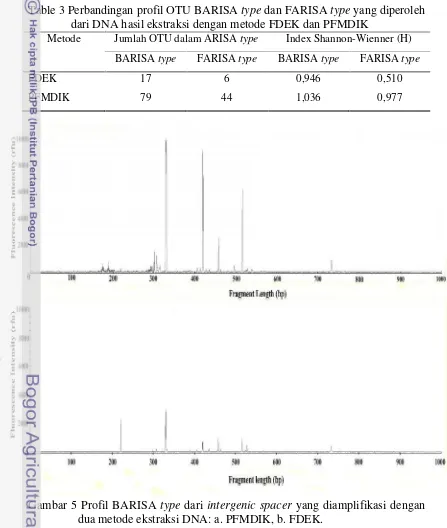
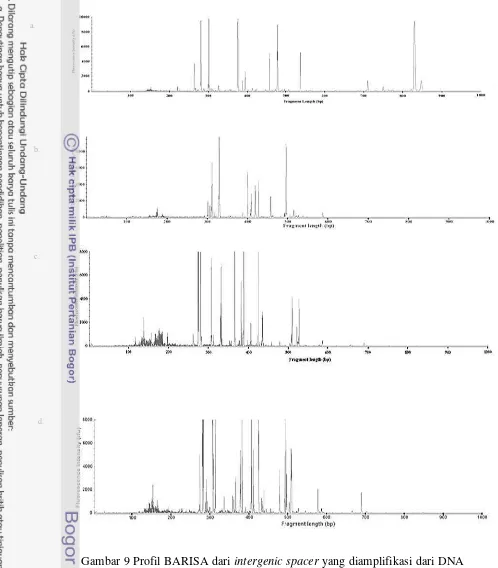
Validasi metode terbaik perlu dilakukan dengan melihat profil ARISA (Gambar 5 dan 6) yang dihasilkan oleh kedua metode. Metode PFMDIK lebih memberikan gambaran komunitas OTU mikroba pada tempe secara lebih baik (Tabel 3).
ProfilAutomated Ribosomal Intergenic Spacer Analysis(ARISA).
Nilai keragaman Index Shannon-Wienner (H’) yang lebih tinggi untuk keragaman OTU pada profil BARISA type maupun FARISA type diperoleh dari metode PFMDIK. Jumlah OTU dalam ARISAtype yang lebih baik juga diperoleh dengan metode PFMDIK (Tabel 3). OTU dalam ARISA type yang lebih bervariasi ditemukan dengan metode ekstraksi PFMDIK dibandingkan dengan metode FDEK (Gambar 5,6).
Table 3 Perbandingan profil OTU BARISAtypedan FARISAtypeyang diperoleh dari DNA hasil ekstraksi dengan metode FDEK dan PFMDIK
Metode Jumlah OTU dalam ARISAtype Index Shannon-Wienner (H)
BARISAtype FARISAtype BARISAtype FARISAtype
FDEK 17 6 0,946 0,510
PFMDIK 79 44 1,036 0,977
Gambar 5 Profil BARISA type dari intergenic spacer yang diamplifikasi dengan dua metode ekstraksi DNA: a. PFMDIK, b. FDEK.
a.
Gambar 6 Profil FARISA type dari daerah intergenic spacer yang diamplifikasi dengan dua metode ekstraksi DNA: a. PFMDIK, b. FDEK
Reprodusibilitas metode juga perlu dilakukan untuk melihat apakah metode yang digunakan menimbulkan bias. Ulangan analisis ARISA dilakukan untuk tiga tempe yang diproduksi selama tiga hari pada satu produsen. Metode PFMDIK memberikan gambaran profil ARISA reprodusibel dan peak dengan ukuran OTU yang sama dihasilkan untuk tiga hari ulangan (Gambar 7).
a.
Gambar 7 Profil BARISA type (tiga ulangan) dari intergenic spacer yang diamplifikasi dari DNA hasil ekstraksi dengan metode PFMDIK. a.
b.
Gambar 8 Profil FARISA type (tiga ulangan) dari intergenic spacer yang diamplifikasi dari DNA hasil ekstraksi dengan metode PFMDIK. a.
b.
Analisis Profil ARISATypeTempe dari Sejumlah Produsen Tempe
Variasi Proses Pembuatan Tempe dari sejumlah Produsen Tempe. Tempe diproduksi melalui proses fermentasi kacang kedelai dengan menggunakan inokulum sebagai starter. Walaupun menghasilkan produk yang sama dan umumnya dilakukan oleh produsen dengan latar belakang asal daerah Jawa, namun proses produksi tempe itu sendiri sangat bervariasi antar satu produsen dengan produsen yang lain.
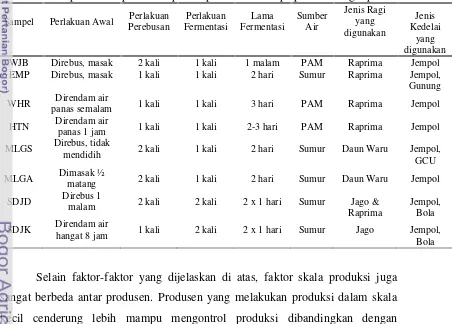
Proses pengolahan tempe dilakukan dengan cara yang sangat berbeda dengan urutan proses berbeda. Perlakuan awal terhadap biji kacang kedelai sampai perlakuan fermentasi dilakukan secara berbeda. Perlakuan awal pada produsen umumnya dimulai dengan proses perebusan kacang kedelai baik sampai matang maupun hanya setengah matang. Walau demikian beberapa produsen lebih memilih untuk merendam biji kacang kedelai saja sebagai tahap awal proses. Proses perebusan dilakukan secara berbeda pula. Pada produsen tertentu, proses perebusan dilakukan sebelum perendaman dan sesudah perendaman. Pada produsen lain proses perebusan hanya dilakukan sebelum perendaman atau sesudah perendaman saja. Lama proses perendaman juga sangat bervariasi antar produsen dan berkisar antara beberapa jam hingga semalam (Tabel 4).
menggunakan sumber air dari Perusahaan Air Minum (PAM) sedangkan produsen lain menggunakan sumber air dari sumur. Proses fermentasi tempe yang dilakukan oleh produsen di Sidoarjo juga berbeda dari produsen lain. Proses fermentasi dilakukan dua kali yaitu di dalam wadah yang tertutup rapat dan fermentasi di atas rak (Tabel 4). Proses ini dilakukan dengan harapan mendapatkan tempe dengan fungi yang tumbuh seperti kapas. Tekstur tempe yang dihasilkan akan terlihat lebih mudah hancur dan bila diproses dengan menggoreng, tempe akan menyusut. Tekstur ini berbeda dengan tekstur tempe dari malang yang padat karena kedelai difermentasi dengan ditindih oleh marmer atau batu bata. Tahapan proses dan kultur starter yang berbeda tentu saja sangat berpengaruh terhadap hasil akhir bervariasi karena membuka peluang yang berbeda bagi masuknya mikroba dalam proses fermentasi tempe.
Tabel 4 Profil perbedaan perlakuan proses pembuatan tempe pada berbagai produsen
Sampel Perlakuan Awal Perlakuan Perebusan Perlakuan Fermentasi Lama Fermentasi Sumber Air Jenis Ragi yang digunakan Jenis Kedelai yang digunakan WJB Direbus, masak 2 kali 1 kali 1 malam PAM Raprima Jempol EMP Direbus, masak 1 kali 1 kali 2 hari Sumur Raprima Jempol, Gunung
WHR Direndam air
panas semalam 1 kali 1 kali 3 hari PAM Raprima Jempol
HTN Direndam air
panas 1 jam 1 kali 1 kali 2-3 hari PAM Raprima Jempol
MLGS Direbus, tidak
mendidih 2 kali 1 kali 2 hari Sumur Daun Waru Jempol, GCU
MLGA Dimasak ½
matang 2 kali 1 kali 2 hari Sumur Daun Waru Jempol
SDJD Direbus 1
malam 2 kali 2 kali 2 x 1 hari Sumur Jago & Raprima
Jempol, Bola
SDJK Direndam air
hangat 8 jam 1 kali 2 kali 2 x 1 hari Sumur Jago Jempol, Bola
produsen seperti EMP, MLGA dan SDJD agak kurang memperhatikan kebersihan lingkungan proses produksi. Beberapa tahapan pengolahan bahkan dilakukan di daerah dengan peluang kontaminasi tinggi seperti pada WC. Bahan baku kedelai yang digunakan juga bervariasi. Selain itu untuk mengurangi bahan baku dan untuk alasan produk yang lebih baik pemakaian bahan tambahan seperti beras, tepung beras dan jagung putih dilakukan. Penggunaan bahan tambahan akan berpengaruh terhadap komunitas mikroba yang terdapat dalam proses fermentasi dan produk akhir.
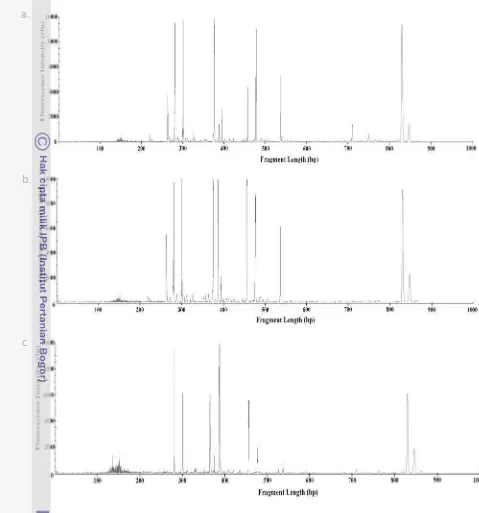
Gambar 9 Profil BARISA dariintergenic spacer yang diamplifikasi dari DNA hasil ekstraksi dari beberapa tempe a. EMP (Bogor), b. WJB (Bogor), c. MLGA (Malang), dan d. MLGS (Malang).
a.
b.
c.
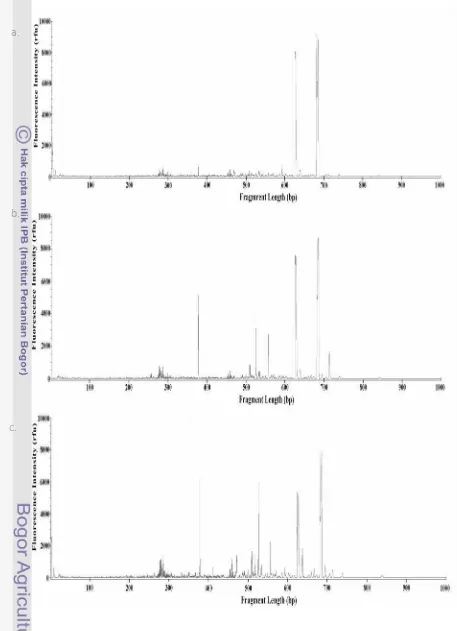
Gambar 10 Profil BARISA dari intergenic spaceryang diamplifikasi dari DNA hasil ekstraksi dari beberapa tempe a. HTN (Ambon), b. WHR (Ambon), c. SDJD (Sidoarjo) dan d. SDJK (Sidoarjo). a.
b.
c.
Gambar 11 Profil FARISA dari intergenic spacer yang diamplifikasi dari DNA hasil ekstraksi dari beberapa tempe a. EMP (Bogor), b. WJB (Bogor), c. MLGA (Malang), dan d. MLGS (Malang). a.
b.
c.
Gambar 12 Profil FARISA dari intergenic spacer yang diamplifikasi dari DNA hasil ekstraksi dari beberapa tempe a. HTN (Ambon), b. WHR (Ambon), c. SDJD (Sidoarjo) dan d. SDJK (Sidoarjo).
Profil OTU ARISA type sangat terbatas untuk menduga kelimpahan dan dominansi spesies, walaupun demikian keragaman OTU dapat dihitung untuk melihat pengaruh perbedaan proses fermentasi terhadap profil ARISA (Yannarell a.
b.
c.
dan Triplett 2005). Profil ARISAtypeyang diperoleh selanjutnya dianalisis untuk melihat keragaman OTU dalam komunitas mikroba pada tempe. Keragaman OTU yang dianalisis berdasarkan profil BARISA type dan FARISA type (Tabel 5) menunjukkan bahwa keragaman OTU tertinggi dalam komunitas pada BARISA
type yang ditunjukkan oleh nilai H’ (Index Shannon-Wienner) ditemukan pada
[image:54.595.103.512.0.829.2]produsen di wilayah Malang dan Sidoarjo. Keragaman OTU dalam komunitas fungi pada tempe WJB dan HTN tergolong rendah dan keragaman OTU fungi yang tinggi diperoleh pada tempe di daerah Malang (MLGA, MLGS), tempe SDJK di Sidoarjo dan tempe EMP di Bogor. Index Simpson (D) untuk semua komunitas fungi relatif rendah. Nilai D (Index Simpson) OTU bakteri pada Tabel 5 menunjukkan bahwa OTU komunitas bakteri lebih kecil dari nilai D (Index Simpson) OTU fungi. Hal ini berarti OTU komunitas bakteri lebih beragam dibandingkan OTU fungi. Data ini mendukung nilai indeks H’ dan menunjukkan adanya keragaman OTU pada profil ARISA.
Tabel 5 Keragaman OTU pada BARISA type dan FARISA type dari Berbagai Tempe Sampel Asal Sampel BARISA FARISA Index Shannon-Wienner (H’)
Index Simpson (D) Jumlah OTU dalam “BARISA type” Index Shannon-Wienner (H’)
Index Simpson (D) Jumlah OTU dalam “FARISA type”
WJB Bogor 1,34 0,11 83 0,88 0,22 36
EMP Bogor 1,17 0,09 46 1,46 0,11 131
HTN Ambon 1,09 0,11 37 0,71 0,30 29
WHR Ambon 1,22 0,11 66 1,09 0,21 46
MLGS Malang 1,61 0,04 104 1,62 0,05 145
MLGA Malang 1,53 0,05 93 1,57 0,06 145
SDJD Sidoarjo 1,57 0,04 90 1,39 0,08 104
SDJK Sidoarjo 1,62 0,04 91 1,50 0,07 124
menunjukkan bahwa ada tidaknya OTU bakteri dalam suatu profil BARISA menyebabkan terbentuknya dua kelompok besar yang terbentuk yaitu kelompok tempe EMP dan kelompok dengan tujuh tempe lain (Gambar 13). Pada kelompok dengan tujuh tempe, terlihat bahwa kelompok ini membentuk dua kelompok utama lagi dimana komunitas bakteri pada tempe WHR telihat sama dengan tempe MLGA. Komunitas bakteri pada tempe HTN lebih memiliki kecenderungan kemiripan dengan tepe WHR dan MLGA. Tempe SDJD dan SDJK memiliki komunitas bakteri yang mirip. Tempe MLGS juga memiliki kecederungan kemiripan komunitas dengan tempe SDJD dan SDJK.
Gambar 13 Pohon filogenetik hubungan keterkaitan delapan komunitas bakteri pada delapan tempe berdasarkanSorensen’s Similarity coefficient
Profil FARISAtype (Gambar 14) menunjukkan adanya dua pengelompokan yang utama yaitu kelompok dengan tempe HTN dan tujuh tempe lain. Pengelompokan tujuh tempe yang ada menunjukkan adanya dua sub pengelompokkan lagi yang menunjukkan kemiripan komunitas fungi pada tempe. Tempe MLGA memiliki komunitas yang lebih mirip dengan tempe EMP sedangkan tempe SDJD memiliki kemiripan komunitas fungi dengan tempe SDJK. Komunitas MLGS memiliki kemiripan komunitas yang sama dengan keempat tempe di atas. Tempe WJB sebaliknya lebih memiliki kemiripan komuitas dengan tempe WHR.
Gambar 14 Pohon filogenetik hubungan keterkaitan delapan komunitas fungi pada delapan tempe berdasarkanSorensen’s Similarity coefficient
Bila komunitas fingerprinting BARISAtypedan FARISAtype dibandingkan dan akan dipilih sebagai barcode tempe, maka profil BARISA terlihat lebih diskriminatif. Fingerprinting BARISA type lebih diskriminatif memilah tempe berdasarkan asal produsen tempe karena keragaman OTU BARISA type yang
lebih tinggi. Nilai koefisien Sorensen’s komunitas bakteri yang relative lebih kecil
dibandingkan komunitas fungi menunjukkan bahwa OTU bakteri yang beragam masih mampu memilah tempe secara lebih diskriminatif. Keragaman yang rendah
pada komunitas fungi disebabkan karena ada tumpang tindih karena adanya OTU yang berulang dalam komunitas OTU FARISA type dibandingkan OTU pada bakteri (Lampiran 2 dan 3). Kesamaan OTU yang muncul menunjukkan bahwa proses fermentasi tempe diperantarai oleh kelompok fungi yang sama.
Analisis Gen 16S rRNA yang Berasal dari Komunitas Bakteri pada Tempe Identifikasi Bakteri yang Melimpah pada Produsen yang Berbeda. Identifikasi spesies bakteri yang ada pada tempe, perlu dilakukan dengan pendekatan pembuatan pustaka gen 16S rRNA. Identifikasi ini bertujuan untuk melihat apakah komposisi spesies bakteri yang terlibat dalam proses fermentasi pada produsen benar-benar berbeda sesuai hasil yang tergambarkan pada Gambar 13. Tiga puluh koloni E. coli DH5α yang membawa gen 16S rRNA dari total komunitas bakteri dan terinsersi pada vector pGEM-T Easy selanjutnya dipilih. Insert gen 16S rRNA selanjutnya diambil dan disekuensing (Lampiran 6). Komposisi bakteri pada tempe dari produsen SDJD dan EMP benar-benar berbeda. Bakteri pada tempe dari produsen SDJD lebih didominasi olehKlebsiella dan uncultured Klebsiella (Gambar15). Tempe pada produsen EMP lebih didominasi oleh bakteri dari genus Acetobacter dan Lactobacillus (Gambar 16). Acetobacter merupakan bakteri yang baru pernah dilaporkan terlibat pada proses fermentasi tempe.
Perbedaan komunitas bakteri ini tentu saja akan berimplikasi terhadap karakteristik tempe yang dihasilkan. Berdasarkan hasil penelitian ini, diduga bahwa bakteri turut berperan dalam perbedaan karakteristik, komposisi Gizi, dan flavor pada tempe.
Analisis Profil ARISA Isolat Tunggal Bakteri, Suatu Upaya Analisis Keamanan Pangan.
[image:58.595.119.518.78.352.2]Profil ARISA Isolat Bacillus.Bakteri seperti Bacillus cereusmerupakan bakteri yang bersifat patogen karena menghasilkan toksin. Analisis keamanaan pangan perlu dilakukan untuk melihat kemiripan bakteri yang ditemukan pada tempe dengan bakteri dari ATCC (American type culture collection). Hasil menunjukkan bahwa isolat Bacillus GR9 yang telah teridentifikasi sebagai Bacillus cereusberdasarkan sekuen gen 16S rRNA-nya ternyata merupakan isolat yang berbeda secara genetik dengan Bacillus cereus ATCC10876 (Tabel 6, Gambar 17). Isolat BacillusGR9 menunjukkan profil BARISA type dengan OTU yang lebih banyak dibandingkan dengan Bacillus cereus ATCC10876 (Tabel 6). Hasil yang diperoleh juga menunjukkan adanya OTU dengan ukuran berbeda yang tidak ditemukan pada kedua isolat. Perbedaan ini merupakan ciri pembeda isolat dan OTU yang sama dapat menjadi ciri spesifik bakteri dari genusBacillus.
Tabel 6 Ukuran profil BARISA isolat tunggalBacillus
BacillusGR9 Bacillus cereusATCC10876 Ukuran OTU dalam
‘BARISAtype’
Luas Area Ukuran OTU
dalam‘BARISA
type’
Luas Area
226, 03 ± 0,02 58704,33 ± 7158,24 225,93 ± 0,03 55136,33 ± 2596,88
227,11 ± 0,12 89288,33 ± 7281,73 226,93 ± 0,02 7810 ± 258,28
454,63 ± 0,00 13881,67 ± 3855,40 451,97 ± 0,04 9164,67 ± 1058,82
455,68 ± 0,01 12245,67 ± 3564,63
Profil ARISA Isolat Klebsiella. Secara medis bakteri dari genus Klebsiella merupakan bakteri yang membahayakan kesehatan karena berkontribusi terhadap timbulnya penyakit paru-paru. Hasil menunjukkan bahwa walaupun Klebsiella 135 asal tempe telah teridentifikasi sebagai Klebsiella pneumoniae, namun secara genetik bakteri ini berbeda dengan Klebsiella pneumoniae ATCC35657 (Tabel 7, Gambar 18). Klebsiella 135 menunjukkan profil BARISA type yang sangat berbeda dari Klebsiella pneumoniae ATCC35657 dalam hal jumlah maupun ukuran OTU yang ada (Tabel 7). Analisis ini memang memberi gambaran yang sangat berbeda namun konfirmasi untuk jaminan keamanan pangan masih perlu dilakukan dengan melihat keragaman schyzotypeyang lebih diskriminatif dan deteksi adanya gen patogen spesifik pada bakteri-bakteri ini.
Tabel 7 Ukuran profil BARISA isolat tunggalKlebsiella
Klebsiella 135 Klebsiella pneumonia ATCC35657
Ukuran‘BARISA
type’
Luas Area Ukuran‘BARISA
type’
Luas Area
287,48 ± 0,06 37690,33 ± 5467,20 280,10 ± 0,00 14433,67 ± 1038,05
430,83 ± 0,14 106966,67 ± 4292,73 423,04 ± 0,00 44127,33 ± 3038,69
507,88 ± 0,17 6108,33 ± 491,73 431,13 ± 0,01 13959,00 ± 917,20
508,96 ± 0,19 23325,67 ± 1879,59 507,84 ± 0,04 13181 ± 1085,27
515,80 ± 0,23 29186,33 ± 1880,76 509,63 ± 0,06 2523,00 ± 2071,58
516,81 ± 0,21 11531,00 ± 1460,341
PEMBAHASAN
Optimasi Metode Ekstraksi DNA Mikroba di Tempe
Kuantitas dan Kualitas DNA. Secara ideal, metode ekstraksi DNA haruslah merupakan metode yang sederhana, cepat dan efisien. Memilih metode ekstraksi seringkali harus mempertimbangkan biaya yang dikeluarkan, hasil optimal yang diperoleh dan terbuangnya substansi yang dapat berpengaruh terhadap proses PCR. Walaupun demikian, metode yang dipilih haruslah metode yang benar-benar baik untuk sampel yang spesifik dengan kriteria utama yang harus dipenuhi didasarkan pada kualitas dan kuantitas DNA yang dihasilkan (Jara et al. 2008). Kuantitas dan kualitas DNA bersifat sangat kritis untuk interpretasi analisis komunitas mikroba (Thakuriaet al. 2008).
Metode FDEK merupakan metode ekstraksi DNA yang relatif murah, tetapi metode ini tidak memberikan hasil lisis yang baik. Hal ini terlihat dari rendemen DNA yang lebih rendah dan kemurnian DNA yang berfluktuatif. Hasil sebaliknya menunjukkan bahwa walaupun lebih mahal, ekstraksi DNA menggunakan metode PFMDIK menghasilkan DNA dengan kualitas yang lebih baik untuk nilai rasio A260/280 walaupun tidak mencapai nilai standar 1.8 (Sambroek et al. 1989). Metode PFMDIK mampu memberikan rendemen dan kemurnian DNA dengan rasio A260/280 yang tinggi dan menghasilkan dua hingga tiga kali konsentrasi DNA yang lebih banyak dibandingkan dengan metode FDEK. Kontaminasi protein pada ekstrak DNA dengan metode PFMDIK lebih rendah daripada dengan metode FDEK. Metode FDEK menghasilkan rasio relatif rendah untuk nilai rasio A260/280 dan konta