DETEKSI Papaya ringspot virus ASAL TANAMAN PEPAYA
(Carica papaya L.) BERDASARKAN TEKNIK REVERSE
TRANSCRIPTION-POLYMERASE CHAIN REACTION
GITA SRI LESTARI
DEPARTEMEN PROTEKSI TANAMAN
FAKULTAS PERTANIAN
PERNYATAAN MENGENAI SKRIPSI DAN SUMBER
INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Deteksi Papaya ringspot virus Asal Tanaman Pepaya (Carica papaya L.) Berdasarkan Teknik
Reverse Transcription-Polymerase Chain Reaction adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Juli 2014
Gita Sri Lestari
ABSTRAK
GITA SRI LESTARI. Deteksi Papaya ringspot virus Asal Tanman Pepaya (Carica papaya L.) Berdasarkan Teknik Reverse Transcription-Polymerase Chain Reaction. Dibimbing oleh SRI HENDRASTUTI HIDAYAT.
Papaya ringspot virus (PRSV) merupakan anggota genus Potyvirus yang dapat menyebabkan penyakit penting pada tanaman pepaya dan beberapa tanaman dari famili Cucurbitaceae. Penyebaran PRSV di Indonesia masih sangat terbatas, bahkan PRSV masih tergolong OPTK kategori A1 berdasarkan Peraturan Menteri Pertanian Nomor.93/Permentan/OT.140/12/2011. Penelitian ini dilakukan untuk mendeteksi PRSV yang menyebabkan penyakit bercak cincin pada tanaman pepaya di Bogor, Jawa Barat dan untuk menyeleksi primer yang dapat mengamplifikasi DNA target secara konsisten dengan teknik RT-PCR. Penelitian diawali dengan kegiatan survei lapangan dan pengambilan contoh tanaman sakit, diikuti dengan deteksi menggunakan metode RT-PCR dan penularan secara mekanis pada tanaman pepaya sehat. Metode deteksi PRSV di laboratorium meliputi ekstraksi RNA total, sintesis cDNA, dan amplifikasi DNA menggunakan tiga jenis primer yaitu primer universal Potyvirus MJ1/MJ2 yang akan mengamplifikasi protein selubung Potyvirus dan dua pasang primer spesifik PRSV yaitu PRSV1298/PRSV1942 dan PRSV326/PRSV800 yang akan mengamplifikasi berturut-turut bagian gen Nib (nuclear inclusion protein) dan gen protein selubung PRSV. Pertanaman pepaya di lapangan menunjukkan gejala mosaik pada bagian daun, tangkai daun, batang dan buah dengan tingkat kejadian penyakit 20% hingga 100%. Semua sampel tanaman sakit dari Bogor berhasil ditularkan melalui penularan secara mekanis, walaupun hanya menunjukkan gejala mosaik ringan. Sampel tanaman sakit asal Bogor terbukti terinfeksi PRSV berdasarkan hasil deteksi dengan metode RT-PCR. Primer spesifik PRSV326/PRSV800 dapat mengamplifikasi DNA target dengan ukuran 475 bp dengan baik dan dapat disarankan sebagai primer untuk mendeteksi PRSV.
ABSTRACT
GITA SRI LESTARI. Detection of Papaya ringspot virus on Papaya (Carica papaya
L.) Based on Reverse Transcription-Polymerase Chain Reaction Technique.
Supervised by SRI HENDRASTUTI HIDAYAT
Papaya ringspot virus (PRSV) is a member of Potyvirus genus, it causes important disease on papaya and some species of Cucurbitaceae family. The spread of PRSV in Indonesia is still very limited, even PRSV is still classified as quarantine pest based on the Decree of Minister of Agriculture No.93/ Permentan /OT.140/12/2011. The aims of this research was to detect PRSV from papaya cultivation showing ringspot symptoms in Bogor, West Java and to determine the best primers for RT-PCR-based detection method. Research activities involved field survey and sample collection, followed by detection using Reverse-Transcription Polymerase Chain Reaction (RT-PCR) method and transmission by mechanical inoculation method to healthy papaya plants. Detection of PRSV in the laboratory involved total RNA extraction, cDNA synthesis, and DNA amplification using three primer pairs, i.e. universal Potyvirus MJ1/MJ2 which amplified Potyvirus coat protein, and two pairs of specific primers PRSV1298/PRSV1942 and PRSV326/PRSV800 which amplified the Nib gene (nuclear inclusion protein) and the PRSV coat protein gene, respectively. Mosaic symptoms on leaves, stems and fruits was observed in the field with disease incidence 20% to100%. All samples from Bogor was successfully transmitted by mechanical inoculation although most of them developed only mild symptoms. Detection using RT-PCR confirmed the infection of PRSV in all samples from Bogor. Amplificationn of DNA target was best achieved using PRSV-specific primer PRSV326/PRSV800 with 475 bp DNA fragmen as amplification products. Therefore, these primers are recommended for detection of PRSV.
DETEKSI Papaya ringspot virus ASAL TANAMAN PEPAYA
(Carica papaya L.) BERDASARKAN TEKNIK REVERSE
TRANSCRIPTION-POLYMERASE CHAIN REACTION
GITA SRI LESTARI
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Pertanian
pada
Departemen Proteksi Tanaman
DEPARTEMEN PROTEKSI TANAMAN
FAKULTAS PERTANIAN
INSTITUT PERTANIAN BOGOR
BOGOR
Judul Usulan : Deteksi Papaya ringspot virus Asal Tanaman Pepaya (Carica papaya) Berdasarkan Teknik Reserve Transcription- Polymerase Chain Reaction
Nama Mahasiswa: Gita Sri Lestari NRP : A34100050
Disetujui oleh
Prof Dr Ir Sri Hendrastuti Hidayat MSc Dosen Pembimbing
Diketahui oleh
Dr Ir Abdjad Asih Nawangsih MSi Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas berkat rahmat dan karunia-Nya sehingga skripsi ini dapat diselesaikan. Penelitian dengan judul Deteksi Papaya ringspot virus Asal Tanaman Pepaya (Carica papaya L.) Berdasarkan Teknik Reverse Transcription-Polymerase Chain Reaction ini dilaksanakan dari bulan Februari hingga Juni 2014.
Penulis mengucapkan terima kasih kepada Prof Dr Ir Sri Hendrastuti Hidayat MSc selaku dosen pembimbing akademik dan dosen pembimbing skripsi yang telah memberikan bimbingan selama kegiatan belajar mengajar serta pengarahan, saran, dan motivasi selama penelitian dan penulisan skripsi. Penulis juga menyampaikan terima kasih kepada Prof Dr Ir Dadang MSc selaku dosen penguji tamu yang telah memberikan banyak saran dalam proses penulisan skripsi. Terima kasih juga kepada seluruh staf pengajar IPB atas ilmu dan pengalaman berharga yang telah diberikan.
Penulis mengucapkan terima kasih juga kepada rekan-rekan di Laboratorium Virologi Tumbuhan atas kebersamaannya Kakak Sari Nurulita dan Kakak Nurul yang telah memberi bantuan dan saran selama pelaksanaan penelitian dan penulisan skripsi. Ungkapan terima kasih juga disampaikan kepada teman-teman Susilawati, Wirhatazia Enbia LC, Rizky Marcheria, Egi Puspita Sari yang telah membantu penulis dalam mengumpulkan data dan seluruh rekan-rekan Proteksi Tanaman angkatan 47.
Kepada ayah, ibu, kakak, dan adik yang telah mencurahkan kasih sayang, doa, dan dukungan baik moril maupun materil, karya ini semoga menjadi persembahan kecil dari ananda. Penulis berharap semoga hasil penelitian ini nantinya dapat memberikan manfaat bagi dunia pendidikan dan ilmu pengetahuan.
DAFTAR ISI
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 3
Manfaat Penelitian 3
BAHAN DAN METODE 4
Tempat dan Waktu Penelitian 4
Metode Penelitian 4
Pengambilan Sampel Tanaman Pepaya Bergejala 4 Penghitungan Kejadian Penyakit di Lapangan 4
Persiapan Media Tanam dan Tanaman Pepaya 4
Penularan Virus Secara Mekanis 4
Deteksi PRSV dengan Teknik RT-PCR 5
HASIL DAN PEMBAHASAN 9
Gejala dan Kejadian Penyakit pada Tanaman Pepaya di Lapangan 9
Inokulasi Virus Secara Mekanis 12
Deteksi PRSV dengan MetodeRT-PCR 14
SIMPULAN 20
SARAN 20
DAFTAR TABEL
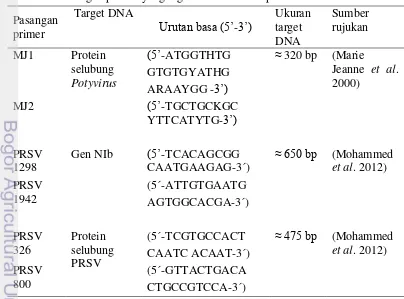
1 Komposisi bahan RT-PCR 6
2 Komposisi bahan PCR 7
3 Pasangan primer yang digunakan untuk amplifikasi DNA PRSV 7 4 Hasil pengamatan tingkat kejadian penyakit di daerah Bogor 9
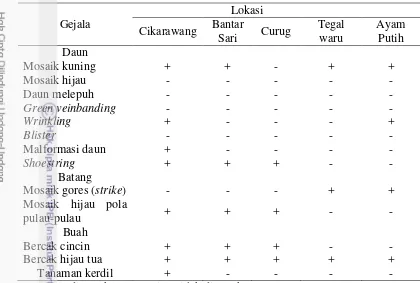
5 Variasi gejala pada tanaman pepaya 10
DAFTAR GAMBAR
1 Gejala pada daun, batang atau tangkai daun, dan buah pada berbagai lokasi (a) mosaik pada daun dan batang, bercak cincin pada buah di Desa Tegal Waru, (b) mosaik pada daun, tangkai daun, dan bercak hijau tua pada buah di Desa Cikarawang, (c) daun seperti tali sepatu (shoestring), mosaik pada batang, dan bercak cincin pada buah di Desa Bantar Sari, (d) daun seperti tali sepatu (shoestring), mosaik pada batang, bercak cincin pada buah di Desa Curug, (e) mosaik kuning, mosaik ringan pada batang, mosaik pada buah di Desa Ayam
Putih 11
2 Gejala infeksi PRSV hasil inokulasi isolat Nanggroe Aceh Darussalam pada tanaman pepaya varietas Orange Lady; (a) daun sehat, (b) mosaik kuning dan pinggiran daun mengeriting, (c) malformasi daun muda dan daun menyerupai tali sepatu (shoestring),
(d) mosaik bergores pada batang , (e) tanaman kerdil 13 3 Gejala infeksi PRSV hasil inokulasi isolat; (a) kontrol, (b) Nanggroe
Aceh Darussalam (c), Desa Tegal Waru (d), Desa Cikarawang (e) Desa Bantar Sari, (f) Curug, (g) Desa Ayam Putih, (h) tinggi tanaman hasil inokulasi, kontrol (h1); kontrol (h2); Desa Tegal Waru,Bogor (h3); Desa Situgede, Bogor (h3); Desa Bantar Sari, Bogor (h4); Desa Curug, Bogor (h5); Nanggroe Aceh Darussalam
(h6); Desa Ayam Putih, Kebumen (h7) 14
4 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2), PRSV326 /PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Nanggroe Aceh Darussalam. Pada masing-masing gel agarosa disertakan DNA (M), kontrol
positif DNA (+) dan kontrol negatif tanpa DNA (-) 15 5 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2), PRSV326/PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Tegal Waru, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol
positif DNA (+) dan kontrol negatif tanpa DNA (-) 16 6 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
PRSV326/PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Cikarawang, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol
positif DNA (+) dan kontrol negatif tanpa DNA (-) 16 7 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2), PRSV326/PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Bantar Sari, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol
positif DNA (+) dan kontrol negatif tanpa DNA (-) 17 8 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2), PRSV326 /PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Curug, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif
DNA (+) dan kontrol negatif tanpa DNA (-) 17
9 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2), PRSV326 /PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Ayam Putih, Kebumen. Pada masing-masing gel agarosa disertakan DNA (M),
kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-) 18 10 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
mengunakan primer spesifik PRSV326/PRSV800. Sampel DNA berasal dari tanaman hasil penularan secara mekanis masing-masing isolat: Nanggroe Aceh Darussalam (sebagai kontrol positif) (A); Tegal Waru, Bogor (B); Cikarawang, Bogor (C); Bantar Sari, Bogor (D); Curug, Bogor (E); Ayam Putih, Kebumen (F). Penanda DNA
1
PENDAHULUAN
Latar Belakang
Pepaya (Carica papaya L.) merupakan tanaman yang berasal dari daerah beriklim tropis di Amerika dan penyebaran awalnya diduga berada didaerah sekitar Meksiko bagian selatan dan Nikaragua (Balitbu 2008). Indonesia adalah negara produsen buah pepaya nomor tiga di dunia setelah India dan Brazil pada tahun 2009-2012. Produksi buah pepaya cenderung meningkat dari tahun ke tahun hingga mencapai 906 312 ton pada tahun 2012 (FAOSTAT 2014).
Di Indonesia pepaya dikonsumsi sebagai buah segar karena bermanfaat bagi pelancar sistem pencernaan dan kandungan gizi yang tinggi. Pepaya kaya akan antioksidan (karoten, vitamin C, flavonoid), vitamin B (folat dan asam pantotenat), mineral (potasium dan magnesium), dan serat. Pepaya juga menghasilkan enzim papain yang banyak digunakan dalam kebutuhan industri untuk bahan pelunak daging, farmasi, produk kecantikan, kosmetik serta berperan sebagai stabilizer
pada pembuatan minuman bir (UF 2012) .
Pepaya tumbuh dan berkembang di daerah beriklim tropis dan subtropis (da Silva et al.2007). Di Indonesia tanaman pepaya umumnya tumbuh menyebar dari dataran rendah hingga dataran tinggi mencapai 1 000 m di atas permukaan laut. Secara umum tanaman pepaya dapat tumbuh pada berbagai jenis tanah dengan kisaran pH 6.5 sampai 7, dengan curah hujan 1 500 hingga 2 000 mm per tahun (Balitbu 2008). Budi daya tanaman pepaya banyak mendapat tantangan karena gangguan dari berbagai organisme pengganggu tanaman (OPT), baik dari golongan hama maupun patogen. Beberapa hama penting yang menyebabkan kerusakan pada tanaman pepaya yaitu dari golongan tungau (Polyphagotarsonemus latus, Tetranychus telarius, Brevipalpus ponicis), kutudaun (Myzus persicae), kutu putih (Aonidiella orientalis), lalat (Bactrocera dorsalis) dan trips (Trips tabaci) (Balitbu 2008). Patogen yang dilaporkan menginfeksi tanaman pepaya berasal dari golongan cendawan (Phytophthora palmivora, Collectotrichum gloesporiodes, Pythium sp., Oidium caricae,
Botryodiplodia theobromae) (da Silva et al. 2007), bakteri (Erwinia papayae) (Deptan 2006), dan virus (Papaya lethal yellowing virus, Papaya meleira virus,
Papaya apical necrosis virus, dan Papaya ringspot virus) (da Silva et al. 2007).
Papaya ringspot virus (PRSV) penyebab penyakit bercak cincinmerupakan salah satu penyakit penting yang menjadi faktor pembatas produksi pepaya secara global (Tripathi et al. 2008; Gonsalves et al. 2010). PRSV merupakan virus dari famili Potyviridae dan genus Potyvirus. PRSV terdiri dari dua strain berbeda yaitu strain W dan P yang dapat dibedakan secara biologi berdasarkan kemampuannya menginfeksi tanaman inang. PRSV-W hanya menginfeksi tanaman dari famili Cucurbitaceae, sedangkan PRSV-P menginfeksi tanaman dari famili Caricaceae dan Cucurbitaceae (Bateson et al. 2002; Tripathi et al. 2008; Gonsalves et al. 2010). Penularan virus secara alami di lapangan dibantu oleh beberapa spesies kutudaun secara non-persisten. Virus ini juga dapat ditularkan secara mekanis dan bukan merupakan patogen terbawa benih (Tripathi et al. 2008; Gonsalves et al. 2010).
2
(veinclearing), shoestring (daun seperti tali sepatu) pada daun yang baru muncul, warna hijau tua disepanjang tulang daun (green veinbanding), tanaman kerdil, bewarna hijau tua dan sedikit cekung berbentuk cincin pada buah, dan terdapat banyak strike oily-looking (mosaik seperti garis) pada batang. Buah yang dihasilkan setelah tanaman terinfeksi biasanya berukuran kecil, menunjukkan bekas seperti berlumut, bercak berbentuk cincin, terlihat tonjolan-tonjolan tidak rata, produksi buah menurun dan buah memiliki rasa yang tidak enak karena kandungan gula berkurang hingga 50%. Tanaman yang terinfeksi pada usia muda akan menjadi kerdil dan tidak akan menghasilkan buah (Agrios 2005; Tripathi et al. 2008; Gonsalves et al. 2010).
Infeksi PRSV pada tanaman pepaya pertama dilaporkan di Hawai pada tahun 1949 (Gonsalves et al. 2010). Strain PRSV yang sama kemudian dilaporkan di Saipan, Kepulauan Mariana Utara dan Guam, Polinesia (Davis et al. 2005) dan Afrika Utara (Diallo et al. 2007). Strain PRSV yang menginfeksi tanaman dari famili Cucurbitaceae (PRSV-W) dilaporkan di Australia 1991 dan Sudan pada tahun 2012 (Gonsalves et al. 2010; Mohammed et al.2012).
Di Indonesia PRSV merupakan OPT Karantina (OPTK) kategori A1 berdasarkan Peraturan Menteri Pertanian Nomor.93/Permentan/OT.140/12/2011, karena keberadaannnya belum dilaporkan. Walaupun demikian terdapat informasi yang menyatakan bahwa PRSV telah ada di Indonesia yaitu di Daerah Istimewa Yogyakarta serta sikuen basa nukleotida PRSV asal Indonesia telah terdaftar pada
GenBank dengan nomor aksesi AF374865.1. Pada tahun 2012 dilaporkan keberadaan PRSV di daerah Nanggroe Aceh Darussalam yang memperkuat keberadaan PRSV di Indonesia (Hidayat et al. 2012). Penelitian terkait survei pada pertanaman pepaya di daerah lain diperlukan untuk mengetahui daerah penyebaran PRSV. Metode deteksi yang tepat sangat diperlukan untuk memastikan keberadaan PRSV di wilayah Negara Indonesia.
Deteksi dan identifikasi virus dapat dilakukan berdasarkan karakter biologi dan molekuler. Deteksi berdasarkan karakter biologi dapat dilakukan melalui pengamatan gejala pada tanaman inang atau melalui penularan dengan cara penyambungan, inokulasi mekanis, atau oleh vektor. Deteksi dan identifikasi berdasarkan karakter molekuler umumnya dilakukan dengan dua cara, yaitu berdasarkan sifat protein dengan uji serologi dan sifat asam nukleat (Agrios 2005). Identifikasi berdasarkan gejala merupakan data pertama yang diperlukan untuk identifikasi virus. Namun, identifikasi berdasarkan gejala saja sering membingungkan karena gejala yang timbul dipengaruhi oleh strain virus, jenis tanaman dan faktor lingkungan (Akin 2006). Diperlukan metode identifikasi dan deteksi baik secara serologi maupun secara molekuler
3
Tujuan Penelitian
Penelitian ini bertujuan untuk mendeteksi PRSV yang menyebabkan penyakit bercak cincin pada pepaya di Bogor dengan teknik reverse transcription-polymerase chain reaction (RT-PCR), serta melakukan seleksi primer yang dapat digunakan untuk deteksi dengan teknik RT-PCR.
Manfaat Penelitian
4
BAHAN DAN METODE
Tempat dan Waktu Penelitian
Survei penyakit dan pengambilan sampel tanaman sakit dilakukan di daerah pertanaman pepaya di Bogor. Identifikasi virus dilakukan di Laboratorium Virologi Tumbuhan, Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor. Penelitian dilaksanakan dari bulan Februari 2014 hingga Juni 2014.
Metode Penelitian
Pengambilan Sampel Tanaman Pepaya Bergejala
Sampel tanaman diambil dan diperoleh dari kebun pepaya di Bogor, yaitu (1) Desa Cikarawang, Kecamatan Dramaga (2) Desa Bantar Sari, Kecamatan Rancabungur (3) Desa Curug, Kecamatan Bogor Barat. Melalui korespondensi diperoleh sampel tanaman dari Desa Tegal Waru, Kecamatan Ciampea, Bogor dan Desa Ayam Putih, Kecamatan Bulus Pesantren, Kabupaten Kebumen. Sampel yang diambil adalah bagian tanaman (daun, batang, buah) yang menunjukkan gejala penyakit berupa mosaik, malformasi dan bercak cincin (ringspot).
Deskripsi gejala dan dokumentasi dengan kamera digital dilakukan untuk masing-masing sampel lapangan. Sampel daun, batang dan buah dari lapangan dibawa ke laboratorium, dibersihkan, dipotong kecil-kecil dan ditimbang sebanyak 0.1 g, lalu dibungkus dan disimpan pada suhu -80 ˚C atau langsung digunakan untuk bahan RT-PCR atau penularan virus ke tanaman sehat.
Penghitungan Kejadian Penyakit di Lapangan
Pengamatan tanaman di lapangan dilakukan secara visual yaitu dengan mengamati gejala pada bagian batang, daun, dan buah tanaman pepaya. Pengamatan kejadian penyakit di lapangan dilakukan terhadap setiap pohon yang ditemui pada masing-masing kebun pepaya. Penghitungan kejadian penyakit di lapangan mengikuti rumus sebagai berikut :
KP =
dengan KP, kejadian penyakit; n, jumlah tanaman yang menunjukkan gejala; N, jumlah tanaman yang diamati.
Persiapan Media Tanam dan Tanaman Pepaya
Media tanam yang digunakan terdiri atas campuran tanah dan pupuk kandang steril dengan perbandingan 2:1 (b:b) dan ditempatkan pada polybag
berukuran 20 cm x 25 cm. Tanaman pepaya yang digunakan untuk uji penularan adalah tanaman pepaya varietas Orange lady dan varietas Calina.
Penularan Virus Secara Mekanis
5 gejala yang muncul dan periode inkubasi virus, serta pemurnian virus dari tanaman sakit yang diperoleh dari lapangan. Sebagai kontrol positif pada percobaan penularan digunakan isolat PRSV asal Nanggroe Aceh Darussalam yang merupakan koleksi Laboratorium Virologi Tumbuhan, Institut Pertanian Bogor.
Penularan virus secara mekanis dilakukan dengan pengolesan cairan perasan (sap) tanaman sakit pada pepaya varietas Orange lady berumur 42 HST (hari setelah tanam) dan Calina berumur 14 HST. Daun pepaya yang terinfeksi ditimbang sebanyak 0.1 g kemudian digerus dan ditambahkan bufer fosfat (61.5 ml K2HPO4 1M dan 38.5 ml KH2PO4 yang dilarutkan dalam 900 ml aquades) pH 7 yang mengandung 1% β-merkaptoetanol sebanyak 500 µl. Sap tanaman dioleskan pada permukaan daun pepaya yang telah ditaburi karborundum 600 mesh lalu dibilas dengan air untuk menghilangkan sisa karborundum.
Deteksi PRSV dengan Teknik Reverse-Transcription Polymerase Chain
Reaction (RT-PCR)
Metode deteksi dengan RT-PCR terdiri dari tahapan: ekstraksi RNA total, sintesis complementary DNA (cDNA), amplifikasi DNA target, dan visualisasi hasil amplifikasi.
Ekstraksi RNA total. Metode ekstraksi RNA dilakukan secara manual atau menggunakan kit ekstraksi.
Ekstraksi RNA secara manual mengikuti metode CTAB (Doyle dan Doyle 1990). Sebanyak 0.1 g sampel tanaman bergejala (daun, batang, dan kulit buah) digerus menggunakan nitrogen cair dan ditambahkan 500 µl bufer ekstraksi yang mengandung (1% 2-β-merkaptoetanol). Hasil gerusan dimasukkan kedalam tabung mikro 2 ml dan diinkubasi dalam penangas air pada suhu 65 ˚C selama 30 menit dan setiap 10 menit sekali dibolak-balik untuk membantu proses lisis. Setelah 30 menit tabung yang berisi ekstraksi tanaman diangkat dari penangas air dan didiamkan selama 2 menit pada suhu ruang, kemudian ditambahkan 500 µl campuran Chloroform: Isoamilalcohol (24:1). Agar tercampur dengan baik tabung divortek dengan kecepatan tinggi selama 5 menit, kemudian disentrifugasi dengan kecepatan 13 000 rpm selama 11 menit. Supernatan dipindahkan ke tabung baru dengan hati-hati, kemudian ditambahkan Isopropanol (volume sebanding dengan supernatan yang diperoleh). Tabung mikro dibolak-balik sehingga terlihat benang-benang RNA, lalu disentrifugasi dengan kecepatan 13 000 rpm selama 6 menit. Setelah disentrifugasi akan terlihat pelet RNA, campuran supernatan dan Isopropanol dipindahkan secara hati-hati sehingga menyisakan pelet RNA Pelet RNA yang diperoleh dicuci dengan etanol 70% sebanyak 500 µl. Pelet RNA yang telah ditambahkan etanol disentrifugasi dengan kecepatan 13 000 rpm selama 6 menit, etanol dibuang lalu tabung diletakkan secara terbalik diatas tisu selama 15 menit agar pelet kering. Pelet yang diperoleh dilarutkan dalam 50 µl bufer TE 1x (10 mM Tris-HCl pH 8.0 mM EDTA).
Ekstraksi RNA menggunakan kit mengikuti prosedur Thermo Scientific (EU,
Lithuania). Sebanyak 0.1 g sampel tanaman bergejala (daun, batang, dan kulit buah) digerus menggunakan nitrogen cair dan ditambahkan 500 µl Plant RNA
6
pada suhu 56 ˚C, lalu disentrifugasi dengan kecepatan 14 000 rpm selama 5 menit. Supernatan yang diperoleh diambil sebanyak 400 hingga 500 µl secara hati-hati tanpa menyentuh pelet dan dipindahkan pada tabung eppendorf 1.5 ml yang telah berisi 250 µl etanol 96%. Cairan dipindahkan ke collection tube yang telah disisipi purification column, sentrifugasi selama 1 menit dengan kecepatan 11 000 rpm. Cairan dibuang dan disisipkan purification column pada collection tube
lainnya. Sebanyak 700 µl wash buffer (WB1) ditambahkan ke dalam collection tube yang telah disisipi purification column yang sama, sentrifugasi selama 1 menit dengan kecepatan 11 000 rpm. Cairan dibuang dan purification column
dipindahkan pada pada tube bersih 2 ml. Sebanyak 500 µl wash buffer (WB2) ditambahkandan dan disentrifugasi selama 1 menit dengan kecepatan 11 000 rpm dilakukan dua kali ulangan. Cairan dibuang dan pindahkan purification column ke Rnase-free 1.5 ml collection tube. Sebanyak 50 µl nuclease free water
ditambahkanpada bagian tengah purification column, sentrifugasi selama 1 menit dengan kecepatan 11 000 rpm. purification column dibuang dan RNA total disimpan pada suhu -80 ˚C.
Sintesis complementary DNA (cDNA). Produk ekstraksi RNA total digunakan sebagai template untuk sitesis cDNA. Sintesis cDNA terjadi melalui proses transkripsi balik RNA dengan menggunakan enzim transkriptase MMuLV (Moloney Murine Leukimia Virus (Tabel 1). Molekul cDNA tersebut digunakan sebagai cetakan dalam proses PCR.
Tabel 1 Komposisi bahan reaksi transkripsi balik RT-PCR
Komponen Volume (µl)*
Setiap reaksi RT diinkubasi berturut-turut pada suhu 65 ˚C selama 5 menit,
37 ˚C selama 60 menit, dan 70 ˚C selama 10 menit. Produk RT-PCR selanjutnya
digunakan dalam tahapan amplifikasi PCR.
7 Tabel 2 Komposisi bahan PCR
Komponen
Beberapa primer yang digunakan dalam proses PCR yaitu primer universal Potyvirus MJ1/MJ2, dan dua pasang primer spesifik PRSV yaitu PRSV1298 /PRSV1942 dan PRSV 326/PRSV800 (Tabel 3). Program amplifikasi untuk primer universal mengacu pada metode Hidayat et al. (2012), terdiri atas 45 siklus melalui beberapa tahap yaitu diawali dengan perlakuan pradenaturasi pada 94 ˚C selama 3 menit, denaturasi (fase pemisahan utas DNA) pada suhu 96 ˚C selama 30
detik, penempelan primer pada suhu 61 ˚C selama 1 menit, ekstensi 72 ˚C selama
1 menit, dilanjutkan dengan ekstensi final pada 72 ˚C selama 2 menit. Program
amplifikasi untuk primer spesifik Nib (PRSV1298/PRSV1942) dan CP (PRSV326/PRSV800) terdiri atas 40 siklus, pradenaturasi 94 ˚C selama 5 menit, denaturasi pada suhu 94 ˚C selama 1 menit, penempelan pada suhu 52 ˚C selama 1 menit, ekstensi 72 ˚C selama 1 menit, ekstensi final 72 ˚C selama 7 menit.
8
Visualisasi Hasil Amplifikasi dengan RT-PCR.
9
HASIL DAN PEMBAHASAN
Gejala dan Kejadian Penyakit pada Tanaman Pepaya di Lapangan
Pengamatan yang dilakukan pada kebun pepaya di tiga desa di Bogor, Jawa Barat menunjukkan bahwa pertanaman pepaya memperlihatkan gejala penyakit yang disebabkan oleh virus berupa mosaik pada daun, batang, tangkai daun dan buah dengan tingkat kejadian penyakit 20% hingga 100% (Tabel 4). Gejala mosaik yang ditemukan berupa mosaik ringan hingga berat disertai beberapa variasi gejala lainnya (Tabel 5). Penghitungan kejadian penyakit di daerah Kebumen, Jawa tengah tidak dilakukan karena sampel diperoleh melalui korespondensi.
Tabel 4 Tingkat Kejadian penyakit bercak cincin pada tanaman pepaya di daerah Bogor, Jawa Barat
*Pengamatan dan penghitungan kejadian penyakit dilakukan hanya pada satu areal pertanaman pepaya
10
Tabel 5 Variasi gejala pada tanaman pepaya di lapangan Gejala
(+), gejala ditemukan; (-), gejala tidak ditemukan
Gejala mosaik kuning ringan pada daun ditemukan di desa Tegal Waru, mosaik sedang di desa Cikarawang dan mosaik berat di desa Ayam putih. Gejala mengeriting (wrinkling) ditemukan di desa Cikarawang dan Ayam Putih. Gejala seperti tali sepatu (shoestring) ditemukan pada tanaman pepaya desa Cikarawang, Bantar sari dan Curug. Gejala mosaik pada batang dan tangkai daun ditemukan pada tanaman pepaya disetiap desa dengan pola yang berbeda. Gejala mosaik berupa mosaik hijau tua dengan pola seperti pulau-pulau pada batang ditemukan di desa Cikarawang, Bantar Sari dan Curug, sedangkan gejala mosaik gores (strike) pada batang ditemukan di desa Tegal Waru dan Ayam Putih. Gejala bercak hijau tua dan bercak cincin pada buah ditemukan pada tanaman pepaya di tiga desa yaitu desa Cikarawang, Bantar sari, dan Curug sedangkan pada desa Tegal waru dan desa Ayam putih gejala pada buah hanya berupa bercak hijau tua (Gambar 1).
11
Gambar 1 Gejala pada daun,batang atau tangkai daun, dan buah pada berbagai lokasi (a) mosaik pada daun dan batang, bercak cincin pada buah di desa Tegal Waru, (b) mosaik pada daun, tangkai daun, dan bercak hijau tua pada buah di desa Cikarawang, (c) daun seperti tali sepatu (shoestring), mosaik pada batang, dan bercak cincin pada buah di desa Bantar Sari, (d) daun seperti tali sepatu (shoestring), mosaik pada batang, bercak cincin pada buah di desa Curug, (e) mosaik kuning, mosaik ringan pada batang, mosaik pada buah di desa Ayam putih Kondisi lingkungan pertumbuhan tanaman yang berbeda-beda berpengaruh pada fisiologi dan proses metabolisme yang terjadi dalam jaringan tanaman,
a
b
c
d
12
sehingga mempengaruhi multiplikasi dan replikasi virus di dalam jaringan tanaman. Faktor lingkungan yang mempengaruhi pertumbuhan tanaman, yaitu intensitas penyinaran cahaya matahari, suhu, dan nutrisi. Intensitas penyinaran matahari yang tinggi dapat menurunkan tingkat kerentananan tanaman. Suhu yang tinggi dapat mengurangi gejala virus, karena proses replikasi terhambat. Kombinasi intensitas cahaya yang tinggi dan suhu yang tinggi meningkatkan pertumbuhan tanaman dan menurunkan tingkat infeksi virus (Walkey 1991). Inokulasi Virus Secara Mekanis
Gejala mosaik yang ditemukan di lapangan dapat disebabkan oleh infeksi beberapa virus, oleh sebab itu identifikasi virus tidak bisa dilakukan berdasarkan deskripsi gejala saja. Untuk mendapatkan gejala yang khas dari setiap infeksi virus, maka perlu dilakukan inokulasi virus pada tanaman sehat dan diamati karakteristik gejalanya. Pada penelitian ini dilakukan inokulasi secara mekanis menggunakan isolat asal Nanggroe Aceh Darussalam pada tanaman pepaya varietas orange lady (Gambar 2). Penularan mekanis juga dilakukan pada pepaya varietas Calina menggunakan semua sampel tanaman sakit. Semua tanaman pepaya varietas Calina menunjukkan gejala setelah inokulasi mekanis, kecuali tanaman yang diinokulasi dengan sampel virus dari Desa Ayam Putih, Kebumen. Gejala yang muncul terlihat 12 hari sampai 14 hari setelah inokulasi yaitu pada bagian apikal daun atau daun termuda. Gonzalez dan Trujillo (2005) melaporkan periode inkubasi yang sama untuk hasil inokulasi mekanis PRSV yaitu 14 hari, sementara Agrios (2005) melaporkan periode inkubasi PRSV berkisar antara 2 hingga 3 minggu setelah inokulasi.
Hasil penularan isolat Nanggroe Aceh Darussalam pada pepaya varietas
13
Gambar 2 Gejala infeksi PRSV hasil inokulasi isolat Nanggroe Aceh pada tanaman pepaya varietas orange lady; (a) daun sehat, (b) mosaik kuning dan pinggiran daun mengeriting, (c) malformasi daun muda dan daun menyerupai tali sepatu (shoestring), (d) mosaik bergores pada batang, (e) tanaman kerdil
Terdapat beberapa variasi gejala yang ditunjukkan oleh tanaman pepaya varietas Calina hasil penularan menggunakan isolat asal Nanggroe Aceh Darussalam, Bogor dan Kebumen. Tanaman yang diinokulasi isolat asal Nanggroe Aceh Darussalam menunjukkan gejala mosaik yang sangat jelas, pinggiran daun mengeriting, serta tanaman menjadi kerdil (Gambar 3b,h4), mosaik ringan terdapat pada tanaman yang diinokulasi isolat Bogor (Gambar 3 c-f), mosaik tidak terlihat pada tanaman yang diinokulasikan isolat Kebumen. Hal tersebut dapat terjadi karena perbedaan sifat virulensi suatu virus, yang umumnya terjadi sebagai mutasi pada gen virulensi virus. Mutasi gen dapat mengakibatkan terjadinya perubahan fungsi gen, sehingga dapat menyebabkan perubahan gejala yang muncul atau virulensi virus (Manzila et al. 2012).
a b c
14
Gambar 3 Gejala infeksi PRSV hasil inokulasi isolat; kontrol (a), Nanggroe
Aceh Darussalam (b), desa Tegal Waru (c), desa Cikarawang (d), desa Bantar Sari (e), desa Curug (f), desa Ayam Putih (g). Tinggi tanaman hasil inokulasi (h), kontrol (h1); desa Tegal Waru,Bogor (h2); desa Situgede, Bogor (h3); desa Bantar Sari, Bogor (h4); desa Curug, Bogor (h5); Nanggroe Aceh Darussalam (h6); desa Ayam Putih, Kebumen (h7).
Perbandingan gejala tersebut menunjukkan bahwa isolat asal Nanggroe Aceh Darussalam sangat virulen dibandingkan isolat Bogor, sedangkan isolat asal Kebumen diduga tidak terinfeksi oleh PRSV. Untuk memastikan bahwa gejala yang muncul merupakan infeksi PRSV, maka dilakukan deteksi secara molekuler menggunakan metode RT-PCR.
Deteksi PRSV dengan Metode Reverse-Transcription Polymerase Chain
Reaction (RT-PCR)
Bagian tanaman pepaya (daun, tangkai daun, buah) yang menunjukkan gejala menjadi sampel untuk mendeteksi PRSV secara molekuler dengan teknik RT-PCR, menggunakan primer universal Potyvirus MJ1/MJ2 dan dua pasangan primer spesifik yaitu PRSV1298/PRSV1942 dan PRSV 326/PRSV800.
Primer universal dan spesifik dipilih karena mengamplifikasi daerah genom virus yang berbeda, namun memiliki tingkat konservasi yang tinggi. Pasangan primer MJ1/MJ2 dirancang untuk mendeteksi protein selubung Potyvirus dengan produk amplifikasi berukuran ≈320 bp (Marie-Jeanne et al. 2000). Primer ini telah banyak digunakan untuk mendeteksi dan mengidentifikasi virus dari kelompok
Potyvirus. Primer ini digunakan oleh Grisoni et al. (2006) untuk mendeteksi
Potyvirus pada tanaman vanila, Babu et al. (2011) mendeteksi Dasheen mosaic virus (DsMV) pada Colocasia esculeta di India, dan Hidayat et al.( 2012) mendeteksi PRSV pada tanaman pepaya asal Nanggroe Aceh Darussalam.
a c d
e f g
1 2 3 4 5 6 7
15 Pasangan primer spesifik PRSV326/PRSV800 dan PRSV1298/PRSV1942 dirancang untuk mengamplifikasi berturut-turut bagian gen selubung protein PRSV dengan ukuran ≈ 475 bp dan bagian gen Nib PRSV dengan ukuran ≈ 650 bp (Mohammed et al. 2012). Gen selubung protein berfungsi dalam enkapsidasi RNA, penularan melalui vektor, kemampuan patogenesis, dan perpindahan dari sel ke sel. Gen Nib berfungsi dalam replikasi virus (Gonsalves et al. 2010)
Amplifikasi menggunakan masing-masing pasangan primer universal dan spesifik memberikan hasil yang berbeda untuk masing-masing sampel (Gambar 4 sampai dengan 9). Pasangan primer MJ1/MJ2 dan PRSV 1298/PRSV1942 hanya berhasil mengamplifikasi DNA virus isolat Nanggroe Aceh Darussalam, namun tidak berhasil mengamplifikasi DNA virus isolat Bogor dan Kebumen. Pasangan primer PRSV326/PRSV800 berhasil mengamplifikasi DNA virus isolat Nanggroe Aceh Darussalam dan Bogor, namun tidak berhasil mengamplifikasi isolat asal Kebumen. Keberhasilan amplifikasi dengan primer spesifik PRSV 326/PRSV800, mengindikasikan daerah dengan konservasi yang tinggi pada bagian yang diamplifikasi oleh pasangan primer tersebut, sehingga penggunaan primer spesifik PRSV326/PRSV800 lebih disarankandalam deteksi PRSV dengan teknik RT-PCR.
Gambar 4 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2) dan PRSV326 /PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Nanggroe Aceh Darussalam. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-)
320 650
475 bp
M + d b - M + d b - M + d b -
16
Gambar 5 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2) dan PRSV326/PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Tegal Waru, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-)
Gambar 6 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2) dan PRSV326/PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Cikarawang, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-)
475 bp
M + - b d u M + - b d u M + - b d u
1 2 3
475 bp
M + - b d u M + - b d u M + - b d u
17
Gambar 7 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2) dan PRSV326/PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Bantar Sari, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-)
Gambar 8 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2) dan PRSV326 /PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Curug, Bogor. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-)
475 bp
M + - b d u M + - b d u M + - b d u
1 2 3
475 bp
M + - b d u M + - b d u M + - b d u
18
Gambar 9 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR
menggunakan primer MJ1/MJ2 (1), PRSV1298/PRSV1942 (2) dan PRSV326 /PRSV800 (3). Sampel DNA berasal dari sampel daun (d), batang (b), buah (u) yang diperoleh dari Desa Ayam Putih, Kebumen. Pada masing-masing gel agarosa disertakan DNA (M), kontrol positif DNA (+) dan kontrol negatif tanpa DNA (-)
Deteksi menggunakan teknik PCR membuktikan infeksi PRSV pada sampel tanaman dari Nanggroe Aceh Darussalam dan Bogor, sementara infeksi PRSV tidak terbukti pada sampel tanaman asal Kebumen. Diduga terdapat perbedaan genetik antara PRSV isolat Nanggroe Aceh Darussalam dengan PRSV isolat Bogor karena isolat asal Nanggroe Aceh Darussalam berhasil diamplifikasi dengan baik menggunakan pasangan primer PRSV326/PRSV800. Selain itu, isolat Nanggroe Aceh Darussalam diduga memiliki virulensi dan konsentrasi yang lebih tinggi dibandingkan isolat-isolat PRSV Bogor berdasarkan keparahan gejala yang terjadi pada tanaman hasil inokulasi.
Sampel tanaman yang tidak teramplifikasi dapat disebabkan oleh beberapa faktor, diantaranya kualitas dan kuantitas DNA yang kurang baik, tidak terdapat kesesuaian antara basa nukleotida target dengan basa nukleotida penyusun primer, virus yang diamplifikasi bukan merupakan virus target (Padmalatha dan Prasad 2006), belum tercapainya optimasi reaksi dan kondisi penyimpanan sampel yang kurang baik mengakibatkan DNA terdegradasi, sehingga primer tidak mampu mengamplifikasi DNA virus yang ada pada sampel (Grisoni et al.2006).
Hasil amplifikasi DNA sampel tanaman yang dideteksi langsung dari lapangan lebih tipis dibandingkan amplifikasi DNA menggunakan tanaman hasil inokulasi. Hal tersebut menunjukkan bahwa terjadi peningkatan konsentrasi virus setelah dilakukan inokulasi pada tanaman (Gambar 10). Hasil amplifikasi ini juga membuktikan sesuai prinsip Postulat Koch, yaitu gejala mosaik pada tanaman pepaya di lapangan disebabkan oleh infeksi PRSV.
19
Gambar 10 Amplifikasi DNA Papaya ringspot virus dengan teknik RT-PCR mengunakan primer spesifik PRSV326/PRSV800. Sampel DNA berasal dari tanaman hasil penularan secara mekanis masing-masing isolat: Nanggroe Aceh Darussalam (sebagai kontrol positif) (A); Tegal Waru, Bogor (B); Cikarawang, Bogor (C); Bantar Sari, Bogor (D); Curug, Bogor (E); Ayam Putih, Kebumen (F). Penanda DNA (M), kontrol negatif tanpa DNA (-)
M A - B C D E F
20
SIMPULAN
Pertanaman pepaya pada beberapa desa di daerah Bogor, Jawa Barat terbukti terinfeksi PRSV berdasarkan uji penularan secara mekanis dan deteksi menggunakan teknik RT-PCR. Hasil penularan secara mekanis menunjukkan bahwa tingkat virulensi isolat-isolat PRSV dari daerah Bogor lebih rendah dibandingkan isolat PRSV dari Nanggroe Aceh Darussalam. Pasangan primer PRSV326/PRSV800 berhasil mengamplifikasi bagian gen protein selubung semua sampel positif PRSV secara konsisten sehingga dapat disarankan untuk digunakan dalam deteksi PRSV dengan teknik RT-PCR. Penelitian ini memperkuat mengenai keberadaan PRSV di wilayah di Indonesia, sehingga status PRSV sebagai OPTK kategori A1 perlu ditinjau ulang.
SARAN
21
DAFTAR PUSTAKA
Agrios GN. 2005. Plant Pathology. 5th ed. London (GB): Academic Press. Akin HM. 2006. Virologi Tumbuhan. Yogyakarta (ID): Kanisius.
[Balitbu] Balai Penelitian Tanaman Buah Tropika. 2008. Pengelolaan Kebun Pepaya Sehat. Solok (ID): Balai Penelitian Tanaman Buah Tropika.
Babu B, Hegde V, Makeshkumar T, Jeeva ML. (2011). Detection and identification of Dasheen mosaic virus infecting Colocasia esculenta in India. Indian J.Virol. [Internet] [diunduh 2014 Juli 11]; 22(1):59-62. doi: 10.1007/s13337-011-0030-7.
Bateson MF, Lines RE, Revill P, Chaleeprom W, Ha CH, Gibbs AJ, Dale JL. 2002. On the evolution and molecular epidemiologi of the potyvirus Papaya ringspot virus. Journal of General Virology. [Internet] [diunduh 2013 Nov 9]; 83:2575-2585. Tersedia pada : http://eprints.qut.edu.au.
Da Silva JA T, Roshid Z, Nhut DT, Sivakumar D, Gera A, Souza MT Jr, Tennat PF. 2007. Papaya (Carica papaya L.) biology and biotechnology. Tree and Forestry Science and Biotechnology. [Internet] [diunduh 2013 Des 27]; 1(1):47-73. Tersedia pada: http://agrisave.com/ bibliotecal/agricola.
Davis RI, Mu L, Maireroa N, Wigmore WJ, Grisoni M, Bateson MF, Thomas JE. 2005. First records of papaya strain of Papaya ringspot virus (PRSV-P) in French Polynesia and the Cook Island. Australasian Plant Pathology. [Internet] [diunduh 2014 April 22]; 34(1):125-126. doi: 10.1071/AP04094. [Deptan] Departemen Pertanian 2006. Pengenalan dan pengendalian hama dan
penyakit penting tanaman pepaya. Agro Inovasi. [Internet] [diunduh 2014 Juni 24]. Tersedia pada: http://www.litbang.deptan.go.id/download /one/33/ file/Bagian-1.pdf.
Diallo HA, Monger W, Kouassi N, Yoro DT, Jones P. 2007. First report of
Papaya ringspot virus infecting papaya in Cote d’lvoire. Plant Pathology. [Internet] [diunduh 2014 Mai 15]; 26(4):718. doi: 10.1111/j.1365-3059. 2007.0 1593.x.
FAOSTAT [Food and Agriculture Organization of The United Nation]. 2014. Crop production [Internet] [diunduh 2014 Juni 12] Tersedia pada: http:// faostat. fao.org/site/339/default.aspx.
Gonsalves D, Tripathi S, Carr J.B, Suzuki J.Y. 2010. Papaya ringspot virus [Internet]. St. Paul (US): The American Phytophatological Society; [diunduh 2013 Mar 23]. Tersedia pada: http:// www.apsnet.org.
Grisoni M, Moles M, Farreyrol K, Rassaby L, Davis R, Pearson M. (2006) Identification of potyvirus infecting vanilla by direct sequencing of a short RT-PCR amplicon. Plant Pathology. [Internet] [diunduh 2014 Juli 11]; 55:523-529. doi: 10.1111/j.1365-3059.2006.01397.x.
Hidayat SH, Nurulita S, Wiyono S. 2012. Infeksi Papaya ringspots virus pada tanaman pepaya di Provinsi Nanggroe Aceh Darussalam. Jurnal Fitopatologi Indonesia. 8(6):184-187.
22
Marie-Jeanne V, Loos R, Peyre J, Alliot B, Signoret P. 2000. Differentiation of poaceae Potyvirus by reverse transcription polymerase chain reaction and retriction analysis. J Phytopathol. 148(3):141-151. doi: 10.1046/j.1439-0434.2000.00473.x.
Matthews REF. 1981. Plant virology. Ed ke-2. London (GB): Academic Press. Miftakhurohmah, Suastika G, Damayanti TA. (2013). Deteksi secara serologi dan
molekuler beberapa jenis virus yang berasosiasi dengan penyakit mosaik tanaman nilam (Pogostemon cablin Benth). Jurnal Littri. [Internet] [diunduh 2014 Juli 11]; 19(3):130-138. Tersedia pada: perkebunan.litbang. deptan.go.id.
Mohammed H, Manggil A, Zicca S, Hussein El A, Tomassol. 2012. First report of
Papaya ringspot virus in pumpkin in Sudan. New Disease Reports. [Internet] [diunduh 2014 Maret 8]; 26:26. doi: 10.5197/j.2044-0588.2012. 026.026.
Padmalatha,K dan Prasad MNV. 2006. Optimization of DNA isolation and PCR protocol for RAPD analysis of selected medicinal and aromatic plants of conservation on concern from Penisular India. Afr.J. Biotechnol. [Internet] [diunduh 2014 Agustus 29]; 5:230-234. Tersedia pada: https://tspace.Libra ry.utoronto.ca.
Tripathi S, Suzuki JY, Ferreira SA, Gonsalves D. 2008. Papaya ringspot virus-P: characteristics, pathogenicity, sequence variability and control. Molecular Plant Pathology. [Internet] [diunduh 2013 September 4]; 9(3):269-280. doi: 10.1111/J.1364-3703.2008.00467.X.
[UF] University of Florida. 2012. An overview of global papaya production, trade, and consumption [Internet] [diunduh 2014 Mai 19]. Tersedia pada: http://edis.ifas.ufl.edu/fe913.
23
RIWAYAT HIDUP
Penulis dilahirkan di Pasaman, Sumatera Barat pada tanggal 14 Maret 1992, sebagai anak ketiga dari empat bersaudara keluarga Gimono dan Ahtimar S.Pdi. Penulis menyelesaikan pendidikan di MA Negri 2 Padang, Sumatera Barat pada tahun 2010, dan pada tahun yang sama diterima di Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB.
Selama menempuh pendidikan di IPB, penulis menjadi asisten praktikum Pengelolaan dan Pemanfaatan Pestisida dalam Proteksi Tanaman tahun ajaran 2014, asisten praktikum mata kuliah Penyakit Benih Pascapanen pada tahun ajaran 2014, dan Dasar Perlindungan Tanaman Program D3 pada tahun ajaran 2014.
Penulis juga aktif mengikuti kegiatan kepanitiaan dan organisasi di IPB yaitu, anggota divisi Kewirausahaan Himpunan Mahasiswa Proteksi Tanaman (HIMASITA) pada tahun 2011, anggota divisi Medis Migratoria Departemen Proteksi Tanaman 2011, panitia divisi PDD acara National Plant Protection Event