PEMODELAN BIPLOT PADA KLASIFIKASI FRAGMEN
METAGENOM DENGAN K-MERS SEBAGAI EKSTRAKSI
CIRI DAN PROBABILISTIC NEURAL NETWORK
SEBAGAI CLASSIFIER
FERDINAN ANDREAS MANGASI SIMAMORA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Pemodelan Biplot pada Klasifikasi Fragmen Metagenom dengan K-Mers sebagai Ekstraksi Ciri dan Probabilistic Neural Network sebagai Classifier adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
FERDINAN ANDREAS MANGASI SIMAMORA. Pemodelan Biplot pada Klasifikasi Fragmen Metagenom dengan K-Mers sebagai Ekstraksi Ciri dan Probabilistic Neural Network sebagai Classifier. Dibimbing oleh AGUS BUONO
Metagenom adalah studi terhadap material genetika yang diambil langsung dari sumber lingkungan organisme dengan melakukan prosedur sekuensing deoxyribonucleic acid. Sekuensing dilakukan pada sekumpulan genom yang sudah tercampur dengan beragam organisme lain pada lingkungan tersebut. Hal ini menyebabkan proses identifikasi organisme diperlukan untuk mencegah kesalahan perakitan fragmen antar organisme. Penelitian ini bertujuan untuk mengklasifikasikan fragmen metagenom pada tingkat genus dengan algoritme Probabilistic Neural Network dan k-mers sebagai pengekstraksi cirinya. Pemodelan biplot digunakan untuk mereduksi ciri dari hasil k-mers yang memiliki dimensi cukup besar. Penelitian ini menggunakan 3 genus mikroorganisme yang dibagi menjadi 2 kelompok dataset yaitu organisme dikenal dan organisme belum dikenal. Panjang fragmen yang digunakan untuk setiap dataset ialah 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Dari hasil penelitian ini didapatkan akurasi terbaik sebesar 98.10% untuk data organisme dikenal dan 92.80% untuk data organisme belum dikenal. Pemodelan biplot berhasil mereduksi 68.75% ciri fragmen dengan efisiensi waktu pengklasifikasian sebesar 12%.
Kata kunci: metagenom, DNA, k-mers, biplot, PNN.
ABSTRACT
FERDINAN ANDREAS MANGASI SIMAMORA. Biplot Modelling on Metagenome Fragment Classification using K-Mers as Feature Extraction and Probabilistic Neural Network as Classifier. Supervised by AGUS BUONO.
Metagenomics is the study of the genetic material taken directly from the organism's source environment by performing deoxyribonucleic acid sequencing procedure. Sequencing was performed on a set of genomes that have been mixed with a variety of other organisms in the environment. This led to the identification of the organism is required to prevent fragment assembly error between organisms. This study objectives to classify metagenome fragments at the genus level with Probabilistic Neural Network algorithms and k-mers as the features extraction. Biplot modelling is used to reduce features of the k-mers results that have dimensions large enough. This study uses 3 genuses of microorganisms which are divided into 2 dataset groups, namely known organisms and unknown organisms. The length of fragments used for each dataset is 500 bp, 1 kbp, 5 kbp, and 10 kbp. From the results of this study, the best accuracy for the known organisms was 98.10% and 92.80% for unknown organisms. Biplot modelling managed to reduce 68.75% fragment features with classification efficiency of 12%.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
PEMODELAN BIPLOT PADA KLASIFIKASI FRAGMEN
METAGENOM DENGAN K-MERS SEBAGAI EKSTRAKSI
CIRI DAN PROBABILISTIC NEURAL NETWORK
SEBAGAI CLASSIFIER
FERDINAN ANDREAS MANGASI SIMAMORA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Penguji:
Judul Skripsi : Pemodelan Biplot pada Klasifikasi Fragmen Metagenom dengan K-Mers Sebagai Ekstraksi Ciri Dan Probabilistic Neural Network Sebagai Classifier
Nama : Ferdinan Andreas Mangasi Simamora NIM : G64124016
Disetujui oleh
Dr Ir Agus Buono, MSi MKom Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Juli 2014 ini ialah bioinformatika, dengan judul Pemodelan Biplot pada Klasifikasi Fragmen Metagenom dengan K-Mers Sebagai Ekstraksi Ciri Dan Probabilistic Neural Network Sebagai Classifier.
Terima kasih penulis ucapkan kepada Bapak Dr Ir Agus Buono, Msi MKom selaku pembimbing yang telah mengarahkan penulis dalam menyelesaikan penelitian ini, serta Bapak Dr Wisnu Ananta Kusuma, ST MT dan Bapak Toto Haryanto, SKom MSi selaku dosen penguji yang telah memberi saran. Di samping itu, penghargaan penulis sampaikan kepada rekan-rekan Ekstensi Ilkom angkatan 7 terutama rekan-rekan satu bimbingan yang telah saling membantu dalam menyelesaikan penelitian ini. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vii
DAFTAR GAMBAR vii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 3
METODE 3
Penyiapan Data 4
Ekstraksi Ciri (K-Mers) 4
Reduksi Ciri (Biplot) 5
Pembagian Data 6
Klasifikasi 6
Pengujian, Evaluasi, dan Analisis Hasil 8
Lingkungan Implementasi 8
HASIL DAN PEMBAHASAN 9
Penyiapan Data 9
Ekstraksi Ciri (K-Mers) 10
Reduksi Ciri (Biplot) 11
Pembagian Data 16
Rangkaian Percobaan 16
Evaluasi dan Analisis Seluruh Hasil Percobaan 16
SIMPULAN DAN SARAN 24
Simpulan 24
Saran 25
DAFTAR PUSTAKA 25
LAMPIRAN 26
DAFTAR TABEL
1 Dataset organisme dikenal 9
2 Dataset organisme belum dikenal 10
3 Nilai akurasi terbaik dan waktu eksekusi klasifikasi pada pengujian
organisme dikenal 17
4 Confusion matrix untuk pengujian terbaik pada pengujian organisme
dikenal 17
5 Perbandingan waktu eksekusi klasifikasi antara fragmen dengan biplot
(terbaik) dan tanpa biplot 18
6 Waktu eksekusi klasifikasi fragmen dengan menggunakan biplot
(terbaik) pada beberapa percobaan 18
7 Waktu eksekusi klasifikasi fragmen tanpa menggunakan biplot pada
beberapa percobaan 18
8 Perbandingan akurasi klasifikasi antara fragmen dengan biplot (terbaik) dan tanpa biplot pada pembagian data latih 60%:40% 19 9 Nilai akurasi klasifikasi fragmen menggunakan biplot (terbaik)
dengan pembagian data latih 60%:40% pada beberapa percobaan 20 10 Nilai akurasi klasifikasi fragmen tanpa menggunakan biplot dengan
pembagian data latih 60%:40% pada beberapa percobaan 20 11 Perbandingan akurasi klasifikasi antara fragmen dengan biplot
(terbaik) dan tanpa biplot pada pembagian data latih 80%:20% 21 12 Nilai akurasi klasifikasi fragmen menggunakan biplot (terbaik)
dengan pembagian data latih 80%:20% pada beberapa percobaan 21 13 Nilai akurasi klasifikasi fragmen tanpa menggunakan biplot dengan
pembagian data latih 80%:20% pada beberapa percobaan 21 14 Nilai akurasi dan waktu eksekusi klasifikasi pada dimensi pengujian
organisme belum dikenal 23
15 Confusion matrix untuk pengujian terbaik pada pengujian organisme
belum dikenal 23
16 Nilai akurasi klasifikasi fragmen organisme belum dikenal pada
beberapa percobaan 23
DAFTAR GAMBAR
1 Diagram alur metode penelitian 3
2 Ekstraksi ciri k-mers 5
3 Biplot pada 2 sumbu koordinat 6
4 Arsitektur PNN 7
5 Plot hasil akar ciri fragmen 500 bp dari dekomposisi nilai singular
pada dimensi akar ciri = 2 11
6 Plot hasil akar ciri fragmen 1 kbp dari dekomposisi nilai singular pada
dimensi akar ciri = 2 12
7 Plot hasil akar ciri fragmen 5 kbp dari dekomposisi nilai singular pada
dimensi akar ciri = 2 12
8 Plot hasil akar ciri fragmen 10 kbp dari dekomposisi nilai singular
9 Biplot fragmen 500 bp dari dekomposisi nilai singular pada dimensi
akar ciri = 2 dengan mengambil 10 peubah 14
10 Biplot fragmen 1 kbp dari dekomposisi nilai singular pada dimensi
akar ciri = 2 dengan mengambil 10 peubah 14
11 Biplot fragmen 5 kbp dari dekomposisi nilai singular pada dimensi
akar ciri = 2 dengan mengambil 10 peubah 15
12 Biplot fragmen 10 kbp dari dekomposisi nilai singular pada dimensi
akar ciri = 2 dengan mengambil 10 peubah 15
13 Plot perbandingan waktu eksekusi klasifikasi antara fragmen dengan
biplot terbaik (■) dan tanpa biplot (♦) 19
14 Plot perbandingan akurasi klasifikasi antara fragmen dengan biplot terbaik dan tanpa biplot pada pembagian data latih 60%:40% 20 15 Plot perbandingan akurasi klasifikasi antara fragmen dengan biplot
terbaik dan tanpa biplot pada pembagian data latih 80%:20% 22 16 Plot perbandingan akurasi klasifikasi antara fragmen organisme
dikenal dengan organisme belum dikenal 24
DAFTAR LAMPIRAN
1 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 10 peubah dan pembagian data latih dan data uji 60%:40% 26 2 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 20
peubah dan pembagian data latih dan data uji 60%:40% 26 3 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 30
peubah dan pembagian data latih dan data uji 60%:40% 27 4 Nilai akurasi pada dimensi akar ciri yang berbeda dengan semua
peubah (tanpa biplot) dan pembagian data latih dan data uji 60%:40% 27 5 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 10
peubah dan pembagian data latih dan data uji 80%:20% 28 6 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 20
peubah dan pembagian data latih dan data uji 80%:20% 28 7 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 30
peubah dan pembagian data latih dan data uji 80%:20% 29 8 Nilai akurasi pada dimensi akar ciri yang berbeda dengan semua
peubah (tanpa biplot) dan pembagian data latih dan data uji 80%:20% 29 9 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang
berbeda dengan h = 1, n = 10 peubah, dan pembagian data latih dan
data uji 60%:40% 30
10 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 20 peubah, dan pembagian data latih dan
data uji 60%:40% 30
11 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 30 peubah, dan pembagian data latih dan
data uji 60%:40% 31
12 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, semua peubah (tanpa biplot), dan pembagian
13 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 10 peubah, dan pembagian data latih dan
data uji 80%:20% 32
14 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 20 peubah, dan pembagian data latih dan
data uji 80%:20% 32
15 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 30 peubah, dan pembagian data latih dan
data uji 80%:20% 33
16 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, semua peubah (tanpa biplot), dan pembagian
PENDAHULUAN
Latar Belakang
Penelitian pada bidang material genetika organisme biasanya dilakukan dengan membudidayakan organisme yang ingin diteliti pada laboratorium dan melakukan penyejajaran sekuens untuk menyusun struktur protein dengan metode pemodelan de novo. Proses ini dilakukan untuk mengekstrak informasi urutan DNA yang berisi informasi genetika dari organisme. Akan tetapi, dari sekian banyak mikroorganisme yang ada di dunia hanya 1% yang dapat dibudidayakan. Untuk mikroorganisme selainnya harus dilakukan dengan mengambil sampel secara langsung dari lingkungan organisme tersebut. Oleh karena itu, metagenom berperan sebagai sebuah bidang studi yang mengkaji material genetika yang diambil langsung dari sumber lingkungan organisme dengan melakukan prosedur sekuensing DNA pada sekumpulan genom yang tercampur pada lingkungan tersebut (Kusuma dan Akiyama 2011).
Genom adalah set lengkap molekul-molekul DNA dalam setiap sel dari organisme hidup yang diturunkan dari generasi ke generasi. Deoxyribonucleic acid (DNA) bertindak sebagai cetakan dari unit kehidupan karena memiliki kemampuan mengodekan informasi penting untuk menghasilkan protein-protein yang dibutuhkan pada proses seluler. DNA berbentuk rantai ganda dari molekul sederhana (nukleotida) yang diikat bersama-sama dalam struktur helix yang dikenal dengan double helix. Nukleotida tersebut tersusun atas 4 basa nitrogen yaitu adenin, timin, guanin, dan sitosin yang dapat direpresentasikan dalam abjad A, T, G, dan C yang membentuk pasangan basa (base pairs/bp) dengan basa komplemennya (de Carvalho 2003).
Proses yang dilakukan pada metagenom ialah lisis dan ekstraksi DNA, kloning dan konstruksi, dan sequencing DNA dari hasil kloning tersebut. Tahap sequencing menghasilkan fragmen-fragmen metagenom yang saling tercampur dan overlap antar organisme. Kondisi overlap ini diperlukan sebagai kunci penyambung fragmen metagenom yang sejenis pada saat proses perakitan. Namun setiap organisme memiliki peluang kondisi overlap yang serupa dengan organisme lain yang berbeda jenis. Hal tersebut mengakibatkan proses identifikasi fragmen diperlukan untuk mencegah kesalahan perakitan fragmen antar organisme. Untuk tingkatan taksonomi pada hewan mulai dari tertinggi hingga terbawah yaitu terdiri dari Kingdom, Phyllum (filum), Class (kelas), Ordo (bangsa), Familia (suku), Genus (marga), dan Species (jenis).
Pada tahun 2008, Wu melakukan penelitian klasifikasi terhadap fragmen metagenom dengan menggunakan k-mers yang dipadukan dengan PCA. Wu menggunakan 4 metode klasifikasi sebagai perbandingan yaitu linear, quadratic, n-nearest neighbor, dan decision tree. Dari penelitian tersebut diperoleh bahwa akurasi terbaik dihasilkan oleh pengklasifikasi linear. Tidak hanya menghasilkan akurasi yang paling baik, pengklasifikasi tersebut memiliki kompleksitas komputasi yang cukup baik diterapkan pada dimensi fitur yang besar (Wu 2008).
2
yang merepresentasikan organisme latih dan organisme uji dengan panjang fragmen 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Akurasi yang dicapai dari penelitian tersebut yaitu 78% untuk panjang fragmen 500 bp, 80% untuk panjang fragmen 1 kbp, 86% untuk panjang fragmen 5 kbp, dan 87% untuk panjang fragmen 10 kbp.
Elliyana (2014) juga melakukan penelitian klasifikasi fragmen metagenom dengan ruang lingkup yang sama dengan Kusuma dan Akiyama. Metode klasifikasi yang digunakan ialah algoritme KNN dengan spaced k-mers sebagai pengekstraksi ciri. Nilai akurasi yang dicapai yaitu 86.11% untuk panjang fragmen 500 bp, 91.77% untuk panjang fragmen 1 kbp, 96.60% untuk panjang fragmen 5 kbp, dan 97.96% untuk panjang fragmen 10 kbp pada nilai k = 3. Dari kedua penelitian tersebut terlihat bahwa panjang fragmen dan tingkat akurasi pengklasifikasian memiliki korelasi positif.
Untuk penelitian kali ini akan dilakukan pengklasifikasian fragmen metagenom menggunakan algoritme Probabilistic Neural Network (PNN) dan k-mers sebagai ekstraksi fitur. Namun k-mers memiliki kecenderungan untuk menghasilkan dimensi yang cukup besar. Hal ini disebabkan nilai k yang digunakan menjadi pangkat sebagai representasi jumlah kombinasi basa. Oleh karena itu, untuk mengatasi hal tersebut biplot digunakan sebagai pereduksi ekstraksi ciri hasil k-mers. Data organisme yang digunakan pada penelitian ini merujuk pada data penelitian Kusuma dan Akiyama (2011) untuk kemudian hasilnya akan dianalisis.
Perumusan Masalah
Berdasarkan latar belakang penelitian yang telah diuraikan sebelumnya, masalah yang akan diteliti sebagai berikut:
1 Bagaimana hasil pengklasifikasian fragmen metagenom pada tingkat genus dengan menggunakan algoritme PNN pada penelitian ini?
2 Bagaimana pengaruh biplot terhadap hasil akurasi pengklasifikasian?
Tujuan Penelitian
Penelitian ini bertujuan untuk:
1 Melakukan klasifikasi fragmen metagenom pada tingkat genus dengan algoritme PNN, k-mers sebagai pengekstraksi ciri, dan biplot sebagai pereduksi cirinya.
2 Mendapatkan hasil akurasi dari hasil penelitian.
3 Menganalisis pengaruh biplot terhadap hasil akurasi pengklasifikasian.
Manfaat Penelitian
Hasil penelitian ini diharapkan dapat bermanfaat untuk:
1 Membantu peneliti dalam mengidentifikasi dan mengklasifikasi fragmen metagenom sesuai dengan tingkat taksonomi untuk genus yang ditentukan. 2 Mendapatkan algoritme yang lebih baik dalam melakukan pengidentifikasian
3 3 Mengembangkan teknik pengklasifikasian fragmen metagenom dengan
penerapan biplot.
Ruang Lingkup Penelitian
Ruang lingkup penelitian ini meliputi:
1 Data yang digunakan merujuk referensi data dari penelitian Kusuma dan Akiyama (2011). Data terdiri dari 2 kelompok dataset yang dibangkitkan oleh perangkat lunak MetaSim dengan format FASTA yang merepresentasikan organisme dikenal dan organisme belum dikenal.
2 Panjang fragmen untuk setiap dataset organisme meliputi 500 bp, 1 kbp, 5 kbp, 10 kbp. Dataset yang digunakan diambil dari genus Agrobacterium, Bacillus, dan Staphylococcus.
3 Sekuens DNA direpresentasikan sebagai 4 karakter A, T, G, dan C yang mewakili basa nitrogen adenin, timin, guanin, dan sitosin.
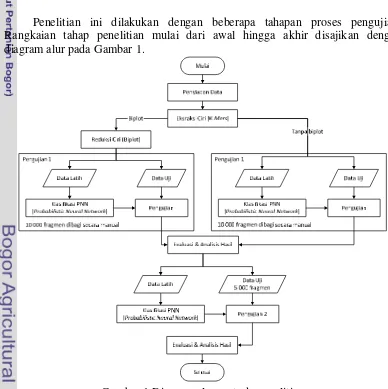
METODE
Penelitian ini dilakukan dengan beberapa tahapan proses pengujian. Rangkaian tahap penelitian mulai dari awal hingga akhir disajikan dengan diagram alur pada Gambar 1.
4
Pada tahap awal data fragmen metagenom disiapkan terlebih dahulu menjadi 2 kelompok dataset yaitu organisme dikenal dan organisme belum dikenal. Setelah itu dilakukan ekstraksi ciri metagenom menggunakan k-mers untuk semua dataset. Hasil ekstraksi ciri dari organisme dikenal dipersiapkan menjadi 2 skema yaitu tanpa reduksi dan direduksi dengan menggunakan biplot. Selanjutnya pembagian data dibagi menjadi data latih dan data uji dilakukan secara manual dengan komposisi 60%:40% dan 80%:20%. Data organisme yang sudah dibagi kemudian diklasifikasi menggunakan algoritme PNN.
Hasil pengujian tersebut dianalisis untuk disimpulkan data latih mana yang terbaik untuk digunakan sebagai data penguji di pengujian kedua dengan dataset organisme belum dikenal. Setelah itu data organisme belum dikenal diklasifikasikan menggunakan algoritme klasifikasi yang sama untuk hasilnya dianalisis kemudian.
Penyiapan Data
Data yang digunakan berupa 2 kelompok dataset yang dibangkitkan oleh perangkat lunak MetaSim dengan format FASTA yang merepresentasikan organisme dikenal sebagai pengujian pertama dan organisme belum dikenal sebagai pengujian kedua.
Organisme dikenal berupa 10 spesies dari 3 genus dengan 4 panjang fragmen berbeda. Sedangkan dataset organisme belum dikenal menggunakan 9 spesies dari 3 genus yang sama dengan 4 panjang fragmen berbeda. Hal yang membedakan antara organisme dikenal dan belum dikenal ialah pada tingkat spesiesnya yang berbeda-beda. Panjang fragmen terdiri dari 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Jumlah fragmen untuk dataset organisme dikenal dan organisme belum dikenal masing-masing adalah 10 000 dan 5 000.
Ekstraksi Ciri (K-Mers)
Tahapan ini dilakukan untuk mendapatkan fitur/ciri yang akan digunakan dalam klasifikasi dan pengujian. Ekstraksi ciri dilakukan dengan membaca frekuensi dari kombinasi nukleotida yang mungkin terbentuk dengan menggunakan k-mers untuk k = 3.
5
Gambar 2 Ekstraksi ciri k-mers
Reduksi Ciri (Biplot)
Analisis biplot diperkenalkan oleh Gabriel (1971). Analisis ini bertujuan menggambarkan hubungan antarpeubah serta perbandingan antarobjek yang disajikan dalam grafik 2 dimensi maupun 3 dimensi (Gabriel 1971). Analisis biplot menggunakan teknik penguraian nilai singular (singular value decomposition/SVD) terhadap matriks data (Buono 2014). Untuk matrix data nXp
yang ada dikomposisikan pada Persamaan 1 berikut:
nXp = nUr x rLr x r(AT)p (1)
Dalam hal ini r adalah pangkat matriks X, sedangkan U, L, dan A ditentukan berdasarkan akar ciri dan vektor ciri matrix XTX sehingga berlaku:
λ1>λ2> …>λr>λr+1>λn
dengan vektor ciri yang bersesuaian dengan akar ciri ke-i adalah vi. Matriks U, L,
dan A di atas dirumuskan pada Persamaan 2, 3, dan 4 berikut: A = [v1 v2 v3 … vr] (2)
L = diagonal λi =
λ1 … 0
0 … 0
0 … λr
(3)
U = μ1μ2 … μr dengan μi = 1
λi
Xvi (4)
Berdasarkan dekomposisi nilai singular di atas maka setiap matriks data nXp
dirumuskan sebagai Persamaan 5 berikut:
X=ULAT=G H (5) dengan nilai G dan H pada Persamaan 6 dan 7 berikut:
G = U = g1g2… gn T (6) H = LAT = h1h2… hp (7)
6
hi = 1
n-1hi (8) gi = √n-1 gi (9)
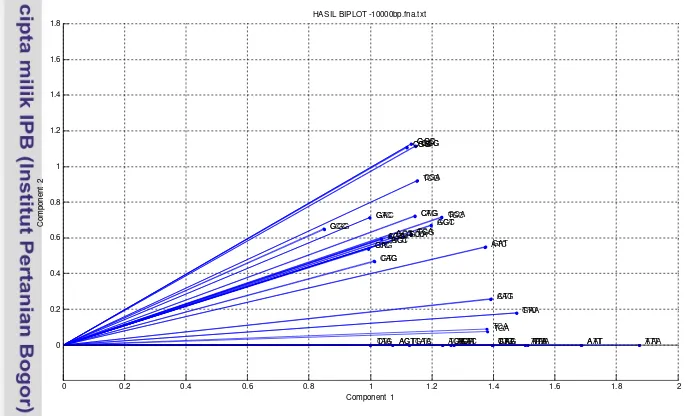
Biplot pada penelitian ini bertujuan untuk mereduksi ciri/peubah. Oleh karena itu, untuk setiap nilai H diplotkan ke dalam bentuk biplot (Gambar 3) untuk menggambarkan karakteristik setiap peubah. Semakin panjangnya garis peubah dari titik pusat ke titik koordinatnya mengindikasikan semakin besar pula pengaruhnya sebagai pembeda antarkelas.
Gambar 3 Biplot pada 2 sumbu koordinat
Pembagian Data
Pembagian data latih dan data uji pada organisme dikenal dilakukan secara manual dengan komposisi 60%:40% dan 80%:20%. Total data organisme dikenal yang digunakan sebanyak 10 000 dengan komposisi 10 spesies berbeda dalam 3 genus. Pembagian dilakukan berimbang dengan mengambil komposisi yang sama untuk setiap kelasnya.
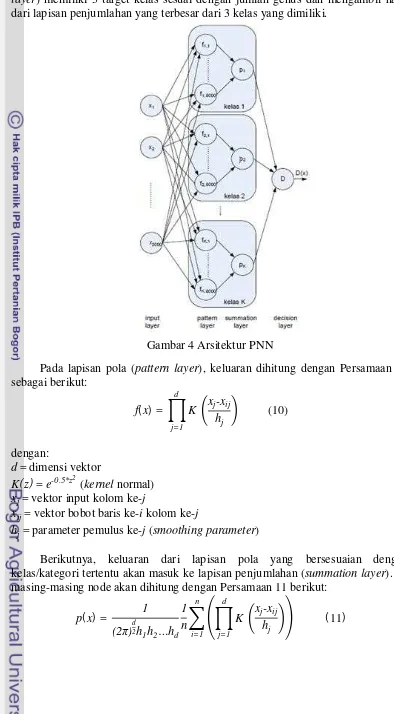
Klasifikasi
Arsitektur PNN yang memiliki bagian lapisan masukan, pola, penjumlahan, dan keluaran (Specht 1990) yang disajikan pada Gambar 4. Pada penelitian ini lapisan masukan berupa hasil ekstraksi data uji. Lapisan pola (pattern layer) hanya menggunakan satu model PNN yaitu dengan nilai parameter pemulus (h) tetap. Lapisan penjumlahan akan memroses hasil dari setiap lapisan pola per kelasnya dan mendapatkan nilai akhir untuk setiap kelas. Lapisan output (decision
7 layer) memiliki 3 target kelas sesuai dengan jumlah genus dan mengambil hasil dari lapisan penjumlahan yang terbesar dari 3 kelas yang dimiliki.
Gambar 4 Arsitektur PNN
Pada lapisan pola (pattern layer), keluaran dihitung dengan Persamaan 10 sebagai berikut:
f(x) = K xj-xij hj d
j=1
(10)
dengan:
d=dimensi vektor
K(z) =e-0.5*z2 (kernel normal)
xj=vektor input kolom ke-j
xij=vektor bobot baris ke-i kolom ke-j
hj=parameter pemulus ke-j (smoothing parameter)
Berikutnya, keluaran dari lapisan pola yang bersesuaian dengan kelas/kategori tertentu akan masuk ke lapisan penjumlahan (summation layer). Di masing-masing node akan dihitung dengan Persamaan 11 berikut:
p(x) = 1 (2π)
d 2h
1h2…hd
1
n K
xj-xij
hj
d
j=1 n
i=1
8 dengan:
d=dimensi vektor input
n=jumlah data latih di suatu kategori xj= vektor data uji ke-j
xij=data latih ke-i untuk ciri j
Dengan menjabarkan nilai kernel pada lapisan pola ke dalam lapisan penjumlahan maka didapatkan Persamaan 12 berikut:
p(x) = 1
xij=data latih ke-i untuk ciri-j (Specht 1990)
Terakhir, pada lapisan output (decision layer) akan ditetapkan hasil akhir kelas yang didapatkan dengan membandingkan setiap nilai dari hasil lapisan penjumlahan. Untuk nilai yang paling besar ditetapkan sebagai kelas.
Pengujian, Evaluasi, dan Analisis Hasil
Pengujian dilakukan dengan melakukan klasifikasi pada fragmen uji organisme dikenal dan organisme belum dikenal. Hasil tersebut kemudian dibandingkan dengan kelas hasil klasifikasi, apakah fragmen uji benar diklasifikasi atau salah diklasifikasi. Alat ukur pengklasifikasian yang digunakan ialah confusion matrix. Hasil penelitian diukur menggunakan tingkat akurasi dari fragmen metagenom yang diuji dan diamati. Nilai akurasi dihitung dengan Persamaan 13.
Akurasi = ∑data uji yang benar diklasifikasi
∑data uji x 100% (13)
Lingkungan Implementasi
Lingkungan implementasi yang digunakan pada penelitian ini adalah sebagai berikut:
1 Perangkat keras berupa notebook:
AMD Athlon Neo X2 Dual Core Processor
Memori 2 GB
Harddisk kapasitas 250 GB
Monitor dengan resolusi 1280 x 800 piksel
2 Perangkat keras virtual (virtual machine) untuk membantu perhitungan pengklasifikasian:
9
Memori 2 GB
Harddisk kapasitas 60 GB
Monitor dengan resolusi 1280 x 800 piksel 3 Perangkat lunak:
Sistem operasi Microsoft Windows XP Professional (perangkat keras notebook)
Sistem operasi Microsoft Windows 7 Professional (perangkat keras virtual)
Simulator metagenom MetaSim 0.9.1
Bloodshed Dev-C++
CodeBlocks 12.11
Matlab 7.7 (R2008b)
HASIL DAN PEMBAHASAN
Penyiapan Data
Penelitian ini menggunakan fragmen metagenom dari 3 genus, yaitu Agrobacterium, Bacillus, dan Staphylococcus. Fragmen tersebut dibagi menjadi kelompok dataset organisme dikenal dan organisme belum dikenal. Pembagian data organisme dapat dilihat pada Tabel 1 dan Tabel 2.
Tabel 1 Dataset organisme dikenal
Spesies Kelas Genus
Agrobacterium radiobacter K48 chromosome 2 Agrobacterium Agrobacterium tumefaciens str. C58 chromosome circular
Agrobacterium vitis S4 chromosome 1
Bacillus amyloliquefaciens FZB42 Bacillus Bacillus anthracis str. ‘Ames Ancestor’
Bacillus cereus 03BB102
Bacillus pseudofirmus OF4 chromosome
Staphylococcus aureus subsp. Sureus JH1 Staphylococcus Staphylococcus epidermidis 1228 chromosome
10
Tabel 2 Dataset organisme belum dikenal
Spesies Kelas Genus
Agrobacterium radiobacter K48 chromosome 1 Agrobacterium Agrobacterium tumefaciens str. C58 chromosome linear
Agrobacterium vitis S4 chromosome 2
Bacillus pumilus SAFR-032 Bacillus
Bacillus subtilis subsp. subtilis str. 16B chromosome Bacillus thuringiensis str. Al Hakam chromosome
Staphylococcus carnosus subsp. carnosus TM300 chromosome Staphylococcus Staphylococcus lugdunensis HKU09-01 chromosome
Staphylococcus saprophyticus subsp. saprophyticus ATCC 15305
Organisme belum dikenal diambil dari spesies yang berbeda dari organisme dikenal namun masih berada dalam satu genus. Organisme tersebut untuk sementara diasumsikan tidak dikenal dan belum diketahui kelas genusnya. Hal ini dimaksudkan sebagai skema pengujian kedua yang akan dilakukan untuk melakukan validasi pengklasifikasian.
Jumlah fragmen yang diambil dari organisme dikenal sebanyak 10 000 fragmen dari 10 spesies, sedangkan dari organisme belum dikenal diambil sebanyak 5 000 fragmen dari 9 spesies. Jumlah fragmen antara tiap kelas tidak berimbang. Panjang fragmen yang digunakan untuk kedua dataset tersebut sama, yaitu 500 bp, 1 kbp, 5 kbp, dan 10 kbp dengan 4 basa penyusunnya yaitu A, G, C, dan T.
Ekstraksi Ciri (K-Mers)
Proses k-mers akan menghasilkan ciri dari fragmen dengan melakukan pembacaan terhadap susunan fragmen dengan mengambil k-substring. Nilai k yang digunakan pada penelitian ini adalah 3. Dengan demikian proses pembacaan k-mers akan menghasilkan 43 kombinasi basa yaitu sebanyak 64 basa untuk setiap pembacaan satu fragmen metagenom. Basa-basa tersebut menjadi peubah yang akan digunakan untuk pengklasifikasian.
Pembacaan fragmen dilakukan terhadap dataset organisme dikenal dan organisme belum dikenal. Oleh karena itu, hasil ekstraksi ciri dari organisme dikenal akan menghasilkan matriks berukuran 10 000 x 64 dan untuk organisme belum dikenal akan menghasilkan matriks berukuran 5 000 x 64 untuk setiap panjang fragmen. Matriks tersebut berisi informasi frekuensi dari setiap pembacaan sekuen basa.
11 Reduksi Ciri (Biplot)
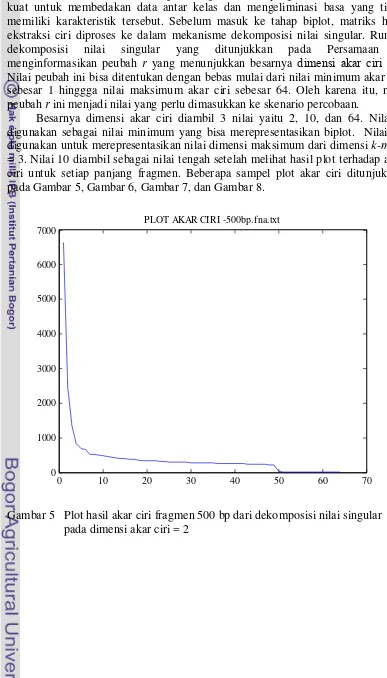
Reduksi ciri dilakukan dengan mengambil basa yang memiliki karakteristik kuat untuk membedakan data antar kelas dan mengeliminasi basa yang tidak memiliki karakteristik tersebut. Sebelum masuk ke tahap biplot, matriks hasil ekstraksi ciri diproses ke dalam mekanisme dekomposisi nilai singular. Rumus dekomposisi nilai singular yang ditunjukkan pada Persamaan 1, menginformasikan peubah r yang menunjukkan besarnya dimensi akar ciri (λ). Nilai peubah ini bisa ditentukan dengan bebas mulai dari nilai minimum akar ciri sebesar 1 hinggga nilai maksimum akar ciri sebesar 64. Oleh karena itu, nilai peubah r ini menjadi nilai yang perlu dimasukkan ke skenario percobaan.
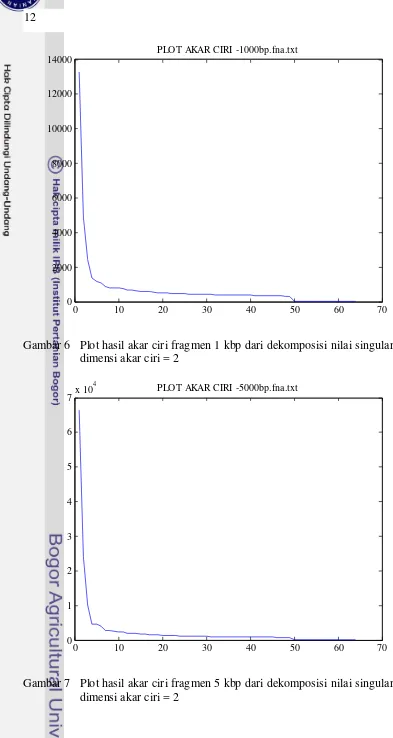
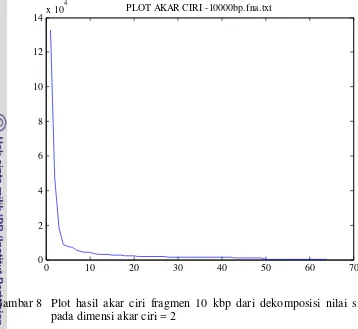
Besarnya dimensi akar ciri diambil 3 nilai yaitu 2, 10, dan 64. Nilai 2 digunakan sebagai nilai minimum yang bisa merepresentasikan biplot. Nilai 64 digunakan untuk merepresentasikan nilai dimensi maksimum dari dimensi k-mers = 3. Nilai 10 diambil sebagai nilai tengah setelah melihat hasil plot terhadap akar ciri untuk setiap panjang fragmen. Beberapa sampel plot akar ciri ditunjukkan pada Gambar 5, Gambar 6, Gambar 7, dan Gambar 8.
Gambar 5 Plot hasil akar ciri fragmen 500 bp dari dekomposisi nilai singular pada dimensi akar ciri = 2
0 10 20 30 40 50 60 70
0 1000 2000 3000 4000 5000 6000 7000
12
Gambar 6 Plot hasil akar ciri fragmen 1 kbp dari dekomposisi nilai singular pada dimensi akar ciri = 2
Gambar 7 Plot hasil akar ciri fragmen 5 kbp dari dekomposisi nilai singular pada dimensi akar ciri = 2
0 10 20 30 40 50 60 70
0 2000 4000 6000 8000 10000 12000 14000
PLOT AKAR CIRI -1000bp.fna.txt
0 10 20 30 40 50 60 70
0 1 2 3 4 5 6 7x 10
4
13
Gambar 8 Plot hasil akar ciri fragmen 10 kbp dari dekomposisi nilai singular pada dimensi akar ciri = 2
Mulai dari nilai akar ciri 1 sampai 10 karakteristiknya cenderung menurun tajam. Kemudian dari nilai akar ciri 10 hingga 64 plot cenderung menurun stabil dan tidak memiliki perbedaan yang cukup signifikan. Hal ini terjadi pada plot akar ciri untuk setiap panjang fragmen percobaan. Oleh karena itu, nilai 10 dianggap sebagai nilai tengah yang bisa mencakup keempat percobaan untuk diujikan.
Hasil dari dekomposisi nilai singular akan digunakan pada Persamaan 5 untuk menghasilkan 2 variabel biplot yaitu variabel G dan H. Variabel G merepresentasikan baris (biasanya objek) dan variabel H merepresentasikan kolom (biasanya peubah).
Biplot pada penelitian ini bertujuan untuk mereduksi ciri/peubah. Untuk setiap nilai H diplotkan ke dalam bentuk biplot untuk menggambarkan karakteristik setiap peubah. Jika percobaan yang dilakukan menggunakan 2 peubah r yang menunjukkan besarnya dimensi akar ciri (λ) maka dimensi matriks H yang dihasilkan sebesar 2 x 64. Matriks H kemudian dilakukan operasi transpose untuk merepresentasikannya sebagai titik koordinat pada 64 peubah.
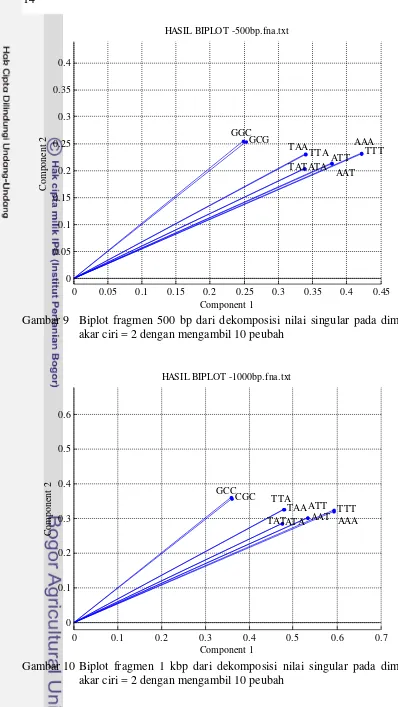
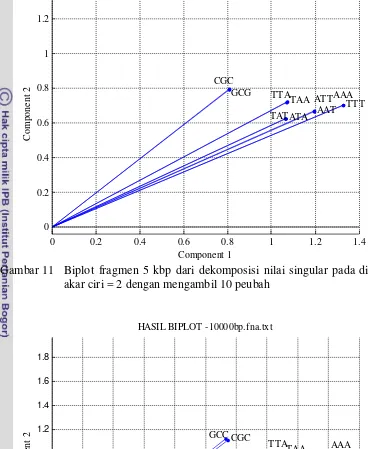
Semakin panjang garis peubah dari titik pusat ke titik koordinatnya maka mengindikasikan semakin besar pula pengaruhnya sebagai pembeda antar kelas. Reduksi ciri dilakukan dengan mengambil n-buah garis terpanjang. Nilai n ini ikut dimasukkan ke skenario percobaan untuk diteliti. Besarnya nilai n diambil 3 nilai yaitu 10, 20, dan 30 peubah. Keputusan ini diambil dengan tujuan untuk meneliti pengaruh reduksi ciri yang dilakukan lebih dari 50% komposisi awal peubah terhadap hasil pengklasifikasian. Beberapa contoh biplot dengan mengambil 10 peubah terpanjang disajikan pada Gambar 9, Gambar 10, Gambar 11, dan Gambar 12.
0 10 20 30 40 50 60 70
0 2 4 6 8 10 12 14x 10
4
14
Gambar 9 Biplot fragmen 500 bp dari dekomposisi nilai singular pada dimensi akar ciri = 2 dengan mengambil 10 peubah
Gambar 10 Biplot fragmen 1 kbp dari dekomposisi nilai singular pada dimensi akar ciri = 2 dengan mengambil 10 peubah
15
Gambar 11 Biplot fragmen 5 kbp dari dekomposisi nilai singular pada dimensi akar ciri = 2 dengan mengambil 10 peubah
Gambar 12 Biplot fragmen 10 kbp dari dekomposisi nilai singular pada dimensi akar ciri = 2 dengan mengambil 10 peubah
16
Pembagian Data
Ciri yang sudah direduksi melalui tahap biplot selanjutnya dibagi menjadi data latih dan data uji sebelum masuk ke tahap pengklasifikasian. Pembagian data latih dan data uji pada organisme dikenal dilakukan secara manual dengan komposisi 60%:40% dan 80%:20%. Total data organisme dikenal yang digunakan sebanyak 10 000 dengan komposisi 10 spesies berbeda dalam 3 genus. Pembagian dilakukan berimbang dengan mengambil komposisi yang sama untuk setiap kelasnya sehingga didapatkan 6 000 data latih dan 4 000 data uji untuk komposisi 60%:40% dan 8 000 data latih dan 2 000 data uji untuk komposisi 80%:20% dari 3 kelas genus.
Rangkaian Percobaan
Dari beberapa metode yang sudah diuraikan sebelumnya ditemukan beberapa parameter yang perlu diteliti pengaruhnya sehingga dimasukkan ke dalam skenario percobaan. Parameter tersebut meliputi:
1 Peubah r sebagai besaran dimensi akar ciri (λ) pada SVD. Untuk peubah ini ditetapkan menggunakan 3 nilai yaitu 2, 10, dan 64.
2 Peubah n sebagai banyaknya peubah yang digunakan dari hasil reduksi ciri. Untuk peubah ini ditetapkan menggunakan 3 nilai yaitu 10, 20, dan 30 peubah.
3 Persentase pembagian data latih dan data uji. Untuk persentase ini ditetapkan menggunakan 2 nilai yaitu 60%:40% dan 80%:20%.
Pengklasifikasian PNN yang ditunjukkan pada Persamaan 10 juga memiliki parameter pemulus (h) ditentukan secara bebas dan akan sangat berpengaruh terhadap hasil akhir akurasi klasifikasi. Dari beberapa kali percobaan akhirnya ditentukan 6 nilai yang cukup mencakup kombinasi percobaan untuk diteliti yaitu nilai h = 1, 8, 9, 10, 11, dan 20.
Percobaan yang akan dilakukan mengombinasikan keempat parameter tersebut sehingga didapatkan 120 percobaan untuk setiap panjang fragmen. Hasil dari 120 percobaan dilampirkan pada Lampiran 1 hingga Lampiran 8.
Evaluasi dan Analisis Seluruh Hasil Percobaan
Dari serangkaian kombinasi percobaan yang telah dilakukan dapat disimpulkan poin-poin sebagai berikut:
1 Hasil akurasi terbaik didapatkan dari percobaan dengan pembagian data latih 80%:20%, menggunakan 20 peubah, dengan besaran dimensi akar ciri r = 10 (SVD), dan nilai h pada klasifikasi PNN sebesar 10. Rata-rata akurasi yang dihasilkan sebesar 93.08%.
17 3 Percobaan dengan pembagian data latih 80%:20% menghasilkan akurasi yang
lebih baik daripada pembagian data latih 60%:40% untuk setiap percobaan. Oleh karena itu, dapat disimpulkan nilai akurasi dan banyaknya pembagian data latih memiliki korelasi positif.
4 Pada percobaan data latih dengan 10 peubah, klasifikasi menghasilkan nilai akurasi yang sama mulai dari dimensi akar ciri (r) sebesar 10 hingga 64 untuk semua parameter pemulus klasifikasi (h).
5 Penentuan reduksi ciri terbaik tidak dapat dilakukan dengan sembarang. Semakin banyak peubah dari hasil reduksi yang digunakan tidak menjamin hasil akurasi yang lebih baik. Hal ini terlihat dari percobaan menggunakan n = 30 peubah dengan pembagian data latih 60%:40%. Untuk semua besarnya dimensi vektor nilai akurasi pada percobaan 5 kbp lebih besar dibandingkan dengan percobaan dengan menggunakan 10 kbp.
6 Nilai h yang terbaik pada parameter PNN berbeda-beda untuk setiap pembagian data latih. Untuk pembagian data latih 60%:40%, nilai h mencapai maksimum pada nilai 8, sedangkan untuk pembagian data latih 80%:20% mencapai maksimum lebih variatif antara 8, 10, dan 20.
7 Besarnya dimensi akar ciri (r) yang maksimum pada dekomposisi nilai singular tidak menjamin nilai akurasi yang terbaik. Hal ini ditunjukkan oleh setiap percobaan yang menggunakan dimensi akar ciri 10 memiliki nilai akurasi lebih besar dibanding dimensi akar ciri 64.
Hasil percobaan data latih terbaik ditampilkan pada Tabel 3 dan Tabel 4 dengan menunjukkan nilai akurasi, waktu eksekusi klasifikasi, dan confusion matrix. Tabel 3 Nilai akurasi terbaik dan waktu eksekusi klasifikasi pada pengujian
organisme dikenal
18
Sebagai perbandingan efisiensi waktu antara reduksi menggunakan biplot terbaik dengan tanpa biplot disajikan hasil percobaan yang telah dilakukan pada Tabel 5.
Tabel 5 Perbandingan waktu eksekusi klasifikasi antara fragmen dengan biplot (terbaik) dan tanpa biplot
Untuk melihat variasi waktu eksekusi klasifikasi antara fragmen dengan menggunakan biplot terbaik dan tanpa biplot dilakukan 4 percobaan tambahan lain dengan mengubah susunan data latih dan data uji. Hasil dari percobaan variasi ini disajikan pada Tabel 6 dan Tabel 7.
Tabel 6 Waktu eksekusi klasifikasi fragmen dengan menggunakan biplot (terbaik) pada beberapa percobaan
Tabel 7 Waktu eksekusi klasifikasi fragmen tanpa menggunakan biplot pada beberapa percobaan
19
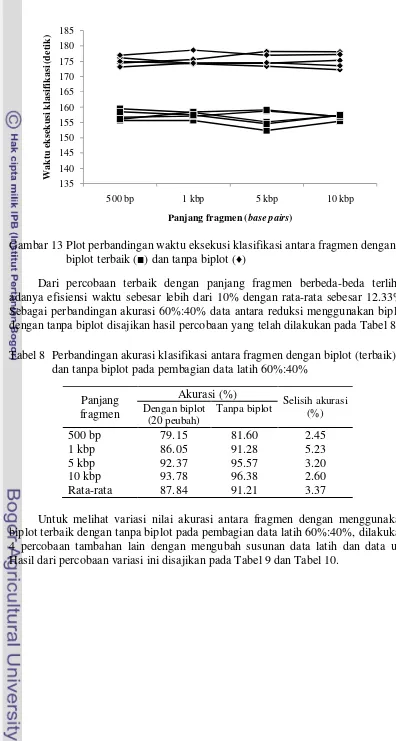
Gambar 13 Plot perbandingan waktu eksekusi klasifikasi antara fragmen dengan biplot terbaik (■) dan tanpa biplot (♦)
Dari percobaan terbaik dengan panjang fragmen berbeda-beda terlihat adanya efisiensi waktu sebesar lebih dari 10% dengan rata-rata sebesar 12.33%. Sebagai perbandingan akurasi 60%:40% data antara reduksi menggunakan biplot dengan tanpa biplot disajikan hasil percobaan yang telah dilakukan pada Tabel 8. Tabel 8 Perbandingan akurasi klasifikasi antara fragmen dengan biplot (terbaik)
dan tanpa biplot pada pembagian data latih 60%:40% Panjang
Untuk melihat variasi nilai akurasi antara fragmen dengan menggunakan biplot terbaik dengan tanpa biplot pada pembagian data latih 60%:40%, dilakukan 4 percobaan tambahan lain dengan mengubah susunan data latih dan data uji. Hasil dari percobaan variasi ini disajikan pada Tabel 9 dan Tabel 10.
135
20
Tabel 9 Nilai akurasi klasifikasi fragmen menggunakan biplot (terbaik) dengan pembagian data latih 60%:40% pada beberapa percobaan
Panjang
Tabel 10 Nilai akurasi klasifikasi fragmen tanpa menggunakan biplot dengan pembagian data latih 60%:40% pada beberapa percobaan
Panjang
Hasil perbandingan beberapa percobaan tersebut divisualisasikan dalam bentuk diagram garis pada Gambar 14.
Gambar 14 Plot perbandingan rata-rata nilai akurasi klasifikasi beberapa
percobaan antara fragmen dengan biplot terbaik dan tanpa biplot pada pembagian data latih 60%:40%
21 Tabel 11 Perbandingan akurasi klasifikasi antara fragmen dengan biplot (terbaik)
dan tanpa biplot pada pembagian data latih 80%:20% Panjang
Untuk melihat variasi nilai akurasi antara fragmen dengan menggunakan biplot terbaik dengan tanpa biplot pada pembagian data latih 80%:20%, dilakukan 4 percobaan tambahan lain dengan mengubah susunan data latih dan data uji. Hasil dari percobaan variasi ini disajikan pada Tabel 12 dan Tabel 13.
Tabel 12 Nilai akurasi klasifikasi fragmen menggunakan biplot (terbaik) dengan pembagian data latih 80%:20% pada beberapa percobaan
Panjang
Tabel 13 Nilai akurasi klasifikasi fragmen tanpa menggunakan biplot dengan pembagian data latih 80%:20% pada beberapa percobaan
Panjang
22
Gambar 15 Plot perbandingan rata-rata nilai akurasi klasifikasi beberapa
percobaan antara fragmen dengan biplot terbaik dan tanpa biplot pada pembagian data latih 80%:20%
Sebelum melanjutkan ke pengujian kedua, nilai anomali akurasi yang muncul pada setiap percobaan dengan parameter h = 1 diteliti lebih lanjut dengan melakukan 4 percobaan tambahan. Percobaan tambahan dilakukan dengan mengambil fragmen baru secara acak dengan panjang fragmen dan jumlah data fragmen yang sama. Hasil dari percobaan tersebut dilampirkan pada Lampiran 9 hingga Lampiran 16.
Hasil percobaan dengan data fragmen baru acak menunjukkan bahwa untuk setiap percobaan memiliki pola yang sama, yaitu adanya kecenderungan nilai akurasi yang menurun mulai dari panjang fragmen 5 kbp hingga 10 kbp. Kesimpulan yang diambil dari percobaan tersebut ialah nilai anomali tidak disebabkan oleh kombinasi data latih yang kurang baik, namun disebabkan oleh faktor yang lain.
Specht (1990) mengungkapkan bahwa nilai akurasi klasifikasi PNN yang dihasilkan tergantung dari ketepatan penggunaan nilai parameter pemulus (h) yang mendasari fungsi kepekatan peluang. Nilai h yang kecil mengakibatkan pola acak individu dalam semakin berpengaruh, sedangkan nilai h yang besar menujukkan sebaliknya dengan pola yang umum. Oleh karena itu, sesuai formula PNN pada Persamaan 12, nilai anomali pada klasifikasi sangat dipengaruhi oleh variasi individu dalam kelas untuk panjang fragmen 5 kbp hingga 10 kbp. Semakin panjang fragmennya semakin tinggi juga variasi antar kelas yang menyebabkan anomali hasil akurasi.
Data latih terbaik dari pengujian pertama selanjutnya digunakan sebagai data latih untuk menguji dataset organisme belum dikenal di pengujian kedua. Fragmen organisme belum dikenal juga direduksi dengan menggunakan peubah yang sama dengan yang digunakan oleh data latihnya. Dari hasil percobaan tersebut didapatkan hasil akurasi, waktu eksekusi klasifikasi, dan confusion matrix yang ditunjukkan pada Tabel 14 dan Tabel 15.
23 Tabel 14 Nilai akurasi dan waktu eksekusi klasifikasi pada dimensi pengujian
organisme belum dikenal
Tabel 15 Confusion matrix untuk pengujian terbaik pada pengujian organisme belum dikenal
Untuk melihat variasi nilai akurasi antara fragmen organisme belum dikenal maka dilakukan 4 percobaan tambahan lain dengan mengubah susunan data latih dan data uji. Hasil dari percobaan variasi ini disajikan pada Tabel 16.
Tabel 16 Nilai akurasi klasifikasi fragmen organisme belum dikenal pada beberapa percobaan
24
Gambar 16 Plot perbandingan rata-rata nilai akurasi klasifikasi beberapa
percobaan antara fragmen organisme dikenal dengan organisme belum dikenal
Pengujian pertama dan kedua menunjukkan hasil yang cukup baik. Nilai minimum akurasi didapatkan sebesar 85.45% pada pengujian pertama dan 80.46% pada pengujian kedua. Untuk akurasi maksimum didapatkan sebesar 98.10% pada pengujian pertama dan 92.80% pada pengujian kedua. Selisih di antara pengujian pertama dan kedua pun kecil dan kurang lebih berkisar di 5%.
Efisiensi waktu pengklasifikasian yang dihasilkan oleh biplot menunjukkan nilai sebesar lebih dari 10% untuk setiap panjang fragmen dengan rata-rata sebesar 12%. Hasil ini dirasakan cukup baik karena hanya menggunakan 20 peubah dari total 64 peubah dengan persentase reduksinya sebesar 68.75%.
Dari confusion matrix yang didapatkan, terlihat bahwa kesalahan klasifikasi lebih banyak terjadi pada genus Bacillus dan Staphylococcus. Hal ini disebabkan oleh masih dekatnya kekerabatan antara kedua genus tersebut yang masih berada dalam satu ordo.
SIMPULAN DAN SARAN
Simpulan
Kombinasi percobaan antara k-mers sebagai ekstraksi ciri, biplot sebagai pereduksi ciri, dan PNN sebagai classifier disimpulkan baik dalam mengklasifikasikan fragmen metagenom. Percobaan fragmen metagenom terpendek berhasil mengklasifikasikan 85.45% organisme dikenal dan 80.46% organisme belum dikenal. Percobaan fragmen terpanjang berhasil mengklasifikasikan 98.10% organisme dikenal dan 92.80% organisme belum dikenal.
Biplot yang berperan sebagai pereduksi ciri fragmen metagenom pada penelitian ini juga bekerja dengan baik. Nilai-nilai akurasi tersebut dihasilkan
25 dengan menggunakan 20 peubah hasil reduksi dari 64 peubah yang ada. Terdapat efisiensi waktu pengklasifikasian yang dihasilkan yaitu sebesar lebih dari 10% untuk setiap panjang fragmen dengan rata-rata sebesar 12%.
Saran
Percobaan ini menggunakan menggunakan 3 parameter yang nilainya bebas ditentukan yaitu peubah r sebagai besaran dimensi akar ciri (λ) pada SVD, peubah n sebagai banyaknya peubah yang digunakan hasil dari reduksi ciri, dan parameter pemulus (h) pada klasifikasi PNN. Ketiga nilai parameter tersebut sangat berpengaruh terhadap hasil akurasi pengklasifikasian dan ditentukan dengan nilai praduga penelitian. Untuk mendapatkan hasil akurasi terbaik dibutuhkan parameter terbaik yang sesuai. Oleh karena itu, perlu digali lebih dalam karakteristik dari dekomposisi nilai singular, biplot, dan parameter pemulus PNN dengan penelitian lebih lanjut. Dari penelitian ini belum ditemukan karakteristik dan korelasi dari ketiga parameter tersebut.
Untuk menguji realibilitas biplot sebagai pereduksi ciri yang baik akan lebih baik diujicobakan pada data dengan dimensi yang lebih besar yaitu menggunakan k-mers dengan nilai k yang lebih besar dari 3.
DAFTAR PUSTAKA
Buono A. 2014. Lecture note kuliah magister matematika dan statistika ilmu komputer. Bogor (ID): Institut Pertanian Bogor.
de Carvalho Jr SA. 2003. Sequence alignment algorithms [tesis]. London (GB): University of London.
Elliyana F. 2014. Klasifikasi fragmen metagenom menggunakan fitur spaced n-mers dan k-nearest neighbour [skripsi]. Bogor (ID): Institut Pertanian Bogor. Gabriel KR. 1971. The biplot graphic display of matrices with application to
principal component analysis. Biometrika. 58(3):453-467.doi:10.1093/biomet/58.3.453.
Kusuma WA, Akiyama Y. 2011. Metagenome fragment binning based on characterization vector. Di dalam: Proceedings of International Conference on Bioinformatics and Biomedical Technology; 2011 Mar 25-27; Sanya, China. hlm 50-54.
Rosen G, Garbarine E, Caseiro D, Polikar R, Sokhansanj B. 2008. Metagenome fragment clasification using n-mer frequency profiles. Advances in Bioinformatics. 2008(1):1-12.doi:10.1155/2008/205969.
Specht DF. 1990. Probabilistic neural networks. Neural Networks. 3(1):109-118.doi:10.1016/0893-6080(90)90049-Q.
26
Lampiran 1 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 10 peubah dan pembagian data latih dan data uji 60%:40%
Panjang
Lampiran 2 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 20 peubah dan pembagian data latih dan data uji 60%:40%
27 Lampiran 3 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 30
peubah dan pembagian data latih dan data uji 60%:40% Panjang
fragmen
Akurasi (%)
h = 1 h = 8 h = 9 h = 10 h = 11 h = 20
Dimensi akar ciri (λ) = 2
500 bp 77.58 77.55 76.53 75.75 75.35 74.63 1 kbp 84.33 87.78 87.83 87.33 87.03 81.00 5 kbp 31.83 93.87 93.77 93.70 93.60 94.25 10 kbp 30.33 93.25 93.25 93.18 93.10 92.93 Rata-rata 56.01 88.11 87.84 87.49 87.27 85.70
Dimensi akar ciri (λ) = 10
500 bp 77.98 77.33 76.58 76.15 75.53 75.03 1 kbp 85.18 88.23 88.10 87.98 87.40 81.33 5 kbp 31.88 94.67 94.62 94.55 94.50 94.40 10 kbp 30.40 93.50 93.53 93.48 93.45 93.10 Rata-rata 56.36 88.43 88.21 88.04 87.72 85.96
Dimensi akar ciri (λ) = 64
500 bp 77.65 77.23 76.50 76.03 75.53 74.65 1 kbp 85.18 88.23 88.10 87.98 87.40 81.33 5 kbp 31.83 94.07 94.02 94.00 94.10 94.47 10 kbp 30.38 93.48 93.48 93.48 93.48 93.18 Rata-rata 56.26 88.25 88.02 87.87 87.62 85.91
Lampiran 4 Nilai akurasi pada dimensi akar ciri yang berbeda dengan semua peubah (tanpa biplot) dan pembagian data latih dan data uji 60%:40%
Panjang fragmen
Akurasi (%)
28
Lampiran 5 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 10 peubah dan pembagian data latih dan data uji 80%:20%
Panjang
Lampiran 6 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 20 peubah dan pembagian data latih dan data uji 80%:20%
29 Lampiran 7 Nilai akurasi pada dimensi akar ciri yang berbeda dengan n = 30
peubah dan pembagian data latih dan data uji 80%:20% Panjang
fragmen
Akurasi (%)
h = 1 h = 8 h = 9 h = 10 h = 11 h = 20
Dimensi akar ciri (λ) = 2
500 bp 79.05 83.70 83.60 83.55 83.80 84.10 1 kbp 84.55 89.85 90.80 91.25 91.55 91.05 5 kbp 29.40 96.30 96.25 96.20 96.25 97.15 10 kbp 28.40 98.65 98.60 98.60 98.60 98.55 Rata-rata 55.35 92.13 92.31 92.40 92.55 92.71
Dimensi akar ciri (λ) = 10
500 bp 79.25 83.65 83.90 83.45 83.55 84.45 1 kbp 85.40 90.20 91.15 91.80 91.95 91.05 5 kbp 29.15 96.95 97.00 96.90 96.85 97.50 10 kbp 28.40 98.15 98.15 98.10 98.10 98.00 Rata-rata 55.55 92.24 92.55 92.56 92.61 92.75
Dimensi akar ciri (λ) = 64
500 bp 78.10 83.60 83.70 83.60 83.95 84.35 1 kbp 85.40 90.20 91.15 91.80 91.95 91.05 5 kbp 29.10 96.60 96.45 96.50 96.55 97.20 10 kbp 28.25 97.95 97.95 97.95 97.95 97.90 Rata-rata 55.21 92.09 92.31 92.46 92.60 92.63
Lampiran 8 Nilai akurasi pada dimensi akar ciri yang berbeda dengan semua peubah (tanpa biplot) dan pembagian data latih dan data uji 80%:20%
Panjang fragmen
Akurasi (%)
30
Lampiran 9 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 10 peubah, dan pembagian data latih dan data uji 60%:40%
31 10 kbp 31.73 31.98 31.55 32.10 32.08
Rata-rata 57.34 57.02 57.44 56.72 57.08
Lampiran 11 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 30 peubah, dan pembagian data latih dan data uji 60%:40%
Panjang fragmen
Akurasi (%)
1 2 3 4 5
Dimensi akar ciri (λ) = 2
500 bp 77.58 77.75 77.90 77.28 76.53 1 kbp 84.33 85.05 83.85 84.30 84.25 5 kbp 31.83 31.19 32.58 30.95 31.60 10 kbp 30.33 30.45 30.43 30.45 30.95 Rata-rata 56.01 56.11 56.19 55.74 55.83
Dimensi akar ciri (λ) = 10
500 bp 77.98 78.25 77.78 78.05 75.78 1 kbp 85.18 84.88 85.10 84.33 84.63 5 kbp 31.88 31.07 32.43 31.05 31.60 10 kbp 30.40 30.38 30.30 30.58 30.98 Rata-rata 56.36 56.14 56.40 56.00 55.75
Dimensi akar ciri (λ) = 64
500 bp 77.65 78.08 77.23 77.55 77.38 1 kbp 85.18 84.88 85.10 84.43 85.15 5 kbp 31.83 31.09 32.38 30.90 31.50 10 kbp 30.38 30.35 30.13 30.53 30.88 Rata-rata 56.26 56.10 56.21 55.85 56.23
Lampiran 12 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, semua peubah (tanpa biplot), dan pembagian data latih dan data uji 60%:40%
Panjang fragmen
Akurasi (%)
1 2 3 4 5
32
Lampiran 13 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 10 peubah, dan pembagian data latih dan data uji 80%:20%
33 10 kbp 28.95 28.74 28.95 28.95 29.54
Rata-rata 56.28 55.75 56.10 55.35 55.76
Lampiran 15 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, n = 30 peubah, dan pembagian data latih dan data uji 80%:20%
Panjang fragmen
Akurasi (%)
1 2 3 4 5
Dimensi akar ciri (λ) = 2 500 bp 79.05 78.60 78.15 78.19 78.95 1 kbp 84.55 85.21 86.10 86.16 84.69 5 kbp 29.40 28.95 29.35 29.45 28.75 10 kbp 28.40 27.99 28.35 28.45 29.04 Rata-rata 55.35 55.19 55.49 55.56 55.36
Dimensi akar ciri (λ) = 10
500 bp 79.25 78.90 78.50 78.84 76.55 1 kbp 85.40 85.91 87.40 86.41 84.69 5 kbp 29.15 28.85 29.30 29.60 28.70 10 kbp 28.40 28.09 28.35 28.30 29.04 Rata-rata 55.55 55.44 55.89 55.79 54.74
Dimensi akar ciri (λ) = 64
500 bp 78.10 80.15 77.90 78.54 77.35 1 kbp 85.40 85.91 87.40 85.71 85.34 5 kbp 29.10 28.85 29.30 29.60 28.70 10 kbp 28.25 28.04 28.40 28.35 29.04 Rata-rata 55.21 55.74 55.75 55.55 55.11
Lampiran 16 Nilai anomali akurasi beberapa percobaan pada dimensi akar ciri yang berbeda dengan h = 1, semua peubah (tanpa biplot), dan pembagian data latih dan data uji 80%:20%
Panjang fragmen
Akurasi (%)
1 2 3 4 5
34