ABSTRAK
AGUNG LUTHFI FAUZAN
. Karakteristik genotipe hibrida hasil persilangan 3
strain ikan nila
Oreochromis niloticus
dengan metode
Random Amplified
Polymorphic DNA
(RAPD). Dibimbing oleh Dinar Tri Soelistyowati dan Rudhy
Gustiano.
Penelitian ini bertujuan untuk mengetahui keragaman genetik dan kekerabatan
hibrida dari hasil persilangan resiprok tiga strain ikan nila (BEST, Nirwana, dan
Red
NIFI) dengan menggunakan metode
Random Amplified Polymorphic DNA
(RAPD
)
. Hasil penelitian menunjukkan bahwa hibridisasi intraspesifik strain ikan
nila meningkatkan keragaman genetik sebesar 27,93% yaitu pada persilangan
antara jantan Nirwana dengan betina BEST dan 27,23% dengan betina
Red
NIFI.
Berdasarkan analisis kekerabatan diketahui kedekatan Nirwana dan
Red
NIFI
dengan kelompok hibrid, dibandingkan dengan BEST. Jarak genetik terbesar
adalah 0,6 antara Nirwana dengan BEST.
ABSTRACT
AGUNG LUTHFI FAUZAN.
Characteristic of the genotypes hybrids resulted
from reciprocal cross of the three strains Nile tilapine using Random Amplified
Polymorphic DNA. Supervised by Dinar Tri Soelistyowati and Rudhy Gustiano.
Objectives of this study were to observe the genetic variability and relationship
among hybrids of three strains (BEST, Nirwana, and Red NIFI) using Random
Amplified Polymorphic DNA (RAPD). The results showed that hybridization
enabled to increase genetic variability respectedly on progeny between male
Nirwana with female BEST (27.93%) as well as between male Nirwana with
female Red NIFI (27.23%). Based on relationship analysis there were indication
that Nirwana and Red NIFI were closer to the hybrid groups, compared to BEST
one. The greatest genetic distance was the hybrids of Nirwana x BEST (0.6).
I.
PENDAHULUAN
Perikanan budidaya menjadi target utama pemerintah dalam upaya
meningkatkan hasil perikanan nasional. Indonesia ditargetkan menjadi produsen
hasil perikanan dan kelautan terbesar pada Tahun 2015 oleh Menteri Kelautan dan
Perikanan terutama produksi perikanan budidaya dicanangkan meningkat sebesar
353%. Salah satu komoditas unggulan budidaya yang ditargetkan adalah ikan nila.
Ikan nila (
Oreochromis niloticus
) merupakan jenis ikan introduksi dari negara
Taiwan yang sudah lama dibudidayakan secara luas di Indonesia sejak Tahun
1969 karena mudah penanganannya, tidak ada kendala reproduksi, dapat
mencapai ukuran tubuh relatif besar, toleransi terhadap lingkungan yang luas,
harga relatif murah dan rasa dagingnya enak.
Ikan nila sebagai komoditas budidaya yang tersebar disebagian besar
wilayah di Indonesia menunjukkan kecenderungan penurunan performa fenotipik
yaitu pertumbuhan lambat, tingkat kematian tinggi, dan ukuran individu kecil. Hal
tersebut diduga terkait dengan
potensial fitness
dari sumber genetik populasi yang
tidak optimal atau bahkan menurun karena sistem rekrutmen calon induk dan
persilangannya tidak terpola yang mengakibatkan penghanyutan sejumlah alel
(
genetic drift
) secara terus menerus sehingga terjadi penurunan keragaman
genetik. Penurunan keragaman genetik akibat hilangnya alel-alel potensial pada
populasi ikan dari hasil perbenihan dapat mengakibatkan terhambatnya laju
pertumbuhan dan rendahnya ketahanan ikan terhadap serangan penyakit dan
perubahan lingkungan (Leary
et al.
, 1985
dalam
Moria
et al.
, 2005).
2
Identifikasi genotipe dapat dilakukan dengan beberapa teknik, diantaranya
adalah menggunakan marka
Random Amplified Polymorphic DNA
(RAPD)
dengan teknik PCR. Marka RAPD adalah sekuens DNA polimorfik yang
dipisahkan oleh gel elektroforesis setelah proses PCR menggunakan satu primer
oligonukleotida pendek secara acak. RAPD sangat baik digunakan untuk
mendeteksi polimorfisme gen dalam jumlah besar karena primer oligonukleotida
bisa mendata semua genom yang memiliki situs ikatan dalam reaksi PCR
(Mulyasari, 2007).
II. BAHAN DAN METODE
Ikan Uji
Ikan uji yang digunakan adalah ikan nila hibrida hasil persilangan resiprok 3 strain BEST,
Nirwana dan Red NIFI koleksi Balai Riset Perikanan Budidaya Air Tawar Sempur, Bogor.
Rancangan persilangan resiprok selengkapnya disajikan pada Tabel 1.
Tabel 1. Rancangan persilangan resiprok 3 strain ikan nila
Jantan
BEST
Nirwana
Red NIFI
Betina
BEST
BB
NB
RB
Nirwana
BN
NN
RN
Red NIFI
BR
NR
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
(BESTxBEST), RN (Red NIFIxNirwana)
Ekstraksi DNA
Metode yang digunakan dalam ekstraksi dan pemurnian genom DNA berdasarkan prosedur
Fermentas dengan menggunakan Genomic DNA Purification KIT. Tahapan kerja yang dilakukan
meliputi:
Ekstraksi DNA dilakukan dengan membersihkan organ sirip dari larutan fiksatif (alkohol 70%),
dengan cara direndam dan dirotasi ke dalam aquades, hingga sampel berada pada dasar wadah.
Sampel diambil, dipotong kecil-kecil, lalu 20-25 mg sampel dengan 400 (l lysis solution
dimasukkan dalam tabung eppendorf 1,5 ml, dicampur menggunakan vortex sampai homogen
selama 20 detik, kemudian diinkubasi pada suhu 65 °C selama 5 menit. Selanjutnya chloroform
sebanyak 600 (l dimasukkan, lalu dicampur menggunakan vortex selama 20 detik, disentrifuse
pada kecepatan 10.000 rpm selama 2 menit.
Precipitation solution dilakukan dengan mencampurkan 720 (l H2O dengan 80 (l diambil dari
10x konsentrasi solution. Supernatan dipindahkan ke tabung eppendorf baru yang berisi 800 (l
precipitation solution, dicampur menggunakan vortex pada suhu ruang selama 2 menit.
Disentrifuse pada kecepatan 10.000 rpm selama 2 menit hingga DNA mengendap. Supernatan
yang mengandung DNA diambil dan dikeringkan. Setelah kering supernatan ditambahkan 100 (l
NaCl (1,2 M). Etanol absolut dingin sebanyak 300 (l dimasukkan, kemudian endapan DNA
diinkubasi pada suhu -20 °C selama 10 menit. Selanjutnya disentrifuse pada kecepatan 10.000
rpm selama 3-4 menit. Etanol dalam tabung eppendorf dibuang kemudian dibilas dengan etanol
dingin 70%, atau setelah kering dapat langsung ditambahkan 100 (l H2O.
Keberadaan DNA genom dapat diketahui dengan cara elektroforesis yakni menambahkan 3 (l
DNA hasil ekstraksi dengan 1 (l loading dye pada gel agarose 1%. Media elektroforesis atau gel
agarose 1% dibuat dengan agarose dimasukkan ke dalam tabung erlenmeyer sebanyak 0,30 g
dalam 30 ml larutan Tris-Borate-EDTA (TBE 1%). Kemudian batu magnetic stirer dimasukan
untuk mengaduk larutan, tabung ditutup dengan aluminium foil. Tabung erlenmeyer diletakkan
hingga berwarna bening, selanjutnya dituang ke dalam cetakan ukuran dan sisir (comb) dipasang
untuk membentuk sumur (well). Gel yang telah membeku dapat langsung digunakan untuk
elektroforesis atau disimpan dengan direndam dalam larutan TBE 1%.
Setelah DNA dan loading dye dimasukkan dalam sumur gel, selanjutnya bak elektroforesis
dialirkan listrik dengan tegangan 100 V dengan kuat arus yang terprogram secara otomatis.
Proses elektroforesis dihentikan setelah DNA bermigrasi dari kutub negatif ke kutub positif
mencapai tiga per empat bagian panjang gel. Hasil elektroforesis diwarnai dengan merendam gel
dalam larutan ethidium bromide dengan konsentrasi 5 ppm selama 20 menit. Keberadaan DNA
pada gel dapat dilihat dengan menggunakan ultraviolet transilluminator dan didokumentasikan
dengan film polaroid.
Random Amplified Polymorphic DNA (RAPD)
Jenis primer yang digunakan dalam penelitian ini disajikan pada Tabel 2 sebagai berikut.
Tabel 2. Jenis primer yang digunakan dalam RAPD-PCR
No.
Kode Primer
Urutan basa (5’–3’)
Panjang nukleotida
G+C (%)
1.
OPA-02
TGC CGA GCT G
10-mer
2.
OPA-03
AGT CAG CCA C
10-mer
60%
3.
OPC-05
GTC CCG ACG A
10-mer
70%
Amplifikasi DNA dilakukan menggunakan metode RAPD-PCR dengan komposisi bahan: 1 (l
primer, 3 (l DNA, 12,5 (l 2x PCR Master Mix dan 8,5 (l H2O nuklease free; dengan total volume
25 (l dicampur menjadi 1 unit. Seluruh bahan dicampur dengan cara memasukkannya ke dalam
tabung eppendorf 0,5 ml kemudian dihomogenkan menggunakan vorteks selama 20 detik dan
diputar menggunakan spin sampai tidak ada gelembung. Selanjutnya dimasukkan dalam
thermocycler dengan siklus sebanyak 35 cycle, yaitu: satu siklus denaturasi awal pada suhu 94
°C selama 2 menit, 34 siklus selanjutnya terdiri atas denaturasi pada suhu 94 °C selama 1 menit,
annealing 36 °C selama 1 menit dan extention72 °C selama 2 menit. Final extention pada suhu
72 °C selama 7 menit. Hasil RAPD-PCR dilihat melalui elektroforesis atau disimpan di dalam
lemari pendingin dengan suhu 4 °C. Untuk satu sampel ikan, proses PCR ini dilakukan sebanyak
jumlah primer yang digunakan (3 primer). Setiap satu kali proses PCR, jumlah primer yang
digunakan sebanyak 1 primer, hingga ketiga primer selesai digunakan.
Elektroforesis
Setelah didapatkan DNA hasil amplifikasi, selanjutnya dilakukan tahapan elektroforesis dengan
cara menambahkan 2 (l loading dye dengan 7 (l DNA hasil amplifikasi pada gel agarose 1,5%,
kemudian dimasukkan pada sumur elektroforesis. Salah satu sumur diisi dengan marker atau
akuades. Selanjutnya dilakukan tahapan running dan staining, lalu diamati di atas ultraviolet
transilluminator dan didokumentasikan dengan film polaroid.
Analisis Data
Tingkat polimorfisme dan heterozigositas dianalisa dengan menggunakan descriptive statistics
(Miller, 1997) serta analisis statistik menggunakan exact test for population differentiation
(Raymond & Rousset, 1995 dalam Miller, 1997) dengan menggunakan program TFPGA (Tools
for Population Genetic Analyses). Kekerabatan antar populasi dianalisis dengan menggunakan
jarak genetik, berdasarkan program UPGMA Wright (1978) modifikasi Rogers (1972) dalam
Miller (1997) dari software TFPGA. Data yang dihasilkan dari penggunaan program tersebut
III.
HASIL DAN PEMBAHASAN
3.1
Hasil
3.1.1
Profil RAPD
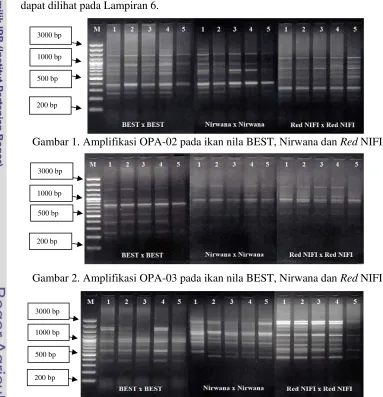
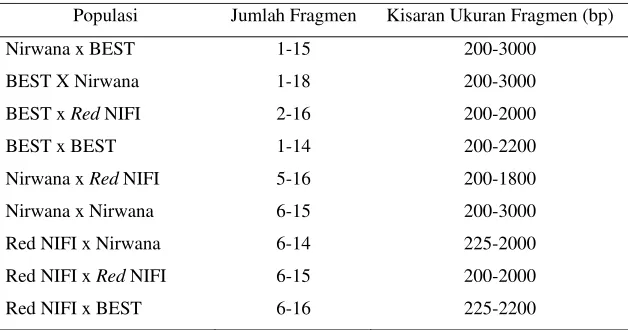
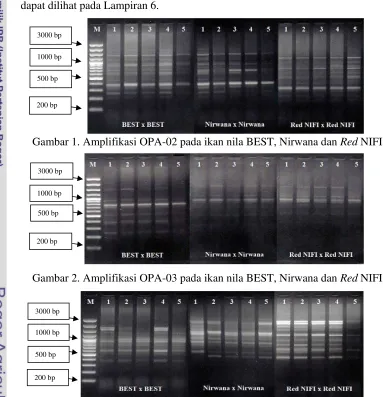
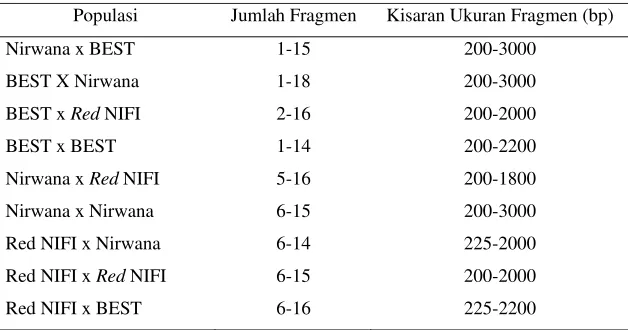
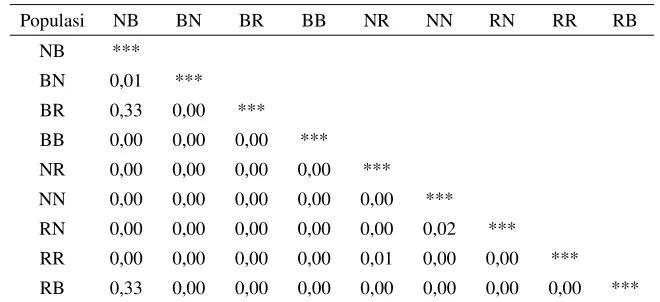
Keragaman profil RAPD meliputi jumlah dan ukuran fragmen DNA. Hasil
amplifikasi dengan menggunakan 3 primer (OPA-02, OPA-03 dan OPC-05)
menunjukkan hasil yang bervariasi, data selengkapnya disajikan pada Tabel 3.
Amplifikasi DNA pada 3 populasi
true breeding
(BEST, Nirwana dan
Red
NIFI)
ikan nila uji dapat dilihat pada Gambar 1-3. Sedangkan untuk hasil hibridisasinya
dapat dilihat pada Lampiran 6.
Gambar 1. Amplifikasi OPA-02 pada ikan nila BEST, Nirwana dan
Red
NIFI
Gambar 2. Amplifikasi OPA-03 pada ikan nila BEST, Nirwana dan
Red
NIFI
Gambar 3. Amplifikasi OPC-05 pada ikan nila BEST, Nirwana dan
Red
NIFI
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
8
Tabel 3. Profil DNA hasil persilangan resiprok 3 strain ikan nila
Populasi Jumlah Fragmen Kisaran Ukuran Fragmen (bp)
Nirwana x BEST 1-15 200-3000
BEST X Nirwana 1-18 200-3000
BEST x Red NIFI 2-16 200-2000
BEST x BEST 1-14 200-2200
Nirwana x Red NIFI 5-16 200-1800
Nirwana x Nirwana 6-15 200-3000
Red NIFI x Nirwana 6-14 225-2000
Red NIFI x Red NIFI 6-15 200-2000
Red NIFI x BEST 6-16 225-2200
Jumlah dan ukuran fragmen DNA pada 9 populasi ikan nila hasil hibridisasi
menunjukkan hasil yang bervariasi. Untuk 3 populasi
true breeding
yaitu BEST
memiliki jumlah dan ukuran fragmen DNA berkisar 1-14 dan 200-2200 bp,
Nirwana (6-15, 200-3000 bp), dan
Red
NIFI (6-15, 200-2000 bp). Sedangkan
hasil hibridisasinya yaitu NirwanaxBEST (1-15, 200-3000 bp), BESTxNirwana
(1-18, 200-3000 bp), BESTx
Red
NIFI (2-16, 200-2000 bp), Nirwanax
Red
NIFI
(6-15, 200-1800 bp),
Red
NIFIxNirwana (6-14, 225-2000 bp),
Red
NIFIxBEST
(6-16, 225-2200 bp).
3.1.1
Polimorfisme
Persentase polimorfisme dianalisis menggunakan program TFPGA
(Lampiran 1). Data persentase polimorfisme selengkapnya disajikan pada Tabel 4.
Tabel 4. Persentase polimorfisme hasil persilangan resiprok 3 strain ikan nila
Jantan
BEST Nirwana Red NIFI
Betina
BEST 20,31% 27,93% 15,13%
Nirwana 19,80% 15,41% 10,46%
Red NIFI 26,91% 27,23% 17,04%
9
(27,93%) dan terendah pada populasi
Red
NIFIxNirwana (10,46%), sedangkan
pada
true breeding
yaitu populasi NirwanaxNirwana (15,41%).
3.1.2
Heterozigositas
Keragaman genetik suatu populasi ikan dapat diketahui melalui nilai
heterozigositas intrapopulasi (Lampiran 3). Nilai heterozigositas bervariasi dari
0,04 hingga 0,12. Pada populasi
true breeding
menunjukkan tingkat
heterozigositas yang lebih rendah dari pada populasi hasil persilangannya yaitu
BESTx
Red
NIFI (0,12) dan Nirwanax
Red
NIFI (0,11). Data selengkapnya
disajikan pada Tabel 5.
Tabel 5. Tingkat heterozigositas hasil persilangan resiprok 3 strain ikan nila
Jantan
BEST Nirwana Red NIFI
Betina
BEST 0,08 0,09 0,06
Nirwana 0,07 0,07 0,04
Red NIFI 0,12 0,11 0,06
3.1.3
Uji Perbandingan Berpasangan F
stSecara statistik dengan menggunakan uji perbandingan berpasangan
F
st(Lampiran 4) menunjukkan terdapat perbedaan genetik secara nyata antara
populasi hasil hibridisasi kecuali populasi NirwanaxBEST, BESTx
Red
NIFI dan
Red
NIFIxBEST (Tabel 6).
Tabel 6. Nilai P dari uji perbandingan berpasangan
F
stdari rata-rata 3 primer
Populasi NB BN BR BB NR NN RN RR RB
NB ***
BN 0,01 ***
BR 0,33 0,00 ***
BB 0,00 0,00 0,00 ***
NR 0,00 0,00 0,00 0,00 ***
NN 0,00 0,00 0,00 0,00 0,00 ***
RN 0,00 0,00 0,00 0,00 0,00 0,02 ***
RR 0,00 0,00 0,00 0,00 0,01 0,00 0,00 ***
RB 0,33 0,00 0,00 0,00 0,00 0,00 0,00 0,00 ***
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
10
3.1.4
Jarak Genetik
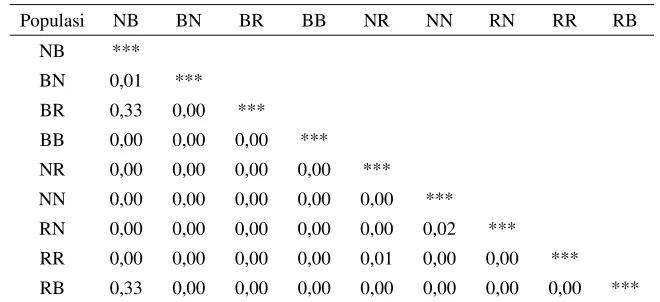
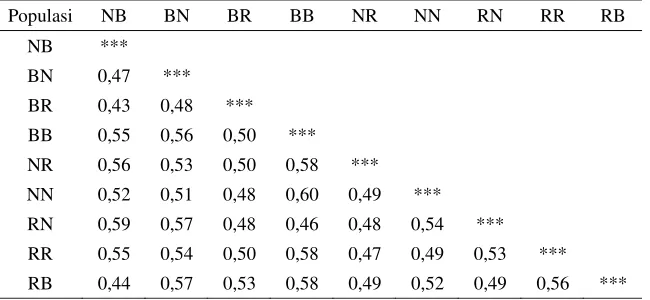
Jarak genetik interpopulasi menunjukkan perbedaan dan dapat
menggambarkan status kekerabatan antar populasi ikan nila. Jarak genetik
interpopulasi ketiga strain dan hasil hibridisasi intraspesifik 3 populasi ikan
disajikan pada Tabel 7. Jarak genetik tertinggi adalah antara populasi
BESTxBEST dengan NirwanaxNirwana sebesar 0,60, sedangkan jarak genetik
terendah adalah 0,43 yaitu antara populasi NirwanaxBEST dengan BESTx
Red
NIFI. Secara umum, jarak genetik 9 populasi ikan nila uji memiliki kisaran yang
tinggi yaitu antara 0,43-0,60.
Tabel 7. Jarak genetik antara 9 populasi ikan nila dari rata-rata 3 primer
Populasi NB BN BR BB NR NN RN RR RB
NB ***
BN 0,47 ***
BR 0,43 0,48 ***
BB 0,55 0,56 0,50 ***
NR 0,56 0,53 0,50 0,58 ***
NN 0,52 0,51 0,48 0,60 0,49 ***
RN 0,59 0,57 0,48 0,46 0,48 0,54 ***
RR 0,55 0,54 0,50 0,58 0,47 0,49 0,53 ***
RB 0,44 0,57 0,53 0,58 0,49 0,52 0,49 0,56 ***
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
(BESTxBEST), RN (Red NIFIxNirwana)
11
0.60 0.45 0.30 0.15 0.00
NB
BR
BN
RB
NR
RR
NN
BB
RN
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
(BESTxBEST), RN (Red NIFIxNirwana)
Gambar 4. Dendrogram 9 populasi ikan nila hasil hibridisasi
12
3.2
Pembahasan
Profil RAPD pada populasi
true breeding
(BEST, Nirwana dan
Red
NIFI)
memiliki karakter yang berbeda. Panjang dan ukuran fragmen DNA pada populasi
BESTxBEST lebih tinggi yaitu 1-14 dan 200-2200 bp dari pada pupulasi
NirwanaxNirwana (6-15, 200-3000 bp), dan
Red
NIFI (6-15, 200-2000 bp).
Sedangkan untuk hasil hibridisasinya, pada beberapa populasi menunjukkan
panjang dan ukuran fragmen DNA yang lebih tinggi, yaitu populasi
NirwanaxBEST (1-15, 200-3000 bp), BESTxNirwana (1-18, 200-3000 bp),
BESTx
Red
NIFI (2-16, 200-2000 bp). Hasil tersebut menunjukkan bahwa
hibridisasi dapat meningkatkan keragaman genetik yang dilihat dari panjang dan
ukuran fragmen DNA pada masing-masing populasi ikan uji.
Tingkat polimorfisme antara ketiga populasi
true breeding
yang tertinggi
adalah pada populasi BESTxBEST sebesar 20,31% lebih tinggi dibandingkan
populasi NirwanaxNirwana (15,41% ) dan
Red
NIFIx
Red
NIFI (17,04%). Sedang
pada hasil hibridisasinya menunjukkan polimorfisme yang lebih tinggi dari
true
breedingnya
yaitu pada populasi NirwanaxBEST sebesar 27,93%, populasi
Nirwanax
Red
NIFI (27,23%) dan populasi BESTx
Red
NIFI (26,91%). Hal
tersebut menunjukkan bahwa proses hibridisasi dapat meningkatkan keragaman
genetik ikan nila. Sedangkan pada populasi hibrid
Red
NIFIxBEST,
Red
NIFIxNirwana, dan BESTxNirwana menunjukkan persentase polimorfisme yang
lebih rendah yaitu 15,13%, 10,46% dan 19,80%. Penyebab rendahnya persentase
polimorfisme pada jenis ikan budidaya antara lain karena aktivitas seleksi induk,
terjadinya
inbreeding
,
genetic drift
dan fenomena
bottleneck effect
yang
merupakan akibat dari tidak terpenuhinya
gene pool
yang tidak lengkap
konfigurasinya pada populasi budidaya tersebut. Hal ini banyak ditemui pada
sistem budidaya ikan/udang di Indonesia yang disebabkan keterbatasan jumlah
induk yang digunakan dalam memproduksi benih sehingga tidak memenuhi
standar
Ne
(
effective breeding number
) (Kirpichnikov, 1981; Kapunsckinski dan
Jacobson, 1987; Tave, 1993)
13
perubahan genetik pada populasi ikan melalui persilangan hibridisasi. Strain
BEST, Nirwana dan
Red
NIFI memiliki proses seleksi yang berbeda. Ikan nila
BEST merupakan ikan hasil pemuliaan untuk karakter keunggulan pertumbuhan
yang dihasilkan melalui proses selama 4 (empat) tahun penelitian oleh Tim
Peneliti dari Balai Riset Perikanan Budidaya Air Tawar Bogor (Arifin, 2008).
Beberapa keunggulan strain ikan nila BEST diantaranya s
urvival rate
(SR)
sebesar 90% dibandingkan nila lokal (60-70%). Laju pertumbuhan 4,85%, lebih
tinggi dibandingkan 2 nila unggulan lainnya, yaitu: Gesit (4,5%) dan Nirwana
(3,93%),
food convertion ratio
(FCR) sebesar 1,1 (Anmi, 2009). Ikan nila
Nirwana merupakan hasil seleksi famili dengan sumber genetik berasal dari ikan
nila GIFT (
Genetic Improvement Farm Tilapia
) dan nila GET (
Genetically
Enchanced Tilapia
) dari Philipina. Menurut Sumantadinata (2006), bahwa
pertumbuhan bobot ikan nila Nirwana meningkat sekitar 45% pada generasi ke-3
(F3) dibandingkan dengan populasi awalnya (Anonim, 2007). Nila
Red
NIFI
dikenal juga sebagai nila merah atau nirah. Ikan ini kemungkinan merupakan hasil
persilangan antara
Tilapia mossambicus
(mujair) atau
Oreochromis niloticus
(nila) dengan
Oreochromis honorum
,
Oreochromis aureus
, atau
Oreochromis zilii
(Amri, 2008).
Tingkat heterozigositas populasi ikan nila hasil hibridisasi berkisar antara
0,04-0,12. Kisaran tersebut sebanding dengan tingkat heterozigositas pada
populasi ikan air tawar di alam, misalnya populasi ikan batak (
Tor sorro
) berkisar
0,08–0,09 (Asih
et al.
, 2006). Pada populasi BESTx
Red
NIFI dan Nirwanax
Red
NIFI sedikit lebih tinggi dibandingkan hasil persilangan lainnya; yaitu sebesar
0,12 dan 0,11. Hasil tersebut menunjukkan bahwa proses hibridisasi dapat
meningkatkan keragaman genetik dan nilai heterozigositas. Pada dasarnya,
hibridisasi adalah memanfaatkan sifat heterosis karena sifat dominan dan
heterozigositas pada banyak lokus (Tave, 1993) atau interaksi dari alel pada lokus
(Kapunsckinski dan Jacobson, 1987).
14
hal ini program seleksi untuk suatu sifat yang dilakukan cukup lama dapat
menurunkan potensi sifat genetik atau dapat menurunkan keragaman genetiknya.
Seleksi merupakan suatu teknik untuk memperbaiki sifat yang terukur
(
quantitative trait
). Prinsip dasar seleksi adalah mengeksploitasi sifat
addictive
dari alel-alel pada semua lokus yang mengontrol sifat terukur untuk memperbaiki
suatu populasi (Kirpichnikov, 1981; Falconer, 1981; Tave, 1993; Gjedrem, 2005).
Rendahnya keragaman genetik diduga juga dapat disebabkan jumlah induk
atau cara yang digunakan dalam pemijahan terbatas (rekrutmen induk tidak
terkontrol) atau penggunaan induk dalam waktu lama. Hal tersebut didukung oleh
pernyataan Secara umum penggunaan jumlah induk yang terbatas dalam setiap
pemijahan dapat mengakibatkan
inbreeding
. Dalam kegiatan pemijahan, sejumlah
induk dipasangkan untuk memenuhi nilai pemijahan efektif dengan jumlah
pasangan induk lebih besar dari 50 pasang (
Ne
>50 pasang), sehingga dapat
memperkecil terjadinya silang dalam (
inbreeding
) (Tave, 1995). Khusus pada ikan
nila induk yang digunakan minimal 30 pasang (Tave, 1995).
Nilai yang diperoleh dari analisis keragaman berpasangan (
F
st) dengan
15
Berdasarkan jarak genetik, populasi NirwanaxBEST dengan BESTx
Red
NIFI (0,43) mempunyai hubungan kekerabatan yang lebih dekat dibandingkan
dengan populasi hibrid lainnya. Semakin dekat jarak genetik maka persilangan
bisa menurunkan mutu keturunannya, yaitu meningkatkan homozigositas (Suryani
et al.
, 2001). Semakin tinggi tingkat kemiripan genetik sebaliknya, semakin besar
jarak genetik interpopulasi, maka populasi tersebut memiliki komponen genetik
yang berbeda (Pandin, 2000).
IV.
KESIMPULAN DAN SARAN
4.1
Kesimpulan
Hibridisasi intraspesifik strain ikan nila meningkatkan polimorfisme genetik
27,93% pada populasi hibrid antara jantan Nirwana dengan betina BEST, dan
27,23% dengan
Red
NIFI. Berdasarkan dendrogram
jarak genetik interpopulasi,
menunjukkan bahwa Nirwana dan
Red
NIFI membentuk kelompok bersama
dengan populasi hibrid yang terpisah dengan BEST. Jarak genetik populasi hibrid
terbesar adalah 0,6 antara Nirwana dengan BEST.
4.2
Saran
Kegiatan penelitian selanjutnya untuk mengetahui keragaman genetik dan
status kekerabatan ikan nila dengan menggunakan metode RAPD disarankan
menggunakan lebih dari 3 primer untuk meningkatkan deskripsi keragaman
genetik ikan uji.
KARAKTERISTIK GENOTIPE HIBRIDA HASIL
PERSILANGAN 3 STRAIN IKAN NILA
Oreochromis niloticus
DENGAN METODE
RANDOM AMPLIFIED POLYMORPHIC DNA
(RAPD)
AGUNG LUTHFI FAUZAN
SKRIPSI
DEPARTEMEN BUDIDAYA PERAIRAN
FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR
BOGOR
ABSTRAK
AGUNG LUTHFI FAUZAN
. Karakteristik genotipe hibrida hasil persilangan 3
strain ikan nila
Oreochromis niloticus
dengan metode
Random Amplified
Polymorphic DNA
(RAPD). Dibimbing oleh Dinar Tri Soelistyowati dan Rudhy
Gustiano.
Penelitian ini bertujuan untuk mengetahui keragaman genetik dan kekerabatan
hibrida dari hasil persilangan resiprok tiga strain ikan nila (BEST, Nirwana, dan
Red
NIFI) dengan menggunakan metode
Random Amplified Polymorphic DNA
(RAPD
)
. Hasil penelitian menunjukkan bahwa hibridisasi intraspesifik strain ikan
nila meningkatkan keragaman genetik sebesar 27,93% yaitu pada persilangan
antara jantan Nirwana dengan betina BEST dan 27,23% dengan betina
Red
NIFI.
Berdasarkan analisis kekerabatan diketahui kedekatan Nirwana dan
Red
NIFI
dengan kelompok hibrid, dibandingkan dengan BEST. Jarak genetik terbesar
adalah 0,6 antara Nirwana dengan BEST.
ABSTRACT
AGUNG LUTHFI FAUZAN.
Characteristic of the genotypes hybrids resulted
from reciprocal cross of the three strains Nile tilapine using Random Amplified
Polymorphic DNA. Supervised by Dinar Tri Soelistyowati and Rudhy Gustiano.
Objectives of this study were to observe the genetic variability and relationship
among hybrids of three strains (BEST, Nirwana, and Red NIFI) using Random
Amplified Polymorphic DNA (RAPD). The results showed that hybridization
enabled to increase genetic variability respectedly on progeny between male
Nirwana with female BEST (27.93%) as well as between male Nirwana with
female Red NIFI (27.23%). Based on relationship analysis there were indication
that Nirwana and Red NIFI were closer to the hybrid groups, compared to BEST
one. The greatest genetic distance was the hybrids of Nirwana x BEST (0.6).
KARAKTERISTIK GENOTIPE HIBRIDA HASIL
PERSILANGAN 3 STRAIN IKAN NILA
Oreochromis niloticus
DENGAN METODE
RANDOM AMPLIFIED POLYMORPHIC DNA
(RAPD)
AGUNG LUTHFI FAUZAN
SKRIPSI
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Perikanan pada
Program Studi Teknologi & Manajemen Perikanan Budidaya
Departemen Budidaya Perairan,
Fakultas Perikanan dan Ilmu Kelautan,
Institut Pertanian Bogor
DEPARTEMEN BUDIDAYA PERAIRAN
FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR
BOGOR
Judul Skripsi
: Karakteristik genotipe hibrida hasil persilangan 3 strain
ikan nila
Oreochromis niloticus
dengan metode
Random
Amplified Polymorphic DNA
(RAPD)
Nama Mahasiswa
: Agung Luthfi Fauzan
Nomor Pokok
: C14060923
Disetujui
Pembimbing
I
Pembimbing
II
Dr. Ir. Dinar Tri Soelystiowati, DEA
Dr. Ir. Rudhy Gustiano, M.Sc
NIP 19611016 198403 2 001
NIP 19610803 198903 1 006
Tanggal Pengesahan:
Diketahui
Dekan Fakultas Perikanan dan Ilmu Kelautan
PERNYATAAN MENGENAI SKRIPSI
DAN SUMBER INFORMASI
Dengan ini saya menyampaikan bahwa Skripsi yang berjudul:
KARAKTERISTIK GENOTIPE HIBRIDA HASIL PERSILANGAN 3
STRAIN IKAN NILA Oreochromis niloticus DENGAN METODE RANDOM
AMPLIFIED POLYMORPHIC DNA (RAPD)
adalah benar merupakan hasil karya yang belum diajukan dalam bentuk apapun
kepada perguruan tinggi manapun. Semua sumber data dan informasi yang berasal
atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain
telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka di bagian akhir
skripsi ini.
Bogor, November 2010
DAFTAR RIWAYAT HIDUP
Penulis dilahirkan di Situbondo, Jawa Timur tanggal 15 Juni 1988 dari Ayah
Sunardi dan Ibu Hartati Sutji Rahayu. Penulis merupakan anak pertama dari empat
bersaudara.
Pendidikan formal yang dilalui penulis adalah TK Dharma Wanita, SDN 3
Setail pada tahun 1994-2000 dilanjutkan di SLTP N 1 Genteng, Banyuwangi pada
tahun 2000-2003, kemudian SMU DARUL ULUM 2 BPPT, Jombang pada tahun
2003 lulus tahun 2006. Penulis melanjutkan pendidikan tinggi di IPB melalui jalur
Undangan Seleksi Masuk Institut Pertanian Bogor pada tahun yang sama dan memilih
program studi Teknologi dan Manajemen Perikanan Budidaya, Departemen Budidaya
Perairan, Fakultas Perikanan dan Ilmu Kelautan.
Selama mengikuti perkuliahan, penulis pernah magang di Balai Benih Ikan,
Cianjur dan Praktek Lapangan Akuakultur di Balai Besar Riset Perikanan Budidaya
Air Laut (BBRPBL) Gondol, Bali. Penulis juga pernah menjadi asisten mata kuliah
Biologi Laut semester ganjil 2007/2008 dan semester genap 2009/2010, Dasar-Dasar
Akuakultur semester ganjil 2008/2009, Dasar-Dasar Genetika Ikan semester genap
2009/2010, Manajemen Budidaya Air Tawar semester ganjil 2010/2011 serta Industri
Perbenihan Organisme Akuatik semester ganjil 2010/2011. Selain itu penulis juga
pernah menjadi pengurus Dewan Perwakilan Mahasiswa (DPM) FPIK IPB periode
2007/2008. Penulis juga pernah menjadi anggota
Fisheries Diving Club
(FDC) FPIK
IPB. Tugas akhir dalam pendidikan tinggi diselesaikan dengan menulis skripsi yang
berjudul “
Karakteristik genotipe hibrida hasil persilangan 3 strain ikan nila
Oreochromis niloticus dengan metode Random Amplified Polymorphic DNA
DAFTAR ISI
Halaman
DAFTAR TABEL
...
iv
DAFTAR GAMBAR
... v
DAFTAR LAMPIRAN
... vi
I.
PENDAHULUAN
...
1
II.
BAHAN DAN METODE
...
3
2.1
Ikan
Uji
...
3
2.2
Ekstraksi
DNA
...
3
2.3
Random Amplified Polymorphic DNA
(RAPD) ...
4
2.4
Elektroforesis
...
5
2.5
Analisis
Data
...
6
III.
HASIL DAN PEMBAHASAN
...
7
3.1
Hasil
...
7
3.1.1
Profil
RAPD
...
7
3.1.2 Polimorfisme ... 8
3.1.3
Heterozigositas
...
9
3.1.4 Uji Perbandingan Berpasangan
F
st...
9
iv
DAFTAR TABEL
Halaman
1. Rancangan persilangan resiprok 3 strain ikan nila ...
3
2. Jenis primer yang digunakan dalam RAPD-PCR ...
5
3. Profil DNA hasil persilangan resiprok 3 strain ikan nila ...
8
4. Persentase polimorfisme hasil persilangan resiprok 3 strain ikan nila ....
8
5. Tingkat heterozigositas hasil persilangan resiprok 3 strain ikan nila ...
9
6. Nilai P dari uji perbandingan berpasangan
F
stdari rata-rata 3 primer ...
9
v
DAFTAR GAMBAR
Halaman
vi
DAFTAR LAMPIRAN
Halaman
1.
Persentase polimorfisme ikan nila dengan 3 primer ...
20
2.
Profil DNA 9 populasi ikan nila hasil hibridisasi ...
20
3.
Tingkat heterozigositas ikan nila dengan 3 primer ...
21
4.
Nilai P dari uji perbandingan berpasangan
F
stdari rata-rata 3 primer ....
21
I.
PENDAHULUAN
Perikanan budidaya menjadi target utama pemerintah dalam upaya
meningkatkan hasil perikanan nasional. Indonesia ditargetkan menjadi produsen
hasil perikanan dan kelautan terbesar pada Tahun 2015 oleh Menteri Kelautan dan
Perikanan terutama produksi perikanan budidaya dicanangkan meningkat sebesar
353%. Salah satu komoditas unggulan budidaya yang ditargetkan adalah ikan nila.
Ikan nila (
Oreochromis niloticus
) merupakan jenis ikan introduksi dari negara
Taiwan yang sudah lama dibudidayakan secara luas di Indonesia sejak Tahun
1969 karena mudah penanganannya, tidak ada kendala reproduksi, dapat
mencapai ukuran tubuh relatif besar, toleransi terhadap lingkungan yang luas,
harga relatif murah dan rasa dagingnya enak.
Ikan nila sebagai komoditas budidaya yang tersebar disebagian besar
wilayah di Indonesia menunjukkan kecenderungan penurunan performa fenotipik
yaitu pertumbuhan lambat, tingkat kematian tinggi, dan ukuran individu kecil. Hal
tersebut diduga terkait dengan
potensial fitness
dari sumber genetik populasi yang
tidak optimal atau bahkan menurun karena sistem rekrutmen calon induk dan
persilangannya tidak terpola yang mengakibatkan penghanyutan sejumlah alel
(
genetic drift
) secara terus menerus sehingga terjadi penurunan keragaman
genetik. Penurunan keragaman genetik akibat hilangnya alel-alel potensial pada
populasi ikan dari hasil perbenihan dapat mengakibatkan terhambatnya laju
pertumbuhan dan rendahnya ketahanan ikan terhadap serangan penyakit dan
perubahan lingkungan (Leary
et al.
, 1985
dalam
Moria
et al.
, 2005).
2
Identifikasi genotipe dapat dilakukan dengan beberapa teknik, diantaranya
adalah menggunakan marka
Random Amplified Polymorphic DNA
(RAPD)
dengan teknik PCR. Marka RAPD adalah sekuens DNA polimorfik yang
dipisahkan oleh gel elektroforesis setelah proses PCR menggunakan satu primer
oligonukleotida pendek secara acak. RAPD sangat baik digunakan untuk
mendeteksi polimorfisme gen dalam jumlah besar karena primer oligonukleotida
bisa mendata semua genom yang memiliki situs ikatan dalam reaksi PCR
(Mulyasari, 2007).
II. BAHAN DAN METODE
Ikan Uji
Ikan uji yang digunakan adalah ikan nila hibrida hasil persilangan resiprok 3 strain BEST,
Nirwana dan Red NIFI koleksi Balai Riset Perikanan Budidaya Air Tawar Sempur, Bogor.
[image:33.612.41.543.61.750.2]Rancangan persilangan resiprok selengkapnya disajikan pada Tabel 1.
Tabel 1. Rancangan persilangan resiprok 3 strain ikan nila
Jantan
BEST
Nirwana
Red NIFI
Betina
BEST
BB
NB
RB
Nirwana
BN
NN
RN
Red NIFI
BR
NR
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
(BESTxBEST), RN (Red NIFIxNirwana)
Ekstraksi DNA
Metode yang digunakan dalam ekstraksi dan pemurnian genom DNA berdasarkan prosedur
Fermentas dengan menggunakan Genomic DNA Purification KIT. Tahapan kerja yang dilakukan
meliputi:
Ekstraksi DNA dilakukan dengan membersihkan organ sirip dari larutan fiksatif (alkohol 70%),
dengan cara direndam dan dirotasi ke dalam aquades, hingga sampel berada pada dasar wadah.
Sampel diambil, dipotong kecil-kecil, lalu 20-25 mg sampel dengan 400 (l lysis solution
dimasukkan dalam tabung eppendorf 1,5 ml, dicampur menggunakan vortex sampai homogen
selama 20 detik, kemudian diinkubasi pada suhu 65 °C selama 5 menit. Selanjutnya chloroform
sebanyak 600 (l dimasukkan, lalu dicampur menggunakan vortex selama 20 detik, disentrifuse
pada kecepatan 10.000 rpm selama 2 menit.
Precipitation solution dilakukan dengan mencampurkan 720 (l H2O dengan 80 (l diambil dari
10x konsentrasi solution. Supernatan dipindahkan ke tabung eppendorf baru yang berisi 800 (l
precipitation solution, dicampur menggunakan vortex pada suhu ruang selama 2 menit.
Disentrifuse pada kecepatan 10.000 rpm selama 2 menit hingga DNA mengendap. Supernatan
yang mengandung DNA diambil dan dikeringkan. Setelah kering supernatan ditambahkan 100 (l
NaCl (1,2 M). Etanol absolut dingin sebanyak 300 (l dimasukkan, kemudian endapan DNA
diinkubasi pada suhu -20 °C selama 10 menit. Selanjutnya disentrifuse pada kecepatan 10.000
rpm selama 3-4 menit. Etanol dalam tabung eppendorf dibuang kemudian dibilas dengan etanol
dingin 70%, atau setelah kering dapat langsung ditambahkan 100 (l H2O.
Keberadaan DNA genom dapat diketahui dengan cara elektroforesis yakni menambahkan 3 (l
DNA hasil ekstraksi dengan 1 (l loading dye pada gel agarose 1%. Media elektroforesis atau gel
agarose 1% dibuat dengan agarose dimasukkan ke dalam tabung erlenmeyer sebanyak 0,30 g
dalam 30 ml larutan Tris-Borate-EDTA (TBE 1%). Kemudian batu magnetic stirer dimasukan
untuk mengaduk larutan, tabung ditutup dengan aluminium foil. Tabung erlenmeyer diletakkan
hingga berwarna bening, selanjutnya dituang ke dalam cetakan ukuran dan sisir (comb) dipasang
untuk membentuk sumur (well). Gel yang telah membeku dapat langsung digunakan untuk
elektroforesis atau disimpan dengan direndam dalam larutan TBE 1%.
Setelah DNA dan loading dye dimasukkan dalam sumur gel, selanjutnya bak elektroforesis
dialirkan listrik dengan tegangan 100 V dengan kuat arus yang terprogram secara otomatis.
Proses elektroforesis dihentikan setelah DNA bermigrasi dari kutub negatif ke kutub positif
mencapai tiga per empat bagian panjang gel. Hasil elektroforesis diwarnai dengan merendam gel
dalam larutan ethidium bromide dengan konsentrasi 5 ppm selama 20 menit. Keberadaan DNA
pada gel dapat dilihat dengan menggunakan ultraviolet transilluminator dan didokumentasikan
dengan film polaroid.
Random Amplified Polymorphic DNA (RAPD)
Jenis primer yang digunakan dalam penelitian ini disajikan pada Tabel 2 sebagai berikut.
Tabel 2. Jenis primer yang digunakan dalam RAPD-PCR
No.
Kode Primer
Urutan basa (5’–3’)
Panjang nukleotida
G+C (%)
1.
OPA-02
TGC CGA GCT G
10-mer
2.
OPA-03
AGT CAG CCA C
10-mer
60%
3.
OPC-05
GTC CCG ACG A
10-mer
70%
Amplifikasi DNA dilakukan menggunakan metode RAPD-PCR dengan komposisi bahan: 1 (l
primer, 3 (l DNA, 12,5 (l 2x PCR Master Mix dan 8,5 (l H2O nuklease free; dengan total volume
25 (l dicampur menjadi 1 unit. Seluruh bahan dicampur dengan cara memasukkannya ke dalam
tabung eppendorf 0,5 ml kemudian dihomogenkan menggunakan vorteks selama 20 detik dan
diputar menggunakan spin sampai tidak ada gelembung. Selanjutnya dimasukkan dalam
thermocycler dengan siklus sebanyak 35 cycle, yaitu: satu siklus denaturasi awal pada suhu 94
°C selama 2 menit, 34 siklus selanjutnya terdiri atas denaturasi pada suhu 94 °C selama 1 menit,
annealing 36 °C selama 1 menit dan extention72 °C selama 2 menit. Final extention pada suhu
72 °C selama 7 menit. Hasil RAPD-PCR dilihat melalui elektroforesis atau disimpan di dalam
lemari pendingin dengan suhu 4 °C. Untuk satu sampel ikan, proses PCR ini dilakukan sebanyak
jumlah primer yang digunakan (3 primer). Setiap satu kali proses PCR, jumlah primer yang
digunakan sebanyak 1 primer, hingga ketiga primer selesai digunakan.
Elektroforesis
Setelah didapatkan DNA hasil amplifikasi, selanjutnya dilakukan tahapan elektroforesis dengan
cara menambahkan 2 (l loading dye dengan 7 (l DNA hasil amplifikasi pada gel agarose 1,5%,
kemudian dimasukkan pada sumur elektroforesis. Salah satu sumur diisi dengan marker atau
akuades. Selanjutnya dilakukan tahapan running dan staining, lalu diamati di atas ultraviolet
transilluminator dan didokumentasikan dengan film polaroid.
Analisis Data
Tingkat polimorfisme dan heterozigositas dianalisa dengan menggunakan descriptive statistics
(Miller, 1997) serta analisis statistik menggunakan exact test for population differentiation
(Raymond & Rousset, 1995 dalam Miller, 1997) dengan menggunakan program TFPGA (Tools
for Population Genetic Analyses). Kekerabatan antar populasi dianalisis dengan menggunakan
jarak genetik, berdasarkan program UPGMA Wright (1978) modifikasi Rogers (1972) dalam
Miller (1997) dari software TFPGA. Data yang dihasilkan dari penggunaan program tersebut
III.
HASIL DAN PEMBAHASAN
3.1
Hasil
3.1.1
Profil RAPD
[image:38.612.112.495.260.657.2]Keragaman profil RAPD meliputi jumlah dan ukuran fragmen DNA. Hasil
amplifikasi dengan menggunakan 3 primer (OPA-02, OPA-03 dan OPC-05)
menunjukkan hasil yang bervariasi, data selengkapnya disajikan pada Tabel 3.
Amplifikasi DNA pada 3 populasi
true breeding
(BEST, Nirwana dan
Red
NIFI)
ikan nila uji dapat dilihat pada Gambar 1-3. Sedangkan untuk hasil hibridisasinya
dapat dilihat pada Lampiran 6.
Gambar 1. Amplifikasi OPA-02 pada ikan nila BEST, Nirwana dan
Red
NIFI
Gambar 2. Amplifikasi OPA-03 pada ikan nila BEST, Nirwana dan
Red
NIFI
Gambar 3. Amplifikasi OPC-05 pada ikan nila BEST, Nirwana dan
Red
NIFI
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
8
Tabel 3. Profil DNA hasil persilangan resiprok 3 strain ikan nila
Populasi Jumlah Fragmen Kisaran Ukuran Fragmen (bp)
Nirwana x BEST 1-15 200-3000
BEST X Nirwana 1-18 200-3000
BEST x Red NIFI 2-16 200-2000
BEST x BEST 1-14 200-2200
Nirwana x Red NIFI 5-16 200-1800
Nirwana x Nirwana 6-15 200-3000
Red NIFI x Nirwana 6-14 225-2000
Red NIFI x Red NIFI 6-15 200-2000
Red NIFI x BEST 6-16 225-2200
Jumlah dan ukuran fragmen DNA pada 9 populasi ikan nila hasil hibridisasi
menunjukkan hasil yang bervariasi. Untuk 3 populasi
true breeding
yaitu BEST
memiliki jumlah dan ukuran fragmen DNA berkisar 1-14 dan 200-2200 bp,
Nirwana (6-15, 200-3000 bp), dan
Red
NIFI (6-15, 200-2000 bp). Sedangkan
hasil hibridisasinya yaitu NirwanaxBEST (1-15, 200-3000 bp), BESTxNirwana
(1-18, 200-3000 bp), BESTx
Red
NIFI (2-16, 200-2000 bp), Nirwanax
Red
NIFI
(6-15, 200-1800 bp),
Red
NIFIxNirwana (6-14, 225-2000 bp),
Red
NIFIxBEST
(6-16, 225-2200 bp).
3.1.1
Polimorfisme
Persentase polimorfisme dianalisis menggunakan program TFPGA
(Lampiran 1). Data persentase polimorfisme selengkapnya disajikan pada Tabel 4.
Tabel 4. Persentase polimorfisme hasil persilangan resiprok 3 strain ikan nila
Jantan
BEST Nirwana Red NIFI
Betina
BEST 20,31% 27,93% 15,13%
Nirwana 19,80% 15,41% 10,46%
Red NIFI 26,91% 27,23% 17,04%
[image:39.612.133.383.526.604.2]9
(27,93%) dan terendah pada populasi
Red
NIFIxNirwana (10,46%), sedangkan
pada
true breeding
yaitu populasi NirwanaxNirwana (15,41%).
3.1.2
Heterozigositas
[image:40.612.136.379.296.377.2]Keragaman genetik suatu populasi ikan dapat diketahui melalui nilai
heterozigositas intrapopulasi (Lampiran 3). Nilai heterozigositas bervariasi dari
0,04 hingga 0,12. Pada populasi
true breeding
menunjukkan tingkat
heterozigositas yang lebih rendah dari pada populasi hasil persilangannya yaitu
BESTx
Red
NIFI (0,12) dan Nirwanax
Red
NIFI (0,11). Data selengkapnya
disajikan pada Tabel 5.
Tabel 5. Tingkat heterozigositas hasil persilangan resiprok 3 strain ikan nila
Jantan
BEST Nirwana Red NIFI
Betina
BEST 0,08 0,09 0,06
Nirwana 0,07 0,07 0,04
Red NIFI 0,12 0,11 0,06
3.1.3
Uji Perbandingan Berpasangan F
stSecara statistik dengan menggunakan uji perbandingan berpasangan
F
st(Lampiran 4) menunjukkan terdapat perbedaan genetik secara nyata antara
populasi hasil hibridisasi kecuali populasi NirwanaxBEST, BESTx
Red
NIFI dan
Red
NIFIxBEST (Tabel 6).
Tabel 6. Nilai P dari uji perbandingan berpasangan
F
stdari rata-rata 3 primer
Populasi NB BN BR BB NR NN RN RR RB
NB ***
BN 0,01 ***
BR 0,33 0,00 ***
BB 0,00 0,00 0,00 ***
NR 0,00 0,00 0,00 0,00 ***
NN 0,00 0,00 0,00 0,00 0,00 ***
RN 0,00 0,00 0,00 0,00 0,00 0,02 ***
RR 0,00 0,00 0,00 0,00 0,01 0,00 0,00 ***
RB 0,33 0,00 0,00 0,00 0,00 0,00 0,00 0,00 ***
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
[image:40.612.127.457.511.662.2]10
3.1.4
Jarak Genetik
[image:41.612.133.458.275.425.2]Jarak genetik interpopulasi menunjukkan perbedaan dan dapat
menggambarkan status kekerabatan antar populasi ikan nila. Jarak genetik
interpopulasi ketiga strain dan hasil hibridisasi intraspesifik 3 populasi ikan
disajikan pada Tabel 7. Jarak genetik tertinggi adalah antara populasi
BESTxBEST dengan NirwanaxNirwana sebesar 0,60, sedangkan jarak genetik
terendah adalah 0,43 yaitu antara populasi NirwanaxBEST dengan BESTx
Red
NIFI. Secara umum, jarak genetik 9 populasi ikan nila uji memiliki kisaran yang
tinggi yaitu antara 0,43-0,60.
Tabel 7. Jarak genetik antara 9 populasi ikan nila dari rata-rata 3 primer
Populasi NB BN BR BB NR NN RN RR RB
NB ***
BN 0,47 ***
BR 0,43 0,48 ***
BB 0,55 0,56 0,50 ***
NR 0,56 0,53 0,50 0,58 ***
NN 0,52 0,51 0,48 0,60 0,49 ***
RN 0,59 0,57 0,48 0,46 0,48 0,54 ***
RR 0,55 0,54 0,50 0,58 0,47 0,49 0,53 ***
RB 0,44 0,57 0,53 0,58 0,49 0,52 0,49 0,56 ***
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
(BESTxBEST), RN (Red NIFIxNirwana)
11
0.60 0.45 0.30 0.15 0.00
NB
BR
BN
RB
NR
RR
NN
BB
RN
Keterangan: NB (NirwanaxBEST), BR (BESTxRed NIFI), BN (BESTxNirwana), RB (Red
NIFIxBEST), NR (NirwanaxRed NIFI), RR (Red NIFIxRed NIFI), NN (NirwanaxNirwana), BB
[image:42.612.155.492.86.277.2](BESTxBEST), RN (Red NIFIxNirwana)
Gambar 4. Dendrogram 9 populasi ikan nila hasil hibridisasi
12
3.2
Pembahasan
Profil RAPD pada populasi
true breeding
(BEST, Nirwana dan
Red
NIFI)
memiliki karakter yang berbeda. Panjang dan ukuran fragmen DNA pada populasi
BESTxBEST lebih tinggi yaitu 1-14 dan 200-2200 bp dari pada pupulasi
NirwanaxNirwana (6-15, 200-3000 bp), dan
Red
NIFI (6-15, 200-2000 bp).
Sedangkan untuk hasil hibridisasinya, pada beberapa populasi menunjukkan
panjang dan ukuran fragmen DNA yang lebih tinggi, yaitu populasi
NirwanaxBEST (1-15, 200-3000 bp), BESTxNirwana (1-18, 200-3000 bp),
BESTx
Red
NIFI (2-16, 200-2000 bp). Hasil tersebut menunjukkan bahwa
hibridisasi dapat meningkatkan keragaman genetik yang dilihat dari panjang dan
ukuran fragmen DNA pada masing-masing populasi ikan uji.
Tingkat polimorfisme antara ketiga populasi
true breeding
yang tertinggi
adalah pada populasi BESTxBEST sebesar 20,31% lebih tinggi dibandingkan
populasi NirwanaxNirwana (15,41% ) dan
Red
NIFIx
Red
NIFI (17,04%). Sedang
pada hasil hibridisasinya menunjukkan polimorfisme yang lebih tinggi dari
true
breedingnya
yaitu pada populasi NirwanaxBEST sebesar 27,93%, populasi
Nirwanax
Red
NIFI (27,23%) dan populasi BESTx
Red
NIFI (26,91%). Hal
tersebut menunjukkan bahwa proses hibridisasi dapat meningkatkan keragaman
genetik ikan nila. Sedangkan pada populasi hibrid
Red
NIFIxBEST,
Red
NIFIxNirwana, dan BESTxNirwana menunjukkan persentase polimorfisme yang
lebih rendah yaitu 15,13%, 10,46% dan 19,80%. Penyebab rendahnya persentase
polimorfisme pada jenis ikan budidaya antara lain karena aktivitas seleksi induk,
terjadinya
inbreeding
,
genetic drift
dan fenomena
bottleneck effect
yang
merupakan akibat dari tidak terpenuhinya
gene pool
yang tidak lengkap
konfigurasinya pada populasi budidaya tersebut. Hal ini banyak ditemui pada
sistem budidaya ikan/udang di Indonesia yang disebabkan keterbatasan jumlah
induk yang digunakan dalam memproduksi benih sehingga tidak memenuhi
standar
Ne
(
effective breeding number
) (Kirpichnikov, 1981; Kapunsckinski dan
Jacobson, 1987; Tave, 1993)
13
perubahan genetik pada populasi ikan melalui persilangan hibridisasi. Strain
BEST, Nirwana dan
Red
NIFI memiliki proses seleksi yang berbeda. Ikan nila
BEST merupakan ikan hasil pemuliaan untuk karakter keunggulan pertumbuhan
yang dihasilkan melalui proses selama 4 (empat) tahun penelitian oleh Tim
Peneliti dari Balai Riset Perikanan Budidaya Air Tawar Bogor (Arifin, 2008).
Beberapa keunggulan strain ikan nila BEST diantaranya s
urvival rate
(SR)
sebesar 90% dibandingkan nila lokal (60-70%). Laju pertumbuhan 4,85%, lebih
tinggi dibandingkan 2 nila unggulan lainnya, yaitu: Gesit (4,5%) dan Nirwana
(3,93%),
food convertion ratio
(FCR) sebesar 1,1 (Anmi, 2009). Ikan nila
Nirwana merupakan hasil seleksi famili dengan sumber genetik berasal dari ikan
nila GIFT (
Genetic Improvement Farm Tilapia
) dan nila GET (
Genetically
Enchanced Tilapia
) dari Philipina. Menurut Sumantadinata (2006), bahwa
pertumbuhan bobot ikan nila Nirwana meningkat sekitar 45% pada generasi ke-3
(F3) dibandingkan dengan populasi awalnya (Anonim, 2007). Nila
Red
NIFI
dikenal juga sebagai nila merah atau nirah. Ikan ini kemungkinan merupakan hasil
persilangan antara
Tilapia mossambicus
(mujair) atau
Oreochromis niloticus
(nila) dengan
Oreochromis honorum
,
Oreochromis aureus
, atau
Oreochromis zilii
(Amri, 2008).
Tingkat heterozigositas populasi ikan nila hasil hibridisasi berkisar antara
0,04-0,12. Kisaran tersebut sebanding dengan tingkat heterozigositas pada
populasi ikan air tawar di alam, misalnya populasi ikan batak (
Tor sorro
) berkisar
0,08–0,09 (Asih
et al.
, 2006). Pada populasi BESTx
Red
NIFI dan Nirwanax
Red
NIFI sedikit lebih tinggi dibandingkan hasil persilangan lainnya; yaitu sebesar
0,12 dan 0,11. Hasil tersebut menunjukkan bahwa proses hibridisasi dapat
meningkatkan keragaman genetik dan nilai heterozigositas. Pada dasarnya,
hibridisasi adalah memanfaatkan sifat heterosis karena sifat dominan dan
heterozigositas pada banyak lokus (Tave, 1993) atau interaksi dari alel pada lokus
(Kapunsckinski dan Jacobson, 1987).
14
hal ini program seleksi untuk suatu sifat yang dilakukan cukup lama dapat
menurunkan potensi sifat genetik atau dapat menurunkan keragaman genetiknya.
Seleksi merupakan suatu teknik untuk memperbaiki sifat yang terukur
(
quantitative trait
). Prinsip dasar seleksi adalah mengeksploitasi sifat
addictive
dari alel-alel pada semua lokus yang mengontrol sifat terukur untuk memperbaiki
suatu populasi (Kirpichnikov, 1981; Falconer, 1981; Tave, 1993; Gjedrem, 2005).
Rendahnya keragaman genetik diduga juga dapat disebabkan jumlah induk
atau cara yang digunakan dalam pemijahan terbatas (rekrutmen induk tidak
terkontrol) atau penggunaan induk dalam waktu lama. Hal tersebut didukung oleh
pernyataan Secara umum penggunaan jumlah induk yang terbatas dalam setiap
pemijahan dapat mengakibatkan
inbreeding
. Dalam kegiatan pemijahan, sejumlah
induk dipasangkan untuk memenuhi nilai pemijahan efektif dengan jumlah
pasangan induk lebih besar dari 50 pasang (
Ne
>50 pasang), sehingga dapat
memperkecil terjadinya silang dalam (
inbreeding
) (Tave, 1995). Khusus pada ikan
nila induk yang digunakan minimal 30 pasang (Tave, 1995).
Nilai yang diperoleh dari analisis keragaman berpasangan (
F
st) dengan
15
Berdasarkan jarak genetik, populasi NirwanaxBEST dengan BESTx
Red
NIFI (0,43) mempunyai hubungan kekerabatan yang lebih dekat dibandingkan
dengan populasi hibrid lainnya. Semakin dekat jarak genetik maka persilangan
bisa menurunkan mutu keturunannya, yaitu meningkatkan homozigositas (Suryani
et al.
, 2001). Semakin tinggi tingkat kemiripan genetik sebaliknya, semakin besar
jarak genetik interpopulasi, maka populasi tersebut memiliki komponen genetik
yang berbeda (Pandin, 2000).
IV.
KESIMPULAN DAN SARAN
4.1
Kesimpulan
Hibridisasi intraspesifik strain ikan nila meningkatkan polimorfisme genetik
27,93% pada populasi hibrid antara jantan Nirwana dengan betina BEST, dan
27,23% dengan
Red
NIFI. Berdasarkan dendrogram
jarak genetik interpopulasi,
menunjukkan bahwa Nirwana dan
Red
NIFI membentuk kelompok bersama
dengan populasi hibrid yang terpisah dengan BEST. Jarak genetik populasi hibrid
terbesar adalah 0,6 antara Nirwana dengan BEST.
4.2
Saran
Kegiatan penelitian selanjutnya untuk mengetahui keragaman genetik dan
status kekerabatan ikan nila dengan menggunakan metode RAPD disarankan
menggunakan lebih dari 3 primer untuk meningkatkan deskripsi keragaman
genetik ikan uji.
DAFTAR PUSTAKA
Abdallah, H. H., M. Elnaldy, A. Obeida, and H. Itriby. 2004. Genetic Diversity of
Nile Tilapia Populations Revealed by Randomly Amplyfied Polymorphic
DNA (RAPD).
Aquaculture Research
35:587-593.
Amri, K., dan Khairuman. 2008. Syarat Hidup Ikan Nila. Jakarta. Agromedia
Pustaka. www.hobiikan.blogspot.com [2 Januari 2010]
Anonim, 2007. Ahli IPB Temukan Varietas Baru Ikan Nirwana.
www.Kapanlagi.com [8 Agustus 2010]
Anmi, L. T. 2009. Nila BEST Sumbang 2 Kali Lipat. www.trubus-online.co.id
[8 Agustus 2010]
Arifin, O. Z. 2005. Polimorfisme mt-DNA Keturunan Pertama (F1) dalam Seleksi
Famili Ikan Nila (
Oreochromis niloticus
) di BPBI Wanayasa Jawa Barat.
Tesis
. Program Pasca Sarjana IPB. Bogor.
_________. 2008. NILA BEST (
Bogor Enhanched Strain Tilapia
).
www.blogspot.com [8 Agustus 2010]
Asih, S., E. Nugroho, dan Mulyasari. 2006. Penentuan Variasi Genetik Ikan batak
(
Tor sorro
) dari Sumatera Utara dengan Metode Analisis Random
Amplified Polymorphism DNA (RAPD).
Kumpulan Hasil Riset Tahun
2006
. Balai Riset Perikanan Budidaya Air Tawar. 1-9.
Dunham, R. E. 1995. The Distibution of Genetically Improved Aquatic Organims
to Global Food Security. Intl. Conf. On
Sustainable Contribition of
Fisheries to Food Securityy
. KC/FI/Tech. FAO. Rome. 111 pp.
Falconer, D. S. 1981. Introduction to Quantitative Genetics, 2nd edition.
Longman, New York.
Gjedrem, T. 2005. Selection and Breeding Programs in Aquaculture.
AKVAFORSk, Institut of Aquaculture Research As. Springer Dordrecht,
Netherland. 364 pp.
Kapunsckinski, A.R. and L.D. Jacobson. 1987. Genetic guidelines for fisheries
management. University of Minnesota, USA. 66 pp.
Kirpichnikov, V. S. 1981. Genetic Bases of Fish Selection. Spinger-Verlag, New
York.
18
Moria, S.B., Haryanti, I.G.N. Permana, dan B. Susanto. 2005. Karakteristik
Genetik Induk Rajungan
Portunus pelagicus
dari Beberapa Perairan Melalui
Analisis RFLP Mt-DNA. Jurnal Penelitian Perikanan Indonesia, 11(5):
57-62.
Mulyasari. 2007. Beberapa Teknik Penentuan Variasi Genetik Pada Ikan Untuk
Proses Pemuliaan.
Media Aquaculture, Vol 2
: 37-40.
Pandin, D. S., 2000. Kemiripan Genetik Populasi Kelapa Dalam Mapanget Tenga,
Bali, Palu dan Sawarna Berdasarkan Penanda RAPD.
Tesis
. Program
Pascasarjana. Institut Pertanian Bogor. Bogor.
Suryani, S. A. M. P., Sukoso, dan Sugama. 2001. Hubungan Kekerabatan Tiga
Spesies Ikan Kerapu Sunu (
Plectropomus
spp.) Atas Dasar Variasi Genetik.
Biosain
. 1 (3): 99-107.
Tave, D. 1993. Genetics for Fish Managers. The AVI Publ. Comp. Inc. NY, USA.
418 pp.
_______. 1995. Selective Breeding Programmes for Medium-sized Fish Farms.
FAO Fisheries Technical Paper
. No. 352. Rome, FAO.
20
Lampiran 1. Persentase polimorfisme ikan nila dengan 3 primer
Populasi Keterangan Primer Rata-rata
OPA-02 OPA-03 OPC-05
Nirwana x BEST Polimorfisme 22.58% 52.38% 8.82% 27.93%
Jumlah sampel 10 10 10
BEST x Nirwana Polimorfisme 16.13% 28.57% 14.71% 19.80%
Jumlah sampel 10 10 10
BEST x Red NIFI Polimorfisme 32.26% 19.05% 29.41% 26.91%
Jumlah sampel 10 10 10
BEST x BEST Polimorfisme 12.90% 33.33% 14.70% 20.31%
Jumlah sampel 10 10 10
Nirwana x Red NIFI Polimorfisme 35.48% 28.57% 17.64% 27.23%
Jumlah sampel 10 10 10
Nirwana x Nirwana Polimorfisme 16.13% 9.52% 20.58% 15.41%
Jumlah sampel 10 10 10
Red NIFI x Nirwana Polimorfisme 6.45% 19.05% 5.88% 10.46%
Jumlah sampel 10 10 10
Red NIFI x Red NIFI Polimorfisme 9.68% 23.81% 17.64% 17.04%
Jumlah sampel 10 10 10
Red NIFI x BEST Polimorfisme 19.35% 14.28% 11.76% 15.13%
Jumlah sampel 10 10 10
Jumlah sampel 90 90 90
Lampiran 2. Profil DNA 9 populasi ikan nila hasil hibridisasi
Populasi Jumlah
Fragmen
Kisaran Ukuran
Fragmen (bp)
Nirwana x BEST 1-15 200-3000
BEST X Nirwana 1-18 200-3000
BEST x Red NIFI 2-16 200-2000
BEST x BEST 1-14 200-2200
Nirwana x Red NIFI 5-16 200-1800
Nirwana x Nirwana 6-15 200-3000
Red NIFI x Nirwana 6-14 225-2000
Red NIFI x Red NIFI 6-15 200-2000
21
Lampiran 3. Tingkat heterozigositas ikan nila dengan 3 primer
Populasi Keterangan Primer Rata-rata
OPA-02 OPA-03 OPC-05
Nirwana x BEST Heterozigositas 0.1085 0.1099 0.0416 0.0867
Jumlah sampel 10 10 10
BEST x Nirwana Heterozigositas 0.0766 0.0737 0.0677 0.0727
Jumlah sampel 10 10 10
BEST x Red NIFI Heterozigositas 0.1494 0.0808 0.1181 0.1161
Jumlah sampel 10 10 10
BEST x BEST Heterozigositas 0.0422 0.1233 0.0713 0.0789
Jumlah sampel 10 10 10
Nirwana x Red NIFI Heterozigositas 0.1577 0.0967 0.0771 0.1105
Jumlah sampel 10 10 10
Nirwana x Nirwana Heterozigositas 0.0788 0.0465 0.0701 0.0651
Jumlah sampel 10 10 10
Red NIFI x Nirwana Heterozigositas 0.0287 0.0520 0.0287 0.0365
Jumlah sampel 10 10 10
Red NIFI x Red NIFI Heterozigositas 0.0386 0.0787 0.0735 0.0636
Jumlah sampel 10 10 10
Red NIFI x BEST Heterozigositas 0.0732 0.0640 0.0559 0.0644
Jumlah sampel 10 10 10
Jumlah sampel 90 90 90
Lampiran 4. Nilai P dari uji perbandingan berpasangan
F
stdari rata-rata 3 primer
Populasi 1 2 3 4 5 6 7 8 9
1 *****
2 0.0068 *****
3 0.3299 0.0000 *****
4 0.0002 0.0015 0.0000 *****
5 0.0000 0.0000 0.0000 0.0000 *****
6 0.0000 0.0010 0.0000 0.0000 0.0009 *****
7 0.0000 0.0000 0.0000 0.0000 0.0000 0.0230 *****
8 0.0000 0.0000 0.0000 0.0000 0.0063 0.0027 0.0000 *****
22
Lampiran 5. Jarak genetik 9 populasi ikan nila
Populasi 1 2 3 4 5 6 7 8 9
1 *****
2 0.4716 *****
3 0.4324 0.4827 *****
4 0.5501 0.5569 0.4969 *****
5 0.5567 0.5290 0.5044 0.5834 *****
6 0.5209 0.5067 0.4774 0.6020 0.4855 *****
7 0.5907 0.5651 0.4807 0.4630 0.4845 0.5382 *****
8 0.5545 0.5391 0.5033 0.5766 0.4703 0.4931 0.5330 *****
9 0.4406 0.5657 0.5289 0.5779 0.4864 0.5167 0.4866 0.5633 *****
Lampiran 6. Amplifikasi DNA pada hibrida hasil persilangan 3 strain ikan nila
Amplifikasi OPA-02 pada nila Red NIFI x Nirwana dan Nirwana x Red NIFI
Amplifikasi OPA-02 pada nila Nirwana x BEST dan BEST x Nirwana
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
23
Amplifikasi OPA-02 pada nila BEST x Red NIFI dan Red NIFI x BEST
Amplifikasi OPA-03 pada nila Red NIFI x Nirwana dan Nirwana x Red NIFI
Amplifikasi OPA-03 pada nila Nirwana x BEST dan BEST x Nirwana
Amplifikasi OPA-03 pada nila BEST x Red NIFI dan Red NIFI x BEST
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
24
Amplifikasi OPC-05 pada nila Red NIFI x Nirwana dan Nirwana x Red NIFI
Amplifikasi OPC-05pada nila Nirwana x BEST dan BEST x Nirwana
Amplifikasi OPC-05 pada nila BEST x Red NIFI dan Red NIFI x BEST
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
3000 bp
1000 bp
500 bp
200 bp
25
Lampiran 7.
Descriptive Statistics
OPA-02
7/23/2010 9:03:27 AM
Analysis of C:\TFPGA\NILA-A2
Data set contains genotypes of individuals sampled from populations.
Organism Type: Diploid Marker Type: Dominant H-W Equilibrium Assumed.
Allele frequencies estimated based on the square root
of the frequency of the null (recessive) genotype.
************************************ ********************
RESULTS FOR EACH POPULATION.
POPULATION 1
Locus 1 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 2 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 3 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 4 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 5 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 6 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 7 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 8 # obs. at locus= 10
Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 9 # obs. at locus= 10 Heterozygosity: 0.4944
Heterozygosity (unbiased): 0.5204 Heterozygosity (direct count): 0.4944
Locus 10 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 11 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 12 # obs. at locus= 10 Heterozygosity: 0.4944
Heterozygosity (unbiased): 0.5204 Heterozygosity (direct count): 0.4944
Locus 13 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 14 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 15 # obs. at locus= 10 Heterozygosity: 0.4649
Heterozygosity (unbiased): 0.4894 Heterozygosity (direct count): 0.4649
Locus 16 # obs. at locus= 10 Heterozygosity: 0.3492
Heterozygosity (unbiased): 0.3676 Heterozygosity (direct count): 0.3492
Locus 17 # obs. at locus= 10 Heterozygosity: 0.4649
Heterozygosity (unbiased): 0.4894 Heterozygosity (direct count): 0.4649
Locus 18 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 19 # obs. at locus= 10 Heterozygosity: 0.0000
26
Locus 20 # obs. at locus= 10 Heterozygosity: 0.4325
Heterozygosity (unbiased): 0.4552 Heterozygosity (direct count): 0.4325
Locus 21 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 22 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 23 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 24 # obs. at locus= 10 Heterozygosity: 0.4944
Heterozygosity (unbiased): 0.5204 Heterozygosity (direct count): 0.4944
Locus 25 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 26 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 27 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 28 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 29 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 30 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 31 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000
Heterozygosity (direct count): 0.0000
---
Results over all loci
Ave. sample size: 10.0000 Ave. heterozygosity: 0.1031
Ave. heterozygosity (unbiased): 0.1085 Ave. heterozygosity (direct count): 0.1031 % polymorphic loci (no criterion): 22.5806 % polymorphic loci (99% criterion): 22.5806
% polymorphic loci (95% criterion): 22.5806
---
POPULATION 2
Locus 1 # obs. at locus= 10 Heterozygosity: 0.4325
Heterozygosity (unbiased): 0.4552 Heterozygosity (direct count): 0.4325
Locus 2 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.0000 Heterozygosity (direct count): 0.0000
Locus 3 # obs. at locus= 10 Heterozygosity: 0.0000
Heterozygosity (unbiased): 0.00