KLASIFIKASI ENZIM PROTEIN MENGGUNAKAN METODE
K-NEAREST NEIGHBOR DAN ANALISIS KOMPONEN
UTAMA SEBAGAI PEREDUKSI CIRI
JEFRI HANRIKO SAPUTRA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi Enzim
Protein Menggunakan Metode
K-Nearest Neighbor
dan Analisis Komponen
utama Sebagai Pereduksi Ciri adalah benar karya saya dengan arahan dari komisi
pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi
mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan
maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan
dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut
Pertanian Bogor.
Bogor, September 2015
Jefri Hanriko Saputra
ABSTRAK
JEFRI HANRIKO SAPUTRA. Klasifikasi Enzim Protein Menggunakan Metode
K-Nearest Neighbor
dan Analisis Komponen utama. Dibimbing oleh TOTO
HARYANTO.
Enzim adalah suatu protein yang berfungsi sebagai biokatalisator dan
mempunyai bentuk globular. Enzim merupakan biokatalisator yang aktif, sebab
hanya dengan jumlah yang sedikit pada kondisi yang tepat, dapat mengatur
jalannya reaksi kimia tertentu. Tujuan penelitian ini adalah untuk melakukan
klasifikasi enzim berdasarkan 6 kelas yang ditentukan oleh
Enzyme Commission
.
Data fasta sekuen protein enzim akan dilakukan ekstraksi fitur. Fitur yang
digunakan adalah 470 fitur yang digunakan pada penelitian Rao
et al
(2009). Data
yang digunakan adalah data fasta yang berjumlah 3000 data, masing-masing 500
data untuk 6 kelas. Metode yang digunakan dalam penelitian ini adalah
K-Nearest
Neighbor
sebagai metode klasifikasi pada enzim dan
Principal Component
Analysis
digunakan untuk mereduksi dimensi fitur. Penelitian ini menunjukkan
sensitivitas rata-rata tertinggi sebesar 0.79 pada K-NN dengan PCA 85% dan PCA
90%.
Kata kunci: enzim, K-NN, PCA, protein
ABSTRACT
JEFRI HANRIKO SAPUTRA. Protein Enzyme Classification Using K-Nearest
Neighbor Method and Principal Component Analysis As Dimension Reductant.
Supervised by TOTO HARYANTO.
Enzyme is a protein that, serves as biocatalyst and has a globular shape.
Enzymes are active biocatalyst, because with only small amounts in the right
conditions, can set the course of a particular chemical reaction. The purpose of
this research is to classify the enzyme based on 6 classes determined by Enzyme
Commission. The feature of enzyme protein sequences extracted from the fasta
data. The features used are 470 features used in from previous research. The
number of enzyme data used is 3000 fasta data. There are 6 classes with 500 data
for each class. The method used is K-Nearest Neighbor as classification method
on enzyme and Principal Component Analysis is used to reduce feature
dimension. This research shows the highest average sensitivity of 0.79 in the
K-NN with PCA85% and PCA90%.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI ENZIM PROTEIN MENGGUNAKAN METODE
K-NEAREST NEIGHBOR DAN ANALISIS KOMPONEN
UTAMA SEBAGAI PEREDUKSI CIRI
JEFRI HANRIKO SAPUTRA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Penguji: Dr Eng Wisnu Ananta Kusuma, ST MT
Muhammad Abrar Istiadi, SKom MKom
Judul Skripsi : Klasifikasi Enzim Protein Menggunakan Metode K-Nearest
Neighbor dan Analisis Komponen Utama Sebagai Pereduksi Ciri
Nama
: Jefri Hanriko Saputra
NIM
: G64114001
Disetujui oleh
Toto Haryanto, SKom MSi
Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi MKom
Ketua Departemen
PRAKATA
Alhamdulillahi R
abbil ‘alamin
, puji dan syukur penulis panjatkan kepada
Allah
Subhanahu wa t
a’ala
atas segala karunia-Nya sehingga karya ilmiah ini
berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak
bulan Desember 2013 ini ialah klasifikasi enzim protein, dengan judul :
Klasifikasi Enzim Protein Menggunakan Metode K-Nearest Neighbor dan
Analisis Komponen Utama Sebagai Pereduksi Ciri.
Terima kasih penulis ucapkan kepada seluruh pihak yang telah berperan
dalam penelitian ini, yaitu:
1
Ayahanda Jhondri Arizon, ibunda Hanifa, dan keluarga atas doa, semangat, dan
dorongan kepada penulis sehingga dapat menyelesaikan penelitian ini.
2
Bapak Toto Haryanto, SKom MSi selaku pembimbing, yang telah memberikan
arahan, ide, masukan, dan dukungan kepada penulis.
3
Bapak Dr Eng Wisnu Ananta Kusuma, ST MT dan Bapak Muhammad Abrar
Istiadi, SKom MKom yang telah bersedia menjadi penguji, dan memberikan
saran yang berharga sehingga tulisan ini menjadi lebih baik dari sebelumnya.
4
Seluruh staf pengajar Ilmu Komputer IPB yang telah memberikan ilmu semasa
perkuliahan.
5
Rekan-rekan Ilmu Komputer IPB yang saling menyemangati selama
pengerjaan penelitian di tahun yang sama.
6
Seluruh rekan satu bimbingan yang tidak dapat disebutkan satu persatu dan
pihak-pihak lainnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2015
DAFTAR ISI
DAFTAR TABEL
vi
DAFTAR GAMBAR
vi
DAFTAR LAMPIRAN
vi
PENDAHULUAN
11
Latar Belakang
11
Perumusan Masalah
2
Tujuan Penelitian
2
Manfaat Penelitian
2
Ruang Lingkup Penelitian
2
METODE
3
Pengumpulan Data
3
Ekstraksi Ciri
3
Normalisasi Data
5
Principal Components Analysis (PCA)
5
K-Fold Cross Validation
6
K-Nearest Neighbor
6
Evaluasi
6
Ruang Lingkup Pengembangan
7
HASIL DAN PEMBAHASAN
7
Pengumpulan data
7
Ekstraksi Ciri
7
Normalisasi
11
Principal Components Analysis (PCA)
11
K-Fold Cross Validation
11
Hasil Klasifikasi KNN
12
Analisis Hasil
12
SIMPULAN DAN SARAN
16
Simpulan
16
Saran
16
DAFTAR PUSTAKA
16
LAMPIRAN
18
DAFTAR TABEL
1
Daftar persebaran fitur
4
2
Matriks konfusi
6
3
Asam amino penyusun protein
8
4
Distribusi asam amino dua gram
8
5
Distribusi grup pertukaran dua gram
9
6
Komposisi atomik asam amino
10
7
Hasil reduksi PCA
11
8
Hasil sensitivitas rata-rata
12
9
Matriks konfusi kelas
hydrolase
KNN PCA85
k
=3 k-fold 1
12
10
Matriks konfusi kelas
isomerase
KNN PCA85
k
=3 k-fold 1
13
11
Matriks konfusi kelas
ligase
KNN PCA85
k
=3 k-fold 1
13
12
Matriks konfusi kelas
lyase
KNN PCA85
k
=3 k-fold 1
13
13
Matriks konfusi kelas
oxydoreductase
KNN PCA85
k
=3 k-fold 1
13
14
Matriks konfusi kelas
transferase
KNN PCA85
k
=3 k-fold 1
14
15
Nilai akurasi, sensitivitas dan spesifisitas KNN PCA85
k
=3 k-Fold 1
14
16
Matriks konfusi kelas
hydrolase
KNN PCA90
k
=3 k-fold 2
14
17
Matriks konfusi kelas
isomerase
KNN PCA90
k
=3 k-fold 2
14
18
Matriks konfusi kelas
ligase
KNN PCA90
k
=3 k-fold 2
14
19
Matriks konfusi kelas
lyase
KNN PCA90
k
=3 k-fold 2
15
20
Matriks konfusi kelas
oxydoreductase
KNN PCA90
k
=3 k-fold 2
15
21
Matriks konfusi kelas
transferase
KNN PCA90
k
=3 k-fold 2
15
22
Nilai akurasi, sensitivitas dan spesifisitas KNN PCA90
k
=3 k-Fold 2
15
DAFTAR GAMBAR
1
Tahapan penelitian
3
2
Isoelectric point 3WKL
9
3
Berat molekul 3WKL
11
DAFTAR LAMPIRAN
1
Data kelas
hydrolase
17
2
Data kelas
isomerase
20
3
Data kelas
ligase
23
4
Data kelas
lyase
26
5
Data kelas
oxydoreductase
29
PENDAHULUAN
Latar Belakang
Protein terdapat di dalam semua sistem kehidupan dan merupakan suatu
komponen seluler utama yang menyusun setengah dari berat kering sel. Setiap sel
mengandung mengandung ratusan protein yang berbeda-beda dan tiap jenis sel
mengandung beberapa protein yang khas bagi sel tersebut. Sebagian besar protein
disimpan di dalam jaringan otot dan beberapa organ tubuh lainnya, sedangkan
sisanya terdapat di dalam darah. Istilah protein yang dikemukakan pertama kali
oleh pakar kimia Belanda, G.J.Mulder pada tahun 1939, berasal dari bahasa
Yunani
’proteios’
.
Proteios
mempu
nyai arti “yang pertama” atau “yang paling
utama”. Protein memiliki peranan penting pada organisme yaitu dalam struktur,
fungsi dan reproduksi.
Enzim adalah suatu kelompok protein yang menjalankan dan mengatur
perubahan-perubahan kimia dalam sistem biologi. Zat ini dihasilkan oleh
organ-organ pada makhluk hidup, yang secara katalitik menjalankan berbagai reaksi,
seperti pemecahan hidrolisis, oksidasi, reduksi, isomerasi, adisi, transfer radikal
dan terkadang pemutusan rantai karbon. Kebanyakan enzim yang terdapat di
dalam alat-alat atau organ-organ organisme hidup berupa larutan koloidal dalam
cairan tubuh, seperti air ludah, darah, cairan lambung dan cairan pankreas. Enzim
juga terdapat di bagian dalam sel. Hal ini terikat erat dengan protoplasma. Enzim
juga ada di dalam mitikondria dan ribosom.
Oleh
International Commission on Enzymes
, enzim secara sistematis
diklasifikasikan menjadi enam kelompok besar, menurut reaksi yang dikatalisi.
Enam kelompok besar tersebut adalah
Hydrolase
,
Isomerase
,
Ligase
,
Lyase
,
Oxydoreductase
dan
Transferase
.
Hydrolase
bertugas dalam pemisahan ikatan
C-O, C-N atau C-S dengan penambahan H
2O pada ikatan.
Isomerase
bertugas dalam
pemindahan gugus di dalam molekul induk untuk menghasilkan bentuk isomatik.
Ligase
pembentukan ikatan C-C, C-S, C-O dan C-N disertai penguraian ikatan
berenergi tinggi seperti ATP.
Lyase
penambahan gugus ke ikatan rangkap atau
pembentukan ikatan rangkap.
Oxydoreductase
bertugas dalam pemindahan
elektron dari satu senyawa ke suatu akseptor dan
Transferase
bertugas
dalampemindahan sebuah gugus fungsional, misalnya gugus amino, metil atau
fosfat (Mark
et al.
1996).
Rao
et al
. (2009) melakukan penelitian klasifikasi superfamily pada protein
menggunakan 479 buah fitur yang didapat dari mengekstrak sekuen fasta protein
tersebut. Penelitian Rao
et al.
menggunakan 490 protein yang termasuk dalam
tiga kelas yaitu 195
esterase
, 155
lipase
, dan 140
cytochrome
. Metode yang
digunakan adalah metode
Probabilistic Neural Network
dengan hasil akurasi
98.2%, spesifisitas 98,4%, sensitivitas 98,7% pada kelas
esterase
. Akurasi 98.7%,
spesifisitas 99,3%, sensitivitas 96,1% pada kelas
lipase
. akurasi 96.7%,
spesifisitas 97,2%, sensitivitas 93,2% pada kelas
cytochrome
.
2
menggunakan algoritme
k-nearest neighbor
. Akurasi pada organisme dikenal dari
fold
terbaik dengan menggunakan PCA 95% untuk panjang fragmen 0.5 Kbp
sampai10 Kbp berkisar antara 91.6% sampai 99,9%.
Penelitian ini melakukan klasifikasi enzim dengan menggunakan 470 fitur
dari 479 fitur yang berasal dari penelitian Rao
et al.
(2009) dan menggunakan
metode penelitian yang digunakan oleh Simangunsong yaitu
k-nearest neighbor
sebagai metode klasifikasi dan
principal component analysis
untuk mereduksi
fitur.
Perumusan Masalah
Berbekalkan Permasalahan yang akan menjadi bahan analisis dalam
penelitian ini adalah:
1
Berapa nilai akurasi klasifikasi enzim dengan menggunakan 470 fitur
penelitian Rao
et al.
(2009)?
2
Berapa akurasi yang diperoleh jika menggunakan metode KNN dengan PCA
dan tanpa seleksi fitur?
3
Bagaimana pengaruh nilai
k
pada metode KNN
yang digunakan terhadap hasil
klasifikasi?
Tujuan Penelitian
Tujuan dari penelitian ini adalah:
1
Mengklasifikasikan enzim dengan hanya menggunakan sekuen enzim.
2
Melakukan menerapkan klasifikasi
K-nearest neighbor
dan
principal
component analysis
dalam mengklasifikasikan enzim protein.
Manfaat Penelitian
Penelitian ini diharapkan dapat memberikan informasi mengenai akurasi
metode
K-Nearest Neighbor
dengan menggunakan 470 fitur Rao
et al.
(2009) dan
dapat melakukan klasifikasi enzim dengan lebih mudah.
Ruang Lingkup Penelitian
Ruang lingkup penelitian ini meliputi:
1
Data yang digunakan ialah fasta sekuen enzim yang berasal dari situs
http://rcsb.org.
2
Fitur yang digunakan hanya 470 fitur dari total 479 fitur Rao
et al.
(2009)
3
Data sekuen fasta enzim yang dipilih hanya masuk dalam salah satu dari enam
kelas klasifikasi dan tidak ada yang ganda
4
Data yang digunakan untuk pelatihan sebanyak 2400 data fasta yang termasuk
dalam 6 kelas. Tiap kelas berjumlah 400 data fasta.
3
METODE
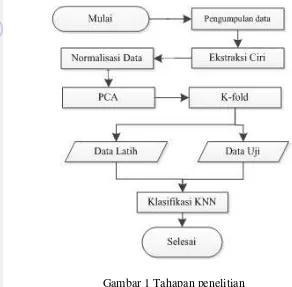
Penelitian ini dilakukan dengan beberapa tahap, yaitu pengumpulan data,
ekstraksi ciri, normalisasi data, melakukan PCA, membagi data dengan teknik
K-fold menjadi 2 bagian yaitu data latih dan data uji. Lalu melakukan klasifikasi
dengan metode K-Nearest Neighbor. Tahapan pada penelitian ini diGambarkan
pada Gambar1.
Gambar 1 Tahapan penelitian
Pengumpulan Data
Data yang digunakan adalah subtrat data fasta protein yang didapatkan dari
situs www.rcsb.org. yang berjumlah 3000 data. Terdapat 6 kelas dengan yaitu
Hydrolase
,
Isomerase
,
Ligase
,
Lyase
,
Oxydoreductase
dan
Transferase
dengan
tiap kelas berjumlah 500 data yang dipilih secara acak. Data keenam kelas
terdapat pada Lampiran 1 sampai Lampiran 6.
Ekstraksi Ciri
4
Tabel 1 Daftar persebaran fitur
Deksripsi fitur Jumlah fitur
Distribusi asam amino 20
Distribusi asam amino dua gram 400
Distribusi grup pertukaran 6
Distribusi grup pertukaran dua gram 36
Isoelectric point (pI) 1
Panjang sekuen 1
Berat molekular 1
Komposisi atomik 5
Total fitur 470
.
Distribusi Asam Amino
Sistem Asam amino merupakan unit dasar struktur protein. Suatu asam
amino α terdiri dari gugus amino, gugus karboksil, atom H dan gugus R tertentu
yang semuanya terikat pada atom karbon α. Atom karbon ini disebut α
karena
bersebelahan dengan gugus karboksil(asam). Gusgus R menyatakan rantai
samping (Mutiara Indah Sari 2007). Terdapat 20 jenis rantai samping yang
bervariasi dalam bentuk dan ukuran.
Distibusi Asam Amino Dua Gram
Pada distribusi asam amino dua gram, tiap jenis asam amino akan
dipasangkan dengan sebuah asam amino sehingga menjadi dua sekuen. Sehingga
jumlah fitur pada distribusi sam amino dua gram berjumlah 20 x 20 atau 400 fitur
(Rao
et al.
2009)
Distribusi Grup Pertukaran
Distribusi pertukaran grup. Dari 20 jenis asam amino tersebut,
dikelompokan menjadi 6 grup. 6 grup tersebut ditentukan berdasarkan kemiripan
tinggi dalam proses evolusinya. (Rao
et al.
2009)
Distribusi Grup Pertukaran Dua Gram
Distribusi pertukaran grup dua gram, didapat dengan mengkombinasikan 6
buah grup tersebut dalam 2 sekuen. Sehingga jumlah fitur pada distribusi grup
pertukaran adalah 36 (Rao
et al.
2009).
Isoelectric Point (pI)
Isoelectric point
adalah pH yang mana molekul tidak membawa muatan
listrik atau bermuatan nol. Isoelectric point dapat dihitung menggunakan
kalkulator
isoelectric
point
pada
situs
http://www.bioinformatics.org/sms2/protein_iep.html
.
Panjang Sekuen
5
Berat Molekular
Berat molekular merupakan berat dari suatu molekul. Berat suatu molekul
dapat dihitung dengan menjumlahkan massa setiap atom berdasarkan rumus
molekulnya. Berat molekular point dapat dihitung menggunakan kalkulator pada
situs
http://www.bioinformatics.org/sms/prot_mw.html
.
Komposisi Atomik
Komposisi atomic merupakan komposisi atom dari molekul protein. Protein
terbentuk oleh asam amino. Pada asam amino hanya terdapat 5 macam atom
yaitu : atom karbon(C), atom hidrogen(H), atom nitrogen(N), atom oksigen(O),
dan atom sulfur(S).
Normalisasi Data
Pada penelitian ini normalisasi data dilakukan dengan menggunakan teknik
normalisasi min-max. Untuk Normalisasi data dilakukan untuk membuat data
hanya bernilai dari 0 sampai satu. Normalisasi dilakukan dengan cara berikut
(Shalabi 2006):
�′
=
� � − � ��− � �� � −
� �
+
� �
(1)
�′
: Nilai data setelah normalisasi
�
: Nilai data awal yang akan dinormalisasikan
maxA
: Nilai data awal terbesar
minA
: Nilai data awal terkecil
newmaxA : Nilai data maksimum setelah dinormalisasi (nilainya 1)
newminA
: Nilai data minimum setelah dinormalisasi (nilainya 0)
Principal Components Analysis (PCA)
PCA adalah teknik yang biasa digunakan untuk mereduksi dimensi data dan
tetap menjaga nilai infromasi penting dari data tersebut Peubah hasil transformasi
PCA merupakan kombinasi linier dari peubah asli dan tersusun berdasarkan
infromasi kandungnya yang disebut sebagai vector
eigen
atau nilai komponen
utama (Abdi, William 2010)
Data matriks kovarian
S
dihitung dengan menggunakan persamaan :
=
1+
�
� �− �
=1
�
�− �
(2)
S
: matrik kovarian
n
: unit sampel
Xi
: jumlah vektor
X
: rata-rata vektor
6
Setelah ditemukan nilai matriks kovarian, ditentukan nilai eigen. Nilai
eigen diurutkan dari teerbesar sampai terkecil. Dalam penelitian ini digunakan
nilai kontribusi 80%, 85%, dan 90%.
K-Fold Cross Validation
K-fold Cross Validation
adalah metode pembagian sebuah kelompok data
yang akan dibagi ke dalam data latih dan data uji . Pembuatan partisi dilakukan
dengan cara melakukan pengolahan data sebanyak k kali dengan menggunakan k-
1 data latih dan sisanya data uji. Akurasi didapat dari rata-rata seluruh k
percobaan (Zhang dan Wu 2011). Pada penelitian ini akan digunakan 5 fold.
Dengan data uji berjumlah 600 data dan data latih berjumlah 2400 data.
K-Nearest Neighbor
K-Nearest Neighbor
(K-NN) adalah suatu metode yang menggunakan
algoritme
supervised
yang mana hasil dari
query instance
yang baru
klasifikasikan berdasarkan mayoritas dari kategori pada
k
-NN. Tujuan dari
algoritme ini adalah menglasifikasi objek baru berdasarkan atribut dan
training
sample.
Pada penelitian nilai
k
yang digunakan adalah bilangan ganji dari 3
hingga 21. Digunakan bilangan ganjil sebagai
k
untuk memperkecil kemungkinan
terjadinya dua kelas atau lebih yang mempunyai jumlah nilai
k
sama. Jika terjadi
dua kelas atau lebih memiliki jumlah nilai
k
yang sama maka kelas yang dipilih
merupakan kelas yang memiliki nilai jarak
euclidian
yang terdekat.
Jarak dengan data tetangga dihitungan dengan jarak
euclidian
dengan
persamaan sebagai berikut :
( , ) =
(
�−
�)
2�=1
(3)
d
: jarak data uji ke data pembelajaran
x
i: data uji ke-i
yi
: data pembelajaran ke-i
n
: banyak data
Evaluasi
Penelitian diuji dengan menghitung akurasi, sensitivitas dan spesifisitas.
Perhitungan akurasi sensitivitas dan spesifisitas dilakukan dengan menggunakan
matriks konfusi. Persamaan dan Tabel 2 di bawah ini digunakan untuk
menghitung akurasi, sensitivitas dan spesifisitas (Akobeng 2007). Matriks yang
akan dibuat sejumlah kelas yang ada yaitu 6 buah.
Tabel 2 Matriks konfusi
Kelas aktual
Kelas prediksi
A (class positif)
¬ A (class negatif)
A(tes positif)
TP
FN
7
Akurasi
=
TP
TN
FP
FN
TN
TP
x 100%
Sensitivitas
=
TP FNTP
x 100%
Spesifisitas =
TN FPTN
x 100%
Ruang Lingkup Pengembangan
Penelitian ini diimplementasikan menggunakan spesifikasi perangkat keras
dan lunak sebagai berikut:
Perangkat Keras :
•
Spesifikasi perangkat keras yang digunakan adalah:
•
Intel Core i5 CPU @ 1.6 GHz., ~2,3GHz.
•
Harddisk
500 GB.
•
Memori 2 GB.
Perangkat Lunak :
•
Sistem operasi
Windows
7.
•
XAMPP
•
Matlab
•
PHP
HASIL DAN PEMBAHASAN
Pengumpulan data
Data yang digunakan adalah data fasta enzim yang didapat dari situs
www.rcsb.org
. pada penelitian ini terdapat 6 kelas
Hydrolase
,
Isomerase
,
Lyase
,
Lyase
,
Oxydoreductase
dan
Transferase
. Masing-masing kelas diambil 500 data,
sehingga total data yang akan digunakan adalah 3000. Karena memungkinkan
untuk satu enzim bisa masuk lebih dari satu kelas. Maka data yang diambil yang
hanya termasuk dalam satu dari enam kelas yang ada.
Ekstraksi Ciri
8
Distribusi Asam Amino
Pada protein ada 20 jenis rantai samping. Distribusi asam amino merupakan
banyaknya kemunculan dari 20 jenis rantai samping yang ada pada suatu protein.
Nama para asam amino penyusun protein bisa dilihat pada Tabel 3.
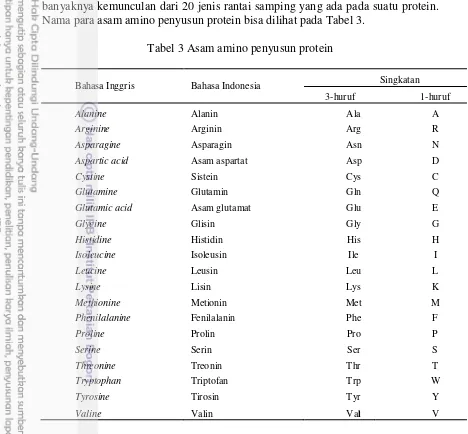
Tabel 3 Asam amino penyusun protein
Bahasa Inggris Bahasa Indonesia Singkatan 3-huruf 1-huruf
Alanine Alanin Ala A
Arginine Arginin Arg R
Asparagine Asparagin Asn N
Aspartic acid Asam aspartat Asp D
Cystine Sistein Cys C
Glutamine Glutamin Gln Q
Glutamic acid Asam glutamat Glu E
Glycine Glisin Gly G
Histidine Histidin His H
Isoleucine Isoleusin Ile I
Leucine Leusin Leu L
Lysine Lisin Lys K
Methionine Metionin Met M
Phenilalanine Fenilalanin Phe F
Proline Prolin Pro P
Serine Serin Ser S
Threonine Treonin Thr T
Tryptophan Triptofan Trp W
Tyrosine Tirosin Tyr Y
Valine Valin Val V
Distibusi Asam Amino Dua Gram
Pada distribusi asam amino dua gram, tiap jenis asam amino akan
dipasangkan dengan sebuah asam amino sehingga menjadi dua sekuen. Sehingga
jumlah fitur berjumlah 20 x 20 atau 400 fitur (Rao
et al.
2009) yang ditunjukan
pada Tabel 4.
Tabel 4 Distribusi asam amino dua gram
A R N D … P S T W Y V A AA AR AN AD … AP AS AT AW AY AV R RA RR RN RD … RP RS RT RW RY RV N NA NR NN ND … NP NS NT NW NY NV
… … … …
9
Distribusi Grup Pertukaran
Distribusi pertukaran grup. Dari 20 jenis asam amino tersebut,
dikelompokan menjadi enam grup. Yang ditentukan berdasarkan kemiripan tinggi
dalam proses evolusinya. (Rao
et al.
2009). enam grup itu adalah :
o
e
1 = {H,R,K}
o
e
2 = {D,E,N,Q}
o
e
3 ={C}
o
e
4 = {A,G,P,S,T}
o
e
5 = {I,L,M,V}
o
e
6 = {F,Y,W}.
Distribusi Grup Pertukaran Dua Gram
Distribusi pertukaran grup dua gram, didapat dengan mengkombinasikan 6
buah grup tersebut dalam 2 sekuen. Sehingga jumlah fitur pada distribusi grup
pertukaran adalah 36 (Rao
et al.
2009).
Tabel 5 Distribusi grup pertukaran dua gram
e1
e2
e3
e4
e5
e6
e1
e1e1
e1e2
e1e3
e1e4
e1e5
e1e6
e2
e2e1
e2e2
e2e3
e2e4
e2e5
e2e6
e3
e3e1
e3e2
e3e3
e3e4
e3e5
e3e6
e4
e4e1
e4e2
e4e3
e4e4
e4e5
e4e6
e5
e5e1
e5e2
e5e3
e5e4
e5e5
e5e6
e6
e6e1
e6e2
e6e3
e6e4
e6e5
e6e6
Panjang Sekuen
Panjang sekuen yang dimaksud ialah panjangnya rantai suatu molekul
protein. Rantai molekul protein dibentuk oleh beberapa asam amino. Panjang
sekuen adalah jumlah banyaknya asam amino yang membentuk protein. Panjang
sekuen nilainya satu fitur.
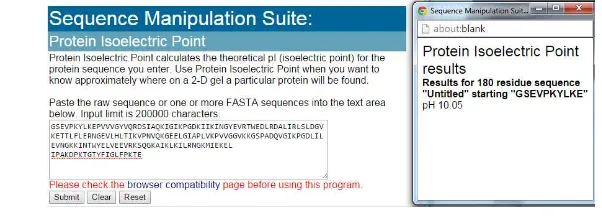
Isoelectric Point (pI)
Isoelectric point
adalah pH yang mana molekul tidak membawa muatan
listrik atau bermuatan nol. Jumlah fitur hanya satu fitur yaitu nilai yang
dikeluarkan berupa pH yang besarnya antara nol dan satu. Contoh hasil
isoelectric
point
pada data fasta 3WKL pada Gambar 2.
10
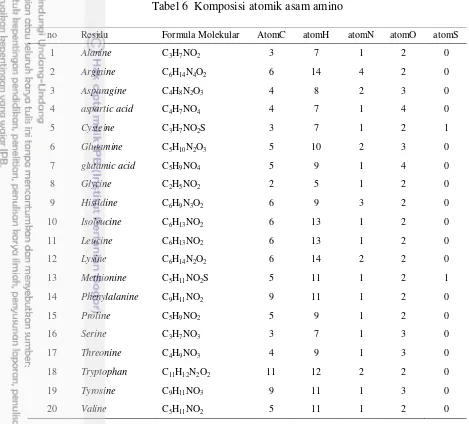
Komposisi Atomik
Komposisi atomic merupakan komposisi atom dari molekul protein. Protein
terbentuk oleh asam amino. Pada asam amino hanya terdapat 5 macam atom yaitu,
atom karbon(C), atom hidrogen(H), atom nitrogen(N), atom oksigen(O), dan atom
sulfur(S). fitur yang berada pada komposisi atomik berjumlah 5 (Mathews
et al.
2013).
Tabel 6 Komposisi atomik asam amino
no Residu Formula Molekular AtomC atomH atomN atomO atomS
1 Alanine C3H7NO2 3 7 1 2 0
2 Arginine C6H14N4O2 6 14 4 2 0
3 Asparagine C4H8N2O3 4 8 2 3 0
4 aspartic acid C4H7NO4 4 7 1 4 0
5 Cysteine C3H7NO2S 3 7 1 2 1
6 Glutamine C5H10N2O3 5 10 2 3 0
7 glutamic acid C5H9NO4 5 9 1 4 0
8 Glycine C2H5NO2 2 5 1 2 0
9 Histidine C6H9N3O2 6 9 3 2 0
10 Isoleucine C6H13NO2 6 13 1 2 0
11 Leucine C6H13NO2 6 13 1 2 0
12 Lysine C6H14N2O2 6 14 2 2 0
13 Methionine C5H11NO2S 5 11 1 2 1
14 Phenylalanine C9H11NO2 9 11 1 2 0
15 Proline C5H9NO2 5 9 1 2 0
16 Serine C3H7NO3 3 7 1 3 0
17 Threonine C4H9NO3 4 9 1 3 0
18 Tryptophan C11H12N2O2 11 12 2 2 0
19 Tyrosine C9H11NO3 9 11 1 3 0
20 Valine C5H11NO2 5 11 1 2 0
Berat Molekular
11
Gambar 3 Berat molekul 3WKL
Normalisasi
Pada tahap normalisasi akan dilakukan normalisasi min-max. normalisasi
bertujuan untuk membuat nilai fitur antara nol dan satu (Shalabi 2006). Hal ini
dilakukan untuk menyeimbangkan nilai fitur. Misal nilai fitur pada panjang
sekuen bisa mencapai angka ribuan dan nilai fitur isoelectric point adalah pH
yaitu hanya berkisar 0 sampai 14. Hal ini dapat menyebabkan nilai fitur isoelectric
point tidak berarti jika dibandingkan panjang sekuen jika dilakukan klasifikasi
dengan metode
K-Nearest Neighbor
.
Principal Components Analysis (PCA)
Semua data yang selesai diekstraksi membentuk matriks 3000 x 470. 3000
untuk jumlah data dan 470 untuk jumlah fitur. Dilakukan PCA 80% PCA 85%
dan PCA 90% untuk mengurangi nilai fitur. Nilai kontribusi yang digunakan
untuk tiap PCA adalah yang paling mendekati dengan nilainya seperti yang
ditunjukan pada Tabel 7.
Tabel 7 Hasil reduksi PCA
PCA Nilai Kontribusi Jumlah Ciri 80% 80.17 136 85% 85.00 166 90% 90.09 207
K-Fold Cross Validation
12
Hasil Klasifikasi KNN
Pada tahap Klasifikasi menggunakan metode KNN data yang digunakan
dalam klasifikasi adalah :
data ekstraksi ciri awal (sebelum di PCA).
data ekstraksi ciri PCA 80%.
data PCA 85%.
data PCA 90%.
Tiap set data tersebut dilakukan klasifikasi dengan menggunakan metode
KNN dengan k=bilangan ganjil dari 3 sampai 21.hasil percobaan klasifikasi KNN
bisa dilihat pada Tabel 8.
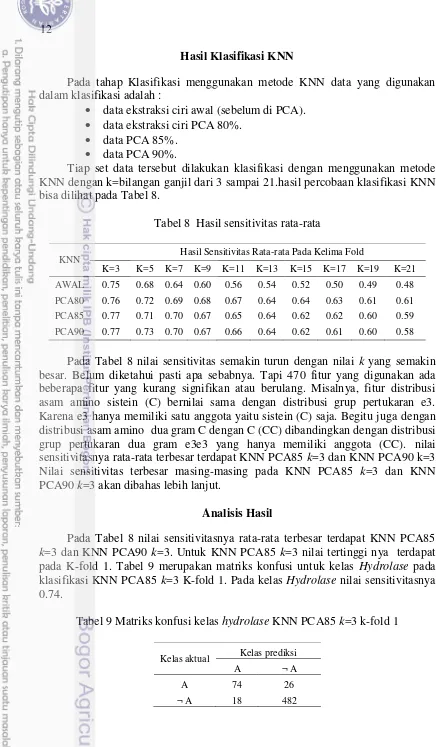
Tabel 8 Hasil sensitivitas rata-rata
KNN Hasil Sensitivitas Rata-rata Pada Kelima Fold
K=3 K=5 K=7 K=9 K=11 K=13 K=15 K=17 K=19 K=21 AWAL 0.75 0.68 0.64 0.60 0.56 0.54 0.52 0.50 0.49 0.48 PCA80 0.76 0.72 0.69 0.68 0.67 0.64 0.64 0.63 0.61 0.61 PCA85 0.77 0.71 0.70 0.67 0.65 0.64 0.62 0.62 0.60 0.59 PCA90 0.77 0.73 0.70 0.67 0.66 0.64 0.62 0.61 0.60 0.58
Pada Tabel 8 nilai sensitivitas semakin turun dengan nilai
k
yang semakin
besar. Belum diketahui pasti apa sebabnya. Tapi 470 fitur yang digunakan ada
beberapa fitur yang kurang signifikan atau berulang. Misalnya, fitur distribusi
asam amino sistein (C) bernilai sama dengan distribusi grup pertukaran e3.
Karena e3 hanya memiliki satu anggota yaitu sistein (C) saja. Begitu juga dengan
distribusi asam amino dua gram C dengan C (CC) dibandingkan dengan distribusi
grup pertukaran dua gram e3e3 yang hanya memiliki anggota (CC). nilai
sensitivitasnya rata-rata terbesar terdapat KNN PCA85
k
=3 dan KNN PCA90 k=3.
Nilai sensitivitas terbesar masing-masing pada KNN PCA85
k
=3 dan KNN
PCA90
k
=3 akan dibahas lebih lanjut.
Analisis Hasil
Pada Tabel 8 nilai sensitivitasnya rata-rata terbesar terdapat KNN PCA85
k
=3 dan KNN PCA90
k
=3. Untuk KNN PCA85
k
=3 nilai tertinggi nya terdapat
pada K-fold 1. Tabel 9 merupakan matriks konfusi untuk kelas
Hydrolase
pada
klasifikasi KNN PCA85
k
=3 K-fold 1. Pada kelas
Hydrolase
nilai sensitivitasnya
0.74.
Tabel 9 Matriks konfusi kelas
hydrolase
KNN PCA85
k
=3 k-fold 1
13
Tabel 10 merupakan untuk kelas
Isomerase
pada klasifikasi KNN PCA85
k
=3 K-fold 1. Pada kelas
Isomerase
nilai sensitivitasnya 0.77.
Tabel 10 Matriks konfusi kelas
isomerase
KNN PCA85
k
=3 k-fold 1
Kelas aktual Kelas prediksi A ¬ A A 77 23 ¬ A 27 473
Tabel 11 merupakan untuk kelas
Ligase
pada klasifikasi KNN PCA85
k
=3
K-fold 1. Pada kelas
Ligase
nilai sensitivitasnya 0.86.
Tabel 11 Matriks konfusi kelas
ligase
KNN PCA85
k
=3 k-fold 1
Kelas aktual Kelas prediksi A ¬ A A 86 14 ¬ A 24 476
Tabel 12 merupakan untuk kelas
Lyase
pada klasifikasi KNN PCA85
k
=3
K-fold 1. Pada kelas
Lyase
nilai sensitivitasnya 0.85.
Tabel 12 Matriks konfusi kelas
lyase
KNN PCA85
k
=3 k-fold 1
Kelas aktual Kelas prediksi A ¬ A A 85 15 ¬ A 26 474
Tabel 13 merupakan untuk kelas
Oxydoreductase
pada klasifikasi KNN
PCA85
k
=3 K-fold 1. Pada kelas
Lyase
nilai sensitivitasnya 0.79.
Tabel 13 Matriks konfusi kelas
oxydoreductase
KNN PCA85
k
=3 k-fold 1
Kelas aktual Kelas prediksi A ¬ A A 79 21 ¬ A 14 486
Tabel 14 merupakan untuk kelas
Transferase
pada klasifikasi KNN PCA85
14
Tabel 14 Matriks konfusi kelas
transferase
KNN PCA85
k
=3 k-fold 1
Kelas aktual Kelas prediksi A ¬ A A 72 28 ¬ A 18 482
Nilai akurasi, sensitivitas dan spesifisitas pada klasifikasi KNN PCA85
k
=3
K-fold satu bisa dilihat pada Tabel 15.
Tabel 15 Nilai akurasi, sensitivitas dan spesifisitas KNN PCA85
k
=3 k-Fold 1
Hydrolase Isomerase Ligase Lyase Oxydoreductase Transferase rataan
Akurasi 0.93 0.92 0.94 0.93 0.94 0.92 0.93 Sensitivitas 0.74 0.77 0.86 0.85 0.79 0.72 0.79 Spesifisitas 0.96 0.95 0.95 0.95 0.97 0.96 0.96
Tabel 16 merupakan matriks konfusi untuk kelas
Hydrolase
pada klasifikasi
KNN PCA90
k
=3 K-fold 2. Pada kelas
Hydrolase
nilai sensitivitasnya 0.75.
Tabel 16 Matriks konfusi kelas
hydrolase
KNN PCA90
k
=3 k-fold 2
Kelas aktual Kelas prediksi A ¬ A A 75 25 ¬ A 20 480
.
Tabel 17 merupakan matriks konfusi untuk kelas
Isomerase
pada klasifikasi
KNN PCA90
k
=3 K-fold 2. Pada kelas
Isomerase
nilai sensitivitasnya 0.86.
Tabel 17 Matriks konfusi kelas
isomerase
KNN PCA90
k
=3 k-fold 2
Kelas aktual Kelas prediksi A ¬ A A 86 14 ¬ A 23 477
Tabel 18 merupakan matriks konfusi untuk kelas
Ligase
pada klasifikasi
KNN PCA90
k
=3 K-fold 2. Pada kelas
Ligase
nilai sensitivitasnya 0.83.
Tabel 18 Matriks konfusi kelas
ligase
KNN PCA90
k
=3 k-fold 2
15
Tabel 19 merupakan matriks konfusi untuk kelas
Lyase
pada klasifikasi
KNN PCA90 k=3 K-fold 2. Pada kelas
Lyase
nilai sensitivitasnya 0.81.
Tabel 19 Matriks konfusi kelas
lyase
KNN PCA90 k=3 k-fold 2
Kelas aktual Kelas prediksi A ¬ A A 81 19 ¬ A 32 468
Tabel 20 merupakan matriks konfusi untuk kelas
Oxydoreductase
pada
klasifikasi KNN PCA90
k
=3 K-fold 2. Pada kelas
Oxydoreductase
nilai
sensitivitasnya 0.82.
Tabel 20 Matriks konfusi kelas
oxydoreductase
KNN PCA90
k
=3 k-fold 2
Kelas aktual Kelas prediksi A ¬ A A 82 18 ¬ A 11 489
Tabel 21 merupakan matriks konfusi untuk kelas
Transferase
pada
klasifikasi KNN PCA90
k
=3 K-fold 2. Pada kelas
Transferase
nilai
sensitivitasnya 0.66.
Tabel 21 Matriks konfusi kelas
transferase
KNN PCA90
k
=3 k-fold 2
Kelas aktual Kelas prediksi A ¬ A A 66 34 ¬ A 18 482
Nilai akurasi, sensitivitas dan spesifisitas pada klasifikasi KNN PCA85
k
=3
K-fold satu bisa dilihat pada Tabel 15.
Tabel 22 Nilai akurasi, sensitivitas dan spesifisitas KNN PCA90
k
=3 k-Fold 2
Hydrolase Isomerase Ligase Lyase Oxydoreduktase Transferase
16
Dari kedua hasil klasifikasi KNN yang memiliki sensitivitas terbesar. Kelas
transferase memiliki nilai sensitivitas yang terkecil dibandingkan dengan lima
kelas lainnya. Hal ini belum diketahui penyebabnya.
SIMPULAN DAN SARAN
Simpulan
Penelitian ini berhasil penerapan K-NN dan PCA dengan penggunaan 470
fitur Rao
et al.
(2009) dalam mengklasifikasikan enzim. Penggunaan PCA 85%
dan 90% menghasilkan klasifikasi KNN pada enzim dengan nilai sensitivitas
rata-rata tertinggi 0.79 pada nilai
k
=3.
Saran
Beberapa saran untuk penelitian selanjutnya yaitu:
1
Mengoptimasikan fitur, dengan menambah atau mengurangi fitur
2
Menggunakan
classifier
yang lain untuk mengetahui apakah
classifier
lain
meningkatkan nilai akurasi, sensitivitas dan spesifitas.
DAFTAR PUSTAKA
Abdi H, Williams LJ. 2010. Principal component analysis. Wiley Interdisciplinary
Reviews:
Computational Statistics
2. 2: 433
–
459.
Akobeng AK. 2007. Understanding Diagnostic Tests 1: Sensitivitas, Spesifisitas,
and Predicting Values
. Foundation Acta Paediatrica
2006, pp.338-341.
Marks DB, Marks AD, Smith CM. 1996.
Biokimia Kedokteran Dasar: Sebuah
Pendekatan Klinis
. Pendit BU, penerjemah; Suyono J, Sadikin V, Mandera
LI, editor. Jakarta (ID): Penerbit EGC. Terjemahan dari:
Basic Medical
Biochemistry: A Clinical Approach
.
Mathews CK, Van Holde KE, Appling DR, Anthony-Cahill SJ
et al
. 2013.
Biochemistry
. ED ke-4. Toronto(US). Pearson.
Rao PN, Devi TU, Kladhar D, Sridhar G, RAO AP. 2009. A Probabilistic Neural
Network Approach for Protein Superfamily Classification.
Journal of
Theoretical and Applied Information Technologi
.
Sari MI. 2007.
Struktur Protein
. Fakultas Kedokteran, Universitas Sumatra Utara.
Shalabi LA, Shaaban Zyad, Kasasbeh B. 2006. Data Mining: A Preprocessing
Engine.
Journal of Computer of Science
. 2(9):735-739, 2006.
Shmueli G, Patel NR, Bruce PC. 2005.
Data Mining in Excel: Lecture Notes and
Cases
. Arlington (US): Resampling Stats, inc.
Simangunsong, VFR. 2015. Klasifikasi fragmen metagenon menggunakan
Principal Component Analysis dan K-Nearest Neighbor [skripsi].
Bogor(ID):Institut Pertanian Bogor.
17
Lampiran 1 Data kelas
hydrolase
18
Lanjutan
19
Lanjutan
20
Lampiran 2 Data kelas
isomerase
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
21
Lanjutan
22
Lanjutan
23
Lampiran 3 Data kelas
ligase
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
24
Lanjutan
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
25
Lanjutan
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
26
Lampiran 4 Data kelas
lyase
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
27
Lanjutan
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
28
Lanjutan
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
29
Lampiran 5 Data kelas
oxydoreductase
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
30
Lanjutan
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
31
Lanjutan
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
32
Lampiran 6 Data kelas
transferase
No PDB ID No PDB ID No PDB ID No PDB ID No PDB ID
33
Lanjutan
34
Lanjutan
35
RIWAYAT HIDUP