IKAN TUNA MATA BESAR (
Thunnus obesus
)
HASIL TANGKAPAN
TUNA LONGLINE
YANG DIDARATKAN DI BENOA
BUDI NUGRAHA
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis ”Studi Tentang Genetika Populasi Ikan Tuna Mata Besar (Thunnus obesus) Hasil Tangkapan Tuna Longline yang Didaratkan di Benoa” adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, Agustus 2009
Budi Nugraha
BUDI NUGRAHA. Study of Population Genetic of Bigeye Tuna (Thunnus Obesus) Caught by Tuna Longline Landed at Benoa. Under direction of MULYONO S. BASKORO, ANWAR BEY PANE, and ESTU NUGROHO.
Bigeye tuna (Thunnus obesus) is one of Family Scombridae and one of tuna export commodity in Indonesia beside yellowfin tuna (T. albacares) and southern bluefin tuna (T. maccoyii). The utilization of tuna resource, especially bigeye tuna in Indian Ocean from year to year tended to increase, therefore the tuna resources in this ocean have indicated over fishing. It is needed a correctly management concept. This concept will be executed effective if made available by data about population condition of bigeye tuna with the accurate and clear definition. One of the methods can be applied to determine the population condition of fish and structure genetics with the high accuration storey by polymorphism DNA; and mitochondria DNA (mtDNA) which are trusted very relevant to the study. Method of diversity genetics measurement for genotype can be conducted with the DNA analysis. Study on population genetic of bigeye tuna from Indian Ocean in Indonesia have never been done. This is important study topic of genetics population of bigeye tuna caught by tuna longline landed in Benoa. The objectives of this study are to obtain information on genetics diversity among population and population structure of bigeye tuna in Indian Ocean of southern Java and Nusa Tenggara. Data of composite haplotype analyses to get the genetics parameter, population structure and relationship of phylogenetic between populations: level of genetics diversity is measured by haplotype diversity index; population relationship determined by genetics distance parameter; degree of difference of molecular haplotype among population is calculated by using Analysis of Molecular Varian (AMOVA) and Fst test; relationship of phylogenetic among population depicted in the form of dendrogram through the clustering assess the genetics distance according to average thread method. Calculation conducted constructively using software TFPGA (Tools for Population Genetics Analysis). The results showed that the value of haplotype diversity from bigeye tuna population have variation between 0,5578-0,8136. The value of haplotype diversity was relatively high, indicated that the condition of bigeye tuna population was undisturbed. The average of genetic distance among sample group was 0,2572. Distance of closest genetic was between sample group 2 and 5. The sample groups were divided become two population groups, the first group was bigeye tuna from sample group 2, 5 and 1, and the second group was from sample group 3 and 4.
RINGKASAN
BUDI NUGRAHA. Studi Tentang Genetika Populasi Ikan Tuna Mata Besar (Thunnus Obesus) Hasil Tangkapan Tuna Longline yang Didaratkan di Benoa. Dibimbing oleh MULYONO S. BASKORO, ANWAR BEY PANE, dan ESTU NUGROHO.
Tuna mata besar atau bigeye tuna (Thunnus obesus) merupakan salah satu komoditi ekspor perikanan tuna yang paling utama di Indonesia selain tuna sirip kuning (T. albacares) dan tuna sirip biru selatan (T. maccoyii). Seiring dengan meningkatnya permintaan pasar dari tahun ke tahun, maka semakin tinggi pula eksploitasi terhadap jenis tuna mata besar, sehingga di perairan Samudera Hindia, sudah terindikasi lebih tangkap atau mendekati titik jenuh. Oleh karena itu diperlukan suatu konsep manajemen yang tepat, dalam jangka panjang dapat menjamin hasil tangkapan yang menguntungkan tetapi kelestarian sumber daya tetap terjaga. Konsep ini akan terlaksana efektif apabila tersedia data tentang kondisi populasi tuna mata besar dengan definisi yang jelas dan akurat. Salah satu metode yang dapat diterapkan untuk menentukan kondisi populasi ikan dan struktur genetiknya dengan tingkat akurasi yang tinggi adalah didasarkan pada polimorfisme DNA; dan DNA mitokondria dipercaya sangat relevan bagi studi tersebut. Penelitian genetika populasi ikan tuna mata besar di Indonesia dari perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara belum pernah dilakukan. Hal di atas, kiranya yang mendasari perlunya diadakan penelitian mengenai genetika populasi ikan tuna mata besar hasil tangkapan tuna longline
yang didaratkan di Benoa. Penelitian ini bertujuan untuk mendapatkan informasi keragaman genetik dan struktur populasi ikan tuna mata besar dari perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara.
Hasil amplifikasi D-Loop mtDNA pada ikan tuna mata besar dengan menggunakan primer Pro-5, dan 12SAR menghasilkan fragmen DNA berukuran sekitar 1.500 bp pada semua sampel ikan tuna mata besar. Keragaman jumlah situs dan ukuran fragmen restriksi yang diperoleh dari hasil restriksi mtDNA dengan empat enzim adalah 18 tipe restriksi yaitu Taq I dan Hin6 I dengan empat tipe restriksi A, B, C dan D, Afa I dan Mbo I dengan lima tipe restriksi A, B, C, D dan E. Berdasarkan tipe restriksi mtDNA tersebut, enzim Afa I dan Mbo I dengan lima tipe restriksi merupakan enzim yang paling sensitif mendeteksi perbedaan panjang fragmen terpotong dibandingkan enzim Taq I dan Hin6 I.
Analisis komposit haplotipe menghasilkan 23 komposit haplotipe pada seluruh kelompok sampel. Jumlah terendah yang diamati adalah pada kelompok sampel 5 (7 komposit haplotipe), sedangkan jumlah tertinggi terdapat pada kelompok sampel 1 (12 komposit haplotipe). Nilai keragaman haplotipe bervariasi antara 0,5578 (kelompok sampel 5) hingga 0,8136 (kelompok sampel 4). Komposit haplotipe BBAB tertinggi ditemukan pada kelompok sampel 5 (65%), selanjutnya berurutan adalah kelompok sampel 2 (54%), kelompok sampel 1 (50%) dan kelompok sampel 4 (30%), dan komposit haplotipe ABAB (43%) pada kelompok sampel 3.
sampel 3 dan 4, kelompok sampel 3 dengan kelompok sampel 4 dan 5 serta kelompok sampel 4 dengan kelompok sampel 5. Tidak ada perbedaan yang nyata antara kelompok sampel 1 dengan 2 dan 5 serta kelompok sampel 2 dengan kelompok sampel 5.
Jarak genetik rata-rata antara kelompok sampel ikan tuna mata besar adalah sekitar 0,2572. Ikan tuna mata besar dari kelompok sampel 1 memiliki jarak genetik yang jauh dengan kelompok sampel 3, tetapi memiliki jarak genetik yang dekat dengan kelompok sampel 2 dan 5. Kelompok sampel 2 memiliki jarak genetik yang jauh dengan kelompok sampel 3, tetapi memiliki jarak genetik yang dekat dengan kelompok sampel 5. Kelompok sampel 3 memiliki jarak genetik yang jauh dengan kelompok sampel 5, tetapi memiliki jarak genetik yang dekat dengan kelompok sampel 4. Dari semua kelompok sampel, ikan tuna mata besar dari kelompok sampel 3 dan 5 memiliki jarak genetik terjauh dengan nilai 0,6621, sedangkan jarak genetik terdekat adalah antara kelompok sampel 2 dan 5 yaitu sebesar 0,0383. Nilai jarak genetik yang rendah antara kelompok sampel 1 dan 2, 1 dan 5, dan 2 dan 5 menunjukkan kedekatan kelompok sampel-kelompok sampel tersebut.
Dendrogram yang dibentuk berdasarkan jarak genetik menunjukkan bahwa kelompok sampel ikan tuna mata besar yang diamati dapat dibagi menjadi dua kelompok, yaitu kelompok pertama terdiri dari ikan tuna mata besar yang berasal dari kelompok sampel 2, 5 dan 1, sedangkan kelompok kedua yang berasal dari kelompok sampel 3 dan 4.
Analisis terhadap kelima kelompok sampel ikan tuna mata besar di Samudera Hindia menunjukkan bahwa keragaman genetik yang dimiliki relatif tinggi, namun pada kelompok sampel 5 mempunyai keragaman genetik yang rendah. Rendahnya keragaman genetik pada kelompok sampel 5 diduga karena daerah penangkapannya terletak di perairan ZEE Indonesia, dimana daerah tersebut sudah terindikasi lebih tangkap. Berdasarkan temuan tersebut di atas, manajemen perikanan tuna yang dapat dilakukan sebaiknya mencakup tujuan jangka pendek (terkait dengan penangkapan berlebih) dan tujuan jangka panjang dalam program konservasi, sehingga kelangsungan sumber daya dengan hasil tangkapan yang optimal dapat tercapai. Beberapa hal yang perlu dilakukan agar kedua tujuan tersebut dapat tercapai diantaranya adalah dengan menjadikan daerah penangkapan di perairan ZEE Indonesia sebagai daerah tertutup bagi penangkapan untuk sementara waktu tertentu, membatasi produksi (kuota) atau jumlah tangkapan yang diperbolehkan, membatasi upaya penangkapan dengan tidak menambah izin dan sekaligus mengurangi jumlah kapal penangkap ikan di perairan tersebut, pengawasan terhadap kapal-kapal tuna longline yang melakukan
IUU fishing di perairan ZEE Indonesia, dan melakukan kerjasama dengan organisasi pengelolaan perikanan regional khususnya perikanan tuna di Samudera Hindia yaitu IOTC agar Indonesia dapat berperan lebih aktif dalam mengelola perikanan tuna di Samudera Hindia.
© Hak Cipta milik IPB, tahun 2009
Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
IKAN TUNA MATA BESAR (
Thunnus obesus
)
HASIL TANGKAPAN
TUNA LONGLINE
YANG DIDARATKAN DI BENOA
BUDI NUGRAHA
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Mayor Teknologi Perikanan Tangkap
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Studi Tentang Genetika Populasi Ikan Tuna Mata Besar (Thunnus obesus) Hasil Tangkapan Tuna Longline yang Didaratkan di Benoa
Nama Lengkap : Budi Nugraha
NRP : C451070071
Disetujui
Komisi Pembimbing
Prof. Dr. Ir. Mulyono S. Baskoro, M.Sc. Ketua
Dr. Ir. Anwar Bey Pane, DEA. Dr. Ir. Estu Nugroho, M.Sc.
Anggota Anggota
Diketahui
Koordinator Mayor Dekan Sekolah Pascasarjana Teknologi Perikanan Tangkap
Dr. Ir. M. Fedi A. Sondita, M.Sc. Prof. Dr. Ir. Khairil A. Notodiputro, M.S.
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Juli 2008 ini ialah genetika populasi, dengan judul “Studi Tentang Genetika Populasi Ikan Tuna Mata Besar
(Thunnus Obesus) Hasil Tangkapan Tuna Longline yang Didaratkan di
Benoa”.
Terima kasih yang sebesar-besarnya penulis sampaikan kepada:
1. Prof. Dr. Ir. Mulyono S. Baskoro, M.Sc., Dr. Ir. Anwar Bey Pane, DEA. dan Dr. Ir. Estu Nugroho, M.Sc. selaku Komisi Pembimbing, yang telah membimbing dan mengarahkan penelitian ini.
2. Kepala Pusat Riset Perikanan Tangkap dan Kepala Balai Riset Perikanan Laut, atas perkenannya untuk tugas belajar dan kesempatan mendapatkan biaya pendidikan.
3. Dr. Ir. M. Fedi A. Sondita, M.Sc. selaku Ketua Koordinator Mayor Teknologi Perikanan Tangkap, atas bimbingan, arahan, dorongan semangat dan bantuannya selama penulis belajar di Mayor Teknologi Perikanan Tangkap. 4. Iskandariah, S.Pi. dari Balai Riset Perikanan Budidaya Air Tawar Bogor yang
telah membantu melakukan analisis di laboratorium.
5. Teman-teman angkatan 2007 yang telah memberi dorongan dan semangat selama perkuliahan.
6. Istri dan anak-anak tercinta, atas dorongan semangat, kesabaran serta doa yang tulus.
7. Ayah dan ibu, atas segala doa dan kasih sayangnya.
8. Semua pihak yang tidak dapat disebutkan satu persatu, yang telah memberi dukungan selama tugas belajar.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2009
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 21 Maret 1973 dari Ayah Pepen Suparna dan Ibu Enok Rohaeni. Penulis merupakan putra pertama dari tiga bersaudara. Telah dikaruniai seorang putra, Luthfi Dirsya Nugraha dan seorang putri, Khairani Lathifah Nugraha, buah pernikahan dengan Linda Rakhmawati.
Pendidikan sarjana ditempuh pada tahun 1992 pada Jurusan Pemanfaatan Sumberdaya Perikanan, Fakultas Perikanan, Institut Pertanian Bogor dan lulus pada tahun 1996. Pada tahun 2003 mulai bekerja sebagai staf peneliti pada Balai Riset Perikanan Laut Jakarta. Pada tahun 2007 mendapat kesempatan mengikuti program pendidikan Pascasarjana di Institut Pertanian Bogor pada Mayor Teknologi Perikanan Tangkap, melalui program pendidikan Badan Riset Kelautan dan Perikanan Departemen Kelautan dan Perikanan, yang dibiayai oleh dana APBN.
xi
2.4 RFLP (Restriction Fragment Length Polymorphism) ... 14
2.5 PCR (Polymerase Chain Reaction) ... 15
3.3.4 Restriksi mtDNA dan Visualisasi Hasil Restriksi ... 23
3.4 Analisis Data ... 24
4 HASIL DAN PEMBAHASAN ... 25
4.1 Sampel Ikan Tuna Mata Besar ... 25
4.2 Keragaman Genetik Ikan Tuna Mata Besar ... 27
4.2.1 Amplifikasi dan Pemotongan dengan Enzim Restriksi ... 27
4.2.2 Keragaman Haplotipe (Haplotype Diversity) ... 30
4.2.3 Jarak Genetik ... 33
4.3 Struktur Populasi ... 36
5 KESIMPULAN DAN SARAN ... 42
5.1 Kesimpulan ... 42
5.2 Saran ... 42
DAFTAR PUSTAKA ... 44
xiii
DAFTAR TABEL
Halaman
1 Kondisi perikanan tuna periode 1995-2005: berat, hook rate dan CPUE
hasil tangkapan tuna didaratkan PT. Perikanan Samodra Besar Benoa Bali . 2
2 Estimasi hasil tangkapan tuna yang didaratkan di Benoa tahun 2000-2003.. 10
3 Produksi tuna segar dan beku kapal tuna longline di Benoa ... 10
4 Jumlah dan nilai ekspor tuna dari Benoa tahun 2000-2005 ... 11
5 Jumlah kapal tuna longline di Indonesia pada tahun 2000-2003 ... 11
6 Jumlah kapal tuna longline di Benoa tahun 1997-2005 ... 12
7 Daerah penangkapan tuna longline di perairan Indonesia ... 13
8 Komposisi sampel ikan tuna mata besar pada setiap kelompok sampel ... 20
9 Posisi dan ukuran panjang kelompok sampel ikan tuna mata besar ... 25
10 Distribusi tipe restriksi pada lima kelompok sampel ikan tuna mata besar . 29 11 Distribusi frekuensi menurut komposit haplotipe pada lima kelompok sampel ikan tuna mata besar ... 31
12 Keragaman lima kelompok sampel ikan tuna mata besar berdasarkan metode jarak berpasangan (Fst) ... 34
DAFTAR GAMBAR
Halaman
1 Kerangka pemikiran penelitian genetika populasi ikan tuna mata besar ... 6
2 Ikan tuna mata besar (Thunnus obesus) ... 8
3 Peta penyebaran ikan tuna mata besar di dunia ... 9
4 Lokasi pengambilan sampel ikan tuna mata besar ... 20
5 Pemotongan sirip ekor untuk sampel genetik ... 22
6 Sebaran ukuran panjang ikan sampel tuna mata besar ... 26
7 Fragmen tunggal mtDNA hasil amplifikasi PCR ikan tuna mata besar ... 27
8 Tipe restriksi dengan enzim Taq I: A B C D ... 28
9 Tipe restriksi dengan enzim Hin6 I: A B C D ... 28
10 Tipe restriksi dengan enzim Afa I: A B C D E ... 28
11 Tipe restriksi dengan enzim Mbo I: A B C D E ... 29
12 Dendrogram hubungan kekerabatan (filogeni) lima kelompok sampel ikan tuna mata besar ... 35
xv
DAFTAR LAMPIRAN
Halaman
1 Analisis deskriptif pada lima kelompok sampel ikan tuna mata besar ... 51
2 Analisis jarak genetik pada lima kelompok sampel ikan tuna mata besar ... 56
3 Analisis keragaman lima kelompok sampel ikan tuna mata besar ... 57
DAFTAR ISTILAH
: Merupakan satu jenis penanda molekular yang banyak dipakai dalam penelitian dan diagnostik biologi molekular.
Asam deoksiribonukleat
atau Deoxyribonucleic Acid (DNA)
: Sejenis asam nukleat yang tergolong biomolekul utama penyusun berat kering setiap organisme.
Daerah penangkapan atau
fishing ground
: Suatu daerah perairan tempat ikan berkumpul dimana penangkapan ikan telah dilakukan.
Elektroforesis : Merupakan suatu teknik pemisahan senyawa yang bermuatan dengan meletakkannya pada suatu medan listrik.
Endonuklease : Enzim yang memotong ikatan fosfodiester di dalam molekul asam nukleat.
Enzim : Suatu protein pengkatalis reaksi kimia tertentu.
Enzim restriksi : Enzim yang dihasilkan oleh bakteri pembelah molekul DNA asing pada situs rekognisi oligonukleotida tertentu. Enzim restriksi digunakan secara luas dalam teknologi DNA rekombinan.
Fenotif atau fenotipe : Sifat yang tampak dalam makhluk hidup.
Fitness : Ukuran kemampuan makhluk hidup untuk
bertahan terhadap kondisi lingkungan. Semakin besar fitness semakin mampu bertahan. Fitness
dalam genetik dinyatakan dalam keragaman genetik.
Genetika : Ilmu yang mempelajari sifat-sifat keturunan (hereditas) serta segala seluk beluknya secara ilmiah.
xvii
Hasil tangkapan per satuan unit upaya (catch per unit effort-CPUE)
: Jumlah hasil tangkapan yang diambil per unit alat tangkap.
Jumlah tangkapan yang
diperbolehkan
: Jumlah maksimum sumber daya ikan yang boleh ditangkap di wilayah pengelolaan perikanan Republik Indonesia dengan memperhatikan kelestarian sumber daya ikan.
Keragaman genetik : Merupakan variasi genetik di dalam setiap spesies yang mencakup aspek biokimia, struktur, dan sifat organisme yang diturunkan secara fisik dari induknya dan dibentuk dari DNA.
Komisi Tuna Samudera
Hindia atau Indian Ocean Tuna Commission (IOTC)
: Merupakan salah satu organisasi perikanan regional di bawah naungan FAO PBB dengan area kewenangan mencakup keseluruhan Samudera Hindia dan bagian utara Laut Antartika.
Mitokondria DNA (mtDNA)
: Merupakan rantai DNA yang terletak di bagian sel yang bernama mitokondria.
Monomorfik : Keadaan suatu lokus dengan alel yang paling umum dijumpai mencapai frekuensi lebih dari 0,95 sehingga lokus tersebut seakan-akan hanya mempunyai sebuah alel.
Pasangan basa atau base
pairs (bp)
: Dua nukleotida dalam RNA atau DNA yang saling komplementer yang terhubung oleh ikatan hidrogen.
Pengelolaan perikanan : Semua upaya, termasuk proses yang terintegrasi dalam pengumpulan informasi, analisis, perencanaan, konsultasi, pembuatan keputusan, alokasi sumber daya ikan, dan implementasi serta penegakan hukum dari peraturan perundang-undangan di bidang perikanan, yang dilakukan oleh pemerintah atau otoritas lain yang diarahkan untuk mencapai kelangsungan produktivitas sumber daya hayati perairan dan tujuan yang telah disepakati.
Polimorfisme Panjang Berkas Restriksi atau
Restriction Fragment Length Polymorphism
(RFLP)
: Merupakan penanda molekul yang pertama kali ditemukan dan digunakan. Penggunaannya dimungkinkan semenjak orang menemukan enzim endonuklease restriksi (RE), suatu kelas enzim yang mampu mengenal dan memotong seurutan pendek basa DNA (biasanya 4-6 urutan basa).
Populasi : Sekumpulan makhluk hidup sejenis yang
menempati tempat yang sama dalam waktu yang sama.
Rawai tuna atau tuna
longline
: Rawai yang khusus untuk menangkap ikan-ikan tuna. Alat tangkap ini merupakan pengembangan teknik pada perikanan pancing (line fishing) dimana satu unitnya terdiri atas pelampung (float) dan tali pelampung (float line), tali utama (main line) dengan sejumlah tali cabang yang berpancing (branch line).
: Merupakan suatu teknik atau metode perbanyakan (replikasi) DNA secara enzimatik tanpa menggunakan organisme.
Struktur populasi : Pengelompokkan hewan dalam sebuah populasi yang didasarkan atas jenis kelamin, umur dan lain-lain.
Wilayah Pengelolaan
Perikanan Republik Indonesia (WPP-RI)
: Perairan Indonesia, Zona Ekonomi Eksklusif Indonesia, dan sungai, danau, waduk, rawa, dan genangan air lainnya yang dapat diusahakan serta lahan pembudidayaan ikan yang potensial di wilayah RI.
Zona ekonomi eksklusif
Indonesia (ZEEI)
1
PENDAHULUAN
1.1 Latar Belakang
Tuna mata besar (Thunnus obesus) atau lebih dikenal dengan bigeye tuna
adalah salah satu anggota Famili Scombridae dan merupakan salah satu komoditi ekspor perikanan tuna yang paling utama di Indonesia selain tuna sirip kuning atau madidihang (Thunnus albacares) dan tuna sirip biru selatan (Thunnus maccoyii). Ekspor tuna mata besar pada umumnya dalam bentuk segar dan beku. Berdasarkan data Dinas Perikanan Provinsi Bali (2002) dan Pelabuhan Perikanan Samudera Jakarta (PPSJ) Muara Baru (2002) diacu dalam Proctor et al. (2003) dilaporkan bahwa pada tahun 2002 tuna yang diekspor dalam bentuk segar dan beku sekitar 18.011,5 ton dari Bali dan 17.471 ton dari Muara Baru dengan negara tujuan Jepang, Amerika, Inggris dan lain-lain.
Pemanfaatan sumber daya tuna, terutama tuna mata besar di perairan Samudera Hindia dari tahun ke tahun cenderung terus meningkat. Hal ini terindikasi dengan semakin bertambahnya jumlah armada yang beroperasi di wilayah perairan tersebut. Direktorat Jenderal Perikanan Tangkap (2005) mencatat bahwa ada sekitar 6.547 unit kapal tuna longline di Indonesia pada tahun 2003. Jumlah tersebut meningkat dibandingkan pada tahun 2002 yang hanya berjumlah 2.264 unit. Diduga jumlah kapal tuna longline yang beroperasi di seluruh perairan Indonesia sekitar 1.400 unit, dimana kira-kira 1.200 beroperasi di Samudera Hindia (Pusat Riset Perikanan Tangkap 2002).
Seiring dengan meningkatnya permintaan pasar dari tahun ke tahun, maka semakin tinggi pula eksploitasi terhadap jenis tuna mata besar, sehingga di perairan Samudera Hindia, utamanya di daerah-daerah penangkapan ikan armada
tangkapan yang menguntungkan (sustainable yield) tetapi kelestarian sumber daya (spawning stock) tetap terjaga. Konsep ini akan terlaksana efektif apabila tersedia data tentang kondisi populasi tuna mata besar dengan definisi yang jelas dan akurat.
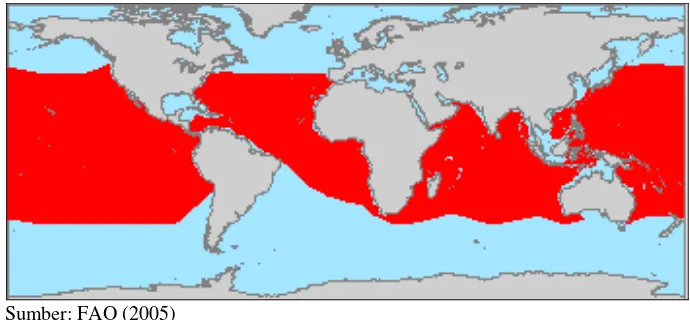
Tabel 1 Kondisi perikanan tuna periode 1995-2005: berat, hook rate dan CPUE hasil tangkapan tuna didaratkan PT. PSB Benoa Bali
Tahun Berat ikan rata-rata (kg) Hook rate CPUE (kg/trip)
1995 32 0,86 280
1996 30 0,84 291
1997 27,5 0,91 282
1998 27 0,87 266
1999 26 0,62 189
2000 27 0,60 177
2001 24 0,72 210
2002 25 0,72 234
2003 30 0,52 199
2004 27 0,48 171
2005 29 0,45 172
Sumber: PT. PSB (2006) diacu dalam Kosasih (2007)
Salah satu metode yang dapat diterapkan untuk menentukan kondisi populasi ikan dan struktur genetiknya dengan tingkat akurasi yang tinggi adalah didasarkan pada polimorfisme DNA; dan DNA mitokondria (mtDNA) yang dipercaya sangat relevan bagi studi tersebut. Informasi lain yang dapat diketahui adalah keragaman genetik, perubahan molekuler dalam genom (mutasi) serta hubungan filogenetik di antara populasi.
3
lain, (1) menduga hubungan kekerabatan, (2) menduga variasi genetik, (3) monitoring kemurnian benih hibrida, dan (4) memilah komponen genetik dari karakter kuantitatif (Sianipar 2003). RAPD merupakan satu jenis penanda molekular yang banyak dipakai dalam penelitian dan diagnostik biologi molekular.
Genetika populasi adalah cabang genetika yang membahas transmisi bahan genetik pada ranah populasi. Dari objek bahasannya, genetika populasi dapat dikelompokkan sebagai cabang genetika yang berfokus pada pewarisan genetik (Anonim 2008a). Pola pewarisan suatu sifat tidak selalu dapat dipelajari melalui percobaan persilangan buatan. Pada tanaman keras atau hewan-hewan dengan daur hidup panjang seperti gajah, misalnya, suatu persilangan baru akan memberikan hasil yang dapat dianalisis setelah kurun waktu yang sangat lama. Pola pewarisan sifat pada organisme-organisme yang memiliki daur hidup panjang harus dianalisis menggunakan data hasil pengamatan langsung pada populasi yang ada (Anonim 2008b). Genetika populasi ikan tuna dengan cakupan geografis dari seluruh laut Indonesia mempunyai struktur yang berbeda-beda sebagai akibat adanya keterbatasan laju migrasi gen (Permana et al. 2007).
Terpilihnya Benoa sebagai daerah penelitian karena Benoa merupakan salah satu pelabuhan perikanan di Bali dimana produksi perikanan tunanya cukup besar dibandingkan Pelabuhan Perikanan Samudera Jakarta (PPSJ) Muara Baru dan Pelabuhan Perikanan Samudera Cilacap (PPSC). Total hasil tangkapan tuna yang didaratkan di Pelabuhan Benoa sebanyak 26.748 ton pada tahun 2002 (Proctor et al. 2003). Produksi ini lebih banyak dibandingkan dengan PPSJ Muara Baru yang hanya sebesar 17.471 ton (PPSJ Muara Baru 2002, diacu dalam Proctor et al. 2003) dan PPSC yang hanya 1.489 ton (PPSC 2002, diacu dalam Proctor et al. 2003). Pelabuhan Benoa berkembang menjadi pusat perikanan tuna disebabkan oleh dekatnya pelabuhan ini dengan daerah penangkapan (fishing ground) yang berada di Samudera Hindia dan dekat dengan bandar udara sehingga memudahkan dalam melakukan ekspor ikan tuna, terutama tuna segar (fresh tuna).
(Nugroho et al. 2006), ikan kakap merah (Lutjanus malabaricus) yang berasal dari beberapa daerah penangkapan di Pantai Utara Jawa dan Laut Jawa bagian timur (Suwarso 2002) dan tuna sirip kuning dari daerah Bali, Maluku Utara dan Sulawesi Utara (Permana et al. 2007) serta ikan lele (Famili Claridaee) dari wilayah Asia Tenggara (Sudarto 2003). Penelitian genetika populasi ikan tuna mata besar yang berasal dari perairan Samudera Hindia telah dilakukan oleh Appleyard et al. (2002) dan Chiang et al. (2008), namun belum pernah dilakukan oleh peneliti Indonesia terutama dari perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara yang merupakan perairan daerah-daerah penangkapan ikan armada tuna longline PT. PSB belum pernah dilakukan.
Hal-hal di atas, kiranya yang mendasari perlunya diadakan penelitian mengenai genetika populasi ikan tuna mata besar hasil tangkapan tuna longline
yang didaratkan di Benoa.
1.2 Perumusan Masalah
Klarifikasi tentang struktur populasi di alam merupakan informasi yang penting untuk pengelolaan pendugaan populasi. Pengumpulan informasi atau data dasar genetik dari suatu spesies merupakan syarat awal yang diperlukan untuk menentukan keragaman (variasi) genetik atau kekerabatan yang dimiliki. Dengan diketahuinya keragaman genetik masing-masing spesies, akan sangat membantu baik untuk membuat suatu kebijakan dalam pengelolaan maupun konservasi dari sumber-sumber genetik di alam, termasuk ikan tuna.
Keragaman genetik merupakan suatu informasi penting untuk evaluasi, dalam jangka pendek, mengenai fitness individu maupun untuk jangka panjang, kelangsungan hidup dari suatu populasi. Terdapat pendapat yang beranggapan bahwa penyebaran ikan tuna mata besar sangat luas dengan kemampuan migrasi yang tinggi sehingga komoditas ini dianggap sebagai milik umum.
5
1.3 Tujuan Penelitian
Penelitian ini bertujuan untuk mendapatkan informasi keragaman genetik dan struktur populasi ikan tuna mata besar dari perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara.
1.4 Manfaat Penelitian
Manfaat penelitian ini adalah memberikan informasi mengenai keragaman genetik dan struktur populasi serta terbentuknya data dasar genetik ikan tuna mata besar dari perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara sehingga dapat dipergunakan sebagai bahan kajian untuk pengelolaannya.
1.5 Hipotesis
Hipotesis yang diajukan adalah terdapat perbedaan secara genotip ikan tuna mata besar hasil tangkapan tuna longline dari perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara.
1.6 Kerangka Pemikiran
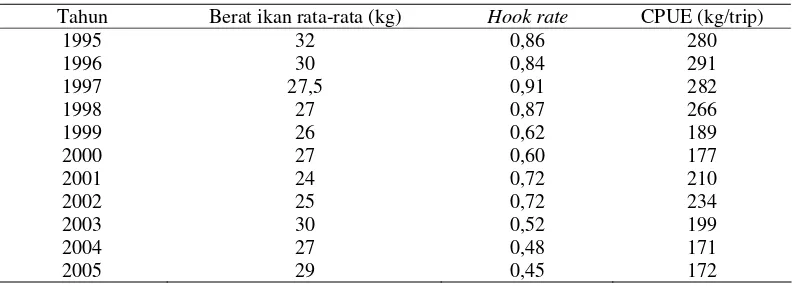
Permasalahan yang ada dalam pengelolaan sumber daya ikan tuna mata besar di Indonesia, terutama di Samudera Hindia adalah belum diketahuinya kondisi populasi spesies tersebut dan belum terkumpulnya informasi atau data dasar genetik dari spesies tersebut, sehingga tidak tersedianya informasi mengenai keragaman genetik dan struktur populasi dari spesies di lokasi tersebut. Kondisi populasi dan data dasar genetik ikan tuna mata besar dapat diprediksi melalui pendekatan dengan menggunakan analisis DNA. Analisis DNA dapat dilakukan dengan menggunakan metode Restriction Fragment Length Polymorphism
Gambar 1 Kerangka pemikiran penelitian genetika populasi ikan tuna mata besar. Data dasar genetik Permasalahan spesies ikan tuna mata besar
Metode RFLP DNA mitokondria
Pengelolaan perikanan tuna mata besar Keragaman genetik
Kondisi populasi
Analisis DNA
2
TINJAUAN PUSTAKA
2.1 Biologi Ikan Tuna Mata Besar
Menurut Saanin (1986), klasifikasi ikan tuna mata besar adalah sebagai berikut :
dunia : Animalia sub dunia : Metazoa filum : Chordata sub filum : Vertebrata kelas : Pisces sub kelas : Teleostei ordo : Percomorphi sub ordo : Scombroidae
famili : Scombridae genus : Thunnus
species : Thunnus obesus
Sumber: FAO (2005)
Gambar 2 Ikan tuna mata besar (Thunnus obesus).
Menurut Fukofuka dan Itano (2006), ikan tuna mata besar mempunyai ciri-ciri luar sebagai berikut :
• Sirip ekor mempunyai lekukan yang dangkal pada pusat celah sirip ekor; • Pada ikan dewasa matanya relatif besar dibandingkan dengan tuna-tuna yang
lain;
• Profil badan seluruh bagian dorsal dan ventral melengkung secara merata; • Sirip dada pada ikan dewasa, 1/4-1/3 kali fork length (FL);
• Sirip dada pada anak ikan tuna (yuwana) lebih panjang dan selalu melewati belakang sebuah garis yang digambar di antara tepi-tepi anterior sirip punggung kedua dan sirip anal;
• Ikan-ikan tuna mata besar dengan ukuran <75 cm (10 kg) mempunyai sirip dada yang lebih panjang dari pada ikan tuna sirip kuning dari ukuran-ukuran yang sebanding;
• Ikan-ikan yuwana sering mempunyai 7-10 strip-strip yang berwarna putih dan tidak terputus-putus, menyilang tegak lurus pada sisi-sisi bagian bawah, jauh lebih sedikit dibandingkan dengan ikan tuna sirip kuning.
9
Papua. Semua jenis tuna terdapat di Indonesia kecuali tuna sirip biru utara dan tuna sirip hitam, karena tuna sirip biru utara menghuni Samudera Pasifik dan Atlantik, sedangkan tuna sirip hitam hanya terdapat di Samudera Atlantik (Uktolseja 1988).
Sumber: FAO (2005)
Gambar 3 Peta penyebaran ikan tuna mata besar di dunia.
2.2 Hasil Tangkapan Ikan Tuna Mata Besar Didaratkan di Benoa
Pengertian hasil tangkapan didaratkan adalah jumlah ikan dari satu atau lebih spesies ataupun hewn air lainnya yang tertangkap oleh suatu kegiatan operasi penangkapan dan didaratkan di suatu tempat pendaratan yang merupakan
fishing base suatu perusahaan penangkapan atau di suatu pelabuhan perikanan. Hasil tangkapan terbagi menjadi 2, yaitu hasil tangkapan utama (HTU) dan hasil tangkapan sampingan (HTS). Hasil tangkapan sampingan (HTS) dapat diartikan sebagai hasil tangkapan yang tertangkap selain hasil tangkapan utama dan bukan merupakan target spesies. Beverly et al. (2003) menyatakan bahwa HTS adalah hasil tangkapan yang tidak diinginkan namun tertangkap secara kebetulan selama operasi penangkapan dengan tuna longline. Penanganan HTS terbagi 2, yaitu disimpan karena memiliki nilai ekonomis tinggi (by-product) dan dibuang karena tidak memiliki nilai ekonomis (discard).
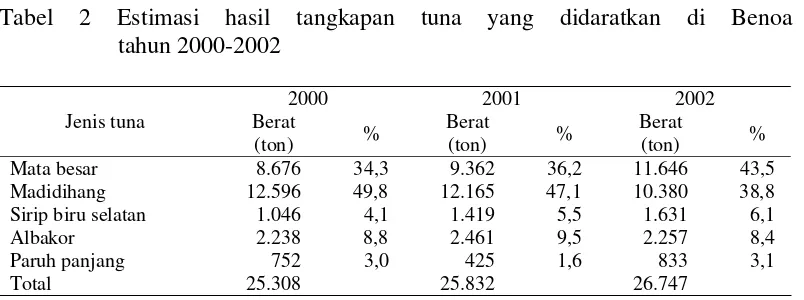
Indonesia adalah salah satu negara perikanan tuna yang penting di Samudera Hindia. Potensi sumberdaya tuna di wilayah Samudera Hindia termasuk ZEEI 200 mil sebesar 216.275 ton/tahun atau 39,4% dari total potensi tuna Indonesia yaitu sebesar 548.387 ton/tahun (Uktolseja et al. 1997). Komisi Tuna Samudera Hindia (Indian Ocean Tuna Commission, IOTC) memperkirakan hasil tangkapan tuna dan sejenisnya (tuna like species) dari Indonesia di Samudera Hindia sebesar 177.384 ton pada tahun 2000 (Herrera 2002, diacu dalam Proctor et al. 2003). Hasil tangkapan tuna mata besar yang didaratkan di Benoa hingga tahun 2002 diperkirakan mencapai 11.646 ton atau 43,5% dari total hasil tangkapan tuna yaitu sebesar 26.747 ton (Tabel 2) (Proctor et al. 2003).
Tabel 2 Estimasi hasil tangkapan tuna yang didaratkan di Benoa tahun 2000-2002 Madidihang 12.596 49,8 12.165 47,1 10.380 38,8 Sirip biru selatan 1.046 4,1 1.419 5,5 1.631 6,1 Albakor 2.238 8,8 2.461 9,5 2.257 8,4 Paruh panjang 752 3,0 425 1,6 833 3,1
Total 25.308 25.832 26.747
Sumber: Proctor et al. (2003)
Sebagian besar ikan tuna, termasuk ikan tuna mata besar hasil tangkapan
tuna longline yang berbasis di Benoa dijual dalam bentuk segar dan beku. Produksi tuna segar dan beku pada tahun 2005 sebesar 13.184 ton. Produksi ini mengalami penurunan dibandingkan pada tahun 2000 yaitu sebesar 15.700 ton (Tabel 3) (Asosiasi Tuna Longline Indonesia, ATLI 2006, diacu dalam Kosasih 2007).
Tabel 3 Produksi tuna segar dan beku kapal tuna longline di Benoa
Tahun Segar (ton) Beku (ton) Total (ton)
11
ATLI (2006) diacu dalam Kosasih (2007) melaporkan bahwa ekspor tuna dari Benoa semenjak tahun 2000-2005 mengalami penurunan. Penurunan yang sangat signifikan terjadi pada tahun 2005 dimana hanya 9.776 ton tuna yang diekspor dibandingkan pada tahun 2000 yang berjumlah 18.758 ton (Tabel 4).
Tabel 4 Jumlah dan nilai ekspor tuna dari Benoa tahun 2000-2005
Tahun Jumlah ekspor (ton) Nilai ekspor (US$)
2000 18.758 82.444.848
2001 17.590 72.595.501
2002 17.847 73.656.163
2003 17.545 72.838.508
2004 12.460 58.896.455
2005 9.776 41.135.741 Sumber: ATLI (2006) diacu dalam Kosasih (2007)
Berdasarkan data statistik perikanan tangkap tahun 2005 jumlah kapal tuna longline pada tahun 2003 berjumlah 6.547 unit, sedangkan pada tahun 2002 hanya berjumlah 2.264 unit atau mengalami kenaikan sebesar 189,18%. Jumlah kapal
tuna longline di Benoa sejak tahun 1997-2004 terus mengalami kenaikan. Pada tahun 1997 jumlah kapal tuna longline di Benoa sebanyak 459 unit, bertambah jumlahnya pada tahun 2004 hingga mencapai 700 unit. Pada tahun 2005 jumlah kapal tuna longline menurun seiring dengan meningkatnya harga bahan bakar minyak (BBM) (Tabel 5) (ATLI 2006, diacu dalam Kosasih 2007).
Tabel 5 Jumlah kapal tuna longline di Benoa tahun 1997-2005
Tahun Jumlah kapal (unit)
1997 459 1998 460 1999 485 2000 537 2001 562 2002 705 2003 701 2004 700 2005 668 Sumber: ATLI (2006) diacu dalam Kosasih (2007)
dibandingkan kapal yang mempunyai ukuran <60 GT (Tabel 6) (ATLI 2006,
diacu dalam Kosasih 2007).
Tabel 6 Ukuran kapal tuna longline dan jenis lainnya di Benoa tahun 2005
Ukuran kapal Tuna longline Alat tangkap lain Total
0-30 GT 172 87 259
31-60 GT 117 17 134
>60 GT 379 40 419
Jumlah 668 144 812
Sumber: ATLI (2006) diacu dalam Kosasih (2007)
Beberapa daerah di Indonesia yang merupakan daerah penangkapan ikan tuna antara lain adalah Laut Banda, Laut Maluku dan perairan selatan Jawa terus menuju timur. Begitu pula di perairan selatan dan barat Sumatera serta perairan lainnya (Gunarso 1998).
Menurut Uktolseja et al. (1991) tuna hampir didapatkan menyebar di seluruh perairan Indonesia. Di Indonesia bagian barat meliputi Samudera Hindia, sepanjang pantai utara dan timur Aceh, pantai barat Sumatera, selatan Jawa, Bali dan Nusa Tenggara. Di perairan Indonesia bagian timur meliputi Laut Banda, Laut Flores, Laut Halmahera, Laut Maluku, Laut Sulawesi, perairan Pasifik di sebelah utara Irian Jaya dan Selat Makasar.
Menurut Wudianto et al. (2003) daerah penangkapan kapal tuna longline
yang berasal dari Cilacap dan Benoa yaitu di perairan selatan Jawa Tengah antara 108-118o BT dan 8-22o LS dimana sebagian besar (>70%) melakukan penangkapan di luar perairan ZEEI. Penangkapan di luar perairan ZEEI Samudera Hindia diduga karena kelimpahan tuna di perairan ZEEI semakin sedikit. Hal ini terlihat dari laju pancing (hook rate) di perairan ZEEI makin kecil dan berat ikan yang tertangkap juga makin kecil. Pada awal-awal perkembangan tuna longline
(1970-an), laju pancing berkisar antara 1,15-2,16. Pada tahun 1999 turun mencapai 0,67. Begitu pula dengan berat ikan yang tertangkap terus turun dari 37 kg/ekor pada tahun 1973 menjadi 26 kg/ekor pada tahun 1999 (Pusat Riset Perikanan Tangkap 2002).
13
Tabel 7 Daerah penangkapan tuna longline di perairan Indonesia
Bulan Daerah penangkapan
Januari barat Sumatera, selatan NTB/NTT, Laut Flores, Laut Banda bagian timur Pebruari barat Sumatera, selatan Jawa/Bali/NTB/NTT dan relatif kecil di Laut Banda Maret barat Sumatera, selatan Jawa/Bali/NTB/NTT dan relatif kecil di Laut Banda April barat Sumatera, selatan Jawa/Bali/NTB/NTT, Laut Flores dan di Laut Banda
cukup padat
Mei terutama di selatan Jawa/Bali/NTB dan Laut Banda Juni selatan Jawa/Bali/NTB dan Laut Banda
Juli selatan Jawa/Bali, lepas pantai NTB/NTT dan sedikit di Laut Banda Agustus sedikit di barat Sumatera dan selatan Jawa dan Laut Banda
September barat Sumatera, selatan Jawa, lepas pantai NTB dan Laut Banda.cukup padat Oktober selatan Jawa/Bali/NTB/NTT dan Laut Banda
November barat Sumatera, sedikit di selatan Jawa/NTB, Laut Flores dan Laut Banda Desember selatan NTB/NTT dan Laut Banda serta Laut Flores
2.3 DNA (Deoxyribonuleic Acid) Mitokondria
DNA mitokondria sangat potensial digunakan untuk pengamatan hubungan genetik antar spesies maupun di dalam spesies (intra spesies yang memiliki hubungan dekat). Brown (1983) diacu dalam Arifin (2005) menyatakan bahwa peranan DNA mitokondria dalam studi keragaman genetik dan biologi populasi pada hewan cukup besar, karena DNA mitokondria memiliki derajat polimorfisme yang tinggi serta hubungan yang jelas antara poplimorfisme dengan substansi basa-basa penyusun genomnya.
DNA mitokondria, berbeda dengan organel sel lainnya, mitokondria memiliki materi genetik sendiri yang karakteristiknya berbeda dengan materi genetik di inti sel. Mitokondria, sesuai dengan namanya, merupakan rantai DNA yang terletak di bagian sel yang bernama mitokondria. DNA mitokondria memiliki ciri-ciri yang berbeda dari DNA nukleus ditinjau dari ukuran, jumlah gen, dan bentuk. Di antaranya adalah memiliki laju mutasi yang lebih tinggi, yaitu sekitar 10-17 kali DNA inti. Selain itu DNA mitokondria terdapat dalam jumlah banyak (lebih dari 1.000 kopi) dalam tiap sel, sedangkan DNA inti hanya berjumlah dua kopi. DNA inti merupakan hasil rekombinasi DNA kedua orang tua sementara DNA mitokondria hanya diwariskan dari ibu (maternally inherited) (Anonim 2008c).
memberikan hasil yang lebih kuat dibandingkan dengan analisis protein karena analisis DNA mitokondria dapat mendeteksi semua keragaman genetik yang bernilai bagi pemahaman identifikasi stok (Ferris dan Berg 1987, diacu dalam
Rina 2001). Hal ini dapat dilihat dari perbandingan hasil-hasil penelitian dengan menggunakan DNA mitokondria dan analisis protein pada ikan-ikan American oyster (Crassostrera virginica), kepiting (Limulus polyphemus), ikan teleostei laut (Fundulus heteroclitus dan Theragra chalcogramma) ikan teleostei air tawar (Stizestedion vitreum) serta ikan anadromus American shad (Alosa sapidissima) yang menyimpulkan bahwa sub divisi populasi lebih besar terlihat pada analisis dengan DNA mitokondria dibandingkan dengan protein (Ward dan Grewe 1995,
diacu dalam Rina 2001).
Daerah D-loop atau dikenal juga dengan nama daerah kontrol (control region) merupakan bagian dari DNA mitokondria yang sangat spesifik. Analisis DNA mitokondria pada D-loop telah digunakan untuk menduga keragaman genetik dan struktur populasi ikan japanese flounder (Paralichthys alovaceus) oleh Fujii dan Nishida (1997).
2.4 RFLP (Restriction Fragment Length Polymorphism)
RFLP adalah suatu metode untuk melihat perbedaan profil dan panjang DNA yang dipotong dengan enzim restriksi endonuklease yang sama tetapi pada individu yang berbeda dalam suatu populasi (Stansfield 1991, diacu dalam Ayu 2005). Metode RFLP digunakan untuk mengetahui polimorfisme DNA pada wilayah tertentu dengan melihat tipe atau pola pemotongan DNA dengan menggunakan bantuan enzim restriksi (Sianipar 2003).
15
Suatu fragmen restriksi yang polimorfik (RFLP) akan muncul apabila DNA dari individu yang berbeda dalam populasi memberikan profil fragmen yang berbeda saat DNA-nya terpotong menggunakan enzim restriksi endonuklease yang sama (Stansfield 1991, diacu dalam Sunandar 2008). RFLP telah digunakan pada genom mitokondria di beberapa situs restriksi antara lain 16S rRNA (ribosomal ribonucleic acid) dan CO (cytochrome oxidase)-I (Mathews et al. 2002, diacu dalam Sunandar 2008) dan area D-loop (Nugroho et al. 2002).
Penelitian Bouchon et al. (1994) diacu dalam Rina (2001) memperlihatkan bahwa analisis mtDNA dengan RFLP telah dapat memperlihatkan variasi DNA pada dua spesies udang penaeid (Penaeus monodon Fab dan P. japonicus Bate). Selain itu, Tabata dan Mizuta (1997) diacu dalam Rina (2001) melakukan penelitian untuk mendapatkan hasil yang lebih rinci pada populasi ikan red sea bream (Pagrus major) dengan analisis RFLP mtDNA pada daerah D-loop.
2.5 Polymerase Chain Reaction (PCR)
PCR atau dalam Bahasa Indonesia dikenal dengan istilah reaksi berantai polimerase merupakan suatu teknik atau metode perbanyakan (replikasi) DNA secara enzimatik tanpa menggunakan organisme. Dengan teknik ini, orang dapat menghasilkan DNA dalam jumlah besar dalam waktu singkat sehingga memudahkan berbagai teknik lain yang menggunakan DNA (Anonim 2009a). Erlich (1989) diacu dalam Rina (2001) menyatakan bahwa PCR adalah sebuah metode in vitro yang digunakan untuk mensintesa sekuen DNA tertentu secara enzimatis dengan menggunakan dua primer oligonukleotida yang menghibridasi pita yang berlawanan dan mengapit daerah target DNA.
dengan satu molekul DNA, dapat diperbanyak dua kali lipat DNA dalam satu siklus suhu denaturation, annealing dan extension.
Sekuens primer DNA merupakan faktor kunci yang menentukan berhasil atau tidaknya suatu PCR, karena primer ini sebagai awal dimulainya proses amplifikasi DNA. Jika primer langsung menempel pada susunan basa nukleotida pada sekuens DNA, maka proses berikutnya akan mudah bekerja dengan baik. umumnya suatu primer DNA memiliki panjang antara 20-24 pasangan basa (basepairs), baik primer yang berukuran panjang atau pendek keduanya umum digunakan dalam proses PCR (Baker dan Birt 2000, diacu dalam Sunandar 2008). Primer DNA yang digunakan sebaiknya memiliki kesamaan sekuens atau spesifik dengan target template DNA
2.6 Keragaman Genetik
Keragaman genetik merupakan hirarki yang paling rendah dalam tingkatan keragaman hayati. Keragaman hayati mencakup area yang meliputi keragaman habitat, komunitas, populasi sampai dengan spesies. Keragaman genetik merupakan cerminan keragaman di dalam spesies yang secara umum disebut subspesies. Terminologi sumber daya genetik diartikan untuk merefleksikan adanya keragaman genetik di dalam satu spesies sampai pada tingkat DNA (Soewardi 2007).
Lebih lanjut Soewardi (2007) menyebutkan bahwa keragaman genetik merupakan bagian dari keragaman hayati (biodiversity) yang memiliki pengertian yang lebih luas, yakni keragaman struktural dan fungsional dari kehidupan pada tingkat komunitas dan ekosistem, populasi, spesies dan genetik. Oleh karena itu dalam rangka mempertahankan keragaman hayati, sumberdaya genetik memiliki peran penting karena semakin beragam sumberdaya genetik, akan semakin tahan populasi tersebut untuk hidup dalam jangka yang lama serta semakin tinggi daya adaptasi terhadap perubahan lingkungan semakin besar. Disamping itu, keragaman genetik juga merupakan kunci penting dalam memelihara keberlanjutan dan meningkatkan produktivitas dari suatu spesies.
17
terpisah jauh secara geografis atau pengaruh tingkah laku seperti migrasi dan waktu memijah. Secara umum keragaman genetik suatu populasi akan mempengaruhi respon populasi terhadap seleksi alam dan seleksi buatan yang dilakukan oleh manusia untuk memenuhi kebutuhannya. Populasi dengan keragaman genetik yang tinggi memiliki peluang hidup yang lebih baik. Hal ini dikarenakan setiap gen memiliki respon yang berbeda-beda terhadap kondisi lingkungan, sehingga dengan dimilikinya berbagai macam gen dari individu-individu di dalam populasi maka berbagai perubahan lingkungan yang ada akan dapat direspons lebih baik. Beberapa studi menunjukkan bahwa karakteristik genetik suatu populasi ikan di alam pada umumnya menunjukkan adanya heterogenitas spasial, bahkan pada jarak yang sangat dekat (Ryman dan Utter 1987).
Pembentukan struktur genetika populasi suatu jenis ikan dipengaruhi berbagai faktor. Faktor-faktor tersebut dapat menambah maupun mengurangi keragaman genetik. Faktor-faktor yang menyebabkan penambahan gen atau meningkatkan keragaman genetik antara lain faktor mutasi dan imigrasi, sedangkan faktor-faktor yang menurunkan keragaman genetik antara lain seleksi alami dan penghanyutan genetik (genetic drift) (Gardner et al. 1991, diacu dalam
Soewardi 2007). Menurut Soelistyawati (1996), keragaman genetik juga dipengaruhi oleh perpindahan materi genetik antar dua populasi yang berbeda tempat.
Ada beberapa metode untuk mengukur keragaman genetik di dalam suatu atau antar populasi. Menurut Chambers dan Bayless (1983) diacu dalam Imron (1998), ada tujuh cara yaitu pengukuran asam inti, sekuensing protein, elektroforesis, imunologi, kromosom, hubungan antar lokus, morfometrik dan studi breeding; sedangkan menurut Allendorf dan Phelp (1981) diacu dalam
adalah DNA mitokondria, DNA figerprinting, amplifikasi DNA dengan PCR dan sekuensing protein dan DNA mikrosatelit. Metode untuk mengukur keragaman genotip yang sekarang ini banyak digunakan oleh para ahli genetika salah satunya adalah DNA mitokondria. Pengukuran tingkat DNA ini mempunyai hasil yang lebih akurat (Ryman dan Utter 1987).
2.7 Pengukuran Jarak Genetik
Unit stok, sekelompok individu atau sub kelompok dari suatu spesies yang memiliki kesamaan dalam struktur atau pola genetik, dapat dipelajari berdasarkan frekuensi genetik dari setiap gen yang terlibat dalam ekspresi fenotipik. Pada tingkat molekular (DNA) ikan laut menunjukkan keragaman genetik walaupun dalam derajat yang lebih rendah dibanding ikan air tawar baik pada level supraspesifik maupun taksa kelompok individu (populasi dan subpopulasi) dimana pada tingkat protein (studi allozyme) tidak terlihat (Suwarso 2002).
Berdasarkan sifat polimorfisme DNA mitokondria, keragaman genetik populasi dapat diketahui oleh dua ukuran divergensi yaitu divergensi di dalam populasi (intrapopulasi) dan divergensi antar populasi (interpopulasi). Selanjutnya dikatakan oleh Hartl (1980) bahwa divergensi interpopulasi diperoleh berdasarkan parameter jarak genetik (genetic distance) dan analisis statistik (sampling varian) terhadap perbedaan situs restriksi. Jarak genetik merupakan ukuran perbedaan genetik antar populasi karena mutasi, seleksi, persilangan acak dan penghanyutan gen yang akan mengubah genetic make up populasi; dan migrasi akan menyebabkan terjadinya evolusi.
Keragaman intrapopulasi dinyatakan dengan parameter diversitas haplotipe atau diversitas nukleon (h), banyaknya nukleomorf (unit polimorfisme pada nukleon yang terdapat dalam bentuk pola situs restriksi), jumlah rata-rata perbedaan situs restriksi, jumlah segresi situs retriksi atau jumlah situs retriksi polimorfisme dalam sejumlah sampel nukleon. Nukleon merupakan suatu segmen DNA, identik dengan gen dalam DNA inti, yang dicirikan oleh peta situs restriksi atau jumlah dan ukuran frgamen DNA (Nei dan Tajima 1981).
19
akurat, selain itu telah melahirkan interes baru dalam pengujian tentang hipotesis mekanisme evolusi. Algoritma dasar dalam rekonstruksi pohon filogenetik didasarkan pada asumsi bahwa bila satu gen pada sepasang spesies atau populasi berevolusi secara clocklike fashion dengan derajat divergensi gen menyimpang selama t generasi, maka diperkirakan gen terpisah dari nenek moyang umumnya selama ½ generasi (Hartl dan Clark 1997, diacu dalam Suwarso 2002). Asumsi ini dipakai dalam menerapkan metode rekonstruksi pohon filogenetik yang didasarkan pada ukuran jarak genetik.
3.1 Waktu dan Tempat Penelitian
Pengambilan sampel ikan tuna mata besar dilakukan pada bulan Juli-September 2008 berlokasi di perairan Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara. Ikan tuna mata besar yang dijadikan sampel merupakan hasil tangkapan kapal tuna longline milik PT. Perikanan Samodra Besar.
Tiga ratus sampel ikan tuna mata besar diambil dari hasil tangkapan kapal
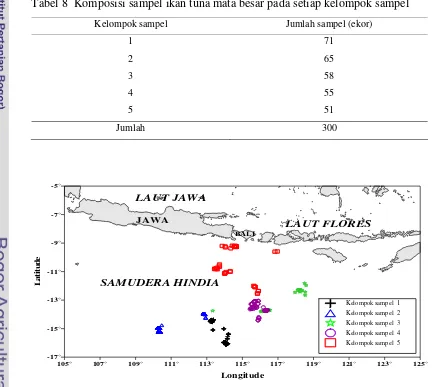
tuna longline yang beroperasi di Samudera Hindia selatan Jawa dan Nusa Tenggara (Tabel 8). Lokasi pengambilan sampel terletak pada koordinat 09o 11’-16o07’ LS dan 110o15’-118o35’ BT (Gambar 4).
Tabel 8 Komposisi sampel ikan tuna mata besar pada setiap kelompok sampel
Kelompok sampel Jumlah sampel (ekor)
1 71
105° 107° 109° 111° 113° 115° 117° 119° 121° 123° 125°
Longitude
Gambar 4 Lokasi pengambilan sampel ikan tuna mata besar.
21
Pelaksanaan analisis sampel dilaksanakan pada bulan Oktober-November 2008 di Laboratorium Genetika Balai Riset Perikanan Budidaya Air Tawar Bogor.
3.2 Bahan dan Alat
Bahan utama yang digunakan dalam penelitian adalah sampel potongan-potongan sirip ekor ikan tuna mata besar (Thunnus obesus).
Alat yang dipakai dalam pengambilan sampel sirip meliputi: meteran, timbangan, pisau, botol sampel dan alat-alat tulis. Bahan dan alat yang digunakan dalam penelitian sampel di laboratorium antara lain taq ready to go, agarose gel,
TBE buffer, 100 bp (base pairs) ladder marker, loading dye, tube 1.500 μl, tube
600 μl, tip 10 μl, tip 100 μl, tip 1.000 μl, larutan proteinase kinase, ethidium bromide, primer Pro-5 (CAC GAC GTT GTA AAA CGA CCT ACC YCY AAC TCC CAA AGC), dan primer 12SAR (GGA TAA CAA TTT CAC ACA GGG CAT AGT GGG GTA TCT AAT CC), serta enzim-enzim TaqI, Afa I, Mbo I dan
Hin6 I.
3.3 Metode Penelitian 3.3.1 Penentuan Sampel
Ikan uji atau sampel yang digunakan dalam penelitian ini adalah tuna mata besar yang tertangkap oleh kapal tuna longline dari Samudera Hindia sebelah selatan Jawa dan Nusa Tenggara dan didaratkan di Benoa, Bali. Sampel diambil dari 5 kapal contoh (tuna longline) yang diikuti. Ikan sampel yang diambil bagian sirip ekornya diukur panjang dan beratnya, kemudian dicatat lokasi tertangkapnya.
Gambar 5 Pemotongan sirip ekor untuk sampel genetik.
3.3.2 Ekstraksi DNA
DNA ikan diekstraksi dari potongan sirip dengan menggunakan metode sebagai berikut :
Sebanyak 5-10 mg potongan sirip ikan dimasukkan ke dalam mikrotube 1,5 ml yang telah berisi 500 μl larutan urea. Adapun komposisi larutan urea terdiri dari :
Urea : 48,048 gr
NaCl : 1,461 gr
1 molar tris (pH 7,5) : 2 ml
10% SDS : 10 ml
Bahan-bahan tersebut diencerkan dengan aquades sampai 200 ml, lalu disaring. Kemudian ke dalam mikrotube ditambahkan 10 μl proteinase kinase (20 mg/ml), diaduk dengan menggunakan vortex, lalu disimpan dalam inkubator
waterbath selama 3-24 jam pada suhu 37 oC. Setelah inkubasi selesai, ke dalam mikrotube ditambahkan 500 μl equilibrated phenol, kemudian diaduk kembali dengan menggunakan vortex. Setelah itu disentrifuse dengan kecepatan 10.000 rpm selama 10 menit. Lapisan supernatant yang terbentuk di lapisan atas diambil dengan menggunakan mikropipet dan dimasukkan ke dalam mikrotube baru.
23
terdapat DNA. Selanjutnya larutan yang diperoleh disentrifuse dengan kecepatan 10.000 rpm selama 10 menit sampai terbentuk endapan putih (pellet). Jika pellet belum terbentuk, maka harus disentrifuse kembali dengan kecepatan 12.000 rpm selama 5 menit.
Setelah pellet terbentuk, larutan di atasnya dibuang. Kemudian pellet dikeringkan pada suhu ruangan dan ditambahkan 100 μl DNA rehydration solution. Hasil ekstraksi selanjutnya disimpan pada suhu 4 oC sebelum digunakan pada tahap selanjutnya.
3.3.3 Amplifikasi Daerah mtDNA
Primer yang digunakan untuk mengamplifikasi sekuens mitokondria
D-loop adalah:
- primer Pro-5 (CAC GAC GTT GTA AAA CGA CCT ACC YCY AAC TCC CAA AGC), dan
- primer 12SAR (GGA TAA CAA TTT CAC ACA GGG CAT AGT GGG GTA TCT AAT CC).
Pengamplifikasian dilakukan menggunakan metode Polymerize Chain Reaction (PCR) dengan komposisi reaksi yang terdiri dari: 3 μl DNA, 2 μl masing-masing primer, 18 μl nuclease free water, dan pure taq DNA dengan total volume keseluruhannya 25 μl.
Siklus PCR yang diterapkan dalam amplifikasi adalah: - lit. temperatur 105 oC;
- satu siklus denaturasi pada suhu 95 oC selama 2 menit;
- 35 siklus penggandaan yang terdiri dari 95 oC selama 1 menit, 45 oC selama 1 menit dan 75 oC selama 2,5 menit; selanjutnya;
- satu siklus terakhir pada suhu 72 oC selama 10 menit; - kemudian disimpan dalam suhu 4 oC selama 5 menit.
DNA hasil PCR diambil sebanyak 3 μl dan ditambahkan dengan 3 μl
3.3.4 Restriksi mtDNA dan Visualisasi Hasil Restriksi
Sekuens mtDNA yang diperoleh kemudian direstriksi dengan menggunakan
endonuklease sesuai dengan prosedur: 3 μl DNA + 2 μl buffer + 0,3 μl enzim restriksi + 14,7 μl nuclease free water, setelah itu disentrifuse selama 15 detik dan kemudian diinkubasi pada suhu 37 oC selama 24 jam. Hasil restriksi kemudian dipisahkan secara elektroforesis dengan menggunakan gel agarose 2-3% dalam
Tris-Boric-EDTA (TBE) buffer dan diamati dengan UV illuminator serta dicetak gambarnya dengan polaroid.
3.4 Analisis Data
Data komposit haplotipe dianalisis untuk mendapatkan parameter genetik, struktur populasi dan hubungan filogenetik antar populasi:
- Tingkat keragaman genetik diukur berdasarkan indek keragaman haplotipe (h) dihitung dengan memanfaatkan data distribusi-frekuensi haplotipe (nukleomorf) berdasarkan Nei dan Tajima (1981).
- Kekerabatan antar populasi ditentukan berdasarkan parameter Jarak Genetik (Nei 1972) dan analisis statistik terhadap perbedaan situs restriksi. Jarak Genetik dihitung menurut Nei (1978).
- Derajat perbedaan molekuler haplotipe di antara populasi diduga dengan menggunakan Analysis of Moleculer Varians (AMOVA) dan uji jarak berpasangan (Fst).
- Hubungan filogenetik di antara populasi digambarkan dalam bentuk dendrogram melalui clustering nilai jarak genetik menurut metode jarak rata-rata.
4
HASIL DAN PEMBAHASAN
4.1 Sampel Ikan Tuna Mata Besar
Pada dasarnya ikan tuna genus Thunnus dapat dibagi dalam kelompok besar berdasarkan penyebarannya secara geografis, yaitu kelompok yang hidup di perairan dingin atau beriklim sedang (temperate species) seperti ikan tuna sirip biru utara (Thunnus thynnus), ikan tuna sirip biru selatan (T. maccoyii) dan albakora (T. alalunga), dan kelompok yang hidup di perairan tropis (tropical species) seperti ikan tuna sirip kuning atau madidihang (T. albacares). Ikan tuna mata besar (T. obesus) mempunyai sifat kedua kelompok tersebut, karena ikan tuna mata besar dapat hidup di daerah yang sangat luas mulai dari daerah tropis hingga daerah beriklim sedang (Suzuki 1988, diacu dalam Sumadhiharga 2001).
Posisi geografis pengambilan sampel sirip ikan tuna mata besar yang juga merupakan daerah penangkapan ikan tuna mata besar secara tepat disajikan pada Gambar 4 dan Tabel 9. Lokasi daerah penangkapan kapal tuna longline yang diamati berada di perairan Samudera Hindia sebelah selatan Jawa Timur, Bali hingga ke Nusa Tenggara. Sebagian besar lokasi penangkapan di sebelah selatan lintang 13o LS, dimana wilayah ini merupakan perairan laut bebas karena sudah di luar ZEE Indonesia.
Tabel 9 Posisi dan ukuran panjang kelompok sampel ikan tuna mata besar
Kelompok sampel Posisi geografis pengambilan sampel Ukuran ikan (cm) 1 113o13'-114o12' BT 14o23'-16o07' LS 61-139 2 110o15'-112o59' BT 13o59'-15o13' LS 86-155 3 113o21'-118o35' BT 11o49'-13o48' LS 73-156 4 115o26'-116o25' BT 13o03'-14o25' LS 65-145 5 113o26'-116o58' BT 09o11'-12o32' LS 102-178
Atlantik dan populasi dari Samudera Hindia dan Samudera Pasifik. Hal ini dibuktikan oleh hasil penelitiannya yang mengindikasikan bahwa sampel ikan tuna mata besar dari Samudera Atlantik secara genetik berbeda dari sampel ikan tuna mata besar dari Samudera Hindia dan Samudera Pasifik. Selain itu, hasil penelitian genetik ikan tuna mata besar yang diambil dari 5 (lima) lokasi pengambilan sampel di perairan Samudera Hindia (Indonesia, Madagaskar, Seychelles, barat Samudera Hindia dan bagian barat Australia) yang dilakukan oleh Appleyard et al (2002) mengindikasikan bahwa tidak terdapat perbedaan yang sangat nyata antara sampel ikan tuna mata besar yang dikumpulkan dari sebelah timur dan barat Samudera Hindia. Hal ini diperkuat oleh penelitian Chiang et al. (2008) yang mengambil sampel dari 4 lokasi di perairan Samudera Hindia (Kepulauan Cocos, Seychelles, sebelah tenggara dan baratdaya Samudera Hindia) yang menyatakan bahwa ikan tuna mata besar dari Samudera Hindia merupakan satu populasi.
Ukuran ikan tuna mata besar yang diambil siripnya untuk dianalisis genetiknya berkisar 60-180 cm (FL) dengan modus 100-110 cm (Gambar 6). Sebagian besar (>90%) ikan tuna mata besar tersebut telah matang gonad. Hal ini sesuai dengan hasil penelitian Nootmorn (2004) yang memperkirakan ukuran pertama kali matang gonad (size at fist maturity) ikan tuna mata besar di Samudera Hindia yaitu 88,08 cm untuk betina dan 86,85 cm untuk jantan.
0
60-69 70-79 80-89 90-99 100-109 110-119 120-129 130-139 140-149 150-159 160-169 170-179
FL (cm)
27
4.2 Keragaman Genetik Ikan Tuna Mata Besar
4.2.1 Amplifikasi dan Pemotongan dengan Enzim Restriksi
Hasil amplifikasi D-Loop mtDNA pada ikan tuna mata besar dengan menggunakan primer Pro-5 (CAC GAC GTT GTA AAA CGA CCT ACC YCY AAC TCC CAA AGC), dan primer 12SAR (GGA TAA CAA TTT CAC ACA GGG CAT AGT GGG GTA TCT AAT CC) menghasilkan fragmen DNA berukuran sekitar 1.500 bp (base pairs) pada semua sampel ikan tuna mata besar (Gambar 7).
Gambar 7 Fragmen tunggal mtDNA hasil amplifikasi PCR ikan tuna mata besar.
Dengan menggunakan primer ini terlihat hanya ada satu pita DNA pada gel agaros dengan perkiraan ukuran 1.500 bp. Hal ini mengindikasikan bahwa primer yang digunakan merupakan primer spesifik untuk ikan tuna mata besar.
Keragaman jumlah situs dan ukuran fragmen restriksi (RFLP) yang diperoleh dari hasil restriksi mtDNA dengan empat enzim adalah 18 tipe restriksi yaitu Taq I dan Hin6 I dengan empat tipe restriksi A, B, C dan D (Gambar 8 dan 9), Afa I dan Mbo I dengan lima tipe restriksi A, B, C, D dan E (Gambar 10 dan 11). Berdasarkan tipe restriksi mtDNA tersebut, enzim Afa I dan Mbo I dengan lima tipe restriksi merupakan enzim yang paling sensitif mendeteksi perbedaan panjang fragmen terpotong dibandingkan enzim Taq I dan Hin6 I.
Penggunaan empat enzim restriksi dalam penelitian ini guna mengukur keragaman genetik suatu populasi telah menunjukkan suatu variabilitas yang tinggi, walaupun idealnya lebih banyak enzim lebih baik. Rekapitulasi identifikasi
500 bp 1.000 bp 1.500 bp
tipe restriksi mtDNA teramplifikasi hasil digesti dengan empat enzim (Taq I, Afa I, Mbo I dan Hin6 I) disajikan pada Tabel 10.
Gambar 8 Tipe restriksi dengan enzim Taq I: A B C D.
Gambar 9 Tipe restriksi dengan enzim Hin6 I: A B C D.
29
Gambar 11 Tipe restriksi dengan enzim Mbo I: A B C D E.
Tabel 10 Distribusi tipe restriksi pada lima kelompok sampel ikan tuna mata besar
Enzim Tipe
Enzim Taq I menghasilkan tipe restriksi A monomorfik pada kelompok sampel 5, tiga tipe restriksi (A, B, C) pada kelompok sampel 1 dan empat tipe restriksi (A, B, C, D) pada kelompok sampel 4. Tipe A dan B ditemukan pada kelompok sampel 2 dan 3, dimana pada kelompok sampel 2 tipe B lebih dominan, sedangkan pada kelompok sampel 3 tipe A dan B hampir sama.
berbeda situs restriksi. Tipe B dominan ditemukan pada semua kelompok sampel. Tipe C hanya ditemukan di kelompok sampel 1, tipe D di kelompok sampel 4 dan tipe E di kelompok sampel 5.
Enzim Mbo I menghasilkan 2 hingga 4 tipe restriksi, dimana tipe A dominan di semua kelompok sampel. Dua tipe restriksi yang sama yaitu tipe A dan C ditemukan pada kelompok sampel 2 dan 5, tiga tipe restriksi pada kelompok sampel 1 dan 4, namun berbeda situs restriksi, dan empat tipe restriksi hanya pada kelompok sampel 3.
Enzim Hin6 I menghasilkan dua tipe restriksi yang sama pada kelompok sampel 1 dan 2, dan tiga tipe restriksi yang sama pada kelompok sampel 3, 4 dan 5. Tipe B dominan hampir ditemukan pada semua kelompok sampel, kecuali pada kelompok sampel 2.
Hasil pemotongan yang menunjukkan ukuran panjang fragmen berbeda akan memberikan tipe pemotongan (haplotipe) yang berbeda pula. Tipe pemotongan yang berbeda pada setiap individu dalam suatu populasi maupun antara populasi dapat disebabkan oleh terjadinya pergantian, penambahan atau hilangnya basa tertentu pada urutan pasangan basa D-Loop mtDNA-nya sehingga enzim tertentu tidak memotong pada situs yang sama. Hal ini mengakibatkan terjadinya pergeseran situs pemotongan. Dengan demikian dapat dikatakan bahwa terdapat perbedaan urutan pasangan basa pada individu yang mempunyai tipe pemotongan basa yang berbeda. Hal ini mengindikasikan adanya keragaman genetik di dalam populasi dan antara populasi.
4.2.2 Keragaman Haplotipe (Haplotype Diversity)
Hasil pemotongan produk PCR dengan menggunakan empat enzim restriksi menghasilkan 23 komposit haplotipe mtDNA D-Loop region. Tipe komposit haplotipe yang diperoleh tersaji pada Tabel 11. Setiap enzim memiliki tipe restriksi (tipe pemotongan) yang berbeda.
31
antara 0,5578 (kelompok sampel 5) hingga 0,8136 (kelompok sampel 4) (Tabel 11 dan Lampiran 1).
Tabel 11 Distribusi frekuensi menurut tipe komposit haplotipe pada lima kelompok sampel ikan tuna mata besar
Frekuensi haplotipe (%)
haplotipe 0,7048 0,6693 0,7232 0,8136 0,5578
kelompok sampel yaitu kelompok sampel 1, 2, 3 dan 4. Komposit haplotipe BBAB dan BBCB merupakan haplotipe utama (major composite haplotypes) karena keduanya terdapat pada lima kelompok sampel, sedangkan ABAB merupakan haplotipe umum (common haplotype) karena terdapat pada empat kelompok sampel dari lima kelompok sampel.
Selain komposite haplotipe utama dan umum, kelima kelompok sampel juga dicirikan dengan komposit haplotipe unik (unique composite haplotypes), yaitu komposit haplotipe yang terdapat pada satu kelompok sampel tetapi tidak terdapat pada kelompok sampel yang lainnya. Komposit haplotipe AAAA, BABA, CBAA, BCAA, ABAD, BADA, CBEB, BDAA, DBAA, DBAB, BAAC dan BEAB adalah komposit haplotipe unik yang masing-masing hanya terdapat pada satu kelompok sampel yang berbeda. Komposit haplotipe AAAA, BABA, CBAA dan BCAA hanya ditemukan pada kelompok sampel 1, ABAD hanya ditemukan pada kelompok sampel 2, BADA hanya ditemukan pada kelompok sampel 3, CBEB, BDAA, DBAA, DBAB dan BAAC hanya ditemukan pada kelompok sampel 4 dan BEAB pada kelompok sampel 5.
Berdasarkan tipe haplotipe BBAB dan BBCB, yang ditemukan pada semua kelompok sampel, menunjukkan kedekatan secara geografis. Dengan demikian dapat diduga secara genetik kelima kelompok sampel tersebut memiliki asal stok yang sama.
Nilai keragaman haplotipe yang diperoleh berkisar dari 0,5578 pada kelompok sampel 5 hingga 0,8136 pada kelompok sampel 4 (Tabel 10). Nilai keragaman haplotipe, secara umum sangat tergantung pada ukuran sampel (Nei 1981; Tabata dan Mizuta 1997, diacu dalam Rina 2001).
33
komposit haplotipe ikan tuna sirip kuning hasil penelitian Permana et al. (2007) dari perairan Bali, Sulawesi Utara dan Maluku Utara memiliki 15 komposit haplotipe.
Nilai keragaman genetik (haplotipe) rata-rata adalah 0,6937. Nilai ini lebih rendah dibandingkan nilai keragaman rata-rata ikan tuna sirip kuning yaitu 0,857 (Permana et al. 2007), namun lebih tinggi jika dibandingkan dengan jenis ikan laut perenang cepat lainnya seperti ikan hiu (0,64) (Heist 1999, diacu dalam
Permana et al. 2007). Graves dan McDowell (1994) diacu dalam Tabata et al. (1997) menjelaskan bahwa keragaman haplotipe keseluruhan mtDNA ikan striped marlin adalah 0,82 dan Avise et al. (1989) diacu dalam Tabata et al. (1997) menyatakan bahwa keragaman haplotipe keseluruhan mtDNA untuk beberapa ikan berada dalam kisaran 0,473-0,998. Relatif tingginya keragaman haplotipe pada ikan tuna mata besar ini memberikan indikasi bahwa keadaan populasinya belum banyak terganggu khususnya kelompok sampel 4 dan 3. Selain itu, keadaan ini juga menunjukkan bahwa ikan tuna mata besar mempunyai tingkat migrasi yang lebih tinggi dibandingkan ikan air laut lainnya sehingga peluang untuk adanya persilangan dengan populasi yang lainnya semakin besar pula (Wild 1994,
diacu dalam Permana et al. 2007).
Keragaman haplotipe terkecil, yaitu 0,5578 terdapat pada ikan tuna mata besar kelompok sampel 5 dan yang tertinggi, yaitu 0,8136 pada kelompok sampel 4. Fenomena ini mengindikasikan bahwa ikan tuna mata besar dari kelompok sampel 5 mempunyai keragaman genetik yang rendah dan memiliki ukuran yang lebih kecil dibandingkan empat kelompok sampel lainnya. Leary et al. (1985)
diacu dalam Ayu (2005), menyatakan bahwa rendahnya keragaman genetik akan mengakibatkan munculnya sifat-sifat negatif, antara lain menurunnya pertumbuhan, keragaman ukuran, kestabilan perkembangan organ, tingkat kelangsungan hidup, serta adaptasi terhadap perubahan lingkungannya.
4.2.3 Jarak Genetik
sampel 3 dan 4, kelompok sampel 3 dengan kelompok sampel 4 dan 5 serta kelompok sampel 4 dengan kelompok sampel 5. Tidak ada perbedaan yang nyata antara kelompok sampel 1 dengan 2 dan 5 serta kelompok sampel 2 dengan kelompok sampel 5. Hasil uji keragaman antara lima kelompok sampel ikan tuna mata besar dengan metode jarak berpasangan (Fst) disajikan pada Tabel 12.
Tabel 12 Keragaman lima kelompok sampel ikan tuna mata besar berdasarkan metode jarak berpasangan (Fst)
Kelompok sampel 1 2 3 4 5
Jarak genetik dan dendrogram hubungan kekerabatan antar kelompok sampel (filogeni) pada lima kelompok sampel ikan tuna mata besar menurut metode UPGMA menggunakan program TFPGA disajikan pada Tabel 13, Gambar 12 dan Lampiran 2. Makin kecil nilai jarak genetik yang diperoleh, maka makin dekat pula keragaman kedua kelompok sampel tersebut, demikian juga sebaliknya.
Tabel 13 Jarak genetik antara ikan tuna mata besar
Kelompok sampel 1 2 3 4 5